Overview
Fuente: Dominique R. Bollino1, Eric A. Legenzov2, Tonya J. Webb1
1 Departamento de Microbiología e Inmunología, Facultad de Medicina de la Universidad de Maryland y el Centro Integral del Cáncer Marlene y Stewart Greenebaum, Baltimore, Maryland 21201
2 Centro de Ingeniería y Tecnología Biomédica, Facultad de Medicina de la Universidad de Maryland, Baltimore, Maryland 21201
La microscopía de fluorescencia confocal es una técnica de imagen que permite una mayor resolución óptica en comparación con la microscopía de epifluorescencia convencional de "campo ancho". Los microscopios confocales son capaces de lograr una resolución óptica x-y mejorada a través del "escaneo láser", normalmente un conjunto de espejos controlados por voltaje (galvanómetro o espejos "galvo") que dirigen la iluminación láser a cada píxel de la muestra a la vez. Lo que es más importante, los microscopios confocales logran una resolución z-axial superior mediante el uso de un agujero para eliminar la luz de foco procedente de lugares que no están en el plano z que se está escaneando, lo que permite al detector recopilar datos de un plano z especificado. Debido a la alta resolución z alcanzable en la microscopía confocal, es posible recopilar imágenes de una serie de planos z (también llamados z-stack) y construir una imagen 3D a través de software.
Antes de discutir el mecanismo de un microscopio confocal, es importante considerar cómo interactúa una muestra con la luz. La luz se compone de fotones, paquetes de energía electromagnética. Un fotón que afecta a una muestra biológica puede interactuar con las moléculas que componen la muestra de una de cuatro maneras: 1) el fotón no interactúa y pasa a través de la muestra; 2) el fotón se refleja / dispersa; 3) el fotón es absorbido por una molécula y la energía absorbida se libera como calor a través de procesos conocidos colectivamente como descomposición no radiativa; y 4) el fotón se absorbe y la energía se emite rápidamente como un fotón secundario a través del proceso conocido como fluorescencia. Una molécula cuya estructura permite la emisión de fluorescencia se llama fluoróforo. La mayoría de las muestras biológicas contienen fluoróforos endógenos insignificantes; por lo tanto, los fluoróforos exógenos deben utilizarse para resaltar las características de interés en la muestra. Durante la microscopía de fluorescencia, la muestra se ilumina con luz de la longitud de onda adecuada para la absorción por el fluoróforo. Al absorber un fotón, se dice que un fluoróforo está "emocionado" y el proceso de absorción se conoce como "excitación". Cuando un fluoróforo renuncia a la energía en forma de fotón, el proceso se conoce como "emisión", y el fotón emitido se llama fluorescencia.
El haz de luz utilizado para excitar un fluoróforo está enfocado por la lente objetivo de un microscopio y converge en un "punto focal" donde se centra al máximo. Más allá del punto focal, la luz vuelve a divergir. Las vigas de entrada y salida se pueden visualizar como un par de conos que se tocan en el punto focal (consulte la figura 1, panel izquierdo). El fenómeno de la difracción impone un límite a la tensión con la que se puede enfocar un haz de luz : el haz en realidad se centra en un punto de tamaño finito. Dos factores determinan el tamaño del punto focal: 1) la longitud de onda de la luz, y 2) la capacidad de recolección de luz de la lente objetivo, que se caracteriza por su apertura numérica (NA). El "punto" focal se extiende no sólo en el plano x-y, sino también en la dirección z, y en realidad es un volumen focal. Las dimensiones de este volumen focal definen la resolución máxima alcanzable por imágenes ópticas. Aunque el número de fotones es mayor dentro del volumen focal, las trayectorias de luz cónicas por encima y por debajo del foco también contienen una densidad más baja de fotones. Cualquier fluoróforo en el camino de luz puede ser excitado. En la microscopía de epifluorescencia convencional (de campo ancho), la emisión de fluoróforos por encima y por debajo del plano focal contribuye a la fluorescencia fuera de foco (un "fondo de superficie"), que reduce la resolución y el contraste de la imagen, como se demuestra en la Figura 1, con el cubo rojo que representa la emisión de fluoróforo por encima del plano focal (estrella roja) que resulta en fluorescencia desenfocada (arriba a la derecha). Este problema se mejora en microscopía confocal, debido a la utilización de un agujero. (Figura 2, abajo a la derecha). Como se muestra en la Figura 3, el agujero permite que las emisiones procedentes del lugar focal lleguen al detector (izquierda), al tiempo que bloquea la fluorescencia desenfocada (derecha) de llegar al detector, mejorando así tanto la resolución como el contraste.
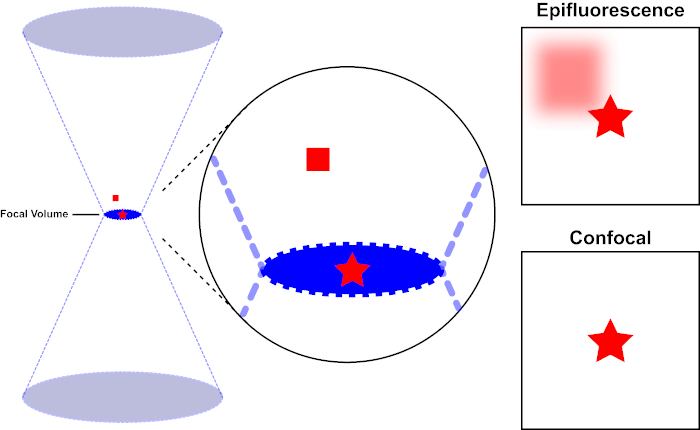
Figura 1. Resolución óptica de epifluorescencia frente a microscopía confocal. Haga clic aquí para ver una versión más grande de esta figura.
El haz de luz utilizado para excitar un fluoróforo está enfocado por la lente objetivo de un microscopio y converge en un volumen focal y luego diverge (izquierda). La estrella roja representa el plano focal de una muestra que se está siendo imagenda mientras que el cuadrado rojo representa la emisión de fluoróforo por encima del plano focal. Al capturar una imagen de esta muestra utilizando un microscopio epifluorescente, la emisión desde el cuadrado rojo fuera de foco será visible y contribuirá a un "fondo brumoso" (arriba a la derecha). Los microscopios confocales tienen un agujero que impide la detección de la luz emitida fuera del plano focal, eliminando el "fondo brumoso" (abajo a la derecha).
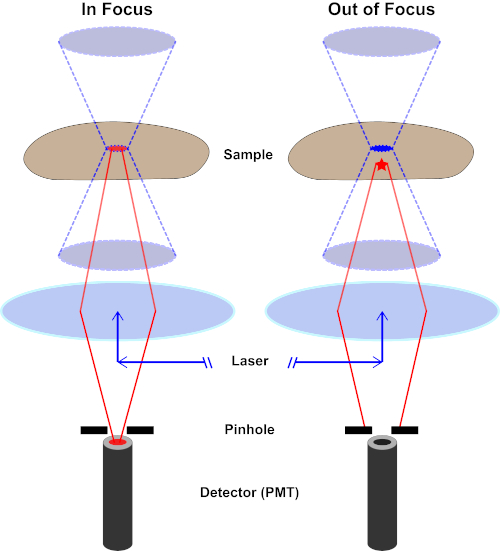
Figura 2. Efecto agujero en microscopía confocal. Haga clic aquí para ver una versión más grande de esta figura.
Aunque la intensidad más alta de la luz de excitación se encuentra en el punto focal de la lente (izquierda, óvalo rojo), otras partes de la muestra que no están en el punto focal (derecha, estrella roja) obtendrán luz y flúor. Para evitar que la luz emitida por estas regiones fuera de foco llegue al detector, hay una pantalla con un agujero delante del detector. Sólo la luz de enfoque (izquierda) que emite desde el plano focal es capaz de viajar a través del agujero y llegar al detector. La luz desenfocada (derecha) está bloqueada con el agujero y no llega al detector.
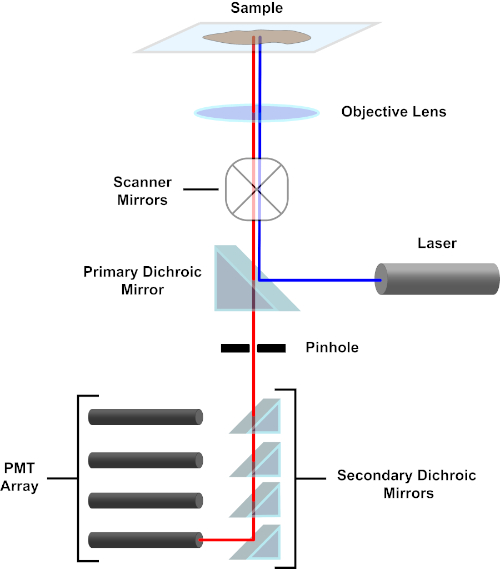
Figura 3. Componentes principales de un microscopio de escaneo láser confocal. Haga clic aquí para ver una versión más grande de esta figura.
En aras de la simplicidad, la descripción mecanicista de un microscopio confocal se limitará a la de la Nikon Eclipse Ti A1R. Aunque puede haber pequeñas diferencias técnicas entre diferentes microscopios confocales, el A1R sirve bien como un buen modelo para describir la función del microscopio confocal. El haz de luz de excitación, producido por una serie de láseres de diodos, se refleja en el espejo dicrosico primario en el objetivo, que centra la luz en la muestra que se está realizando. El espejo dicoico primario refleja selectivamente la luz de excitación mientras permite que la luz en otras longitudes de onda pase a través. A continuación, la luz se encuentra con los espejos de escaneo que barren el haz de luz a través de la muestra de una manera x-y, iluminando un solo píxel (x,y) a la vez. La fluorescencia emitida por los fluoróforos en el píxel iluminado es recogida por la lente objetivo y pasa a través del espejo dicrromico primario para llegar a una serie de tubos fotomultiplicadores (PMP). Los espejos dicoicos secundarios dirigen la luz de emisión a la PMT apropiada. La luz de excitación dispersa por la muestra de nuevo en el objetivo se refleja en el espejo dicroro primario hacia la muestra, y así se impide que entre en la detección camino de luz y alcanzando los PMT (véase el cuadro 3). Esto permite cuantificar la fluorescencia relativamente débil sin contaminación por la luz dispersa del haz de luz de excitación, que suele ser órdenes de magnitud más intensas que la fluorescencia. Debido a que el agujero bloquea la luz desde fuera del volumen focal, la luz que llega al detector proviene de un plano zestrecho y seleccionado. Por lo tanto, las imágenes se pueden recoger de una serie de planos zadyacentes; esta serie de imágenes se conoce a menudo como una 'z-pila'. Mediante el uso del software adecuado, se puede procesar una z-stack para generar una imagen 3D de la muestra. Una ventaja particular de la microscopía confocal es la capacidad de distinguir la localización subcelular de la tinción. Por ejemplo, la diferenciación entre la tinción de membrana de la tinción intracelular, que es muy difícil con la microscopía de epifluorescencia convencional (1, 2, 3).
La preparación de muestras es una faceta importante de las imágenes confocales. Una fuerza de las técnicas de microscopía óptica es la flexibilidad para crear imágenes de células vivas o fijas. Al intentar producir imágenes 3D, debido al número de imágenes que se deben adquirir para una pila z, la dificultad de mantener la salud celular, y el movimiento de las células vivas y sus orgánulos, el uso de células fijas es típico. El procedimiento de fijación y tinción de células para fluorescencia confocal es similar al utilizado convencionalmente en inmunofluorescencia. Después del cultivo en diapositivas de cámara o en cubreobjetos, las células se fijan usando paraformaldehyde para preservar la morfología celular. La unión de anticuerpos no específica se bloquea mediante albúmina de suero bovino, leche o suero normal. Con el fin de mantener la especificidad de los anticuerpos secundarios, la solución utilizada no debe proceder de la misma especie en la que se generaron los anticuerpos primarios. Las células se incuban con anticuerpos primarios que unen el antígeno de interés. Al etiquetar varios objetivos celulares, los anticuerpos primarios deben derivarse cada uno de una especie diferente. Los anticuerpos que etiqueten un antígeno están unidos por anticuerpos secundarios conjugados con fluoróforos. Se deben seleccionar anticuerpos secundarios conjugados con fluoróforos para que sean compatibles con las longitudes de onda de excitación láser disponibles en el microscopio confocal. Al visualizar múltiples antígenos, los espectros de excitación/emisión de los fluoróforos deben diferir lo suficiente para que sus señales puedan ser discriminadas por análisis microscópicos. La muestra manchada se monta en un portaobjetos para obtener imágenes. Se utiliza un medio de montaje para prevenir el fotoblanqueo y la deshidratación de muestras. Si lo desea, se puede utilizar un medio de montaje que contenga una contramancha nuclear (por ejemplo, DAPI o Hoechst) (4).
En el siguiente protocolo, los fibroblastos de ratón transinfectados para expresar CD1d (LCD1) se tiñieron con anticuerpos que reconocieron CD1d y CD107a (LAMP-1). CD1d es un importante complejo de histocompatibilidad 1 (MHC 1) -como receptor presente en la superficie del antígeno que presenta antígenos lipídicos. LAMP-1 (proteína de membrana asociada a la islosomal-1) es una proteína transmembrana presente principalmente en las membranas lisosomal. Para una correcta presentación del antígeno, CD1d se trafica a través del compartimento lisosomal de pH bajo, por lo que LAMP-1 se utiliza como marcador del compartimento lisosomal para este protocolo. Al sondear las células LCD1 con anti-CD1d y anti- LAMP-1 que se produjeron en diferentes especies, se pueden utilizar anticuerpos secundarios con fluoróforos únicos para determinar la localización de cada proteína en la célula y si CD1d está presente en el LAMP-1 positivo compartimentos lisosomal.
Procedure
1. Materiales
Búferes
- Tampón de lavado: 1 salina estéril con fosfato (PBS) sin calcio o magnesio
- Búfer de fijación: 1% paraformaldehído en PBS
- Búfer de permeabilización: 0.1% Triton X-100 en PBS
- Buffer de bloqueo: 1% albúmina sérica bovina en PBS
- Medio de crecimiento celular: DMEM complementado con 10% de suero bovino fetal (FBS), penicilina/estreptomicina y L-glutamina
Equipo
- Capucha de flujo laminar
- Incubadora humedecida (37oC, 5% CO2)
- Microscopio de escaneo láser confocal; aquí, Nikon Eclipse Ti láser
Materiales y reactivos
- Diapositivas de cultivo celular por cámara
- Medios de montaje anti-fade con DAPI (para tinción de núcleos)
- Vidrio de la cubierta del microscopio
- Toallitas de tarea delicadas
- Pipettors y puntas
Reactivos específicos de ensayo
- Células adherentes (células primarias o líneas celulares); aquí, se utilizaron fibroblastos de ratón transtrófilos con CD1d (LCD1).
- Anticuerpos primarios para detectar objetivos celulares; aquí, se utilizaron cd107a antiratón de rata (LAMP-1) y ratón antiratón CD1d.
- Anticuerpos secundarios conjugados con fluoróforos específicos de los isotipos de anticuerpos primarios; aquí se utilizaron IgG anti-rata conjugada con Alexafluor 488 y anti-ratón IgG conjugado con Alexafluor 647.
2. Protocolo
Preparación para la tinción de anticuerpos
Células de sembrado
- Resuspender las células de interés en los medios de crecimiento.
- Luego, la semilla de 500 l de la suspensión de la célula / por pozo en los pozos de un portaobjetos de cámara de 4 pozos. (Aquí, las células LCD1 se sembraron a 2.5x105 células/cámara en 500 s de medios de crecimiento. La densidad de sembración puede variar entre líneas celulares).
- Incubar el tobogán de la cámara durante la noche en una incubadora deCO2 del 5% a 37oC, para permitir que las células se adhieran al vidrio.
- Al día siguiente, aspirar los medios de cada poca y luego lavar las células 1X con 500 l de PBS.
Fijación
- Para fijar las células, agregue una solución de paraformaldehído de 500 l 1% en cada poca y incubadurante durante 15 minutos a temperatura ambiente.
- Después de la incubación, recoja el paraformaldehído en un contenedor de residuos líquidos peligrosos apropiado.
- Luego, lave las células 3 veces con 500 l de PBS para eliminar los restos del fijador.
Permeabilización
- Permeabilizar las células incubando con un tampón/bien de permeabilización de 500 l durante 15 minutos a temperatura ambiente.
- Luego, lave las células brevemente 3 veces con 500 l de PBS.
Bloqueo
- Incubar las células de cada pozo con un tampón de bloqueo de 500 ml durante 1 hora a 4oC, para bloquear la unión de anticuerpos no específicos.
Incubación de anticuerpos primarios
- Aspirar el búfer de bloqueo de las cámaras deslizantes.
- A continuación, agregue 500 s de solución de anticuerpos primarios diluidas a las células. (Aquí, anti-CD107a (LAMP-1) se diluyó 1:500 y anti-CD1d se utilizó sin diluir (1H6 anticuerpo monoclonal fue amablemente proporcionado por el Dr. Randy Brutkiewicz)).
- Incubar los toboganes durante la noche a 4oC.
Nota: Si sondea más de un objetivo, asegúrese de que los anticuerpos primarios sean isotipos diferentes. Las concentraciones recomendadas de anticuerpos varían entre los fabricantes y deben valorarse antes de su uso.
Incubación Secundaria de Anticuerpos
- Aspirar la solución primaria de anticuerpos de los pozos.
- Lave las cámaras del pozo 4 veces con 500 oL de PBS.
- A continuación, añadir 500 s de la solución de anticuerpos secundarios diluidas a cada poca. (Aquí, tanto los anticuerpos secundarios- anti-ratón IgG Alexafluor 647 y anti-rata IgG Alexafluor 488 se diluyeron 1:2000 en el búfer de bloqueo).
- Incubar a temperatura ambiente durante 1 h en la oscuridad.
- Después de la incubación, aspirar la solución secundaria de anticuerpos.
- Lavar las cámaras 4 veces con 500 oL de PBS para eliminar cualquier anticuerpo secundario no unido.
Nota: Las concentraciones recomendadas de anticuerpos varían entre los fabricantes y deben valorarse antes de su uso. Si se sondea más de un objetivo, los anticuerpos secundarios deben conjugarse con diferentes fluoróforos con espectros únicos de excitación/emisión. También tenga en cuenta la excitación/emisión de la contramancha nuclear (es decir, DAPI) mientras selecciona los fluoróforos. La selección de fluoróforos puede verse afectada por la configuración láser del microscopio confocal utilizado. La configuración láser de la máquina dictará qué fluoróforos son adecuados para el experimento.
3. Montaje de Coverslips
- En primer lugar, retire cuidadosamente las cámaras de la corredera.
- Luego, sostenga la diapositiva en ángulo sobre una delicada limpieza de tareas y retire el líquido de los bordes sin tocar las células.
- Agregue 1 gota de medio de montaje antidescoloro, que contenga la mancha nuclear DAPI, en cada sección de las células.
- A continuación, coloque un recubre de 20 mm x 60 mm sobre la diapositiva sosteniéndolo sobre los bordes con las yemas de los dedos. (Evite la formación de burbujas sobre las células, ya que interfieren con las imágenes).
- Limpie cualquier medio de montaje adicional en los lados con una delicada limpieza de tareas y guarde los portaobjetos en la oscuridad a temperatura ambiente hasta una semana.
4. Imágenes confocales
Muestras de imagen en un microscopio de escaneo láser confocal. Para los datos mostrados en la Figura 2, la Nikon Eclipse Ti A1R se utilizó con el software NIS Elements Advanced Research. En la siguiente sección se detalla el procedimiento para capturar imágenes utilizando el software antes mencionado.
- Para comenzar a crear imágenes de las celdas, abra el software 'NIS Elements Advanced Research' haciendo clic en el icono de'NIS software.'
- A continuación, en la ventana de control haga clic en 'TiPad' pestaña y elegir el objetivo deseado para la imagen. (Aquí, se utilizó el primer objetivo 40x).
- Cargue la diapositiva con celdas en el escenario y céndala debajo de la lente.
- Ahora, en la pestaña 'A1plus Compact GUI', configure los láseres apropiados para los fluoróforos utilizados. Haga clic en el símbolo de engranaje para abrir el menú de ajustes espectrales y de tinte y seleccionar los canales necesarios y configurar el láser para cada canal.
- A continuación, seleccione las emisiones apropiadas en el menú desplegable bajo el primer espejo dicroico.
- A continuación, en 'A1plus Compact GUI' ventana, haga clic en 'Ch. Series' para configurar la serie de canales de línea, que configura si los láseres utilizados se disparará en la muestra simultáneamente o secuencialmente. (Aquí, se eligieron pasadas secuenciales, comenzando con el canal 1, seguido por el canal 2, luego 4).
- Después de eso, comience a escanear haciendo clic en el icono ' Punta deflecha' en la parte superior. En este punto mientras la imagen está en vivo, bajo 'A1plus Compact GUI' ventana, haga clic en la escala deslizante y modificar el tamaño del agujero para asegurar la limitación de la luz de enfoque. (Aquí, se utilizó el ajuste más bajo disponible (0,5).
- A continuación, ajuste los ajustes 'alto voltaje' y 'offset' bajo cada láser a los niveles apropiados, mediante el uso de las escalas deslizantes para permitir la detección de la tinción específica mientras se limita cualquier posible tinción de fondo. Si hay disponible una muestra de tinción positiva, comience por tomar imágenes de esta muestra para cada canal para asegurarse de que los ajustes del láser producen proporciones óptimas de señal a ruido.
Precaución: la alta intensidad del láser durante períodos prolongados puede causar fotoblanqueo. - Después de establecer los valores óptimos de HV y desplazamiento para cada láser, haga clic en la pestaña 'ND Acquisition' y luego seleccione el icono 'Z' para configurar los parámetros para la serie z. A continuación, mientras se adquiere una imagen en vivo de la muestra, primero establezca la parte inferior encontrando la parte inferior de la imagen y haciendo clic en el botón 'inferior' luego encontrar la posición superior de la muestra y haga clic en el botón 'superior'. Establezca el tamaño del paso escribiendo específicamente el tamaño de paso preferido en el valor de m para cada paso o especificando cuántos pasos totales se necesitan.
- Una vez establecidos los parámetros de la serie z, seleccione la resolución de tamaño/píxel deseada de la imagen. Para ello, haga clic en la ventana 'A1plus Compact GUI' y debajo del icono 'size' seleccione la resolución deseada. Para disminuir el ruido de la imagen, se puede seleccionar el menú desplegable junto al símbolo 'á' para promediar el número seleccionado de imágenes.
- Ahora, haga clic en la pestaña'Ejecutar ahora' en el menú 'Nd Acquisition' para comenzar a tomar imágenes de la muestra.
- Una vez completada la imagen, guarde la imagen haciendo clic en 'Archivo', luego 'Guardar como', que exportará el archivo de imagen con la extensión '.nd2'. Por último, repita el proceso para cada una de las otras muestras.
La microscopía de fluorescencia confocal es una técnica de imagen especializada para la localización de una proteína o antígeno de interés en una muestra de células o tejidos etiquetando el antígeno con un colorfluorescente conjugado por anticuerpos y detectando la señal fluorescente. Ofrece una mayor resolución espacial que la microscopía de fluorescencia de campo ancho, con la ayuda de dos agujeros colocados en los planos focales de la lente objetivo, dándole el nombre de confocal. Permite a los usuarios visualizar la tinción a un nivel subcelular, como la diferenciación entre la tinción de membrana superficial de la tinción intracelular.
Un microscopio confocal sigue un principio básico similar al de un microscopio de fluorescencia clásico. El haz de una fuente de luz, generalmente un láser para confocal, se refleja en un espejo dicroroico y se centra en una lente objetiva en la muestra. Esta luz excita a los fluoróforos para emitir una longitud de onda diferente, que viaja de vuelta a través de la lente objetivo y el espejo dicroico a una cámara o ocular.
La resolución mejorada de un microscopio confocal se debe principalmente a la presencia de dos agujeros, que son agujeros muy pequeños para que la luz pase a través de las trayectorias de excitación y emisión de luz. Los agujeros se colocan estratégicamente en el plano focal de la lente objetivo. Ahora, cambiemos a un esquema de vista lateral de la disposición del microscopio para revisar la trayectoria de la luz. Después de pasar a través del agujero de excitación, el haz de luz de excitación tiene el efecto de originarse desde un punto focal, lo que permite que la lente objetivo para luego enfocar la luz a un punto en la muestra, así. El haz de emisión desde este punto focal converge en el agujero de emisión, lo que le permite pasar. Ahora, durante la excitación, los fluoróforos dentro del camino de luz, por encima y por debajo del punto focal, también están ligeramente excitados. Mientras que la luz de emisión que se origina desde el punto focal pasa a través del agujero, las emisiones de los puntos desenfocados convergen antes o después del agujero de emisión, y por lo tanto se bloquean, lo que resulta en una fluorescencia de fondo reducida.
El ciclo de detección de excitación-emisión debe repetirse para cada punto de imagen de la región de interés, lo que se puede hacer de varias maneras diferentes. Por ejemplo, el escaneo láser confocal utiliza espejos de escaneo galvanómetro, que desvían la luz de excitación en diferentes ángulos. Por lo tanto, barriendo el haz de luz a través de la muestra en el plano XY. El confocal de disco giratorio utiliza un disco con una matriz de agujeros, que gira para desplazar la disposición de los agujeros. Esto permite a los usuarios iluminar varios puntos de imagen pequeños en la muestra cada vez, cubriendo gradualmente toda el área a medida que el disco gira. Como resultado de los agujeros, la imagen XY en el detector representa un plano Z estrecho. Por lo tanto, las imágenes se pueden recopilar de una serie de planos Z consecutivos, a menudo denominados pila Z. A partir de estas imágenes, un software adecuado puede generar una representación 3D del patrón de señal de fluorescencia en la muestra.
En este protocolo, observará inmunomanchación de fibroblastos de ratón, seguido de imágenes en un microscopio confocal para visualizar diferencialmente una proteína de superficie celular y una proteína lisosomal.
Para empezar, utilizando técnicas estériles, resuspender las células de interés en 500 microlitros de medios de crecimiento por pozo, y luego sembrarlas en los pozos de un tobogán de cámara de cuatro pozos. Aquí, estamos usando fibroblastos de ratón que fueron transinfectados para expresar la molécula que presenta antígenos, CD1d. Para permitir que las células se adhieran al vidrio, coloque el deslizamiento de la cámara en una incubadora de dióxido de carbono del 5% a 37 grados centígrados e incubadurante durante la noche. Por la mañana, aspirar los medios de cada poca, y luego lavar las células una vez con 500 microlitros de PBS durante unos segundos.
Para fijar las células, agregue 500 microlitros de 1% de solución de paraformaldehído en cada pocal e incubar durante 15 minutos a temperatura ambiente. Después de la incubación, recoja el paraformaldehído en un contenedor de residuos líquidos peligrosos apropiado, y luego retire cualquier resto del fijador lavando las células tres veces con PBS durante unos segundos.
Para permitir la penetración de anticuerpos en las células, agregue 500 microlitros de tampón de permeabilización a cada poca, e incubar en el banco durante 15 minutos a temperatura ambiente. Después de la permeabilización, lave las células brevemente tres veces con 500 microlitros de PBS. A continuación, agregue 500 microlitros de tampón de bloqueo a cada poca, e incubar durante una hora a cuatro grados Celsius para evitar la unión inespecífica de anticuerpos.
Preparar los anticuerpos primarios, anti-CD1d y anti-LAMP-1, a concentraciones de trabajo adecuadas. Luego, aspirar el amortiguador de los pozos y cubrir las células en cada pozo con 500 microlitros de solución de anticuerpos primarios diluidas y luego incubar la diapositiva en una superficie plana durante la noche a cuatro grados centígrados. A la mañana siguiente, diluir los anticuerpos secundarios, en este caso un anticuerpo anti-ratón y anti-rata con etiquetas fluorescentes distintas, en el bloqueo del tampón a las concentraciones de trabajo adecuadas. A continuación, aspirar la solución primaria de anticuerpos de los pozos y luego lavar las células cuatro veces con 500 microlitros de PBS. Luego, añadir 500 microlitros de la solución de anticuerpos secundarios diluidas a cada pocal, e incubar a temperatura ambiente durante una hora en la oscuridad. Después de la incubación, aspirar la solución secundaria de anticuerpos y lavar los pozos cuatro veces con 500 microlitros de PBS para eliminar cualquier anticuerpo secundario no unido.
Para montar las muestras después del lavado final, desmonte cuidadosamente y retire las cámaras de la diapositiva. Para eliminar el PBS residual, sostenga la diapositiva en un ángulo sobre una toallita de tarea delicada y retire el líquido de los bordes sin tocar las celdas. Una vez que se elimina el exceso de PBS, agregue una gota de medio de montaje antidescoloro, que contiene la mancha nuclear DAPI, en cada sección de las células. A continuación, tome un resbalón de 20 por 60 milímetros, y usando sólo las yemas de los dedos comience a bajar el cubreobjetos lentamente en cada borde, teniendo cuidado de evitar la formación de burbujas sobre las células. Limpie cualquier medio de montaje adicional en las diapositivas con una delicada limpieza de tareas y guarde las diapositivas en la oscuridad a temperatura ambiente durante un máximo de una semana.
Para comenzar a crear imágenes de las celdas, primero haga clic en el icono del software NIS en el escritorio. Una vez en la ventana de control, haga clic en la pestaña TiPad en la parte superior y elija el objetivo deseado para la creación de imágenes. Luego, cargue la diapositiva con células en el escenario y céndala debajo de la lente. A continuación, en la pestaña A1plus Compact GUI junto a la pestaña TiPad, configure los láseres adecuados para los fluoróforos utilizados. Haga clic en el símbolo de engranaje para abrir el menú de ajustes espectrales y de tinte. Una vez abierto el menú de ajustes espectrales y de tinte, seleccione los canales necesarios y ajuste el láser para cada canal. A continuación, seleccione las emisiones adecuadas en el menú desplegable bajo el primer espejo dicroico. A continuación, en la ventana de A1plus Compact GUI, haga clic en Ch.Series para configurar la serie de canales de línea, que configura si los láseres utilizados se dispararán en la muestra simultáneamente o secuencialmente.
Después de eso, comience a escanear haciendo clic en el icono de punta de flecha en la parte superior. En este punto, mientras la imagen está activa, en la ventana de la GUI de A1plus Compact, haga clic en la escala deslizante y modifique el tamaño del agujero para asegurar la limitación de la luz desenfocada. A continuación, ajuste los ajustes de alto voltaje y desplazamiento debajo de cada láser a los niveles adecuados mediante el uso de las escalas deslizantes para permitir la detección de la tinción específica mientras se limita cualquier posible tinción de fondo. Si hay disponible una muestra de tinción positiva, comience por tomar imágenes de esta muestra para cada canal para asegurarse de que los ajustes del láser producen proporciones óptimas de señal a ruido. Después de establecer los valores óptimos de HV y desplazamiento para cada láser, haga clic en la pestaña ND Acquisition y, a continuación, seleccione el icono Z para configurar los parámetros de la serie z.
A continuación, mientras adquiere una imagen en vivo de la muestra, primero establezca la parte inferior encontrando la parte inferior de la imagen y haciendo clic en el botón inferior. A continuación, busque la posición superior de la muestra y haga clic en el botón superior. Establezca el tamaño del paso escribiendo específicamente el tamaño de paso preferido en las micras para cada paso o especificando cuántos pasos totales se necesitan. Para seleccionar el tamaño/resolución de píxeles deseado de la imagen, haga clic en la ventana GUI de Aiplus Compact y, en el icono de tamaño, seleccione la resolución deseada.
Para reducir el ruido de la imagen, puede seleccionar el menú desplegable junto al símbolo theta para promediar el número seleccionado de imágenes. Después de esto, haga clic la pestaña Run Now en el menú ND Acquisition para comenzar a tomar imágenes de la muestra. Una vez completada la creación de imágenes, guarde la imagen haciendo clic en archivo y, a continuación, guarde como, que exportará el archivo de imagen con la extensión dot-nd2. Por último, repita el proceso para cada una de las otras muestras.
En este experimento, los fibroblastos de ratón que expresaban el gen de la glicoproteína superficial CD1d fueron fijos, inmunomanchados e imágenes en un microscopio confocal. Esta imagen muestra una sola sección de una pila Z en la ampliación 40X, donde CD1d se tiñe en rojo. La muestra fue corinado con LAMP-1, un marcador lisosomal, en verde. La mancha nuclear DAPI se utilizó para mostrar los núcleos de las células.
En una imagen compuesta donde se fusionan los tres canales diferentes, la apariencia de amarillo resulta de la superposición de los canales rojo y verde, e indica un área donde CD1d y LAMP-1 se colocalizan en los lisosomas. Las áreas donde sólo hay un color indican la presencia de CD1d o LAMP-1 sin colocalización. Esta imagen muestra una representación 3D de las celdas construidas a partir de imágenes capturadas en la pila z y este método habilitó la construcción de una vista lateral de este grupo de celdas. Esta siguiente imagen muestra un segmento de la pila z con un aumento de 100X, lo que demuestra los patrones de expresión de estas dos proteínas con mayor detalle. El cuadro delineado rosa en el lado derecho de la imagen muestra la sección transversal de la coordenada X designada por la línea rosa en la imagen, que representa la vista lateral en la línea rosa. Del mismo modo, el cuadro azul delineado en la parte inferior de la imagen muestra la sección transversal de la coordenada Y designada por la línea azul en la imagen, que representa la vista frontal en la línea azul. La representación 3D de la imagen de la pila z permite a los usuarios ver la imagen en 3D, visualizando todos los planos x, y y z. Esto se puede utilizar para estudiar la colocalización de las diferentes manchas en diferentes regiones dentro de la célula.
Subscription Required. Please recommend JoVE to your librarian.
Results
En este experimento, los fibroblastos de ratón que expresaban el gen de la glicoproteína superficial CD1d fueron fijos, inmunomanchados e imágenes en un microscopio confocal. Una imagen representativa obtenida usando el protocolo antedicho se muestra en la Figura 4. En el panel superior de A, se presentan imágenes de un solo canal que muestran el patrón de tinción de cada objetivo individual. Estas imágenes comprenden una sola sección (rebanada) de la pila z capturada. El panel derecho muestra la tinción DAPI de los núcleos de las células. Los paneles centrales muestran CD1d teñido en rojo y LAMP-1, un marcador lisosomal, teñido en verde. El panel izquierdo es una imagen compuesta donde se combinan los tres canales diferentes. La apariencia de amarillo resulta de la superposición de los canales rojo y verde, e indica un área donde CD1d y LAMP-1 se co-localizan. Los resultados de la tinción confirman que CD1d está localizado en los compartimentos endosomales LAMP-1+. También hay áreas donde sólo hay un color, lo que indica la presencia de CD1d o LAMP-1 sin co-localización. El panel inferior de A muestra una representación 3D de las celdas construidas a partir de imágenes capturadas en la pila z.
El panel B muestra una rebanada de la pila z con un aumento de 100x que demuestra los patrones de expresión de estas dos proteínas con mayor detalle. El cuadro delineado rosa en el lado derecho de la imagen muestra la sección transversal de la coordenada x designada por la línea rosa en la imagen, que representa la vista lateral en la línea rosa. Del mismo modo, el cuadro azul delineado en la parte inferior de la imagen muestra la sección transversal de la coordenada Y designada por la línea azul en la imagen, que representa la vista frontal en la línea azul. La representación 3D de la imagen de la pila z permite a los usuarios ver la imagen en 3D, visualizando todos los planos x, y y z.

Figura 4: Manchado de CD1d y LAMP1. Haga clic aquí para ver una versión más grande de esta figura.
A, panel superior: Las células LCD1 fueron fijas, permeabilizadas y teñidas con anticuerpos a CD1d (rojo) y LAMP-1 (verde, un marcador del compartimento lisosomal). DAPI (azul, se utilizó para visualizar el núcleo). La fusión (panel izquierdo) muestra que CD1d está localizado en el compartimento endosomal/lisosomal positivo LAMP-1 (amarillo).
A, panel inferior: representación 3D de las mismas celdas en el panel superior. Las imágenes se adquirieron utilizando un objetivo de inmersión en aceite de 40x en la Nikon Eclipse Ti,utilizando el software NIS Elements Advanced Research.
B: Imagen de 100x de celdas LCD1d teñidas como en A, con información de pila para una coordenada y particular (denotada por la línea azul) en la parte inferior de la imagen (caja azul). La información de pila para una coordenada X determinada (denotada por la línea rosa) se muestra en el lado derecho de la imagen (caja rosa).
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
La tinción fluorescente confocal es un procedimiento relativamente simple que da como resultado imágenes de muestras de muy alta calidad que se preparan de una manera similar a la de la microscopía de fluorescencia convencional. En resumen, las muestras se fijan, se permeabilizan y luego se bloquean. Se permite que los anticuerpos primarios contra una proteína o proteínas de interés se unan, y luego se utilizan anticuerpos secundarios conjugados con fluoróforos para visualizar la tinción. La microscopía de fluorescencia confocal tiene aplicaciones en muchas áreas de investigación. Por ejemplo, al manchar marcadores de orgánulos subcelulares junto con una proteína de interés, se puede utilizar la microscopía confocal para determinar las ubicaciones subcelulares de diversas proteínas. En comparación con la microscopía de fluorescencia convencional, las imágenes confocales pueden distinguir de manera más eficaz entre la superficie celular y la ubicación intracelular de una proteína. Además, las imágenes confocales también se pueden utilizar para determinar si dos proteínas se colocalizan dentro de la célula. Aunque no se describe en este protocolo, la microscopía de fluorescencia confocal también se puede realizar en células vivas para detectar cambios dinámicos.

Vídeo 1: Vídeo creado en el software NIS Elements Advanced Research, destacando la capacidad de moverse a través de la representación 3D de las imágenes. Haga clic aquí para ver este video (Haga clic con el botón derecho para descargar).
Subscription Required. Please recommend JoVE to your librarian.
References
- Claxton, N. S., Fellers, T. J. and Davidson, M. W. Laser scanning confocal microscopy. Department of Optical Microscopy and Digital Imaging, National High Magnetic Field Laboratory, Florida State University, 37 p., Unpublished (2010). Available at- http://www.vertilon.com/pdf/PP6207.pdf.
- Ojcius, D. M., Niedergang, F., Subtil, A., Hellio, R. and Dautry-Varsat, A. Immunology and the confocal microscope. Research in Immunology, 147 (3),175-88 (1996).
- Paddock, S. W. and Eliceiri K. W. Laser scanning confocal microscopy: history, applications, and related optical sectioning techniques. Methods in Molecular Biology, 1075, 9-47 (2014).
- Hoff. F. How to prepare your specimen for immunofluorescence microscopy. Philipps University Marburg, Institute of Cytobiology and Cytopathology, Germany. (2015) Available at- http://www.leica-microsystems.com.




