Summary
Xenopus ectoderma embrionale è diventato un modello attraente per gli studi di polarità cellulare. Un saggio è descritta, in cui la distribuzione subcellulare delle proteine fluorescenti è valutata in cellule ectoderma. Questo protocollo aiuterà a trattare questioni relative al controllo spaziale della segnalazione.
Abstract
Polarità delle cellule è una proprietà fondamentale delle cellule eucariotiche che viene dinamicamente regolato da fattori intrinseci ed estrinseci, sia durante lo sviluppo embrionale 1, 2. Una delle vie di segnalazione coinvolte nel presente regolamento è la via Wnt, che viene utilizzato più volte durante l'embriogenesi e critica per la malattia umana 3, 4, 5. Molteplici componenti molecolari di questa via di segnalazione coordinatamente regolano in maniera spazialmente limitato, ma i meccanismi alla base non sono pienamente compresi. Xenopus cellule embrionali epiteliali è un ottimo sistema per studiare la localizzazione subcellulare delle diverse proteine di segnalazione. Proteine di fusione fluorescenti sono espressi in embrioni di Xenopus da microiniezione di RNA, gli espianti ectodermiche sono preparati e la localizzazione delle proteine viene valutato epifluorescenza. In questo protocollo sperimentale descriviamo come localizzazione subcellulare di Diversin, una proteina citoplasmatica che è stato implicato nella segnalazione e polarità determinazione delle cellule 6, 7 viene visualizzato in Xenopus cellule ectodermiche per lo studio di trasduzione del segnale Wnt 8. Coespressione di un ligando Wnt o un recettore Frizzled altera la distribuzione di Diversin fuse con il rosso proteina fluorescente, RFP, e reclute alla membrana cellulare in maniera polarizzata 8, 9. Questo protocollo ex vivo dovrebbe essere un'utile aggiunta a studi in vitro su colture di cellule di mammifero, in cui il controllo spaziale del segnale è diversa da quella del tessuto intatto ed è molto più difficile da analizzare.
Protocol
1. Fecondazione in vitro di uova di Xenopus
- Ottenere le uova da rane femminile che sono stati iniettati con gonadotropina corionica umana (400 unità / rana) 12 ore prima dell'esperimento.
- Mettere le uova in una piccola quantità (0.5-1 ml) di soluzione 1 x Marc Ringer modificati (MMR) 10 per le uova e fecondare in vitro con un piccolo pezzo di testicolo sezionato. Dopo 2-3 minuti, aggiungere 0,1 x MMR per coprire tutta la superficie delle uova. In 20 minuti, uova cappotto gelatina viene rimosso da 3% cisteina - HCL (portata a pH 8 con idrossido di sodio). Le uova vengono lavate con 0,1 x MMR tre volte e lasciato in un freddo incubatore (13 ° C) per preparazioni iniettabili.
- Uova fecondate sono permesso di sviluppare a 2-4 stadio cellulare. Per preparazioni iniettabili, gli embrioni vengono trasferiti nella soluzione contenente il 3% Ficoll, 0,5 x MMR.
2. RNA Microiniezione
- RNA vengono sintetizzate dai modelli di DNA linearizzato utilizzando mMessage mMachine kit (Ambion) e diluita con acqua RNase-free alla concentrazione stock di 0,1-1 mg / mL. Dosi ottimali di RNA per preparazioni iniettabili sono determinati in esperimenti pilota. RNA per Diversin-RFP, il marcatore membrana GFP-CAAX Frizzled e 8 sono utilizzati al 0,1-1 ng per iniezione.
- Gli aghi da iniezione sono preparati con un estrattore ago da una capillare e poi con una smerigliatrice ago. Prima dell'iniezione, ogni ago è calibrato con acqua per espellere 10 nl di iniezione di liquido per.
- Per preparazioni iniettabili, gli embrioni sono posti su un piatto di plastica in una grande goccia del 3% Ficoll, 0,5 x MMR. Una microlitri di soluzione RNA è risucchiata in un ago da iniezione con Narishige microinjector. 10 nl di RNA viene iniettato in blastomeri animali embrioni di 8 cellule 2-3 volte. Gli embrioni iniettati vengono trasferiti in un pozzo di 12-pozzetti.
3. Preparazione ectodermica Espianti
- Quando gli embrioni iniettati raggiungere presto stadio di gastrula, vengono trasferiti in una soluzione 0,6 x MMR in un piatto di 3 centimetri di plastica rivestiti con 1% di agarosio. Membrana vitellina viene rimosso manualmente con un paio di pinze. Espianti ectodermica sono asportati dagli embrioni utilizzando un ago di tungsteno e un hairloop.
- Espianti ectodermica vengono trasferiti in un flacone di vetro e fissato con 3,7% di formaldeide in soluzione salina tamponata con fosfato (PBS) per 30 minuti. Espianti fissi vengono lavati con PBS per tre volte (10 minuti ciascuno). DAPI è incluso nel terzo lavaggio a macchia nuclei.
- Espianti sono montati su un vetrino. Due strisce di nastro adesivo sono attaccati alla slitta e la espianti sono posti tra le due strisce. Dal momento che la superficie esterna di espianti è pigmentata, la parte interna del espianti deve essere rivolto l'obiettivo microscopio. Aggiungere 2-3 gocce 20 ml di soluzione di montaggio (70% glicerolo in PBS di cui 25 mg / ml DABCO, anti-sbiadimento reagente) 11. Mettere un coprioggetti sopra.
4. Imaging di Espianti al microscopio a fluorescenza
- I campioni sono visti al microscopio Zeiss Axioplan a fluorescenza con filtri appropriati.
- Le immagini sono scattate con l'attaccamento Apotome di visualizzare uno specifico piano da diversi espianti indipendenti.
5. Criosezionamento
Criosezionamento è un modo alternativo per visualizzare la distribuzione delle proteine fluorescenti nella cella e più applicabile per immunocolorazione. Nella fase 10, gli embrioni sono fissati per 1-2 ore con fissativo Dent (20% DMSO, 80% metanolo), lavate con PBS, e incorporate nel 15% soluzione di saccarosio pesce gelatin/15% 11. Gli embrioni congelati sono rapidamente integrati in ghiaccio secco e criosezioni sono generate su Leica criostato. Sezioni che comprendono le cellule ectodermiche che ereditano RNA iniettato e loro prodotti proteici tradotte. Le sezioni mantenere fluorescenza e può essere immunoistochimica con anticorpi specifici e poi etichettati con anticorpi secondari coniugati con fluorescenza. I nuclei sono colorati con DAPI. I mezzi di montaggio sono gli stessi come descritto sopra. L'imaging può essere eseguita come descritto sopra.
6. Rappresentante dei risultati:
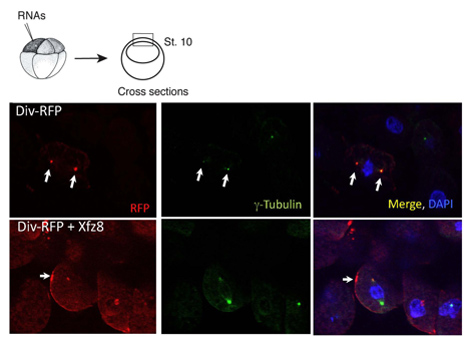
Figura 1. Recettore Frizzled reclute Diversin alla membrana cellulare. Cellule che esprimono Ectoderma 8 Frizzled (Xfz8) e Div-RFP RNA rivelare Div-RFP a livello della membrana cellulare, al posto del centrosoma (come rivelato da g-tubulina co-scarico). Lo schema dell'esperimento è al top, una tipica sezione trasversale è mostrato.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Abbiamo usato il protocollo di cui sopra per caratterizzare la localizzazione subcellulare di Diversin. In espianti polo animale, Diversin-RFP è stata rilevata accanto al nucleo e colocalized con g-tubulina, un marker centrosomica, in criosezioni (Figura 1). Una volta che la localizzazione subcellulare di una proteina è identificato, costruisce la cancellazione può essere generata per stabilire quali domini di proteine sono necessarie e sufficienti per la localizzazione subcellulare. Usando questo approccio, i domini di localizzazione centrosomica Diversin sono stati mappati al centro e nella regione carbossi-terminale domini della proteina, entrambi contenenti il motivo a spirale-coil 8.
Lo stesso protocollo può essere utilizzato negli studi, nei quali la localizzazione delle proteine è alterata in risposta alla segnalazione. Abbiamo scoperto che le proteine Wnt secrete agire per riposizionare Div-RFP puntata alle strutture adiacenti alla membrana cellulare, mentre FZ8 reclute Div-RFP alle patch membrana cellulare (Figura 1). Abbiamo inoltre scoperto che il dominio carbossi-terminale non è essenziale per la membrana di reclutamento di per sé, ma necessari per l'assunzione a membrana polarizzata.
In sintesi, il protocollo sperimentale sopra vi aiuterà a diversi studi di interazioni proteina-proteina e localizzazione delle proteine ai diversi compartimenti cellulari dopo la stimolazione delle cellule con fattori di crescita specifici o proteine di segnalazione.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Nessun conflitto di interessi dichiarati.
Acknowledgments
La ricerca nel laboratorio Sokol è patrocinata dal Institues Nazionale di Salute.
References
- Gurdon, J. B. Embryonic induction --- molecular aspects. Development. 99, 285-306 (1987).
- Principles of Developmental Genetics. Moody, S. A. , Academic Press. (2007).
- Clevers, H. Wnt/beta-catenin signaling in development and disease. Cell. 127, 469-480 (2006).
- Klaus, A., Birchmeier, W. Wnt signalling and its impact on development and cancer. Nat Rev Cancer. 8, 387-398 (2008).
- Gordon, M. D., Nusse, R. Wnt signaling: multiple pathways, multiple receptors, and multiple transcription factors. J Biol Chem. 281, 22429-22433 (2006).
- Schwarz-Romond, T., Asbrand, C., Bakkers, J., Kuhl, M., Schaeffer, H. J., Huelsken, J., Behrens, J., Hammerschmidt, M., Birchmeier, W. The ankyrin repeat protein Diversin recruits Casein kinase Iepsilon to the beta-catenin degradation complex and acts in both canonical Wnt and Wnt/JNK signaling. Genes Dev. 16, 2073-2084 (2002).
- Moeller, H., Jenny, A., Schaeffer, H. J., Schwarz-Romond, T., Mlodzik, M., Hammerschmidt, M., Birchmeier, W. Diversin regulates heart formation and gastrulation movements in development. Proc Natl Acad Sci U S A. 103, 15900-15905 (2006).
- Itoh, K., Jenny, A., Mlodzik, M., Sokol, S. Y. Centrosomal localization of Diversin and its relevance to Wnt signaling. J. Cell Sci. 122, 3791-3798 (2009).
- Itoh, K., Jacob, J., Sokol, Y. S. A role for Xenopus Frizzled 8 in dorsal development. Mech Dev. 74, 145-157 (1998).
- Newport, J., Kirschner, M. A major developmental transition in early Xenopus embryos: I. characterization and timing of cellular changes at the midblastula stage. Cell. 30, 675-686 (1982).
- Itoh, K., Brott, B. K., Bae, G. U., Ratcliffe, M. J., Sokol, S. Y. Nuclear localization is required for Dishevelled function in Wnt/beta-catenin signaling. J Biol. 4, 3-3 (2005).



