Introduction
Insekter spiller en viktig rolle ved overføring av mat-relaterte sykdommer, fordi de kan spre patogener til næringsmidler eller næringsmiddelkontaktflater og redskaper 1. Blant insekter, fluer, kakerlakker, og maur utvise atferd som favoriserer spredning av matbårne patogener. Disse atferd inkluderer en forening med råtnende saken, nekter og avføring, endophily (inn bygninger), og synanthropy (samboer med mennesker) 2. .. Matbårne patogener som Salmonella spp, Listeria monocytogenes, Campylobacter spp, Escherichia coli O157: H7, og medlemmer av slekten Cronobacter (tidligere Enterobacter sakazakii) har blitt rapportert å bli sendt av insekter 3-5. Synanthropic skitt fluer mekanisk spre matbårne bakterier ved å overføre patogener fra sine forurensede kroppsoverflater. Imidlertid kan tilstedeværelsen av matbårne patogene i fordøyelseskanalen av fluer være opptil tre gangerstørre enn det som ble observert på sine kroppsoverflater (kropp, hode, ben og vinger) 5. Matbårne patogener kan også forbli i flua fordøyelsessystemet kanalen for et større tidsrom enn på kroppsoverflaten 6,7 og i noen tilfeller, er de i stand til å formere seg, kolonisere flua fordøyelseskanalen 4,8,9. Dette øker vektorpotensialet av fluer fordi de kan ytterligere spre matbårne patogener gjennom avføring og oppstøt 10,11.
I dag er det bedre overvåkningssystemer som er i stand til å oppdage matbårne sykdomsutbrudd raskere. Mens du utfører matbårne utbrudd undersøkelser, offentlige helsemyndigheter se etter mat som kan være kilden (e) eller kjøretøy (er) for infeksjon. Etterforskerne kan også utføre en miljøvurdering av anlegget (eller anlegg) som er involvert for å finne ut hvordan maten var forurenset og kan samle inn prøver som en del av etterforskningen 12. Despite den enorme mengden av vitenskapelig litteratur om insekter som bærere av matbårne patogener, linking insekter som vektorer av patogenet som forårsaker en bestemt matbåren sykdom utbrudd har vært utfordrende. Dette er hovedsakelig fordi insekter ikke blir aseptisk samlet som en del av miljø prøvetaking programmer under matbårne utbrudd undersøkelser. Å inkludere insekter, spesielt de som utviser atferd som favoriserer spredning av matbårne patogener, som en del av en miljøprøvetaking prosedyre, en standardisert, rask, følsom og pålitelig protokoll for å oppdage matbårne patogener fra en enkelt insekt må være på plass.
Tradisjonelle plette teknikker for påvisning av matbårne patogene fra insekter er arbeidskrevende og avhengig av den konkurrerende vekst av mål-bakterier i forskjellige dyrkningsmedier for å overvinne den raske veksten av det medfødte kommensal bakterieflora av insekt. De fleste av studiene som har forbundet insekter med bacterial patogener har øket følsomheten for fremgangsmåten ved å blande sammen flere insekter i stedet for identifisering av nærværet av patogener i en per individ basis. Således har disse studier ikke skille hoveddelen av insekt hvor patogener ble funnet 13-18. Evnen til å identifisere om matbårne patogener er plassert på kroppsoverflaten eller i fordøyelseskanalen til et individ insekt er viktig da dette kan ha epidemiologiske implikasjoner og kan føre til ulike løsningsstrategier. Som mekaniske vektorer, fluer som landet på mat for en kort tid kan bare overføre lave nivåer av bakterier fra sin kroppsoverflate, mens de fluer som regurgitate og defekt på mat øke sannsynligheten for overføring av patogener ved potensielt høyere nivåer av infeksjon. Følgelig er det viktig å estimere forekomsten av matbårne patogene per en individuell insekt og å skille hoveddelen av at insekt hvor den bakterielle p athogen ligger.
Selv om bruken av kultur-uavhengige metoder for å detektere matbårne patogene i økende grad blir gjennomført, har de ikke vært kommersielt anvendt for å detektere matbårne patogener fra en enkelt insekt. Foreløpig er det validert molekylære protokoller som er kommersielt tilgjengelig for rask påvisning av matbårne patogener fra matvarer som blir brukt av industri og offentlige instanser. Disse metodene omfatter DNA-baserte systemer for påvisning av patogener i en rekke matvareprøver. Selv om molekyl protokoller er raskere enn tradisjonelle metoder kontaktflate, er anriking av prøven fremdeles nødvendig for å oppnå følsomhetsnivået for 10 to kolonidannende enheter (CFU) av det bakterielle patogenet nødvendig i polymerase kjedereaksjon (PCR) -baserte Metode 19. Dessuten er isolering av rene bakteriekolonier fra PCR-positive prøvene er nødvendig for å bekrefte patogenet ved hjelp av egnede metoder.
innhold "> Formålet med denne protokollen er å standardisere en kommersielt tilgjengelig PCR-basert system som brukes til å detektere patogener fra mat og miljøprøver for deteksjon av matbårne bakterier fra kroppen overflate og fordøyelseskanalen av et enkelt fly, og å isolere dem videre patogener fra samples.The følsomhet av protokollen beskrevet her ble først kalibrert med lab-oppdratt voksen husfluer (Musca domestica) som ble eksperimentelt matet med serielle fortynninger av hver bakteriell patogen. Den standardiserte protokollen ble senere brukt til å kartlegge 100 villfanget flyr for tilstedeværelsen av matbårne patogener fra sine kroppsoverflater og / eller fordøyelses kanalene. Dette vil standardiserte protokollen tillater folkehelse laboratorier for å oppdage helse truslene fra insekter, noe som åpner for muligheten for å samle dem som en del av miljøprøvetakingsprogram når du utfører matbåren utbrudds undersøkelser.Protocol
1. Innsamling av fluer
- Samle individuelle fluer ved hjelp av sterile entomologiske feie garn. Sette garn i en kjøligere og overføre dem til laboratoriet.
2. Disseksjon av Fluer
- Immobilisere aseptisk innsamlede fluer ved å plassere dem ved -20 ° C i 5 - 7 min.
- Ved hjelp av sterile pins sted en flue i et sterilt rør inneholdende 2 ml 1 ml forvarmet (37 ° C) bufret peptonvann (BPW). Bland forsiktig ved å vende røret i 2 min. Det er viktig at hele kroppen av fluen være i kontakt med mediet, slik at bakterieflora tilstede på kroppsoverflaten (S) av flue vil bli overført til BPW (BPW-S). Merke røret med et nummer og kroppsdel av flua (dvs. 1S).
MERK: Vennligst se tabell over spesifikke reagenser / Utstyr for en detaljert beskrivelse av materialer og reagenser som er nevnt i denne protokollen. - Ved hjelp av steril tang fjerne fly fra BPW-S media og overføringdet til et tomt og rent 2 ml tube til overflaten desinfisere fly. Inkuber røret som inneholder BPW-S medier ved 37 ° C under gjennomføring av desinfeksjon og disseksjon protokoll.
- Surface-desinfisere fly ved å dyppe den i en ml 70% etanol for 1 min, etterfulgt av en skylling med sterilt destillert vann før dyppe den i 1 ml nylaget 0,05% (v / v) kloroppløsning. Skyll tre ganger med sterilt destillert vann. Overføre vann fra siste skylling til en autoklaveres 2 ml tube.
MERK: Kast væske hver gang ved hjelp av en 1000 mL mikropipette eller ved å snu røret, noe som gjør at fly er fortsatt inne i røret. Bland forsiktig ved å snu opp ved hvert trinn av det overflatedesinfeksjonsprosessen. - For å evaluere effektiviteten av renseprosessen, overføre 100 ul av vannet fra siste skylling i en trypticase soya-agar (TSA) plate og spres ved hjelp av en steril L-formet engangs sprederen. Inkuber platen ved 37 ° C i 24 timer. Etter icubation, registrere tilstedeværelsen av eventuelle bakteriekolonier.
MERK: Tilstedeværelsen av bakteriekolonier på TSA plater indikerer en ineffektiv overflatedesinfeksjonsprosessen. Hvis dette skjer, bør tilstedeværelsen av matbårne patogener kun rapporteres på kroppsoverflaten av flua fordi krysskontaminering mellom kroppsoverflaten og fordøyelsessystemet kanalen ikke kan utelukkes.
- Surface-desinfisere fly ved å dyppe den i en ml 70% etanol for 1 min, etterfulgt av en skylling med sterilt destillert vann før dyppe den i 1 ml nylaget 0,05% (v / v) kloroppløsning. Skyll tre ganger med sterilt destillert vann. Overføre vann fra siste skylling til en autoklaveres 2 ml tube.
- Etter overflate desinfisering av fly, overføre den til et stykke Autoclaved papir håndkle for å fjerne overflødig vann og deretter til et sterilt 60 mm disponibel petriskål.
- Plasser petriskål under en dissekere omfang og identifisere fly til artsnivå ved hjelp av dikotome tastene for tovingede familier 20,21.
- Ved hjelp autoklaveres fin spiss tang dra forsiktig i anus og hele fordøyelsessystemet kanalen (A) ut av fly og aseptisk overføre den til en annen steril 2 ml tube inneholder 1 ml forvarmet (37 ° C) BPW med 0,5 mm zirkonia / silika perler (BPW-A). Merk the rør med samme nummer som er valgt for de enkelte utlegger og hoveddelen av det fly (dvs., 1A).
- Bland røret som inneholder BPW-A grundig i 5 - 10 min ved å bruke en celle disruptor. Inkuber ved 36 ± 1 ° C, mens de utfører resten av protokollen.
- Til bilag og / eller oppbevare prøven for langsiktig, plasserer resten av flua i en ren 2 ml tube og tilsett 1 - 2 ml 95% etanol.
3. Primær og Sekundær berikelse
- Merke alle primære og sekundære berikelse rør inneholdende media i henhold til den eksempelnummer, og hoveddelen av fly.
- Under en steril hette, overføre 300 ul BPW-S (overflate) til sterile 2 ml rør som inneholder følgende media:
- For Salmonella, må du bruke en ml forvarmet (42 ° C) BPW. Inkuber i et resirkulerende vannbad ved 42,5 ° C i 22 - 24 timer. For sekundær berikelse, overføre 100 mL av anriket BPW til 400 & #181; l av forvarmet (37 ° C), hjerne-hjerte-infusjon (BHI) tidligere plassert i sterile klase rør. Inkuber ved 37 ° C i 3 timer.
- For Cronobacter, bruke 1 ml forvarmet (37 ° C) BPW med novobiocin (10 mg / l; Wallace, M., personlig kommunikasjon). Alternativt kan du bruke en ml av R & F Enterobacter sakazakii anrikningsbuljong med supplement (vancomycin og cefsulodin) som primær berikelse. Inkuber ved 37 ° C i 22-26 timer. For sekundær anrikning, overføre 100 ul av anriket BPW med novobiocin i 400 ul av forvarmet (37 ° C) BHI-buljong som tidligere er lagt inn i sterile klase rør. Inkuber ved 37 ° C i 3 timer.
- For L. monocytogenes, bruke 1 ml nylaget RT 24 Listeria anrikningsbuljong (24 LEB) med selektiv supplement. Inkuber ved 37 ° C i 44 ± 5 timer. Ingen sekundære anrikning er nødvendig for påvisning av L. monocytogenes. Gjenta trinn 3.2.1 - 3.2.3 ovenfor bruker røret merket som BPW-A.
4. Utarbeidelse av PCR-basert system for Amplification og Påvisning av Target matbårne Patogen
Trinnene 4-8 bruke en kommersiell PCR cycler / detektor system, en datamaskin arbeidsstasjon, og klar til bruk kits til skjermen for Salmonella (Salmonella to standard analyse kit), Cronobacter arter (E. sakazakii standard analyse kit), og Listeria monocytogenes (L. monocytogenes 24E analysen kit). Standardanalyser bruke PCR end-point deteksjon. Hvert sett inneholder PCR-ready tabletter med en interkalerende fargestoff som avgir en fluorescens-signal ved binding til dobbeltkjedet DNA. Signalet blir tatt i løpet av deteksjons fase av PCR-system program, å generere et smeltekurve som tolkes av programvaren som positive eller negative.
- Forbered reagenser og utstyr som spesifisert av produseresrer protokoll per hvert mål matbåren patogen.
MERK: Protokollene for påvisning Salmonella og Cronobacter krever en ett-trinns lyse prosedyre mens protokoll for påvisning av L. monocytogenes krever en to-trinns prosedyre lyse (se avsnitt 5 og 6, henholdsvis). - Slå på automatisert varmeblokk velge det bestemte programmet for målpatogenet. Alternativt, hvis varme blokkene er manuell, setter temperaturen til 37 ° C (for Salmonella, Cronobacter spp, og L. monocytogenes.) Eller til 55 ± 2 ° C (for L. monocytogenes del 2 av lyse - se trinn 6.2) og 95 ± 3 ° C.
- Pass på at kjøleblokkene har blitt nedkjølt O / N, ellers slappe dem ved 2 - 8 ° C i minst 2 timer.
- Bruke dataprogram av PCR-basert gjenkjenning system, lage et rack fil etter produsentens anvisninger.
- Merke og ordne klyngeRørene inneholdende den lyse reagens i stativet, i henhold til stativet fil.
- Initialisere PCR-basert gjenkjenning system instrument.
5. Utfør Lysis for påvisning av Salmonella og Cronobacter
- Klargjør lysis-reagens ved å tilsette 150 ul av protease til en 12 ml flaske med lyseringsbuffer.
- Overfør 200 ul lyserings reagens til hvert av de tidligere merkede klase rør.
MERK: Cluster rør som inneholder lysereagensen som kan lagres ved 2 - 8 ° C i opptil 2 uker. - Bruke lange pipettespisser, overføre 20 mL av sekundære beriket prøver (se trinn 3.2.1 og 3.2.2) til tilsvarende klynge rør som inneholder 200 mL av lysis reagens. Bruk nye pipettespisser for hver prøve.
MERK: Hold rør fra primær- og sekundær berikelse i kjøleskapet (Salmonella) eller ved romtemperatur (Cronobacter) for ytterligere bekreftelse analyse av PCR-positive / negative prøver. Forbered negative kontroller ved å legge til 20 mikroliter sterilt BHI media til klase rør som inneholder 200 mL av lysis reagens. - Forbered positive kontroller ved å legge til 20 pl O / N bakteriekulturer (dyrket i BHI) av alle kjente Salmonella eller Cronobacter belastning å klase rør som inneholder 200 mL av lysis reagens.
- Cap klase rør og sikre tett med capping verktøyet.
- Plasser rack av klase rør i den automatiserte varmeblokken etter å ha valgt den spesifikke program for målpatogenet. Alternativt inkuber klase rør ved 37 ± 2 ° C i 20 min, etterfulgt av inkubasjon ved 95 ± 3 ° C i 10 min. Til slutt overfører klynge rør til kjøleblokker (2 - 8 ° C) i 5 min.
MERK: Cluster rør inneholdende lysat kan lagres ved -20 ° C i opp til 2 uker.
6. Utfør Lysis for påvisning av L. monocytogenes
- Utfør del en avlyse som følger:
- Legg 1,8 ml sterilt avionisert vann til flaske helt tint lyserende midlet 1.
MERK: Oppbevar lyseringsmiddel en ved 2 - 8 ° C til den er klar til bruk. Etter åpning og fortynning, butikken på RT (20 - 30 ° C) i inntil seks måneder. - Kombiner lysemidler 1 og 2 i en 4: 1-forhold (40 ul fortynnet lyseringsmiddel og en 10 pl av lyseringsmiddel 2 per hver prøve). Overfør 50 ul av de kombinerte lyserende midler til å klynge rør. Bruk massen innen 4 timer.
- Legg 500 ul av primær anriket prøve (se trinn 3.2.3) til klyngen rør inneholdende 50 ul av de kombinerte lyserende midler.
- Forbered en negativ kontroll ved å legge 500 mikroliter sterilt 24 LEB til 50 mL av den kombinerte lyserende midler.
- Forberede en positiv kontroll ved å legge 500 mL av O / N L. monocytogenes kultur dyrket i 24 LEB til 50 ul av de kombinerte lyserende midler.
- Cap klase rør, bland forsiktig og stedi varmeblokk ved 37 ± 1 ° C i 30 min.
MERK: Hold rør fra primær berikelse i kjøleskapet i ytterligere bekreftelse analyse av PCR-positive / negative prøver.
- Legg 1,8 ml sterilt avionisert vann til flaske helt tint lyserende midlet 1.
- Utføre en del 2 av lysis som følger:
- Forberede lysereagensen som beskrevet i trinn 5.1 og 5.2.
- Ved hjelp av lange pipetter overføre 20 mL av del én lysat til klase rør som inneholder 200 mL av lysis reagens. Bruk nye pipettespisser for hver prøve.
- Cap klase rør og sikre tett med capping verktøyet.
- Sted klase rør i automatisert oppvarming blokk velge den spesifikke program for L. monocytogenes. Alternativt inkuber klase rør ved 55 ± 2 ° C i 30 minutter, etterfulgt av inkubasjon ved 95 ± 3 ° C i 10 min. Til slutt overfører klynge rør til kjøleblokker (2 - 8 ° C) i 5 min.
MERK: Cluster rør inneholdende lysat kan lagres ved -20 ° C i opp til 2 uker.
7. Hydrate PCR-Ready Tabletter
- Velg en kjølt (4 ° C) PCR kjøleblokk og plassere en PCR rør stativ over innsatsen.
- Plassere tilsvarende PCR-rør inneholdende PCR-ready tabletter (inkludert i hvert sett) for målet matbårne patogene i holderen, i henhold til stativet fil.
- Bruke decapping verktøy, nøye fjerne dekslene fra PCR-rør. Forkaste caps og kontrollere at hver tube inneholder en tablett.
- Transfer 50 III (for Salmonella og Cronobacter) eller 30 pl (for L. monocytogenes) av lysat til spesifikke PCR-rørene. Bruk nye optiske caps og sikre fast på PCR-rør ved hjelp av tildekking verktøyet.
MERK: Når du har lagt lysatet til PCR-klar tabletter, prøvene må være kjølt ved 2 - 8 ° C inntil lastet inn i PCR-basert gjenkjenning system. De PCR-rørene kan sentrifugert ved 2500 xg i noen sekunder for å sikre at hele volumet er i den nederste than tube. - Laste PCR rør i PCR cycler / deteksjon system instrument ved å åpne instrumentet skuff.
- Plasser rack av PCR-rørene i brønnene i skuffen, og kontroller at rørene sitter riktig.
- Lukk skuff og initiere program som beskrevet av produsentens protokoll.
MERK: PCR-basert instrument har forhåndsinnstilt sykkelparametre for hver matbåren patogen. - Kontroller at PCR sykling statuslinjen viser en blå bar som indikerer at forsterkning delen av programmet er i gang.
MERK: For standard PCR-analyser, behandlingstiden av hele programmet (forsterkning og deteksjon) tar orden ca. 3 - 3,5 timer å fullføre.
8. Gjennomgå resultatene
- Når behandlingen er fullført, følger instruksjonene på skjermen fra PCR-basert system instrument for å fjerne prøver og gjennomgang resultater.
- Hvis målet matbårne patogene er til stede i prøven (entenoverflate eller fordøyelseskanalen av flua) brønnen er rød med en "pluss" tegn (positiv). Hvis patogenet er fraværende, er vel grønn med en "minus" tegn (negativ).
- Dersom brønnen er gul med en rød strek over midten, indikerer det en signalfeil.
9. Isolering av bakterielle patogener fra PCR-positive resultater
- Velg rør fra den primære (for L. monocytogenes) eller sekundær (for Salmonella og Cronobacter) berikelse av disse prøvene som var PCR-positive. Også tilfeldig velge 3 - 5% av prøvene som ble PCR-negative og gjør som følger:
- For Salmonella:
- Tilsett 100 ul av den sekundære berikelse media til 10 ml av Rappaport-Vassiliadis (RV) medium og 1 ml tetrationat (TT) kokes. Inkuber rørene ved 42,5 ° C i et resirkulerende vannbad i 22 - 24 timer.
- Etter inkubasjon strek en 3 mm løkkefull (10 ul) av hver, en RVd TT media på vismut sulfitt (BS) agar, xylose lysin desoksykolat (XLD) agar, og Hektoen ente (HE) agar. Inkuber platene ved 35 ± 1 ° C i 22 - 24 timer.
- Etter inkubasjon undersøke plater for tilstedeværelse av Salmonella typiske kolonier på hvert medium. Hvis ingen isolerte kolonier kan oppnås etter flere sub-dyrkning av fremgangsmåten, bør prøven som negative og rapportere som en falsk positiv for PCR-basert system.
MERK: For typiske Salmonella kolonier på bestemte media se 22. Velg fem presumptive typiske Salmonella kolonier og subkultur dem på BS, XLD, eller HE til rene kulturer av isolerte / enkeltkolonier oppnås. - Velg en ren koloni og identifisere presumptive Salmonella ved hjelp av biokjemiske kommersielle tester som VITEK to identitetskort eller API biokjemiske identifikasjonssystem, etter produsentens anvisninger.
- For Cronobacter: <ol>
- Strek en 3 mm løkkefull (10 pl) av sekundær berikelse media på to plater av kromogeniske kulturmedier som R & F Enterobacter sakazakii (Cronobacter) kromogen plating medium, og / eller ChromID sakazakii Agar. Inkuber platene ved 35 ° C i 22 - 24 timer.
- Etter inkubasjon undersøke plater for nærværet av typiske Cronobacter kolonier (blå-svart til blå-grå). Velg fem presumptive Cronobacter kolonier og subkultur dem på R & F Enterobacter sakazakii (Cronobacter) kromogen plating medium, ChromID sakazakii Agar, eller TSA til rene kulturer av isolerte / enkeltkolonier oppnås.
MERK: Hvis ingen isolerte kolonier kan oppnås etter flere under-dyrking skritt, vurdere prøven som negativ og rapportere som en falsk positiv for PCR-basert gjenkjenning system. - Velg en ren koloni og identifisere presumptive Cronobacter ved hjelp av biokjemiske kommer-sielle tester som VITEK to identitetskort eller API 20E biokjemisk identifikasjon system, etter produsentens anvisninger.
- Serie en 3 mm løkkefull (10 ul) av den primære anrikning media på to plater av glans Listeria agar (bla). Inkuber platene ved 36 ± 1 ° C i 22-26 timer.
- Etter inkubasjon undersøke platene for tilstedeværelsen av presumptive L. monocytogenes (blå-grønn) kolonier. Velg fem presumptive L. monocytogenes kolonier og subkultur dem på BLA til å skaffe rene kulturer av isolerte / enkeltkolonier. Re-negative plater inkuberes ved 36 ± 1 ° C i ytterligere 22-26 timer.
- Velg en ren koloni og identifisere presumptive L. monocytogenes ved hjelp av kommersielle biokjemiske tester som VITEK to identitetskort eller API Listeria biokjemiske identifikasjonssystem, følger produsentens jeginstruksjonene angis.
Representative Results
Denne protokollen ble først kalibrert på et sett av lab-alet husfluer som ble eksperimentelt matet i 24 timer med væskeflue mat (2% pulvermelk) inneholdende seriefortynninger (februar 10 til august 10 CFU / ml) av C. sakazakii, S. ente, L. monocytogenes, eller C. jejuni (n = 21 for hver bakteriell patogen). Anrikningsmedier samt inkubasjonstider og temperaturer ble justert for hver matbårne patogene til PCR-basert system var i stand til å gjenkjenne de laveste nivåene av bakterier (10 2 CFU / ml) fra kroppsoverflaten og fordøyelseskanalen av en enkelt eksperimentelt matet fly. Bruke anrikningsmedier og vilkårene som er beskrevet i protokollen delen, PCR-basert system oppdaget C. sakazakii, S. ente, og L. monocytogenes fra kroppsoverflaten av 100% av fluene ble matet med bakterielle Inokula> 10 3 CFU / ml (Figur 1a). Når fluene ble matet med 102 CFU / ml, andelen av påvisning av C. sakazakii, S. ente, og L. monocytogenes fra sin kroppsoverflate var 100%, 66% og 33%, respektivt (figur 1A). PCR-basert system også oppdaget disse tre matbårne patogener fra fordøyelseskanalen av fluer matet med alle bakteriekonsentrasjon på prosenter ≥33% (figur 1B). Imidlertid påvisning av C. jejuni ble bare oppnådd når lab-alet fluer ble eksperimentelt matet med flytende føde inneholdende den høyeste bakterieinokulum (10 8 CFU / ml). Derfor C. jejuni ble ekskludert fra gruppen av matbårne patogener som kan testes fra individ synanthropic skitt fluer bruker denne PCR-basert gjenkjenning system.
Med denne standardiserte protokollen, var vi i stand til å fastslå utbredelsen av Cronobacter spp., S. ente, og L. monocytogenes fra kroppsoverflaten og / eller the fordøyelsessystemet kanalen på 100 vill fluer som var individuelt og aseptisk fanget fra søppelcontainer område av ti urbane restauranter ligger i hovedstadsområdet i Washington, DC 5 samlet skitt fluer var representative for minst seks arter inkludert M. domestica (47%), Lucilia cuprina (33%), L. sericata (14%), Cochliomyia macellaria (2%), Sarcophaga haemorrhoidalis (2%), og Ophyra leucostoma (1%). Ett fly ble identifisert bare til familienivå (Anthomyiidae; 1%). Overflaten-desinfeksjon protokollen var effektiv på å unngå kryssforurensning mellom kroppsdeler på flua fordi ingen bakterievekst ble observert på TSA plater for vann fra siste desinfeksjon skylling av hver enkelt fly. Dermed kan det skilles mellom matbårne bakterier tilstede på kroppsdeler fra hvert fly.
Ingen falske positiver ble registrert fra prøver av kroppsoverflaten og fordøyelseskanalen avindividuelle fluer ved bruk av denne kommersielle PCR-basert system for påvisning av S. ente og L. monocytogenes, og bekreftelse av levedyktige patogener på agar plater var i enighet med PCR-positive resultater. Det var imidlertid ikke mulig å isolere rene kulturer av Cronobacter spp. fra alle PCR-positive prøver. Derfor påvisning av dette patogenet ved PCR-basert Systemet viste falske positiver fra kroppsoverflaten (50%; 9/18) og fordøyelsessystemet kanalen (48%; 16/33) av enkeltvillfanget fluer. Tilfeldig utvalgte PCR-negative prøvene som ble belagt på bestemte medier, bekreftet fravær av matbårne patogener. Derfor ble ingen falske negativer detektert fra en hvilken som helst av prøvene ved bruk av denne kommersielle PCR-basert system for å detektere Cronobacter spp., S. ente, eller L. monocytogenes.
Bare de PCR-positive prøver der patogenet ble isolert og bekreftet ble betraktet som positiveog inkludert for statistisk analyse. Den samlede nærvær av matbårne patogener i fordøyelseskanalen av villfanget skitt fluer var betydelig høyere enn på kroppsoverflaten (χ 2 = 6,8772, df = 1, p = 0,0087). 22% av fordøyelses kanalene og 8% av kroppsoverflater av innsamlede vill fluer var positive for minst en av de tre matbårne patogene (figur 2). Samlet, utbredelsen av Cronobacter spp. verken på kroppsoverflater eller fordøyelses kanalene i innsamlede fluer var statistisk høyere (19%; Fishers eksakte test p = 0,0165) enn utbredelsen av S. ente (7%) og L. monocytogenes (4%). Det ble imidlertid ingen statistiske forskjeller observeres når du utfører parvise sammenligninger mellom kroppsdeler fra fluene for hver bakteriell patogen (Figur 3; Fishers eksakte test p = 0,1464, p = 0,1184, og p = 0,6212 for Cronobacterspp., S. ente, og L. monocytogenes, henholdsvis). Ingen av fluene var positiv for alle tre evaluerte patogener. Men tre av fluer (to L. cuprina og ett L. sericata) gjennomført Salmonella spp. og L. monocytogenes på overflaten eller i fordøyelseskanalen.
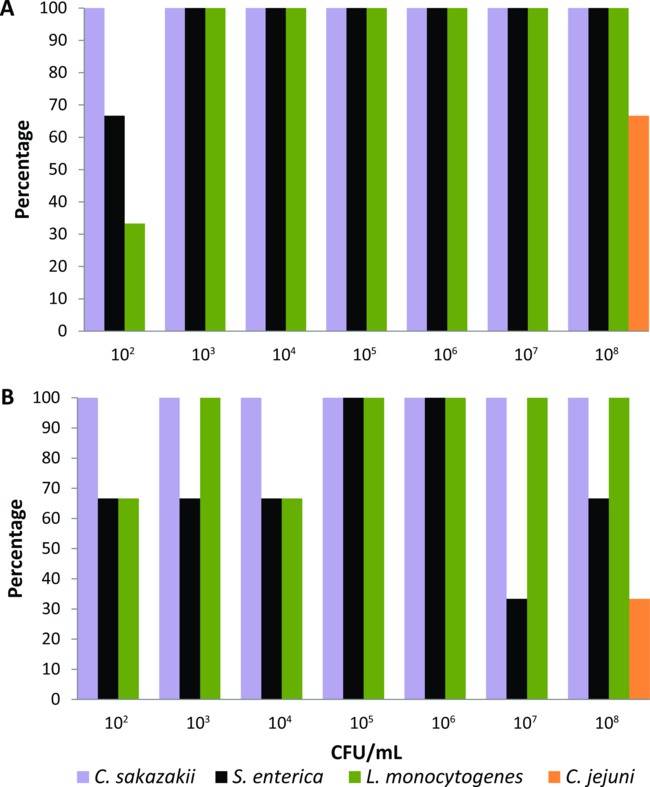
Figur 1. Deteksjons nivåer av Cronobacter sakazakii, Salmonella ente, Listeria monocytogenes, og Campylobacter jejuni fra (A) kroppsoverflaten og (B), fordøyelseskanal individuelle lab-oppdrettet huset fluer matet med flytende mat som inneholder forskjellige bakterie inocula (n = 21 for hver bakteriell patogen, n = 3 for hver bakteriekonsentrasjon).
Figur 2. Andel av kroppsoverflater og fordøyelses kanalene i enkelte fluer funnet positive for noen av de mål matbårne patogener.

Figur 3. Prevalens av Cronobacter spp., Salmonella ente, og Listeria monocytogenes fra kroppsoverflaten og fordøyelseskanalen av synanthropic villfanget fluer. P-verdiene som er rapportert er fra parvise sammenligninger mellom kroppsoverflaten og fordøyelsessystemet kanalen for hver bakteriell patogen (Fishers eksakte test, p-verdi <0,05 indikerer statistisk signifikans). Copyright © American Society for Microbiology, Journal of Applied and Environmental Microbiology 78 (22): 7891-902, 2012. doi: 10,1128 / AEM.02195-12.
Discussion
Tidligere studier som har påvist matbårne patogener fra ville insekter har brukt et stort utvalg av protokoller som kanskje ikke har den nødvendige informasjonen til å nøyaktig vurdere næringsmiddelrelatert risiko for tilstedeværelsen av en enkel flue i matvarer eller mat-relaterte miljøer 13,15, 23,24. Her viste vi at ved hjelp av denne standardiserte protokollen, er det mulig å påvise og isolere Cronobacter spp., S. ente, og L. monocytogenes fra kroppsoverflaten og fordøyelseskanalen av enkle fluer fanget i naturen. Fordi insekter kan bære lavt antall målet matbåren patogen og høyt antall andre urfolk microbiota 25,26, denne protokollen krever primære (og noen ganger sekundær) berikelse av prøvene i spesifikke kultur media for å øke sensitiviteten for påvisning av målet matbåren patogen . Resultater fra PCR-basert gjenkjenning system ble oppnådd i løpet av ca 30 timer (for DeteDette skjer av Cronobacter spp. og S. ente) og 48 timer (for påvisning av L. monocytogenes) etter først å behandle prøvene. Dermed er denne protokollen pålitelig samt rask og følsom nok til å screene et enkelt fly for tilstedeværelse av matbårne patogene.
Bekreftelse av PCR-positive resultater, og isolering av levedyktige bakterier er en del av standard prosedyre for mange laboratorier. I tillegg, for epidemiologi formål er rene bakteriekulturer fra PCR-positive prøver som kreves for ytterligere å bekrefte og serotype av matbårne patogene ved hjelp av biokjemiske, immunologiske eller genetiske metoder. Selv om ingen falske positiver ble observert når det oppdages S. ente og L. monocytogenes fra kroppsdeler fra enkle villfanget fluer, ved hjelp av denne protokollen, fant vi opp til en 50% rente av falske positive for Cronobacter spp. Dette tyder på at PCR-basert gjenkjenning system for slekten Cronobacteh kan kryssreagere med andre Enterobacteriaceae tilstede blant de svært komplekse microbiota båret av fluer. Således, isolering og rensing av rene kolonier av slekten Cronobacter fra PCR-positive prøvene krever mer selektiv plettering enn evaluert de andre patogener.
Denne protokollen har primært blitt standardisert for å skjerme enkeltvillfanget fluer for tilstedeværelsen av Cronobacter spp., S. ente, og L. monocytogenes ved hjelp av en kommersiell PCR-basert gjenkjenning system. Imidlertid ble denne protokollen også lett tilpasses for å screene kroppsdeler av enkelt fluer for tilstedeværelsen av andre matbårne patogener som enterohemorrhagiske E. coli O157: H7 (enten ved hjelp av E. coli O157: H7 MP standard assay kit eller E. coli O157: H7 i sanntid analysen kit) og Shiga-toxigenic E. coli (STEC) gruppe (ved hjelp av real-time STEC suite), innhenting av følsomhet> 80% (avpublisered data). Dessuten kan denne protokoll potensielt være innrettet til å detektere matbårne patogener fra andre insekter som er kjent vektorer av sykdommer (kakerlakker og maur), men mer forskning på dette området er nødvendig.
Matbåren sykdom utbrudd undersøkelser er svært dynamisk og består av en multi-stegs prosess som kan variere i forhold til den konkrete situasjonen og det lokale miljøet blir undersøkt 12,27. Disse undersøkelser er viktige fordi de gir umiddelbar offentlig helsevern ved å hindre fremtidige sykdommer. I tillegg kan disse undersøkelsene belyse nye mekanismer som matbårne mikroorganismer er spredt, og ta opp viktige spørsmål som fører til nye områder for forskning 28. Etterforskningsmetoder, samt standardiserte, raske, og sensitive protokoller er nødvendige for å påvise matbårne patogener fra enkelte insekter. Denne standardiserte protokollen åpner muligheten til aseptisk samle insekter som fluer, which kan vektor den matbåren bakteriell patogen, som en del av en miljøprøveprogram. Den epidemiologiske opplysninger som kan oppnås av dette ville være til nytte i å konstruere et riktig bilde av mekanismene for overføring av matbårne patogener av insekter (dvs. lengden på eksponeringstiden: et fly ved landing versus fluer landing, avføring, og gulpet opp).
Til slutt, selv om den kommersielle PCR-basert deteksjonssystem som er beskrevet her er praktisk å bruke, og forenkler PCR-amplifisering og visualisering av en genus-nivå amplikon, er det på ingen måte den eneste passende system. Lysatet fra anrikede prøver kan alternativt brukes til å screene med hensyn til nærvær av matbårne patogener ved hjelp av offentlig tilgjengelige artsspesifikk primer-parene. Imidlertid bør deteksjonsfølsomhet påvises før deres bruk.
Materials
| Name | Company | Catalog Number | Comments |
| Bismuth sulfite (BS) agar | Fisher Scientific | R452402 | *Multiple suppliers. |
| Brain heart infusion (BHI) broth | Becton, Dickson and Company | 299070 | *Pre-warmed to 37°C. Multiple suppliers. |
| Brilliance Listeria agar (BLA) | Fisher Scientific | CM1080B | *Multiple suppliers. |
| Buffered peptone water (BPW) | Becton, Dickson and Company | 212367 | *Pre-warmed to 37°C or 42°C. Multiple suppliers. |
| Brilliance Cronobacter agar (Druggan-Forsythe-Iversen formulation/DFI) | Fisher Scientific | CM1055B | *Multiple suppliers. |
| chromID Sakazakii Agar | bioMérieux | 43741 | *Call for information: 800.682.2666 |
| R & F Enterobacter sakazakii (Cronobacter) Chromogenic Plating Medium | R & F Laboratories | Various | *Call for information: +1.630.969.530 |
| R & F Enterobacter sakazakiii Enrichment Broth and supplement | R & F Laboratories | Various | *Call for information: +1.630.969.530 |
| Hektoen enteric (HE) agar | Fisher Scientific | OXCM0419B | *Multiple suppliers. |
| 24 Listeria enrichment broth (24LEB) | Oxoid | CM1107 | *Freshly prepared at room-temperature. Multiple suppliers. |
| Listeria selective enrichment supplement | Oxoid | SR0243 | *Multiple suppliers. |
| Novobiocin | Fisher Scientific | OXSR0181E | *Multiple suppliers. Store at 2-8 °C |
| Vancomycin hydrochloride hydrate | Sigma Aldrich | 861987 | Store at 2-8 °C |
| Cefsulodin sodium salt hydrate | Sigma Aldrich | C8145 | Store at 2-8 °C |
| Rappaport-Vassiliadis (RV) medium | Fisher Scientific | CM0669B | *Multiple suppliers. |
| Tetrathionate (TT) Broth | Becton, Dickson and Company | 249120 | *Multiple suppliers. |
| Trypticase soy agar (TSA) | Becton, Dickson and Company | 236930 | *Multiple suppliers. |
| Xylose lysine desoxycholate (XLD) agar | Becton, Dickson and Company | 221284 | *Multiple suppliers. |
| API Biochemical identification system | bioMérieux | Various | *Call for information: +1.800.682.2666 |
| VITEK 2: Product Safety | bioMérieux | Various | *Call for information: +1.800.682.2667 |
| BAX System Q7 | DuPont | ||
| BAX E. sakazakii Standard assay kit | DuPont | D11801836 | |
| BAX L. monocytogenes 24E assay kit | DuPont | D13608125 | |
| BAX Salmonella 2 Standard assay kit | DuPont | D14368501 | |
| Capping tool | DuPont | D11677028 | |
| Decapping tool | DuPont | D11134095 | |
| PCR tube rack/holder | DuPont | D12701663 | |
| Featherweight forceps, wide tip | BioQuip | 4750 | Sterilize before use. Multiple suppliers. |
| Fine point, straight tip forceps | BioQuip | 4731 | Sterilize before use. Multiple suppliers. |
| Zirconia/silica beads, 0.5 mm | Bio Spec Products, Inc. | 11079105z | Multiple suppliers. |
| Petri dishes - 60X15mm | Fisher Scientific | 08-772B | Multiple suppliers. |
| Disposable inoculating loops, 10µL | Fisher Scientific | 22-363-606 | Multiple suppliers. |
| L-shaped cell spreaders | Fisher Scientific | 14-665-230 | Multiple suppliers. |
| Microcentrifuge tubes, 2 ml | Fisher Scientific | Various | Sterilize before use when needed. Secure lid is preferred. Multiple suppliers. |
| Cluster tubes | Fisher Scientific | 05-500-13 | |
| Cluster tubes caps | Fisher Scientific | 05-500-23 | |
| Sodium hypochlorite (Liquid chlorine bleach) | *Dilute to 0.05% with water. Multiple suppliers. | ||
| Sterile deionized water | Multiple suppliers. | ||
| Sterile distilled water | Multiple suppliers. | ||
| Ethyl alcohol 190 proof | *Dilute to 70% with water when needed. Multiple suppliers. | ||
| Genie cell disruptor, 120V - for 1.5ml and 2.0ml microtubes | Scientific Industries, Inc. | SI-D238 | Multiple suppliers. |
| Heating block | Multiple suppliers. | ||
| Cooling block | Multiple suppliers. | ||
| Recirculating water bath | Multiple suppliers. | ||
| Stereo microscope | Multiple suppliers. | ||
| Centrifuge | Multiple suppliers. | ||
| Incubator | Multiple suppliers. |
References
- Voeller, J. G. Wiley Handbook of Science and Technology for Homeland Security. , Wiley Inc. 1-16 (2008).
- Olsen, A. R., Gecan, J. S., Ziobro, G. C., Bryce, J. R. Regulatory action criteria for filth and other extraneous materials V. Strategy for evaluating hazardous and nonhazardous filth. Regulatory Toxicology and Pharmacology. 33 (3), 363-392 (2001).
- Hald, B., Skovgård, H., Pedersen, K., Bunkenborg, H. Influxed insects as vectors for Campylobacter jejuni and Campylobacter coli in Danish broiler houses. Poultry Science. 87 (7), 1428-1434 (2008).
- Kobayashi, M., et al. Houseflies: Not simple mechanical vectors of enterohemorrhagic Escherichia coli O157:H7. American Journal of Tropical Medicine and Hygiene. 61 (4), 625-629 (1999).
- Pava-Ripoll, M., Pearson, R. E. G., Miller, A. K., Ziobro, G. C. Prevalence and relative risk of Cronobacter spp., Salmonella spp., and Listeria monocytogenes associated with the body surfaces and guts of individual filth flies. Applied and Environmental Microbiology. 78 (22), 7891-7902 (2012).
- Hewitt, C. G. Houseflies and how they spread disease. , Cambridge University Press. (1912).
- Rochon, K., Lysyk, T. J., Selinger, L. B. Retention of Escherichia coli by house fly and stable fly (Diptera:Muscidae) during pupal metamorphosis and eclosion. Journal of Medical Entomology. 42 (3), 397-403 (2005).
- Greenberg, B., Kowalski, J. A., Klowden, M. J. Factors affecting the transmission of Salmonella by flies: natural resistance to colonization and bacterial interference. Infection and Immunity. 2 (6), 800-809 (1970).
- Mramba, F., Broce, A. B., Zurek, L. Vector competence of stable flies, Stomoxys calcitrans L. (Diptera:Muscidae), for Enterobacter sakazakii. Journal of Vector Ecology. 32 (1), 134-139 (2007).
- Ekdahl, K., Normann, B., Andersson, Y. Could flies explain the elusive epidemiology of campylobacteriosis. Bmc Infectious Diseases. 5 (11), 11 (2005).
- Nayduch, D., Noblet, G. P., Stutzenberger, F. J. Vector potential of houseflies for the bacterium Aeromonas caviae. Medical and Veterinary Entomology. 16 (2), 193-198 (2002).
- Multistate and Nationwide Foodborne Outbreak Investigations: A Step-by-Step Guide. , CDC. Atlanta, GA. Available from: http://www.cdc.gov/foodsafety/outbreaks/investigating-outbreaks/investigations/index.html (2013).
- Sievert, K., Messler, S., Klimpel, S., Feffer, K. Comprehensive study on the occurrence and distribution of pathogenic microorganisms carried by synanthropic flies caught at different rural locations in Germany. Journal of Medical Entomology. 46 (5), 1164-1166 (2009).
- Hernández-Escareño, J. J., et al. Presence of Enterobacteriaceae, Listeria spp., Vibrio spp. and Staphylococcus spp. in house fly (Musca domestica L.), collected and macerated from different sites in contact with a few animals species. Revista Cientifica-Facultad De Ciencias Veterinarias. 22 (2), 128-134 (2012).
- Mian, L. S., Maag, H., Tacal, J. V. Isolation of Salmonella from muscoid flies at commercial animal establishments in San Bernardino county, California. Journal of Vector Ecology. 27 (1), 82-85 (2002).
- Olsen, A. R., Hammack, T. S. Isolation of Salmonella spp. from the housefly, Musca domestica L., and the dump fly, Hydrotaea aenescens (Wiedemann) (Diptera:Muscidae), at caged-layer houses. Journal of Food Protection. 63 (7), 958-960 (2000).
- Holt, P. S., Geden, C. J., Moore, R. W., Gast, R. K. Isolation of Salmonella enterica serovar Enteritidis from houseflies (Musca domestica) found in rooms containing Salmonella serovar Enteritidis-challenged hens. Applied and Environmental Microbiology. 73 (19), 6030-6035 (2007).
- Mramba, F., Broce, A., Zurek, L. Isolation of Enterobacter sakazakii from stable flies, Stomoxys calcitrans L. (Diptera:Muscidae). Journal of Food Protection. 69 (3), 671-673 (2006).
- Koyuncu, S., Andersson, M. G., Haggblom, P. Accuracy and Sensitivity of Commercial PCR-Based Methods for Detection of Salmonella enterica in Feed. Applied and Environmental Microbiology. 76 (9), 2815-2822 (2010).
- Gagné, R. Insect and mite pests in food, an illustrated. 1, U.S. Department of Agriculture and U.S. Department of Health and Human Service. 269-296 (1991).
- Greenberg, B. Flies and disease, Vol I. Ecology, classification and biotic associations. , Princeton University Press. (1971).
- Wallace, H. A., Jacobson, A., Hammack, T. S. Chapter 5 Salmonella. Bacteriological Analytical Manual. , Available from: http://www.fda.gov/downloads/Food/FoodScienceResearch/UCM244774.pdf (2007).
- Barro, N., Aly, S., Tidiane, O. C. A., Sababenedjo, T. A. Carriage of bacteria by proboscises, legs, and feces of two species of flies in street food vending sites in Ouagadougou, Burkina Faso. Journal of Food Protection. 69 (8), 2007-2010 (2006).
- Ugbogu, O. C., Nwachukwu, N. C., Ogbuagu, U. N. Isolation of Salmonella and Shigella species from house flies (Musca domestica L.) in Uturu, Nigeria. African Journal of Biotechnology. 5 (11), 1090-1091 (2006).
- Brownlie, J. C., Johnson, K. N. Symbiont-mediated protection in insect hosts. Trends in Microbiology. 17 (8), 348-354 (2009).
- Feldhaar, H. Bacterial symbionts as mediators of ecologically important traits of insect hosts. Ecological Entomology. 36 (5), 533-543 (2011).
- Foodborne disease outbreaks: Guidelines for investigation and control. , World Health Organization. Available from: http://www.who.int/foodsafety/publications/foodborne_disease/outbreak_guidelines.pdf (2008).
- Wright, A. P., Gould, L. H., Mahon, B., Sotir, M. J., Tauxe, R. V. Overview of the Impact of Epidemic-Assistance Investigations of Foodborne and Other Enteric Disease Outbreaks, 1946-2005. American Journal of Epidemiology. 174, S23-S35 (2011).



