Introduction
Insekter spiller en vigtig rolle i transmissionen af fødevarerelaterede sygdomme, fordi de kan sprede patogener til fødevarer eller kontakt med fødevarer overflader og redskaber 1. Blandt insekter, fluer, kakerlakker og myrer udviser adfærd, der fremmer udbredelsen af fødevarebårne patogener. Disse adfærdsmønstre omfatter en forening med rådnende sag, nægter og afføring, endophily (ind bygninger), og synanthropy (samlevende med mennesker) 2. .. Fødevarebårne patogener, såsom Salmonella spp, Listeria monocytogenes, Campylobacter spp, Escherichia coli O157: H7, og medlemmer af slægten Cronobacter (tidligere Enterobacter sakazakii) er blevet rapporteret, at indberette insekter 3-5. Synanthropic skidtfluer mekanisk spredt fødevarebårne bakterier ved at overføre patogener fra deres forurenede kropsoverflader. Tilstedeværelsen af fødevarebårne patogener i fordøjelseskanalen af fluer kan imidlertid være op til tre gangestørre end den observeret på deres krop overflader (krop, hoved, ben og vinger) 5. Fødevarebårne patogener kan også forblive i flue fordøjelseskanalen for en større længde af tid end på kropsoverfladen 6,7 og i nogle tilfælde er de i stand til at formere sig, kolonisere flue fordøjelseskanalen 4,8,9. Dette øger vektor potentiale fluer, fordi de yderligere kan sprede fødevarebårne patogener gennem afføring og opstød 10,11.
I dag er der bedre overvågningssystemer, der kan hurtigere at opdage fødevarebårne sygdomme udbrud. Under udførelsen fødevarebårne udbrud undersøgelser, den offentlige sundhed embedsmænd kigge efter de fødevarer, der kan være den kilde (r) eller køretøj (er) for infektion. Efterforskere kan også udføre en miljøvurdering af anlægget (eller anlæg), der deltager for at finde ud af, hvordan maden var forurenet, og kan indsamle prøver som led i undersøgelsen 12. DespITE den store mængde af videnskabelig litteratur vedrørende insekter som bærere af fødevarebårne patogener, der forbinder insekter som vektorer af patogenet forårsager en bestemt fødevarebårne sygdomme udbrud har været udfordrende. Det er primært fordi insekter ikke bliver aseptisk indsamlet som led i miljø- prøveudtagning programmer i fødevarebårne udbrud undersøgelser. Hvis du vil medtage insekter, især dem, der udviser adfærd, der fremmer udbredelsen af fødevarebårne patogener, som led i en procedure miljøprøver, en standardiseret, hurtig, følsom og pålidelig protokol til at påvise fødevarebårne patogener fra en enkelt insekt skal være på plads.
Traditionelle plating teknikker til påvisning af fødevarebårne patogener fra insekter er besværlige og afhænger af konkurrencedygtig vækst af de målrettede bakterier i forskellige dyrkningsmedier til at overvinde den hurtige vækst i den medfødte kommensale mikrobiota af insektet. De fleste af de undersøgelser, der er forbundet insekter med bacterial patogener har øget følsomhed af fremgangsmåden ved at samle flere insekter stedet for at identificere tilstedeværelsen af patogener på en per individuel basis. Således har disse undersøgelser ikke skelner kroppen del af insekt, hvor patogener blev fundet 13-18. Evnen til at identificere, om fødevarebårne patogener er placeret på kroppens overflade eller i fordøjelseskanalen af en individuel insekt er vigtig, da dette kan have epidemiologiske konsekvenser, og kan føre til forskellige afbødningsstrategier. Som mekaniske vektorer, fluer, der lander på mad for en kort tid, kan kun overføre lave niveauer af bakterier fra deres krop overflade, disse fluer, gylpe og defecate på fødevarer øge sandsynligheden for overførsel patogener på potentielt højere niveauer af infektion. Derfor er det vigtigt at vurdere forekomsten af fødevarebårne patogener per individuel insekt- og at differentiere kropsdel at insekt hvor det bakterielle p athogen er placeret.
Selvom brugen af kultur-uafhængige metoder til påvisning af fødevarebårne patogener i stigende grad ved at blive gennemført, er de ikke blevet kommercielt anvendt til at påvise fødevarebårne patogener fra en enkelt insekt. I øjeblikket er der valideret molekylære protokoller, der er kommercielt til rådighed for hurtig påvisning af fødevarebårne patogener fra fødevarer, der bliver brugt af industrien og reguleringsorganer. Disse metoder omfatter DNA-baserede systemer til detektion af patogener i en række fødevarer prøver. Selv molekylære protokoller er hurtigere end traditionelle plating metoder, er der stadig behov berigelse af prøven for at opnå følsomheden af 10 2 kolonidannende enheder (CFU) af den bakterielle patogen nødvendig polymerasekædereaktion (PCR) -baserede metoder 19. Derudover er isolation af rene bakteriekolonier fra PCR-positive prøver, der kræves for at bekræfte patogenet ved anvendelse af passende metoder.
indhold "> Formålet med denne protokol er at standardisere en kommercielt tilgængelig PCR-baseret system, der anvendes til at detektere patogener fra fødevarer og miljøprøver til påvisning af fødevarebårne bakterier fra kroppens overflade og fordøjelseskanalen af en enkelt flue og yderligere isolere de patogener fra samples.The følsomhed af protokollen beskrevet her blev først kalibreret med lab-opdrættet voksen hus fluer (Musca domestica), der blev eksperimentelt fodret med serielle fortyndinger af hver bakterielt patogen. Den standardiserede protokol blev efterfølgende anvendt til at undersøge 100 indfanget flyver for tilstedeværelsen af fødevarebårne patogener fra deres krop overflader og / eller fordøjelseskanal. vil denne standardiseret protokol tillade folkesundheden laboratorier til påvisning af sundhedsmæssige trusler fra insekter, der giver mulighed for muligheden for at indsamle dem som en del af miljøprogram prøveudtagning, når du udfører fødevarebårne undersøgelser af udbrud.Protocol
1. Indsamling af fluer
- Saml individuelle fluer ved hjælp af sterile entomologiske sweep redskaber. Læg garn i en køler og overføre dem til laboratoriet.
2. Dissektion af fluer
- Immobilisere aseptisk indsamlede fluer ved at placere dem ved -20 ° C i 5-7 min.
- Brug steril pincet sted et fly i et sterilt 2 ml rør indeholdende 1 ml foropvarmet (37 ° C) bufret peptonvand (BPW). Bland røret forsigtigt ved inversion for 2 min. Det er vigtigt, at hele kroppen af fluen være i kontakt med mediet, således at mikrobiota stede på kroppens overflade (S) af fluen vil blive overført til BPW (BPW-S). Mærk røret med et nummer og kropsdel af fluen (dvs. 1S).
BEMÆRK: Se venligst Tabel over specifikke reagenser / Udstyr til en detaljeret beskrivelse af materialer og reagenser, der er nævnt i denne protokol. - Brug steril pincet fjerne flyve fra BPW-S-medier og overførseldet til en tom og ren 2 ml rør til overfladen desinficere fluen. Inkubér rør indeholdende BPW-S-medium ved 37 ° C, mens de udfører desinfektion og dissektion protokol.
- Overflade-desinficere fluen ved at nedsænke det i 1 ml 70% ethanol i 1 minut efterfulgt af en skylning med sterilt destilleret vand, før nedsænkning i 1 ml frisk fremstillet 0,05% (v / v) blegemiddelopløsning. Skyl 3 gange med sterilt destilleret vand. Overfør vand fra den sidste skylning i en autoklaveret 2 ml rør.
BEMÆRK: Smid væsken hver gang ved hjælp af en 1.000 pi mikropipette eller ved at vende røret, og sørg for fluen forbliver inde i røret. Bland forsigtigt ved inversion på hvert trin i overfladen-desinfektion proces. - For at vurdere effektiviteten af desinfektionsprocessen, overføre 100 pi af vandet fra den sidste skylning til en trypticase soja-agar (TSA) plade og spredes ved hjælp af en steril L-formet engangs sprederen. Inkubér pladen ved 37 ° C i 24 timer. Efter icubation, registrere tilstedeværelsen af eventuelle bakteriekolonier.
BEMÆRK: Tilstedeværelsen af bakteriekolonier på TSA-plader viser en ineffektiv overflade-desinfektion proces. Hvis dette sker, bør tilstedeværelsen af fødevarebårne patogener kun indberettes på kroppen overflade af fluen, fordi krydsforurening mellem kroppens overflade og fordøjelseskanalen ikke kan udelukkes.
- Overflade-desinficere fluen ved at nedsænke det i 1 ml 70% ethanol i 1 minut efterfulgt af en skylning med sterilt destilleret vand, før nedsænkning i 1 ml frisk fremstillet 0,05% (v / v) blegemiddelopløsning. Skyl 3 gange med sterilt destilleret vand. Overfør vand fra den sidste skylning i en autoklaveret 2 ml rør.
- Efter overfladebehandlet desinfektion fluen, overføre det til et stykke autoklaveret køkkenrulle for at fjerne overskydende vand og derefter til en steril 60 mm engangs petriskål.
- Placer petriskålen under et dissekere omfang og identificere fluen til artsniveau hjælp dikotome nøgler til tovingede familier 20,21.
- Brug autoklaveres fin spids pincet forsigtigt trække anus og hele fordøjelseskanalen (A) ud af fluen og aseptisk overdrager den til andre sterilt 2 ml rør indeholdende 1 ml forvarmet (37 ° C) BPW med 0,5 mm zirconia / silica perler (BPW-A). Label the rør med samme nummer valgt for den enkelte flue og kropsdelen af fluen (dvs. 1A).
- Bland rør indeholdende BPW-A grundigt i 5 - 10 min ved hjælp af en celle disruptor. Der inkuberes ved 36 ± 1 ° C, mens de udfører resten af protokollen.
- At voucher og / eller opbevare prøven for langsigtet, placere resten af fluen i et rent 2 ml rør og tilsæt 1 - 2 ml 95% ethanol.
3. Primær og Sekundær Berigelse
- Mærk alle rør primære og sekundære berigelse holdige medier ifølge prøvenummer og kropsdelen af fluen.
- Under en steril hætte, overføre 300 pi BPW-S (overflade) til sterilt 2 ml rør indeholdende følgende medier:
- For Salmonella, bruge 1 ml forvarmet (42 ° C) BPW. Inkuber i et recirkulerende vandbad ved 42,5 ° C i 22-24 timer. For sekundær berigelse, overføre 100 pi beriget BPW til 400 & #181 l forvarmet (37 ° C) Brain Heart Infusion (BHI) bouillon tidligere anbringes i sterile klynge rør. Der inkuberes ved 37 ° C i 3 timer.
- For Cronobacter Brug 1 ml forvarmet (37 ° C) BPW med novobiocin (10 mg / l; Wallace, M., personlig kommunikation). Alternativt kan du bruge 1 ml af R & F Enterobacter sakazakii berigelsesbouillon med tillæg (vancomycin og cefsulodin) som primær berigelse. Der inkuberes ved 37 ° C i 22-26 timer. For sekundær berigelse, overføre 100 pi beriget BPW med novobiocin til 400 pi forvarmet (37 ° C) BHI bouillon tidligere placeret i sterile klynge rør. Der inkuberes ved 37 ° C i 3 timer.
- For L. monocytogenes, brug 1 ml frisk fremstillet RT 24 Listeria berigelsesbouillon (24 LEB) med selektiv supplement. Der inkuberes ved 37 ° C i 44 ± 5 timer. Ingen sekundær berigelse kræves til påvisning af L. monocytogenes. Gentag trin 3.2.1 - 3.2.3 ovenfor under anvendelse af røret mærket som BPW-A.
4. Fremstilling af PCR-baserede system for amplifikation og detektion af Target Fødevarebåren Pathogen
Trin 4-8 bruger en kommerciel PCR cycler / detektor-system, en computer arbejdsstation, og klar-til-brug kits til screening for Salmonella (Salmonella 2 standard assay kit), Cronobacter arter (E. sakazakii standard assay kit), og Listeria monocytogenes (L. monocytogenes 24E assay kit). Standard analyser anvender PCR endepunkt detektion. Hvert sæt indeholder PCR-ready tabletter med en interkalerende farvestof, der udsender et fluorescenssignal ved binding til dobbeltstrenget DNA. Signalet fanget under detektion fase af PCR-system program, der genererer en smeltekurve, der fortolkes af softwaren som positiv eller negativ.
- Forbered reagenser og udstyr som specificeret af produRER protokol pr hvert mål fødevarebårne patogener.
BEMÆRK: Protokollerne til påvisning af Salmonella og Cronobacter kræver en et-trins lyseprocedure henhold til protokollen til påvisning L. monocytogenes kræver en totrins lysis (se afsnit 5 og 6, henholdsvis). - Tænd for automatiseret varmeblok vælge et særprogram for målet patogen. Alternativt hvis varme blokke er manuel, sæt temperaturen til 37 ° C (for Salmonella, Cronobacter spp, og L. monocytogenes.) Eller til 55 ± 2 ° C (for L. monocytogenes del 2 af lysis - se trin 6.2) og 95 ± 3 ° C.
- Sørg for, at køle- blokke er blevet nedkølet O / N, ellers chill dem ved 2 - 8 ° C i mindst 2 timer.
- Ved hjælp af computersoftware af PCR-baserede påvisning systemet oprette en rack-fil efter producentens anvisninger.
- Label og arrangere klyngerør indeholdende lysis reagens i reolen ifølge rack fil.
- Initialiser PCR-baseret detektionssystem instrument.
5. Udfør Lysis til påvisning af Salmonella og Cronobacter
- Forbered lysis reagens ved tilsætning af 150 pi af protease til en 12 ml flaske lysisbuffer.
- Overfør 200 pi lysis reagens til hver af de tidligere mærkede klynge rør.
BEMÆRK: Cluster rør indeholdende lysis reagens kan opbevares ved 2 - 8 ° C i op til 2 uger. - Brug lange pipettespidser, overførsel 20 pi sekundære berigede prøver (se trin 3.2.1 og 3.2.2) til tilsvarende klynge rør indeholdende 200 pi lysis reagens. Brug nye pipettespidser for hver prøve.
BEMÆRK: Hold rør fra primær og sekundær berigelse i køleskab (Salmonella) eller ved stuetemperatur (Cronobacter) for yderligere bekræftelse analyse af PCR-positive / negative prøver. Forbered negative kontroller ved tilsætning af 20 pi sterilt BHI medie til klynge rør indeholdende 200 pi lysis reagens. - Forberedes positive kontroller ved at tilsætte 20 pi O / N-bakteriekulturer (dyrket i BHI) af alle kendte Salmonella eller Cronobacter stamme til klynge rør indeholdende 200 pi lysis reagens.
- Cap klynge rør og sikre tæt hjælp capping værktøj.
- Placer rack af klynge rør i den automatiserede varmeblokken efter valg særprogram for målet patogen. Alternativt inkuberes klynge rør ved 37 ± 2 ° C i 20 minutter, efterfulgt af inkubering ved 95 ± 3 ° C i 10 min. Endelig overføre klynge rør til afkøling blokke (2 - 8 ° C) i 5 minutter.
BEMÆRK: Cluster rør indeholdende lysatet kan opbevares ved -20 ° C i op til 2 uger.
6. Udfør Lysis til påvisning af L. monocytogenes
- Udfør første del aflysis som følger:
- Tilføj 1,8 ml sterilt deioniseret vand til flasken fuldt optøet lyserende middel 1.
BEMÆRK: Store lyserende middel 1 ved 2 - 8 ° C indtil den er klar til brug. Efter åbning og fortynding, opbevar ved stuetemperatur (20 - 30 ° C) i op til 6 måneder. - Kombiner lyserende midler 1 og 2 i en 4: 1 ratio (40 pi fortyndet lyserende middel 1 og 10 pi lyserende middel 2 per hver prøve). Transfer 50 pi af de kombinerede lyserende midler til at klynge rør. Brug blandingen inden 4 timer.
- Der tilsættes 500 pi primær beriget prøve (se trin 3.2.3) til klyngen rør indeholdende 50 pi af de kombinerede lyserende midler.
- Forbered en negativ kontrol ved tilsætning af 500 pi sterilt 24 LEB til 50 pi af de kombinerede lyserende midler.
- Forbered en positiv kontrol ved at tilsætte 500 pi af O / N L. monocytogenes dyrket i 24 LEB til 50 pi af de kombinerede lyserende midler.
- Sæt låg på klynge rør, bland forsigtigt og stedi varmeblok ved 37 ± 1 ° C i 30 minutter.
BEMÆRK: Hold rør fra primær berigelse i køleskabet i yderligere bekræftelse analyse af PCR-positive / negative prøver.
- Tilføj 1,8 ml sterilt deioniseret vand til flasken fuldt optøet lyserende middel 1.
- Udfør del 2 i lyse som følger:
- Forbered lysereagens som anvist i trin 5.1 og 5.2.
- Brug lange pipettespidser overføre 20 pi del lysat til klynge rør indeholdende 200 pi lysis reagens. Brug nye pipettespidser for hver prøve.
- Cap klynge rør og sikre tæt hjælp capping værktøj.
- Placer klynge rør i automatiseret varmeblok Valg af særprogrammet for L. monocytogenes. Alternativt inkuberes klynge rør ved 55 ± 2 ° C i 30 minutter, efterfulgt af inkubering ved 95 ± 3 ° C i 10 min. Endelig overføre klynge rør til afkøling blokke (2 - 8 ° C) i 5 minutter.
BEMÆRK: Cluster rør indeholdende lysatet kan opbevares ved -20 ° C i op til 2 uger.
7. Hydrate PCR-Ready Tablets
- Vælg en afkølet (4 ° C) PCR køleblok og placere et PCR-rør rack over indsatsen.
- Placer tilsvarende PCR-rør indeholdende PCR-ready tabletter (inkluderet med hvert kit) for målet fødevarebårne patogener i holderen ifølge rack fil.
- Ved hjælp af decapping værktøj, forsigtigt fjerne hætterne fra PCR-rør. Kassér hætterne og kontrollere, at hvert rør indeholder en tablet.
- Overfør 50 pi (for Salmonella og Cronobacter) eller 30 pi (for L. monocytogenes) af lysat til specifikke PCR-rør. Brug nye optiske hætter og sikre tæt på PCR-rør ved hjælp af capping værktøj.
OBS: Efter tilsætning lysatet til PCR-klar tabletter, prøverne skal forblive nedkøles til 2 - 8 ºC, indtil indlæst i PCR- baserede detektionssystem. De PCR-rør kan centrifugeres ved 2500 xg i et par sekunder for at sikre, at den fulde mængde i bunden than rør. - Læg de PCR-rør til PCR variator / detection system instrument ved at åbne instrumentet skuffen.
- Placer rack af PCR-rør i brøndene i skuffen, og kontroller, at rørene sidder korrekt.
- Luk skuffen og initiere programmet som beskrevet af producenten protokol.
BEMÆRK: PCR-instrument er forudindstillet cykling parametre for hver fødevarebåren patogen. - Kontroller, at PCR cykling statuslinjen viser en blå bjælke angiver, at forstærkningen del af programmet kører.
BEMÆRK: standard PCR-assays, behandlingstiden for det fulde program (amplifikation og detektion) tager cirka 3-3,5 timer at fuldføre.
8. Anmeldelse Resultater
- Efter behandlingen er færdig, skal du følge skærmen fra PCR-baseret system instrument til at udtage prøver, og resultaterne af gennemgangen.
- Hvis målet fødevarebårne patogen er til stede i prøven (entenoverflade eller fordøjelseskanalen af fluen) brønden er rød med en "plus" tegn (positiv). Hvis patogenet er fraværende, og er grønt med en minus "tegn (negativ).
- Hvis brønden er gul med en rød bar over midten, indikerer et signal fejl.
9. Isolering af bakterielle patogener fra PCR-positive resultater
- Vælg rør fra den primære (for L. monocytogenes) eller sekundær (for salmonella og Cronobacter) berigelse af prøverne, der var PCR-positive. Også, skal du vælge tilfældigt 3-5% af prøverne, der var PCR-negative og gør følgende:
- For Salmonella:
- Tilsæt 100 pi af den sekundære berigelse medier til 10 ml Rappaport-Vassiliadis (RV) medium og 1 ml tetrathionat (TT) bouillon. Inkuber rørene ved 42,5 ° C i et recirkulerende vandbad i 22-24 timer.
- Efter inkubation streak en 3 mm løkkefuld (10 pi) af hver, RV end TT medier på bismuth sulfit (BS) agar, xylose lysin desoxycholat (XLD-agar), og Hektoen enterisk (HE) agar. Pladerne inkuberes ved 35 ± 1 ° C for 22-24 timer.
- Efter inkubation undersøges pladerne for tilstedeværelse af typiske Salmonella kolonier på hvert medie. Hvis der ikke kan opnås nogen isolerede kolonier efter flere sub-dyrkning skridt, overveje prøven som negativ og rapportere som en falsk positiv for PCR-baseret system.
BEMÆRK: For typiske Salmonella kolonier på specifikke medier se 22. Vælg fem formodede typiske Salmonella kolonier og videredyrke dem på BS, XLD, eller han indtil rene kulturer af isolerede / enkelt kolonier opnået. - Vælg en ren koloni og identificere formodede Salmonella ved hjælp biokemiske kommercielle tests såsom VITEK 2 id-kort eller API biokemisk identifikationssystem, efter fabrikantens anvisninger.
- For Cronobacter: <ol>
- Streak en 3 mm løkkefuld (10 pi) af det sekundære berigelse medier på to plader af kromogene dyrkningsmedier som F & F Enterobacter sakazakii (Cronobacter) kromogent plating medium og / eller ChromID sakazakii Agar. Pladerne inkuberes ved 35 ° C i 22-24 timer.
- Efter inkubation undersøger plader for tilstedeværelsen af typiske Cronobacter kolonier (blå-sort til blå-grå). Vælg 5 formodede Cronobacter kolonier og videredyrke dem på F & F Enterobacter sakazakii (Cronobacter) kromogent plating medium, ChromID sakazakii Agar eller TSA indtil rene kulturer af isolerede / enkelt kolonier opnået.
BEMÆRK: Hvis der ikke kan opnås nogen isolerede kolonier efter flere sub-dyrkning skridt, overveje prøven som negativ og rapportere som en falsk positiv for PCR-baserede påvisning system. - Vælg en ren koloni og identificere formodede Cronobacter ved hjælp biokemisk indusielle tests såsom VITEK 2 id-kort eller API 20E biokemisk identifikationssystem, efter fabrikantens anvisninger.
- Træk en 3 mm løkkefuld (10 pi) af den primære berigelse medier på to plader af Brilliance Listeria agar (BLA). Pladerne inkuberes ved 36 ± 1 ° C for 22-26 timer.
- Efter inkubation undersøges pladerne for tilstedeværelse af formodede L. monocytogenes (blå-grøn) kolonier. Vælg 5 formodede L. monocytogenes kolonier og videredyrke dem på BLA indtil opnåelse rene kulturer af isolerede / enkelte kolonier. Re-inkuber negative plader ved 36 ± 1 ° C i yderligere 22 til 26 timer.
- Vælg en ren koloni og identificere formodede L. monocytogenes ved hjælp kommercielle biokemiske tests såsom VITEK 2 id-kort eller API Listeria biokemisk identifikationssystem, efter fabrikantens instructions.
Representative Results
Denne protokol blev først kalibreret på et sæt af lab-opdrættet hus fluer, der blev eksperimentelt fodret i 24 timer med flydende flue fødevarer (2% mælkepulver) indeholdende seriefortyndinger (Februar 10-August 10 CFU / ml) af C. sakazakii, S. enterica, L. monocytogenes eller C. jejuni (n = 21 for hvert bakterielt patogen). Berigelse medier samt inkubationstider og temperaturer blev justeret for hver fødevarebåren patogen indtil PCR-baserede system var i stand til at opdage de laveste niveauer af bakterier (10 2 CFU / ml) fra kroppens overflade og fordøjelseskanalen af en enkelt eksperimentelt fodret flyve. Brug af medier berigelse og betingelserne i protokollen afsnittet PCR-baserede system opdaget C. sakazakii, S. enterica og L. monocytogenes fra kropsoverfladen 100% af fluer fodret med bakterielle podestoffer> 10 3 CFU / ml (figur 1A). Når fluer blev fodret med 102 CFU / ml, procentdelen af påvisning af C. sakazakii, S. enterica og L. monocytogenes fra kroppen overflade var 100%, 66% og 33%, (figur 1A). PCR-baserede system også opdaget disse tre fødevarebårne patogener fra fordøjelseskanalen af fluer fodret med alle bakterielle koncentrationer på procenter ≥33% (figur 1B). Imidlertid påvisning af C. jejuni blev kun opnået, når lab-opdrættet fluer blev eksperimentelt fodret med flydende fødevarer, der indeholder den højeste bakterielle inokulum (10 8 CFU / ml). Derfor C. jejuni blev udelukket fra gruppen af fødevarebårne patogener, kunne testes fra individuel synanthropic skidtfluer hjælp af denne PCR-baseret påvisningssystem.
Med denne standardiseret protokol, var vi i stand til at bestemme forekomsten af Cronobacter spp., S. enterica, og L. monocytogenes fra kroppens overflade og / eller the fordøjelseskanalen af 100 vilde fluer, der var individuelt og aseptisk fanget fra den dumpster område på ti restauranter byområder beliggende i hovedstadsområdet Washington, DC 5 Samlede filth fluer var repræsentative for mindst seks arter, herunder M. domestica (47%), Lucilia cuprina (33%), L. sericata (14%), Cochliomyia macellaria (2%), Sarcophaga haemorrhoidalis (2%), og Ophyra leucostoma (1%). En flue blev kun identificeret til familie niveau (Anthomyiidae; 1%). Overfladen desinfektion protokol var effektiv til at undgå krydskontaminering mellem kropsdele af fluen, fordi ingen bakterievækst blev observeret på TSA plader til vand fra den sidste desinfektion skylning enkelte flue. Således kunne der skelnes mellem fødevarebårne bakterier på kroppen dele af hver flue.
Ingen falske positiver blev påvist fra prøver af kroppens overflade og fordøjelseskanalindividuelle fluer når dette kommercielle PCR-baseret system til påvisning af S. enterica og L. monocytogenes, og bekræftelsen af levedygtige patogener på agarplader var i overensstemmelse med PCR-positive resultater. Men det var ikke muligt at isolere rene kulturer af Cronobacter spp. fra alle PCR-positive prøver. Derfor er detektion af dette patogen ved PCR-baserede Systemet viste falske positiver fra kroppens overflade (50%, 18/09) og fordøjelseskanalen (48%; 16/33) af enkelt vild fanget fluer. Tilfældigt udvalgte PCR-negative prøver, der blev belagt på specifikke medier, bekræftede fraværet af fødevarebårne patogener. Derfor blev der ikke falske negativer påvist i nogen af prøverne ved brug af denne kommercielle PCR-baseret system til påvisning Cronobacter spp., S. enterica eller L. monocytogenes.
Kun de PCR-positive prøver, hvor patogenet blev isoleret og bekræftet blev betragtet som positiveog medtaget til statistisk analyse. Den samlede forekomst af fødevarebårne patogener i fordøjelseskanalen af vilde-fanget filth fluer var betydeligt højere end på kroppen overflade (χ 2 = 6,8772, df = 1, p = 0,0087). 22% af fordøjelseskanal og 8% af kroppens overflader af indsamlede vilde fluer var positive for mindst én af de tre fødevarebårne patogener (figur 2). Samlet forekomsten af Cronobacter spp. på enten kroppen overflader eller fordøjelseskanal indsamlede fluer var statistisk højere (19%; Fishers eksakte test p = 0,0165) end forekomsten af S. enterica (7%) og L. monocytogenes (4%). Der blev imidlertid ikke statistiske forskelle observeret ved udførelse parvise sammenligninger mellem kropsdele af fluerne for hver bakterielt patogen (figur 3; Fishers eksakte test p = 0,1464, p = 0,1184, og p = 0,6212 for Cronobacterspp., S. enterica, og Listeria monocytogenes, henholdsvis). Ingen af de fluer var positive for alle tre patogener evalueret. Men tre af de fluer (to L. cuprina og en L. sericata) gennemføres Salmonella spp. og L. monocytogenes på overfladen eller i fordøjelseskanalen.
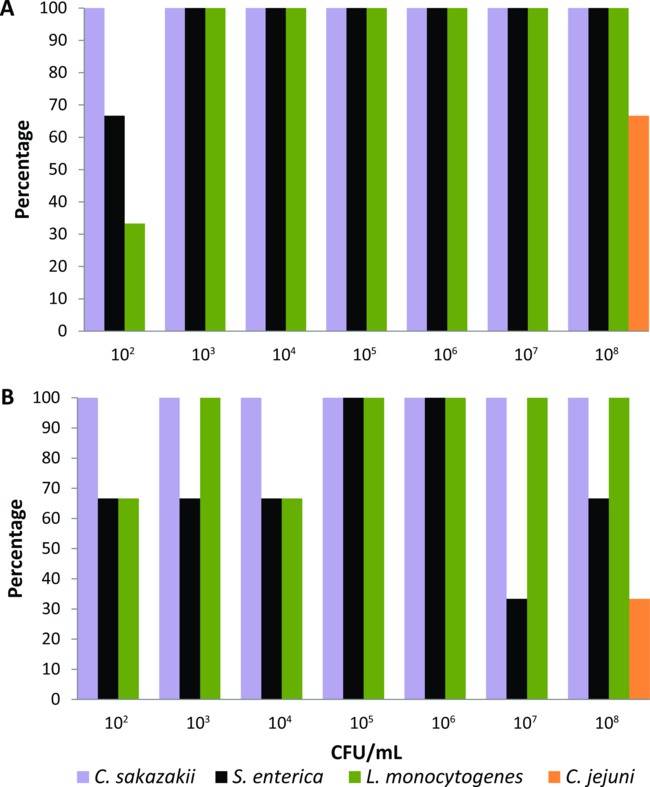
Figur 1. Detection niveauer af Cronobacter sakazakii, Salmonella enterica, Listeria monocytogenes, og Campylobacter jejuni fra (A) kroppens overflade og (B) fordøjelseskanalen af individuelle lab-opdrættet hus fluer fodret med flydende fødevarer, der indeholder forskellige bakterielle podestoffer (n = 21 for hver bakterielt patogen, n = 3 for hver bakteriel koncentration).
Figur 2. Andel af kroppens overflader og fordøjelseskanal individuelle fluer fundet positive for nogen af de mål fødevarebårne patogener.

Figur 3. Udbredelsen af Cronobacter spp., Salmonella enterica og Listeria monocytogenes fra kroppens overflade og fordøjelseskanalen af synanthropic vilde-fanget fluer. P-værdier er rapporteret er fra parvise sammenligninger mellem kropsoverfladen og fordøjelseskanalen for hver bakterielt patogen (Fishers eksakte test, p-værdi <0,05 indikerer statistisk signifikans). Copyright © American Society for Microbiology, Journal of Applied and Environmental Microbiology 78 (22): 7891-902, 2012. doi: 10,1128 / AEM.02195-12.
Discussion
Tidligere undersøgelser, der har påvist fødevarebårne patogener fra vilde insekter har brugt mange forskellige protokoller, der måske ikke indeholde de nødvendige oplysninger til præcist at vurdere den fødevarerelaterede risiko for forekomst af en enkelt flue i fødevarer eller fødevarerelaterede miljøer 13,15, 23,24. Her har vi vist, at ved hjælp af denne standardiserede protokol, er det muligt at detektere og isolere Cronobacter spp., S. enterica og L. monocytogenes fra kroppens overflade og fordøjelseskanalen af enkelte fluer fanget i naturen. Fordi insekter kan bære lave antal af målet fødevarebårne patogen og høje antal andre indfødte mikrobiota 25,26, protokollen kræver primære (og til tider sekundær) berigelse af prøverne i bestemte dyrkningsmedier for at øge følsomheden af påvisning af målet fødevarebåren patogen . Resultaterne fra PCR-baseret påvisningssystem blev opnået inden for cirka 30 timer (for detektion af Cronobacter spp. og S. enterica) og 48 timer (til påvisning af Listeria monocytogenes) efter oprindeligt behandling af prøverne. Således er denne protokol er pålidelig samt hurtig og følsom nok til at screene et enkelt flue for tilstedeværelsen af fødevarebårne patogener.
Bekræftelse af PCR-positive resultater og isolering af levedygtige bakterier er en del af standardprocedure, af mange laboratorier. Desuden for Epidemiologi formål, rene bakteriekulturer fra PCR-positive prøver skal yderligere bekræfte og serotype af fødevarebåren patogen hjælp biokemiske, immunologiske eller genetiske metoder. Selv om der ikke falske positiver blev observeret, når påvisning S. enterica og L. monocytogenes fra kropsdele af enkelt vild fanget fluer, ved hjælp af denne protokol, fandt vi op til 50% på falske positiver for Cronobacter spp. Dette antyder, at PCR-baseret påvisningssystem for slægten CronobactER kan krydsreagere med andre Enterobacteriaceae stede blandt de meget komplekse mikroflora bæres af fluer. Således, isolering og oprensning af rene kolonier af slægten Cronobacter fra PCR-positive prøver kræver mere selektiv udpladning end de andre patogener evalueret.
Denne protokol er primært blevet standardiseret til at screene individuelle vilde-fanget fluer for tilstedeværelsen af Cronobacter spp., S. enterica og L. monocytogenes anvender et kommercielt PCR-baseret påvisningssystem. Men denne protokol også nemt tilpasses til at screene kropsdele af enkelte fluer for tilstedeværelsen af andre fødevarebårne patogener, såsom enterohæmoragisk E. coli O157: H7 (under anvendelse af enten E. coli O157: H7 MP standard assay kit eller E. coli O157: H7 realtid assay kit) og shiga-toksigene E. coli (STEC) gruppe (ved hjælp af real-time STEC suite), opnåelse følsomheder> 80% (Afpublicered data). Desuden kan denne protokol potentielt indrettet til at detektere fødevarebårne patogener fra andre insekter, der er kendt vektorer af sygdomme (kakerlakker og myrer), men der er behov for mere forskning på dette område.
Fødevarebårne sygdomme undersøgelser af udbrud er meget dynamisk og omfatter en multi-trins proces, der kan variere alt efter den konkrete situation og det lokale miljø, som undersøges 12,27. Disse undersøgelser er vigtige, fordi de giver øjeblikkelig beskyttelse ved at forhindre fremtidige sygdomme folkesundheden. Derudover kan disse undersøgelser belyse nye mekanismer, hvormed fødevarebårne mikroorganismer spredes, og rejser vigtige spørgsmål, der fører til nye områder for forskning 28. Efterforskningsteknikker samt standardiserede, hurtige og følsomme protokoller er nødvendige til påvisning af fødevarebårne patogener fra de enkelte insekter. Denne standardiserede protokol åbner mulighed for aseptisk samle insekter som fluer, which kan vektor den fødevarebårne bakterielle patogen, som en del af et miljøprogram prøveudtagning. De epidemiologiske oplysninger, der kan opnås fra dette ville være nyttige i at konstruere et præcist billede af de mekanismer for overførsel af fødevarebårne patogener af insekter (dvs. længden af eksponeringstid: en flue ved at lande versus fluer landing, afføring, og regurgitating).
Endelig, selv om den kommercielle PCR-baseret påvisning her beskrevne system er praktisk at anvende og forenkler PCR-amplifikation og visualisering af en slægt-niveau amplicon, er det på ingen måde den eneste passende system. Af lysatet fra berigede prøver kunne alternativt anvendes til at screene for tilstedeværelsen af fødevarebårne patogener ved hjælp af offentligt tilgængelige artsspecifikke primerpar. Imidlertid bør detektionsfølsomheden påvises forud for deres anvendelse.
Materials
| Name | Company | Catalog Number | Comments |
| Bismuth sulfite (BS) agar | Fisher Scientific | R452402 | *Multiple suppliers. |
| Brain heart infusion (BHI) broth | Becton, Dickson and Company | 299070 | *Pre-warmed to 37°C. Multiple suppliers. |
| Brilliance Listeria agar (BLA) | Fisher Scientific | CM1080B | *Multiple suppliers. |
| Buffered peptone water (BPW) | Becton, Dickson and Company | 212367 | *Pre-warmed to 37°C or 42°C. Multiple suppliers. |
| Brilliance Cronobacter agar (Druggan-Forsythe-Iversen formulation/DFI) | Fisher Scientific | CM1055B | *Multiple suppliers. |
| chromID Sakazakii Agar | bioMérieux | 43741 | *Call for information: 800.682.2666 |
| R & F Enterobacter sakazakii (Cronobacter) Chromogenic Plating Medium | R & F Laboratories | Various | *Call for information: +1.630.969.530 |
| R & F Enterobacter sakazakiii Enrichment Broth and supplement | R & F Laboratories | Various | *Call for information: +1.630.969.530 |
| Hektoen enteric (HE) agar | Fisher Scientific | OXCM0419B | *Multiple suppliers. |
| 24 Listeria enrichment broth (24LEB) | Oxoid | CM1107 | *Freshly prepared at room-temperature. Multiple suppliers. |
| Listeria selective enrichment supplement | Oxoid | SR0243 | *Multiple suppliers. |
| Novobiocin | Fisher Scientific | OXSR0181E | *Multiple suppliers. Store at 2-8 °C |
| Vancomycin hydrochloride hydrate | Sigma Aldrich | 861987 | Store at 2-8 °C |
| Cefsulodin sodium salt hydrate | Sigma Aldrich | C8145 | Store at 2-8 °C |
| Rappaport-Vassiliadis (RV) medium | Fisher Scientific | CM0669B | *Multiple suppliers. |
| Tetrathionate (TT) Broth | Becton, Dickson and Company | 249120 | *Multiple suppliers. |
| Trypticase soy agar (TSA) | Becton, Dickson and Company | 236930 | *Multiple suppliers. |
| Xylose lysine desoxycholate (XLD) agar | Becton, Dickson and Company | 221284 | *Multiple suppliers. |
| API Biochemical identification system | bioMérieux | Various | *Call for information: +1.800.682.2666 |
| VITEK 2: Product Safety | bioMérieux | Various | *Call for information: +1.800.682.2667 |
| BAX System Q7 | DuPont | ||
| BAX E. sakazakii Standard assay kit | DuPont | D11801836 | |
| BAX L. monocytogenes 24E assay kit | DuPont | D13608125 | |
| BAX Salmonella 2 Standard assay kit | DuPont | D14368501 | |
| Capping tool | DuPont | D11677028 | |
| Decapping tool | DuPont | D11134095 | |
| PCR tube rack/holder | DuPont | D12701663 | |
| Featherweight forceps, wide tip | BioQuip | 4750 | Sterilize before use. Multiple suppliers. |
| Fine point, straight tip forceps | BioQuip | 4731 | Sterilize before use. Multiple suppliers. |
| Zirconia/silica beads, 0.5 mm | Bio Spec Products, Inc. | 11079105z | Multiple suppliers. |
| Petri dishes - 60X15mm | Fisher Scientific | 08-772B | Multiple suppliers. |
| Disposable inoculating loops, 10µL | Fisher Scientific | 22-363-606 | Multiple suppliers. |
| L-shaped cell spreaders | Fisher Scientific | 14-665-230 | Multiple suppliers. |
| Microcentrifuge tubes, 2 ml | Fisher Scientific | Various | Sterilize before use when needed. Secure lid is preferred. Multiple suppliers. |
| Cluster tubes | Fisher Scientific | 05-500-13 | |
| Cluster tubes caps | Fisher Scientific | 05-500-23 | |
| Sodium hypochlorite (Liquid chlorine bleach) | *Dilute to 0.05% with water. Multiple suppliers. | ||
| Sterile deionized water | Multiple suppliers. | ||
| Sterile distilled water | Multiple suppliers. | ||
| Ethyl alcohol 190 proof | *Dilute to 70% with water when needed. Multiple suppliers. | ||
| Genie cell disruptor, 120V - for 1.5ml and 2.0ml microtubes | Scientific Industries, Inc. | SI-D238 | Multiple suppliers. |
| Heating block | Multiple suppliers. | ||
| Cooling block | Multiple suppliers. | ||
| Recirculating water bath | Multiple suppliers. | ||
| Stereo microscope | Multiple suppliers. | ||
| Centrifuge | Multiple suppliers. | ||
| Incubator | Multiple suppliers. |
References
- Voeller, J. G. Wiley Handbook of Science and Technology for Homeland Security. , Wiley Inc. 1-16 (2008).
- Olsen, A. R., Gecan, J. S., Ziobro, G. C., Bryce, J. R. Regulatory action criteria for filth and other extraneous materials V. Strategy for evaluating hazardous and nonhazardous filth. Regulatory Toxicology and Pharmacology. 33 (3), 363-392 (2001).
- Hald, B., Skovgård, H., Pedersen, K., Bunkenborg, H. Influxed insects as vectors for Campylobacter jejuni and Campylobacter coli in Danish broiler houses. Poultry Science. 87 (7), 1428-1434 (2008).
- Kobayashi, M., et al. Houseflies: Not simple mechanical vectors of enterohemorrhagic Escherichia coli O157:H7. American Journal of Tropical Medicine and Hygiene. 61 (4), 625-629 (1999).
- Pava-Ripoll, M., Pearson, R. E. G., Miller, A. K., Ziobro, G. C. Prevalence and relative risk of Cronobacter spp., Salmonella spp., and Listeria monocytogenes associated with the body surfaces and guts of individual filth flies. Applied and Environmental Microbiology. 78 (22), 7891-7902 (2012).
- Hewitt, C. G. Houseflies and how they spread disease. , Cambridge University Press. (1912).
- Rochon, K., Lysyk, T. J., Selinger, L. B. Retention of Escherichia coli by house fly and stable fly (Diptera:Muscidae) during pupal metamorphosis and eclosion. Journal of Medical Entomology. 42 (3), 397-403 (2005).
- Greenberg, B., Kowalski, J. A., Klowden, M. J. Factors affecting the transmission of Salmonella by flies: natural resistance to colonization and bacterial interference. Infection and Immunity. 2 (6), 800-809 (1970).
- Mramba, F., Broce, A. B., Zurek, L. Vector competence of stable flies, Stomoxys calcitrans L. (Diptera:Muscidae), for Enterobacter sakazakii. Journal of Vector Ecology. 32 (1), 134-139 (2007).
- Ekdahl, K., Normann, B., Andersson, Y. Could flies explain the elusive epidemiology of campylobacteriosis. Bmc Infectious Diseases. 5 (11), 11 (2005).
- Nayduch, D., Noblet, G. P., Stutzenberger, F. J. Vector potential of houseflies for the bacterium Aeromonas caviae. Medical and Veterinary Entomology. 16 (2), 193-198 (2002).
- Multistate and Nationwide Foodborne Outbreak Investigations: A Step-by-Step Guide. , CDC. Atlanta, GA. Available from: http://www.cdc.gov/foodsafety/outbreaks/investigating-outbreaks/investigations/index.html (2013).
- Sievert, K., Messler, S., Klimpel, S., Feffer, K. Comprehensive study on the occurrence and distribution of pathogenic microorganisms carried by synanthropic flies caught at different rural locations in Germany. Journal of Medical Entomology. 46 (5), 1164-1166 (2009).
- Hernández-Escareño, J. J., et al. Presence of Enterobacteriaceae, Listeria spp., Vibrio spp. and Staphylococcus spp. in house fly (Musca domestica L.), collected and macerated from different sites in contact with a few animals species. Revista Cientifica-Facultad De Ciencias Veterinarias. 22 (2), 128-134 (2012).
- Mian, L. S., Maag, H., Tacal, J. V. Isolation of Salmonella from muscoid flies at commercial animal establishments in San Bernardino county, California. Journal of Vector Ecology. 27 (1), 82-85 (2002).
- Olsen, A. R., Hammack, T. S. Isolation of Salmonella spp. from the housefly, Musca domestica L., and the dump fly, Hydrotaea aenescens (Wiedemann) (Diptera:Muscidae), at caged-layer houses. Journal of Food Protection. 63 (7), 958-960 (2000).
- Holt, P. S., Geden, C. J., Moore, R. W., Gast, R. K. Isolation of Salmonella enterica serovar Enteritidis from houseflies (Musca domestica) found in rooms containing Salmonella serovar Enteritidis-challenged hens. Applied and Environmental Microbiology. 73 (19), 6030-6035 (2007).
- Mramba, F., Broce, A., Zurek, L. Isolation of Enterobacter sakazakii from stable flies, Stomoxys calcitrans L. (Diptera:Muscidae). Journal of Food Protection. 69 (3), 671-673 (2006).
- Koyuncu, S., Andersson, M. G., Haggblom, P. Accuracy and Sensitivity of Commercial PCR-Based Methods for Detection of Salmonella enterica in Feed. Applied and Environmental Microbiology. 76 (9), 2815-2822 (2010).
- Gagné, R. Insect and mite pests in food, an illustrated. 1, U.S. Department of Agriculture and U.S. Department of Health and Human Service. 269-296 (1991).
- Greenberg, B. Flies and disease, Vol I. Ecology, classification and biotic associations. , Princeton University Press. (1971).
- Wallace, H. A., Jacobson, A., Hammack, T. S. Chapter 5 Salmonella. Bacteriological Analytical Manual. , Available from: http://www.fda.gov/downloads/Food/FoodScienceResearch/UCM244774.pdf (2007).
- Barro, N., Aly, S., Tidiane, O. C. A., Sababenedjo, T. A. Carriage of bacteria by proboscises, legs, and feces of two species of flies in street food vending sites in Ouagadougou, Burkina Faso. Journal of Food Protection. 69 (8), 2007-2010 (2006).
- Ugbogu, O. C., Nwachukwu, N. C., Ogbuagu, U. N. Isolation of Salmonella and Shigella species from house flies (Musca domestica L.) in Uturu, Nigeria. African Journal of Biotechnology. 5 (11), 1090-1091 (2006).
- Brownlie, J. C., Johnson, K. N. Symbiont-mediated protection in insect hosts. Trends in Microbiology. 17 (8), 348-354 (2009).
- Feldhaar, H. Bacterial symbionts as mediators of ecologically important traits of insect hosts. Ecological Entomology. 36 (5), 533-543 (2011).
- Foodborne disease outbreaks: Guidelines for investigation and control. , World Health Organization. Available from: http://www.who.int/foodsafety/publications/foodborne_disease/outbreak_guidelines.pdf (2008).
- Wright, A. P., Gould, L. H., Mahon, B., Sotir, M. J., Tauxe, R. V. Overview of the Impact of Epidemic-Assistance Investigations of Foodborne and Other Enteric Disease Outbreaks, 1946-2005. American Journal of Epidemiology. 174, S23-S35 (2011).



