Introduction
Insekter spelar en viktig roll i överföringen av kostrelaterade sjukdomar eftersom de kan sprida patogener till livsmedel eller matkontaktytor och redskap en. Bland insekter, flugor, kackerlackor och myror uppvisar beteenden som gynnar spridningen av livsmedelsburna patogener. Dessa beteenden inkluderar en förening med ruttnande materia, avfall och avföring, endophily (in byggnader), och synanthropy (sambo med människor) 2. .. Foodborne patogener såsom Salmonella spp, Listeria monocytogenes, Campylobacter spp, Escherichia coli O157: H7, och medlemmar av släktet Cronobacter (tidigare Enterobacter sakazakii) har rapporterats som skall överföras via insekter 3-5. Synanthropic filth flugor mekaniskt sprida livsmedelsburna bakterier genom att överföra patogener från sina förorenade huden. Emellertid kan närvaron av livsmedelsburna patogener i matsmältningskanalen av flugor vara upp till tre gångerstörre än den som observerades på deras kroppsytor (kropp, huvud, ben och vingar) 5. Foodborne patogener kan också stanna kvar i flugans matsmältningskanalen för en större tid än på kroppsytan 6,7 och i vissa fall, kan de föröka sig, kolonisera flugans tarmkanalen 4,8,9. Detta ökar vektorpotentialen av flugor, eftersom de kan spridas vidare livsmedelsburna patogener genom avföring och uppstötningar 10,11.
Numera är det förbättrade övervakningssystem som är i stånd att detektera foodborne sjukdomsutbrott snabbare. När de utför livsmedelsburna undersökningar av sjukdomsutbrott folkhälsotjänstemän leta efter mat som kan vara källan (er) eller fordon (s) för infektion. Utredarna kan också utföra en miljöbedömning av anläggningen (eller anläggningar) inblandade att ta reda på hur maten var förorenad och kan samla in prover som en del av utredningen 12. Despite den stora mängd vetenskaplig litteratur om de insekter som bärare av livsmedelsburna patogener, länka insekter som vektorer för patogenen orsakar en viss livsmedelsburna sjukdomar utbrott har varit utmanande. Detta beror främst på insekter inte blir aseptiskt samlas in som en del av miljöprovtagningsprogram under livsmedelsburna undersökningar av sjukdomsutbrott. Om du vill inkludera insekter, särskilt de som uppvisar beteenden som gynnar spridningen av livsmedelsburna patogener, som en del av ett provtagningsförfarande miljö, ett standardiserat, snabb, känslig och tillförlitlig protokoll för att upptäcka livsmedelsburna patogener från en enda insekt måste vara på plats.
Traditionella plätering tekniker för detektion av livsmedelsburna patogener från insekter är arbetskrävande och beror på konkurrens tillväxt målbakterien i olika odlingsmedier för att övervinna den snabba tillväxten av den medfödda kommensala floran i insekten. De flesta av de studier som har associerade insekter med bacterial patogener har ökat känsligheten av metoden genom att samla ihop flera insekter snarare än att identifiera närvaron av patogener på en per individuell basis. Således gjorde dessa studier inte differentiera kroppsdel av insekten där patogener påträffades 13-18. Förmågan att identifiera huruvida livsmedelsburna patogener är placerade på kroppsytan eller i matsmältningskanalen hos en individ insekt är viktigt eftersom detta kan ha epidemiologiska implikationer och kan leda till olika begränsningsstrategier. Som mekaniska vektorer, flugor att landa på mat för en kort tid får endast överföra låga nivåer av bakterier från sin kroppsyta, medan de flugor som spy och bajsa på maten öka sannolikheten för överföring patogener på potentiellt högre infektion. Följaktligen är det viktigt att uppskatta förekomsten av en livsmedelsburen patogen per individuell insekt och differentiera kroppsdelen av denna insekt där den bakteriella p athogen ligger.
Även om användningen av kulturoberoende metoder för att upptäcka livsmedelsburna patogener alltmer genomförs, de har inte använts kommersiellt för att upptäcka livsmedelsburna patogener från en enda insekt. För närvarande finns det validerade molekylära protokoll som är kommersiellt tillgängliga för snabb detektion av livsmedelsburna patogener från livsmedel som används av industrin och tillsynsmyndigheter. Dessa metoder innefattar DNA-baserade system för detektion av patogener i en mängd olika livsmedelsprover. Även molekylära protokoll är snabbare än traditionella plating metoder, är anrikning av provet fortfarande krävs för att erhålla känslighetsnivån 10 2 kolonibildande enheter (CFU) av bakteriell patogen behövs polymerase chain reaction (PCR) -baserade metoder 19. Dessutom behövs isolering av rena bakteriekolonier från PCR-positiva prov för att bekräfta patogenen med lämpliga metoder.
innehåll "> Syftet med detta protokoll är att standardisera en kommersiellt tillgänglig PCR-baserat system som används för att upptäcka patogener från livsmedels- och miljöprover för upptäckt av livsmedelsburna bakterier från kroppsytan och matsmältningskanalen av en enda fluga och att ytterligare isolera dem patogener från samples.The känslighet av protokollet som beskrivs här var kalibreras först med lab-uppfödda vuxen husflugor (Musca domestica) som var experimentellt utfodrats med seriespädningar av varje bakteriell patogen. Det standardiserade protokoll användes därefter för att kartlägga 100 vildfångad flugor för förekomst av livsmedelsburna patogener från sina kroppsytor och / eller matsmältnings kanaler. Denna standardiserat protokoll tillåter folkhälso laboratorier för att upptäcka hälsorisker förknippade med insekter, vilket gör det möjligt att samla in dem som en del av miljöprovtagningsprogram när du utför livsmedelsburna undersökningar av sjukdomsutbrott.Protocol
1. Insamling av flugor
- Samla enskilda flugor använder sterila entomologiska svep nät. Sätt näten i ett svalare och överföra dem till labbet.
2. Dissekering av flugor
- Immobilisera aseptiskt insamlade flugor genom att placera dem vid -20 ° C 5 - 7 min.
- Använda steril pincett plats en fluga i ett sterilt 2 ml rör innehållande 1 ml förvärmda (37 ° C) buffrat peptonvatten (BPW). Blanda röret försiktigt genom inversion under 2 min. Det är väsentligt att hela kroppen av flugan vara i kontakt med media så att mikrobiota närvarande på kroppsytan (S) av flugan kommer att överföras till BPW (BPW-S). Märk röret med ett nummer och kroppsdel av flugan (dvs., 1S).
OBS: Se Tabell över specifika reagenser / Utrustning för en detaljerad beskrivning av material och reagenser som nämns i detta protokoll. - Använda steril tång bort flugan från BPW-S media och överföringden till en tom och ren 2 ml rör till ytan desinficera flugan. Inkubera röret innehållande BPW-S media vid 37 ° C under utförandet av desinfektion och dissektion protokoll.
- Utanpå desinficera flugan genom nedsänkning i 1 ml 70% etanol under 1 minut, följt av en sköljning med sterilt destillerat vatten före neddoppning i en ml av nyframställd 0,05% (volym / volym) blekmedelslösning. Skölj tre gånger med sterilt destillerat vatten. Överför vatten från den sista sköljningen till en autoklaverad 2 ml rör.
OBS: Kasta vätskan varje gång genom att använda en 1000 l mikropipett eller genom att vända röret och se flugan kvar inuti röret. Blanda försiktigt genom inversion vid varje steg i ytan-desinfektionsprocessen. - För att utvärdera effektiviteten av desinficeringsprocessen, överför 100 pl av vattnet från den sista sköljningen till ett tryptikas soja agar (TSA) platta och sprids med hjälp av en steril L-formad engångsspridare. Inkubera plattan vid 37 ° C under 24 h. Efter icubation, registrera förekomst av bakteriekolonier.
OBS: Förekomsten av bakteriekolonier på TSA-plattor indikerar en ineffektiv yta-desinfektionsprocessen. Om detta inträffar bör förekomsten av livsmedelsburna patogener endast rapporteras på kroppsytan av flugan eftersom korskontaminering mellan kroppsytan och matsmältningskanalen inte kan uteslutas.
- Utanpå desinficera flugan genom nedsänkning i 1 ml 70% etanol under 1 minut, följt av en sköljning med sterilt destillerat vatten före neddoppning i en ml av nyframställd 0,05% (volym / volym) blekmedelslösning. Skölj tre gånger med sterilt destillerat vatten. Överför vatten från den sista sköljningen till en autoklaverad 2 ml rör.
- Efter utanpå desinficering flugan, överföra den till en bit autoklaveras pappershandduk för att avlägsna överflödigt vatten och sedan till en steril 60 mm disponibel petriskål.
- Placera petriskål under ett dissekera omfattning och identifiera flugan till artnivå använder dikotoma nycklar för Dipteran familjer 20,21.
- Använda autoklave fin spets pincett försiktigt dra anus och hela matsmältningskanalen (A) ur flugan och aseptiskt överföra det till en annan steril 2 ml rör innehållande 1 ml förvärmda (37 ° C) BPW med 0,5 mm zirconia / kiseldioxid pärlor (BPW-A). Etikett the rör med samma nummer väljs för den enskilda fluga och kroppsdelen av flugan (dvs 1A).
- Blanda röret med BPW-En noggrant under 5 - 10 min med hjälp av en cell disruptor. Inkubera vid 36 ± 1 ° C medan du utför resten av protokollet.
- Att verifikationen och / eller förvara provet för långsiktig, placera resten av flugan i ett rent 2 ml rör och till 1 - 2 ml 95% etanol.
3. Primär och Sekundär Berikning
- Märk alla primära och sekundära anriknings rör innehållande medier enligt provnummer och kroppen en del av flugan.
- Under en steril huva, överföra 300 pl BPW-S (yta) för att sterila 2 ml rör innehållande följande medier:
- För Salmonella, använd 1 ml förvärmda (42 ° C) BPW. Inkubera i en recirkulerande vattenbad vid 42,5 ° C under 22-24 h. För sekundär anrikning, överföra 100 l anrikat BPW till 400 & #181; l förvärmda (37 ° C) hjärna-hjärta-infusion (BHI) buljong som tidigare placerats i sterila kluster rör. Inkubera vid 37 ° C under 3 h.
- För Cronobacter, använd 1 ml förvärmda (37 ° C) BPW med novobiocin (10 mg / L; Wallace, M., personlig kommunikation). Alternativt kan du använda 1 ml i R & F Enterobacter sakazakii anrikningsbuljong med tillägg (vankomycin och cefsulodin) som primär anrikning. Inkubera vid 37 ° C under 22-26 h. För sekundär anrikning, överföra 100 pl anrikat BPW med novobiocin till 400 pl förvärmda (37 ° C) BHI buljong som tidigare placerats i sterila kluster rör. Inkubera vid 37 ° C under 3 h.
- För L. monocytogenes, använd 1 ml nyberedd RT 24 Listeria anrikningsbuljong (24 LEB) med selektiv tillägg. Inkubera vid 37 ° C under 44 ± 5 h. Ingen sekundär anrikning krävs för detektion av L. monocytogenes. Upprepa steg 3.2.1 - 3.2.3 ovan att använda röret märkt som BPW-A.
4. Beredning av PCR-baserat system för amplifiering och detektion av Target Foodborne Pathogen
Steg 4-8 använder en kommersiell PCR cycler / detektorsystem, en datorarbetsplats, och färdiga att använda byggsatser till skärmen för salmonella (Salmonella 2 standardanalyssats), Cronobacter arter (E. sakazakii standardanalyssats), och Listeria monocytogenes (L. monocytogenes 24E analyssats). Standardanalyser använda PCR-ändpunktsdetektering. Varje sats innehåller PCR-färdiga tabletter med ett infogat färgämne som avger en fluorescenssignal när bindning till dubbelsträngat DNA. Signalen fångas under fasen av PCR systemprogrammet upptäckt, generera en smältkurva som tolkas av programvaran som positiva eller negativa.
- Förbered reagenser och den utrustning som anges av fabrikationsfelrer protokoll per varje mål livsmedelsburna patogener.
OBS: De protokoll för att upptäcka salmonella och Cronobacter kräver ett steg lys förfarande medan protokoll för detektering L. monocytogenes kräver en tvåstegsprocess lys förfarandet (se avsnitt 5 och 6, respektive). - Slå på automatiserade värmeblocket väljer ett särskilt program för målet patogenen. Alternativt, om värmeblocken är manuell, ställa temperaturer till 37 ° C (för salmonella, Cronobacter spp, och L. monocytogenes.) Eller till 55 ± 2 ° C (för L. monocytogenes del 2 av lys - se steg 6.2) och 95 ± 3 ° C.
- Se till att kylningsblocken har varit kylda O / N, annars kyla dem vid 2-8 ° C under minst 2 h.
- Med hjälp av datorn mjukvaran av PCR-baserade detekteringssystemet, skapar ett rack fil enlighet med tillverkarens instruktioner.
- Märk och ordna klusterRören innehållande lyseringsreagenset i racket, enligt kuggstången filen.
- Initiera PCR-baserade system för upptäckt instrument.
5. Utför Lys för detektion av Salmonella och Cronobacter
- Förbered lyseringsreagenset genom tillsats 150 pl av proteas till en 12 ml flaska av lysbuffert.
- Överför 200 l av lysisreagens till var och en av de tidigare märkta klusterrören.
OBS: Kluster rör innehållande lyseringsreagenset kan förvaras vid 2-8 ° C i upp till 2 veckor. - Använda långa pipettspetsar, överföra 20 pl sekundära berikade prover (se steg 3.2.1 och 3.2.2) till motsvarande kluster rör innehållande 200 ul lysisreagens. Använd nya pipettspetsar för varje prov.
OBS: Håll rören från primär och sekundär anrikning i kylskåp (Salmonella) eller vid RT (Cronobacter) för ytterligare bekräftelse analys av PCR-positiva / negativa prover. Förbered negativa kontroller genom att tillsätta 20 l sterilt BHI media till kluster rör innehållande 200 ul lysisreagens. - Bered positiva kontroller genom att tillsätta 20 pl O / N bakteriekulturer (odlas i BHI) av någon känd Salmonella eller Cronobacter stam till kluster rör innehållande 200 ul lysisreagens.
- Cap klusterrören och säkra ordentligt med täckverktyget.
- Placera racket av kluster rör i den automatiserade värmeblocket efter val av det särskilda programmet för målet patogenen. Alternativt inkubera kluster rören vid 37 ± 2 ° C under 20 min, följt av inkubation vid 95 ± 3 ° C under 10 min. Slutligen, överföra klusterrören till kyla block (2 - 8 ° C) i 5 min.
OBS: Kluster rör innehållande lysatet kan lagras vid -20 ° C i upp till 2 veckor.
6. Utför Lys för detektion av L. monocytogenes
- Utför del ett avlys på följande sätt:
- Lägg 1,8 ml sterilt avjoniserat vatten till flaska helt tinade lyseringsmedel 1.
OBS: Affär lyse agent 1 vid 2 - 8 ° C tills den ska användas. Efter öppning och utspädande, lagra vid RT (20-30 ° C) i upp till 6 månader. - Kombinera lysemedel 1 och 2 i en 4: 1-förhållande (40 fil utspätt lyseringsmedel 1 och 10 pl av lyseringsmedel 2 per varje prov). Överför 50 ìl av de kombinerade lyserande agenter att klustra rören. Använd blandningen inom 4 h.
- Lägg 500 l av primär anrikat prov (se steg 3.2.3) till klustret röret innehållande 50 pl av de kombinerade lyserande medel.
- Förbered en negativ kontroll genom att tillsätta 500 l sterilt 24 LEB till 50 pl de kombinerade lyserande medel.
- Förbered en positiv kontroll genom att lägga 500 l O / N L. monocytogenes kultur odlas i 24 LEB till 50 pl de kombinerade lyserande medel.
- Cap klusterrören, blanda försiktigt och platsi uppvärmningsblock vid 37 ± 1 ° C under 30 minuter.
OBS: Håll rören från primär anrikning i kylskåp i ytterligare bekräftelse analys av PCR-positiva / negativa prover.
- Lägg 1,8 ml sterilt avjoniserat vatten till flaska helt tinade lyseringsmedel 1.
- Utför del 2 av lys enligt följande:
- Förbered lyseringsreagenset enligt instruktionerna i steg 5.1 och 5.2.
- Använda långa pipettspetsar överför 20 pl del ett lysat till kluster rör innehållande 200 ul lysisreagens. Använd nya pipettspetsar för varje prov.
- Cap klusterrören och säkra ordentligt med täckverktyget.
- Placera kluster rör i automatiserad värmeblock väljer ett särskilt program för L. monocytogenes. Alternativt, inkubera klusterrören vid 55 ± 2 ° C i 30 min, följt av inkubation vid 95 ± 3 ° C i 10 min. Slutligen, överföra klusterrören till kyla block (2 - 8 ° C) i 5 min.
OBS: Kluster rör innehållande lysatet kan lagras vid -20 ° C i upp till 2 veckor.
7. Hydrate PCR-Ready Tablets
- Välj en kyld (4 ° C) PCR kylblocket och placera en PCR-rör rack över insatsen.
- Placera motsvarande PCR-rör innehållande PCR-färdiga tabletter (medföljer varje kit) för målet livsmedelsburna patogener i hållaren, enligt rack filen.
- Med hjälp av decapping verktyget försiktigt bort locken från PCR-rör. Kasta locken och kontrollera att varje rör innehåller en tablett.
- Överför 50 jil (för Salmonella och Cronobacter) eller 30 | il (för L. monocytogenes) av lysat till specifika PCR-rör. Använd nya optiska lock och säkra stadigt på PCR-rören med hjälp av tak verktyget.
OBS: Efter tillsats av lysatet till PCR-redo tabletter, prov bevaras kylda vid 2-8 ° C innan den har laddats in i den PCR-baserade detekteringssystemet. De PCR-rör kan centrifugeras vid 2500 xg under några sekunder för att säkerställa att hela volymen är i botten than rör. - Ladda PCR-rören i PCR cycler / detektionssystem instrument genom att öppna instrumentlådan.
- Placera racket av PCR-rör i brunnarna i lådan och kontrollera att rören sitter korrekt.
- Stäng lådan och initiera programmet som beskrivs av tillverkarens protokoll.
OBS: PCR-baserade instrument har förinställt cykelparametrar för varje livsmedelsburen patogen. - Kontrollera att PCR-cykling statusfältet visas ett blått fält indikerar att förstärkningen delen av programmet är igång.
OBS: För standard PCR-analyser, behandlingstiden för hela programmet (amplifiering och detektion) tar ca 3-3,5 timmar att slutföra.
8. Granska Resultat
- Efter behandlingen är klar, följ instruktionerna på skärmen från PCR-baserat system instrument för att ta prover och granskningsresultat.
- Om målet foodborne patogenen är närvarande i provet (antingenyta eller matsmältningskanalen av flugan) brunnen är rött med en "plus" tecken (positivt). Om patogenen är frånvarande, är väl grön med en "minus" tecken (negativ).
- Om brunnen är gul med ett rött streck över centrum, betyder det en signal fel.
9. Isolering sjukdomsalstrande bakterier från PCR-positiva resultat
- Välj rören från den primära (för L. monocytogenes) eller sekundär (för Salmonella och Cronobacter) anrikning av dessa prover som var PCR-positiva. Också, slumpmässigt välj 3 - 5% av proverna som var PCR-negativa och gör så här:
- För Salmonella:
- Lägg 100 pl av den sekundära anrikningsmedier till 10 ml Rappaport-Vassiliadis (RV) medium och till ett ml tetrationat (TT) buljong. Inkubera rören vid 42,5 ° C i ett recirkulerande vattenbad för 22-24 tim.
- Efter inkubation, strimma en 3 mm ögla (10 pl) av varje, RV ettd TT media på vismut sulfit (BS) agar, xylos lysin desoxycholat (XLD) agar, och Hektoen enter (HE) agar. Inkubera plattorna vid 35 ± 1 ° C under 22-24 h.
- Efter inkubation, undersök plattorna för närvaro av typiska Salmonella kolonier på varje media. Om inga isolerade kolonier kan erhållas efter flera sub-odling steg, anser provet som negativa och rapportera som ett falskt positivt för PCR-baserade system.
OBS: För typiska Salmonella kolonier på specifika medier ser 22. Välj fem presumtiva typiska Salmonella kolonier och subkultur dem på BS, XLD, eller HE tills rena kulturer av isolerade / enkla kolonier erhålls. - Välj en ren koloni och identifiera presumtiva Salmonella genom biokemiska kommersiella tester såsom VITEK 2 Legitimation eller API biokemiska identifikationssystem, enligt tillverkarens anvisningar.
- För Cronobacter: <ol>
- Streak 3 mm ögla (10 pl) av den sekundära anrikningsmedia på två plattor av kromogena odlingsmedia som R & F Enterobacter sakazakii (Cronobacter) kromogent plätering medium och / eller ChromID sakazakii Agar. Inkubera plattorna vid 35 ° C under 22-24 h.
- Efter inkubation, undersök plattorna för närvaro av typiska Cronobacter kolonier (blå-svart till blå-grå). Välj fem presumtiva Cronobacter kolonier och subkultur dem på R & F Enterobacter sakazakii (Cronobacter) kromogent plätering medium, ChromID sakazakii Agar, eller TSA tills rena kulturer av isolerade / enkla kolonier erhålls.
OBS: Om inga isolerade kolonier kan erhållas efter flera sub-odling steg, anser provet som negativa och rapportera som ett falskt positivt för PCR-baserade detektionssystem. - Välj en ren koloni och identifiera presumtiva Cronobacter genom biokemisk kommersiellella tester såsom VITEK 2 Legitimation eller API 20E biokemiska identifieringssystem, efter tillverkarens anvisningar.
- Serie en 3 mm ögla (10 | il) av den primära anrikningsmedia på två plattor av briljans Listeria agar (BLA). Inkubera plattorna vid 36 ± 1 ° C under 22-26 h.
- Efter inkubation, undersöka plattor för förekomst av presumtiva L. monocytogenes (blågröna) kolonier. Välj 5 presumtiva L. monocytogenes kolonier och subkultur dem på BLA tills erhålla rena kulturer av isolerade / enkla kolonier. Åter inkubera negativa plattorna vid 36 ± 1 ° C under ytterligare 22 till 26 h.
- Välj en ren koloni och identifiera presumtiva L. monocytogenes med hjälp kommersiella biokemiska tester såsom VITEK 2 Legitimation eller API Listeria biokemiska identifieringssystem, enligt tillverkarens jagnstructions.
Representative Results
Detta protokoll var först kalibreras på en uppsättning lab-uppfödda husflugor som experimentellt utfodras under 24 timmar med flytande fluga livsmedel (2% pulvermjölk) som innehåller serieutspädningar (10 februari-10 Augusti CFU / ml) av C. sakazakii, S. enterica, L. monocytogenes, eller C. jejuni (n = 21 för varje bakteriell patogen). Anriknings medier samt inkubationstider och temperaturer justerades för varje livsmedelsburna patogener tills PCR-baserade systemet kunde upptäcka de lägsta nivåerna av bakterier (10 2 CFU / ml) från kroppsytan och matsmältningskanalen av en enda experimentellt matas flyga. Använda anrikningsmedier och villkor som beskrivs i protokollet avsnittet upptäckte PCR-baserat system C. sakazakii, S. enterica, och L. monocytogenes från kroppsytan på 100% av flugor som utfodrats med bakteriella inokulat> 10 3 CFU / ml (Figur 1a). När flugor matades med 102 CFU / ml, den procentuella andelen av detektion av C. sakazakii, S. enterica, och L. monocytogenes från deras kroppsyta var 100%, 66%, och 33% respektive (Figur 1A). PCR-baserade systemet upptäcktes även dessa tre livsmedelsburna patogener från matsmältningskanalen av flugor som utfodrats med alla bakteriekoncentrationer på procentsatser ≥33% (Figur 1B). Emellertid detektion av C. jejuni endast uppnås när lab-uppfödda flugor experimentellt matas med flytande livsmedel som innehåller den högsta bakterieinokulat (10 8 CFU / ml). Följaktligen, C. jejuni uteslöts ur gruppen av livsmedelsburna patogener som kunde testas från individ synanthropic filth flugor använder denna PCR-baserade system för upptäckt.
Med denna standardiserat protokoll, kunde vi fastställa förekomsten av Cronobacter spp., S. enterica, och L. monocytogenes från kroppsytan och / eller the matsmältningskanalen 100 vilda flugor som var för sig och aseptiskt fångats från dumpster området tio urbana restauranger som ligger i huvudstadsregionen i Washington, DC 5 Samlade Filth flugor var representativt för minst sex arter, inklusive M. domestica (47%), Lucilia cuprina (33%), L. sericata (14%), Cochliomyia macellaria (2%), Sarcophaga haemorrhoidalis (2%), och Ophyra Leucostoma (1%). En fluga identifierades endast till familjenivå (Blomsterflugor, 1%). Ytan-desinfektion protokoll var effektivt att undvika korskontaminering mellan kroppsdelar flyga eftersom ingen bakterietillväxt observerades på TSA-plattor för vatten från den senaste desinfektionen skölj av varje enskild fluga. Således kan man skilja mellan livsmedelsburna bakterier på kroppsdelar av varje fluga.
Inga falska positiva upptäcktes från prover av kroppsytan och matsmältningskanalen avenskilda flugor när du använder denna kommersiella PCR-baserat system för detektion av S. enterica och L. monocytogenes och bekräftelse av livskraftiga patogener på agarplattor var överens med PCR-positiva resultat. Det var emellertid inte möjligt att isolera rena kulturer av Cronobacter spp. från alla PCR-positiva prover. Följaktligen detekteringen av denna patogen genom PCR-baserade Systemet visade falska positiva från kroppsytan (50%; 18/09) och matsmältningskanalen (48%; 16/33) av enstaka vildfångade flugor. Slumpvis utvalda PCR-negativa prover som pläterade på specifika medier, bekräftade frånvaron av livsmedelsburna patogener. Därför togs inga falska negativa detekteras från något av proverna vid användning av denna kommersiella PCR-baserat system för att detektera Cronobacter spp., S. enterica, eller L. monocytogenes.
Endast de PCR-positiva prov där patogenen isolerades och bekräftade ansågs positivtoch ingår för statistisk analys. Den totala förekomsten av livsmedelsburna patogener i matsmältningskanalen av vildfångade Filth flugor var betydligt högre än på kroppsytan (χ 2 = 6,8772, df = 1, p = 0,0087). 22% av de alimentära kanaler och 8% av kroppsytor insamlade vilda flugor var positiva för åtminstone en av de tre livsmedelsburna patogener (Figur 2). Sammantaget prevalensen av Cronobacter spp. på antingen kroppsytor eller matsmältnings kanalerna i insamlade flugor var statistiskt högre (19%; Fishers exakta test p = 0,0165) än förekomsten av S. enterica (7%) och L. monocytogenes (4%). Dock inga statistiska skillnader observerades när du utför parvisa jämförelser mellan kroppsdelar flugorna för varje bakteriell patogen (Figur 3; Fishers exakta test p = 0,1464, p = 0,1184 och p = 0,6212 för Cronobacterspp., S. enterica, och L. monocytogenes, respektive). Ingen av flugorna var positiva för alla tre patogener utvärderade. Men tre av flugor (två L. cuprina och en L. sericata) genom Salmonella spp. och L. monocytogenes på ytan eller i matsmältningskanalen.
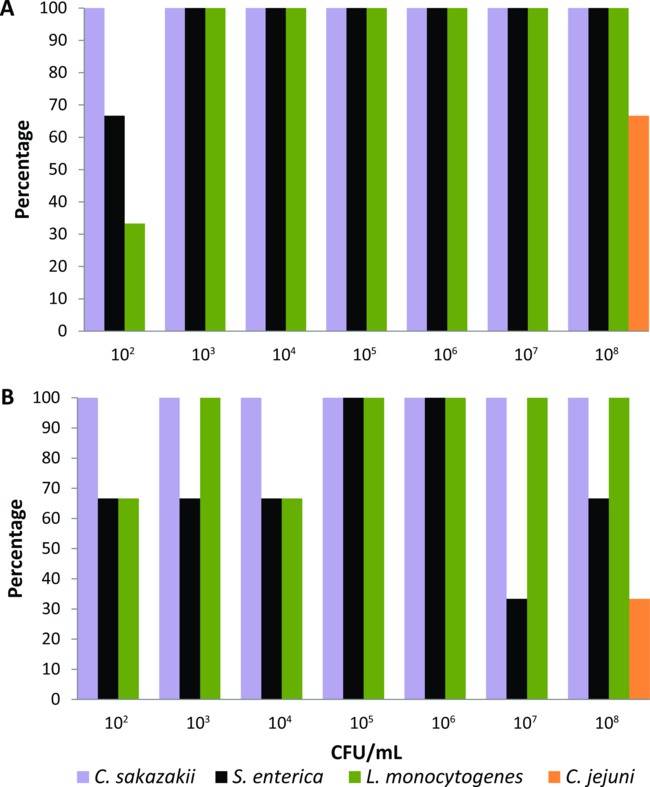
Figur 1. Detektionsnivåer Enterobacter sakazakii, Salmonella enterica, Listeria monocytogenes, och Campylobacter jejuni från (A) kroppsytan och (B) matsmältningskanalen individuella lab-uppfödda husflugor matas med flytande livsmedel som innehåller olika bakterie inokulat (n = 21 för varje bakteriell patogen, n = 3 per varje bakteriekoncentration).
Figur 2. Andel kroppsytor och matsmältnings kanalerna i enskilda flugor hittade positivt för någon av de mål livsmedelsburna patogener.

Figur 3. Förekomst av Cronobacter spp., Salmonella enterica och Listeria monocytogenes från kroppsytan och matsmältningskanalen av synanthropic vildfångade flugor. P-värden som rapporteras är från parvisa jämförelser mellan kroppsytan och matsmältningskanalen för varje bakteriell patogen (Fishers exakta test, p-värde <0,05 indikerar statistisk signifikans). Copyright © American Society for Microbiology, Journal of Applied and Environmental Microbiology 78 (22): 7891-902, 2012. doi: 10,1128 / AEM.02195-12.
Discussion
Tidigare studier som har upptäckts livsmedelsburna patogener från vilda insekter har använt en stor variation av protokoll som inte kan innehålla den information som behövs för att korrekt bedöma maten relaterad risk för förekomst av en enda fluga i livsmedel eller livsmedelsrelaterade miljöer 13,15, 23,24. Här visade vi att användningen av denna standardiserade protokollet, är det möjligt att detektera och isolera Cronobacter spp., S. enterica, och L. monocytogenes från kroppsytan och matsmältningskanalen av enstaka flugor som fångats i vilt. Eftersom insekter kan bära lågt antal av målet livsmedelsburna patogener och ett stort antal andra inhemska mikroorganismer 25,26, kräver detta protokoll primära (och ibland sekundär) anrikning av proverna i specifika odlingsmedier för att öka känsligheten för upptäckt av målet livsmedelsburna patogener . Resultat från PCR-baserade system för upptäckt erhölls inom ca 30 tim (för DETEInsatser av Cronobacter spp. och S. enterica) och 48 h (för detektion av L. monocytogenes) efter initialt bearbeta prover. Således är detta protokoll tillförlitlig samt snabb och känslig nog för att screena en enda fluga för förekomst av livsmedelsburna patogener.
Bekräftelse av PCR-positiva resultat och isolering av livsdugliga bakterier är en del av det standardförfarande för många laboratorier. Dessutom, för epidemiologiska ändamål, är rena bakteriekulturer från PCR-positiva prov som krävs för att ytterligare bekräfta och serotyp av livsmedelsburna patogener använder biokemiska, immunologiska eller genetiska metoder. Även om inga falska positiva observerades vid detektering S. enterica och L. monocytogenes från kroppsdelar av enstaka vildfångade flugor, med hjälp av detta protokoll, fann vi upp till en 50% av antalet falska positiva för Cronobacter spp. Detta antyder att det PCR-baserade systemet för släktet Cronobact detekteringser kan korsreagera med andra Enterobacteriaceae närvarande bland de mycket komplexa floran bärs av flugor. Således, isolering och rening av rena kolonier av släktet Cronobacter från PCR-positiva prover kräver mer selektiv plätering än de andra patogener utvärderades.
Detta protokoll har främst standardiserats för att screena individuella vildfångade flugor för förekomst av Cronobacter spp., S. enterica, och L. monocytogenes använder en kommersiell PCR-baserade system för upptäckt. Dock var detta protokoll också enkelt anpassas till skärmen kroppsdelar av enstaka flugor för förekomst av andra livsmedelsburna patogener som enterohemorragisk E. coli O157: H7 (antingen med E. coli O157: H7 MP standardanalys kit eller E. coli O157: H7 realtidsanalys kit) och shiga toxinbildande E. coli (STEC) grupp (med användning av realtids STEC suite), erhålla känslig> 80% (Avpubliceraed uppgifter). Dessutom kan detta protokoll potentiellt anpassas för att upptäcka livsmedelsburna patogener från andra insekter som är kända vektorer sjukdomar (kackerlackor och myror), men det behövs mer forskning på området.
Foodborne utredningar sjukdom utbrotts är mycket dynamisk och omfattar en flerstegsprocess som kan variera beroende på den specifika situationen och den lokala miljön utreds 12,27. Dessa undersökningar är viktiga eftersom de ger omedelbart skydd för folkhälsan genom att förebygga framtida sjukdomar. Dessutom kan dessa undersökningar klarlägga nya mekanismer genom vilka livsmedelsburna mikroorganismer sprids, och väcker viktiga frågor som leder till nya områden för forskning 28. Utredningsteknik samt standardiserade, snabba och känsliga protokoll är nödvändiga för att upptäcka livsmedelsburna patogener från enskilda insekter. Denna standardiserat protokoll öppnar möjligheten att aseptiskt samla insekter som flugor, which kan vektor den livsmedelsburen bakteriell patogen, som en del av en miljöprovtagningsprogram. Den epidemiologiska information som kan vinna på detta skulle vara till nytta för att konstruera en korrekt bild av mekanismerna för överföring av livsmedelsburna patogener av insekter (dvs, längd exponeringstid: en fluga genom att landa kontra flugor landning, avföring, och regurgitating).
Slutligen, trots att den kommersiella PCR-baserade system för upptäckt som beskrivs här är praktiskt att använda och förenklar PCR-amplifiering och visualisering av ett släkte-nivå amplikon, är det ingalunda den enda lämpliga systemet. Lysatet från anrikade prover skulle alternativt kunna användas för att screena för närvaron av livsmedelsburna patogener genom att använda allmänt tillgängliga artspecifika primerpar. Dock bör detektionskänsligheten påvisas innan de används.
Materials
| Name | Company | Catalog Number | Comments |
| Bismuth sulfite (BS) agar | Fisher Scientific | R452402 | *Multiple suppliers. |
| Brain heart infusion (BHI) broth | Becton, Dickson and Company | 299070 | *Pre-warmed to 37°C. Multiple suppliers. |
| Brilliance Listeria agar (BLA) | Fisher Scientific | CM1080B | *Multiple suppliers. |
| Buffered peptone water (BPW) | Becton, Dickson and Company | 212367 | *Pre-warmed to 37°C or 42°C. Multiple suppliers. |
| Brilliance Cronobacter agar (Druggan-Forsythe-Iversen formulation/DFI) | Fisher Scientific | CM1055B | *Multiple suppliers. |
| chromID Sakazakii Agar | bioMérieux | 43741 | *Call for information: 800.682.2666 |
| R & F Enterobacter sakazakii (Cronobacter) Chromogenic Plating Medium | R & F Laboratories | Various | *Call for information: +1.630.969.530 |
| R & F Enterobacter sakazakiii Enrichment Broth and supplement | R & F Laboratories | Various | *Call for information: +1.630.969.530 |
| Hektoen enteric (HE) agar | Fisher Scientific | OXCM0419B | *Multiple suppliers. |
| 24 Listeria enrichment broth (24LEB) | Oxoid | CM1107 | *Freshly prepared at room-temperature. Multiple suppliers. |
| Listeria selective enrichment supplement | Oxoid | SR0243 | *Multiple suppliers. |
| Novobiocin | Fisher Scientific | OXSR0181E | *Multiple suppliers. Store at 2-8 °C |
| Vancomycin hydrochloride hydrate | Sigma Aldrich | 861987 | Store at 2-8 °C |
| Cefsulodin sodium salt hydrate | Sigma Aldrich | C8145 | Store at 2-8 °C |
| Rappaport-Vassiliadis (RV) medium | Fisher Scientific | CM0669B | *Multiple suppliers. |
| Tetrathionate (TT) Broth | Becton, Dickson and Company | 249120 | *Multiple suppliers. |
| Trypticase soy agar (TSA) | Becton, Dickson and Company | 236930 | *Multiple suppliers. |
| Xylose lysine desoxycholate (XLD) agar | Becton, Dickson and Company | 221284 | *Multiple suppliers. |
| API Biochemical identification system | bioMérieux | Various | *Call for information: +1.800.682.2666 |
| VITEK 2: Product Safety | bioMérieux | Various | *Call for information: +1.800.682.2667 |
| BAX System Q7 | DuPont | ||
| BAX E. sakazakii Standard assay kit | DuPont | D11801836 | |
| BAX L. monocytogenes 24E assay kit | DuPont | D13608125 | |
| BAX Salmonella 2 Standard assay kit | DuPont | D14368501 | |
| Capping tool | DuPont | D11677028 | |
| Decapping tool | DuPont | D11134095 | |
| PCR tube rack/holder | DuPont | D12701663 | |
| Featherweight forceps, wide tip | BioQuip | 4750 | Sterilize before use. Multiple suppliers. |
| Fine point, straight tip forceps | BioQuip | 4731 | Sterilize before use. Multiple suppliers. |
| Zirconia/silica beads, 0.5 mm | Bio Spec Products, Inc. | 11079105z | Multiple suppliers. |
| Petri dishes - 60X15mm | Fisher Scientific | 08-772B | Multiple suppliers. |
| Disposable inoculating loops, 10µL | Fisher Scientific | 22-363-606 | Multiple suppliers. |
| L-shaped cell spreaders | Fisher Scientific | 14-665-230 | Multiple suppliers. |
| Microcentrifuge tubes, 2 ml | Fisher Scientific | Various | Sterilize before use when needed. Secure lid is preferred. Multiple suppliers. |
| Cluster tubes | Fisher Scientific | 05-500-13 | |
| Cluster tubes caps | Fisher Scientific | 05-500-23 | |
| Sodium hypochlorite (Liquid chlorine bleach) | *Dilute to 0.05% with water. Multiple suppliers. | ||
| Sterile deionized water | Multiple suppliers. | ||
| Sterile distilled water | Multiple suppliers. | ||
| Ethyl alcohol 190 proof | *Dilute to 70% with water when needed. Multiple suppliers. | ||
| Genie cell disruptor, 120V - for 1.5ml and 2.0ml microtubes | Scientific Industries, Inc. | SI-D238 | Multiple suppliers. |
| Heating block | Multiple suppliers. | ||
| Cooling block | Multiple suppliers. | ||
| Recirculating water bath | Multiple suppliers. | ||
| Stereo microscope | Multiple suppliers. | ||
| Centrifuge | Multiple suppliers. | ||
| Incubator | Multiple suppliers. |
References
- Voeller, J. G. Wiley Handbook of Science and Technology for Homeland Security. , Wiley Inc. 1-16 (2008).
- Olsen, A. R., Gecan, J. S., Ziobro, G. C., Bryce, J. R. Regulatory action criteria for filth and other extraneous materials V. Strategy for evaluating hazardous and nonhazardous filth. Regulatory Toxicology and Pharmacology. 33 (3), 363-392 (2001).
- Hald, B., Skovgård, H., Pedersen, K., Bunkenborg, H. Influxed insects as vectors for Campylobacter jejuni and Campylobacter coli in Danish broiler houses. Poultry Science. 87 (7), 1428-1434 (2008).
- Kobayashi, M., et al. Houseflies: Not simple mechanical vectors of enterohemorrhagic Escherichia coli O157:H7. American Journal of Tropical Medicine and Hygiene. 61 (4), 625-629 (1999).
- Pava-Ripoll, M., Pearson, R. E. G., Miller, A. K., Ziobro, G. C. Prevalence and relative risk of Cronobacter spp., Salmonella spp., and Listeria monocytogenes associated with the body surfaces and guts of individual filth flies. Applied and Environmental Microbiology. 78 (22), 7891-7902 (2012).
- Hewitt, C. G. Houseflies and how they spread disease. , Cambridge University Press. (1912).
- Rochon, K., Lysyk, T. J., Selinger, L. B. Retention of Escherichia coli by house fly and stable fly (Diptera:Muscidae) during pupal metamorphosis and eclosion. Journal of Medical Entomology. 42 (3), 397-403 (2005).
- Greenberg, B., Kowalski, J. A., Klowden, M. J. Factors affecting the transmission of Salmonella by flies: natural resistance to colonization and bacterial interference. Infection and Immunity. 2 (6), 800-809 (1970).
- Mramba, F., Broce, A. B., Zurek, L. Vector competence of stable flies, Stomoxys calcitrans L. (Diptera:Muscidae), for Enterobacter sakazakii. Journal of Vector Ecology. 32 (1), 134-139 (2007).
- Ekdahl, K., Normann, B., Andersson, Y. Could flies explain the elusive epidemiology of campylobacteriosis. Bmc Infectious Diseases. 5 (11), 11 (2005).
- Nayduch, D., Noblet, G. P., Stutzenberger, F. J. Vector potential of houseflies for the bacterium Aeromonas caviae. Medical and Veterinary Entomology. 16 (2), 193-198 (2002).
- Multistate and Nationwide Foodborne Outbreak Investigations: A Step-by-Step Guide. , CDC. Atlanta, GA. Available from: http://www.cdc.gov/foodsafety/outbreaks/investigating-outbreaks/investigations/index.html (2013).
- Sievert, K., Messler, S., Klimpel, S., Feffer, K. Comprehensive study on the occurrence and distribution of pathogenic microorganisms carried by synanthropic flies caught at different rural locations in Germany. Journal of Medical Entomology. 46 (5), 1164-1166 (2009).
- Hernández-Escareño, J. J., et al. Presence of Enterobacteriaceae, Listeria spp., Vibrio spp. and Staphylococcus spp. in house fly (Musca domestica L.), collected and macerated from different sites in contact with a few animals species. Revista Cientifica-Facultad De Ciencias Veterinarias. 22 (2), 128-134 (2012).
- Mian, L. S., Maag, H., Tacal, J. V. Isolation of Salmonella from muscoid flies at commercial animal establishments in San Bernardino county, California. Journal of Vector Ecology. 27 (1), 82-85 (2002).
- Olsen, A. R., Hammack, T. S. Isolation of Salmonella spp. from the housefly, Musca domestica L., and the dump fly, Hydrotaea aenescens (Wiedemann) (Diptera:Muscidae), at caged-layer houses. Journal of Food Protection. 63 (7), 958-960 (2000).
- Holt, P. S., Geden, C. J., Moore, R. W., Gast, R. K. Isolation of Salmonella enterica serovar Enteritidis from houseflies (Musca domestica) found in rooms containing Salmonella serovar Enteritidis-challenged hens. Applied and Environmental Microbiology. 73 (19), 6030-6035 (2007).
- Mramba, F., Broce, A., Zurek, L. Isolation of Enterobacter sakazakii from stable flies, Stomoxys calcitrans L. (Diptera:Muscidae). Journal of Food Protection. 69 (3), 671-673 (2006).
- Koyuncu, S., Andersson, M. G., Haggblom, P. Accuracy and Sensitivity of Commercial PCR-Based Methods for Detection of Salmonella enterica in Feed. Applied and Environmental Microbiology. 76 (9), 2815-2822 (2010).
- Gagné, R. Insect and mite pests in food, an illustrated. 1, U.S. Department of Agriculture and U.S. Department of Health and Human Service. 269-296 (1991).
- Greenberg, B. Flies and disease, Vol I. Ecology, classification and biotic associations. , Princeton University Press. (1971).
- Wallace, H. A., Jacobson, A., Hammack, T. S. Chapter 5 Salmonella. Bacteriological Analytical Manual. , Available from: http://www.fda.gov/downloads/Food/FoodScienceResearch/UCM244774.pdf (2007).
- Barro, N., Aly, S., Tidiane, O. C. A., Sababenedjo, T. A. Carriage of bacteria by proboscises, legs, and feces of two species of flies in street food vending sites in Ouagadougou, Burkina Faso. Journal of Food Protection. 69 (8), 2007-2010 (2006).
- Ugbogu, O. C., Nwachukwu, N. C., Ogbuagu, U. N. Isolation of Salmonella and Shigella species from house flies (Musca domestica L.) in Uturu, Nigeria. African Journal of Biotechnology. 5 (11), 1090-1091 (2006).
- Brownlie, J. C., Johnson, K. N. Symbiont-mediated protection in insect hosts. Trends in Microbiology. 17 (8), 348-354 (2009).
- Feldhaar, H. Bacterial symbionts as mediators of ecologically important traits of insect hosts. Ecological Entomology. 36 (5), 533-543 (2011).
- Foodborne disease outbreaks: Guidelines for investigation and control. , World Health Organization. Available from: http://www.who.int/foodsafety/publications/foodborne_disease/outbreak_guidelines.pdf (2008).
- Wright, A. P., Gould, L. H., Mahon, B., Sotir, M. J., Tauxe, R. V. Overview of the Impact of Epidemic-Assistance Investigations of Foodborne and Other Enteric Disease Outbreaks, 1946-2005. American Journal of Epidemiology. 174, S23-S35 (2011).



