Introduction
Myxovirus Widerstand (Mx) Proteine sind ein wichtiger Teil der angeborenen Immunabwehr gegen virale Pathogene. Diese Proteine sind groß dynamin artigen GTPasen, die durch Typ I und Typ III Interferone induziert werden. Die entsprechenden Mx Gene in fast allen Wirbeltieren vorhanden sind in einer oder mehreren Kopien und deren Genprodukte hemmen ein breites Spektrum von Viren, einschließlich Orthomyxoviridae (z. B. Influenza-Virus), Rhabdoviridae (z. B. vesikuläres Stomatitis-Virus), Bunyaviridae (z. , La Crosse Virus) und Retroviridae (zB Human Immunodeficiency Virus-1) 1-4. Es ist unklar, wie diese Proteine erkennen, wie ein breites Spektrum von Viren, ohne ersichtlichen gemeinsamen primären Sequenzmotive in dieser Viren. Analyse der Interaktion von Mx-Proteine mit den viralen Ziele, die möglicherweise mit höherer Ordnung Komplexe mit anderen Wirtszellfaktoren, wird dazu beitragen, die molekularen Mechanismen zu verstehen that in der Rüstungswettlauf zwischen Viren und ihren Wirten entwickelt.
Das Zusammenspiel von Säugetier Mx Proteine und virale Ziele wurde sehr ausgiebig für den menschlichen M · A untersucht. Menschliche MxA können die Replikation vieler Viren, einschließlich der Orthomyxoviren Influenza A und Thogoto Virus zu hemmen. M · A bindet die Thogoto Virus Ribonukleoproteinkomplexe (vRNPs) und damit ihre Kern Eintrag, der in Block 5 der Infektion führt zu verhindern. Diese Wechselwirkung zwischen MxA und Thogoto Virus vRNPs wurde mit Co-Sedimentation und Co-Immunopräzipitation 6-9 gezeigt. Wie Mx-Proteine hemmen Influenza-A-Viren ist weniger klar. Ein Hauptproblem ist, dass es nicht einfach ist, eine Wechselwirkung zwischen einem Mx-Protein und einem Influenza-Genprodukt zu demonstrieren. Ein Bericht zeigte eine Interaktion zwischen Mensch M · A und der NP-Protein in Influenza A-Virus infizierten Zellen 10. Diese Wechselwirkung kann nur durch Zusammen immunopr gezeigtecipitation wenn die Zellen mit dem Vernetzungsmittel Dithiobis (succinimidylpropionat) vor der Lyse behandelt wurden, was darauf hindeutet, dass die Wechselwirkung transiente und / oder schwach. Neuere Studien haben gezeigt, daß der Differenz Mx Empfindlichkeit verschiedener Influenza-A-Stämme wird durch die Entstehung des NP-Proteins 11,12 bestimmt. Im Einklang mit dieser, können Influenza-A-Viren zum Teil aus Mx Kontrolle entziehen durch Mutation spezifische Reste in der NP-Protein 13. Dies deutet darauf hin, dass das Hauptziel des Influenza-A-Viren, die für Wirts Mx das NP-Protein, in vRNP Komplexe wahrscheinlich NP montiert. Jedoch keines dieser neuere Studien zeigten eine Interaktion zwischen Influenza NP oder vRNPs und entweder menschlichen oder Maus MxA Mx1.
Kürzlich haben wir gezeigt, zum ersten Mal, eine Wechselwirkung zwischen dem Influenza NP und der Maus Mx1-Protein mit einer optimierten Zusammen Immunpräzipitationsprotokoll 14, die hier im Detail beschrieben wird. Im allgemeinen co-immunoprecipitation ist einer der am häufigsten verwendeten biochemischen Ansätzen zur Protein-Protein-Wechselwirkungen zu untersuchen. Diese Technik wird oft als Alternative Techniken, beispielsweise Hefe-Zwei-Hybrid bevorzugt, da sie es ermöglicht, Protein-Protein-Wechselwirkungen in ihrer natürlichen Umgebung zu untersuchen. Co-Immunfällung kann endogen exprimierten Proteine durchgeführt werden, wenn Antikörper gegen die Proteine von Interesse zur Verfügung. Alternativ können die Proteine von Interesse in die Zelle durch Transfektion oder Infektion exprimiert werden, und ein Affinitäts-Tag verwendet werden. Zusätzlich zu den oben genannten Vorteilen hat die beschriebene Coimmunpräzipitation Protokoll ermöglicht die Detektion von schwachen und / oder transienten Protein-Interaktionen. Der Hauptbestandteil in dieser optimierten Protokolls ist die Zugabe von N-Ethylmaleimid (NEM) in der Zell-Lyse-Puffer. NEM ist ein Alkylierungsmittel, das mit freien Thiolgruppen wie gegenwärtig Cysteine reagiert, bei einem pH von 6,5-7,5, um eine stabile Thio-Ester zu bilden(Abbildung 1). Bei höheren pH kann NEM auch mit Aminogruppen reagieren oder Hydrolyse 15 zu unterziehen. NEM wird typischerweise verwendet, um freie Thiolgruppen zu blockieren, um die Bildung einer Disulfidbindung zu verhindern oder zu hemmen, die enzymatische Aktivität. Zum Beispiel wird häufig verwendet, um NEM desumoylating Enzyme, die Cystein-Proteasen sind blockieren. In der beschriebenen Co-Immunpräzipitationsprotokoll wurde NEM anfänglich in dem Lysepuffer enthalten, da es war berichtet worden, dass die Sumoylierung von Influenzaproteine können die Wechselwirkungen zwischen viralen Proteinen 16 beeinflussen. Unerwarteterweise ist der Zusatz von NEM erwiesen Taste, um die Wechselwirkung zwischen Influenza NP und Maus Mx1 durch Co-Immunpräzipitation dokumentiert sein. Es ist unklar, warum die Zugabe von NEM ist von entscheidender Bedeutung, um die NP-Mx1 Interaktion zu erkennen. Möglicherweise ist die Interaktion zu vorübergehenden und / oder schwach. NEM könnte die Interaktion, beispielsweise zu stabilisieren, unter Wahrung einer bestimmten Konformation Mx1, ein virales Protein oder sogar eine unbekannte dritte Komponente. Eine derartige stabilisierende Wirkung von NEM ist zuvor beobachtet worden, beispielsweise für das Zusammenwirken zwischen der Ribonukleotidreduktase M1 und sein Inhibitor Gemcitabin (F2dC) 17. Mx1 und NP beide enthalten mehrere Cystein-Reste, die durch NEM modifiziert werden könnte. Zum Beispiel wird eine aktuelle Studie von Rennie et al., Dass ein stielfrei M · A-Variante enthält drei Lösungsmittel ausgesetzt Cysteinreste, die von Iodacetamid geändert werden können. Mutagenierten Reste Serine beeinflusste nicht die enzymatische Aktivität des MxA, aber verhindert disulfidvermittelte Aggregation 18. Da diese Cysteine in Mx1 erhalten, bedeutet dies, dass die analogen Cysteine in Mx1 durch NEM und als solche beeinflussen dessen Konformation oder Löslichkeit modifiziert werden. Außerdem könnte NEM beeinflussen auch die GTPase-Aktivität von Mx1, die für die Anti-Influenza-Aktivität Mx1 ist, und dadurch die Wechselwirkung zwischen Mx1 und NP stabilisieren. Allerdings ist eine direkte Wirkung von NEM auf der GTPase activität der Mx1 ist unwahrscheinlich, da NEM ist auch erforderlich, um die Interaktion zwischen Influenza NP und GTPase inaktive Mutanten des Mx1 Protein 14 zu erkennen. Es ist klar, ist mehr Forschung notwendig, um die Wirkung von NEM auf der NP-Mx1 Interaktion zu entwirren.
Zusammenfassend ermöglicht die beschriebene Coimmunpräzipitation Protokoll, um die Wechselwirkung zwischen der antiviralen Mx1-Protein und seine virales Ziel, das Influenza-NP-Protein zu untersuchen. Dieses Protokoll kann auch verwendet werden, um andere schwache bzw. transiente Interaktionen, die auf die Stabilisierung der spezifischen Proteinzustände hängen untersuchen. Protein-Protein-Wechselwirkung, die auf spezifische Konformationen abhängig sind zuvor beschrieben worden, beispielsweise Calcium-bindende Proteine wie Calmodulin 19. Schließlich könnte die vorteilhafte Rolle der NEM auch andere Methoden, die Protein-Protein-Wechselwirkungen, wie Ko-Sedimentation Assays verwendet werden.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Hinweis: Die nach der Transfektion und Co-Immunopräzipitation Protokoll wird für einen 9 cm Petrischale Format etabliert. Andere Formate sind auch nach der Skalierung des Protokolls möglich.
1. Seeding die humane embryonale Nierenzellen (HEK) 293T-Zellen
- Samen der HEK293T Zellen einen Tag vor der Transfektion mit 1,2 x 10 6 Zellen pro 9 cm Petrischale in 12 ml Dulbeccos modifiziertem Eagle-Medium (DMEM) mit 10% fötalem Kälberserum, 2 mM L-Glutamin, 0,4 mM Natriumpyruvat ergänzt, 0,1 mM nicht-essentiellen Aminosäuren, 100 U / ml Penicillin und 0,1 mg / ml Streptomycin.
- Wachsen die Zellen 16 Stunden bei 37 ° C und 5% CO 2.
- Sichtprüfung der Morphologie und der Lebensfähigkeit der Zellen mit einem umgekehrten Lichtmikroskop vor der Transfektion. Die Zellen müssen subkonfluenten für optimale Transfektionseffizienz sein.
2. Calciumphosphat-Transfektion von HEK293T Zellen
Notiz: Verwenden 0,5-1 ug pCAXL-NP oder leer pCAXL Plasmid in Kombination mit 1-3 ug pCAXL-Mx1 pro 9 cm Schale. Verwenden eine gleiche Menge an Gesamt Plasmid DNA in allen Proben; einstellen mit leeren Plasmid, wenn nötig.
- Bereiten Sie die folgenden Transfektion Puffer:
- Bereiten Tris-EDTA (TE) mit Konzentrationen von 1,0 mM Tris-HCl pH 8,0 und 0,1 mM EDTA pH 8,0.
- Bereiten BS / HEPES mit Konzentrationen von 25 mM HEPES (5,96 g / L, 4- (2-Hydroxyethyl) -1-piperazinethansulfonsäure), 274 mM NaCl (16 g / L), 10 mM KCl (0,74 g / L), 1,5 mM NaHPO 4 · 12H 2 O (0,5 g / l) und 11,1 mM Dextrose (2 g / L). PH-Wert auf 7,05.
- Bereiten CaCl 2 / HEPES mit Konzentrationen von 1,25 M CaCl 2 · 2H 2 O (183,8 g / l) und 125 mM HEPES (29,79 g / l). PH-Wert auf 7,05 mit NaOH.
- Warm die Transfektion Puffer bei 37 ° C vor dem Gebrauch.
- Bereiten Sie die Plasmid Proben hergestellt durch Verdünnen der Plasmid-DNA in 600 ul TE.Diese Mischungen sind in Vertiefungen einer Platte mit 6 Vertiefungen.
- Fügen Sie 150 ul CaCl 2 / HEPES in einer tropfenweise Art und Weise an die Plasmid-Proben und mischen durch Pipettieren 3 mal nach oben und unten.
- Bereiten Sie die Transfektionslösung von tropfenweiser Zugabe der Plasmid-Lösung (TE + DNA + CaCl 2 / HEPES, 750 ul) zu 750 ul der BS / HEPES-Puffer in einer frischen Platte mit 6 Vertiefungen vorgesehen sind. Verteilen Sie die Plasmid-Lösung gleichmäßig über die gesamte Vertiefung, die BS / HEPES-Puffer.
- Schüttle die Transfektionslösung auf einem Plattenschüttler für 90 sec bei 1.000 UpM.
- Inkubieren der Mischung für 5 min bei Raumtemperatur.
- Fügen Sie den Transfektionslösung (1,5 ml) tropfenweise zu den Zellen. Verwenden Sie einen P1000 Mikropipette, die Transfektionslösung auf die Zellen tropfen. Zerstreuen Sie die Mischung über den gesamten 9 cm Petrischale und schütteln Sie die Platte ganz sanft.
- Die Zellen für 6 Stunden bei 37 ° C und 5% CO 2. Dann entfernen Sie das Medium durch Absaugen und wi sofort zu ersetzenth 12 ml frisch, vorgewärmte Medium. Die frisches Medium zu den Zellen hinzu sanft auf Zellablösung zu verhindern. Dazu halten Sie die Spitze der Pipette an der Seite des Brunnens und drücken Sie sie vorsichtig aus dem Medium.
- Die Zellen für weitere 16 h bei 37 ° C und 5% CO 2.
3. Co-Immunpräzipitation
Hinweis: Führen Sie die Co-Immunopräzipitation 24 Stunden nach der Transfektion.
- Vorbereitung des geringen Salz Lysepuffer und hohen Salzwaschpuffer.
- Wird eine Stammlösung von 2 M N-Ethylmaleimid (NEM) durch Wiegen der Menge an NEM und in absolutem Ethanol gelöst wird. Bereiten Sie die NEM-Stammlösung frisch vor Gebrauch.
ACHTUNG: NEM ist sehr giftig, vorzubereiten und diese Stammlösung in einem Abzug. - Bereiten niedrigen Salz Lysepuffer in einer Konzentration von 50 mM Tris-HCl pH 8, 150 mM NaCl, 5 mM Ethylendiamintetraessigsäure (EDTA), 1% NP40 und eine Protease-Inhibitor-Cocktail (aufzulösen 1 Tabellet in 50 ml Lyse-Puffer). Hinzufügen NEM bis zu einer Endkonzentration von 25 mM (dh 1:80 verdünnt). Halten Sie auf dem Eis nach der Zugabe der Protease-Inhibitoren und NEM.
Hinweis: Achten Sie stets darauf, die Protease-Inhibitoren und NEM vor Gebrauch frisch. - Vorbereiten einer Hochsalzwaschpuffer in Konzentrationen von 50 mM Tris-HCl pH 8, 500 mM NaCl, 5 mM EDTA und 1% NP40. Beachten Sie, dass der hohe Salzwaschpuffer nicht NEM enthalten.
- Wird eine Stammlösung von 2 M N-Ethylmaleimid (NEM) durch Wiegen der Menge an NEM und in absolutem Ethanol gelöst wird. Bereiten Sie die NEM-Stammlösung frisch vor Gebrauch.
- Herstellung von Zelllysaten.
- Entfernen des Mediums und Waschen der Zellen mit 2 ml eiskalter phosphatgepufferter Kochsalzlösung (PBS). Sehr sanft fügen Sie den Waschpuffer, wie HEK293T-Zellen lösen leicht.
- Entfernen Sie die PBS und fügen Sie 600 ul eiskaltem salzarme Lysepuffer pro 9 cm Petrischale.
- Die Inkubation erfolgt während 20 Minuten auf Eis. Stellen Sie sicher, daß die Platten horizontal gehalten, um eine vollständige Bedeckung der Plattenoberfläche mit Lysepuffer gewährleisten. Schütteln Sie die Platten alle 5 min.
- Sammeln des Zelllysats in einem 1,5 ml MikrozentrifugenRohr und Zentrifuge für 3 Minuten bei 4 ° C und 16.000 × g, um die unlösliche Fraktion zu pelletieren.
- Übertragen Sie die lösliche Fraktion, dh dem Zelllysat in ein frisches 1,5 ml Mikrozentrifugenröhrchen und auf Eis halten. Unmittelbar mit dem Co-Immunpräzipitationsprotokoll fortzusetzen, um eine Dissoziation der interagierenden Proteine zu verhindern. Führen alle folgenden Schritte so weit wie möglich auf Eis oder bei 4 ° C, um die proteolytische Aktivität in den Lysaten zu begrenzen.
- Erzeugung von Immunkomplexen.
Anmerkung: In diesem Schritt wird das Protein von Interesse mit dem entsprechenden Antikörper gebunden ist. Um die NP-Mx1 Interaktion zu studieren, mit einem Maus-Anti-NP monoklonaler Antikörper.- Für jede Probe mischen 135 ul Lysat mit 2 & mgr; l Anti-NP-monoklonalen Antikörper und 113 ul niedrigen Salz Lysepuffer (Gesamtvolumen 250 ul). Den übrigen Lysat bei -20ºC für eine weitere Analyse, wie Western-Blotting, um die Expressionsniveaus des vermuteten intera dokumentierenRésol Partner in den transfizierten Zellen.
Anmerkung: Alternativ, die Proteinkonzentration des Lysats (zB mit Bradford-Reagens) und mit einem Betrag von Gesamtprotein, beispielsweise 400 & mgr; g für jeden Lysats. - Inkubieren des Antikörper-Lysat Mischung für 3 Stunden auf einem sich drehenden Rad bei 4 ° C. Dieser Schritt kann auf eine Inkubation über Nacht verlängert werden.
- Für jede Probe mischen 135 ul Lysat mit 2 & mgr; l Anti-NP-monoklonalen Antikörper und 113 ul niedrigen Salz Lysepuffer (Gesamtvolumen 250 ul). Den übrigen Lysat bei -20ºC für eine weitere Analyse, wie Western-Blotting, um die Expressionsniveaus des vermuteten intera dokumentierenRésol Partner in den transfizierten Zellen.
- Vorbereitung der Protein G-Kügelchen.
Anmerkung: Die Protein-G-Kügelchen werden versandt und in 20% Ethanol zur Konservierung gespeichert. Die Wulst-Slurry besteht normalerweise aus 50% Perlen und diese Perlen gewaschen werden müssen, bevor sie verwendet werden, um die Immunkomplexen immunpräzipitieren.- Benutzen 50 ul Perlen, also 100 ul Kügelchen-Aufschlämmung, für jede Probe. Die Menge der Kügelchen für alle Proben in der Co-Immunpräzipitationstest in einem Rohr erforderlich waschen. Schneiden Sie die Spitze eines 1 ml Pipettenspitze zu Pipetten der Wulst-Slurry zu erleichtern.
- Zentrifugieren Sie die Protein G Kügelchen-Aufschlämmung bei 8.000 xg und 4 ° C für 30 sec. Entfernen Sie die Ethanollösung und das gleiche Volumen des geringen Salz Lysepuffer. Zentrifugieren Sie die Protein G Kügelchen-Aufschlämmung bei 8.000 xg und 4 ° C für 30 Sekunden und entfernen Sie den Überstand vorsichtig. Wiederholen Sie diesen Waschschritt 3 mal.
Hinweis: Die salzarme Lysepuffer verwendet, um die Perlen zu waschen muss nicht Protease-Inhibitoren oder NEM enthalten. - Schätzen Sie das Volumen von Protein G-Kügelchen, und fügen Sie ein gleiches Volumen geringer Salz Lysepuffer, eine neue 50% Perlen Schlamm in geringen Salz Lysepuffer zu machen.
- Für jede Probe wurden dann 100 ul der Wulst-Slurry in einem frischen 1,5-ml-Mikrozentrifugenröhrchen und Lagerung auf Eis bis zum Gebrauch. Sei vorsichtig, um die Wulst-Aufschlämmung vor dem Teilen zu resuspendieren, da diese Perlen schneller sedimentieren am Boden des Rohrs.
- Immunpräzipitation der Immunkomplexen durch Protein G-Kügelchen und ihre Elution.
- Vor der Verwendung der Protein G-Kügelchen für die Immunpräzipitation, Zentrifugieren Sie alle Röhrchen 30 s bei 8.000 xg und 4 ° C undDurch visuelle Überprüfung, dass es eine gleiche Menge an Perlen in allen Proben vorhanden. Falls erforderlich die Menge an Perlen in einigen der Proben und Zentrifuge wieder. Die Überstände verwerfen. Achten Sie darauf, um die pelletierten Protein G-Kügelchen zu stören.
- Kurz zentrifugieren Immunkomplexen (dh., Lysaten mit Antikörper, 250 ul) für 30 sec bei 8.000 x g und 4 ° C, um die gesamte Probe zu dem Boden des Röhrchens zu sammeln. Übertragen Sie die Immunkomplexen auf die Protein G-Kügelchen (50 ul).
- Inkubiere 60 min auf einem sich drehenden Rad bei 4 ° C. Inkubieren Sie nicht diese Immunkomplexe länger als 75 min mit den Perlen, um unspezifische Bindung von Proteinen an die Protein-G-Perlen zu verringern.
- Zentrifugieren Protein G-Kügelchen (mit gebundenen Immunkomplexe) für 30 sec bei 8.000 x g und 4 ° C und Entfernen der Überstände. Achten Sie darauf, um die pelletierten Protein G-Kügelchen zu stören. Optional: Speichern dieses Überstände bei 4 ° C oder -20 ° C für eine spätere Analyse,beispielsweise um die Menge an ungebundenen Proteins abzuschätzen.
- Für etwa 5 min mit 900 ul Hochsalz Lysepuffer waschen die Protein G-Kügelchen. Stellen Sie sicher, dass die Beads vollständig im Waschpuffer für optimale Wasch suspendiert. Zentrifugieren Sie die Protein G-Kügelchen für 30 Sekunden bei 8.000 xg und 4 ° C und entsorgen Sie die Überstände. Wiederholen Sie diesen Waschschritt 4 mal. Achten Sie darauf, um die pelletierten Protein G-Kügelchen zu stören, um den Verlust von immunpräzipitierten Material vermeiden.
- Nach dem letzten Waschschritt werden 50 ul 2x Laemmli Probenpuffer zu den Kügelchen und erhitzt die Suspension 10 min bei 95 ° C, um die (Co) immunpräzipitierten Proteine zu eluieren.
- Herstellung von 10 ml 6x Laemmli-Puffer mit 1 g Natriumdodecylsulfat, 3,5 ml Glycerin, 3,5 ml 1 M Tris-HCl pH 6,8 und 420 & mgr; l β-Mercaptoethanol. Einzustellen, um ein Gesamtvolumen von 10 ml durch Zugabe von destilliertem Wasser. Verdünnen Sie 3-mal in destilliertem Wasser auf 2x Laemmli-Puffer zu erhalten.
ACHTUNG: &# 946; -Mercaptoethanol giftig ist, herzustellen und zu verwenden Laemmli-Puffer in einem Abzug.
- Herstellung von 10 ml 6x Laemmli-Puffer mit 1 g Natriumdodecylsulfat, 3,5 ml Glycerin, 3,5 ml 1 M Tris-HCl pH 6,8 und 420 & mgr; l β-Mercaptoethanol. Einzustellen, um ein Gesamtvolumen von 10 ml durch Zugabe von destilliertem Wasser. Verdünnen Sie 3-mal in destilliertem Wasser auf 2x Laemmli-Puffer zu erhalten.
- Nach dem Erhitzen Zentrifuge die Protein G-Kügelchen für 30 Sekunden bei 8.000 xg und die Proben bei 4 ° C (kurzfristig) oder -20 ° C (langfristig) zu speichern.
4. Analysieren Sie den (Mit-) immunpräzipitierten Proteine
- Visualisierung der in dem Zelllysat und Eluat der Co-Immunopräzipitation von SDS-PAGE und Western-Blotting 20 21,22 vorhandenen Proteine. In der Regel laden Hälfte des Laemmli Eluat auf Gel. Achten Sie darauf, um die pelletierten Protein G-Kügelchen zu stören, wenn die Entnahme von Proben für die Gelbeladung. Mx1 und NP-Expression wurden mit einem anti-Mx1 und Anti-NP-Antikörper bzw. 14 offenbart. Die Banden wurden mit HRP-basierten Chemilumineszenz und einer Röntgenfilmentwickler detektiert.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
N-Ethylmaleimid ist eine organische Verbindung, die verwendet werden können, um irreversibel ändern freien Thiolgruppen, beispielsweise zur Cysteinproteasen (Figur 1) zu hemmen.
Die antivirale Mx1-Protein hemmt die Influenza-A-Virus-Replikation durch die Interaktion mit dem viralen Nukleoprotein. Die hier beschriebene optimierte Co-Immunopräzipitation-Protokoll ermöglicht, diese NP-Mx1 Interaktion zu studieren. HEK293T-Zellen wurden mit Expressionsvektoren für die antivirale Mx1-Protein in Abwesenheit oder Gegenwart des Influenza-NP-Protein transfiziert. Als nächstes wird das NP-Protein war heruntergezogen von Gesamtzell-Lysaten mit einem NP-spezifischen monoklonalen Antikörpers, Fig. 2 zeigt, daß die Mx1-Protein ist nur co-immunpräzipitiert in Gegenwart coexprimiert NP. Mögliche unspezifische Co-Immunpräzipitation von Mx1 in Abwesenheit von NP wird entweder durch unspezifische Pull-down des Mx1-Protein durch die Anti-NP-Antikörper oder durch unspezifische Bindung von Mx1 die verursachteProtein G-Kügelchen. Daher enthalten immer eine negative Kontrolle, diese unspezifischen Coimmunpräzipitation beurteilen. 3 zeigt, dass die NP-Mx1 Wechselwirkung kann nur in Gegenwart von NEM detektiert werden. In diesem Experiment wurde die nichtspezifische Bindung von Mx1 den Protein G-Kügelchen wurde mit einer Kontroll Co-Immunpräzipitations-Reaktion in Abwesenheit von Anti-NP-Antikörper bewertet.
Dieses Protokoll kann auch verwendet werden, um die Wechselwirkung zwischen Mx1 und das Influenza NP-Protein aus infizierten Zellen oder aus Virionen isoliert untersuchen. Für diese Anwendung wurde das obige Protokoll leicht durch Kombinieren Lysaten Mx1 exprimierenden Zellen mit Lysaten, die das virale Protein NP vor Beginn der Co-Immunpräzipitationsprotokoll angepasst. Abbildung 4 zeigt die Co-Immunpräzipitation von Mx1 mit NP isoliert aus transfizierten Zellen, infizierte Zellen oder Virionen.
Zusammenfassend zeigen diese Ergebnisse, dass diese Co-Immunopräzipitation protocol kann verwendet werden, um die Interaktion zwischen antivirales Protein und dessen virales Ziel zu untersuchen.
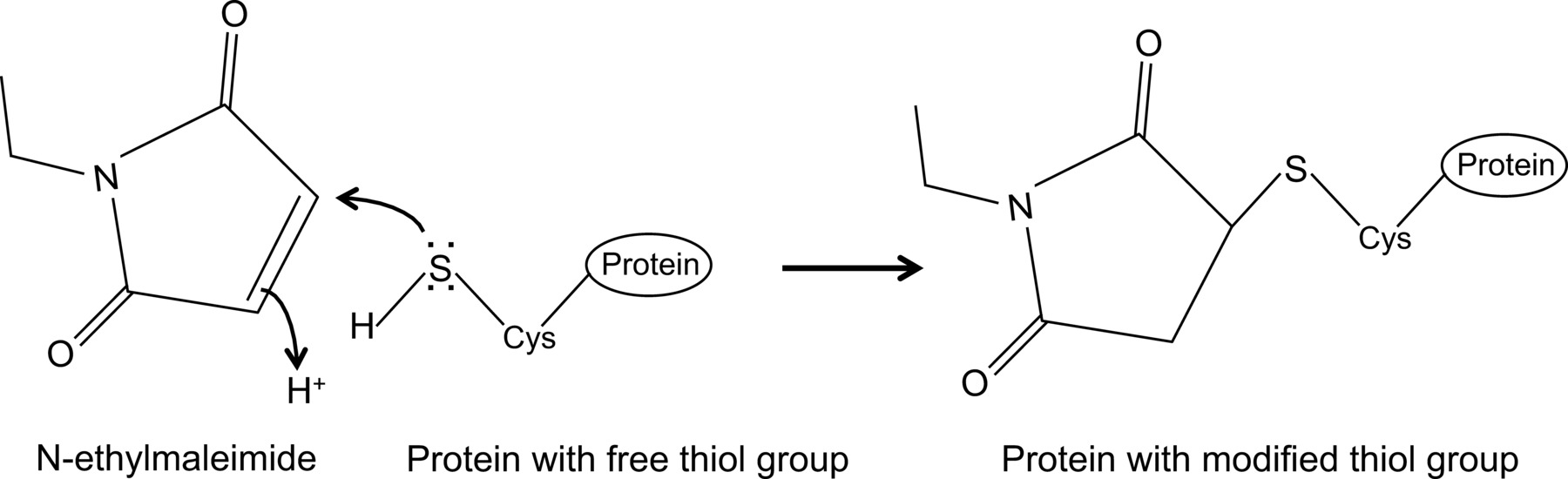
Abb. 1: Die irreversible Veränderung der freien Thiolgruppen von N-Ethylmaleimid Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.

Fig. 2: Mx1 Wechselwirkung mit NP Westernblot-Analyse von Co-Immunpräzipitations-Experiment mit zwei Proben: eines, bei dem sowohl Mx1 und NP vorhanden sind, und einen Steueraufbau in dem das NP-Protein fehlt. NP wurde mit anti-NP und NP und Mx1 immunpräzipitiert wurden mittels Western Blot sichtbar gemacht. Diese Zahl hat sich f verändert wurdeROM 14.

Abbildung 3: N-Ethylmaleimid ist wichtig zu erkennen, die NP - Mx1 Wechselwirkung Westernblot-Analyse von Co-Immunpräzipitations-Experiment, in Gegenwart oder Abwesenheit von N-Ethylmaleimid durchgeführt.. NP wurde mit anti-NP und Mx1 und NP immunpräzipitiert wurden mittels Western Blot sichtbar gemacht.

Abb. 4: ein Steuer Lysat (kein vRNPs), transfiziert vRNPs, vRNPs aus infizierten Zellen oder vRNPs von Influenza A Virionen isoliert: Mx1 interagiert mit NP isoliert aus infizierten Zellen oder Virionen Lysate enthalten Mx1 wurden mit Lysaten, die NP aus verschiedenen Quellen kombiniert . Nach dem Mischen der Lysaten co-immunopreNiederschläge mit Anti-NP wurde durchgeführt und Mx1 und NP wurden mittels Western Blot sichtbar gemacht.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Untersuchung der Wechselwirkung zwischen der antiviralen Proteine und ihre virale Ziele ist sehr wichtig, um die Details des antiviralen Mechanismus dieser Proteine zu verstehen. Dies kann neue Erkenntnisse darüber, wie Viren und ihren Wirten co-entwickelt geben und die Grundlage für die Entwicklung neuer antiviraler Strategien. Die hier beschriebene optimierte Coimmunpräzipitation Protokoll ermöglicht, die Interaktion zwischen der Maus Mx1-Protein und seine virales Ziel, das Influenza-NP-Protein zu untersuchen. Der wichtigste Aspekt dieses Protokolls ist der Zusatz von NEM in Lysepuffer, wie der NP-Mx1 Wechselwirkung ist nicht nachweisbar in der Abwesenheit von NEM (Abbildung 3). Bisher ist es nicht bekannt, warum die Anwesenheit von NEM ist wesentlich, um diese Wechselwirkung zu detektieren. Jedoch könnte dieses Protokoll nützlich, andere schwache und / oder transienten Interaktionen, die auf die Stabilisierung der spezifischen Proteinzustände hängen zu studieren, insbesondere wenn Cysteine beteiligt sind.
Einwichtige Beschränkung von Co-Immunfällungstests im allgemeinen ist das Vorhandensein von spezifischen und hochwertigen Antikörper, konformative Epitope in einer der Wechselwirkungspartner mit hoher Affinität erkennen. Die NP-Mx1 Interaktion nicht mit unseren im Haus produziert Anti-Mx1 polyklonalen Antiserums nachgewiesen werden. Dieses Antiserum immunpräzipitiert auch Influenza NP-Protein, auch in Abwesenheit von Mx1. Darüber hinaus ist die Anti-NP-monoklonaler Antikörper, der verwendet wurde, erkennt das NP-Protein des A / Puerto Rico / 8/34 Influenza-Stamm, aber leider ist es nicht geeignet, Pull-Down-NP von Virusstämme. Eine weitere Einschränkung dieses Coimmunpräzipitation Protokoll ist das nichtspezifische Bindung von Mx1 die Protein G-Kügelchen. Letztere Bindung kann durch Erhöhung der Salzkonzentration im Waschpuffer und die Verringerung der Kontaktzeit zwischen dem Lysat und dem Protein G-Kügelchen zu überwinden. Zudem ist die Verringerung der Menge an Perlen pro Immunpräzipitation verwendetReaktion von 50 ul bis 25 ul, weiter zu verringern unspezifische Bindung von Mx1 zu diesen Perlen. Im Allgemeinen ist die unbeabsichtigte Pull-down von kontaminierenden Proteinen durch nicht-spezifische Bindung an das Protein G-Kügelchen verursacht wird, kann auch durch andere Strategien reduziert werden. Beispielsweise können diese Proteine während einer vorher klare Schritt, bei dem diese Proteine durch Inkubation des Lysates mit Protein G-Kügelchen in der Abwesenheit von Antikörper entfernt entnommen werden. Die verunreinigenden Proteine werden dann zusammen mit den Kügelchen entfernt und das pre-geklärte Lysat wird für Co-Immunopräzipitation verwendet. Diese Strategie ist nur vorteilhaft, wenn die verunreinigenden Proteine anders als die Protein (e) untersucht werden. Alternativ können die unspezifischen Bindungsstellen auf dem Protein G-Kügelchen könnten mit BSA blockiert werden. Jedoch ist diese Strategie nur empfohlen, wenn die Perlen zuerst mit dem Antikörper beschichtet ist, wie BSA kann auch die Ausfällung von Immunkomplexen (dh reduzierte Antikörperbindung durch die beschichtete Kügelchen) zu reduzieren. Takenzusammen, ist es sehr wichtig, um geeignete Kontrollen enthalten, die dem Protein G-Kügelchen oder an die verwendeten Antikörper auszuschließen unspezifische Bindung der Proteine von Interesse.
Das beschriebene Co-Immunopräzipitation Protokoll modifiziert werden, um die Wechselwirkung von Mx1 mit dem NP-Protein in einer anderen Umgebung vorhanden sind, zB in infizierten Zellen oder in gereinigter vRNPs studieren. Mx1 hemmt die Expression der viralen Proteine einschließlich NP, bei Influenzavirus-Infektion 23. Daher ist es technisch extrem schwierig, die NP-Mx1 Wechselwirkung Mx1 transfizierten Zellen, die nachfolgend infiziert werden studieren. Trotzdem ist das Co-Immunpräzipitations-Protokoll auch nach dem Kombinieren Lysaten Mx1 exprimierenden Zellen und infizierten Zellen, was erfolgreiche Erfassung des NP-Mx1 Wechselwirkung (Figur 4) durchgeführt werden. Da der entscheidende Ziel von NEM nicht bekannt ist, wurde NEM während der Lyse der beiden Zellpopulationen gegeben. Falls gewünscht, kann der pH-Wertder Lysepuffer kann geändert werden. Die hier beschriebenen Versuche werden bei pH 8 (wie in Turan et al. 10) durchgeführt, aber die NP-Mx1 Co-Immunpräzipitation wurde auch erfolgreich mit einem pH 7.2 Lyse-Puffer durchgeführt. Eigentlich ein pH-Wert von 7,2 ist bevorzugt, da bei diesem pH-NEM reagiert nur mit freien Thiolgruppen und dieser pH erhöht auch die Ausbeute der Extraktion des Mx1-Protein. Darüber hinaus könnte die Protein G-Kügelchen durch Protein-A-Kügelchen ersetzt werden, abhängig von den Wirtsarten, aus denen die verwendeten Antikörper abgeleitet werden. Allerdings sind Reinigungsergebnisse mit Protein G-Kügelchen erhalten. Schließlich kann dieses Protokoll auch verwendet, um die Interaktion zwischen Mx1 und anderen Influenzaproteine, zB PB2 14 zu untersuchen. In diesem Fall wird ein V5 epitopmarkiert PB2 verwendet wurde, die im Prinzip mit im Handel erhältlichen anti-V5 Agarose Affinitätsgel kombiniert werden könnte.
In zukünftigen Experimenten könnte dieses Protokoll wertvoll für die Regionen in Mx1 identifizieren undNP, die für das NP-Mx1 Interaktion sind. Ein solcher Bereich in Mx1 könnte Schleife L4, wie diese Schleife ist gezeigt worden, kritisch für die Interaktion zwischen MxA und Thogoto Virus NP 8,9 sein. Wenn geeignete NP-spezifische Antikörper verfügbar sind, werden dieses Protokoll auch erlauben zu bestimmen, ob die erhöhte Empfindlichkeit der Vogelgrippe-A-Stämme für Mx1 korreliert mit einer stärkeren NP-Mx1 Interaktion oder nicht, die dazu beitragen könnten, den Mechanismus der Mx Empfindlichkeit zu verstehen. Darüber hinaus ist die Interaktion zwischen Mx1 und Komponenten von Viren an andere als die Orthomyxoviridae Familien angehören, können ebenfalls durch diese angepasst Coimmunpräzipitation Protokolls. Schließlich weitere Untersuchungen zur Wirkung von NEM auf dem NP-Mx1 entwirren sehr wertvoll, um diese spezifische Interaktion besser verstehen zu können, sondern auch, um einen Einblick in die breiteren Anwendungsmöglichkeiten der Zugabe dieser Verbindung während der Zelllyse und Co-Immunopräzipitation gewinnen.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen.
Acknowledgments
Diese Arbeit wurde von FWO-Vlaanderen, der IOF Projekt IOF10 / StartT / 027 und der Universität Gent Sonderforschungsstipendium BOF12 / GOA / 014 unterstützt.
Materials
| Name | Company | Catalog Number | Comments |
| DMEM high glucose | Gibco | 52100-047 | |
| N-Ethylmaleimide | Sigma | E-3876 | Toxic |
| Igepal CA-630 | Sigma | I-30212 | also known as NP40 |
| Protease inhibitor cocktail | Roche | 11 873 580 001 | |
| anti-NP monoclonal antibody | NIH Biodefense and Emerging Infections Research Resources Repository | NR-4282 | ascites blend of clones A1 and A3 |
| anti-RNP polyclonal serum | NIH Biodefense and Emerging Infections Research Resources Repository | NR-3133 | directed against A/Scotland/840/74 (H3N2) |
| Protein G Sepharose 4FF | GE Healthcare | 17-0618-01 | |
| Hyperfilm ECL 18 x 24 cm | GE Healthcare | 28-9068-36 | |
| ECL western blotting substrate | Pierce | 32106 |
References
- Verhelst, J., Hulpiau, P., Saelens, X. Mx proteins: antiviral gatekeepers that restrain the uninvited. Microbiol Mol Biol Rev. 77 (4), 551-566 (2013).
- Goujon, C., et al. Human MX2 is an interferon-induced post-entry inhibitor of HIV-1 infection. Nature. 502 (7472), 559-562 (2013).
- Kane, M., et al. MX2 is an interferon-induced inhibitor of HIV-1 infection. Nature. 502 (7472), 563-566 (2013).
- Liu, Z., et al. The interferon-inducible MxB protein inhibits HIV-1 infection. Cell Host Microbe. 14 (4), 398-410 (2013).
- Kochs, G., Haller, O. Interferon-induced human MxA GTPase blocks nuclear import of Thogoto virus nucleocapsids. Proc Natl Acad Sci U S A. 96 (5), 2082-2086 (1999).
- Flohr, F., Schneider-Schaulies, S., Haller, O., Kochs, G. The central interactive region of human MxA GTPase is involved in GTPase activation and interaction with viral target structures. FEBS Lett. 463 (1-2), 24-28 (1999).
- Kochs, G., Haller, O. GTP-bound human MxA protein interacts with the nucleocapsids of Thogoto virus (Orthomyxoviridae). J Biol Chem. 274 (7), 4370-4376 (1999).
- Mitchell, P. S., et al. Evolution-guided identification of antiviral specificity determinants in the broadly acting interferon-induced innate immunity factor MxA. Cell Host Microbe. 12 (4), 598-604 (2012).
- Patzina, C., Haller, O., Kochs, G. Structural requirements for the antiviral activity of the human MxA protein against Thogoto and influenza A virus. J Biol Chem. 289 (9), 6020-6027 (2014).
- Turan, K., et al. Nuclear MxA proteins form a complex with influenza virus NP and inhibit the transcription of the engineered influenza virus genome. Nucleic Acids Res. 32 (2), 643-652 (2004).
- Dittmann, J., et al. Influenza A virus strains differ in sensitivity to the antiviral action of Mx-GTPase. J Virol. 82 (7), 3624-3631 (2008).
- Zimmermann, P., Manz, B., Haller, O., Schwemmle, M., Kochs, G. The viral nucleoprotein determines Mx sensitivity of influenza A viruses. J Virol. 85 (16), 8133-8140 (2011).
- Manz, B., et al. Pandemic influenza A viruses escape from restriction by human MxA through adaptive mutations in the nucleoprotein. PLoS Pathog. 9 (3), e1003279 (2013).
- Verhelst, J., Parthoens, E., Schepens, B., Fiers, W., Saelens, X. Interferon-inducible protein Mx1 inhibits influenza virus by interfering with functional viral ribonucleoprotein complex assembly. J Virol. 86 (24), 13445-13455 (2012).
- Brewer, C. F., Riehm, J. P. Evidence for possible nonspecific reactions between N-ethylmaleimide and proteins. Anal Biochem. 18 (2), 248-255 (1967).
- Wu, C. Y., Jeng, K. S., Lai, M. M. The SUMOylation of matrix protein M1 modulates the assembly and morphogenesis of influenza A virus. J Virol. 85 (13), 6618-6628 (2011).
- Chen, Z., Zhou, J., Zhang, Y., Bepler, G. Modulation of the ribonucleotide reductase M1-gemcitabine interaction in vivo by N-ethylmaleimide. Biochem Biophys Res Commun. 413 (2), 383-388 (2011).
- Rennie, M. L., McKelvie, S. A., Bulloch, E. M., Kingston, R. L. Transient dimerization of human MxA promotes GTP hydrolysis, resulting in a mechanical power stroke. Structure. 22 (10), 1433-1445 (2014).
- Gifford, J. L., Walsh, M. P., Vogel, H. J. Structures and metal-ion-binding properties of the Ca2+-binding helix-loop-helix EF-hand motifs. Biochem J. 405 (2), 199-221 (2007).
- Separating Protein with SDS-PAGE. , JoVE. Available from: http://www.jove.com/science-education/5058/separating-protein-with-sds-page (2014).
- Gallagher, S., Chakavarti, D. Immunoblot analysis. J Vis Exp. (16), (2008).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Pavlovic, J., Haller, O., Staeheli, P. Human and mouse Mx proteins inhibit different steps of the influenza virus multiplication cycle. J Virol. 66 (4), 2564-2569 (1992).



