Introduction
Resistência myxovirus (Mx) proteínas são uma parte importante da defesa imune inata contra patógenos virais. Estas proteínas são grandes GTPases dinamina-like que são induzidos por tipo I e tipo III interferons. Os genes Mx correspondentes estão presentes em quase todos os vertebrados em uma ou várias cópias e dos seus produtos de genes inibir uma vasta variedade de vírus, incluindo Orthomyxoviridae (por exemplo., O vírus da gripe), Rhabdoviridae (por exemplo., O vírus da estomatite vesicular), Bunyaviridae (por exemplo. , La Crosse vírus) e Retroviridae (por exemplo, vírus da imunodeficiência humana-1) 1-4. Não está claro como estas proteínas reconhecer uma ampla gama de vírus tais, sem qualquer sequência primária compartilhada aparente motivos na estes vírus. Analisando a interação de proteínas Mx com seus alvos virais, potencialmente envolvendo complexos de ordem superior com outros fatores de célula hospedeira, irá ajudar a compreender os mecanismos moleculares tchapéu evoluíram na corrida armamentista entre os vírus e seus hospedeiros.
A interação entre as proteínas de mamíferos e Mx alvos virais tem sido estudado mais extensivamente para MxA humano. MxA humano pode inibir a replicação de muitos vírus, incluindo o vírus influenza A orthomyxovirus e vírus Thogoto. MxA liga os complexos de ribonucleoproteína vírus Thogoto (vRNPs), impedindo assim a sua entrada nuclear, o que resulta no bloqueio da infecção 5. Esta interacção entre MXA e Thogoto vRNPs vírus tem sido demonstrado com a co-sedimentação e de co-imunoprecipitação experiências 6-9. Como proteínas Mx dificultar vírus da gripe A é menos clara. Um problema principal é que não é fácil de demonstrar uma interacção entre uma proteína Mx e um produto de gene da influenza. Um relatório demonstrou uma interação entre MxA humana e da proteína NP em influenza A células infectadas pelo vírus 10. Essa interação só poderia ser demonstrado por co-immunoprecipitation se as células terem sido tratadas com as de reticulação reagentes ditiobis (propionato de succinimidilo), antes de lise, sugerindo que a interacção é transitória e / ou fraco. Estudos mais recentes têm mostrado que a sensibilidade diferencial Mx de estirpes diferentes de influenza A é determinado pela origem da proteína NP 11,12. Em linha com esta, os vírus da gripe A pode escapar do controle, em parte, Mx por mutação resíduos específicos na proteína NP 13. Isto sugere que o principal alvo dos vírus influenza para acolhimento Mx é a proteína NP, provavelmente NP montados em complexos vRNP. No entanto, nenhum desses estudos mais recentes demonstraram uma interacção entre a gripe NP ou vRNP e MxA quer humano ou de ratinho Mx1.
Recentemente, demonstrou, pela primeira vez, uma interacção entre o NP de influenza e da proteína Mx1 rato com um protocolo de co-imunoprecipitação optimizado 14, a qual é descrita aqui em detalhe. Em geral, a co-immunoprecipitation é uma das abordagens bioquímicas mais freqüentemente utilizados para investigar as interações proteína-proteína. Esta técnica é muitas vezes preferido ao longo técnicas alternativas, por exemplo, de leveduras duplamente híbrido, uma vez que permite investigar interacções proteína-proteína no seu ambiente natural. Co-imunoprecipitação pode ser levada a cabo em proteínas expressas endogenamente, se os anticorpos contra as proteínas de interesse estão disponíveis. Alternativamente, as proteínas de interesse podem ser expressos na célula através de transfecção ou infecção e um tag de afinidade pode ser usado. Além das vantagens acima mencionadas, o protocolo de co-imunoprecipitação descrita permite a detecção de interacções proteína fracos e / ou transientes. O principal componente neste protocolo optimizado é a adição de N-etilmaleimida (NEM) em tampão de lise celular. NEM é um reagente de alquilação, que reage com grupos tiol livres tais como presentes em cisteínas, a um pH de 6,5-7,5, para formar uma tio-éster estável(Figura 1). A pH mais elevado, a NEM também pode reagir com grupos amino ou sofrer hidrólise 15. NEM é normalmente utilizado para bloquear os grupos tiol livres, a fim de evitar a formação de ligações dissulfureto ou inibir a actividade enzimática. Por exemplo, a NEM é muitas vezes usado para bloquear enzimas desumoylating, que são proteases de cisteína. No protocolo de co-imunoprecipitação descrita, NEM foi inicialmente incluído no tampão de lise, porque tinha sido relatado que o sumoilação de proteínas de influenza podem influenciar a interacção entre as proteínas virais 16. Inesperadamente, a adição de NEM provou ser a chave para documentar a interacção entre NP de influenza e rato Mx1 por co-imunoprecipitação. Não está claro porque a adição de NEM é crucial para detectar a interacção NP-Mx1. Possivelmente, a interacção é muito transiente e / ou fraco. NEM poderia estabilizar a interacção, por exemplo, ao preservar uma conformação específica de Mx1, uma proteína viral, ou ainda um terceiro compo desconhecidonente. Tal efeito estabilizante de NEM foi observada antes, por exemplo, para a interacção entre a ribonucleótido-redutase M1 e o seu inibidor gemcitabina (F2dC) 17. Mx1 e NP ambos contêm múltiplos resíduos de cisteína que poderiam ser modificados por NEM. Por exemplo, um estudo recente da Rennie et al. Demonstraram que uma stalkless MxA variante contém três resíduos de cisteína expostos solventes que podem ser modificados por iodoacetamide. Mutação destes resíduos para serines não influenciou a atividade enzimática da MxA, mas impediu a agregação mediada por dissulfeto 18. À medida que estas cisteínas são conservadas em Mx1, isto sugere que os análogos em cisteínas Mx1 pode ser modificada por NEM e, como tal influência sua conformação ou solubilidade. Além disso, a NEM também pode afectar a actividade de GTPase de Mx1, que é essencial para a actividade anti-gripe de Mx1, e assim estabilizar a interacção entre Mx1 e NP. No entanto, um efeito directo de NEM na GTPase actividade de Mx1 é improvável, como NEM também é necessária para detectar a interação entre a gripe NP e GTPase mutantes inativos da proteína Mx1 14. Claramente, é necessária mais investigação para desvendar o efeito de NEM na interação NP-Mx1.
Em resumo, o protocolo de co-imunoprecipitação descrita permite estudar a interacção entre a proteína antiviral Mx1 e seu alvo viral, a proteína NP da gripe. Este protocolo poderia também ser utilizada para estudar outras interacções fracas ou transientes que dependem da estabilização de conformações de proteínas específicas. Interacção proteína-proteína que dependem conformações específicos têm sido descritos antes, por exemplo, para as proteínas de ligação ao cálcio, tais como a calmodulina 19. Finalmente, o papel benéfico de NEM também pode ser usado noutros métodos que detectam interacções proteína-proteína, tais como ensaios de co-sedimentação.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Nota: O seguinte protocolo de transfecção e co-imunoprecipitação é estabelecida para um formato de placa de Petri 9 cm. Outros formatos também são possíveis após a escala do protocolo.
1. Sementeira do rim embrionário humano (HEK) Células 293T
- Semente da células HEK293T um dia antes da transfecção a 1,2 x 10 6 culas por 9 centímetros de Petri em 12 ml de meio de Eagle modificado por Dulbecco (DMEM) suplementado com 10% de soro fetal de vitela, 2 mM de L-glutamina, 0,4 mM de Na-piruvato, 0,1 aminoácidos não essenciais mM, 100 U / ml de penicilina e 0,1 mg / ml de estreptomicina.
- Crescer as células 16 horas a 37 ° C e 5% de CO 2.
- Inspeccione visualmente a morfologia e a viabilidade das células com um microscópio de luz invertida antes da transfecção. As células necessitam de ser sub-confluente para a eficiência de transfecção óptima.
2. O cálcio-fosfato de transfecção de células HEK293T
Nota: Use 0,5-1 mg de pCAXL-NP ou vazio plasmídeo pCAXL em combinação com 1-3 mg de pCAXL-Mx1 por 9 centímetros prato. Use uma quantidade igual de DNA total plasmídeo em todas as amostras; ajustar com plasmídeo vazio, se necessário.
- Preparar os seguintes tampões de transfecção:
- Prepare de Tris-EDTA (TE) com concentrações de 1,0 mM de Tris-HCl, pH 8,0 e EDTA 0,1 mM, pH 8,0.
- Prepare BS / HEPES com concentrações de 25 mM de HEPES (5,96 g / L, 4- (2-hidroxietil) -1-piperazinoetanossulfónico), NaCl 274 mM (16 g / l), KCl 10 mM (0,74 g / L), NaHPO 4 1,5 mM · 12H 2 O (0,5 g / L) e 11,1 mM de dextrose (2 g / L). Ajustar o pH a 7,05.
- Prepare CaCl2 / com as concentrações de HEPES 1,25 M CaCl 2 2H 2 O (183,8 g / L) e tampão HEPES 125 mM (29,79 g / L). Ajustar o pH a 7,05 com NaOH.
- Aquecer os buffers de transfecção, a 37 ° C antes da utilização.
- Preparar as amostras diluindo o plasmídeo de ADN de plasmídeo em 600 ul de TE.Preparar estas misturas em poços de uma placa de 6 poços.
- Adicionar 150 mL de CaCl2 / HEPES de forma sábia soltar para as amostras de plasmídeos e misture pipetando três vezes para cima e para baixo.
- Preparar a solução de transfecção por adição gota a gota, a solução de plasmídeo (DNA TE + + CaCl 2 / HEPES; 750 ul) a 750 ul de tampão de BS / HEPES fornecidos em uma nova placa de 6 poços. Distribuir a solução de plasmídeo uniformemente sobre o poço de tampão contendo completa BS / HEPES.
- Agitar a solução de transfecção sobre um agitador de placas durante 90 segundos a 1000 RPM.
- Incubar a mistura durante 5 minutos à temperatura ambiente.
- Adicionar a solução de transfecção (1,5 ml) gota a gota às células. Usar uma micropipeta P1000 a escorrer a solução de transfecção nas células. Dispersar a mistura sobre a completa nove centímetros de Petri e agitar a placa muito suavemente.
- Incubar as células a 37 ° C e 5% de CO 2 durante 6 h. Em seguida, retire o meio por aspiração e substitua imediatamente with 12 ml de meio fresco, pré-aquecido. Adicionar cuidadosamente a meio fresco às células para evitar a separação de células. Para isto, segurar a ponta da pipeta contra o lado do poço e suavemente empurrar para fora do meio.
- Incubar as células durante mais 16 horas adicionais a 37 ° C e 5% de CO 2.
3. Co-imunoprecipitação
Nota: Execute o co-imunoprecipitação 24 horas após a transfecção.
- Preparação do tampão de baixo lise sal e tampão de lavagem de alta sal.
- Prepara-se uma solução estoque de 2 M de N-etilmaleimida (NEM) pesando a quantidade de NEM e dissolvendo-o em etanol absoluto. Preparar a solução estoque NEM fresco antes de usar.
CUIDADO: NEM é muito tóxico, preparar e usar esta solução estoque em um exaustor. - Preparar tampão de lise de sal baixo a concentrações de 50 mM Tris-HCl pH 8, 150 mM de NaCl, 5 mM de ácido etilenodiaminotetracético (EDTA), 1% de NP40 e um cocktail de inibidores de protease (1 dissolver mesat em 50 ml de tampão de lise). Adicionar NEM até uma concentração final de 25 mM (isto é, diluir 1:80). Mantenha no gelo após a adição dos inibidores da protease e NEM.
Nota: Sempre adicionar os inibidores da protease e NEM imediatamente antes da utilização. - Preparar um tampão de lavagem de alto teor salino a concentrações de 50 mM Tris-HCl pH 8, NaCl 500 mM, EDTA 5 mM e 1% de NP40. Note-se que o tampão de lavagem de alto teor salino não contém NEM.
- Prepara-se uma solução estoque de 2 M de N-etilmaleimida (NEM) pesando a quantidade de NEM e dissolvendo-o em etanol absoluto. Preparar a solução estoque NEM fresco antes de usar.
- Preparação de lisados celulares.
- Remover o meio e lava-se as células com 2 ml de fosfato de gelo frio tamponado salino (PBS). Adicionar muito suavemente o tampão de lavagem, como células HEK293T desprendem facilmente.
- Retire o PBS e adicionar 600 mL de tampão de lise sal baixo gelada por 9 centímetros de Petri.
- Incubar as placas durante 20 min em gelo. Certifique-se de que as placas são mantidas horizontal, para assegurar a cobertura completa da superfície da placa com tampão de lise. Agitar suavemente as placas a cada 5 minutos.
- Recolhe-se o lisado celular numa microcentrífuga de 1,5 mltubo e centrifuga-se durante 3 min a 4 ° C e 16.000 xg para sedimentar a fracção insolúvel.
- Transferir a fracção solúvel, isto é, o lisado de células para um novo tubo de microcentrífuga de 1,5 ml e manter em gelo. Imediatamente continuar com o protocolo de co-imunoprecipitação, para evitar a dissociação das proteínas que interagem. Executar todas as etapas seguintes, tanto quanto possível em gelo ou a 4 ° C para limitar a actividade proteolítica nos lisados.
- Geração de complexos imunes.
Nota: Neste passo, a proteína de interesse está ligado pelo anticorpo apropriado. Para estudar a interacção NP-Mx1, utilizar um anticorpo monoclonal anti-NP do rato.- Para cada amostra, misturar 135 ul de lisado com 2 ul de anticorpo monoclonal anti-NP e 113 ul de tampão de lise de sal baixo (volume total de 250 uL). Armazenar o lisado remanescente à temperatura de -20 ° C para posterior análise, tais como Western blotting, para documentar os níveis de expressão da intera supôsparceiros ction nas células transfectadas.
Nota: Em alternativa, medir a concentração de proteína do ligado (por exemplo, com o reagente de Bradford) e utilizar uma quantidade fixa de proteína total, por exemplo, 400 ug, para cada lisado. - Incubar a mistura de anticorpo-lisado durante 3 horas numa roda giratória a 4 ° C. Este passo pode ser estendido para uma incubação de um dia para o outro.
- Para cada amostra, misturar 135 ul de lisado com 2 ul de anticorpo monoclonal anti-NP e 113 ul de tampão de lise de sal baixo (volume total de 250 uL). Armazenar o lisado remanescente à temperatura de -20 ° C para posterior análise, tais como Western blotting, para documentar os níveis de expressão da intera supôsparceiros ction nas células transfectadas.
- Preparação dos grânulos de proteína G.
Nota: As esferas de proteína G são transportados e armazenados em etanol a 20% para a preservação. O grânulo-lama é normalmente constituída por 50% de partículas e estes grânulos têm de ser lavadas antes de serem usadas para imunoprecipitar os complexos imunes.- Utilizar 50 ul de pérolas, isto é, 100 ul de talão-lama, para cada amostra. Lava-se a quantidade de esferas necessárias para todas as amostras do ensaio de co-imunoprecipitação de um tubo. Corte a ponta de uma ponta de pipeta de 1 ml para facilitar a pipetagem do grânulo-lama.
- Centrifugar a proteína G talão-lama em 8.000 xg e 4 ° C durante 30 seg. Remover a solução de etanol e adicionar um volume igual de tampão de lise de baixa sal. Centrifugar a proteína G do grânulo-suspensão a 8000 xg e 4 ° C durante 30 seg e o sobrenadante remover suavemente. Repita este passo de lavagem 3 vezes.
Nota: O tampão de lise de baixa sal usado para lavar as pérolas não necessitam de conter inibidores da protease ou NEM. - Estimar o volume de proteína G pérolas e adicione um volume igual de tampão baixo lise sal para fazer um novo 50% grânulo-lama em tampão baixo lise sal.
- Para cada amostra, transferir 100 ul de talão-suspensão em um tubo de 1,5 ml fresco microcentrífuga e armazenar em gelo até à utilização. Ter cuidado para voltar a suspender o grânulo-lama antes de se dividir, como estas pérolas sedimentam rapidamente para o fundo do tubo.
- Immunoprecipitation dos complexos imunes pela proteína G pérolas e sua eluição.
- Antes de utilizar as esferas de proteína G para a imunoprecipitação, todos os tubos de centrifugação de 30 segundos a 8000 xg e 4 ° C everificar, por inspecção visual de que existe uma quantidade igual de pérolas presente em todas as amostras. Se for necessário ajustar a quantidade de grânulos em algumas das amostras e centrifugar novamente. Descartar o sobrenadante. Tenha cuidado para não perturbar as proteínas G perla peletizadas.
- Resumidamente, centrifugar os complexos imunes (isto é., Os lisados com anticorpo, 250 ul) durante 30 segundos a 8000 xg e 4 ° C para recolher a amostra completa, na parte inferior do tubo. Transferir os complexos imunes para as esferas de proteína G (50 uL).
- Incubar 60 min sobre uma roda giratória a 4 ° C. Não incubar estes complexos imunitários mais do que 75 minutos com os grânulos para reduzir a ligação não específica de proteínas para as esferas de proteína G.
- Centrifuga-se as esferas de proteína G (com complexos imunes ligados) durante 30 segundos a 8000 xg e 4 ° C e remove-se os sobrenadantes. Tenha cuidado para não perturbar as proteínas G perla peletizadas. Opcional: armazenar estes sobrenadantes a 4 ° C ou -20 ° C para posterior análise,por exemplo, para estimar a quantidade de proteína não ligada.
- Lavam-se as esferas de proteína G para aproximadamente 5 min com 900 ul de tampão de lise de sal elevado. Certifique-se de que as pérolas são completamente ressuspenso no tampão de lavagem para a lavagem óptima. Centrifuga-se as esferas de proteína G durante 30 segundos a 8000 xg e 4 ° C e desprezar o sobrenadante. Repita este passo de lavagem 4 vezes. Tenha cuidado para não perturbar as esferas de proteína G peletizadas para evitar a perda de material imunoprecipitado.
- Após o último passo de lavagem, adicionar 50 ul de tampão de amostra Laemmli 2x para os grânulos e aquecer a suspensão durante 10 min a 95 ° C para eluir as (co) proteínas imunoprecipitadas.
- Preparar 10 ml de tampão de Laemmli 6x com 1 g de dodecil sulfato de sódio, 3,5 ml de glicerol, 3,5 ml de 1 M de Tris-HCl a pH 6,8 e 420 ul de β-mercaptoetanol. Ajustar para um volume total de 10 ml por adição de água destilada. Dilui-se 3 vezes em água destilada para obter tampão Laemmli 2x.
CUIDADO: &# 946; mercaptoetanol é tóxico, preparar e usar tampão Laemmli em um exaustor.
- Preparar 10 ml de tampão de Laemmli 6x com 1 g de dodecil sulfato de sódio, 3,5 ml de glicerol, 3,5 ml de 1 M de Tris-HCl a pH 6,8 e 420 ul de β-mercaptoetanol. Ajustar para um volume total de 10 ml por adição de água destilada. Dilui-se 3 vezes em água destilada para obter tampão Laemmli 2x.
- Após o aquecimento, centrifugar os grânulos de proteína G durante 30 segundos a 8000 xg e armazenar as amostras a 4 ° C (a curto prazo) ou -20 ° C (a longo prazo).
4. Analisar o (co-) imunoprecipitou Proteínas
- Visualizar as proteínas presentes no ligado celular e o eluato de co-imunoprecipitação por SDS-PAGE e western blotting 20 21,22. Tipicamente carregar metade do eluato em gel de Laemmli. Tenha cuidado para não perturbar as esferas proteína G peletizadas quando recolha de amostras para carregamento de gel. Mx1 e expressão NP foram revelados com um anti-Mx1 e anticorpo anti-NP, respectivamente 14. As bandas foram detectadas com base quimioluminescência-HRP e um programador de película de raios-X.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
N-etilmaleimida é um composto orgânico que pode ser utilizado para modificar irreversivelmente grupos tiol livres, por exemplo, para inibir as proteases de cisteína (Figura 1).
A proteína antiviral Mx1 inibe a replicação do vírus da gripe A, interagindo com a nucleoproteína virai. O protocolo de co-imunoprecipitação otimizado aqui descrito permite estudar essa interação NP-Mx1. Células HEK293T foram transfectadas com vectores de expressão para a proteína Mx1 antiviral na ausência ou presença da proteína NP da gripe. Em seguida, a proteína NP foi puxada para baixo a partir de lisados de células totais com um anticorpo monoclonal específico para NP. A Figura 2 mostra que a proteína Mx1 é apenas co-imunoprecipitada na presença de co-expressa NP. Possível co-imunoprecipitação inespecífica de Mx1 na ausência de NP é causado por qualquer inespecífica suspenso da proteína Mx1 pelo anticorpo anti-NP ou por ligação não específica de Mx1 para ogrânulos de proteína G. Portanto, sempre incluir um controlo negativo para avaliar essa co-imunoprecipitação não específica. A Figura 3 mostra que a interacção NP-Mx1 só pode ser detectada na presença de NEM. Nesta experiência, a ligação não específica de Mx1 para as esferas de proteína G foi avaliada com uma reacção de controlo de co-imunoprecipitação, na ausência de anticorpo anti-NP.
Este protocolo pode também ser utilizado para estudar a interacção entre a proteína e Mx1 NP da gripe isolados a partir de células infectadas ou de viriões. Para esta aplicação, o protocolo anterior foi ligeiramente adaptado combinando os lisados de células que expressam Mx1 com lisados contendo a proteína viral NP antes do início do protocolo de co-imunoprecipitação. A Figura 4 mostra a co-imunoprecipitação de Mx1 com NP isolado a partir de células transfectadas, células infectadas ou virions.
Em conclusão, estes resultados mostram que este prot co-imunoprecipitaçãoOCOL pode ser utilizado para estudar a interacção entre uma proteína anti-viral e o seu alvo viral.
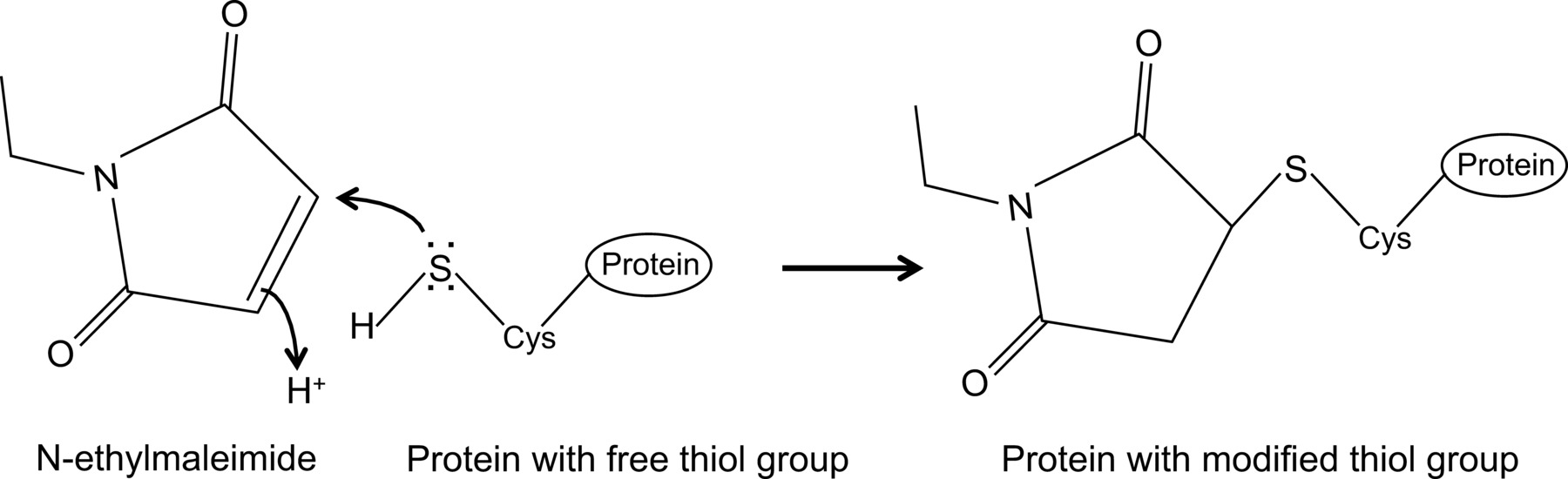
Figura 1:. Alteração irreversível de grupos tiol livres por N-ethylmaleimide Por favor, clique aqui para ver uma versão maior desta figura.

Análise Mx1 interage com NP Western blot de uma experiência de co-imunoprecipitação com duas amostras:: Figura 2. Um em que tanto Mx1 e NP estão presentes e uma instalação de controlo, em que a proteína NP está ausente. NP foi imunoprecipitada com anticorpos anti-NP e NP e Mx1 foram visualizados por Western blotting. Esta figura foi modificado from 14.

Figura 3: N-etilmaleimida é importante para detectar a NP - interacção Mx1 análise de Western blot de uma experiência de co-imunoprecipitação realizado na presença ou ausência de N-etilmaleimida.. NP foi imunoprecipitada com anticorpos anti-NP e NP Mx1 e foram visualizados por Western blotting.

Figura 4:. Mx1 interage com NP isolado a partir de células infectadas ou lisados de viriões contendo Mx1 foram combinadas com lisados contendo NP a partir de diferentes fontes: um lisado de controlo (sem vRNPs), transfectadas vRNPs, vRNPs partir de células infectadas ou vRNPs isoladas a partir de viriões A da gripe . Depois de se misturar os lisados, co-immunoprecipitation com anti-NP foi realizado e Mx1 e NP foram visualizados por Western blotting.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
O estudo da interacção entre proteínas antivirais e seus alvos virais é muito importante para compreender os detalhes do mecanismo antiviral destas proteínas. Isso pode dar novos insights sobre como os vírus e seus hospedeiros co-evoluído e ser a base para o desenvolvimento de novas estratégias antivirais. O protocolo de co-imunoprecipitação optimizado descrito aqui permite o estudo da interacção entre a proteína de rato e a sua Mx1 alvo viral, a proteína NP da gripe. O aspecto mais importante deste protocolo é a adição de NEM em tampão de lise, como a interacção NP-Mx1 não é detectável na ausência de NEM (Figura 3). Até à data, não se sabe por que a presença de NEM é essencial para detectar esta interacção. No entanto, este protocolo pode ser útil para estudar outras interacções fracas e / ou transientes, que dependem da estabilização de conformações de proteínas específicas, especialmente se as cisteínas estão envolvidas.
Umlimitação importante de ensaios de co-imunoprecipitação, em geral, é a disponibilidade de anticorpos específicos e de alta qualidade que reconhecem epítopos conformacionais de um dos parceiros de interacção com elevada afinidade. A interação NP-Mx1 não pôde ser demonstrado com o nosso em casa produzido anti-soro policlonal anti-Mx1. Este anti-soro também imunoprecipita a proteína NP de influenza, mesmo na ausência de Mx1. Além disso, o anticorpo monoclonal anti-NP, que foi usado, reconhece a proteína NP do conversor A / Puerto Rico / 8/34 da gripe estirpe, mas, infelizmente, não é adequado para puxar para baixo NP de estirpes de vírus da gripe aviária. Outra limitação deste protocolo de co-imunoprecipitação é a ligação não específica de Mx1 à proteína G perla. A última ligação pode ser superada pelo aumento da concentração de sal no tampão de lavagem e reduzir o tempo de contacto entre o lisado e a proteína G grânulos. Além disso, a redução da quantidade de grânulos usados para cada imunoprecipitaçãoreacção de 50 ul e 25 ul, pode diminuir ainda mais a ligação não específica de Mx1 para estes grânulos. Em geral, a não intencional suspenso de proteínas contaminantes, causada por ligação não específica à proteína G grânulos, pode também ser reduzida por outras estratégias. Por exemplo, estas proteínas pode ser removido durante um passo de pré-clara, em que estas proteínas são removidos por incubação do ligado com pérolas de proteína G na ausência de anticorpo. As proteínas contaminantes são removidos juntamente com os grânulos e o lisado pré-limpo é utilizado para co-imunoprecipitação. Esta estratégia só é vantajoso se as proteínas contaminantes são diferentes da proteína (s) em estudo. Alternativamente, os locais de ligação não específicos na proteína G grânulos poderiam ser bloqueados com BSA. No entanto, esta estratégia só é recomendado se os grânulos são primeiro revestidas com o anticorpo, como BSA, também pode reduzir a precipitação de complexos imunes (isto é, ligação de anticorpo reduzido pelas-grânulos revestidos). Tomadoem conjunto, é muito importante incluir controlos adequados para excluir ligação não específica das proteínas de interesse para as esferas de proteína G ou com os anticorpos utilizados.
O protocolo de co-imunoprecipitação descrito pode ser modificado para estudar a interacção de Mx1 com a proteína NP presentes num ambiente diferente, por exemplo, em células infectadas ou em vRNPs purificadas. MX1 inibe a expressão de proteínas virais, incluindo NP, durante a infecção do vírus da gripe 23. Portanto, é tecnicamente extremamente difícil de estudar a interacção de NP-Mx1 em células Mx1 transfectadas que são subsequentemente infectados. Ainda assim, este protocolo de co-imunoprecipitação, também pode ser realizada depois de combinar os lisados de células que expressam Mx1 e células infectadas, permitindo a detecção da interacção bem sucedida NP-Mx1 (Figura 4). Dado que o objectivo fundamental de NEM não é conhecido, a NEM foi adicionada durante a lise de ambas as populações de células. Se desejado, o pHo tampão de lise pode ser alterado. As experiências aqui descritas são realizadas a pH 8 (como no Turan et al. 10), mas a co-imunoprecipitação de NP-Mx1 também foi realizada com sucesso com um tampão de lise, pH 7,2. Na verdade, um pH de 7,2 é preferido, porque neste NEM pH reage exclusivamente com grupos tiol livres e este valor de pH também aumenta o rendimento da extracção da proteína Mx1. Além disso, as esferas de proteína G pode ser substituído por pérolas de proteína A, dependendo das espécies hospedeiras a partir do qual os anticorpos utilizados são derivados. No entanto, os resultados mais limpas são obtidos com esferas de proteína G. Finalmente, este protocolo pode também ser utilizado para estudar a interacção entre Mx1 e outras proteínas de influenza, por exemplo, PB2 14. Neste caso, um epitopo tag PB2 V5 foi utilizado, o que poderia, em princípio, ser combinada com gel de afinidade de agarose anti-V5 disponível comercialmente.
Em experiências futuras, este protocolo pode ser valiosa para identificar as regiões em Mx1 eNP que são importantes para a interacção NP-Mx1. Uma tal região em Mx1 poderia ser L4 laço, como este circuito tem sido mostrado para ser crítico para a interacção entre MxA e vírus Thogoto NP 8,9. Se anticorpos específicos-NP adequados estejam disponíveis, este protocolo permitirá também para determinar se o aumento da sensibilidade das cepas de gripe aviária para Mx1 correlaciona-se com uma maior interacção NP-Mx1 ou não, o que pode ajudar a entender o mecanismo de sensibilidade Mx. Além disso, a interacção entre Mx1 e componentes de vírus que pertencem a outras famílias que o Orthomyxoviridae, pode também abranger por este protocolo de co-imunoprecipitação adaptado. Por fim, mais estudos para desvendar o efeito de NEM na NP-Mx1 será muito valiosa para entender melhor essa interação específica, mas também para ganhar a introspecção nas aplicações mais amplas do que a adição deste composto durante a lise celular e co-imunoprecipitação experimentos.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Os autores declaram que não têm interesses financeiros concorrentes.
Acknowledgments
Este trabalho foi apoiado por FWO-Vlaanderen, o projeto IOF IOF10 / StarTT / 027 e da Universidade de Ghent Especial Research Grant BOF12 / GOA / 014.
Materials
| Name | Company | Catalog Number | Comments |
| DMEM high glucose | Gibco | 52100-047 | |
| N-Ethylmaleimide | Sigma | E-3876 | Toxic |
| Igepal CA-630 | Sigma | I-30212 | also known as NP40 |
| Protease inhibitor cocktail | Roche | 11 873 580 001 | |
| anti-NP monoclonal antibody | NIH Biodefense and Emerging Infections Research Resources Repository | NR-4282 | ascites blend of clones A1 and A3 |
| anti-RNP polyclonal serum | NIH Biodefense and Emerging Infections Research Resources Repository | NR-3133 | directed against A/Scotland/840/74 (H3N2) |
| Protein G Sepharose 4FF | GE Healthcare | 17-0618-01 | |
| Hyperfilm ECL 18 x 24 cm | GE Healthcare | 28-9068-36 | |
| ECL western blotting substrate | Pierce | 32106 |
References
- Verhelst, J., Hulpiau, P., Saelens, X. Mx proteins: antiviral gatekeepers that restrain the uninvited. Microbiol Mol Biol Rev. 77 (4), 551-566 (2013).
- Goujon, C., et al. Human MX2 is an interferon-induced post-entry inhibitor of HIV-1 infection. Nature. 502 (7472), 559-562 (2013).
- Kane, M., et al. MX2 is an interferon-induced inhibitor of HIV-1 infection. Nature. 502 (7472), 563-566 (2013).
- Liu, Z., et al. The interferon-inducible MxB protein inhibits HIV-1 infection. Cell Host Microbe. 14 (4), 398-410 (2013).
- Kochs, G., Haller, O. Interferon-induced human MxA GTPase blocks nuclear import of Thogoto virus nucleocapsids. Proc Natl Acad Sci U S A. 96 (5), 2082-2086 (1999).
- Flohr, F., Schneider-Schaulies, S., Haller, O., Kochs, G. The central interactive region of human MxA GTPase is involved in GTPase activation and interaction with viral target structures. FEBS Lett. 463 (1-2), 24-28 (1999).
- Kochs, G., Haller, O. GTP-bound human MxA protein interacts with the nucleocapsids of Thogoto virus (Orthomyxoviridae). J Biol Chem. 274 (7), 4370-4376 (1999).
- Mitchell, P. S., et al. Evolution-guided identification of antiviral specificity determinants in the broadly acting interferon-induced innate immunity factor MxA. Cell Host Microbe. 12 (4), 598-604 (2012).
- Patzina, C., Haller, O., Kochs, G. Structural requirements for the antiviral activity of the human MxA protein against Thogoto and influenza A virus. J Biol Chem. 289 (9), 6020-6027 (2014).
- Turan, K., et al. Nuclear MxA proteins form a complex with influenza virus NP and inhibit the transcription of the engineered influenza virus genome. Nucleic Acids Res. 32 (2), 643-652 (2004).
- Dittmann, J., et al. Influenza A virus strains differ in sensitivity to the antiviral action of Mx-GTPase. J Virol. 82 (7), 3624-3631 (2008).
- Zimmermann, P., Manz, B., Haller, O., Schwemmle, M., Kochs, G. The viral nucleoprotein determines Mx sensitivity of influenza A viruses. J Virol. 85 (16), 8133-8140 (2011).
- Manz, B., et al. Pandemic influenza A viruses escape from restriction by human MxA through adaptive mutations in the nucleoprotein. PLoS Pathog. 9 (3), e1003279 (2013).
- Verhelst, J., Parthoens, E., Schepens, B., Fiers, W., Saelens, X. Interferon-inducible protein Mx1 inhibits influenza virus by interfering with functional viral ribonucleoprotein complex assembly. J Virol. 86 (24), 13445-13455 (2012).
- Brewer, C. F., Riehm, J. P. Evidence for possible nonspecific reactions between N-ethylmaleimide and proteins. Anal Biochem. 18 (2), 248-255 (1967).
- Wu, C. Y., Jeng, K. S., Lai, M. M. The SUMOylation of matrix protein M1 modulates the assembly and morphogenesis of influenza A virus. J Virol. 85 (13), 6618-6628 (2011).
- Chen, Z., Zhou, J., Zhang, Y., Bepler, G. Modulation of the ribonucleotide reductase M1-gemcitabine interaction in vivo by N-ethylmaleimide. Biochem Biophys Res Commun. 413 (2), 383-388 (2011).
- Rennie, M. L., McKelvie, S. A., Bulloch, E. M., Kingston, R. L. Transient dimerization of human MxA promotes GTP hydrolysis, resulting in a mechanical power stroke. Structure. 22 (10), 1433-1445 (2014).
- Gifford, J. L., Walsh, M. P., Vogel, H. J. Structures and metal-ion-binding properties of the Ca2+-binding helix-loop-helix EF-hand motifs. Biochem J. 405 (2), 199-221 (2007).
- Separating Protein with SDS-PAGE. , JoVE. Available from: http://www.jove.com/science-education/5058/separating-protein-with-sds-page (2014).
- Gallagher, S., Chakavarti, D. Immunoblot analysis. J Vis Exp. (16), (2008).
- Eslami, A., Lujan, J. Western blotting: sample preparation to detection. J Vis Exp. (44), (2010).
- Pavlovic, J., Haller, O., Staeheli, P. Human and mouse Mx proteins inhibit different steps of the influenza virus multiplication cycle. J Virol. 66 (4), 2564-2569 (1992).



