Summary
Il protocollo immunofluorescenza indiretta descritto in questo articolo consente il rilevamento e la localizzazione delle proteine nella ghiandola mammaria del mouse. Un metodo completo è dato per la preparazione di campioni della ghiandola mammaria, per eseguire immunoistochimica, per l'immagine delle sezioni di tessuto al microscopio a fluorescenza, e di ricostruire le immagini.
Abstract
L'immunofluorescenza indiretta è utilizzato per rilevare e localizzare le proteine di interesse in un tessuto. Il protocollo qui presentato descrive un metodo semplice e completo per la rilevazione di proteine immunitario, mouse allattamento ghiandola mammaria essere preso come esempio. Un protocollo per la preparazione dei campioni di tessuto, soprattutto per quanto riguarda la dissezione del mouse di ghiandola mammaria, la fissazione dei tessuti e sezionamento tessuti congelati, sono dettagliati. Un protocollo standard per eseguire immunofluorescenza indiretta, tra cui un passo di recupero dell'antigene opzionale, è anche presentato. L'osservazione delle sezioni di tessuto etichettati così come l'acquisizione di immagini e post-trattamenti sono anche indicati. Questa procedura fornisce una panoramica completa, dalla raccolta di tessuto animale per la localizzazione cellulare di una proteina. Anche se questo metodo generale può essere applicato ad altri campioni di tessuto, deve essere adattato ad ogni coppia anticorpo tessuto / primary studiato.
Introduction
La ghiandola mammaria è un atipico organo esocrina mammiferi la cui principale funzione è quella di produrre latte per nutrire i neonati. Lo sviluppo del tessuto mammario si verifica soprattutto dopo la nascita ed è caratterizzata da un processo unico in cui l'epitelio invade lo stroma circostante. Questo tessuto subisce molti cambiamenti (crescita, differenziazione e regressione), soprattutto durante la vita adulta, in concomitanza con variazioni di stato riproduttivo (Figura 1). Oltre alla morfologia generale del tessuto, le proporzioni dei vari tipi di cellule, nonché la loro disposizione all'interno della ghiandola mammaria cambiare radicalmente durante lo sviluppo 1-5.
Durante la vita embrionale, l'epitelio mammario deriva da linee latte mammarie, che è definita da un lieve ispessimento e stratificazione della ectoderma, tra gli arti anteriori e posteriori su ciascun lato della linea mediana intorno al giorno embrionale 10.5 (E10.5) (Figura 1A ).Sulla E11.5, la linea del latte scompone in singoli placodi, simmetricamente posizionati lungo la linea di latte mammaria in posizioni riproducibili, e il mesenchima circostante inizia a condensare. I placodi cominciano ad affondare più in profondità nel derma e mesenchima mammaria organizza a strati concentrici intorno al bocciolo mammaria (E12.5-E14.5). A partire dal E15.5, dell'epitelio mammario, comincia a proliferare e allungare a formare il germoglio principale che spinge attraverso il mesenchima mammaria verso il cuscinetto adiposo. Il germoglio primario sviluppa un lume cavo con un'apertura per la pelle, contrassegnata dalla formazione della guaina capezzolo. Sul E18.5, il condotto di allungamento è diventato il cuscinetto adiposo e si è ramificata in un piccolo sistema duttale arborized inglobato nel cuscinetto adiposo. Lo sviluppo è essenzialmente arrestato e la ghiandola mammaria rudimentale rimane morphogenetically quiescente fino alla pubertà. Nel embrione maschio, l'attivazione dei recettori degli androgeni porta alla degenerazione dei germogli, che scompaionoda E15.5. Come di E18, sviluppo mammario cessa fino alla pubertà 6-9.
Alla nascita, la ghiandola mammaria ospita un sistema duttale rudimentale che allunga e rami lentamente (una crescita isometrica). All'inizio della pubertà, strutture sferiche situate alle estremità dei condotti chiamati gemme estremità terminale (TEBS), sono formate da uno strato esterno di cellule del cappuccio e un nucleo interno multistrato di cellule (cellule del corpo). Queste strutture sono altamente proliferative e infiltrano il tessuto stromale circostante in risposta a stimoli ormonali. La proliferazione all'interno dei risultati TEBS in allungamento duttale, accoppiato con ramificazione morfogenesi. Questo processo porta alla creazione di una rete arborized epiteliale di base che emana dal capezzolo (Figura 1B, pubertà). A ~ 10-12 settimane dopo la nascita, quando l'epitelio ha invaso tutto il cuscinetto di grasso, la sua espansione si arresta e il TEBS scomparire. Sviluppo duttale subisce quindi cambiamenti dinamici, vale a dire, successive la proliferazione e la regressione delle cellule epiteliali secondo cicli estrali 10 (Figura 1B, adulti).
Dall'inizio di gestazione, il tessuto mammario subisce importante crescita e cambiamenti morfologici per preparare l'allattamento. L'epitelio mammario ampiamente proliferano e differenziare, portando ad una ramificata rete altamente tubulo-alveolari. Allo stesso tempo, le cellule epiteliali mammarie (MECS) diventano polarizzata e in grado di sintetizzare e secernere prodotti lattiero-caseari. MECs organizzano in numerose strutture alveolari (acini), che sono circondati da cellule mioepiteliali contrattili e incorporati in uno stroma composta da tessuto connettivo e adiposo, vasi sanguigni e terminazioni nervose (Figura 1B, gravidanza). Inoltre, il lato basale MECs è in stretto contatto con la membrana basale (matrice extracellulare) e le interazioni tra queste due entità strettamente regolano sia la funzione morfogenesi e secretoria della mammellary epitelio 11-13.
Tutti questi processi si basano sull'azione di vari segnali ambientali, di cui i più importanti sono hormones14, fattori paracrini e la matrice extracellulare. Ad esempio, progesterone induce vasta ramificazione laterale 15 e alveologenesis che, in combinazione con prolattina (PRL) 16,17, promuove e mantiene la differenziazione degli alveoli. Oltre a steroidi e PRL18, citochine e vie di segnalazione associate allo sviluppo (Wnt e Notch vie di segnalazione) sono anche coinvolti nell'impegno lignaggio mammaria e di sviluppo 19-21. Al termine della gravidanza, i MECs luminali iniziano a produrre un latte ricco di proteina nota come colostro nel lume degli alveoli. Inoltre, il progesterone agisce sulla permeabilità epiteliale e dato che le giunzioni strette sono ancora aperte, il colostro si trova anche nel sangue materno.
Dopo il parto, il Mammary epitelio occupa quasi tutto il volume della ghiandola mammaria e è altamente organizzato (Figura 2, mammario). Unità latte produzione, ovvero alveoli (Figura 2, alveoli), sono formate da un monostrato di polarizzati mammarie epiteliali cellule secretorie (MESCs), con la loro membrana plasmatica apicale delimita il lume. Alveoli si dispongono in lobuli che sono raggruppati in lobi collegati a condotti che drenano il latte con l'ambiente esterno (Figura 2, del lobo). Allattamento si verifica, ad esempio., MESCs iniziano a secernere abbondanti quantità di latte, innescato principalmente dal calo di ormoni della placenta (principalmente progesterone) (Figura 1B, allattamento). Geni delle proteine del latte sono attivati in un corso a tempo temporale definito che va dalla gravidanza al allattamento 9,22,23, soprattutto in risposta a ipofisaria PRL rilasciato al momento dell'allattamento. Allo stesso tempo, i contatti tra MESCs e la matrice extracellulare, sia stimolano proteine del latte syntESI attraverso i segnali che sono mediati attraverso le interazioni tra integrine e laminina 24,25 cellulari, e sopprimono l'apoptosi in MESCs 26,27. Queste vie di segnalazione provocano l'attivazione di proteine del latte gene promoters 28 attraverso l'attivazione della trascrizione specifici fattori di 29. Contatti cellula-cellula sono importanti anche per alcuni aspetti della differenziazione tra cui l'istituzione della polarità apicale e la secrezione vettoriale di prodotti lattiero-caseari. Giunzioni strette rapidamente stretti dopo l'inizio della lattazione e MESCs finemente orchestrano l'assorbimento di molecole dal sangue così come la sintesi, il trasporto e la secrezione di componenti del latte, in risposta alle esigenze nutrizionali dei neonati. Al momento della suzione, la contrazione delle cellule mioepiteliali circondano gli alveoli si verifica in risposta a ossitocina e porta alla emissione del latte attraverso i condotti e nel capezzolo. Il latte è un liquido che contiene proteine complessa (principalmentecaseine), zuccheri (principalmente lattosio), lipidi e minerali, come pure molecole bioattive come immunoglobuline A (IgA), fattori di crescita e ormoni. Sono sintetizzati caseine, assemblati in strutture supramolecolari, ossia micelle di caseina, trasportate lungo la via secretoria, e poi rilasciato per esocitosi, cioè, la fusione delle vescicole secretorie-caseina contenente (SV) con la membrana plasmatica apicale di MESC (Figura 2).
Traffico intracellulare si basa su scambi materiali tra compartimenti membranose e coinvolge solubile N-ethylmaleimide sensibile Fusion (NSF) Allegato Protein (SNAP) Receptor (SNARE) 30,31. La famiglia delle proteine SNARE è suddivisa in SNAREs vescicolari (v-lacci), presenti nella membrana della vescicola e SNAREs bersaglio (t-lacci), localizzati sulle membrane di destinazione. Con sfreccia tra loro domini coiled-coil, V e t-SNARE assemblare per formare un complesso altamente stabile fascio quattro elica, indicati come thcomplesso SNARE e. Questo complesso promuove la fusione di due doppi strati lipidici opposti gradualmente portarli in prossimità 30,32. In seguito, i complessi SNARE sono dissociate dalla adenosina trifosfatasi NSF e le sue proteine adattatore Snap e SNARE proteine sono riciclati di nuovo al loro compartimento di origine 33. È interessante notare che, ogni proteina SNARE risiede prevalentemente in compartimenti cellulari distinti e rullante accoppiamento possono contribuire alla specificità degli eventi di fusione intracellulari 34. Precedenti studi suggeriscono che almeno Protein 23 (SNAP23) e vescicole associata a membrana di proteine 8 (VAMP8), e sintaxine (Stx) sinaptosomiali associata -7 e -12 svolgono un ruolo nella caseina esocitosi 35,36. Queste proteine sono state trovate anche in associazione con la frazione lipidica del latte, vale a dire, di grasso del latte (globuli MFGs) 37. Il modello prevalente corrente postula che gocce lipidiche citoplasmatici (CLD) sono formati dall'accumulo di l neutroipids (soprattutto trigliceridi ed esteri di steroli) e colesterolo deriva dalla dieta materna tra i due volantini del reticolo endoplasmatico (ER) membrane 38-41. Grandi CLD si formano, almeno in parte, dalla fusione di CLD piccole mentre viene trasportato al lato apicale MESCs dove vengono rilasciati come MFGs (1-10 micron di diametro) per gemmazione, essendo avvolto dalla membrana plasmatica apicale MESC 40-42. Allattamento cessa dopo cuccioli sono svezzati e le MESCs progressivamente muoiono per apoptosi, che porta alla regressione del tessuto mammario a uno stato puberale (Figura 1B, involuzione).
Immunofluorescenza (IF) è un metodo di laboratorio di analisi comune usato in quasi tutti gli aspetti della biologia, sia nella ricerca e nella diagnostica clinica. SE tecniche possono essere eseguite su sezioni di tessuto (immunoistochimica, IHC) o cellulari (immunocitochimica, ICC) campioni. Questo approccio potente si basa sull'utilizzo di fluorescente-anticorpi marcati che specificamente si legano (direttamente o indirettamente) all'antigene di interesse, permettendo così la visualizzazione della sua distribuzione tissutale tramite microscopia a fluorescenza. Segnali di fluorescenza per lo più dipendono dalla qualità e la concentrazione degli anticorpi e corretta manipolazione del campione. Un protocollo semplice immunofluorescenza indiretta (IIF) viene presentato per rilevare prodotti lattiero-caseari (caseine e MFGs) e proteine coinvolte nella secrezione di prodotti lattiero-caseari (butirofilina (BTN1), rullante proteine) su sezioni congelate di tessuto mammario del mouse (Figura 3). Anche se questo protocollo offre una panoramica completa IHC, che vanno dalla raccolta di tessuto per l'immagine dopo il trattamento, critica e passaggi facoltativi, nonché alcune raccomandazioni tecniche vengono anche presentati e discussi.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Topi CD1 sono stati allevati presso l'INRA (UE0907 IERP, Jouy-en-Josas, Francia). Tutti gli aspetti etici della cura degli animali conformi alle relative linee guida ei requisiti di licenza stabiliti dal Ministero dell'Agricoltura francese. Le procedure utilizzate sono state approvate dal comitato locale di etica (accordo 12/097 dal Comethea Jouy-en-Josas / AgroParisTech).
1. ghiandola mammaria preparazione del campione
- Mouse ghiandola mammaria dissezione
- Euthanize topi al giorno 10 di lattazione per dislocazione cervicale e appuntare l'animale verso il basso con l'addome rivolto verso l'alto.
- Bagnare la zona ventrale con etanolo e asciugare con un tovagliolo di carta.
- Utilizzando pinze, tirare la pelle addominale tra le due zampe posteriori e fare un'incisione (solo attraverso la pelle) di circa 1 cm con le forbici affilate. A partire da questa prima incisione, quindi utilizzare le forbici per tagliare la pelle fino al collo sul mouse. Tirare la pelle lontano dal peritoneo e pin lungo un lato della pelle alla volta, allungandolo insegnato.
- Raccogliere il addominale e le inguinali ghiandole mammarie spingendole dalla pelle con un tampone e infine tirando o tagliando via dal peritoneo.
Nota: In questo passaggio Carmine colorazione può essere eseguita per visualizzare l'epitelio mammario entro tutta la ghiandola 43. Questo approccio può essere utile per analizzare la morfologia globale della ghiandola mammaria in varie condizioni fisiologiche (stadi di sviluppo, le malattie, in vivo trattamenti). - Togliere il linfonodo si trova all'incrocio tra la addominale e le ghiandole inguinali 44.
- Fissaggio tessuto mammario
- Tagliare il tessuto mammario in 3 mm 3 frammenti con un bisturi e sciacquare immediatamente questi frammenti in una soluzione salina tampone fosfato (PBS), pH 7,4, al fine di eliminare il più possibile il latte.
- Asciugare rapidamente i frammenti su una cartaasciugamano e metterli in una soluzione di PBS freddo contenente 4% paraformaldeide (PFA, HCHO, 32% soluzione di formaldeide, ATTENZIONE) per 10 a 15 minuti in ghiaccio.
Nota: Questo è il tempo sufficiente per consentire la successiva analisi su sezioni di tessuto mammario da IIF36 e / o ibridazione in situ 45. Tuttavia, come fissativi aldeidi penetrano piuttosto lentamente a pezzi di tessuto (~ 1-3 mm per ora), questo può essere prorogato per garantire una fissazione ottimale del campione di tessuto. In alternativa, riparare i tessuti in vivo mediante perfusione un animale anestetizzato con una soluzione fissativa (non specificato nel presente studio).
- Infusione di saccarosio
- Sciacquare rapidamente i frammenti mammari in PBS freddo e immergerli in una soluzione PBS freddo contenente 40% di saccarosio (D-saccarosio, C12H22O11, Mr 342,3 g / mol) per 16 a 48 ore a 4 ° C sotto leggero scuotimento.
- Tissue incorporamento
Nota: In questo passaggio, frammenti mammaria può essere ri-tagliata per rendere frammenti più piccoli (2-3 mm 3) O per regolare la loro forma.- Correttamente etichettare gli stampi di plastica e riempire un terzo del volume dello stampo con OCT, mantenuta a RT. Mettere un frammento (2-3 mm 3) di tessuto mammario per stampo e coprire con mescola ottobre
- Mettere gli stampi sulla superficie del azoto liquido (su un foglio di alluminio o usando un setaccio metallico) e lasciare il prodotto di congelare.
Nota: Deve diventare solido e bianco prima di immergere lo stampo in azoto liquido.
- Conservare i campioni congelati a -80 ° C fino a quando vengono eseguite sezioni di tessuto.
2. congelato sezionamento dei tessuti
Nota: Un criostato, che è essenzialmente un microtomo in freezer, è necessario fare sezioni di tessuto congelato. Una temperatura più bassa è spesso richiesto per i tessuti grassi o ricchi di lipidi, come ghiandola mammaria vergine.
- Regolare la temperatura del criostato a -26 ° C e attendere che si ha stabizato. Mantenere il blocco di tessuto congelato a -26 ° C durante l'intera procedura di sezionamento. Evitare assolutamente scongelamento del tessuto in qualsiasi momento durante la procedura.
- Raffreddare la lametta, supporto di taglio, il dispositivo anti-rollio ed il pennello a -26 ° C mettendoli in criostato per almeno 10 min. Anche mettere una casella scorrevole all'interno del criostato per essere in grado di memorizzare vetrini delle sezioni sono fatti.
- Correttamente etichettare i vetrini che verranno utilizzati per raccogliere le sezioni di tessuto e mantenerle a temperatura ambiente; altrimenti sezioni di tessuto non aderirvi. Togliere il campione dalla muffa all'interno del criostato.
Nota: Utilizzando vetrini con carica positiva notevolmente favorire l'adesione delle sezioni di tessuto congelato fresche a causa della maggiore attrazione elettrostatica. - Coprire la superficie di un disco di tessuto metallico con OCT (mantenuta a RT) e spingere il campione congelato su di esso. Posizionare il bagnato montare all'interno del criostato e lasciarlo coolo per almeno 15 minuti.
- Posizionare il supporto bagnato nel supporto del disco del criostato. Regolare lo spessore del taglio di 5-6 micron e, se possibile, utilizzare un nuovo lama affilata o almeno modificare l'area sulla lama utilizzata per tagliare ogni campione, poiché alcuni tessuti sarà rapidamente noioso.
- Regolare la posizione del dispositivo anti-rollio sulla lama di rasoio mediante tagli del mezzo di montaggio fino a quando le fette sono formate in modo uniforme e corretto. Idealmente, il dispositivo anti-rollio si scavalcare la lama di rasoio di circa 1 mm.
- Una volta che le impostazioni siano corrette, eseguire sezioni di tessuto girando la ruota in un movimento continuo uniforme. A meno che la temperatura è ideale, una sezione di tessuto sarà, per sua natura, cerca di rannicchiarsi.
- Utilizzare un pennello per afferrare e manovrare la sezione attraverso il palco in modo da mettere come desiderato sul vetrino. Utilizzare la spazzola per pulire i resti eventualmente presenti sul blocco tessuti congelati e / o la lama di rasoio.
- Tirarela sezione di tessuto verso l'utente ed evitare premendolo sul palco criostato. Evitare di premere la sezione di tessuto sulla scena criostato in quanto può portare alla adesione della fetta del tessuto sulla scena, e quindi l'impossibilità di recuperare con il vetrino.
- Recupero sezioni di tessuto uno per uno prendendoli alla superficie di un vetrino, tenendolo sopra la sezione e inclinare fino a toccare la sezione di tessuto.
Nota: Le sezioni di tessuto aderiscano rapidamente al vetro caldo a causa di attrazione statica. Se più sezioni di tessuto sono posti sullo stesso vetrino, fare attenzione a non sovrapporre loro e con lo spazio abbastanza per essere in grado di racchiuderli singolarmente in un cerchio idrofoba (vedi paragrafo 3.1.1.).
3. immunofluorescenza indiretta
- Individuazione sezioni
- Utilizzare una penna barriera idrofobica per disegnare un cerchio intorno al tessuto idrofobico montato-slide. Lasciate che il cerchio asciugare per circa 1 minuti a temperatura ambiente. Tracciare una linea intorno alla temissione sezioni con un pennarello indelebile nero fine così, ma sul lato del vetrino opposto a quello in cui le sezioni di tessuto sono.
Nota: Questo cerchio è idrorepellente e acetone- e alcool-solubile. Si prevede quindi una barriera alle soluzioni acquose utilizzati durante la procedura IHC e riduce il volume dei reagenti richiesti. - Reidratare sezioni di tessuto coprendoli con una goccia di ~ 250 microlitri di PBS per alcuni minuti a temperatura ambiente. Fissare sezioni di tessuto coprendoli con ~ 250 ml di una soluzione PFA 3% appena preparato in PBS per 10 a 15 minuti.
Nota: Facoltativamente in questo caso, utilizzare una soluzione di aldeide quenching (cloruro di ammonio 50 mM (NH 4 Cl, Mr 53,5 g / mol) in PBS 0,1 M glicina o (C 2 H 5 NO 2, Mr 75,07 g / mol) in PBS ) per arrestare la reazione di fissazione. Semplice e abbondante lavaggio PBS è generalmente sufficiente per eliminare l'aldeide reagito.
- Utilizzare una penna barriera idrofobica per disegnare un cerchio intorno al tessuto idrofobico montato-slide. Lasciate che il cerchio asciugare per circa 1 minuti a temperatura ambiente. Tracciare una linea intorno alla temissione sezioni con un pennarello indelebile nero fine così, ma sul lato del vetrino opposto a quello in cui le sezioni di tessuto sono.
- Recupero antigene (opzionale)
- Posizionare la soluzione AR (Tris 100 mm (C 4 H 11 NO 3, il sig 121.14) 5% di urea (NH 2 CONH 2, il sig 60.06) pH 9.6) in un bicchiere. Il volume di soluzione AR deve essere sufficiente a coprire completamente i vetrini collocato in un supporto di vetro.
- Preriscaldare la soluzione AR a 95 ° C controllando la temperatura con un termometro e quindi posizionare i vetrini in un rack adatto, immergere la cremagliera nel buffer caldo, copertura per limitare l'evaporazione e incubare per 10 min a 95 ° C.
- Rimuovere il becher dal bagnomaria e lasciare i vetrini per altri 10 min nel buffer.
- Risciacquare sezioni di tessuto con PBS (~ 250 microlitri / sezione) e saturare con una soluzione3% di albumina di siero bovino (BSA, ~ 250 microlitri / sezione) in PBS per almeno 30 minuti a temperatura ambiente.
- Mettere 30-50 microlitri di anticorpo primario diluito in PBS contenente 2% BSA in ogni sezione di tessuto.
Nota: Questo volume è sufficiente per formare una goccia che copre completamente la sezione di tessuto. - Posizionare lo stesso volume di diluente (2% BSA in PBS) da solo su una sezione di tessuto per eseguire un controllo negativo senza anticorpo primario.
- Includere sistematicamente questo controllo negativo in ogni esperimento IHC ed eseguire per ciascun anticorpo secondario utilizzato per stimare lo sfondo dell'esperimento (etichettatura non specifica dovuta alla anticorpo secondario e / o il tessuto auto-fluorescenza). Altri tipi di controlli positivi o negativi possono essere eseguite anche per garantire la specificità del etichettatura (vedi la discussione).
- Porre i vetrini in una scatola umidificata O / N a 4 ° C.
Nota: Gli anticorpi primari utilizzati sono stati topo monoclonale anti-citocheratina8 (CK8, diluizione 1:50), mouse monoclonale anti-citocheratina 14 (CK14, diluizione 1:50), policlonale di coniglio caseina anti-topo (# 7781, 1:50 diluizione, generosamente fornito da MC Neville, dell'Università del Colorado Salute Sciences Center, CO, USA), policlonale di coniglio anti-BTN1 (1: 300 di diluizione, generosamente fornito da IH Mather, Dipartimento di Scienze Animali e aviaria, University of Maryland, College Park, MD, USA), policlonale di coniglio anti-Stx6 ( 01:50 diluizione, generosamente fornito da S. Tooze, Cancer Research UK, London Research Institute, Londra, Regno Unito) e policlonale di coniglio anti-VAMP4 (diluizione 1:50). - Lavare accuratamente sezioni di tessuto con PBS almeno quattro volte per 10 minuti a temperatura ambiente.
- Diluire l'anticorpo secondario appropriata (IgG anti-coniglio di capra coniugato con rodamina (H + L), 1: 300 diluizione) in PBS contenente 2% BSA, posizionare 30-50 ml di questa soluzione in tutte le sezioni di tessuto, e incubare per 1,5 ore a RT.
- Poiché fluorocromi sono molecole sensibili alla luce, non lo fannoesporre sezioni di tessuto alla luce fino alla loro analisi. Per IFI su sezioni di tessuto, favorire anticorpi secondari accoppiati a un fluoroforo rosso da membrane cellulari tendono a generare un auto-fluorescenza verde che possono interferire con basso etichettatura. Inoltre, la scelta di un anticorpo secondario fluoroforo accoppiati rosso permette l'etichettatura concomitante di lipidi neutri (vedi sotto).
- Lavare accuratamente le sezioni di tessuto con PBS almeno quattro volte per 10 minuti a temperatura ambiente.
- Per alcuni esperimenti, eseguire post-fissazione incubando i campioni con 2% PFA diluito in PBS per 10 minuti a RT per stabilizzare i ponteggi antigene / anticorpo. Tuttavia, questo passo può essere soppressa in molti casi.
- Per visualizzare CLD e MFGs, colore lipidi neutri incubando sezioni di tessuto in 30-50 ml di una soluzione PBS contenente 3 mg / ml di BODIPY 493/ 503 per 10 minuti a RT. Rapidamente sciacquare sezioni di tessuto due volte con PBS.
- Controcolorare DNA nucleare con 30-50 ml di una soluzione di PBS contenente 3 mM di DAPI (4-6-diamidino-2-phenylindole, 5 mg / ml soluzione madre) per 10 minuti a RT. Lavare le sezioni di tessuto due volte con PBS prima del montaggio dei vetrini per l'osservazione.
- Rimuovere PBS e una goccia di mezzo di montaggio per ogni sezione di tessuto.
- Collocare un lato del vetrino ad un angolo contro la slitta, il contatto con il bordo esterno della goccia liquido e abbassare il coperchio lentamente, evitare bolle d'aria. Lasciare che il liquido per diffondere tra il vetrino e vetrino per alcuni minuti e poi rimuovere l'eccesso di mezzo di montaggio con un tovagliolo di carta.
- Sigillare la polizza di copertura al vetrino con le sezioni smalto e del tessuto conservare a 4 ° C per impedire la loro esposizione alla luce fino osservazione.
4. Fluorescenza Osservazione e Image Acquisition
Nota: Un microscopio a fluorescenza dotato di una fotocamera controllato dal software di acquisizione immagini è tenuto ad osservare i risultati IHC.
- Prima di acquisire immagini, controllare l'intensità della etichettatura e valutare il fondo dell'esperimento, cercando in controlli negativi. Acquisire le immagini di ciascuna etichetta fluorescente (canale di colore) individualmente.
- Acquisire tutte le immagini, compresi quelli dei controlli corrispondenti, nelle stesse condizioni (esposizione e le impostazioni generali) per ogni canale di colore.
- Microscopia convenzionale
- Eseguire epifluorescenza con un microscopio dotato di filtri standard per isotiocianato di fluorescina (FITC, verde), rodamina (rosso) e DAPI (blu) emissioni, × 20 × 63 (-immersione in olio, NA 1.3) obiettivi e una telecamera ad DP50.
- Microscopia confocale
- Eseguire la microscopia confocale con un micropiviale equipaggiata con il software ZEN, utilizzando × 20 × 63 a (-immersione in olio, NA 1.4) gli obiettivi e le 488- e 568 nm di eccitazione lunghezze d'onda del laser.
Trattamento 5. Immagine
Nota: Tutte le immagini post-trattamenti vengono eseguiti utilizzando il software gratuito ImageJ (http://imagej.nih.gov/ij/).
- Sovrapporre immagine (merge)
- Aprire le immagini acquisite in ogni canale che verrà combinato (File / Apri). Se si lavora con 8 bit in scala di grigi, attribuire colore artificiale per ciascun canale utilizzando la tabella di ricerca (tabelle di immagine / di ricerca).
- Generare il quadro composito da immagini in scala di grigi o colorati utilizzando il comando "Unisci i canali" (Immagine / colori / Merge Canali) e poi attribuire un colore per ogni canale.
- Eseguire immagine pile sovrapposizione nello stesso modo, aprendo pile acquisite in ogni canale che verrà combinato (File / Apri) e utilizzando il comando "Unisci canali &# 8221; (Immagine / colori / Merge Canali) per attribuire un colore per ogni canale. Salvare lo stack composita come una sequenza di immagini o come un film (vedi paragrafo 5.4).
- Immagine pila Z proiezione
- Utilizzare la funzione Z di proiezione (Immagine / Pila / Zproject, intensità massima) per fornire una visione a due dimensioni di tutte le immagini di una serie di immagini da proiettandole lungo l'asse perpendicolare al piano dell'immagine (asse z). L'opzione "Maximum Intensity" crea un'immagine in cui ogni pixel contiene il valore massimo su tutte le immagini della pila. Ciò genera una singola immagine che permette la visualizzazione di tutti i colorazione osservata attraverso l'intera serie di immagini per un particolare canale o dopo la sovrapposizione di più canali.
- Immagine proiezione abbinata 3D
- Utilizzare il comando di proiezione 3D (progetto Immagine / Pila / 3D, più luminoso Point, asse y) per generare una sequenza di proiezioni di un volume rotante su un piano. La resa visiva di surfaces e le strutture interne dipende sia il metodo di proiezione (più vicino punto, punto più luminoso (usato qui), o valore medio-) ed i parametri di visualizzazione selezionati. Ogni frame della sequenza animata è il risultato di sbalzo da un diverso angolo di visualizzazione.
- Ruotare l'immagine creata in 3D intorno a ciascuno dei tre assi ortogonali (l'asse y è stato qui selezionato). Salvare la sequenza di prodotto come una singola immagine o un filmato.
- Serie di immagini per la conversione di film
- Aprire una serie di immagini (File / Apri) e salvarlo come un film in formato .AVI utilizzando il comando "AVI" (File / Salva con nome / AVI).
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
La ghiandola mammaria è una ghiandola sottocutanea situato lungo la struttura ventrale sia del torace e dell'addome nei roditori. La posizione dei cinque coppie di ghiandole del mouse durante la gestazione è mostrata in Figura 4. La morfologia della ghiandola mammaria cambia drasticamente durante il suo sviluppo, riflettendo modifiche funzionali richieste per prepararsi alla piena lattazione (Figura 1B). Negli animali vergini o nullipare, la ghiandola mammaria è costituito da un epitelio duttale scarsamente ramificati incorporato all'interno di un stroma grasso sottile che può essere difficile da vedere. Fin dall'inizio della gravidanza, i prolifera epitelio mammario e si espande, con conseguente più grandi ghiandole mammarie che diventano più facile vedere e rimuovere (Figura 4). Durante l'allattamento, il tessuto mammario è più spessa e appare più bianco per la presenza di latte. Solo ghiandole mammarie addominali e inguinali sono raccolti perché cervicale e toracico Glan mammariads sono meno facilmente rimossi a causa della loro stretta associazione con i muscoli. Per alcuni esperimenti, cuccioli possono essere separati dalla lattazione femmina 4-6 hr prima del sacrificio per limitare la secrezione latte MESCs 46,47.
Identificazione di mioepiteliali mammarie e cellule epiteliali
Cellule mioepiteliali contrattili circondano gli alveoli possono essere distinte da MESCs luminali attraverso l'uso di anticorpi diretti contro marcatori specificamente espressi da ciascuno di questi tipi cellulari. Nella ghiandola mammaria, i marcatori attualmente utilizzati sono citocheratine (CK). CK sono una grande famiglia di proteine citoplasmatiche che polimerizzano a formare filamenti intermedi del citoscheletro (10 nanometri di diametro in media) in tessuti epiteliali. I filamenti intermedi sono estremamente stabili e forniscono un supporto meccanico per l'architettura cellulare, e organizzano tessuti contribuendo ad adesione cellula-cellula e cellula-connettivale basaleinterazioni dei tessuti. I sottoinsiemi di CKs espressi dalle cellule epiteliali dipendono principalmente dal tipo di epitelio, lo stadio di sviluppo e il suo stato di differenziazione. Inoltre, questo vale anche per le controparti maligne del epiteli. Così, questi marcatori sono strumenti semplici e preziosi per caratterizzare popolazioni di cellule in un tessuto in condizioni fisiologiche e sono utilizzati per la diagnosi dei tumori e caratterizzazione in patologia chirurgica 48.
Nel normale ghiandola mammaria, cellule mioepiteliali e MESCs luminali possono essere distinti in base alla loro espressione differenziale di CK14 e CK8, rispettivamente (Figura 5). Questi marcatori citoplasmatici vengono rilevati in sezioni mammarie di topi in allattamento dopo PFA fissazione e AR. Le immagini sono state acquisite con un microscopio a epifluorescenza convenzionali. CK8 sembra essere distribuito in tutto il citoplasma delle MESCs luminale (Figura 5, CK8). Si noti che il fondo rosso osservared per il controllo negativo senza anticorpo primario (figura 5, -Ig1) è principalmente dovuta alla sezione di tessuto di piegatura, come suggerisce l'etichettatura DNA blu, che mostra diversi strati di nuclei (figura 5, -Ig1, nuclei). CK14 è specificamente osservata in cellule myoepithelial piatte e allungate situate alla base degli alveoli (Figura 5), CK14. Un altro modo comune per identificare le cellule mioepiteliali è quello di rilevare actina del muscolo liscio alfa (un - SMA) presenti in queste cellule contrattili (vedi Figura 4 a 49).
Rilevamento dei prodotti lattiero-caseari del mouse
Dopo il parto, le MESCs completamente differenziati iniziano a produrre abbondanti quantità di latte. Componenti di latte sono secreti dalle vie distinte 40,50. Micelle di caseina sono secreti dalle esocitosi di SV Golgi-derivati, mentre i lipidi vengono rilasciati come MFGs dalla gemmazione della pl apicaleasma membrana MESCs (figura 2, mammaria cellule epiteliali secretoria). Per alcuni esperimenti, i cuccioli sono separati dalla femmina 4-6 hr prima di raccogliere le ghiandole mammarie, in modo da rallentare la secrezione lattea 46,47. In queste condizioni, la membrana plasmatica apicale MESCs e il contenuto del lume può essere facilmente osservato, che non è il caso durante l'allattamento poiché alveoli sono contratti e il lume sono chiusi. Inoltre, rallentando la secrezione è essenziale anche quando si studia proteine coinvolte nel traffico di membrana, come trappole. Infatti, Snares ciclo tra donatore e compartimenti accettori e la loro localizzazione subcellulare è difficile da determinare in quanto l'etichettatura è spesso diffusa quando fatturato membrana è alto, vale a dire., Durante l'allattamento. Pertanto, rallentando la secrezione lattea rimuovendo i cuccioli fornisce condizioni adeguate per studiare la localizzazione intracellulare di SNARE quando il t- e V SNARE risiedono preferenzialmente nel donatoree compartimento accettore, rispettivamente (vedi sotto).
Figura 6 mostra la localizzazione delle caseine nella ghiandola mammaria topo allattamento al giorno 10 di lattazione, in presenza (Figura 6, p +) o in assenza (Figura 6, -p) di cuccioli. Le sezioni di tessuto sono state osservate sia da microscopia convenzionale epifluorescenza (le tre colonne sulla destra) e microscopia confocale (Figura 6, colonna di sinistra). Durante latte, caseine sembrano essere principalmente accumulata nella regione apicale (Figura 6, + p, frecce). Microscopia confocale rivela che le caseine sono presenti anche, sebbene in misura minore, al lato basale MESCs in presenza di cuccioli (figura 6, + p, frecce), che non possono essere chiaramente riscontrabili in microscopia convenzionale (figura 6, caseine, confrontare Pannelli sinistro e destro). Infatti, in largo campo epifluorescenza, la fluorescenza emessa dal campione (sfondo fluorescence) passa attraverso il volume eccitato e altera la risoluzione degli oggetti osservati nel piano focale dell'obiettivo (out-of-focus fluorescenza). Ciò è particolarmente vero per i campioni di spessore (spessore superiore a 2 micron). Microscopia confocale permette di ottenere immagini di alta qualità da campioni preparati per epifluorescenza, come la profondità di campo può essere controllata e fluorescenza di fondo escluso dal piano focale. Inoltre, in presenza di cuccioli (Figura 6, + p), il lume degli alveoli sono abbastanza chiuso e il lato apicale MESCs è meglio osservata in assenza di cuccioli (Figura 6), -p, quando il lume alveoli si dilata a causa dell'accumulo di prodotti lattiero-caseari. Quando la secrezione lattea è rallentato, caseine anche appaiono accumulati sotto la membrana plasmatica apicale (figura 6, -p, punte di freccia), e sono chiaramente osservati a lato basale di MESCs (Figura 6, -p, frecce). I controlli negativi senza ANTIB primarioody non ha mostrato alcuna etichettatura (Figura 6, -Ig1).
Caseari possono essere facilmente rilevati co-combinando IHC delle caseine e controcolorazione lipide neutro di CLD e MFGs (Figura 7). Le sezioni di tessuto sono stati ripresi come Z-stack mediante microscopia confocale, che sono stati di post-trattamento con ImageJ per produrre proiezioni Z o proiezioni 3D per ogni (Figura 7, caseine, lipidi) o tutti i canali di colore (Figura 7, fusione). Le sequenze di immagini prodotte sono state salvate come immagini singole (Figure 7 e 8) o filmati (vedi Film complementari).
Sebbene alcuni etichettatura stato osservato dalla loro parte basale, caseine erano per lo più accumulati sul lato apicale delle MESCs (figura 7, + p), come già descritto quando le femmine non venivano separati dai cuccioli (Figura 6, + p). CLD sono anche localizzate principalmente nella regione apicale del MESCs, mentre più grande segretoMFGs Ed sono presenti nel lume degli alveoli. Si noti che caseine e MFGs sono facilmente visibili nel lume di alveoli in assenza di cuccioli (figura 7, confronta + p e -p). Caseine non co-localizzare con CLD o MFGs in una di queste condizioni in quanto la sovrapposizione dei due canali di colore non produce etichettatura giallo (Figura 7, unire le immagini). Tuttavia, pila immagine post-trattamenti dimostrano che circondano le caseine MFGs secreti nel lume degli alveoli, suggerendo che queste proteine possono interagire con il MFG (Figura 7, unire le immagini). Notare la differenza delle immagini prodotte da ogni post-trattamento utilizzato (Figura 7, confrontare Zproj e 3D proj per ciascun canale di colore).
Rilevazione di butirofilina, una proteina marker di MFGs.
BTN1 è una delle principali proteine associate con MFGs nel latte 51. Questa proteina transmembrana è mainly localizzata a livello della membrana plasmatica apicale delle MESCs e di conseguenza è trovato sulla superficie del MFG dopo la sua uscita per gemmazione 52. Figura 8 mostra che al giorno 10 di lattazione, BTN1 è localizzata principalmente a livello della membrana plasmatica apicale e, in misura minore, nella regione apicale MESCs. BTN1 circonda anche i MFGs presenti nel lume di alveoli, così come alcune delle CLD apicali (figura 8, proj 3D merge, punte di freccia). I risultati sono mostrati come una singola immagine estratta dall'immagine Z-stack acquisita (Figura 8, immagine) o come vista 3D generata con il comando proiezione 3D di ImageJ, come descritto in precedenza (Figura 8, proj 3D). Si noti che una singola immagine può essere sufficiente per osservare la distribuzione apicale della proteina, ma l'associazione spaziale BTN1 con MFGs secreti o apicale CLD si osserva solo dopo la ricostruzione 3D della Z-stack (Figura 8 confronta immagine BTN1 e 3D proj merge pictUres). La Z-stack può anche essere ricostruita come film per dare una migliore vista spaziale della distribuzione della proteina. L'immagine Z-stack acquistato per BTN1 solo (filmati di complemento 1 e 3) o sovrapposti con gli altri due canali di colore (fondersi, film supplementari 2 e 4) sono indicati come esempi. Lo Z-stack può essere letto immagine per immagine dall'alto verso il basso (film di complemento 1 e 2) o come una vista rotante (asse y) della proiezione in 3D di tutta la pila di immagini (film supplementari 3 e 4 ).
Rilevazione di due proteine SNARE: Stx6 e VAMP4
Come precedentemente accennato, SNAREs sono proteine di membrana che scorrere le membrane donatore e accettore. E 'quindi meglio rallentare fatturato membrana associata alla elevata attività secretoria delle MESCs separando le femmine da cuccioli prima di raccogliere la ghiandola mammaria quando si studiaqueste proteine. Stx6 e VAMP4 sono stati entrambi descritti come associati con la rete trans-Golgi 53,54. Tuttavia, queste proteine SNARE possono anche svolgere un ruolo importante a livello di altri compartimenti cellulari, come i granuli di secreto (Stx6) 55,56 e l'apparato di Golgi (VAMP4) 57. Precedenti studi suggeriscono che le proteine SNARE svolgono un ruolo nella secrezione di caseina 35,36. Durante l'allattamento, Stx6 e VAMP4 si trovano nella regione sub-apicale di MESCs. Stx6 viene osservata tra il nucleo e la membrana apicale MECs, corrispondente al Golgi e la rete trans-Golgi (Figura 9, Stx6), ed è anche presente, anche se in misura minore, in caseina contenente SV 36. VAMP4 è anche localizzato nella regione sub-apicale MESCs, ma l'etichettatura sembra essere più puntiformi e si accumula sotto la membrana plasmatica apicale (Figura 9, VAMP4) a causa della sua associazione con entrambe CLD e caseina contaSV ining 36. Controllo negativo senza anticorpo primario non ha dato luogo ad alcuna etichettatura.

Figura 1. Topo sviluppo della ghiandola mammaria durante la vita embrionale e adulta. (A) La ghiandola mammaria di topo cominciano a svilupparsi intorno giorno embrionale 10 (E10) dai ectodermica (azzurro) linee di latte (di colore rosa). A E11.5, placodi formano simmetricamente lungo la linea del latte mammaria e mesenchima circostante (blu scuro) inizia a condensare. I placodi invaginate per formare gemme (E12.5-E14.5) e, E15.5, l'epitelio mammario (rosa), proliferano e allungare a formare il germoglio principale che spinge attraverso il mesenchima mammaria verso il cuscinetto di grasso (verde chiaro ). Si forma un lume cavi e si apre per dare origine al capezzolo (viola). Sul E18.5, l'epitelio mammario forma un Rudimentastruttura ramificata ry collegato all'esterno. Adattato da 6 con il permesso di Macmillan Publishers Ltd: Nature Reviews Genetics, diritto d'autore 2007. (B) Durante la pubertà, l'epitelio mammario (viola) entra in una fase di crescita significativa (esteso allungamento, biforcazione e ramificazioni laterali). All'inizio della gestazione, ampia e rapida proliferazione nonché ramificazione laterale verificarsi, portando alla notevole espansione dell'epitelio mammario, che invade completamente l'intero pad grasso mammario. L'epitelio mammario raggiunge uno stato funzionale altamente differenziata durante l'allattamento quando MESCs luminali secernono grandi quantità di latte. Quando l'allattamento cessa dopo lo svezzamento, i evolventi ghiandola mammaria. MESCs vengono rimossi per apoptosi e fagocitosi, portando alla scomparsa delle strutture lobulo-alveolare che vengono sostituiti da tessuto adiposo. Adattato da schema 1 di http://brisken-lab.epfl.ch/research e il capitolo 2.2. http://tvmouse.ucdavis.edu/bcancercd/22/index.html. Cliccate qui per vedere una versione più grande di questa figura.
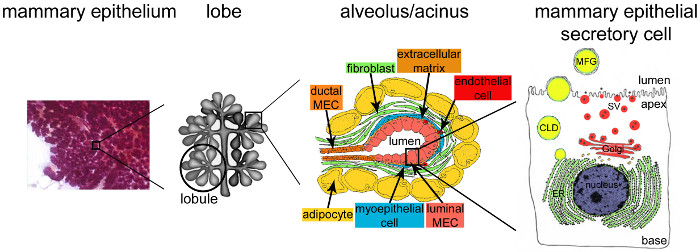
Figura 2. Architettura della ghiandola mammaria durante l'allattamento. Durante l'allattamento, il pieno sviluppo e l'epitelio molto ramificata (viola) rappresenta la stragrande maggioranza del tessuto mammario. Il tessuto epiteliale è formata da strutture tubulo-alveolare incorporati in uno stroma contenente vari tipi di cellule (fibroblasti, adipociti, cellule muscolari lisce, sangue e vasi linfatici e terminazioni nervose). MESCs sono organizzate in strutture o alveoli acinar, assemblati in lobuli che formano lobi. Ogni alveolo è una unità funzionale produttrice di latte che è collegato a una rete altamente ramificata di canali lobulare e interlobulari, permettendo così il latte da dpiovuto verso l'esterno. Ciascun alveolo è delimitato da un monostrato di MESCs polarizzati, il lato apicale che confina con un lume centrale. Il lato basale della MESCs è in stretto contatto con una matrice extracellulare e cellule mioepiteliali contrattili. Prodotti lattiero-caseari vengono rilasciati sul lato apicale di MESCs. Latte Major (caseine) sono secreti come micelle di caseina (punti neri) di esocitosi di vescicole secretorie Golgi-derivati (SVS), mentre i lipidi sono rilasciati come globuli di grasso del latte (MFGs) per gemmazione della membrana plasmatica apicale di MESCs. CLD: citoplasmatica lipidi droplet; ER: reticolo endoplasmatico; MEC: cellule epiteliali mammarie. Adattato dal capitolo 2.2. http://tvmouse.ucdavis.edu/bcancercd/22/index.html., Fig. 02 www.cellbiol.net/ste/alpHERCEPTIN1.php, fig. 26-02 a 58, e da 50. Cliccate qui per vedere una versione più grande di questa figura.
.within-page = "always"> 

Figura 3. Procedura sperimentale per eseguire immunofluorescenza indiretta su sezioni congelate di ghiandola mammaria del mouse. La ghiandola mammaria è raccolto da un topo femmina CD1 al giorno 10 di lattazione. Il tessuto mammario viene tagliato in piccoli frammenti che sono stati corretti con paraformaldeide e infuso in saccarosio, prima di essere incorporato in OCT e snap-congelato. I campioni ghiandola mammaria vengono poi tagliati in sezioni sottili congelate e preparate per IIF mediante incubazione con anticorpi secondari successivi primari e fluorocromi coniugati, rispettivamente. Dopo il montaggio, i campioni vengono analizzati con un microscopio a fluorescenza, permettendo l'acquisizione di immagini che possono successivamente essere post-trattata./53179/53179fig3large.jpg "Target =" _ blank "> Clicca qui per vedere una versione più grande di questa figura.

. Figura 4. anatomica posizione della ghiandola mammaria di topo sinistra: vista ventrale del sistema del mouse mammaria nella fase finale della gravidanza. A destra: la localizzazione e l'aspetto della ghiandola mammaria nella fase finale della gravidanza nel topo. Si noti che durante l'allattamento, le ghiandole mammarie sono più spessi e sembrano più bianca per la presenza di latte negli alveoli. Adattato da http://ctrgenpath.net/static/atlas/mousehistology/Windows/femaleu/mousemammgldiagram.html e http://www.pathbase.net/Necropsy_of_the_Mouse/index.php?file=Chapter_3.html. Cliccate qui per visualizzare una versione più grande di questa figura.
-together.within-page = "always"> 
Figura 5. Individuazione delle cellule epiteliali luminali e cellule mioepiteliali basali nella ghiandola mammaria del mouse. MESCs Luminal e cellule mioepiteliali sono identificati dalla IIF nella ghiandola mammaria del mouse al giorno 10 di lattazione, in base alla loro espressione di CK-8 e CK-14 , rispettivamente. DNA nucleare era macchiato con DAPI (blu). Le immagini sono state acquisite con un microscopio a epifluorescenza convenzionali. Il quadro composito (merge) mostra la sovrapposizione di etichettatura corrispondente alle caseine (rosso) e nuclei (blu), rispettivamente. -Ig1, Controllo negativo senza anticorpo primario. Gli asterischi indicano lumen. Scala bar = 100 micron. Cliccate qui per vedere una versione più grande di questa figura.
e 6 "src =" / files / ftp_upload / 53179 / 53179fig6.jpg "/>
Figura 6. Localizzazione cellulare della caseina nella ghiandola mammaria del mouse. Le caseine sono rilevati da IIF nella ghiandola mammaria del mouse al giorno 10 di lattazione. La ghiandola mammaria è stato raccolto dalle femmine in presenza (+ p) o in assenza (-p) di cuccioli. Le immagini sono state acquisite con un convenzionale (a destra del pannello, caseine, nuclei e si fondono) o confocale (caseine (rosso), pannello di sinistra) microscopio a fluorescenza. In entrambe le condizioni, caseine (rosso) vengono rilevati nella regione apicale (frecce) e più o meno al basale MESCs (frecce). I controlli negativi senza anticorpi primari non mostrano alcuna etichettatura (-Ig1). DNA nucleare è macchiata con DAPI (blu). Il quadro composito (merge) mostra la sovrapposizione di etichettatura corrispondente alle caseine (rosso) e nuclei (blu), rispettivamente. Gli asterischi indicano lumen. Barra di scala = 100 micron per le immagini epifluorescenza (pannello di destra, caseine, nuclei, si fondono) e = 10 &# 181;. M per le immagini confocale (colonna di sinistra) Cliccate qui per vedere una versione più grande di questa figura.
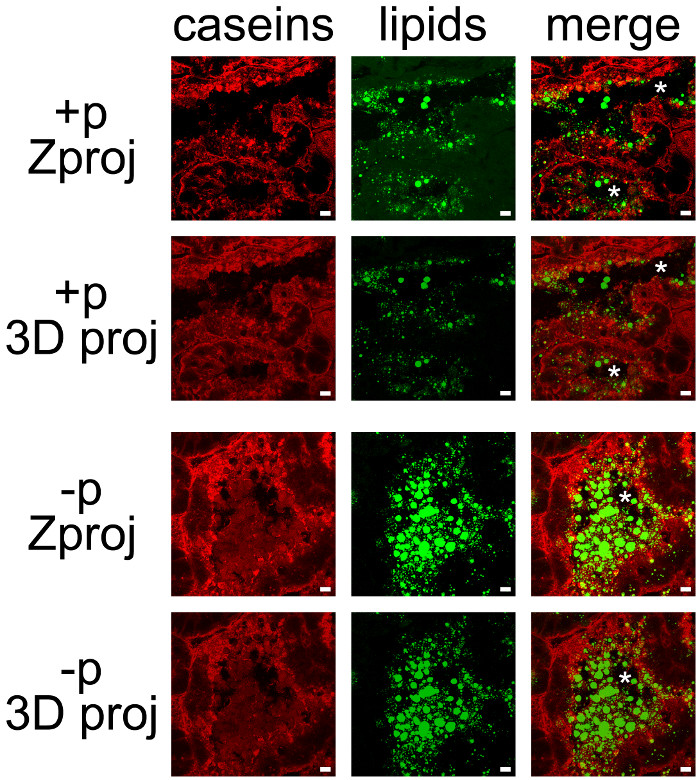
Figura 7. Localizzazione cellulare di prodotti lattiero-caseari nella ghiandola mammaria del mouse. Caseine (rosso) vengono rilevati dalla IIF nella ghiandola mammaria del mouse al giorno 10 di lattazione in presenza (+ p) o in assenza (-p) di cuccioli. Lipidi neutri (CLD e MFGs) sono di contrasto con BODIPY 493/503 (verde). Le immagini composite (unione) mostrano la sovrapposizione dei due etichettature. Le immagini sono state acquisite come Z-stack con un microscopio confocale. Z-stack sono stati post-trattati con ImageJ per generare proiezioni Z (Zproj) o proiezioni in 3D (asse y) (proj 3D) dell'intero stack in ogni canale per entrambi (merge). Gli asterischi indicano lumen. Barra di scala= 10 micron. Cliccate qui per vedere una versione più grande di questa figura.

Figura 8. Localizzazione cellulare di butirofilina e lipidi nella ghiandola mammaria del mouse. BTN1 (rosso) viene rilevato da IIF in ghiandola mammaria mouse giorno 10 di lattazione in assenza dei cuccioli. Lipidi neutri (CLD e MFGs) e DNA nucleare sono di contrasto con BODIPY 493/503 (verde) e DAPI (blu), rispettivamente. Le immagini sono state acquisite con un microscopio confocale come immagine Z-stack. I risultati sono mostrati come una singola immagine estratta dalla pila di immagini (immagine, BTN1, lipidi, nuclei e si fondono) o dopo il trattamento con ImageJ per generare una vista 3D (asse y) di tutta la pila di immagini (proj 3D, BTN1 , lipidi, nuclei, si fondono). Le immagini composite (unione) mostrano lasovrapposizione dei tre canali di colore. -Ig1, Controllo negativo senza anticorpo primario. Gli asterischi indicano lumen. Barra di scala = 10 micron. Cliccate qui per vedere una versione più grande di questa figura.

Figura 9. Localizzazione cellulare di due proteine SNARE nel topo ghiandola mammaria. Syntaxin 6 (Stx6) e VAMP4 (V4) vengono rilevati dalla IIF nella ghiandola mammaria del mouse al giorno 10 di lattazione. Le immagini sono state acquisite con un convenzionale (conv) epifluorescenza o un confocale (LSM) microscopio. Le immagini composite (unione) mostrano la sovrapposizione delle etichette osservata per ogni proteina SNARE (rosso) e per il DNA nucleare contrastate con DAPI (falsi colori verde), rispettivamente. -Ig1, Controllo negativo senza anticorpo primario. Asteriscos indicano lumen. Scala bar = 10 micron per le immagini confocale e = 100 micron per le immagini epifluorescenza. Cliccate qui per vedere una versione più grande di questa figura.


Tabella 1. Immunoistochimica guida alla risoluzione.

Film complementare 1. Localizzazione del butirofilina nella ghiandola mammaria del mouse. BTN1 (rosso) viene rilevato da IIF nella ghiandola mammaria del mouse al giorno 10 di lattazione. Le immagini sono state acquisite con un co microscopio nfocal come Z-stack e post-trattato con ImageJ per generare un film. Lo Z-stack viene letto dall'alto verso il basso. Clicca qui per vedere questo video.

Film complementare 2. Localizzazione di butirofilina e lipidi neutri nella ghiandola mammaria del mouse. BTN1 (rosso) viene rilevato da IIF in ghiandola mammaria del mouse a giorno 10 di lattazione. Lipidi neutri (CLD e MFGs) e DNA nucleare sono di contrasto con BODIPY 493/503 (verde) e DAPI (blu), rispettivamente. Immagini sono state acquisite con un microscopio confocale come Z-stack per ciascun canale di colore e sono stati post-trattati con ImageJ per generare un Z-stack composita che sovrappone i tre canali di colore. La risultante Z-stack composito viene letto dall'alto verso il basso.https://www.jove.com/files/ftp_upload/53179/supvid2.mp4 "target =" _ blank "> Clicca qui per vedere questo video.
Film complementare 3. La localizzazione del butirofilina nella ghiandola mammaria del mouse. BTN1 (rosso) viene rilevato da IIF nella ghiandola mammaria del mouse al giorno 10 di lattazione. Le immagini sono state acquisite con un microscopio confocale come Z-stack e post-trattati con ImageJ (proiezione 3D) per generare una rotazione (asse y) vista spaziale del etichettatura BTN1. Cliccate qui per vedere il video.

Film complementare 4. La localizzazione di butirofilina e neutri lipidi nel topo maghiandola mmary. BTN1 (rosso) viene rilevato da IIF nella ghiandola mammaria del mouse al giorno 10 di lattazione. Lipidi neutri (CLD e MFGs) e DNA nucleare sono di contrasto con BODIPY 493/503 (verde) e DAPI (blu), rispettivamente. Immagini sono state acquisite con un microscopio confocale come Z-stack per ciascun canale di colore e sono stati post-trattati con ImageJ per generare un Z-stack composita che sovrappone i tre canali di colore. ImageJ (proiezione 3D) è stato ulteriormente utilizzato per generare una rotazione (asse y) vista spaziale del Z-stack composito. Cliccate qui per vedere il video.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
IHC è un metodo relativamente semplice e diretto sperimentale di localizzare l'antigene in sezioni di tessuto, che dipende principalmente sulle specifiche interazioni epitopi-anticorpo. Anche se un gran numero di protocolli sono utilizzati per localizzare una proteina IIF, il nucleo di queste procedure è quasi sempre la stessa. Tuttavia, vi sono alcuni aspetti critici che possono influenzare fortemente il risultato e deve quindi essere ottimizzati per ciascuno studio individuale IHC. L'aspetto più impegnativo di questo approccio consiste nel determinare le migliori condizioni sperimentali, cioè., Quelli generando un segnale forte e specifico per l'antigene di interesse. Le variabili che devono essere considerate per la progettazione sperimentale e l'ottimizzazione sono: (1) il tipo di antigene (specie, livelli di espressione, localizzazione subcellulare); (2) il tipo epitopo (sequenza, la conformazione, putativi modificazioni post-traduzionali); (3) preparazione dei campioni (inclusione in paraffina o per sezioni congelate); (4) il fissaggio method (perfusione o immersione); (5) il fissativo utilizzato (formaldeide, alcool o acetone); (6) il reagente bloccante utilizzato (siero normale, BSA o latte non grasso); (7) il passo AR; (8) il metodo di rilevamento (diretto o indiretto); (8) il tipo di anticorpo primario (monoclonale o policlonale); (9) l'anticorpo secondario (specie e etichetta); (10) controcolorazioni (nucleare e o altro etichettatura / compartimento cellulare); e (11), il mezzo di montaggio (vedi Tabella 1 per i dettagli). La fissazione e la procedura di blocco, almeno, richiedono l'ottimizzazione di ulteriori fattori quali la concentrazione, pH, temperatura, tempo di incubazione e diluente.
Il primo aspetto cruciale riguarda la preparazione dei campioni di tessuto, che è strettamente legata al metodo di fissazione, che a sua volta influenza la qualità dei risultati. Ad esempio, pezzi di tessuto possono essere fissi o meno prima incorporamento. Questo passaggio può anche dipendere dal metodo di inserimento prescelto, vale a dire, OCT vs. paraffina, Che si volte dipende l'anticorpo primario utilizzato. Fissaggio del tessuto può essere eseguita in vivo mediante perfusione un animale anestetizzato con una soluzione fissativo. Questo metodo è utile per conservare gli antigeni studiando tessuti intatti ma potrebbe non essere sufficiente a fissare il tessuto di interesse. In questo caso, piccoli pezzi di tessuto (non più spesso di 10 mm) possono essere immersi nella soluzione fissativo. Tessuto congelato può essere preparato immergendo il tessuto in azoto liquido o isopentano e snap-congelamento è altamente raccomandato per la successiva rilevazione di modificazioni post-traduzionali come fosforilazione. Tuttavia, a differenza dei tessuti inclusi in paraffina, congelamento non è adeguata per la conservazione a lungo termine dei tessuti a causa della formazione di cristalli di ghiaccio all'interno delle cellule che possono alterare la morfologia subcellulare. Una volta tagliato, sezioni di tessuto congelato possono essere conservati a -80 ° C fino a 1 anno. In ogni caso, la preparazione dei campioni di tessuto è un compromesso tra il mantenimento del tessuto/ architettura cellulare e preservare l'integrità epitopo.
Dal momento che altera la composizione chimica dei tessuti, è fondamentale per ottimizzare le condizioni di fissaggio per evitare sia incompleta (sotto di fissazione) ed eccessiva (overfixation) di fissazione.
Infatti, underfixation può ridurre il segnale specifico promuovendo la degradazione proteolitica di alcuni antigeni. D'altra parte, può alterare overfixation dell'etichettatura specifica mascherando l'epitopo o generare un forte background non specifico. Così, oltre alla scelta della soluzione fissativo, altri parametri come il tempo di incubazione, la temperatura ed il pH influiscono fissaggio del tessuto. Sebbene PFA è il fissativo più utilizzato per IHC, esso non può essere considerato come un fissativo "universale". PFA induce proteina-proteina e proteina-acido nucleico legami incrociati e può quindi artificiosamente modificare l'epitopo (overfixation) e quindi impedire la sua recoriconosci- dal anticorpo primario. Tuttavia, l'epitopo può essere ulteriormente smascherato mediante tecniche AR (vedi sotto). PFA può anche essere adatta per il rilevamento di determinati antigeni, come è stato mostrato di indurre la traslocazione di alcune proteine fosforilate dalla membrana al citoplasma. In tali casi, PFA deve essere sostituito da opportuni fissativi alternativi come l'alcool. Diversamente PFA, alcoli come metanolo o etanolo non mascherare epitopi quanto consentono il fissaggio del tessuto sostituendo molecole d'acqua nei tessuti. Questo può portare alla precipitazione delle proteine e quindi impedire l'interazione anticorpo / epitopo causa di cambiamenti conformazionali. È opinione diffusa che alcoli non penetrano e pertanto non mantengono morfologia tissutale e PFA. L'acetone è un altro fissativo alternativa, che è comunemente usato quando si lavora con, sezioni di tessuto snap-congelati non fissati. Tuttavia, l'acetone è un forte agente disidratante e può portare alla precipitazione irreversibile delle proteine dei tessuti.
Per alcuni antigeni, un ulteriore passaggio di AR può essere richiesto per ottenere un buon segnale, soprattutto se il fissativo induce il cambiamento conformazionale o altera la carica elettrostatica dell'epitopo (mascheramento dell'epitopo). Metodi AR mirano a invertire questi processi per ripristinare l'immunoreattività dell'epitopo e la successiva interazione con l'anticorpo primario. Metodi AR basano principalmente su due approcci: (1) proteasi indotta recupero epitopo, cioè con enzimi come proteinasi K, tripsina o pepsina, che unirà peptidi che maschera l'epitopo; e (2) il recupero epitopi-calore indotto, vale a dire, con un forno a microonde, pentole a pressione, vapore verdura, autoclavi o bagni di acqua. Quest'ultimo approccio è particolarmente tempo-, a temperatura, buffering e pH-sensibile, e le condizioni ottimali deve essere determinato empiricamente (un esempio è fornito nella sezione Protocol). In alternativa, l'affinità di un anticorpo per il suo antigene può essere miglioratamodificando il pH o la concentrazione di cationi del diluente dell'anticorpo.
Un passo permeabilizzazione a volte è necessario per ottenere un buon segnale per un epitopo intracellulare nelle sezioni di tessuto di spessore, in particolare per l'antigene colorazione nucleare. Ciò può essere ottenuto in vari modi mediante: (1) alcoli o acetone come fissativi; o (2) detergenti come Triton, NP-40 (0,1-0,2% in PBS, 10 min), digitonina, saponina o Tween 20 (0,2-0,5% per 10 a 30 minuti) dopo fissazione PFA. Tuttavia, la scelta del detergente dipende dalla localizzazione cellulare dell'epitopo rilevato. Infatti, detergenti aggressivi, come Triton-X100, che solubilizzano membrane cellulari, sono adatti per il rilevamento di epitopi nucleare, ma può portare a segnalare alterazione su l'estrazione di alcune proteine membranose. L'uso di detergenti lievi (saponina e Tween 20) sono più adatti per la rivelazione di epitopi citoplasmatici.
Il secondo passo fondamentale è la blocking di colorazione aspecifica. Il legame di un anticorpo al suo epitopo bersaglio è governata da forze intermolecolari (interazioni esempio, idrofobiche e ionici, legame a idrogeno). Così, interazioni di anticorpi primari e / o secondari con altre proteine rispetto ai loro antigeni bersaglio possono provocare colorazione aspecifica. Questo genera sfondo elevata fluorescenza, che impedisce la visualizzazione della proteina di interesse (basso rapporto segnale / rumore). Blocco reagenti riduce le interazioni non specifiche senza compromettere specifica interazione anticorpo / epitopo. Una procedura comune consiste di incubazione sezioni di tessuto con siero normale inattivato al calore o di BSA. Quando si utilizza un siero normale, deve essere della stessa specie di quella del animale ospite dell'anticorpo secondario o da una specie non correlate. In tutti i casi, il reagente blocco selezionato deve essere aggiunta ai diluenti per gli anticorpi primari e secondari. Inoltre, l'uso di detergenti non ionici come Triton X-100, Tween 20 o saponina aiuta a ridurre le interazioni non specifiche.
Il terzo e probabilmente più importante parametro è la selezione anticorpo primario e ottimizzazione. Ovviamente, la scelta migliore è un anticorpo di alta qualità con una minima reattività crociata. Come anticorpi monoclonali di solito presentano elevata affinità e specificità per un singolo epitopo, sono i migliori strumenti per discriminare un particolare membro di una famiglia di proteine con l'identità di alta sequenza. Tuttavia, l'interazione anticorpo / epitopo può essere compromessa se l'epitopo bersaglio ha perso il suo stato conformazionale nativo o quando l'accesso al epitopo è impedito da interazioni con altre proteine, modificazioni post-traduzionali, temperatura, pH, la fissazione e concentrazione salina. In questi casi, gli anticorpi policlonali sono più adatti come riconoscono epitopi diversi della stessa proteina. Inoltre, essi sono spesso più stabili rispetto agli anticorpi monoclonali in un ampio intervallo di pH e concentrazione salina.Studi preliminari devono definire le condizioni di incubazione appropriate, cioè, lavorando diluizione (anticorpo monoclonale: 5-25 mg / ml, anticorpo policlonale: 1,7-15 mg / ml), il tempo di incubazione, diluenti e temperatura, che deve essere determinato empiricamente per ogni anticorpo primario. Questi parametri devono essere ottimizzati per determinare le condizioni che producono il segnale ottimale con basso rumore di fondo. La specificità di etichettatura è favorita da tempi di incubazione più lunghi a temperature più basse (es., 4 ° C vs RT).
La scelta per eseguire il rilevamento diretto o indiretto spesso dipende dal livello di espressione dell'antigene. Ad esempio, un epitopo altamente espresso può semplicemente essere rilevato con un anticorpo primario fluorocromo coniugato, permettendo così un facile e semplice colorazione multicolore evitando possibili sfondo non specifico dovuto all'utilizzo di un anticorpo secondario. Tuttavia, diretto se maggio genera un segnale basso ad un costo più elevato, e può sometimes essere difficile, l'anticorpo marcato quando non sono disponibili in commercio. Viceversa, IIF è più sensibile per rilevare basse epitopi espressi come il segnale generato è più intenso dovuta all'interazione di almeno due anticorpi secondari marcati (sollevata contro la specie ospite anticorpo primario) con l'anticorpo primario (amplificazione). Inoltre, una vasta gamma di anticorpi secondari coniugati a vari fluorofori sono disponibili in commercio, relativamente poco costoso e qualità controllata. Tuttavia, questo approccio può indurre cross-reattività e richiede quindi di scegliere con attenzione gli anticorpi primari che non sono prodotte nella stessa specie o di differenti isotipi durante l'esecuzione di esperimenti con più di etichettatura. IIF anche richiede a volte passaggi di blocco in più e deve includere controlli negativi sistematici (vedi sotto). Amplificazione può essere ulteriormente ottenuto utilizzando un anticorpo secondario coniugato con biotina e fluorescente avidina o streptavidina (quattro biotins vincolato al molecule). Tuttavia, questo metodo di amplificazione richiede passaggi aggiuntivi per evitare legami non specifici e non può essere adattato per la colorazione di alcuni tessuti (fegato, rene, cuore, cervello, polmone e allattamento ghiandola mammaria) per la presenza di elevati livelli di biotina endogena . Tuttavia, la biotina endogena possono essere bloccati pre-incubazione del campione con avidina e successivamente con biotina prima dell'incubazione con l'anticorpo primario. La scelta dei fluorocromi coniugati, che sono piccole molecole chimiche con la proprietà di emettere luce quando viene eccitato dalla luce di una lunghezza d'onda più corta, dipende principalmente dal tipo di apparecchiature microscopio disponibili.
Se correttamente progettati per limitare sia cross-reattività tra gli anticorpi e di crossover tra le proprietà spettrali dei fluorocromi utilizzati, a base di immunofluorescenza IHC permette la visualizzazione simultanea di più bersagli cellulari.
L'ultima criticapunto relativo esperimenti IHC riguarda i controlli positivi e negativi che devono essere eseguite per sostenere la validità della colorazione, per identificare artefatti sperimentali e per l'accurata interpretazione dei risultati. Alcuni tessuti mostrano sfondo fluorescente alto (denominato autofluorescenza) che potrebbe portare ad una cattiva interpretazione dei risultati. Così, sezioni di tessuto devono essere rispettate sia sotto fluorescenza e in campo chiaro illuminazione prima di iniziare l'esperimento IHC. Un controllo negativo che omette l'anticorpo primario deve sistematicamente essere incluso in ogni esperimento IHC per garantire che un potenziale legame dell'anticorpo secondario non specifico è trascurabile e non oscurare o simile al pattern di colorazione specifica. Un controllo isotipo può essere eseguita quando si lavora con un anticorpo primario monoclonale sostituendolo con un anticorpo non-immune dello stesso isotipo (ad esempio, IgG1, IgG2a, IgG2b, IgM) alla stessa concentrazione. Questo controllo permette di estimate la colorazione aspecifica, che può essere dovuta alle interazioni di anticorpi con il campione. Per dimostrare il legame di un anticorpo al suo antigene specifico, un controllo assorbimento può essere realizzata in due modi pre-incubando l'anticorpo primario: (1) con la sua immunogeno solubile (10: 1 rapporto molare) O / N a 4 ° C ; e (2) con cellule o sezioni di tessuto che esprimono l'epitopo di interesse, ma che differiscono dal tessuto studiato (per esempio, vedere la Figura 4B in 59). In entrambi i casi, il conseguente impoverimento dell'anticorpo primario dovrebbe portare a poca o nessuna colorazione. Un altro tipo di controllo può essere fatto utilizzando un anticorpo primario irrilevante, cioè., Diretto contro un epitopo che presenta una localizzazione cellulare che è diverso dal epitopo di interesse (nucleare vs. citoplasmatica). L'anticorpo irrilevante deve essere dello stesso isotipo e specie dell'anticorpo primario di interesse. Ulteriori controlli per esperimenti IHC possono includere l'uso di campioni da tissUES noti per esprimere (animali transgenici) o meno (animali knock-out), l'epitopo di interesse. Questo può fornire un utile riferimento e contribuire a ottimizzare la procedura IHC.
Una limitazione principale di tecniche IF è che possono essere applicati solo a fisso (morti) e / o di cellule permeabilizzate, sia procedura potenzialmente indurre artefatti. Altre limitazioni di questo approccio sono dovute all'uso di un microscopio per l'osservazione dei campioni. In primo luogo, come la risoluzione ottica di epifluorescenza e confocale microscopi è limitata, ubicazione o co-locazione delle proteine rilevate non dovrebbero essere troppo interpretato. In secondo luogo, photobleaching, vale a dire. dissolvenza dell'intensità di fluorescenza nel tempo quando esposta alla luce, è essenzialmente dovuto alla generazione di specie reattive dell'ossigeno nel campione su eccitazione di fluorescenza che, a sua volta, porta alla distruzione fotochimica del fluoroforo. Photobleaching può essere ridotto: a) mantenere i campioni al riparo dallaluce durante l'esperimento IF e stoccaggio fino alla loro osservazione; b) utilizzando un agente antifade (specie reattive dell'ossigeno spazzini) nel mezzo di montaggio; c) ridurre l'intensità e / o durata della luce di eccitazione; d) aumentare la concentrazione di fluorofori o utilizzando una bassa concentrazione di un fluorocromo con alta efficienza quantica-; ed e) con fluorofori robuste che sono meno inclini a photobleaching (es. Alexa Fluors, Seta Fluors o DyLightFluors). In terzo luogo, autofluorescenza è spesso dovuto alla presenza di coenzimi flavina (FAD e FMN: assorbimento, 450 nm, emissione, 515 nm) e nucleotidi piridinici ridotti (NADH: assorbimento, 340 nm, emissione, 460 nm) in cellule di mammifero. Inoltre, l'uso di aldeidi, particolarmente glutaraldeide, per fissare i campioni, può portare a livelli elevati di autofluorescenza. Questo può essere minimizzato lavando i campioni con 0,1% sodio boroidruro in PBS prima incubazione anticorpo e / o selezionando sonde e filtri ottici che maximize il segnale di fluorescenza rispetto al autofluorescenza. In quarto luogo, la fluorescenza sovrapposizione (anche definito spurgo-through, incrociato o diafonia) è principalmente dovuto alle emissioni proprietà spettrali dei fluorofori come spesso mostrano molto ampie larghezze di banda, diversi profili spettrali asimmetrici, nonché varie lunghezze d'onda di picco di emissione ed il numero di maxima. Fluorescenza sovrapposizione si verifica quando si lavora con diversi fluorofori (etichettatura multicolore) e si caratterizza per l'emissione di un fluoroforo nel canale (filtro) di un'altra fluoroforo. Artefatti Bleed-through devono essere ridotti al minimo in quanto spesso complicano l'interpretazione dei risultati IF, in particolare nel caso di co-localizzazione o studi quantitativi. Come il bilanciamento delle emissioni fluorofori può essere leggermente migliorata solo con il procedimento IF, sanguinare-through è principalmente essere ridotto al momento dell'acquisizione delle immagini utilizzando un filtro ottimizzato set fluorescenza e / o rilevatore fotomoltiplicatore al fine di adeguatamente SEPARmangiato i profili spettrali dei fluorofori. A questo proposito, la microscopia confocale è adatto all'imaging multicolore come consente la differenziazione spettri di emissione di fluorescenza di singole fluorofori indirizzando ciascun segnale di un particolare canale di rilevamento. Inoltre, microscopia confocale permette di regolare il guadagno, fotomoltiplicatore tensione o della potenza del laser per i singoli canali di rilevamento per l'acquisizione sequenziale (solo uno fluoroforo alla volta) di etichettatura. Idealmente, i controlli con etichetta singola devono essere eseguite per quantificare il sanguinare-through ed eventualmente rimuoverlo computazionale. Un controllo senza anticorpi secondari (controllo di sfondo) può essere preparato per impostare i limiti di guadagno e offset del segnale di ciascun canale per l'acquisizione dell'immagine ottimale. Può essere utilizzato anche per l'elaborazione post-acquisizione di sfondo immagine corretta (autofluorescenza).
In conclusione, il metodo descritto fornisce un semplice protocollo standard per facili realizatisu di immunostaining su sezioni ghiandola mammaria. Tuttavia, le principali fasi di un esperimento IHC devono essere ottimizzate per ciascun antigene / anticorpo coppia per visualizzare colorazione specifica e per minimizzare non specifici segnali di fondo. Il metodo descritto comprende anche diversi metodi di base per la post-trattamento della maggior parte delle immagini ottenute. Immunolocalizzazione Basato sulla fluorescenza è un metodo potente con una vasta gamma di applicazioni dalla localizzazione cellulare di un antigene alla diagnosi. Nuovi progressi in questi approcci saranno raggiunti con il futuro sviluppo di nuovi fluorofori, dispositivi di acquisizione e tecniche di microscopia, di immagine precedentemente non osservate i dettagli delle strutture e dei processi biologici.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Gli autori dichiarano di non avere interessi finanziari in competizione.
Acknowledgments
Gli autori sono grati per l'impianto nucleo di imaging INRA MIMA2 (INRA, UMR1198, Jouy-en-Josas) e al personale dell'unità IERP (UE 0907, INRA, Jouy-en-Josas) per la cura degli animali e le strutture. Vorremmo anche ringraziare IH Mather, MC Neville e S. Tooze per averci fornito antibodie molto utile.
Materials
| Name | Company | Catalog Number | Comments |
| Dissection | |||
| Pins | |||
| Ethanol | |||
| Scissors | |||
| Scalpel and adapted blades | |||
| Ice | |||
| Towel paper | |||
| Tissue sample preparation | Company | Catalog Number | Comments/Description |
| Phosphate Buffered Saline (pH7.4) | Sigma | P-3813 | |
| Paraformaldehyde (PFA, 32% EM grade, 100 ml) | Electron Microscopy Sciences | 15714-S | personnal protection equipment required WARNING: this product will expose you to Formaldehyde Gas, a chemical known to cause cancer |
| OCT compound/Tissue Tek | Sakura | 4583 | |
| Sucrose (D-saccharose) | VWR | 27480.294 | |
| Plastic molds | Dominique Dutscher | 39910 | |
| Liquid nitrogen | |||
| Cryostat/sample support | Leica | CM3050S | |
| Razor blades (SEC35) | Thermo Scientific | 152200 | |
| Slide box | |||
| Glass slides Superfrost/Superfrost Ultra Plus | Thermo Scientific | 10143560W90/1014356190 | |
| Brushes | |||
| IHC | Company | Catalog Number | Comments/Description |
| Super Pap Pen | Sigma | Z377821-1EA | |
| Permanent marker (black) | |||
| 50 mM NH4Cl in PBS | Sigma | A-0171 | |
| 0.1 M glycine in PBS | VWR | 24403.367 | |
| Antigen Retrieval solution: Tris 100 mM 5% urea pH9.6 | |||
| Heater (up to 100°C) | |||
| Bovine Serum Albumin (BSA) | Sigma | A7906-100G | |
| Vectashield (anti-fading mounting medium) without DAPI/with DAPI | Vector Laboratories | H-1000/H-1200 | |
| Glass coverslips 22x50mm (microscopy grade) | VWR | CORN2980-225 | |
| Nail polish | |||
| Primary antibodies | Company | Catalog Number | Comments/Description |
| Rabbit anti-mouse caseins (#7781; 1:50 dilution) | generously gifted by M.C. Neville (University of Colorado Health Sciences Center, USA) |
||
| Mouse anti-cytokeratin 8 (CK8, clone 1E8, 1:50 dilution) | Biolegend (Covance) | MMS-162P | |
| Mouse anti-cytokeratin 14 (CK14, cloneLL002, 1:50 dilution) | Thermo Scientific | MS-115-P0/P1 | |
| Rabbit anti-butyrophilin (1:300 dilution) | generously gifted by I.H. Mather (Department of Animal and Avian Sciences University of Maryland College Park, USA) | ||
| Rabbit anti-Stx6 (1:50 dilution) | generously gifted S. Tooze (Cancer Research UK, London Research Institute, London, UK) |
||
| Rabbit anti-VAMP4 (1:50 dilution) | Abcam | ab3348 | |
| Secondary antibodies | Company | Catalog Number | Comments/Description |
| Rhodamine-conjugated goat anti-rabbit IgG (H + L) (1:300 dilution) | Jackson ImmunoResearch Laboratories | 111-025-003 | |
| Counterstains | Company | Catalog Number | Comments/Description |
| Bodipy 493/503 | Life Technologies (Molecular Probes) | D-3922 | |
| DAPI (4-6-diamidino-2-phenylindole) | Life Technologies (Molecular Probes) | D-1306 | |
| Observation/Image capture | Company | Catalog Number | Comments/Description |
| conventional fluorescence microscope | Leica Leitz DMRB microscope |
Standard filters for FITC, Rhodamine and DAPI emissions, ×63 oil-immersion objective (NA 1.3), DP50 imaging camera (Olympus), CellˆF software (Olympus) |
|
| Laser Scanning Microscope (confocal microscopy) | Zeiss LSM 510 microscope |
Plan-Apochromat ×63 oil-immersion objective (NA 1.4), CLSM 510 software, Confocal facilities, MIMA2 Platform, INRA Jouy-en-Josas, France, http://mima2.jouy.inra.fr/mima2) | |
| Image treatment | Company | Catalog Number | Comments/Description |
| ImageJ 1.49k software | Free software |
References
- Watson, C. J., Khaled, W. T. Mammary development in the embryo and adult: a journey of morphogenesis and commitment. Development. , 135-995 (2008).
- Smith, G. H. Experimental mammary epithelial morphogenesis in an in vivo model: evidence for distinct cellular progenitors of the ductal and lobular phenotype. Breast Cancer Res Treat. 39, 21-31 (1996).
- Van Keymeulen, A., et al. Distinct stem cells contribute to mammary gland development and maintenance. Nature. 479, 189-193 (2011).
- Oakes, S. R., Gallego-Ortega, D., Ormandy, C. J. The mammary cellular hierarchy and breast cancer. Cell Mol Life Sci. 71, 4301-4324 (2014).
- Visvader, J. E., Stingl, J. Mammary stem cells and the differentiation hierarchy: current status and perspectives. Genes & development. 28, 1143-1158 (2014).
- Robinson, G. W. Cooperation of signalling pathways in embryonic mammary gland development. Nat Rev Genet. 8, 963-972 (2007).
- Cowin, P., Wysolmerski, J. Molecular mechanisms guiding embryonic mammary gland development. Cold Spring Harb Perspect Biol. 2, a003251 (2010).
- Brisken, C., O'Malley, B. Hormone action in the mammary gland. Cold Spring Harb Perspect Biol. 2, a003178 (2010).
- Gjorevski, N., Nelson, C. M. Integrated morphodynamic signalling of the mammary gland. Nat Rev Mol Cell Biol. 12, 581-593 (2011).
- Daniel, C. W., Smith, G. H. The mammary gland: a model for development. J Mammary Gland Biol Neoplasia. 4, 3-8 (1999).
- Howlett, A. R., Bissell, M. J. The influence of tissue microenvironment (stroma and extracellular matrix) on the development and function of mammary epithelium. Epithelial Cell Biol. 2, 79-89 (1993).
- Edwards, G., Streuli, C. Signalling in extracellular-matrix-mediated control of epithelial cell phenotype. Biochem Soc Trans. 23, 464-468 (1995).
- Hennighausen, L., Robinson, G. W. Think globally, act locally: the making of a mouse mammary gland. Genes & development. 12, 449-455 (1998).
- Topper, Y. J., Freeman, C. S. Multiple hormone interactions in the developmental biology of the mammary gland. Physiol Rev. 60, 1049-1106 (1980).
- Brisken, C., et al. A paracrine role for the epithelial progesterone receptor in mammary gland development. Proc Natl Acad Sci U S A. 95, 5076-5081 (1998).
- Ormandy, C. J., Binart, N., Kelly, P. A. Mammary gland development in prolactin receptor knockout mice. J Mammary Gland Biol Neoplasia. 2, 355-364 (1997).
- Oakes, S. R., Rogers, R. L., Naylor, M. J., Ormandy, C. J. Prolactin regulation of mammary gland development. J Mammary Gland Biol Neoplasia. 13, 13-28 (2008).
- Hennighausen, L., Robinson, G. W. Information networks in the mammary gland. Nat Rev Mol Cell Biol. 6, 715-725 (2005).
- Kouros-Mehr, H., Werb, Z. Candidate regulators of mammary branching morphogenesis identified by genome-wide transcript analysis. Dev Dyn. 235, 3404-3412 (2006).
- Khaled, W. T., et al. The IL-4/IL-13/Stat6 signalling pathway promotes luminal mammary epithelial cell development. Development. 134, 2739-2750 (2007).
- Asselin-Labat, M. L., et al. Gata-3 is an essential regulator of mammary-gland morphogenesis and luminal-cell differentiation. Nat Cell Biol. 9, 201-209 (2007).
- Barcellos-Hoff, M. H., Aggeler, J., Ram, T. G., Bissell, M. J. Functional differentiation and alveolar morphogenesis of primary mammary cultures on reconstituted basement membrane. Development. 105, 223-235 (1989).
- Robinson, G. W., McKnight, R. A., Smith, G. H., Hennighausen, L. Mammary epithelial cells undergo secretory differentiation in cycling virgins but require pregnancy for the establishment of terminal differentiation. Development. 121, 2079-2090 (1995).
- Streuli, C. H., Bissell, M. J. Mammary epithelial cells, extracellular matrix, and gene expression. Cancer Treat Res. 53, 365-381 (1991).
- Streuli, C. H., et al. Laminin mediates tissue-specific gene expression in mammary epithelia. J Cell Biol. 129, 591-603 (1995).
- Boudreau, N., Sympson, C. J., Werb, Z., Bissell, M. J. Suppression of ICE and apoptosis in mammary epithelial cells by extracellular matrix. Science. 267, 891-893 (1995).
- Pullan, S., et al. Requirement of basement membrane for the suppression of programmed cell death in mammary epithelium. J Cell Sci. 109 (Pt 3), 631-642 (1996).
- Schmidhauser, C., Bissell, M. J., Myers, C. A., Casperson, G. F. Extracellular matrix and hormones transcriptionally regulate bovine beta-casein 5' sequences in stably transfected mouse mammary cells. Proc Natl Acad Sci U S A. 87, 9118-9122 (1990).
- Streuli, C. H., et al. Stat5 as a target for regulation by extracellular matrix. J Biol Chem. 270, 21639-21644 (1995).
- Sollner, T., et al. SNAP receptors implicated in vesicle targeting and fusion. Nature. 362, 318-324 (1993).
- Jahn, R., Scheller, R. H. SNAREs--engines for membrane fusion. Nat Rev Mol Cell Biol. 7, 631-643 (2006).
- Weber, T., et al. SNAREpins: minimal machinery for membrane fusion. Cell. 92, 759-772 (1998).
- Sollner, T., Bennett, M. K., Whiteheart, S. W., Scheller, R. H., Rothman, J. E. A protein assembly-disassembly pathway in vitro that may correspond to sequential steps of synaptic vesicle docking, activation, and fusion. Cell. 75, 409-418 (1993).
- McNew, J. A. Regulation of SNARE-mediated membrane fusion during exocytosis. Chem Rev. 108, 1669-1686 (2008).
- Wang, C. C., et al. VAMP8/endobrevin as a general vesicular SNARE for regulated exocytosis of the exocrine system. Mol Biol Cell. 18, 1056-1063 (2007).
- Chat, S., et al. Characterisation of the potential SNARE proteins relevant to milk product release by mouse mammary epithelial cells. Eur J Cell Biol. 90, 401-413 (2011).
- Reinhardt, T. A., Lippolis, J. D. Bovine milk fat globule membrane proteome. J Dairy Res. 73, 406-416 (2006).
- Robenek, H., et al. Butyrophilin controls milk fat globule secretion. Proc Natl Acad Sci U S A. 103, 10385-10390 (2006).
- Fujimoto, T., Ohsaki, Y., Cheng, J., Suzuki, M., Shinohara, Y. Lipid droplets: a classic organelle with new outfits. Histochem Cell Biol. 130, 263-279 (2008).
- Mather, I. H., Keenan, T. W. Origin and secretion of milk lipids. J Mammary Gland Biol Neoplasia. 3, 259-273 (1998).
- Heid, H. W., Keenan, T. W. Intracellular origin and secretion of milk fat globules. Eur J Cell Biol. 84, 245-258 (2005).
- McManaman, J. L., Russell, T. D., Schaack, J., Orlicky, D. J., Robenek, H. Molecular determinants of milk lipid secretion. J Mammary Gland Biol Neoplasia. 12, 259-268 (2007).
- de Assis, S., Warri, A., Cruz, M. I., Hilakivi-Clarke, L. Changes in mammary gland morphology and breast cancer risk in rats. Journal of visualized experiments : JoVE. , (2010).
- Plante, I., Stewart, M. K., Laird, D. W. Evaluation of mammary gland development and function in mouse models. Journal of visualized experiments : JoVE. , (2011).
- Galio, L., et al. MicroRNA in the ovine mammary gland during early pregnancy: spatial and temporal expression of miR-21, miR-205, and miR-200. Physiol Genomics. 45, 151-161 (2013).
- Linzell, J. L., Peaker, M. The effects of oxytocin and milk removal on milk secretion in the goat. J Physiol. 216, 717-734 (1971).
- Knight, C. H., Peaker, M., Wilde, C. J. Local control of mammary development and function. Rev Reprod. 3, 104-112 (1998).
- Walid, M. S., Osborne, T. J., Robinson, J. S. Primary brain sarcoma or metastatic carcinoma? Indian J Cancer. 46, 174-175 (2009).
- Hue-Beauvais, C., et al. Localisation of caveolin in mammary tissue depends on cell type. Cell Tissue Res. 328, 521-536 (2007).
- McManaman, J. L., Neville, M. C. Mammary physiology and milk secretion. Adv Drug Deliv Rev. 55, 629-641 (2003).
- Mather, I. H., Jack, L. J. A review of the molecular and cellular biology of butyrophilin, the major protein of bovine milk fat globule membrane. J Dairy Sci. 76, 3832-3850 (1993).
- Heid, H. W., Winter, S., Bruder, G., Keenan, T. W., Jarasch, E. D. Butyrophilin, an apical plasma membrane-associated glycoprotein characteristic of lactating mammary glands of diverse species. Biochim Biophys Acta. 728, 228-238 (1983).
- Bock, J. B., Klumperman, J., Davanger, S., Scheller, R. H. Syntaxin 6 functions in trans-Golgi network vesicle trafficking. Mol Biol Cell. 8, 1261-1271 (1997).
- Tran, T. H., Zeng, Q., Hong, W. VAMP4 cycles from the cell surface to the trans-Golgi network via sorting and recycling endosomes. J Cell Sci. 120, 1028-1041 (2007).
- Wendler, F., Page, L., Urbe, S., Tooze, S. A. Homotypic fusion of immature secretory granules during maturation requires syntaxin 6. Mol Biol Cell. 12, 1699-1709 (2001).
- Wendler, F., Tooze, S. Syntaxin 6: the promiscuous behaviour of a SNARE protein. Traffic. 2, 606-611 (2001).
- Shitara, A., et al. VAMP4 is required to maintain the ribbon structure of the Golgi apparatus. Mol Cell Biochem. 380, 11-21 (2013).
- Thibault, C., Levasseur, M. C. La reproduction chez les mammifères et l'homme. , INRA Editions. 928 (2001).
- Truchet, S., Wietzerbin, J., Debey, P. Mouse oocytes and preimplantation embryos bear the two sub-units of interferon-gamma receptor. Mol Reprod Dev. 60, 319-330 (2001).



