Introduction
Antilichamen kunnen vouwen en functioneren in de intracellulaire omgeving zijn veelbelovend gereedschap voor zowel onderzoek en therapeutische toepassingen. Ze hebben het vermogen om eiwitten te moduleren door te binden aan een doeleiwit in cellen voorkomen eiwit-eiwit interacties verstoren eiwit-nucleïnezuur interacties of substraattoegang enzymen 1-5 voorkomen.
Hoewel antilichamen veel mogelijkheden voor intracellulaire toepassingen, engineering van hen om juiste vouwing en oplosbaarheid in de intracellulaire omgeving, terwijl de mogelijkheid om te binden aan een doelwit antigeen uitdaging. De reducerende cytoplasmatische omgeving voorkomt de vorming van de disulfide bindingen normaliter voor de stabiele vouwen van full-length antilichamen en antilichaamfragmenten, waaronder enkele keten variabel fragment (scFv) antilichamen 6,7. Een aantal gerichte evolutie benaderingen zijn gebruikt om antilichamen met h engineeroge affiniteiten voor target antigenen 8-10. Deze benaderingen vaak gebruikt faagdisplay, gist oppervlakte-of bacteriële oppervlakte-grote libraries van antilichamen 11-13 screenen. Deze werkwijzen zijn krachtig en effectief voor het identificeren van antilichamen die binden aan doelen, maar ze zijn afhankelijk van de secretieroute eiwitten die wordt weergegeven 14-16 vervoeren. De secretieroute translocatie ongevouwen eiwitten in het cytoplasma vermindering in het endoplasmatisch reticulum lumen in gist of in het periplasma van bacteriën. De eiwitten vouw onder oxiderende omstandigheden en worden weergegeven op het celoppervlak of verpakt in faagdeeltjes te screenen op bindingsaffiniteit 17,18. Hierdoor antilichamen die middels deze technieken niet noodzakelijkerwijs goed vouwen in de cytoplasma en intracellulaire oplosbaarheid moeten vaak afzonderlijk worden gemanipuleerd wanneer de antilichamen worden gebruikt voor intracellulaire toepassingen.
Verbeterende doeltreffendheid van technische antilichamen die goed gevouwen in het cytoplasma, we eerder gemeld het succes van MAD-TRAP (membraan-verankerde display Tat-gebaseerde herkenning associëren eiwitten), een werkwijze voor het screenen van een antilichaam scFv bibliotheek die Escherichia coli binnen- membraan-display 19. Bacteriële binnenste membraan weergave afhankelijk van de twin-arginine translocatie (Tat) pathway voor het transporteren van geëxposeerde antilichamen, in tegenstelling tot andere gemeenschappelijke weergavemethoden de secretieroute gebruiken. De Tat pathway bevat een mechanisme voor kwaliteitscontrole waardoor slechts oplosbaar, correct gevouwen eiwitten worden vervoerd van de E. coli cytoplasma, over het binnenste membraan, en in het periplasma 20,21. Overexpressie Tat substraten (bijv., Eiwitten gericht op de Tat pathway met een N-eindstandige fusie met het Tat signaalpeptide ssTorA) die goed gevouwen in het cytoplasma vormen een langlevende translocatie tussenproduct de N-terminus in het cytoplasma en de C-terminus in het periplasma 19. Dit maakt weergave van correct gevouwen Tat substraten, waaronder antilichaamfragmenten, aan de periplasmatische zijde van het E. coli binnenste membraan. Na verwijdering van het buitenmembraan door enzymatische digestie sferoplasten produceren worden antilichamen blootgesteld aan de extracellulaire ruimte (figuur 1). Hierdoor kan Tat substraten op het binnenmembraan worden gescreend op binding aan een specifiek doel. Belangrijker is het benutten van de Tat pathway van celoppervlak scherm zorgt ervoor dat alleen de antilichamen in de bibliotheek die goed gevouwen in het cytoplasma wordt ondervraagd voor binding, het gelijktijdig engineering van bindingsaffiniteit en intracellulaire vouwen. In dit protocol beschrijven we hoe u een scFv-bibliotheek op de E. tonen coli binnenste membraan, pan de bibliotheek tegen een doelantigeen, en het uitvoeren van een tweede scherm om de meest veelbelovende bestanddelen van de bibliotheek te identificeren. Terwijl we ons richten het protocol inzake scFvs, kan de methode worden toegepast op engineering van een eiwit waarvan de toepassing vereist binding en intracellulaire vouwen.
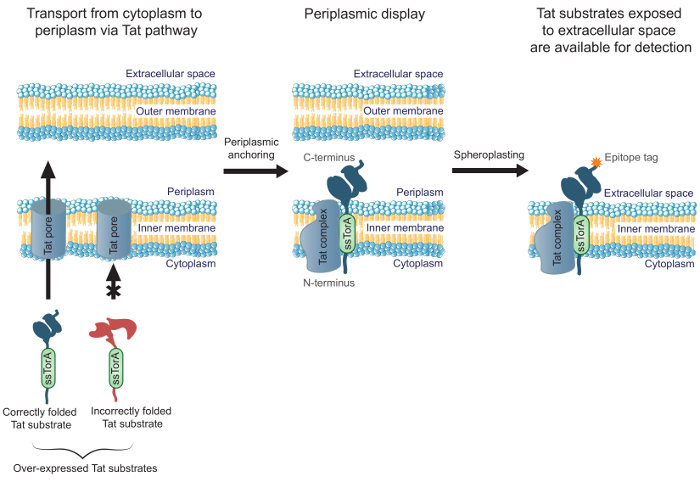
Figuur 1. Tat binnenste membraan-display. In E. coli, scFv antilichamen die tot expressie worden gebracht als een fusie met het ssTorA signaalsequentie correct gevouwen en in het cytoplasma getransporteerd over het binnenmembraan. Een translocatie tussenvormen, waarbij de scFvs verankerd in de binnenmembraan van de N-terminus in het cytoplasma en de C-terminus in het periplasma. E. coli buitenmembraan enzymatisch gedigereerd sferoplasten vormen, waarbij het verankerde antilichamen blootgesteld aan de extracellulaire ruimte en beschikbaar voor detectie door toepassing van een antilichaam dat bindt aan de C-terminaal gesmolten epitooplabel het geëxposeerd antilichaam.load / 54583 / 54583fig1large.jpg "target =" _ blank "> Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
1. Bereid de scFv bibliotheek als een fusie met het ssTorA Signal Sequence
- Zorg voor een deoxyribonucleïnezuur (DNA) bibliotheek met varianten van een scFv-gen.
OPMERKING: De bibliotheek kan ook gemaakt worden met behulp van een geschikte wijze om diversiteit in het gehele scFv gen of gerichte domeinen genereren 22 (bijvoorbeeld het derde complementariteitsbepalende gebieden, CDR3.). - Plaats de DNA-bibliotheek in de EMB plasmide (figuur 2) onder toepassing van standaard moleculaire kloneringswerkwijzen 23.
Opmerking: Dit plasmide brengt scFvs als een genetische fusie met het ssTorA signaalsequentie (N-terminaal scFv) en de FLAG-epitoop-tag (C-terminal naar het scFv). Het ontwerp van het plasmide voor binnenmembraan weergave is eerder 19 beschreven. De EMB plasmide is verkrijgbaar bij de auteurs.

Figuur 2. Binnen membraan weergave plasmide (EMB) kaart (stap 1,2 tot 1,3). Dit plasmide bevat een lac-promotor, oorsprong van replicatie ColE1 en een chloramfenicol resistentiegen. De ingevoegde scFv gen wordt gefuseerd aan het ssTorA signaalsequentie de scFv richten op de Tat pathway en een FLAG epitoop tag, met alle drie in hetzelfde leesraam. Restrictie-enzymplaatsen worden aangegeven. Een bibliotheek ingevoegd tussen de Xbal en Notl restrictie-enzymplaatsen, de grootte van het plasmide 2219 bp plus de grootte van het scFv. Klik hier om een grotere versie van deze figuur te zien.
- Transformeer het plasmide DNA dat de bibliotheek in MC4100 E. coli cellen 23. Herstellen en te groeien deze bacteriële vorm van de bibliotheek. Centrifugeer bij 4000 xg gedurende 15 minuten bij kamertemperatuur om de cellen te verzamelen. Verwijder het supernatant en resuspendeer de verzamelde cellenin 25% glycerol in Luria-Bertani (LB) media. Store fracties bij -80 ° C totdat het nodig is, of gaat u naar stap 2.
LET OP: Het protocol is geverifieerd met MC4100 cellen, hoewel andere E. coli-stammen wordt ook verwacht dat compatibel is met het protocol te zijn. Elektroporatie is de voorkeurswerkwijze voor transformatie, vanwege de hoge transformatie-efficiëntie. De bibliotheek zal kenmerkend bestaan uit ten minste 10 9 scFv varianten in dit stadium en elk aliquot moet voldoende cellen bevatten, die de bibliotheek onder 100-voudig.
2. Druk de Bibliotheek en Bereid Sferoplasten
- Dooi een hoeveelheid van de bacteriële bibliotheek (van stap 1,3) bij kamertemperatuur en voeg het monster aan een kolf die 100 ml LB medium met 20 ug / ml chlooramfenicol (Cm). Groeien 3 uur bij 37 ° C en 225 rpm in een schudder geïncubeerd.
- Na 3 uur, verwijder de kolf uit de 37 ° C geïncubeerd shaker. Sta expressie van het scFv bibliotheekdoorgaan O / N voor 15 tot 22 uur bij 20 ° C en 225 rpm in een schudder geïncubeerd.
LET OP: geen inductor is nodig bij gebruik van de EMB plasmide, als promotor is lekkende. Merk op dat MC4100 cellen niet overexpressie het Lac repressor (en Lacl wordt niet gevonden op het plasmide).

Figuur 3. E.coli cellen en sferoplasten. (A) E. coli cellen zijn cilindrisch van vorm. (B) Na spheroplasting gebruik EDTA en lysozym, het buitenmembraan van E. coli cellen bezwijkt en de resulterende sferoplasten zijn bolvormig. Differentiële interferentie contrast (DIC) microscopie beelden werden verkregen met een 100x objectief op een omgekeerde microscoop. Zoom clik hier om een grotere versie van deze figuur te bekijken.
- Bereid de bibliotheek sferoplasten.
OPMERKING: Sferoplasten worden gevormd door het scheuren van de buitenmembraan van E. coli en zijn bolvormig (Figuur 3).- Bereid de benodigde buffers.
OPMERKING: Alle buffers moeten steriel zijn.- Bereid 1 x met fosfaat gebufferde zoutoplossing (PBS, pH 7,4) door het oplossen van 8 g NaCl, 0,2 g KCl, 1,44 g Na 2 HPO 4 en 0,24 g KH 2PO 4 in gedestilleerd H 2 O tot een eindvolume van 1000 ml. Houd op het ijs.
- Bereid PBS met 0,1% (w / v) bovine serum albumine (BSA) door het oplossen van 0,2 g BSA in 200 ml 1 x PBS. Houd op het ijs.
- Bereid de fractionering buffer (FB) door mengen van 7,5 ml steriel gefiltreerde 1 M sucrose, 1 ml 1 M Tris buffer (pH 8,0) en 1,5 ml gedestilleerd H 2 O. Houd op het ijs.
- Bereid 1 mM ethyleendiaminetetraazijnzuur (EDTA) door toevoeging van 30 ul of 0,5 M EDTA tot 14,97 ml gedestilleerd H 2 O.
- Bereid 0,5 M MgCl2 door oplossen 4,76 g MgCl 2 in 100 ml gedestilleerd H 2 O. Houd op het ijs.
- Neem de kolf uit de schudinrichting en meet de optische dichtheid (OD) bij 600 nm met een spectrofotometer om de celdichtheid te bepalen. Bereken de hoeveelheid geïnduceerde kweek nodig zodat elk monster spheroplasting heeft 1 x 10 10 cellen.
OPMERKING: De benadering van een OD 600 van 1 wijst op een concentratie van 10 9 cellen / ml voor E. coli kan worden gebruikt 24. - Centrifugeer de berekende hoeveelheid geïnduceerde kweek in een 1,5 ml microcentrifuge buis bij 12.000 x g bij kamertemperatuur gedurende 5 minuten. Maak minstens twee monsters indien een vraagstuk met monstervoorbereiding.
- Verwijder het supernatant van de kweken gecentrifugeerd en resuspendeer elke cel pellet in 100 ul ijskoude FB. Centrifugeer bij 12.000 xg bij kamertemperatuur gedurende 1 minuut en verwijder het supernatant door pipetteren. Resuspendeer elk pellet in 350 pl ijskoude FB aangevuld met 3,5 pl van 10 mg / ml lysozym.
- vortex langzaam elke buis onder toevoeging druppelsgewijs 700 ui 1 mM EDTA, en incubeer de buizen bij kamertemperatuur gedurende 20 minuten onder langzaam draaien op een buis rotator om de monsters te mengen. Verwijder de buizen uit de rotator, voeg 50 ul ijskoude 0,5 M MgCl2 aan elke buis en incubeer ze op ijs gedurende 10 min. Centrifugeer de buizen bij 11.000 xg bij 4 ° C gedurende 10 minuten.
- Isoleer de sferoplast pellet.
- Gebruik een micropipet met een tip 1 ml langzaam omhoog te trekken deel van pellet. Terwijl de buis onder een hoek met de opening direct boven een nieuwe 1,5 ml buis, Til de pipetpunt uit de supernatant en schuif de pellet in de nieuwe buis.
- Als een aanzienlijke hoeveelheid supernatant wordt overgebracht naar de nieuwe buis, verwijdert u deze door pipetteren. Als de pellet is niet stevig enough over te dragen, re-centrifuge bij 11.000 xg gedurende 2 minuten en poging pellet isolatie weer.
- Resuspendeer de pellet in sferoplast elke buis 1 ml ijskoude 1 x PBS. Alternate tussen pipetteren en langzaam vortexen op een vortex houder totdat de pellet volledig geresuspendeerd. Niet monsters afhouden van het ijs langer dan 2 minuten per keer, en terugkeren naar de ijs gedurende ten minste 5 minuten voor het verwijderen van het ijs opnieuw. Houd de sferoplasten bij 4 ° C (gedurende maximaal 2 dagen) tot gebruik voor panning in stap 4.
- Bereid de benodigde buffers.
3. Immobiliseer het doelantigeen op Magnetic Beads
- Biotinyleren het doelantigeen in vivo tijdens recombinante productie in E. coli-cellen. U kunt ook gebruik maken van chemische conjugatie 25 of de aankoop doelwitantigeen die al is gebiotinyleerd, en ga verder met stap 3.2.
- Voeg 816 g bicine tot 50 ml water tot 10 × bicine buffer te maken. Verdun de buffer tot 1 x met gedestilleerd H2O en verhit tot 50 ° C. Voeg 14,7 mg biotine aan 12 ml van de verwarmde 1 × bicine buffer tot een biotine oplossing die 5 mM biotine in 10 mM bicine buffer maken. Bewaren bij -20 ° C totdat het nodig is.
- Drukken en biotinyleren het doeleiwit met de pAK400cb-BCCP plasmide 26, die de productie van het doelantigeen maakt als fusie met het biotine carboxyl dragereiwit (BCCP).
LET OP: E. coli cellen native biotinyleren BCCP, waardoor de noodzaak om te zuiveren en chemisch biotinyleren het doeleiwit voorafgaand aan immobilisatie op met streptavidine beklede parels. De inheemse E. coli biotineligase BirA volstaat biotinyleren het fusie-eiwit.- Grow E. coli die het biotinylatie plasmide (met het doelantigeen ingevoegd als een fusie aan het N-uiteinde van BCCP) O / N 15 tot 18 uur in 5 ml LB medium aangevuld met 20 ug / ml Cm bij 37 ° C onder schudden bij 225 rpm.
- Meet de OD bij 600 nm onder toepassing van een spectrofotometer en het berekenen van de hoeveelheid kweek nodig (V add) aan subcultuur in een startende OD van 0,05 in 25 ml vers LB-medium met 20 ug / ml Cm met de vergelijking: voeg = (0,05 x 25 ml) / (OD 600-0,05), waarbij 600 OD de optische dichtheid van de O / N kweek en add V het volume van de O / N kweek toe te voegen aan de verse LB. Subcultuur en groeien tot een OD van 0,5-0,8 per geïncubeerd schudinrichting bij 37 ° C en 225 rpm.
- Add isopropyl β-D-1-thiogalactopyranoside tot een uiteindelijke concentratie van 100 pM en biotine tot een uiteindelijke concentratie van 5 uM. Induceren expressie in een schudinrichting geïncubeerd 15 tot 22 uur bij 20 ° C en 225 rpm.
- Oogst bacteriën door centrifugeren bij 4000 xg bij 4 ° C gedurende 10 minuten. Verwijder het supernatant. Bewaar de pellet bij -20 ° C tot aan gebruik.
- Toevoegen1 ml van een cel lysis detergens per 0,2 g celpellet. Resuspendeer door pipetteren en voorzichtig zwenken gedurende 20 minuten om de cellen te lyseren. Na lysis, centrifugeer bij 16.000 xg en 4 ° C gedurende 20 min. Pipetteer de oplosbare lysaat (supernatans) in een nieuwe 1,5 ml buis.
- Gebruik een 3 kDa moleculair gewicht cutoff kolom om de niet-gebonden biotine verwijderen. Pipetteer het lysaat in de kolom en centrifugeer bij 20 ° C volgens de instructies van de fabrikant. Wassen met 1 x PBS tot het biotine in het lysaat werd 100-voudig verdund en het volume van het gewassen lysaat is gelijk aan het oorspronkelijke volume van het lysaat. Breng het lysaat naar een nieuwe buis.
- Immobiliseer het gebiotinyleerde doelantigeen op met streptavidine beklede magnetische korrels.
- Bereid 1 x PBS en 1 x PBS met 0,1% (w / v) BSA zoals beschreven in stap 2.3.1.
- Bereid de magnetische bolletjes.
Opmerking: Dit vereist het gebruik van een magnetisch scheidingsrek.- Resuspendeer streptavidin-gecoate magnetische korrels in het oorspronkelijke buisje. Hetzij vortex gedurende ten minste 30 seconden of roteren gedurende 5 min.
- Transfer 7-10 x 10 9 kralen aan een 1,5 ml buis.
Opmerking: Het volume dat nodig is afhankelijk van de kraal concentratie door de fabrikant zijn. - Plaats de buis met de kralen op de magneet rek voor 2 minuten om de kralen te verzamelen over de zijkant van de buis. Met de buis nog steeds op de magneet, verwijder voorzichtig het supernatant door pipetteren zonder verstoring van de kralen.
- Te wassen, verwijdert u de tube van de magneet, en resuspendeer de kralen in 1 ml van 1 x PBS door pipetteren zonder het genereren van bubbels. Zet de buis naar de magneet voor 2 min om de kralen te verzamelen, en verwijder voorzichtig de supernatant door pipetteren. Herhaal dit proces nog twee keer voor een totaal van drie wassingen. Zorg ervoor dat er geen vloeistof overblijft in de buis na de laatste wasbeurt.
- Voeg het lysaat met het gebiotinyleerde antigeen aan de magnetische beads.
- Haal de buis uit de magneet en resuspendeer de kralen in 1 ml lysaat (uit stap 3.1.5). Incubeer bij kamertemperatuur gedurende 30 min onder zachtjes draaien.
- Plaats de buis op de magneet gedurende 3 min bij de antigeen beklede parels te verzamelen. Was de beklede korrels vijf maal met 1 x PBS met 0,1% BSA op dezelfde wijze als beschreven in stap 3.2.2.3 tot 3.2.2.4. Na de laatste wasbeurt, resuspendeer de kralen in 1 x PBS met 0,1% BSA tot hetzelfde volume gebruikt in stap 3.2.2.2.
- Als het geïmmobiliseerde doelantigeen stabiel is bij 4 ° C, bewaar de beklede korrels bij 4 ° C totdat het nodig is voor panning. Anders gaat u naar stap 4.
4. Scherm het scFv bibliotheek door panning tegen de Target Antigen (figuur 4)

Figuur 4. Panning (Stap 4). Antigeen-beklede magnetische korrels are geïncubeerd met sferoplasten expressie antilichaambibliotheek varianten. Plasmide DNA uit bead-gebonden sferoplasten wordt teruggewonnen en gebruikt om een sub-bank die wordt gescreend met de ELISA-gebaseerde secundaire screening genereren. Overeenkomstige protocol stappen worden genoteerd. Klik hier om een grotere versie van deze figuur te bekijken.
- Incubeer de beklede kralen met sferoplasten.
- Gebruik een sferoplast verhouding van ongeveer 5 parel: 1. Voeg 4 x 10 9 sferoplasten en 8 x 10 8 kralen in een steriele 15 ml buis.
OPMERKING: Stel dat geen cellen werden verloren tijdens de spheroplasting proces, zodat de concentratie nog 1 x 10 10 sferoplasten / ml. - Voeg 1 x PBS met 0,1% BSA om het totale volume 4 ml wordt. Aliquot in vier 1,5 ml buizen met elk 1 ml. Incubeer de reacties bij 4 ° C gedurende 5 uur onder zachtjes draaien.
- Gebruik een sferoplast verhouding van ongeveer 5 parel: 1. Voeg 4 x 10 9 sferoplasten en 8 x 10 8 kralen in een steriele 15 ml buis.
- Prepare de kraal gebonden sferoplasten voor polymerasekettingreactie (PCR).
- Plaats de panning reageerbuisjes op de magneet gedurende 3 min. Verwijder het supernatant door pipetteren, en was de kraal gebonden sferoplasten vier maal met ijskoude 1 x PBS met 0,1% BSA op dezelfde wijze als beschreven in stap 3.2.2.3 tot 3.2.2.4. Resuspendeer de kraal gebonden sferoplasten in elke buis in 25 pl gedestilleerd H2O Bewaar de kralen bij -20 ° C of ga verder met stap 4.3.
- Voer gehele plasmide-PCR on-bead gebonden sferoplasten de plasmiden die de genen voor bead gebonden scFvs amplificeren.
- Het verkrijgen van primers met de volgende sequenties: 5'CCAACTCTTTTTCCGAAGGTAACTG3 '(voorwaartse primer) en 5'TAGCTCTTGATCCGGCAAACAAA3' (reverse primer).
Opmerking: Deze zullen end-to-end op tegenoverliggende strengen van de EMB plasmide (figuur 2) binden en zijn ontworpen om te hybridiseren een gemeenschappelijk kenmerk van EMB, zodat amplificatie optreedt ongeacht descFv variante sequentie. - Fosforyleren de primers.
OPMERKING: Zonder fosforylering, herligatie zal optreden. Primers kunnen ook worden besteld met 5'-fosforylering, in plaats van het gebruik van deze methode fosforylering in dit protocol.- In een 0,5 ml buis opstelling een fosforyleringsreactie de voorwaartse PCR-primer zoals beschreven in tabel 1. Herhaal deze procedure voor de tegengestelde primer.
- Incubeer de reacties bij 37 ° C gedurende 1 uur. incubeer ze vervolgens bij 65 ° C gedurende 20 minuten om de T4 polynucleotidekinase (PNK) deactiveren. Bewaar de gefosforyleerde primers bij -20 ° C.
- PCR uitvoeren.
- In een PCR buis Maak de PCR zoals beschreven in tabel 2.
LET OP: Meerdere reacties kunnen voorbereid voor een hogere opbrengst zijn. Het ongebruikte bead-gebonden sferoplasten kunnen worden opgeslagen bij -20 ° C. - Verwarm de PCR reacties bij 98 ° C gedurende 15 min in een thermische cycler voor volledige lysis te verzekeren van de sferoplasten. Verwijder de buizen uit de thermische cycler en voeg 0,5 pl van een high fidelity polymerase elk. Terugvoerbuizen de thermische cycler en start met het programma in Tabel 3.
- Verzamel de PCR producten geschikt. Bewaar bij -20 ° C of ga verder met stap 4.4.
- In een PCR buis Maak de PCR zoals beschreven in tabel 2.
- Het verkrijgen van primers met de volgende sequenties: 5'CCAACTCTTTTTCCGAAGGTAACTG3 '(voorwaartse primer) en 5'TAGCTCTTGATCCGGCAAACAAA3' (reverse primer).
Tabel 1. PNK fosforyleringsreactie (Stap 4.3.2.1).
| Reagens | Volume (pl) |
| Gedestilleerd H 2 O | 15 |
| 10x T4 DNA ligase reactiebuffer | 2 |
| 100 pM primer | 2 |
| T4 polynucleotidekinase (PNK) | 1 |
| Reagens | Volume (pl) |
| Gedestilleerd H 2 O | 28.5 |
| 5x High-fidelity polymerase buffer | 10 |
| 10 M Gefosforyleerd voorwaartse primer | 2.5 |
| 10 uM gefosforyleerde reverse primer | 2.5 |
| 40 mM dNTP mix (10 mM elk dNTP) | 1 |
| -Bead gebonden sferoplasten | 5 |
Tabel 3. PCR-programma (Stap 4.3.3.2).
| Stap | Temperatuur (° C) | Tijd (min: sec) | Aantal cycli |
| Initial denatureren | 98 | 00:30 | 1 |
| denatureren | 98 | 00:10 | 35 |
| gloeien | 69 | 00:30 | |
| Uitbreiding | 72 | 00:30 per kb | |
| laatste verlenging | 72 | 06:00 | 1 |
| Greep | 12 | Eindeloos | 1 |
- Opnieuw circularize heel plasmide-PCR producten en het gebruik geligeerd product te transformeren MC4100 E. coli-cellen.
- Zuiver het PCR-product door het uitvoeren van de PCR-reactie op een agarosegel 23, kleuring van het DNA in de gel 23, en met eengel cleanup kit om de gelineariseerde plasmide zuiveren volgens de door de fabrikant geleverde instructies. Meet de concentratie onder toepassing van een spectrofotometer bij 260 nm. Bewaar het gezuiverde fragment bij -20 ° C totdat het nodig is, of ga verder met stap 4.4.2.
- Opnieuw circularize plasmide uit het PCR-product.
- Intermoleculaire ligatie van het PCR-product te voorkomen, voert de ligatiereactie met een lage concentratie 27 van 1 ng / ul PCR product. Bereken het volume nodig voor het bereiden van een 800 gl ligatiereactie bij deze concentratie.
- Bereid de ligatiereactie op ijs. In een buis, voeg het volume berekend in stap 4.4.2.1 van het PCR product, 80 pi 10 x DNA ligase buffer en gedestilleerd H 2 O tot 800 pl. Voeg 4 pi T4-DNA-ligase, en zij plaatst buizen bij 16 ° C in een waterbad of thermische cycler. Incubeer bij 16 ° CO / N voor 14 tot 18 uur. Bewaar de voltooide ligatiereacties bij -20 ° Ctotdat het nodig is, of ga verder met stap 4.4.3.
- Plaats de ligatie reactie op een verwarmingsblok bij 65 ° C gedurende 15 minuten aan hitte-inactiveren DNA ligase. Maak dan gebruik van een microdialyse membraan of DNA opruimen kit om de-zout het geligeerde DNA. Bewaren bij 20 ° C of ga verder met stap 4.4.4.
- Gebruik de gehele warmte geïnactiveerd,-de gezouten ligatieproduct te transformeren MC4100 E. coli cellen 23. Bereiden glycerol voorraden, zoals beschreven in stap 1.3, van de cellen met de resulterende gepand subbank en bewaar monsters bij -80 ° C.
- Herhaal stap 4 in zijn geheel met een bekende hoeveelheid uit stap 4.4.4 een tweede panning op de subbank doen.
NB: Een tweede panning helpt verrijken bibliotheek bestanddelen die goed binden aan het doelantigeen 19.
5. Voer een tweede scherm met behulp van een Enzyme-linked immunosorbent assay methode om veelbelovende klonen voor verdere karakterisering identificeren (Figuur 5) </ P>

Figuur 5. ELISA-gebaseerde secundaire screening (Stap 5). (A) bibliotheek varianten van de subbank verrijkte tijdens panning geïnoculeerd in afzonderlijke putjes van een kweekplaat voor groei en expressie. (B) Een ELISA plaat gecoat met doelantigeen. (C) De bibliotheek varianten worden gescreend met behulp van de ELISA-gebaseerde secundaire scherm in het protocol beschreven. Na analyse van de gegevens die zijn verkregen uit de secundaire scherm, zijn varianten van belang geselecteerd en gekarakteriseerd verder. Overeenkomstige protocol stappen worden genoteerd. Klik hier om een grotere versie van deze figuur te bekijken.
- Dooi een buis van de gepand subbank (uit stap 4.4.4) en de plaat op LB agarplaten. Plate verschillende verdunningenbij concentraties zo laag zijn individuele kolonies (bijvoorbeeld 10 2 - 6 tot 10-voudige verdunningen). Incubeer de platen gedurende 15-18 uur bij 37 ° C. Bewaar de platen bij 4 ° C of ga verder met stap 5.2.
- Cultuur en induceren kolonies van de gepand subbank. Voer alle stappen onder steriele omstandigheden. Gebruik een multichannel pipet te stappen met 96 putjes.
- Voeg 200 gl LB met 20 ug / ml Cm in elk putje van een ronde bodem 96-well kweekplaat.
- Kies een individuele kolonie van de agarplaat met een pipetpunt, plaats de tip in het eerste putje van de 96-well plaat en Roer inoculeren. Gebruik een nieuwe tip voor elk putje. Inoculeren een kolonie in elk putje. Als controle, ten minste één steriliteit controleputje zonder kolonie geïnoculeerd.
- Herhaal stap 5.2.1 en 5.2.2 tot enkele 96-well platen te enten.
- Plaats de 96 putjes op een microplaat schudinrichting bij 310 rpm. Incubeer bij 376; C gedurende 20 tot 24 uur om de scFvs te drukken.
- Voor elke cultuur plaat bereid in stap 5.2, jas een 96-well ELISA plaat met het doelantigeen.
- Verdun gezuiverd doelantigeen tot een geschikte concentratie (bijvoorbeeld 1 ug / ml tot 4 ug / ml) in 1 x PBS om de bekledingsoplossing maken. Voeg 5 ml bekledingsoplossing per 96-well plaat.
OPMERKING: De juiste concentratie is afhankelijk van het antigeen worden gebruikt en moet worden aangepast. - Voeg 50 ul van de bekledingsoplossing aan elk putje van een 96 putjes hoogbindende clear polystyreen ELISA-plaat. klop de plaat op het stationaire oppervlak zodat het gehele oppervlak van elk putje is bekleed. Herhaal dit voor elke plaat. Incubeer de platen bij 4 ° CO / N.
- Verdun gezuiverd doelantigeen tot een geschikte concentratie (bijvoorbeeld 1 ug / ml tot 4 ug / ml) in 1 x PBS om de bekledingsoplossing maken. Voeg 5 ml bekledingsoplossing per 96-well plaat.
- Kopie van de kolonies van de 96 putjes op agar platen.
- Plaats een steriele polystyreen replicator in de putjes van een kweekplaat met een kleine hoeveelheid vloeibare zee verzamelenid. Til de replicator en over te dragen aan een 15 cm LB-agar plaat zodanig dat alle tips de plaat raken. Zodra de vloeistof heeft overgedragen, til de replicator recht omhoog. Herhaal dit voor elke cultuur plaat.
- Label de agar plaat met de correcte oriëntatie, zodat de resultaten van het tweede scherm in de 96-well plaat kan worden gekoppeld aan de juiste gerepliceerde kolonie op de plaat of verdere karakterisering gewenst. Groeien bij 37 ° C gedurende 15 tot 18 uur, en bewaar bij 4 ° C totdat het nodig is.
- Voer de secundaire scherm ELISA.
- Bereid de blokkerende oplossing door 2% (w / v) melkpoeder in 1 x PBS. Leeg de bekledingsoplossing van de ELISA-platen. Voeg 100 ul van de blokkerende oplossing aan elk putje. Incubeer bij kamertemperatuur gedurende ten minste 2 uur, of blok O / N bij 4 ° C.
- Bereid de wasbuffer door het toevoegen van polysorbaat 20 om een eindconcentratie van 0,05% in 1 x PBS. Maak 250 ml per ELISA-plaat.
- Voeg 20 ul van eengeconcentreerd cellysis wasmiddel aan elk putje van de ronde-bodem kweekplaat, en incubeer de petrischaal op een microplaat schudinrichting bij kamertemperatuur gedurende 15 tot 20 minuten. Begin de lysis tegelijkertijd dat het blokkeren van de ELISA-platen is voltooid, zodat de lysis en de wasstap 5.5.4 gelijktijdig kunnen worden uitgevoerd.
- Leeg de blokkerende oplossing uit de ELISA-platen. Was de geblokkeerde ELISA-platen vier keer met 200 ui wasbuffer per well per wasbeurt. Leeg de wasbuffer uit de putjes.
- Breng 50 pl van elk putje van de cellyse plaat om de overeenkomstige putje van de ELISA plaat, met een nieuwe tip voor elk putje. Incubeer de ELISA plaat bij kamertemperatuur gedurende 1-2 uur.
- Bereid het antilichaam oplossing voor gebonden scFvs te detecteren.
- Gebruik een mierikswortel peroxidase (HRP) geconjugeerd primair antilichaam dat bindt aan de FLAG epitoop tag gefuseerd met de bibliotheek scFvs.
- Verdun het antilichaam aan de geschikte verdunning gebruikt in een ELISA (zie leverancier en #39; s aanbevelingen) in 2% (w / v) melkpoeder in 0,05% polysorbaat 20 in 1 x PBS. Bereid 5 ml voor elke plaat.
- Was de ELISA platen vier maal met wasbuffer zoals beschreven in stap 5.5.4.
- Voeg 50 ul van de oplossing met antilichaam aan elk putje van de ELISA plaat. Incubeer gedurende 1-2 uur bij KT.
- Bereid de HRP substraat door het oplossen van o-fenyleendiamine dihydrochloride (OPD) tabletten in gedestilleerd H2O per protocol van de fabrikant met vermijding licht. Bereid 20 ml per ELISA-plaat.
- Bereid 3 MH 2 SO 4 door het verdunnen van geconcentreerd H 2 SO 4 met gedestilleerd H 2 O als dat nodig is. Bereid 5 ml per ELISA-plaat.
Voorzichtig: H 2 SO 4 is een sterk zuur. Zorg ervoor dat u de juiste persoonlijke beschermingsmiddelen te dragen. - Was de ELISA platen viermaal gewassen met wasbuffer, zoals beschreven in stap 5.5.4.
- Incubeer ELISA platen met de HRP Substrate.
- Voeg 200 ul van het HRP substraat aan elk putje. Om blootstelling aan licht te minimaliseren, voegen de ondergrond een ELISA-plaat in een tijd, en wikkel met aluminiumfolie alvorens over te gaan naar de volgende plaat. Incubeer de platen gedurende 30 tot 60 min bij kamertemperatuur in het donker.
- Na de eerste 30 minuten, controleer de platen donker worden van het substraat en incubeer langer indien nodig te visualiseren ontwikkeling van kleur.
- Voeg 50 ul van 3 MH 2 SO 4 aan elk putje om de reactie te blussen. Met behulp van een andere tip voor elk putje, meng de oplossing in de putten door voorzichtig en neer te pipetteren zonder schuimen. Consistentie en verzadiging voorkomen, voeg H 2 SO 4 snel en zorgvuldig alle ELISA-platen vóór het mengen van de oplossing voor elke plaat.
- Meet de absorptie van de oplossing in de putjes van elke plaat bij 492 nm met een plaatlezer.
- Analyseer de absorptiegegevens identificeren scFv varianten die exhibHet veelbelovende binding signalen en karakteriseren van deze veelbelovende scFvs. Selecteer scFvs die absorptie signalen boven het achtergrondsignaal en hoger dan het gemiddelde signaal op elke plaat vertonen.
OPMERKING: De absorptie niveau afhankelijk van de eigenschappen van het antigen en anti-FLAG antilichaam dat, samen met de kracht van de scFv varianten die werden geïsoleerd uit de screening.
Representative Results
De intracellulaire eiwitvouwing kwaliteitscontrole mechanisme van de Tat pathway van E. coli beperkt transport over de binnenste celmembraan eiwitten die goed gevouwen in het reducerende cytoplasmatische milieu. Door overexpressie een fusie van een scFv met de ssTorA signaalsequentie (de signaalsequentie van het eiwit TORA, dat van nature wordt getransporteerd door de Tat pathway 20), wordt translocatie geblokkeerd, waardoor weergave van scFvs op het binnenste membraan 19. Na enzymatische verstoring van de buitenmembraan zijn de geëxposeerde antilichamen beschikbaar gesteld voor het screenen op antigeenbindingsactiviteit. De mogelijkheid om te profiteren van de Tat pathway van scFv weergave werd aangetoond door Karlsson et al. 19 (figuur 6). De scFv antilichamen scFv13 en scFv13.R4 werden gefuseerd aan ofwel de natieve sequentie ssTorA of een gemodificeerde ssTorA dat de arginine-arginine residu pair erkend mistde Tat pathway. scFv13.R4 werd ontwikkeld door Martineau et al. van scFv13 door middel van vier ronden van gerichte evolutie en is bekend om goed vouwen in de cytoplasma 9. Dit scFv werd op het binnenste membraan, maar alleen uitgedrukt als een fusie met de natieve signaalsequentie ssTorA (figuur 6). In tegenstelling, scFv13 niet goed gevouwen cytoplasmatisch 9, zodat het niet goed op het binnenste membraan, ongeacht de signaalsequentie waaraan het is gefuseerd. Bovendien, als de scFvs werden tot expressie gebracht in cellen die de TatC eiwit, een essentieel onderdeel van het Tat machine 20,28 ontbrak, vertoning werd niet waargenomen, waaruit het belangrijke verband tussen binnenste membraan display en de Tat pathway. Deze resultaten demonstreren dat alleen eiwitten die de Tat signaalpeptide bevatten en die correct in het cytoplasma worden gevouwen worden op het binnenste membraan, waardoor transport door de Tat pathway te functioneren als een scherm voor intracellulaire folding.

Figuur 6. Detectie van scFvs weergegeven op het binnenste membraan. Flowcytometrie-analyse werd uitgevoerd om de weergave van slecht gevouwen scFv13 en goed gevouwen scFv13.R4 detecteren op het binnenste membraan. scFvs werden gefuseerd met natieve ssTorA of ssTorA (KK), waarbij de Arg-Arg pair in ssTorA sequentie werd gemodificeerd om Lys-Lys. Het C-terminale FLAG-epitoop tags op de scFvs werden gedetecteerd met een fluoresceïne isothiocyanaat (FITC) geconjugeerd anti-FLAG antilichaam. Cellen zonder TatC eiwit (ΔtatC) en ssTorA-scFv13 zonder de FLAG tag werden getest als controles. M geeft de mediane fluorescentie waarde. Overgenomen uit referentie 19 met toestemming. Klik hier om een grotere versie van deze figuur te bekijken.
19. Om dit aan te tonen, een voor fouten gevoelige PCR bibliotheek op basis van scFv13, dat een lage bindingsaffiniteit voor β-galactosidase is, is gepand tegen het doelantigeen β-galactosidase Display en panning werkwijze beschreven in het protocol. scFv 1-4 werd geïsoleerd na één ronde van mutagenese en panning en hadden hogere bindingsaffiniteit voor p-galactosidase dan scFv13 (figuur 7A) en een hogere oplosbaarheid cytoplasma (Figuur 7B).
Een nieuwe bibliotheek op basis van scFv 1-4, werd gemaakt met voor fouten gevoelige PCR en panning van deze tweede generatie bibliotheek tegenβ-galactosidase werd uitgevoerd met een modificatie van het protocol beschreven. De panning tegen β-galactosidase voor de tweede ronde van ontwikkeling werd uitgevoerd in de aanwezigheid van gezuiverde, oplosbare scFv 14 als concurrent van de waarschijnlijkheid isoleren klonen met hogere affiniteit dan scFv 1-4 verbeteren. Na deze tweede ronde van mutagenese en panning, scFv en scFv 2-1 2-3 werden geïsoleerd met behulp van de ELISA-gebaseerde secundaire screening. Deze scFvs niet alleen hadden hogere bindingsaffiniteit voor β-galactosidase dan scFv13, maar vertoonden betere binding dan de eerste ronde scFv kloon 1-4. scFv 2-1 vertoonden β-galactosidase binding vergelijkbaar met die van scFv13.R4 (figuur 7A). scFv 2-3 toont ook een verdere toename van cytoplasmatische oplosbaarheid vergeleken met scFv 14, aandacht voor de gelijktijdige engineering van oplosbaarheid en antigeenbindende. Aangezien affiniteit en oplosbare expressie van de scFvs worden gescreend voor het gelijktijdig, is het mogelijk dat een geselecteerde scFv is moderate oplosbaarheid maar hoge binding of vice versa. Bijvoorbeeld, scFv 2-1 een lagere expressie dan oplosbare scFv 2-3, maar vertoont hogere bindingsaffiniteit voor p-galactosidase.

Figuur 7. Target-binding en cytoplasmatische expressie van scFv varianten die middels binnenste membraan display. (A) scFvs werden uitgedrukt in het cytoplasma van E. coli-cellen (bijv., zonder dat de ssTorA signaalsequentie) met een hexahistidine (6 x His) tag en gezuiverd met behulp van nikkel-nitrilotriazijnzuur spin-kolommen. De binding van het gezuiverde scFvs aan P-galactosidase werd gemeten met een ELISA. Gezuiverde scFvs werden op-β-galactosidase beklede ELISA platen en gebonden scFvs werden gedetecteerd met een anti-6 x His antilichaam. De gegevens zijn een gemiddelde van zes herhalingen, en de fout balk toont standaardafwijking van het gemiddelde.(B) De oplosbare en onoplosbare fracties van cellysaten van cellen die het cytoplasmatisch scFvs werden geanalyseerd met een Western blot geprobed met een anti-6 x His antilichaam. Totale eiwitconcentratie werd gebruikt voor het laden van de monsters te normaliseren. Herdrukt (A) en aangepast (B) uit referentie 19 met toestemming. Klik hier om een grotere versie van deze figuur te bekijken.
Discussion
Engineering van antilichamen voor cytoplasmatische activiteit is een moeilijke taak vanwege het reducerende milieu van het cytoplasma, die de vorming van stabiliserende disulfidebindingen 6,7 belemmert. Dit veroorzaakt de meeste antilichamen cytoplasmatisch inactief tenzij zij zijn ontworpen voor stabiliteit en oplosbaarheid in het cytoplasma, naast het feit ontworpen voor bindingsaffiniteit. De bestaande methoden van faagdisplay, bacteriële oppervlakte-en gist oppervlakte-methoden gebruiken allemaal de secretieroute 14-16 voor de weergave van gemanipuleerde antilichamen, maar deze werkwijzen hebben geen middelen om intracellulaire vouwen ingenieur. Antilichamen gemanipuleerd middels binnenste membraan beeldscherm hebben cytoplasmatische stabiliteit en oplosbaarheid verbeterd doordat het vouwen kwaliteitscontrole van de Tat pathway voorkomt translocatie van antilichamen die slecht gevouwen en stabiel in het cytoplasma. Deze methode vereenvoudigt het iteratief proces engineering intracellulaire antilichamen voor een affiniteitnd oplosbaarheid, de twee eigenschappen zijn gemanipuleerd in één stap. Hoewel deze methode is ontworpen voor engineering antilichamen met oplosbaarheid in het reducerende intracellulaire omgeving, kan het ook worden toegepast op technische antilichamen functioneren in niet-reducerende omstandigheden, omdat de eiwitten ontworpen met behulp van deze methode behouden hun vouwen in de oxiderende omgeving van het periplasma.
Hoewel deze techniek vereenvoudigt het proces van engineering van antilichamen met een hoge affiniteit en hoge cytoplasma oplosbaarheid, een aantal beperkingen zijn belangrijk om te overwegen bij het gebruik van dit protocol. Bij de analyse van secundaire screening ELISA signalen te identificeren veelbelovende scFv varianten, de drempel voor veeleisende tussen potentieel interessante varianten en die adequate antigeenbindend niet kan vertonen waarschijnlijk niet duidelijk tot na verscheidene klonen verder gekarakteriseerd. Het is belangrijk om te zoeken naar verbeterde binding via ouderlijke antilichaam; echter,een abnormaal hoog signaal kan indicatief aviditeit 29 of aggregatie effecten 30, een uitdaging die niet uniek is voor de binnenste membraan weergave screening aanpak. Een belangrijke beperking te onthouden bij gebruik van dit protocol is het onvermogen om sferoplasten herstellen na panning, die immers niet-levende (ongepubliceerde gegevens). Dit vereist de DNA-amplificatie en transformatie stappen om de antilichaam coderend plasmiden herstellen.
Verschillende kritische stappen van het protocol in te schakelen de simultaneous engineering van het vouwen en binden van antilichamen. Voor screening succesvol te zijn, moet de scFv-bank wordt gescreend worden uitgedrukt als een fusie met het ssTorA signaalpeptide. Zonder deze procedure stelt antilichamen niet worden gericht op de Tat pathway en bijgevolg niet worden getransloceerd naar het periplasma 19. Bovendien is het noodzakelijk dat een C-terminale epitoop tag gefuseerd met de antilichamen om detectie van de geëxposeerde antilichamen toe in de prullenbakding assays. Duidelijk, E. coli stam gebruikt om de scFvs moet ook de nodige Tat pathway machines drukken, maar dit geldt voor de gebruikte E. coli.
Wijzigingen in dit protocol zijn mogelijk om zijn potentieel te verbeteren om antilichamen met de gewenste eigenschappen te isoleren. Een subtractieve panning stap kan voorafgaand aan panning tegen het doelwit antigeen aan de scFv bibliotheek van niet-gewenste bestanddelen afbreken worden afgerond. De bibliotheek sferoplasten kunnen worden geïncubeerd met magnetische bolletjes gecoat met BCCP alleen of bekleed met een niet-gewenste eiwit, en de sferoplasten die binden aan de kralen kunnen worden verwijderd voor het screenen van de resterende ongebonden sferoplasten voor binding aan het gewenste doel. Zoals in het Representatieve resultaten, een werkwijze ter verbetering van de affiniteit van een geïsoleerde scFv is een oplosbare concurrent in de panning reactie te concurreren met de informatie op de sferoplasten scFvs omvatten. Omdat de oplosbare compingetitor is een gezuiverd eiwit, geen DNA wordt geamplificeerd uit, dus alleen sequenties van de informatie op de sferoplasten scFvs wordt de PCR-reactie worden gewonnen. Daarnaast kan deze werkwijze worden uitgebreid naar andere typen techniek antilichamen of niet-antilichaam bindende eiwitten.
E. coli binnenste membraan beeldscherm is een krachtig platform voor engineering antilichamen met hoge affiniteit en hoge niveaus van intracellulaire oplosbaarheid. Deze methode is bijzonder geschikt voor efficiënte engineering van antilichamen zijn ontworpen om te functioneren in de intracellulaire omgeving. Deze intracellulaire antilichamen worden reeds onderzocht als potentiële geneesmiddelen in een aantal gebieden, met inbegrip van neurodegeneratieve ziekten, kanker en virale infecties 31. Deze techniek wordt meer algemeen gebruik van intracellulaire antilichamen als hulpmiddelen voor onderzoek en geneeskunde op deze gebieden en elk ander gebied waarop bestuderen een eiwitdoel in situ gewenst mogelijk.
Materials
| Name | Company | Catalog Number | Comments |
| scFv library | Varies | A suitable scFv library should be obtained from a commercial or academic source. | |
| MC4100 E. coli cells | Coli Genetic Stock Center | 6152 | Cells need to be chemically competent or electrocompetent, depending on the selected transformation method. |
| Glycerol | Fisher Scientific | BP229-4 | |
| Difco dehydrated culture media LB Broth, Miller (Luria-Bertani) | BD | 244610 | |
| Chloramphenicol (Cm) | Fisher Scientific | BP904-100 | |
| Sodium chloride (NaCl) | Fisher Scientific | BP358-1 | |
| Potassium chloride (KCl) | Fisher Scientific | BP366-500 | |
| Sodium phosphate, dibasic (Na2HPO4) | Fisher Scientific | BP332-500 | |
| Potassium phosphate, monobasic (KH2PO4) | Fisher Scientific | BP362-500 | |
| Bovine serum albumin (BSA) | Fisher Scientific | BP9706-100 | |
| Sucrose | Fisher Scientific | BP220-1 | |
| Tris base | Fisher Scientific | BP1521 | |
| Ethylenediaminetetraacetic acid (EDTA), 0.5 M | Fisher Scientific | BP2482-500 | |
| Magnesium chloride (MgCl2) | Fisher Scientific | BP214-500 | |
| Lysozyme | Sigma Aldrich | L3790-10X1ML | |
| Vortex mixer | VWR | 97043-564 | |
| Bicine | Fisher Scientific | BP2646100 | |
| D-Biotin | Fisher Scientific | BP232-1 | |
| Isopropyl β-D-1-thiogalactopyranoside | Fisher Scientific | BP1755-1 | |
| BugBuster Master Mix (cell lysis detergent) | EMD Millipore | 71456 | |
| Vivaspin 2 MWCO, 3,000 daltons | GE Healthcare Sciences | 28932240 | |
| Target antigen | Varies | N/A | Purified target antigen may be purchased or produced/purified. |
| Dynabeads MyOne Streptavidin T1 | Invitrogen | 65601 | |
| Dynamag-2 magnet | Invitrogen | 12321D | |
| Tube rotator | VWR | 13916-822 | |
| PCR primers | IDT | N/A | Primer sequences are as described in the protocol. |
| 10x T4 DNA ligase reaction buffer | New England BioLabs | B0202S | |
| T4 Polynucelotide kinase (PNK) | New England BioLabs | M0201S | Make sure the T4 ligase buffer used in the primer phosphorylation reaction contains 1 mM ATP. |
| 5x Phusion HF buffer pack | New England BioLabs | B0518S | |
| Deoxynucleotide (dNTP) solution mix, 10 mM each dNTP | New England BioLabs | N0447L | |
| Phusion DNA polymerase | New England BioLabs | M0530S | Other high-fidelity polymerases may be used as an alternative, but the annealing temperature in Table 3 must be adjusted. |
| C1000 Touch thermal cycler with dual 48/48 fast reaction module | Bio-Rad | 185-1148 | |
| Agarose | Promega | V3121 | |
| SYBR Safe DNA gel stain | Invitrogen | S33102 | |
| Wizard SV gel and PCR clean-up system | Promega | A9281 | |
| T4 DNA ligase | New England BioLabs | M0202S | |
| Microdialysis membrane filter | EMD Millipore | VSWP04700 | |
| Agar | BD | 214030 | |
| 96-well polystyrene round-bottom cell culture plates | VWR | 10062-902 | |
| Costar general polystyrene assay plate lids | Corning | 3931 | |
| Microtitre plate shaker | VWR | 12620-926 | |
| Costar 96 well EIA/RIA Easy Wash clear flat bottom polystyrene high bind microplate | Corning | 3369 | |
| Bel-blotter polycarbonate 96-well replicating tool | Bel-Art Products | 378760002 | |
| Instant nonfat dry milk | Quality Biological | A614-1000 | |
| Tween 20 (polysorbate 20) | Fisher Scientific | BP337-500 | |
| PopCulture reagent (concentrated cell lysis detergent) | EMD Millipore | 71092-3 | |
| Monoclonal ANTI-FLAG M2-Peroxidase(HRP) antibody produced in mouse | Sigma Aldrich | A8592 | |
| SigmaFast OPD | Sigma Aldrich | P9187-50SET | |
| Sulfuric acid (H2SO4), 10 N solution | Fisher Scientific | SA200-1 | |
| Reynolds Wrap aluminum foil | VWR | 89079-075 | |
| BioTek Epoch microplate spectrophotometer | Fisher Scientific | 11120570 |
References
- Biocca, S., Pierandrei-Amaldi, P., Campioni, N., Cattaneo, A. Intracellular immunization with cytosolic recombinant antibodies. Biotechnology (NY). 12 (4), 396-399 (1994).
- Chen, S. Y., Bagley, J., Marasco, W. A. Intracellular antibodies as a new class of therapeutic molecules for gene-therapy. Hum. Gene Ther. 5 (5), 595-601 (2008).
- Gargano, N., Biocca, S., Bradbury, A., Cattaneo, A. Human recombinant antibody fragments neutralizing human immunodeficiency virus type 1 reverse transcriptase provide an experimental basis for the structural classification of the DNA polymerase family. J Virol. 70 (11), 7706-7712 (1996).
- Mhashilkar, A. M., et al. Inhibition of HIV-1 Tat-mediated LTR transactivation and HIV-1 infection by anti-Tat single chain intrabodies. Embo J. 14 (7), 1542-1551 (1995).
- Strube, R. W., Chen, S. Y. Characterization of anti-cyclin E single-chain Fv antibodies and intrabodies in breast cancer cells: enhanced intracellular stability of novel sFv-F-c intrabodies. J. Immunol. Meth. 263 (1-2), 149-197 (2002).
- Mössner, E., Koch, H., Plückthun, A. Fast selection of antibodies without antigen purification: adaptation of the protein fragment complementation assay to select antigen-antibody pairs. J. Mol. Biol. 308 (2), 115-122 (2001).
- Wörn, A., et al. Correlation between in vitro stability and in vivo performance of anti-GCN4 intrabodies as cytoplasmic inhibitors. J Biol Chem. 275 (4), 2795-2803 (2000).
- Knappik, A., Plückthun, A. Engineered turns of a recombinant antibody improve its in vivo folding. Protein Eng. 8 (1), 81-89 (1995).
- Martineau, P., Jones, P., Winter, G. Expression of an antibody fragment at high levels in the bacterial cytoplasm. J Mol Biol. 280 (1), 117-127 (1998).
- Steipe, B., Schiller, B., Plückthun, A., Steinbacher, S. Sequence statistics reliably predict stabilizing mutations in a protein domain. J. Mol. Biol. 240 (3), 188-192 (1994).
- Daugherty, P. S. Protein engineering with bacterial display. Curr Opin Struct Biol. 17 (4), 474-480 (2007).
- Lener, M., et al. Diverting a protein from its cellular location by intracellular antibodies. Eur. J. Biochem. 267 (4), 1196-1205 (2000).
- Lynch, S. M., Zhou, C., Messer, A. An scFv intrabody against the nonamyloid component of α-synuclein reduces intracellular aggregation and toxicity. J. Mol. Biol. 377 (1), 136-147 (2008).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr. Opin. Struc. Biol. 17 (4), 467-473 (2007).
- Kieke, M. C., et al. Selection of functional T cell receptor mutants from a yeast surface-display library. Proc. Natl. Acad. Sci. USA. 96 (10), 5651-5656 (1999).
- Steiner, D., Forrer, P., Stumpp, M. T., Pluckthun, A. Signal sequences directing cotranslational translocation expand the range of proteins amenable to phage display. Nat. Biotechnol. 24, 823-831 (2006).
- Pugsley, A. P. The complete general secretory pathway in gram-negative bacteria. Microbiol. Rev. 57 (1), 50-108 (1993).
- Rapoza, M. P., Webster, R. E. The filamentous bacteriophage assembly proteins require the bacterial SecA protein for correct localization to the membrane. J. Bacteriol. 175 (6), 1856-1859 (1993).
- Karlsson, A. J., et al. Engineering antibody fitness and function using membrane-anchored display of correctly folded proteins. J. Molec. Biol. 416 (1), 94-107 (2012).
- DeLisa, M. P., Tullman, D., Georgiou, G. Folding quality control in the export of proteins by the bacterial twin-arginine translocation pathway. Proc Natl Acad Sci U S A. 100 (10), 6115-6120 (2003).
- Fisher, A. C., Kim, W., DeLisa, M. P. Genetic selection for protein solubility enabled by the folding quality control feature of the twin-arginine translocation pathway. Protein Sci. 15 (3), 449-458 (2006).
- Maynard, J., Georgiou, G. Antibody engineering. Annu Rev Biomed Eng. 2, 339-376 (2000).
- Green, M. R., Sambrook, J. Molecular Cloning: A Laboratory Manual. 1, Fourth, Cold Spring Harbor Laboratory Press. (2012).
- Milo, R., Jorgensen, P., Moran, U., Weber, G., Springer, M. BioNumbers--the database of key numbers in molecular and cell biology. Nucleic Acids Res. 38, D750-D753 (2010).
- Hermanson, G. T. Bioconjugate Techniques. , Third, Elsevier/Academic Press. (2013).
- Tayapiwatana, C., Chotpadiwetkul, R., Kasinrerk, W. A novel approach using streptavidin magnetic bead-sorted in vivo biotinylated survivin for monoclonal antibody production. J Immunol Methods. 317 (1-2), 1-11 (2006).
- Zhu, G., Song, L., Lippard, S. J. Visualizing inhibition of nucleosome mobility and transcription by cisplatin-DNA interstrand crosslinks in live mammalian cells. Cancer Res. 73 (14), 4451-4460 (2013).
- Bogsch, E. G., et al. An essential component of a novel bacterial protein export system with homologues in plastids and mitochondria. J. Biol. Chem. 273, 18003-18006 (1998).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng. Des. Sel. 28 (10), 339-350 (2015).
- Garber, K. Bispecific antibodies rise again. Nat. Rev. Drug. Discov. 13 (11), 799-801 (2014).
- Marschall, A. L., Dübel, S., Böldicke, T. Specific in vivo knockdown of protein function by intrabodies. MAbs. 7 (6), 1010-1035 (2015).



