Summary
İki bitki organelleri DNA zenginleştirme yönteminin karşılaştırılması ve optimizasyonu sunulmaktadır: geleneksel diferansiyel santrifüjleme ve metilasyon durumuna dayalı toplam gDNA'nın fraksiyonlanması. Ortaya çıkan DNA miktarını ve kalitesini değerlendirir, kısa okunmuş yeni nesil dizilemede performans gösteririz ve uzun okunan tek moleküllü dizilemede kullanım potansiyelini tartışırız.
Abstract
Bitki organelleri genomları, kompleks yapıları ve / veya alt genomik parçaları oluşturmak için eşleştirme veya rekombinasyona uğrayabilen büyük, tekrarlayan elementler içerir. Organeller genomları aynı zamanda belirli bir hücre veya doku türünün (heteroplasmi) katkı maddelerinde bulunur ve alt tiplerin bolluğu, gelişim boyunca veya stres altındayken (alt stoikometrik kayma) değişebilir. Organelar genom yapısını ve fonksiyonunu daha iyi anlamak için yeni nesil sıralama (NGS) teknolojileri gereklidir. Geleneksel sıralama çalışmaları organellar DNA elde etmek için çeşitli yöntemler kullanır: (1) Başlangıç dokusu büyük miktarda kullanılırsa, homojenize edilir ve diferansiyel santrifüjleme ve / veya degrade saflaştırma tabi tutulur. (2) Daha az miktarda doku kullanılırsa ( yani, tohum, malzeme veya alan sınırlıysa), yeterli DNA elde etmek için (1) 'de olduğu gibi aynı işlem gerçekleştirilir ve bunu takiben tam genom amplifikasyon uygulanır. (3) Biyoenformatik analiz seq için kullanılabilirToplam genomik DNA'yı ve organelar okumaları ayrıştırmak. Bütün bu yöntemlerin kendi zorlukları ve zıvanaları vardır. (1) 'te bu kadar büyük miktarda başlangıç dokusu elde etmek zor olabilir; (2) 'de, bütün genom amplifikasyonu bir sıralama ön yargı getirebilir; Ve (3) de, nükleer ve organel genomları arasındaki homoloji, toplanma ve analizi etkileyebilir. Büyük nükleer genomlara sahip bitkilerde, biyoinformatik analizler için sekanslama maliyetlerini ve sekans karmaşıklığını azaltmak için organellar DNA'yı zenginleştirmek avantajlıdır. Burada, toplam genomik DNA'yı nükleer ve organellar fraksiyonlara ayırmak için, geleneksel diferansiyel santrifüj yöntemini, adapte edilmiş bir CpG-metil açılım yaklaşımı olan dördüncü bir yöntemle karşılaştırabiliriz. Her iki yöntem de, organellar sekanslar için oldukça zenginleştirilmiş DNA olan NGS için yeterli miktarda DNA üretir; buna rağmen, mitokondriya ve kloroplastlarda farklı oranlarda bulunur. Buğday yaprak dokusu için bu yöntemlerin optimizasyonunu sunmak ve önemli avantajları tartışmak veÖrnek giriş, protokol kolaylığı ve akışaşağı uygulama bağlamında her yaklaşımın avantajları.
Introduction
Genom dizilimi, önemli bitki özelliklerinin altında yatan genetik temeli incelemek için güçlü bir araçtır. Çoğu genom dizilimi çalışması genlerin çoğunluğu çekirdekte yer aldığı için nükleer genom içeriğine odaklanır. Bununla birlikte, mitokondri (ökaryotlarda) ve plastidler (bitkilerde, özel form, kloroplast, fotosentezde çalışır) de dahil olmak üzere organellar genomları, organizmanın gelişimi, stres tepkisi ve genel uyum için gerekli olan önemli genetik bilgilere katkıda bulunur 1 . Organelar genomları nükleer genom dizilimi için toplam DNA ekstraksiyonlarında tipik olarak yer alır, ancak DNA ekstraksiyonundan önce organel sayıları azaltma yöntemleri de kullanılır 2 . Birçok çalışma, organellar genomlarını 3 , 4 , 5 ve 6 numaralı gruplara toplamak için toplam gDNA özütlemelerinden sıralama sonuçlarını kullandı.Xref "> 6 , 7. Bununla birlikte, çalışmanın amacı organellar genomlara odaklanmaktır, toplam gDNA'yı kullanmak sıralamaya ilişkin maliyetleri arttırır çünkü birçok okuma, özellikle büyük nükleer genomlara sahip bitkilerde çekirdek DNA dizileri için" kaybolur " Ayrıca, organellar sekansların nükleer genom ve organeller arasında kopyalanması ve aktarılması nedeniyle, dizileme okumalarının doğru haritalama pozisyonunu uygun genoma çevirmek bioinformatik olarak zorlayıcıdır 2 , 8. Organelar genomlarının nükleer genomdan saflaştırılması bir Bu problemleri azaltmak için strateji. Mitokondri ve kloroplastlar arasındaki homoloji bölgeleriyle eşleşen okumaları ayırmak için daha fazla biyoenformatik stratejiler kullanılabilir.
Birçok bitki türünden gelen organellar genomları sıralanırken, organellar genom çeşitliliğinin genişliği hakkında pek az şey bilinmektedirYabani popülasyonlarda veya ekili yetiştirme havuzlarında kullanılabilir. Organel genomları, aynı zamanda, tekrar sıraları 9 arasında rekombinasyon için önemli yapısal yeniden düzenlenmeye uğramayan dinamik moleküller olduğu bilinmektedir. Dahası, organellar genomun çok sayıda kopyası her bir organel içinde bulunur ve her hücrede birden fazla organel bulunur. Bu genomların tüm kopyaları aynı değildir, bu heteroplasmi olarak bilinir. Kanonik resimde aksine "ana çevrelerinde," alt-genomik daire, doğrusal kromozomlar, doğrusal konkatamerler ve dallanmış yapılar 10 de dahil olmak üzere organel genom yapılar, daha karmaşık bir görüntü için hemen giderek artan kanıtlar bulunmaktadır. Bitki organelleri genomlarının toplanması, nispeten büyük boyutları ve önemli ters çevrilmiş ve doğrudan tekrarlamalarıyla daha da karmaşıktır.
Organelar izolasyonu, DNA saflaştırması ve sonraki genom için geleneksel protokoller E dizilimi sıklıkla hantaldır ve başlangıç noktasında 11 , 12 , 13 , 14 , 15 , 16 , 17 olması gereken yüzlerce gramdan daha fazla genç yaprak dokusundan birkaç gram yukarıya kadar doku girişinin büyük miktarlarını gerektirir. Bu, doku sınırlı olduğunda organellar genom sıralamasını erişilmez hale getirir. Bazı durumlarda, tohum miktarları, nesiller bazında veya geçiş yoluyla korunması gereken erkek steril hatlarda sıralanması gerektiğinde sınırlıdır. Bu durumlarda, organellar DNA saflaştırılabilir ve sonra bütün genom amplifikasyonuna tabi tutulabilir. Bununla birlikte, bütün genom amplifikasyonu, önemli yapısal sıralama önyargılarını ortaya çıkarabilir; bu, yapısal değişiklikleri, alt-genomik yapıları ve heteroplasmi seviyelerini değerlendirirken özel bir problemdir> 18. Kısa okunan sıralama teknolojileri için kütüphane hazırlığındaki son gelişmeler, tam genom amplifikasyonundan kaçınmak için düşük giriş engellerini aşmıştır. Örneğin Illumina Nextera XT kütüphane hazırlık kiti, girdi 19 olarak kullanılmak üzere 1 ng kadar az DNA'ya izin verir. Bununla birlikte, PacBio veya Oxford Nanopore sıralama teknolojileri gibi uzun okunan sıralama uygulamaları için standart kütüphane hazırlıkları, hala organellar genom dizilimi için zorluk oluşturabilen nispeten yüksek miktarda giriş DNA'sı gerektirir. Son zamanlarda, girdi miktarlarını azaltmak ve mikrogram-DNA miktarlarının elde edilmesinin zor olduğu örneklerde genom dizilimini kolaylaştırmak için yeni ve kullanıcı tarafından yapılmış uzun okunur sıralama protokolleri geliştirildi. 20 , 21 . Ancak, bu kütüphane preparatlarına beslemek için yüksek molekül ağırlıklı, saf organellar fraksiyonlarının elde edilmesi bir sorun teşkil etmektedir.
AradıkO Tam genom amplifikasyonuna ihtiyaç duymadan NGS için uygun olan organellar DNA zenginleştirme ve izolasyon yöntemlerini karşılaştırır ve optimize eder. Özellikle, amacımız, bir yaprak alt örneği gibi sınırlı başlangıç malzemelerinden yüksek molekül ağırlıklı organellar DNA'yı zenginleştirmek için en iyi uygulamaların belirlenmesi idi. Bu çalışma, organellar DNA'yı zenginleştirmek için yöntemlerin karşılaştırmalı bir analizini sunmaktadır: (1) (2) ticari olarak mevcut bir DNA CpG-metil bağlama alanı proteini çekme yaklaşımının kullanılmasına dayanan bir DNA fraksiyonasyon protokolüne karşı (2) değiştirilmiş, geleneksel bir diferansiyel santrifüj protokolü 22 bitki dokusuna uygulanır 23 . Organel DNA'nın buğday yaprağı dokusundan izole edilmesi için, diğer bitkiler ve doku türlerine kolayca uzatılabilecek en iyi uygulamaları önermekteyiz.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Organel İzolasyon ve DNA Ekstraksiyonu için Bitki Malzemelerinin Üretimi
- Buğday fidelerinin standart büyümesi
- Kök başına 4-6 tohum ile küçük, kare kaplarda vermikülit tohumları tohumlar. 16 saat ışık devri, 23 ºC gündüz / 18 ºC gece bir sera veya büyüme odasına aktarın.
- Bitkiler her gün su ister. Çimlenme üzerine ve çimlenmeden sonraki 7. günde ¼ çay kaşığı granül 20-20-20 NPK gübresi ile bitkiler dölleyin.
- Buğday fidelerinin alternatif etiyolasyonu
- Adım 1.1'i izleyin, ancak kapları 8 saat boyunca 16 saat / 18ºC'de 23 ° C'de koyu renk büyütme odasında yerleştirin. Alternatif olarak, seralardaki bitkileri örtün ( örn., Bir saklama kabı ile, ancak uygun havalandırma korunmalıdır).
- Büyüme ve doku toplama
- Bitkileri 12 - 14 gün boyunca büyütün. Çoğu buğday genotipi içinS, 75-100 fidan, farklı santrifüj yöntemini kullanarak iki organelar çıkarım için yeterli 10-12 g doku verir (bölüm 2); Nükleer DNA'dan organelleri fraksiyon haline getirmek için DNA CpG-metilasyona dayalı açılma yaklaşımını kullanırsak, yalnızca bir bitki gereklidir (bölüm 3).
- Diferansiyel santrifüj yaklaşımı kullanılıyorsa, dokuyu taze olarak toplayın ve bölüm 2'de açıklandığı gibi numunelerin işlenmesine hemen başlayın.
- CpG-metil açılma yaklaşımı kullanılıyorsa, genç yaprak dokusunun 20 mg'lık kesitlerini mikro santrifüj tüplerine hasat edin (standart büyümüş veya etilenmiş dokuyu kullanın, Temsilcisi Sonuçları'na bakın ). Sıvı nitrojende sıkıştırın ve kullanıma kadar -80 ºC'de dondurun. 3. bölümde anlatıldığı gibi, DNA'nın pulldown fraksiyonlamasına devam edin.
2. Yöntem # 1: Diferansiyel Santrifüj (DC) Kullanarak DNA Ekstraksiyonu
NOT: Farklılıkerential santrifüj protokolü iki organellere izole ancak mitokondri 17, 24 için zenginleştirmek üzere koşulları optimize iki yayınlardan modifiye edilmiştir. Elde edilen protokol daha az zaman alır ve daha önceki yöntemlere kıyasla daha az toksik kimyasal kullanır. Spesifik olarak, STE ekstraksiyon tamponuna polivinilpirolidon (PVP) eklenmesi ve sodyum florür (NaF) içeren NETF tamponundaki son yıkama adımının ortadan kaldırılması da dahil olmak üzere, tamponlar ve yıkama adımlarında değişiklikler yaptık.
Dikkat: Bu tampon 2-mercaptoethanol (BME) içerdiğinden, STE tamponunun hazırlanması ve kullanılması, uygun bir kişisel koruma ekipmanıyla kimyasal bir davlumbaz altında yapılmalıdır.
- Başlamadan önce yapılacaklar
- Tüm ekipmanın son derece temiz olduğundan emin olun ve otoklavlanabilir herhangi bir cihazı otoklavlayın ( örneğin, taşlama silindiri, yüksek hızlı merkezTüpler vb. ).
NOT: Çapraz bulaşmayı önlemek için pipetleme gerektiren tüm adımlar için filtre uçları önerilir. - Gerekli ekipman ve reaktiflerin listesini görün ve Yöntem # 1 için gerekli tamponları ve çalışma stoklarını hazırlayın ( Tablo 1 ). Kriyojenik öğütme bloklarını -20 ºC'ye, rotorları ve tamponları 4 ºC'ye soğutun, mikrosantrifüjü 4 ºC'ye getirin ve 37 ºC su banyosu açın.
- Tüm ekipmanın son derece temiz olduğundan emin olun ve otoklavlanabilir herhangi bir cihazı otoklavlayın ( örneğin, taşlama silindiri, yüksek hızlı merkezTüpler vb. ).
- Organellerin izolasyonu
- 5 g taze dokuyu alın ve buz gibi soğutulmuş bir behergusta soğuk, steril suyla durulayın.
NOT: Santrifüjler, davlumbazlar vb. Ile yapılan tüm işlemler ve nakliye esnasında örnekleri daima buzda tutun . Alternatif olarak, protokolü gerçekleştirmek için yeterli alana ve ekipmana erişim varsa soğuk bir odada çalışın. - Makas kullanarak yaprak dokusunu ~ 1 cm'lik parçalar halinde doğrudan iki seramik öğütme içeren 50 mL tüp içine kessilindirler.
NOT: Çapraz bulaşmayı önlemek için örnekler arasındaki makasları temizleyin veya değiştirin. - Doku homojenleştiricisi yoksa, harç ve havlu kullanın ve adımlar 2.2.4 - 2.2.9'un yerine geçmek için izleyin.
- Yaprak dokusunu buz üzerinde önceden soğutulmuş harca kesin. Örnekleri 15 mL STE'de 2 - 3 dakika öğüttürün (davlumbazda).
- Önceden ıslak, steril bir filtreleme bezi (yaklaşık 22 ila 25 mikron gözenek boyutu, ayrıntılar için ana protokole bakın) içeren bir huni yoluyla tamponu (harç içindeki doku bırakın) başka bir 50 mL tüp içine boşaltın . Harca ve havaneye ilave 10 mL STE ekleyin ve tekrar homojenize edin.
- Homojenleştirilmiş dokuyu ve tamponu aynı huniye dökün. Harç ve havluyu 10 mL STE ile durulayın ve huniye dökün. Mümkün olduğunca çok sıvı almak için filtrasyon bezini huniye sıkıştırın ve sıkıştırın.
NOT: Çapraz bulaşmayı önlemek için örnekler arasındaki eldivenleri değiştirin. Profesyonelle devam etAdım 2.2.10'da
- Her 50 mL tüpe 20 mL STE (duman kabininde) ilave edin.
- Numuneleri bir doku öğütme aparatı içinde önceden soğutulmuş kriyojenik öğütme bloklarına yerleştirin ve 1,750 rpm'de 2 x 30 saniye boyunca öğüttürün. Örnek pozisyonlarını döndürün ve numuneleri öğütücüler arasında yaklaşık 1 dakika buz üzerine yerleştirin.
NOT: Bu basamakta bir havan ve havaneli karıştırıcı, blender veya diğer doku öğütücü / homojenleştirici cihaz kullanılabilir. Bununla birlikte, her yöntem elde edilen DNA kalitesini farklı derecelerde etkileyecektir ve bu nedenle, downstream uygulamalarla devam etmeden önce DNA uzunluğu ve kalitesi değerlendirilmelidir. - Buz içine yerleştirilmiş temiz bir 50 mL tüpe bir huni yerleştirin. Huni içine bir kat filtreleme bezi yerleştirin ve 5 mL STE ile önceden ıslatın. Akışı atmayın.
- Homojenleştirilmiş dokuyu huniye boşaltın. 15 mL STE ile öğütme tüpünü durulayın, özetleyin ve tüpü ters çevirerek duvarları ve kapağı durulayın ve huniye dökünEl.
- Seramik taşları dikkatlice çıkarın ve sıkın ve filtreleme bezi huniye sıkıştırın.
NOT: Çapraz bulaşmayı önlemek için örnekler arasındaki eldivenleri değiştirin. - Dökülmesini önlemek için tüp kapaklarını parafilm ile sarın. 4 ºC'de 10 dakika süreyle 2000 x g'de santrifüjleyin.
- Dikkatlice serolojik bir pipet kullanarak süpernatantı aspire edin (pelleti rahatsız etmemek için) ve 50 mL yüksek hızlı santrifüj tüpüne yerleştirin (tüplerde sıkı conta contaları yoksa dökülmeyi önlemek için tüp kapaklarını parafilm ile sarın). Peletleri atın.
- Tüpleri STE kullanarak 0.1 g içinde dengeleyin ve elde edilen süpernatanı 18.000 xg ve 4 ° C'de 20 dakika santrifüjleyin. Tüpleri dengelemek için, buz üzerinde küçük bir buz bulup buz koyun, teraziyi dara alın ve örnekleri soğuk tutmak için buz üzerinde tartın. Alternatif olarak, soğuk bir odada bir terazi ve davlumbaz kullanın.
- Süpernatanı atın. Peleğe 1 mL ST ekleyip nazikçe tekrar askıya alYumuşak bir fırça. 24 mL ST (son hacim 25 mL) ilave edin ve karışımı / girdaplı ( yani, tüm sıvıyı çıkarmak için tüpün yan tarafındaki boya fırçasına basın).
- ST'yi kullanarak tüpleri 0.1 g içinde dengeleyin. 18.000 xg'de ve 4ºC'de 20 dakika boyunca santrifüjleyin. Bu arada DNaseI solüsyonunu hazırlayın (stok ve çalışma solüsyonu tarifleri için Tablo 1'e bakın). Her numune için 1.5 mL'lik bir tüpte 200 μL'lik bir alikuot yapın.
- Süpernatantı atın, tüpü bloke edin ve yumuşak bir fırça kullanarak ST'nin 300 μL'sindeki peletini (hala yüksek hızda bir santrifüj tüpünde) yeniden askıya alın. Boya fırçasını, DNASI çözeltisinin 200 μL'ini içeren önceden hazırlanmış 1.5 mL tüp içine yerleştirin ve fırçaya yapışmış pelletlerin tümünü çıkarmak için fırça döner. DNaseI çözeltisini yüksek hızlı santrifüj tüpüne pipetleyin ve karıştırmak için hafifçe döndürünüz.
- 37 ° C'de 30 dakika su banyosunda inkübe edin (yoğuşmayı önlemek için tüpün üst kısmına sarın parafilmG kapağa koyun). İnkübasyon sırasında 2 kez dönüp hafifçe karıştırın.
- Pellet karışımını, geniş bir delikli bir pipet ucu kullanarak tüpün içinden yavaşça pipetleyin ve 1.5 mL düşük bağlama tüpüne yerleştirin. Yüksek hızda santrifüj tüpüne 500 mcL 400 mM EDTA, pH 8.0 ekleyin ve tüpün içindeki artık pelet miktarını elde etmek için yavaşça pipetleyin. EDTA'yı topak karışımı olarak aynı 1.5 mL'lik düşük bağlama tüpüne aktarın ve ters çevirme ile hafifçe karıştırın.
- 4 ºC'de 20 dakika boyunca 18,000 xg'de santrifüjleyin. Süpernatantı atın, tüpü silin ve DNA izolasyonu için bir kerede kullanın. Gerekirse, topakları -20 ºC'de dondurun, ancak artık DNaseI, hemen işleme tabi tutulmazsa, numune DNA'sını bozabildiğinden verim düşüşüyle sonuçlanabilir.
- 5 g taze dokuyu alın ve buz gibi soğutulmuş bir behergusta soğuk, steril suyla durulayın.
- Ticari sütun bazlı bir yaklaşım kullanarak izole organellerden DNA ekstraksiyonu
NOT: Tam protokol 25 için kit el kitabına bakın ve değişiklikler için aşağıya bakın. PrDoğrudan organel izolasyonundan DNA ekstraksiyonuna geçilmesi tercih edilir. Tekrarlanan dondurma ve çözme, DNA fragman boyutlarını azaltacak ve kalıntı DNaseI ile DNA bozunumuna neden olacaktır. Vortekslemeyi veya kuvvetli pipetlemeyi sınırlayın, çünkü DNA'yı kesebilir. DNA geri kazanımını en üst düzeye çıkarmak için düşük bağlamalı mikrosantrifüj tüplerinin kullanılması önerilir.- DNA özütleme prosedürü
NOT: Tamponların düzgün bir şekilde yapıldığından / depolandığından ve döner sütun prosedürlerinin anlaşıldığından emin olmak için, ayrıntılı ticari protokol 25'i okuyun.- Pelet ile doğrudan tüpe 180 μL Tampon ATL ekleyin (daha önce donmuş ve tezgah üstünde oda sıcaklığına kadar dengelenmişse çözülmüştür).
- Kit el kitabındaki "DNA Saflaştırmaları İçin Dokulardan DNA Arındırma" protokolünde 3. adıma geçin, aşağıdaki değişikliklerle birlikte: 3. adımda 30 dakikalık bir lizis, isteğe bağlı RNaz A sindirimini ve elüsyon için 3 x 200 μL AE ( Her biri bir şeyeParate tüp ve daha sonra elüsyonları birleştirin).
- QPCR için bir alikuot (en az 20 μL) kaydedin (adım 4.1'e bakın). Konsantre olmadan önce miktarı ölçmek için, yüksek hassasiyetli miktar tespiti için ek olarak 1 μL'lik bir tasarruf yapın.
- İsterseniz, örnek konsantrasyonu ile devam edin.
- DNA özütleme prosedürü
- Ticari filtre üniteleri ile numune konsantrasyonu
NOT: Daha fazla bilgi için ticari protokol 26'ya bakınız. Aşağı akım kullanımına bağlı olarak, örnek konsantrasyonu gerçekleştirmek gerekli olmayabilir ( örn., Son nokta PCR ve qPCR uygulamaları için). Bununla birlikte, NGS kütüphanesi inşası için, DNA ekstraksiyonu sonrasında elde edilen seyreltilmiş organellar DNA'nın konsantre edilmesi büyük olasılıkla olacaktır.- Konsantrasyon sütun prosedürü
- Dijital bir analitik terazi üzerinde temiz bir tartı kağıdı üzerine boş filtre ünitesini (bir tüp içermez) dikkatlice önceden tartın (bkz. Tablo 2 ). Ağırlığı kaydedin.
- pi sayısıBirleştirilen elüsyonları filtre ünitesine koyun ve dikkatle tekrar tartın.
NOT: Ticari el kitabı 26 , filtre ünitesinin maksimum hacminin 500 mcL olduğunu söyler, ancak birime aynı anda taşması olmadan üniteye 575 mcL'ye kadar eklenebilir. - Dolu filtre ünitesini bir tüpe (kolonlarla birlikte sağlanmış) dikkatlice yerleştirin. İstenilen konsantre hacmi elde etmek için istenilen süre için 500 xg'de santrifüjleyin. ~ 575 μL'lik bir numune hacmi için, 20 dakikalık bir sıkma genellikle 15-30 μL'lik bir konsantrasyon hacmiyle sonuçlanır.
- Filtre ünitesini tüpten çıkarın ve tekrar tartın. İstenen konsantre hacme ulaşılmış olup olmadığını belirlemek için tabloyu kullanın. Değilse tekrar 500 xg'de daha kısa bir süre için santrifüjleyin ve tekrar tartın; Istenilen konsantre hacme ulaşılana kadar tekrarlayın.
- Filtre ünitesinin üst kısmına yeni bir tüp (kolonlarla birlikte verilir) yerleştirin ve tersine çevirin. Co taşımak için 1000 xg'da 3 dakika boyunca santrifüjleyinTüpün içine konsantre ol.
- Kurtarılan hacmi belirleyin. Filtre tutma sebebiyle bu genellikle hesaplanan hacimden 3-5 μL daha düşük olacaktır. Aşırı konsantrasyon halinde, istenen hacmi elde etmek için steril su veya TE ile seyreltin.
- DNA'yı yüksek hassaslıkta niceleme kullanarak ölçün (üretici talimatlarına göre).
- Konsantrasyon sütun prosedürü
3. Yöntem # 2: Toplam Genomik DNA'dan Organelar DNA'yı zenginleştirmek için Metil-fraksiyonlama (MF) Yaklaşımı
NOT: Bu protokol, bitkiler ve mantarlar 27 ve ticari Microbiome DNA Zenginleştirme Kiti protokolü 28 için kullanıcı tarafından geliştirilmiş Genomik İpucu Kiti DNA ekstraksiyon protokolünden değiştirildi. Teorik olarak, yüksek molekül ağırlıklı DNA üreten herhangi bir DNA izolasyon protokolü, pulldown için kullanılabilir. Kısa okunan sıralama için, aşağı yukarı> 15 kb'lik fragmanlar veren herhangi bir ekstraksiyon, açılan yerde kullanım için yeterlidir. Merak ettimNg-okuma dizilemesi, daha büyük fragmanlar istenebilir. Bu nedenle, bu protokolü yüksek molekül ağırlıklı DNA üretmek için optimize ettik.
- Toplam DNA izolasyonu
NOT: Gerekli ekipman ve reaktiflerin listesine bakınız ve Yöntem # 2 için gerekli tamponları ve çalışma stoklarını hazırlayınız ( Tablo 1 ). Liziz tamponu çalışma solüsyonu yapmak için liziz tampon maddesine parçalayıcı enzimler ekleyin. Termomikeri açın ve 37 ° C'ye ayarlayın. Su banyosunu 50 ° C'ye getirin ve QF tamponunu banyoya yerleştirin. Dondurucuda% 70 EtOH yerleştirin ve mikrosantrifüjü 4 ° C'ye ayarlayın.- Ticari DNA ekstraksiyon sütunları kullanılarak toplam DNA ekstraksiyonu
NOT: Başlamadan önce, yerçekimi akış anyon değişim kolonları kullanımına ilişkin ayrıntılı bilgi için ticari el kitabı 29 okundu. Sütunlar, özel bir rafta kurulabilir veya sağlanan plastik halkaları kullanarak tüplerin üzerine yerleştirilebilir. G dahil olmak üzere tüm adımlarEnomik uçlar, yerçekimi akışı ile ilerlemesine izin verilmeli ve artık sıvı geçilmemelidir.- 2 mL'lik tüpler için tasarlanmış elle tutulan öğütme kalemi kullanarak 2 mL'lik düşük bağlama tüpünde sıvı nitrojende 20 mg donmuş doku öğüt.
- 2 mL liziz tamponu çalışma solüsyonu ekleyin (tüpler çok dolacaktır).
- 300 rpm'de hafif çalkalamayla 1 saat boyunca 37 ° C'de bir termomiksöre kuluçkalayın. Bir termomiksör yoksa, bir ısı bloğunda inkübe edin ve 15 dakikada bir hafifçe titreşerek karıştırın, uygun bir alternatiftir.
- 4 uL RNaz A (100 mg / mL, 200 μg / mL nihai konsantrasyon) ilave edin. Melez bir çalkantı ile 300 rpm'de karıştırılarak 37 ° C'de 30 dakika boyunca bir termomikserde inkübe edin.
- 80 μL proteinaz K (20 mg / mL, 0.8 mg / ml son konsantrasyon) ilave edin, karıştırmak için tersine çevirin ve 300 rpm'de yavaşça çalkalayarak 50 ° C'de 2 saat boyunca bir termomikserde inkübe edin.
- 4 ° C'de 20 dakika boyunca santrifüjleyin ve 1Çözülemez enkazın pelet haline getirilmesi için 5,000 xg.
- Numuneler santrifüj ederken, kolonları 1 mL Tampon QBT ile dengeleyin ve kolonun yerçekimi akışı ile boşaltılmasına izin verin.
- Denkleştirilen kolona numuneyi derhal uygulamak (peleti önlemek) ve kolondan tamamen akmasına izin vermek için geniş çaplı bir pipet ucu kullanın. Numune bulutlu hale gelirse, sütuna uygulamadan önce tekrar filtreleyin veya santrifüjleyin (ayrıntılar için ticari el kitabına bakın 29 ).
- Numune reçineye tamamen girdikten sonra, kolonu 4 x 1 mL Tampon QC ile yıkayın.
- Kolonu, temiz, 2 mL, düşük bağlamalı mikrosantrifüj tüpü üzerine asın. Genomik DNA'yı, 50 ° C'de önceden ısıtılmış 0.8 mL Tampon QF ile elute edin.
- Elde edilen DNA'ya 0.56 mL (0.7 hacim elüsyon tamponu) oda sıcaklığında izopropanol ekleyerek DNA'yı çöktürün.
- Inversiyon (10X) ile karıştırın ve 15,000 xg ve 4 ° C'de 20 dakika için hemen santrifüjleyin. BakımSerbest, gevşek bağlanmış pelet bozmadan süpernatantı tamamen çıkarın.
- Santrifüjlenen DNA pelletini 1 mL'lik soğuk% 70 etanol ile yıkayın. 15,000 xg ve 4 ° C'de 10 dakika boyunca santrifüjleyin.
- Peleti bozmadan süpernatantı dikkatlice çıkarın (bu adımla da dikkatli olun). Hava ile 5-10 dakika kurutun ve DNA'yı 0.1 mL elüsyon tamponu (EB) içine tekrar süspansiyon haline getirin. DNA gece boyunca oda sıcaklığında eritilir. DNA'yı kesebilecek pipetleme işleminden kaçının.
- Yüksek duyarlıklı bir DNA kantifikasyon testi kullanarak numuneleri ölçün (üreticinin talimatlarına göre).
- Ticari DNA ekstraksiyon sütunları kullanılarak toplam DNA ekstraksiyonu
- Metillenmiş ve metilleştirilmemiş DNA'nın boncuk bazlı fraksiyonasyonu
NOT: Yakın tarihli bir yayın, insan IgG Fc fragmanına (MBD2-Fc proteini) kaynaştırılmış bir CpG'ye spesifik metil bağlama alanı proteinini fraksiyona doğru kullanan bir açılma yaklaşımından yararlanan ticari olarak temin edilebilir bir kitin 28 kullanımını göstermiştirBitki organelleri genomlarını (metilleştirilmemiş) nükleer genomdan (yüksek metillenmiş) içeriği yedi 23 . Buğday numunelerindeki fraksiyonasyon etkinliği daha önce bu ticari MF kiti 28 kullanılarak test edilmemiştir.- Başlamadan önce yapılacaklar
- Taze olarak% 80 etanol (reaksiyon başına en az 800 μL) hazırlayın. Buz üzerinde çözülmesi için 5x bağ / yıkama tamponu ayarlayın ve numune başına 5 mL 1x tampon hazırlayın (protokol esnasında steril, nükleaz içermeyen su ile 5X tamponu seyreltin ve buz üzerinde tutun).
- MBD2-Fc proteinine bağlı manyetik boncukları hazırlayın
- Gerekli boncuk seti sayısını hazırlayın. Reaksiyonları 160 ve 320 μL boncuk gerektiren 1 ila 2 μg toplam girdi DNA'sı arasında kullanılacak şekilde ölçeklendirin. Aşağıda listelenen reaksiyonların 1 μg toplam girdi DNA'sı için olduğunu ve bu yüzden 160 μL boncuk gerektirdiğini unutmayın. Reaksiyonları ihtiyaçlara göre ölçeklendirin.
- Geniş çaplı ipuçları kullanarak, Protein A Manyetik B'yi yavaşça pipetleyinHomojen bir süspansiyon oluşturmak için yukarı ve aşağı çamurdan arındırın. Alternatif olarak, boncuk tüpünü 4 ° C'de 15 dakika yavaşça döndürün.
NOT: Boncukları vorteks vermeyin. - Üretici talimatlarına göre yönergeleri takip edin 28 .
- Metillenmiş nükleer DNA'yı yakalayın
- Her bir örnek için, 160 μL MBD2-Fc-bağlı manyetik boncuk içeren bir tüpe 1 μg giriş DNA'sı ekleyin.
- 1x nihai bir konsantrasyon için DNA girdi örneğinin hacmi göz önüne alındığında uygun olarak 5x bağ / yıkama tamponu ekleyin (eklemek için 5x bağ / yıkama tamponu hacmi (μL) = giriş DNA'sının hacmi (μL) / 4). Geniş çaplı pipet ucu kullanarak karıştırmak için numuneyi birkaç kez pipetleyin.
- Tüpleri 15 dakika boyunca oda sıcaklığında döndürün. Örnekleri geniş çaplı bir pipet ucu ile hafifçe pipetleyin ve örneklerin boncuk yığılmasını önlemek için kuluçka boyunca 2-3 kez fiske vurun.
NOT: pipetleme ve flickiMetilize DNA'nın etkili bir şekilde indirilmesini sağlamak için kritik önem taşır.
- Zenginleştirilmiş, metil olmayan organel DNA'yı toplayın
- DNA ve MBD2-Fc'ye bağlı manyetik boncuk karışımını içeren tüpü kısaca döndürün. Boncukları tüpün yanına toplamak için tüpü manyetik bir rafa en az 5 dakika boyunca yerleştirin. Çözüm açıkça görülmelidir.
- Geniş çaplı ipuçları kullanarak, boncukları rahatsız etmeden temizlenmiş yüzer malzemeyi dikkatlice çıkarın. Süpernatantı (metilleştirilmemiş, organelleri zenginleştirilmiş DNA'yı içerir), temiz, düşük bağlayıcı, 2 mL'lik mikrosantrifüj tüpüne aktarın. Bu örneği -20 veya -80 ° C'de saklayın veya saflaştırma için doğrudan adım 3.2.6'ya geçin.
- Elde edilen nükleer DNA, MBD2-Fc'ye bağlı manyetik boncuklardan alındı
- Nükleer fraksiyon da istenirse, MBD2-Fc bağlı manyetik boncuk nükleer DNA Zehir için üretici talimatları 28 izleyin; Adım 3.2.7'de açıklandığı gibi arındırın.
- Boncuk esaslı nükleik asit arıtımı
- Temizleme boncuklarının oda sıcaklığında olduğundan ve iyice karıştırıldıklarından emin olun. Protokolü, MF kiti el kitabı 28'deki talimatlara göre uygulayın.
NOT: Örnek şimdi NGS kütüphane inşası için veya başka bir aşağı doğru analiz için kullanılabilir.
- Temizleme boncuklarının oda sıcaklığında olduğundan ve iyice karıştırıldıklarından emin olun. Protokolü, MF kiti el kitabı 28'deki talimatlara göre uygulayın.
4. Örnekleme ve Kalite Kontrolü
- Organik zenginleşmeyi değerlendirmek için qPCR tahlili
NOT: Burada listelenen qPCR reaksiyonu ve analiz parametreleri, bir Roche LightCycler 480'de kullanılmak üzere tasarlanmıştır ve farklı ekipman ve reaktifler için ayarlanması gerekebilir. QPCR kullanılamazsa, burada tarif edilen aynı primerler ve koşulları kullanarak, numune saflığının kalitatif bir ölçümü olarak, bir agaroz jel üzerinde son nokta PCR ve görselleştirme kullanılabilir. Amplicon boyutları tüm primer setleri için ~ 150 bp olacaktır. Primer sekans için Tablo 3'e bakınız.Ces ve eşleştirmeler.- QPCR reaksiyonu ayarı
- Tek tek bir 20 uL qPCR reaksiyonu oluşturmak için, dikkatlice bir 96-kuyucuklu qPCR plakasının tek bir kuyusuna pipetleyin: 2 x SYBR Green I Master'ın 10 uL; 2 uL 10 uM ileri ve geri primer karışımı (0.5 uM'lik bir son konsantrasyon için); 2 uL şablon (standart eğri aralığı dahilinde); ve steril, nükleaz içermeyen H2 O. 6 uL pipetleme hatalarını azaltmak için, şablon dışındaki tüm tepkime bileşenleri ile bir ana karışımı yapmak için tercih edilir. Ana karışığı qPCR plakasına ekleyin ve daha sonra ilgi şablonunu her kuyuya ekleyin. Her örnek için üç teknik kopya pipetleme hatasının etkilerini en aza indirgemek için yapılmalıdır.
NOT: Nihai olarak, nükleer organellerin kantifikasyon çevrimlerine oranı örnekler arasında karşılaştırılarak konsantrasyonda ufak farklar kabul edilebilir. Bununla birlikte, DNA konsantrasyonları yaklaşık olarak ea aralığında olmalıdırDiğerini. - Plakayı yüksek kaliteli bir qPCR sızdırmazlık filmi ile mühürleyin. Örnekleri hafifçe vorteksleyin, kabarcıkların oluşmasını önlemek için özen gösterin. Numuneyi toplamak ve herhangi bir küçük kabarcığı ortadan kaldırmak için plakayı 4 ° C'de 2 dakika kısaca döndürün.
- Plakayı makineye yükleyin. QPCR programını aşağıda listelenen kurallara uygun olarak çalıştırın.
- Tek tek bir 20 uL qPCR reaksiyonu oluşturmak için, dikkatlice bir 96-kuyucuklu qPCR plakasının tek bir kuyusuna pipetleyin: 2 x SYBR Green I Master'ın 10 uL; 2 uL 10 uM ileri ve geri primer karışımı (0.5 uM'lik bir son konsantrasyon için); 2 uL şablon (standart eğri aralığı dahilinde); ve steril, nükleaz içermeyen H2 O. 6 uL pipetleme hatalarını azaltmak için, şablon dışındaki tüm tepkime bileşenleri ile bir ana karışımı yapmak için tercih edilir. Ana karışığı qPCR plakasına ekleyin ve daha sonra ilgi şablonunu her kuyuya ekleyin. Her örnek için üç teknik kopya pipetleme hatasının etkilerini en aza indirgemek için yapılmalıdır.
- QPCR reaksiyon parametreleri
NOT: Bunlar amplifikasyon aşamasının tavlama çevrimi haricinde varsayılan parametrelerdir. Kullanılanlar bu protokolde sunulan primerlerden farklıysa, bu ayarı belirli primerlere uyacak şekilde ayarlayın.- 4.4 ° C / s'lik bir rampa hızı ile 95 ° C'de 5 dakika ön inkübe edin.
- 4.4 ° C / s'lik bir rampa hızı ile (1) 95 ° C'de 10 saniye boyunca 45 amplifikasyon döngüsü gerçekleştirin; (2) 20 ° C için 60 ° C, 2.2 ° C / s'lik bir rampa hızı; Ve (3) 72 ° C'de 10 saniye boyunca, 4.4 ° C / s'lik bir rampa hızı ile (veri (3) sırasında elde edilen veriler).
- Optio kullanın4.4 ° C / s'lik bir rampa hızı ile 5 s boyunca 95 ° C'de nal erime eğrisi çevrimi; 65 ° C'de 1 dakika boyunca, 2.2 ° C / s'lik bir rampa hızı ile; Ve 97 ° C, sürekli bir alım modu.
- 30 ° C'de 40 ° C'lik bir soğutma çevrimi kullanın ve 1.5 ° C / s'lik bir rampa hızı kullanın.
- Deney parametreleri
- SYBR şablonunu seçin. Deneme düğmesindeki program parametrelerini kontrol edin. Bir kez plaka yüklendiğinde, deneme başlatılabilir ve ayarlar test çalışırken ayarlanabilir.
- Numune editörünü kullanarak numuneler atayın. İş akışı olarak Abs Quant'i seçin ve örnekleri bilinmeyen, standartlar veya negatif kontroller olarak belirleyin. Belirlemek çoğaltır ve her replikasyonun ilki örnek adlarını girin. Standartlara konsantrasyon ve birimler ekleyin.
- Analiz için alt grupları kurun; Bunlar alt set editöründe atanır.
- Analiz için, "yeni analiz oluştur" listesinden Abs Quant / 2nd Derivative Max'i seçin.Harici olarak kaydedilen standart eğrisi (varsa) içe aktarın ve daha sonra hesaplamayı yapın; Rapor seçilen bilgileri içerecektir.
- Kopya sayısının veya konsantrasyonunun belirlenmesi için kesin kesin nicelikleme yapmak için, test edilen örneği temsil eden standart bir eğri kullanın ( örn ., Yukarıdaki yöntemlerden izole edilen organellar DNA). Standart bir eğri hazırlamak için gereken mitokondriyal DNA miktarı, makul bir miktarda doku ile elde edilemeyeceğinden, yazılım tarafından sağlanan kopya sayısı hesaplamalarını kullanmayın; bunun yerine göreli zenginleşmeyi belirlemek için geçiş noktası (Cp) değerlerini inceleyin Örneklerde nükleer DNA ile karşılaştırıldığında organellar. Bu göreli miktarları toplam genomik DNA'yla karşılaştırın (bkz. Temsilci Sonuçlar ). Tam ışık yetiştirilmiş, iki haftalık buğday fidelerinden toplam genomik DNA'nın beş 1: 10 dilüsyonunda primer verimliliklerini test edin (bu rapordaki temsili verimliliklerŞekil 2'deki efsane).
- QPCR reaksiyonu ayarı
- Pulsed-field jel elektroforezi (PFGE)
NOT: Bu protokol, yüksek molekül ağırlıklı DNA'yı çözmek için PFGE'yi gerçekleştirmek için üretici talimatlarına dayanmaktadır. Malzeme Tablosuna bakın .- Jel ve örneklerin hazırlanması
- Jel ve numune hazırlama ile ilgili yönergeleri izleyin ve mevcut sisteme uyarlayın.
- Parametreleri çalıştır
- Elektroforez sistemini kurmak için yönergeleri izleyin ve aşağıdaki parametreleri kullanın: başlangıç geçiş süresi 2 s, son geçiş süresi 13 sn, çalışma süresi 15 saat ve 16 dakika, V / cm 6 ve dahil açılı 120 ° .
- Jel lekesi ve görüntüsü
- Jel, seçilen bir boya ile örtün ( örn. Etidyum bromür veya uygun bir alternatif) ve uygun bir jel dokümantasyon sistemi ile görüntü yapın.
- Jel ve örneklerin hazırlanması
- DNA Kitaplığı Hazırlık Kiti için üreticinin talimatlarına göre 1 ng DNA kullanın.
- Tek bir koşu ile sıralama için barkod ve havuz örnekleri. Üreticinin yönergelerine göre sıralamayı gerçekleştirin.
NOT: Havuzlama ve sıralama parametreleri, ilgi türüne, istenen kapsama seviyesine ve kütüphaneleri sıralamak için kullanılan platforma bağlı olarak değiştirilebilir. Örneğin, bir HiSeq şeridi MiSeq şeridine kıyasla çok daha fazla çıktı, bu nedenle çok daha fazla numune multiplekslenebiliyor. Organelar genomlarının kapsama seviyelerinin aşağı akım analizi için yeterli olup olmadığını belirlemek için daha küçük bir örnek alt kümesini sıralayın.- Veriler için gerekli kırpma ve filtreleme boyutunu belirlemek için FastQC 31'i kullanarak okuma kalitesini inceleyin.
- Ham okumaları, Trimmomatic 32 veya başka bir benzer program kullanarak süzün ve filtreleyin. Aşağıdaki ayarları kullanın: ILLUMINACLIP 2:30:10 (adaptörleri çıkarmak için), LİDER 3, DORSE 3, SLIDINGWINDOW 4:10 ve MINLEN 100.
- Kaliteli filtrelenmiş ve adaptör ile kesilmiş eşleştirilmiş uçlu (PE) okumaları, Çin Bahar mitokondriyeline (NCBI Referans Dizisi NC_007579.1 33 ), kloroplasta (NCBI Referans Sırası NC_002762.1 34 ) ve nükleer 35 referans genomlarına Bowtie2 36 , Aşağıdaki ayarlarla: -I 0 -X 800 - hassas.
- Sam uyum dosyalarını bam formatına (samtools) dönüştürür ve bam dosyalarını sıralar. Bam dosyalarını, geneli kapsayan kapsama alanını ve bedtools ile taban başına kapsamı hesaplamak için kullanın. Sonuçları R-plot fonksiyonuyla görselleştir.
- Başlamadan önce yapılacaklar
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Bu yazıda sunulan protokoller, organellar DNA'sını bitki dokusundan zenginleştirmek için iki farklı yöntem tarif eder. Burada sunulan koşullar, buğday dokusu için optimizasyonu yansıtır. Protokollerde, gerekli doku girişi ve DNA çıktısı için önemli adımların bir karşılaştırması Şekil 1'de açıklanmaktadır. Test ettiğimiz DC protokolünün basamakları, daha önce tarif edilenlere benzer koşulları izlemektedir ( Şekil 1A ). Hasat edilen doku taze işlenmeli ve sağlam organelleri izole etmek için diferansiyel santrifüj ve / veya gradyanlara tabi tutulmalıdır. Nükleer DNA, organeller yıkılmadan önce yok edilir ve nihayet, DNA ayrıştırılır ve aşağı doğru uygulamalar için kullanılır. Buna karşılık, MF protokolünde, bitki dokusu hasat edilebilmekte ve kullanılmadan önce saklanabilmektedir ve sağlam organellere ihtiyaç duyulmamaktadır. Bunun yerine, nükleer ve organellar DNA toplam gDNA'dan DNA'nın metilasyon durumu. Her iki protokol kabaca eşit miktarda organellar DNA üretir ( Şekil 1C ). Tek bir bitkiden küçük bir numune kullanılabilir olduğundan doku girdisine göre toplam organellar DNA çıkışı açısından, doku sınırlı olduğunda MF protokolü avantajlıdır ve bitki, daha ileri analiz için büyümesine izin verilebilir. Tipik olarak, DC protokollerinde, birçok fidenin hava dokularına ihtiyaç vardır ve bu bitkiler atılır. Bununla birlikte, DC yöntemi, MF yaklaşımı ile mümkün olmayan, bir organel türü için diğerini zenginleştirmek üzere optimize edilebilir. MF yaklaşımında daha az elle çalışma zamanı olmasına rağmen, her bir protokol için toplam sürenin kabaca eşit olduğu belirtilmelidir.
Her iki yöntem de farklı oranlarda olan Mitokondri ve Plastid Dizileri İle Organel DNA İçin Zenginleşir:
"Çok düşük oranda saflaştırılmış organellar DNA, her iki yöntemden de elde edilir (~ 50-100 ng seviyesinde; Şekil 1C ). Hem DC hem de MF'den izole edilen DNA'daki organellar genom zenginleştirmesi ve nükleer genom kirlenmesi düzeylerini değerlendirmek için Yöntemleri kullanılarak, bir qPCR tahlili kullanılmıştır Bu tahlilde, üç amplikonun ( yani, nükleer spesifik, ACTIN ; mitokondrial spesifik, NAD3 ; Kloroplasta spesifik, PSBB ) toplam genomik DNA'da değerlendirildi ve organellar DNA fraksiyonu her iki yöntemden elde edildi ( Şekil 2 ). Niceleme döngüsü (C q ) değerleri her numune için incelendi ( Şekil 2A ) ve C q hedef amplifikasyonun flüoresansının arka plan flüoresans seviyesinin üzerinde arttığı PCR döngüsü olarak tanımlandığından C q ve hedef bolluğun ters ilişki. İçindeDC örnek NAD3 ve psbB C q sırasıyla ~ 17 ve ~ 15 döngü önce (bir Cı q ~ 36 vardır) aktine göre (Cı-Q değerleri ile zenginleştirme seviyeleri için Şekil 2B) vardır. Bu, sırasıyla NAD3 ve PSBB için teorik 167.181 ve 47.790 kat zenginleştirme, DC örneğindeki ACTIN'e göre ( Şekil 2B , hesaplama için Şekil 2'nin efsanesine bakınız). Toplam genomik DNA örneğinde, ACTIN'e göre NAD3 ve PSBB'nin kat zenginleşmeleri sırasıyla yalnızca 158 ve 10,701'dir . Organelar genomlarının, hücre başına nükleer genomdan 37 daha fazla kopya sayısına sahip olduğu ve organellerin toplam genomik DNA'daki nükleik amplikon sayısıyla karşılaştırıldığında, organellar amplikonlann daha yüksek bir miktarının bulunması şaşırtıcı değildir 37 .R hücresi, doku türüne veya gelişme evresine 38 , 39 bağlı olarak farklılık gösterebilir. Genel olarak, veriler, beklenen mitokondri için DC yönteminin tercihli olarak zengin olduğunu göstermektedir; çünkü santrifüj hızları, mitokondriyayı seçici olarak izole etmek ve nükleer ve kloroplast "kontaminasyonu" azaltmak için optimize edilmiştir.MF total gDNA'sının metil olmayan fraksiyonu hem organelüler amplikonların önemli derecede zenginleştiğini gösterir ve bu hedeflerin yerli nispi miktarlarını muhafaza etmesi beklenir. Metillenmemiş fraksiyonunda aktine NAD3 ve psbB göreceli olarak kat zenginleşme, sırasıyla 20.551 ve 1.703.253, olan (Şekil 2A ve 2B). Metilatlanmış fraksiyonda ACTIN'e nazaran NAD3 ve PSBB'nin kat zenginleşmeleri sırasıyla 31 ve 823'türBu MBD2-Fc proteini, metillenmiş nükleer DNA'nın indirilmesinde son derecede etkilidir. Kloroplast amplikon toplam genomik DNA (~ 6 Cq önce), metile fraksiyonu (~ 5 ° C q önce) mitokondriyal amplikon daha yüksek miktarını ve metile edilmemiş bölümünü (~ 6 Cq önce) örnekleri vardır, bu düşündürmektedir Bu amplikonların doğal miktarı, MDB2 açılımı ile önemli ölçüde değişmemiştir. Burada, bu genomların spesifik dizilimine olan ilgiden ötürü, metilleştirilmemiş (organellar) fraksiyon üzerine odaklanıyoruz. Bununla birlikte, nükleer genom birincil ilgi ise, MF ve metilatlanmış fraksiyonun ardışık dizilişi, organellar DNA "kontaminasyonu" nun azaltılması nedeniyle, toplam genomik DNA dizilemesinden çok daha yüksek bir nükleer genom kapsama alanı sağlayacaktır.
Eğer qPCR yoksa, bitiş noktası PCR (qPCR ile aynı primerleri kullanarak) kaliteyi sağlarOrganellar saflığının değerlendirilmesi. Bu durumda, saf organelar DNA örnekleri, mitokondriyal ve plastid amplikonları için amplifikasyon gösterecek, ancak agaroz jel üzerinde nükleer amplikonun saptanabilir bir amplifikasyonu göstermezken, toplam genomik DNA, önceki üç çalışmada gösterildiği gibi, her üç primer kümesi için amplifikasyon gösterir. 11 , 12 .
Her iki yöntemden de izole edilen organellar DNA, NGS için uygundur:
Kesilmiş ve temizlenmiş PE dizilim okumaları (bkz. Adım 4.3) önceden yayınlanmış buğday organelleri referans genomları ile eşleştirildi ve her bir örneği haritalamak için kullanılan okuma miktarı 800.000 ila 1.100.000 okuma arasında değişiyordu ( Şekil 3I ). De novo Illumina diziliminin haritalanmasından elde edilen sonuçlar mevcut buğday kloroplastını ve mitokondri genomlarını qPCR res ile tutarlı olarak okurDC yöntemiyle, mitokondriyal DNA'da zenginleştirilmiş DNA üreten ( Şekil 3A ve 3B , okunanların ~% 80 ve ~% 10'u sırasıyla mitokondriyal (mt) ve kloroplast (cp) genomlarına eşlenir) ve MF yöntemi ( Şekil 3A ve 3B , ~% 20 ve okunanların ~% 80'i mt ve cp genomlarına sırasıyla) doğal zenginliğini yansıtan DNA üretirler. Her iki yöntemde de, her iki buğday organelleri genomunun teorik kapsamı (hesaplama için Şekil 3'ün efsanesine bakınız), 100X kapsamı aşar (ve MF yönteminden metil olmayan kısımdaki kloroplast genomu için ~ 2.000X kapsama aralığındadır), hatta 12 kütüphane multiplekslendiğinde ( Şekil 3C ve 3D ; bu analizde yer alan 6 kütüphane, ayrı bir analiz için ilave 6 kütüphane ile toplandı, toplam 12 kütüphaneTek bir dizilim şeridinde toplanır). Kapsamın daha ayrıntılı bir görünüşü, belirli derinliklerde ve aynı zamanda taban başına kapsama düzeylerinde kapsanan genomun fraksiyonunu inceleyerek elde edildi ( Şekil 3E- I ). MF yöntemi için taban tabaka başına ortalama kapsam, mt genomu için ~ 300 - 450X, cp genomu için ise 4000 - 5,000X idi. DC yöntemi için sırasıyla, mt ve cp genomları için, taban başı ortalama kapsama alanı ~ 900 - 1,300 ve ~ 500 - 700X idi. Bununla birlikte, hem mt hem de cp genomlarının çok düşük veya yüksek kapsama alanına sahip küçük bir kısmı vardı ve bu her iki yöntemden de türetilen organellar DNA'da görüldü ( Şekil 3I ). Ortalamadan daha yüksek olan bölgeler, muhtemelen organellar genomlar arasındaki homoloji bölgelerine karşılık gelir ve kapsama alanı düşük olan bölgeler, sıraladığımız çeşitler ile yayınlanan referanslar arasındaki SNP'leri veya diğer küçük varyasyonları gösterebilir. Bu düşünceyi desteklemek için, bu yükselmelerBüyük ihtimalle, MF yönteminden türetilen mt DNA'sı ( Şekil 3E ve 3I ) için, bu yöntemde cp genomunun yüksek kapsamı nedeniyle, en belirgin olmuştur. Açıklanamayan bir şekilde, cp genomunun kapsamı DC yönteminde ( Şekil 3G ve 3H ) MF yönteminde daha pürüzlüdür; bu, cp DNA boyunca MBD2-Fc açılımında hafif yanlılıklara neden olabilir. Bunun nedenini belirlemek için daha fazla deney yapılması gerekecektir. Ne olursa olsun, mt ve cp genomları, belirli bir derinlikte sıralanan genom fraksiyonunun incelenmesiyle gösterilebilen, her iki yöntemle de kapsama alanı bakımından eksikti ve kapsama alanlarının eksikliği yok ( Şekil 3E -3H ). Buna ek olarak, her iki genoma ait kapsam seviyeleri, varyant analizi gibi aşağı doğru analiz için yeterli kabul edilir. Nadir varyantların analizi için gerekli görülürse, numunenin azaltılmasıBirleştirilmiş numuneler daha büyük kapsama alanı sağlayacaktır. Alternatif olarak, HiSeq kitaplıkları şu an PE300 MiSeq kütüphanelerinin aksine PE150 uzunluğunda sınırlandığı için, dizi uzunluğunun bir fedakarlıkta olmasına rağmen, daha geniş bir dizi derinliğe erişirken HiSeq şeridine bir numara daha toplanabilir.
Bir haritalama yaklaşımı kullanarak nükleer genom kontaminasyon düzeylerini incelemek amacıyla, PE okuma haritalama kategorileri incelendi. PE okumaları, çeşitli konfigürasyonlarda referans genoma eşlenebilir. 1 ve 2 okumaları, iki arkadaş arasındaki belirli bir "beklenen" mesafeyle (kütüphanenin ortalama ekleme boyutuna ve genellikle haritalama yazılımında bir girdi parametresi olarak belirtilen) baştan başa modaya referansla hizalandığında ), Bu PE okumalarının "uyumlu" olarak eşlendiği söylenir. Buna karşılık, "uyumsuz" haritalama, eşlerin daha az veya daha fazla beklenen ile eşleştiği durumdur.Referans gene veya diğer konfigürasyonlarda (kafa-kuyruk veya kuyruk-kuyruk) eşleme. Eğer sadece bir arkadaşın referans genomu ile hizalanması halinde, o PE'nin okunduğunun ne uyumlu ya da uyuşmayan şekilde referans genomu gösterdiği söylenir. Üç okuma haritalama kategorisinde, PE okumaları referans genomuna bir veya birkaç kez hizalanabilir.
Hem DC hem de MF tarafından izole edilmiş organellar DNA için, mitokondriyal genomun okuma harfi ağırlıklı olarak uyumlu bir zaman kategorisinde ( Şekil 4A ) bulunmuştur ( Şekil 4A ), kloroplast genomuna eş zamanlı olarak eşit oranlarda eş zamanlı olarak bir kez morfik eşlemeli olarak eşlenik olarak daha fazla Bir zaman ( Şekil 4B ), muhtemelen kloroplast genomunda bulunan büyük ters çevrilmiş tekrarlardan ve ayrıca son derece yüksek kapsama düzeylerine bağlı olarak. Bununla birlikte, nükleer genoma eşlenen daha az PE okuması ve büyük orandaNe uyumlu ne de uyumsuz moda ( yani, sadece bir eşin haritalamasını yapabiliyor). Bunlar büyük olasılıkla nükleer genomdaki organeller genomları veya yanlış bir şekilde bir araya getirilmiş bölgelere homolog dizilere "hedef dışı" haritalandırılmasıdır. Ayrıca QPCR sonuçları ile yansıtılmaktadır sadece çok küçük bir miktar, (<% 5) DC MF yöntemi (Şekil 4C) izole organel DNA nükleer genom kirlenme düşük seviyelerini gösteren, buna paralel olarak nükleer genomu eşleştirilmiş okur (Şekil 2A ). Çin Bahar etiolasyonsuz dokulardan MBD2-Fc çekme işleminden sonra çekirdek fraksiyonu, aynı zamanda, açılmanın metilleştirilmemiş DNA'nın çıkarılmasında ne kadar etkili olduğunu belirlemek üzere dizildi. Nükleer fraksiyondan türetilmiş kütüphanedeki okumaların% 1'den azı, organellar referans genomlara eşlenirken, tüm okunanların% 45'i nükleer genom ile eşleştirilmiştir ( Şekil 4 ). Bununla birlikte, çoğu okuma uyumsuz bir şekilde haritalandı, wBuğday nükleer referans genomundaki yanlış montajın ve parçalanmanın yüksek seviyelerini büyük olasılıkla yansıtır. Ne olursa olsun sonuçlar, MBD2-Fc açılmasının metilleştirilmemiş organel DNA'nın metilasyona uğramış nükleer DNA'dan çıkarılmasında oldukça etkili olduğunu düşündürmektedir. Bu metodlardan kaynaklanan organellerin zenginleştirilmiş DNA'nın mitokondri ve kloroplast sekanslarının bir karışımı içerdiğinden ve bu organellerin arasındaki eski gen aktarımından kaynaklanan sekans benzerlikleri genomlarında kaldığından, okumaların spesifik Genomlar biyoinformatik olarak çözülmelidir.
Yaprak Dokusunun Etiolasyonu, Organolektif Bereketlerini Değerlendirememektedir:
Geleneksel olarak etyol dokular bitki mitokondriyal DNA izolasyonu için, fenolik ve nişasta seviyelerini düşürmek için ekstraktiyete müdahale edebilmek için tercih edilirN veya aşağı akım uygulamaları 13 . Organelar genom zenginleştirme düzeylerinin büyüme koşulları tarafından değiştirilip geliştirilemeyeceğini belirlemek için, hem etyollanmış hem de etil olmayan dokular MF protokolüne ve dizilişine tabi tutuldu. İlginçtir, etiyolasyon, etilleştirilmemiş koşullarla karşılaştırıldığında, organellar referans genomlarına ( Şekil 3A ve 3B ) veya taban başına kapsama alanına ( Şekil 3I ) eşlenen okumaların yüzdesini önemli ölçüde değiştirmedi. Organelar DNA'yı diferansiyel santrifüj kullanarak hem etiyole edilmiş hem de etil olmayan dokuları da izole ettik ve qPCR'yi kullanarak farklı dokular arasında zenginleşme açısından çok az fark bulundu (veriler gösterilmemiştir). Bu, fizyolojik açıdan anlamlı olan, etilasyona uğramamış dokuların, organelar dizileme çalışmaları için, zenginleştirmenin kayda değer bir değişikliğiyle kullanılabileceğini düşündürmektedir.
Kalite Kontrolü bunu öneriyorMF DNA, Uzun Okunan Dizileme İçin En Uygunudur:
Uzun süren okunan sıralama, araştırmacılar tarafından daha erişilebilir hale geldiğinde, yüksek molekül ağırlıklı DNA izolasyonu gittikçe önem kazanmaktadır. Organelar DNA'yı yalın ve kalitesiz yöntemlerle izole etmek için PFGE kullanıldı. Toplam genomik DNA tipik olarak PFGE'de yaygın bir bulaşma olarak göç eder ve molekül ağırlığı protokol ve DNA'nın depolandığı ve ekstraksiyon sonrası nasıl ele alındığı ile belirlenir. Genomik uçlarla izole edilen toplam genomik DNA, PFGE kullanılarak doğrulanan 50 kb'yi aşmalıdır ( Şekil 5 , şerit 2). Genomik uçlardan elde edilen toplam genomik DNA, organel DNA'dan nükleer fraksiyonlamaya yönelik Mikrobik Zenginleştirme Kitine girdi olarak kullanılır. Fraksiyonasyondan sonra elde edilen nükleer fraksiyon boyut olarak azalır, ancak yaklaşık 50 kb merkezli kalır ( Şekil 5 , şerit 4). Bu su değilMBD2-Fc-bağlı boncuklardan elüsyon olarak nükleer fraksiyonun nispeten sert kullanımının ısı ve proteinaz K sindirimini gerektirmesi nedeniyle, şaşırtıcıdır. Sınırlı kütleden ötürü, organellar fraksiyonu PFGE üzerinde çalışılmadı, ancak TapeStation ile yapılan analizler DNA> 50 kb'yi gösterdi (veriler gösterilmemiştir). Diferansiyel santrifüj ile elde edilen organellar DNA'sı ~ 20 kb'lik ortalama bir kütleye sahiptir, büyük olasılıkla uzatılmış organellar izolasyon protokolü ve daha sonra kolon bazlı DNA özütleme ve konsantrasyonundan kaynaklanmaktadır. Gradyan esaslı organellar izolasyon ve alternatif DNA özütleme yöntemleri, daha büyük DNA fragmanı boyutlarını koruyabilir. Ne olursa olsun, bu protokolde elde edilen boyutun DNA'sı, kütüphane hazırlığı sırasında özen gösterilirse 10 veya 15 kb dizilim okumaları üretmek için kullanılabilir.
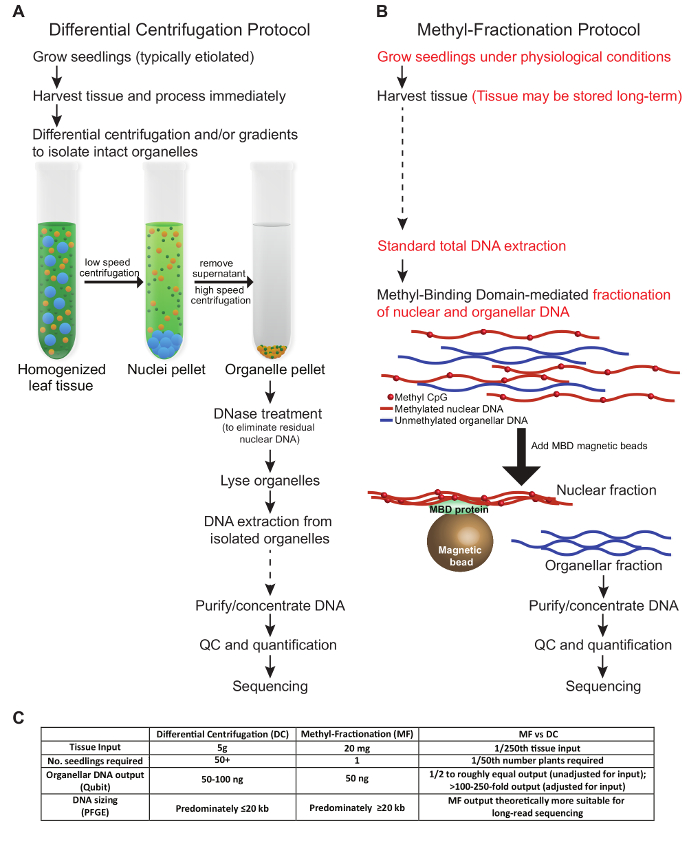
Şekil 1: İki Meto'nun Karşılaştırmalı Bir GörünümüBitki Organelleri DNA'sını zenginleştirecek ds. Geleneksel bir DC protokolü ( A ), MF protokolü ( B ) ile karşılaştırılmıştır. Örneklerin dondurulması ve çözülmesinden kaçınılması önerilir; Bununla birlikte, numunelerin uzun vadeli depolanabildiği adımlar kesik çizgili oklarla ( A ve B ) gösterilmektedir. Protokoller arasındaki temel farklar kırmızı renkle vurgulanır ( B ). ( C ) Tabloda doku girişi, gerekli bitki sayısı, DNA çıkışı ve elde edilen DNA boyutu bakımından yöntemler karşılaştırılmaktadır. Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.

Şekil 2: İki Yöntemle İzole Edilen Organellar DNA'da Nükleer DNA Kirliliğinin Değerlendirilmesi. (
(B) Tablo (A) 'da grafikte gösterilir Cq değerleri ve aktine organel amplikonlar nisbetle kat zenginleştirme gösterir. * Katlama zenginleştirmesi = 2 (Cq ACTIN - Cq Hedef) . Formül, her primer seti için 2 mükemmel bir verimlilik varsayar, çünkü minör deviatIyonu önemsizdir ve hesaplama ve genel eğilim üzerinde çok az etkili olur ( ACTIN = 1.961, NAD3 = 1.95 ve PSBB = 1.989). Primer etkinlikleri, toplam genomik DNA'nın beş 1: 10 dilüsyon serisiyle standart bir eğri yapılarak değerlendirildi. Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.

Şekil 3: Okunan Haritalama ve Kloroplast ve Mitokondrial Genomların Teorik Kapsamı. Mitokondrial ( A ) veya kloroplast ( B ) Çin Bahar referans genomlarına eşlenen okumaların yüzdesi. Çin Bahar mitokondrial ( C ) veya kloroplast ( D ) referans geno'nun ilgili teorik kapsamıMes, toplam okunan sayıları ve farklı genomlara eşleme okuma yüzdesini kullanarak hesaplanan sırasıyla 450 ve 135 kb genom boyutları varsayılır. Organellar DNA için MF yönteminden ( E ve G ) veya DC yönteminden ( F ve H ) genom çapında dağıtım. Veri panel E - H Çin bahar solmuş örnek olan, ancak tüm diğer numuneler için benzeri bir eğilim göstermiştir. ( I ) Paneller A - D' deki tüm numuneler için taban başına ortalama, en düşük ve en yüksek kapsama alanı. "E" içeren örnek etiketler etiyolasyonlu örnekleri belirtir ve "NE" etiyolasyona uğramamış örnekleri belirtir. DC, diferansiyel santrifüj yöntemiyle izole edilen DNA'yı ve Unmetile ise, MBD2-Fc (MF protokolü) ile indirgeme işleminden sonra metilleştirilmemiş fraksiyonda bulunan DNA'yı belirtir. "Chris" etiketli numuneler, buğday Triticum aestivum'u belirtirChris. ' CS buğday Triticum aestivum 'Çin Baharı örneklerini belirtir. Not: Organeller ve nükleer genomların yanı sıra organellar genomları arasındaki antik gen transferinden kaynaklanan kloroplast, mitokondri ve nükleer genomlar arasındaki dizi benzerliği nedeniyle, ham okumaların küçük bir yüzdesi birden fazla genoma işaret edebilir. Ek olarak, organellar referans genomuna eşlenmeyen oklar bu şekilde gösterilmez. Dolayısıyla, burada gösterilen yüzdeler ( A ve B ) toplam% 100 değildir. Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.

Şekil 4: Buğday Nükleer Genomuna PE Eşlemeli Okundu. PE kategorilerinin yüzdesi Mitokondriyal (A) , kloroplast (B) veya nükleer (C) Çin Bahar referans genomlarına eşleme tiplerini okuyun. - E, etiyolasyon örnekleri ve - NE, etilen olmayan örnekleri belirtir. DC, diferansiyel santrifüj yöntemiyle izole edilmiş DNA'yı, Unmetile ise MF protokolünde MBD2-Fc ile indirgeme sonrasında metilleştirilmemiş fraksiyonda bulunan DNA'yı, ve Metillenmiş, MBD2-Fc indirgemesinden sonra nükleer fraksiyonu belirtir. "Chris" etiketli örnekler buğday Triticum aestivum 'Chris'i belirtir. CS, Triticum aestivum 'Çin Baharı' buğday numunelerini belirtir. Eşleştirilmemiş oklar gösterilmiyor. Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.
Oad / 55528 / 55528fig5.jpg "/>
Şekil 5: PFGE'yi kullanarak DNA kalitesinin incelenmesi. Buğday toplam genomik DNA'sı (şerit 2), diferansiyel santrifüjden (şerit 3) elde edilen buğday organelleri DNA'sı ve MBD2-Fc açılma yaklaşımı ile (şerit 4) MF'den sonraki nükleer fraksiyon (şerit 4)% 1 agaroz jeli üzerinde PFGE'ye Bir işaretleyici olarak kullanılan 1 kb'lik merdiven (kulvarlar 1 ve 5). Bu rakamın daha büyük bir versiyonunu görmek için lütfen tıklayınız.
| Tampon Adı | Yemek tarifi | notlar | Yöntem |
| STE Arabelleği | 400 mM sükroz, 50 mM Tris pH 7.8, 20 mM EDTA pH 8.0,% 0.6 (ağ / hac) polivinilpirolidon (PVP),% 0.2 (ağ / hac) sığır serumu albümini (BSA), 0% 1 (h / h) β-merkaptoetanol (BME) | Yalnızca sukroz, Tris ve EDTA içeren tampon karışımı bir ay öncesine kadar yapılabilir ve 4 ° C'de tutulabilir. PVP, BSA ve BME, kullanımdan hemen önce gerekli tampon miktarının bir miktarına taze eklenmelidir. | Yöntem # 1 |
| ST tamponu | 400 mM sükroz, 50 mM Tris pH 7.8,% 0.6 (ağ / hac) polivinilpirolidon (PVP),% 0.1 (ağ / hac) sığır serum albümini (BSA) | Yalnızca sukroz ve Tris içeren tampon karışımı bir ay öncesine kadar yapılabilir ve 4 ° C'de tutulabilir. ST tamponunun EDTA veya BME içermediğini ve daha düşük bir konsantrasyonda BSA içerdiğini unutmayın. | Yöntem # 1 |
| DNaz stoğu | 2 mg / ml DNaz, 0.15 M NaCl içinde, stok yoğunluğu 2 mg / ml'ye | -20 ° C'de 200 ul alikotlar saklayın. DNase çalışma solüsyonunu (örnek başına 200 ul DNase solüsyonu) hazırlamak için bkz.Aşağıdaki Tablo 1. DNase sindiriminin tüm ayrıntıları için aşağıdaki protokolün tamamını inceleyin. DNase çalışma solüsyonu taze hazırlanmalıdır. DNaz reaksiyonunu durdurmak için 400 mM EDTA pH 8.0 çözeltisi gereklidir (reaksiyonu durdurmak için nihai konsantrasyon 0.2 M EDTA'dır, ayrıntılar için tam protokol bakınız). | Yöntem # 1 |
| DNaz çalışma solüsyonu | ST Tamponu içinde 0.25 mg / ml DNase ve 20 mM MgCl2 | Numune başına taze, 200 ul hazırlayın. Gösterilen konsantrasyonlar nihai reaksiyon hacmi için, bu yüzden karışımı: 62.5 ul, 2 mg / ml Dnase (500 nihai ul reaksiyon hacmi göre) 4 ul (200 ul DNaz çözelti hacmine göre) 1 M MgCl2, ST tamponu 133.5 ul için 200 ul son hacim. | Yöntem # 1 |
| Liziz Tamponu | 20 mM EDTA pH 8.0; 10 mM Tris pH 7.9; 500 mM Guanidin-HCl; 200 mM NaCI; % 1 Triton X-100; 0.5 mg / ml lizan enzimleriTrichoderma harzianum | Enzimleri parçalama haricindeki bütün malzemeleri karıştırın ve oda sıcaklığında saklayın. Lizing enzimleri derhal kullanılmak üzere küçük bir alikotta taze olarak eklenmelidir. | Yöntem # 2 |
Tablo 1: Ev yapımı tamponların ve çalışma stoklarının tarifleri.
| Konsantrasyon Çalışma Sayfası | |||||||
| ÖRNEK ADI | Boş Cihaz Ağırlığı (g) | Dolu Cihazın Ağırlığı (g) | Dolum Hacmi (ul, doldurulmuş eksi boş ağırlıklar) | Birinci Devirden Sonra Ağırlık (20 dak *, g) | Birinci Devirden Sonra Hacim (ul, doluEksi boş ağırlıklar) | Ağırlık 2. Döndükten Sonra (X min *, g) | 2. Sikme Sonrası Hacim (ul, dolu eksi boş ağırlıkları) |
| Gerçek kurtarılan hacmin, hesaplanmış hacimden birkaç ul daha düşük olacağını unutmayın. | |||||||
Tablo 2: Konsantrasyon Çalışma Sayfası.
| isim | Genom Özgüllüğü | Gen Sırası Kaynağı | Sıra (5 '- 3') |
| Ta_ACTIN - F | Nükleer | Gramen İskelesi IWGSC_CSS_1AS_scaff_3272162: 10,663-12,557 | CAGGTATCGCTGACCGTATGA |
| Ta_ACTIN - R | Nükleer | Yukarıdaki ile aynı | GAAGGTAGGGCTGAACAAGAAAC |
| Ta_NAD3 - F | mitokondrial | NCBI üyeliği EU534409.1 | GGTGATGCCAGAAGTCGTTT |
| Ta_NAD3 - R | mitokondrial | Yukarıdaki ile aynı | CAGATCAATCTTGTTAGGAGGTACTG |
| Ta_PSBB - F | kloroplast | NCBI üyeliği KJ592713.1 | GCTACCTTTGCTTTGCTCTTCT |
| Ta_PSBB - R | kloroplast | Yukarıdaki ile aynı | GCTGCCTGTTTCCTTGTAGTT |
Tablo 3: qPCR Astarlarının Listesi.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Bugüne kadar, çoğu organellar sıralama çalışmaları, geleneksel DC yöntemleri üzerine odaklanarak spesifik DNA'yı zenginleştirir. Çeşitli bitkilerden organelleri izole etme yöntemleri tanımlanmıştır; yosun 40 ; Buğday 15 ve yulaf 11 gibi monokotlar; Ve arabidopsis 11 , ayçiçeği 17 ve kolza tohumu 14 gibi dikotlar. Çoğu protokol 13 , 14 , 15 , 16 , 17 yaprak dokularına odaklanırken bazıları tohumlar da dahil olmak üzere çeşitli doku türleri için uyarlanmıştır 11 . Protoplastlardan organellerin izolasyonu da gösterilmiştir 41 . Bununla birlikte, bu, tüm sistemlere uymaz ve ilgilenilen doku sınırlı olduğunda mümkün değildir. Bu orgaNellar izolasyon yöntemleri fizyolojik çalışmalar gibi spesifik deneyler için bozulmamış organelleri kurtarmak için tasarlanmıştır. Bu protokoller hantaldır ve spesifik organellar fraksiyonları izole etmek için çok etkili olan, ancak büyük bir doku girişi gerektiren ( örn., Sakkaroz veya Percoll gradyanları gibi) yoğunluk gradyentlerinin kullanılmasını gerektirir; buna bağlı olarak, 5 g'dan fazla kilogramın üstünde Doku tipi). Bununla birlikte, DC yöntemi, spin hızlarını ve yoğunluk gradyanlarını değiştirerek, mitokondri ya da kloroplast gibi spesifik hücresel fraksiyonları zenginleştirmek üzere optimize edilebilir. Buna karşılık, MF yaklaşımı daha az başlangıç materyali (20 mg) gerektirir, ancak mitokondriyal ve plastid DNA'lar, DNA ekstraksiyonu için kullanılan doku içindeki görece bolluklarına göre mevcut olacaktır. Bununla birlikte, MF protokolü karışık organellar DNA'yı izole etmek için alternatif bir yaklaşım sunar ve özellikle az miktarda dokuyla başlamak için faydalıdır.
T o organel izolasyon Aşağıdaki örnek, saflığı analiz, bugüne kadar en çalışma, sadece son nokta, PCR ve jel elektroforezi 11, 12 kullanır. Bu, numune saflığının adil nitel bir ölçüsüdür. Bununla birlikte, düşük amplifikasyon seviyeleri bir agaroz jel üzerinde görselleştirilemeyebilir. QPCR 14 gibi daha niceliksel kalite kontrol önlemleri içeren az sayıda rapor bulunmaktadır. Her iki yöntemden de izole edilen DNA numunesinin saflığının nicel bir değerlendirmesi için, numunede ne kadar nükleer DNA'nın kalacağını ve kloroplast DNA'ya nispi oranlarda mitokondriyel olduğunu belirlemek için qPCR ve sıralama kullanıldı. Burada değerlendirilen her iki yöntem de nükleer DNA'nın atılmasında etkili. Her iki yöntem de farklı oranlarda olmasına rağmen mitokondriyal ve kloroplast DNA karışımını üretir.
Karanlıkta büyüyen bitkiler (etiyolasyon), fenoliklerin azalması nedeniyle organellerin izolasyonunu kolaylaştırmaya yardımcı olduğu bildirilmektedirRef "> 13. Ancak, bu karşılaştırmada, ışıkla yetiştirilen numuneler üzerinde etyollanmış doku ile çalışmanın kayda değer bir avantaj bulamadık. Işıklandırılmış olarak ihtisaslaşmış kloroplastların oranı daha yüksek olmasına rağmen, toplam plastid sayısı, Kloroplast genomuna okuma oranına yansıyan, farklı ışık koşullarında değişmez Bu nedenle, farklı dokulardaki heterolog mikroorganizmaların değerlendirilmesi ya da farklı stres altındaki ya da ekspresyon analizleri için aşağı akım fonksiyonel analizleri için, genomik sekanslama yapılması önerilir. Fizyolojik olarak uygun koşullar altında yetiştirilen bitkiler.
Kısa okunan sıralama teknolojileri ile uygulama için, burada karşılaştırılan her iki teknik de, yeterli DNA miktarı ve kalitesini sağlar. Bununla birlikte, tek moleküllü sıralama uygulamaları için> 20 kb'lik uzun okuma elde etmek için daha yüksek kaliteli DNA gereklidir. Örneğin, ideal olarak> 1 μg saf orga20 kb'lik ekleme kütüphanesi hazırlıkları için dahili, düşük girdi protokolleri için molekül ağırlığı> 20 kb olan nellar buğday DNA'sı gereklidir 42 . Yeni geliştirilmiş, düşük girişli protokoller DNA gereksinimlerini ( örneğin, 50 ng veya daha da az 20 ) azaltabilir, ancak meydan okuma, kütüphane hazırlıklarına giren yüksek kaliteli, yüksek molekül ağırlıklı DNA'ya sahip olmaya devam etmektedir. Küçük fragmanların SMRTbell'e yerleştirilmesi ve kütüphanenin 43 boyut dağılımını ortadan kaldırması DNA'nın çoğunluğunun> 20 kb olması zorunludur. Evde DNA ekstraksiyon protokolleri ve DNA ekstraksiyonu için bir dizi ticari protokol denedik (gösterilmedi). Buğday yaprağı dokusu için, DNA miktarı ve kalitesi, özellikle uzunluk arasındaki en iyi denge, ticari bir kit 27 , 29 kullanılarak elde edildi. Bitki türlerine ve ilgilenilen dokuya bağlı olarak, alternatiVe ekstraksiyon protokolleri eşit derecede uygun veya daha verimli olabilir. Yine de, boyutunun> 50 kb yüksek molekül ağırlığı genomik DNA'sının ekstraksiyonunun ardından MBD2-Fc açılma yaklaşımı 28 ile fraksiyonlamanın, sınırlı başlangıç materyalinden uzun okunan sekanslama yapılmasına müsait olduğuna karar verdik. Gelecekteki çalışma, uzun eklemeli kütüphane hazırlanması ve daha sonradan uzun okunan sıralama için fraksiyonlamayı takiben gereken başlangıç maddesinin sınırlarını test etmelidir. Kritik olarak, bu yaklaşım DNA'yı bütün genom amplifikasyonuna gerek duymadan uzun okunan sekanslama için uygun olan tek bir yaprağın alt örneğinden izole etmek için sağlam bir yöntem sağlayabilir. Bu yaklaşımın ek doku türlerine kolayca adapte edilebileceğini ve diğer bitki türlerine genel olarak uygulanabilir olacağını öngörüyoruz. Doku miktarlarının sınırlandığı durumlarda (örneğin, geçiş çizelgesindeki bireysel nesillerdeki sıralama veya daha nadir doku türleri) özellikle yararlı olacaktır.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yazarlar, rekabet eden çıkarlarının olmadığını beyan ettiler.
Bu yayındaki ticari isimler veya ticari ürünlerin açıklamaları, yalnızca spesifik bilgi sağlamak amacıyla verilmiştir ve ABD Tarım Bakanlığı'nın tavsiyesi veya onayını içermez. USDA eşit fırsat sağlayıcısı ve işverendir.
Acknowledgments
Birleşik Devletler Tarım-Tarımsal Araştırma Servisi ve Ulusal Bilim Vakfı'ndan (IOS 1025881 ve IOS 1361554) fon sağlandığını kabul etmek istiyoruz. R. Caspers'a sera bakımı ve bitki bakımı için teşekkür ederiz. Ayrıca, Illumina kütüphanesi hazırlıklarının ve sıralama işlemlerinin yapıldığı Minnesota Üniversitesi Genomik Merkezi'ne teşekkür ediyoruz. Dergi editörlerinin yorumları ve el yazmalarımızı daha da güçlendiren dört anonim gözden geçiren için de minnettarız. OECD'ye, Japonya'daki meslektaşları ile ortak projeler için bu protokolleri entegre etmek için SK'ye bir burs için teşekkür ediyoruz.
Materials
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol (beta-mercaptoethanol; BME) | Sigma Aldrich | M3148-100ml | |
| 2-propanol (Isopropyl alcohol/isopropanol), bioreagent | Sigma Aldrich | I9516 | |
| agarose, Bio-Rad Cetified Megabase agarose | Bio-Rad | 1613108 | |
| analytical balance | Mettler Toledo | AB54-S | |
| balance | Mettler Toledo | PB1502-S | |
| bovine serum albumin (BSA) | Sigma Aldrich | B4287-25G | |
| Ceramic grinding cylinders, 3/8in x 7/8in | SPEX SamplePrep | 2183 | |
| Cryogenic Blocks compatible with tissue homogenizer for holding 50 mL tubes | SPEX SamplePrep | 2664 | |
| DNaseI | Sigma | DN25 | |
| ethanol, absolute | Decon Laboratories | 2716 | |
| Ethylenediamine Tetraacetic Acid (EDTA), 0.5 M Solution, pH 8.0 | Fisher | BP2482-500 | |
| gel imaging system | |||
| gel stain | Such as GelRed or Ethidium Bromide | ||
| grinding pestle, wide tip for 2 mL conical tubes | |||
| Guanidine-HCl, 8 M solution | ThermoFisher | 24115 | |
| LightCycler 480 SYBR Green I Master | Roche | 4707516001 | |
| liquid nitrogen | |||
| Lysing enzymes from Trichoderma harzianum | Sigma | L1412 | |
| Magnesium Chloride | G Bioscience | 24115 | |
| magnetic rack | ThermoFisher | A13346 | |
| microcentrifuge tubes, LoBind 1.5 mL | Eppendorf | 22431021 | |
| microcentrifuge tubes, standard nuclease-free 1.5 mL | Eppendorf | ||
| microcentrifuge, refrigerated | Sorvall | Legend X1R | Or equivalent product, must be capable of reaching at least 18,000 x g with rotors for 50 mL tubes, Oak Ridge tubes, and 1.5 mL tubes |
| microcentrifuge, room temperature | Eppendorf | 5424 | Or equivalent product, must be capable of reaching at least 18,000 x g with rotor for 1.5 mL and 2 mL microcentrifuge tubes |
| Microcon DNA Fast Flow Centrifugal Filter Units | EMD Millipore | MRCFOR100 | |
| Miracloth, 1 square per sample cut to fit funnel | EMD Millipore | 475855 | |
| NEBNext Microbiome DNA Enrichment Kit | New England Biolabs | E2612L | |
| parafilm | Parafilm M | PM992 | |
| plastic pots and trays | |||
| polyvinylpyrrolidone (PVP) | Fisher | BP431-100 | |
| Proteinase K | Qiagen | 19131 | |
| Pulsed-Field Gel Electrophoresis rig (e.g. CHEF DR III) | Bio-Rad | 1703697 | |
| purification beads, Agencourt AMpureXP beads | Beckman Coulter | A63881 | |
| QIAamp DNA Mini Kit | Qiagen | 51304 | |
| Qiagen 20/g Genomic Tip DNA Extraction Kit | Qiagen | 10223 | |
| Qiagen Buffer EB (elution buffer) | Qiagen | 19086 | |
| Qiagen DNA Extraction Buffer Set | Qiagen | 19060 | |
| QiaRack | Qiagen | 19015 | |
| qPCR machine (e.g. Roche Light Cycler 480) | Roche | ||
| qPCR plate sealing film | Roche | 4729757001 | |
| qPCR plate, 96 well plate | Roche | 4729692001 | |
| Qubit assay tubes | Life Technologies | Q32856 | |
| Qubit Broad Spectrum assay kit | Life Technologies | Q32850 | |
| Qubit High Sensitivity assay kit | Life Technologies | Q32851 | |
| RNaseA | Qiagen | 19101 | |
| Serological pipettes (20 mL) and pipet-aid | Fisher | 13-678-11 | |
| Small funnels, 1 per sample | |||
| Sodium Chloride | Ambion | AM9759 | |
| Soft paintbrush, 2 per sample | |||
| SPEX SamplePrep 2010 Geno/Grinder or another type of tissue homogenizer | SPEX SamplePrep | Or another comparable tissue homogenizer. If you do not have access to a tissue homogenizer, then grinding in a pre-chilled mortar and pestle will suffice (see protocol for details). However, a homogenizer will give more consistent results and total homogenization time is reduced. | |
| Sucrose | Omnipure | 8550 | |
| TBE | |||
| thermomixer | |||
| Tris | Sigma | T2819-100ml | |
| Triton X-100 | Promega | H5142 | |
| tube rotater | |||
| tubes, 50 mL conical polypropylene | Corning | 352070 | |
| tubes, 50 mL high-speed polypropylene | ThermoScientific/Nalgene | 3119-0050 | e.g. Nalgene Oakridge tubes or equivalent |
| vermiculite | |||
| water bath | |||
| water, sterile and certified Nuclease-free | Fisher | 1481 | |
| water, sterile milliQ |
References
- Liberatore, K. L., Dukowic-Schulze, S., Miller, M. E., Chen, C., Kianian, S. F. The role of mitochondria in plant development and stress tolerance. Free Radic Biol Med. 100, 238-256 (2016).
- Samaniego Castruita, J. A., Zepeda Mendoza, M. L., Barnett, R., Wales, N., Gilbert, M. T. Odintifier--A computational method for identifying insertions of organellar origin from modern and ancient high-throughput sequencing data based on haplotype phasing. BMC Bioinformatics. 16 (232), 1-13 (2015).
- Zhang, T., Zhang, X., Hu, S., Yu, J. An efficient procedure for plant organellar genome assembly, based on whole genome data from the 454 GS FLX sequencing platform. Plant Methods. 7 (38), 1-8 (2011).
- Wambugu, P. W., Brozynska, M., Furtado, A., Waters, D. L., Henry, R. J. Relationships of wild and domesticated rices (Oryza AA genome species) based upon whole chloroplast genome sequences. Sci Rep. 5 (13957), 1-9 (2015).
- Iorizzo, M., et al. De novo assembly of the carrot mitochondrial genome using next generation sequencing of whole genomic DNA provides first evidence of DNA transfer into an angiosperm plastid genome. BMC Plant Biol. 12 (61), 1-17 (2012).
- Park, S., et al. Complete sequences of organelle genomes from the medicinal plant Rhazya stricta (Apocynaceae) and contrasting patterns of mitochondrial genome evolution across asterids. BMC Genomics. 15 (405), 1-18 (2014).
- Skippington, E., Barkman, T. J., Rice, D. W., Palmer, J. D. Miniaturized mitogenome of the parasitic plant Viscum scurruloideum is extremely divergent and dynamic and has lost all nad genes. Proc Natl Acad Sci U S A. 112 (27), E3515-E3524 (2015).
- Wicke, S., Schneeweiss, G. M. Chapter 1. Next Generation Sequencing in Plant Systematics. Hörandl, E., Appelhans, M. , Koeltz Scientific Books. (2015).
- Sloan, D. B. One ring to rule them all? Genome sequencing provides new insights into the 'master circle' model of plant mitochondrial DNA structure. New Phytol. 200 (4), 978-985 (2013).
- Woloszynska, M. Heteroplasmy and stoichiometric complexity of plant mitochondrial genomes--though this be madness, yet there's method in't. J Exp Bot. 61 (3), 657-671 (2010).
- Ahmed, Z., Fu, Y. B. An improved method with a wider applicability to isolate plant mitochondria for mtDNA extraction. Plant Methods. 11 (56), 1-11 (2015).
- Ejaz, M., et al. Comparison of small scale methods for the rapid and efficient extraction of mitochondrial DNA from wheat crop suitable for down-stream processes. Genet Mol Res. 13 (4), 10320-10331 (2014).
- Eubel, H., Heazlewood, J. L., Millar, A. H. Isolation and subfractionation of plant mitochondria for proteomic analysis. Methods Mol Biol. 355, 49-62 (2007).
- Hao, W., Fan, S., Hua, W., Wang, H. Effective extraction and assembly methods for simultaneously obtaining plastid and mitochondrial genomes. PLoS One. 9 (9), e108291 (2014).
- Pomeroy, M. K. Studies on the respiratory properties of mitochondria isolated from developing winter wheat seedlings. Plant Physiol. 53 (4), 653-657 (1974).
- Taylor, N. L., Stroher, E., Millar, A. H. Arabidopsis organelle isolation and characterization. Methods Mol Biol. 1062, 551-572 (2014).
- Triboush, S. O., Danilenko, N. G., Davydenko, O. G. A method for isolation of chloroplast DNA and mitochondrial DNA from Sunflower. Plant Mol Biol Rep. 16 (2), 183-189 (1998).
- Pinard, R., et al. Assessment of whole genome amplification-induced bias through high-throughput, massively parallel whole genome sequencing. BMC Genomics. 7 (216), 1-21 (2006).
- Lamble, S., et al. Improved workflows for high throughput library preparation using the transposome-based Nextera system. BMC Biotechnol. 13 (104), 1-10 (2013).
- Raley, C., et al. Preparation of next-generation DNA sequencing libraries from ultra-low amounts of input DNA: Application to single-molecule, real-time (SMRT) sequencing on the Pacific Biosciences RS II. bioRxiv. , (2014).
- Tsai, Y. C., et al. Resolving the Complexity of Human Skin Metagenomes Using Single-Molecule Sequencing. MBio. 7 (1), e01948 (2016).
- Feehery, G. R., et al. A method for selectively enriching microbial DNA from contaminating vertebrate host DNA. PLoS One. 8 (10), e76096 (2013).
- Yigit, E., Hernandez, D. I., Trujillo, J. T., Dimalanta, E., Bailey, C. D. Genome and metagenome sequencing: Using the human methyl-binding domain to partition genomic DNA derived from plant tissues. Appl Plant Sci. 2 (11), 1-6 (2014).
- Noyszewski, A. K., et al. Accelerated evolution of the mitochondrial genome in an alloplasmic line of durum wheat. BMC Genomics. 15 (67), 1-16 (2014).
- Qiagen. QIAamp DNA Mini and Blood Mini Handbook. , 5th ed, Available from: https://www.qiagen.com/ch/resources/ (2016).
- E.M. Corporation. User Guide: Microcon Centrifugal Filter Devices. , Available from: http://www.emdmillipore.com/US/en/product/Microcon-DNA-Fast-Flow-Centrifugal-Filter-Unit-with-Ultracel-membrane,MM_NF-MRCF0R100 (2013).
- Qiagen. User developed protocol: Isolation of genomic DNA from plants and filamentous fungi using the QIAGEN Genomic-tip - (EN). , Available from: https://www.qiagen.com/ch/resources/ (2001).
- New England BioLabs, Inc.. NEBNext Microbiome DNA Enrichment Kit: Instruction Manual Version 4.0. , Available from: http://www.neb.com/~/media/Catalog/All-Products/371BCB5A557C462D95D1E45E15BBFEA3/Datacards or Manuals/E2612Manual.pdf (2015).
- Qiagen. QIAGEN Genomic DNA Handbook. , Available from: https://www.qiagen.com/ch/resources/ (2012).
- PacificBiosciences. Guidelines for Using the BIO-RAD® CHEF Mapper® XA Pulsed Field Electrophoresis System. , Available from: http://www.pacb.com/wp-content/uploads/Unsupported-Guidelines-Using-BIO-RAD-CHEFMapper-XA-Pulsed-Field-Electrophoresis.pdf (2016).
- Andrews, S. FastQC: A quality control tool for high throughput sequence data. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Ogihara, Y., et al. Structural dynamics of cereal mitochondrial genomes as revealed by complete nucleotide sequencing of the wheat mitochondrial genome. Nucleic Acids Res. 33 (19), 6235-6250 (2005).
- Ogihara, Y., et al. Structural features of a wheat plastome as revealed by complete sequencing of chloroplast DNA. Mol Genet Genomics. 266 (5), 740-746 (2002).
- International Wheat Genome Sequencing Consortium (IWGSC). A chromosome-based draft sequence of the hexaploid bread wheat (Triticum aestivum) genome. Science. 345 (6194), (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nat Methods. 9 (4), 357-359 (2012).
- Bendich, A. J. Why do chloroplasts and mitochondria contain so many copies of their genome? Bioessays. 6 (6), 279-282 (1987).
- Kumar, R. A., Oldenburg, D. J., Bendich, A. J. Changes in DNA damage, molecular integrity, and copy number for plastid DNA and mitochondrial DNA during maize development. J Exp Bot. 65 (22), 6425-6439 (2014).
- Ma, J., Li, X. Q. Organellar genome copy number variation and integrity during moderate maturation of roots and leaves of maize seedlings. Curr Genet. 61 (4), 591-600 (2015).
- Lang, E. G., et al. Simultaneous isolation of pure and intact chloroplasts and mitochondria from moss as the basis for sub-cellular proteomics. Plant Cell Rep. 30 (2), 205-215 (2011).
- Tobin, A. K. Subcellular fractionation of plant tissues. Isolation of chloroplasts and mitochondria from leaves. Methods Mol Biol. 59, 57-68 (1996).
- PacificBiosciences. Procedure & Checklist - 10 kb to 20 kb Template Preparation and Sequencing with Low (100 ng) Input DNA. , Available from: http://www.pacb.com/wp-content/uploads/Procedure-Checklist-10-20kb-Template-Preparation-and-Sequencing-with-Low-Input-DNA.pdf (2015).
- PacificBiosciences. Template Preparation and Sequencing Guide. , Available from: http://www.pacb.com/wp-content/uploads/2015/09/Guide-Pacific-Biosciences-Template-Preparation-and-Sequencing.pdf (2014).



