Summary
Immunolabeling metoder för att analysera distinkta populationer av mikrotubuli i hjärnans utveckling zebrafiskar beskrivs här, som är allmänt tillämpliga på andra vävnader. Det första protokollet beskriver en optimerad metod för immunolabeling stabil och dynamisk mikrotubuli. Det andra protokollet ger en metod för att bild och kvantifiera begynnande mikrotubuli specifikt.
Abstract
Mikrotubuli (MTs) är dynamisk och sköra strukturer som är utmanande att bilden i vivo, särskilt i ryggradsdjur embryon. Här presenteras Immunolabeling metoder för att analysera distinkta populationer av MTs i utvecklingsländer neuralrörsdefekter zebrafiskar embryot. Medan fokus ligger på nervvävnad, är denna metod allmänt tillämplig på andra vävnader. Förfarandena är optimerade för tidigt till mitten av-somitogenesis-stegs embryon (1 somite till 12 thoraxsegmenten), men de kan anpassas till en rad andra stadier med relativt små justeringar. Det första protokollet ger en metod för att bedöma rumslig distribution av stabila och dynamiska MTs och utföra en kvantitativ analys av dessa populationer med bildbehandling programvara. Detta synsätt kompletterar befintliga verktyg bild mikrotubulära dynamics och distribution i realtid, med transgena linjerna eller övergående uttryck märkta konstruktioner. Sådana verktyg är verkligen mycket användbara, men de inte skiljer lätt mellan dynamisk och stabil MTs. Förmågan att bild och analysera dessa distinkta mikrotubulära populationer har viktiga konsekvenser för förstå mekanismerna bakom cell polarisering och morfogenes. Det andra protokollet beskriver en teknik för att analysera begynnande MTs specifikt. Detta åstadkoms genom att fånga de novo tillväxt egenskaperna för MTs över tid, efter mikrotubulära depolymerization med den drog-nocodazole och en återhämtningsperiod efter drogen blekt. Denna teknik ännu inte har tillämpats till studien av MTs hos zebrafiskar embryon, men är en värdefull analys för att undersöka funktionen i vivo av proteiner inblandade i mikrotubuli församling.
Introduction
Mikrotubuli (MTs) är polymerer av α - och β-tubulin att monterar in linjär protofilaments, varav flera kombineras för att bilda ett ihåligt rör1,2. MTs är polariserade strukturer, med snabbväxande plus ändar och långsamt växande minus ändar som är förankrade på centrosome eller andra mikrotubulära-organisera center (MTOC)3. De novo MT bildandet initieras av kärnbildning på γ-tubulin ring komplexa (γ-TURC), vilket ger en mall för MT församling4. I någon cell, i kan två populationer av MTs skiljas det vänden över i olika takt. Dynamiska MTs utforska deras cellulära miljön genom att växla mellan faser av tillväxt och krympning i en process som kallas dynamisk instabilitet5. Till skillnad från dynamiska MTs, stabil MTs är icke växande och har en längre halveringstid än dynamisk MTs6.
Decennier av forskning i cellbiologi har tillhandahållit ett sofistikerat utbud av verktyg att studera MT struktur och funktion och resulterade i en stor kropp av kunskap om dessa cytoskeletal element. Till exempel, MTs spela en central roll i upprättande och underhåll av cell polaritet, som beror inte bara på deras inneboende polaritet, men också till differential subcellulär fördelningen av stabil kontra dynamiska MTs7, 8. däremot långt mindre förstås om MT arkitektur och funktion i mer komplex tredimensionell (3-D) miljöer, såsom ryggradsdjur embryot, delvis på grund av utmaningen som imaging MT cytoskelettet med hög upplösning. Trots denna begränsning, den senaste generationen av GFP-uttryckande transgena linjer etiketten MTs eller övergående uttryck för fluorescently-taggade MT markörer har ökat vår förståelse av de dynamiska förändringar som MTs genomgå och deras mobilnät och utvecklande roll i zebrafiskar embryot. Hela MT nätverket kan avbildas i transgena linjerna i vilken tubulin är antingen direkt märkt9 eller tubulin polymerer märks indirekt genom att använda MT-associerade proteiner Doublecortin-liknande-Kinas (Dclk) eller Ensconsin (EMTB)10, 11. Andra linjer (och konstruktioner) har genererats som möjliggör bedömning av MT inneboende polaritet av specifikt märkning MT plus ändar eller centrosome-förankrade minus slutar11,12,13, 14. kraften i dessa verktyg ligger i förmågan att studera MT dynamik i live, utveckla organismer. Sådana studier visat, till exempel fysisk och dynamisk distribution av MTs i viss cellpopulationer, orienteringen av mitotiska spindlar i vävnader genomgår morfogenes (en indikator på planet av celldelning), polariteten av MT polymeren När det gäller processer såsom cell töjning och migration, och MT tillväxttakt bestäms av komet hastighet9,13,15. Begränsning av dessa verktyg är att de inte lätt diskriminera mellan stabila och dynamiska MT populationer.
Teckning från rika cellbiologi litteratur, beskrivs immunolabeling metoder att bilden stabil och dynamisk MTs i zebrafiskar embryot här, som är ett komplement till användningen av transgena linjerna. Den utbredda användningen av sådana immunolabeling metoder i zebrafisk har hämmats något av svårigheten att bevara MT under förfarandet för fixering. Protokoll 1 beskriver en optimerad metod för immunolabeling totalt, dynamisk, och stabil MTs i tvärsnitt av utveckla zebrafisk bakhjärnan. Dessutom beskrivs en enkel metod som använder kommersiellt tillgänglig programvara för att kvantifiera dessa MT populationer. Stabil MTs särskiljs från dynamiska MTs baserat på flera post-translationella modifieringar av α-tubulin, såsom acetylering och detyrosination, som ackumuleras på stabil MTs över tid16,17. I zebrafiskar embryot uppstår acetylering på strålkroppen och axonal MTs men inte på stabil interphase MTs18, begränsa användbarheten av denna markör till en delmängd av stabiliserad MTs. Däremot verkar detyrosination förekomma på alla stabila MTs i zebrafiskar embryo18. Denna post-translationella modifieringen exponerar karboxi-terminal glutaminsyra α-tubulin (detyrosinated tubulin)18 och kan upptäckas med hjälp av anti-Glu-tubulin19. Även detyrosination förekommer företrädesvis på stabil MTs, indikerar experimentella bevis att denna post-translationella modifieringen är ett resultat av, snarare än en orsak till, MT stabilitet16. Ömsesidiga MT befolkningen består av dynamiska MTs, utmärks med en antikropp, anti-Tyr-tubulin, som uttryckligen erkänner den tyrosinated formen av α-tubulin19. Efter immunolabeling med dessa markörer och confocal imaging, kvantitativ analys av MTs (längd, antal och relativa överflöd) kan utföras i definierade regioner i utvecklingsländerna neuralrörsdefekter. En strömlinjeformad metod finns här för att utföra denna analys med hjälp av 3D-bildbehandling programvara. Denna metod kan tillämpas för att hantera frågor om morfogenes och inrättande eller mognaden av cell polaritet20. Faktiskt, utarbetandet av polariserade matriser av stabil MTs medföljer många utvecklande händelser, inklusive ljusmätare morfogenes21, epithelialization av celler i utvecklingsländer neuralrörsdefekter18 och axon bildandet8.
Protokoll 2 beskriver en in-vivo -anpassning av en cellbiologi analysen att analysera MTs under deras församlingen fas (kärnbildning/anchorage och tillväxt)22,23. Begynnande MTs är nucleated på centrosome och därefter förankrade subdistal bihang av mor centriol23. En metod att analysera begynnande MT återväxt efter depolymerization beskrivs. Detta protokoll ger detaljer om nocodazole behandling till depolymerize MTs, förfarandet drog blekt och efterbehandling återhämtningsperioden. MT re-tillväxt övervakas vid regelbundna intervalls post bortspolning av immunolabeling med markörer för totala MTs (anti β-tubulin) tillsammans med markörer för centrosome (anti γ-tubulin) och nucleus (4', 6-diamidin-2-fenylindol (DAPI)), enligt de allmänna förfarandena beskrivs i protokoll 1. MT depolymerization steget i detta protokoll är viktigt eftersom det gör bedömning av de novo MT tillväxt snarare förlängning av redan existerande MTs. Denna teknik skiljer sig därför från andra offentliggjorda förfaranden att mäta MT tillväxttakter (i frånvaro av depolymerization) med hjälp av en plus spets märkaren såsom slutet bindande protein 3 smält till grönt fluorescerande protein (EB3-GFP), som visas i Tran et al., 201211. Dessutom denna analys är särskilt användbart för att analysera embryon defekt i de novo MT församling, såsom tidigare rapporterade NEDD1 mutanter som rekrytering av γ-tubulin till centrosome är nedsatt, vilket leder till ofullständig neuralrörsdefekter bildandet och neuronala defekter24.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
etik uttalande: förfaranden som beskrivs nedan Följ University of Maryland Baltimore County djurvård riktlinjer.
1. analys av stabila och dynamiska MTs använder Immunolabeling (protokoll 1)
- manuell dechorionation av embryon före fixering
- Erhåll lekt färska embryon genom att hälla bort överflödigt system vatten och sedan samla återstående embryon till en plast petriskål (se Tabell för material).
- Ta bort eventuellt skräp från system vatten och överföra embryon till en ny maträtt fylld med embryo medium (se Tabell för material) att säkerställa att embryona utvecklas i en ren miljö.
- De embryon som utvecklas till ett önskat Stadium i en temperaturkontrollerad inkubator på 28,5 ° C.
- Plats embryon yngre än 24 h efter befruktning (hpf) i en glasskål före dechorionation.
Obs: Dechorionate embryon före fixering att maximera snabb penetration av fixativ och bevara MT integritet. Användning embryo medium i stället för system vatten att ge den ytterligare Ca 2 + krävs under dechorionation. - Manuellt ta bort chorions från embryon medan i petriskål, med fina tången i dissekera Mikroskop.
- Nypa ett litet område i runda, genomskinliga chorion som omger embryot med ett par pincett och försiktigt dra tången isär att skapa en bristning i membranet.
- Förstora öppnandet genom att försiktigt bända på brusten chorion använda pincett. Var noga med att inte röra embryot med tången som det kunde brista.
- Fixering av stegvis embryon
- överföring iscensatt, dechorionated embryon till 1,5 mL centrifugrör. Ta bort så mycket embryo medium som möjligt med hjälp av ett glas Pasteur-pipett.
Obs: Utför fixering och läkemedel behandlingar på ung (mid-somitogenesis) embryon, före bildandet av neurala centers medla smärta förnimmelsen, som kräver inget tilläggsupplysningar förfarande att lindra smärta under dödshjälp. Utvecklingsstadier är enligt definitionen i Kimmel et al., 1995 25. De 4-5 och 11-12 somite stadierna användes för att få bilder för figurerna 2 och 3. - Förbereda 4% PARAFORMALDEHYD (PFA) / MT församlingen buffert (MAB) fixativ (se Tabell för material) genom att kombinera 1 mL 8% PFA per 1 mL 2 X MAB och lägga 2 µL 100% Triton x-100 per 1 mL totalvolym.
FÖRSIKTIGHET: Använd handskar vid hantering lösningar innehållande PFA och Triton x-100, som är hudirriterande. - Fix embryon i 1 mL 4% PFA/MAB fixativ för 5 min på 28,5 ° C. Aspirera fixeringsvätskan med en pipett, ersätta det med 1 mL färsk fixativ och Inkubera under 3 h i rumstemperatur (RT) på en rocker.
Obs: Prover måste fastställas snabbt till deras biologiska temperatur (28.5 ° C för zebrafisk) förhindra temperatur-anhörigen MT depolymerization.
- överföring iscensatt, dechorionated embryon till 1,5 mL centrifugrör. Ta bort så mycket embryo medium som möjligt med hjälp av ett glas Pasteur-pipett.
- Aspirera fixativ och tillsätt 1 mL 1 x Tris-buffrad koksaltlösning med NP40 (TBS-NP40)-buffert. Skaka försiktigt på RT på en rocker tre gånger för 5 min varje. Lagra embryon vid 4 ° C i 1 mL färsk 1 X TBS-NP40 för mer än 7 dagar.
FÖRSIKTIGHET: Använd handskar vid hantering av lösningar som innehåller NP-40, hudirriterande. - Sectioning embryon för immunolabeling
- värme RT 4% låg smältpunkt (LMP) agaros inbäddning medium i en stängd behållare tills lösningen blir klar med en värmeplatta inställd på 50 ° C placerade nära dissekera Mikroskop . Förvara behållaren stängd mellan prover och uppvärmd under hela inbäddning processen (steg 1.4.4-1.4.6).
- Överföring embryon från 1,5 mL Centrifugera rören till en petriskål med glas pipett och fyll den med 1 X TBS-NP40.
- Ta bort stor äggula cellerna från somitogenesis skede embryon (4-5 och 11-12 thoraxsegmenten) i petriskål med fin pincett under förstoringen av en dissekera Mikroskop 26. Håll embryot svans knopp med ett par pincett och skala bort äggula cellerna med det andra paret för att bevara hindbrain vävnaden. Överföra de-yolked embryona till ett område med äggula smuts petriskål.
Obs: Bädda in embryon i agaros-fyllda mögel individuellt för att förhindra för tidig härdning LMP Agarens. - Fylla en 12 mm x 5 x 3 mm väl av snittningen mögel med 200 µL smälte LMP agaros med hjälp av en mikropipett. Utför de steg 1.4.5.-1.4.6. snabbt (inom 20 s fylla formen) att bädda in embryot innan LMP Agarens kyler till RT och stelnar.
- Använd fin pincett för att överföra en de-yolked embryon av tailbud från petriskål till agaros-fyllda formen mot dess spetsiga änden under en dissekera Mikroskop.
- Använd fin pincett att orientera embryot in i formen så att vibratome skär i önskad planet. Skapa tvärgående sektioner genom att orientera embryot så att hindbrain vävnaden går parallellt med längden av mögel med dess dorsala ytan mot kanten och dess främre ytan inför avsmalnande regionen slutet. Upprepa steg 1.4.4-1.4.6 för återstående embryona.
- Tillåt den agaros inbäddning för att stelna i 5 min på RT.
- Generera 40 µm sektioner av högsta axeln Agarens inbäddade embryon (steg 1.4.1-1.4.7) med en vibratome med snittningen skålen fylld med 1 x TBS-NP40. Överföra delar av intresse till en 24-well platta i 500 µL 1 x TBS-NP40 med fin pincett. Placera delarna av endast ett embryo per brunn.
Obs: Se om du vill referera 18 för mer information. Säkerställa att sektioner förblir återfuktad på alla gånger i minst 250 µL buffert och rock vid låg hastighet (10-25 rpm) för resterande åtgärder för att förhindra separation från agaros inbäddning. Rengöringsmedel finns i blockering och tvätta lösningar bör minska ytspänningen i det flytande mediet och tillåta nedsänkning av avsnitten. Kontrollera att sektioner kvar i brunnarna under och efter alla manipulationer. Använd försiktighet för att förhindra oavsiktligt kasta sektioner under tvättar.
- Ta bort bufferten och tillsätt 500 µL blockerande lösning. Rock för minst 1 h på RT.
Obs: Använd en blockerande lösning som innehåller 5% sera från värdarter av varje sekundär antikropp som ska användas (se Tabell för material). - Inkubera i 300 µL primära antikroppar utspätt i blockerande buffert för 36-72 timmar vid 4 ° C på en rocker. Tvätta två gånger i 600 µL 1 x TBS-NP40 på en rocker för 30 min vardera, på RT.
Obs: Dubbel-etikett sektioner genom ruvning i primära antikroppar mot totala MTs (anti β-tubulin eller anti α-tubulin) och stabil MTs (anti-Glu-tubulin) eller dynamiska MTs (anti Tyr tubulin). Välj primär antikroppar som har tagits upp i annan värdarter när dubbel märkning för total och post-translationally modifierad α-tubulin populationer. Se Tabell för material för antikropp utspädningar. - Inkubera i 300 µL av fluorophore-konjugerad sekundära antikroppar utspätt i blockerande buffert på en rocker för 16-24 h, vid 4 ° C i mörker. Tvätta två gånger i 600 µL 1 x TBS-NP40 på en rocker för 30 min vardera, på RT.
Obs: Linda flera skålen som innehåller sekundära antikroppar i folie från denna punkt och framåt och efter varje manipulering att förhindra härdning. Välj sekundära antikroppar som reagerar med den värd immunglobulin av primär antikropp. Välj sekundär antikropp fluorophores som har separata, icke-överlappande utsläpp spectra. Se Tabell för material för antikropp utspädningar. - Inkubera embryon i 500 µL DAPI lösning på en rocker för 30 min, vid RT. Wash tre gånger i TBS-NP40 rocking på RT för 5 min varje.
Obs: Nuclear märkning ger cellulär sammanhanget för MT kvantifiering utförs i steg 1.12. - Placera en droppe monteringsmedium med anti fade agent på mitten av en dammfri bild. Använd fina pincett för att överföra sektioner till montering medium droplet-programmet. Placera ett dammfritt täckglas ovanpå provet. Lagra bilder i en torr, mörk och sval plats, insvept i folie, tills imaging utförs.
Obs: Kretsande avsnitten på baksidan av bilden med en spetsig permanenta märkpenna före imaging hjälper till att identifiera avsnitten när du använder mikroskopet. - Confocal Imaging
- Mount sektioner på en inverterad laserscanning confocal Mikroskop livsmedelsförpackningen bilden till scenen med den täckglas inför målet. Bestämma lämpliga optiken (mål, laser och kanalinställningar som få och offset) på en kontroll bild och hålla dem konsekvent mellan prover 27. Undvika oversaturating pixlar för att förhindra dataförluster.
- Fånga Z-stackar confocal bilder med kanalinställningar för den valda sekundär antikropp fluorophores och Spara bild filer 27. Förvärva Z-staplar för varje avsnitt.
Obs: Replikera de parametrar som används för att få bilderna i figurerna 2 och 3 med hjälp av följande förvärv inställningar: mode = XYZ; objektiva förstoring = 63 X olja nedsänkning lins; objektiva numeriska bländaröppningen = 1,4; Z-steg = 0.1 µm; Z-djup = 16,23 µm. Använd följande kanalinställningar: DAPI excitation med 20% UV-range laser, utsläpp filtrera intervallet = 430-480 nm, Fotomultiplikator (PMT) vinst = 525 V och PMT offset =-1.72%. 448 nm fluorophore (se Tabell för material) excitation med 20% 488 nm laser, utsläpp filtrera intervallet = 493-573 nm, PMT vinst = 689 V och PMT offset = -0,2%. 594 nm fluorophore excitation med 32% 594 nm laser, utsläpp filtrera intervallet = 608-706 nm, PMT vinst = 768 V och PMT offset = -6,8%. - Spara raw-datafiler med unikt, beskrivande filnamn och skapa en kopia för redigering i bild analysprogram.
- Sammanställning av Z-stackar för att Visa maximal projektioner
- öppna data fil kopia med offentlig domän 3-D bild analys programvara (t.ex., ImageJ). Kontrollera att varje kanal visas som en enskild bildsekvens (Z-stackar).
- Dela bild kanaler med hjälp av följande meny sekvens: “ bilder/färg/Split kanaler ”.
- Skapa en sammanslagen bild markeringsområde kanalerna sevärdheter genom att använda följande meny sekvens: " bilder/färg/Merge kanaler. " Välj 594 nm, 488 nm och DAPI kanaler vara falska färgade röda, gröna och blå, respektive. Kontrollera " skapa komposit " och välj " OK " 28.
Obs: Utelämna DAPI kanal för att bättre förmedla information specifik för MTs i en maximal projektion som i figur 2 och 3, genom bara välja falska färger för de andra två kanalerna. - Undersöka den sammanslagna Z-stacken och anteckna inledande och avslutande positionerna för de inre bästa Z-plan för alla synliga kanaler. Avfärda de yttersta Z-plan som vanligtvis har suboptimala signal på grund av de ojämna ytorna av avsnittet. Se referens 29 för detaljer.
- Visualisera den sammanslagna Z-stacken som en 2D-bild genom att utföra en maximal intensitet projektion av Z-stacken använder följande 3D-bild analys menyn sekvens: " bilder/Stack/Z-projekt. " ange start- och slutdatum positioner av de inre bästa Z-plan från steg 1.11.3 som den " Start skiva " och " Stop slice, " respektive. Välj " Max intensitet " eftersom den projektionstyp och klicka " OK ". Se referens 28 för mer detaljer.
- Analysera MT märkning
- Öppna programvaran kommersiell 3D-bild analys. Välj " skapa bibliotek " och ange ett beskrivande namn för bildbiblioteket. Klicka " skapa. " dra RAW-filer som genereras från mikroskopet confocal in i biblioteket. Större filer kräver mer tid att överföra.
- Välj en fil att analysera. Välja " Extended fokus " från den " vyn " menyn för att Visa kanal sammanslagna bilden i huvudfönstret.
- Justera tröskelvärde genom att dra skjutreglaget verktyget för varje kanal till vänster eller höger tills bakgrunden signalen sänks och den sanna signalen är robust. Observera att varje kanal visar en sann signal för molekyl märkt (t.ex., DAPI-kanal som visar avlånga eller mitotiska kärnor men inte auto-fluorescens från cytoplasman eller agaros).
- Välj det " Freehand regionen av intresse (ROI) " verktyg och beskriva regionen av intresse som skall analyseras. Välj den " åtgärder " fliken följt av " Beskär till markering " att beskära bilden. Spara den beskurna bildfilen under ett nytt namn. Klicka på den " mätningar " fliken för att skapa protokollet för filtrering specifika objekt som är relevanta för 3D-analys.
- Drar " hitta objekt " till fönstret protokoll. Byta namn på det första protokollet " DAPI. " Välj DAPI kanal i listmenyn. Dra följande inställningar till protokollet DAPI och placera dem nedan " hitta objekt " i följande ordning (tabell 1): " fylla hål i ObjectsŔ " Separat röra ObjectsŔ " Utesluta objekt av SizeŔ " Utesluter inte röra ROIs ".
Obs: Målet med inställningarna i tabell 1 är att först ange en tröskel som ignorerar signaler vars fördelning och storlek är oförenliga med en storlek på objekt som analyseras. Till exempel eliminera en signal inte tillräckligt stor för att vara en kärna när räknar kärnor. - Genomför sekvensen (steg 1.12.9) att montera återstående filter för β-tubulin och andra markörer som använder inställningarna i tabell 1.
- Välj " åtgärd " på botten av varje protokoll. Välja " intensitet och volymmätning " och " skelett Längd " för alla tubulin märkning, men endast förstnämnda för signalen DAPI.
- Rita en ROI runt regionen skall mätas. Observera måtten under den " Sammanfattning " fliken efter programvaran bearbetar regionen. Kopiera data och spara dem till en fungerande kalkylblad, som i tabell 2. Skapa en säkerhetskopia av kalkylbladet för senare analys.
- Välj mätningar av intresse (till exempel längden på MT bundle, antal MT buntar/kärnan, som avslöjades med olika markörer) i kalkylbladet och analysera för att avgöra genomsnitt för varje grupp.
Obs: Den genomsnittliga MT bunt längden = summan av de ' menar skelett längd för β-tubulin ' för varje embryo dividerat med det totala antalet embryon. Se rad 20 i tabell 2. Formatera kalkylbladet så att variabler och experimentella grupper återgivningsegenskaper enkelt.
2. De Novo MT församlingen Assay (protokoll 2)
- konstruktion och test flera genomflöde apparaten ( figur 1) 2 dagar före experimentet.
Obs: Apparaten möjliggör samtidiga bortspolning av flera experimentella grupper efter nocodazole behandling med leveranser från Tabell för material. Silikon sealer kräver minst 24 h i torktid innan det utgör ingen toxicitet risk för embryon.- Split ett 50 mL centrifugrör i halv på längden, med en jigg eller bandsåg.
- Skär 7 semi-kretsar, med en radie av 3 cm, ur 70 µm nylon mesh och trimma dem att passa tätt till en hälften av Splits Centrifugera röret. Limma de semi-kretsarna i det parallellt med 10 mL gradering markeringarna med hjälp av akvarium-säker silicon sealer centrifugröret. Låta enheten torka i 2 dagar och skölj genom blötläggning i en bägare vatten för 2-3 h.
- Linje i topp (gängad) slutet av skurna centrifugalröret med modellera så att höjden av vätskan kvar i genomflöde enheten har ett djup på ¼ tum ( figur 1, 2 och 3).
- Förbereda blekt apparaten genom att ta bort kolven från en 50 mL spruta och infoga 12 inches av fina slangar i spetsen. Skjut slangen så långt det går och tätning runt gemensamma använder modellering lera.
- Våt pre mesh med embryo medium för att låta vätskan att köra igenom hela genomflöde enheten. Vinkel på enheten is så att vätska pooler i alla fack men ändå tömmer ut på framsidan där lera fälgen är belägen. Använda en ring stativ, upphäva blekt apparaten ovanför enhetens genomflöde på is ( figur 1).
- Chill 200 mL embryo medium på is och häll nog in i blekt apparaten så att alla luftbubblor är avmarkerade och att flödet är cirka 7 mL/min. justera flödet genom att ändra höjden på sprutan.
- Enzymatiskt dechorionate embryon
- göra en fungerande lösning av icke-specifika proteas genom spädning av 1 mL 10 mg/mL ospecifika proteas lager i 20 mL embryo medium.
- Utför kemiska dechorionation på embryon 1 h före tidpunkt när de förväntas nå önskad utvecklingsstadiet. Digest chorions genom att avlägsna embryot från 100 mm petriskålar som innehåller iscensatt embryon och tillsats av 20 mL ospecifika proteas arbetslösning.
- Inkubera embryon vid 37 ° C i 5 min.
Obs: Inte överstiga 5 min eller använda en högre koncentration av icke-specifika proteas lösning, eftersom detta kommer att resultera i embryon faller isär en gång behandlade med nocodazole. - Snabbt Pipettera ut icke-specifik proteas och fylla rätter med cirka 25 mL embryo medium. Upprepa en gång.
- Med 1 mL glas pipett överföring embryon yngre än 24 hpf till glas rätter för att skydda dem från skador.
- Komplett dechorionation genom att manuellt ta bort chorions med ett par fina pincetten, som beskrivs i steg 1.1.5.
- Placera glas petriskålar som innehåller dechorionated embryon i en inkubator på 28,5 ° C i minst 30 min tills de når önskad utvecklingsstadiet.
- Depolymerize befintliga MTs
- förbereda en fungerande lösning av 5 µg/mL nocodazole genom att kombinera 50 µL 1 mg/mL lager nocodazole med 10 mL is kallt embryo medium.
FÖRSIKTIGHET: Använd handskar vid hantering av nocodazole, en hudirriterande. - Utbyta embryo medlet av gruppen nocodazole behandling med 10 mL kallt nocodazole fungerande lösning. Placera petriskålar på is en lämplig tid för utvecklingsstadiet (exempelvis 1 h för 4-5 somite embryon). Avsätta kontrollgrupp embryon i en petriskål på is som skall fastställas tillsammans med blekt prover i steg 2.3.4.1.
- Överföring embryon med eld spegelglas 1 mL pipett till genomflöde utrustningen och med separata fack för varje experimentella gruppen. Starta den nocodazole bortspolning av hälla is kallt embryo medium in i toppen av 50 mL sprutan.
Obs: Använd minst 30 embryon per experimentella gruppen. Experimentella grupper kunde bestå av kontroll embryon eller en mängd morpholino eller RNA-injiceras embryon. Blekt kräver sammanlagt ca 150 mL embryo medium ska läggas var 8-10 min. blekt nocodazole medan man fortsätter att hämma MT tillväxt med is. Att hålla embryon på is är avgörande för framgången för denna analys eftersom MTs är instabila vid kalla temperaturer och kyla fördröjer utvecklingen i tidiga embryon. - Tillåta MTs till återföds efter 20 min av blekt på RT genom att överföra embryon till glas petriskålar som innehåller varma (28.5 ° C) embryo medium med eld polerade 1 mL glas pipett. Så snart embryona överförs, starta en timer.
- Fixa kontroll och blekt embryona på 1 min, 5 min och 10 min med pipett cirka 10 embryon till en 1,5 mL Centrifugera röret fylls med 1 mL 4% PFA/MAB fix (28.5 ° C) och följ anvisningarna i steg 1.2.3.
- förbereda en fungerande lösning av 5 µg/mL nocodazole genom att kombinera 50 µL 1 mg/mL lager nocodazole med 10 mL is kallt embryo medium.
- Förbereda prover för immunolabeling som beskrivs i avsnitt 1.3-1.5.
- Immunolabel flytande sektioner och bild embryon som beskrivs i avsnitt 1.6-1.10 med följande ändringar till primär antikropp specifikationer: användning 1: 500 kanin anti-γ-tubulin och 1: 200 musen anti-β-tubulin.
- Process och analysera bilder med 3D-bild analys programvara, som beskrivs i steg 1.12.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Analys av stabila och dynamiska MTs med immunolabeling
I protokoll nr 1avslöjas fördelningen av MT delpopulationer under tidig (neurala kölen) och sena (neurala stav) utvecklingsstadier neuralrörsdefekter, med Glu-tubulin och Tyr-tubulin som markörer för stabila och dynamiska MTs, respektive. Dynamiska MTs dominerar i hindbrain skede neurala kölen (4-5 thoraxsegmenten) (figur 2A-D). Som kölen utvecklas till neurala staven (11-12 thoraxsegmenten), ett skede av förbättrad epithelialization, är kvalitativt färre MTs immunreaktiva med anti-Tyr-tubulin antikroppen (figur 2E-H), särskilt i ventrala staven. Däremot Glu-tubulin är spridda och punktuell hela neurala kölen (figur 3A-D), men anrikas i ventrala neurala stången längs MT skrifter (figur 3E-H). Pilspetsar peka på specifika MT buntar eller strukturer där märkning ökas.
Även om både anti-Glu-tubulin och anti-Tyr-tubulin antikroppar producerades i samma värdart (förhindra en dubbel märkning experiment), indikerar dessa resultat att stabila och dynamiska MT markörer sällan överlappar i zebrafisk bakhjärnan. För det första har den ventrala neurala rod stabilare (figur 3F) än dynamisk (figur 2F) MTs. Trenden är omvänd i dorsala neurala staven, överensstämmer med en modell av zebrafisk neurulation där den dorsala vävnaden förblir dynamiska tills neuralrörsdefekter är bildas20. För det andra, medan mitotiska spindlar är fullt märkta med Tyr-tubulin antikroppen i neurala kölen (figur 2D, pilspetsar), bara basen av spindeln, sammanfaller med centrosome, är märkt med stabilitet markören () Glu-tubulin Figur 3 D, pilspetsar). Β-tubulin immunofluorescens, gemensamma för båda analyser, informerar experimenter fördelningen av alla MTs och ger en grund för att avskeda icke-specifik märkning.
Mäta objekt med 3D-bild analys programvara resulterar i en stor mängd data som ordnas i en praktisk tabell (tabell 2). För att göra längd, antal och området mätningar, använder vi bara en delmängd av de data som är tillgängliga för att analysera. En av komponenterna i de data som vi inte ytterligare analysera är antalet objekt identifieras. Detta nummer används som en intern kvalitetskontroll, som numret inte ska varierar kraftigt mellan som sektioner och förhållandet mellan atomkärnor MTS bör stanna liknande i en enda behandling skick. En avvikare är en indikator som antingen analysen behöver köras igen med justerade filter eller som bilden heter alltför dåligt för att analysera. Således bör alla avvikare bilder vara reanalyzed med justerade inställningar. Avsnittet avvikare bör undersökas för tecken på dålig märkning eller fysiska skador som kan resultera i ovanligt objekt räknas. När analysen är klar och kvalitet kontrolleras, användbar information kan återvinnas från rådata som genomsnittlig längd av totala MTs och stabil MTs eller förhållandet mellan stabil MTs till totala MTs (tabell 3). Utöver dessa mätningar, kan många andra mätvärden erhållas med hjälp av 3D-analys bildbehandlingsprogram som kan användas för att dra slutsatser om MTs eller deras förhållande till andra cellulära strukturer (nucleus, centrosome, etc.).
De novo MT församlingen assay
Nocodazole behandling depolymerizes MTs resulterar i diffusa märkning (figur 4A, 4 D, och 4 G). Som MTs återföds, de sträcker sig från centrosome (figur 4B, 4E, och 4 H), men detta kan inte vara självklart i ett enda plan på grund av deras icke-plana banor (figur 4C, 4F, och 4I). Vissa bildbehandlingsprogram för analys är dock kan mäta längder i 3-D, möjliggör en bedömning av MT tillväxt efter den nocodazole blekt (tabell 4). En viktig iakttagelse som kan erhållas från datamängden i tabell 4 är att den genomsnittliga längden på MTs förefaller öka över tid efter den nocodazole blekt i alla regioner av neuralrörsdefekter analyseras. Som nämnts ovan, kan andra typer av mätvärden erhålls från 3D-bild analys programvara ge cellulära sammanhang för att tolka MT data (till exempel förhållandet mellan MTs per kärna).
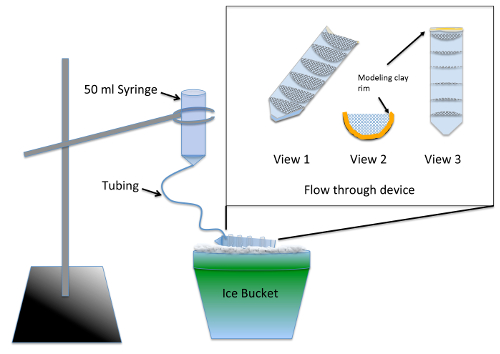
Figur 1 : Illustration av blekt apparater för de novo MT församlingen assay. Infällt är en närbild av genomströmmande enheten gjort från mesh limmas i en 50 mL centrifugrör skuren på längden. Mesh compartmentalizes genomflöde enheten så att flera experimentella grupper kan bearbetas samtidigt. Under användning, embryo medium läggs till sprutan och rinner långsamt genom slangen att fylla genomflöde enheten, som ger en konstant Skölj alla experimentella grupper. Klicka här för att se en större version av denna siffra.

Figur 2: användning av immunolabeling till bild dynamiska MTs. Dechorionated embryon fastställdes vid lämpliga skeden (4-5 i A-D och 12-13 thoraxsegmenten i E-H), tvären sektioneras genom hindbrain och immunolabeled med antikroppar mot β-tubulin (grön i A och E) för att markera alla MTs och tyrosinated α-tubulin (röd i B och F) för att avslöja dynamiska MT populationer. Högdynamiska MTs kan ses i de sammanslagna bilderna (C, G) och deras högre förstoringar (D, H) som områden där gul etikett är synlig (pilspetsar i D, H). Skala barer = 25 µm (A-C och E-G) och 10 µm (D och H). Klicka här för att se en större version av denna siffra.

FIGUR 3: användning av immunolabeling att bilden stabil MTs. Dechorionated embryon har åtgärdats sektioneras genom hindbrain och immunolabeled vid lämpliga skeden (4-5 thoraxsegmenten i A -D och 12-13 thoraxsegmenten i E-H). Stabil MTs är märkta med antikroppar mot detyrosinated form av α-tubulin (Glu-tubulin) (röd i B och F) medan totala MTs var visualiserat med en allmän β-tubulin antikropp (grön i A och E). Röda och gula signaler i sammanslagna bilder (C, G) och deras högre förstoringar (D, H) representerar områden av hög MT stabilitet (pilspetsar i D, H). Skala barer = 25 µm (A-C och E-G) och 10 µm (D och H). Klicka här för att se en större version av denna siffra.

Figur 4: användning av immunolabeling till bild begynnande MTs. Dechorionated embryon var fast på 4-5 thoraxsegmenten och tvären sektioneras genom bakhjärnan. Avsnitten var immunolabeled med β-tubulin (D, E, och F) att markera växande MTs och γ-tubulin (A, Boch C) att markera den kärnbildning punkt/centrosome. En dorsal region av neuralrörsdefekter är förpackade i (A, D; B, E och C-F) och visat på högre förstoring (G, H, jag, respektive) att avslöja atomkärnor (DAPI, blå), centrioles (γ-tubulin, röda) och totalt MTs (β-tubulin, grön). Vit pilspetsar: colocalization av MTs och centrioles; gula pilspetsar: den andra centriol i en cell är synlig. Skala barer = 25 µm (A-F) och 10 µm (G-I). Klicka här för att se en större version av denna siffra.

Tabell 1: Standardinställningar för att filtrera objekt i 3D-bild analys programvara.

Tabell 2: representativa raw uppgifter som erhållits med hjälp av 3D-bildanalys programvara för att analysera stabil MTs. Varje kolumn representerar mätningar från ett enda avsnitt. Min: minsta mätning; Max: största mätning; SD: standardavvikelse; SE: standardfelet.

Tabell 3: exempel på datamängder som kan erhållas från 3-D bild analys programvara för att kvantifiera stabil MTs. Välj mått av genomsnittlig längd totalt (β-tubulin) och stabila (Glu-tubulin) MTs beräknas genom att ta medelvärdet av den genomsnittliga skeletala längden för relevanta etiketten från alla prover (se tabell 2) och förhållandet mellan stabil för att Summa () MTs GLU-tubulin streck per β-tubulin ränder) beräknas genom att ta antalet genomsnittliga β-tubulin dividerat med genomsnittliga Glu-tubulin räkningen.

Tabell 4: Exempel på datamängder som kan erhållas från 3D-bild analys programvara för att analysera de novo MT församling. Representativa resultat från de novo MT församling experimentera, jämföra datamängder som erhålls för tre återställningspunkter tid (1, 5 och 10 min) efter nocodazole blekt. För varje tidpunkt visas mätningar som erhållits för nukleära greve, centrioles (γ-tubulin puncta), antal totala MTs (β-tubulin streck), för utvalda regioner av den avbildade analyseras (tvärsnitt av utveckla neuralrörsdefekter).
Subscription Required. Please recommend JoVE to your librarian.
Discussion
För närvarande finns det många metoder för imaging MT dynamics i tidiga zebrafiskar utveckling, alltifrån live avbildning av märkta molekyler till immunolabeling av fast vävnad11,12,13,14. Även om MTs i en enda cell kan existera i en dynamisk eller stabil stater, är epithelialization en process där MTs successivt stabiliseras med tiden. Använda markörer för stabila och dynamiska MTs erbjuder ett sätt att visualisera detta fenomen. Metoden presenteras här utnyttjar kraften av 3D-bildprogram att kvantifiera övergången från dynamisk till stabil MT populationer i ett tvärsnitt av embryonala zebrafiskar vävnad. I protokoll 2används metoden att märka en distinkt befolkning på begynnande MTs och följ övertiden kärnbildning och tillväxt.
MTs är notoriskt svåra att bilden i deras infödda staten på grund av deras benägenhet att depolymerize. Således är en viktig del av denna metod snabb fixering av MTs hela hela embryot. Detta uppnås genom att starta fixering vid fysiologiska temperaturer och använda en buffert som både stabiliserar MTs och ökar permeabiliteten i embryot. Fixering tiden är också viktigt eftersom förkortade fixeringen misslyckas att arrestera MTs medan överdriven fixering kan maskera epitoper, stör antikropp bindande. Den föreslagna fixering tid 3-4 h arbetar med embryon som är i mitten av gastrulation upp till 24 h efter befruktningen. Embryon yngre i slutet av tidsskalan bör fastställas för närmare 3 h medan äldre embryon kan behöva hela 4 h. Även med korrekt fixering, kommer MTs depolymerize med tiden så snittning och immunolabeling måste ske inom en vecka efter fastställande.
När vävnaden är ordentligt fast, kan det uppstå problem med immunolabeling. Det vanligaste problemet som uppstått har varit dålig penetration genom centrum av vävnad, särskilt, om för många avsnitt inkuberas i samma brunnen. Öka koncentrationen av den primära antikropp och inkubation tid för primära och sekundära antikroppar, kombinerat med ökande tvätt för att förbättra permeabilisering av embryon kommer att förbättra de flesta immunolabeling problem. Om antikroppar märkning misslyckas på grund av fixering frågor eller immunolabeling problem, är det möjligt att fastställa orsaken genom att undersöka antikroppens märkning mönster. Dålig fixering kommer att resultera i intensiv märkning i membranet och diffusa märkning i cytoplasman, medan överdriven fixering kommer att resultera i svag märkning som behåller MT arkitekturen. Dålig penetration av antikroppen, dock visas som områden i mitten av vävnaden utan märkning.
Förmågan att analysera MT bilder på ett meningsfullt sätt är beroende av hög kvalitet imaging. För att fånga MT längd i 3-D, bör minimistorleken möjligt för målet och numeriska bländaröppningen Z-steg användas. Bilder som visas här fångades med en 63 X olja emersion målet med 1,4 numeriska bländaröppningen producerar följande: pixel = 240 nm, Z-steg = 0.1 µm, Z-stack storlek = 16.252 µm. Eftersom en enda MT bredd är 25 nm, ungefär 10 gånger nedan gränsen för upplösning för ett ljusmikroskop, detta metriska inte kan vara noggrant mätt med denna teknik. I stället kan bara MT längder lika med eller större än den minsta pixelstorleken uppnåeligt i alla tre dimensioner mätas. Linje eller ram i genomsnitt kan förbättra MT signal definitionen. MT analys bör reserveras för högkvalitativa sektioner. Medan vävnad med dålig fixering inte kan avbildas och analyseras, milda overfixation kan kompenseras genom att försiktigt öka laser intensiteten och vinna för att upptäcka svaga signalen samtidigt bibehålla en bra dynamiskt omfång. Stackars antikropp penetration, kan medan inte optimal, korrigeras genom att begränsa bild förvärvandet väl märkta regionerna, vilket resulterar i imaging ett tunnare avsnitt (5-10 µm). Hög bakgrund från märkning kan kompenseras genom att justera filterinställningarna. Om någon av dessa justeringar görs, är det dock nödvändigt att kontrollera att tröskeln filter acceptabelt på varje plan i Z-stacken.
3D-bild analys programvara tillåter försöksledaren att kvantifiera MT längd, area, vinkel, överflöd och andra mätvärden i 3D-rymden av fasta vävnadssnitt. Den metod som beskrivs här ger riktningar för att erhålla sådana uppgifter med hjälp av kommersiellt tillgängliga program. Dock kan filtrering modulerna anpassas till offentlig domän programvara förstärkt med relevanta plugins eller makron, att göra analys tillgänglig för alla. Raw-bilder måste vara thresholded att undvika inklusive bakgrund och icke-specifika signaler i kvantifieringen före analys. När analysen är klar och data överförs till ett fungerande kalkylblad, kan många slutsatser göras från datauppsättningen. En av de beräkningar som gjorts här var Glu-tubulin streck per β-tubulin ränder, eller förhållandet mellan stabil MTs totala MTS där 1 representerar att hela MT cytoskelettet var stabiliserad i en ROI. Om experimenter vill komplettera sina kvantitativa data, generera en polerad tagged image format (TIFF) är bild med skala barer enkel med 3D-bild analys programvara.
Denna analys kan den funktionella analysen av proteiner inblandade i MT församling, i vivo. Om immunolabeling utförs på alternerande seriell avsnitt, kunde detta protokoll användas för att studera dynamisk och stabil MTs i samma embryot. I framtiden, gör ändringar, till exempel ökad rengöringsmedel eller förändrad inbäddning vinklar att användningen av dessa metoder för äldre embryon och ett bredare utbud av anatomiska frågor.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Författarna har något att avslöja.
Acknowledgments
Mikroskopet confocal köptes med medel från den amerikanska National Science Foundation (NSF), grant #DBI-0722569. Forskningen har med stöd av den amerikanska nationella institut för hälsa och nationella institutet av allmänna medicinska vetenskaper (NIH/NIGMS) grant #GM085290 och U.S. avdelning av försvar (DOD) grant #W81XWH-16-1-0466 tilldelas R.M. Brewster. E. Vital stöddes av ett bidrag till UMBC från Howard Hughes Medical Institute genom före college och Grundutbildning Science Education Program, bevilja #52008090. S.P. Brown stöddes av en amerikanska Institutionen för utbildning GAANN gemenskap, en Meyerhoff Graduate Fellowship finansieras av NIH/NIGMS bidrag #GM055036 och en forskning Assistantship finansieras av US DOD grant #W81XWH-16-1-0466.
Materials
| Name | Company | Catalog Number | Comments |
| Agarose | Used to treat petridishes. Prepare 1% agarose by heating a solution of 1 gram agarose per 100 ml 1X embryo medium in a microwave until polymerized. |
||
| Kpipes | Sigma | P7643 | |
| NaCl | Sigma | S7653 | |
| Tris-HCl | Sigma | T3253-500G | |
| KCl | Sigma | P9333-500G | |
| CaCl2·2H2O | Sigma | C5080 | |
| NP-40 | American Bioanalyticals | AB01424 | |
| EGTA | Sigma | E3889-25G | |
| MgCl2 | Sigma | M2670-500G | |
| Bovine serum albumin (BSA) | Fisher | BP1605 | |
| Triton-x | American Bioanalyticals | AB02025 | |
| Anti-Fade mounting medium | Invitrogen | P10144 | |
| Mouse anti-β-tubulin | Developmental studies Hybridoma Bank | E7 | 1/200 |
| Rabbit anti-γ-tubulin | Genetex | GTX113286 | 1/500 |
| Rabbit anti-α-tubulin | Genetex | GTX108784 | 1/1000* |
| Rabbit anti-detyrosinated-tubulin | Millipore | AB3201 | 1/200-1/1000* Titrate antibody with first use of new lot. |
| Rabbit anti-tyrosinated-tubulin | Millipore | ABT171 | 1/500 |
| Mouse anti-centrin | Millipore | 04-1624 | 1/1000 |
| Goat 488 anti-rabbit | Thermofisher | A11008 | 1/500 |
| Goat 594 anti-rabbit | Thermofisher | A11012 | 1/500 |
| Goat 594 anti-mouse | Thermofisher | A11005 | 1/500 |
| Goat 488 anti-mouse | Thermofisher | A11001 | 1/500 |
| Vibratome | Vibratome | 1500 | |
| Forceps | World Precision Instruments | 555227F | |
| 100 mm petri dish | Cell treat | 229693 | |
| 35 mm petri dish | Cell treat | 229638 | |
| 50 ml falcon tube | Fisher | 14-432-22 | |
| Woven nylon mesh 70 um | Amazon.com | B0043D1SZG | |
| Micropipette | Gilson | F123602 | |
| Glass pipette | Fisher | NC-999363-9 | |
| Aquarium sealant | Amazon.com, by MarineLand | Silicone Sealer 1 oz (Tube) | |
| Ring stand | Fisher | 14-675BO | |
| Microbore PTFE Tubing, 0.022"ID | Cole-Parmer | WU-06417-21 | |
| Modeling clay | Amazon.com | Sargent Art 22-4000 | Any wax or oil based non-toxic modeling clay will suffice |
| Clamp | Fisher | 02-215-466 | |
| 60ml syringe | Fisher | 14-820-11 | |
| Embryo medium (E3) | 34.8 g NaCl 1.6 g KCl 5.8 g CaCl2·2H2O 9.78 g MgCl2·6H2O To prepare a 60X stock, dissolve the ingredients in H2O, to a final volume of 2 L. Adjust the pH to 7.2 with NaOH. Autoclave. To prepare 1X medium, dilute 16.5 mL of the 60X stock to 1 L. |
||
| Blocking Solution | 50 ml TBS-NP-40 2.5 ml normal goat serum 1 g BSA 625 µl Triton-X |
||
| TBS-NP-40 (pH 7.6) | 155 mM NaCl 10 mM Tris HCl 0.1% NP-40 |
||
| 2x MAB (pH6.4) | 160 mM KPIPES 10 mM EGTA 2 mM MgCl2 |
||
| Commercial 3-D Image processing Software | PerkinElmer | Volocity (V 6.2) | |
| Dry block heater | VWR | 12621-108 | Used as a hot plate to melt agarose in Protocol 1. |
| Dissecting Microscope | Leica | MZ12 | |
| Confocal Microscope | Leica | SP5 | |
| Flat embedding mold | emsdiasum.com | BEEM 70904-01 | |
| Public domain image processing software | NIH | ImageJ (V 1.5) | |
| * Success varies by lot number | |||
References
- Akhmanova, A., Steinmetz, M. O. Tracking the ends: a dynamic protein network controls the fate of microtubule tips. Nat Rev Mol Cell Biol. 9 (4), 309-322 (2008).
- Conde, C., Cáceres, A. Microtubule assembly, organization and dynamics in axons and dendrites. Nat Rev Neurosci. 10 (5), 319-332 (2009).
- Kaverina, I., Straube, A. Regulation of cell migration by dynamic microtubules. Semin Cell Dev Biol. 22 (9), 968-974 (2011).
- Kollman, J. M., Merdes, A., Mourey, L., Agard, D. A. Microtubule nucleation by γ-tubulin complexes. Nat Rev Mol Cell Biol. 12 (11), 709-721 (2011).
- Howard, J., Hyman, A. A. Growth, fluctuation and switching at microtubule plus ends. Nat Rev Mol Cell Biol. 10 (8), 569-574 (2009).
- Schulze, E., Kirschner, M. Dynamic and stable populations of microtubules in cells. J Cell Biol. 104 (2), 277-288 (1987).
- Gundersen, G. G., Kalnoski, M. H., Bulinski, J. C. Distinct populations of microtubules: Tyrosinated and nontyrosinated alpha tubulin are distributed differently in vivo. Cell. 38 (3), 779-789 (1984).
- Li, R., Gundersen, G. G. Beyond polymer polarity: how the cytoskeleton builds a polarized cell. Nat Rev Mol Cell Biol. 9 (11), 860-873 (2008).
- Asakawa, K., Kawakami, K. A transgenic zebrafish for monitoring in vivo microtubule structures. Dev Dyn Off Publ Am Assoc Anat. 239 (10), 2695-2699 (2010).
- Wühr, M., Tan, E. S., Parker, S. K., Detrich, H. W., Mitchison, T. J. A model for cleavage plane determination in early amphibian and fish embryos. Curr Biol CB. 20 (22), 2040-2045 (2010).
- Tran, L. D., Hino, H., et al. Dynamic microtubules at the vegetal cortex predict the embryonic axis in zebrafish. Development. 139 (19), 3644-3652 (2012).
- Butler, R., Wood, J. D., Landers, J. A., Cunliffe, V. T. Genetic and chemical modulation of spastin-dependent axon outgrowth in zebrafish embryos indicates a role for impaired microtubule dynamics in hereditary spastic paraplegia. Dis Model Mech. 3 (11-12), 743-751 (2010).
- Yoo, S. K., Lam, P. -Y., Eichelberg, M. R., Zasadil, L., Bement, W. M., Huttenlocher, A. The role of microtubules in neutrophil polarity and migration in live zebrafish. J Cell Sci. 125 (23), 5702-5710 (2012).
- Andersen, E. F., Halloran, M. C. Centrosome movements in vivo correlate with specific neurite formation downstream of LIM homeodomain transcription factor activity. Development. 139 (19), 3590-3599 (2012).
- Lee, S. -J. Dynamic regulation of the microtubule and actin cytoskeleton in zebrafish epiboly. Biochem Biophys Res Commun. 452 (1), 1-7 (2014).
- Bulinski, J. C., Gundersen, G. G. Stabilization and post-translational modification of microtubules during cellular morphogenesis. BioEssays. 13 (6), 285-293 (1991).
- Magiera, M. M., Janke, C. Chapter 16 - Investigating Tubulin Posttranslational Modifications with Specific Antibodies. Methods Cell Biol. 115, 247-267 (2013).
- Hong, E., Jayachandran, P., Brewster, R. The polarity protein Pard3 is required for centrosome positioning during neurulation. Dev Biol. 341 (2), 335-345 (2010).
- Westermann, S., Weber, K. Post-translational modifications regulate microtubule function. Nat Rev Mol Cell Biol. 4 (12), 938-948 (2003).
- Jayachandran, P., Olmo, V. N., et al. Microtubule-associated protein 1b is required for shaping the neural tube. Neural Develop. 11, 1 (2016).
- Nam, S. -C. Role of Tau, a microtubule associated protein, in Drosophila photoreceptor morphogenesis. Genes N Y N 2000. 54 (11), 553-561 (2016).
- Abal, M., Piel, M., Bouckson-Castaing, V., Mogensen, M., Sibarita, J. -B., Bornens, M. Microtubule release from the centrosome in migrating cells. J Cell Biol. 159 (5), 731-737 (2002).
- Delgehyr, N., Sillibourne, J., Bornens, M. Microtubule nucleation and anchoring at the centrosome are independent processes linked by ninein function. J Cell Sci. 118 (8), 1565-1575 (2005).
- Manning, J. A., Lewis, M., Koblar, S. A., Kumar, S. An essential function for the centrosomal protein NEDD1 in zebrafish development. Cell Death Differ. 17 (8), 1302-1314 (2010).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn Off Publ Am Assoc Anat. 203 (3), 253-310 (1995).
- Beck, A. P., Watt, R. M., Bonner, J. Dissection and Lateral Mounting of Zebrafish Embryos: Analysis of Spinal Cord Development. JoVE J Vis Exp. (84), e50703 (2014).
- FÖldes-Papp, Z., Demel, U., Tilz, G. P. Laser scanning confocal fluorescence microscopy: an overview. Int Immunopharmacol. 3 (13-14), 1715-1729 (2003).
- Ferreira, T., Rasband, W. S. ImageJ User Guide - IJ 1.46. , Available from: https://imagej.nih.gov/ij/docs/guide/ (2010).
- Z-functions - ImageJ. , Available from: https://imagej.net/Z-functions (2017).



