Summary
Immunolabeling methoden voor het analyseren van verschillende populaties van microtubuli in de ontwikkelende hersenen van de zebravis zijn hier beschreven, die over het algemeen van toepassing zijn op andere weefsels. Het eerste protocol schetst een geoptimaliseerde methode voor immunolabeling stabiele en dynamische microtubuli. Het tweede protocol biedt een methode om beeld en kwantificeren van ontluikende microtubuli specifiek.
Abstract
Microtubuli (MTs) zijn dynamisch en broos structuren die uitdagend om te afbeelding in vivo, met name in gewervelde embryo's zijn. Immunolabeling methoden worden hier beschreven om te analyseren verschillende populaties van MTs in de ontwikkelingslanden neurale buis van de zebravis embryo. Terwijl de nadruk op zenuwweefsel ligt, geldt deze methode in grote lijnen voor andere weefsels. De procedures zijn geoptimaliseerd voor vroeg tot midden-somitogenesis-fase embryo's (1 Somiet tot 12 somieten), maar ze aangepast aan een scala aan andere etappes met relatief kleine aanpassingen worden kunnen. Het eerste protocol biedt een methode voor het beoordelen van de ruimtelijke spreiding van stabiele en dynamische MTs en een kwantitatieve analyse van deze populaties met beeld-verwerkende software uit te voeren. Deze aanpak vormt een aanvulling op bestaande hulpprogramma's voor afbeelding microtubulus dynamiek en distributie in real-time, met behulp van transgene lijnen of voorbijgaande uitdrukking van tagged constructies. Inderdaad, dergelijke hulpmiddelen zijn zeer nuttig, maar ze maken geen gemakkelijk onderscheid tussen dynamische en stabiele MTs. De mogelijkheid om beeld en analyseren van de populaties van deze verschillende microtubulus heeft belangrijke implicaties voor het begrijpen van mechanismen ten grondslag liggen aan de cel polarisatie en voedselproductie. Het tweede protocol schetst een techniek voor het analyseren van ontluikende MTs specifiek. Dit wordt bereikt door het vastleggen van de DOVO groei eigenschappen van MTs na verloop van tijd na microtubulus depolymerization met de nocodazole van de drug en een herstelperiode na drug wassen. Deze techniek heeft nog niet vereffend op de studie van MTs in zebrafish embryo's, maar is een waardevolle assay voor het onderzoeken van de functie in vivo van eiwitten die betrokken zijn bij de vergadering van de microtubuli.
Introduction
Microtubuli (MTs) zijn polymeren van α - en β-tubuline dat tot lineaire protofilaments samenvoegen, waarvan er verscheidene combineren om te vormen van een holle buis1,2. MTs zijn gepolariseerde structuren, met snelgroeiende plus uiteinden en langzaam groeiende minus uiteinden die zijn verankerd in de centrosoom of andere microtubulus-organiseren center (MTOC)3. DOVO MT vorming wordt geïnitieerd door nucleatie op de complexe γ-tubuline-ring (γ-TURC), waarmee een sjabloon voor MT vergadering4. In een bepaalde cel kunnen twee populaties van MTs worden onderscheiden die draaien tegen verschillende tarieven. Dynamische MTs verkennen hun cellulaire omgeving door te schakelen tussen de fasen van groei en krimp in een proces dat bekend staat als dynamische instabiliteit5. In tegenstelling tot dynamische MTs, stabiele MTs zijn niet-groeiende en hebben een langere halfwaardetijd dan dynamische MTs6.
Decennia van onderzoek in de celbiologie heeft geboden een verfijnde allerlei tools om te studeren MT structuur en functie en resulteerde in een grote hoeveelheid kennis over deze cytoskeletal elementen. Bijvoorbeeld, MTs spelen een centrale rol bij de totstandbrenging en het onderhoud van de polariteit van de cel, die toe te schrijven niet alleen aan hun intrinsieke polariteit, maar ook voor de differentiële subcellular verdeling van stabiel versus dynamische MTs7, is 8. daarentegen veel minder is begrepen over MT architectuur en functie in meer complexe driedimensionale (3-D) omgevingen, zoals het gewervelde embryo, gedeeltelijk als gevolg van de uitdaging van het cytoskelet van de MT op hoge resolutie beeldvormende. Niettegenstaande deze beperking, de recente generatie GFP-uiting te geven aan transgene lijnen dat label MTs of voorbijgaande expressie van fluorescently-tagged MT markers toegenomen ons begrip van de dynamische veranderingen die MTs ondergaan en hun cellulaire en Developmental rol in de zebravis embryo. Het hele MT-netwerk kan worden beeld in transgene lijnen in welke tubuline is ofwel rechtstreeks gelabelde9 of tubuline polymeren niet indirect worden aangeduid met behulp van MT-geassocieerde eiwitten Doublecortin-like-kinase (Dclk) of Ensconsin (EMTB)10, 11. Andere lijnen (en constructies) zijn gegenereerd die beoordeling van MT intrinsieke polariteit inschakelen door specifiek labeling MT plus eindigt of centrosoom-verankerd minus uiteinden11,12,13, 14. de kracht van deze tools ligt in de mogelijkheid te bestuderen van MT dynamiek in leven, ontwikkeling van organismen. Dergelijke studies is gebleken dat, bijvoorbeeld, de ruimtelijke en dynamische verdeling van MTs in specifieke cel populaties, de oriëntatie van de mitotische spindles in weefsels morfogenese (een indicator van het vliegtuig van celdeling), ondergaan de polariteit van het MT-polymeer met betrekking tot processen zoals cel rek en migratie, en MT groeitempo op jaarbasis bepaald door komeet snelheid9,13,15. De beperking van deze tools is dat ze niet gemakkelijk tussen stabiele en dynamische MT populaties discrimineren.
Tekening uit de rijke cel biologie literatuur, worden immunolabeling methoden om het imago van stabiele en dynamische MTs in de zebravis embryo hier beschreven, die zijn een aanvulling op het gebruik van transgene lijnen. Het wijdverbreide gebruik van dergelijke methodes van de immunolabeling in de zebravis enigszins belemmerd door de moeilijkheid in het MT integriteit behouden tijdens de procedure fixatie. Protocol 1 schetst een geoptimaliseerde methode voor immunolabeling totaal, dynamische, en stabiele MTs in grensoverschrijdende secties van de ontwikkelingslanden zebrafish hindbrain. Bovendien, een eenvoudige methode met behulp van commercieel beschikbare software wordt beschreven om het kwantificeren van de populaties van deze MT. Stabiele MTs onderscheiden zich van dynamische MTs gebaseerd op verschillende posttranslationele modificaties van α-tubuline, zoals acetylation en detyrosination, die op stabiele MTs in de tijd16,17 accumuleren. In het embryo van de zebravis, vindt acetylation plaats op Ciliaire en axonale MTs maar niet op stabiele interfase MTs18, beperking van het nut van deze markering tot een subset van gestabiliseerde MTs. Daarentegen lijkt detyrosination optreden op alle stabiele MTs in de zebravis embryo18. Deze posttranslationele wijziging blootstelt de glutaminezuur carboxy-terminal van α-tubuline (detyrosinated tubuline)18 en kan worden opgespoord met behulp van anti-Glu-tubuline19. Hoewel detyrosination bij voorkeur op stabiele MTs plaatsvindt, experimenteel bewijs geeft aan dat deze posttranslationele wijziging een gevolg van, in plaats van een doodsoorzaak, MT stabiliteit16. De wederzijdse MT bevolking, samengesteld uit dynamische MTs, onderscheidt zich met behulp van een antilichaam, anti-Tyr-tubuline, die specifiek de tyrosinated vorm van α-tubuline19herkent. Na immunolabeling met deze markeringen en confocal beeldvorming, de kwantitatieve analyse van MTs (lengte, aantal en relatieve overvloed) kan worden uitgevoerd in bepaalde regio's van de ontwikkeling van de neurale buis. Een gestroomlijnde methode is hier geboden voor het uitvoeren van deze analyse met behulp van 3D-beeldverwerking software. Deze methode kan worden toegepast om vragen te stellen met betrekking tot voedselproductie en de inrichting of de rijping van cel polariteit20. Inderdaad, de uitwerking van gepolariseerde arrays van stabiele MTs begeleidt veel developmental evenementen, waaronder fotoreceptor morfogenese21, epithelialization van cellen in de ontwikkelingslanden neurale buis18 en axon vorming8.
Protocol 2 beschrijft een in vivo -bewerking van een cel biologie assay voor het analyseren van MTs tijdens hun vergadering fase (nucleatie/anchorage en groei)22,23. Ontluikende MTs zijn nucleated op de centrosoom en vervolgens verankerd aan de subdistal aanhangsels van de moeder centriool23. Een methode voor het analyseren van de ontluikende MT hergroei na depolymerization wordt beschreven. Dit protocol bevat details over de behandeling van de nocodazole aan de depolymerize van MTs, de drug washout-procedure en de herstelperiode na de behandeling. MT uitgroei is gecontroleerd op regelmatige intervals post wassen door immunolabeling met markeringen voor totale MTs (anti β-tubuline) naast markers voor het centrosoom (anti γ-tubuline) en kern (4', 6-diamidino-2-phenylindole (DAPI)), volgens de algemene procedures beschreven in Protocol 1. De MT depolymerization stap van dit protocol is essentieel omdat hierdoor beoordeling van DOVO MT groei en uitbreiding van bestaande MTs niet. Deze techniek verschilt dus van andere gepubliceerde procedures voor het meten van MT groeicijfers (in afwezigheid van depolymerization) met behulp van een plus tip marker zoals einde bindende eiwit 3 gesmolten tot groen fluorescent proteïne (EB3-GFP), zoals in Tran et al., 201211. Bovendien, deze bepaling is met name handig voor het analyseren van de embryo's met gebreken in DOVO MT vergadering, zoals de eerder gemelde NEDD1 mutanten waarin rekrutering van γ-tubuline tot de centrosoom bijzondere waardevermindering heeft ondergaan is, wat resulteert in onvolledige de formatie van de neurale buis en neuronale gebreken24.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
ethische verklaring: de procedures beschreven hieronder volgen de Universiteit van Maryland Baltimore County dierenverzorgers richtsnoeren.
1. analyse van stabiele en dynamische MTs met behulp van Immunolabeling (Protocol 1)
- Manual dechorionation van embryo's voorafgaand aan de fixatie
- verkrijgen voortgebracht vers embryo's door gieten uit overtollige systeem water en vervolgens resterende embryo's te verzamelen in een plastic petrischaal (Zie Tabel of Materials).
- Verwijderen puin van het systeem water en overdracht van embryo's naar een nieuwe schotel gevuld met embryo medium (Zie Tabel van materialen) om ervoor te zorgen dat de embryo's in een schone omgeving ontwikkelen.
- Laten de embryo's te ontwikkelen naar het gewenste werkgebied in een temperatuurgevoelig incubator op 28,5 ° C.
- Plaats embryo's jonger dan 24 h na bevruchting (hpf) in een glazen schaal vóór dechorionation.
Opmerking: Dechorionate embryo's voorafgaand aan de fixatie te maximaliseren snelle penetratie van de fixeer en behouden MT-integriteit. Gebruik medium van het embryo in plaats van systeem water zodat de extra Ca 2 + vereist tijdens dechorionation.
De chorions verwijderen - handmatig van embryo's in de petrischaal, met behulp van fijne pincet onder een Microscoop ontleden.
- Een klein gebied in de ronde, transparante chorion die het embryo met een paar pincet en zachtjes trekken pincet uit elkaar omsluit aan het maken van een breuk in het membraan van knijpen.
- Vergroten de opening door fijn nieuwsgierige op de gescheurde chorion met pincet. Wees voorzichtig niet te raken van het embryo met de verlostang als het kon scheuren.
- Fixatie van gefaseerde embryo's
- overdracht geënsceneerd, dechorionated embryo's 1,5 mL centrifuge buizen. Verwijder zo veel embryo medium mogelijk met behulp van een glazen pipet van Pasteur.
Opmerking: Fixatie en drug behandelingen op embryo's young (midden-somitogenesis), voorafgaand aan de vorming van de neurale centra bemiddelen pijn sensatie, waarvoor geen additonal procedure om pijn te verlichten tijdens euthanasie uitvoeren. Ontwikkelingsstadia zijn zoals gedefinieerd in Kimmel et al.., 1995 25. De 4-5 en 11-12 Somiet fasen werden gebruikt voor het verkrijgen van beelden voor figuren 2 en 3. - Bereiden 4% paraformaldehyde (PFA) /MT vergadering buffer (MAB) fixatief (Zie Tabel van materialen) door het combineren van 1 mL 8% PFA per 1 mL 2 X MAB en het toevoegen van 2 µL 100% Triton X-100 per totale volume 1 mL.
Let op: Draag handschoenen tijdens het verwerken van oplossingen die PFA en Triton X-100, die huid irriterende stoffen zijn bevatten. - Fix embryo's in 1 mL 4% PFA/MAB fixeerspray voor 5 min op 28,5 ° C. Aspirate de fixeer met een pipet, vervangen door 1 mL verse fixeer en incubeer gedurende 3 uur bij kamertemperatuur (RT) op een rocker.
Opmerking: Monsters moeten worden vastgesteld snel bij hun biologische temperatuur (28,5 ° C voor zebravis) om te voorkomen dat de temperatuur-afhankelijke MT depolymerization.
- overdracht geënsceneerd, dechorionated embryo's 1,5 mL centrifuge buizen. Verwijder zo veel embryo medium mogelijk met behulp van een glazen pipet van Pasteur.
- Gecombineerd fixeer en voeg 1 mL 1 x Tris-gebufferde zoutoplossing met NP40 (TBS-NP40) buffer. Zachtjes doorroeren op RT op een rocker drie keer voor elke 5 min. Opslaan van de embryo's bij 4 ° C in 1 mL vers 1 X TBS-NP40 voor niet meer dan 7 dagen.
Let op: Draag handschoenen bij het verwerken van oplossingen met NP-40, een huid irriterende. - Sectioning embryo's voor immunolabeling
- Heat RT 4% lage smeltpunt (LMP) agarose medium insluiten in een ongeopende verpakking totdat de oplossing wordt duidelijk met een warmhoudplaat ingesteld op 50 ° C geplaatst dichtbij een ontleden Microscoop . Houd de container gesloten tussen monsters en verwarmd tijdens het hele proces insluiten (stappen 1.4.4-1.4.6).
- Overdracht embryo's uit de 1,5 mL Centrifugeer buizen naar een petrischaal met behulp van een precisiepipet glas en vul deze met 1 X TBS-NP40.
- Verwijder de grote dooier cellen uit de somitogenesis fase embryo's (4-5 en 11-12 somieten) in de petrischaal met fijne pincet onder de vergroting van een ontleden Microscoop 26. Houd het embryo door de staart bud met een paar pincet en schil weg de cellen van de dooier met het andere paar oog op het behoud van het hindbrain weefsel. De de-yolked embryo's overbrengen in een gebied van de petrischaal vrij van vuil van de dooier.
Opmerking: Het insluiten van embryo's in de agarose gevulde mal individueel om te voorkomen dat vroegtijdige verharding van de LMP agarose. - Vulling een 12 x 5 mm x 3 mm goed van de vectorafbeeldingsbestanden schimmel met 200 µL gesmolten LMP agarose met behulp van een micropipet. Het uitvoeren van de stappen 1.4.5.-1.4.6. snel (binnen 20 s van de mal vullen) embryo insluiten voordat de LMP agarose aan RT afkoelt en stolt.
- Fijne pincet gebruiken om een de-yolked embryo door de tailbud van de petrischaal naar de agarose gevulde mal richting de taps toelopende einde onder een Microscoop ontleden.
- Gebruik fijne pincet om te oriënteren van het embryo in de mal, zodanig dat de vibratome in het gewenste vlak snijdt. Transversale secties maken door het oriënteren van het embryo, zodat het hindbrain weefsel loopt parallel aan de lengte van de schimmel met de dorsale oppervlak tegenover de rand en de gerichte het einde taps toelopende regio anterior oppervlak. Herhaal stappen 1.4.4-1.4.6 voor de resterende embryo.
- Toestaan het agarose insluiten om te stollen voor 5 min op RT.
- Genereren 40 µm secties van de langste as van het agarose ingesloten embryo's (stappen 1.4.1-1.4.7) met behulp van een vibratome met de vectorafbeeldingsbestanden schotel gevuld met 1 x TBS-NP40. Secties van belang overbrengen in een 24-well plaat in 500 µL 1 x TBS-NP40 met fijne pincet. Plaats van de secties van slechts één embryo per putje.
Nota: Verwijs om te verwijzen naar 18 voor meer informatie. Erop toezien dat secties gehydrateerd op alle tijden in ten minste 250 µL van buffer en rock bij lage snelheid (10-25 rpm) voor de resterende stappen blijven om te voorkomen dat de scheiding van het agarose insluiten. Detergentia presenteren het tijdelijk blokkeren en wassen oplossingen moeten verlagen de oppervlaktespanning van de vloeistof en onderdompeling van de secties. Controleer of secties in de putten tijdens en na alle manipulaties blijven. Wees voorzichtig om te voorkomen dat per ongeluk teruggooi secties tijdens wasbeurten.
- Verwijderen van de buffer en voeg 500 µL van het blokkeren van de oplossing. Rock gedurende ten minste 1 uur op RT.
Opmerking: Gebruik een blokkerende oplossing die bevat 5% sera uit de host-soort van elke secundair antilichaam worden gebruikt (Zie Tabel of Materials). - Incubate in 300 µL primaire antilichamen verdund in blokkeerbuffer voor 36-72 uur bij 4 ° C op een rocker. Wassen tweemaal in 600 µL 1 x TBS-NP40 op een rocker voor 30 minuten elk, op RT.
Opmerking: Double-label secties door broeden in primaire antilichamen tegen totale MTs (anti β-tubuline of anti-α-tubuline) en stabiele MTs (anti-Glu-tubuline) of dynamische MTs (anti-Tyr tubuline). Selecteer primaire antilichamen die naar voren zijn gebracht in andere host soorten als α-tubuline populaties dubbele etikettering voor totaal en post-translationally gewijzigd. Raadpleeg Tabel van materialen voor antilichaam verdunningen. - Incubate in 300 µL van fluorophore-geconjugeerde secundaire antilichamen verdund in blokkeerbuffer op een rocker voor 16-24 uur, bij 4 ° C in het donker. Wassen tweemaal in 600 µL 1 x TBS-NP40 op een rocker voor 30 minuten elk, op RT.
Opmerking: Wrap de multi goed schotel met secundair antilichaam in folie vanaf dit moment en na elke manipulatie om te voorkomen dat het blussen. Selecteer secundaire antilichamen die met de host-immunoglobuline van het primaire antilichaam reageren. Kies secundair antilichaam fluorophores moeten afzonderlijke, niet-overlappende emissie spectra. Raadpleeg Tabel van materialen voor antilichaam verdunningen. - Broeden de embryo's 500 µL van DAPI oplossing op een rocker gedurende 30 minuten, bij RT. Wash driemaal in TBS-NP40 schommelen op RT voor elke 5 min.
Opmerking: Nucleaire labeling biedt cellulaire context voor de kwantificering van de MT uitgevoerd in stap 1.12. - Breng een druppel van montage medium met anti-fade agent in het midden van een dia stofvrij. Gebruik fijne pincet secties overbrengen naar de montage middellange druppel. Plaats een stofvrije dekglaasje aan op de top van het monster. Dia's opslaan in een droge, donkere en koele plek, verpakt in folie, totdat imaging wordt uitgevoerd.
Opmerking: Cirkelen de secties aan de achterkant van de dia met behulp van een schone-tipped permanent marker vóór imaging zal helpen om te identificeren secties bij het gebruik van de Microscoop. - Confocal Imaging
- Mount secties op een omgekeerde laser confocal microscoop scannen door het aanbrengen van de dia naar het werkgebied met het dekglaasje aan geconfronteerd met de doelstelling. Bepalen van de juiste optica (doelstelling, laser en de montages van het kanaal, zoals krijgen en gecompenseerd) op de dia van een besturingselement en houd ze consistente tussen monsters 27. Te voorkomen dat de pixels om gegevensverlies te voorkomen oversaturating.
- Z-stacks confocal beelden met behulp van de montages van het kanaal voor de geselecteerde secundair antilichaam fluorophores vastleggen en opslaan van de afbeelding bestanden 27. Z-stacks voor elke sectie verwerven.
Opmerking: De gehanteerde parameters voor de beelden in de figuren 2 en 3 verkrijgen met behulp van de volgende overname-instellingen repliceren: modus = XYZ; objectieve vergroting = 63 X olie-immersie objectief; objectieve numerieke diafragma = 1.4; Z-stap = 0.1 µm; Z-diepte = 16.23 µm. Gebruik de volgende montages van het kanaal: DAPI excitatie met 20% UV-range laser, emissie filter bereik = 430-480 nm, fotomultiplicator (PMT) winst = 525 V, en bet offset =-1.72%; 448 nm fluorophore (Zie Tabel van materialen) excitatie met 20% 488 laser bij de nm, emissie filter bereik = 493-573 nm, bet winst = 689 V, en bet offset = -0,2%; 594 nm fluorophore excitatie met 32% 594 laser bij de nm, emissie filter bereik = 608-706 nm, bet winst = 768 V, en bet offset =-6.8%. - Ruwe databestanden opslaan met unieke, beschrijvende bestandsnamen en een kopie voor bewerking in de software van de analyse van de afbeelding maken.
- Compilatie van Z-stacks voor het weergeven van maximale prognoses
- openen van het kopiëren van bestanden van gegevens met behulp van publieke domein 3D-afbeelding analysesoftware (bijvoorbeeld ImageJ). Controleer elk kanaal wordt weergegeven als een reeks afzonderlijke afbeeldingen (Z-stacks).
- Afbeelding kanalen splitsen met behulp van de volgende menu-reeks: “ beelden/kleur/Split kanalen ”.
- Maakt een samengevoegde afbeelding door te bedekken de kanalen van belang met behulp van de volgende menu-reeks: " beelden/kleur/Merge kanalen. " Selecteer de 594 nm, 488 nm en DAPI kanalen als valse rood, groen en blauw, respectievelijk. Controleer " maken composiet " en selecteer " OK " 28.
Opmerking: De DAPI kanaal te brengen beter detail specifiek voor MTs in een maximale projectie zoals in Figuur 2 en 3, door alleen valse kleuren voor de andere twee kanalen selecteren weglaten. - Onderzoekt de samengevoegde Z-stack en noteer de begin- en eindposities van de innerlijke beste Z-vliegtuigen voor alle zichtbare kanalen. Ontslaan de buitenste Z-vliegtuigen, die meestal suboptimaal signaal als gevolg van de ongelijke oppervlakken van de sectie hebben. Als u verwijst naar de 29 voor meer informatie verwijzen.
- Visualiseren de samengevoegde Z-stack als een enkele 2-D-afbeelding door het uitvoeren van een projectie van maximale intensiteit van de Z-stack met behulp van de volgende reeks van 3D-afbeeldingen analyse menu: " beelden/Stack/Z-project. " invoeren van de begin- en eindpositie van de innerlijke beste Z-vliegtuigen vanaf stap 1.11.3 als de " Start segment " en " Stop segment, " respectievelijk. Selecteer " Max intensiteit " als de projectie type en klik op " OK ". Als u verwijst naar 28 voor meer details verwijzen.
- Analyseren MT labeling
- de commerciële 3D-afbeelding analysesoftware niet openen. Selecteer " bibliotheek maken " en bieden een beschrijvende naam voor de beeldbank. Klik op " maken. " sleept u raw-afbeeldingsbestanden gegenereerd op basis van de confocal microscoop in de bibliotheek. Grotere bestanden meer tijd nodig om te overdracht.
- Selecteer een bestand om te analyseren. Kies " uitgebreid Focus " van de " weergave " menu om het beeld kanaal-samengevoegd in het hoofdvenster. De drempelmethode aanpassen
- door te slepen van de tool van de schuifregelaar voor elk kanaal naar links of naar rechts totdat de achtergrond signaal wordt verminderd en het ware signaal robuust is. Observeren dat elk kanaal geeft een ware signaal voor het molecuul aangeduid (b.v., DAPI kanaal tonen langwerpige of mitotische kernen maar niet auto-fluorescentie van het cytoplasma of agarose).
- Selecteer de " Freehand regio van belang (ROI) " gereedschap en een overzicht van de regio van belang om te worden geanalyseerd. Selecteer de " acties " tabblad gevolgd door " gewas aan selectie " bijsnijden van de afbeelding. Sla het bestand van de bijgesneden afbeelding onder een nieuwe naam. Klik op de " metingen " tabblad maken het protocol voor het filteren van specifieke objecten die relevant zijn voor de 3-D analyse.
- Drag " objecten vinden " naar het protocol-venster. Naam van het eerste protocol " DAPI. " Selecteer de DAPI-kanaal in het dropdownmenu. De volgende instellingen bij het protocol van de DAPI Sleep en plaats ze hieronder " objecten vinden " in de volgende volgorde (tabel 1): " vullen gaten in de ObjectsŔ " Aparte ObjectsŔ aan te raken " Uitsluiten objecten door SizeŔ " Uitsluiten niet aanraken van ROIs ".
Opmerking: Het doel van de instellingen in tabel 1 moeten eerst een drempel vaststellen die signalen om de teruggooi waarvan de distributie en grootte in strijd zijn met de grootte van objecten worden geanalyseerd. Bijvoorbeeld, een signaal niet groot genoeg om een kern te wanneer tellen kernen elimineren. - Uitvoeren de volgorde (stap 1.12.9) te monteren de resterende filters voor β-tubuline en andere markeringen met de instellingen in tabel 1.
- Selecteer " maatregel " aan de onderkant van elk protocol. Kies " intensiteit en volumemeting " en " skelet lengte " voor alle tubuline labeling, maar alleen de voormalige voor het signaal van de DAPI.
- Tekent een ROI rond de regio te meten. Observeren van de metingen onder de " samenvatting " tab nadat de software de regio verwerkt. De gegevens kopiëren en deze opslaan op een werkbare spreadsheet, zoals in tabel 2. Maak een back-up van het werkblad voor een latere anayse.
- Selecteer metingen van belang (bijvoorbeeld lengte van MT bundel, aantal MT bundels/kern, zoals geopenbaard met verschillende markeringen) in het werkblad en analyseren om te bepalen van de gemiddelden voor elke groep.
Opmerking: De gemiddelde MT bundel lengte = de som van het ' skelet lengte voor β-tubuline betekenen ' voor elk embryo gedeeld door het totale aantal embryo's. Verwijzen naar rij 20 van tabel 2. Het werkblad zodat de variabelen en experimentele groepen zijn gemakkelijk opgenomen in een grafiek opmaken.
2. DOVO MT vergadering Assay (Protocol 2)
- Construct en test de multi goed doorstroom toestellen ( Figuur 1) 2 dagen voorafgaand aan het experiment.
Opmerking: Het apparaat kunt gelijktijdige wegspoeling van meerdere experimentele groepen na de nocodazole behandeling met behulp van leveranties uit Tabel van materialen. Siliconen sealer vereist ten minste 24 uur van droogtijd voordat het vormt geen risico van toxiciteit voor het embryo's.- Een centrifugebuis 50 mL in tweeën te splitsen in de lengte, met behulp van een mal of lintzaag.
- Cut 7 halve cirkels, met een straal van 3 cm, uit 70 µm nylon gaas en trim laten passen strak in één helft van de splitsing Centrifugeer buis. Lijm de halve cirkels in de centrifugebuis parallel aan de 10 mL gradatie markeringen met behulp van aquarium-safe silicon sealer. Het apparaat te drogen voor 2 dagen en spoel door inweken in een bekerglas water voor 2-3 h.
- Lijn van de bovenste (schroefdraad) uiteinde van de centrifugebuis gesneden met het modelleren van klei, zodanig dat de hoogte van de vloeistof bewaard in de doorstroom apparaat een diepte van ¼ inch ( Figuur 1, 2 en 3 heeft).
- Bereid het wassen-apparaat door het verwijderen van de zuiger van een 50 mL injectiespuit en invoegen van 12 inch van fijne buizen in de tip. Duw de buis in zover het zal gaan en afdichting rond het gewricht met behulp van modellering klei.
- Natte vooraf de Maas embryo medium gebruik zodat de vloeistof om het gehele doorstroom apparaat te doorlopen. Hoek het apparaat op het ijs, zodat die vloeistof in alle compartimenten maar toch zwembaden leegt uit de voorkant waar de rand van de klei zich bevindt. Met behulp van een ring-tribune, schorten het wassen apparaat boven het apparaat doorstroming op ijs ( Figuur 1).
- Chill 200 mL van embryo medium op ijs en giet genoeg op het wassen apparaat om ervoor te zorgen dat alle luchtbellen zijn uitgeschakeld en dat het debiet ongeveer 7 mL/min. het debiet aanpassen is door het veranderen van de hoogte van de injectiespuit.
- Enzymatisch dechorionate embryo's
- Maak een werkoplossing van aspecifieke protease door verder verdunnen 1 mL 10 mg/mL aspecifieke protease voorraad in 20 mL embryo middellange.
- De chemische dechorionation uitvoeren op embryo's 1 uur vóór het timepoint wanneer ze worden verwacht om de gewenste ontwikkelings fase te bereiken. Digest chorions door het verwijderen van embryo medium van 100 mm petrischalen met geënsceneerd embryo's en het toevoegen van 20 mL van de werkoplossing aspecifieke protease.
- Incubate embryo's bij 37 ° C gedurende 5 minuten
Opmerking: Niet meer bedragen dan 5 min of gebruik een hogere concentratie van aspecifieke protease oplossing, aangezien dit zal resulteren in de embryo's vallen uit elkaar eenmaal behandeld met nocodazole. - Snel Pipetteer uit aspecifieke protease en bijvullen van de gerechten met ongeveer 25 mL embryo medium. Eenmaal herhaald.
- Met behulp van een 1-mL glazen pipet, overdracht embryo's jonger dan 24 hpf aan glas gerechten om hen te beschermen tegen schade.
- Complete dechorionation door het handmatig verwijderen van chorions met behulp van een paar fijne pincet, zoals beschreven in stap 1.1.5.
- Plaatsen van glas petrischalen met embryo's die dechorionated in een incubator 28,5 ° C gedurende ten minste 30 minuten tot ze de gewenste ontwikkelingsstadium.
- Depolymerize de bestaande MTs
- bereiden een werkende oplossing van 5 µg/mL nocodazole door het combineren van 50 µL 1 mg/mL voorraad nocodazole met 10 mL ijs koud embryo medium.
Let op: Gebruik handschoenen bij het verwerken van nocodazole, een huid irriterende. - Wisselen het embryo medium van de nocodazole behandelde groep met 10 mL koude nocodazole werken. Plaats petrischalen op ijs voor een geschikte tijd voor de ontwikkelings fase (bijvoorbeeld, 1 h voor 4-5 Somiet embryo's). Onbehandeld controlegewas embryo's in een petrischaal gereserveerd op ijs vast te stellen naast wassen monsters in stap 2.3.4.1.
- Overdracht embryo's met behulp van een precisiepipet spiegelglas van 1 mL brand het doorstroom apparaat, met aparte vakken voor elke experimentele groep. Start de nocodazole wassen door gieten ijs koud embryo medium in de top van de spuit 50 mL.
Opmerking: Gebruik ten minste 30 embryo's per experimentele groep. Experimentele groepen kon bestaan uit controle embryo's of een verscheidenheid van morfolino of RNA-geïnjecteerd embryo's. Het wassen vereist een totaal van ongeveer 150 mL van embryo medium moet worden toegevoegd elke 8-10 min. wassen de nocodazole terwijl het voortdurend voor de remming van de groei van de MT met ijs. Houden van de embryo's op ijs is van essentieel belang voor het welslagen van deze test omdat MTs unstable bij koude temperaturen en koude de ontwikkeling in vroege embryo's vertraagt. - Toestaan MTs te regrow na 20 min van wassen op RT door overdracht van embryo's naar glas petrischalen met warm (28,5 ° C) embryo medium gebruik een brand gepolijst 1 mL glazen pipet. Zodra de embryo's worden overgedragen, start een timer.
- Fix van de embryo's controle- en wassen op 1 min, 5 min en 10 min door ongeveer 10 embryo's pipetteren in een 1,5 mL Centrifugeer buis gevuld met 1 mL 4% PFA/MAB fix (28,5 ° C) en volg de aanwijzingen in stap 1.2.3.
- bereiden een werkende oplossing van 5 µg/mL nocodazole door het combineren van 50 µL 1 mg/mL voorraad nocodazole met 10 mL ijs koud embryo medium.
- Voorbereiding van monsters voor immunolabeling zoals beschreven in 1.3-1.5 afdelingen.
- Immunolabel zwevende secties en afbeelding embryo's als beschreven in secties 1.6-1.10 met de volgende wijzigingen aan de specificaties van het primaire antilichaam: gebruik 1:500 konijn anti-γ-tubuline en 1:200 muis anti-β-tubuline.
- Proces en analyseren van beelden met behulp van de software van de analyse van de 3D-afbeelding, zoals beschreven in stap 1.12.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Analyse van stabiele en dynamische MTs met behulp van immunolabeling
In Protocol 1, wordt de verdeling van de MT subpopulatie tijdens de vroege (neurale Kiel) en late (neurale rod) stadia van ontwikkeling van de neurale buis onthuld, met behulp van Glu-tubuline en Tyr-tubuline als markeringen voor stabiele en dynamische MTs, respectievelijk. Dynamische MTs overheersen in de hindbrain de neurale Kiel stadium (4-5 somieten) (Figuur 2A-D). Als de kiel zich tot de neurale staaf (11-12 somieten), een fase van verbeterde epithelialization ontwikkelt, zijn kwalitatief minder MTs immunoreactive met het anti-Tyr-tubuline antilichaam (Figuur 2E-H), met name in de ventrale staaf. In tegenstelling, Glu-tubuline is verspreid en punctate in de neurale Kiel (Figuur 3A-D), maar is verrijkt met de ventrale neurale staaf langs MT traktaten (Figuur 3E-H). Pijlpunten wijs specifieke MT bundels of structuren waar etikettering wordt verhoogd.
Hoewel zowel anti-Glu-tubuline en anti-Tyr-tubuline antilichamen werden geproduceerd in de dezelfde soort van de host (het voorkomen van een dubbele labeling experiment), deze resultaten wijzen erop dat stabiele en dynamische MT markeringen zelden in de zebravis hindbrain overlappen. Ten eerste, is de ventrale neurale staaf heeft meer stabiel (Figuur 3F) dan dynamische (Figuur 2F) MTs. De trend wordt omgekeerd in de dorsale neurale staaf, consistent met een model van zebravis neurulatie waarin de dorsale weefsel dynamische blijft totdat de neurale buis is gevormd20. In de tweede plaats terwijl mitotische spindels zijn volledig gelabeld met de Tyr-tubuline antilichaam in de neurale Kiel (alleenFiguur 2D, pijlpunten), alleen de basis van de spindel, samenvallen met de centrosoom, is gemarkeerd met de stabiliteit markering Glu-tubuline ( Figuur 3 D, pijlpunten). Β-tubuline immunofluorescentie, gemeen hebben beide tests, de experimentator van de verdeling van alle MTs informeert en vormt de basis om te sluiten niet-specifieke etikettering.
Meten van objecten met behulp van de software van de analyse van de 3D-afbeelding resulteert in een grote hoeveelheid gegevens die kunnen worden geordend in een handige tabel (tabel 2). Lengte, aantal en oppervlakte-metingen maken, gebruiken wij alleen een subset van de gegevens die beschikbaar is voor het analyseren. Een van de onderdelen van de gegevens die wij doen niet verder analyseren is het aantal objecten geïdentificeerd. Dit nummer wordt gebruikt als een interne kwaliteitscontrole, zoals het nummer niet variëren moet tussen zoals secties, en de verhouding tussen de kernen aan MTs qua een voorwaarde één behandeling blijven moet. Een uitschieter is een indicator dat de analyse moet worden met aangepaste filters opnieuw moet worden uitgevoerd of dat de afbeelding heet te slecht om te analyseren. Aldus, moeten alle beelden van de uitbijter worden reanalyzed met aangepaste instellingen. De uitschieter sectie moet worden onderzocht op tekenen van slechte labeling of fysieke schade die zou kunnen in ongewone object graven resulteren. Zodra de analyse is voltooid en kwaliteit gecontroleerd, nuttige informatie kan worden teruggevorderd van de ruwe gegevens zoals gemiddelde lengte van totale MTs en stabiele MTs of de verhouding van stabiele MTs aan totale MTs (tabel 3). Naast deze metingen, kan veel andere metriek worden verkregen met behulp van de 3D-afbeelding analysesoftware die kan worden gebruikt om de gevolgtrekkingen over MTs of hun relatie vestigen op andere cellulaire structuren (nucleus, centrosoom, enz.).
DOVO MT vergadering assay
De behandeling van nocodazole depolymerizes MTs wat resulteert in diffuse labeling (Figuur 4A, 4 D, en 4 G). Zoals de MTs regrow, ze uit te breiden van het centrosoom (Figuur 4B, 4E, en 4 H), echter kan dit niet worden duidelijk in een enkel vliegtuig als gevolg van hun niet-vlakke trajecten (Figuur 4C, 4F, en 4I). Toch zijn sommige afbeelding analysesoftware kan meten lengtes in 3D, waardoor een evaluatie van MT groei na het wassen van de nocodazole (tabel 4). Een belangrijke opmerking dat worden uit de dataset in tabel 4 verkregen kan is dat de gemiddelde lengte van MTs lijkt te verhogen na verloop van tijd na het wassen van de nocodazole in alle regio's van de neurale buis geanalyseerd. Zoals hierboven vermeld, kunnen andere soorten statistieken die zijn verkregen van de software van de analyse van de 3D-afbeelding cellulaire context om te interpreteren van de MT-gegevens (bijvoorbeeld ratio van MTs per nucleus).
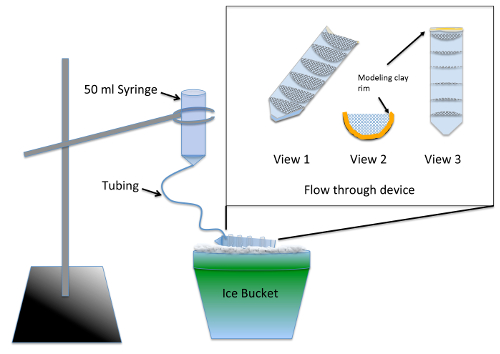
Figuur 1 : Illustratie van wassen toestellen voor DOVO MT vergadering assay. De inzet is een close-up van de doorstroom apparaat gemaakt van mesh gelijmd in een 50 mL centrifugebuis snijden in de lengte. De Maas compartmentalizes de doorstroom apparaat zodanig dat meerdere experimentele groepen tegelijkertijd kunnen worden verwerkt. Tijdens het gebruik, embryo medium wordt toegevoegd aan de spuit en langzaam stroomt door de buis te vullen de doorstroom apparaat, bieden een constante spoeling aan alle experimentele groepen. Klik hier voor een grotere versie van dit cijfer.

Figuur 2: gebruik van immunolabeling om het imago van dynamische MTs. Dechorionated embryo's werden vastgesteld op passende fasen (4-5 in A-D en 12-13 somieten in E-H), dwars door de hindbrain, en immunolabeled met antilichamen tegen β-tubuline (groen in A en E) zijn gesegmenteerd ter gelegenheid van alle MTs en tyrosinated α-tubuline (rood in B en F) te onthullen van dynamische MT populaties. Hoogdynamische MTs kunnen worden gezien in de samengevoegde afbeeldingen (C, G) en hun hogere vergrotingen (D, H) als gebieden waar gele label is zichtbaar (pijlpunten in D, H). Schaal bars = 25 µm (A-C en E-G) en 10 µm (D en H). Klik hier voor een grotere versie van dit cijfer.

Figure 3: gebruik van immunolabeling om het imago van stabiele MTs. Dechorionated embryo's werden vastgesteld, gesegmenteerd via de hindbrain, en immunolabeled in passende stadia (4-5 somieten in A -D en 12-13 somieten in E-H). Stabiele MTs worden aangeduid met antistoffen tegen de detyrosinated vorm van α-tubuline (Glu-tubuline) (rode in B en F) terwijl de totale MTs waren gevisualiseerd met een algemene β-tubuline antilichaam (groen in A en E). Rode en gele signalen in samengevoegde afbeeldingen (C, G) en hun hogere vergrotingen (D, H) vertegenwoordigen gebieden van hoge MT stabiliteit (pijlpunten in D, H). Schaal bars = 25 µm (A-C en E-G) en 10 µm (D en H). Klik hier voor een grotere versie van dit cijfer.

Figuur 4: gebruik van immunolabeling om het imago van de ontluikende MTs. Dechorionated embryo's waren vastgesteld op 4-5 somieten en dwars door de hindbrain worden gesegmenteerd. Secties waren immunolabeled met β-tubuline (D, E, en F) ter gelegenheid van de groeiende MTs en γ-tubuline (A, Ben C) ter gelegenheid van de nucleatie punt/centrosoom. Een dorsale regio van de neurale buis is boxed in (A, D; B, E en C-F) en op hogere vergroting (G, H, ik, respectievelijk) te onthullen van kernen (DAPI, blauw), centrioles (γ-tubuline, rood) en totaal MTs (β-tubuline, groen). Pijlpunten White: colocalization van MTs en centrioles; gele pijlpunten: de tweede centriool van een cel zichtbaar is. Schaal bars = 25 µm (A-F) en 10 µm (G-ik). Klik hier voor een grotere versie van dit cijfer.

Tabel 1: Standaardinstellingen voor het filteren van objecten in de software van de analyse van de 3D-afbeelding.

Tabel 2: vertegenwoordiger rauwe gegevensset verkregen met behulp van 3D-afbeelding analyse software voor het analyseren van stabiele MTs. Elke kolom vertegenwoordigt metingen van één afdeling. Min: kleinste meting; Max: grootste meting; SD: de standaarddeviatie van; SE: standaardfout.

Tabel 3: voorbeelden van datasets die kunnen worden verkregen van 3-D beeld analysesoftware om te kwantificeren stabiele MTs. Selecteer metingen van de gemiddelde lengte van totaal (β-tubuline) en stabiele (Glu-tubuline) MTs berekend door het nemen van het gemiddelde van de gemiddelde skelet lengte voor het desbetreffende label van alle monsters (Zie tabel 2) en de verhouding van stabiel als u wilt optellen (MTs Glu-tubulin strepen per β-tubuline strepen) berekend door het nemen van de gemiddelde β-tubuline telling gedeeld door het gemiddelde aantal Glu-tubuline.

Tabel 4: Voorbeelden van datasets die kan worden verkregen bij de software van de analyse van de 3D-afbeelding te analyseren DOVO MT vergadering. Representatieve resultaten van de vergadering van DOVO MT experimenteren, vergelijken datasets verkregen voor drie herstel tijd punten (1, 5 en 10 min) na nocodazole wassen. Voor elk punt van tijd, worden metingen verkregen voor nucleaire tellen, centrioles (γ-tubuline puncta), aantal totale MTs (β-tubuline strepen), weergegeven voor geselecteerde regio's van de verbeelde geanalyseerd (doorsnede van de ontwikkeling van de neurale buis).
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Er zijn momenteel vele methoden voor imaging-MT dynamiek in de vroege ontwikkeling van de zebravis, variërend van levende beeldvorming van tagged moleculen tot immunolabeling van vaste weefsel11,12,13,14. Hoewel MTs in één cel kunnen bestaan in dynamische of stabiele staten, is epithelialization een proces waarin de MTs is geleidelijk gestabiliseerd na verloop van tijd. Met behulp van merkers voor stabiele en dynamische MTs biedt een manier om dit verschijnsel te visualiseren. De methode die hier gepresenteerd borduurt voort op de kracht van 3D-imaging software te kwantificeren van de overgang van dynamische naar stabiele MT populaties in een dwarsdoorsnede van embryonale zebrafish weefsel. In Protocol 2, is de methode gebruikt om een aparte populatie van ontluikende MTs label en volg hun overwerk nucleatie en groei.
MTs zijn notoir moeilijk om het imago in hun geboortestaat vanwege hun neiging tot depolymerize. Het belangrijkste onderdeel van deze methode is dus snelle fixatie van MTs gedurende het gehele embryo. Dit wordt bereikt door de fixatie op fysiologische temperaturen te starten en met behulp van een buffer die zowel de MTs stabiliseert en de permeabiliteit van het embryo verhoogt. De tijd van de fixatie is ook belangrijk omdat beknot fixatie niet in tot arrestatie MTs slaagt terwijl overmatige fixatie epitopes, bemoeien met antilichaam binding kan maskeren. De tijd van de voorgestelde fixatie van 3-4 h werkt met embryo's die in Mid gastrulatie tot 24 uur na bemesting zijn. Embryo's jonger eind van de tijdschaal moeten worden vastgesteld voor dichter tot 3 h terwijl oudere embryo's de hele 4 uur wellicht. Zelfs met goede fixatie, zal de MTs depolymerize met tijd dus segmenteren en immunolabeling binnen één week na de vaststelling plaatsvinden moeten.
Zodra het weefsel naar behoren is vastgesteld, kunnen problemen optreden met immunolabeling. Het meest voorkomende probleem is al slechte penetratie door het midden van het weefsel, vooral als te veel secties worden geïncubeerd in de zelfde put. Verhoging van de concentratie van het primaire antilichaam en incubatie tijd voor primaire en secundaire antilichamen, gecombineerd met de verhoging van de detergentia ter verbetering van de permeabilization van de embryo's zal verzachten de meeste van de problemen van de immunolabeling. Als het antilichaam labeling mislukt als gevolg van fixatie vragen of immunolabeling problemen, het is mogelijk om de oorzaak door onderzoek van het antilichaam labeling patroon. Slechte fixatie zal resulteren in een intens labelen in het membraan en diffuse labelen in het cytoplasma, terwijl overmatige fixatie zal leiden tot zwakke labeling die behoudt MT het platform. Slechte penetratie van het antilichaam, wordt echter weergegeven als gebieden in het midden van het weefsel zonder de labeling.
De mogelijkheid om het analyseren van MT beelden op een zinvolle manier is afhankelijk van hoogwaardige beeldvorming. Wilt vastleggen MT lengte met 3D-opmaak, moet de minimumgrootte mogelijk voor de doelstelling en de numerieke aperture Z-stap worden gebruikt. Afbeeldingen hier getoond werden gevangen met een 63 X olie emersion doelstelling met 1.4 numerieke diafragma produceren de volgende: pixel = 240 nm, Z-stap = 0.1 µm, Z-stackgrootte = 16.252 µm. Omdat de breedte van een enkele MT 25 nm, ongeveer 10 keer onder de limiet van resolutie voor een lichte Microscoop, dit metrische kan niet nauwkeurig worden gemeten met behulp van deze techniek. In plaats daarvan kunnen enige MT lengtes gelijk is aan of groter is dan de minimale pixelgrootte haalbaar in alle drie dimensies worden gemeten. Lijn en/of frame gemiddeld kan de definitie van MT signaal verbeteren. MT analyse moet worden gereserveerd voor hoge kwaliteit secties. Terwijl weefsel met slechte fixatie niet kan worden beeld en geanalyseerd, milde overfixation kunnen worden gecompenseerd door het zorgvuldig het verhogen van de intensiteit van de laser en krijgen om op te sporen het zwakke signaal met behoud van een goede dynamisch bereik. Arme antilichaam penetratie, kan terwijl niet optimaal is, worden gecorrigeerd door Beeldacquisitie goed gelabelde regio's, resulterend in een dunnere sectie (5-10 µm) imaging te beperken. Hoge achtergrond van etikettering kan worden gecompenseerd door de filterinstellingen aan te passen. Als een van deze aanpassingen worden gedaan, het is echter noodzakelijk om te verifiëren dat de drempel van de filters aanvaardbaar op elk vliegtuig van de Z-stack.
De software van de analyse van de 3D-afbeelding staat de experimentator te kwantificeren MT lengte, gebied hoek, overvloed en andere statistieken in de 3D-ruimte van vaste weefselsecties. De hier beschreven methode geeft aanwijzingen voor het verkrijgen van dergelijke gegevens met behulp van commercieel beschikbare software. De filter modules kunnen echter worden aangepast aan publiek domein-software verbeterd met relevante plugins en/of macro's, analyse voor iedereen beschikbaar maken. Vóór de analyse dient de raw beelden thresholded te vermijden zoals achtergrond en niet-specifieke signalen in de kwantificering. Zodra de analyse is voltooid en de gegevens overbrengt naar een werkbaar werkblad, kunnen vele gevolgtrekkingen worden gemaakt van de gegevensset. Een van de hier gemaakte berekeningen was Glu-tubuline strepen per β-tubuline strepen, of de verhouding van stabiele MTs aan totale MTs waar 1 staat dat het hele MT cytoskelet werd gestabiliseerd in een ROI. Als de experimentator wenst aan te vullen van hun kwantitatieve gegevens, het genereren van een (TIFF) gepolijst tagged image file format is afbeelding met schaal bars moeiteloos met de software van de analyse van de 3D-afbeelding.
Deze bepaling laat de functionele analyse van eiwitten die betrokken zijn bij de assemblage van de MT, in vivo. Als immunolabeling op afwisselende seriële secties wordt uitgevoerd, kan dit protocol worden gebruikt om de studie van dynamische en stabiele MTs in het hetzelfde embryo. Wijzigingen zoals meer detergentia of gewijzigde insluiten hoeken kunnen in de toekomst het gebruik van deze methoden voor oudere embryo's en een breder scala van anatomische vragen.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
De auteurs hebben niets te onthullen.
Acknowledgments
De confocal microscoop werd gekocht met geld van de Amerikaanse National Science Foundation (NSF), subsidie #DBI-0722569. Het onderzoek werd gesteund door de Amerikaanse nationale instituten van gezondheid/nationale Institute of General Medical Sciences (NIH/NIGMS) subsidie #GM085290 en Amerikaanse departement van defensie (DOD) subsidie #W81XWH-16-1-0466 toegekend aan R.M. Brewster. E. Vital werd gesteund door een subsidie naar UMBC van het Howard Hughes Medical Institute via de pre college en Undergraduate Science Education Program, verlenen van #52008090. S.P. Brown werd ondersteund door een Amerikaanse departement van onderwijs GAANN Fellowship, een Meyerhoff Graduate Fellowship gefinancierd door subsidie van het NIH/NIGMS, #GM055036, en een onderzoek assistentschap gefinancierd door de V.S. DOD subsidie #W81XWH-16-1-0466.
Materials
| Name | Company | Catalog Number | Comments |
| Agarose | Used to treat petridishes. Prepare 1% agarose by heating a solution of 1 gram agarose per 100 ml 1X embryo medium in a microwave until polymerized. |
||
| Kpipes | Sigma | P7643 | |
| NaCl | Sigma | S7653 | |
| Tris-HCl | Sigma | T3253-500G | |
| KCl | Sigma | P9333-500G | |
| CaCl2·2H2O | Sigma | C5080 | |
| NP-40 | American Bioanalyticals | AB01424 | |
| EGTA | Sigma | E3889-25G | |
| MgCl2 | Sigma | M2670-500G | |
| Bovine serum albumin (BSA) | Fisher | BP1605 | |
| Triton-x | American Bioanalyticals | AB02025 | |
| Anti-Fade mounting medium | Invitrogen | P10144 | |
| Mouse anti-β-tubulin | Developmental studies Hybridoma Bank | E7 | 1/200 |
| Rabbit anti-γ-tubulin | Genetex | GTX113286 | 1/500 |
| Rabbit anti-α-tubulin | Genetex | GTX108784 | 1/1000* |
| Rabbit anti-detyrosinated-tubulin | Millipore | AB3201 | 1/200-1/1000* Titrate antibody with first use of new lot. |
| Rabbit anti-tyrosinated-tubulin | Millipore | ABT171 | 1/500 |
| Mouse anti-centrin | Millipore | 04-1624 | 1/1000 |
| Goat 488 anti-rabbit | Thermofisher | A11008 | 1/500 |
| Goat 594 anti-rabbit | Thermofisher | A11012 | 1/500 |
| Goat 594 anti-mouse | Thermofisher | A11005 | 1/500 |
| Goat 488 anti-mouse | Thermofisher | A11001 | 1/500 |
| Vibratome | Vibratome | 1500 | |
| Forceps | World Precision Instruments | 555227F | |
| 100 mm petri dish | Cell treat | 229693 | |
| 35 mm petri dish | Cell treat | 229638 | |
| 50 ml falcon tube | Fisher | 14-432-22 | |
| Woven nylon mesh 70 um | Amazon.com | B0043D1SZG | |
| Micropipette | Gilson | F123602 | |
| Glass pipette | Fisher | NC-999363-9 | |
| Aquarium sealant | Amazon.com, by MarineLand | Silicone Sealer 1 oz (Tube) | |
| Ring stand | Fisher | 14-675BO | |
| Microbore PTFE Tubing, 0.022"ID | Cole-Parmer | WU-06417-21 | |
| Modeling clay | Amazon.com | Sargent Art 22-4000 | Any wax or oil based non-toxic modeling clay will suffice |
| Clamp | Fisher | 02-215-466 | |
| 60ml syringe | Fisher | 14-820-11 | |
| Embryo medium (E3) | 34.8 g NaCl 1.6 g KCl 5.8 g CaCl2·2H2O 9.78 g MgCl2·6H2O To prepare a 60X stock, dissolve the ingredients in H2O, to a final volume of 2 L. Adjust the pH to 7.2 with NaOH. Autoclave. To prepare 1X medium, dilute 16.5 mL of the 60X stock to 1 L. |
||
| Blocking Solution | 50 ml TBS-NP-40 2.5 ml normal goat serum 1 g BSA 625 µl Triton-X |
||
| TBS-NP-40 (pH 7.6) | 155 mM NaCl 10 mM Tris HCl 0.1% NP-40 |
||
| 2x MAB (pH6.4) | 160 mM KPIPES 10 mM EGTA 2 mM MgCl2 |
||
| Commercial 3-D Image processing Software | PerkinElmer | Volocity (V 6.2) | |
| Dry block heater | VWR | 12621-108 | Used as a hot plate to melt agarose in Protocol 1. |
| Dissecting Microscope | Leica | MZ12 | |
| Confocal Microscope | Leica | SP5 | |
| Flat embedding mold | emsdiasum.com | BEEM 70904-01 | |
| Public domain image processing software | NIH | ImageJ (V 1.5) | |
| * Success varies by lot number | |||
References
- Akhmanova, A., Steinmetz, M. O. Tracking the ends: a dynamic protein network controls the fate of microtubule tips. Nat Rev Mol Cell Biol. 9 (4), 309-322 (2008).
- Conde, C., Cáceres, A. Microtubule assembly, organization and dynamics in axons and dendrites. Nat Rev Neurosci. 10 (5), 319-332 (2009).
- Kaverina, I., Straube, A. Regulation of cell migration by dynamic microtubules. Semin Cell Dev Biol. 22 (9), 968-974 (2011).
- Kollman, J. M., Merdes, A., Mourey, L., Agard, D. A. Microtubule nucleation by γ-tubulin complexes. Nat Rev Mol Cell Biol. 12 (11), 709-721 (2011).
- Howard, J., Hyman, A. A. Growth, fluctuation and switching at microtubule plus ends. Nat Rev Mol Cell Biol. 10 (8), 569-574 (2009).
- Schulze, E., Kirschner, M. Dynamic and stable populations of microtubules in cells. J Cell Biol. 104 (2), 277-288 (1987).
- Gundersen, G. G., Kalnoski, M. H., Bulinski, J. C. Distinct populations of microtubules: Tyrosinated and nontyrosinated alpha tubulin are distributed differently in vivo. Cell. 38 (3), 779-789 (1984).
- Li, R., Gundersen, G. G. Beyond polymer polarity: how the cytoskeleton builds a polarized cell. Nat Rev Mol Cell Biol. 9 (11), 860-873 (2008).
- Asakawa, K., Kawakami, K. A transgenic zebrafish for monitoring in vivo microtubule structures. Dev Dyn Off Publ Am Assoc Anat. 239 (10), 2695-2699 (2010).
- Wühr, M., Tan, E. S., Parker, S. K., Detrich, H. W., Mitchison, T. J. A model for cleavage plane determination in early amphibian and fish embryos. Curr Biol CB. 20 (22), 2040-2045 (2010).
- Tran, L. D., Hino, H., et al. Dynamic microtubules at the vegetal cortex predict the embryonic axis in zebrafish. Development. 139 (19), 3644-3652 (2012).
- Butler, R., Wood, J. D., Landers, J. A., Cunliffe, V. T. Genetic and chemical modulation of spastin-dependent axon outgrowth in zebrafish embryos indicates a role for impaired microtubule dynamics in hereditary spastic paraplegia. Dis Model Mech. 3 (11-12), 743-751 (2010).
- Yoo, S. K., Lam, P. -Y., Eichelberg, M. R., Zasadil, L., Bement, W. M., Huttenlocher, A. The role of microtubules in neutrophil polarity and migration in live zebrafish. J Cell Sci. 125 (23), 5702-5710 (2012).
- Andersen, E. F., Halloran, M. C. Centrosome movements in vivo correlate with specific neurite formation downstream of LIM homeodomain transcription factor activity. Development. 139 (19), 3590-3599 (2012).
- Lee, S. -J. Dynamic regulation of the microtubule and actin cytoskeleton in zebrafish epiboly. Biochem Biophys Res Commun. 452 (1), 1-7 (2014).
- Bulinski, J. C., Gundersen, G. G. Stabilization and post-translational modification of microtubules during cellular morphogenesis. BioEssays. 13 (6), 285-293 (1991).
- Magiera, M. M., Janke, C. Chapter 16 - Investigating Tubulin Posttranslational Modifications with Specific Antibodies. Methods Cell Biol. 115, 247-267 (2013).
- Hong, E., Jayachandran, P., Brewster, R. The polarity protein Pard3 is required for centrosome positioning during neurulation. Dev Biol. 341 (2), 335-345 (2010).
- Westermann, S., Weber, K. Post-translational modifications regulate microtubule function. Nat Rev Mol Cell Biol. 4 (12), 938-948 (2003).
- Jayachandran, P., Olmo, V. N., et al. Microtubule-associated protein 1b is required for shaping the neural tube. Neural Develop. 11, 1 (2016).
- Nam, S. -C. Role of Tau, a microtubule associated protein, in Drosophila photoreceptor morphogenesis. Genes N Y N 2000. 54 (11), 553-561 (2016).
- Abal, M., Piel, M., Bouckson-Castaing, V., Mogensen, M., Sibarita, J. -B., Bornens, M. Microtubule release from the centrosome in migrating cells. J Cell Biol. 159 (5), 731-737 (2002).
- Delgehyr, N., Sillibourne, J., Bornens, M. Microtubule nucleation and anchoring at the centrosome are independent processes linked by ninein function. J Cell Sci. 118 (8), 1565-1575 (2005).
- Manning, J. A., Lewis, M., Koblar, S. A., Kumar, S. An essential function for the centrosomal protein NEDD1 in zebrafish development. Cell Death Differ. 17 (8), 1302-1314 (2010).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn Off Publ Am Assoc Anat. 203 (3), 253-310 (1995).
- Beck, A. P., Watt, R. M., Bonner, J. Dissection and Lateral Mounting of Zebrafish Embryos: Analysis of Spinal Cord Development. JoVE J Vis Exp. (84), e50703 (2014).
- FÖldes-Papp, Z., Demel, U., Tilz, G. P. Laser scanning confocal fluorescence microscopy: an overview. Int Immunopharmacol. 3 (13-14), 1715-1729 (2003).
- Ferreira, T., Rasband, W. S. ImageJ User Guide - IJ 1.46. , Available from: https://imagej.nih.gov/ij/docs/guide/ (2010).
- Z-functions - ImageJ. , Available from: https://imagej.net/Z-functions (2017).



