ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
संकुल नियमित रूप से प्रतिरिक्ति लघु palindromic दोहराता/CRISPR जुड़े प्रोटीन 9 (CRISPR/Cas9) प्रणाली आनुवंशिक इंजीनियरिंग के लिए एक आशाजनक उपकरण प्रदान करता है, और transgenes के लक्षित एकीकरण की संभावना को खोलता है । हम एक समरूपता-मध्यस्थता अंत में शामिल होने का वर्णन (HMEJ)-आधारित रणनीति vivo में कुशल डीएनए लक्षित एकीकरण के लिए और लक्षित जीन CRISPR का उपयोग कर चिकित्सा/Cas9.
Abstract
एक होनहार जीनोम संपादन मंच के रूप में, CRISPR/Cas9 प्रणाली कुशल आनुवंशिक हेरफेर के लिए महान क्षमता है, विशेष रूप से transgenes के लक्षित एकीकरण के लिए । हालांकि, मुताबिक़ पुनर्संयोजन (मानव संसाधन) और गैर-मुताबिक़ अंत में शामिल होने के विभिंन indel उत्परिवर्तनों की कम दक्षता के कारण (NHEJ)-गैर-विभाजन कोशिकाओं में आधारित रणनीतियों, vivo जीनोम संपादन में एक बड़ी चुनौती बनी हुई है । यहां, हम एक समरूपता मध्यस्थता अंत में शामिल होने का वर्णन (HMEJ)-आधारित CRISPR/Cas9 प्रणाली vivo सटीक लक्षित एकीकरण में कुशल के लिए. इस प्रणाली में, लक्षित जीनोम और दाता वेक्टर समरूपता शस्त्र युक्त (~ 800 बीपी) एकल गाइड आरएनए (sgRNA) लक्ष्य अनुक्रम द्वारा पार्श्व CRISPR/Cas9 द्वारा सट रहे हैं । यह HMEJ आधारित रणनीति माउस zygotes में कुशल transgene एकीकरण, साथ ही vivo मेंहेपैटोसाइट्स में प्राप्त होता है । इसके अलावा, एक HMEJ आधारित रणनीति fumarylacetoacetate hydrolase के सुधार के लिए एक कुशल दृष्टिकोण प्रदान करता है (फह) हेपैटोसाइट्स में उत्परिवर्तन और बचाता है फह-कमी प्रेरित जिगर की विफलता चूहों । एक साथ ले लिया, लक्षित एकीकरण पर ध्यान केंद्रित, इस HMEJ आधारित रणनीति आनुवंशिक रूप से संशोधित पशु मॉडल और लक्षित जीन चिकित्सा की पीढ़ी सहित आवेदनों की एक किस्म के लिए एक आशाजनक उपकरण प्रदान करता है ।
Introduction
सटीक, लक्षित जीनोम संपादन अक्सर आनुवंशिक रूप से संशोधित पशु मॉडल और नैदानिक उपचार के उत्पादन के लिए आवश्यक है । बहुत प्रयास कुशल लक्षित जीनोम संपादन, जिंक फिंगर nuclease (ZFN), प्रतिलेखन उत्प्रेरक जैसे प्रभाव nucleases (TALENs), और CRISPR/Cas9 प्रणालियों के रूप में के लिए विभिंन रणनीतियों को विकसित करने के लिए किया गया है । इन रणनीतियों के जीनोम में लक्षित डीएनए डबल-किनारा टूटता है (DSB) बनाने के लिए, और आंतरिक डीएनए की मरंमत प्रणाली का लाभ ले, जैसे मुताबिक़ पुनर्संयोजन (मानव संसाधन)1,2, microhomology-मध्यस्थता अंत में शामिल होने (MMEJ)3 , 4 , 5, और गैर मुताबिक़ अंत में शामिल होने (NHEJ)6,7,8 के लक्षित एकीकरण प्रेरित करने के लिए transgenes1,9। मानव संसाधन आधारित रणनीति वर्तमान में सबसे अधिक इस्तेमाल किया जीनोम संपादन दृष्टिकोण है, जो सेल लाइनों में बहुत ही कुशल है, लेकिन देर S/G2 चरण में अपनी प्रतिबंधित घटना के कारण गैर-विभाजित कोशिकाओं के लिए आसानी से सुलभ नहीं है । इस प्रकार, मानव संसाधन आधारित रणनीति vivo जीनोम संपादन में के लिए लागू नहीं है । हाल ही में, NHEJ आधारित रणनीति कुशल जीन दस्तक के लिए माउस के ऊतकों में8में विकसित किया गया था । फिर भी, NHEJ आधारित विधि आमतौर पर जंक्शनों पर indels परिचय, यह मुश्किल सटीक जीनोम संपादन उत्पंन करने के लिए, खासकर जब में फ्रेम संलयन जीन8का निर्माण करने की कोशिश कर रहा । MMEJ आधारित लक्षित एकीकरण सटीक जीनोम संपादन करने में सक्षम है । हालांकि, यह केवल विनय पिछले रिपोर्ट5में लक्षित एकीकरण दक्षता बढ़ जाती है । इसलिए, vivo में सटीक लक्षित एकीकरण की दक्षता में सुधार तत्काल व्यापक चिकित्सीय अनुप्रयोगों के लिए3की जरूरत है ।
हाल ही में प्रकाशित एक काम में, हम एक समरूपता-मध्यस्थता अंत में शामिल होने का प्रदर्शन किया (HMEJ) आधारित रणनीति है, जो सभी विट्रो में और vivo मेंदोनों की रिपोर्ट की रणनीतियों में उच्चतम लक्षित एकीकरण दक्षता दिखाया10। यहां, हम HMEJ प्रणाली की स्थापना के लिए एक प्रोटोकॉल का वर्णन है, और भी एकल गाइड आरएनए (sgRNA) वैक्टर ब्याज की जीन लक्ष्यीकरण और दाता वैक्टर sgRNA लक्ष्य साइटों और समरूपता हथियारों के ~ 800 बीपी को लक्षित करने का निर्माण (चित्रा 1) . इस प्रोटोकॉल में, हम भी डीएनए में चूहे की पीढ़ी के लिए विस्तृत कदम का वर्णन-चूहों में और vivo मेंऊतकों में लक्षित एकीकरण के लिए संक्षिप्त कदम. इसके अलावा, HMEJ आधारित रणनीति के एक सबूत-अवधारणा के अध्ययन के फह उत्परिवर्तन और बचाव फहसही करने के लिए अपनी क्षमता का प्रदर्शन किया- -जिगर की विफलता चूहों, जो आगे अपनी चिकित्सीय क्षमता से पता चला.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
शंघाई संस्थानों में जैविक विज्ञान (कैस) के लिए जैव चिकित्सा अनुसंधान नैतिकता समिति द्वारा पशु विषयों सहित सभी प्रक्रियाओं को अनुमोदित किया गया है ।
1. दाता Plasmids की डिजाइन
-
sgRNA का चयन
- लक्ष्य क्षेत्र11,12,13,14,15पर sgRNAs का पूर्वानुमान लगाने के लिए ऑनलाइन CRISPR डिज़ाइन टूल का उपयोग करें । Cdx2 लोकस के लिए, डिजाइन छह अलग sgRNAs (Cdx2-sgRNA1 ~Cdx2-sgRNA6) के आसपास उच्च रैंक और कम क्षमता के साथ बंद लक्ष्य (चित्रा 1ए)16।
- Linearize 2 µ जी के Cas9-सीएमवी-EGFP अभिव्यक्ति वैक्टर और sgRNA द्वारा BbsI पाचन (1 µ एल के लिए 2 ज में 37 ° c के एक अंतिम एकाग्रता में 1 U/BbsI l की मात्रा में 20 µ l). तो 1 × ताे बफर में 1% agarose जेल के साथ जेल शुद्धि किट के द्वारा उत्पाद शुद्ध ।
- sgRNA oligonucleotides के 10 µ एल में एक जोड़ी के मिश्रण 1 × टी-4 डीएनए ligase बफर के अंतिम एकाग्रता करने के लिए 50 µ एम. के लिए 95 डिग्री सेल्सियस से 25 डिग्री सेल्सियस के तापमान में परिवर्तन की दर के साथ एक तापमान ढाल का उपयोग oligo समाधान की मशीन 5 ° c/5 मिनट (95 ° c 5 मिनट के लिए , तो 5 मिनट के लिए 90 ° c, 5 मिनट के लिए 85 ° c, आदि, जो ओलिगोस्पर्मिया ऐनी होगा ।
- annealed उत्पाद के 4 µ एल, 2 µ एल के 1 µ एल के साथ रैखिक वेक्टर के 10 ligase एल में 1 × टी-4 डीएनए µ बफर, और फिर ligase 22 डिग्री सेल्सियस के लिए 1-2 एच (चित्रा 1बी) के लिए मिश्रण ।
-
सर्वेयर sgRNA की nuclease परख
नोट: sgRNA के लिए इस्तेमाल किया दस्तक में प्रयोग की लक्ष्यीकरण दक्षता सर्वेयर nuclease परख द्वारा मूल्यांकन (भी T7 endonuclease मैं (T7EI) परख के रूप में जाना जाता है)17। उच्च डीएनए दरार दक्षता और sgRNA काटने साइट और स्टॉप codon के बीच एक कम दूरी के साथ sgRNA का चयन करें ।- Transfect Cas9-sgRNA-EGFP अभिव्यक्ति वैक्टर N2a सेल में DMEM किट द्वारा 10% भ्रूण गोजातीय सीरम, 1% पीएसजी, और 1% गैर आवश्यक एमिनो एसिड के साथ पूरक लाइनों में संस्कृति अभिकर्मक ( सामग्री की तालिकादेखें) । 5% सह में 37 डिग्री सेल्सियस पर transfected कोशिकाओं की मशीन2।
- मशीन के 48 घंटे के बाद, एक नियंत्रण के रूप में गैर transfected कोशिकाओं का उपयोग प्रतिदीप्ति-सक्रिय सेल छँटाई (FACS) द्वारा 5,000 transfected कोशिकाओं (GFP+) इकट्ठा).
- 2-5 µ एल oflysis बफर में एकत्र कोशिकाओं को पचाने (0.1% ट्राइटन एक्स-100, 0.1% के बीच 20, और 100 µ g/एमएल Proteinase k) 56 ° c पर 30 मिनट के लिए, और फिर निष्क्रिय Proteinase k 95 ° c पर 10 मिनट के लिए ।
- निर्माता के प्रोटोकॉल का उपयोग करके नेस्टेड पीसीआर (तालिका 1) द्वारा नमूना बढ़ाना । पीसीआर प्रॉडक्ट्स का साइज 300-500 बीपी तय है ।
- डीएनए पोलीमरेज़ के साथ lysis उत्पाद के 1 µ एल और sgRNA लक्ष्य साइट (0.1 µ एम, अंतिम एकाग्रता) (तालिका 1) के आसपास के अनुक्रम को पहचानने बाहरी प्राइमरों की एक जोड़ी मिश्रण, और 20 µ एल की मात्रा में प्राथमिक पीसीआर प्रदर्शन.
- 5 मिनट के लिए 95 ° c पर डीएनए पोलीमरेज़ सक्रिय करें, और 30 के लिए 95 ° c 30 एस के लिए, 60 ° c 30 s के लिए, और 72 ° c 24 s (1 min/1 kb) के लिए, 72 ° c पर 5 मिनट के लिए एक अंतिम विस्तार के साथ प्राथमिक पीसीआर प्रदर्शन ।
- प्राथमिक पीसीआर उत्पाद के 1 µ एल और नेस्टेड इनर प्राइमरों की एक जोड़ी का उपयोग कर माध्यमिक पीसीआर प्रदर्शन करते हैं ।
- स्वभाव और पुनः-ऐनी 300-600 शुद्ध पीसीआर उत्पाद के एनजी 20 µ में 1 × T7EI प्रतिक्रिया बफर के एल (50 मिमी NaCl, 10 मिमी Tris-एचसीएल, 10 मिमी MgCl2, 1 मिमी डीटीटी पीएच 7.9) 5 डिग्री सेल्सियस की दर के साथ 95 डिग्री सेल्सियस से 25 डिग्री सेल्सियस के तापमान का एक ढाल का उपयोग कर 5/
- annealed पीसीआर उत्पादों के लिए T7EI एंजाइम के 1 µ एल जोड़ें और 2 एच के लिए 37 डिग्री सेल्सियस पर पचा । फिर 1 × ताे में 2% agarose जेल पर पाचन उत्पाद चलाएँ 40 मिनट के लिए 120 V में बफर जब तक टुकड़े अलग कर रहे हैं ( सामग्री की तालिकादेखें).
- ImageJ का उपयोग करने के लिए कट और बिना खतना के डीएनए के बैंड तीव्रता निर्धारित करते हैं । पहले रिपोर्ट की गई विधियों का उपयोग करते हुए indel आवृत्ति की गणना करें ( चित्र 1C).
-
दाता वेक्टर का निर्माण
नोट: Cdx2 जीन के लिए HMEJ दाता वैक्टर उत्पंन करने के लिए, एक दाता डीएनए का निर्माण (800 बीपी एचएएल-p2A-mCherry-800 बीपी मलक) दोनों सिरों पर 23 nt Cdx2-sgRNAs लक्ष्यीकरण अनुक्रम के साथ पार्श्व (चित्रा 1डी और चित्रा 1ई) । लक्ष्य अनुक्रम के पाम मुताबिक़ हाथ के अंत से सटे था । गिब्सन विधानसभा HMEJ दाता क्लोनिंग के लिए सिफारिश की है ।- बढ़ाना 800 bp बाएं समरूपता बांह (एचएएल) के साथ आगे प्राइमर-1F (से 15-20 nt ओवरलैप अनुक्रम वेक्टर, 23 nt Cdx2-sgRNA लक्ष्यीकरण अनुक्रम, और के बारे में 20 nt अनुक्रम से एचएएल) और रिवर्स प्राइमर-1R (15-20 से युक्त बीपी ओवरलैप अनुक्रम p2A-mCherry और एचएएल से 20 nt अनुक्रम के बारे में) 0.1 µ m अंतिम एकाग्रता में माउस जीनोमिक डीएनए का उपयोग कर 200 एनजी/µ l (चित्रा 1डी, तालिका 1).
- आगे प्राइमर के साथ p2A-mCherry सम्मिलन टुकड़ा बढ़ाना-2F (से 15-20 nt ओवरलैप अनुक्रम एचएएल से और के बारे में 20 nt अनुक्रम प्रविष्टि अंश से) और रिवर्स प्राइमर-2R (15-20 nt से आच्छादित अनुक्रम से और के बारे में 20 nt अनुक्रम प्रविष्टि टुकड़ा) 0.1 µ एम अंतिम एकाग्रता में जीनोमिक डीएनए या रिपोर्टर दृश्यों के साथ प्लाज्मिड का उपयोग कर 100 एनजी/µ l या 30 एनजी/µ एल (चित्रा 1डी, तालिका 1) ।
- आगे प्राइमर के साथ 800 बीपी सही समरूपता बांह (हर) बढ़ाना-3F (15-20 nt से आच्छादित अनुक्रम वेक्टर से, 23 nt Cdx2-sgRNA लक्ष्यीकरण अनुक्रम, और के बारे में 20 nt अनुक्रम से हर) और रिवर्स प्राइमर-3R (15-20 nt युक्त से ओवरलैप अनुक्रम p2A-mCherry और हर के बारे में 20 nt अनुक्रम पर) 0.1 µ m अंतिम एकाग्रता में माउस जीनोमिक डीएनए का उपयोग कर 200 एनजी/µ एल (चित्रा 1डी, तालिका 1) ।
- 1 × ताे बफर में 1% agarose जेल पर सभी पीसीआर उत्पादों को चलाएं और जेल निष्कर्षण किट द्वारा अपेक्षित आकार के पीसीआर उत्पादों को शुद्ध करें, निर्माता के निर्देशों (तालिका 1) के अनुसार ।
- डाइजेस्ट KpnI और XbaI द्वारा एक के साथ एक निर्माण वेक्टर की कैलरी एनजी । मिश्रण 2 µ एल में रैखिक सदिश का l 30-40 एनजी/µ l के साथ तीन पीसीआर परिवर्धित टुकड़े (1 µ एल के लिए प्रत्येक, $२०० एनजी/2x गिब्सन मिश्रण में µ एल) । 60 मिनट के लिए 50 डिग्री सेल्सियस पर मिश्रण को 10 µ एल के लिए अंतिम मात्रा को समायोजित करने के लिए H2O जोड़ें ।
- सभी इकट्ठे उत्पाद के साथ सक्षम ई. कोलाई कोशिकाओं को बदलने और निर्माता के निर्देशों के अनुसार डीएनए निष्कर्षण किट द्वारा प्लाज्मिड निर्माणों को निकालने. डीएनए अनुक्रमण द्वारा HMEJ दाता सत्यापित करें ।
2. HMEJ-आधारित विधि का उपयोग कर माउस भ्रूण में जीनोम संपादन
-
Cas9 mRNA का उत्पादन
- Cas9 mRNA तैयारी के लिए, पीसीआर प्रवर्धन द्वारा Cas9 कोडिंग क्षेत्र के लिए T7 प्रमोटर अनुक्रम जोड़ने के लिए उपयुक्त प्राइमरी जोड़ी तालिका 1में सूचीबद्ध का उपयोग कर । 1 × उच्च निष्ठा डीएनए पोलीमरेज़ मिश्रण करने के लिए वेक्टर व्यक्त करने के Cas9 के एनजी-0.1 µ मीटर और 20 के एक अंतिम एकाग्रता में प्राइमर Cas9 F/R जोड़ें । अंतिम खंड को 50 µ l को एच2O के साथ समायोजित करें ।
- 5 मिनट के लिए 95 ° c पर डीएनए पोलीमरेज़ सक्रिय करें, और के लिए 95 ° c पर 36 चक्र के लिए पीसीआर प्रदर्शन 30 s, 60 ° c के लिए 30 s, और 68 ° c 4 मिनट के लिए (1 min/1 kb), 68 ° c पर एक अंतिम विस्तार के साथ 10 मिनट के लिए.
- शुद्ध T7-Cas9 पीसीआर उत्पाद के लिए इन विट्रो प्रतिलेखन (IVT), और फिर mRNA प्रतिलेखन किट द्वारा 0.5-1 µ जी डीएनए टाइप 20 µ एल की कुल मात्रा में 37 ° c के लिए, निर्माता के निर्देशों के अनुसार ( सामग्री की तालिकादेखें).
- मिश्रण करने के लिए DNase के 1 µ l जोड़ें 15 मिनट के लिए 37 डिग्री सेल्सियस पर डीएनए टेम्पलेट को दूर करने के लिए. एक पाली-37 डिग्री सेल्सियस पर 45 मिनट के लिए एक पूंछ जोड़ें और Cas9 mRNA द्वारा आरएनए शुद्धि किट, निर्माता के निर्देशों के अनुसार पुनर्प्राप्त करें ( सामग्री की तालिकादेखें) ।
-
sgRNA का उत्पादन
- ऊपर के रूप में उच्च निष्ठा डीएनए पोलीमरेज़ के साथ एक T7 प्रमोटर द्वारा संचालित sgRNA टेम्पलेट उत्पन्न करते हैं । एक sgRNA टेंपलेट के रूप में वेक्टर युक्त पाड़ चुनें । इस्तेमाल किया प्राइमरों तालिका 1में सूचीबद्ध हैं ।
- T7-sgRNA पीसीआर उत्पाद को शुद्ध और के लिए टेम्पलेट के रूप में 0.5-1 µ g डीएनए का उपयोग करें इन विट्रो में एक लघु आरएनए प्रतिलेखन किट का उपयोग sgRNA के लिए 37 ° c में 6 h के लिए 20 µ l की कुल मात्रा, निर्माता के निर्देशों के अनुसार ( सामग्री की तालिका देखें < /c11 >).
- मिश्रण करने के लिए DNase के 1 µ एल जोड़ें और 15 मिनट के लिए 37 ° c पर मशीन जारी रखने के लिए डीएनए टेंपलेट हटा दें । आरएनए शुद्धि किट द्वारा sgRNAs को शुद्ध करें ( सामग्री की तालिकादेखें) ।
- RNase-मुक्त पानी में 500 एनजी/µ एल के लिए sgRNA को पतला करें और नमूनों को 3 महीने तक − 80 डिग्री सेल्सियस पर स्टोर करें ।
नोट: CRISPR ribonucleoproteins (RNPs) एक बेहतर काटने दक्षता18,19,20के साथ एक वैकल्पिक प्रतिस्थापन कर रहे हैं ।
-
भ्रूण संग्रह, microinjection और इन विट्रो संस्कृति
- Superovulate female B6D2F1 (C57BL/6 × DBA2J) चूहों (7-8 सप्ताह पुरानी) गर्भवती घोड़ी से सीरम गोनॉडोट्रॉफिन (PMSG), जिसके बाद मानव chorionic गोनॉडोट्रॉफिन (एचसीजी) 48 एच बाद में । एचसीजी इंजेक्शन के बाद, B6D2F1 पुरुषों के साथ घर महिलाओं रात भर ।
- सह2 संज्ञाहरण, एचसीजी इंजेक्शन के बाद 24 एच द्वारा महिलाओं बलिदान । M2 माध्यम में (प्रत्येक महिला के लिए 30-50 भ्रूण के साथ) उनके oviducts से निषेचित भ्रूण ले लीजिए ।
- निषेचित भ्रूण (एक दिवसीय इंजेक्शन के लिए लगभग 300 अंडे) KSOM मध्यम (5.55 g/L NaCl में रखें, ०.१९ g/l KCl, 0.05 g/l KH2PO4, 0.05 g/l MgSO4• 7H2हे, 0.04 g/l ग्लूकोज, 1.12 g/l सोडियम स्तनपान, 2.1 g/l NaHCO3 , 0.02 g/L सोडियम पाइरूवेट, 0.25 g/l CaCl2• 2H2O, ०.००४ g/l EDTA, ०.१४६ g/l l-glutamine, और 1 g/l गोजातीय सीरम एल्ब्युमिन) 37 ° c पर 5% CO2के साथ एक मशीन में ।
- Mix Cas9 mRNA (100 एनजी/µ एल), sgRNA (50 एनजी/µ एल), और HMEJ दाता सदिश (100 एनजी/µ एल), और एच2ओ जोड़ने के लिए अंतिम मात्रा को समायोजित करने के लिए 10 µ एल मिश्रण बर्फ पर रखो ।
- एक Micropipette खींचने का उपयोग (पैरामीटर्स: हीट, 74; खींचो, 60; वेग, 80; समय/देरी, 200; दबाव, 300) केशिका सुई खींचो (बाहरी व्यास 1.0 मिमी, रेशा के साथ ०.७८ मिमी । सामग्री की तालिकादेखें) । वाणिज्यिक सुई microinjection के लिए एक वैकल्पिक प्रतिस्थापन होगा ।
- मिश्रण की एक छोटी बूंद में अच्छी तरह से परिभाषित pronuclei के साथ zygotes के कोशिका द्रव्य में एक संभावित मात्रा सुई HEPES-CZB 5 µ जी/एमएल cytochalasin बी युक्त लगातार प्रवाह सेटिंग्स (चित्रा2 ए) के साथ एक microinjector का उपयोग (देखें सामग्री की तालिका)21.
नोट: zygotes के प्रत्येक समूह 20-30 मिनट के भीतर इंजेक्शन किया जाना चाहिए । Cytochalasin बी इंजेक्शन के बाद माउस युग्मनज की व्यवहार्यता को बढ़ा सकता है । वैकल्पिक रूप से, microinjection पीजो प्रणाली के साथ संचालित किया जा सकता है, के रूप में पहले से ही वर्णित22। - संस्कृति KSOM मध्यम में 37 डिग्री सेल्सियस से कम 5% सह के तहत2 में इंजेक्शन zygotes प्रतिदीप्ति अवलोकन के लिए 3.5 दिनों के बाद ब्लास्टोसिस्ट चरण तक (आंकड़े २ बी और 2सी).
-
भ्रूण हस्तांतरण और चूहों की पीढ़ी
- मेट मद इंजेक्शन के रूप में उसी दिन vasectomized ICR नर चूहों के साथ ICR मादा चूहे ।
- संस्कृति 2 में इंजेक्शन zygotes-सेल चरण में 37 ° c के तहत 5% सह2, और स्थानांतरण 25-30 2-0.5 दिन पोस्ट coitum (डीपीसी) में pseudopregnant ICR महिलाओं के oviducts में सेल भ्रूण । प्राप्तकर्ता माताओं 19.5 डीपीसी में पिल्ले उद्धार ।
-
माउस genotyping
- एक डीएनए निष्कर्षण किट का उपयोग कर पैर की अंगुली या पूंछ के नमूनों से माउस जीनोमिक डीएनए निकालें, निर्माता के निर्देशों के अनुसार ( सामग्री की तालिकादेखें).
- की पहचान 5 ' और ' 3 दस्तक के जंक्शन-जीनोमिक डीएनए के 200-400 एनजी का उपयोग करने के लिए एक टेंपलेट के रूप में स्पेक्ट्रोमेट्री द्वारा मापा पीसीआर amplification प्रदर्शन ।
- 5 मिनट के लिए 95 ° c पर डीएनए पोलीमरेज़ सक्रिय करें, और के लिए 95 डिग्री सेल्सियस पर 38 चक्र के लिए पीसीआर प्रदर्शन 30 एस, 60 ° c के लिए 30 s, और 72 ° c 1 मिनट के लिए (1 min/1 kb), 72 डिग्री सेल्सियस पर एक अंतिम विस्तार के साथ 10 मिनट के लिए. 5 ' जंक्शन के लिए, एचएएल के ऊपर आगे प्राइमर का उपयोग करें, दस्तक में टुकड़ा (p2A-mCherry) पर रिवर्स एक के साथ. 3 ' जंक्शन के रूप में, दस्तक पर आगे प्राइमर का उपयोग करें-टुकड़ा में (p2A-mCherry), हर (तालिका 1) के बहाव पर रिवर्स एक के साथ ।
- 1% agarose जेल पर पीसीआर प्रोडक् ट की 6 µ l को 1 × ताे बफ़र में चलाएं और अपेक्षित अंश आकार के लिए जांचें । फिर डीएनए अनुक्रमण (चित्रा 2डी) द्वारा उन्हें सत्यापित करें.
3. HMEJ-हेपैटोसाइट्स में Vivo जीनोम संपादन में आधारित
- प्लेस प्राप्तकर्ता C57BL/6J माउस (8 सप्ताह) एक निरोधक उपकरण में और भट्ठा के माध्यम से पूंछ रखो ।
- खारा समाधान के 2 मिलीलीटर में HMEJ दाता वैक्टर (30 µ g) और spCas9 एक्सप्रेशन वैक्टर (30 µ ग्राम) मिलाएं । नियंत्रण प्रयोग के लिए, HMEJ दाता वैक्टर (30 µ ग्राम) खारा समाधान (चित्रा 3) के 2 मिलीलीटर में निलंबित ।
- 70% इथेनॉल के साथ माउस पूंछ साफ । पूंछ नस में सुई डालें और 5-7 एस के भीतर प्लाज्मिड डीएनए समाधान इंजेक्षन. सुई निकालें और निरोधक डिवाइस से माउस छोड़ें ।
- इंजेक्शन के बाद 5-9 दिनों के बाद सह द्वारा चूहों2 संज्ञाहरण बलिदान । Perfuse के साथ transcardially चूहों को 0.9% खारा, 4% paraformaldehyde एक सिकुड़नेवाला पंप का उपयोग कर के बाद, और 4 डिग्री सेल्सियस पर रात भर जिगर ठीक ।
- निर्जलीकरण 30% सुक्रोज रात भर का उपयोग कर ऊतक, जब तक यह ट्यूब के नीचे तक डूब ।
- धारा 10 जिगर के नमूनों के लिए µm की मोटाई पर जमे हुए ऊतक ।
- 0.1 M फॉस्फेट में तीन बार वर्गों कुल्ला-बफर (पंजाब) और प्राथमिक एंटीबॉडी के साथ उन्हें गर्मी: खरगोश विरोधी mCherry (5% NGS में पतला) 4 डिग्री सेल्सियस पर रातोंरात.
- धो वर्गों पंजाब में तीन बार, और फिर उंहें माध्यमिक एंटीबॉडी के साथ मशीन: Cy3-AffiniPure बकरी विरोधी खरगोश आईजीजी के लिए कमरे के तापमान पर 2 घंटे के लिए एक कक्षीय शेखर पर ।
- Counterstain 20 मिनट के लिए DAPI के साथ वर्गों और आगे प्रतिदीप्ति अवलोकन के लिए ग्लास स्लाइड पर ग्लिसरीन के साथ माउंट (चित्रा 3बी) ।
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
माउस भ्रूण में HMEJ आधारित जीनोम संपादन: को HMEJ की क्षमता में दस्तक-परिभाषित माउस zygotes में विधि आधारित है, हम Cas9 mRNA, वितरित sgRNA माउस Cdx2 है, जो HMEJ के पिछले zygotes को एक p2A-mCherry रिपोर्टर जीन फ्यूज डिजाइन किया गया था में codon जीन और Cdx2 दाता लक्ष्यीकरण जीन (चित्रा 2ए) । इंजेक्शन zygotes संस्कृति में blastocysts में विकसित की है । के लिए दस्तक में दक्षता का मूल्यांकन, हम एक फ्लोरोसेंट माइक्रोस्कोप के साथ mCherry प्रतिदीप्ति का विश्लेषण किया, और हमने पाया कि blastocysts प्राप्त HMEJ दाताओं के 12.9% mCherry, जो सख्ती trophectoderm में व्यक्त किया गया था के लिए सकारात्मक थे (आंकड़े बी , 2c). पीसीआर पॉजिटिव चूहों अनुक्रमण करके, हम यह भी पाया कि सभी की जांच की एकीकरण की घटनाओं में फ्रेम एकीकरण दोनों 5 ' और 3 ' जंक्शनों (चित्रा 2डी) में सटीक थे ।
HMEJ-वयस्क ऊतकों और HMEJ-मध्यस्थता जीन थेरेपी में आधारित जीनोम संपादन: जांच करने के लिए कि क्या HMEJ आधारित जीनोम संपादन वयस्क ऊतकों में लागू किया जा सकता है, हम mCherry कैसेट डाला सही रोकने के codon से पहले Actb जीन के transducing Actb-HMEJ के लिए constructs C57/B6J माउस जिगर द्वारा पूंछ-नस hydrodynamic इंजेक्शन (चित्रा 3ए) । इंजेक्शन के 7 दिनों के बाद, हमने पाया कि transfected हेपैटोसाइट्स के लगभग आधे जिगर वर्गों पर दाग के रूप में mCherry व्यक्त (चित्रा 3बी) ।
जीन थेरेपी के लिए एक HMEJ आधारित रणनीति का उपयोग करने की संभावना का पता लगाने के लिए, हम fumarylacetoacetate hydrolase (फह) की कमी चूहों कार्यरत हैं । फह-/- माउस एक अच्छी तरह से स्थापित वंशानुगत tyrosinemia प्रकार मैं (HTI) माउस मॉडल है, जो फह जीन के 5 एक्सॉन में एक प्रविष्टि टुकड़ा बंदरगाह, निंनलिखित अनुक्रम23में frameshift उत्परिवर्तनों के कारण । फह-/ चूहों को बनाए रखने के लिए, हम tyrosine catabolic मार्ग के ऊपर के एक अवरोधक के साथ फह-/ चूहों का इलाज किया, 2-(2-नाइट्रो-4-trifluoromethylbenzoyl)-1, 3-cyclohexanedione (NTBC)24. यहां हम बाहर सेट को देखने के लिए कि क्या MMEJ-और HMEJ मध्यस्थता जीन सुधार फहमें फह उत्परिवर्तन बचाव सकता है-/ हम hydrodynamically इंजेक्शन Cas9 के साथ एक साथ निर्माण फह-MMEJ या फह-HMEJ constructs, सम्मिलित करने के लिए डिज़ाइन किया गया फह सीडीएनए के एक्सॉन 5 करने के लिए intron 4 में फह जीन, करने के लिए फह-/ माउस जिगर ( चित्र 3 ग). इंजेक्शन के बाद एक सप्ताह, NTBC जिगर की क्षति (चित्रा 3सी) प्रेरित करने के लिए वापस ले लिया गया था । NTBC की वापसी के बाद, फह-सही हेपैटोसाइट्स के फह-/ चूहों को प्राप्त करने वाले फह-HMEJ और Cas9 construction ने MMEJ-आधारित विधि से अधिक प्रभावी प्रसार दिखाया (चित्रा 3डी ).

चित्र 1 : HMEJ-मध्यस्थता इन विट्रो मेंलक्षित एकीकरण ।
(क) sgRNAs के चयन के लिए प्रायोगिक योजना: छः विभिन्न sgRNAs (Cdx2-sgRNA1 ~Cdx2-sgRNA6) के आसपास के पड़ाव codon के लिए एक उच्च पद और ऑफ-टारगेट क्षमता वाले Cdx2 लोकस को ऑनलाइन CRISPR डिजाइन के आधार पर चुना गया उपकरण. protospacer आसंन आकृति (पाम) अनुक्रम लाल रंग में है । (ख) प्रायोगिक डिजाइन: Cas9-सीएमवी-GFP अभिव्यक्ति plasmids व्यक्त sgRNA, Cas9, और GFP N2a कोशिकाओं में पेश किया गया. GFP+ कोशिकाओं को सर्वेयर परख के लिए 3 दिन में हल किया गया । (ग) Cdx2 लक्ष्यीकरण के लिए सर्वेयर परख: 6 विभिन्न sgRNAs को सर्वेयर परख के लिए तैयार किया गया. सामान्य N2a कोशिका जीनोमिक डीएनए नियंत्रण के रूप में कार्य करता है. *, sgRNA के लिए इस्तेमाल किया Cdx2-2a-mCherry दस्तक में प्रयोग । (घ) गिब्सन विधानसभा का उपयोग HMEJ दाताओं के निर्माण की योजनाबद्ध सिंहावलोकन । (ङ) HMEJ के योजनाबद्ध सिंहावलोकन- Cdx2 लोकस पर मध्यस्थता जीन लक्ष्यीकरण रणनीति. HAL/मलक, वाम/सही समरूपता बांह; त्रिकोण, sgRNA लक्ष्य साइटों; की/या, बाहरी आगे/ यदि/आईआर, इनर आगे/ पिछली रिपोर्ट10से संशोधित चित्र । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।

चित्र 2 : HMEJ के माध्यम से माउस भ्रूण में जीनोम संपादन-मध्यस्थता लक्षित एकीकरण
(क) microinjection की प्रयोगात्मक योजना: Cas9 mRNA का एक मिश्रण (100 एनजी/µ एल), sgRNA (50 एनजी/µ एल), और दाता plasmids (100 एनजी/µ एल) माउस zygotes में इंजेक्ट किया गया । (ख) HMEJ रणनीति द्वारा संपादित माउस भ्रूण के प्रतिनिधि प्रतिदीप्ति छवियां । बार, 20 µm. (ग) में दस्तक-क्षमता mCherry+ blastocysts के प्रतिशत से संकेत दिया । प्रत्येक बार के ऊपर संख्या, कुल blastocysts गिना । (घ) Cdx2 लोकस में जीन संपादित चूहों का अनुक्रम विश्लेषण. पीसीआर उत्पाद 5 ' और 3 ' जंक्शन साइटों से परिवर्धित अनुक्रम थे । ऊपरी, समरूपता बांह; बैंगनी, p2A; लाल, mCherry; मलक या एचएएल, दाएं या बाएं मुताबिक़ बांह । डैश्ड रेखाएं स्पष्टता के लिए छोड़ा गया क्षेत्र चिह्नित करते हैं । पिछली रिपोर्ट10से संशोधित चित्र । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।

चित्र 3 : HMEJ-मध्यस्थता vivo मेंलक्षित एकीकरण ।
(एक) hydrodynamic पूंछ नस इंजेक्शन का योजनाबद्ध सिंहावलोकन । plasmids व्यक्त दाता अनुक्रम और sgRNA का एक मिश्रण है, और plasmids व्यक्त spCas9 hydrodynamic पूंछ नस इंजेक्शन के माध्यम से जिगर को दिया गया । (B) प्रतिनिधि इम्यूनोफ्लोरेसेंस की छवियां हेपैटोसाइट्स । लिवर सेक्शन में 7 दिन पोस्ट इंजेक्शन एकत्र किए गए । स्केल बार, 50 µm. GFP, transfected कक्ष । (ग) या तो MMEJ के Plasmids-या HMEJ मध्यस्थता जीन प्रतिस्थापन सीडीएनए इंजेक्शन द्वारा एक्सॉन में 5 से 14 intron के फह फह में शामिल करने के लिए डिज़ाइन किया गया रणनीति फह जीन के hydrodynamic में वितरित किया गया -/ NTBC on: फह-/ चूहों को NTBC जल पर बनाए रखा गया; NTBC बंद: NTBC पानी की वापसी (NTBC वापसी के पहले दिन के रूप में परिभाषित किया गया था 0 दिन, जो इंजेक्शन के बाद 7 दिन है) । (घ) फह immunohistochemistry से यकृत वर्गों के दाग- फह-/ चूहों MMEJ या HMEJ plasmids के साथ इंजेक्शन. स्केल बार, 100 µm । पिछली रिपोर्ट्स5,10से संशोधित आंकड़ा । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।
Subscription Required. Please recommend JoVE to your librarian.
Discussion
HMEJ दाता plasmids के निर्माण में सबसे महत्वपूर्ण कदम हैं: (1) उच्च डीएनए दरार दक्षता और sgRNA काटने साइट और स्टॉप codon के बीच कम दूरी के साथ sgRNA का चयन, और (2) HMEJ दाता का उचित निर्माण । CRISPR/Cas9-दोनों transgene दाता सदिश पर मध्यस्थता दरार (sgRNA लक्ष्य साइटों और ~ 800 बीपी समरूपता हथियार युक्त) और लक्षित जीनोम vivo में कुशल और सटीक लक्षित एकीकरण के लिए आवश्यक है । HMEJ-आधारित विधि का उपयोग कर चूहों में दस्तक की पीढ़ी के सबसे महत्वपूर्ण कदम हैं: (१) Cas9 mRNA और sgRNA की उच्च गुणवत्ता की तैयारी (कोई अध Cas9 mRNA और sgRNA में मौजूद नहीं है), और (२) उच्च कोटि के HMEJ दाता प्लाज्मिड की तैयारी. प्लाज्मिड भ्रूण विकास पर कोई विषाक्त प्रभाव से पता चलता है ।
हाल ही में, एक NHEJ आधारित विधि भी vivo जीनोम संपादन8 में कुशल के लिए सूचित किया गया था । फिर भी, indel उत्परिवर्तनों के विभिंन प्रकार आमतौर पर जंक्शनों पर प्रेरित किया गया, के रूप में पिछले रिपोर्टों8में वर्णित है, यह मुश्किल सटीक एकीकरण प्राप्त करने के लिए बना । यहां, HMEJ आधारित रणनीति हम ऊपर वर्णित मुश्किल से किसी भी indel उत्परिवर्तनों के साथ सटीक लक्षित एकीकरण दिखाया । इस प्रकार, एक HMEJ आधारित रणनीति (जैसे एक बिंदु उत्परिवर्तन के रूप में) सही एक है, जो NHEJ के लिए लागू नहीं है विधि के साथ एक रूपांतरित अनुक्रम की जगह के लिए एक आदर्श मंच हो सकता है ।
मोज़ेक भ्रूण में जीन संपादन के लिए एक बड़ी समस्या है । mRNA के बजाय एक पूर्व भ्रूण चरण में Cas9 प्रोटीन के इंजेक्शन transgene को प्राप्त कर सकते हैं-में एक सेल स्टेज पर मोज़ेक बिना । नैदानिक अनुप्रयोगों के लिए, CRISPR/Cas9 प्रणालियों के वयस्क ऊतकों में प्रसव अभी भी चुनौतीपूर्ण है ।
HMEJ आधारित जीनोम संपादन के कई भविष्य संभावित उपयोग कर रहे हैं । यह आनुवंशिक रूप से संशोधित पशु मॉडल उत्पन्न करने के लिए इस्तेमाल किया जा सकता है । अपने उच्च दस्तक पर विचार-भ्रूण में दक्षता में, इस विधि काफी जानवर आनुवंशिक रूप से संशोधित पशु मॉडल पैदा करने के लिए आवश्यक संख्या कम हो सकता है, और विशेष रूप से गैर मानव रहनुमा आनुवंशिक मॉडल पैदा करने की संभावना को खोलता है । HMEJ आधारित जीनोम का संपादन वंश का पता लगाने के वयस्क ऊतकों, जो विशेष रूप से पशु मॉडलों के लिए उपयोगी है में व्यक्तिगत कोशिका प्रकार कर सकते हैं, के बाद से वहां ऐसी गैर के रूप में उपलब्ध पशु मॉडल, मानव रहनुमाओं की कमी है । यह लक्षित जीन चिकित्सा के लिए इस्तेमाल किया जा सकता है: एक HMEJ आधारित रणनीति के सबसे आकर्षक आवेदन क्लिनिक का उपयोग करता है के लिए जीन थेरेपी है । इस अध्ययन में, हम वंशानुगत tyrosinemia प्रकार मैं चूहों hydrodynamic इंजेक्शन के द्वारा संकेत वैक्टर के फह उत्परिवर्तन सही । हालांकि, CRISPR/Cas9 प्रणाली के वयस्क ऊतकों में प्रसव अभी भी नैदानिक उपयोग के लिए प्रमुख तकनीकी चुनौती है, के रूप में hydrodynamic इंजेक्शन रोगियों में प्रदर्शन की संभावना नहीं है । वर्तमान में, प्रसव की रणनीति के आगे सुधार तत्काल क्लिनिक में इस HMEJ-आधारित पद्धति का अनुवाद करने से पहले की जरूरत है ।
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
लेखकों का खुलासा करने के लिए कुछ नहीं है ।
Acknowledgments
यह काम कैस रणनीतिक प्राथमिकता अनुसंधान कार्यक्रम (XDB02050007, XDA01010409), नेशनल Hightech आर एंड डी कार्यक्रम (863 कार्यक्रम; 2015AA020307), चीन के राष्ट्रीय प्राकृतिक विज्ञान फाउंडेशन (NSFC अनुदान ३१५२२०३७, ३१५००८२५, ३१५७१५०९, के द्वारा समर्थित किया गया था, ३१५२२०३८), चीन युवा हजार प्रतिभा कार्यक्रम (HY करने के लिए), चीनी विज्ञान अकादमी के परियोजना के माध्यम से तोड़, शंघाई सिटी साइंस और प्रौद्योगिकी परियोजना (16JC1420202 HY) की समिति, चीन के विज्ञान और प्रौद्योगिकी मंत्रालय (MOST; 2016YFA0100500) ।
Materials
| Name | Company | Catalog Number | Comments |
| pX330 | Addgene | 42230 | |
| pAAV vector | Addgene | 37083 | |
| pX260 | Addgene | 42229 | |
| AAV_Efs_hSpCas9_NLS_FLAG-SV40 | Addgene | 97307 | AAV vector for encoding a human codon-optimized SpCas9 driven by EFs promoter |
| AAV_Actb HMEJ donor_U6_sgRNA_EF1a_GFP_polyA | Addgene | 97308 | HMEJ donor for fusing a p2A-mCherry reporter to mouse Actb. EGFP driven by EF1a promoter and U6-driven sgRNAs targeting Actb. AAV backbone. |
| AAV_Cdx2 HMEJ donor | Addgene | 97319 | HMEJ donor for fusing a p2A-mCherry reporter to mouse Cdx2. |
| Lipofectamine 3000 Transfection Reagent | Life Technology | L3000015 | |
| Nuclease-Free Water | Life Technologies | AM9930 | |
| Bbs I | New England Biolabs | R0539S | |
| NEB Buffer 2 | New England Biolabs | B7002S | |
| T7 endonuclease I | New England Biolabs | M0302L | |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621L | |
| Plasmid EndoFree-Midi Kit | Qiagen | 12143 | |
| MMESSAGE MMACHINE T7 ULTRA | Life Technologies | AM1345 | |
| MEGACLEAR KIT 20 RXNS | Life Technologies | AM1908 | |
| MEGASHORTSCRIPT T7 KIT 25 RXNS | Life Technologies | AM1354 | |
| Flaming/Brown Micropipette Puller | Sutter Instrument | P-97 | Micropipette Puller (parameters: heat, 74; pull, 60; velocity, 80; time/delay, 200; pressure, 300) |
| Borosilicate glass | Sutter Instrument | B100-78-10 | type of capillaries (outer diameter 1.0 mm, inner diameter 0.78 mm with filament) |
| FemtoJet microinjector | Eppendorf | ||
| Freezing microtome | Leica | CM1950-Cryostat | thickness of 40 μm for brain, 10 μm for liver |
| Rabbit anti-mCherry | GeneTex | ||
| Cy3-AffiniPure Goat Anti-Rabbit IgG | Jackson Immunoresearch | ||
| DMEM | Gibco | 11965092 | |
| FBS | Gibco | 10099141 | |
| NEAA | Gibco | 11140050 | |
| Pen,Strep,Glutamine | Gibco | 10378016 | |
| Gel Extraction Kit | Omega | D2500-02 | |
| FACS | BD AriaII | ||
| PMSG | Ningbo Sansheng Medicine | S141004 | |
| HCG | Ningbo Sansheng Medicine | B141002 | |
| Cytochalasin B | Sigma | CAT#C6762 | |
| KSOM+AA with D-Glucose and Phenol Red | Millipore | CAT#MR-106-D | |
| M2 Medium with Phenol Red | Millipore | CAT#MR-015-D | |
| Mineral oil | Sigma |
References
- Yang, H., et al. Generation of Mice Carrying Reporter and Conditional Alleles by CRISPR/Cas-Mediated Genome Engineering. Cell. 154 (6), 1370-1379 (2013).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nature Biotechnology. 29 (8), 731-734 (2011).
- Nakade, S., et al. Microhomology-mediated end-joining-dependent integration of donor DNA in cells and animals using TALENs and CRISPR/Cas9. Nature Communications. 5, 5560 (2014).
- Hisano, Y., et al. Precise in-frame integration of exogenous DNA mediated by CRISPR/Cas9 system in zebrafish. Scientific reports. 5, 8841 (2015).
- Yao, X., et al. Cas9 - Mediated Precise Targeted Integration In Vivo Using a Double Cut Donor with Short Homology Arms. EBioMedicine. , (2017).
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome research. 24 (1), 142-153 (2014).
- Maresca, M., Lin, V. G., Guo, N., Yang, Y. Obligate ligation-gated recombination (ObLiGaRe): custom-designed nuclease-mediated targeted integration through nonhomologous end joining. Genome Research. 23 (3), 539-546 (2013).
- Suzuki, K., et al. In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration. Nature. 540 (7631), 144-149 (2016).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Yao, X., et al. Homology-mediated end joining-based targeted integration using CRISPR/Cas9. Cell Research. 27 (6), 801-814 (2017).
- Han, D. W., et al. Direct reprogramming of fibroblasts into epiblast stem cells. Nature Cell Biology. 13 (1), 66-71 (2011).
- Han, D. W., et al. Direct Reprogramming of Fibroblasts into Neural Stem Cells by Defined Factors. Cell Stem Cell. , (2012).
- Ambasudhan, R., et al. Direct reprogramming of adult human fibroblasts to functional neurons under defined conditions. Cell Stem Cell. 9 (2), 113-118 (2011).
- Sparman, M., et al. Epigenetic reprogramming by somatic cell nuclear transfer in primates. Stem Cells. 27 (6), 1255-1264 (2009).
- Schatten, G., Mitalipov, S. Developmental biology: Transgenic primate offspring. Nature. 459 (7246), 515-516 (2009).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Quadros, R. M., et al. Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome Biology. 18 (1), 92 (2017).
- Park, K. E., et al. Targeted Gene Knockin in Porcine Somatic Cells Using CRISPR/Cas Ribonucleoproteins. International journal of molecular sciences. 217 (6), (2016).
- Woo, J. W., et al. DNA-free genome editing in plants with preassembled CRISPR-Cas9 ribonucleoproteins. Nature biotechnology. 33 (11), 1162-1164 (2015).
- Harms, D. W., et al. Mouse Genome Editing Using the CRISPR/Cas System. Current protocols in human genetics. 83, 11-27 (2014).
- Yang, H., Wang, H., Jaenisch, R. Generating genetically modified mice using CRISPR/Cas-mediated genome engineering. Nature protocols. 9 (8), 1956-1968 (2014).
- Grompe, M., et al. Loss of Fumarylacetoacetate Hydrolase Is Responsible for the Neonatal Hepatic-Dysfunction Phenotype of Lethal Albino Mice. Genes & development. 7 (12), 2298-2307 (1993).
- Paulk, N. K., et al. Adeno-associated virus gene repair corrects a mouse model of hereditary tyrosinemia in vivo. Hepatology. 51 (4), 1200-1208 (2010).
Tags
आनुवंशिकी अंक 133 CRISPR/Cas9 लक्षित एकीकरण समरूपता-मध्यस्थ अंत में शामिल होने Vivo में भ्रूण आनुवंशिक रूप से संशोधित चूहों Hydrodynamic इंजेक्शनErratum
Formal Correction: Erratum: CRISPR/Cas9-mediated Targeted Integration In Vivo Using a Homology-mediated End Joining-based Strategy
Posted by JoVE Editors on 03/10/2021.
Citeable Link.
An erratum was issued for: Studying TGF-β Signaling and TGF-β-induced Epithelial-to-mesenchymal Transition in Breast Cancer and Normal Cells. The phrases "surveyor assay" and "Surveyor Nuclease" have been updated to "T7E1 assay" to " T7 endonuclease I" respectively.
Step 1.2 in the Protocol has been updated from:
- Surveyor nuclease assay of sgRNA
NOTE: The targeting efficiency of the sgRNA used for the knock-in experiment is evaluated by surveyor nuclease assay (also known as T7 endonuclease I (T7EI) assay)17. Select the sgRNA with high DNA cleavage efficiency and a low distance between the sgRNA cutting site and the stop codon.
to:
- T7 endonuclease assay of sgRNA
NOTE: The targeting efficiency of the sgRNA used for the knock-in experiment is evaluated by T7 endonuclease (T7EI) assay17. Select the sgRNA with high DNA cleavage efficiency and a low distance between the sgRNA cutting site and the stop codon.
Figure 1 in the Representative Results has been updated from:
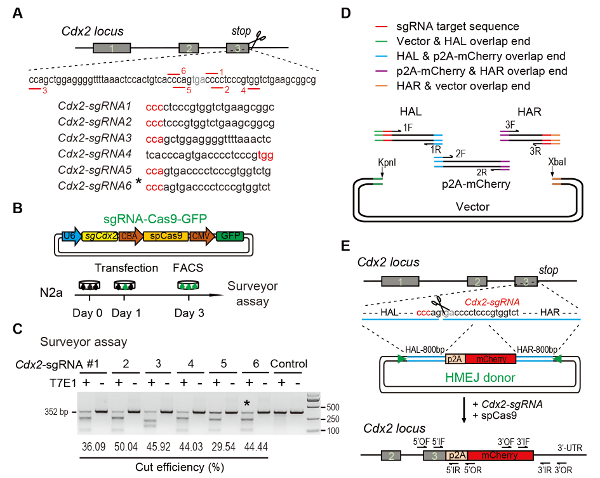
Figure 1: HMEJ-mediated targeted integration in vitro.
(A) Experimental scheme for selection of sgRNAs: Six different sgRNAs (Cdx2-sgRNA1~Cdx2-sgRNA6) around the stop codon of the Cdx2 locus with a higher rank and off-target potential were chosen based on online CRISPR design tool. The protospacer adjacent motif (PAM) sequence is in red. (B) Experimental design: The Cas9-CMV-GFP expression plasmids expressing sgRNA, Cas9, and GFP were introduced into N2a cells. GFP+ cells were sorted at day 3 for surveyor assay. (C) Surveyor assay for Cdx2 targeting: 6 different sgRNAs were designed for surveyor assay. Normal N2a cell genomic DNA serves as control. *, the sgRNA used for Cdx2-2A-mCherry knock-in experiment. (D) Schematic overview of construction of HMEJ donors using Gibson assembly. (E) Schematic overview of HMEJ-mediated gene targeting strategy at Cdx2 locus. HAL/HAR, left/right homology arm; triangles, sgRNA target sites; OF/OR, outer forward/reverse primer; IF/IR, inner forward/reverse primer. Figure modified from previous report10. Please click here to view a larger version of this figure.
to:
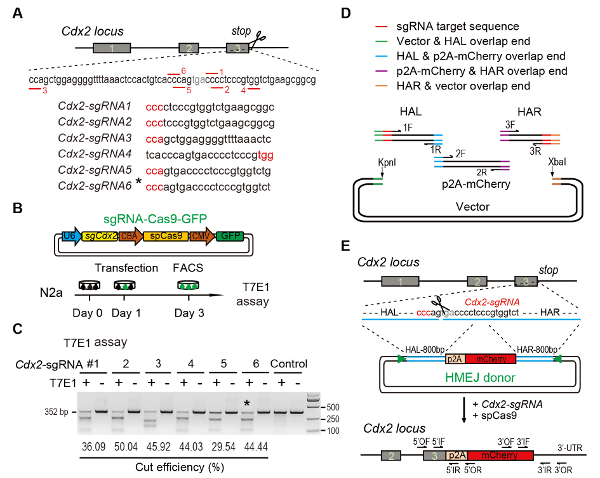
Figure 1: HMEJ-mediated targeted integration in vitro.
(A) Experimental scheme for selection of sgRNAs: Six different sgRNAs (Cdx2-sgRNA1~Cdx2-sgRNA6) around the stop codon of the Cdx2 locus with a higher rank and off-target potential were chosen based on online CRISPR design tool. The protospacer adjacent motif (PAM) sequence is in red. (B) Experimental design: The Cas9-CMV-GFP expression plasmids expressing sgRNA, Cas9, and GFP were introduced into N2a cells. GFP+ cells were sorted at day 3 for T7EI assay. (C) T7EI assay for Cdx2 targeting: 6 different sgRNAs were designed for T7EI assay. Normal N2a cell genomic DNA serves as control. *, the sgRNA used for Cdx2-2A-mCherry knock-in experiment. (D) Schematic overview of construction of HMEJ donors using Gibson assembly. (E) Schematic overview of HMEJ-mediated gene targeting strategy at Cdx2 locus. HAL/HAR, left/right homology arm; triangles, sgRNA target sites; OF/OR, outer forward/reverse primer; IF/IR, inner forward/reverse primer. Figure modified from previous report10. Please click here to view a larger version of this figure.



