ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
באופן קבוע באשכולות interspaced חלבון קצר palindromic חזרה/CRISPR הקשורים 9 (CRISPR/Cas9) מערכת מספקת כלי מבטיח עבור הנדסה גנטית, ופותחת לו את האפשרות של שילוב יישוב של transgenes. אנו מתארים קצה בתיווך הומולוגיה להצטרף (HMEJ)-המבוססת על אסטרטגיה עבור DNA יעיל ממוקד אינטגרציה ויוו , ממוקד ג'ין טיפולים באמצעות CRISPR/Cas9.
Abstract
כפלטפורמה מבטיח הגנום העריכה, מערכת CRISPR/Cas9 יש פוטנציאל גדול עבור הנדסה גנטית יעיל, במיוחד לשילוב יישוב של transgenes. עם זאת, בשל יעילות נמוכה רקומבינציה הומולוגית (HR) ואת מוטציות שונות ויאסין של קצה שאינם הומולוגיים להצטרף (NHEJ)-מבוסס אסטרטגיות-חלוקת תאים, ויוו הגנום עריכה נשאר אתגר גדול. כאן, אנו מתארים את קצה בתיווך הומולוגיה להצטרף (HMEJ)-CRISPR/Cas9 מערכת לשילוב יעיל ויוו מדויק יישוב מבוססת. במערכת זו, הגנום יישוב, התורם וקטור המכיל הומולוגיה זרועות (~ 800 bp) ולצדו היעד RNA (sgRNA) מדריך בודד רצפים הם ביקע מאת CRISPR/Cas9. אסטרטגיה זו מבוססת על HMEJ משיגה שילוב יעיל transgene העכבר מופרות, וכן hepatocytes ויוו. יתר על כן, אסטרטגיה מבוסס-HMEJ מציעה גישה יעילה עבור תיקון של fumarylacetoacetate ההידרולז (Fah) מוטציה ב- hepatocytes ואת מציל Fah-מחסור המושרה כשל בכבד עכברים. יחדיו, התמקדות ממוקד אינטגרציה, אסטרטגיה זו מבוססת-HMEJ מספק כלי מבטיח עבור מגוון של יישומים, כולל דור של מודלים מהונדס גנטית וטיפולים ג'ין יישוב.
Introduction
עריכה מדויקת, ממוקד הגנום נדרש לעיתים קרובות לייצור מודלים מהונדס גנטית וטיפולים רפואיים. הרבה מאמץ לפתח אסטרטגיות שונות עבור יעיל הגנום יישוב עריכה, כגון אבץ אצבע נוקלאז (ZFN), nucleases אפקטור כמו מפעיל שעתוק (TALENs), ומערכות CRISPR/Cas9. אסטרטגיות אלה ליצור מעברי כפול-גדיל DNA יישוב (DSB) הגנום, ולנצל מהותי מערכות תיקון ה-DNA, כגון1,רקומבינציה הומולוגית (HR)2, בתיווך microhomology סוף מצטרף (MMEJ)3 , 4 , 5ולאחר סיום שאינם הומולוגיים להצטרף (NHEJ)6,7,8 כדי לעודד אינטגרציה יישוב של1,transgenes9. האסטרטגיה מבוססת-HR נמצא כעת הכי נפוץ הגנום עריכה הגישה, אשר יעילה מאוד של שורות תאים, אבל לא נגיש ללא חלוקת תאים בשל התרחשותו מוגבלת בשלב S/G2 מאוחר. לפיכך, האסטרטגיה מבוססת-HR אינה ישימה עבור ויוו עריכה הגנום. לאחרונה, האסטרטגיה מבוססת NHEJ פותחה עבור ג'ין יעיל טוק ב- העכבר רקמות8. למרות זאת, השיטה מבוססת NHEJ מציג בדרך כלל indels בצמתים, ומקשה להפיק הגנום מדויק עריכה, במיוחד כאשר מנסים לבנות במסגרת פיוז'ן גנים8. שילוב יישוב מבוסס-MMEJ הוא מסוגל הגנום מדויק עריכה. עם זאת, רק בצניעות מגבירה את יעילות שילוב יישוב דוחות קודמים5. לכן, שיפור היעילות של אינטגרציה יישוב מדויק ויוו דרושה עבור יישומים טיפולית רחבה3.
בעבודה שפורסמה לאחרונה, הפגנו קצה בתיווך הומולוגיה להצטרף (HMEJ)-המבוססת על אסטרטגיה, אשר הראו את היעילות הגבוהה ביותר אינטגרציה יישוב כל דיווח אסטרטגיות בשני במבחנה , ויוו10. כאן, אנו מתארים את פרוטוקול להקמתה של מערכת HMEJ, הבנייה של הווקטורים RNA (sgRNA) יחיד-מדריך מיקוד הגן של עניין התורם המומנט מחסה אתרי היעד sgRNA ו ~ 800 bp הומולוגיה נשק (איור 1) . ב פרוטוקול זה, אנו גם מתארים את השלבים המפורטים עבור הדור של ה-DNA טוק-אין עכברים של צעדים קצרים לשילוב יישוב רקמות ויוו. יתר על כן, מחקר הוכחה הרעיון של האסטרטגיה מבוססת HMEJ הוכיח את יכולתו לתקן Fah מוטציה והצלה Fah- / - כבד כשל עכברים, אשר חשף עוד יותר את הפוטנציאל הטיפולי.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
כל ההליכים לרבות נושאים בעלי חיים אושרו על ידי ועדת האתיקה במחקר ביו-מכוני שנגחאי מדע ביולוגי (CA).
1. עיצוב של התורם פלסמידים
-
מבחר sgRNA
- להשתמש באינטרנט כלי עיצוב CRISPR כדי לחזות sgRNAs המטרה אזור11,12,13,14,15. על מיקומה Cdx2 , עיצוב 6 sgRNAs שונים (Cdx2-sgRNA1 ~Cdx2-sgRNA6) סביב התחנה codon עם פוטנציאל העליונים והתחתונים דרגה גבוהה יותר את המטרות (איור 1א')16.
- Linearize µg 2 Cas9-CMV-EGFP ביטוי מקומם על ידי sgRNA על ידי BbsI עיכול (1 µL של BbsI עבור 2 h ב 37 ° C-ריכוז סופי של U/µL 1 בנפח של 20 µL). ואז לטהר את המוצר על ידי ג'ל ערכת טיהור עם ג'ל agarose 1% 1 × טה למאגר.
- לערבב זוג sgRNA oligonucleotides ב µL 10 1 × T4 DNA ליגאז המאגר כדי ריכוז סופי של 50 מיקרומטר. דגירה הפתרון oligo באמצעות הדרגתי בטמפרטורה של 95 מעלות צלזיוס עד 25 ° C עם קצב שינוי טמפרטורה של 5 ° C/5 דקות (95 מעלות צלזיוס למשך 5 דקות ואז 90 º C למשך 5 דקות, 85 מעלות צלזיוס במשך 5 דקות, וכו '.), אשר anneal את oligos.
- לערבב 4 µL של annealed מוצר, µL 2 של וקטור ליניארית עם µL 1 של T4 DNA ליגאז ב µL 10 1 × T4 DNA ליגאז מאגר, ואז מאתרים ומפסיקים ב 22 מעלות צלזיוס במשך 1-2 h (איור 1B).
-
מודד נוקלאז וזמינותו של sgRNA
הערה: יעילות מיקוד sgRNA המשמש את הניסוי בטוק מחושבת על ידי מודד נוקלאז assay (הידוע גם בשם T7 endonuclease אני (T7EI) assay)17. בחר את sgRNA עם יעילות גבוהה של ביקוע DNA, מרחק נמוך בין האתר חיתוך sgRNA על stop codon.- Transfect Cas9-sgRNA-EGFP ביטוי וקטורים לתוך שורות תאים N2a בתרבית ב DMEM בתוספת 10% סרום שור עוברית, 1% פריז סן ז'רמן, ו- 1% שאינם חיוניים חומצת אמינו לפי תקנים קיט (ראה טבלה של חומרים). דגירה התאים transfected ב 37 ° C 5% CO2.
- לאחר 48 שעות של דגירה, לאסוף 5,000 transfected תאים (GFP+) על ידי תא לפעיל על-ידי קרינה פלואורסצנטית מיון (FACS) באמצעות תאים שאינם transfected כפקד.
- לעכל את התאים שנאספו במאגר 2-5 µL oflysis (0.1% X-100 טריטון, 0.1% Tween 20, ו- 100 µg/mL Proteinase K) ב 56 מעלות צלזיוס למשך 30 דקות ולאחר מכן חום בטל proteinase K ב 95 מעלות צלזיוס למשך 10 דקות.
- להגביר את המדגם PCR מקוננים (טבלה 1) באמצעות הפרוטוקול של היצרן. הגודל של מוצרי ה-PCR מוגדר כ 300-500 bp.
- לערבב 1 µL של פירוק מוצר עם ה-DNA פולימראז וזוג תחל החיצוני זיהוי הרצף סביב אתר היעד sgRNA (0.1 מיקרומטר, הריכוז הסופי) (טבלה 1), ולבצע את ה-PCR העיקרי באמצעי אחסון של 20 µL.
- להפעיל את ה-DNA פולימראז ב 95 מעלות צלזיוס למשך 5 דקות ולבצע PCR העיקרי של 30 מחזורים ב 95 ° C ל 30 s, 60 ° C ל 30 s, ו- 72 מעלות צלזיוס במשך 24 s (1דקות/1 kb), עם סיומת הסופי ב-72 מעלות במשך 5 דקות.
- לבצע את ה-PCR משני באמצעות µL 1 של מוצר ה-PCR ראשי וזוג תחל פנימי מקוננים.
- Denature, anneal מחדש ng 300-600 של מוצר ה-PCR מטוהרים µL 20 1 × T7EI התגובה מאגר (50 מ מ NaCl, 10 מ מ טריס-HCl, 10 מ מ MgCl2, 1 מ מ DTT pH 7.9) באמצעות הדרגה של טמפרטורה מ 95 ° C עד 25 ° C, בקצב של 5 ° C/5 דקות.
- להוסיף 1 µL של האנזים T7EI מוצרי ה-PCR annealed, תקציר ב 37 מעלות צלזיוס במשך שעתיים. לאחר מכן להפעיל את המוצר עיכול על 2% ג'ל agarose 1 × מאגר טה-120 V למשך 40 דקות עד השברים מופרדים (ראה טבלה של חומרים).
- השתמש ImageJ כדי לקבוע עוצמות הלהקה גזור והחובות נימולים. לחשב תדירות ויאסין בשיטות כאמור דיווח9 (איור 1C).
-
בניית תורמת וקטורית
הערה: כדי ליצור HMEJ התורם וקטורים עבור ג'ין Cdx2 , לבנות תורם דנ א (800 bp האל-p2A-mCherry-800 bp הר) תמר מוקף עם 23 nt Cdx2-sgRNAs מיקוד רצף בשני קצותיו ( איור 1Eואיור 1D ). . פאם רצף היעד היה סמוך לסוף הזרוע הומולוגיים. גיבסון הרכבה מומלצת HMEJ תורם, שיבוט.- להגביר את הזרוע השמאלית הומולוגיה 800 bp (HAL) עם פריימר לפנים-1F (המכיל 15-20 nt חפיפה הרצף של וקטור, 23 nt רצף מיקוד Cdx2-sgRNA, כ-20 nt הרצף של האל), להפוך פריימר-1R (המכיל רצף חפיפה bp 15-20 מ p2A-mCherry, כ-20 nt הרצף של האל)-מיקרומטר 0.1 הריכוז הסופי באמצעות העכבר הדנ א ב 200 ng/µL (איור 1ד, טבלה 1).
- להגביר את השבר ההכנסה p2A-mCherry עם פריימר לפנים-2F (מכיל 15-20 nt חפיפה הרצף של האל והרצף nt כ-20 מן ההכנסה פרגמנט) ולהפוך פריימר-2R (המכיל 15-20 nt חפיפה הרצף של הר והרצף nt כ-20 מ המקטע ההכנסה) ב 0.1 מיקרומטר הסופי הריכוז באמצעות דנ א גנומי או פלסמיד עם הכתב רצפים 100 ng/µL או 30 ng/µL (איור 1ד, טבלה 1).
- להגביר את הזרוע הומולוגיה נכון 800 bp (הר) עם פריימר לפנים-3F (המכיל 15-20 nt חפיפה הרצף של וקטור, 23 nt רצף מיקוד Cdx2-sgRNA, כ-20 nt הרצף של הר) הפוכה (המכיל 15-20 nt חפיפה הרצף של פריימר-3R p2A-mCherry, כ-20 nt הרצף של הר)-מיקרומטר 0.1 הריכוז הסופי באמצעות העכבר הדנ א ב 200 ng/µL (איור 1ד, טבלה 1).
- לרוץ כל המוצרים PCR על 1% agarose ג'ל במאגר טה × 1, לטהר את המוצרים ה-PCR של הגודל הצפוי על ידי ג'ל ערכת חילוץ, לפי הוראות היצרן (טבלה 1).
- לעכל 50-100 ננוגרם של הווקטור לבנות עם KpnI ו XbaI. לערבב 2 µL של וקטור ליניארית ב- 30-40 ng/µL עם שלושה PCR מוגבר קטעים (1 µL עבור כל, 100-200 ng/µL) 2 x מיקס גיבסון. הוסף H2O כדי לכוונן את עוצמת הקול הסופי כדי 10 µL.Incubate המיקס ב 50 מעלות צלזיוס למשך 60 דקות.
- שינוי צורה המוסמכת e. coli תאים עם כל מוצר שהורכב לחלץ פלסמיד בונה על-ידי ערכת חילוץ הדנ א על פי הוראות היצרן. לאמת את התורם HMEJ על ידי רצפי DNA.
2. הגנום עריכה בעוברי העכבר באמצעות השיטה מבוססת-HMEJ
-
הייצור של ה-mRNA Cas9
- הכנה Cas9 mRNA, להוסיף הרצף יזם T7 כדי Cas9 קידוד האזור על ידי הגברה PCR באמצעות צמד פריימר מתאים המפורטים בטבלה1. להוסיף את המפתח Cas9 F/R-ריכוז סופי של 0.1 מיקרומטר ו- 20 ng של Cas9 להביע וקטור דיוק גבוה × 1 DNA פולימראז מיקס. לכוון את עוצמת הקול הסופי כדי µL 50 עם H2O.
- להפעיל את ה-DNA פולימראז ב 95 מעלות צלזיוס למשך 5 דקות ולבצע PCR של מחזורים 36 ב 95 ° C ל 30 s, 60 ° C ל 30 s, ו- 68 מעלות צלזיוס למשך 4 דקות (1דקות/1 kb), עם סיומת הסופי ב 68 מעלות צלזיוס למשך 10 דקות.
- לטהר המוצר PCR T7-Cas9 עבור במבחנה שעתוק (IVT) ולאחר מכן לתמלל 0.5-1 µg DNA על-ידי ערכת שעתוק mRNA ב 37 מעלות צלזיוס במשך 8 שעות בהנפח הכולל של 20 µL, לפי הוראות היצרן (ראה טבלה של חומרים).
- להוסיף 1 µL של DNase לתערובת כדי להסיר את תבנית ה-DNA ב 37 מעלות צלזיוס במשך 15 דקות הוסף זנב poly-A למשך 45 דקות ב 37 מעלות צלזיוס ולשחזר את mRNA Cas9 על-ידי ערכת טיהור RNA, לפי הוראות היצרן (ראה טבלה של חומרים).
-
הפקה של sgRNA
- ליצור את תבנית sgRNA מונחה על ידי יזם T7 עם דיוק גבוה DNA פולימראז כמו לעיל. לבחור לגרדום sgRNA וקטורית כתבנית. צבעי יסוד בשימוש מפורטים בטבלה 1.
- לטהר את המוצר T7-sgRNA PCR ולהשתמש 0.5-1 µg DNA כתבנית עבור במבחנה שעתוק של sgRNA באמצעות ערכת שעתוק RNA קצר ב 37 מעלות צלזיוס במשך 6-אייץ ' ב הנפח הכולל של 20 µL, לפי הוראות היצרן (ראה טבלה של חומרים < / c11 >).
- להוסיף 1 µL של DNase לתערובת וממשיכים הדגירה ב 37 מעלות צלזיוס למשך 15 דקות להסיר את תבנית ה-DNA. לטהר את sgRNAs על-ידי ערכת טיהור RNA, כמו לעיל (ראה טבלה של חומרים).
- לדלל את sgRNA ל 500 ng/µL במים נטולי RNase ולאחסן את הדגימות-−80 ° C עד 3 חודשים.
הערה: CRISPR ribonucleoproteins (RNPs) הם ההחלפה אלטרנטיבית עם יותר חיתוך יעילות18,19,20.
-
העובר אוסף, microinjection ו- in vitro לקשרי תרבות
- Superovulate הנשי B6D2F1 (C57BL/6 × DBA2J) עכברים (בן 7-8 שבועות) על ידי סוסה הרה סרום גונדוטרופין (PMSG), ואחריו גונדוטרופין כוריוני אנושי (hCG) 48 שעות מאוחר יותר. אחרי הזריקה hCG, הבית נקבות עם זכרים B6D2F1 בין לילה.
- להקריב את הנשים על ידי CO2 הרדמה, 24 שעות לאחר הזרקת hCG. לאסוף את העוברים מופרית שלהם oviducts (עם 30-50 העוברים על כל נקבה) M2 בינוני.
- המקום העוברים מופרית (כ- 300 ביצים להזרקה יום אחד) לתוך KSOM בינוני (5.55 g/L NaCl, 0.19 g/L אשלגן כלורי, 0.05 g/L ח'2PO4, 0.05 g/L MgSO4•7H2O, 0.04 g/L גלוקוז, 1.12 g/L סודיום לקטט, 2.1 g/L NaHCO3 , 0.02 נקודות g/L נתרן פירובט, 0.25 g/L CaCl2•2H2O, EDTA 0.004 g/L, 0.146 g/L-גלוטמין ו 1 g/L שור אלבומין) ב 37 מעלות צלזיוס בתוך אינקובטור עם 5% CO2.
- מיקס Cas9 mRNA (100 ng/µL), sgRNA (50 ng/µL), HMEJ תורם וקטור (100 ng/µL), ולהוסיף H2O כדי לכוונן את עוצמת הקול הסופי µL 10. לשים את התערובת על קרח.
- משוך נימי מחטים (הקוטר החיצוני 1.0 מ מ, קוטר פנימי 0.78 מ מ עם נימה) באמצעות של פולר Micropipette (פרמטרים: חום, 74 משוך, 60; מהירות, 80; השהיה, 200 /; לחץ, 300. עיין בטבלה של חומרים). מחטים מסחרי תהיה ההחלפה חלופי עבור microinjection.
- להחדיר נפח סביר של התערובת לתוך הציטופלסמה של מופרות pronuclei מוגדרים היטב ב- droplet של HEPES-CZB בינוני המכיל µg/mL 5 cytochalasin B באמצעות microinjector של קבוע לזרום הגדרות (איור 2א) (ראה טבלה של חומרים)21.
הערה: כל קבוצה של מופרות צריך להיות מוזרק בתוך 20-30 דקות Cytochalasin B יכול להגביר את הכדאיות של העכבר זיגוטה לאחר ההזרקה. לחלופין, microinjection יכול להיות מופעל עם מערכת piezo, כפי שתואר לעיל22. - תרבות של מופרות מוזרק KSOM בינוני ב 37 מעלות צלזיוס מתחת 5% CO2 עד הבלסטוציסט שלב אחרי 3.5 ימים להשגחה קרינה פלואורסצנטית (דמויות 2B ו 2C).
-
העובר והעברת הדור של עכברים
- חבר estrous ICR עכברים נקבה עם עיקור עכברים זכרים ICR באותו יום כמו הזרקה.
- תרבות של מופרות מוזרק לתוך הבמה 2 תאים ב 37 ° C תחת 5 CO2, והעברת 25-30 2-תא עוברי לתוך oviducts של הנקבות ICR pseudopregnant ב coitum פוסט יום 0.5 (dpc). אמהות הנמענים לספק הגורים 19.5 dpc.
-
Genotyping העכבר
- תמצית דנ א גנומי העכבר מדגימות הבוהן או הזנב באמצעות ערכת חילוץ דנ א, לפי הוראות היצרן (ראה טבלה של חומרים).
- זיהוי הצומת 5' ו 3' טוק-in אירועים באמצעות 200-400 ננוגרם של דנ א גנומי נמדדת UV/מול מסות כתבנית כדי לבצע את amplification ה-PCR.
- להפעיל את ה-DNA פולימראז ב 95 מעלות צלזיוס למשך 5 דקות ולבצע PCR של 38 מחזורים ב 95 ° C ל 30 s, 60 ° C ל 30 s, ו- 72 מעלות צלזיוס במשך 1 דקה (1דקות/1 kb), עם סיומת הסופי ב-72 מעלות למשך 10 דקות. 5' לצומת, לשימוש של פריימר לפנים-הזרם של ה-HAL, עם אחד הפוכה על השבר טוק-ב (p2A-mCherry). לגבי 3' צומת, השתמש את פריימר לפנים על השבר טוק-ב (p2A-mCherry), עם אחד הפוכה על במורד הזרם של הר (טבלה 1).
- הפעל µL 6 של המוצר PCR על ג'ל agarose 1% 1 × טה מאגר ובדוק גודל המקטע הצפוי. אז לאמת אותם על ידי ה-DNA רצף (איור 2D).
3. HMEJ מבוסס In Vivo הגנום עריכה Hepatocytes
- הנח C57BL/6J הנמען העכבר (שבוע 8) התקן ריסון ולשים את הזנב דרך הסדק.
- מערבבים HMEJ התורם וקטורים (30 µg) ו- spCas9 ביטוי וקטורים (30 µg) ב 2 מ ל תמיסת מלח. עבור בקרת הניסוי להשעות HMEJ התורם וקטורים (30 µg) ב 2 מ ל תמיסת מלח (איור 3א).
- לנקות את זנב העכבר עם 70% אתנול. הוסף המחט לתוך הזנב הווריד ולהזריק את פתרון ה-DNA פלסמיד בתוך 5-7 ס הסר את המחט, שחרר את לחצן העכבר מהתקן ריסון.
- להקריב עכברים על ידי הרדמה2 CO לאחר 5-9 ימים לאחר ההזרקה. Perfuse את transcardially עכברים עם סליין 0.9%, ואחריה paraformaldehyde 4% באמצעות משאבה סחרור, את הכבד בן לילה ב 4 º C.
- מייבשים את הרקמה באמצעות 30% סוכרוז למשך הלילה, עד שהיא שוקעת לתחתית של התחתית.
- סעיף הרקמה קפוא-עובי של 10 מיקרומטר לדוגמאות בכבד.
- לשטוף את הסעיפים 3 פעמים ב 0.1 M באגירה פוספט (PB), דגירה אותם עם נוגדן ראשוני: ארנב אנטי-mCherry (מדולל ב- 5% המיתרים) לילה ב 4 º C.
- לשטוף את סעיפים 3 פעמים ב PB ולאחר מכן דגירה אותם עם נוגדנים משניים: Cy3-AffiniPure עז ארנב אנטי אג עבור 2 h בטמפרטורת החדר על תפקודי לב / נשימה.
- Counterstain הסעיפים עם דאפי כעשרים דקות ו הר עם גליצרין בשקופיות זכוכית להשגחה נוספת קרינה פלואורסצנטית (איור 3ב).
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
מבוססי HMEJ הגנום עריכה בהעכבר עוברי: כדי להגדיר את היעילות טוק-אין של השיטה מבוססת HMEJ העכבר מופרות, נמסר לנו Cas9 ה-mRNA, sgRNA לעבר הגן Cdx2 , התורם HMEJ לתוך עכבר מופרות, שנועד נתיך של הגן כתב p2A-mCherry codon האחרון של Cdx2 גנים (איור 2א). מופרות מוזרק התפתחה blastocysts בתרבות. כדי להעריך את היעילות בטוק, ניתחנו את זריחה mCherry עם מיקרוסקופ פלואורסצנטי, ומצאנו כי 12.9% של blastocysts קבלת HMEJ תורמים היו חיוביים עבור mCherry, אשר בא לידי בקפדנות trophectoderm (דמויות 2B 2 ג). על ידי קביעת רצף העכברים PCR חיובית, מצאנו גם כי כל בדק שילוב אירועים היו שילובי במסגרת מדויקת ובצמתים 5' והן 3' (איור 2D).
הגנום מבוססי HMEJ עריכה של רקמות בוגרות, HMEJ בתיווך ריפוי גנטי: לחקור אם עריכת מבוססי HMEJ הגנום יכול להיות מיושם ברקמות למבוגרים, הכנסנו את הקלטת mCherry ממש לפני stop codon של ג'ין Actb מאת transducing Actb- HMEJ בונה כדי כבדי העכבר C57/B6J על ידי וריד הזנב הזרקת hydrodynamic (איור 3א). אחרי 7 ימים של זריקות, מצאנו כי כמעט לחצי hepatocytes transfected הביעה mCherry כפי צבעונית על הסעיפים הכבד (איור 3ב).
לבחון את האפשרות של שימוש אסטרטגיית מבוססי HMEJ עבור טיפול גנטי, אנחנו מועסקים fumarylacetoacetate ההידרולז (Fah)-עכברים לקוי. Fah- / - העכבר הוא tyrosinemia תורשתי ומבוססת סוג דגם עכבר (ההטי), אשר בנמלים של ההכנסה שרסיס אקסון 5 של הגן Fah , גרימת מוטציות frameshift רצף הבאים23. כדי לשמור על עכברים- / - Fah, התייחסנו Fah- / - העכברים עם מעכב של הזרם של מסלול קטבולי טירוזין, (NTBC) 2-(2-nitro-4-trifluoromethylbenzoyl)-1,3-cyclohexanedione24. כאן יצאנו לראות אם תיקון הגנים MMEJ ו HMEJ-בתיווך יכול להציל את המוטציה Fah Fah- / - העכבר. הזרקנו hydrodynamically Cas9 לבנות יחד עם Fah- MMEJ או Fah- HMEJ בונה, שנועדו להוסיף Fah cDNA של אקסון 5 עד 14 לתוך אינטרון 4 של ג'ין Fah , Fah - / - עכבר (כבדים איור 3 C)-שבוע לאחר הזריקה, NTBC היה מסוגר כדי לגרום נזק לכבד (איור 3ג'). לאחר הנסיגה של NTBC, Fah-hepatocytes המתוקן של העכברים Fah- / - קבלת Fah- HMEJ וקבועים Cas9 הראה הפצה יעילה יותר מאשר בשיטה המבוססת MMEJ (איור 3D ).

איור 1 : בתיווך HMEJ ממוקד אינטגרציה במבחנה.
ערכת ניסיוני (A) עבור מבחר sgRNAs: 6 sgRNAs שונים (Cdx2-sgRNA1 ~Cdx2-sgRNA6) סביב stop codon של מיקומה Cdx2 עם גבוה דרגה, את-יעד פוטנציאלי היו נבחר בהתבסס על עיצוב CRISPR באינטרנט כלי. הרצף מוטיב סמוכים (פאם) protospacer הוא אדום. תכנון ניסויים (B): Cas9-CMV-GFP ביטוי פלסמידים לבטא sgRNA, Cas9 ו- GFP הוכנסו לתוך התאים N2a. GFP+ התאים היו מיון ביום 3 עבור מודד וזמינותו. (ג) מודד assay Cdx2 הכוונת: 6 sgRNAs שונות תוכננו עבור מודד וזמינותו. רגיל N2a תא הדנ א משמש בקרה. *, sgRNA משמש Cdx2-2A-mCherry טוק-אין ניסוי. (D) סקירה סכמטי של בניית HMEJ תורמים באמצעות הרכבה גיבסון. (E) סקירה סכמטי של HMEJ בתיווך גן מיקוד אסטרטגיה-לוקוס Cdx2 . זרוע הומולוגיה האל/הר, שמאלה/ימינה; משולשים, sgRNA אתרי היעד; של / OR, פריימר לפנים/הפוכה החיצוני; אם / IR, פריימר לפנים/הפוכה הפנימי. דמות שונה הדוח הקודם10. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 2 : הגנום עריכה בהעכבר העוברים דרך בתיווך HMEJ ממוקד אינטגרציה
(א) תכנית ניסויית של microinjection: תערובת של Cas9 mRNA (100 ng/µL), sgRNA (50 ng/µL), ושל התורמים פלסמידים (100 ng/µL) היו מוזרק העכבר מופרות. (B) זריחה נציג תמונות של העכבר עוברי נערך על ידי אסטרטגיה HMEJ. בר, 20 מיקרומטר. (ג) בטוק יעילות שצוין על-ידי אחוז mCherry+ blastocysts. מספר מעל כל עמודה, הכולל blastocysts נספרים. (ד) רצף ניתוח של עכברים נערך ג'ין-לוקוס Cdx2 . מוצרי ה-PCR מוגבר מאתרי צומת 5' ו 3' היו רציף. הומולוגיה זרוע; סגול, p2A; . רד, mCherry; הר או האל, זרוע הומולוגי ימינה או שמאלה. קווים מקווקווים מסמנים את האזור מושמט עבור בהירות. דמות שונה הדוח הקודם10. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 3 : בתיווך HMEJ ממוקד אינטגרציה ויוו.
(א) סקירה סכמטי של הזרקת וריד הזנב hydrodynamic. תערובת של פלסמידים לבטא התורם רצף, sgRNA, פלסמידים לבטא spCas9 נשלחו אל הכבד דרך הזרקה וריד הזנב hydrodynamic. (B) תמונות immunofluorescence הנציגה של hepatocytes. הסעיפים הכבד היו שנאספו 7 ימים שלאחר ההזרקה. סולם בר, 50 מיקרומטר. ה-GFP, transfected תאים. פלסמידים (C) או MMEJ או HMEJ-בתיווך גנים החלפת אסטרטגיה שנועדה להכניס אינטרון 4 לייעו Fah Fah cDNA של אקסון 5 עד 14 נשלחו לתוך Fah- / - כבדי העכבר באמצעות הזרקת hydrodynamic. NTBC ב: עכברים Fah- / - היו מתוחזקים על המים NTBC; NTBC הנחה: נסיגה של מים NTBC (היום הראשון של נסיגה NTBC מוגדר כ- 0, הוא יום היום ה-7 לאחר ההזרקה). (ד) Fah אימונוהיסטוכימיה מכתים סעיפים כבד מן Fah- / - עכברים מוזרק עם פלסמידים MMEJ או HMEJ. סולם בר, 100 מיקרומטר. דמות שונה מ דוחות קודמים5,10. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
השלבים הקריטיים ביותר בבנייה של פלסמידים התורם HMEJ הם: (1) מבחר sgRNA עם יעילות גבוהה של ביקוע DNA והמרחק נמוך בין אתר חיתוך sgRNA, stop codon ובניית (2) נכונה HMEJ התורם. CRISPR/Cas9-מתווכת המחשוף בנתיב התורם הן transgene (מכיל אתרי היעד sgRNA והזרועות הומולוגיה bp ~ 800) ואת הגנום יישוב נחוץ לשם שילוב יעיל ומדויק יישוב ויוו. השלבים הקריטיים ביותר של הדור של טוק-אין עכברים באמצעות השיטה מבוססת-HMEJ הם: (1) הכנת באיכות גבוהה של Cas9 mRNA ו- sgRNA (ניוון לא קיימת ב- Cas9 mRNA ו- sgRNA), (2) הכנת פלסמיד התורם HMEJ באיכות גבוהה. פלסמיד מראה ללא תופעות רעילות על התפתחות.
לאחרונה, שיטה המבוססת על NHEJ גם דווח כי עבור יעיל ויוו הגנום עריכה8. עם זאת, סוגים שונים של מוטציות ויאסין היו בדרך כלל המושרה בצמתים, כפי שמתואר הקודם דוחות8, ומקשה על מנת להשיג שילוב מדויק. כאן, האסטרטגיה מבוססת-HMEJ שאנו המתואר לעיל הראה שילוב יישוב מדויק עם מוטציות ויאסין בקושי יש. לפיכך, אסטרטגיית מבוסס-HMEJ יכול להיות לפלטפורמה אידיאלית החלפת רצף מוטציה (כגון מוטציה) עם הנכונה, אשר אינה ישימה עבור שיטה המבוססת NHEJ.
פסיפס היא בעיה מרכזית עבור ג'ין עריכה של העוברים. הזרקה של חלבון Cas9 במקום ה-mRNA בשלב העוברי מוקדם יותר עשויה להשיג transgene טוק-in בשלב תא אחד בלי פסיפס. עבור יישומים קליניים, משלוח של מערכות CRISPR/Cas9 לתוך רקמות בוגרות הוא עדיין מאתגר.
יש הרבה שימושים פוטנציאליים עתידיים של הגנום מבוססי HMEJ עריכה. זה יכול לשמש כדי ליצור מודלים מהונדס גנטית. בהתחשב שלה בטוק יעילות גבוהה בעוברי, שיטה זו יכול להפחית באופן משמעותי את מספר בעלי חיים לצורך יצירת מודלים מהונדס גנטית, במיוחד פותחת את האפשרות של יצירת דגמים גנטיים שאינם בני אדם הפרימטים. עריכת מבוססי HMEJ הגנום יכול סוגי תאים בודדים עקבות השושלת ברקמות למבוגרים, אשר שימושי במיוחד עבור מודלים של בעלי חיים, מאז יש חוסר של מודלים בבעלי חיים זמינים, כגון קופים. זה יכול לשמש יישוב ג'ין טיפולים: היישום האטרקטיביים ביותר של אסטרטגיית מבוססי HMEJ הוא טיפול גנטי עבור הקליניקה משתמשת. במחקר זה, אנחנו תיקן את המוטציה Fah מסוג tyrosinemia תורשתי אני עכברים באמצעות הזרקת hydrodynamic של הווקטורים המצוין. משלוח של מערכת CRISPR/Cas9 לתוך רקמות בוגרות זאת, עדיין האתגר הטכני העיקרי לשימוש קליני, כמו הזרקת hydrodynamic לא סביר שיש לבצע בחולים. כיום, נוסף של האסטרטגיה משלוח בדחיפות צורך בשיפור לפני שיטה זו מבוססת על HMEJ לתרגם המרפאה.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
המחברים אין לחשוף.
Acknowledgments
עבודה זו נתמכה על ידי רשויות אישורים אסטרטגי עדיפות תכנית המחקר (XDB02050007, XDA01010409), ה-Hightech R הלאומית & תוכנית D (תוכנית 863; 2015AA020307), קרן מדעי הטבע הלאומי של סין (NSFC מעניקה 31522037, 31500825, 31571509, 31522038), סין הנוער אלפי התוכנית כישורים (להיי), או באמצעות פרויקט של האקדמיה הסינית למדעים, שנחאי העיר ועדת המדע והטכנולוגיה פרויקט (16JC1420202 להיי), משרד המדע, הטכנולוגיה של סין (רוב; 2016YFA0100500).
Materials
| Name | Company | Catalog Number | Comments |
| pX330 | Addgene | 42230 | |
| pAAV vector | Addgene | 37083 | |
| pX260 | Addgene | 42229 | |
| AAV_Efs_hSpCas9_NLS_FLAG-SV40 | Addgene | 97307 | AAV vector for encoding a human codon-optimized SpCas9 driven by EFs promoter |
| AAV_Actb HMEJ donor_U6_sgRNA_EF1a_GFP_polyA | Addgene | 97308 | HMEJ donor for fusing a p2A-mCherry reporter to mouse Actb. EGFP driven by EF1a promoter and U6-driven sgRNAs targeting Actb. AAV backbone. |
| AAV_Cdx2 HMEJ donor | Addgene | 97319 | HMEJ donor for fusing a p2A-mCherry reporter to mouse Cdx2. |
| Lipofectamine 3000 Transfection Reagent | Life Technology | L3000015 | |
| Nuclease-Free Water | Life Technologies | AM9930 | |
| Bbs I | New England Biolabs | R0539S | |
| NEB Buffer 2 | New England Biolabs | B7002S | |
| T7 endonuclease I | New England Biolabs | M0302L | |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621L | |
| Plasmid EndoFree-Midi Kit | Qiagen | 12143 | |
| MMESSAGE MMACHINE T7 ULTRA | Life Technologies | AM1345 | |
| MEGACLEAR KIT 20 RXNS | Life Technologies | AM1908 | |
| MEGASHORTSCRIPT T7 KIT 25 RXNS | Life Technologies | AM1354 | |
| Flaming/Brown Micropipette Puller | Sutter Instrument | P-97 | Micropipette Puller (parameters: heat, 74; pull, 60; velocity, 80; time/delay, 200; pressure, 300) |
| Borosilicate glass | Sutter Instrument | B100-78-10 | type of capillaries (outer diameter 1.0 mm, inner diameter 0.78 mm with filament) |
| FemtoJet microinjector | Eppendorf | ||
| Freezing microtome | Leica | CM1950-Cryostat | thickness of 40 μm for brain, 10 μm for liver |
| Rabbit anti-mCherry | GeneTex | ||
| Cy3-AffiniPure Goat Anti-Rabbit IgG | Jackson Immunoresearch | ||
| DMEM | Gibco | 11965092 | |
| FBS | Gibco | 10099141 | |
| NEAA | Gibco | 11140050 | |
| Pen,Strep,Glutamine | Gibco | 10378016 | |
| Gel Extraction Kit | Omega | D2500-02 | |
| FACS | BD AriaII | ||
| PMSG | Ningbo Sansheng Medicine | S141004 | |
| HCG | Ningbo Sansheng Medicine | B141002 | |
| Cytochalasin B | Sigma | CAT#C6762 | |
| KSOM+AA with D-Glucose and Phenol Red | Millipore | CAT#MR-106-D | |
| M2 Medium with Phenol Red | Millipore | CAT#MR-015-D | |
| Mineral oil | Sigma |
References
- Yang, H., et al. Generation of Mice Carrying Reporter and Conditional Alleles by CRISPR/Cas-Mediated Genome Engineering. Cell. 154 (6), 1370-1379 (2013).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nature Biotechnology. 29 (8), 731-734 (2011).
- Nakade, S., et al. Microhomology-mediated end-joining-dependent integration of donor DNA in cells and animals using TALENs and CRISPR/Cas9. Nature Communications. 5, 5560 (2014).
- Hisano, Y., et al. Precise in-frame integration of exogenous DNA mediated by CRISPR/Cas9 system in zebrafish. Scientific reports. 5, 8841 (2015).
- Yao, X., et al. Cas9 - Mediated Precise Targeted Integration In Vivo Using a Double Cut Donor with Short Homology Arms. EBioMedicine. , (2017).
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome research. 24 (1), 142-153 (2014).
- Maresca, M., Lin, V. G., Guo, N., Yang, Y. Obligate ligation-gated recombination (ObLiGaRe): custom-designed nuclease-mediated targeted integration through nonhomologous end joining. Genome Research. 23 (3), 539-546 (2013).
- Suzuki, K., et al. In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration. Nature. 540 (7631), 144-149 (2016).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Yao, X., et al. Homology-mediated end joining-based targeted integration using CRISPR/Cas9. Cell Research. 27 (6), 801-814 (2017).
- Han, D. W., et al. Direct reprogramming of fibroblasts into epiblast stem cells. Nature Cell Biology. 13 (1), 66-71 (2011).
- Han, D. W., et al. Direct Reprogramming of Fibroblasts into Neural Stem Cells by Defined Factors. Cell Stem Cell. , (2012).
- Ambasudhan, R., et al. Direct reprogramming of adult human fibroblasts to functional neurons under defined conditions. Cell Stem Cell. 9 (2), 113-118 (2011).
- Sparman, M., et al. Epigenetic reprogramming by somatic cell nuclear transfer in primates. Stem Cells. 27 (6), 1255-1264 (2009).
- Schatten, G., Mitalipov, S. Developmental biology: Transgenic primate offspring. Nature. 459 (7246), 515-516 (2009).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Quadros, R. M., et al. Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome Biology. 18 (1), 92 (2017).
- Park, K. E., et al. Targeted Gene Knockin in Porcine Somatic Cells Using CRISPR/Cas Ribonucleoproteins. International journal of molecular sciences. 217 (6), (2016).
- Woo, J. W., et al. DNA-free genome editing in plants with preassembled CRISPR-Cas9 ribonucleoproteins. Nature biotechnology. 33 (11), 1162-1164 (2015).
- Harms, D. W., et al. Mouse Genome Editing Using the CRISPR/Cas System. Current protocols in human genetics. 83, 11-27 (2014).
- Yang, H., Wang, H., Jaenisch, R. Generating genetically modified mice using CRISPR/Cas-mediated genome engineering. Nature protocols. 9 (8), 1956-1968 (2014).
- Grompe, M., et al. Loss of Fumarylacetoacetate Hydrolase Is Responsible for the Neonatal Hepatic-Dysfunction Phenotype of Lethal Albino Mice. Genes & development. 7 (12), 2298-2307 (1993).
- Paulk, N. K., et al. Adeno-associated virus gene repair corrects a mouse model of hereditary tyrosinemia in vivo. Hepatology. 51 (4), 1200-1208 (2010).
Tags
גנטיקה גיליון 133 CRISPR/Cas9 אינטגרציה יישוב בתיווך הומולוגיה סוף שהצטרף In Vivo העובר מהונדסים גנטית עכברים הזרקה HydrodynamicErratum
Formal Correction: Erratum: CRISPR/Cas9-mediated Targeted Integration In Vivo Using a Homology-mediated End Joining-based Strategy
Posted by JoVE Editors on 03/10/2021.
Citeable Link.
An erratum was issued for: Studying TGF-β Signaling and TGF-β-induced Epithelial-to-mesenchymal Transition in Breast Cancer and Normal Cells. The phrases "surveyor assay" and "Surveyor Nuclease" have been updated to "T7E1 assay" to " T7 endonuclease I" respectively.
Step 1.2 in the Protocol has been updated from:
- Surveyor nuclease assay of sgRNA
NOTE: The targeting efficiency of the sgRNA used for the knock-in experiment is evaluated by surveyor nuclease assay (also known as T7 endonuclease I (T7EI) assay)17. Select the sgRNA with high DNA cleavage efficiency and a low distance between the sgRNA cutting site and the stop codon.
to:
- T7 endonuclease assay of sgRNA
NOTE: The targeting efficiency of the sgRNA used for the knock-in experiment is evaluated by T7 endonuclease (T7EI) assay17. Select the sgRNA with high DNA cleavage efficiency and a low distance between the sgRNA cutting site and the stop codon.
Figure 1 in the Representative Results has been updated from:
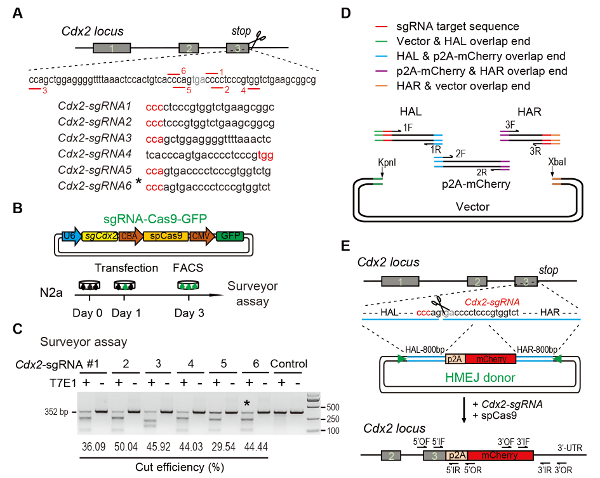
Figure 1: HMEJ-mediated targeted integration in vitro.
(A) Experimental scheme for selection of sgRNAs: Six different sgRNAs (Cdx2-sgRNA1~Cdx2-sgRNA6) around the stop codon of the Cdx2 locus with a higher rank and off-target potential were chosen based on online CRISPR design tool. The protospacer adjacent motif (PAM) sequence is in red. (B) Experimental design: The Cas9-CMV-GFP expression plasmids expressing sgRNA, Cas9, and GFP were introduced into N2a cells. GFP+ cells were sorted at day 3 for surveyor assay. (C) Surveyor assay for Cdx2 targeting: 6 different sgRNAs were designed for surveyor assay. Normal N2a cell genomic DNA serves as control. *, the sgRNA used for Cdx2-2A-mCherry knock-in experiment. (D) Schematic overview of construction of HMEJ donors using Gibson assembly. (E) Schematic overview of HMEJ-mediated gene targeting strategy at Cdx2 locus. HAL/HAR, left/right homology arm; triangles, sgRNA target sites; OF/OR, outer forward/reverse primer; IF/IR, inner forward/reverse primer. Figure modified from previous report10. Please click here to view a larger version of this figure.
to:
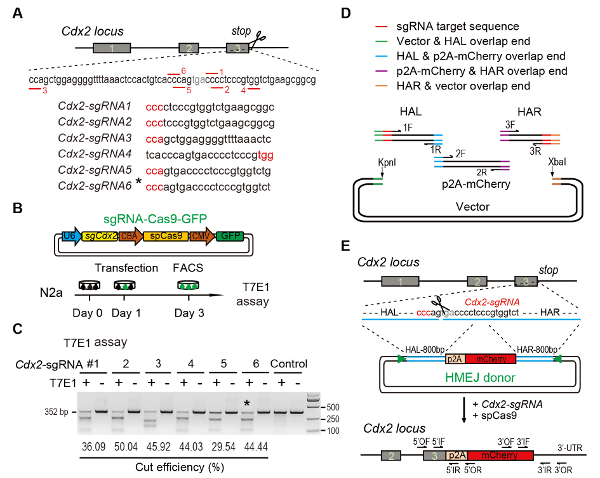
Figure 1: HMEJ-mediated targeted integration in vitro.
(A) Experimental scheme for selection of sgRNAs: Six different sgRNAs (Cdx2-sgRNA1~Cdx2-sgRNA6) around the stop codon of the Cdx2 locus with a higher rank and off-target potential were chosen based on online CRISPR design tool. The protospacer adjacent motif (PAM) sequence is in red. (B) Experimental design: The Cas9-CMV-GFP expression plasmids expressing sgRNA, Cas9, and GFP were introduced into N2a cells. GFP+ cells were sorted at day 3 for T7EI assay. (C) T7EI assay for Cdx2 targeting: 6 different sgRNAs were designed for T7EI assay. Normal N2a cell genomic DNA serves as control. *, the sgRNA used for Cdx2-2A-mCherry knock-in experiment. (D) Schematic overview of construction of HMEJ donors using Gibson assembly. (E) Schematic overview of HMEJ-mediated gene targeting strategy at Cdx2 locus. HAL/HAR, left/right homology arm; triangles, sgRNA target sites; OF/OR, outer forward/reverse primer; IF/IR, inner forward/reverse primer. Figure modified from previous report10. Please click here to view a larger version of this figure.



