ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Kümelenmiş düzenli olarak kısa palindromik tekrarlar/ilişkili CRISPR protein 9 (CRISPR/Cas9) interspaced sistem genetik mühendisliği için umut verici bir araçtır ve hedeflenen entegrasyonu transgenes olasılığı kadar açılır. Biz (HMEJ) katılmadan homoloji-aracılı son tarif-verimli DNA Tümleştirme vivo içinde hedef ve gen tedavileri CRISPR/Cas9 kullanarak hedef için strateji tabanlı.
Abstract
Bir umut verici genom düzenleme platformu olarak, CRISPR/Cas9 sistem transgenes hedeflenen entegrasyonu için özellikle verimli genetik manipülasyon için büyük potansiyele sahiptir. Ancak, homolog rekombinasyon (HR) düşük verimliliğini ve çeşitli Indel mutasyonlar (NHEJ) katılmadan homolog olmayan sonu nedeniyle-sigara bölen hücreleri, in vivo genom kalır düzenleme stratejileri büyük bir meydan okuma dayalı. Burada, biz (HMEJ) katılmadan homoloji-aracılı son tarif-esaslı CRISPR/Cas9 sistem verimli vivo içinde kesin hedeflenen tümleştirme için. Bu sistemde, vektör hedeflenen genom ve donör içeren homoloji silah (~ 800 bp) dizileri CRISPR/Cas9 tarafından i ciddi tek Kılavuzu RNA (sgRNA) hedef tarafından çevrili. Bu HMEJ tabanlı strateji verimli transgene Tümleştirme fare zigotları yanı sıra tetkikine vivo içindeelde eder. Ayrıca, bir HMEJ tabanlı strateji fumarylacetoacetate hidrolaz (Fah) mutasyon tetkikine düzeltilmesi için verimli bir yaklaşım sunuyor ve Fahkurtarır-eksikliği bağlı Karaciğer yetmezliği fareler. Birlikte ele alındığında, odaklanan entegrasyonu hedefleyen, bu HMEJ tabanlı strateji uygulamaları, üretimi hedeflenen gen terapileri ve genetiği değiştirilmiş hayvan modelleri de dahil olmak üzere çeşitli için umut verici bir araç sağlar.
Introduction
Kesin, hedeflenen genom düzenleme kez klinik tedavi ve genetiği değiştirilmiş hayvan modelleri üretmek için gereklidir. Düzenleme, çinko parmak nükleaz (ZFN), gibi transkripsiyon harekete geçirmek gibi efektör enzimler (TALENs), verimli hedeflenen genom için çeşitli stratejiler geliştirmek için çok çaba yapılmış ve CRISPR/Cas9 sistemleri. Bu stratejiler hedeflenen DNA çift iplikli kırılmalara (DSB) genom oluşturmak ve homolog rekombinasyon (HR)1,2, microhomology-aracılı sonunda (MMEJ)3 katılma gibi içsel DNA onarım sistemleri yararlanmak , 4 , 5ve homolog bitiş transgenes1,9hedeflenen Tümleştirme ikna etmek için (NHEJ)6,7,8 katılmadan. İK tabanlı strateji şu anda en sık kullanılan düzenleme yaklaşımı, hücre hatlarında çok etkili, ama geç S/G2 aşamasında kısıtlı onun oluşumu nedeniyle olmayan bölme hücrelere kolayca erişilebilir değil genom olduğunu. Böylece, insan kaynakları tabanlı strateji genom vivo içinde düzenleme için geçerli değildir. Son zamanlarda, NHEJ tabanlı strateji verimli gen knock bileşeninde fare doku8için geliştirilmiştir. Yine de, NHEJ tabanlı yöntem genellikle hassas genom düzenleme, özellikle çerçeve füzyon genlerin8oluşturmak çalışırken oluşturmak üzere kavşaklar, indels sunar. Hedeflenen bütünleşmesidir kesin genom düzenleyebilen MMEJ tabanlı. Ancak, sadece mütevazi önceki raporlar5hedeflenen entegrasyon verimliliği artırır. Bu nedenle, kesin hedeflenen entegrasyon içinde vivo verimliliğini artırmak acilen geniş tedavi edici uygulamaları3için gereklidir.
Kısa bir süre önce bir eser bulunmaktadır (HMEJ) katılmadan homoloji-aracılı sonunda gösterdi-strateji, tüm bildirilen stratejileri vitro ve in vivo10içinde en yüksek hedefli entegrasyon verimliliği gösterdi dayalı. Burada, biz HMEJ sisteminin kurulması için bir iletişim kuralı tanımlamak ve aynı zamanda faiz ve donör gen hedefleme tek-Kılavuzu RNA (sgRNA) vektörel çizimler inşaat vektörler barındıran sgRNA hedef siteleri ve ~ 800 bp homoloji silah (şekil 1) . Bu protokol için biz de ayrıntılı adımlar için hedeflenen doku içinde vivoWuppertal'de DNA çakma fareler ve kısa adımlar oluşturmada açıklar. Ayrıca, HMEJ tabanlı strateji bir kanıtı-of-concept çalışma Fah mutasyon düzeltmek ve daha fazla terapötik potansiyelini ortaya Fah- / - Karaciğer yetmezliği fare, kurtarmak için onun yetenek gösterdi.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Hayvan konular da dahil olmak üzere tüm yordamları Biyomedikal Araştırma Etik Komitesi, Shanghai Enstitüleri tarafından biyolojik bilim (CA) için onaylanmıştır.
1. donör plazmid tasarımını
-
SgRNA seçimi
- Online CRISPR tasarım araçları hedef bölge11,12,13,14,15sgRNAs tahmin etmek için kullanın. Cdx2 odağı için altı farklı sgRNAs tasarım (Cdx2-sgRNA1 ~Cdx2-sgRNA6) stop kodonu ile daha yüksek rütbe ve daha düşük potansiyel kapalı-(şekil 1A)16hedefler.
- Cas9-CMV-EGFP ifade vektörel çizimler ve sgRNA 2 µg BbsI sindirim (20 µL hacmi 1 U/µL son bir konsantrasyon, 37 ° C'de 2 h için BbsI, 1 µL) tarafından linearize. Sonra ürün jel arıtma kiti ile % 1'özel jel 1 × TAE arabelleği tarafından arındırmak.
- Mix 1 × T4 DNA ligaz arabelleği 50 µM. son bir konsantrasyon için 10 µL sgRNA oligonucleotides bir çift kuluçkaya bir sıcaklık değişim oranı, 5 ° C/5 dk (95 ° C 5 min için ile 95 ° c ile 25 ° C arasında bir sıcaklık gradyanı kullanarak oligo çözüm o zaman 90 ° C 5 dk, 5 dk, vbiçin 85 ° C.), hangi oligos TAV.
- Mix 4 µL olan ürün, doğrusallaştırılmış vektör 2 µL 1 × T4 DNA ligaz arabelleği, 10 µL T4 DNA ligaz 1 µL ile komplementer ve 22 ° c için 1-2 h (şekil 1B) ligate.
-
Arazi nükleaz tahlil sgRNA
Not: Çakma deneme için kullanılan sgRNA hedefleme verimliliğini surveyor nükleaz tahlil tarafından değerlendirilir (diğer bir de ben (T7EI) T7 endonükleaz tahlil)17. Yüksek DNA bölünme verimlilik ve sgRNA kesme sitesi ve kodonu arasında düşük bir mesafe ile sgRNA seçin.- Cas9-sgRNA-EGFP ifade vektörel çizimler N2a hücre satır içine transfect %1 PSG ve % 1 gerekli olmayan amino asit transfection tarafından DMEM % 10 fetal sığır serum ile desteklenmiş olarak kültürlü, kit ( Tablo malzemelerigörmek). Transfected hücreleri % 5 CO237 ° C'de kuluçkaya.
- 5.000 transfected hücreleri (GFP+) floresan aktif hücre (FACS) sıralama, kuluçka 48 saat sonra toplamak olmayan transfected hücreler bir denetim olarak kullanma.
- 2-5 µL oflysis tampon (%0.1 Triton X-100, % 0,1 ara 20 ve 100 µg/mL İndinavir K) 30 dk 56 ° C'de kendine hakim hücre sindirmek ve ısı İndinavir K 95 ° c 10 dk sonra devre dışı.
- Üreticinin iletişim kuralını kullanarak iç içe geçmiş PCR (Tablo 1) tarafından örnek yükseltmek. PCR ürünlerinin boyutu 300-500 bp için ayarlanır.
- Mix 1 µL DNA polimeraz ve dış astar sgRNA hedef site (0,1 µM, son konsantrasyonu) çevresinde sıra tanıma bir çift ile lizis ürünün (Tablo 1) ve birincil PCR 20 µL hacmindeki gerçekleştirin.
- DNA polimeraz 95 ° c 5 min için etkinleştirmek ve 30 95 ° C'de 30 döngüleri için birincil PCR gerçekleştirmek s, 30 60 ° C s ve 72 ° C 24 s (1 dk./1 kb), 5 min için 72 ° C'de son uzantılı.
- Birincil PCR ürününün 1 µL ve iç içe geçmiş iç astar bir çift kullanarak ikincil PCR gerçekleştirin.
- Denatüre ve 300-600 ng 1 × T7EI reaksiyon arabelleği (50 mM NaCl, 10 mM Tris-HCl, 10 mM MgCl2, 1 mM DTT pH 7,9) 20 µL arıtılmış PCR ürününün yeniden tavlama sıcaklığı 95 ° c ila 25 ° C bir degrade, 5 ° C/5 dk bir oranla kullanarak.
- Tavlanmış PCR ürünleri ve sindirmek için 2 h 37 ° C'de T7EI enzim 1 µL ekleyin. Sonra parçaları ( Tablo malzemelerigörmek) ayrılır kadar sindirim ürün % 2 özel jel 1 × TAE arabelleği 120 V 40 min için çalıştırın.
- ImageJ grup yoğunluklarda kesim ve saf DNA belirlemek için kullanın. Daha önce yöntemlerle Indel frekans9 (şekil 1C) bildirilen hesaplayın.
-
Donör vektör inşaatı
Not: Cdx2 gen HMEJ donör vektörel çizimler oluşturmak için bir donör DNA oluşturmak (800 bp HAL-p2A-mCherry-800 bp HAR) 23 ile çevrili nt Cdx2-sgRNAs hedef sıra iki ucunda (şekil 1D ve şekil 1E). Hedef sıra PAM homolog kol ucuna bitişik. Gibson derleme klonlama HMEJ donör için önerilir.- 800 bp sol homoloji kol (HAL) ile ileri astar-1F yükseltmek (15-20 nt örtüşme vektör, 23 serisinden içeren nt Cdx2-sgRNA hedefleme sırası ve yaklaşık 20 nt serisinden HAL) ve ters astar-1R (15-20 bp örtüşme serisinden içeren p2A-mCherry ve yaklaşık 20 nt serisinden HAL) 0,1 µM son konsantrasyonu 200 ng/µL (şekil 1D, Tablo 1) fare genomik DNA'yı kullanarak.
- P2A-mCherry ekleme parçası ile ileri astar-2F (15-20 nt örtüşme serisinden HAL ve yaklaşık 20 nt serisinden ekleme parçası içeren) yükseltmek ve astar-2R (15-20 nt örtüşme serisinden HAR ve yaklaşık 20 nt serisinden içeren ters ekleme parçası) 0,1 µM son konsantrasyonu genomik DNA veya plazmid muhabir serileri 100 ng/µL veya 30 ng/µL (şekil 1D, Tablo 1) ile kullanarak.
- 800 bp doğru homoloji kol (HAR) ile ileri astar 3F yükseltmek (15-20 nt örtüşme vektör, 23 serisinden içeren nt Cdx2-sgRNA hedefleme sırası ve HAR yaklaşık 20 nt serisinden) ve ters astar-3R (15-20 nt örtüşme serisinden içeren p2A-mCherry ve HAR yaklaşık 20 nt serisinden) 0,1 µM son konsantrasyonu 200 ng/µL (şekil 1D, Tablo 1) fare genomik DNA'yı kullanarak.
- PCR ürünlerinin % 1'özel jel 1 × TAE arabelleği çalıştırın ve beklenen boyutu PCR ürünlerinin jel ekstraksiyon kiti tarafından üreticinin yönergelerine göre (Tablo 1) arındırmak.
- 50-100 ng KpnI ve XbaI ile bir yapı vektörünün sindirmek. Mix 2 µL 30-40 ng/µL üç PCR ile de doğrusallaştırılmış Yöneyin Gibson mix x 2 parçaları (1 µL için her, 100-200 ng/µL) güçlendirilmiş. Son 10 µL.Incubate 50 ° c 60 dk için mix için ayarlamak için H2O ekleyin.
- Yetkili E. coli dönüşümü hücreleri monte ürün ve özü plazmid DNA ekstraksiyon kiti üreticinin yönergelerine uygun olarak oluşturur. HMEJ donör tarafından DNA sıralama doğrulayın.
2. genom HMEJ tabanlı yöntemi kullanma fare embriyo düzenleme
-
Üretim Cas9 mRNA
- Cas9 mRNA hazırlık için Tablo 1' de listelenen uygun astar çiftini kullanarak PCR güçlendirme tarafından bölge desteği T7 organizatörü sıra Cas9 için ekleyin. Astar Cas9 F/R 0,1 µM son bir konsantrasyon ve 20 eklemek Cas9, ng vektör 1 × yüksek sadakat DNA polimeraz karışımı için ifade. H2o 50 µL için son ses seviyesini
- DNA polimeraz 95 ° c 5 min için etkinleştirmek ve PCR 36 devredir 30 95 ° c gerçekleştirmek s, 30 60 ° C s ve 68 ° C 4 dakika (1 dk./1 kb), 10 dk 68 ° C'de son uzantılı.
- T7-Cas9 PCR ürünü vitro transkripsiyon (IVT) için arındırmak ve 0.5-1 uyarlamak µg DNA mRNA transkripsiyon kiti için üreticinin yönergelerine göre 20 µL toplam hacmi 8 h 37 ° C'de tarafından (bkz. Tablo malzeme).
- Dnaz 1 µL 15 dakika Ekle Poli-A kuyruk 37 ° C'de 45 dakika süreyle 37 ° C'de DNA şablonunu kaldırmak ve Cas9 mRNA RNA arıtma paketi tarafından üreticinin yönergelerine göre kurtarmak için karışıma ekleyin ( Tablo malzemelerigörmek).
-
SgRNA imalatı
- T7 organizatörü yüksek sadakat DNA polimeraz ile tahrik sgRNA şablonu oluşturun. Vektör şablon olarak içeren bir sgRNA iskele seçin. Kullanılan astar Tablo 1' de listelenmiştir.
- T7-sgRNA PCR ürünü arındırmak ve 0.5-1 kullanmak µg DNA için üreticinin yönergelerine göre 20 µL toplam hacmi 6 h 37 ° C'de bir kısa RNA transkripsiyonu kit kullanarak sgRNA, vitro transkripsiyonu için şablon olarak ( malzemeler tablo bkz < / c11 >).
- Dnaz 1 µL karışıma ekleyin ve kuluçka DNA şablonunu kaldırmak 15 dakika 37 ° C'de devam edin. SgRNAs RNA arıtma paketi tarafından arındırmak (bakınız Tablo reçetesi) Yukarıdaki gibi.
- SgRNA 500 ng/µL RNase free su için sulandırmak ve örnekleri −80 ° C de 3 aya kadar saklayın.
Not: Alternatif bir ikame daha iyi kesme verimliliği18,19,20CRISPR ribonucleoproteins (RNPs) vardır.
-
Embriyo koleksiyonu, mikroenjeksiyon ve vitro kültürü
- 48 saat sonra insan Korionik gonadotropin (hCG) tarafından takip kadın B6D2F1 (C57BL/6 × DBA2J) fareler (7-8 haftalık) tarafından hamile kısrak serum gonadotropin (PMSG), superovulate. Sonra hCG enjeksiyonu, B6D2F1 erkek ev kadınları gecede.
- Dişiler tarafından CO2 anestezi, 24 h hCG enjeksiyonu sonra kurban. Döllenmiş embriyo (30-50 embriyolar her kadın için ile) onların oviducts M2 orta toplamak.
- Yer KSOM orta içine döllenmiş embriyo (yaklaşık 300 yumurta için bir günlük enjeksiyon) (5,55 g/M NaCl, 0,19 g/L KCl, 0,05 g/L KH2PO4, 0,05 g/L MgSO4•7H2O, 0,04 g/M glikoz, 1.12 g/L sodyum laktat, 2.1 g/L NaHCO3 , 0,02 g/L sodyum pyruvate, 0, 25 g/L CaCl2•2H2O, 0,004 g/M EDTA, 0,146 g/L L-glutamin ve 1 g/L sığır serum albümin) bir kuluçka %5 CO237 ° C'de.
- Mix Cas9 mRNA (100 ng/µL), sgRNA (50 ng/µL) ve HMEJ donör vektör (100 ng/µL) ve için 10 µL. son ses seviyesini ayarlamak için H2O koymak karışımı buz ekleyin.
- Çek kapiller iğneler (dış çap 1.0 mm, iç çapı ile filaman 0,78 mm) bir Micropipette çektirmenin kullanarak (Parametreler: ısı, 74 çekme, 60; hız, 80; / gecikme, 200; basınç, 300. Malzemelerin tabloyabakın). Ticari iğneler mikroenjeksiyon için alternatif bir ikame olurdu.
- Karışımı olası bir birim zigotları sitoplazma HEPES-CZB orta 5 µg/mL cytochalasin bir microinjector sabiti ile kullanarak B içeren bir damlacık içinde iyi tanımlanmış pronuclei ile enjekte akış ayarları (Şekil 2A) (bkz: Tablo malzemelerin)21.
Not: Her grup zigotları enjekte edilebilir 20-30 dk. içinde Cytochalasin B enjeksiyon sonra fare zigot canlılık artırabilir. Alternatif olarak, mikroenjeksiyon22daha önce açıklandığı gibi piezo sistemi ile çalıştırılabilir. - KSOM orta altında %5 CO2 blastosist kadar sahne Floresans gözlem (2B rakamlar ve 2C) için 3,5 gün sonra 37 ° C'de enjekte zigotları kültür.
-
Embriyo transferi ve fareler nesil
- Estrous ICR dişi fareler vasectomized ICR erkek fareler ile enjeksiyon olarak aynı gün dostum.
- 37 ° C altında %5 CO2ve transfer 25-30 2 hücreli embriyo içine oviducts pseudopregnant ICR kadın 0.5 gün sonrası coitum (dpc), 2-hücre aşamada içine enjekte zigotları kültür. Alıcı anneler yavrularını 19.5 dpc teslim.
-
Fare Genotipleme
- Fare genomik DNA bir DNA ekstraksiyon kiti kullanarak ayak veya kuyruk örnekleri üreticinin yönergelerine göre ayıklamak ( Tablo malzemelerigörmek).
- PCR amplification gerçekleştirmek için bir şablon olarak UV/vis spektrometresi tarafından ölçülen genomik DNA'ın 200-400 ng kullanarak çakma olayların 5' ve 3' kavşak tanımlayın.
- DNA polimeraz 95 ° c 5 min için etkinleştirmek ve PCR 38 devredir 30 95 ° c gerçekleştirmek s, 30 60 ° C s ve 72 ° C için 1 dk (1 dk./1 kb), 10 dk 72 ° C'de son uzantılı. 5' birleşme, için ileri astar, HAL, çakma parçası (p2A-mCherry) üzerinde geriye doğru bir akış yukarı kullanın. 3' olarak Kavşağı, çakma parçası (p2A-mCherry), ileri astar ters bir HAR (Tablo 1) ile aşağı kullanmak.
- 6 µL PCR ürününün % 1'özel jel 1 × TAE arabellek ve onay beklenen parça boyutu için çalıştırın. Daha sonra onları (Şekil 2D) sıralama DNA tarafından doğrulayın.
3. HMEJ-Based In Vivo genom tetkikine içinde düzenleme
- Bir yasaklama cihazda yer alıcı C57BL/6J fare (8 hafta) ve kuyruk slit koymak.
- HMEJ donör vektörel çizimler (30 µg) ve spCas9 ifade vektörler (30 µg) 2 mL tuzlu su çözeltisi içinde karıştırın. Denetim deney için HMEJ donör vektörel çizimler (30 µg) 2 mL serum fizyolojik çözüm (şekil 3A) askıya alma.
- Fare kuyruğu % 70 etanol ile temizleyin. Kuyruk içine iğne damar ve 5-7 s. plazmid DNA çözümünüzde enjekte Ekle iğneyi çıkarın ve yasaklama aygıttan fare düğmesini serbest bırakın.
- Fareler CO2 anestezi tarafından enjeksiyon sonra 5-9 gün sonra kurban. Fareler transcardially % 0,9 serum fizyolojik ile sıvı, Peristaltik pompa kullanarak % 4 paraformaldehyde tarafından takip ve karaciğer gecede 4 ° C'de düzeltmek
- Tüp altına batmadan kadar % 30 Sükroz gecede, kullanarak doku kurutmak.
- Donmuş doku karaciğer örnekleri için 10 µm kalınlığında, bölüm.
- Sonraki bölümlerde fosfat tamponlu 0.1 M (PB) üç kez durulama ve onları ile birincil antikor kuluçkaya: tavşan anti-mCherry (% 5'seyreltilmiş NGS) gecede 4 ° C'de
- Bölümlerde üç kez PB yıkama ve sonra onları ile ikincil antikor kuluçkaya: Cy3-AffiniPure keçi Anti-tavşan IgG orbital Çalkalayıcı üzerinde oda sıcaklığında 2 h için.
- 20 dk için DAPI bölümlerle ve montajı ile gliserin cam slaytlara daha da floresan gözlem (şekil 3B) için counterstain.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Fare embriyo genom HMEJ tabanlı düzenleme: HMEJ tabanlı yöntemi çakma verimliliğini fare zigotları tanımlamak için biz Cas9 mRNA, p2A-mCherry muhabir gene Cdx2 son kodonu ile sigorta için tasarlanmı Cdx2 gen ve HMEJ donör fare zigotları hedefleme sgRNA teslim gen (Şekil 2A). Blastokistlerin kültür içine enjekte zigotları geliştirdi. Çakma etkinliğini değerlendirmek için mCherry floresan floresan mikroskop ile analiz ettik ve biz HMEJ bağış almaya blastokistlerin % 12.9 kesinlikle trophectoderm içinde ifade edildi mCherry için pozitif bulunmuştur (rakamlar 2B 2 C). PCR olumlu fareler sıralama tarafından tüm hassas çerçeve entegrasyonlar 5' ve hem 3' kavşak (Şekil 2D) edildi Tümleştirme olaylar incelendiğinde de bulduk.
Yetişkin doku ve HMEJ-aracılı gen terapisi genom HMEJ tabanlı düzenleme: Genom HMEJ tabanlı düzenleme yetişkin dokularda uygulanabilir olup olmadığını araştırmak için biz hemen önce kodonu Actb gen mCherry kaset kuyruk-ven tarafından Actb- HMEJ yapıları C57/B6J fare karaciğer için transducing tarafından eklenen hidrodinamik enjeksiyon (şekil 3A). İğne 7 gün sonra biz neredeyse yarısı transfected Hepatosit karaciğer bölümler (şekil 3B) lekeli gibi mCherry ifade bulundu.
Gen tedavisi için bir HMEJ tabanlı strateji kullanma imkanı keşfetmek için fumarylacetoacetate hidrolaz (Fah) istihdam-eksik fareler. Fah- / - fare iyi kurulmuş bir kalıtsal tyrosinemia tip ı exon 5 Aşağıdaki sıra23' frameshift mutasyonlar neden Fah genin bir ekleme parçası limanlar (HTI) fare modeli var. Fah- / - fareler korumak için tirozin katabolik yolu, 2-(2-nitro-4-trifluoromethylbenzoyl)-1,3-cyclohexanedione (NTBC)24ters yönde bir inhibitörü ile Fah- / - fareler tedavi ettik. Burada biz MMEJ ve HMEJ aracılı gen düzeltme Fah mutasyon Fah- / - Mouse kurtarmak olup olmadığını görmek için yola çıktı. Hydrodynamically Cas9 yapı enjekte ile birlikte Fah- MMEJ veya Fah- HMEJ yapıları, exon 5-14 intron Fah geni, 4 içine Fah cDNA eklemek için tasarlanmış/Fah- / - için fare karaciğer () Şekil 3 C). bir hafta sonra enjeksiyon, karaciğer hasarı (şekil 3C) ikna etmek için NTBC çekildi. NTBC, Fahçekilmesinden sonra-düzeltilmiş tetkikine Fah- HMEJ ve Cas9 yapıları alma Fah- / - farelerin gösterdi daha etkili yayılma yöntemi (şekil 3D MMEJ göre daha ).

Resim 1 : HMEJ-aracılı hedef Tümleştirme vitro.
(A)deneysel düzeni sgRNAs seçimi için: altı farklı sgRNAs (Cdx2-sgRNA1 ~Cdx2-sgRNA6) bir daha yüksek rütbe ve hedef kapalı potansiyeli olan Cdx2 locus kodonu etrafında seçilmiş online CRISPR tasarım dayalı idi aracı. Protospacer bitişik motifi (PAM) kırmızı sırasıdır. (B) deneysel tasarım: Cas9-CMV-GFP ifade plazmid sgRNA, Cas9 ve GFP ifade N2a hücre içine getirilmiştir. GFP+ hücreleri günde 3 surveyor tahlil için sıralanmış. (C) Surveyor tahlil Cdx2 hedeflemek için: 6 farklı sgRNAs arazi tahlil için tasarlanmıştır. Normal N2a hücre genomik DNA denetimi olarak hizmet vermektedir. *, Cdx2için kullanılan sgRNA-2A-mCherry çakma deney. (D) HMEJ bağış Gibson derleme kullanarak inşaatı şematik bakış. (E) HMEJ-aracılı gen Cdx2 locus strateji hedefleme şematik bakış. HAL/HAR, sol/sağ homoloji kol; üçgenler, sgRNA hedef siteleri; VE / veya, dış ileri/geri astar; Eğer / IR, iç ileri/geri astar. Şekil değiştiren önceki rapor10. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Resim 2 : Fare embriyo HMEJ-aracılı ile genom düzenleme hedef Integration
Mikroenjeksiyon(a)deneysel düzeni: Cas9 mRNA karışımı (100 ng/µL), sgRNA (50 ng/µL) ve donör plazmid (100 ng/µL) fare zigotları enjekte. (B) temsilcisi Floresans görüntüler fare embriyo HMEJ strateji tarafından düzenlenmiş. Bar, 20 verimliliği µm. (C) çakma blastokistlerin mCherry+ yüzdeyle belirtti. Sayı her çubuğun, toplam blastokistlerin sayılır. (D) sıra Cdx2 odağı, gen düzenlenmiş farelerin analiz. PCR ürünleri 5' ve 3' kavşak sitelerden güçlendirilmiş sıralı. Homoloji koluna; mor, p2A; kırmızı, mCherry; HAR veya HAL, sağ veya sol homolog kol. Kesik çizgiler netlik için atlanmış bölge işaretleyin. Şekil değiştiren önceki rapor10. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.

Şekil 3 : HMEJ-aracılı hedef entegrasyon içinde vivo.
(A)hidrodinamik kuyruk ven enjeksiyon şematik bakış. Donör sırası ve sgRNA ifade plazmit ve spCas9 ifade plazmid karışımı hidrodinamik kuyruk ven enjeksiyon yoluyla karaciğere teslim. (B) temsilcisi ayirt tetkikine görüntülerini. Karaciğer bölümleri toplanan 7 gün sonrası enjeksiyon vardı. Ölçek çubuğu, 50 µm. GFP, hücre transfected. (C) plazmid exon 5-14 Fah cDNA intron Fah geni 4 eklemek için tasarlanmış ya MMEJ ya da HMEJ aracılı gen değiştirme strateji Fah- / - fare karaciğer hidrodinamik enjeksiyonu ile teslim edildi. NTBC Tarih: Fah- / - fareler tutulan NTBC su; NTBC kapalı: NTBC su çekilmesi (ilk gün NTBC çekildiği gün 0, 7 gün sonra enjeksiyon olarak tanımlanmıştır). (D) Fah immünhistokimya, MMEJ veya HMEJ Plasmid'ler ile enjekte Fah- / - fare karaciğer bölümleri boyama. Önceki raporlar5,10' dan ölçek çubuğu, 100 µm. şekil değiştiren. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
HMEJ donör plazmid yapımında en önemli adım vardır: (1) yüksek DNA bölünme verimlilik ve düşük arasındaki mesafeyi sgRNA kesme makinası ve kodonu, HMEJ donör (2) uygun inşası ile sgRNA yelpazesi. CRISPR/Cas9-aracılı bölünme (sgRNA hedef siteleri ve ~ 800 bp homoloji silah içeren) her iki transgene donör vektör Tarih ve hedeflenen genom verimli ve hassas hedeflenen entegrasyon için gerekli içinde vivo. Çakma HMEJ tabanlı yöntemi kullanma fare nesil en kritik adımlar şunlardır: (1) yüksek kalitesi (hiçbir dejenerasyon var Cas9 mRNA ve sgRNA) sgRNA ve Cas9 mRNA hazırlanması ve (2) yüksek kalite HMEJ donör plazmid hazırlanması. Plazmid embriyonik geliştirme üzerinde hiçbir toksik etkileri gösterir.
Son zamanlarda, bir NHEJ tabanlı yöntemi için verimli vivo içinde genom8düzenleme bildirildi. Yine de, Indel mutasyonlar türleri genellikle kavşak hassas entegrasyonu sağlamak üzere önceki raporlar8' de açıklandığı gibi indüklenen. Burada, yukarıda açıklanan HMEJ tabanlı strateji neredeyse hiç Indel mutasyonlar ile kesin hedeflenen entegrasyon gösterdi. Böylece, HMEJ tabanlı bir strateji ile NHEJ tabanlı yöntemi için geçerli değildir doğru bir mutasyona uğramış bir sıra (örneğin, bir nokta mutasyon) eski yerine koymak için ideal bir platform olabilir.
Mozaizm gen embriyo düzenlemek için büyük bir sorundur. Bir daha erken embriyonik evre at Cas9 proteinin mRNA yerine enjeksiyon transgene knock bileşenini mozaizm olmadan bir hücre aşamada elde edebilir. Klinik uygulamalarda, CRISPR/Cas9 sistemleri teslim yetişkin dokuların içine hala meydan okuyor.
Genom HMEJ tabanlı düzenleme birçok gelecekteki potansiyel kullanımı vardır. Genetiği değiştirilmiş hayvan modelleri oluşturmak için kullanılabilir. Çakma yüksek verimlilik embriyo olarak göz önüne alındığında bu yöntem genetiği değiştirilmiş hayvan modelleri oluşturmak için gerekli hayvan sayısı önemli ölçüde azaltabilir ve özellikle insan dışı primat genetik modellerini üreten olasılığını açar. Genom HMEJ tabanlı düzenleme insan dışı primatlar gibi mevcut hayvan modellerin eksikliği olduğundan hayvan modellerinde, özellikle yararlı olan soy izleme bireysel hücre tipleri yetişkin dokularda olabilir. Hedeflenen gen tedavileri için kullanılabilir: klinik kullanımlar için en cazip bir HMEJ tabanlı strateji gen tedavisi uygulamasıdır. Bu çalışmada, biz Fah mutasyon kalıtsal tyrosinemia türü düzeltildi ben belirtilen vektörlerin hidrodinamik enjeksiyonla fareler. Ancak, hidrodinamik enjeksiyon hastalarda yapılması pek mümkün olduğu gibi yetişkin dokuların içine CRISPR/Cas9 sisteminin teslim hala klinik kullanımı için büyük teknik sorun olduğunu. Şu anda, daha fazla dağıtım stratejisi geliştirilmesi acilen kliniğe bu yöntemi HMEJ göre tercüme önce gereklidir.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Yazarlar ifşa gerek yok.
Acknowledgments
Bu eser CAS stratejik öncelik araştırma programı (XDB02050007, XDA01010409), ulusal Hightech R & D programı (863 Program; 2015AA020307), Çin Ulusal doğal Bilim Vakfı tarafından desteklenmiştir (verir NSFC 31522037, 31500825, 31571509, 31522038), Çin Gençlik bin yetenekleri programı (için HY), ara Çin bilim, Shanghai şehir Komitesi, bilim ve teknoloji Projesi (16JC1420202 HY için), Bilim Bakanlığı ve Çin teknoloji (çoğu; 2016YFA0100500).
Materials
| Name | Company | Catalog Number | Comments |
| pX330 | Addgene | 42230 | |
| pAAV vector | Addgene | 37083 | |
| pX260 | Addgene | 42229 | |
| AAV_Efs_hSpCas9_NLS_FLAG-SV40 | Addgene | 97307 | AAV vector for encoding a human codon-optimized SpCas9 driven by EFs promoter |
| AAV_Actb HMEJ donor_U6_sgRNA_EF1a_GFP_polyA | Addgene | 97308 | HMEJ donor for fusing a p2A-mCherry reporter to mouse Actb. EGFP driven by EF1a promoter and U6-driven sgRNAs targeting Actb. AAV backbone. |
| AAV_Cdx2 HMEJ donor | Addgene | 97319 | HMEJ donor for fusing a p2A-mCherry reporter to mouse Cdx2. |
| Lipofectamine 3000 Transfection Reagent | Life Technology | L3000015 | |
| Nuclease-Free Water | Life Technologies | AM9930 | |
| Bbs I | New England Biolabs | R0539S | |
| NEB Buffer 2 | New England Biolabs | B7002S | |
| T7 endonuclease I | New England Biolabs | M0302L | |
| NEBuilder HiFi DNA Assembly Master Mix | New England Biolabs | E2621L | |
| Plasmid EndoFree-Midi Kit | Qiagen | 12143 | |
| MMESSAGE MMACHINE T7 ULTRA | Life Technologies | AM1345 | |
| MEGACLEAR KIT 20 RXNS | Life Technologies | AM1908 | |
| MEGASHORTSCRIPT T7 KIT 25 RXNS | Life Technologies | AM1354 | |
| Flaming/Brown Micropipette Puller | Sutter Instrument | P-97 | Micropipette Puller (parameters: heat, 74; pull, 60; velocity, 80; time/delay, 200; pressure, 300) |
| Borosilicate glass | Sutter Instrument | B100-78-10 | type of capillaries (outer diameter 1.0 mm, inner diameter 0.78 mm with filament) |
| FemtoJet microinjector | Eppendorf | ||
| Freezing microtome | Leica | CM1950-Cryostat | thickness of 40 μm for brain, 10 μm for liver |
| Rabbit anti-mCherry | GeneTex | ||
| Cy3-AffiniPure Goat Anti-Rabbit IgG | Jackson Immunoresearch | ||
| DMEM | Gibco | 11965092 | |
| FBS | Gibco | 10099141 | |
| NEAA | Gibco | 11140050 | |
| Pen,Strep,Glutamine | Gibco | 10378016 | |
| Gel Extraction Kit | Omega | D2500-02 | |
| FACS | BD AriaII | ||
| PMSG | Ningbo Sansheng Medicine | S141004 | |
| HCG | Ningbo Sansheng Medicine | B141002 | |
| Cytochalasin B | Sigma | CAT#C6762 | |
| KSOM+AA with D-Glucose and Phenol Red | Millipore | CAT#MR-106-D | |
| M2 Medium with Phenol Red | Millipore | CAT#MR-015-D | |
| Mineral oil | Sigma |
References
- Yang, H., et al. Generation of Mice Carrying Reporter and Conditional Alleles by CRISPR/Cas-Mediated Genome Engineering. Cell. 154 (6), 1370-1379 (2013).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nature Biotechnology. 29 (8), 731-734 (2011).
- Nakade, S., et al. Microhomology-mediated end-joining-dependent integration of donor DNA in cells and animals using TALENs and CRISPR/Cas9. Nature Communications. 5, 5560 (2014).
- Hisano, Y., et al. Precise in-frame integration of exogenous DNA mediated by CRISPR/Cas9 system in zebrafish. Scientific reports. 5, 8841 (2015).
- Yao, X., et al. Cas9 - Mediated Precise Targeted Integration In Vivo Using a Double Cut Donor with Short Homology Arms. EBioMedicine. , (2017).
- Auer, T. O., Duroure, K., De Cian, A., Concordet, J. P., Del Bene, F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome research. 24 (1), 142-153 (2014).
- Maresca, M., Lin, V. G., Guo, N., Yang, Y. Obligate ligation-gated recombination (ObLiGaRe): custom-designed nuclease-mediated targeted integration through nonhomologous end joining. Genome Research. 23 (3), 539-546 (2013).
- Suzuki, K., et al. In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration. Nature. 540 (7631), 144-149 (2016).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Yao, X., et al. Homology-mediated end joining-based targeted integration using CRISPR/Cas9. Cell Research. 27 (6), 801-814 (2017).
- Han, D. W., et al. Direct reprogramming of fibroblasts into epiblast stem cells. Nature Cell Biology. 13 (1), 66-71 (2011).
- Han, D. W., et al. Direct Reprogramming of Fibroblasts into Neural Stem Cells by Defined Factors. Cell Stem Cell. , (2012).
- Ambasudhan, R., et al. Direct reprogramming of adult human fibroblasts to functional neurons under defined conditions. Cell Stem Cell. 9 (2), 113-118 (2011).
- Sparman, M., et al. Epigenetic reprogramming by somatic cell nuclear transfer in primates. Stem Cells. 27 (6), 1255-1264 (2009).
- Schatten, G., Mitalipov, S. Developmental biology: Transgenic primate offspring. Nature. 459 (7246), 515-516 (2009).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Cong, L., et al. Multiplex Genome Engineering Using CRISPR/Cas Systems. Science. 339 (6121), 819-823 (2013).
- Quadros, R. M., et al. Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome Biology. 18 (1), 92 (2017).
- Park, K. E., et al. Targeted Gene Knockin in Porcine Somatic Cells Using CRISPR/Cas Ribonucleoproteins. International journal of molecular sciences. 217 (6), (2016).
- Woo, J. W., et al. DNA-free genome editing in plants with preassembled CRISPR-Cas9 ribonucleoproteins. Nature biotechnology. 33 (11), 1162-1164 (2015).
- Harms, D. W., et al. Mouse Genome Editing Using the CRISPR/Cas System. Current protocols in human genetics. 83, 11-27 (2014).
- Yang, H., Wang, H., Jaenisch, R. Generating genetically modified mice using CRISPR/Cas-mediated genome engineering. Nature protocols. 9 (8), 1956-1968 (2014).
- Grompe, M., et al. Loss of Fumarylacetoacetate Hydrolase Is Responsible for the Neonatal Hepatic-Dysfunction Phenotype of Lethal Albino Mice. Genes & development. 7 (12), 2298-2307 (1993).
- Paulk, N. K., et al. Adeno-associated virus gene repair corrects a mouse model of hereditary tyrosinemia in vivo. Hepatology. 51 (4), 1200-1208 (2010).
Tags
Genetik sayı: 133 CRISPR/Cas9 hedeflenen Tümleştirme homoloji-aracılı son katılma In Vivo embriyo genetik fareler hidrodinamik enjeksiyon modifiyeErratum
Formal Correction: Erratum: CRISPR/Cas9-mediated Targeted Integration In Vivo Using a Homology-mediated End Joining-based Strategy
Posted by JoVE Editors on 03/10/2021.
Citeable Link.
An erratum was issued for: Studying TGF-β Signaling and TGF-β-induced Epithelial-to-mesenchymal Transition in Breast Cancer and Normal Cells. The phrases "surveyor assay" and "Surveyor Nuclease" have been updated to "T7E1 assay" to " T7 endonuclease I" respectively.
Step 1.2 in the Protocol has been updated from:
- Surveyor nuclease assay of sgRNA
NOTE: The targeting efficiency of the sgRNA used for the knock-in experiment is evaluated by surveyor nuclease assay (also known as T7 endonuclease I (T7EI) assay)17. Select the sgRNA with high DNA cleavage efficiency and a low distance between the sgRNA cutting site and the stop codon.
to:
- T7 endonuclease assay of sgRNA
NOTE: The targeting efficiency of the sgRNA used for the knock-in experiment is evaluated by T7 endonuclease (T7EI) assay17. Select the sgRNA with high DNA cleavage efficiency and a low distance between the sgRNA cutting site and the stop codon.
Figure 1 in the Representative Results has been updated from:
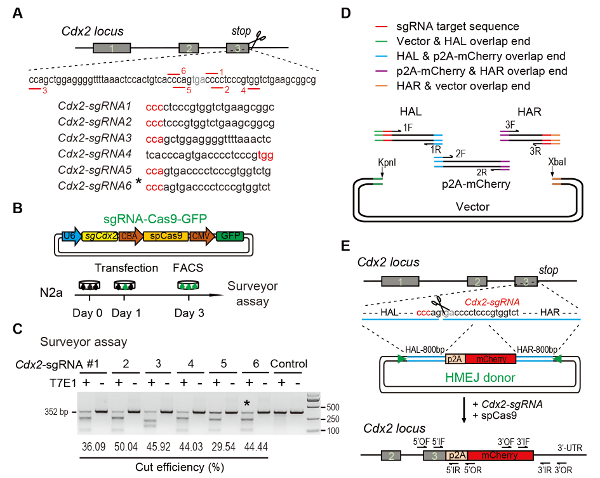
Figure 1: HMEJ-mediated targeted integration in vitro.
(A) Experimental scheme for selection of sgRNAs: Six different sgRNAs (Cdx2-sgRNA1~Cdx2-sgRNA6) around the stop codon of the Cdx2 locus with a higher rank and off-target potential were chosen based on online CRISPR design tool. The protospacer adjacent motif (PAM) sequence is in red. (B) Experimental design: The Cas9-CMV-GFP expression plasmids expressing sgRNA, Cas9, and GFP were introduced into N2a cells. GFP+ cells were sorted at day 3 for surveyor assay. (C) Surveyor assay for Cdx2 targeting: 6 different sgRNAs were designed for surveyor assay. Normal N2a cell genomic DNA serves as control. *, the sgRNA used for Cdx2-2A-mCherry knock-in experiment. (D) Schematic overview of construction of HMEJ donors using Gibson assembly. (E) Schematic overview of HMEJ-mediated gene targeting strategy at Cdx2 locus. HAL/HAR, left/right homology arm; triangles, sgRNA target sites; OF/OR, outer forward/reverse primer; IF/IR, inner forward/reverse primer. Figure modified from previous report10. Please click here to view a larger version of this figure.
to:
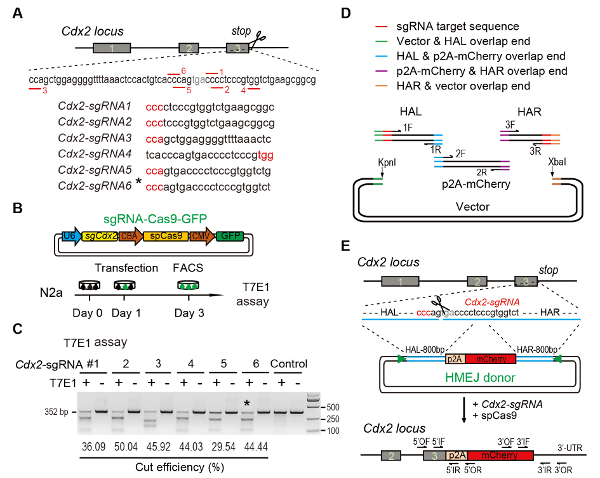
Figure 1: HMEJ-mediated targeted integration in vitro.
(A) Experimental scheme for selection of sgRNAs: Six different sgRNAs (Cdx2-sgRNA1~Cdx2-sgRNA6) around the stop codon of the Cdx2 locus with a higher rank and off-target potential were chosen based on online CRISPR design tool. The protospacer adjacent motif (PAM) sequence is in red. (B) Experimental design: The Cas9-CMV-GFP expression plasmids expressing sgRNA, Cas9, and GFP were introduced into N2a cells. GFP+ cells were sorted at day 3 for T7EI assay. (C) T7EI assay for Cdx2 targeting: 6 different sgRNAs were designed for T7EI assay. Normal N2a cell genomic DNA serves as control. *, the sgRNA used for Cdx2-2A-mCherry knock-in experiment. (D) Schematic overview of construction of HMEJ donors using Gibson assembly. (E) Schematic overview of HMEJ-mediated gene targeting strategy at Cdx2 locus. HAL/HAR, left/right homology arm; triangles, sgRNA target sites; OF/OR, outer forward/reverse primer; IF/IR, inner forward/reverse primer. Figure modified from previous report10. Please click here to view a larger version of this figure.



