Summary
सिंथेटिक जीव विज्ञान गैर-विहित अमीनो एसिड के सह-शोधों के सम्मिलन का उपयोग कर अभूतपूर्व गुणों के साथ प्रोटीन के इंजीनियरिंग सक्षम बनाता है. यहां, हम प्रस्तुत कैसे एक वर्णक्रमीय लाल-उपंयास प्रतिदीप्ति स्पेक्ट्रोस्कोपी गुणों के साथ एक GFP प्रकार fluorophore के संस्करण स्थानांतरित, "गोल्ड" फ्लोरोसेंट प्रोटीन (GdFP), शब्द का उत्पादन किया है चयनात्मक दबाव निगमन (SPI) के माध्यम से ई. कोलाई में उत्पादित ।
Abstract
फ्लोरोसेंट प्रोटीन जीवन विज्ञान के लिए मौलिक उपकरण हैं, विशेष रूप से जीवित कोशिकाओं के प्रतिदीप्ति माइक्रोस्कोपी के लिए । जबकि जंगली प्रकार और Aequorea विक्टोरिया (avGFP) से हरी फ्लोरोसेंट प्रोटीन के इंजीनियर वेरिएंट के रूप में अच्छी तरह से अंय प्रजातियों से homologs के रूप में पहले से ही ऑप्टिकल स्पेक्ट्रम के बड़े हिस्से को कवर, एक वर्णक्रमीय अंतर के पास अवरक्त क्षेत्र में रहता है, के लिए जिसमें avGFP आधारित fluorophores उपलब्ध नहीं हैं । लाल-फ्लोरोसेंट प्रोटीन (FP) वेरिएंट काफी एकाधिक आणविक प्रजातियों के वर्णक्रमीय unएनीमोन के लिए toolkit का विस्तार होता है, लेकिन स्वाभाविक रूप से होने वाली लाल-कोरल या समुद्र से व्युत्पंन एफपीएस खिसकाया कम प्रतिदीप्ति क्वांटम उपज है और अवर फोटो-avGFP वेरिएंट की तुलना में स्थिरता । आगे और हेरफेर और chromophore की संयुग्मित प्रणाली के संभव विस्तार दूर लाल वर्णक्रमीय क्षेत्र के लिए भी 20 विहित अमीनो आनुवंशिक कोड द्वारा निर्धारित एसिड की प्रदर्शनियों द्वारा सीमित है । इन सीमाओं को दूर करने के लिए, सिंथेटिक जीव विज्ञान आगे वर्णक्रमीय लाल chromophore त्रय में गैर विहित अमीनो एसिड की प्रविष्टि के माध्यम से स्थानांतरण प्राप्त कर सकते हैं । हम उपंयास वर्णक्रमीय गुणों के साथ इंजीनियर avGFP वेरिएंट के लिए SPI के आवेदन का वर्णन । प्रोटीन अभिव्यक्ति एक tryptophan-auxotrophic ई. कोलाई तनाव में और उपयुक्त इण्डोल पुरोगामी के साथ विकास के मीडिया का सप्लीमेंट द्वारा किया जाता है । कोशिकाओं के अंदर, इन अग्रदूतों इसी tryptophan अनुरूप करने के लिए परिवर्तित कर रहे है और UGG codons के जवाब में राइबोसोमल मशीनरी द्वारा प्रोटीन में शामिल किया । टीआरपी के प्रतिस्थापन-६६ में बढ़ाया "सियान" avGFP के संस्करण (ECFP) एक इलेक्ट्रॉन द्वारा 4-aminotryptophan GdFP में परिणाम एक १०८ एनएम स्टोक्स पारी और एक जोरदार लाल स्थानांतरित उत्सर्जन अधिकतम (५७४ एनएम) की विशेषता है, जबकि जा रहा है से अधिक स्थिर इसके पूर्ववर्ती ECFP. अवशेष-गैर विहित एमिनो एसिड के विशिष्ट शामिल जन स्पेक्ट्रोमेट्री द्वारा विश्लेषण किया है । GdFP के स्पेक्ट्रोस्कोपी गुण समय-हल प्रतिदीप्ति स्पेक्ट्रोस्कोपी जीवन विज्ञान में आनुवंशिक रूप से इनकोडिंग एफपीएस के मूल्यवान अनुप्रयोगों में से एक के रूप में की विशेषता है ।
Introduction
के बाद से जेलिफ़िश Aequorea विक्टोरिया (avGFP) में १९६२1 में हरी फ्लोरोसेंट प्रोटीन की खोज और अंय eukaryotic कोशिकाओं में १९९४2 में पहली heterologous अभिव्यक्ति, GFP परिवार के फ्लोरोसेंट प्रोटीन बन गए है जीवन विज्ञान में अत्यधिक मूल्यवान उपकरण और लक्ष्य । व्यापक आनुवंशिक और आणविक इंजीनियरिंग प्रजातियों के समायोजन-विशिष्ट codon उपयोग, तह का त्वरण, बेहतर परिपक्वता, वृद्धि की चमक, oligomerization की रोकथाम और वर्णक्रमीय और photochemical गुणों की सिलाई शामिल जिसमें reversibly photoswitch3,4,5,6की क्षमता शामिल है । GFP अपने 4-(p-hydroxybenzylidene) imidazolidin-5-एक (HBDI) chromophore से अपनी प्रतिदीप्ति बकाया है । बाद के तथाकथित chromophore एमिनो एसिड के त्रय (Ser-65/Tyr-66/Gly-67 से avGFP में) के गठन के बाद पेप्टाइड रीढ़ की एक अतिरिक्त आबंध बॉण्ड के निर्माण के तहत आणविक ऑक्सीजन के प्रभाव के तहत7। resonantly स्थिर संयुग्मित प्रणाली अपने आणविक वातावरण के साथ गतिशील रूप से सूचना का आदान प्रदान, दिखाई रेंज और इन प्रोटीन की विशेषता हरी प्रतिदीप्ति में अवशोषण के लिए अनुमति देता है ।
chromophore त्रय के भीतर, एक खुशबूदार एमिनो एसिड की उपस्थिति अनिवार्य है । हालांकि, मानक अमीनो एसिड की प्रदर्शनियां केवल चार खुशबूदार अवशेषों (उसकी, Phe, टीआरपी और Tyr) शामिल हैं । यह पारंपरिक mutagenesis के दृष्टिकोण को काफी अधिक लाल-avGFP वेरिएंट को प्राप्त करने की सीमा सबसे लाल के सापेक्ष स्थानांतरित प्राकृतिक एफपीएस जैसे DsRed8 से Discosoma striata coralimorphs या mKate/mNeptune से9 समुद्र एनेमोन Entacmaea quadricolor. इसलिए, ६०० एनएम के ऊपर ऑप्टिकल स्पेक्ट्रम का सुदूर लाल और निकट-अवरक्त भाग विरल GFP वेरिएंट द्वारा कवर किया जाता है । यह, ज़ाहिर है, प्रतिदीप्ति सूक्ष्म दृष्टिकोण है कि एक ही समय में कई fluorophore प्रजातियों के वर्णक्रमीय मल्टीप्लेक्स की आवश्यकता के लिए एक गंभीर सीमा है । उदाहरण के लिए, लंबी तरंग दैर्ध्य मार्करों भी गहरी ऊतक इमेजिंग10के लिए सेटिंग्स में 700-1000 एनएम के बीच त्वचा ऊतक के कम अवशोषण शासन का उपयोग करने के लिए आवश्यक हैं ।
फ्लोरोसेंट प्रोटीन avGFP से व्युत्पंन स्पेक्ट्रोस्कोपी गुण और उनके chromophores11के रासायनिक प्रकृति के आधार पर कई वर्गों में विभाजित हैं । इसके त्रय Ser के साथ-65/Tyr-66/Gly-67, वाइल्ड-टाइप chromophore के बीच एक equilibrated मिश्रण के रूप में मौजूद है न्यूट्रल, phenolic फॉर्म (λmax = ३९५ एनएम, ε = २१,००० मीटर-1सेमी-1) और anionic phenolate फॉर्म (λmax = ४७५ एनएम, ε = ७,१०० मीटर -1cm-1), और उत्सर्जन स्पेक्ट्रम ५०८ एनएम पर एक एकल चोटी का प्रदर्शन । Ser के हाइड्रॉक्सिल समूह-६५ महत्वपूर्ण महत्व का है, क्योंकि यह chromophore आसपास के क्षेत्र में एक एच बांड Glu-२२२ (दूरी: ३.७ Å), जो इस ionization के carboxylate को बढ़ावा देता है दान । वर्ग मैं एक anionic phenolate chromophore की विशेषता है, के रूप में EGFP में (Phe-64-लियू/Ser-65-के. λमैक्स = ४८८ एनएम, ε = ३५,६०० एम-1सेमी-1, λem = ५०९ एनएम) । Ser के कारण-६५--(ाला, Gly) प्रतिस्थापन, तटस्थ phenol फार्म के ३९५ एनएम उत्तेजना चोटी दबा दिया है और anionic phenolate के 470-475 एनएम चोटी पांच से छह गुना बढ़ाया है और ४९० एनएम के लिए स्थानांतरित कर दिया । द्वितीय श्रेणी में एक तटस्थ phenolic chromophore के साथ प्रोटीन शामिल हैं, के रूप में नीलमणि-GFP में । यहां, एक-२०३-इले प्रतिस्थापन लगभग पूरी तरह से ४७५ एनएम उत्तेजना को दबा, केवल चोटी छोड़ने ३९९ एनएम । चूँकि anionic chromophore ठीक से solvated नहीं हो सकता, इसलिए उसका तटस्थ रूप इष्ट है. कक्षा III "पीला" फ्लोरोसेंट वेरिएंट EYFP (शामिल हैं; Ser-65-Gly/वैल-68-लियू/Ser-72-ाला/-203-Tyr; λmax ε = ५१४ एनएम, ε = 84600 M-1cm-1, λ = ५२७ एनएम) एक खुशबूदार पक्ष श्रृंखला और phenolate, के रूप में के बारे में π-stacking बातचीत के साथ-२०३-अपने (टीआरपी, Phe, Tyr) प्रतिस्थापन, जो अप करने के लिए नेतृत्व 20 एनएम लाल-स्थानांतरित उत्सर्जन maxima (-२०३-Tyr) । इसके अलावा प्रतिस्थापन (Gln-६९-Lys) एक और 1-2 एनएम लाल पाली में परिणाम ५२९ एनएम, सबसे लाल खिसक avGFP संस्करण जाना जाता है11। एक इण्डोल (Tyr-६६-टीआरपी) के लिए phenol के आदान-प्रदान वर्ग चतुर्थ बनाता है, के रूप में सियान-फ्लोरोसेंट ECFP (Ser-65-Tyr/λmax1 = ४३४ एनएम, ε = २४,८०० मीटर-1सेमी-1; λmax2 = ४५२ एनएम, ε = २३,६०० एम-1सेमी-1 ; λem1 = ४७७ एनएम, λem2 = ५०४ एनएम) । भारी इण्डोल के आवास शायद अंय द्वारा सक्षम है, क्षतिपूरक उत्परिवर्तनों । ECFP उत्तेजना और उत्सर्जन maxima तटस्थ या anionic chromophores के साथ प्रोटीन के उन लोगों के बीच गिर जाते हैं । कक्षा V प्रोटीन phenol के स्थान पर एक imidazole बंदरगाह (Tyr-६६-अपने), जैसे, EBFP की तरह नीले फ्लोरोसेंट प्रोटीन । छठी कक्षा एक phenol-फिनाइल एक्सचेंज द्वारा तटस्थ chromophore फार्म का विशेष रूप से एहसान, जो फलस्वरूप सबसे अधिक नीला-स्थानांतरित उत्तेजना और उत्सर्जन चोटी पदों (३६० एनएम और ४४२ एनएम, क्रमशः) की ओर जाता है द्वारा उत्पादित है ।
शास्त्रीय साइट निर्देशित mutagenesis उपंयास avGFP chromophore वेरिएंट के उत्पादन के लिए विशेष रूप से उपयुक्त है, 65-67 tripeptide के परिवर्तन और 20 विहित अमीनो एसिड के फ्रेम में अवशेषों बातचीत से । इन संभावनाओं को और अधिक विस्तार किया जा सकता है जब गैर-विहित वेरिएंट खुशबूदार अमीनो एसिड के दौरान पेश कर रहे हैं राइबोसोमल प्रोटीन संश्लेषण12. सिद्धांत रूप में, वहां दो तरीके से यह पूरा कर रहे हैं । पहली रणनीति प्रोटीन अनुवाद मशीनरी के सब्सट्रेट सहिष्णुता पर निर्भर करता है, विशेष रूप से संबंधित एमिनो एसिड अनुरूप के प्रति aminoacyl-tRNA synthetases (aaRSs). उच्च दक्षता के साथ इस लक्ष्य को प्राप्त करने के लिए, auxotrophic ई. कोलाई अभिव्यक्ति उपभेदों कार्यरत है कि इसी प्राकृतिक एमिनो एसिड संश्लेषित करने में असमर्थ हैं । यह उपयुक्त गैर विहित अमीनो एसिड (ncaas) या संस्कृति माध्यम के लिए उसके पूर्ववर्ती जोड़कर बाद के प्रतिस्थापन की अनुमति देता है । इस रणनीति को भी चयनात्मक दबाव निगमन (SPI)13,14के रूप में जाना जाता है, अवशेषों विशिष्ट प्रतिस्थापन, जो ncAA के वैश्विक निगमन में परिणाम में सक्षम बनाता है । दूसरी रणनीति बंद codon शमन tRNAs जो इंजीनियर aaRS एंजाइमों द्वारा ncAA के साथ आरोप लगाया जाता है का उपयोग करता है । के readthrough में यह परिणाम में फ्रेम codons बंद करो और अनुमति देता है साइट विशेष ncAA शामिल । नतीजतन, stop codon दमन (SCS) की इस विधि आनुवंशिक कोड के विस्तार की ओर जाता है15। mutagenesis के माध्यम से, एक बंद codon वांछित साइट पर लक्ष्य जीन में रखा गया है । सिद्धांत रूप में, SPI भी रिकॉमबिनेंट पेप्टाइड्स और एक अद्वितीय ncAA अधिष्ठापन असर प्रोटीन बनाने के लिए इस्तेमाल किया जा सकता है, यह देखते हुए कि दुर्लभ विहित अमीनो एसिड के रूप में मिले या टीआरपी प्रतिस्थापन के लिए चुना जाता है । Trp के साथ, SPI दृष्टिकोण 4-F सहित एनालॉग की एक बड़ी विविधता के साथ काम करने के लिए दिखाया गया है, 5-एफ-और 6-एफ-टीआरपी, 7-aza-टीआरपी, 4-oh-और 5-oh-टीआरपी, साथ ही 4-, और 5-NH2-टीआरपी या सम β (thienopyrrolyl) alanine डेरिवेटिव्स16 ,17,18,19,20. इस प्रकार, SPI गैर द्वारा GFP chromophores के खुशबूदार अमीनो एसिड की जगह के लिए अत्यधिक लाभप्रद हो सकता है-विहित वेरिएंट आगे दर्जी स्पेक्ट्रा और स्टोक्स इन एफपीएस बदलाव की संभावना का पता लगाने के लिए । सभी प्रोटीन अनुक्रम संशोधनों के लिए के रूप में, FP तह और chromophore परिपक्वता के साथ संगतता प्रयोग परीक्षण किया जाना चाहिए ।
इस काम में, हम वर्ग चतुर्थ ECFP21, जो जंगली प्रकार avGFP Tyr, इसके chromophore त्रय के भीतर एक टीआरपी अवशेषों के बजाय वहन करती है का उपयोग । SPI का प्रयोग, इस टीआरपी-६६ (और टीआरपी-५७, ECFP में ही अंय टीआरपी अवशेषों) 4 द्वारा प्रतिस्थापित-अमीनो-टीआरपी है । इलेक्ट्रॉन की उपस्थिति-4-अमीनो-chromophore भीतर टीआरपी के एमिनो समूह दान एक दूर लाल स्थानांतरित उत्तेजित राज्य प्रोटॉन स्थानांतरण (ESPT) एक १०८ एनएम स्टोक्स बदलाव के साथ संपंन की प्रतिध्वनि स्थिरीकरण एहसान । इस ' गोल्ड ' फ्लोरोसेंट प्रोटीन (GdFP) सभी avGFP-व्युत्पंन प्रोटीन के बीच प्रतिदीप्ति अधिकतम (५७४ एनएम) की सबसे बड़ी लाल बदलाव के साथ संस्करण का गठन किया । हम SPI द्वारा GdFP प्रोटीन उत्पादन की विधि का वर्णन है और जन स्पेक्ट्रोस्कोपी द्वारा परिणामी संशोधित प्रोटीन के अनिवार्य विश्लेषण के लिए प्रोटोकॉल प्रदान करते हैं । इसके अलावा, हम बताएंगे कि कैसे GdFP का उपयोग किया जा सकता है और समय में विश्लेषण-प्रतिदीप्ति स्पेक्ट्रोस्कोपी दृष्टिकोण हल ।
Protocol
1. टीआरपी के परिवर्तन-auxotrophic ई. कोलाई
- एक टीआरपी के रासायनिक या electrocompetent कोशिकाओं (५० µ l) रूपांतरण-auxotrophic ई. कोलाई तनाव, उदा. ATTC ४९९८० (WP2, उत्परिवर्ती ई. कोलाई तनाव बी/आर22से व्युत्पंन), के साथ 1 µ एल के एक 1 एनजी/µ एल pQE के जलीय समाधान-80L His6-ECFP गर्मी सदमे या प्लाज्मिड, क्रमशः का उपयोग electroporation । विवरण के लिए कृपया जौव विज्ञान शिक्षा डाटाबेस23,24 को देखें ।
नोट: अभिव्यक्ति वेक्टर pQE-80L His6-ECFP encodes एक N-टर्मिनली 6x अपने-टैग ECFP21 लाख ऑपरेटर के साथ एक बैक्टीरियल T5 प्रमोटर द्वारा संचालित । इसके आगे एक AmpR चयन मार्कर और प्रतिकृति के colE1 मूल (pQE-80L वेक्टर रीढ़ अनुक्रम पर पाया जा सकता है: https://www.qiagen.com/mx/resources/resourcedetail?id=c3b71572-4d82-4671-a79b-96357fe926d1&lang=en & reसुझाएं = true) । His6-ECFG जंगली प्रकार के प्रोटीन के सैद्धांतिक आणविक वजन (chromophore परिपक्वता के बाद25) है २८३०३.९२ डा. अनुवादित लक्ष्य प्रोटीन अनुक्रम के रूप में इस प्रकार है (अपने-टैग को रेखांकित किया है, सदिश-बोल्ड में अनुक्रम व्युत्पंन): MRGSHHHHHHजी एसMVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTWGVQCFSRYPDHMK
QHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYISHNVYITADKQKNGIKANFKIRHNIEDGS
VQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYK । - प्लेट पौंड-आगर प्लेटों (तालिका 1) 10 ग्राम/L ग्लूकोज, १०० µ g/एमएल एम्पीसिलीन के साथ पूरक और ३७ डिग्री सेल्सियस रात भर में प्लेटें पर बदल कोशिकाओं ।
2. रिकॉमबिनेंट प्रोटीन एक्सप्रेशन
-
रात भर संस्कृति ई. कोलाई ATCC ४९९८० pQE-80L His6-ECFP
- पौंड मध्यम (तालिका 1के 5 मिलीलीटर तैयार; एक बाँझ पिपेट टिप या टीका लूप का उपयोग कर एक आगर प्लेट से एक अकेले कॉलोनी के साथ एरोबिक विकास और inoculate के लिए 10 ग्राम/एल ग्लूकोज, १०० µ g/एमएल एम्पीसिलीन) में एक बंजर 14 मिलीलीटर polystyrene संस्कृति ट्यूब के साथ पूरक ।
नोट: नए सिरे से रूपांतरित कक्षों से कालोनियों का उपयोग अनुशंसित है । बैक्टीरियल कालोनियों के साथ प्लेटें (१.२ कदम से.) 4 डिग्री सेल्सियस पर कई दिनों के लिए संग्रहित किया जा सकता है । - रात में 200-250 rpm पर एक कक्षीय शेखर में ३७ डिग्री सेल्सियस पर कोशिकाओं को मशीन ।
- पौंड मध्यम (तालिका 1के 5 मिलीलीटर तैयार; एक बाँझ पिपेट टिप या टीका लूप का उपयोग कर एक आगर प्लेट से एक अकेले कॉलोनी के साथ एरोबिक विकास और inoculate के लिए 10 ग्राम/एल ग्लूकोज, १०० µ g/एमएल एम्पीसिलीन) में एक बंजर 14 मिलीलीटर polystyrene संस्कृति ट्यूब के साथ पूरक ।
-
वन्य-प्रकार ECFP की अभिव्यक्ति
- Inoculate 10 मिलीलीटर ताजा पौंड मध्यम (तालिका 1; 10 g/L ग्लूकोज, १०० µ g/एमएल एम्पीसिलीन) के साथ पूरक १०० µ एल के साथ रातोंरात संस्कृति के एक १०० मिलीलीटर Erlenmeyer कुप्पी में. २०० rpm पर एक कक्षीय शेखर में ३७ डिग्री सेल्सियस पर कुप्पी की मशीन ।
नोट: वैकल्पिक रूप से, इस चरण में किया जा सकता 10 मिलीलीटर NMM19 मध्यम (तालिका 1) १०० µ g/l एम्पीसिलीन और ०.५ mM L-tryptophan के साथ पूरक (विकल्प, इण्डोल उपयोग किया जा सकता है) । - ६०० एनएम (आयुध डिपो६००) पर ऑप्टिकल घनत्व को मापने हर 20 मिनट. अधिमानी 1 सेमी की एक पथ की लंबाई के साथ एक cuvette का उपयोग कर एक spectrophotometer में ६०० एनएम (ओडी६००) में विलुप्त होने का निर्धारण करके कोशिका घनत्व को मापने । हमेशा कोई संदर्भ निष्पादित करें इसी संस्कृति माध्यम का उपयोग माप । नमूनों को पतला और 0.1-0.8 का माप मान प्राप्त करने के लिए अच्छी तरह से नमूनों को मिलाएं, फिर कमजोर पड़ने वाले कारक का उपयोग करके६०० की गणना करें । विवरण के लिए, कृपया पिछले प्रकाशन 26देखें ।
- एक आयुध डिपो 0.5-0.8 (लगभग 2-3 टीका के बाद एच) के६०० मूल्य तक पहुंचने पर, नमूना लेने के लिए "से पहले प्रेरण" एसडीएस के लिए पृष्ठ (सोडियम dodecyl सल्फेट polyacrylamide जेल ट्रो, 4 कदम) ।
- ०.५ mM IPTG करने के लिए तरल संस्कृति का समायोजन करके लक्ष्य प्रोटीन अभिव्यक्ति प्रेरित (isopropyl β-D-1-thiogalactopyranoside, 1 एम स्टॉक समाधान से) और यह एक कक्षीय शेखर में 30 डिग्री सेल्सियस पर में यह मशीन २०० rpm पर 4-8 एच के लिए ।
नोट: सियान फ्लोरोसेंट प्रोटीन सामांयतः ३७ डिग्री सेल्सियस से नीचे तापमान पर व्यक्त कर रहे है27। - एसडीएस-पृष्ठ (चरण 4.) के लिए "अभिव्यक्ति के बाद" नमूना लें ।
- ५,००० x g और 4 डिग्री सेल्सियस पर 10 मिनट के लिए केंद्रापसारक द्वारा बैक्टीरियल कोशिकाओं फसल ।
- supernatant को त्याग कर छोड़ दें और-20 ° c या-८० ° c लक्ष्य प्रोटीन शुद्धि तक सेल छर्रों फ्रीज ।
- Inoculate 10 मिलीलीटर ताजा पौंड मध्यम (तालिका 1; 10 g/L ग्लूकोज, १०० µ g/एमएल एम्पीसिलीन) के साथ पूरक १०० µ एल के साथ रातोंरात संस्कृति के एक १०० मिलीलीटर Erlenmeyer कुप्पी में. २०० rpm पर एक कक्षीय शेखर में ३७ डिग्री सेल्सियस पर कुप्पी की मशीन ।
-
उत्पादन GdFP के लिए SPI
- Inoculate NMM19 मध्यम (तालिका 1) के 10 मिलीलीटर १०० µ जी के साथ पूरक है/एमएल एम्पीसिलीन, 15 µ एम tryptophan और एक १०० मिलीलीटर µ कुप्पी में रातोंरात संस्कृति के 10 Erlenmeyer एल और एक कक्षीय शेखर में रात भर संस्कृति की कुप्पी मशीन में २०० rpm ।
नोट: ई. कोलाई और SPI की खेती के लिए रासायनिक रूप से परिभाषित मीडिया की एक किस्म उपलब्ध है । इसके अलावा NMM इस्तेमाल किया, MOPS मध्यम28, ग्लूकोज खनिज लवण मध्यम29, डेविस ंयूनतम मध्यम30, M9 ंयूनतम मध्यम31, या GMML३२ इस्तेमाल किया जा सकता है । - अगले दिन, माप आयुध डिपो६०० हर 30 मिनट तक मूल्य केवल परिवर्तन से कम ०.०५ से अधिक 30 मिनट । पठारी मान लगभग १ होना चाहिए.
नोट: ± ०.३ इकाइयों द्वारा विचलन स्वीकार्य हैं । जीवाणु तनाव और मध्यम उपयोग के आधार पर, प्रारंभिक tryptophan एकाग्रता (step. 2.3.1) समायोजन की आवश्यकता हो सकती है । - एसडीएस-पृष्ठ (चरण 4.) के लिए "प्रेरण से पहले" नमूना ले लो ।
- ५,००० x g और 4 डिग्री सेल्सियस पर 10 मिनट के लिए केंद्रापसारक द्वारा बैक्टीरियल कोशिकाओं फसल । supernatant का त्याग कर दें ।
- NMM19 मध्यम के 10 मिलीलीटर में १०० µ जी/एमएल एम्पीसिलीन के साथ एक १०० मिलीलीटर Erlenmeyer कुप्पी में कोशिकाओं को पुनः निलंबित और 1 मिमी के एक अंतिम एकाग्रता के लिए 4-अमीनो-इण्डोल जोड़ें ५० mm शेयर समाधान का उपयोग कर । २०० rpm पर एक कक्षीय शेखर में 30 डिग्री सेल्सियस में 30 मिनट के लिए मशीन जारी रखें ।
नोट: इस कदम के कारण एम्पीसिलीन की कम रासायनिक स्थिरता की सिफारिश की है और सेलुलर 4 अमीनो-इण्डोल के ऊपर सुनिश्चित करता है । - ०.५ मिमी की एक अंतिम एकाग्रता के लिए IPTG जोड़कर लक्ष्य प्रोटीन अभिव्यक्ति प्रेरित 1 एम स्टॉक का उपयोग कर और एक कक्षीय शेखर में 30 डिग्री सेल्सियस पर रात भर नमूना २०० rpm में ।
नोट: सियान फ्लोरोसेंट प्रोटीन सामांयतः ३७ डिग्री सेल्सियस से नीचे तापमान पर व्यक्त कर रहे है27। - अगले दिन, माप आयुध डिपो६००।
- एसडीएस-पृष्ठ (चरण 4.) के लिए "अभिव्यक्ति के बाद" नमूना लें ।
- ५,००० x g और 4 डिग्री सेल्सियस पर 10 मिनट के लिए केंद्रापसारक द्वारा बैक्टीरियल कोशिकाओं फसल और supernatant त्यागें ।
- मामले में इस तरह के एक पोत केंद्रापसारक के लिए इस्तेमाल नहीं किया गया था, एक रंग का उपयोग कर एक ५० मिलीलीटर शंकु polystyrene ट्यूब में सेल गोली हस्तांतरण । लक्ष्य प्रोटीन शुद्धि जब तक-20 ° c या-८० ° c पर सेल गोली फ्रीज ।
- Inoculate NMM19 मध्यम (तालिका 1) के 10 मिलीलीटर १०० µ जी के साथ पूरक है/एमएल एम्पीसिलीन, 15 µ एम tryptophan और एक १०० मिलीलीटर µ कुप्पी में रातोंरात संस्कृति के 10 Erlenmeyer एल और एक कक्षीय शेखर में रात भर संस्कृति की कुप्पी मशीन में २०० rpm ।
3. लक्ष्य प्रोटीन शोधन के माध्यम से मैटीरियल धातु आयन संबध क्रोमैटोग्राफी (IMAC)
-
बैक्टीरियल सेल lysis
- गल बर्फ पर सेल गोली 10-20 मिनट के लिए ।
- बर्फ पर बर्फ ठंडा बाध्यकारी बफर (तालिका 1) के 5 मिलीलीटर का उपयोग कर एक ५० मिलीलीटर शंकु polystyrene ट्यूब में सेल गोली reसस्पेंड ।
- जोड़ें 20 µ एल के ५० मिलीग्राम/एमएल lysozyme, 20 µ एल के 1 मिलीग्राम/एमएल DNase मैं, और 20 µ एल के 1 मिलीग्राम/एमएल RNase ए ट्यूब बंद, 5 बार पलटने से धीरे मिश्रण, और 30 मिनट के लिए बर्फ पर रखने के लिए ।
नोट: आंशिक कक्ष व्यवधान lysozyme द्वारा catalyzed के रूप में होता है । - लाइसे एक अल्ट्रासाउंड homogenizer टिप का उपयोग कर sonification द्वारा कोशिकाओं में 3 मिनट के तीन चक्र का उपयोग कर एक 15 मिलीलीटर polystyrene ट्यूब के साथ कीचड़ बर्फ से ठंडा 2 पल्स के एस, ठहराव के 4 एस और ४५% आयाम ।
नोट: वैकल्पिक रूप से, उच्च दबाव homogenization इस्तेमाल किया जा सकता है, उदा, १४,००० साई में 20 चक्र. यदि आवश्यक हो, ंयूनतम साधन मात्रा तक पहुंचने के लिए बाध्यकारी बफर का उपयोग कर पतला । इसके अलावा, प्रोटीन निष्कर्षण रिएजेंट सेल व्यवधान के लिए इस्तेमाल किया जा सकता है । उदाहरण के लिए सामग्री तालिका देखें । - 15000-18000 x g, 4 ° c पर 30 मिनट के लिए नमूना केंद्रापसारक ।
- एक ताजा ट्यूब में supernatant स्थानांतरण और तरल मात्रा नीचे ध्यान दें ।
- एक 5 मिलीलीटर प्लास्टिक Luer लॉक सिरिंज और एक polyvinylidene फ्लोराइड (PVDF) सिरिंज फिल्टर का उपयोग कर एक ०.४५ µm सिरिंज फिल्टर के माध्यम से समाधान फ़िल्टर.
- एसडीएस-पृष्ठ (चरण 4.) के लिए "lysate" का नमूना लें ।
- ddH2O (पूर्व lysate के रूप में बराबर मात्रा) में सेल मलबे गोली reसस्पेंड ।
- एसडीएस-पृष्ठ (चरण 4.) के लिए नमूना "गोली" ले लो ।
-
IMAC शुद्धि
- निर्माता के निर्देशों के अनुसार 1 मिलीलीटर पैक या स्व-पैक IMAC FPLC (fast प्रोटीन लिक्विड क्रोमैटोग्राफी) स्तंभ का उपयोग करें । स्तंभ equilibration के लिए बाइंडिंग बफ़र (तालिका 1) का उपयोग करें और साथ ही वॉश चरण के लिए जो इस प्रकार है कि कक्ष lysate को स्तंभ पर लागू किया गया है ।
- लीजिए और GdFP के साथ पूल eluate भागों जो दिखाई हल्के सुनहरे रंग से पहचाना जा सकता है ।
नोट: वैकल्पिक रूप से, लक्ष्य प्रोटीन एक स्वचालित FPLC प्रणाली का उपयोग कर एक रैखिक imidazole ढाल (0-250 mM) का उपयोग कर eluted जा सकता है । - ४६६ एनएम (ɛ४६६ nm = २३,७०० M-1 cm-1)३३ रेफरेंस बफर के साथ संदर्भ के रूप में विलुप्त होने गुणांक के लिए साहित्य मूल्य का उपयोग कर प्रोटीन एकाग्रता का निर्धारण । प्रक्रिया पर विवरण के लिए, कृपया पिछले प्रकाशन26देखें ।
- एसडीएस-पृष्ठ के लिए "eluate" का नमूना लें, और Coomassie-धुंधला होने की स्थिति में प्रति लेन प्रोटीन का 1-10 µ g का उपयोग करें ।
नोट: एसडीएस नमूना मात्रा धुंधला विधि और डाई संवेदनशीलता के आधार पर भिन्न हो सकते हैं । - Dialyze डायलिसिस बफर या एमएस बफर के खिलाफ eluate अंश की एक aliquot 5000-10000 की एक आणविक वजन cutoff (MWCO) के साथ एक झिल्ली का उपयोग कर । निर्माता के निर्देशों के अनुसार डायलिसिस झिल्ली तैयार करें । Dialyze एक 1 मिलीलीटर नमूना तीन बार १०० एमएल बफर के खिलाफ कम से 2 ज के लिए । इस कार्यविधि पर विवरण के लिए, कृपया पिछला प्रकाशन३४देखें ।
- भंडारण के लिए, डायलिसिस बफर में-८० डिग्री सेल्सियस में प्रोटीन का नमूना फ्रीज ।
नोट: Aliquots को कम से 6 महीने के लिए स्थिर होना चाहिए ।
4. एसडीएस-पृष्ठ नमूना ई. कोलाई पूरे सेल निकालने की तैयारी
- एक सेल निलंबन के समकक्ष६०० के 1 एमएल के लिए स्थानांतरण = 1 निलंबन (उदा. ५०० µ एल के आयुध डिपो६००= 2) एक १.५ मिलीलीटर microcentrifuge ट्यूब में ।
- फसल ५००० x जी, कमरे के तापमान पर 10 मिनट के लिए केंद्रापसारक द्वारा कोशिकाओं । pipetting द्वारा supernatant को त्यागें ।
- जोड़ें ८० µ एल के ddH2ओ और 20 µ एल के 5x एसडीएस लोडिंग डाई बफर (तालिका 1) के लिए सेल गोली और pipetting द्वारा मिश्रण.
- एक पानी के स्नान या गर्मी ब्लॉक में 5 मिनट के लिए ९५ ° c के लिए हीटिंग से कोशिकाओं स्वभाव । बाद में, कमरे के तापमान के लिए नमूनों शांत ।
- पिछले प्रकाशन३५के अनुसार Coomassie-सना एसडीएस-पृष्ठ के लिए 10 µ l का उपयोग करें ।
नोट: एसडीएस नमूना मात्रा धुंधला विधि और डाई संवेदनशीलता के आधार पर भिन्न हो सकते हैं ।
5. उच्च प्रदर्शन द्वारा बरकरार प्रोटीन मास विश्लेषण तरल क्रोमैटोग्राफी (HPLC) Electrospray Ionization समय के लिए युग्मित-उड़ान मास स्पेक्ट्रोमेट्री (LC-ईएसआई-तोफ-MS)
नोट: HPLC ग्रैडिएंट, सेटिंग्स और बफ़र्स भिंन स्तंभ और उपयोग किए गए उपकरण के आधार पर भिन्न हो सकते हैं । अनुकरणीय उपकरणों के लिए सामग्री तालिका देखें ।
- एमएस बफर के खिलाफ एक नमूना dialyzed से प्रोटीन एकाग्रता का निर्धारण (चरण 3.2.3.) ms बफ़र का उपयोग कर (सामग्री तालिका देखें) संदर्भ के रूप में ।
- प्रोटीन का नमूना पतला करने के लिए ०.१ मिलीग्राम/एमएल ८० µ एल के अंतिम खंड के लिए एमएस बफर का उपयोग कर, सावधान pipetting द्वारा मिश्रण, गिलास डालने के साथ एक एमएस नमूना शीशी में समाधान हस्तांतरण और एक टोपी के साथ बंद करो । शीशी को झाड़कर हवा के बुलबुले निकालें ।
- एमएस बफर की 1 मिलीलीटर के साथ कांच डालने (बफर रिक्त) के बिना एक दूसरा नमूना शीशी भरें ।
- साधन गर्म करने के लिए अनुमति देते हैं । साधन अंशांकन प्रदर्शन । सुनिश्चित करें कि तरल क्रोमैटोग्राफी-ग्रेड सॉल्वैंट्स की पर्याप्त मात्रा में उपलब्ध है (> 100 एमएल) ।
- कार्यक्रम एक रैखिक 20 मिनट HPLC ढाल से 5% करने के लिए ८०% बफर एक (०.१% ddH में फार्म का एसिड2O), बफर बी के साथ संयुक्त (०.१% acetonitrile में फार्म का एसिड).
- ०.३ मिलीलीटर/मिनट के प्रवाह पर HPLC शुरू करें और स्तंभ दबाव स्थिर होने तक प्रतीक्षा करें ।
- नियंत्रण रेखा-ईएसआई-तोफ-एमएस विधि के लिए 5 µ एल के एक नमूना इंजेक्शन मात्रा सेट, एक नमूना चलाने के बाद एक खाली चलाने के लिए एक worklist बनाने के लिए और इसी नमूना शीशी पदों आवंटित. भागो worklist ।
- worklist पूरा होने के बाद, जनरेट किया गया नमूना डेटा फ़ाइल खोलें । deconvolution के लिए कुल आयन वर्तमान (टिक) भूखंड में एक सीमा का चयन करें और एमएस deconvolute अधिकतम एन्ट्रापी deconvolution एल्गोरिथ्म का उपयोग कर स्पेक्ट्रम ।
नोट: प्रयोगात्मक शर्तों के आधार पर, अतिरिक्त प्रजातियों गैर परिपक्व FP या बफर आयन adducts से हो सकता है ।
6. प्रतिदीप्ति जीवनकाल माप और क्षय-जुड़े स्पेक्ट्रा (GdFP के दास)
नोट: समय-समाधान प्रतिदीप्ति स्पेक्ट्रोस्कोपी की इंस्ट्रूमेंटेशन के लिए, कृपया अनुकरणीय उपकरणों के लिए सामग्री तालिका देखें । अवशोषित के रूप में अच्छी तरह के रूप में प्रतिदीप्ति उत्तेजना और फ्लोरोसेंट प्रोटीन के उत्सर्जन स्पेक्ट्रा भी प्रयोगशाला यूवी/विज़ और प्रतिदीप्ति spectrophotometers का उपयोग कर दर्ज किया जा सकता है ।
-
तरंग दैर्ध्य-हल प्रतिदीप्ति GdFP के जीवनकाल माप
- GdFP के एक 1 µ m समाधान के 2 मिलीलीटर को पंजाब के बफर (तालिका 1में) 7 पीएच पर तैयार करें । एक 1 सेमी क्वार्ट्ज cuvette में समाधान भरें ।
- नमूना उत्तेजना के लिए ps-स्पंदित ४७० एनएम लेजर स्थापित करें और ४८८ एनएम लंबे समय से गुजारें उत्सर्जन फिल्टर, और समायोजित ६०० एल/mm बार के कसा हुआ-और तरंग दैर्ध्य-संबंधित एकल फोटॉन गिनती३६ (TWCSPC) तरंग दैर्ध्य शासन के अधिग्रहण के लिए डिटेक्टर 500-700 एनएम ।
- के बारे में २०० x 103 फोटॉनों/s की गिनती दर में प्रतिदीप्ति उत्सर्जन के बारे में जब तक 103 मायने रखता एकल फोटॉन गिनती सॉफ्टवेयर के साथ प्रतिदीप्ति क्षय curves के अधिग्रहण अधिकतम में जमा हो जाती है ।
-
वाद्य प्रतिक्रिया समारोह का मापन३७ (IRF)
- 1 सेमी क्वार्ट्ज के साथ नमूना cuvette बदलें cuvette 1 g/L कोलाइडयन सिलिका (~ २२० एम2/g) के साथ में पंजाब के बफर में पीएच 7 पर ।
नोट: सिलिका सस्पेंशन एक ४०० g/L जलीय सस्पेंशन का उपयोग कर तैयार किया जाता है । - निकालें ४८८ एनएम लंबे समय से गुजारें उत्सर्जन फ़िल्टर और डालने के लिए ग्रे फिल्टर TWCSPC डिटेक्टर पर गिनती की दर को समायोजित करने के लिए नीचे १०० x 103 मायने रखता/
- 16 चैनल TWCSPC डिटेक्टर के चैनल 8 में ४७० एनएम फोटॉनों के अधिग्रहण के लिए कद्दूकस करना समायोजित करें ।
- के बारे में 10 x 103 मायने रखता उत्सर्जन अधिकतम में जमा हो जाती है जब तक IRF अधिग्रहण ।
- परिवर्तित प्रतिदीप्ति क्षय घटता है और वैश्विक फिटिंग३८ प्रोग्राम के साथ ASCII डेटा फ़ाइलों के लिए IRF ।
- जुड़े हुए मापदंडों के रूप में जंमों के साथ तीन घातीय घटकों की एक राशि के एक मॉडल के अनुसार वैश्विक फिट आचरण ।
- भूखंड क्षय संबंधित स्पेक्ट्रा (दास) डेटा विश्लेषण सॉफ्टवेयर के साथ तरंग दैर्ध्य की निर्भरता में व्यक्तिगत क्षय घटकों के आयाम वितरण के रूप में.
- 1 सेमी क्वार्ट्ज के साथ नमूना cuvette बदलें cuvette 1 g/L कोलाइडयन सिलिका (~ २२० एम2/g) के साथ में पंजाब के बफर में पीएच 7 पर ।
Representative Results
चुनिंदा दबाव शामिल करने की तकनीक का प्रयोग, टीआरपी-६६ chromophore त्रय में ECFP (और टीआरपी-५७, ECFP में ही अंय टीआरपी अवशेषों) 4 द्वारा प्रतिस्थापित किया जा सकता है-अमीनो-टीआरपी, जिससे लाल-GdFP अलग वर्णक्रमीय गुणों के साथ स्थानांतरित कर दिया । मास स्पेक्ट्रोमेट्री प्रोटीन में गैर विहित एमिनो एसिड की वांछित stoichiometric एकीकरण प्रदर्शित करने के लिए इस्तेमाल किया जाना चाहिए, चित्रा 1में दिखाया परिणामों के साथ. बाद में, हम माइक्रोस्कोपी से डेटा प्रदान, यूवी की तुलना अवशोषण स्पेक्ट्रोस्कोपी के रूप में के रूप में अच्छी तरह से स्थिर राज्य और समय और तरंग दैर्ध्य-हल प्रतिदीप्ति स्पेक्ट्रोस्कोपी के पीएच निर्भरता पर ध्यान देने के साथ GdFP fluorophore के गुणों की विशेषता के लिए स्पेक्ट्रा.
ECFP में दो टीआरपी अवशेषों के आदान-प्रदान की पुष्टि करने के लिए 4-अमीनो-टीआरपी, मास spectrometric विश्लेषण किया जाता है । चित्रा 1 GdFP के एक प्रतिनिधि deconvoluted ईएसआई एमएस स्पेक्ट्रम से पता चलता है । जबकि वाइल्ड-टाइप ECFP में chromophore परिपक्वता के बाद २८,२८३.९ दा की गणना प्रोटीन द्रव्यमान है, GdFP के इसी मास में २८,३१३.९ डा. GdFP के deconvoluted ईएसआई-एमएस स्पेक्ट्रम २८,३१४.१ ± ०.१ दा है, जो कम 10 पीपीएम से सैद्धांतिक मूल्य से भटक पर एक मुख्य जन चोटी दर्शाती है । 25विश्लेषण के इस प्रकार के लिए ठेठ सटीकता रेंज के भीतर होने के नाते, इस SPI के माध्यम से ncAA के शामिल होने की पुष्टि (जंगली के लिए प्रायोगिक मूल्य प्रकार ECFP: २८,२८३.७ Da) ।
2 चित्रा प्रतिदीप्ति इमेजिंग माइक्रोस्कोपी (CFIM) जीवाणु कोशिकाओं के ECFP, EGFP, EYFP और GdFP पर पंजाबियों बफर में बैक्टीरिया के निलंबन पर व्यक्त की छवियों से पता चलता है । सभी छवियों के बारे में एक नमूना के लिए एक ही ऊर्जा पर एक यूवी उद्देश्य और लेजर उत्तेजना से सुसज्जित एक खुर्दबीन पर प्राप्त कर रहे थे ।
चित्रा 3 ए ई. कोलाई बैक्टीरिया की CFIM छवियों के एक उपरिशाई से पता चलता है GdFP सहित विभिंन एफपीएस व्यक्त, हमेशा बहुत समान उत्तेजना ऊर्जा के साथ निगरानी ( चित्रा 2में के रूप में तरंग दैर्ध्य) । चित्र बी में दिखाया गया FP वेरिएंट की chromophore संरचनाओं से पता चलता है । ECFP की तुलना में GdFP की चमक के संबंध में (प्रतिदीप्ति क्वांटम यील्ड φ = ०.४), EGFP (φ = ०.६) और EYFP (φ = ०.६) यह नोट करना महत्वपूर्ण है कि GdFP के लिए, प्रतिदीप्ति लाइट (30 एनएम) के एक व्यापक अधिग्रहण रेंज अन्य सभी एसपीई के लिए इस्तेमाल किया 20 एनएम के विपरीत में इस्तेमाल किया गया था cies, क्रम में समान मूल्यों के लिए छवियों की तीव्रता को समायोजित करने के लिए । एक थोड़ा कम विलुप्त गुणांक और अद्वितीय photophysical गुणों का एक परिणाम के रूप में एक कम मात्रा उपज के साथ, GdFP की चमक को दिखाया अंय एफपीएस की तुलना में कम है ।
ECFP (चित्रा 3सी) के अवशोषण स्पेक्ट्रम ४३४ एनएम और ४५२ एनएम में दो विशेषता maxima है । इसके विपरीत, GdFP ४६६ एनएम में अधिकतम के साथ एक व्यापक लाल-स्थानांतरित अवशोषण बैंड की विशेषता है । EGFP का अवशोषण आगे लाल है-४८८ एनएम के लिए स्थानांतरित कर दिया । हालांकि, ECFP (४१ एनएम) और EGFP (20 एनएम) की तुलना में GdFP (१०८ एनएम) की बहुत बड़ा स्टोक्स पारी के कारण, GdFP के उत्सर्जन स्पेक्ट्रम सबसे लाल सभी तीन GFP डेरिवेटिव यहां की जांच की स्थानांतरित (चित्रा 3 डी) है. जबकि ECFP के प्रतिदीप्ति उत्सर्जन ४७५ एनएम और ५०५ एनएम में दो विशेषता maxima से पता चलता है, EGFP एक व्यापक मुख्य उत्सर्जन ५०८ एनएम (λअधिकतम) ५४० एनएम पर एक मामूली कंधे के साथ पर नुकीला बैंड है । GdFP के प्रतिदीप्ति के बारे में ५६५ एनएम (λ अधिकतम पर प्रकट होताहै.) (चित्रा 3 डी) । इसके उत्सर्जन स्पेक्ट्रम में वाइल्ड-टाइप ECFP का छोटा सा योगदान होता है जो ४७५ एनएम पर छोटे कंधे के रूप में भी दिखाई देता है । यह छोटा ECFP अंश SPI कार्यविधि के दौरान प्रेरण से पहले संश्लेषित किया गया है, जैसा३३वर्णित है ।
चित्रा 3E GdFP के अवशोषण स्पेक्ट्रम में पीएच-निर्भर परिवर्तन दिखाता है । 8 से 5 के लिए एक पीएच परिवर्तन के लिए, उत्सर्जन अधिकतम लाल और एक मामूली अवशोषण बैंड का विस्तार करने के लिए थोड़ा बदलाव मनाया जाता है । हालांकि, अवशोषण आयाम की कमी पीएच 8 और पीएच 5 के बीच केवल 10% के बारे में है, यह दर्शाता है कि GdFP chromophore के जमीन राज्य संपत्तियों बहुत कमजोर पीएच द्वारा संशोधित कर रहे हैं ।
प्रतिदीप्ति उत्सर्जन एकल फोटॉन गिनती द्वारा मॉनिटर का समय हल चित्रा 4में दिखाया गया है । क्षय ५५० एनएम और ६०० एनएम (चित्रा 4a) पर केन्द्रित चैनल में निगरानी curves ६०० एनएम में ५५० एनएम पर क्षय की तुलना में थोड़ा तेजी से प्रतिदीप्ति क्षय प्रदर्शन । प्रतिदीप्ति क्षय के एक वैश्विक फिट के परिणामों के साथ दो घातीय घटकों में परिणाम प्रतिदीप्ति क्षय घटकों के साथ एक समय स्थिरांक के १.० ns और ३.३ ns (चित्र 4c और D) ।
GdFP के प्रतिदीप्ति उत्सर्जन दृढ़ता से पीएच पर निर्भर करता है, क्योंकि यह GFP परिवार के कई फ्लोरोसेंट प्रोटीन वेरिएंट के लिए विशिष्ट है । चित्रा 4B पीएच 5 और पीएच 8, जो स्पष्ट रूप से कम पीएच पर प्रतिदीप्ति तीव्रता में कमी से पता चलता है के बीच GdFP के प्रतिदीप्ति उत्सर्जन की तुलना करता है, जबकि वर्णक्रमीय विशेषताओं लगातार रहना ।
क्षय- संबंधित स्पेक्ट्रा (DAS)३९ of GdFP (आंकड़ा 4c और D) दो अलग उत्सर्जन बैंड की विशेषता है. धीमी गति से ३.३ एनएस घटक का योगदान अधिक तेजी से घटक (४०%) के मामूली योगदान के साथ ५५० एनएम (६०%)) के आसपास लघु तरंग दैर्ध्य रेंज में अधिक स्पष्ट है । ६०० एनएम पर, दोनों घटकों को एक ही आयाम के बारे में है । पीएच 7 (चित्रा 4c) से पीएच 6 (चित्रा 4d) से एक पारी पर, शायद ही दास की वर्णक्रमीय विशेषताओं को बदलने और वैश्विक फिटिंग दिनचर्या से समय स्थिरांक भी एक ही है (दास समय स्थिरांक की सटीकता ± ०.१५ एनएस के बारे में है) । हालांकि, दो दास घटकों के निरपेक्ष आयाम में अंतर स्पष्ट रूप से स्पष्ट है, जो पूरी तरह से चित्रा 4Bमें एक ही पीएच बदलाव पर कम प्रतिदीप्ति उत्सर्जन आयाम के लिए खातों ।
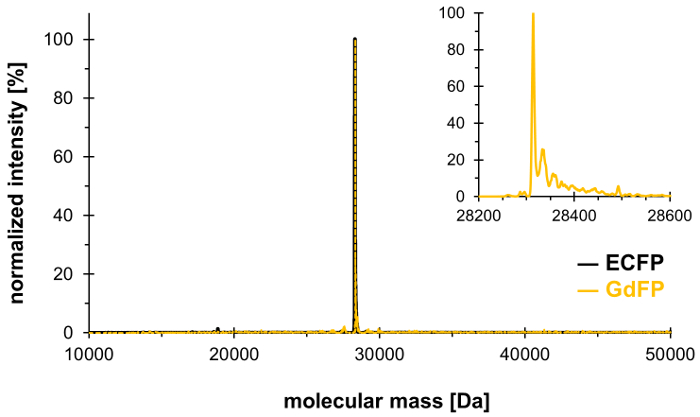
चित्रा 1: GdFP के प्रतिनिधि deconvoluted ईएसआई-एमएस स्पेक्ट्रम । GdFP के ईएसआई-एमएस स्पेक्ट्रम (सोने का रंग, बढ़ाया साजिश इनसेट के रूप में दिखाया गया है) २८३१४.१ दा पर एक मुख्य शिखर से पता चलता है (गणना मूल्य २८३१३.९ दा) । वाइल्ड-टाइप ECFP के लिए स्पेक्ट्रम ब्लैक में दिखाया गया है । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।

चित्रा 2: विभिंन एफपीएस व्यक्त बैक्टीरियल आबादी से फोकल प्रतिदीप्ति माइक्रोस्कोपिक छवियां । छवि प्राप्ति के लिए निम्न तरंग दैर्ध्य सेटिंग्स उपयोग किए गए थे: ECFP (λex = ४५७ एनएम, डिटेक्शन: 461-480 एनएम), EGFP (λex = ४८८ एनएम, डिटेक्शन: 495-515 एनएम), GDFP (λex = ४७६ एनएम, डिटेक्शन: 560-590 एनएम), EYFP (λex = ५१४ एनएम, पता लगाना: 520-530 एनएम) । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।

चित्रा 3: GdFP के वर्णक्रमीय गुण. (क) ECFP, EGFP और GdFP को व्यक्त करने वाले जीवाणु कोशिकाओं के मिश्रण की CFIM छवि पंजाब में बैक्टीरिया के निलंबन के बाद बफर. (ख) GdFP के Chromophore संरचनाओं (4-अमीनो-के साथ ६६ अवशेषों के स्थान पर टीआरपी), माता पिता का ECFP (६६ की स्थिति पर टीआरपी के साथ) और EFGP (Tyr के साथ स्थिति ६६ पर) । (ग) GdFP, ECFP और EGFP के सामान्यीकृत अवशोषण स्पेक्ट्रा की तुलना, जबकि (घ) में, ECFP के सामान्यीकृत प्रतिदीप्ति उत्सर्जन स्पेक्ट्रम (उत्तेजना पर ४३० एनएम) प्रतिदीप्ति और EGFP के GdFP उत्सर्जन स्पेक्ट्रा की तुलना में है (दोनों ४५० एनएम पर उत्साहित) । (ङ) पीएच-अवशोषण स्पेक्ट्रा की निर्भरता (२८० एनएम पर सामान्यीकृत अवशोषण) । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।

चित्रा 4: GdFP का समय-हल प्रतिदीप्ति. (क) GdFP के प्रतिदीप्ति क्षय समय-और तरंग दैर्ध्य-सुलझाया वर्णक्रमीय चैनल में ५५० एनएम और ६०० एनएम (± १२.५ एनएम) उत्तेजना के बाद ४७० एनएम लेजर दालों के साथ केंद्रित में गिनती फोटॉन । वाद्य प्रतिक्रिया फ़ंक्शन (IRF) उपयोग किए गए सेटअप के समय रिज़ॉल्यूशन के बारे में जानकारी प्रदान करता है । (ख) पीएच पर निर्भर GdFP के उत्सर्जन स्पेक्ट्रम की भिंनता (४६० एनएम पर उत्तेजना) । (C, D) क्षय-संबंधित स्पेक्ट्रा (दास) पीएच 7 (ग) और पीएच 6 (D) में GdFP के deconvolution के बाद निर्धारित समय-और तरंग दैर्ध्य-हल प्रतिदीप्ति क्षय और क्षय की वैश्विक फिटिंग के साथ दो घातीय कार्यों के एक वैश्विक सेट द्वारा सभी चैनलों में लिंक्ड समय स्थिरांक । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।

चित्र 5: ECFP (black) और GdFP (gold) chromophores के intramolecular प्रभारी अंतरण की संरचनाएं । ncAA के भाग के रूप में एक एमिनो समूह के अच्छे इलेक्ट्रॉन दाता द्वारा chromophore प्रणाली के आकार में वृद्धि अधिक mesomeric संरचनाओं के गठन के लिए उत्साहित राज्य के अनुनाद स्थिरीकरण प्राप्त करने के लिए सक्षम बनाता है । FP पाड़ को कनेक्शन अंक semicircles के रूप में दिखाए जाते हैं । कृपया यहां क्लिक करें इस आंकड़े का एक बड़ा संस्करण को देखने के लिए ।
| स्टॉक समाधान | एकाग्रता, विलायक | नोट | |
| 20% D-ग्लूकोज | २०० g/L D-ग्लूकोज ddH2O में | एक ०.४५ µm ताकना आकार सिरिंज फिल्टर के माध्यम से निस्पंदन द्वारा बंध्याकरण | |
| इण्डोल | isopropanol में ५० मिमी | ||
| 4-अमीनो-इण्डोल | 20% इथेनॉल में ५० मिमी (१०० मिलीलीटर के अंतिम खंड में 20 मिलीलीटर इथेनॉल ddH2O के साथ भरा हुआ) | ||
| IPTG | १ मी आपल्याकडे ddH२हे | ||
| L-tryptophan | 15 मिमी में भंग ddH2O का उपयोग कर 1 M hcl (चमचे के तहत एचसीएल dropwise जोड़ें जब तक पाउडर dissoved है) | ||
| lysozyme | ddH2O में ५० मिलीग्राम/एमएल | ||
| DNase I | 1 मिलीग्राम/एमएल ddH2में ओ | ||
| RNase एक | 1 मिलीग्राम/एमएल ddH2में ओ | ||
| Amp100 | १०० मिलीग्राम/एमएल एम्पीसिलीन में ddH2ओ | ||
| सोडियम-dodecylsulfate (एसडीएस) | २०० g/L में ddH2हे | ||
| अमोनियम सल्फेट ((NH4)2तो4) | १ मी आपल्याकडे ddH२हे | बंध्याकरण द्वारा autoclaving | |
| पोटेशियम dihydrogen फॉस्फेट (KH2पीओ4) | १ मी आपल्याकडे ddH२हे | बंध्याकरण द्वारा autoclaving | |
| डि-पोटेशियम हाइड्रोजन फॉस्फेट (K2HPO4) | १ मी आपल्याकडे ddH२हे | बंध्याकरण द्वारा autoclaving | |
| मैग्नीशियम सल्फेट (MgSO4) | १ मी आपल्याकडे ddH२हे | बंध्याकरण द्वारा autoclaving | |
| D-ग्लूकोज | १ मी आपल्याकडे ddH२हे | एक ०.४५ µm ताकना आकार सिरिंज फिल्टर के माध्यम से निस्पंदन द्वारा बंध्याकरण | |
| सोडियम क्लोराइड (NaCl) | 5 M मे ddH2हे | बंध्याकरण द्वारा autoclaving | |
| कैल्शियम क्लोराइड (CaCl2) | 1 g/L | एक ०.४५ µm ताकना आकार सिरिंज फिल्टर के माध्यम से निस्पंदन द्वारा बंध्याकरण | |
| आयरन (II) क्लोराइड (FeCl2) | 1 g/L | एक ०.४५ µm ताकना आकार सिरिंज फिल्टर के माध्यम से निस्पंदन द्वारा बंध्याकरण | |
| Thiamine | 10 g/L | एक ०.४५ µm ताकना आकार सिरिंज फिल्टर के माध्यम से निस्पंदन द्वारा बंध्याकरण | |
| बायोटिन | 10 g/L | एक ०.४५ µm ताकना आकार सिरिंज फिल्टर के माध्यम से निस्पंदन द्वारा बंध्याकरण | |
| ट्रेस तत्वों मिश्रण | कॉपर सल्फेट (CuSO4), जिंक क्लोराइड (ZnCl2), मैंगनीज क्लोराइड (MnCl2), अमोनियम molybdate ((NH4)2मू4); प्रत्येक 1 mg/L में ddH2O | एक ०.४५ µm ताकना आकार सिरिंज फिल्टर के माध्यम से निस्पंदन द्वारा बंध्याकरण | |
| 19 अमीनो एसिड मिक्स | 1.) भंग ०.५ g l-फेनिलएलनिन और ०.५ g l-tyrosine में १०० मिलि ddH2O के साथ dropwise के अलावा 1 M HCl के साथ चमचे के नीचे जब तक पाउडर घुल रहा है । | ||
| 2.) शेष एल अमीनो एसिड (एल-tryptophan को छोड़कर) में से प्रत्येक के ०.५ ग्राम बाहर वजन । 22 एमएल के फो 1 मीटर KH2पीओ4 और ४८ एमएल के 1 m K2HPO4के साथ मिक्स करें । ddH2O के बारे में ८०० मिलीलीटर जोड़ें । हलचल जब तक समाधान स्पष्ट हो जाता है । | |||
| 3.) चरण 1 से भंग l-फेनिलएलनिन और l-tyrosine जोड़ें.) और ddH2O के साथ वॉल्यूम को 1 L पर समायोजित करें । | |||
| 4.) एक बोतल शीर्ष फिल्टर इकाई के साथ वैक्यूम निस्पंदन द्वारा एमिनो एसिड मिश्रण निष्फल । | |||
| बफ़र्स और मीडिया | रचना/तयारी | ||
| एसडीएस लोड हो रहा है डाई बफर, 5x केंद्रित | ०.२५ m Tris pH ६.८, ५०% v/v ग्लिसरॉल, ०.२५% w/v bromphenol नीला, ०.५ m didhiothreitol (डीटीटी; वै 5% β-mercaptoethanol), 10% w/v सोडियम-dodecylsulfate (एसडीएस) | ||
| बाइंडिंग बफ़र | ५० मिमी सोडियम dihydrogenphosphate (2PO4), ५०० मिमी NaCl, 10 मिमी imidazole, पीएच 8 | ||
| रेफरेंस बफर | ५० mm सोडियम dihydrogenphosphate (2PO4), ५०० mm NaCl, २५० mm imidazole, पीएच 8 | ||
| डायलिसिस बफर | ५० मिमी सोडियम dihydrogenphosphate (णः2PO4), १५० mm NaCl, १०० मिलीलीटर/L ग्लिसरॉल, पीएच 8 | ||
| MS बफ़र | 10 एमएम Tris-एचसीएल, पीएच 8 | ||
| l-tryptophan (NMM19) के अलावा 19 l-अमीनो एसिड युक्त नई न्यूनतम मध्यम | मिश्रण सभी स्टॉक समाधान निंनलिखित अंतिम सांद्रता प्राप्त करने के लिए: ७.५ mM (NH4)2तो4, १.७ मिमी NaCl, 22 मिमी KH2पीओ4, ५० मिमी कश्मीर2HPO4, 1 मिमी MgSO4, 20 मिमी डी-ग्लूकोज, ५० मिलीग्राम/ 19 अमीनो एसिड मिक्स, 1 µ g/l CaCl2, 1 µ g/l FeCl2, 10 µ g/l thiamine, 10 mg/l बायोटिन, ०.०१ mg/l ट्रेस तत्वों मिश्रण | ||
| पौंड मध्यम | रचना: 10 g/l tryptone, 5 g/l खमीर निकालें, 10 g/l NaCl, pH ७.० में ddH2O | ||
| तैयारी: | |||
| 1.) बाहर ५० ग्राम tryptone, 25 ग्राम खमीर निकालने, 5 जी NaCl एक 1 एल कांच की बोतल में वजन । | |||
| 2.) ddH2O जोड़ें ~ ८०० मिलीलीटर और सरगर्मी के तहत घटकों को भंग । | |||
| 3.) पीएच को मापने और 1 मीटर एचसीएल या 1 मीटर NaOH, यदि आवश्यक हो तो के अलावा dropwise द्वारा पीएच 7 को समायोजित करें । 1 L तक ddH2O जोड़ें । | |||
| 4.) autoclaving द्वारा निष्फल, बाद में मात्रा घटाने के लिए जाँच करें और यदि आवश्यक हो तो क्षतिपूर्ति करने के लिए बाँझ ddH2जोड़ें । उपयोग करने तक 4 डिग्री सेल्सियस पर स्टोर । | |||
| पौंड आगर प्लेट्स | रचना: 10 g/l tryptone, 5 g/l खमीर निकालें, 10 g/l NaCl, 15 g/l आगर-आगर, पीएच ७.० में ddH2हे | ||
| तैयारी: | |||
| 1.) ५० ग्राम tryptone, 25 ग्राम खमीर निकालने, 5 ग्राम NaCl, ७.५ ग्राम आगर-आगर एक 1 एल ग्लास बोतल में बाहर तौलना । | |||
| 2.) जोड़ें ddH2O अप करने के लिए ५०० मिलीलीटर और सरगर्मी के तहत घटकों को भंग । | |||
| 3.) पीएच को मापने और 1 मीटर एचसीएल या 1 मीटर NaOH, यदि आवश्यक हो तो के अलावा dropwise द्वारा पीएच 7 को समायोजित करें । 1 L तक ddH2O जोड़ें । | |||
| 4.) autoclaving द्वारा निष्फल, बाद में मात्रा घटाने के लिए जाँच करें और बाँझ ddH2हे को क्षतिपूर्ति करने के लिए, यदि आवश्यक जोड़ें । (नोट: पौंड आगार प्लेटों की तैयारी के लिए उपयोग करने तक 4 डिग्री सेल्सियस पर संग्रहित किया जा सकता है । ध्यान से पिघल जम आगर एक माइक्रोवेव का उपयोग) | |||
| 5.) जब समाधान अभी भी गर्म है (30-40 ° c), १०० µ g/एमएल के अंतिम एकाग्रता के लिए एम्पीसिलीन जोड़ें | |||
| 6.) 5 कदम से तरल के बारे में 15 मिलीलीटर डालो.) बाँझ शर्तों के तहत एक बाँझ 10 सेमी पेट्री डिश में । जब आगर जम जाता है, प्लेटें 4 डिग्री सेल्सियस पर 1 सप्ताह के लिए उपयोग तक संग्रहित किया जा सकता है । | |||
| फास्फेट-बफर खारा (पंजाब) | रचना: १३७ मिमी NaCl, २.७ मिमी KCl, 10 मिमी ना2HPO4, १.८ मिमी KH2पीओ4, 1 मिमी CaCl2, ०.५ मिमी MgCl2, पीएच 7. autoclaving या निस्पंदन द्वारा निष्फल । | ||
तालिका 1: स्टॉक समाधान और बफ़र ।
Discussion
बहुत उच्च ncaa शामिल क्षमता को प्राप्त करने के लिए, auxotrophy आधारित SPI विधि चयापचय इंजीनियर मेजबान कोशिकाओं है, जो ncAA के इसी प्राकृतिक समकक्ष संश्लेषित करने में सक्षम नहीं है के उपयोग पर निर्भर करता है । ई. कोलाईके लिए, इस तरह के उपभेदों आसानी से उपलब्ध हैं । यहां तक कि एक ही प्रोटीन में एकाधिक ncaas के एक साथ शामिल व्यवहार्य multiauxotrophic उपभेदों का उपयोग कर रहा है । अवशेषों-प्रतिस्थापन के विशिष्ट मोड और रासायनिक प्रदर्शनों की तुलना में इसी तरह के रासायनिक सादृश्यों को प्रतिबंधित किया जा रहा कमियां के रूप में देखा जा सकता है । फिर भी, प्रोटीन वेरिएंट की एक बड़ी संख्या प्राकृतिक बैक्टीरियल अनुवाद तंत्र के रूप में उत्पादित किया जा सकता है कई एमिनो एसिड अनुरूप सहन । उदाहरण के लिए, अधिक से अधिक ५० ncaa प्रोटीन में शामिल किया जा सकता है इन विट्रो अनुवाद का उपयोग कर, आनुवंशिक कोड के सभी codons के बारे में ७३% के लिए लेखांकन के लिए reपून४०के लिए उपलब्ध है । इसके अलावा, SPI भी लक्ष्य प्रोटीन४१के कुशल multisite लेबलिंग की अनुमति दे सकते हैं । सिद्धांत रूप में, SPI पद्धति ई. कोलाईतक ही सीमित नहीं है, लेकिन किसी भी अंय मेजबान में काम कर सकते है और विहित 20 अमीनो एसिड में से प्रत्येक के लिए, बशर्ते कि auxotrophic उपभेदों और परिभाषित खेती मीडिया उपलब्ध हैं । उदाहरण के लिए, दो methionine अनुरूप, azidohomoalanine (अहा) और homopropargylglycine (Hpg), व्यावसायिक रूप से उपलब्ध है और विविध जीवों में प्रोटीन और proteomes लेबलिंग के लिए इस्तेमाल किया । इसके अलावा, अहा intracellularly उत्पादन किया जा सकता है और बाद में प्रोटीन में शामिल४२। इस ncAA विशेष रूप से Tirrel और सहकर्मियों द्वारा विकसित के रूप में क्लिक करें रसायन विज्ञान के रूप में bioorthogonal conjugations के लिए उपयुक्त है: उदाहरण के लिए, संयंत्र ऊतक में Arabidopsis थालियाना, में Bombyx मोरी लार्वा४३, Drosophila कोशिकाओं४४, लार्वा zebrafish४५ साथ ही स्तनधारी कोशिकाओं ंयूरॉंस४६सहित, प्रोटीन अहा४७,४८के साथ लेबल किया जा सकता है । इसी तरह, trp एनालॉग को सफलतापूर्वक trp में रोगाणुरोधी पेप्टाइड्स में शामिल किया गया था-auxotrophic Lactococcus लैकि्टस उपभेदों४९. SPI भी Xenobiology५०,५१के क्षेत्र के लिए उपयोगी है, जो बुनियादी रासायनिक मेकअप के लिए विकल्प की पड़ताल है जीवन का । उदाहरण के लिए, ई. कोलाई५२ और B. सबटिलिस५३पर पिछले काम पर आधारित है, एक ई. कोलाई तनाव हाल ही में चयनात्मक के बजाय thienopyrrole का उपयोग करने के लिए दबाव के साथ एक विकासवादी रणनीति द्वारा विकसित किया गया था इण्डोल, आनुवंशिक कोड५४में thienopyrrole-alanine द्वारा tryptophan के proteome-वाइड प्रतिस्थापन में जिसके परिणामस्वरूप । आम तौर पर, विहित एमिनो एसिड टीआरपी, जो एक एकल triplet (UGG) द्वारा इनकोडिंग है, प्रोटीन इंजीनियरिंग के लिए एक होनहार लक्ष्य इण्डोल रसायन विज्ञान है, जो कई रासायनिक रूपांतरों प्रदान करता है के अमीर पहलुओं के कारण प्रस्तुत करता है । हाल ही में, और SPI आधारित शामिल करने के लिए एक विकल्प के रूप में, एक उपंयास SCS मंच टीआरपी अनुरूप साइट को शामिल करने में सक्षम-विशेष रूप से दोनों बैक्टीरियल और eukaryotic मेजबान में बताया गया है५५। यह आगे vivo ncAA-आधारित प्रोटीन इंजीनियरिंग, वर्णक्रमीय गुणों के परिवर्तन सहित में उपकरण बॉक्स का विस्तार ।
auxotrophic अभिव्यक्ति मेजबान के उपयोग के अलावा, SPI प्रोटोकॉल सख्त किण्वन की स्थिति की आवश्यकता है, दोनों लक्ष्य अभिव्यक्ति समय और माध्यम की संरचना के संदर्भ में आदेश उच्च ncAA निगमन दक्षता और लक्ष्य प्रोटीन यील्ड तक पहुंचने के लिए ५६. खेती रासायनिक परिभाषित ंयूनतम मीडिया है, जो अनिवार्य रूप से प्रमुख लवण नाइट्रोजन (अमोनियम नमक) और कार्बन (डी ग्लूकोज), विटामिन और तत्वों का पता लगाने के लिए सूत्रों के अलावा शामिल का उपयोग कर आयोजित किया जाता है । हालांकि सख्ती से आगे auxotrophies के अभाव में आवश्यक नहीं है, शेष अमीनो एसिड (20-n, यदि n अमीनो एसिड को प्रतिस्थापित किया जा रहे हैं) सामांयतः जीवाणु विकास को बढ़ावा देने के लिए जोड़ रहे है५७। लक्ष्य प्रोटीन अभिव्यक्ति की प्रेरण से पहले एक प्रारंभिक विकास चरण के दौरान, प्रतिस्थापित किया जा करने के लिए n विहित अमीनो एसिड सांद्रता सीमित में जोड़ रहे हैं । सेलुलर विकास आय जब तक लक्षित आवश्यक अमीनो एसिड समाप्त हो रहे हैं, के रूप में प्रयोग एक स्थिर आयुध डिपो६००द्वारा संकेत दिया । बाद में, संस्कृति माध्यम ताजा माध्यम है कि घट एमिनो एसिड की कमी है और प्रचुर मात्रा में सांद्रता में ncAA शामिल द्वारा प्रतिस्थापित किया जाता है । इस प्रोटोकॉल में दिखाए गए के रूप में tryptophan सादृश्यों के राइबोसोमल के लिए, एक इण्डोल अनुरूप खिलाया जाता है, जो tryptophan tryptophan५८द्वारा व्युत्पंन इसी सिंथेस में परिवर्तित intracellularly हो जाता है । अगले, लक्ष्य प्रोटीन अभिव्यक्ति प्रेरित है । इस स्तर पर, कोशिकाओं को लघुगणकीय वृद्धि के अंत के करीब हैं, कुल सेल संख्या और फिटनेस के बीच एक संतुलन के रूप में । उपस्थिति और विहित अमीनो की निगमन के रूप में जंगली प्रकार के प्रोटीन का उत्पादन करने के लिए नेतृत्व करेंगे, यह सुनिश्चित करने के लिए महत्वपूर्ण है कि आवश्यक अमीनो एसिड पूरी तरह से प्रेरण से पहले समाप्त हो गया है । इसी तरह, यह, सामांयतः मास स्पेक्ट्रोमेट्री द्वारा लक्ष्य प्रोटीन में ncAA निगमन की दक्षता की जांच करने के लिए अनिवार्य है । विहित अमीनो एसिड की पर्याप्त उपस्थिति के मामले में, खेती की स्थिति को समायोजित करने की जरूरत है, जैसे, प्रारंभिक विकास चरण या बाद की अवधि के लिए आवश्यक अमीनो एसिड (ओं) की एकाग्रता में फेरबदल करके । ncaa की दिशा में कम aaRS गतिविधि के मामले में, अंतर्जात एंजाइम या सह के व्यक्ती एक अलग aaRS की अभिव्यक्ति है, जो ncaa की दिशा में अधिक सक्रिय है,५९आयोजित किया जा सकता है ।
विहित एमिनो एसिड टीआरपी तीन उल्लेखनीय सुविधाओं के साथ संपंन है: (i) प्रोटीन में अपनी प्राकृतिक बहुतायत कम है; (ii) इसके जैव भौतिक और रासायनिक गुण अद्वितीय है (उदा., यह आमतौर पर प्रोटीन और पेप्टाइड्स के आंतरिक प्रतिदीप्ति का प्रमुख मूल है), और (iii) यह जैव रासायनिक बातचीत और कार्यों की एक किस्म के लिए योगदान देता है सहित π-स्टैकिंग, एच-बांडिंग और कटियन-π इंटरैक्शन. इन सभी सुविधाओं को मौलिक टीआरपी → 4 पर बदल रहे है GdFP में अमीनो-टीआरपी प्रतिस्थापन । संदेह से परे, एक "सोने" avGFPs के वर्ग के डिजाइन इंजीनियरिंग दर्जी के लिए एक उल्लेखनीय उदाहरण है-फ्लोरोसेंट प्रोटीन बनाया । अलग वर्णक्रमीय गुण के साथ, एफपीएस mutagenesis और ncAA निगमन के माध्यम से कुछ वर्णक्रमीय खिड़कियों की ओर देखते किया जा सकता है । GdFP के मामले में, इस ECFP chromophore त्रय में निहित इण्डोल अंगूठी के फ्रेम में एक सरल रासायनिक मुद्रा एच → एनएच2 द्वारा पूरा किया जाता है. चित्रा 5 chromophore के भीतर ncAA निगमन के प्रभाव को प्रदर्शित करता है. इलेक्ट्रॉन का परिचय-4 से उत्पंन समूह दान-अमीनो-इण्डोल (intracellularly 4 में परिवर्तित-अमीनो-टीआरपी) mesomeric संरचनाओं की एक किस्म है कि एक स्थिर उत्तेजित राज्य की व्याख्या कर सकते है सक्षम बनाता है । Spectroscopically, इसके बढ़े स्टोक्स बदलाव और विस्तारित संयुग्मित प्रणाली के इन विशिष्ट गुणों से लाल-खिसका प्रतिदीप्ति उत्सर्जन का परिणाम है । के रूप में पहले की रिपोर्ट, GdFP chromophore के भीतर बढ़ाया intramolecular प्रभारी हस्तांतरण अंतर्निहित पीएच के प्रति संवेदनशील है (चित्रा 4B) और एस के बीच द्विध्रुवीय पल में एक बड़ा परिवर्तन के साथ0 जमीन और एस1 उत्तेजित राज्य ECFP३३के सापेक्ष । वैकल्पिक इलेक्ट्रॉन के रूप में समूह दान, tryptophan एक इण्डोल hydroxy समूहों के साथ प्रतिस्थापित अंगूठी असर अनुरूप इस्तेमाल किया जा सकता है, के रूप में मॉडल प्रोटीन barstar४१के साथ एक तुलनात्मक अध्ययन में सूचना दी ।
अवशोषण और GdFP के प्रतिदीप्ति स्पेक्ट्रा ECFP और EGFP की तुलना में व्यापक हैं (चित्र 3 सी और डी). सजातीय अवशोषण और प्रतिदीप्ति बैंड का विस्तार आम तौर पर chromophore में कंपन मोड के कारण होता है और, इसके अलावा, chromophore के युग्मन द्वारा आगे कंपन प्रोटीन में मौजूद मोड के लिए६०। स्थानीय प्रोटीन पर्यावरण युग्मन आरोप chromophore पर स्थानीय द्वारा समर्थित है । प्रोटीन की संरचनात्मक सजातीयता के रूप में vibronic स्पेक्ट्रम के स्थानीय रूपों की ओर जाता है, chromophore के vibronic स्पेक्ट्रा और प्रोटीन के बाकी हिस्सों के बीच इस तरह के युग्मन प्रभारी स्थानीयकरण और mesomeric राज्यों द्वारा समर्थित के रूप में संकेत दिया चित्र 5। इस युग्मन भी बड़े स्टोक्स बदलाव का समर्थन करता है और जरूरी प्रतिदीप्ति क्वांटम उपज कम कर देता है । अंय red-स्थानांतरित एफपीएस की तुलना में, GdFP भी बेहतर प्रोटीन स्थिरता और एकत्रीकरण के लिए एक कम प्रवृत्ति३३,६१,६२दर्शाती है । यह न केवल अंय FP वेरिएंट से रंग में अलग है, लेकिन यह भी एक काफी वृद्धि हुई thermostability और बढ़ाया सहकारी तह३३प्रदर्शित । इसकी प्रतिदीप्ति तीव्रता कम से ९०% ६० डिग्री सेल्सियस करने के लिए हीटिंग पर संरक्षित है, जबकि ECFP प्रतिदीप्ति के बारे में 30% करने के लिए कम है । प्रोटीन में, खुशबूदार एमिनो एसिड अक्सर पक्ष श्रृंखला है, जो सामांयतः है प्रोटीन तृतीयक संरचना पर एक स्थिर प्रभाव है बातचीत के नेटवर्क के लिए योगदान करते हैं । avGFP बंदरगाह ऐसे एक पक्ष श्रृंखला नेटवर्क है, जो खुद chromophore के होते हैं, साथ ही साथ Phe-१६५, अपने-१४८, और Tyr-१४५ । इन साइड चेन न केवल GdFP संरचना३३में काफी कठोर हैं, लेकिन महत्वपूर्ण बात, वे chromophore के साथ hydrophobic संपर्क फार्म । सबसे प्रमुख उपंयास GdFP में पहचान की सुविधा है कि aminated chromophore Phe-१६५ के लिए और अधिक समीपस्थ है । इस बातचीत के अंय ज्ञात avGFPs में एक विशेषता नहीं देखा है । के रूप में दो अवशेषों 3.2-4.5 Å अलग कर रहे हैं, एमिनो खुशबूदार बातचीत भी मौजूद हो सकता है । chromophore के अमीन प्रेरित अनुनाद स्थिरीकरण के साथ एक साथ, इन सबसे अधिक संभावना एक सहकारी फैशन में अमीनो एसिड के इस hydrophobic नेटवर्क को स्थिर । एक अधिक प्रभावी intramolecular प्रभारी स्थानांतरण chromophore की जमीनी स्थिति की तुलना में उत्साहित राज्य में इन बातचीत के द्वारा समर्थित हो सकता है, और यह कम से १०८ एनएम स्टोक्स के लिए आंशिक रूप से खातों पारी३३,६२ .
fluorophore गुणों के तर्कसंगत डिजाइन में, स्थानीय π-प्रणाली के आकार में वृद्धि एक लाल-उत्तेजना तरंग दैर्ध्य में परिणाम की भविष्यवाणी की है । अंगूठे का यह नियम ६६ स्थिति में एमिनो एसिड की श्रृंखला द्वारा माना जाता है तटस्थ chromophores के लिए अग्रणी: Phe (λmax = ३५५ एनएम) < आमा (λma x= ३८६ एनएम) < Tyr (λmax = ३९५ एनएम) < Trp (λmax = ४३६ एनएम)६३. प्रकृति में π-बांड की chromophore की संयुग्मित प्रणाली के इस विस्तार को विभिन्न रणनीतियों से प्राप्त किया गया है । Discosoma striataसे DsRed के लिए, यह एक अतिरिक्त एमिनो एसिड के एकीकरण के द्वारा बढ़ाया है, इस प्रकार λअधिकतम ५७३ एनएम६४स्थानांतरण । Anemonia sulcata से asFP595 (λmax = ५९५ एनएम) का chromophore एक imino समूह द्वारा विस्तारित किया गया था, जो उसके π-सिस्टम६५का विस्तार कर रहा है । GdFP और अंय avFPs के chromophore के बाद से एक ही आकार का है, एक अलग सिद्धांत को विस्तारित DsRed और asFP595 chromophores की सीमा में एक उत्सर्जन तरंग दैर्ध्य आवश्यक जरूरत है । १०८ एनएम के गहन स्टोक्स बदलाव GdFP chromophore, जो फ्लोरोसेंट प्रोटीन के डिजाइन में एक नया photophysical सिद्धांत से पता चलता है की अलग संरचना के लिए जिंमेदार ठहराया है । प्रारंभिक गणना (के रूप में ६२में रिपोर्ट) से पता चला है कि उत्तेजित-राज्य GdFP के chromophore के द्विध्रुवीय क्षण काफी जमीनी राज्य की तुलना में बड़ा है, ECFP के संबंधित मूल्यों के विपरीत । जबकि GdFP के द्विध्रुवीय पल ~ 3 डी से बढ़ जाती है (Debye) एस में ~ 15 d करने के लिए s 1में, ECFP chromophore के लिए परिवर्तन नहीं बल्कि मध्यम था (से ~ 4 डी से ~ 6 डी) । इस प्रकार, GdFP के अद्वितीय गोल्डन प्रतिदीप्ति chromophore के भीतर पर्याप्त intramolecular प्रभारी हस्तांतरण के कारण होता है, जो संभव mesomeric संरचनाओं की विविधता बढ़ जाती है ( चित्रा 5देखें) कि अनुनाद स्थिरीकरण के लिए अनुमति देते हैं । इससे जो उत्सर्जन होता है उससे एनर्जी लेवल कम होता है । उत्तेजना पर द्विध्रुवीय पल में गहरा परिवर्तन का एक परिणाम के रूप में, intramolecular प्रभारी जुदाई chromophore वातावरण की इलेक्ट्रोस्टैटिक क्षमता में परिवर्तन के लिए मुख्य कारण है । आसपास के प्रोटीन मैट्रिक्स, बदले में, chromophore उत्तेजना के बाद प्रभारी वितरण में परिवर्तन करने के लिए समायोजित कर देता है । बाद के संरचनात्मक छूट उत्साहित chromophore के ऊर्जा स्तर को कम करती है, जो अपने चार्ज हस्तांतरण चरित्र के कारण लाल करने के लिए प्रतिदीप्ति स्पेक्ट्रम पाली । एक ही कारण के लिए, बड़े स्टोक्स बदलाव और विकिरण प्रक्रियाओं की बढ़ी दरों की एक परिणाम के रूप में, GdFP की प्रतिदीप्ति क्वांटम उपज ECFP३३की तुलना में कम है ।
उच्च क्वांटम उपज और ECFP और EGFP के छोटे स्टोक्स बदलाव आमतौर पर chromophore के एक कठोर प्रोटीन पर्यावरण के लिए जिंमेदार माना हैं, जो स्वतंत्रता की डिग्री कम कर देता है और, नतीजतन, आंतरिक रूपांतरण उत्साहित राज्य के radiating छूट एहसान करने के लिए ६६. नतीजतन, शेष प्रोटीन मैट्रिक्स को कम युग्मन के साथ अधिक कठोरता से एंबेडेड chromophores के आणविक डिजाइन एक गाइड के रूप में सेवा के लिए आगे लाल उच्च प्रतिदीप्ति क्वांटम उपज के साथ GFP डेरिवेटिव स्थानांतरित हो सकता है । इसलिए, आगे इंजीनियरिंग दृष्टिकोण के लिए लाल रंग का उत्पादन करने के लिए-फ्लोरोसेंट प्रोटीन, π-इलेक्ट्रॉन प्रणाली की वृद्धि और कमजोर युग्मन के साथ एक कठोर chromophore संरचना प्रोटीन पर्यावरण के लिए अत्यधिक वांछनीय है । इस तरह के संशोधनों को भी या तो सीधे GFP में शुरू किया जा सकता है आधारित chromophores या chromophore आसपास के क्षेत्र में वांछित ncaas के स्थान से ।
Disclosures
लेखकों की घोषणा है कि वे कोई प्रतिस्पर्धा वित्तीय हितों की है ।
Acknowledgments
यह काम जर्मन रिसर्च फाउंडेशन द्वारा समर्थित किया गया था (उत्कृष्टता के क्लस्टर "Catalysis में अवधारणाओं को एकीकृत) T.F. और N.B. के लिए और संघीय शिक्षा और विज्ञान मंत्रालय के द्वारा (BMBF कार्यक्रम" HSP २०२० ", टू-WIMIplus परियोजना SynTUBio) को एफ.-J.S.
Materials
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| 4-aminoindole | Sigma-Aldrich | 525022 | |
| acetonitrile | VWR | HiPerSolv CHROMANORM ULTRA for LC-MS, 83642 | LC-MS grade required |
| agar-agar | Carl Roth | 5210 | |
| ammonium molybdate ((NH4)2MoO4) | Sigma-Aldrich | 277908 | |
| ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| ampicillin sodium salt | Carl Roth | K029 | |
| biotin | Sigma-Aldrich | B4501 | |
| bromophenol blue | Sigma-Aldrich | B0126 | |
| calcium chloride (CaCl2) | Sigma-Aldrich | C5670 | |
| colloidal silica | Sigma-Aldrich | Ludox HS-40, 420816 | |
| Coomassie Brillant Blue R 250 | Carl Roth | 3862 | |
| copper sulfate (CuSO4) | Carl Roth | CP86.1 | |
| D-glucose | Carl Roth | 6780 | |
| di-sodium hydrogen phosphate (Na2HPO4) | Carl Roth | X987 | |
| di-potassium hydrogen phosphate (K2HPO4) | Carl Roth | P749.1 | |
| 1,4-dithiothreitol (DTT) | Carl Roth | 6908 | |
| DNase I | Sigma-Aldrich | D5025 | |
| ethanol | Carl Roth | 9065.1 | |
| formic acid | VWR | HiPerSolv CHROMANORM for LC-MS, 84865 | LC-MS grade required |
| glycerol | Carl Roth | 3783 | |
| imidazole | Carl Roth | X998 | |
| indole | Sigma-Aldrich | I3408 | |
| iron(II) chloride (FeCl2) | Sigma-Aldrich | 380024 | |
| isopropanol | Carl Roth | AE73.1 | |
| isopropyl β-D-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I6758 | |
| lysozyme | Sigma-Aldrich | L6876 | |
| magnesium chloride (MgCl2) | Carl Roth | KK36.1 | |
| magnesium sulfate (MgSO4) | Carl Roth | 8283.2 | |
| manganese chloride (MnCl2) | Sigma-Aldrich | 63535 | |
| β-mercaptoethanol | Carl Roth | 4227.3 | |
| potassium chloride (KCl) | Carl Roth | 6781.3 | |
| potassium dihydrogen phosphate (KH2PO4) | Sigma-Aldrich | P5655 | |
| RNase A | Carl Roth | 7156 | |
| sodium chloride (NaCl) | Carl Roth | P029 | |
| sodium dihydrogen phosphate (NaH2PO4) | Carl Roth | T879 | |
| sodium dodecyl sulphate (NaC12H25SO4) | Carl Roth | 0183 | |
| thiamine | Sigma-Aldrich | T4625 | |
| Tris(hydroxymethyl)-aminomethane (Tris) | Carl Roth | 5429 | |
| Tris hydrochloride (Tris-HCl) | Sigma-Aldrich | 857645 | |
| tryptone | Carl Roth | 8952 | |
| yeast extract | Carl Roth | 2363 | |
| zinc chloride (ZnCl2) | Sigma-Aldrich | 229997 | |
| Name | Company | Catalog Number | Comments |
| amino acids | |||
| L-alanine | Sigma-Aldrich | A7627 | |
| L-arginine | Sigma-Aldrich | A5006 | |
| L-asparagine | Sigma-Aldrich | A8381 | |
| L-aspartic acid | Sigma-Aldrich | A0884 | |
| L-cysteine | Sigma-Aldrich | C7352 | |
| L-glutamic acid | Sigma-Aldrich | G2128 | |
| L-glutamine | Sigma-Aldrich | G3126 | |
| L-glycine | Sigma-Aldrich | G7126 | |
| L-histidine | Sigma-Aldrich | H8000 | |
| L-isoleucine | Sigma-Aldrich | I2752 | |
| L-leucine | Sigma-Aldrich | L8000 | |
| L-lysine | Sigma-Aldrich | L5501 | |
| L-methionine | Sigma-Aldrich | M9625 | |
| L-proline | Sigma-Aldrich | P0380 | |
| L-phenylalanine | Sigma-Aldrich | P2126 | |
| L-serine | Sigma-Aldrich | S4500 | |
| L-threonine | Sigma-Aldrich | T8625 | |
| L-tryptophan | Sigma-Aldrich | T0254 | |
| L-tyrosine | Sigma-Aldrich | T3754 | |
| L-valine | Sigma-Aldrich | V0500 | |
| Name | Company | Catalog Number | Comments |
| Lab materials | |||
| 0.45 µm syringe filter with PVDF membrane | Carl Roth | CCY1.1 | |
| 1.5 mL microcentrifuge tubes | Eppendorf | 30120086 | |
| conical polystyrene (Falcon) tubes, 50 mL | Fisher Scientific | 14-432-22 | |
| Luer-Lock syringe 5 mL | Carl Roth | EP96.1 | |
| dialysis membrane, Molecular Weight Cut-Off (MWCO) 5,000 | Spectrum Medical Industries | Spectra/Por MWCO 5000 dialysis membrane, 133198 | |
| Immobilized Metal ion Affinity Chromatography (IMAC) column 1 mL, Ni-NTA | Macherey Nagel | Protino series, 745410.5 | |
| petri dishes (polystyrene, sterile) | Carl Roth | TA19 | |
| pQE-80L plasmid vector | Qiagen | no longer available | replaced by N-terminus pQE Vector set Cat No./ID: 32915 |
| protein extraction reagent BugBuster | EMB Millipore | 70921-4 | |
| round-bottom polystyrene tubes, 14 mL | Fisher Scientific | Corning Falcon, 14-959-1B | |
| Trp-auxotrophic E. coli strain | ATCC | ATCC 49980 | Bridges BA et al., Chem Biol Interact., 1972, 5(2):77-84; see main text for alternatives |
| Name | Company | Catalog Number | Comments |
| Mass Spectrometry equipment | |||
| mass spectrometer for LC-ESI-TOF-MS | Agilent | Agilent 6530 Accurate-Mass QTOF | coupled with Infinity LC system |
| mass spectrometry data analysis software | Agilent | MassHunter Qualitative Analysis software v. B.06.00 | |
| High-Performance Liquid Chromatography (HPLC) column for LC-ESI-TOF-MS | Sigma-Aldrich | Supelco Discovery BIO Wide Pore C5 HPLC column, 3 µm particle size, 10 cm x 2.1 mm | |
| HPLC autosampler vials 1.5 mL | Sigma-Aldrich | Supelco 854165 | with conical 0.1 mL glass inserts, screw caps and septa |
| Name | Company | Catalog Number | Comments |
| General equipment | |||
| benchtop centrifuge for 1.5 mL Eppendorf tubes | Eppendorf | 5427 R | |
| cooling centrifuge for 50 mL Falcon tubes | Eppendorf | 5810 R | |
| high pressure microfluidizer for bacterial cell disruption | Microfluidics | LM series with “Z” type chamber | |
| peristaltic pump for LC | GE Healthcare | P-1 | |
| Fast Protein Liquid Chromatography (FPLC) system | GE Healthcare | ÄKTA pure 25 L | |
| orbital shaker for bacterial cultivation | Infors HT | Minitron | |
| UV/Vis spectrophotometer | Biochrom | ULTROSPEC 2100 | |
| ultrasonic homogenizer for bacterial cell disruption | Omnilab | Bandelin SONOPULS HD 3200, 5650182 | with MS72 sonifier tip |
| Name | Company | Catalog Number | Comments |
| Fluorescence spectroscopy equipment | |||
| ps-pulsed laser 470 nm | Picoquant GmbH | PDL-470 | |
| time- and wavelength-correlated single photon counting (TWSPC) acquisition software | Picoquant GmbH | SymPhoTime 64 | |
| time- and wavelength-correlated single photon counting (TWSPC) detector | Picoquant GmbH | PML-16C | 16 spectral channels, to be selected by grating settings |
| single photon counting software | Picoquant GmbH | SPCM 9.75 | |
| global fitting software | Picoquant GmbH | SPC2Glo(R) | |
| fluorescence decay data analysis software | Picoquant GmbH | FluoFit program | |
| data analysis software | OriginLab Inc. | Origin 9.2 | |
| neutral density filter set | Schott | NG1 to NG11 | (400 - 650 nm, transmission 50 %, 20%, 10 %, 5 %) |
| 488 nm long-pass emission filter | AHF Analysentechnik | AHF-488 | |
| quartz cuvette | Thorlabs GmbH | CV10Q1400 | 1 cm pathlength |
References
- Shimomura, O., Johnson, F. H., Saiga, Y. Extraction, Purification and Properties of Aequorin, a Bioluminescent Protein from the Luminous Hydromedusan, Aequorea. J Cell Compar Physl. 59 (3), 223-239 (1962).
- Chalfie, M., Tu, Y., Euskirchen, G., Ward, W. W., Prasher, D. C. Green fluorescent protein as a marker for gene expression. Science. 263 (5148), 802-805 (1994).
- Andresen, M., et al. Structure and mechanism of the reversible photoswitch of a fluorescent protein. P Natl Acad Sci USA. 102 (37), 13070-13074 (2005).
- Andresen, M., et al. Structural basis for reversible photoswitching in Dronpa. P Natl Acad Sci USA. 104 (32), 13005-13009 (2007).
- Brakemann, T., et al. A reversibly photoswitchable GFP-like protein with fluorescence excitation decoupled from switching. Nat Biotechnol. 29 (10), 942-947 (2011).
- Kremers, G. -J., Gilbert, S. G., Cranfill, P. J., Davidson, M. W., Piston, D. W. Fluorescent proteins at a glance. J Cell Sci. 124 (Pt 2), 157-160 (2011).
- Shimomura, O. Structure of the chromophore of aequorea 0. shimomura green fluorescent protein. FEBS Lett. 104 (2), 220-222 (1979).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N. G., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Shcherbo, D., et al. Bright far-red fluorescent protein for whole-body imaging. Nat Methods. 4 (9), 741-746 (2007).
- Shcherbakova, D. M., Subach, O. M., Verkhusha, V. V. Red fluorescent proteins: advanced imaging applications and future design. Angew Chem Int Edit. 51 (43), 10724-10738 (2012).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Curr Protein Pept Sc. 9 (4), 338-369 (2008).
- Wang, L., Xie, J., Deniz, A. A., Schultz, P. G. Unnatural amino acid mutagenesis of green fluorescent protein. J Org Chem. 68 (1), 174-176 (2003).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Sharma, N., Furter, R., Kast, P., Tirrell, D. A. Efficient introduction of aryl bromide functionality into proteins in vivo. FEBS Lett. 467 (1), 37-40 (2000).
- Liu, C. C., Schultz, P. G. Adding new chemistries to the genetic code. Annu Rev Biochem. 79, 413-444 (2010).
- Twine, S. M., Murphy, L., Phillips, R. S., Callis, P., Cash, M. T., Szabo, A. G. The Photophysical Properties of 6-Azaindole. J Phys Chem B. 107 (2), 637-645 (2003).
- Lepthien, S., Hoesl, M. G., Merkel, L., Budisa, N. Azatryptophans endow proteins with intrinsic blue fluorescence. P Natl Acad Sci USA. 105 (42), 16095-16100 (2008).
- Budisa, N., et al. Probing the role of tryptophans in Aequorea victoria green fluorescent proteins with an expanded genetic code. Biol Chem. 385 (2), 191-202 (2004).
- Ross, J. B., et al. Spectral enhancement of proteins: biological incorporation and fluorescence characterization of 5-hydroxytryptophan in bacteriophage lambda cI repressor. P Natl Acad Sci USA. 89 (24), 12023-12027 (1992).
- Soumillion, P., Jespers, L., Vervoort, J., Fastrez, J. Biosynthetic incorporation of 7-azatryptophan into the phage lambda lysozyme: Estimation of tryptophan accessibility, effect on enzymatic activity and protein stability. Protein Eng Des Sel. 8 (5), 451-456 (1995).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Bridges, B. A., Mottershead, R. P., Rothwell, M. A., Green, M. H. L. Repair-deficient bacterial strains suitable for mutagenicity screening: tests with the fungicide captain. Chem Biol Interact. 5 (2), 77-84 (1972).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: The Heat Shock Method. J Vis Exp. , (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: Electroporation. J Vis Exp. , (2017).
- Grigorenko, B. L., Krylov, A. I., Nemukhin, A. V. Molecular modeling clarifies the mechanism of chromophore maturation in the green fluorescent protein. J Am Chem Soc. , (2017).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to the Spectrophotometer. J Vis Exp. , (2017).
- Goedhart, J., et al. Structure-guided evolution of cyan fluorescent proteins towards a quantum yield of 93%. Nat Commun. 3, 751 (2012).
- Neidhardt, F. C., Bloch, P. L., Smith, D. F. Culture medium for enterobacteria. J Bacteriol. 119 (3), 736-747 (1974).
- Hörnsten, E. G. On culturing Escherichia coli on a mineral salts medium during anaerobic conditions. Bioprocess Eng. 12 (3), 157-162 (1995).
- Davis, B. D. The Isolation of Biochemically Deficient Mutants of Bacteria by Means of Penicillin. P Natl Acad Sci USA. 35 (1), 1-10 (1949).
- Sambrook, J., Russell, D. W. Molecular Cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY, USA. (2001).
- Wang, Y. -S., et al. The de novo engineering of pyrrolysyl-tRNA synthetase for genetic incorporation of L-phenylalanine and its derivatives. Mol Biosyst. 7 (3), 714-717 (2011).
- Bae, J. H., et al. Expansion of the genetic code enables design of a novel "gold" class of green fluorescent proteins. J Mol Biol. 328 (5), 1071-1081 (2003).
- JoVE Science Education Database. Dialysis: Diffusion Based Separation. J Vis Exp. , Cambridge, MA. (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Separating Protein with SDS-PAGE. J Vis Exp. , (2017).
- Petrásek, Z., et al. Excitation energy transfer from phycobiliprotein to chlorophyll d in intact cells of Acaryochloris marina studied by time- and wavelength-resolved fluorescence spectroscopy. Photoch Photobio Sci. 4 (12), 1016-1022 (2005).
- Kolber, Z. S., Barkley, M. D. Comparison of approaches to the instrumental response function in fluorescence decay measurements. Anal Biochem. 152 (1), 6-21 (1986).
- Pelet, S., Previte, M. J. R., Laiho, L. H., So, P. T. C. A fast global fitting algorithm for fluorescence lifetime imaging microscopy based on image segmentation. Biophys J. 87 (4), 2807-2817 (2004).
- Loefroth, J. E. Time-resolved emission spectra, decay-associated spectra, and species-associated spectra. J Phys Chem. 90 (6), 1160-1168 (1986).
- Hartman, M. C. T., Josephson, K., Lin, C. -W., Szostak, J. W. An expanded set of amino acid analogs for the ribosomal translation of unnatural peptides. PLoS One. 2 (10), e972 (2007).
- Budisa, N., et al. Global replacement of tryptophan with aminotryptophans generates non-invasive protein-based optical pH sensors. Angew Chem Int Edit. 41 (21), 4066-4069 (2002).
- Ma, Y., Biava, H., Contestabile, R., Budisa, N., di Salvo, M. L. Coupling bioorthogonal chemistries with artificial metabolism: intracellular biosynthesis of azidohomoalanine and its incorporation into recombinant proteins. Molecules. 19 (1), 1004-1022 (2014).
- Teramoto, H., Kojima, K. Incorporation of Methionine Analogues Into Bombyx mori Silk Fibroin for Click Modifications. Macromol Biosci. 15 (5), 719-727 (2015).
- Deal, R. B., Henikoff, J. G., Henikoff, S. Genome-wide kinetics of nucleosome turnover determined by metabolic labeling of histones. Science. 328 (5982), 1161-1164 (2010).
- Hinz, F. I., Dieterich, D. C., Tirrell, D. A., Schuman, E. M. Non-canonical amino acid labeling in vivo to visualize and affinity purify newly synthesized proteins in larval zebrafish. ACS Chem Neurosci. 3 (1), 40-49 (2012).
- Dieterich, D. C., et al. In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat Neurosci. 13 (7), 897-905 (2010).
- Dieterich, D. C., Link, A. J., Graumann, J., Tirrell, D. A., Schuman, E. M. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). P Natl Acad Sci USA. 103 (25), 9482-9487 (2006).
- Glenn, W. S., et al. Bioorthogonal Noncanonical Amino Acid Tagging (BONCAT) Enables Time-Resolved Analysis of Protein Synthesis in Native Plant Tissue. Plant Physiol. 173 (3), 1543-1553 (2017).
- Zhou, L., et al. Incorporation of tryptophan analogues into the lantibiotic nisin. Amino Acids. 48 (5), 1309-1318 (2016).
- Acevedo-Rocha, C. G., Budisa, N. Xenomicrobiology: a roadmap for genetic code engineering. Microb Biotechnol. 9 (5), 666-676 (2016).
- Agostini, F., Völler, J. -S., Koksch, B., Acevedo-Rocha, C. G., Kubyshkin, V., Budisa, N. Biocatalysis with Unnatural Amino Acids: Enzymology Meets Xenobiology. Angew Chem Int Edit. 56 (33), 9680-9703 (2017).
- Bacher, J. M., Ellington, A. D. Selection and characterization of Escherichia coli variants capable of growth on an otherwise toxic tryptophan analogue. J Bacteriol. 183 (18), 5414-5425 (2001).
- Wong, J. T. Membership mutation of the genetic code: loss of fitness by tryptophan. Pc Natl Acad Sci USA. 80 (20), 6303-6306 (1983).
- Hoesl, M. G., et al. Chemical Evolution of a Bacterial Proteome. Angew Chem Int Edit. 54 (34), 10030-10034 (2015).
- Italia, J. S., et al. An orthogonalized platform for genetic code expansion in both bacteria and eukaryotes. Nat Chem Biol. 13 (4), 446-450 (2017).
- Völler, J. -S., Thi To, T. M., Biava, H., Koksch, B., Budisa, N. Global substitution of hemeproteins with noncanonical amino acids in Escherichia coli with intact cofactor maturation machinery. Enzyme Microb Tech. 106, 55-59 (2017).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Völler, J. -S., Budisa, N. Coupling genetic code expansion and metabolic engineering for synthetic cells. Curr Opin Biotech. 48, 1-7 (2017).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr Opin Chem Biol. 14 (6), 774-780 (2010).
- Somsen, O. J., van Grondelle, R., van Amerongen, H. Spectral broadening of interacting pigments: polarized absorption by photosynthetic proteins. Biophys J. 71 (4), 1934-1951 (1996).
- Kurschus, F. C., Pal, P. P., Bäumler, P., Jenne, D. E., Wiltschi, B., Budisa, N. Gold fluorescent annexin A5 as a novel apoptosis detection tool. Cytom Part A. 75 (7), 626-633 (2009).
- Lepthien, S., Wiltschi, B., Bolic, B., Budisa, N. In vivo engineering of proteins with nitrogen-containing tryptophan analogs. Appl Microbiol Biot. 73 (4), 740-754 (2006).
- Wachter, R. M., Elsliger, M. -A., Kallio, K., Hanson, G. T., Remington, S. J. Structural basis of spectral shifts in the yellow-emission variants of green fluorescent protein. Structure. 6 (10), 1267-1277 (1998).
- Verkhusha, V. V., Lukyanov, K. A. The molecular properties and applications of Anthozoa fluorescent proteins and chromoproteins. Nat Biotechnol. 22 (3), 289-296 (2004).
- Martynov, V. I., Savitsky, A. P., Martynova, N. Y., Savitsky, P. A., Lukyanov, K. A., Lukyanov, S. A. Alternative cyclization in GFP-like proteins family. The formation and structure of the chromophore of a purple chromoprotein from Anemonia sulcata. J Biol Chem. 276 (24), 21012-21016 (2001).
- Piatkevich, K. D., Malashkevich, V. N., Morozova, K. S., Nemkovich, N. A., Almo, S. C., Verkhusha, V. V. Extended Stokes shift in fluorescent proteins: chromophore-protein interactions in a near-infrared TagRFP675 variant. Sci Rep. 3 (1), 1847 (2013).



