Summary
Syntetisk biologi möjliggör konstruktion av proteiner med oöverträffade egenskaper med hjälp av co-translational införande av icke-kanoniska aminosyror. Vi presenterade här, hur en spektralt röd-skiftat variant av en GFP-typ fluorophore med romanen fluorescens spektroskopiska egenskaper, kallas ”guld” fluorescerande protein (GdFP), produceras i E. coli via selektionstryck inkorporering (SPI).
Abstract
Fluorescerande proteiner är grundläggande verktyg för biovetenskap, i synnerhet för fluorescensmikroskopi av levande celler. Medan vildtyp och konstruerade varianter av grönt fluorescerande protein från Aequorea victoria (avGFP) samt homologs från andra arter redan täcker stora delar av det optiska spektrumet, finns en spektral klyfta i nära infraröda området, för som avGFP-baserade fluorophores är inte tillgängliga. Röd-skiftat fluorescerande protein (FP) varianter skulle väsentligen expandera toolkit för spektral minoriteter av flera molekylära arter, men naturligt förekommande röd-skiftat FPs härrör från koraller eller havsanemoner har lägre fluorescens kvantutbyte och sämre foto-stabilitet jämfört med varianter av avGFP. Ytterligare manipulation och eventuell utvidgning av kromoforens konjugerade system mot den mån spektrala-regionen är också begränsad av repertoaren av 20 kanoniska aminosyror som föreskrivs av den genetiska koden. För att övervinna dessa begränsningar, kan syntetisk biologi uppnå ytterligare spektrala röd-shifting via införandet av icke-kanoniska aminosyror i kromofor triaden. Vi beskriver tillämpningen av SPI på ingenjör avGFP varianter med romanen spektrala egenskaper. Proteinuttryck utförs i en tryptofan-auxotrophic E. coli stam och genom att komplettera odlingsmedier med lämplig indol prekursorer. Inuti cellerna, dessa prekursorer konverteras till de motsvarande tryptofan-analoger och införlivas med proteiner ribosomal maskineriet svar på UGG kodon. Byte av Trp-66 i den förbättra ”cyan” varianten av avGFP (ECFP) av en elektron-donera 4-aminotryptophan resulterar i GdFP med en 108 nm Stokes Skift och en starkt röd-skiftat utsläpp maximal (574 nm), medan det är thermodynamically stabilare än dess föregångare ECFP. Rester-specifika införlivandet av icke-kanoniska aminosyran analyseras av masspektrometri. GdFP spektroskopiska egenskaper kännetecknas av tid-löst fluorescens-spektroskopi som en av de värdefulla tillämpningarna av genetiskt kodade FPs i biovetenskap.
Introduction
Sedan upptäckten av grönt fluorescerande protein i maneter Aequorea victoria (avGFP) i 19621 och första heterologa uttrycket i 19942 i andra eukaryota celler, har fluorescerande proteiner i familjen GFP blivit mycket värdefulla verktyg och mål inom life sciences. Omfattande genetisk och molekylär teknik ingår justeringen av artspecifika kodon användning, acceleration av vikning, förbättrad mognad, ökad ljusstyrka, förebyggande av oligomerisering och sömnad av spektral- och fotokemiska inklusive förmågan att reversibelt photoswitch3,4,5,6. GFP varar skyldig dess fluorescens från dess 4-(p- hydroxybenzylidene) imidazolidin-5-en (HBDI) kromofor. Den senare bildas autocatalytically från så kallade kromofor triaden av aminosyror (Ser-65/Tyr-66/Gly-67 i avGFP) efter bildandet av en ytterligare kovalent bindning inom peptid ryggraden under inflytande av molekylärt syre7. Resonantly stabiliserad konjugerade systemet samverkar dynamiskt med dess molekylära miljö, vilket möjliggör upptaget i det synliga området och karakteristiska grön fluorescens av dessa proteiner.
Inom kromofor Triaden är förekomsten av en aromatisk aminosyra obligatoriskt. Dock standard aminosyra repertoaren består av endast fyra aromatiska rester (hans, Phe, Trp och Tyr). Detta begränsar konventionella mutagenes strategier för att uppnå betydligt mer röd-skiftade avGFP varianter i förhållande till de mest röd-skiftat naturliga FPs såsom DsRed8 från Discosoma striata coralimorphs eller mKate/mNeptune9 från havsanemon Entacmaea quadricolor. Därför, den mån och nära infraröda delen av det optiska spektrumet över 600 nm är glest täckt av GFP varianter. Detta är naturligtvis en allvarlig begränsning för fluorescens mikroskopiska metoder som kräver spektral demultiplexning av flera fluorophore arter samtidigt. Till exempel lång våglängd markörer är också nödvändigt att göra använda av systemet låg absorption av hudvävnad mellan 700-1000 nm i inställningarna för djup vävnad imaging10.
Fluorescerande protein som härrör från avGFP är indelade i flera klasser baserat på spektroskopiska egenskaper och kemiska naturen av deras chromophores11. Med dess triad Ser-65/Tyr-66/Gly-67, vildtyp kromoforen finns som en skakad blandning mellan neutral, fenoliska form (λmax = 395 nm, ε = 21.000 M-1cm-1) och anjoniska phenolate form (λmax = 475 nm, ε = 7 100 M -1cm-1), och emissionsspektrum uppvisar en enskild topp på 508 nm. Hydroxylgruppen Ser-65 är av avgörande betydelse, som donerar en H-bond att Glu-222 i kromofor närhet (avstånd: 3,7 Å), som främjar joniseringen av denna bildas. Klass jag kännetecknas av en anjoniska phenolate kromofor, liksom i andra (Phe-64-Leu/Ser-65-Thr; λmax = 488 nm, ε = 35,600 M-1cm-1λem = 509 nm). På grund av Ser-65-Thr(Ala,Gly) substitution, 395 nm excitation toppen av formuläret neutrala fenol är undertryckta och 470-475 nm toppen av de anjoniska phenolate är fem - till six - faldigt ökat och skiftade till 490 nm. Klass II består av proteiner med en neutral fenoliska kromofor, liksom i sapphire-GFP. Här Thr-203-Ile substitution nästan helt undertrycker 475 nm magnetiseringen, lämnar bara toppen på 399 nm. Eftersom anjon kromoforen inte kan vara korrekt solvatiserade, gynnas dess neutrala form. Klass III består av de ”gula” fluorescerande varianterna (EYFP; Ser-65-GLY/val-68-Leu/ser-72-Ala/THR-203-Tyr; Λmax ε = 514 nm, ε = 84, 600 M-1cm-1, λem = 527 nm) med π-stapling växelverkan av en aromatisk sidokedjan och phenolate, som åstadkommits genom Thr-203-His(Trp,Phe,Tyr) substitutioner, som leda till upp till 20 nm röd-skiftat utsläpp maxima (Thr-203-Tyr). Ytterligare ersättning (Gln-69-Lys) resulterar i en annan 1-2 nm röd förskjutning till 529 nm, den mest röd-skiftade avGFP varianten känd11. Utbyte av fenol för en indol (Tyr-66-Trp) skapar klass IV, som i den cyan-fluorescerande ECFP (Ser-65-Thr/Tyr-66-Trp; λmax1 = 434 nm, ε = 24 800 M-1cm-1; λmax2 = 452 nm, ε = 23.600 M-1cm-1 ; Λem1 = 477 nm, λem2 = 504 nm). Inkvartering av den skrymmande indol aktiveras förmodligen som andra, kompenserande mutationer. De ECFP magnetisering och utsläpp maxima faller emellan de av proteiner med neutral eller anjoniska chromophores. Klass V proteiner harbor en Imidazol i stället för fenol (Tyr-66-hans), t.ex., blå fluorescerande proteiner som EBFP. Klass VI är producerad av en fenol-till-fenyl exchange gynnar formuläret neutrala kromofor uteslutande, vilket följaktligen leder till mest blå-skiftat excitation och utsläpp topp positioner (360 nm och 442 nm, respektive).
Klassiskt webbplats riktad mutagenes är särskilt lämplig för produktion av Roman avGFP kromofor varianter, av permutationen av 65-67 tripeptid och samverkande rester i ramen av de 20 aminosyrorna som kanoniska. Dessa möjligheter kan utökas ytterligare när icke-kanoniska varianter av aromatiska aminosyror införs under ribosomala proteinsyntes12. I princip finns det två sätt att åstadkomma detta. Den första strategin bygger på substrat toleransen av protein översättning maskiner, särskilt av aminoacyl-tRNA-synthetases (aaRSs) mot relaterade aminosyra analoger. Att uppnå detta med hög verkningsgrad, auxotrophic E. coli stammar som uttryck är anställda som inte kan syntetisera motsvarande naturliga aminosyran. Detta tillåter utbyte av den senare genom att lägga till lämpliga icke-kanoniska aminosyror (ncAAs) eller prekursorer därav till odlingsmediet. Denna strategi, även känd som selektiv tryck inkorporering (SPI)13,14, kan rester-specifika ersättare, som resulterar i global införlivandet av ncAA. Den andra strategin använder stop kodon suppressor tRNAs som debiteras med ncAA av konstruerad aaRS enzymer. Detta resulterar i en genomläsning av i-frame stop kodon och tillåter platsspecifika ncAA inkorporering. Denna metod av stop kodon suppression (SCS) leder följaktligen till expansionen av den genetiska kod15. Via mutagenes placeras ett stop-kodon i målgenen på önskad plats. I princip kan SPI också användas att skapa rekombinanta peptider och proteiner bär en unik ncAA installation, med tanke på att sällsynta kanoniska aminosyror såsom Met eller Trp väljs för substitution. Med Trp, SPI metoder har visat sig fungera med ett stort utbud av analoger inklusive 4 - F-, 5 - F - och 6-F-Trp, 7-aza-Trp, 4-OH - och 5-OH-Trp, samt 4- och 5-NH2- Trp eller ens β (thienopyrrolyl) alanin derivat16 ,17,18,19,20. SPI kan således vara mycket fördelaktigt för att ersätta aromatiska aminosyror av GFP chromophores av icke-kanoniska varianter att undersöka möjligheten för att ytterligare skräddarsy spectra och Stokes förskjutning av dessa FPs. När det gäller alla protein sequence ändringar prövas förenlighet med FP vikning och kromofor mognad experimentellt.
I detta arbete använder vi klass IV ECFP21, som bär i stället för den vildtyp avGFP Tyr, Trp rester inom dess kromofor triad. Med SPI, ersätts denna Trp-66 (och Trp-57, bara andra Trp återstoden i ECFP) av 4-amino-Trp. Förekomsten av den elektron-donera aminogrupp av 4-amino-Trp inom kromoforen gynnar resonans stabiliseringen av en långt rött-skiftat exciterat tillstånd proton överföring (ESPT) begåvad med en 108 nm Stokes SKIFT. Detta ”gold” fluorescerande protein (GdFP) utgör varianten med den största röda förskjutningen av fluorescensen maximal (574 nm) bland alla avGFP framställda proteiner. Vi beskriva metoden av GdFP proteinproduktion av SPI och ger protokollen för obligatorisk analys av de resulterande modifierade proteinerna av massa spektroskopi. Vidare visar vi hur GdFP kan användas och analyseras i tid-löst fluorescens-spektroskopi metoder.
Protocol
1. omvandling av Trp-auxotrophic E. coli
- Omforma kemiskt eller electrocompetent celler (50 µL) av en Trp-auxotrophic E. coli -stam, t.ex. ATTC 49980 (WP2, mutant från E. coli stam B/R22), med 1 µL av en 1 ng/µL vattenlösning av pQE - 80 L His6-ECFP plasmiden med värme chock eller elektroporation, respektive. Hänvisas till JoVE Science Education Database23,24 för detaljer.
Anmärkning: Uttrycket vektorn pQE - 80L His6-ECFP kodar en N-terminalt 6 x hans-taggade ECFP21 drivs av en bakteriell T5 arrangören med lac operatör. Bär det ytterligare en AmpR urval markör och en colE1 ursprung replikering (pQE - 80 L vektor ryggraden sekvensen kan hittas på: https://www.qiagen.com/mx/resources/resourcedetail?id=c3b71572-4d82-4671-a79b-96357fe926d1&lang=en & automatiska förslag = true). Den teoretiska molekylvikten av proteinet His6-ECFG vildtyp (efter kromofor mognad25) är 28303.92 Da. Översatta mål protein sekvensen är som följer (hans-tag underströk, vektor-derived sekvenser i fetstil): MRGSJennieGSMVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTWGVQCFSRYPDHMK
QHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYISHNVYITADKQKNGIKANFKIRHNIEDGS
VQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYK. - Platta celler på LB-agar plattor (tabell 1) kompletteras med 10 g/L glukos, 100 µg/mL ampicillin och inkubera plattorna vid 37 ° C under natten.
2. rekombinant proteinuttryck
-
Övernattning kultur av E. coli ATCC 49980 pQE - 80 L His6-ECFP
- Förbereda 5 mL LB medium (tabell 1; kompletteras med 10 g/L glukos, 100 µg/mL ampicillin) i en steril 14 mL polystyren kultur tube för aerob tillväxt och Inokulera med en ensamma koloni från en agarplatta med hjälp av en steril pipett spets eller inympning loop.
Obs: Rekommenderas använda kolonier från färska celler. Plattorna med bakteriekolonier (från steg 1.2.) kan lagras vid 4 ° C i flera dagar. - Inkubera cellerna vid 37 ° C i orbitalskak vid 200-250 rpm över natten.
- Förbereda 5 mL LB medium (tabell 1; kompletteras med 10 g/L glukos, 100 µg/mL ampicillin) i en steril 14 mL polystyren kultur tube för aerob tillväxt och Inokulera med en ensamma koloni från en agarplatta med hjälp av en steril pipett spets eller inympning loop.
-
Uttryck för vildtyp ECFP
- Inokulera 10 mL färsk LB medium (tabell 1, kompletterad med 10 g/L glukos, 100 µg/mL ampicillin) med 100 µL av den nattliga kulturen i en 100 mL-Erlenmeyerkolv. Inkubera kolven vid 37 ° C i orbitalskak på 200 rpm.
Obs: Detta steg kan utföras frivilligt i 10 mL NMM19 medium (tabell 1) kompletteras med 100 µg/L ampicillin och 0,5 mM L-tryptofan (alternativt indol kan användas). - Mät den optiska densiteten vid 600 nm (OD600) varje 20 min. prioriterat mäta cell densitet genom att bestämma utrotning på 600 nm (OD600) i en spektrofotometer med en kyvetten med en väglängd av 1 cm. alltid utföra en referens mätning med motsvarande odlingssubstratet. Späd ut proverna och blanda proverna väl för att få ett mätvärde på 0,1-0,8 och sedan beräkna OD600 med utspädningsfaktorn. För detaljer, vänligen se föregående publikation 26.
- När du når ett OD600 värde på 0,5-0,8 (cirka 2-3 timmar efter inympningen) kan ta prov ”före induktion” för SDS-PAGE (sodium dodecyl sulfate polyakrylamid gelelektrofores, steg 4).
- Inducera mål proteinuttryck genom att justera den flytande kulturen till 0,5 mM IPTG (isopropyl β-D-1-thiogalactopyranoside, från 1 M stamlösning) och inkubera det vid 30 ° C i orbitalskak på 200 rpm för 4-8 h.
Obs: Cyan fluorescerande proteiner uttrycks vanligen vid temperaturer under 37 ° C27. - Ta prov ”efter uttrycket” för SDS-PAGE (steg 4.).
- Skörda bakteriecellerna genom centrifugering i 10 min på 5 000 x g och 4 ° C.
- Avlägsna supernatanten genom dekantering och frysa cell pellets vid-20 ° C eller -80 ° C tills målet protein rening.
- Inokulera 10 mL färsk LB medium (tabell 1, kompletterad med 10 g/L glukos, 100 µg/mL ampicillin) med 100 µL av den nattliga kulturen i en 100 mL-Erlenmeyerkolv. Inkubera kolven vid 37 ° C i orbitalskak på 200 rpm.
-
SPI för att producera GdFP
- Inokulera 10 mL NMM19 medium (tabell 1) kompletteras med 100 µg/mL ampicillin, 15 µM tryptofan och 10 µL övernattning kultur i en 100 mL Erlenmeyerkolv och inkubera kultur kolven övernattning på 30 ° C i orbitalskak på 200 rpm.
Obs: Det finns en mängd kemiskt definierade media för odling av E. coli och SPI. Utöver NMM används häri, moppar medium28, glukos-mineral saltar medium29, Davis minimal medium30, M9 minimal medium31eller GMML32 kan användas. - Nästa dag, mäta OD600 varje 30 min tills värdet ändras endast av mindre än 0,05 över 30 min. Platå värdet bör vara cirka 1.
Obs: Avvikelser av ± 0,3 enheter är acceptabelt. Beroende på bakteriestam och medium som används, kan den första tryptofan koncentrationen (steg. 2.3.1) behöva justeras. - Ta prov ”före induktion” för SDS-PAGE (steg 4.).
- Skörda bakteriecellerna genom centrifugering i 10 min på 5 000 x g och 4 ° C. Kassera supernatanten genom dekantering.
- Omsuspendera cellerna i 10 mL NMM19 medium med 100 µg/mL ampicillin till en 100 mL Erlenmeyerkolv och Lägg 4-amino-indol till en slutlig koncentration på 1 mM med 50 mM stamlösning. Fortsätt inkubationstiden för 30 min vid 30 ° C i orbitalskak på 200 rpm.
Obs: Detta steg rekommenderas på grund av den låga kemisk stabiliteten av ampicillin och säkerställer cellernas upptag av 4-amino-indol. - Inducera mål proteinuttryck genom att lägga till IPTG till en slutlig koncentration av 0,5 mM med 1 M lager och inkubera provet över natten vid 30 ° C i orbitalskak på 200 rpm.
Obs: Cyan fluorescerande proteiner uttrycks vanligen vid temperaturer under 37 ° C27. - Nästa dag, mäta OD600.
- Ta prov ”efter uttrycket” för SDS-PAGE (steg 4.).
- Skörda bakteriecellerna genom centrifugering i 10 min på 5 000 x g och 4 ° C och Kassera supernatanten genom dekantering.
- Om sådant fartyg inte användes för centrifugering, över cellpelleten till en 50 mL konisk polystyren tub med hjälp av en spatel. Frysa cellpelleten vid-20 ° C eller -80 ° C tills målet protein rening.
- Inokulera 10 mL NMM19 medium (tabell 1) kompletteras med 100 µg/mL ampicillin, 15 µM tryptofan och 10 µL övernattning kultur i en 100 mL Erlenmeyerkolv och inkubera kultur kolven övernattning på 30 ° C i orbitalskak på 200 rpm.
3. mål Protein rening via immobiliserade metall affinitet jonkromatografi (IMAC)
-
Bakteriell cell lysis
- Tina cellpelleten på is för 10-20 min.
- Återsuspendera cellpelleten i en 50 mL konisk polystyren tub med 5 mL iskallt bindande buffert (tabell 1) på is.
- Tillsätt 20 µL av 50 mg/mL lysozym, 20 µL av 1 mg/mL DNAS jag, 20 µL av 1 mg/mL RNase A. Stäng tuben, blanda försiktigt genom att invertera 5 gånger och hålla den på is i 30 min.
Obs: Partiell cell störningar inträffar som katalyseras av lysozym. - Lysera celler genom att placera mätkolven med hjälp av ett ultraljud Homogenisatorer spets med tre cykler på 3 min i en 15 mL polystyren röret kyls av slush ice med 2 s av puls, 4 s paus och 45% amplitud.
Obs: Alternativt, högt tryck homogenisering kan användas, t.ex., 20 cykler vid 14 000 psi. Vid behov, späd med bindande buffert för att nå minimal instrument volymen. Protein extraktionsreagens kan dessutom användas för cell störningar. Se material tabell för exempel. - Centrifugera provet under 30 minuter vid 15.000-18.000 x g, 4 ° C.
- Över supernatanten till en färsk tub och anteckna vätskevolym.
- Filtrera lösningen genom ett 0,45 µm spruta filter med hjälp av en 5 mL Luer lock Plastspruta och en vinylidenfluoridplast (PVDF) sprutan filter.
- Ta prov ”lysate” för SDS-PAGE (steg 4.).
- Återsuspendera skräp cellpelleten i ddH2O (lika stor volym som f.d. lysate).
- Ta prov ”pellets” för SDS-PAGE (steg 4.).
-
IMAC rening
- Använd en 1 mL färdigförpackade eller själv packade IMAC FPLC (snabb protein vätskekromatografi) kolumn enligt tillverkarens anvisningar. Använda bindande buffert (tabell 1) för kolumnen Jämviktstiden samt när det gäller tvättningen som följer efter cellen lysate har tillämpats till kolumnen.
- Samla och pool eluatet fraktioner med GdFP som kan identifieras av synligt ljus gyllene färg.
Obs: Alternativt målproteinet kan vara elueras med hjälp av en linjär Imidazol-gradient (0-250 mM) med hjälp av ett automatiserat FPLC system. - För att bestämma proteinkoncentration med hjälp av litteratur-värdet för extinktionskoefficient vid 466 nm (ɛ466 nm = 23,700 M-1 cm-1)33 med eluering buffert som referens. För detaljer om förfarandet, hänvisas till föregående publikation26.
- Ta prov ”eluatet” för SDS-PAGE, och använda 1-10 µg av protein per körfält vid Coomassie-färgning.
Obs: SDS prov belopp kan variera beroende på färgningsmetod och färgämne känslighet. - Dialyze en alikvot av eluatet fraktioner mot dialys buffert eller MS buffert med ett membran med en molekylvikt cutoff (MWCO) på 5 000-10 000. Förbereda dialys membran enligt tillverkarens anvisningar. Dialyze en 1 mL prov tre gånger mot 100 mL buffert för minst 2 h. För detaljer om proceduren, hänvisas till föregående publikation34.
- Förvaring, frysa protein prov i dialys buffert vid-80 ° C.
Obs: Alikvoter bör vara stabilt i minst 6 månader.
4. SDS-PAGE provberedning av E. coli hela Cell extrakt
- Överföra en cellsuspension som motsvarar 1 mL OD600 = 1 suspension (t.ex. 500 µL OD600= 2) i ett 1,5 mL mikrocentrifug rör.
- Skörda cellerna genom centrifugering i 10 min på 5000 x g, rumstemperatur. Kassera supernatanten genom pipettering.
- Lägga till 80 µL ddH2O och 20 µL av 5 x SDS lastning dye buffert (tabell 1) i cellpelleten och mix av pipettering.
- Denature cellerna genom uppvärmning till 95 ° C i 5 min i ett vatten bad eller värme block. Därefter svalka proven till rumstemperatur.
- Använd 10 µL för Coomassie-färgade SDS-PAGE enligt föregående publikation35.
Obs: SDS prov belopp kan variera beroende på färgningsmetod och färgämne känslighet.
5. intakt Protein massa analys av högpresterande vätskekromatografi (HPLC) kopplat till elektrospray jonisering Time-of-flight masspektrometri (LC-ESI-TOF-MS)
Obs: HPLC gradient, inställningar och buffertar kan variera beroende på den avskiljande kolumn och instrument som används. Se material tabell för exemplarisk utrustning.
- Bestämma proteinkoncentration från ett prov dialyseras mot MS buffert som beskrivs ovan (steg 3.2.3.) med MS buffert (se material tabell) som referens.
- Späd provet protein till 0,1 mg/mL med MS buffert för en slutlig volym av 80 µL, blanda genom försiktig pipettering, överför lösningen till en MS autosampler injektionsflaska med glas insats och stängs med en mössa. Avlägsna luftbubblor genom att snärta injektionsflaskan.
- Fyll en andra autosampler injektionsflaska utan glas Infoga (buffert tomt) med 1 mL MS buffert.
- Låt instrumentet att värma upp. Utföra instrumentkalibrering. Se till att det finns tillräckliga mängder flytande kromatografi-grade lösningsmedel (> 100 mL).
- Programmera en linjär 20 min HPLC gradient från 5% till 80% buffert en (0,1% myrsyra i ddH2O), kombinerat med buffert B (0,1% myrsyra i acetonitril).
- Starta HPLC vid ett flöde av 0,3 mL/min och vänta tills kolumnen trycket är stabil.
- Ställ en autosampler injektionsvolym 5 µL för LC-ESI-TOF-MS-metoden, skapa en arbetslista för en tom köra följt av en prov körning och tilldela den motsvarande autosampler injektionsflaska positioner. Kör i arbetslistan.
- Öppna den genererade exempeldatafil efter arbetslistan. Markera ett område på totalt ion nuvarande (TIC) tomt för deconvolution och deconvolute MS spektrumet med maximal entropi deconvolution algoritm.
Obs: Beroende på försöksbetingelser, ytterligare arter kan uppstå från icke-mognat FP eller buffert ion addukter.
6. fluorescens livstid mätningar och förfall-associerade Spectra (DAS) av GdFP
Obs: För instrumenteringen av tid-löst fluorescens-spektroskopi, se Tabell för material för exemplarisk utrustning. Absorbans och fluorescens magnetiseringen samt utsläpp spektra av fluorescerande proteiner kan också registreras med hjälp av laboratorium UV/Vis och fluorescens spektrofotometrar.
-
Våglängd-löst fluorescens livstid mätning av GdFP
- Förbered 2 mL av en 1 µM lösning av GdFP av utspädning i PBS-bufferten (tabell 1) vid pH 7. Häll lösningen i en 1 cm kvarts kyvetten.
- Installera ps-pulsade 470 nm laser för provet excitation och 488 nm lång-pass utsläpp filtret och justera 600 L/mm galler av tid - och våglängd-korrelerade enda fotonen räkna36 (TWCSPC) detektor för förvärv av våglängd regimen 500-700 nm.
- Förvärva fluorescens utsläpp räknas uppgå till ca 200 x 103 fotoner/s tills ca 103 räknas ackumuleras i förvärvet högst fluorescens decay kurvorna med single photon counting programvara.
-
Mätning av de instrumentala svar funktion37 (IRF)
- Ersätta den prov kyvetten med en 1 cm kvarts kyvetten fylld med 1 g/L kolloidal kiseldioxid (~ 220 m2g) i PBS-bufferten vid pH 7.
Obs: Den kiseldioxid-suspensionen bereds med en 400 g/L vattensuspension. - Ta bort 488 nm lång-pass utsläpp filter och infoga grå filter för att justera räkna på TWCSPC detektorn till under 100 x 103 räknas/s.
- Justera gallerdurk för förvärvet av 470 nm fotoner i kanal 8 av 16-kanals TWCSPC detektorn.
- Förvärva IRF tills ca 10 x 103 räknas ackumuleras i maximala utsläpp.
- Konvertera fluorescens decay kurvor och IRF ASCII datafiler med globala passande38 program.
- Beteende Global passar enligt en modell av en summa av tre exponentiell komponenter med livstider som länkade parametrar.
- Plot förfall associerade spectra (DAS) som amplitud distributioner av enskilda decay komponenter i beroende av våglängden med data analysprogram.
- Ersätta den prov kyvetten med en 1 cm kvarts kyvetten fylld med 1 g/L kolloidal kiseldioxid (~ 220 m2g) i PBS-bufferten vid pH 7.
Representative Results
Med hjälp av tekniken selektionstryck bolagsordning, kan Trp-66 i kromofor triaden av ECFP (och Trp-57, bara andra Trp återstoden i ECFP) ersättas av 4-amino-Trp, därmed generera den röd-skiftade GdFP med distinkta spektrala egenskaper. Masspektrometri måste användas för att visa önskad stökiometriska integrationen av icke-kanoniska aminosyran i proteinet, med resultat som visas i figur 1. Efteråt, vi tillhandahåller data från mikroskopi, UV-Vis absorption spektroskopi samt steady state och tid - och våglängd-löst fluorescens-spektroskopi att karakterisera egenskaperna för GdFP fluorophore med fokus på pH beroendet av den Spectra.
För att bekräfta utbytet av de två Trp resterna i ECFP av 4-amino-Trp, utförs massa spektrometriska analysen. Figur 1 visar ett representativt deconvoluted ESI-MS spektrum av GdFP. Medan vildtyps-ECFP har en beräknad protein massa 28,283.9 Da efter kromofor mognaden, är motsvarande massa GdFP 28,313.9 Da. Deconvoluted ESI-MS spectrumen av GdFP uppvisar en huvudsakliga massa peak på 28,314.1 ± 0,1 Da, som avviker från det teoretiska värdet av mindre än 10 ppm. Att vara inom intervallet typisk noggrannhet för denna typ av analys25, detta bekräftar införlivandet av ncAA via SPI (experimental värde för vildtyp ECFP: 28,283.7 Da).
Figur 2 visar confocal fluorescensmikroskopi imaging (CFIM) bilder av bakterieceller som uttrycker ECFP, andra, EYFP och GdFP vid resuspension av bakterier i PBS-bufferten. Alla bilder har förvärvats på ett mikroskop som är utrustat med en UV mål och laser magnetiseringen på om samma energi för varje prov.
Figur 3A visar en överlagring av CFIM bilder av E. coli -bakterier som uttrycker olika FPs inklusive GdFP, alltid övervakas med mycket liknande exciteringsenergi (våglängder som i figur 2). Figur 3B visar kromofor strukturer FP varianter visas. När det gäller ljusstyrkan på GdFP jämfört med ECFP (fluorescens quantum avkastning φ = 0,4), andra (φ = 0,6) och EYFP (φ = 0,6) det är viktigt att notera att för GdFP, en bredare förvärv fluorescens ljuset (30 nm) användes i motsats till 20 nm används för alla andra spe Cies, för att justera intensiteten av bilderna till liknande värden. Med en något lägre extinktionskoefficient och en minskad kvantutbyte till följd av unika photophysical egenskaper, är ljusstyrkan på GdFP lägre än andra FPs visas.
Absorptionsspektrum av ECFP (figur 3 c) har två karakteristiska maxima på 434 nm och 452 nm. Däremot GdFP kännetecknas av en bred röd-skiftat absorptionsbandet med maximat på 466 nm. Absorptionen av andra är ytterligare röd-skiftat till 488 nm. På grund av den mycket större Stokes förskjutningen av GdFP (108 nm) jämfört med ECFP (41 nm) och andra (20 nm), utsläpp spectrumen av GdFP är den mest röd-skiftat av alla tre GFP derivat undersökt här (figur 3D). Medan fluorescens utsläpp av ECFP visar två karakteristiska maxima på 475 nm och 505 nm, andra har en bred viktigaste utsläppskällorna bandet topp på 508 nm (λmax) med en liten axel vid 540 nm. Fluorescensen i GdFP visas vid ca 565 nm (λmax.) (Figur 3D). Dess utsläpp spektrum innehåller ett litet bidrag av vildtyp ECFP som syns också som en liten axel på 475 nm. Denna bråkdel ECFP syntetiseras före induktion under förfarandet för SPI, som beskrivs33.
Figur 3E visar pH-beroende ändringarna i absorptionsspektrum av GdFP. För en pH förändring från 8 till 5, maximala utsläpp skiftar något till röda och en liten breddning av absorptionsbandet observeras. Minskning av absorption amplituden är dock bara ca 10% mellan pH 8 och pH 5, vilket indikerar att GdFP kromoforen grundtillståndet egenskaper är mycket svagt av pH.
Tiden löst fluorescens utsläppet övervakas av single photon counting visas i figur 4. Decay kurvorna övervakas i de spektrala kanaler centrerad vid 550 nm och 600 nm (figur 4A) uppvisar en något snabbare fluorescens förfall på 600 nm jämfört med förfalla vid 550 nm. Resultaten av en global passform av fluorescensen förfalla kurvor med två exponentiell komponenter resulterar i två spektralt distinguishable fluorescens decay komponenter med tidskonstanter 1.0 ns och 3,3 ns (figur 4 c och D).
Fluorescens utsläpp av GdFP beror starkt på pH, eftersom det är typiskt för många fluorescerande protein varianter av familjen GFP. Figur 4B jämför fluorescens utsläpp av GdFP mellan pH 5 och pH 8, vilket tydligt visar en minskning i fluorescensintensiteten vid lägre pH, medan de spektrala egenskaperna bo konstant.
Den förfalla-associerade spectra (DAS)39 i GdFP (figur 4 c och D) kännetecknas av två distinkta utsläpp band. Bidraget av de långsamma 3,3 ns komponent är mer uttalad i kort våglängdsområdet omkring 550 nm (60%) med mindre bidrag av komponenten snabbare (40%). På 600 nm, båda komponenterna har ungefär samma amplituden. Vid en övergång från pH 7 (figur 4 c) till pH 6 (figur 4 d), de spektrala egenskaperna för DAS ändra knappast och tid konstanterna från den globala passande rutinen är också den samma (riktigheten av DAS tid konstanterna är ungefär ± 0,15 ns). Dock skillnaden i absoluta amplituderna av de två komponenterna DAS är tydligt, som helt står för reducerad fluorescens utsläpp amplituden vid samma pH förändring i figur 4B.
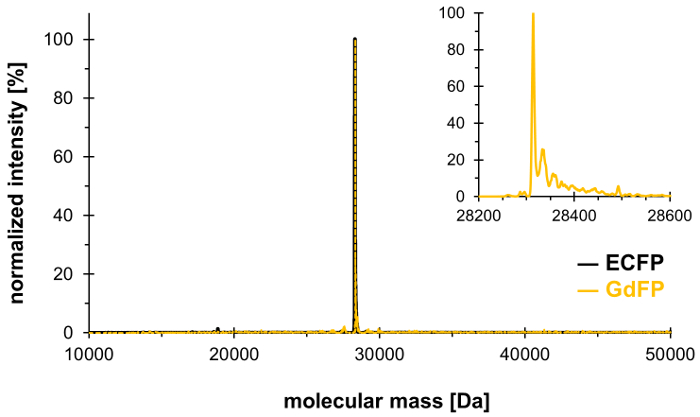
Figur 1: representativa deconvoluted ESI-MS spektrum av GdFP. ESI-MS spectrumen av GdFP (guld färg, förstorade tomt visas som infälld) visar en huvudsakliga topp på 28314.1 Da (beräknat värde 28313.9 Da). Spektrumet för vildtyp ECFP visas i svart. Klicka här för att se en större version av denna siffra.

Figur 2: Confocal fluorescence mikroskopi bilder från bakteriell populationer att uttrycka olika FPs. Följande våglängd inställningar användes för bild förvärv: ECFP (λex = 457 nm, detection: 461-480 nm), andra (λex = 488 nm, detection: 495-515 nm), GDFP (λex = 476 nm, detection: 560-590 nm), EYFP (λex = 514 nm, upptäckt: 520-530 nm). Klicka här för att se en större version av denna siffra.

Figur 3: spektrala egenskaper GdFP. (A), CFIM bild av en blandning av bakterieceller att uttrycka ECFP och andra GdFP efter resuspension av bakterier i PBS-bufferten. (B) kromofor strukturer av GdFP (med 4-amino-Trp i stället för rester 66), parental ECFP (med Trp på position 66) och EFGP (med Tyr på position 66). (C) jämförelse av de normaliserade Absorberingsspectra av GdFP, ECFP och andra, medan i (D), normaliserade fluorescens emissionsspektrum av ECFP (magnetiseringen på 430 nm) jämförs med fluorescens utsläpp spektra av andra och GdFP (båda upphetsad vid 450 nm). (E) pH-beroende av Absorptionsspektra (normaliserade absorption vid 280 nm). Klicka här för att se en större version av denna siffra.

Figur 4: tid-löst fluorescens av GdFP. (A) fluorescens förfalla av GdFP övervakas av tid - och våglängd-löst single photon counting spectral kanaler centrerad vid 550 nm och 600 nm (± 12,5 nm) efter excitation med 470 nm laser pulserar. Funktionen instrumental svar (IRF) ger information om tiden upplösning används inställningen. (B) variant av utsläpp spectrumen av GdFP beroende av pH (magnetiseringen på 460 nm). (C, D) Förruttnelse-associerade spectra (DAS) av GdFP vid pH 7 (C) och pH 6 (D) bestäms efter deconvolution av tid - och våglängd-löst fluorescens sönderfaller och globala montering av sönderfaller i alla kanaler av en global uppsättning två exponentialfunktioner med länkade tidskonstanter. Klicka här för att se en större version av denna siffra.

Figur 5: strukturer av intramolekylära kostnad överföring av ECFP (svart) och GdFP (guld) chromophores. Öka i storlek av kromofor systemet bra elektron donator av en aminogrupp som en del av ncAA kan bildandet av mer mesomera strukturer för att uppnå resonans stabilisering av det exciterat tillståndet. Anslutningen pekar till FP schavotten visas som halvcirklar. Klicka här för att se en större version av denna siffra.
| Stamlösning | koncentration, lösningsmedel | Obs | |
| 20% D-glukos | 200 g/L D-glukos i ddH2O | Sterilisera genom filtrering genom ett 0,45 µm porstorlek storlek spruta filter | |
| indol | 50 mM i isopropanol | ||
| 4-amino-indol | 50 mM i 20% etanol (20 mL etanol i en slutlig volym av 100 mL fylld med ddH2O) | ||
| IPTG | 1 M i ddH2O | ||
| L-tryptofan | 15 mM upplöst i ddH2O använda 1 M HCl (Lägg HCl droppvis under omrörning tills pulvret är dissoved) | ||
| lysozym | 50 mg/mL i ddH2O | ||
| DNAS jag | 1 mg/mL i ddH2O | ||
| RNase A | 1 mg/mL i ddH2O | ||
| Amp100 | 100 mg/mL ampicillin i ddH2O | ||
| natrium-dodecylsulfate (SDS) | 200 g/L i ddH2O | ||
| ammoniumsulfat ((NH4)2SO4) | 1 M i ddH2O | Sterilisera i autoklav | |
| Kaliumdivätefosfat (KH2PO4) | 1 M i ddH2O | Sterilisera i autoklav | |
| di-kalium väte fosfat (K2HPO4) | 1 M i ddH2O | Sterilisera i autoklav | |
| magnesiumsulfat (MgSO4) | 1 M i ddH2O | Sterilisera i autoklav | |
| D-glukos | 1 M i ddH2O | Sterilisera genom filtrering genom ett 0,45 µm porstorlek storlek spruta filter | |
| natriumklorid (NaCl) | 5 M i ddH2O | Sterilisera i autoklav | |
| kalciumklorid (CaCl2) | 1 g/L | Sterilisera genom filtrering genom ett 0,45 µm porstorlek storlek spruta filter | |
| järn(II) klorid (FeCl2) | 1 g/L | Sterilisera genom filtrering genom ett 0,45 µm porstorlek storlek spruta filter | |
| tiamin | 10 g/L | Sterilisera genom filtrering genom ett 0,45 µm porstorlek storlek spruta filter | |
| Biotin | 10 g/L | Sterilisera genom filtrering genom ett 0,45 µm porstorlek storlek spruta filter | |
| spårämnen mix | kopparsulfat CuSO4, zink klorid (ZnCl2), manganese chloride (MnCl2), ammoniummolybdat ((NH4)2MoO4); varje 1 mg/L i ddH2O | Sterilisera genom filtrering genom ett 0,45 µm porstorlek storlek spruta filter | |
| 19 aminosyror mix | 1.) Lös 0,5 g L-fenylalanin och 0,5 g L-tyrosin i 100 ml ddH2O med droppvis tillägg 1 M HCl under omrörning tills pulvret är upplöst. | ||
| 2.) väger ut 0,5 g av varje av de återstående L-aminosyror (förutom L-tryptofan). Blanda med 22 mL fo 1 M KH2PO4 och 48 mL 1 M K2HPO4. Lägga till ddH2O ca 800 mL. Rör tills lösningen blir klar. | |||
| 3.) Lägg den upplösta L-fenylalanin och L-tyrosin från steg 1.) och justera volymen till 1 L med ddH2O. | |||
| 4.) Sterilize aminosyra blandningen av dammsugarfilter med en flaska topp filterenhet. | |||
| Buffertar och Media | Sammansättning eller preparatet | ||
| SDS lastning dye buffert, 5 x koncentrerad | 0,25 M Tris pH 6.8, 50% v/v glycerol, 0,25% w/v bromfenolblått, 0,5 M didhiothreitol (DTT; alternativt 5% β-merkaptoetanol), 10% w/v natrium-dodecylsulfate (SDS) | ||
| bindande buffert | 50 mM natrium dihydrogenphosphate (NaH2PO4), 500 mM NaCl, 10 mM Imidazol, pH 8 | ||
| eluering buffert | 50 mM natrium dihydrogenphosphate (NaH2PO4), 500 mM NaCl, 250 mM Imidazol, pH 8 | ||
| dialys buffert | 50 mM natrium dihydrogenphosphate (NaH2PO4), 150 mM NaCl, 100 mL/L glycerol, pH 8 | ||
| MS buffert | 10 mM Tris-HCl, pH 8 | ||
| nya minimalmedium innehåller 19 L-aminosyror utom L-tryptofan (NMM19) | Blanda alla lager lösningar för att få följande slutliga koncentrationer: 7,5 mM (NH4)2SO4, 1,7 mM NaCl, 22 mM KH2PO4, 50 mM K2HPO4, 1 mM MgSO4, 20 mM D-glukos, 50 mg/L 19 aminosyror blanda, 1 µg/L CaCl21 µg/L FeCl2, 10 µg/L tiamin, 10 mg/L biotin, 0,01 mg/L spårämnen mix | ||
| LB medium | Sammansättning: 10 g/L trypton, 5 g/L jästextrakt, 10 g/L NaCl, pH 7,0 i ddH2O | ||
| Förberedelser: | |||
| (1) väger ut 50 g trypton, 25 g jäst extrakt, 5 g NaCl i en 1 L glasflaska. | |||
| 2.) Lägg till ddH2O till ~ 800 mL och lös upp komponenter under omrörning. | |||
| 3.) Mät pH och justera till pH 7 genom droppvis tillsätta 1 M HCl eller 1 M NaOH, om det behövs. Lägg till ddH2O upp till 1 L. | |||
| 4.) Sterilize genom autoklavering, kolla för volymförlust efteråt och lägga sterila ddH2O att kompensera vid behov. Förvaras vid 4 ° C fram till användning. | |||
| LB agarplattor | Sammansättning: 10 g/L trypton, 5 g/L jästextrakt, 10 g/L NaCl, 15 g/L agar-agar, pH 7,0 i ddH2O | ||
| Förberedelser: | |||
| (1) väger ut 50 g trypton, 25 g jäst extrakt, 5 g NaCl, 7,5 g agar-agar i en 1 L glasflaska. | |||
| 2.) Lägg till ddH2O upp till 500 mL och lös upp komponenter under omrörning. | |||
| 3.) Mät pH och justera till pH 7 genom droppvis tillsätta 1 M HCl eller 1 M NaOH, om det behövs. Lägg till ddH2O upp till 1 L. | |||
| 4.) Sterilize genom autoklavering, kolla för volymförlust efteråt och lägga sterila ddH2O att kompensera, om det behövs. (Obs: LB agar kan lagras vid 4 ° C fram till användning för beredning av LB agarplattor. Försiktigt smälta stelnat agar i mikrovågsugn) | |||
| (5.) när lösningen är fortfarande varmt (30-40 ° C), lägga till ampicillin till en slutlig koncentration på 100 µg/mL | |||
| 6.) Häll ca 15 mL av vätskan från steg 5.) i en steril 10 cm petriskål under sterila förhållanden. När ägarn är stelnat, kan plattor förvaras i 1 vecka vid 4 ° C fram till användning. | |||
| fosfatbuffrad saltlösning (PBS) | Sammansättning: 137 mM NaCl, 2,7 mM KCl, 1,8 mM KH2PO4, 0,5 mM MgCl2, 1 mM CaCl2, 10 mM Na2HPO4, pH 7. Sterilisera i autoklav eller filtrering. | ||
Tabell 1: Lager lösning och buffert.
Discussion
För att uppnå mycket höga ncAA inkorporering effektivitetsvinster, bygger metoden auxotrophy-baserade SPI på användning av metaboliskt bakåtkompilerade värdceller, som inte klarar syntetisera den motsvarande naturliga motsvarigheten i ncAA. För E. coliär sådana stammar lätt tillgängliga. Även samtidiga införlivandet av flera ncAAs i samma protein är möjligt med hjälp av multiauxotrophic stammar. Rester-specifika funktionsläget av ersättning och kemiska repertoaren begränsas till liknande kemiska analoger kan ses som nackdelar. Dock kan ett stort antal protein varianter produceras som den naturliga bakteriella översättning apparaturen tål många aminosyra analoger. Exempelvis kunde mer än 50 ncAAs införlivas med proteiner med hjälp av in vitro- översättning, för cirka 73% av alla kodon av den genetiska koden ska vara tillgänglig för omtilldelning40. SPI kan dessutom också effektiv multisite märkning av de mål protein41. I princip SPI metoden är inte begränsad till E. coli, men kan arbeta i alla andra värdar och för varje av de kanoniska 20 aminosyrorna, förutsatt att auxotrophic stammar och definierade odling medier är tillgängliga. Till exempel finns två metionin analoger, azidohomoalanine (Aha) och homopropargylglycine (Hpg), kommersiellt tillgängliga och används för märkning proteiner och proteom i olika organismer. Aha kan dessutom producerade intracellulärt och därefter införlivas protein42. Detta ncAA är speciellt lämplig för bioorthogonal konjugationer såsom Klicka kemi som utvecklats av Tirrel och medarbetare: till exempel i plantera vävnad av Arabidopsis thaliana, Bombyx mori larver43, Drosophila celler44, larval zebrafiskar45 samt däggdjursceller inklusive nervceller46, proteiner kan märkas med Aha47,48. På samma sätt införlivades framgångsrikt Trp analoger av antimikrobiella peptider i Trp-auxotrophic Lactococcus lactis stammar49. SPI är också användbart för fältet i Xenobiology50,51, som undersöker alternativ till den grundläggande kemiska sammansättningen av liv. Till exempel utvecklades baserat på tidigare verk på E. coli52 och B. subtilis53, en E. coli -stam nyligen av en evolutionär strategi med selektiv tryck att använda thienopyrrole i stället för indol, vilket resulterar i proteomet-wide substitution av tryptofan genom thienopyrrole-alanin i den genetiska kod54. Generellt, den kanoniska aminosyran Trp, som kodas av en enda triplet (UGG), presenterar en lovande måltavla för proteinteknik på grund av de rika aspekterna av indol kemi, som erbjuder många kemiska varianter. Nyligen, och som ett alternativ till SPI-baserade införlivande, rapporterade en roman SCS plattform kapabel att införliva Trp analoger anläggningsvis i både bakteriella och eukaryota värdar har varit55. Detta breddar ytterligare verktygslådan för i vivo ncAA-baserade protein engineering, inklusive ändring av spektrala egenskaper.
Förutom användningen av auxotrophic uttryck värdar kräver SPI protokollet strikt jäsning förhållanden, både vad gäller mål uttryck timing och sammansättningen av mediet för att nå hög ncAA inkorporering effektivitet och protein avkastningsmålet 56. odling utförs med kemiskt definierade minimal media, som i huvudsak innehåller förutom stora salter källorna till kväve (ammoniumsalt) och kol (D-glukos), vitaminer och spårämnen. Även om inte strikt krävs i avsaknad av ytterligare auxotrophies, de återstående aminosyrorna (20 -n, om n aminosyror är bytas) läggs vanligen till främja bakterietillväxt57. Under en initial tillväxtfas före induktion av målet proteinuttryck läggs n kanoniska aminosyror bytas i begränsa halterna. Cellulära tillväxt fortsätter tills de rikta essentiella aminosyrorna är uttömda, så experimentellt indikeras av en stationär OD600. Därefter ersättas odlingssubstratet med färska medium som saknar utarmat aminosyran och innehåller ncAA i riklig koncentrationer. För ribosomal inkorporeringen av tryptofan analoger som visas i detta protokoll, matas en indol analoga, som blir intracellulärt konverteras till motsvarande tryptofan derivatan av tryptofan syntas58. Nästa, target proteinuttryck induceras. I detta skede är cellerna nära slutet av logaritmisk tillväxt, som en balans mellan antalet totala celler och kondition. Som närvaro och införlivandet av de kanoniska amino skulle leda till vildtyp proteinproduktion, är det viktigt att säkerställa att den essentiella aminosyran är helt urladdat före induktion. Likaså är det obligatoriskt att undersöka effektiviteten av ncAA införlivande i målproteinet, vanligen genom masspektrometri. Vid påtaglig närvaro av kanoniska aminosyran odling villkor behöver justeras, t.ex., genom att ändra koncentrationen av den grundläggande amino acid(s) för den första tillväxtfasen eller varaktigheten av den senare. Vid låg aaRS aktivitet mot ncAA, kan överuttryck av enzymet endogen eller samtidig uttryck för en annan aaRS, som är mer aktiv mot ncAA, vara genomförda59.
Kanoniska aminosyran Trp begåvas med tre anmärkningsvärda funktioner: (i) dess naturliga överflöd i proteiner är låg; (ii) dess biofysiska och kemiska egenskaper är unika (t.ex., är det oftast dominerande ursprunget till den inneboende fluorescensen av proteiner och peptider), och (iii) det bidrar till en mängd biokemiska interaktioner och funktioner inklusive Π-stapling, H-bindning och katjon-π interaktioner. Alla dessa funktioner är radikalt vid Trp → 4-amino-Trp substitution GdFP. bortom tvivel, utformningen av en ”guld” klass av avGFPs är ett anmärkningsvärt exempel för tekniska skräddarsydda autofluorescent proteiner. Med distinkta spektrala egenskaper, kan FPs stämmas mot vissa spektrala windows via mutagenes och ncAA inkorporering. Vid GdFP sker detta genom ett enkelt kemiskt utbyte H → NH2 i ramen av indol-ring i ECFP kromofor triaden. Figur 5 visar effekterna av ncAA inkorporering inom kromoforen. Införandet av gruppen elektron-donera med ursprung från 4-amino-indol (intracellulärt konverteras till 4-amino-Trp) möjliggör en mängd mesomera strukturer som kan förklara en stabiliserad exciterat tillstånd. Spectroscopically, dess utvidgade Stokes Skift och röd-skiftat fluorescens utsläpp resultera från dessa distinkta egenskaper det utökade konjugerade systemet. Som rapporterats tidigare, förbättrad intramolekylära kostnad överföring inom GdFP kromoforen är till sin natur känslig för pH (figur 4B) och åtföljs av en större förändring i dipolmoment mellan S0 marken och S1 upphetsad staten i förhållande till ECFP33. Som alternativa elektron-donera grupper, skulle tryptofan analoger bär en indol-ring ersättas med hydroxy grupper kunna användas, som rapporterats i en jämförande studie med den modell protein barstar41.
Absorption och fluorescens spektra av GdFP är breddat jämfört med ECFP och andra (figur 3 c och D). Homogen breddning av banden absorption och fluorescens orsakas vanligtvis av vibrations lägen i kromoforen och dessutom av kopplingen av kromoforen till ytterligare vibrations lägen finns i protein60. Kopplingen till den lokala protein miljön stöds av avgifter lokaliserade på kromoforen. Eftersom det strukturella inhomogenitet av protein leder till lokala variationer av vibronic spektrum, sådan koppling mellan vibronic spektra av kromoforen och resten av proteinet stöds av kostnad utlokaliseringarna och mesomera stater som anges i Figur 5. Denna koppling också stöder stora Stokes övergången och minskar nödvändigtvis fluorescens quantum avkastning. I jämförelse med andra röda-skiftat FPs uppvisar GdFP även förbättrad protein stabilitet och en låg tendens för aggregering33,61,62. Det inte bara skiljer sig i färg från andra FP-varianter men också uppvisar en väsentligt ökad termostabiliteten och förbättrad kooperativa fällbara33. Dess fluorescensintensitet är minst 90% bevaras vid uppvärmning till 60 ° C, medan ECFP fluorescens reduceras till ca 30%. I proteiner bidrar aromatiska aminosyror ofta till nätverk av samverkande sida kedjor, som vanligen har en stabiliserande effekt på proteinets tertiär struktur. avGFP hamnar sådant side chain nätverk, som består av kromoforen själv, samt Phe-165, hans-148, och Tyr-145. Dessa sida kedjor är inte bara ganska styv i de GdFP struktur33, men ännu viktigare, de bildar hydrofoba kontakter med kromoforen. Den mest framstående roman funktionen identifieras i GdFP är att aminated kromoforen är mer proximalt Phe-165. Denna interaktion är en funktion som inte observerats i andra kända avGFPs. De två restsubstanser förekommer 3,2 till 4,5 Å apart, kan amino-aromatiska interaktioner också vara närvarande. Tillsammans med aminering-inducerad resonans stabiliseringen av kromoforen stabilisera dessa troligen denna hydrofoba nätverk av aminosyror i en kooperativ mode. En effektivare intramolekylära kostnad överföring kan stödjas av dessa interaktioner i jämförelse marken glada delstaten kromoforen och åtminstone delvis står för den 108 nm Stokes Skift33,62 .
I rationell design av fluorophore egenskaper förväntas en ökning av storleken på delocalized π-systemet resultera i en röd-skiftat excitation våglängd. Denna tumregel är lydde av serien av aminosyror i position 66 leder till neutrala chromophores: Phe (λmax = 355 nm) < hans (λma x= 386 nm) < Tyr (λmax = 395 nm) < Trp (λmax = 436 nm)63. I naturen, har denna utvidgning av kromoforens konjugerade system av π-obligationer uppnåtts genom olika strategier. För DsRed från Discosoma striataförlängs av integrationen av en ytterligare aminosyra, därmed skifta λmax till 573 nm64. Kromoforen av asFP595 (λmax = 595 nm) från Anemonia sulcata förlängdes med en imingrupp grupp, utvidga dess π-systemet65. Eftersom kromoforen av GdFP och andra avFPs är av samma storlek, måste en annan princip innebära emissionsvåglängden i spänna av den utökade DsRed och asFP595 chromophores. Den djupgående Stokes förskjutningen av 108 nm tillskrivs den tydliga strukturen i GdFP kromoforen, som avslöjar en ny photophysical princip i utformningen av autofluorescent proteiner. Preliminära beräkningar (som rapporterats i 62) har visat att dipolmoment av glada-state kromoforen i GdFP är betydligt större än i grundtillståndet, i motsats till respektive värdena för ECFP. Medan dipolmoment av GdFP ökar från ~ 3 D (Debye) i tillståndet S0 till ~ 15 D i S1, förändringen för ECFP kromoforen var ganska måttlig (från ~ 4 D till ~ 6 D). Således, den unika gyllengula fluorescensen av GdFP orsakas av betydande intramolekylära kostnad överföring inom kromoforen, vilket ökar mängden möjliga mesomera strukturer (se figur 5) som möjliggör resonans stabilisering. Detta minskar energinivån där utsläpp sker. Till följd av djupgående förändringen i dipolmoment vid excitation är de intramolekylära laddningsseparation rektor resonerar för förändringar i elektrostatiska potentialen av kromofor miljön. Omgivande proteinet matrix, justerar i sin tur till förändringar i avgiften fördelningen efter kromofor excitation. Den efterföljande strukturella avslappning sänker energi av glada kromoforen, som skiftar fluorescens spektrumet till röda på grund av dess kostnad överföring karaktär. För samma anledning som en följd av den stora Stokes Skift och förbättrade priser radiationless processer, fluorescens quantum avkastningen av GdFP minskas jämfört med ECFP33.
Den höga kvantutbyte och små Stokes förskjutning av ECFP och andra är vanligtvis tillskrivs en styv protein miljö av kromoforen, vilket minskar antalet frihetsgrader och, följaktligen, inre omvandling att gynna radiative uppmjukning av de exciterat tillståndet 66. följaktligen mer stelt inbäddade chromophores med minskad koppling till återstående protein matrix molekylär design kan fungera som en guide att producera längre röd-skiftat GFP derivat med hög fluorescence kvantutbyte. För ytterligare tekniska metoder för att producera röda-skiftade autofluorescent proteiner, därför utvidgningen av π-elektron och en styv kromofor struktur med svag koppling till protein miljön önskvärt. Sådana ändringar kan också införas antingen direkt i GFP-baserade chromophores eller genom placering av önskad ncAAs i kromofor närhet.
Disclosures
Författarna förklarar att de har inga konkurrerande finansiella intressen.
Acknowledgments
Detta arbete stöds av den tyska forskningsfondens (kluster av Excellence ”Unifying begrepp i katalys) att T.F. och N.B. och av förbundsministeriet för utbildning och vetenskap (BMBF Program” HSP 2020 ”, TU-WIMIplus projekt SynTUBio) till F.-J.S.
Materials
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| 4-aminoindole | Sigma-Aldrich | 525022 | |
| acetonitrile | VWR | HiPerSolv CHROMANORM ULTRA for LC-MS, 83642 | LC-MS grade required |
| agar-agar | Carl Roth | 5210 | |
| ammonium molybdate ((NH4)2MoO4) | Sigma-Aldrich | 277908 | |
| ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| ampicillin sodium salt | Carl Roth | K029 | |
| biotin | Sigma-Aldrich | B4501 | |
| bromophenol blue | Sigma-Aldrich | B0126 | |
| calcium chloride (CaCl2) | Sigma-Aldrich | C5670 | |
| colloidal silica | Sigma-Aldrich | Ludox HS-40, 420816 | |
| Coomassie Brillant Blue R 250 | Carl Roth | 3862 | |
| copper sulfate (CuSO4) | Carl Roth | CP86.1 | |
| D-glucose | Carl Roth | 6780 | |
| di-sodium hydrogen phosphate (Na2HPO4) | Carl Roth | X987 | |
| di-potassium hydrogen phosphate (K2HPO4) | Carl Roth | P749.1 | |
| 1,4-dithiothreitol (DTT) | Carl Roth | 6908 | |
| DNase I | Sigma-Aldrich | D5025 | |
| ethanol | Carl Roth | 9065.1 | |
| formic acid | VWR | HiPerSolv CHROMANORM for LC-MS, 84865 | LC-MS grade required |
| glycerol | Carl Roth | 3783 | |
| imidazole | Carl Roth | X998 | |
| indole | Sigma-Aldrich | I3408 | |
| iron(II) chloride (FeCl2) | Sigma-Aldrich | 380024 | |
| isopropanol | Carl Roth | AE73.1 | |
| isopropyl β-D-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I6758 | |
| lysozyme | Sigma-Aldrich | L6876 | |
| magnesium chloride (MgCl2) | Carl Roth | KK36.1 | |
| magnesium sulfate (MgSO4) | Carl Roth | 8283.2 | |
| manganese chloride (MnCl2) | Sigma-Aldrich | 63535 | |
| β-mercaptoethanol | Carl Roth | 4227.3 | |
| potassium chloride (KCl) | Carl Roth | 6781.3 | |
| potassium dihydrogen phosphate (KH2PO4) | Sigma-Aldrich | P5655 | |
| RNase A | Carl Roth | 7156 | |
| sodium chloride (NaCl) | Carl Roth | P029 | |
| sodium dihydrogen phosphate (NaH2PO4) | Carl Roth | T879 | |
| sodium dodecyl sulphate (NaC12H25SO4) | Carl Roth | 0183 | |
| thiamine | Sigma-Aldrich | T4625 | |
| Tris(hydroxymethyl)-aminomethane (Tris) | Carl Roth | 5429 | |
| Tris hydrochloride (Tris-HCl) | Sigma-Aldrich | 857645 | |
| tryptone | Carl Roth | 8952 | |
| yeast extract | Carl Roth | 2363 | |
| zinc chloride (ZnCl2) | Sigma-Aldrich | 229997 | |
| Name | Company | Catalog Number | Comments |
| amino acids | |||
| L-alanine | Sigma-Aldrich | A7627 | |
| L-arginine | Sigma-Aldrich | A5006 | |
| L-asparagine | Sigma-Aldrich | A8381 | |
| L-aspartic acid | Sigma-Aldrich | A0884 | |
| L-cysteine | Sigma-Aldrich | C7352 | |
| L-glutamic acid | Sigma-Aldrich | G2128 | |
| L-glutamine | Sigma-Aldrich | G3126 | |
| L-glycine | Sigma-Aldrich | G7126 | |
| L-histidine | Sigma-Aldrich | H8000 | |
| L-isoleucine | Sigma-Aldrich | I2752 | |
| L-leucine | Sigma-Aldrich | L8000 | |
| L-lysine | Sigma-Aldrich | L5501 | |
| L-methionine | Sigma-Aldrich | M9625 | |
| L-proline | Sigma-Aldrich | P0380 | |
| L-phenylalanine | Sigma-Aldrich | P2126 | |
| L-serine | Sigma-Aldrich | S4500 | |
| L-threonine | Sigma-Aldrich | T8625 | |
| L-tryptophan | Sigma-Aldrich | T0254 | |
| L-tyrosine | Sigma-Aldrich | T3754 | |
| L-valine | Sigma-Aldrich | V0500 | |
| Name | Company | Catalog Number | Comments |
| Lab materials | |||
| 0.45 µm syringe filter with PVDF membrane | Carl Roth | CCY1.1 | |
| 1.5 mL microcentrifuge tubes | Eppendorf | 30120086 | |
| conical polystyrene (Falcon) tubes, 50 mL | Fisher Scientific | 14-432-22 | |
| Luer-Lock syringe 5 mL | Carl Roth | EP96.1 | |
| dialysis membrane, Molecular Weight Cut-Off (MWCO) 5,000 | Spectrum Medical Industries | Spectra/Por MWCO 5000 dialysis membrane, 133198 | |
| Immobilized Metal ion Affinity Chromatography (IMAC) column 1 mL, Ni-NTA | Macherey Nagel | Protino series, 745410.5 | |
| petri dishes (polystyrene, sterile) | Carl Roth | TA19 | |
| pQE-80L plasmid vector | Qiagen | no longer available | replaced by N-terminus pQE Vector set Cat No./ID: 32915 |
| protein extraction reagent BugBuster | EMB Millipore | 70921-4 | |
| round-bottom polystyrene tubes, 14 mL | Fisher Scientific | Corning Falcon, 14-959-1B | |
| Trp-auxotrophic E. coli strain | ATCC | ATCC 49980 | Bridges BA et al., Chem Biol Interact., 1972, 5(2):77-84; see main text for alternatives |
| Name | Company | Catalog Number | Comments |
| Mass Spectrometry equipment | |||
| mass spectrometer for LC-ESI-TOF-MS | Agilent | Agilent 6530 Accurate-Mass QTOF | coupled with Infinity LC system |
| mass spectrometry data analysis software | Agilent | MassHunter Qualitative Analysis software v. B.06.00 | |
| High-Performance Liquid Chromatography (HPLC) column for LC-ESI-TOF-MS | Sigma-Aldrich | Supelco Discovery BIO Wide Pore C5 HPLC column, 3 µm particle size, 10 cm x 2.1 mm | |
| HPLC autosampler vials 1.5 mL | Sigma-Aldrich | Supelco 854165 | with conical 0.1 mL glass inserts, screw caps and septa |
| Name | Company | Catalog Number | Comments |
| General equipment | |||
| benchtop centrifuge for 1.5 mL Eppendorf tubes | Eppendorf | 5427 R | |
| cooling centrifuge for 50 mL Falcon tubes | Eppendorf | 5810 R | |
| high pressure microfluidizer for bacterial cell disruption | Microfluidics | LM series with “Z” type chamber | |
| peristaltic pump for LC | GE Healthcare | P-1 | |
| Fast Protein Liquid Chromatography (FPLC) system | GE Healthcare | ÄKTA pure 25 L | |
| orbital shaker for bacterial cultivation | Infors HT | Minitron | |
| UV/Vis spectrophotometer | Biochrom | ULTROSPEC 2100 | |
| ultrasonic homogenizer for bacterial cell disruption | Omnilab | Bandelin SONOPULS HD 3200, 5650182 | with MS72 sonifier tip |
| Name | Company | Catalog Number | Comments |
| Fluorescence spectroscopy equipment | |||
| ps-pulsed laser 470 nm | Picoquant GmbH | PDL-470 | |
| time- and wavelength-correlated single photon counting (TWSPC) acquisition software | Picoquant GmbH | SymPhoTime 64 | |
| time- and wavelength-correlated single photon counting (TWSPC) detector | Picoquant GmbH | PML-16C | 16 spectral channels, to be selected by grating settings |
| single photon counting software | Picoquant GmbH | SPCM 9.75 | |
| global fitting software | Picoquant GmbH | SPC2Glo(R) | |
| fluorescence decay data analysis software | Picoquant GmbH | FluoFit program | |
| data analysis software | OriginLab Inc. | Origin 9.2 | |
| neutral density filter set | Schott | NG1 to NG11 | (400 - 650 nm, transmission 50 %, 20%, 10 %, 5 %) |
| 488 nm long-pass emission filter | AHF Analysentechnik | AHF-488 | |
| quartz cuvette | Thorlabs GmbH | CV10Q1400 | 1 cm pathlength |
References
- Shimomura, O., Johnson, F. H., Saiga, Y. Extraction, Purification and Properties of Aequorin, a Bioluminescent Protein from the Luminous Hydromedusan, Aequorea. J Cell Compar Physl. 59 (3), 223-239 (1962).
- Chalfie, M., Tu, Y., Euskirchen, G., Ward, W. W., Prasher, D. C. Green fluorescent protein as a marker for gene expression. Science. 263 (5148), 802-805 (1994).
- Andresen, M., et al. Structure and mechanism of the reversible photoswitch of a fluorescent protein. P Natl Acad Sci USA. 102 (37), 13070-13074 (2005).
- Andresen, M., et al. Structural basis for reversible photoswitching in Dronpa. P Natl Acad Sci USA. 104 (32), 13005-13009 (2007).
- Brakemann, T., et al. A reversibly photoswitchable GFP-like protein with fluorescence excitation decoupled from switching. Nat Biotechnol. 29 (10), 942-947 (2011).
- Kremers, G. -J., Gilbert, S. G., Cranfill, P. J., Davidson, M. W., Piston, D. W. Fluorescent proteins at a glance. J Cell Sci. 124 (Pt 2), 157-160 (2011).
- Shimomura, O. Structure of the chromophore of aequorea 0. shimomura green fluorescent protein. FEBS Lett. 104 (2), 220-222 (1979).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N. G., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Shcherbo, D., et al. Bright far-red fluorescent protein for whole-body imaging. Nat Methods. 4 (9), 741-746 (2007).
- Shcherbakova, D. M., Subach, O. M., Verkhusha, V. V. Red fluorescent proteins: advanced imaging applications and future design. Angew Chem Int Edit. 51 (43), 10724-10738 (2012).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Curr Protein Pept Sc. 9 (4), 338-369 (2008).
- Wang, L., Xie, J., Deniz, A. A., Schultz, P. G. Unnatural amino acid mutagenesis of green fluorescent protein. J Org Chem. 68 (1), 174-176 (2003).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Sharma, N., Furter, R., Kast, P., Tirrell, D. A. Efficient introduction of aryl bromide functionality into proteins in vivo. FEBS Lett. 467 (1), 37-40 (2000).
- Liu, C. C., Schultz, P. G. Adding new chemistries to the genetic code. Annu Rev Biochem. 79, 413-444 (2010).
- Twine, S. M., Murphy, L., Phillips, R. S., Callis, P., Cash, M. T., Szabo, A. G. The Photophysical Properties of 6-Azaindole. J Phys Chem B. 107 (2), 637-645 (2003).
- Lepthien, S., Hoesl, M. G., Merkel, L., Budisa, N. Azatryptophans endow proteins with intrinsic blue fluorescence. P Natl Acad Sci USA. 105 (42), 16095-16100 (2008).
- Budisa, N., et al. Probing the role of tryptophans in Aequorea victoria green fluorescent proteins with an expanded genetic code. Biol Chem. 385 (2), 191-202 (2004).
- Ross, J. B., et al. Spectral enhancement of proteins: biological incorporation and fluorescence characterization of 5-hydroxytryptophan in bacteriophage lambda cI repressor. P Natl Acad Sci USA. 89 (24), 12023-12027 (1992).
- Soumillion, P., Jespers, L., Vervoort, J., Fastrez, J. Biosynthetic incorporation of 7-azatryptophan into the phage lambda lysozyme: Estimation of tryptophan accessibility, effect on enzymatic activity and protein stability. Protein Eng Des Sel. 8 (5), 451-456 (1995).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Bridges, B. A., Mottershead, R. P., Rothwell, M. A., Green, M. H. L. Repair-deficient bacterial strains suitable for mutagenicity screening: tests with the fungicide captain. Chem Biol Interact. 5 (2), 77-84 (1972).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: The Heat Shock Method. J Vis Exp. , (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: Electroporation. J Vis Exp. , (2017).
- Grigorenko, B. L., Krylov, A. I., Nemukhin, A. V. Molecular modeling clarifies the mechanism of chromophore maturation in the green fluorescent protein. J Am Chem Soc. , (2017).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to the Spectrophotometer. J Vis Exp. , (2017).
- Goedhart, J., et al. Structure-guided evolution of cyan fluorescent proteins towards a quantum yield of 93%. Nat Commun. 3, 751 (2012).
- Neidhardt, F. C., Bloch, P. L., Smith, D. F. Culture medium for enterobacteria. J Bacteriol. 119 (3), 736-747 (1974).
- Hörnsten, E. G. On culturing Escherichia coli on a mineral salts medium during anaerobic conditions. Bioprocess Eng. 12 (3), 157-162 (1995).
- Davis, B. D. The Isolation of Biochemically Deficient Mutants of Bacteria by Means of Penicillin. P Natl Acad Sci USA. 35 (1), 1-10 (1949).
- Sambrook, J., Russell, D. W. Molecular Cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY, USA. (2001).
- Wang, Y. -S., et al. The de novo engineering of pyrrolysyl-tRNA synthetase for genetic incorporation of L-phenylalanine and its derivatives. Mol Biosyst. 7 (3), 714-717 (2011).
- Bae, J. H., et al. Expansion of the genetic code enables design of a novel "gold" class of green fluorescent proteins. J Mol Biol. 328 (5), 1071-1081 (2003).
- JoVE Science Education Database. Dialysis: Diffusion Based Separation. J Vis Exp. , Cambridge, MA. (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Separating Protein with SDS-PAGE. J Vis Exp. , (2017).
- Petrásek, Z., et al. Excitation energy transfer from phycobiliprotein to chlorophyll d in intact cells of Acaryochloris marina studied by time- and wavelength-resolved fluorescence spectroscopy. Photoch Photobio Sci. 4 (12), 1016-1022 (2005).
- Kolber, Z. S., Barkley, M. D. Comparison of approaches to the instrumental response function in fluorescence decay measurements. Anal Biochem. 152 (1), 6-21 (1986).
- Pelet, S., Previte, M. J. R., Laiho, L. H., So, P. T. C. A fast global fitting algorithm for fluorescence lifetime imaging microscopy based on image segmentation. Biophys J. 87 (4), 2807-2817 (2004).
- Loefroth, J. E. Time-resolved emission spectra, decay-associated spectra, and species-associated spectra. J Phys Chem. 90 (6), 1160-1168 (1986).
- Hartman, M. C. T., Josephson, K., Lin, C. -W., Szostak, J. W. An expanded set of amino acid analogs for the ribosomal translation of unnatural peptides. PLoS One. 2 (10), e972 (2007).
- Budisa, N., et al. Global replacement of tryptophan with aminotryptophans generates non-invasive protein-based optical pH sensors. Angew Chem Int Edit. 41 (21), 4066-4069 (2002).
- Ma, Y., Biava, H., Contestabile, R., Budisa, N., di Salvo, M. L. Coupling bioorthogonal chemistries with artificial metabolism: intracellular biosynthesis of azidohomoalanine and its incorporation into recombinant proteins. Molecules. 19 (1), 1004-1022 (2014).
- Teramoto, H., Kojima, K. Incorporation of Methionine Analogues Into Bombyx mori Silk Fibroin for Click Modifications. Macromol Biosci. 15 (5), 719-727 (2015).
- Deal, R. B., Henikoff, J. G., Henikoff, S. Genome-wide kinetics of nucleosome turnover determined by metabolic labeling of histones. Science. 328 (5982), 1161-1164 (2010).
- Hinz, F. I., Dieterich, D. C., Tirrell, D. A., Schuman, E. M. Non-canonical amino acid labeling in vivo to visualize and affinity purify newly synthesized proteins in larval zebrafish. ACS Chem Neurosci. 3 (1), 40-49 (2012).
- Dieterich, D. C., et al. In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat Neurosci. 13 (7), 897-905 (2010).
- Dieterich, D. C., Link, A. J., Graumann, J., Tirrell, D. A., Schuman, E. M. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). P Natl Acad Sci USA. 103 (25), 9482-9487 (2006).
- Glenn, W. S., et al. Bioorthogonal Noncanonical Amino Acid Tagging (BONCAT) Enables Time-Resolved Analysis of Protein Synthesis in Native Plant Tissue. Plant Physiol. 173 (3), 1543-1553 (2017).
- Zhou, L., et al. Incorporation of tryptophan analogues into the lantibiotic nisin. Amino Acids. 48 (5), 1309-1318 (2016).
- Acevedo-Rocha, C. G., Budisa, N. Xenomicrobiology: a roadmap for genetic code engineering. Microb Biotechnol. 9 (5), 666-676 (2016).
- Agostini, F., Völler, J. -S., Koksch, B., Acevedo-Rocha, C. G., Kubyshkin, V., Budisa, N. Biocatalysis with Unnatural Amino Acids: Enzymology Meets Xenobiology. Angew Chem Int Edit. 56 (33), 9680-9703 (2017).
- Bacher, J. M., Ellington, A. D. Selection and characterization of Escherichia coli variants capable of growth on an otherwise toxic tryptophan analogue. J Bacteriol. 183 (18), 5414-5425 (2001).
- Wong, J. T. Membership mutation of the genetic code: loss of fitness by tryptophan. Pc Natl Acad Sci USA. 80 (20), 6303-6306 (1983).
- Hoesl, M. G., et al. Chemical Evolution of a Bacterial Proteome. Angew Chem Int Edit. 54 (34), 10030-10034 (2015).
- Italia, J. S., et al. An orthogonalized platform for genetic code expansion in both bacteria and eukaryotes. Nat Chem Biol. 13 (4), 446-450 (2017).
- Völler, J. -S., Thi To, T. M., Biava, H., Koksch, B., Budisa, N. Global substitution of hemeproteins with noncanonical amino acids in Escherichia coli with intact cofactor maturation machinery. Enzyme Microb Tech. 106, 55-59 (2017).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Völler, J. -S., Budisa, N. Coupling genetic code expansion and metabolic engineering for synthetic cells. Curr Opin Biotech. 48, 1-7 (2017).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr Opin Chem Biol. 14 (6), 774-780 (2010).
- Somsen, O. J., van Grondelle, R., van Amerongen, H. Spectral broadening of interacting pigments: polarized absorption by photosynthetic proteins. Biophys J. 71 (4), 1934-1951 (1996).
- Kurschus, F. C., Pal, P. P., Bäumler, P., Jenne, D. E., Wiltschi, B., Budisa, N. Gold fluorescent annexin A5 as a novel apoptosis detection tool. Cytom Part A. 75 (7), 626-633 (2009).
- Lepthien, S., Wiltschi, B., Bolic, B., Budisa, N. In vivo engineering of proteins with nitrogen-containing tryptophan analogs. Appl Microbiol Biot. 73 (4), 740-754 (2006).
- Wachter, R. M., Elsliger, M. -A., Kallio, K., Hanson, G. T., Remington, S. J. Structural basis of spectral shifts in the yellow-emission variants of green fluorescent protein. Structure. 6 (10), 1267-1277 (1998).
- Verkhusha, V. V., Lukyanov, K. A. The molecular properties and applications of Anthozoa fluorescent proteins and chromoproteins. Nat Biotechnol. 22 (3), 289-296 (2004).
- Martynov, V. I., Savitsky, A. P., Martynova, N. Y., Savitsky, P. A., Lukyanov, K. A., Lukyanov, S. A. Alternative cyclization in GFP-like proteins family. The formation and structure of the chromophore of a purple chromoprotein from Anemonia sulcata. J Biol Chem. 276 (24), 21012-21016 (2001).
- Piatkevich, K. D., Malashkevich, V. N., Morozova, K. S., Nemkovich, N. A., Almo, S. C., Verkhusha, V. V. Extended Stokes shift in fluorescent proteins: chromophore-protein interactions in a near-infrared TagRFP675 variant. Sci Rep. 3 (1), 1847 (2013).



