Summary
Syntetisk biologi gjør prosjektering av proteiner med enestående egenskaper med co-translational innsetting av ikke-kanoniske aminosyrer. Her, presenterte vi hvordan en spectrally rød-skiftet variant av en GFP-type fluorophore med romanen fluorescens spektroskopiske egenskaper, kalt "gold" fluoriserende protein (GdFP), er produsert i E. coli via seleksjonspress inkorporering (SPI).
Abstract
Fluorescerende proteiner er grunnleggende verktøy for biovitenskap, spesielt for fluorescens mikroskopi levende celler. Mens vill-type og utviklet varianter av grønne fluorescerende protein fra Aequorea victoria (avGFP) samt homologs fra andre arter allerede dekker store deler av optisk spekteret, fortsatt en spektral gap i nær-infrarøde regionen, for hvilke avGFP-baserte fluorophores er ikke tilgjengelige. Rød-skiftet fluorescerende protein (FP) varianter ville vesentlig utvide et verktøysett for spectral unmixing av flere molekylær arter, men naturlig forekommende rød-skiftet FPs avledet fra koraller eller sjøanemoner har lavere fluorescens quantum avkastning og dårlig bilde-stabilitet sammenlignet med varianter av avGFP. Ytterligere manipulasjon og mulige utvidelsen av chromophore's konjugert systemet mot langt rødt spectral regionen er også begrenset av repertoaret av 20 kanoniske aminosyrer foreskrevet av den genetiske koden. For å overvinne disse begrensningene, kan syntetisk biologi oppnå ytterligere spectral rød-skiftende via innsetting av ikke-kanoniske aminosyrer chromophore triad. Vi beskriver bruken av SPI til ingeniør avGFP varianter med romanen spektrale egenskapene. Protein uttrykk utføres i en tryptofan-auxotrophic E. coli belastning og supplere vekst medier med egnet indol prekursorer. I cellene, er disse prekursorer konvertert til den tilsvarende tryptofan analogs og innlemmet i proteiner av ribosomal maskineriet svar UGG kodon. Utskifting av Trp-66 i den forbedrede "cyan" varianten av avGFP (ECFP) av et elektron-donere 4-aminotryptophan resulterer i GdFP med et 108 nm Stokes Skift og et sterkt rød-skiftet utslipp maksimal (574 nm), samtidig som thermodynamically mer stabilt enn forgjengeren ECFP. Rest-spesifikke innlemmelse av ikke-kanoniske aminosyren analyseres av massespektrometri. Spektroskopiske egenskapene til GdFP er preget av tid-løst fluorescens spektroskopi som en av de verdifulle anvendelser av genetisk kodet FPs i biovitenskap.
Introduction
Siden oppdagelsen av grønne fluorescerende proteinet i maneter Aequorea victoria (avGFP) i 19621 og første heterologous uttrykk i 19942 i andre eukaryotic celler, har fluorescerende proteiner av GFP familien blitt svært verdifulle verktøy og mål i biovitenskap. Omfattende genetiske og molekylære prosjektering inkludert justering av artsspesifikke codon bruk, akselerasjon av folding, forbedret modning, økt lysstyrke, forebygging av oligomerization og skreddersy spectral og fotokjemisk egenskaper inkluderer evnen å reversibel photoswitch3,4,5,6. GFP skylder sin fluorescens fra sin 4-(p- hydroxybenzylidene) imidazolidin-5-en (HBDI) chromophore. Sistnevnte er autocatalytically dannet fra den såkalte chromophore triaden av aminosyrer (Ser-65/Tyr-66/Gly-67 i avGFP) etter dannelsen av en ekstra kovalent binding i peptid ryggraden under påvirkning av molekylære oksygen7. Resonantly stabilisert konjugert systemet kommuniserer dynamisk med sin molekylære miljø, slik at absorpsjon i det synlige området og karakteristiske grønne fluorescens av disse proteinene.
Innen chromophore triaden er tilstedeværelsen av en aromatiske aminosyre obligatorisk. Men omfatter standard aminosyre repertoaret bare fire aromatiske rester (hans Phe, Trp og Tyr). Dette begrenser konvensjonelle mutagenese tilnærminger for å oppnå vesentlig mer rød-skiftet avGFP varianter i forhold til mest rød-skiftet naturlig FPs som DsRed8 fra Discosoma striata coralimorphs eller mKate/mNeptune9 fra sjøanemonen Entacmaea quadricolor. Derfor den langt rødt og nær-infrarøde delen av optisk spekteret over 600 nm er tynt dekket av GFP varianter. Dette er selvfølgelig en alvorlig begrensning for fluorescens mikroskopiske tilnærminger som krever spectral demultiplexing av flere fluorophore samtidig. For eksempel lang-bølgelengde markører er også nødvendig for å gjøre bruk av lav absorpsjon regimet av huden vevet mellom 700-1000 nm i innstillingene for dype vev imaging10.
Fluorescerende proteiner fra avGFP er delt i flere klasser basert på spektroskopiske egenskaper og kjemisk natur deres chromophores11. Med sin triaden Ser-65/Tyr-66/Gly-67, vill-type chromophore eksisterer som en equilibrated blanding mellom skjemaet nøytral, fenoliske (λMaks = 395 nm, ε = 21000 M-1cm-1) og anionic phenolate (λMaks = 475 nm, ε = 7,100 M -1cm-1), og utslipp spektrum utstillinger en enkelt peak på 508 nm. Gruppen hydroksyl Ser-65 er av avgjørende betydning, som det donerer et H-bånd til Glu-222 chromophore i nærheten (avstand: 3.7 Å), som fremmer ionisering av denne carboxylate. Klassen jeg er preget av en anionic phenolate chromophore, som EGFP (Phe-64-Leu/Ser-65-Thr; λMaks = 488 nm, ε = 35 600 M-1cm-1, λem = 509 nm). På grunn av Ser-65-Thr(Ala,Gly) substitusjonsbehandling, 395 nm eksitasjon toppen av skjemaet nøytral fenol er undertrykt og 470-475 nm toppen av den anionic phenolate er fem - til six ganger utvidet og flyttet til 490 nm. Klasse II består av proteiner med en nøytral fenoliske chromophore, som safir-GFP. Her Thr-203-Ile substitusjon nesten helt undertrykker 475 nm magnetisering, forlater bare toppen på 399 nm. Siden anionic chromophore ikke kan være riktig solvated, foretrukket sin nøytrale form. Klasse III består av "gul" fluoriserende variantene (EYFP; Ser-65-Gly/Val-68-leu/ser-72-ala/THR-203-Tyr; ΛMaks ε = 514 nm, ε = 84, 600 M-1cm-1, λem = 527 nm) med π-stabling interaksjon en aromatiske side kjede og phenolate, som forårsaket av Thr-203-His(Trp,Phe,Tyr) erstatninger, som fører til opptil 20 nm rød-skiftet utslipp maxima (Thr-203-Tyr). Ytterligere substitusjonsbehandling (Gln-69-Lys) resultater i en annen 1-2 nm rød Skift til 529 nm, mest rød-skiftet avGFP varianten kjent11. Utveksling av fenol for en indol (Tyr-66-Trp) oppretter klasse IV i cyan-fluorescerende ECFP (Ser-65-Thr/Tyr-66-Trp; λmax1 = 434 nm, ε = 24,800 M-1cm-1; λmax2 = 452 nm, ε = 23,600 M-1cm-1 ; Λem1 = 477 nm, λem2 = 504 nm). Innkvarteringen av store indol er sannsynligvis aktivert av andre, kompenserende mutasjoner. ECFP eksitasjon og utslipp maxima faller innimellom de av proteiner med nøytral eller anionic chromophores. Klassen V proteiner havn en imidazole i stedet for fenol (Tyr-66-hans), f.eks., blå-fluorescerende proteiner som EBFP. Klassen VI er produsert av en fenol-til-fenyl utveksling favoriserer skjemaet nøytrale chromophore utelukkende, som dermed fører til de mest blå-forskjøvet eksitasjon og utslipp peak stillingene (360 nm og 442 nm, henholdsvis).
Klassisk område-rettet mutagenese er spesielt egnet for produksjon av romanen avGFP chromophore varianter, ved Permutasjon av 65-67 tripeptide og samspill rester i rammen av 20 kanoniske aminosyrer. Disse mulighetene kan utvides ytterligere når ikke-kanoniske varianter av aromatiske aminosyrer er introdusert under ribosomal protein syntese12. I prinsippet finnes det to måter å oppnå dette. Første strategi er avhengig av underlaget toleranse av protein oversettelse maskiner, spesielt av aminoacyl-tRNA synthetases (aaRSs) mot relaterte aminosyre analogs. Å oppnå dette med høy effektivitet, auxotrophic E. coli uttrykket stammer er ansatt som ikke klarer å syntetisere den tilsvarende naturlige aminosyren. Dette tillater utskifting av sistnevnte legge til passende ikke-kanoniske aminosyrer (ncAAs) eller forløpere derav til kultur medium. Denne strategien, også kjent som selektiv press inkorporering (SPI)13,14, kan rest-spesifikke erstatninger, som resulterer i globale innlemmelse av ncAA. Den andre bruker stopp codon suppressor tRNAs som belastes med ncAA av konstruert aaRS enzymer. Dette resulterer i readthrough av i-ramme stopp kodon og tillater områdespesifikke ncAA innlemmelse. Derfor fører denne metoden av stopp codon undertrykkelse (SCS) til utvidelse av genetiske koden15. Via mutagenese plasseres en stopp codon i målet genet på ønsket område. I prinsippet kan SPI også brukes til å opprette rekombinant peptider og proteiner bærer en unik ncAA installasjon, gitt at sjeldne kanoniske aminosyrer som Met eller Trp er valgt for ressurserstatting. Med Trp, SPI tilnærminger har vist seg å arbeide med en rekke analogs inkludert 4 - F-, 5 - F - og 6-F-Trp, 7-aza-Trp, 4-OH - og 5-OH-Trp, samt 4- og 5-NH2- Trp eller selv β (thienopyrrolyl) alanin derivater16 ,17,18,19,20. Dermed kan SPI være svært fordelaktig for å erstatte aromatiske aminosyrer av GFP chromophores av ikke-kanoniske varianter å utforske muligheten for å ytterligere tilpasse spectra og Stokes skifte av disse FPs. Som for alle protein sekvens modifikasjoner, må kompatibilitet med FP folding og chromophore modning testes eksperimentelt.
I dette arbeidet bruker vi klasse IV ECFP21, som bærer i stedet for vill-type avGFP Tyr, en Trp rester i sin chromophore triade. Bruker SPI, er denne Trp-66 (og Trp-57, bare andre Trp rester i ECFP) erstattet av 4-amino-Trp. Tilstedeværelsen av elektron-donere amino gruppen av 4-amino-Trp i chromophore favoriserer resonans stabilisering av langt rød-skiftet opphisset tilstand proton transport (ESPT) med et 108 nm Stokes skifte. Denne "gull" fluoriserende protein (GdFP) utgjør varianten med største røde skifte av fluorescens maksimal (574 nm) blant alle avGFP-avledet proteiner. Vi beskriver metoden GdFP protein produksjon av SPI og tilbyr protokollene for obligatorisk analyse av resulterende endret proteiner av masse spectroscopy. Videre, vi viser hvordan GdFP kan brukes og analyseres i tid-løst fluorescens spektroskopi tilnærminger.
Protocol
1. endring av Trp-auxotrophic E. coli
- Transform kjemisk eller electrocompetent celler (50 µL) Trp-auxotrophic E. coli belastning, f.eks. ATTC 49980 (WP2, mutant avledet fra E. coli belastning B/R22), med 1 µL av en 1 ng/µL vandig løsning av pQE - 80 L His6-ECFP plasmider med varme støt eller electroporation, henholdsvis. Se JoVE vitenskap utdanning Database23,24 for detaljer.
Merk: Uttrykket vektoren pQE - 80L His6-ECFP koder en N-terminalt 6 x hans-merket ECFP21 drevet av en bakteriell T5 promoter operatoren lac. Videre bærer en AmpR markøren og en colE1 opprinnelse replikering (pQE - 80 L vektor ryggraden sekvensen kan finnes på: https://www.qiagen.com/mx/resources/resourcedetail?id=c3b71572-4d82-4671-a79b-96357fe926d1&lang=en & autoSuggest = true). Den teoretiske Molekylvekten av His6-ECFG vill-type protein (etter chromophore modning25) er 28303.92 Da. Oversatt målet protein sekvensen er som følger (hans-tag understreket, vektor-avledet sekvenser i fet skrift): MRGSHHHHHHGSMVSKGEELFTGVVPILVELDGDVNGHKFSVSGEGEGDATYGKLTLKFICTTGKLPVPWPTLVTTLTWGVQCFSRYPDHMK
QHDFFKSAMPEGYVQERTIFFKDDGNYKTRAEVKFEGDTLVNRIELKGIDFKEDGNILGHKLEYNYISHNVYITADKQKNGIKANFKIRHNIEDGS
VQLADHYQQNTPIGDGPVLLPDNHYLSTQSALSKDPNEKRDHMVLLEFVTAAGITLGMDELYK. - Plate transformert celler i LB-agar plater (tabell 1) med 10 g/L glukose, 100 µg/mL ampicillin og ruge platene på 37 ° C over natten.
2. rekombinant Protein uttrykk
-
Natten kultur av E. coli ATCC 49980 pQE - 80 L His6-ECFP
- Forberede 5 mL av LB medium (tabell 1; supplert med 10 g/L glukose, 100 µg/mL ampicillin) i et sterilt 14 mL polystyren kultur rør for aerobic vekst og vaksinere med en enkeltvis colony fra en agar tallerken med en bakteriefri pipette tips eller inokuleringen sløyfe.
Merk: Bruke kolonier fra ferske transformert celler anbefales. Platene med bakteriell koloniene (fra trinn 1.2.) kan lagres på 4 ° C i flere dager. - Inkuber cellene på 37 ° C i en orbital shaker på 200-250 rpm over natten.
- Forberede 5 mL av LB medium (tabell 1; supplert med 10 g/L glukose, 100 µg/mL ampicillin) i et sterilt 14 mL polystyren kultur rør for aerobic vekst og vaksinere med en enkeltvis colony fra en agar tallerken med en bakteriefri pipette tips eller inokuleringen sløyfe.
-
Uttrykket av vill-type ECFP
- Vaksinere 10 mL frisk LB medium (tabell 1; supplert med 10 g/L glukose, 100 µg/mL ampicillin) med 100 µL av natten kultur i en 100 mL Erlenmeyer kolbe. Inkuber kolbe på 37 ° C i en orbital shaker på 200 rpm.
Merk: Hvis dette trinnet kan utføres i 10 mL NMM19 medium (tabell 1) med 100 µg/L ampicillin og 0,5 mM L-tryptofan (eventuelt indol kan brukes). - Måle optisk densitet ved 600 nm (OD600) hver 20 min. fortrinnsvis måle celle tetthet ved å bestemme utryddelse på 600 nm (OD600) i et spektrofotometer bruke søppel med banen lengde på 1 cm. alltid utføre en referanse måling ved hjelp av de tilhørende kultur medium. Fortynne prøver og blanding prøvene godt for å få en måling verdi på 0,1 0,8, deretter beregne OD600 med fortynning faktor. For detaljer, se forrige publikasjonen 26.
- På å nå en OD600 verdi på 0,5 0,8 (ca 2-3 h etter inoculation), ta prøve "før induksjon" SDS-siden (natrium dodecyl sulfate polyakrylamid gel geleelektroforese, trinn 4).
- Indusere protein måluttrykk ved å justere flytende kulturen til 0,5 mM IPTG (isopropyl β-D-1-thiogalactopyranoside, fra 1 M lagerløsning) og ruge det ved 30 ° C i en orbital shaker på 200 rpm for 4-8 h.
Merk: Cyan fluorescerende proteiner uttrykkes vanligvis ved temperaturer under 37 ° C27. - Ta prøve "etter uttrykket" SDS-siden (trinn 4.).
- Høste bakterieceller med sentrifugering i 10 min på 5000 x g og 4 ° C.
- Forkast nedbryting av dekantere vin og fryse celle pellets på 20 ° C eller-80 ° C til målet protein rensing.
- Vaksinere 10 mL frisk LB medium (tabell 1; supplert med 10 g/L glukose, 100 µg/mL ampicillin) med 100 µL av natten kultur i en 100 mL Erlenmeyer kolbe. Inkuber kolbe på 37 ° C i en orbital shaker på 200 rpm.
-
SPI for produksjon av GdFP
- Vaksinere 10 mL av NMM19 medium (tabell 1) med 100 µg/mL ampicillin, 15 µM tryptofan og 10 µL av natten kultur i en 100 mL Erlenmeyer kolbe og ruge kultur kolbe overnatting på 30 ° C i en orbital shaker på 200 rpm.
Merk: Det finnes en rekke kjemisk definert medier for dyrking av E. coli og SPI. I tillegg til NMM brukes her, MOPPER middels28, glukose-mineral salter medium29, Davis minimal middels30, M9 minimal middels31eller GMML32 kan brukes. - Neste dag, måle OD600 enhver 30 min før verdien bare endres med mindre enn 0.05 over 30 min. Platået verdien bør være ca 1.
Merk: Avvik av ± 0,3 enheter er akseptable. Avhengig av bakterielle belastningen og mediet som brukes, må den første tryptofan konsentrasjonen (trinn. 2.3.1) justering. - Ta prøve "før induksjon" SDS-siden (trinn 4.).
- Høste bakterieceller med sentrifugering i 10 min på 5000 x g og 4 ° C. Kast nedbryting av dekantere vin.
- Resuspend cellene i 10 mL av NMM19 medium med 100 µg/mL ampicillin i en 100 mL Erlenmeyer kolbe og legge 4-amino-indol til en siste konsentrasjon av 1 mM med 50 mM lagerløsning. Fortsette inkubasjon i 30 min på 30 ° C i en orbital shaker på 200 rpm.
Merk: Dette trinnet er anbefalt på grunn av lav kjemiske stabiliteten av ampicillin og sikrer mobilnettet opptak av 4-amino-indol. - Indusere protein måluttrykk ved å legge IPTG til en siste konsentrasjon på 0,5 mM bruke 1 M og ruge prøven overnatting på 30 ° C i en orbital shaker på 200 rpm.
Merk: Cyan fluorescerende proteiner uttrykkes vanligvis ved temperaturer under 37 ° C27. - Neste dag, måle OD600.
- Ta prøve "etter uttrykket" SDS-siden (trinn 4.).
- Høste bakterieceller med sentrifugering i 10 min på 5000 x g og 4 ° C og kast nedbryting av dekantere vin.
- I tilfelle slike et fartøy ikke ble brukt for sentrifugering, overføre celle pellets til en 50 mL konisk polystyren rør ved hjelp av en slikkepott. Fryse celle pellet på 20 ° C eller-80 ° C til målet protein rensing.
- Vaksinere 10 mL av NMM19 medium (tabell 1) med 100 µg/mL ampicillin, 15 µM tryptofan og 10 µL av natten kultur i en 100 mL Erlenmeyer kolbe og ruge kultur kolbe overnatting på 30 ° C i en orbital shaker på 200 rpm.
3. mål Protein rensing via immobilisert metall Ion affinitet kromatografi (IMAC)
-
Bakteriell celle lysis
- Tine celle pellet på is 10-20 minutter.
- Resuspend celle pellet i et 50 mL konisk polystyren rør med 5 mL av iskalde bindende buffer (tabell 1) på is.
- Legge til 20 µL av 50 mg/mL lysozyme, 20 µL av 1 mg/mL DNase jeg 20 µL av 1 mg/mL RNase A. Lukk røret, bland forsiktig ved å snu 5 ganger og holde det på is 30 min.
Merk: Delvis celle avbrudd oppstår som katalysert av lysozyme. - Lyse cellene av sonification ved hjelp av en ultralyd homogenizer tips bruke tre sykluser av 3 min i en 15 mL polystyren tube avkjølt av slaps isen med 2 s av puls, 4 s av pause og 45% amplitude.
Merk: Alternativt høytrykk homogenisering kan brukes f.eks., 20 sykluser på 14.000 psi. Eventuelt kan du fortynne bruker bindingen buffer for å nå minimal instrument volumet. Protein utvinning reagenser kan dessuten brukes til celle avbrudd. Se materialer tabell eksempler. - Sentrifuger prøven i 30 min 15,000-18,000 x g, 4 ° C.
- Overføre nedbryting i en fersk rør og skrive ned flytende volumet.
- Filtrere løsningen gjennom et 0,45 µm sprøyte filter bruker en 5 mL Luer lås plastsprøyte og filtere polyvinylidene fluor (PVDF) sprøyten.
- Ta prøve "lysate" for SDS-side (trinn 4.).
- Resuspend celle rusk pellet i ddH2O (like volum som tidligere lysate).
- Ta prøven "pellet" for SDS-side (trinn 4.).
-
IMAC rensing
- Bruk en 1 mL ferdigpakkede eller selv pakket IMAC FPLC (rask protein flytende kromatografi) kolonne i henhold til produsentens instruksjoner. Bruk bindingen buffer (tabell 1) for kolonnen balanse samt for vask trinnet som følger etter cellen lysate brukes på kolonnen.
- Samle og basseng eluate brøker med GdFP som kan identifiseres av synlig lys gylden farge.
Merk: Hvis målet protein kan være elut med lineær imidazole gradering (0-250 mM) bruker en automatisert system for FPLC. - Bestemme protein konsentrasjonen med litteratur verdien for utryddelse koeffisient på 466 nm (ɛ466 nm = 23,700 M-1 cm-1)33 med elueringsbufferen som referanse. Informasjon om prosedyren, se forrige publikasjonen26.
- Ta prøven "eluate" for SDS-siden, og bruk 1-10 µg protein per kjørefelt ved Coomassie flekker.
Merk: SDS eksempel beløp kan variere avhengig av flekker metoden og fargestoff følsomhet. - Dialyze en aliquot av eluate fraksjoner mot dialyse buffer eller MS buffer med en membran med en molekylvekt cutoff (MWCO) av 5000-10, 000. Forberede dialyse membran i henhold til produsentens instruksjoner. Dialyze en 1 mL prøve tre ganger mot 100 mL bufferen for minst 2 timer. Informasjon om denne fremgangsmåten, se forrige publikasjonen34.
- For lagring, fryse protein eksempel dialyse buffer på-80 ° C.
Merk: Dele bør være stabil i minst 6 måneder.
4. SDS side eksempel utarbeidelsen av E. coli hele cellen ekstrakt
- Overføre en celle suspensjon tilsvarende 1 mL av OD600 = 1 suspensjon (f.eks. 500 µL OD600= 2) i en 1,5 mL microcentrifuge tube.
- Høste celler med sentrifugering i 10 min 5000 x g, romtemperatur. Kast nedbryting av pipettering.
- Legge til 80 µL ddH2O og 20 µL 5 x eksponeringsmåter lasting fargestoff buffer (tabell 1) cellen pellets og blanding av pipettering.
- Denature cellene ved å varme til 95 ° C i 5 minutter i en vann bad eller varme blokk. Deretter kule prøvene til romtemperatur.
- Bruk 10 µL Coomassie-farget SDS-siden etter forrige publikasjonen35.
Merk: SDS eksempel beløp kan variere avhengig av flekker metoden og fargestoff følsomhet.
5. intakt Protein masse analyse av høy ytelse flytende kromatografi (HPLC) koblet til Electrospray Ionization Time of flight massespektrometri (LC-ESI-TOF-MS)
Merk: HPLC gradering, innstillinger og buffere kan variere avhengig av separasjon kolonne og instrument som brukes. Se materialer tabell for eksemplarisk utstyr.
- Bestemme protein konsentrasjonen fra et utvalg dialyzed mot MS bufferen som beskrevet ovenfor (trinn 3.2.3.) bruker MS buffer (se materialer tabell) som referanse.
- Fortynne protein prøven til 0,1 mg/mL bruker MS buffer for en endelig mengde 80 µL, bland ved forsiktig pipettering, overføre løsningen til MS autosampler ampuller med Glassinnsatsen og lukke den med en lue. Fjerne luftbobler ved å sveipe ampullen.
- Fyll andre autosampler ampuller uten Glassinnsatsen (buffer tomt) med 1 mL av MS buffer.
- For at maskinen skal varme opp. Utføre instrument kalibrering. Kontroller at det finnes tilstrekkelig mengder væske kromatografi førsteklasses løsemidler (> 100 mL).
- Programmet lineær 20 min HPLC gradering fra 5% til 80% bufferen en (0,1% maursyre i ddH2O), kombinert med buffer B (0,1% maursyre i acetonitrile).
- Start HPLC på en flyt på 0,3 mL/min og vent til kolonnen trykket er stabil.
- Angi en autosampler injeksjon volum på 5 µL for metoden LC-ESI-TOF-MULTIPLE Sclerosis, opprette en worklist for en tom kjøre etterfulgt av en prøve-kjøre og tilordne den tilsvarende autosampler medisinglass posisjoner. Kjøre worklist.
- Etter worklist, åpne den genererte eksempeldatafil. Merke et område i totale ion gjeldende (TIC) tomten for deconvolution og deconvolute MS spekteret med maksimum entropi deconvolution algoritmen.
Merk: Avhengig av eksperimentelle forhold, flere arter kan oppstå fra umodne FP eller bufferen ion addukter.
6. fluorescens levetid målinger og forfall-assosiert spektra (DAS) av GdFP
Merk: For instrumentering av tid-løst fluorescens spektroskopi, se Tabellen for materiale for eksemplarisk utstyr. Absorbansen samt fluorescens eksitasjon og utslipp spektra av fluorescerende proteiner kan også registreres med laboratoriet UV/Vis og fluorescens Spektrofotometrene.
-
Bølgelengde-løst fluorescens levetid måling av GdFP
- Forberede 2 mL på 1 µM løsning av GdFP av fortynning i PBS buffer (tabell 1) ved pH 7. Fylle ut løsningen i 1 cm kvarts søppel.
- Installere ps-pulsed 470 nm laser for eksempel eksitasjon og 488 nm lang-pass utslipp filteret, og justere 600 L/mm rist av den tid - og bølgelengde-korrelert enkelt Foton teller36 (TWCSPC) detektor for oppkjøpet av bølgelengde regimet 500-700 nm.
- Erverve fluorescens utslipp med en teller hastighet på ca 200 x 103 fotoner/s til ca 103 teller er akkumulert i oppkjøpet maksimal fluorescens forfall kurver med enkelt Foton telling programvare.
-
Måling av instrumental svar funksjon37 (IRF)
- Erstatte eksempel cuvette med 1 cm kvarts søppel fylt med 1 finans kolloidal silika (~ 220 m2/g) i PBS buffer ved pH 7.
Merk: Silika suspensjon laget av en 400 finans vandig suspensjon. - Fjerne 488 nm lang-pass utslipp filter og sett inn grå filtre for å justere antall på TWCSPC detektoren til under 100 x 103 teller/s.
- Justere rist for kjøp av 470 nm fotoner i kanalen 8 av 16-kanal TWCSPC detektor.
- Få IRF til ca 10 x 103 teller er akkumuleres i utslipp maksimal.
- Konvertere fluorescens forfall kurver og IRF til ASCII datafiler med global passende38 programmet.
- Oppførsel Global passer etter en modell av en sum av tre eksponentiell komponenter med levetid som koblede parametere.
- Tomten forfall knyttet spectra (DAS) som amplituden distribusjoner av personlige forfall komponenter i avhengighet av bølgelengden dataanalyse programvare.
- Erstatte eksempel cuvette med 1 cm kvarts søppel fylt med 1 finans kolloidal silika (~ 220 m2/g) i PBS buffer ved pH 7.
Representative Results
Bruker teknikken av seleksjonspress innlemmelse, kan Trp-66 i chromophore triaden av ECFP (og Trp-57, bare andre Trp rester i ECFP) erstattes av 4-amino-Trp, og dermed generere den rød-skiftet GdFP med forskjellige spektrale egenskapene. Massespektrometri må brukes til å vise ønsket stoichiometric integrering av den ikke-kanoniske aminosyren i protein, med resultater som vises i figur 1. Etterpå vi gi data fra mikroskopi, UV-Vis absorpsjon spektroskopi samt stabil og tid - og bølgelengde-løst fluorescens spektroskopi å karakterisere egenskaper for GdFP fluorophore med fokus på pH avhengigheten av den Spectra.
For å bekrefte utveksling av to Trp rester i ECFP av 4-amino-Trp, er masse spectrometric analyse utført. Figur 1 viser et representativ deconvoluted ESI-MS GdFP. Mens vill-type ECFP har en beregnet protein masse 28,283.9 Da etter chromophore modning, er tilsvarende masse GdFP 28,313.9 Da. Deconvoluted ESI-MS spekteret av GdFP viser en viktigste masse peak på 28,314.1 ± 0,1 Da, som avviker fra den teoretiske verdien av mindre enn 10 ppm. Å være innenfor de typiske nøyaktighet for denne typen analyse25, dette bekrefter inkorporering av ncAA via SPI (eksperimentell verdi for vill-type ECFP: 28,283.7 Da).
Figur 2 viser AC confocal fluorescens tenkelig mikroskopi (CFIM) bilder av bakterielle celler uttrykke ECFP, EGFP, EYFP og GdFP på rørets bakterier i PBS buffer. Alle bildene ble anskaffet på et mikroskop utstyrt med en UV mål og laser eksitasjon på om den samme energien for hvert utvalg.
Figur 3A viser en overlapping av CFIM bilder til E. coli bakterier uttrykke ulike FPs inkludert GdFP, alltid overvåkes med ligner eksitasjon energi (bølgelengder som i figur 2). Figur 3B viser chromophore strukturer av FP variantene vises. Om lysstyrken i GdFP sammenlignet med ECFP (fluorescens quantum avkastning φ = 0,4), EGFP (φ = 0,6) og EYFP (φ = 0,6) det er viktig å merke seg at for GdFP, en bredere oppkjøpet rekke fluorescens lyset (30 nm) ble brukt i motsetning til 20 nm brukes for alle andre arter CIES, for å justere intensiteten av bilder til tilsvarende verdier. Med en litt lavere utryddelse koeffisient og en redusert quantum avkastning av unike photophysical egenskaper, er lysstyrken på GdFP lavere sammenlignet med andre FPs vises.
Absorpsjon spekteret av ECFP (Figur 3 c) har to karakteristiske maxima 434 nm og 452 nm. Derimot GdFP er preget av en bred rød-skiftet absorpsjon band med maksimum på 466 nm. Absorpsjon av EGFP er ytterligere rød-skiftet til 488 nm. Men på grunn av mye større Stokes skifte av GdFP (108 nm) sammenlignet med ECFP (41 nm) og EGFP (20 nm), utslipp spektrum av GdFP er den mest rød-skiftet alle tre GFP derivater undersøkt her (figur 3D). Mens fluorescens utslipp av ECFP viser to karakteristiske maxima 475 nm og 505 nm, EGFP har en bred viktigste utslipp bandet topp på 508 nm (λmax) med en liten skulder på 540 nm. Fluorescens for GdFP vises med 565 nm (λMaks) (Figur 3D). Sine utslipp spektrum inneholder et lite bidrag av vill-type ECFP som også vises som en liten skulder på 475 nm. Denne ECFP brøkdel er syntetisert før induksjon under SPI-prosedyren som beskrevet33.
Figur 3E viser pH-avhengig endringer i absorpsjon spekteret av GdFP. For en pH endring fra 8 til 5, utslipp maksimal seg litt til rød og en liten utvidelse av absorpsjon bandet er observert. Reduksjon av absorpsjon amplituden er imidlertid bare ca 10% mellom pH 8 og pH 5, indikerer at egenskapene bakken staten GdFP chromophore er meget svakt endret av pH.
Tiden løst fluorescens utslipp overvåkes av én Foton teller er vist i Figur 4. Forfall kurvene overvåket i spektral kanaler sentrert på 550 nm og 600 nm (figur 4A) utstillingen en litt raskere fluorescens forfall på 600 nm forhold til forfallet på 550 nm. Resultatene av en global tilpasning av fluorescensen forfalle kurver med to eksponentiell komponenter resulterer i to spectrally skjelnes fluorescens forfall komponenter med tid konstantene av 1.0 ns og 3.3 ns (figur 4C og D).
Fluorescens utslipp av GdFP avhengig sterkt av pH, som det er typisk for mange fluorescerende protein varianter av GFP familien. Figur 4B sammenligner fluorescens utslipp av GdFP mellom pH 5 og pH 8, som tydelig viser en nedgang i fluorescens intensiteten på lavere pH, mens de spektrale egenskapene bo konstant.
Forfall-assosiert spectra (DAS)39 i GdFP (figur 4C og D) er preget av to forskjellige utslipp band. Bidraget fra de langsomme 3.3 ns komponenten er mer uttalt i kort bølgelengdeområde rundt 550 nm (60%) med mindre bidrag av komponenten raskere (40%). På 600 nm, begge komponentene har om samme amplituden. På et skifte fra pH 7 (figur 4C) til pH 6 (Figur 4 d), de spektrale egenskapene i DAS endre knapt tid konstantene globale montering rutiner er også det samme (nøyaktigheten av DAS tid konstantene er om ± 0,15 ns). Men de absolutte amplituder av to DAS-komponenter er tydelig tydelig, som fullt står for redusert fluorescens utslipp amplituden på samme pH skifte i figur 4B.
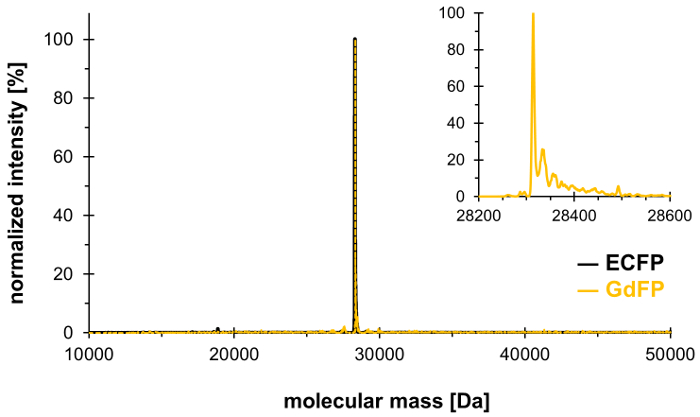
Figur 1: Representative deconvoluted ESI-MS spekteret av GdFP. ESI-MS spekteret av GdFP (gull farge, forstørret tomten vises som innfelte) viser et hoveddisplayet peak på 28314.1 Da (beregnet verdi 28313.9 Da). Spekteret for vill-type ECFP er vist i svart. Klikk her for å se en større versjon av dette tallet.

Figur 2: AC Confocal fluorescens mikroskopi bilder fra bakteriell populasjoner uttrykke ulike FPs. Følgende bølgelengde innstillinger ble brukt for bildeopptak: ECFP (λex = 457 nm, deteksjon: 461-480 nm), EGFP (λex = 488 nm, deteksjon: 495-515 nm), GDFP (λex = 476 nm, deteksjon: 560-590 nm), EYFP (λex = 514 nm, deteksjon: 520-530 nm). Klikk her for å se en større versjon av dette tallet.

Figur 3: spektrale egenskapene til GdFP. (A) CFIM bilde av en blanding av bakterielle celler uttrykke ECFP, EGFP og GdFP etter rørets bakterier i PBS buffer. (B) Chromophore strukturer av GdFP (med 4-amino-Trp i stedet for rester 66), foreldrekontroll ECFP (med Trp posisjonen 66) og EFGP (med Tyr posisjonen 66). (C) sammenligning av de normaliserte absorpsjon spektra av GdFP, ECFP og EGFP, mens i (D), normaliserte fluorescens utslipp spektrum av ECFP (eksitasjon på 430 nm) i forhold til fluorescens utslipp spektra av EGFP og GdFP (begge glade ved 450 nm). (E) pH-avhengighet av absorpsjon spectra (normalisert absorpsjon på 280 nm). Klikk her for å se en større versjon av dette tallet.

Figur 4: tid-løst fluorescens av GdFP. (A) fluorescens forfallet av GdFP overvåkes av gang - og bølgelengde-løst enkelt Foton teller i spektral kanaler sentrert på 550 nm og 600 nm (± 12,5 nm) etter eksitasjon med 470 nm laser pulser. Instrumental responsen funksjon (IRF) gir informasjon om tid oppløsningen for brukes. (B) variant av utslipp spekteret av GdFP avhengig av pH (eksitasjon på 460 nm). (C, D) Forfall-assosiert spektra (DAS) av GdFP ved pH 7 (C) og pH 6 (D) bestemmes etter deconvolution av gang - og bølgelengde-løst fluorescens henfall og globale montering av henfall i alle kanaler av et globalt sett to eksponentialfunksjoner med koblede tid konstantene. Klikk her for å se en større versjon av dette tallet.

Figur 5: strukturer av intramolekylære kostnad av ECFP (svart) og GdFP (gull) chromophores. Økningen i størrelsen på den chromophore systemet av god elektron giveren av en aminosyre som en del av ncAA kan dannelsen av mer mesomeric strukturer for å oppnå resonans stabilisering i glade delstaten. Koblingspunktene til FP stillaset vises som semicircles. Klikk her for å se en større versjon av dette tallet.
| Lagerløsning | konsentrasjon, løsemiddel | Merk | |
| 20% D-glukose | 200 g/L D-glukose i ddH2O | sterilisere ved filtrering gjennom filtere 0,45 µm pore størrelse sprøyte | |
| indol | 50 mM i isopropanol | ||
| 4-amino-indol | 50 mM i 20% etanol (20 mL etanol i et siste volum 100 ml fylt opp med ddH2O) | ||
| IPTG | 1 M ddH2O | ||
| L-tryptofan | 15 mM oppløst i ddH2O bruker 1 M HCl (legge HCl dropwise under omrøring til pulver er dissoved) | ||
| lysozyme | 50 mg/mL i ddH2O | ||
| DNase jeg | 1 mg/mL i ddH2O | ||
| RNase A | 1 mg/mL i ddH2O | ||
| Amp100 | 100 mg/mL ampicillin i ddH2O | ||
| natrium-dodecylsulfate (SDS) | 200 g/L i ddH2O | ||
| ammonium sulfate ((NH4)2SO4) | 1 M ddH2O | sterilisere av autoklavering | |
| kalium dihydrogen fosfat (KH2PO4) | 1 M ddH2O | sterilisere av autoklavering | |
| di-kalium hydrogen fosfat (K2HPO4) | 1 M ddH2O | sterilisere av autoklavering | |
| magnesium sulfat (MgSO4) | 1 M ddH2O | sterilisere av autoklavering | |
| D-glukose | 1 M ddH2O | sterilisere ved filtrering gjennom filtere 0,45 µm pore størrelse sprøyte | |
| natriumklorid (NaCl) | 5 M i ddH2O | sterilisere av autoklavering | |
| veisalt (CaCl2) | 1 g/L | sterilisere ved filtrering gjennom filtere 0,45 µm pore størrelse sprøyte | |
| Iron(II) chloride (FeCl2) | 1 g/L | sterilisere ved filtrering gjennom filtere 0,45 µm pore størrelse sprøyte | |
| Thiamin | 10 g/L | sterilisere ved filtrering gjennom filtere 0,45 µm pore størrelse sprøyte | |
| biotin | 10 g/L | sterilisere ved filtrering gjennom filtere 0,45 µm pore størrelse sprøyte | |
| sporstoffer blanding | kobber sulfat (CuSO4), sink klorid (ZnCl2), mangan klorid (MnCl2), ammonium molybdate ((NH4)2MoO4); hver 1 mg/L i ddH2O | sterilisere ved filtrering gjennom filtere 0,45 µm pore størrelse sprøyte | |
| 19 aminosyrer blanding | 1.) oppløsning 0,5 g L-fenylalanin og 0,5 g L-Tyrosin i 100 ml ddH2O dropwise tillegg 1 M HCl under omrøring til pulver er oppløst. | ||
| 2.) veie ut 0,5 g av hver av de resterende L-aminosyrer (unntatt L-tryptofan). Bland med 22 mL for 1 M KH2PO4 og 48 mL 1 M K2HPO4. Legge til ddH2O ca 800 mL. Rør til løsningen blir klart. | |||
| 3.) Legg den oppløste L-fenylalanin og L-Tyrosin fra trinn 1.) og juster volumet til 1 L med ddH2O. | |||
| 4.) Sterilize aminosyre blandingen av vakuum filtrering med en flaske topp filter enhet. | |||
| Buffere og Media | Sammensetning/forberedelse | ||
| SDS lasting fargestoff buffer, 5 x konsentrert | 0,25 M Tris pH 6.8, 50% v/v glyserol, 0,25% w/v bromphenol blå, 0,5 M didhiothreitol (DTT, alternativt 5% β-mercaptoethanol), 10% w/v natrium-dodecylsulfate (SDS) | ||
| bindende buffer | 50 mM natrium dihydrogenphosphate (NaH2PO4), 500 mM NaCl, 10 mM imidazole, pH 8 | ||
| elueringsbufferen | 50 mM natrium dihydrogenphosphate (NaH2PO4), 500 mM NaCl, 250 mM imidazole, pH 8 | ||
| dialyse buffer | 50 mM natrium dihydrogenphosphate (NaH2PO4), 150 mM NaCl, 100 mL/L glyserol, pH 8 | ||
| MS buffer | 10 mM Tris-HCl, pH 8 | ||
| nye minimal mediet som inneholder 19 L-aminosyrer unntatt L-tryptofan (NMM19) | Bland alle lager løsninger for å få de følgende siste konsentrasjonene: 7,5 mM (NH4)2SO4, 1,7 mM NaCl, 22 mM KH2PO4, 50 mM K2HPO4, 1 mM MgSO4, 20 mM D-glukose, 50 mg/L av 19 aminosyrer mix, 1 µg/L CaCl2, 1 µg/L FeCl2, 10 µg/L Thiamin, 10 mg/L biotin, 0,01 mg/L sporstoffer blanding | ||
| LB medium | Sammensetning: 10 g/L tryptone, 5 finans gjærekstrakt, 10 g/L NaCl, pH 7.0 i ddH2O | ||
| Forberedelse: | |||
| 1.) veie ut 50 g tryptone, 25 g gjærekstrakt, 5 g NaCl i en 1 L glassflaske. | |||
| 2). Legg til ddH2O til ~ 800 mL og oppløse komponenter under omrøring. | |||
| 3.) måle pH og justere pH 7 av dropwise tillegg 1 M HCl eller 1 M NaOH, om nødvendig. Legge til ddH2O til 1 L. | |||
| 4.) Sterilize av autoklavering, sjekker for volum tap og legge til sterilt ddH2O å kompensere om nødvendig. Lagre på 4 ° C før bruk. | |||
| LB agar plater | Sammensetning: 10 g/L tryptone, 5 finans gjærekstrakt, 10 g/L NaCl, 15 finans agar-agar, pH 7.0 i ddH2O | ||
| Forberedelse: | |||
| 1.) veie ut 50 g tryptone, 25 g gjærekstrakt, 5 g NaCl, 7.5 g agar-agar i en 1 L glassflaske. | |||
| 2). Legg til ddH2O opptil 500 mL og oppløse komponenter under omrøring. | |||
| 3.) måle pH og justere pH 7 av dropwise tillegg 1 M HCl eller 1 M NaOH, om nødvendig. Legge til ddH2O til 1 L. | |||
| 4.) Sterilize av autoklavering, sjekker for volum tap og legge sterilt ddH2O å kompensere, om nødvendig. (Merk: LB agar kan lagres på 4 ° C før bruk for utarbeidelse av LB agar plater. Nøye smelte befestet agar bruker mikrobølgeovn) | |||
| 5.) når løsningen er fortsatt varm (30-40 ° C), legge ampicillin til en siste konsentrasjon 100 µg/ml | |||
| 6.) Hell ca 15 mL av væsken fra trinn 5.) i et sterilt 10 cm Petriskål under sterile forhold. Når agar er styrket, kan plater lagres i 1 uke på 4 ° C før bruk. | |||
| fosfat-bufret saltvann (PBS) | Sammensetning: 137 mM NaCl, 2.7 mM KCl, 10 mM Na2HPO4, 1,8 mM KH2PO4, 1 mM CaCl2, 0,5 mM MgCl2, pH 7. Sterilisere autoklavering eller filtrering. | ||
Tabell 1: Lager løsning og buffer.
Discussion
For å oppnå høy ncAA innlemmelse effektivitet, avhengig auxotrophy-baserte SPI metoden av bruken av metabolically utviklet vertsceller som ikke kan syntetisere det tilsvarende naturlige motstykket i ncAA. E. colier slike stammer lett tilgjengelig. Selv samtidige inkorporering av flere ncAAs til samme protein er mulig med multiauxotrophic stammer. Rest-spesifikke modus erstatning og kjemiske repertoaret begrenses til lignende kjemisk analogs kan bli sett på som ulemper. Likevel kan et stort antall proteiner varianter produseres som naturlig bakteriell oversettelse apparatet tåler tallrike aminosyre analogs. For eksempel kan mer enn 50 ncAAs bli innlemmet i proteiner bruker i vitro oversettelse, utgjør ca 73% av alle kodon av genetiske koden skal være tilgjengelig for overføring40. Videre kan SPI også tillate effektiv multisite merking av målet protein41. I prinsippet SPI metodikken er ikke begrenset til E. coli, men kan arbeide i en annen vertsmaskin og for hver av de kanoniske 20 aminosyrene, forutsatt at auxotrophic stammer og definerte dyrking medier er tilgjengelige. For eksempel er to metionin analogs, azidohomoalanine (Aha) og homopropargylglycine (Hpg), kommersielt tilgjengelig og brukes for merking proteiner og proteomes i ulike organismer. Aha kan i tillegg produsert intracellulært og deretter innlemmet i protein42. Denne ncAA er spesielt egnet for bioorthogonal conjugations som Klikk kjemi som utviklet av Tirrel og kolleger: eksempelvis i plante vev av Arabidopsis thaliana, i Bombyx mori Larvene43, Drosophila celler44, larver sebrafisk45 samt pattedyrceller inkludert nerveceller46, proteiner kan være merket med Aha47,48. Tilsvarende ble Trp analogs vellykket innlemmet i antimikrobielle peptider i Trp-auxotrophic Lactococcus lactis stammer49. SPI er også nyttig for feltet i Xenobiology50,51, som utforsker alternativer til grunnleggende kjemiske make-up av livet. For eksempel ble basert på tidligere arbeider på E. coli52 og B. subtilis53, en E. coli belastning nylig utviklet av en evolusjonær strategi med seleksjonspress å bruke thienopyrrole i stedet for indol, noe som resulterer i proteom hele substitusjon av tryptofan av thienopyrrole-alanin i den genetiske koden54. Vanligvis presenterer den kanoniske aminosyren Trp, som er kodet av en enkelt triplett (UGG), et lovende mål protein engineering på grunn av de rike fasetter av Indolkjemi, som tilbyr mange kjemiske variasjoner. Nylig, og som et alternativ til SPI-baserte innlemmelse, rapportert en roman SCS plattform i stand til å innlemme Trp analogs site-specifically i både bakteriell og eukaryotic verter har vært55. Dette utvider ytterligere verktøykassen av i vivo ncAA-basert protein engineering, inkludert endring av spektrale egenskapene.
I tillegg til bruk av auxotrophic uttrykk verter krever SPI-protokollen strenge gjæring forhold, både når det gjelder målet uttrykk timing og sammensetningen av medium for å nå høy ncAA innlemmelse effektivitet og målet protein avkastning 56. dyrking er gjennomført med kjemisk definerte minimal media, som i hovedsak inneholder dessuten store salter informasjonskilder nitrogen (ammonium salt) og karbon (D-glukose), vitaminer og sporstoffer. Selv om ikke strengt nødvendig i fravær av ytterligere auxotrophies, de resterende aminosyrene (20 -n, hvis n aminosyrer er byttes) legges vanligvis til fremme bakterievekst57. I en innledende vekst fasen før induksjon av protein måluttrykk legges n kanoniske aminosyrer byttes i begrense konsentrasjoner. Mobilnettet vekst fortsetter til de målrettede essensielle aminosyrene er oppbrukt, som eksperimentelt angitt av en stasjonær OD600. Deretter erstattes kultur medium med frisk medium som mangler utarmet aminosyren og inneholder ncAA i rikelig konsentrasjoner. Ribosomal inkorporering av tryptofan analogs som vist i denne protokollen, matet en indol analoge, som intracellulært blir konvertert til den tilsvarende tryptofan deriverte av tryptofan syntase58. Neste er mål protein uttrykk indusert. På dette stadiet er cellene nær slutten av logaritmisk vekst, som en balanse mellom totalt celle nummer og fitness. Som tilstedeværelse og innlemmelse av de kanoniske amino ville føre til vill-type protein produksjon, er det avgjørende for å sikre at den essensielle aminosyren er helt oppbrukt før induksjon. Likeledes er det obligatorisk å undersøke effektiviteten av ncAA inkorporering målet protein, vanligvis ved massespektrometri. Ved betydelig tilstedeværelse av kanoniske aminosyren dyrking betingelser må justeres, f.eksved å endre konsentrasjonen av den viktige amino acid(s) for første vekstfase eller varigheten av sistnevnte. Ved lav aaRS aktivitet mot ncAA, kan overuttrykte av endogene enzym eller co uttrykk for en annen aaRS, som er mer aktiv mot ncAA, være gjennomført59.
Den kanoniske aminosyren Trp er utstyrt med tre bemerkelsesverdige egenskaper: (i) sin naturlige overflod i proteiner er lav; (ii) Biofysiske og kjemiske egenskaper er unike (f.eks., er det vanligvis dominerende opprinnelsen til den iboende fluorescensen proteiner og peptider), og (iii) det bidrar til en rekke biokjemiske interaksjoner og funksjoner inkludert Π-stabling, H-bånd og kasjon-π interaksjoner. Alle disse funksjonene er radikalt forandret på Trp → 4-amino-Trp substitusjon i GdFP. utover tvil, utformingen av en "gull" klasse av avGFPs er en bemerkelsesverdig eksempel på prosjektering skreddersydde autofluorescent proteiner. Med forskjellige spectral egenskaper stilles FPs mot visse spectral Vinduer via mutagenese og ncAA innlemmelse. Ved GdFP oppnås dette ved en enkel kjemiske utveksling H → NH2 i rammen av indol ringen i ECFP chromophore triad. Figur 5 viser effekten av ncAA innlemmelse i chromophore. Innføring av elektron-å gi gruppen fra 4-amino-indol (intracellulært konvertert til 4-amino-Trp) gjør det mulig for en rekke mesomeric strukturer som kan forklare en stabilisert glade tilstand. Tyske, sin forstørret Stokes Skift og rød-skiftet fluorescens utslipp skyldes disse forskjellige egenskaper av utvidet konjugert. Som rapportert tidligere, forbedret intramolekylære kostnad overføring innen GdFP chromophore er iboende følsom for pH (figur 4B) og ledsages av en større endring i dipol øyeblikk mellom S0 bakken og S1 glade tilstand forhold til ECFP33. Som alternativ elektron-donere grupper, kan tryptofan analogs bærer en indol ring erstattet med hydroxy grupper brukes som rapportert i en komparativ studie med modell protein barstar41.
Absorpsjon og fluorescens spektra av GdFP er utvidet forhold til ECFP og EGFP (Figur 3 c og D). Homogen utvide absorpsjon og fluorescens band er vanligvis forårsaket av vibrasjonsmedisin moduser i chromophore og i tillegg av koblingen av chromophore til ytterligere vibrasjonsmedisin moduser i protein60. Koblingen til lokale protein miljøet støttes av kostnader lokalisert på chromophore. Som den strukturelle inhomogeneity av proteinet fører til lokale varianter av vibronic spekteret, slik kopling mellom de vibronic spektra av chromophore og resten av protein støttes av gratis delocalization og mesomeric stater som indikert i Figur 5. Dette støtter også store Stokes Skift og reduserer nødvendigvis fluorescens quantum avkastningen. I forhold til andre rød-skiftet FPs har GdFP selv forbedret protein stabilitet og en lav tendens til aggregering33,61,62. Det ikke bare varierer i farge fra andre FP varianter, men også har en betydelig økt thermostability og forbedret samarbeidsvillig folding33. Fluorescens intensitet er minst 90% bevart ved oppvarming til 60 ° C, mens ECFP fluorescens er redusert til ca 30%. Proteiner bidra aromatiske aminosyrer ofte til nettverk av samspill siden kjeder, som vanligvis har en stabiliserende effekt på protein's tertiær struktur. avGFP havner slikt side kjede nettverk, som består av chromophore selv, så vel som Phe-165, hans-148, og Tyr-145. Siden disse kjedene er ikke bare ganske stive GdFP struktur33, men viktigst, de danner hydrofobe kontakter med chromophore. Den mest fremtredende roman trekk i GdFP er at aminated chromophore er mer proksimale til Phe-165. Dette samspillet er en funksjon ikke observert i andre kjente avGFPs. Som to rester er 3,2-4.5 Å hverandre, kan amino-aromatiske interaksjoner finnes også. Sammen med amination-indusert resonans stabilisering av chromophore stabilisere disse sannsynligvis hydrofobe nettverket av aminosyrer i en samarbeidende mote. En mer effektiv intramolekylære kostnad overføring kan være støttet av disse interaksjoner i glade delstaten i forhold til bakken staten i chromophore og det minst delvis utgjør den 108 nm Stokes Skift33,62 .
Rasjonell design av fluorophore egenskaper, er en økning i størrelsen på delocalized π-systemet spådd for å resultere i en rød-skiftet eksitasjon bølgelengde. Denne tommelfingerregelen er følges av serien av aminosyrer i posisjon 66 fører til nøytrale chromophores: Phe (λMaks = 355 nm) < hans (λma x= 386 nm) < Tyr (λMaks = 395 nm) < Trp (λMaks = 436 nm)63. I naturen, har denne forlengelsen av den chromophore konjugert π-obligasjoner blitt oppnådd av ulike strategier. For DsRed fra Discosoma striataforlenget med integrasjonen av en ekstra aminosyre, dermed skiftende λmax til 573 nm64. Chromophore av asFP595 (λMaks = 595 nm) fra Anemonia sulcata ble forlenget med en imino gruppe, forstørre sin π-systemet65. Siden chromophore av GdFP og andre avFPs er av samme størrelse, må en annen prinsipp innebærer et utslipp bølgelengde i området utvidet DsRed og asFP595 chromophores. Dyp Stokes skifte av 108 nm tilskrives forskjellige strukturen til GdFP chromophore, som avslører et nytt photophysical prinsipp i utformingen av autofluorescent proteiner. Foreløpige beregninger (som rapportert i 62) har vist at dipol øyeblikk glade tilstand chromophore på GdFP er betydelig større enn i bakken staten, i motsetning til verdier i ECFP. Mens dipol øyeblikk av GdFP øker fra ~ 3 D (Debye) i den S0 tilstanden til ~ 15 D i S1, endringen for ECFP chromophore var heller moderate (fra ~ 4 D ~ 6 D). Dermed er den unike golden fluorescensen av GdFP forårsaket av betydelig intramolekylære kostnad overføring innen chromophore, som øker variasjonen av mulig mesomeric strukturer (se figur 5) som tillater resonans stabilisering. Dette reduserer energinivået som utslipp oppstår. Av dyptgripende endringen i dipol øyeblikket på eksitasjon er intramolekylære kostnad separasjon den viktigste grunnen for endringene i elektrostatisk potensial i chromophore miljøet. Omkringliggende protein matrise, justerer igjen endringer i distribusjonen etter chromophore eksitasjon. Den følgende strukturelle avslapningen senker energi nivået av det glade chromophore, som skifter fluorescens spekteret til røde på grunn av sin gratis overføring karakter. For samme grunn av den store Stokes Skift og forbedret radiationless prosesser, fluorescens quantum avkastningen av GdFP er redusert sammenlignet med ECFP33.
Høy quantum avkastning og liten Stokes endring av ECFP og EGFP vanligvis tilskrevet en rigid protein miljø av chromophore, noe som reduserer grader av frihet og dermed intern konvertering å favorisere strålingspådriv avslapning i glade delstaten 66. derfor molekylær utformingen av mer strengt innebygde chromophores med redusert kopling til gjenværende protein matrix kan fungere som en guide til å produsere lenger rød-skiftet GFP derivater med høy fluorescens quantum avkastning. Derfor for ytterligere tekniske tilnærminger til å produsere rød-skiftet autofluorescent proteiner, er utvidelse av π-elektron system og en rigid chromophore struktur med svak kobling til protein miljøet svært ettertraktet. Slike endringer kan også bli introdusert enten direkte inn i GFP-basert chromophores eller plasseringen av ønsket ncAAs chromophore i nærheten.
Disclosures
Forfatterne erklærer at de har ingen konkurrerende økonomiske interesser.
Acknowledgments
Dette arbeidet ble støttet av tysk Research Foundation (Cluster of Excellence "samlende konsepter i katalyse) T.F. og NB og av Federal Utdannings og vitenskap (BMBF Program"HSP 2020", TU-WIMIplus prosjektet SynTUBio) til F.-js
Materials
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| 4-aminoindole | Sigma-Aldrich | 525022 | |
| acetonitrile | VWR | HiPerSolv CHROMANORM ULTRA for LC-MS, 83642 | LC-MS grade required |
| agar-agar | Carl Roth | 5210 | |
| ammonium molybdate ((NH4)2MoO4) | Sigma-Aldrich | 277908 | |
| ammonium sulfate ((NH4)2SO4) | Sigma-Aldrich | A4418 | |
| ampicillin sodium salt | Carl Roth | K029 | |
| biotin | Sigma-Aldrich | B4501 | |
| bromophenol blue | Sigma-Aldrich | B0126 | |
| calcium chloride (CaCl2) | Sigma-Aldrich | C5670 | |
| colloidal silica | Sigma-Aldrich | Ludox HS-40, 420816 | |
| Coomassie Brillant Blue R 250 | Carl Roth | 3862 | |
| copper sulfate (CuSO4) | Carl Roth | CP86.1 | |
| D-glucose | Carl Roth | 6780 | |
| di-sodium hydrogen phosphate (Na2HPO4) | Carl Roth | X987 | |
| di-potassium hydrogen phosphate (K2HPO4) | Carl Roth | P749.1 | |
| 1,4-dithiothreitol (DTT) | Carl Roth | 6908 | |
| DNase I | Sigma-Aldrich | D5025 | |
| ethanol | Carl Roth | 9065.1 | |
| formic acid | VWR | HiPerSolv CHROMANORM for LC-MS, 84865 | LC-MS grade required |
| glycerol | Carl Roth | 3783 | |
| imidazole | Carl Roth | X998 | |
| indole | Sigma-Aldrich | I3408 | |
| iron(II) chloride (FeCl2) | Sigma-Aldrich | 380024 | |
| isopropanol | Carl Roth | AE73.1 | |
| isopropyl β-D-1-thiogalactopyranoside (IPTG) | Sigma-Aldrich | I6758 | |
| lysozyme | Sigma-Aldrich | L6876 | |
| magnesium chloride (MgCl2) | Carl Roth | KK36.1 | |
| magnesium sulfate (MgSO4) | Carl Roth | 8283.2 | |
| manganese chloride (MnCl2) | Sigma-Aldrich | 63535 | |
| β-mercaptoethanol | Carl Roth | 4227.3 | |
| potassium chloride (KCl) | Carl Roth | 6781.3 | |
| potassium dihydrogen phosphate (KH2PO4) | Sigma-Aldrich | P5655 | |
| RNase A | Carl Roth | 7156 | |
| sodium chloride (NaCl) | Carl Roth | P029 | |
| sodium dihydrogen phosphate (NaH2PO4) | Carl Roth | T879 | |
| sodium dodecyl sulphate (NaC12H25SO4) | Carl Roth | 0183 | |
| thiamine | Sigma-Aldrich | T4625 | |
| Tris(hydroxymethyl)-aminomethane (Tris) | Carl Roth | 5429 | |
| Tris hydrochloride (Tris-HCl) | Sigma-Aldrich | 857645 | |
| tryptone | Carl Roth | 8952 | |
| yeast extract | Carl Roth | 2363 | |
| zinc chloride (ZnCl2) | Sigma-Aldrich | 229997 | |
| Name | Company | Catalog Number | Comments |
| amino acids | |||
| L-alanine | Sigma-Aldrich | A7627 | |
| L-arginine | Sigma-Aldrich | A5006 | |
| L-asparagine | Sigma-Aldrich | A8381 | |
| L-aspartic acid | Sigma-Aldrich | A0884 | |
| L-cysteine | Sigma-Aldrich | C7352 | |
| L-glutamic acid | Sigma-Aldrich | G2128 | |
| L-glutamine | Sigma-Aldrich | G3126 | |
| L-glycine | Sigma-Aldrich | G7126 | |
| L-histidine | Sigma-Aldrich | H8000 | |
| L-isoleucine | Sigma-Aldrich | I2752 | |
| L-leucine | Sigma-Aldrich | L8000 | |
| L-lysine | Sigma-Aldrich | L5501 | |
| L-methionine | Sigma-Aldrich | M9625 | |
| L-proline | Sigma-Aldrich | P0380 | |
| L-phenylalanine | Sigma-Aldrich | P2126 | |
| L-serine | Sigma-Aldrich | S4500 | |
| L-threonine | Sigma-Aldrich | T8625 | |
| L-tryptophan | Sigma-Aldrich | T0254 | |
| L-tyrosine | Sigma-Aldrich | T3754 | |
| L-valine | Sigma-Aldrich | V0500 | |
| Name | Company | Catalog Number | Comments |
| Lab materials | |||
| 0.45 µm syringe filter with PVDF membrane | Carl Roth | CCY1.1 | |
| 1.5 mL microcentrifuge tubes | Eppendorf | 30120086 | |
| conical polystyrene (Falcon) tubes, 50 mL | Fisher Scientific | 14-432-22 | |
| Luer-Lock syringe 5 mL | Carl Roth | EP96.1 | |
| dialysis membrane, Molecular Weight Cut-Off (MWCO) 5,000 | Spectrum Medical Industries | Spectra/Por MWCO 5000 dialysis membrane, 133198 | |
| Immobilized Metal ion Affinity Chromatography (IMAC) column 1 mL, Ni-NTA | Macherey Nagel | Protino series, 745410.5 | |
| petri dishes (polystyrene, sterile) | Carl Roth | TA19 | |
| pQE-80L plasmid vector | Qiagen | no longer available | replaced by N-terminus pQE Vector set Cat No./ID: 32915 |
| protein extraction reagent BugBuster | EMB Millipore | 70921-4 | |
| round-bottom polystyrene tubes, 14 mL | Fisher Scientific | Corning Falcon, 14-959-1B | |
| Trp-auxotrophic E. coli strain | ATCC | ATCC 49980 | Bridges BA et al., Chem Biol Interact., 1972, 5(2):77-84; see main text for alternatives |
| Name | Company | Catalog Number | Comments |
| Mass Spectrometry equipment | |||
| mass spectrometer for LC-ESI-TOF-MS | Agilent | Agilent 6530 Accurate-Mass QTOF | coupled with Infinity LC system |
| mass spectrometry data analysis software | Agilent | MassHunter Qualitative Analysis software v. B.06.00 | |
| High-Performance Liquid Chromatography (HPLC) column for LC-ESI-TOF-MS | Sigma-Aldrich | Supelco Discovery BIO Wide Pore C5 HPLC column, 3 µm particle size, 10 cm x 2.1 mm | |
| HPLC autosampler vials 1.5 mL | Sigma-Aldrich | Supelco 854165 | with conical 0.1 mL glass inserts, screw caps and septa |
| Name | Company | Catalog Number | Comments |
| General equipment | |||
| benchtop centrifuge for 1.5 mL Eppendorf tubes | Eppendorf | 5427 R | |
| cooling centrifuge for 50 mL Falcon tubes | Eppendorf | 5810 R | |
| high pressure microfluidizer for bacterial cell disruption | Microfluidics | LM series with “Z” type chamber | |
| peristaltic pump for LC | GE Healthcare | P-1 | |
| Fast Protein Liquid Chromatography (FPLC) system | GE Healthcare | ÄKTA pure 25 L | |
| orbital shaker for bacterial cultivation | Infors HT | Minitron | |
| UV/Vis spectrophotometer | Biochrom | ULTROSPEC 2100 | |
| ultrasonic homogenizer for bacterial cell disruption | Omnilab | Bandelin SONOPULS HD 3200, 5650182 | with MS72 sonifier tip |
| Name | Company | Catalog Number | Comments |
| Fluorescence spectroscopy equipment | |||
| ps-pulsed laser 470 nm | Picoquant GmbH | PDL-470 | |
| time- and wavelength-correlated single photon counting (TWSPC) acquisition software | Picoquant GmbH | SymPhoTime 64 | |
| time- and wavelength-correlated single photon counting (TWSPC) detector | Picoquant GmbH | PML-16C | 16 spectral channels, to be selected by grating settings |
| single photon counting software | Picoquant GmbH | SPCM 9.75 | |
| global fitting software | Picoquant GmbH | SPC2Glo(R) | |
| fluorescence decay data analysis software | Picoquant GmbH | FluoFit program | |
| data analysis software | OriginLab Inc. | Origin 9.2 | |
| neutral density filter set | Schott | NG1 to NG11 | (400 - 650 nm, transmission 50 %, 20%, 10 %, 5 %) |
| 488 nm long-pass emission filter | AHF Analysentechnik | AHF-488 | |
| quartz cuvette | Thorlabs GmbH | CV10Q1400 | 1 cm pathlength |
References
- Shimomura, O., Johnson, F. H., Saiga, Y. Extraction, Purification and Properties of Aequorin, a Bioluminescent Protein from the Luminous Hydromedusan, Aequorea. J Cell Compar Physl. 59 (3), 223-239 (1962).
- Chalfie, M., Tu, Y., Euskirchen, G., Ward, W. W., Prasher, D. C. Green fluorescent protein as a marker for gene expression. Science. 263 (5148), 802-805 (1994).
- Andresen, M., et al. Structure and mechanism of the reversible photoswitch of a fluorescent protein. P Natl Acad Sci USA. 102 (37), 13070-13074 (2005).
- Andresen, M., et al. Structural basis for reversible photoswitching in Dronpa. P Natl Acad Sci USA. 104 (32), 13005-13009 (2007).
- Brakemann, T., et al. A reversibly photoswitchable GFP-like protein with fluorescence excitation decoupled from switching. Nat Biotechnol. 29 (10), 942-947 (2011).
- Kremers, G. -J., Gilbert, S. G., Cranfill, P. J., Davidson, M. W., Piston, D. W. Fluorescent proteins at a glance. J Cell Sci. 124 (Pt 2), 157-160 (2011).
- Shimomura, O. Structure of the chromophore of aequorea 0. shimomura green fluorescent protein. FEBS Lett. 104 (2), 220-222 (1979).
- Shaner, N. C., Campbell, R. E., Steinbach, P. A., Giepmans, B. N. G., Palmer, A. E., Tsien, R. Y. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Shcherbo, D., et al. Bright far-red fluorescent protein for whole-body imaging. Nat Methods. 4 (9), 741-746 (2007).
- Shcherbakova, D. M., Subach, O. M., Verkhusha, V. V. Red fluorescent proteins: advanced imaging applications and future design. Angew Chem Int Edit. 51 (43), 10724-10738 (2012).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Curr Protein Pept Sc. 9 (4), 338-369 (2008).
- Wang, L., Xie, J., Deniz, A. A., Schultz, P. G. Unnatural amino acid mutagenesis of green fluorescent protein. J Org Chem. 68 (1), 174-176 (2003).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Sharma, N., Furter, R., Kast, P., Tirrell, D. A. Efficient introduction of aryl bromide functionality into proteins in vivo. FEBS Lett. 467 (1), 37-40 (2000).
- Liu, C. C., Schultz, P. G. Adding new chemistries to the genetic code. Annu Rev Biochem. 79, 413-444 (2010).
- Twine, S. M., Murphy, L., Phillips, R. S., Callis, P., Cash, M. T., Szabo, A. G. The Photophysical Properties of 6-Azaindole. J Phys Chem B. 107 (2), 637-645 (2003).
- Lepthien, S., Hoesl, M. G., Merkel, L., Budisa, N. Azatryptophans endow proteins with intrinsic blue fluorescence. P Natl Acad Sci USA. 105 (42), 16095-16100 (2008).
- Budisa, N., et al. Probing the role of tryptophans in Aequorea victoria green fluorescent proteins with an expanded genetic code. Biol Chem. 385 (2), 191-202 (2004).
- Ross, J. B., et al. Spectral enhancement of proteins: biological incorporation and fluorescence characterization of 5-hydroxytryptophan in bacteriophage lambda cI repressor. P Natl Acad Sci USA. 89 (24), 12023-12027 (1992).
- Soumillion, P., Jespers, L., Vervoort, J., Fastrez, J. Biosynthetic incorporation of 7-azatryptophan into the phage lambda lysozyme: Estimation of tryptophan accessibility, effect on enzymatic activity and protein stability. Protein Eng Des Sel. 8 (5), 451-456 (1995).
- Heim, R., Tsien, R. Y. Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Curr Biol. 6 (2), 178-182 (1996).
- Bridges, B. A., Mottershead, R. P., Rothwell, M. A., Green, M. H. L. Repair-deficient bacterial strains suitable for mutagenicity screening: tests with the fungicide captain. Chem Biol Interact. 5 (2), 77-84 (1972).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: The Heat Shock Method. J Vis Exp. , (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Bacterial Transformation: Electroporation. J Vis Exp. , (2017).
- Grigorenko, B. L., Krylov, A. I., Nemukhin, A. V. Molecular modeling clarifies the mechanism of chromophore maturation in the green fluorescent protein. J Am Chem Soc. , (2017).
- JoVE Science Education Database. General Laboratory Techniques. Introduction to the Spectrophotometer. J Vis Exp. , (2017).
- Goedhart, J., et al. Structure-guided evolution of cyan fluorescent proteins towards a quantum yield of 93%. Nat Commun. 3, 751 (2012).
- Neidhardt, F. C., Bloch, P. L., Smith, D. F. Culture medium for enterobacteria. J Bacteriol. 119 (3), 736-747 (1974).
- Hörnsten, E. G. On culturing Escherichia coli on a mineral salts medium during anaerobic conditions. Bioprocess Eng. 12 (3), 157-162 (1995).
- Davis, B. D. The Isolation of Biochemically Deficient Mutants of Bacteria by Means of Penicillin. P Natl Acad Sci USA. 35 (1), 1-10 (1949).
- Sambrook, J., Russell, D. W. Molecular Cloning: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. Cold Spring Harbor, NY, USA. (2001).
- Wang, Y. -S., et al. The de novo engineering of pyrrolysyl-tRNA synthetase for genetic incorporation of L-phenylalanine and its derivatives. Mol Biosyst. 7 (3), 714-717 (2011).
- Bae, J. H., et al. Expansion of the genetic code enables design of a novel "gold" class of green fluorescent proteins. J Mol Biol. 328 (5), 1071-1081 (2003).
- JoVE Science Education Database. Dialysis: Diffusion Based Separation. J Vis Exp. , Cambridge, MA. (2017).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Separating Protein with SDS-PAGE. J Vis Exp. , (2017).
- Petrásek, Z., et al. Excitation energy transfer from phycobiliprotein to chlorophyll d in intact cells of Acaryochloris marina studied by time- and wavelength-resolved fluorescence spectroscopy. Photoch Photobio Sci. 4 (12), 1016-1022 (2005).
- Kolber, Z. S., Barkley, M. D. Comparison of approaches to the instrumental response function in fluorescence decay measurements. Anal Biochem. 152 (1), 6-21 (1986).
- Pelet, S., Previte, M. J. R., Laiho, L. H., So, P. T. C. A fast global fitting algorithm for fluorescence lifetime imaging microscopy based on image segmentation. Biophys J. 87 (4), 2807-2817 (2004).
- Loefroth, J. E. Time-resolved emission spectra, decay-associated spectra, and species-associated spectra. J Phys Chem. 90 (6), 1160-1168 (1986).
- Hartman, M. C. T., Josephson, K., Lin, C. -W., Szostak, J. W. An expanded set of amino acid analogs for the ribosomal translation of unnatural peptides. PLoS One. 2 (10), e972 (2007).
- Budisa, N., et al. Global replacement of tryptophan with aminotryptophans generates non-invasive protein-based optical pH sensors. Angew Chem Int Edit. 41 (21), 4066-4069 (2002).
- Ma, Y., Biava, H., Contestabile, R., Budisa, N., di Salvo, M. L. Coupling bioorthogonal chemistries with artificial metabolism: intracellular biosynthesis of azidohomoalanine and its incorporation into recombinant proteins. Molecules. 19 (1), 1004-1022 (2014).
- Teramoto, H., Kojima, K. Incorporation of Methionine Analogues Into Bombyx mori Silk Fibroin for Click Modifications. Macromol Biosci. 15 (5), 719-727 (2015).
- Deal, R. B., Henikoff, J. G., Henikoff, S. Genome-wide kinetics of nucleosome turnover determined by metabolic labeling of histones. Science. 328 (5982), 1161-1164 (2010).
- Hinz, F. I., Dieterich, D. C., Tirrell, D. A., Schuman, E. M. Non-canonical amino acid labeling in vivo to visualize and affinity purify newly synthesized proteins in larval zebrafish. ACS Chem Neurosci. 3 (1), 40-49 (2012).
- Dieterich, D. C., et al. In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat Neurosci. 13 (7), 897-905 (2010).
- Dieterich, D. C., Link, A. J., Graumann, J., Tirrell, D. A., Schuman, E. M. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). P Natl Acad Sci USA. 103 (25), 9482-9487 (2006).
- Glenn, W. S., et al. Bioorthogonal Noncanonical Amino Acid Tagging (BONCAT) Enables Time-Resolved Analysis of Protein Synthesis in Native Plant Tissue. Plant Physiol. 173 (3), 1543-1553 (2017).
- Zhou, L., et al. Incorporation of tryptophan analogues into the lantibiotic nisin. Amino Acids. 48 (5), 1309-1318 (2016).
- Acevedo-Rocha, C. G., Budisa, N. Xenomicrobiology: a roadmap for genetic code engineering. Microb Biotechnol. 9 (5), 666-676 (2016).
- Agostini, F., Völler, J. -S., Koksch, B., Acevedo-Rocha, C. G., Kubyshkin, V., Budisa, N. Biocatalysis with Unnatural Amino Acids: Enzymology Meets Xenobiology. Angew Chem Int Edit. 56 (33), 9680-9703 (2017).
- Bacher, J. M., Ellington, A. D. Selection and characterization of Escherichia coli variants capable of growth on an otherwise toxic tryptophan analogue. J Bacteriol. 183 (18), 5414-5425 (2001).
- Wong, J. T. Membership mutation of the genetic code: loss of fitness by tryptophan. Pc Natl Acad Sci USA. 80 (20), 6303-6306 (1983).
- Hoesl, M. G., et al. Chemical Evolution of a Bacterial Proteome. Angew Chem Int Edit. 54 (34), 10030-10034 (2015).
- Italia, J. S., et al. An orthogonalized platform for genetic code expansion in both bacteria and eukaryotes. Nat Chem Biol. 13 (4), 446-450 (2017).
- Völler, J. -S., Thi To, T. M., Biava, H., Koksch, B., Budisa, N. Global substitution of hemeproteins with noncanonical amino acids in Escherichia coli with intact cofactor maturation machinery. Enzyme Microb Tech. 106, 55-59 (2017).
- Budisa, N., Steipe, B., Demange, P., Eckerskorn, C., Kellermann, J., Huber, R. High-level biosynthetic substitution of methionine in proteins by its analogs 2-aminohexanoic acid, selenomethionine, telluromethionine and ethionine in Escherichia coli. Eur J Biochem. 230 (2), 788-796 (1995).
- Völler, J. -S., Budisa, N. Coupling genetic code expansion and metabolic engineering for synthetic cells. Curr Opin Biotech. 48, 1-7 (2017).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr Opin Chem Biol. 14 (6), 774-780 (2010).
- Somsen, O. J., van Grondelle, R., van Amerongen, H. Spectral broadening of interacting pigments: polarized absorption by photosynthetic proteins. Biophys J. 71 (4), 1934-1951 (1996).
- Kurschus, F. C., Pal, P. P., Bäumler, P., Jenne, D. E., Wiltschi, B., Budisa, N. Gold fluorescent annexin A5 as a novel apoptosis detection tool. Cytom Part A. 75 (7), 626-633 (2009).
- Lepthien, S., Wiltschi, B., Bolic, B., Budisa, N. In vivo engineering of proteins with nitrogen-containing tryptophan analogs. Appl Microbiol Biot. 73 (4), 740-754 (2006).
- Wachter, R. M., Elsliger, M. -A., Kallio, K., Hanson, G. T., Remington, S. J. Structural basis of spectral shifts in the yellow-emission variants of green fluorescent protein. Structure. 6 (10), 1267-1277 (1998).
- Verkhusha, V. V., Lukyanov, K. A. The molecular properties and applications of Anthozoa fluorescent proteins and chromoproteins. Nat Biotechnol. 22 (3), 289-296 (2004).
- Martynov, V. I., Savitsky, A. P., Martynova, N. Y., Savitsky, P. A., Lukyanov, K. A., Lukyanov, S. A. Alternative cyclization in GFP-like proteins family. The formation and structure of the chromophore of a purple chromoprotein from Anemonia sulcata. J Biol Chem. 276 (24), 21012-21016 (2001).
- Piatkevich, K. D., Malashkevich, V. N., Morozova, K. S., Nemkovich, N. A., Almo, S. C., Verkhusha, V. V. Extended Stokes shift in fluorescent proteins: chromophore-protein interactions in a near-infrared TagRFP675 variant. Sci Rep. 3 (1), 1847 (2013).



