Summary
Protein sammenlægning fremkalder cellulære oxidativt stress. Denne protokol beskriver en metode til overvågning af de intracellulære stater af amyloidogenic proteiner og den oxidative stress i forbindelse med dem, ved hjælp af flowcytometri. Metoden bruges til at studere funktionen af opløselige og sammenlægning-tilbøjelige varianter af amyloid-β-peptid.
Abstract
Protein misfoldning og sammenlægning i amyloid konformationer har været relateret til debut og progression af flere neurodegenerative sygdomme. Der er dog stadig lidt information om hvordan uopløselige protein aggregater udøve deres toksiske virkninger in vivo. Simpel prokaryote og eukaryote modelorganismer, såsom bakterier og gær, har bidraget væsentligt til vores nuværende forståelse af mekanismerne bag intracellulære amyloid dannelse, aggregater formering og toksicitet. I denne protokol, er brugen af gær beskrevet som en model til at dissekere forholdet mellem dannelsen af protein aggregater og deres indvirkning på cellulære oxidativt stress. Metoden kombinerer påvisning af intracellulære opløselige/aggregerede status for en amyloidogenic protein med kvantificeringen af cellulære oxidative skader som følge af dens udtryk ved hjælp af flowcytometri (FC). Denne fremgangsmåde er enkel, hurtig og kvantitative. Undersøgelsen viser teknikken ved at sammenholde de cellulære oxidativt stress forårsaget af et stort sæt af amyloid-β peptid varianter med deres respektive iboende sammenlægning tilbøjeligheder.
Introduction
Proteostasis er en grundlæggende faktor for celle fitness og aldersfordelte processer. I celler vedligeholdes protein homøostase af sofistikerede protein kvalitetskontrol netværk har til formål at sikre den korrekte hvorved man genfolder af fejlfoldede protein konformere af chaperoneproteiner og/eller deres målrettede proteolyse med flere godt bevarede mekanismer1 ,2,3,4,5. Et stort antal undersøgelser understøtte forbindelsen mellem symptomdebut og progression af en bred vifte af sygdomme hos mennesker og manglende proteostasis, fører til protein misfoldning og sammenlægning. For eksempel, betragtes tilstedeværelsen af protein indskud som en patologisk kendetegnende for mange neurodegenerative sygdomme som Alzheimers, Parkinsons, og Huntingtons sygdomme6,7,8, prionogenic sygdomme, og ikke-degenerative amyloidoses9. Det er blevet foreslået, at tidlig oligomere og protofibrillar forsamlinger i sammenlægning reaktion er de vigtigste elicitors af cytotoksicitet, etablere afvigende interaktioner med andre proteiner i overfyldte cellulære miljø10. Desuden kan protein indeslutninger (PI) overføres mellem celler, formerings deres giftvirkning11,12. Det kunne derfor være, at dannelsen af PI faktisk kan udgøre en afgiftende mekanisme, der begrænser forekomsten af farlige aggregerede arter til bestemte steder i cellen, hvor de kan behandles eller akkumuleret uden større bivirkninger 13 , 14.
Standard in vitro- biokemiske metoder har givet vigtige indsigter i de forskellige fiskearter, der befolker sammenlægning reaktioner og deres egenskaber15,16. Men de betingelser, der anvendes i disse assays er klart forskellige fra dem, der forekommer inden for cellen, og derfor sætte spørgsmålstegn ved deres fysiologiske betydning. På grund af den bemærkelsesværdige bevarelse af cellulære veje som protein kvalitetskontrol, autophagy eller regulering af cellulære redox stat17,18 blandt eukaryoter19,20,21 ,22,23, spirende gær Saccharomyces cerevisiae (S. cerevisiae) fremstod som en privilegeret simpel cellulære model til at studere de molekylære determinanter for protein sammenlægning og dens tilknyttede cytotoksiske effekt i biologisk relevante miljøer24,25,26.
Protein sammenlægning tilbøjelighed er en funktion i sagens natur kodet i den primære sekvens. Dannelsen af amyloid-lignende strukturer kan således forudsiges baseret på identificering og evaluering af styrken af fremme af sammenlægning regioner i polypeptider27. Men trods succesen med bioinformatic algoritmer til at forudsige in vitro- sammenlægning egenskaberne af protein sekvenser, de er stadig langt fra forecasting, hvordan disse tilbøjeligheder omsættes i vivo cytotoksiske effekt. Undersøgelser, der vedrører forbindelsen mellem den aggregerede stat af et bestemt protein og dens tilknyttede cellulære skader på en systematisk måde kan bidrage til at omgå denne beregningsmæssige begrænsning. Denne forbindelse behandles i den foreliggende undersøgelse, at drage fordel af en lang række varianter af amyloid-β peptid Aβ42 adskiller sig kun i en enkelt rester, men viser en kontinuerlig række sammenlægning tilbøjeligheder i vivo28. Især er en FC-baseret tilgang til at identificere de konformationelle arter tegner sig for den oxidative skader fremkaldes ved sammenlægning-tilbøjelige proteiner i gærceller beskrevet. Metoden, der giver mange fordele såsom enkelhed, høj overførselshastighed kapacitet og præcis kvantitativ måling. Denne fremgangsmåde gjorde det muligt at bekræfte, at PI spiller en beskyttende rolle mod oxidativt stress.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. S. cerevisiae kulturer og Protein udtryk
Bemærk: Aβ varianter udviser forskellige relative sammenlægning tilbøjeligheder på grund af en mutation i et enkelt rester på holdning 19 (Phe19) af Aβ42 peptid (figur 1A). Disse peptid varianter er markeret med grøn fluorescerende proteiner (NGL), der fungerer som en sammenlægning reporter (figur 1)29.
- Omdanne plasmider kodning for 20 Aβ42-NGL varianter i gærceller med en BY4741 forældrenes baggrund (MATa hans3Δ1 leu2Δ0 mødte15Δ0 ura3Δ0)30. Hver plasmid koder for en Aβ42 mutant smeltet til normal god landbrugspraksis af en GSSGSSG linker. Plasmider er pESC(-URA) og omfatter de valgbar markør URA329.
- Forberede den syntetiske komplet medium uden uracil (SC-URA) med følgende komponenter: 1.9 g/L gær nitrogen base uden uracil, 5 g/L ammoniumsulfat og 900 mL af Hedeselskabet2O kombineret med 100 mL 20% (w/v) glukose.
- Sætte en koloni af de transformerede gærceller til 20 mL SC-URA medium indeholdende 2% glukose og vokse kulturer natten over ved 30 ° C under agitation på 210 rpm.
- Den næste dag, vokse nye gær celler kulturer ved at vaccinere 100 µL af overnight kultur til 5 mL frisk SC-URA medium.
- På en OD590 af 0,5, centrifugeres kulturer på 3.000 x g i 4 min., supernatanten fjernes, og resuspend celler i den samme mængde frisk SC-URA medium indeholdende 2% raffinose.
- Der inkuberes ved 30 ° C under agitation på 210 rpm i 30 min. Derefter centrifugeres celler ved 3.000 x g i 4 min og supernatanten. Resuspend celler i frisk SC-URA medium indeholdende 2% galactose for at fremkalde det rekombinante protein udtryk.
- Efter 16 h af inducerende protein udtryk i SC-URA medium indeholdende 2% galactose, høste 1 mL af de inducerede celler i sterile microcentrifuge rør ved centrifugering ved 3.000 x g i 4 min.
Bemærk: De mest relevante forskelle opstår efter en induktion periode af 16 h. Induced gærceller udtrykker Aβ42-NGL varianter kan visualiseres ved Fluorescens mikroskopi til at bestemme den rekombinante protein fordeling inde i cellerne (figur 2B).
2. celle farvning
Bemærk: Frisk ikke-induceret celler er påkrævet som en negativ kontrol at fastlægge en fluorescerende tærskel under en FC analyse.
- Tage de 16 h-induceret gærceller og bestemme deres optiske densitet på 590 nm (OD590). Fortynde dem i en steril fosfatbufferet saltopløsning (PBS) til en OD590 på 0,1.
- Forberede 1 x fosfatbufferet saltopløsning som følger: tilføje 8 g NaCl, 0,2 g af KCl, 1.44 g Na2HPO4, og 0,24 g KH2PO4 til 800 mL af Hedeselskabet2O. fylde løsning op til 1 L af Hedeselskabet2O efter justering til pH 7,4 med HCl. Filter buffer gennem en 0,2 µm filter.
- Overføre de udtrykker celle suspensioner behørigt mærket rør (12 × 75 mm runde-bunden polystyren) og beskytte dem mod lys. Kontroller, at de er klar til at blive lastet på flow forskellige for analyse, sammen med ikke-induceret og ikke-farvede celler.
- Tilføje oxidativt stress sonden (f.eks. CellROX dyb rød) til hver prøve i en endelig koncentration på 5 µM og inkuberes celler ved 30 ° C i 30 min. i mørke.
- Efter inkubationen cellerne vaskes 3 gange med 1 x PBS og resuspend dem i den samme mængde af buffer inden deres analyse af FC.
3. flow flowcytometri analyse
Bemærk: Brug en FC setup med passende lasere og filtre til at registrere normal god landbrugspraksis og oxidativ stress sonde fluorescens signal. En flow Flowcytometret udstyret med en 488 nm blå laser til påvisning af normal god landbrugspraksis og en 635 nm rød laser til påvisning af oxidativt stress sonde fluorescens kan bruges. Erhvervelse af emission fluorescens af normal god landbrugspraksis og oxidativ stress sonden er udført med et 530/30 nm BP filter og en 660/20 BP filteret, henholdsvis (tabel 1).
- Klik på Åbn nye regneark panelet og oprette følgende erhvervelse dot parceller.
- Vælg Scatter Plot værktøj fra værktøjslinjen og oprette et plot med variabler Side Scatter-området (SSC-A) på y-aksen vs frem Scatter-området (FSC-A) i en lineær skala på x-aksen.
- Klik på Scatter Plot værktøj fra værktøjslinjen og Opret et plot med variabler FL1-området (FITC) på x-aksen vs. FL3-området (APC) i en logaritmisk skala på y-aksen.
- Klik på ikonet Indstillinger for instrumenter . Klik på fanen udligning og Angiv alle kompensationsniveauer. Klik på fanen erhvervelse og vælg et samlet antal på 20.000 begivenheder skal registreres.
- Klik på ikonet Strømningshastighed for at skifte flowet til lav.
- Klik på fanen erhverve at begynde at køre ikke-induceret, unstained celler og justere spændingen i Apparatindstillingen for fremad og side scatter indtil befolkningen er distribueret i midten-venstre kvadrant.
- Klik på ikonet Polygon at indstille en region R1 omkring cellen befolkningen undtagen celle debris, og bruge denne låge befolkning P1 = R1 for alle fluorescerende dot plots og histogram repræsentationer (figur 3).
- For at justere den ydelse spænding af fluorescens signal, løbe de unstained celler i FL1-FL3 dot plot, tuning tilvækst i fanen Instrument indstilling indtil cellerne er fordelt i den nederste venstre kvadrant.
- Ændre prøven til induceret cellerne til at måle normal god landbrugspraksis fluorescens. I fanen Instrument indstilling angive gevinst i den FSC-A vs FL1 (FITC) når befolkningen er fordelt i den nederste højre kvadrant. Definere befolkningens positive celle med en gate (Gate P2).
- Ændring af prøven til den ikke-induceret farvede celler til at vise oxidativt stress af fluorescens på en FSC-A vs. FL3 (APC) dot plot. Tune gevinsten, indtil cellen befolkningen er fordelt i venstre øverste kvadrant. Gate befolkningens positive celle i P3.
- Gøre to histogram grunde med ikonet histogrammet repræsenterer celle fluorescens. For at repræsentere fluorescens-intensiteten, Sørg for FL1 (FITC) repræsenterer normal god landbrugspraksis Fluorescens er i x-aksen, og FL3 (APC) repræsenterer Fluorescens er i y-aksen, anvende P2 og P3 befolkninger på en logaritmisk skala, henholdsvis.
Bemærk: Histogram overlejringer er en god måde at illustrere forskelle mellem fluorescens profiler af celler, der udtrykker forskellige Aβ varianter.
4. rekombinante Protein Immunodetection
- Bestem Proteinniveau udtryk ved høst gær kulturer induceret for 16 h ved centrifugering på 4.000 x g for 6 min og gemme celle pellets ved-80 ° C.
- Resuspend de indsamlede celler, der udtrykker rekombinante proteiner til 16 h i PBS. Forberede 200 µL celle suspensioner af hver Aβ42-NGL mutant til en OD590 af 20.
- For total protein fraktion analyse, høste 100 µL af hver mutant ved centrifugering ved 14.000 x g i 20 min. og resuspend celle pellets i den samme mængde gær lysisbuffer Y-PER (50 mM Tris-HCl pH 8.0, 1% DMSO, 200 mM NaCl, 1 mM EDTA der indeholder 10 mg/mL SB3-14 (myr istyl sulfobetaine) suppleret med 1 mM phenylmethylsulfonyl fluorid (PMSF)).
- Inkuber prøver i 20 min. ved stuetemperatur under milde agitation.
- Fastslå ekstrakt proteinkoncentration ved hjælp af Bradford assay. Ilæg op til 5 µg protein ekstrakt af hver prøve på en 15% acrylamid SDS-PAGE elektroforese gel og skamplet på en polyvinylidene xenondifluorid (PVDF) membran på 100 V til 60 min.
- Forberede en Bradford reagens med 100 mg af Coomassie strålende blå G-250 opløses i 50 mL 95% ethanol og tilsættes 100 mL af 85% (w/v) fosforsyre. Derefter, når farven er helt opløst, fortyndes blandingen med ddH2O til 1 L og filtrere det gennem cellulose filtrerpapir lige før brug.
Bemærk: Bradford reagens skal være lys brun. - Forberede en standard af bovint serumalbumin (BSA) spænder fra 5-100 µg af protein i 1.000 µL af Bradford reagens.
- Tilføje 1-10 µL af protein ekstrakt til 1 mL af Bradford reagens og inkuberes blanding ved stuetemperatur i 5 min.
- Absorbansen af standarderne og protein ekstrakt prøver på OD595.
- Til analysen, skal du gøre et plot med absorbans værdier opnået for standarder vs. µg af protein. Baseret på standardkurven, bestemme koncentrationerne af de oprindelige prøver fra mængden af protein, overvejer mængden og fortynding, hvis nogen.
- Forberede en Bradford reagens med 100 mg af Coomassie strålende blå G-250 opløses i 50 mL 95% ethanol og tilsættes 100 mL af 85% (w/v) fosforsyre. Derefter, når farven er helt opløst, fortyndes blandingen med ddH2O til 1 L og filtrere det gennem cellulose filtrerpapir lige før brug.
- Skyl membran med 1 x Tris-bufferet saltvand (TBS), 0,1% Tween 20 (TTBS), og blokere det med 5% (w/v) nonfat tørt mælk i 1 x TTBS.
- For at forberede 1 L af 10 x TBS, opløse 24 g af Tris base og 88 g NaCl i 900 mL H2Odd og pH-værdien til 7,6. Tilføje ddH2O til en endelige mængden af 1 L. En 1 x løsning, Bland 1 del af 10 x lager med 9 dele af Hedeselskabet2O.
- Inkuber membran for 1 h med en primær β-amyloid antistof 6E10 fortyndes 1:1,000 og i 1 time ved stuetemperatur med en sekundær antistof ged anti-mus IgG-HRP konjugat fortyndet 1:10, 000.
- Udvikle membraner med 1 mL Luminata fluorescerende reagens og kvantificere bands af densitometric analyse som beskrevet i trin 5.2.
5. dataanalyse
- For at analysere resultaterne af FC, oprette et bord, der viser den gennemsnitlige fluorescerende intensitet (MFI) og median fluorescens med dens tilsvarende standard fejl og/eller koefficient af varians (CV) for normal god landbrugspraksis fluorescens og oxidativ stress niveauer.
- Klik på fanen inspektør og ikonet statistik til at tilpasse de statistiske data, der er nødvendige for hver kanal.
Bemærk: Når man sammenligner protein varianter, konvertere fluorescens værdier data til en fold-over baggrunden eller opløsning metriske (RD):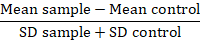
I denne parameter, betyder prøven er den gennemsnitlige fluorescens problem prøver, den gennemsnitlige kontrol er den gennemsnitlige fluorescens af celler, der anvendes som en kontrol og SD er standardafvigelsen for den gennemsnitlige fluorescens. RD faktor er en mere nøjagtig indeks end MFI værdi, da det ikke kun beregner forskellen mellem prøver men også tegner sig for spredning af data. Denne beregning er specielt relevant, når man sammenligner data fra forskellige eksperimenter udført over tid. Når RDs er blevet beregnet, er brug af en statistisk test tilrådeligt at bekræfte betydningen af de konstaterede forskelle mellem protein varianter.
- Klik på fanen inspektør og ikonet statistik til at tilpasse de statistiske data, der er nødvendige for hver kanal.
- Protein immunodetection, kvantificere rekombinante protein niveauer af vestlige skamplet bruger ImageJ software.
- Før densitometri analyse, skal du konvertere de oprindelige raw-billeder af den udviklede membran til JPEG-fil-format og gråtoner tilstand.
- Juster afsnittet Sæt målinger i menuen analyser at Betyde grå værdi.
- Klik på værktøjet rektangel fra ImageJ og definere en region af interesse (ROI) med en ensartet størrelse for bandet densitometri analyse. Centrere hver bandet på membran billedet inde i den oprettede rektangulær ramme og optage måling én efter én ved hjælp af Ctrl + M (kommandoen måling).
- Måle den lokale baggrund støder op til hver enkelt band, anvender den samme ROI område. Efter måling, trækkes den tilsvarende lokale baggrund fra hver enkelt band.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Denne protokol beskriver hvordan til at ansætte en samling af 20 varianter af den Aβ42 peptid hvor Phe19 har været muteret til alle naturlige proteinogeniske aminosyrer28. De teoretiske sammenlægning tilbøjeligheder af disse proteiner kan analyseres ved hjælp af to forskellige bioinformatic algoritmer (AGGRESCAN og TANGO31,32). I begge tilfælde, gør denne analyse en progressiv graduering af sammenlægning tendenser, tilskrive, som hovedregel, de højeste værdier for hydrofobe rester og den laveste opladet og polar dem (figur 1B, 1 C).
For at spore eksperimentelt tilstanden intracellulære sammenlægning af forskellige Aβ42 varianter, kræver den beskrevne eksperiment en transformation af et plasmid, kodning for Aβ42 og smeltet til normal god landbrugspraksis (figur 1A) under kontrol af galaktose-inducerbar GAL1 i S. cerevisiae. Gærceller giver udtryk for Aβ42 varianter efter en induktion periode på 16 h kan visualiseres i et fluorescens mikroskop. Denne visualisering gør det muligt at bekræfte dannelsen af PI i 10 af de 20 varianter fra samlingen analyseret. Antallet og størrelsen af de fluorescerende aggregater afviger fra variant til variant (figur 2). Figur 2A viser PI kvantificering fra i alt 500 celler i hver af de to uafhængige kulturer. Her, vi kan observere en fremragende aftale mellem forudsagt og i vivo sammenlægning egenskaber.
For at tage fat om dannelsen af protein aggregater kan udløse oxidativt stress i denne model cell system, bruger denne protokol FC til at overvåge den cellulære normal god landbrugspraksis fluorescens sammen med fluorescens af fluorogenic sonde som en indikator for reaktive ilt arter (ROS) produktion. Figur 3 illustrerer proceduren og opnået resultater for Localisation og Ile mutanter, der svarer til de administrationsprocedurerne distribuerede og PI-dannende Aβ42 varianter, henholdsvis.
Først, den analyserede celle befolkning er gated (P1) i FSC-A vs. SSC-A dot plot om at fjerne celle efterladenskaber fra analyse (figur 3A). Fluorescens signal af normal god landbrugspraksis vs oxidativt stress sonde med befolkningens P1 er repræsenteret i dot plot grafer hvor grøn fluorescerende celler er gated i P3 (figur 3B). Figur 3 c og 3D vise histogrammer skabt for at få statistiske data for hver udtrykt variant, herunder CV (tabel 2), for at kvantificere gennemsnitlige og median fluorescens af hver fluorescerende markør.
Alle mutanter forventes at være udtrykt på samme niveau, da de adskiller sig i en enkelt aminosyre (0,02% af Aβ42-NGL sekvensen). Dog kan proteostasis maskiner reagere på en differentieret måde på deres særlige sammenlægning tilbøjeligheder. Derfor er udtrykket Proteinniveau i cellulære ekstrakter kvantificeret ved hjælp af en Aβ-specifikke antistof (figur 4). Vi ser, at som en generel tendens, PI-dannende Aβ42 varianter er til stede på et lavere niveau end de resterende administrationsprocedurerne distribuerede i cytosol.
Vigtige forskelle blandt Aβ42 varianter kan observeres, når deres oxidativt stress sonde fluorescens, protein niveauer og normal god landbrugspraksis fluorescens egenskaber er repræsenteret i forhold til deres evne til at danne PI og deres iboende sammenlægning tilbøjeligheder ( Figur 5). Den mere sammenlægning-tilbøjelige PI-dannende varianter fremkalde meget lavere oxidativ stress end deres mere opløselige modparter (figur 5A, 5B). Med disse resultater er der ingen indlysende sammenhæng mellem størrelsen og antallet af aggregater pr. individuel celle (figur 2) og oxidativ stress niveauer. PI-dannende varianter, især mutanter forsynet med Trp og Phe på position 19, er til stede i cellen på et lavere niveau end de homogen måde distribueres i cytosol (figur 5 c, 5 D). En undtagelse til dette er Thr mutant, som udgør mindre end 10% af cellerne PI (figur 1B, 1 C). Dette stemmer overens med det faktum, at de mest sammenlægning-liggende varianter selektivt ryddet af gær kvalitetskontrol nedbrydning maskiner28. Normal god landbrugspraksis fluorescens og protein niveauer korrelerer godt for de mere opløselige Aβ42 former, men ikke for dem, der danner PI (figur 5E, 5F). Dette skyldes sandsynligvis, at i disse cellulære populationer, fluorescens kommer fra to forskellige rum, cytosol og optagelser, og deres relative bidrag til den samlede fluorescens varierer mellem mutanter34.

Figur 1 . Sammenlægning tilbøjelighed analyse for indsamling af Aβ42-NGL fusion konstruktioner fremstillet af mutation af rester på holdning 19 i Aβ42 peptid ved alle naturlige aminosyrer. A. dette billede viser en 3D model af wt Aβ42 sammenvoksede til normal god landbrugspraksis ved en linker (Aβ42, grå; Normal god landbrugspraksis, grønne), baseret på foreløbige 1EMA for grøn fluorescerende proteiner fra Aequorea victoria og 2OTK for Alzheimers Aβ peptid som Phe19 sidekæde af wt Aβ42 er vist med rødt. Modellen er lavet ved hjælp af Pymol software. Rest 19 i Aβ42 rækkefølge indtager en central placering i den centrale hydrofobe klynge. I søjlediagrammer er lavet med to forskellige bioinformatic prædiktorer: B. AGGRESCAN, C. TANGO. Venligst klik her for at se en større version af dette tal.

Figur 2 . Aβ42-normal god landbrugspraksis PI-dannende varianter i S. cerevisiae kulturer induceret for 16 h af udtryk. A. søjlegrafen Angiver procentdelen af celler, der indeholder et andet antal PI beregnes ud fra en i alt 500 fluorescerende celler for hver variant i to biologiske replikater. B. disse billeder er repræsentative fluorescerende mikroskopi billeder af udvalgte Aβ42-NGL varianter (Phe, Ile, Thr, Trp). De blev erhvervet i UV-lys ved hjælp af en excitation filter for normal god landbrugspraksis (450-500 nm) og en emission vifte (515-560 nm). Skalalinjen repræsenterer 10 µm. venligst klik her for at se en større version af dette tal.

Figur 3 . Ordningen for flow flowcytometri (FC) analyse of S. cerevisiae celler, der udtrykker valgte Aβ42-NGL varianter. A. diagram viser gated gærceller (P1) i dot parceller (FSC-A vs. SSC-A) hvor celle efterladenskaber fjernes fra cellen befolkningen. De mikroskopiske billeder af Localisation (ensartet fordelt) og Ile (PI-dannende) varianter er vist ud for FC dot parceller. Skalalinjen repræsenterer 10 µm. B. Disse scatter dot plot billeder repræsenterer normal god landbrugspraksis-A vs. oxidativt stress sonde hvor gated befolkningen (P3) omfatter kun fluorescerende celler, undtagen baggrund signal. Celle frekvens histogrammer er c. normal god landbrugspraksis signal (FITC amplitude) gated fra P1 og D. CellROX (APC amplitude) gated fra P3. Celle erhvervelse blev udført med en flow Flowcytometret. Hver plot repræsenterer 20.000 begivenheder. Q og jeg svarer til Localisation og Ile Aβ42 mutanter, henholdsvis. Venligst klik her for at se en større version af dette tal.

Figur 4 . Kvantificering af cellulære protein niveauer. Denne figur viser Western blotting af total protein fraktioner af Aβ42-NGL mutanter efter 16 h af udtryk i S. cerevisiae. Varianter af PI-dannende er farvet i grønt og dem diffust fordelt på cytosol er farvet i lyse rødt. Venligst klik her for at se en større version af dette tal.

Figur 5 . Cellulære oxidativt stress, intracellulære normal god landbrugspraksis fluorescens og cellulære protein niveauer for Aβ42-NGL mutanter bestemmes efter 16 h af udtryk i S. cerevisiae. Varianter repræsenteret på x-aksen har været bestilt efter deres forudsagte sammenlægning tilbøjeligheder af enten AGGRESCAN (venstre panel) eller TANGO (højre panel). Varianter af PI-dannende er farvet i grønt og de ikke-PI-dannende varianter er farvet i lyse rødt. A. B. disse søjlediagrammer viser de oxidative stress sonde fluorescens værdier opnået ved en FC analyse af Aβ42-NGL mutanter. C. D. disse søjlediagrammer repræsenterer protein niveauer som kvantificeres ved en vestlige skamplet densitometri analyse ved hjælp af ImageJ software. E. F. disse søjlediagrammer viser normal god landbrugspraksis fluorescens værdier efter en FC analyse. Fejllinjer for den oxidative stress sonde og normal god landbrugspraksis fluorescens værdier repræsenterer koefficienten for varians (CV) af FC-gated celler. Fejllinjer for udtryk Proteinniveau repræsenterer ± SE (n = 3). Venligst klik her for at se en større version af dette tal.
| Kanal | Fluorophore | Excitation bølgelængde (nm) | Band Pass Filter |
| FITC | NORMAL GOD LANDBRUGSPRAKSIS | 488 | 530/30 |
| APC | CellRox | 635 | 660/20 |
| PerCP | IP | 488 | 585/42 |
Tabel 1. Laserkilde, band-pass-filtre og fluorophores anvendes i flow Flowcytometret.
| Normal god landbrugspraksis fluorescens | CV | CellROX fluorescens | CV | |
| A | 9316 | 96.4 | 1964 | 127,5 |
| C | 9709 | 91,9 | 1275 | 172.2 |
| D | 11213 | 101.7 | 3443 | 155.8 |
| E | 12256 | 101,1 | 3220 | 152.2 |
| F | 3010 | 96.1 | 1245 | 146.4 |
| G | 11541 | 97.2 | 2947 | 158 |
| H | 7895 | 98,2 | 3582 | 120,8 |
| Jeg | 7365 | 97.2 | 1416 | 141.3 |
| K | 10839 | 100.4 | 3102 | 122.1 |
| L | 7605 | 96,9 | 1401 | 161.8 |
| M | 8149 | 96 | 1308 | 170.4 |
| N | 12741 | 97,5 | 3403 | 134.9 |
| P | 9768 | 102.8 | 2629 | 143.6 |
| Q | 13066 | 91.3 | 3354 | 169,9 |
| RASMUSSEN | 8537 | 101.3 | 2839 | 127,5 |
| S | 12053 | 99.1 | 3313 | 174.3 |
| T | 10615 | 97,7 | 2213 | 107,7 |
| V | 9169 | 96.1 | 1878 | 121.7 |
| W | 1715 | 94,7 | 1531 | 100 |
| Y | 7574 | 94,5 | 1234 | 138 |
Tabel 2. Liste over værdier for normal god landbrugspraksis fluorescens og oxidativ stress sonde fluorescens fremstillet ved FC analyse af gærceller udtrykker Aβ42-NGL mutanter for 16 h. Denne tabel viser de gennemsnitlige fluorescens intensitet (MFI) og CV for hver mutant.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
En bred vifte af sygdomme er forbundet med ophobning af fejlfoldede proteiner i cellulære indskud6,7,8,33. Har gjort mange anstrengelser at optrævle de molekylære mekanismer, der udløser udbrud af disse sygdomme ved hjælp af beregningsmæssige metoder, som ikke tager hensyn protein fusioner, eller in vitro- metoder, hvor proteinkoncentration forbliver konstante under reaktionen. Men inden for cellen, proteiner er konstant syntetiseret og nedbrydes i en overfyldt og uensartet omgivelser. Dette forklarer de hyppige uoverensstemmelser mellem i siliciummangan, in vitro og i vivo sammenlægning egenskaber af amyloidogenic proteiner.
Der er flere grunde til hvorfor simpel cellulær modeller, såsom gær, er et rimeligt valg at studere sammenlægning og tilknyttede toksicitet af proteiner relateret til neurodegenerative sygdomme i en mere biologisk sammenhæng. For eksempel, giver de os mulighed for at analysere virkningen af protein proteinmisfoldning aggregering på en intakt cellulære befolkning ved hjælp af hurtig analyser som gennemført her. Dog bør vi overveje at oxidative niveauer kan variere blandt gærstammer. Således, for at sammenligne mellem stammer, eller endda mellem forskellige proteiner, oxidanter og reduktionsmidler, såsom DTT og diamin, bør anvendes til at etablere en oxidation/reduktion skala.
I eksemplet med illustreret i denne artikel, bioinformatic analyse, cellulære protein kvantificering, er protein lokalisering imaging og samtidige FC analyse af normal god landbrugspraksis aktivitet og oxidativ stress niveauer blevet integreret for at identificere de molekylære arter ansvarlig for oxidative skader fremkaldes ved protein sammenlægning reaktioner. Resultaterne viser, at de mere opløselige Aβ42 varianter, snarere end den mere sammenlægning-tilbøjelige, fremme de højeste oxidativt stress. Dette peger på arternes diffusible bliver de farligere Aβ42 arter in vivo. Lavere toksicitet aggregere varianter synes at reagere både det faktum, at disse konformationer er afsondret i PI og deres privilegerede nedbrydning af protein kvalitet maskiner, hvilket resulterer i lavere protein niveauer end deres opløselige modparter.
Den beskrevne metode er ikke begrænset til analysen af den oxidative stress produceret af Aβ42 aggregerede/opløselige arter og kan også anvendes til at studere en række protein sammenlægning lidelser. Desuden, teknikken kan være nyttigt at overvåge effekten af sammenlægning hæmmere på cellulære oxidativt stress, giver mulighed for at skille sig for yderligere kliniske anvendelser af disse molekyler, der fremmer akkumuleringen af oxidative aktive protein arter. Endelig, så længe en fluorogenic sonde er tilgængelig, tilgangen tilbyder bemærkelsesværdige muligheder til at identificere de molekylære arter, der er ansvarlig for andre toksiske virkninger forbundet med protein sammenlægning i en hurtig en nem måde, giver kvantitative data.
Subscription Required. Please recommend JoVE to your librarian.
Acknowledgments
Materials
| Name | Company | Catalog Number | Comments |
| Yeast cells BY4741 | ATCC | 201388 | Genotype: MATa his3Δ1 leu2Δ0 met15Δ0 ura3Δ0 |
| pESC(-Ura) plasmid | Agilent Genomics | 217454 | Yeast expression plasmid with a Gal promotor. Selectable marker URA3 |
| Yeast Synthetic Drop-out Medium Supplements | Sigma | Y1501 | Powder |
| Yeast Nitrogen Base Without Amino Acids | Sigma | Y0626 | Powder |
| Raffinose | Sigma | R7630 | Powder |
| Glucose | Sigma | G7021 | Powder |
| Galactose | Sigma | G0750 | Powder |
| Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | Solution 10X |
| CellROX Deep Red Reagent | Life Technologies | C10422 | Free radical cell-permeant fluorescent sensor, non-fluorescent while in a reduced state, and exhibits bright fluorescence upon oxidation by reactive oxygen species (ROS), with absorption/emission maxima at 644/665 nm. |
| Y-PER protein extraction reagent | Thermo Scientific | 78990 | Liquid cell lysis buffer |
| Acrylamide/Bis-acrylamide | Sigma | A6050 | Solution |
| Bradford dye reagent | Bio-Rad | 5000205 | Dye reagent for one-step determination of protein concentration |
| β-amyloid antibody 6E10 | BioLegend | 803001 | Mouse IgG1. The epitope lies within amino acids 3-8 of beta amyloid (EFRHDS). |
| Goat anti-mouse IgG-HRP conjugate | Bio-Rad | 1721011 | |
| Membrane Immobilon-P, PVDF | Millipore | IPVH00010 | |
| Luminata forte | Merk | WBLUF0100 | Premixed, ready to use chemiluminescent HRP detection reagent |
| Phenylmethanesulfonyl fluoride solution (PMSF) | Sigma | 93482 | Protease inhibitor. Dissolved at 0.1 M in ethanol |
| FACSCanto flow cytometer | BD Biosciences | 657338 | Equipped with a 488 nm blue laser for the detection of GFP, and 635 nm red laser / 530/30 nm BP filter and 660/20 BP filter |
| Mini Trans-Blot Electrophoresis Transfer cell | Bio-Rad | 1703930 | Protein transference system |
| Mini-PROTEAN Tetra Handcast Systems | Bio-Rad | 1658000FC | Electrophoresis system |
References
- Frydman, J. Folding of newly translated proteins in vivo: the role of molecular chaperones. Annual Review of Biochemistry. 70, 603-647 (2001).
- Hartl, F. U., Bracher, A., Hayer-Hartl, M. Molecular chaperones in protein folding and proteostasis. Nature. 475, 324-332 (2011).
- Wong, E., et al. Molecular determinants of selective clearance of protein inclusions by autophagy. Nature Communications. 3, (2012).
- Winkler, J., Tyedmers, J., Bukau, B., Mogk, A. Chaperone networks in protein disaggregation and prion propagation. Journal of Structural Biology. 179, 152-160 (2012).
- Glickman, M. H., Ciechanover, A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiological Reviews. 82, 373-428 (2002).
- Dobson, C. M. Getting out of shape. Nature. 418, 729-730 (2002).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Aguzzi, A., O'Connor, T. Protein aggregation diseases: pathogenicity and therapeutic perspectives. Nature Reviews Drug Discovery. 9, 237-248 (2010).
- Rapezzi, C., et al. Transthyretin-related amyloidoses and the heart: a clinical overview. Nature Reviews Cardiology. 7, 398-408 (2010).
- Deas, E., et al. Alpha-synuclein oligomers interact with metal ions to induce oxidative stress and neuronal death in Parkinson's disease. Antioxidants & Redox Signaling. 24, 376-391 (2016).
- Brundin, P., Melki, R., Kopito, R. Prion-like transmission of protein aggregates in neurodegenerative diseases. Nature Reviews Molecular Cell Biology. 11, 301-307 (2010).
- Frost, B., Diamond, M. I. Prion-like mechanisms in neurodegenerative diseases. Nature Reviews Neuroscience. 11, 155-159 (2009).
- Arrasate, M., Mitra, S., Schweitzer, E. S., Segal, M. R., Finkbeiner, S. Inclusion body formation reduces levels of mutant huntingtin and the risk of neuronal death. Nature. 431, 805-810 (2004).
- Ross, C. A., Poirier, M. A. Opinion: what is the role of protein aggregation in neurodegeneration? Nature Reviews Molecular Cell Biology. 6, 891-898 (2005).
- Mishra, R., Sjölander, D., Hammarström, P. Spectroscopic characterization of diverse amyloid fibrils in vitro by the fluorescent dye Nile red. Molecular BioSystems. 7, 1232-1240 (2011).
- Klunk, W. E., Jacob, R. F., Mason, R. P. Quantifying amyloid by congo red spectral shift assay. Methods in Enzymology. 309, 285-305 (1999).
- Khurana, V., Lindquist, S. Modelling neurodegeneration in Saccharomyces cerevisiae: why cook with baker's yeast? Nature Reviews Neuroscience. 11, 436-449 (2010).
- Tenreiro, S., Outeiro, T. F. Simple is good: yeast models of neurodegeneration. FEMS Yeast Research. 10, 970-979 (2010).
- Moosavi, B., Mousavi, B., Macreadie, I. G. Yeast model of amyloid-β and Tau aggregation in Alzheimer's disease. Journal of Alzheimer's Disease. 47, 9-16 (2015).
- Tenreiro, S., Munder, M. C., Alberti, S., Outeiro, T. F. Harnessing the power of yeast to unravel the molecular basis of neurodegeneration. Journal of Neurochemistry. 127, 438-452 (2013).
- Yang, J., Hao, X., Cao, X., Liu, B., Nyström, T. Spatial sequestration and detoxification of huntingtin by the ribosome quality control complex. eLife. 5, (2016).
- Braun, R. J., Büttner, S., Ring, J., Kroemer, G., Madeo, F. Nervous yeast: modeling neurotoxic cell death. Trends in Biochemical Sciences. 35, 135-144 (2010).
- Figley, M. D., Gitler, A. D. Yeast genetic screen reveals novel therapeutic strategy for ALS. Rare Diseases. 1, e24420 (2013).
- Cooper, A. A., et al. Alpha-synuclein blocks ER-Golgi traffic and Rab1 rescues neuron loss in Parkinson's models. Science. 313, 324-328 (2006).
- Johnson, B. S., McCaffery, J. M., Lindquist, S., Gitler, A. D. A yeast TDP-43 proteinopathy model: exploring the molecular determinants of TDP-43 aggregation and cellular toxicity. Proceedings of the National Academy of Sciences of the United States of America. 105, 6439-6444 (2008).
- Bharathi, V., et al. Use of ade1 and ade2 mutations for development of a versatile red/white color assay of amyloid-induced oxidative stress in saccharomyces cerevisiae. Yeast. 33, 607-620 (2016).
- Pallarès, I., Ventura, S. Advances in the prediction of protein aggregation propensity. Current Medicinal Chemistry. , (2017).
- Villar-Piqué, A., Ventura, S. Protein aggregation propensity is a crucial determinant of intracellular inclusion formation and quality control degradation. Biochimica et Biophysica Acta. 1833, 2714-2724 (2013).
- Navarro, S., Villar-Piqué, A., Ventura, S. Selection against toxic aggregation-prone protein sequences in bacteria. Biochimica et Biophysica Acta. 1843, 866-874 (2014).
- Morell, M., de Groot, N. S., Vendrell, J., Avilés, F. X., Ventura, S. Linking amyloid protein aggregation and yeast survival. Molecular BioSystems. 7, 1121-1128 (2011).
- Conchillo-Solé, O., et al. AGGRESCAN: a server for the prediction and evaluation of "hot spots" of aggregation in polypeptides. BMC Bioinformatics. 8, 65 (2007).
- Fernandez-Escamilla, A. M., Rousseau, F., Schymkowitz, J., Serrano, L. Prediction of sequence-dependent and mutational effects on the aggregation of peptides and proteins. Nature Biotechnology. 22, 1302-1306 (2004).
- Renner, M., Melki, R. Protein aggregation and prionopathies. Pathologie Biologie.(Paris). 62, 162-168 (2014).
- Carija, A., Navarro, S., de Groot, N. S., Ventura, S. Protein aggregation into insoluble deposits protects from oxidative stress. Redox Biology. 12, 699-711 (2017).