Summary
세균성 세포는 높은 공간 구성 됩니다. 느린 성장 Myxococcus xanthus 셀에이 조직은 시간이 지남에 따라, 여러 세대에 걸쳐 spatiotemporal 고해상도 형광 라이브 셀 이미징에 대 한 설정 개발 되었다. 이 메서드를 사용 하 여 염색체 분리 및 세포 분열에 대 한 중요 한 단백질의 spatiotemporal 역학 결정 수 있습니다.
Abstract
세균성 세포의 형광 라이브 셀 이미징 단백질과 중앙 세포 주기 이벤트 기본 염색체의 공간과 일시적인 역동성의 분석에서 주요 방법입니다. 그러나, 이미지 느린 성장 박테리아 나타냅니다에 이러한 분자의 fluorophores 및 phototoxicity의 photobleaching 때문에 도전 이미지 수집 중입니다. 여기, 우리가 Myxococcus xanthus (이 4-6 h의 세대 시간)의 경우이 한계를 우회 하는 간단한 프로토콜을 설명 합니다. 이 위해, M. xanthus 셀 두꺼운 영양소를 포함 한 천 패드 온도 제어 습 한 환경에서 재배 됩니다. 이러한 조건 하에서 우리가 단일 세포의 성장에 따라 개별 셀의 두 배로 시간을 결정 합니다. 또한, 주요 휴대와 같은 염색체 분리 및 세포 분열 여러 이상 ParB-YFP, FtsZ-GFP, mCherry PomX 등 관련 붙일 레이블된 마커 단백질을 포함 하는 셀의 형광 라이브 셀 이미징으로 이미지로 수 처리 셀 순환입니다. 그 후, 인수 이미지 montages 또는 영화를 생성 하기 위해 처리 됩니다.
Introduction
세균성 세포는 공간적으로 매우 많은 단백질 세포질 구획1,2,,34이내 비대칭으로 지역화 구성 됩니다. 이 지역화 종종 매우 동적 이며, 세포 주기 신호 또는 외부 신호에 대 한 응답에서 시간에 따라 변화. 동등 하 게, 세균성 염색체는 공간적으로 매우 개별 loci 전과 분리 과정5중 특정 subcellular 위치에 배치 되 고 조직. 이 동적 공간 조직, 사업부, 세포 주기 규칙, 차별화, 운동 성, 신호 변환으로 염색체 조직 성장과 분리;에 대 한 중요 하다 따라서, 그것은 세균성 기능의 기본적으로 모든 측면을 영향을 줍니다.
이 세포질 과정의 spatiotemporal 역학 대장균, 새 균의 subtilis, 비 브리 오 cholerae, 및 Caulobacter crescentus 중요 한 봉사와 다른 세균성 종의 다양 한 분석 되 모형 유기 체입니다. 그러나,이 4 개의 종만 거 대 한 세균성 다양성의 작은 스펙트럼을 커버 되며, 아마도 당연히 이러한 종 사이의 큰 계통 발생 거리를 감안할 때, 세포 조직 및 양극 화 메커니즘에 다른 박테리아입니다. 이 결국 세균 세포의 spatiotemporal 역학 기본 일반적인 원리를 추출 수 추가 세균성 종을 공부에 대 한 필요를 발생 시킵니다.
그람 음성 델타 proteobacterium M. xanthus 박테리아6협력과 사회적 행동의 연구에 모델 생물 이다. M. xanthus 엄격한 되 고 그것은 세포 바깥쪽 높은 조정에, 집단 패션과 다른 미생물7먹이 확산 식민지를 형성 하는 영양소, 존재. 영양소 부족에 대응, 셀, 내부 셀의 수천의 구성 된 fruiting 시체의 형성에서 발달 프로그램 시작 막대 모양의 운동 세포 분화에 구형 2 중 포자8. 행동, 즉, 군비 및 fruiting 몸 형성의 두 종류만 고체 표면에 있는 세포에 의해 실행 됩니다. 또한, 두 영양 조건 셀에에서 참여 직접 셀 연락처 운동 성 자극 하거나 받는 사람9,10 독 기능 외부 막 단백질의 교환 등을 포함 하는 프로세스 , LPS11, exopolysaccharides에 인접 하 여 운동 성 자극의 교환12, 셀 및 셀에 의해 신호 세포 표면-정박 단백질13,14신호.
최근, M. xanthus 모델 유기 체 공부 운동 성 규정15, 세포 분열16,,1718및 염색체 조직19 기본 메커니즘에 대 한 될 수 있다 ,,2021. 중요 한 단계는 M. xanthus 세포 주기 관련 붙일 레이블된 단백질16, 를 들고 긴장에 스냅 샷 이미지 또는 짧은 시간 경과 녹음을 사용 하 여 형광 현미경으로 자세히 분석 된 17,18,,1920. 이상적으로, 많은 셀 따라야 한다 단일 셀 해상도와 형광 라이브 셀 이미징 세포 주기 매개 변수에 강력한 양적 데이터를 얻기 위해 하나 이상의 전체 세포 주기 위한 여. 그러나, 이것은의 경우에 도전 M. xanthus 표준 실험실 조건에서 4-6 h는 상대적으로 긴 세대 시간이 고 fluorophores 및 이미지 수집 동안 phototoxicity의 photobleaching 때문에.
여기, 우리 따라 프로토콜 설명 M. xanthus 형광 라이브 셀 적어도 24 h에 대 한 이미징 및 여러 세포 주기를 포함 하 여 단일 셀 해상도 셀. 중요 한 것은, 전체 프로토콜 중 셀 천 패드에 유지 됩니다 및 연락처 종속 활동의 소셜 라이프 스타일에 대 한 필수적인 수 문의 가까이에 M. xanthus. 프로토콜 또한 모니터 모양, 크기, 사단, 및 형광 프로브에 대 한 사용자가 단일 셀 해상도 높은 시간 해상도에 있으며 따라서, 셀 다양성의 정량화 및 세포 주기 이벤트의 상관 관계를 수 있습니다.
Protocol
1. 준비 및의 성장 M. xanthus 긴장
주: 표 1 과 표 2를 참조 하십시오.
- 준비 1 %casitone 국물 (CTT) 성장 매체 1% (w/v) 췌 장 소화의 카 세 인 (예를 들어, Bacto casitone), 10 mM Tris HCl pH 8.0, 1mm KH2포4 pH 7.6, 8mm MgSO422, 보충 대 (50 µ g/mL) 또는 oxytetracycline (10 µ g/mL)입니다. 이후 다른 박테리아와 오염 위험을 줄이기 위해 모든 미디어에 gentamycin (10 µ g/mL)를 추가 M. xanthus 세포가 그것에 자연적으로 저항.
- 1%의 5 mL을 접종 CTT 관련 포함 된 야생의 단일 갓 성장된 식민지와 antibiotic(s) 입력 (WT) DK1622 23, SA4420 (ΔmglA)24, SA4797 (ΔmglA, ΔpomX/PpomZ mCherry pomX )16, SA8241 (ΔmglA, ftsZ+/PnatftsZ-gfp), 또는 SA4749 (ΔmglA, parB+/PnatparB-yfp)에 1 일의 아침입니다.
- 단일 resuspend M. xanthus 1 %CTT 무 균 튜브에 항생제로 보충 하 고 50ml 삼각 플라스 크 1%의 5 mL를 포함 하는 전체 서 스 펜 션 전송의 500 µ L에 식민지 CTT.
참고: 10 배 볼륨 문화 삼각 플라스 크를 사용 하 여 충분 한 aeriation 및 최적의 성장을 보장.
- 단일 resuspend M. xanthus 1 %CTT 무 균 튜브에 항생제로 보충 하 고 50ml 삼각 플라스 크 1%의 5 mL를 포함 하는 전체 서 스 펜 션 전송의 500 µ L에 식민지 CTT.
- 8 세대 (4-6 h의 세대 시간으로 대략 40-48 h) 32 ° C, 220 rpm, 어둠 속에서 떨고에 대 한 셀을 성장. 지 수 성장 단계에 세포를 유지 (OD550 < 1.2) 고정 단계에 도달에서 그들을 방지. 필요한 경우, 0.1의 OD550 에 관련 antibiotic(s) 포함 된 신선한 1 %CTT 매체로 셀 희석-0.2.
참고: 단일 셀 현미경 검사 법에 대 한 한 최적의 세550 은 0.5-0.7. 이 세550, 세포의 충분 한 수는 정량화 세포 매개 변수 통계 분석 허용 하려면 이미지 당 존재.
2입니다. 현미경 샘플의 준비
참고: 현미경으로 볼 수 있도록 셀 현미경 coverslip에 배치 되며 다음 영양소를 포함 하는 agarose 패드에 의해 덮여. coverslip 플라스틱에 붙어 또는 금속 프레임 기계 제공을 지원 합니다. 현미경 검사 법에 대 한 준비, 1 %agarose/TPM/0.2% 대형 패드 CTT 준비 되어야 한다 사전에 2.1-2.3 단계에 설명 된 대로. 또한 여기에 사용 되는 특정 제품에 대 한 테이블의 자료 를 참조 하십시오.
- TPM 버퍼 (10 mM Tris HCl pH 7.6, 1mm KH2포4 pH 7.6, 8mm MgSO4)의 500 mL를 준비 하 고 필터 또는 오토 클레이 브 소독 병 최고의 필터를 사용 하 여.
참고: 살 균 버퍼는 실 온에서 몇 달 동안 저장할 수 있습니다. - 0.2%를 포함 하는 1 %agarose 현미경 솔루션 준비 CTT (믹스 80 mL TPM 버퍼와 1 %CTT 매체의 20 mL와 agarose의 1 g). agarose는 녹은 때까지 전자 레인지에가 열.
참고: 0.2 %CTT 성장 하 고 기아 방지 셀 수 있도록 충분 한. 현미경 매체에서 CTT의 높은 농도 높은 배경 형광 발생 합니다. - 페 트리 접시 녹은 agarose 0.5 c m의 두께 (대 한 11.5 cm x 11.5 cm 광장 페 트리 접시, 녹은 agarose의 약 60 mL가 필요)와 실내 온도 내려 식힙니다.
참고: agarose 패드 저장할 수 습 한 환경에서 4 ° C에서 최대 2 일 동안.- 미리 사용 하기 전에 적어도 15 분 동안 32 ° C에서 1% agarose/TPM/0.2% CTT 패드 따뜻한.
참고: 현미경 검사 법에 대 한 셀을 준비 하려면 다음과 같이 2.4 2.8.
- 미리 사용 하기 전에 적어도 15 분 동안 32 ° C에서 1% agarose/TPM/0.2% CTT 패드 따뜻한.
- 메 마른 유리 coverslip 장소 (60 mm x 22 mm, 두께: 0.7 m m) (그림 1A); 중간에 구멍 있는 플라스틱 또는 금속 프레임에 이 프레임 얇은 coverslip에 대 한 기계적 지원 역할 하 고 현미경 중 드리프트를 줄이는 데 도움이 됩니다. 테이프와 프레임에 coverslip 수정.
- 프레임을 준비 하는 1 m m 두꺼운 금속 격판덮개에서 75 m m × 25 m m 프레임을 잘라 다음 잘라 중간에 적절 한 크기의 구멍 (20 m m × 30 m m이이 실험에서).
- 기 하 급수적으로 성장의 10-20 µ L를 추가 M. xanthus 는 coverslip 셀.
- 셀 이나 시간 경과 기록에 단백질의 추적을 단순화 하기 위해 셀에 표준 표식으로 형광 0.5 µ m 스피어를 추가 합니다.
- TPM 버퍼에서 스피어 1: 100 희석 하 고 몇 개월까지 4 ° C에서 저장 합니다. 사용 하기 전에 철저 하 게 동요 하 고 셀에 희석된 스피어의 5-10 µ L를 추가 합니다.
참고: 여기 스피어는 형광에 있습니다 모든 일반적인, 녹색, 노랑, 파랑과 빨강 형광 채널 사용 되었다.
- TPM 버퍼에서 스피어 1: 100 희석 하 고 몇 개월까지 4 ° C에서 저장 합니다. 사용 하기 전에 철저 하 게 동요 하 고 셀에 희석된 스피어의 5-10 µ L를 추가 합니다.
- 작은 패드에 밖으로 큰 미리 데워 1% agarose/TPM/0.2% CTT 패드 coverslip의 크기 약 잘라내어 셀 (그림 1B) 위에 놓습니다. 1% agarose/TPM/0.2% CTT agarose 패드 증발을 방지 하 고 습 한 환경에서 셀 유지 위에 coverslip을 놓습니다.
참고: 혼자 coverslip 적어도 2 시간에 대 한 중요 한 증발을 방지 합니다. 이상 경과 기록에 대 한 1% agarose/TPM/0.2% CTT 패드와 coverslip 샌드위치 증발을 방지 하기 위하여 파라핀 필름으로 밀봉 한다. - Agarose 패드의 아래쪽에 연결 하는 세포를 15-20 분 동안 32 ° C에 현미경 샘플을 품 어. 다음 시간 경과 현미경 녹음을 시작 합니다.
3. 현미경 및 시간 경과 수집
참고: 여기에 설명 된 프로토콜 자동 초점, 100 X 거꾸로 widefield 현미경을 위해 개발 되었다 / 1.30 나 오일 PH3 목표, X, Y 자동화 단계, sCMOS 카메라, 광원, 녹색 형광에 대 한 필터, 레드-형광, 또는 황색 형광 단백질, 그리고 온도 제어 보육 실입니다. 이 챔버는 일정 온도에서 빛 으로부터 보호 하는 세포를 유지 합니다.
- 미리 열 인큐베이션 챔버와 ~ 1-2 h 32 ° C에 현미경 현미경 검사 법을 시작 하기 전에.
참고: 현미경 설정에 따라 난방 걸릴 수 있습니다 더 이상. 사전 난방 드리프트를 줄이기 위해 필수적 이며, 자동 제어 시스템을 안정화. - 현미경에 전환 하 고 현미경 제어 소프트웨어를 시작 합니다. 올바른 목표를 선택 하 고 올바른 거울과 얻으려고 필터 단계 녹색 형광, 레드-형광, 또는 황색 형광 단백질의 이미지 뿐 아니라 이미지.
참고: 현미경은 일반적으로 현미경 제어 및 이미지 수집을 위한 기본 소프트웨어와 함께 제공 됩니다. 여기는 상용 소프트웨어 현미경 및 이미지 수집을 제어 하 사용 되었다 ( 재료의 표참조). - 목표와 32 ° c.에 미리 incubated 샘플의 하단에 렌즈에 고품질 침수 오일 한 방울을 추가 가장 낮은 가능한 Z 샘플 현미경 스테이지에 배치 하는 경우 대물 렌즈의 손상을 방지 하려면 위치에서 목표를 놓습니다. 현미경 단계에 샘플와 목적으로 "구멍 쪽" 금속 프레임을 배치 합니다. 무대 홀더에서 샘플을 안전 하 게 고정.
- Z-방향으로 가까이 목표 스테이지를 이동 하 여 셀에 초점. 샘플 하단 측면에 기름 방울 하 고 대물 렌즈 느린 단계 이동 접촉. X에서 무대를 이동 / Y 방향으로 여러 개의 단일 셀까지 표시 보기 지역에 셀 초점면에 있을 때. 그 이상의 형광 스피어 나중 인수 이미지를 정렬 하려면 보기 지역에는 다는 것을 확인 하십시오.
참고: 최적의 조건 보기 (2048 x 2048 픽셀 또는 133.1 x 133.1 µ m)의 지역 당 15-30 셀의 셀 밀도 도달 한다. - 필요한 경우 여러 파장 및 무대 위치에서 이미지를 수집 하는 현미경을 허용 하는 시간 경과 실험을 설정 하는 현미경 제어 소프트웨어의 다차원 수집 마법사를 엽니다.
- 기본 탭에서 Timelapse 및 여러 파장을 활성화 합니다. 추가 탭은 윈도우의 왼쪽에 표시 됩니다.
- 디렉터리 선택 인수 이미지를 저장할 컴퓨터의 하드 드라이브에 빈 폴더를 선택 하 고 저장 탭을 클릭 합니다. 증분 기본 이름 파일이 있으면 연속 데이터 집합 이전 것 덮어쓰지 않습니다 되도록 활성화 합니다. 다음 실험 날짜와 스트레인 이름 또는 실험의 제목 이름을 주어 라.
- 시간 매개 변수를 조정 Timelapse 탭을 클릭 하십시오. 지속 시간 24 시간을 설정 하 고 시간 간격 을 20 분으로 설정. 시간 포인트의 번호는 자동으로 변경 됩니다.
참고: 최적의 시간 간격 실험 및 분석 세포 기능에 따라 달라 집니다. 잦은 이미지 인수 photobleaching을 발생할 수 있습니다. 따라서, 시간적 해상도 photobleaching 사이 교환 경험 찾을 수 있어야 합니다. 4-6 h의 2 배 시간에 이미지 수 수 쉽게 획득 5 분 (또는 원하는 경우 심지어 작은 간격)의 간격에 단계 대조 현미경 검사 법. 24 h의 시간-과정을 통해 형광 현미경 이미지 약 15-30 분 간격으로 기록 되어야 원하는 경우. - 파장 탭에서 클릭 변경 하 여 각 시간 지점에서 각 이미지에 대 한 인수를 파장의 수를 선택 합니다.
참고: 각 파장에 대 한 새 탭의 왼쪽에 나타납니다는 다차원 수집 " 마법사와 파장 아래로 위에서 순서에서 취득 될 것 이다. 각 파장에 대 한 수집 설정은 별도로 수정할 수 있습니다. - 위에서 첫 번째 파장 탭을 클릭 합니다. 단계 대조 조명 드롭-다운 목록에서 선택 합니다. 노출 에 대 한 100 ms를 선택 하 고 모든 시간 포인트 취득 드롭-다운 목록에서 선택 합니다. 드롭-다운 목록에서 없음을 선택 하 여 자동 노출 를 비활성화 합니다.
- 각 시간 지점에서 인수 하는 각 파장에 대 한 단계 3.5.5를 반복 합니다. 실험 설정 및 설명 붙일 레이블된 단백질 여기에 대 한 다음 매개 변수 사용 노출: mCherry 융합 단백질에 대 한 250 ms, 200 ms YFP 융해 단백질, 및 GFP 융합 단백질에 대 한 1000 ms.
참고: 각 스트레인 및 형광 단백질에 대 한 최적의 조명 설정은 램프 강도와 각 파장에 대 한 이미지 수집 시간을 변경 하 여 사전에 결정 되어야 합니다. 너무 긴 이미지 수집 시간 phototoxic 효과 증가 하 고 궁극적으로 성장 검거와 세포 죽음으로 이어질 것 이다. 따라서, 이미지 품질 및 세포 생존을 절충을 달성 한다. - 같은 실험에 기록 된 셀의 수를 늘리기 위해 여러 단계 위치에서 이미지를 취득 합니다.
- 여러 단계 위치에서 이미지를 수집, 기본 탭에서 여러 단계 위치 를 활성화 합니다. 단계 탭에서 클릭 하 고 보기의 필드 보고 라이브 버튼을 클릭.
- 관심 (ROI) 영역 보기의 필드에 때까지 X/Y-방향에서 단계를 이동 합니다. 저장 X-및 Y-좌표를 클릭 하 여에 "+" 단계 탭에 이동 무대 다시 X/Y-방향으로 좌표 저장 하는 새로운 투자 수익을 찾을 때까지 다시는 "+"를 클릭 하 여 합니다. 원하는 지역 번호 저장 될 때까지 이동 합니다.
참고: 형광 이미지 수집, 경우에 관심사 (ROIs)의 지구는 너무 가까이 있는지 확인 phototoxicity을 최소화 하기 위해 서로.
- 확인 한 번 더 그 셀에 초점에 다른 저장 된 X-및 Y-위치를 클릭 하 여 있으며 AFC 개최 실험의 과정을 통해 일정 저장 된 Z 위치를 계속 클릭 하 여 하드웨어 자동 초점을 시작 합니다.
- 다차원 수집 마법사에서 취득 을 클릭 하 여 현미경 제어 소프트웨어에서 시간 경과 녹음을 시작 합니다.
참고: 한 창 인수는 각 파장에 대 한 나타나고 있는 추가 창이 다음 그림 획득까지 시간과 획득된 시간 포인트의 수를 표시 하는 나타납니다. - 셀에에서 다는 여전히 초점 후 처음 몇 시간 이미지의 품질을 최대화 하 고 필요한 경우 촛점을 시간 경과 기록에 포인트 확인 하십시오.
4. 생성 시간 경과 영화 및 이미지 정렬
참고: 몇몇 상업 및 무료 소프트웨어 패키지 이미지 수집 및 이미지 분석에 사용할 수 있습니다. 우리는 상업적으로 사용 가능한 소프트웨어를 사용 하 여 ( 재료의 표참조) 여러 사전 설치 된 플러그인과 추가 도구.
- 이미지 분석/처리 소프트웨어가 설치 되어 있는 컴퓨터에서 시간 경과 기록에서 개별 이미지를 저장 합니다.
- 소프트웨어를 시작 하 고 검토 다차원 데이터를 클릭 하 여 스택으로 이미지를 엽니다 | 기본 파일 선택 | 디렉터리 선택. 다차원 데이터와 폴더를 엽니다. 데이터 집합을 확인 하 고 보기; 데이터 집합 시간에서 단일 이미지 포인트 하나 끝날 때까지 표시 됩니다. 활성화 (스택 만들기)에 대 한 파장을 고 해야 하는 스택의 로드 이미지를 클릭 모든 이미지를 선택 합니다. 모든 파장에 대해이 단계를 반복 하 고 완성 된 스택을 저장 합니다.
- (선택 사항) 파일을 사용 하 여 동영상에 필요한 모든 이미지를 엽니다 | 오픈.
참고:이 좋습니다 이미지를 열려고 한 인수 파장에 의해 감속 하지 않을 컴퓨터 계산 능력이 제한 된 경우를 한 번에. 시간 경과 기록, 예를 들어, 시작, 끝, 또는 몇 시간 포인트의 특정 부분, 생략 하는 경우 다음이 조정할 수 있습니다 완성 된 영화. - 드리프트에 대 한 수정 해야 하는 이미지의 더미를 활성화 합니다. 애플 리 케이 션에 의해 정렬 도구를 엽니다 | 자동 맞춤... 확인 스택 이미지와 참조 평면으로 처음 비행기 포인트 에 대 한 원본으로. 소스 스택 버튼으로 스택을 선택 하 고 적용을 클릭 합니다.
참고: 자동 맞춤 시간과 컴퓨팅 파워를 걸릴 것입니다 하지만 드리프트 현미경 설정의 큰 더미를 해결 하는 좋은 방법입니다. 이 자동 정렬 스피어 포함 하지만 또한 그들 없이 작동 수 있습니다 하는 경우에 잘 작동 합니다. - 정렬 된 스택을 저장 합니다.
- ROIs를 사용 합니다.
참고: 시간 경과 형광 현미경 쉽게 컴퓨팅 파워를 많이 채택 하 고이 영화의 다운스트림 처리 속도 데이터 파일의 큰 세트를 만듭니다. 따라서이 좋습니다 ROIs를 식별 하 여 작은 파일 작업을 수행 하는 세포를 분리.- 사각형 영역 도구를 선택 합니다. 단계 대조 이미지에 ROI를 직접 그려서 관심의 셀 주위 ROI를 만듭니다. 그 관심의 셀 표시 되 고 전체 시간 경과 영화 내내 초점이 다는 것을 확인 하십시오.
- 동일한 데이터 집합의 두 번째 파장의 시간 경과 영화를 엽니다. 형광을 단계 대조 사진에서 투자 수익을 전송 하려면 두 번째 파장의 이미지 도구 사용 하 여 전송 지역 지역으로 | 영역 전송. 소스 이미지 와 대상 이미지와 두 번째 파장 dataset으로 위상 대비 데이터 집합을 선택 합니다. 모든 영역 을 선택 하 고 확인을 누릅니다.
- 동일한 데이터 집합에 대 한 인수는 각 파장에 대 한 단계 4.6.2를 반복 합니다.
- ROI를 선택 하 고 편집으로 스택으로 복제 | 복제 | 스택... 또는 Shift + Ctrl + D 키를 누릅니다. 다음 파일을 중복된 스택 저장 | 저장 원본 데이터와 같은 폴더에.
- 4.6.4 단계를 반복 하 여 동일한 데이터 집합에 대 한 인수는 모든 파장의 각 ROI에 대 한
- MOV 또는 AVI 형식의 동영상을 생성 하려면 열고 스택을 통해 동영상 만들기 기능 | 영화. 소스 스택 버튼으로 시간 경과 녹음을 선택 합니다. 출력 형식, 프레임 속도, 프레임 수를 선택 하 고 저장을 클릭 합니다.
Representative Results
M. xanthus 고체 표면에 이동 하는 느린 성장 박테리아 이다. 우리의 실험 설치를 테스트 하려면 우리는 메구미 DK1622 WT 셀과 시간 경과 실험을 수행. 단계 대조 이미지 (그림 2A, B) 24 시간 5 분의 간격으로 인수 했다. 대부분의 셀 그룹에 정렬. 예상 했던 대로, 세포 운동 성 표시 하 고 그룹에서 주로 이동. 더 셀 때때로 이동 방향을 반대로 관찰 합니다. 이러한 결과 WT 셀 테스트 조건 하에서 세포 운동 성 일반적으로 작동 좋습니다. 그러나, 세포는 5 분 마다 기록 됩니다, 경우에 개별 세포의 식별은 어렵습니다. 또한, 세포 운동 이기 때문에, 많은 셀 탈출 하거나 어렵게 연장된 기간에 대 한 셀에 따라 보기의 필드를 입력 합니다.
같은 추적 하려면 M. xanthus 세포 운동 성25위해 필수적 이다, mglA 유전자 라이브 셀 이미징에 의해 세포 주기의 여러 라운드, 개별 긴장을 삭제할 수 있습니다. 이 보기의 필드 밖으로 이동 하는 이미징 프로토콜 중에서 세포를 방지 합니다. 프레임 삭제 시 외에 설명 된 대로 생성 됩니다. 26
예상 했던 대로, 비 운동 ΔmglA 셀 (그림 3), 위상 대비 라이브 셀 이미징에 세포 활성 운동을 표시 되지 않았다. 우리는 성장과 개별 셀의 microcolony 형성 동안에 따라 수 있었다. 이미지 5 분 간격으로 24 시간에 대 한 인수 했다 시간 경과 기록에 따라, 단일 셀 해상도 interdivision 시간 (2 개의 세포 분열 이벤트 사이의 시간)을 계량 가능 했다. ΔmglA 돌연변이의 셀 했다 235 ±의 간 사단 시간 50 분 (n = 97 셀). 약 4 h, interdivision 시간 WT 셀 서 스 펜 션 문화 측정 배로 시간을 비슷합니다. 이 증거를 제공 한다 M. xanthus 셀이 실험 조건 하에서 최적의 성장.
우리의 설정 셀 YFP 표시 된 단백질을 오랜 기간 동안 추적 하는 동안 일반적으로 성장 하는 것을 허용 하는지 여부를 조사 하기 위해 형광 시간 경과와 영상 수행 했습니다 M. xanthus YFP 태그 단백질을 표현 하는 셀. 이 위해, 우리는 복제 (ori)의 기원에 대 한 표식으로 ParB YFP 따라. ParB에서 ParABS 시스템의 구성 요소는 M. xanthus 오 라; 근 위 동위 사이트에 바인딩합니다 따라서, 원본 복제 및 염색체 분리 뒤19,,2021될 수 있습니다. 와 이미지 수집 (위상 대조 및 형광, YFP 채널에서 200 ms 획득 시간) 20 분 마다, 세포 성장, 분할 및 성장 24 h(그림 4)에 표시 됩니다. 녹음의 시작에 ParB YFP 셀(그림 4)의 대부분의 subpolar 지역에서 두 개의 클러스터 형성. 직전 또는 세포 분열 후에, subpolar ParB YFP 중복 된 셀 극에서 클러스터. 두 클러스터 중 약 40-60 분 (그림 4A, B) 후 최종 subpolar 위치를 도달 하는 새로운 셀 극 translocated 두 번째 복사 하는 동안 오래 된 셀 극에 남아 있었다. 이 관측은 얇은 천 패드19를 사용 하 여 짧은 시간 경과 녹음에서 생성 된 이전 데이터와 일치 하 여. 우리는이 실험 설정 허용 느린 성장에 여러 세포 주기에 염색체 분리를 추적 하기 위해 형광 시간 경과 현미경 결론 M. xanthus 세포, perturbing 세포 성장 또는 염색체 분리 기계 없이.
비슷한 실험에서 우리는 시간 경과 형광 현미경으로 세포 분열에 대 한 마커를 따라 하고자 결정 했다. 다른 거의 모든 박테리아와 비슷한 M. xanthus 세포 분열16,,1718FtsZ, 세균성 tubulin 같은 GTPase 필요 합니다. FtsZ는 midcell, 소위 Z 반지, 세포 분열27,28에 필요한 다른 모든 단백질을 보충 하는 데 도움이에 고리 모양의 구조를 형성 한다. M. xanthus, Z-반지와 midcell에서 위치 형성은 3 PomXYZ 단백질16,17에 의해 자극 됩니다. 이 3 개의 단백질 2 개의 딸 세포에서 핵양체의 중간에 "어머니" 세포에 세포 분열의 사이트에서는 핵양체에 걸쳐 전송 염색체 관련 된 복잡 한 양식. 핵양체의 중간 midcell, 염색체 분리, 그리고 여기 전에 PomXYZ 복잡 한 신병 FtsZ 일치 하 고 Z-고리 형성을 자극.
여기, 우리가 먼저 ftsZ gfp를 표현 하는 비 운동 셀 따 랐 다. 때문에 우리 5-fold 노출 시간을 증가 ParB YFP 보다 약한 형광 신호 FtsZ GFP 전반적인 쇼 1 GFP 채널에서 s. 예상 했던 대로, FtsZ GFP의 강한 축적 했다만 midcell에서 관찰 하 고이 지역화 결정 세포 분열 긴축 (그림 5A)의 위치. FtsZ GFP는 주로 더 이상 셀에 midcell에서 클러스터를 형성 했다. 또한이 클러스터 시간이 지남에 강도 증가 분명 했다. 후에 세포 분열, FtsZ GFP 다시 두 딸에 midcell에 축적 된 약 2 시간 후 (그림 5B) 세포 관찰 합니다. 이것은 인구에 있는 세포의 약 50% 스냅 샷 분석16,17에 따라 midcell에서 FtsZ 지역화 표시 찾는 일치.
두 번째 실험에서는 우리가 mCherry pomX표현 24 h에 대 한 비 운동 ΔmglA 셀 따 랐 다. PomXYZ 시스템의 일환으로, PomX midcell16에서 세포 분열을 자극 함으로써 Z-링 형성과 위치, 가이드 수 있습니다. MCherry-PomX의 형광 신호 강한 이며 중요 한 것은 250 양의 형광 채널에 노출 시간을 수 있습니다, 그리고 모든 셀 크기에서 성장 하 고 24 시간 ( 후 microcolonies를 형성 하는 실험의 과정을 통해 세포 분열 이벤트 표시 그림 6A). 이전에 보고 된16, 거의 모든 세포는 mCherry PomX 클러스터 포함. Midcell 및 midcell midcell를 실험 과정 translocated에서 클러스터에 지역화의 대부분. 세포 분열 동안 mCherry PomX 클러스터 클러스터를 받는 각 딸 세포로, 분할 했다. FtsZ-GFP, 반대로 mCherry PomX midcell 80-90%의 세포 주기에서 지역화 하 고 세포 분열 (그림 6B) 후 곧이 위치를 도달 했다.
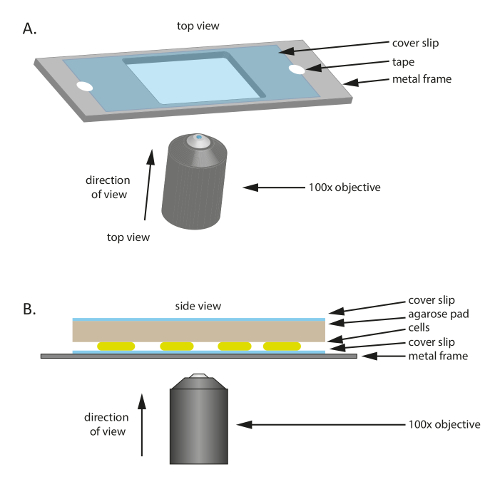
그림 1 :이 연구에서 사용 하는 실험 설정의 도식. (A) A 금속 또는 플라스틱 프레임 샘플에 대 한 지원 역할. coverslip 샘플의 움직임을 줄이기 위해 테이프와 금속 프레임에 고정 됩니다. (B) 측 실험 샘플 설정의 볼. 셀은 coverslip (A)에 표시 된에 장착 됩니다. Agarose 패드 영양분과 세포에 습도 제공 하는 셀 위에 배치 됩니다. Agarose 패드 증발을 줄이기 위해 추가 coverslip 적용 됩니다. 높은 품질의 이미지는 100 X 기름 침수 단계 대조 목적 사용 됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 2 : 단계 대조 WT의 시간 경과 현미경 M. xanthus 셀. 셀 24 h에 대 한 다음, 이미지 마다 5 분 (A)를 인수 했다 동일한 보기의 필드 마다 5 분의 대표 이미지 표시 됩니다. 색된 화살표는 개별 셀의 이동 방향을 나타냅니다. 같은 색 시간이 지남에 동일한 셀을 표시합니다. 숫자는 분을 나타냅니다. 눈금 막대: 5 µ m. (B) 모든 시간 후 보기의 동일한 필드의 이미지가 표시 됩니다. 보기의 동일한 필드 표시 되지만 셀 이동 하기 때문에, 셀 지속적으로 입력 하 고 보기의 필드를 떠나 note. 숫자는 시간에 시간을 나타냅니다. 눈금 막대: 5 µ m. PH: 단계 대조. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 3 : 단계 대조 비 메구미의 시간 경과 현미경 M. xanthus 셀. ΔmglA 셀 24 h. 이미지 마다 5 분을 인수 했다 그리고 모든 시간 후 대표 이미지 표시 됩니다 따라 했다. 선택 된 세포 분열 constrictions 주황색 화살표와 함께 표시 됩니다. 숫자는 시간에 시간을 나타냅니다. PH: 단계 대조입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
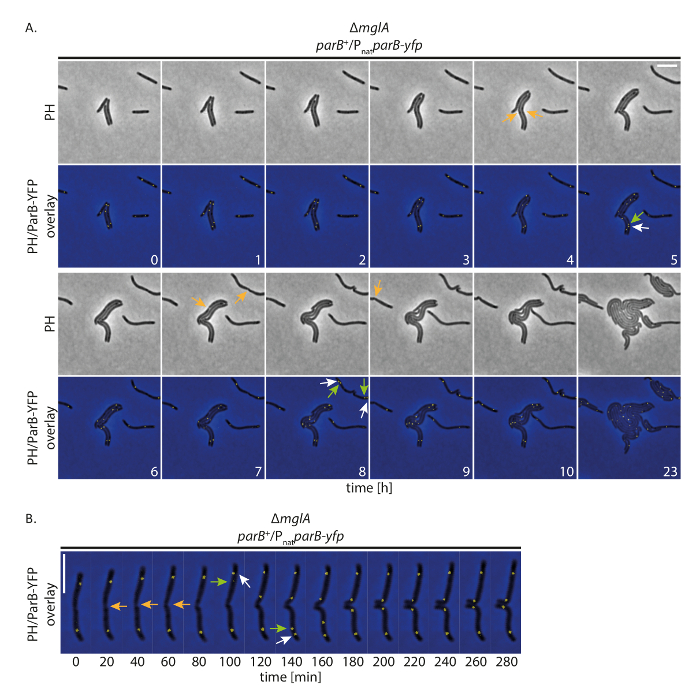
그림 4 : 비 운동에서 ParB YFP의 형광 시간 경과 현미경 M. xanthus 셀. ΔmglA 돌연변이표현 parB-yfp 네이티브 parB (SA4749; ΔmglA; 앞의 셀 parB +/PnatparB-yfp) 24 h에 대 한 위상 대비 및 형광 현미경 검사 법에 선행 되었다. (A) 이미지 마다 20 분을 인수 했다 및 대표 이미지 매시간 10 h 24 h. 이미지가 단계 대조 (PH)에 표시 된 후 동일한 셀 함께 표시 됩니다 때까지 그리고 단계 대조의 오버레이 YFP 신호. 선택한 셀 분할 주황색 화살표와 함께 표시 됩니다. 흰색과 녹색 화살표 translocating 클러스터를 표시 하는 녹색 화살표와 함께 ParB YFP 클러스터 중복 이벤트를 나타냅니다. 숫자는 시간에 시간을 나타냅니다. 눈금 막대: 5 µ m. (B) 이미지 (A)에서 인수 했다 그러나 더 높은 시간 해상도 표시 됩니다. 숫자는 분을 나타냅니다. 화살표 (A)에서 있습니다. 눈금 막대: 5 µ m. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 5 : 비 운동에서 FtsZ GFP의 형광 시간 경과 현미경 M. xanthus 셀. 네이티브 ftsZ (SA8241; ΔmglA;의 존재에 ftsZ gfp 를 표현 하는 ΔmglA 돌연변이의 셀 ftsZ +/PnatftsZ-gfp) 24 h에 대 한 위상 대비 및 형광 현미경 검사 법에 선행 되었다. (A) 이미지 마다 20 분을 인수 했다 및 대표 이미지 10 h까지 매 시간 표시 됩니다, 24 h. 후 동일한 셀 함께 이미지 단계 대조 (PH)에 단계 대조 및 GFP 신호의 오버레이로 표시 됩니다. 선택한 셀 분할 주황색 화살표와 함께 표시 됩니다. 흰색 화살표는 midcell에서 FtsZ GFP 클러스터를 나타냅니다. 숫자는 시간에 시간을 나타냅니다. 눈금 막대: 5 µ m. (B) 이미지 (A)에서 인수 했다 그러나 더 높은 시간 해상도 표시 됩니다. 숫자는 분을 나타냅니다. 녹색 및 흰색 화살표 왼쪽 및 오른쪽 셀에 FtsZ GFP 클러스터를 각각 표시합니다. 주황색 화살표는 세포 분열을 나타냅니다. 눈금 막대: 5 µ m. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 6 : MCherry-PomX 비 운동에서 형광 시간 경과 현미경 M. xanthus 셀. 비 운동 ΔpomX 셀 mCherry-PomX (SA4797;mglAΔ ΔpomX/PpomZ mCherry pomX) 축적 단계 대비 및 형광 현미경 검사 법 (A) 20 분 마다 24 h에 대 한 선행 했다 대표 이미지 10 h까지 매 시간 표시 됩니다, 이미지 단계 대조 (PH)에 단계 대조 및 mCherry의 오버레이로 표시 됩니다 24 h. 후 동일한 셀 함께 신호. 선택한 셀 분할 주황색 화살표와 함께 표시 됩니다. 흰색과 녹색 화살표는 각각 이벤트, 분할 전후 mCherry PomX 클러스터를 나타냅니다. 숫자는 시간에 시간을 나타냅니다. 눈금 막대: 5 µ m. (B) (A)에서 인수 했다 영상과 더 높은 시간 해상도 표시 됩니다. 화살표 (A)에서 있습니다. 눈금 막대: 5 µ m. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
| 세균성 긴장 | 관련 유전자 형1 | 참조 |
| DK1622 | Wildtype | 23 |
| SA4420 | ΔmglA | 24 |
| SA4749 | ΔmglA; parB+/attB:: PnatparB yfp (pAH7) | 이 연구 |
| SA4797 | ΔmglA; ΔpomX / attB::PpomZ mCherry pomX (pAH53) | 16 |
| SA8241 | ΔmglA; ftsZ+/ mxan18-19::PnatftsZ gfp (pDS150) | 이 연구 |
| 괄호에 플라스 미드 표시 유전자 융해를 포함 하 고 게놈에 표시 된 사이트에서 intergated 했다. AttB 사이트에서 통합 된 플라스 미드 또는 mxan18-19 intergenic 지역에서 표현 했다 그들의 네이티브 발기인 (Pnat) 또는 pomZ (PpomZ)의 네이티브 발기인. |
||
표 1:이 연구에 사용 된 세균성 긴장의 목록입니다.
| 플라스 미드 | 관련 특성 | 참조 |
| pAH7 | PnatparB yfp; Mx8 attP; TetR | 19 |
| pAH53 | PpomZ mCherry pomX; Mx8 attP ; KmR | 16 |
| pDS150 1 | PnatftsZ gfp ; mxan18-19 ; TetR | 이 연구 |
| pMR3691 | Vanillate 유도할 수 있는 유전자 발현에 대 한 플라스 미드 | 18 |
| pKA51 | PnatftsZ gfp ; Mx8 attP; TetR | 17 |
| 1 pDS150: pDS150 pKA51 Mx8 attP 사이트 mxan18-19 intergenic 지역으로 대체 되었다의 파생입니다. 이 대 한 mxan18-19 intergenic 지역 Mxan18-19 BsdRI fwd 뇌관으로 pMR3691에서 증폭 되었다 (GCGATCATTGCGCGCCAGACGATAACAGGC)와 Mxan18-19 계 BlpI (GCGGCTGAGCCCGCGCCGACAACCGCAACC)와 pKA51로 복제. |
||
표 2:이 연구에 사용 된 플라스 미드의 목록입니다.
Discussion
형광 라이브 셀 이미징 세균성 세포의 spatiotemporal 역학을 공부 하는 강력한 도구 되고있다. 그러나와 같은 운동 및 느린 성장 박테리아의 시간 경과 형광 현미경 M. xanthus,, 도전 되었으며 짧은 시간 기간에 대해서만 수행 했다. 여기, 우리 제시의 라이브 셀 이미징에 대 한 쉬운--사용 하 고 강력한 방법 M. xanthus 시간 경과 형광 현미경 검사 법에 의해. 이 메서드를 사용 하면 셀과 단일 셀 해상도 세포 주기의 여러 라운드 붙일 레이블된 단백질에 따라 수 있습니다.
느린 성장의 라이브 셀 이미징의 성공에 영향을 주는 몇 가지 필수 구성 요소는 M. xanthus 세포를 포함 하 여: 1) 셀 첨부;에 대 한 단단한 표면 영양분과 산소; 2) 여부 3) 일정 한 습도 온도; 그리고 4) 노출 시간과 이미지 수집 주파수 등 실험 조건의 최적화.
우리의 실험 설정, 영양소 보충 두꺼운 agarose 패드 사용 합니다. 단일 셀을 따라 미세 장치 반대 agarose 두꺼운 패드를 사용 하 여 몇 가지 기본적인 혜택만도 몇 가지 단점이 있다. 첫째, agarose 패드 뿐만 아니라에 대 한 화면을 제공 합니다 M. xanthus 첨부 파일 및 운동 뿐만 아니라 적어도 24 h에 대 한 성장 위한 충분 한 영양소를 세포. 둘째, 스냅 샷된 분석 일반적으로 붙일 레이블된 단백질의 세포내 지역화를 공부 하는 데 사용 이전 agarose 패드16,,1729의 동일한 종류에서 수행 되었다. 따라서, 스냅 샷된 분석에서 데이터는 여기에 설명 된 방법으로 얻은 데이터를 직접 비교할 수 있습니다. 셋째, agarose 패드를 쉽게 수정 하 고 항생제 또는 CuSO4 등 다른 보충 교재와 보충 수 있습니다 그리고 vanillate 일반적으로 위해 사용 되는 유전자 식 유도18,30. 마지막으로, 세포 실험 과정 중 양식 microcolonies 수 있습니다, 때문에이 설정 또한 허용 한다 분석 되 고 특정 매개 변수에서 직접 셀 상호 작용의 효과 공부. 이 점은의 경우 특히 중요 한 M. xanthus 이 박테리아 표시 여러 연락처 종속 상호 작용 때문에. 이 방법의 주요 결점은 실험 조건 실험의 기간에 대 한 사전 설정 됩니다. 대조적으로, 미세 장치는 일반적으로 항생제31예를 들어 추가 하 여 실험 과정 중 실험 조건 변경 허용.
무료 소프트웨어 패키지 (예를 들어, MicrobeJ, Oufti) 자동으로 단일 세포 및 개별 세포 내 단백질 지 방화의 성장을 분석을 사용할 수 있습니다. 그러나,이 소프트웨어는 단일 셀 또는 셀의 작은 그룹의 분석에 적합. 따라서, 그것은 자동으로 여기에 설명 된 24 시간 기록에 대해 생성 된 데이터 분석에 대 한 도전 남아 있습니다.
요약, 우리는 느린 성장 라이브 셀 이미징 수행 하는 쉬운--사용 하 고 재현할 수 프로토콜 설명 M. xanthus 박테리아. 우리는 간단한 영양소 보충 agarose 패드는 적어도 24 시간에 대 한 성장을 유지 하 고 관찰 하 고 분석 단백질 지 방화 및 여러 세대에 걸쳐 단일 셀 해상도와 성장에 대 한 허용 하는 충분 한 보여줍니다.
Disclosures
저자 들은 아무 경쟁 금융 관심사 선언 합니다.
Acknowledgments
이 작품에 의해 독일 연구 위원회 (DFG) Transregio 174 "Spatiotemporal 역학의 세균성 세포의 프레임 워크 내에서 지원 했다" 고 맥스 플랑크 사회.
Materials
| Name | Company | Catalog Number | Comments |
| DMI6000B with AFC | Leica microsystems | 11888945 | Automated inverted widefield fluorescence microscope with adaptive focus control |
| Universal mounting frame | Leica microsystems | 11532338 | Stage holder for different sample sizes |
| HCX PL FLUOTAR 100x/1.30 oil PH3 | Leica microsystems | 11506197 | Phase contrast objective |
| Orca Flash 4.0 camera | Hamamatsu | 11532952 | 4.0 megapixel sCMOS camera for picture aquisition |
| Filter set TXR ET, k | Leica microsystems | 11504170 | Fluorescence filter set, Ex: 560/40 Em: 645/75 |
| Filter set L5 ET, k | Leica microsystems | 11504166 | Fluorescence filter set, Ex: 480/40 Em: 527/30 |
| Filter set YFP ET, k | Leica microsystems | 11504165 | Fluorescence filter set, Ex: 500/20 Em: 535/30 |
| ProScan III | Prior | H117N1, V31XYZEF, PS3J100 | Microscope automation controller with interactive control center |
| EL 6000 light source | Leica microsystems | 11504115 | External fluorescence light source |
| Incubator BLX Black | Pecon | 11532830 | Black incubation chamber surrounding the microscope |
| Tempcontrol 37-2 digital | Leica microsystems | 11521719 | Automated temperature control for incubation chamber |
| Gentmycin sulphate | Carl Roth | 0233.4 | Gentamycin |
| Oxytetracylin dihydrate | Sigma Aldrich | 201-212-8 | Oxytetracyclin |
| Kanamycin sulphate | Carl Roth | T832.3 | Kanamycin |
| Filtropur BT25 0.2 bottle top filter | Sarstedt | 831,822,101 | Bottle top filter for sterilization of buffers |
| Deckgläser | VWR | 630-1592 | Glass cover slip (60 x 22 mm, thickness: 0.7 mm) |
| Seakem LE agarose | Lonza | 50004 | Agarose for microscopy slides |
| Leica Metamorph AF | Leica microsystems | 11640901 | Microscope control software and software for picture analysis |
| Tetraspeck Microsperes, 0.5 µm | ThermoFisher | T7281 | Fluorescent microspheres |
| petri dish | Greiner Bio-one | 688102 | 120 mm x 120 mm x 17 mm squared petri dish for agarose pads |
| BD Bacto Casitone | Becton Dickinson | 225930 | Casitone |
| Parafilm M | VWR | 291-1213 | Parafilm |
| Tris(hydroxymethyl)-aminomethane | Carl Roth | AE15.2 | Tris |
| Magnesium sulphate heptahydrate | Carl Roth | P027.2 | Magnesium sulphate |
| Potassium dihydrogen phosphate p.a. | Carl Roth | 3904.1 | Potassium dihydrogen phosphate |
| 1% CTT medium: 1 % (w/v) BD Bacto™ casitone, 10 mM Tris-HCl ph 8.0, 1 mM potassium phosphate buffer pH 7.6, 8 mM MgSO4 | Cultivation medium for M.xanthus | ||
| TPM buffer: 10 mM Tris-HCl ph 8.0, 1 mM potassium phosphate buffer pH 7.6, 8 mM MgSO4 | Buffer for preparation of microscopy slides for M.xanthus |
References
- Shapiro, L., McAdams, H. H., Losick, R. Why and how bacteria localize proteins. Science. 326 (5957), 1225-1228 (2009).
- Treuner-Lange, A., Søgaard-Andersen, L. Regulation of cell polarity in bacteria. J Cell Biol. 206 (1), 7-17 (2014).
- Laloux, G., Jacobs-Wagner, C. Spatiotemporal control of PopZ localization through cell cycle-coupled multimerization. J Cell Biol. 201, 827-841 (2013).
- Rudner, D. Z., Losick, R. Protein subcellular localization in bacteria. Cold Spring Harb. Perspect. Biol. 2 (4), 000307 (2010).
- Badrinarayanan, A., Le, T. B. K., Laub, M. T. Bacterial chromosome organization and segregation. Annu Rev Cell Dev Biol. 31 (1), 171-199 (2015).
- Munoz-Dorado, J., Marcos-Torres, F. J., Garcia-Bravo, E., Moraleda-Munoz, A., Perez, J. Myxobacteria: Moving, Killing, Feeding, and Surviving Together. Front Microbiol. 7, 781 (2016).
- Berleman, J. E., Kirby, J. R. Deciphering the hunting strategy of a bacterial wolfpack. FEMS Microbiol Rev. 33 (5), 942-957 (2009).
- Konovalova, A., Petters, T., Søgaard-Andersen, L. Extracellular biology of Myxococcus xanthus. FEMS Microbiol. Rev. 34, 89-106 (2010).
- Nudleman, E., Wall, D., Kaiser, D. Cell-to-cell transfer of bacterial outer membrane lipoproteins. Science. 309, 125-127 (2005).
- Vassallo, C. N., et al. Infectious polymorphic toxins delivered by outer membrane exchange discriminate kin in myxobacteria. eLife. 6, 29397 (2017).
- Vassallo, C., et al. Cell rejuvenation and social behaviors promoted by LPS exchange in myxobacteria. Proc Natl Acad Sci USA. 112 (22), 2939-2946 (2015).
- Li, Y., et al. Extracellular polysaccharides mediate pilus retraction during social motility of Myxococcus xanthus. Proc. Natl. Acad. Sci. USA. 100, 5443-5448 (2003).
- Kim, S. K., Kaiser, D. Cell alignment required in differentiation of Myxococcus xanthus. Science. 249, 926-928 (1990).
- Lobedanz, S., Søgaard-Andersen, L. Identification of the C-signal, a contact dependent morphogen coordinating multiple developmental responses in Myxococcus xanthus. Genes Dev. 17, 2151-2161 (2003).
- Schumacher, D., Søgaard-Andersen, L. Regulation of cell polarity in motility and cell division in Myxococcus xanthus. Annu Rev Microbiol. 71 (1), 61-78 (2017).
- Schumacher, D., et al. The PomXYZ proteins self-organize on the bacterial nucleoid to stimulate cell division. Dev Cell. 41 (3), 299-314 (2017).
- Treuner-Lange, A., et al. PomZ, a ParA-like protein, regulates Z-ring formation and cell division in Myxococcus xanthus. Mol Microbiol. 87 (2), 235-253 (2013).
- Iniesta, A. A., Garcia-Heras, F., Abellon-Ruiz, J., Gallego-Garcia, A., Elias-Arnanz, M. Two systems for conditional gene expression in Myxococcus xanthus inducible by isopropyl-beta-D-thiogalactopyranoside or vanillate. J Bacteriol. 194 (21), 5875-5885 (2012).
- Harms, A., Treuner-Lange, A., Schumacher, D., Søgaard-Andersen, L. Tracking of chromosome and replisome dynamics in Myxococcus xanthus. reveals a novel chromosome arrangement. PLoS Genet. 9 (9), 1003802 (2013).
- Iniesta, A. A. ParABS system in chromosome partitioning in the bacterium Myxococcus xanthus. PLoS One. 9 (1), 86897 (2014).
- Lin, L., Osorio Valeriano, M., Harms, A., Søgaard-Andersen, L., Thanbichler, M. Bactofilin-mediated organization of the ParABS chromosome segregation system in Myxococcus xanthus. Nat Commun. 8 (1), 1817 (2017).
- Hodgkin, J., Kaiser, D. Cell-to-cell stimulation of movement in nonmotile mutants of Myxococcus. Proc Natl Acad Sci U S A. 74 (7), 2938-2942 (1977).
- Kaiser, D. Social gliding is correlated with the presence of pili in Myxococcus xanthus. Proc Natl Acad Sci USA. 76 (11), 5952-5956 (1979).
- Miertzschke, M., et al. Structural analysis of the Ras-like G protein MglA and its cognate GAP MglB and implications for bacterial polarity. EMBO J. 30 (20), 4185-4197 (2011).
- Hodgkin, J., Kaiser, D. Genetics of gliding motility in Myxococcus xanthus. (Myxobacterales): Two gene systems control movement. Mol Gen Genet. 171, 177-191 (1979).
- Shi, X., et al. Bioinformatics and experimental analysis of proteins of two-component systems in Myxococcus xanthus. J Bacteriol. 190 (2), 613-624 (2008).
- Bi, E. F., Lutkenhaus, J. FtsZ ring structure associated with division in Escherichia coli. Nature. 354 (6349), 161-164 (1991).
- Lutkenhaus, J., Pichoff, S., Du, S. Bacterial cytokinesis: From Z ring to divisome. Cytoskeleton. 69 (10), 778-790 (2012).
- McLoon, A. L., et al. MglC, a Paralog of Myxococcus xanthus GTPase-Activating Protein MglB, Plays a Divergent Role in Motility Regulation. J Bacteriol. 198 (3), 510-520 (2015).
- Gomez-Santos, N., et al. Comprehensive set of integrative plasmid vectors for copper-inducible gene expression in Myxococcus xanthus. Appl Environ Microbiol. 78 (8), 2515-2521 (2012).
- Treuner-Lange, A., et al. The small G-protein MglA connects to the MreB actin cytoskeleton at bacterial focal adhesions. J Cell Biol. 210 (2), 243-256 (2015).



