Summary
Nous présentons ici un protocole de basé sur la spectrométrie de masse en tandem pour la quantification des antibiotiques fréquemment utilisés dans les unités de soins intensifs, nommément céfépime, méropénem, ciprofloxacine, la moxifloxacine, linézolide et pipéracilline.
Abstract
Il y a une demande croissante pour le suivi thérapeutique pharmacologique des antibiotiques dans de nombreuses installations cliniques, en particulier en ce qui concerne la mise en œuvre de programmes d’intendance aux antibiotiques de l’hôpital.
Dans les travaux en cours, nous présentons un Protocole multiplex haute performance liquid chromatography-spectrométrie de masse (HPCL-MS/MS) pour la quantification du céfépime, méropénem, ciprofloxacine, la moxifloxacine, linézolide et pipéracilline, couramment utilisés antibiotiques dans les unités de soins intensifs. La méthode était déjà complètement validée conformément aux lignes directrices de l’Agence européenne des médicaments.
Après un nettoyage rapide d’échantillon, les analytes sont séparés sur une colonne CLHP en phase inverse C8 en 4 minutes et quantifiés avec stables isotopes marqués internes normes correspondantes dans électrospray (ESI +) spectrométrie de masse en réaction multiples le temps de surveillance (MRM). La méthode présentée utilise une instrumentation simple offrant des conditions chromatographiques uniformes, ce qui permet le suivi quotidien et robuste thérapeutique antibiotique dans les laboratoires cliniques. La courbe d’étalonnage s’étend sur la plage de concentration pharmacocinétiques, donc y compris les montants antibiotiques à proximité de la concentration minimale inhibitrice (CMI) des bactéries sensibles et les concentrations maximales (Cmax) qui sont obtenues avec un bolus schémas d’administration. Sans la nécessité de la dilution du sérum avant le nettoyage de l’échantillon, l’aire sous la courbe pour un antibiotique administré peut être obtenue par le biais de mesures multiples.
Introduction
Bien que les antibiotiques ont révolutionné la pratique de la médecine, des infections bactériennes sévères demeurent des principales causes de morbidité et de mortalité dans les maladies graves1. À cet égard, l’administration rapide d’un anti-infectieux adapté à un dosage adéquat est de l’importance supérieure pour maladie contrôle2.
Un corps croissant d’évidence montre que le traitement empirique avec des antibiotiques à large spectre devient de plus en plus problématique avec la complexité des populations de patients. Ceci est particulièrement vrai pour les unités de soins intensifs (USI), où une énorme variabilité interindividuelle des principaux paramètres pharmacocinétiques de (PK) est fréquemment observée3,4. Par conséquent, les patients ICU courent un risque imminent de niveaux sous thérapeutiques avec le danger d’un succès thérapeutique insuffisant5,6. Là encore, les patients sont inutilement exposés à des concentrations d’antibiotique excessivement élevées pouvant entraîner des événements indésirables graves avec aucun des avantages cliniques7. L’abus d’antibiotiques et de la posologie insuffisante ont également alimenté la diffusion de la résistance aux antibiotique, qui devient une menace croissante pour la santé publique8.
D’améliorer l’utilisation des antibiotiques et de préserver leur effectivenessas longtemps que possible, l’Organisation mondiale de la santé a lancé un plan d’action global sur la résistance antimicrobienne en 20159. Programmes d’intendance antibiotiques constituent une pierre essentielle angulaire d’utilisation prudente des antimicrobiens en national de santé publique stratégies10, aider les cliniciens à améliorer sensiblement la qualité des soins aux patients11 et, en même temps, la réduire la résistance aux antibiotiques12. Antimicrobiens chez chaque patient par le biais de l’application du médicament thérapeutique suivi (TDM) est un instrument clé dans ce contexte13.
À ce jour, disponible dans le commerce TDM essais sont uniquement disponibles pour les antibiotiques glycopeptides et aminoglycosides. La quantification des substances des autres classes nécessite généralement un développement de la méthode interne ou une validation qui peut être encombrante. Nous avons, par conséquent, présenter en détail le protocole pour un dosage basé sur la spectrométrie de masse solide qui peut être utilisé pour la quantification des antibiotiques plus pertinents aux soins intensifs au sein de leurs gammes de concentration pertinente clinique14. La méthode a récemment mis en place dans nos installations de spectrométrie de masse et a été appliquée pour la TDM en réanimation de routine depuis lors. La procédure utilise un cadre analytique simple et direct avec un nettoyage uniforme échantillon, permettant la mise en oeuvre rapide des antibiotiques TDM dans de nombreuses installations avec des capacités de spectrométrie de masse.
Le protocole décrit ici a été optimisé pour la quantification du céfépime, méropénem, ciprofloxacine, la moxifloxacine, linézolide et pipéracilline dans le sérum humain, en utilisant la chromatographie liquide (LC) à dilution isotopique en combinaison avec un tandem mass spectrométrie (MS/MS). Pour la dilution isotopique méthodologie LC-MS/MS, composés marqués isotopes stables sont ajoutés à un échantillon d’intérêt avec une matrice spécifique (p. ex., sérum). Isotopes marqués normes peuvent distinguer de leurs homologues non étiquetées, à savoir l’analyte d’intérêt, en raison de différents poids moléculaires de la molécule naturelle et leurs produits de fragmentation, appelés une transition ion-pour-fille-ion parent. Comme composés marqués isotopes ont un comportement physico-chimique globalement presque identique par rapport à leurs homologues non étiquetées, ils sont des étalons internes idéales pour la SM/SM, ce qui permet une quantification de l’analyte presque indépendante de matrice avec un degré élevé de 15de précision. De nos jours, nombreux stables isotopes marqués normes interne qui peuvent être utilisés pour la quantification de petites molécules, y compris la TDM d’antimicrobiens, sont disponibles dans le commerce.
La séparation chromatographique de l’antibiotique analyser dans le protocole décrit est réalisée avec une colonne en phase inversée C8 alkyl-longueur de la chaîne analytique (100 mm x 2,1 mm, 3 µm taille des particules). Au cours du développement de la méthode, les éléments de la matrice normalisée standard interne pour tous les analytes était de 94,6 % à 105,4 %, avec un coefficient de variation de ≤8.3 %14.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Remarque : Il est recommandé de travailler sous une hotte lors de la manipulation de solvants organiques, comme le méthanol. Préparer tous les tampons et les phases mobiles dans des fioles jaugées. Si non précisés, les solutions peuvent être conservées à température ambiante pendant environ 1 mois après préparation.
1. préparation des étalons et des échantillons de contrôle de la qualité
Remarque : Une fiche d’analyse de données correspondant pour la préparation de solutions de stock et spike est donnée dans le Fichier supplémentaires. Pour des raisons de traçabilité, insérer le fabricant, numéro de catalogue et un numéro de lot de chaque antibiotique dans les colonnes correspondantes. Dissoudre tous les antibiotiques en une chambre froide à 4 ° C et garder le temps de travail le plus court possible.
- Préparation de 100 mL de méthanol à 25 % dans l’eau : pré-remplir une fiole jaugée de 100 mL avec 25 mL de méthanol absolu et remplissez-le à 100 mL d’eau distillée.
- Préparer 10 mL d’acide acétique 200 mM dans l’eau : pré-remplir une fiole jaugée de 10 mL avec 9 mL d’eau de qualité HPLC, ajouter 115 µL d’acide acétique (pureté de 99,5 %, 17,4 M) et ajouter de l’eau distillée jusqu'à 10 mL.
- Préparer 25 mL de méthanol à 25 % dans l’eau avec l’acide acétique 20 mM : pré-remplir une fiole jaugée de 25 mL avec 2,5 mL de la solution d’acide acétique aqueux de 200 mM, ajouter 6,25 mL de méthanol absolu et remplir le flacon de 25 mL d’eau distillée.
- Utiliser une balance de précision pour peser les montants appropriés des antibiotiques dans les tubes coniques 15 mL comme indiqué dans le Fichier supplémentaire dans la colonne poids initial.
- Préparer les solutions des fluoroquinolones, la ciprofloxacine et moxifloxacin dans le méthanol / eau de 25 % dont 20 mM d’acide acétique. Pour ce faire, ajoutez le volume correspondant aux quantités pondérées tel que décrit dans le Fichier supplémentaire dans la colonne « volume final ». Rapidement dissoudre les antibiotiques fluoroquinolones dans un bain à ultrasons pendant 2 min et au vortex intense.
- Préparer les solutions de céfépime, méropénem, linézolide et pipéracilline dans le méthanol / eau de 25 %. Pour ce faire, ajoutez le volume correspondant aux quantités pondérées comme décrit dans le Fichier supplémentaire dans la colonne volume final et dissoudre rapidement les antibiotiques au vortex intense. Dissoudre le Méropénem comme la dernière substance.
- Combiner les solutions mères de tous les antibiotiques tel que décrit dans le tableau correspondant volume de solution mère dans le Fichier supplémentaire pour produire dix fois spike-solutions concentrées.
- Spike neuf volumes de sérum exempt de drogues avec un volume des solutions concentrées spike dix fois pour obtenir les calibrateurs de sérum 0 – 7 et contrôle de qualité (QC) A – D. Par exemple, ajouter 0,5 mL de solution de spike à 4,5 mL de sérum dans un tube en polypropylène 10 mL et il incuber pendant 15 min dans l’entreposage au froid à 4 ° C sur un mélangeur à rouleaux à 50 tr/min.
- Utiliser une pipette à répétition pour générer 100 µL d’extraits des étalons et des QCs dans les tubes en polypropylène de 1,5 mL.
- Stocker les calibrateurs, contrôles qualité et antibiotiques solutions mères à-80 ° C jusqu'à six mois.
- Pour chaque antibiotique, également préparer une solution propre contenant 1 000 mg/L d’un seul antibiotique. Diluer la solution mère correspondante avec un diluant approprié (par exemple, pour la ciprofloxacine, utilisez 25 % méthanol-eau 20 mM d’acide acétique).
Remarque : Les solutions soignées aux antibiotiques sont nécessaires pour l’instrument-tuning seulement.
2. préparation du mélange d’étalons internes
NOTE : Normes internes sont homologues isotopique marqué les analytes d’intérêt qui sont ajoutés à un échantillon pendant le nettoyage de l’échantillon. Comme les étalons internes ont des propriétés physico-chimiques globalement presque identiques à leurs homologues non étiquetées, ils compensent les effets de matrice d’un échantillon donné.
- Préparer 10 mL de méthanol à 50 % dans de l’eau en ajoutant 5 mL de méthanol absolu à une fiole de 10 mL shake et remplissez-le à 10 mL d’eau distillée.
- Préparer 10 mL de méthanol à 50 % dans l’eau, y compris l’acide acétique 20 mM. Pour ce faire, ajouter 1 mL d’acide acétique de 200 mM dans une fiole de 10 mL, ajouter 5 mL de méthanol absolu et remplissez-le à 10 mL d’eau distillée.
- Générer des solutions mères étalons internes (IS) avec 1 000 mg/L directement dans le flacon fourni par le fabricant. Dissoudre le céfépime -13C12D3 sulfate dans l’eau distillée, Méropénem-D6, linézolide-D3et pipéracilline-D5 dans une solution de méthanol-eau de 50 %. Dissoudre la ciprofloxacine-D8 à 50 % méthanol-eau avec 20 mM acétate et moxifloxacin hydrochloride -13C1D3 dans l’eau distillée avec de l’acétate de 20 mM.
- Combiner les solutions mères IS dans un tube en polypropylène de 1,5 mL pour donner un mélange concentré quintuplé, passant de standard interne. Ajouter 10 µL du céfépime -13C12D,3, 10 µL de Méropénem-D6, 1 µL de ciprofloxacine-D8, 2 µL de moxifloxacine chlorhydrate -13C1D3, 2 µL de linézolide-D 3et 10 µL de l’Association pipéracilline-D5 965 µL de méthanol / eau de 25 %.
- Stocker les solutions stocks standards internes et la quintuple mélange concentré de IS à-80 ° C.
3. échantillon stockage
Remarque : Assurez-vous que le sérum est obtenu aussi rapidement que possible et que la chaîne du froid des échantillons congelés est maintenue.
- Collecter le sang dans les tubes de prélèvement de sérum.
- Laissez le caillot de sang pendant 20 à 30 min à température ambiante.
- Séparer le sérum du sang par centrifugation à 2 000 x g pendant 10 min.
- Transférer le surnageant dans un tube propre en polypropylène.
- Stocker le sérum jusqu'à six mois à-80 ° C jusqu'à ce qu’il est analysé. Vous pouvez également stocker les échantillons jusqu'à 3 jours à-20 ° C.
4. tampon de préparation pour la chromatographie
- Pour préparer le formiate d’ammonium 1 M dans l’eau, dissoudre 6,306 g de formiate d’ammonium dans 100 mL d’eau de qualité CLHP en utilisant un ballon de secousse de 100 mL. La solution de stocker jusqu'à 1 mois à 4 ° C.
- Préparer le mobile phase un [formiate d’ammonium 10 mM dans l’acide formique-eau (99.9:0.1 v/v)]. Pré-remplir une fiole jaugée de 1 000 mL avec environ 500 mL d’eau de qualité HPLC, ajouter 1 mL d’acide formique et 10 mL de la solution de formiate d’ammonium 1 M et remplissez-le à 1 000 mL avec l’eau de qualité HPLC. Transférer la phase mobile A dans une bouteille de verre propre et le raccorder au système HPLC. Stocker la phase mobile jusqu'à 2 semaines à température ambiante.
- Préparer la phase mobile méthanol absolu de qualité CLHP-B. de transférer dans une bouteille en verre propre et le raccorder au système HPLC.
- Utiliser du méthanol absolu comme l’aiguille laver solvant et connecter le tuyau correspondant à la bouteille en verre contenant une phase mobile B.
- Générer le sceau et un solvant de purge de l’acide formique-méthanol-eau (7:92.9:0.1, v/v/v). Pré-remplir une fiole jaugée de 1 000 mL avec environ 500 mL d’eau distillée, ajouter 70 mL de méthanol absolu, 1 mL d’acide formique et ajouter de l’eau distillée à 1 000 mL. Transférer le solvant dans une bouteille de verre propre et raccorder avec le système de CLHP.
Remarque : Différents systèmes de l’échantillonneur automatique utilisent une forte et un solvant de lavage aiguille faible. Dans un tel cas, préparer les solutions de lavage selon les recommandations du fabricant. Par exemple, la fort lavage avec de l’alcool de méthanol-eau-leur (70:20:10, v/v/v) et les faibles laver avec eau-méthanol (95 : 5, v/v).
5. instrument Tuning
Remarque : Cette étape est réalisée pour la mise en place de la méthode sur un spectromètre de masse spécifique.
- Diluer l’analyte pur 1 000 mg/L et les solutions étalons internes 01:10 ou au 1/100 dans un mélange de la phase mobile A et B (50/50, v/v), selon l’intensité de signal du détecteur. Programmez le spectromètre de masse avec la fonction autotune ou faire un réglage manuel pour le suivant parent-à-fille ions transitions14: céfépime (481.0 > 167.0/395.7), céfépime -13C12D3 (485,1 > 167,1 / 400,0), Méropénem (384.1 > 114.0/141.0), Méropénem-D6 (390,1 > 114.0/147.2), ciprofloxacine (332,0 > 231.0/245.0), ciprofloxacine-D8 (340.1 > 235.1/249.3), la moxifloxacine (402.0 > 261.0/383.9), la moxifloxacine-13 C1D3 (406,1 > 265.1/388.0), le linézolide (338,0 > 235.0/296.0), le linézolide-D3 (341,1 > 235.1/297.1), pipéracilline (518.0 > 143.0/358.9) et pipéracilline-D5 (523,1 > 142.8/364.1).
- Pour les appareils avec réglage automatique, utilisez la fonction autotune pour ajuster automatiquement la tension et les paramètres de l’entrée de MS à travers les détecteurs.
- Pour les instruments à réglage manuel, régler les paramètres (p. ex., tension de collision et l’énergie de collision) jusqu'à l’optimum (généralement le maximum) intensité du signal est obtenue au détecteur pour chaque ion parent et sa fille. Par exemple, branchez un mélange tee, livrer la phase mobile A et B (50/50, v/v) à 0,5 mL/min et insuffler en permanence la norme antibiotique ou intérieure soignée avec un débit de 0,1 mL/min.
6. HPLC-MS/MS Set-up
NOTE : Comprend le spectromètre de masse, système de CLHP (y compris l’échantillonneur automatique), et le logiciel correspondant dépend du fabricant. Adapter les paramètres de spectromètre de masse et la procédure de lavage selon les recommandations du fabricant.
- Stocker les paramètres de spectromètre de masse à un correspondant 'MS paramétrer le fichier '. Utilisez l’ionisation par électrospray en mode positif (ESI +) pour tous les analytes. Adapter les paramètres de source d’ion de l’instrument utilisé (par exemple, une tension capillaire de 1,5 kV, une source température de 120 ° C, une température de désolvatation de 400 ° C, un débit de gaz de désolvatation de 600 L/h, une tension de lentille de RF de 0,1 V et un temps de pause de 80 ms).
- Spécifiez l’analyte et normes internes ajuster les paramètres (p. ex., tension capillaire, énergie de collision) dans un «fichier MS'.
- Fixer les conditions de l’échantillonneur automatique comme suit dans le 'fichier d’entrée ': la température de l’échantillon à 10 ° C, avec une limite de ± 5 ° C ; la séquence de lavage à 1 x purge-lavage-purge avec une 600 µL purge remplacement de volume.
- Dans ce qui précède 'fichier d’entrée ', régler le débit à 0,4 ou 0,5 mL/min, le temps d’exécution à 4 min, le limiteur de pression à 345 bar et la température de la colonne à 30 ° C, avec une limite de ± 5 ° C. Ajoutez le nom de solvant de phases mobiles A et B et leur valeur A B/93% de 7 %, respectivement.
- Programmer le gradient chromatographique dans le 'fichier d’entrée ' comme suit : 0,00 – 0,10 min avec la phase mobile 7 % B/93% A, 0,11 à 0,60 min avec 65 % de phase mobile B/35% A, 0,61 – 2.10 min avec 95 % de phase mobile B/5% A, 2.11 – 4,00 min avec la phase mobile 7 % B/93% A.
Remarque : Calculer le volume de la colonne extra, le volume de la retenue pour la plate-forme instrumentale et les facteurs de rétention analyte tel que décrit dans l’USP < 621 > chromatographie orientation16.
7. échantillon mesure Master File
Remarque : Avec le 'exemple de fichier maître de mesure ', sérums des patients sont spécifiés, l’analyse HPLC-MS/MS est démarré et l’évaluation des données est effectuée. Deux fichiers de modèle distinct comprenant une paire de contrôle de basse et de haute qualité sont générés ; un modèle inclut QC paire A et C, l’autre paire QC un B et D.
- Créer un nouveau 'exemple de fichier maître de mesure '. Sélectionnez les clauses mentionnées ci-dessus 'MS paramétrer le fichier ', 'fichier MS'et « fichier d’arrivée » (article 6), insérez-les dans chaque ligne de l’échantillon et spécifiez le volume d’injection avec 15 µL.
- Dans l’ordre croissant, ajouter le texte « sample » de calibrateurs 0 – 7 et contrôle de la qualité (CQ) paire a/c ou paire QC b/j.
- Spécifiez le type d’échantillon. Sélectionnez le type d’échantillon « standard » pour des étalons et des « QC » pour les paires de contrôle de la qualité.
- Spécifiez la concentration de chaque substance antibiotique pour étalons correspondants et des contrôles de qualité (voir la feuille de calcul, concentration [µg/mL] Cal 7 – Cal 0, QC a/c ou b/j,).
- Programme de la 'méthode d’évaluation de données '. Utiliser les transitions qui ont été optimisées au cours de l’instrument tuning (section 5). Correspondre à chaque antibiotique avec la norme correspondante isotopique marqué (p. ex., Méropénem - Méropénem-D6).
8. nettoyage et analyse HPLC-MS/MS de l’échantillon
Remarque : Pour chaque lot d’échantillon, un contrôle de qualité jumelé avec une basse et à haute concentration d’antibiotique (QC ca ou QC b/j) est traité et analysé. Entre les différents lots, les échantillons appariés de QC sont utilisés dans une autre séquence (par exemple, le jour 1, sélectionnez le 'exemple de fichier maître de mesure ' y compris QC paire a/c ; le jour 2, sélectionnez la paire QC incluant un b/j. Le traitement des échantillons de sérum est illustré à la Figure 1.
- Préparer l’agent de précipitation 10 % méthyltert-butyl éther dans le méthanol (10 : 90, v/v) (par exemple, inscrire un 25 mL fiole jaugée avec 2,5 mL de méthyltert-butyl éther et remplissez-le à 25 mL avec du méthanol absolu).
- Placer la phase inversée C8 dans la chambre de la colonne. Connectez-le à l’HPLC et spectromètre de masse dans le sens de l’écoulement.
- Générer la liste d’exemples. Ouvrez le correspondant 'exemple de fichier maître de mesure ' modèle et ajouter les sérums des patients censés être traités à la liste. Générer des groupes de jusqu'à 20 échantillons de patients et de leur flanc avec la paire de contrôle de la qualité correspondante.
- Wet-prime le système HPCL utilisant le 'fichier d’entrée ' maitrise des logiciels : régler la fonction de « premier humide » à 50 % de phase mobile A/50% B et humide-premier pendant 2 min avec un débit de 1 mL/min.
- Actualiser la seringue. Pour ce faire, exécutez 6 coups de 600 µL dans le logiciel de commande.
- Equilibrer la colonne phase inverse de C8. En utilisant le logiciel, allumez le flux dans le 'fichier d’entrée ' et rincez-la avec 7 % mobile phase B/93% A un minimum de 5 min, à l’aide d’un débit de 0,5 mL/min. vérifier la température de la colonne de 30 ° C.
- Décongeler les échantillons de patients, une partie aliquote de calibrateurs 0 – 7 et une paire de contrôle de la qualité (A / B ou C/D).
- Avec une pipette à répétition, ajouter 25 µL du mélange standard interne à 100 µL calibrateur, échantillon QC ou sérum de patient dans un tube en polypropylène de 1,5 mL et vortex le tube pendant quelques secondes.
- Incuber le mélange pendant 5 min à température ambiante sur un dispositif trembleur de benchtop (p. ex., à 1 200 tr/min).
- Avec une pipette à répétition, ajouter 150 µL d’un réactif de précipitation au mélange échantillon-interne standard.
- Encore une fois, vortex le tube pendant quelques secondes et il incuber pendant 5 min à température ambiante sur un dispositif trembleur de benchtop (p. ex., à 1 200 tr/min).
- Centrifuger la suspension à 20 000 x g dans une centrifugeuse de table pendant 10 min à 4 ° C.
- Diluer le liquide surnageant 1:3 avec l’eau de qualité HPLC à l’aide d’un flacon en verre avec un insert-micro et charger les échantillons aussi traités dans l’échantillonneur automatique.
- Démarrer manuellement l’analyse HPLC-MS/MS le 'exemple de fichier de contrôle mesure '.
Remarque : Pour une conservation prolongée, bien rincer la colonne analytique selon la recommandation du manufacturier [par exemple, 0,5 mL/min méthanol / eau (50/50, v/v)] pour empêcher l’effondrement de phase.

Figure 1 : représentation schématique du nettoyage échantillon. Précipitation de protéines à la force centrifuge élevée donne une boulette dense et clair surnageant, indiquant que les précipitations protéine était complète. Tout le temps de traitement est environ 30 min, y compris le nettoyage de l’échantillon, la séparation chromatographique et l’analyse MS/MS. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
9. quantification et évaluation de la qualité
- Pour traiter les échantillons, ouvrir le correspondant 'exemple de fichier de contrôle mesure ', sélectionnez les calibrateurs et contrôles de qualité des échantillons de patients et d’évaluer avec le 'méthode de quantification des antibiotiques '.
- Vérifier si les sommets pour un analyte spécifique sont correctement intégrés. Inspecter les pics pour chaque étalon, QC et échantillon de patients et réintégrer manuellement à la ligne de base si nécessaire.
- Étudier la courbe d’étalonnage et déterminer si elle remplit les critères de qualité suivants : a) linéarité sur la gamme d’étalonnage ensemble, b) un étalonnage coefficient r2 > 0,995, c) la déviation de chaque solution étalon ± 15 % de la valeur nominale, à l’exception de la limite inférieure de quantification (PPQM), où il faut ± 20 %.
- Rejeter un étalon ne respecte ne pas les critères susmentionnés et réévaluer la courbe d’étalonnage, y compris l’analyse de régression.
- Étudier les contrôles de qualité et d’examiner si les écarts sont ± 15 % de la valeur nominale.
- Si la concentration d’un échantillon de patients est supérieure à la concentration de l’étalon plus haut, diluer l’échantillon avec de l’eau distillée, jusqu'à 1:5 (par exemple, 100 µL de sérum plus de 400 µL d’eau distillée) avant le nettoyage de l’échantillon. Reperform étapes 8,8 – 8.14 pour cet échantillon spécifique et retraiter il.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
En utilisant le protocole décrit, un chromatogramme typique est représenté sur la Figure 2. Selon l’United States Pharmacopeia (USP) chromatographie lignes directrices16, le volume mort de colonne dans le système actuel a été déterminé avec ~0.22 mL et le volume de la colonne extra (y compris l’injecteur, tuyaux et connecteurs) avec ~0.08 mL, ce qui donne un volume de braquage de ~0.30 mL. Les facteurs de rétention calculée pour tous les analytes étaient 2,8 (pour céfépime) - 4.2 (par pipéracilline).
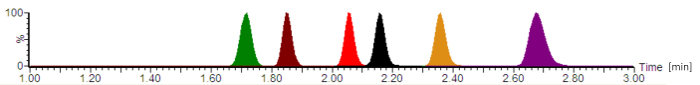
Figure 2 : chromatogramme analytique typique avec des intensités de signal normalisé. Les antibiotiques sont à élution dans l’ordre suivant : céfépime (vert), Méropénem (brun), ciprofloxacine (rouge), la moxifloxacine (noir), linézolide (orange) et pipéracilline (violet). Les temps de rétention, qui ne figurent pas dans les minutes et les symétries de pic d’analyte varient en fonction de la composition exacte des phases mobiles, le débit, la tubulure de la chromatographie et l’âge de la colonne analytique. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Figure 3 a contient une liste de tableau d’exemples pour les échantillons traités, y compris les calibrateurs 0 - 7 (« Kalibrator 0 » - « Kalibrator 7 »), contrôles de qualité et le sérum du patient, qui est indiqué par le nombre d’injection (#) ; l’exemple de texte d’identification (Exemple de texte) ; la concentration mesurée en mg/L (conc.) ; le type de l’échantillon qui est un blanc, norme, contrôle de la qualité ou échantillon de patients (Type) ; la concentration nominale des calibrateurs en mg/L (Conc STD.) ; le temps de rétention analytique (RT) ; la réponse qui est le rapport entre la surface du pic de la zone de l’analyte/pic IS (réponse) ; la déviation de la valeur de la concentration nominale (% Dev) ; la position du flacon (flacon) ; et le temps d’acquisition (Acq.Time). Le paramètre de clé utilisé pour la quantification est la réponse, augmentant graduellement avec la concentration de l’analyte, en raison de la quantité constante de l’étalon interne ajouté isotopes marqués.
Figure 3 b montre la courbe d’étalonnage. Dans la régression, le coefficient de détermination r2 devrait être > 0,995. Le modèle de calibration suivant est utilisé pour tous les analytes décrits dans cette méthode : courbe type = linéaire ; origine = inclus ; pondération = 1 / x ; transformation de l’axe = none. Dans l’exemple donné, la courbe d’étalonnage et les contrôles de qualité remplissent tous les critères de qualité : r2 > 0,995 pour la courbe d’étalonnage et de la déviation des calibrateurs (y compris le QMD) et les échantillons QC se trouve à ± 15 % de la valeur nominale.
Les transitions mesurées ion parent-à-fille (MRM) sont indiquées dans la Figure 3, montrant quatre pics dans le même temps de rétention : les deux pics supérieurs représentent deux transitions qui sont mesurées pour l’analyte d’intérêt, les deux pics inférieurs représentent la transitions de l’étalon interne correspondante isotopes marqués. Pour l’évaluation de la qualité, les sommets de l’analyte dans les fenêtres de temps de rétention respectifs sont vérifiés visuellement et manuellement réintégrés à la ligne de base, lorsque cela est nécessaire.
La concentration minimale inhibitrice (CMI) constitue l’élément central de la TDM antimicrobienne, définissant l’exposition pharmacocinétique qui est nécessaire pour atteindre une cible pharmacocinétique/pharmacodynamique (PK/KD) ratio13,17. En conséquence, les niveaux de concentration cible antibiotiques TDM sont exprimées par rapport à la CMI de l’agent pathogène causal. Étant donné que l’action des antibiotiques bêta-lactamines est fonction du temps, leur efficacité est maximisée grâce à la réalisation des concentrations thérapeutiques qui dépassent le MIC 4 x-5 x (fT > 4-5 x MIC). Face à des agents pathogènes infectieux inconnus, la fourchette de concentration de creux de libre pipéracilline (protéine-indépendant) est, par conséquent, 64 mg/L, correspondant à environ 90 mg/L de pipéracilline total18.
Le premier patient (échantillon #11) a un niveau de cuve sérique élevé satisfaisante de la pipéracilline 83,4 mg/L qui est également suffisante pour problème agents pathogènes, tels que Pseudomonas aeruginosa. Le deuxième patient (échantillon #12) a une concentration d’environ 0,2 mg/L, qui est au-dessous du calibrateur le plus bas (PPQM). Peut-être le patient a récupéré, et l’administration de pipéracilline a été abandonnée. Le résultat « < 0,5 mg / L » est, par conséquent, présenté dans le système d’information hospitalier. Le troisième malade (échantillon #13) a une concentration de creux de pipéracilline faible de seulement 5,3 mg/L, ce qui n’est pas suffisant pour la nette majorité des agents pathogènes. Pour la chimiothérapie antimicrobienne efficace, le dosage devrait être augmenté par le médecin.
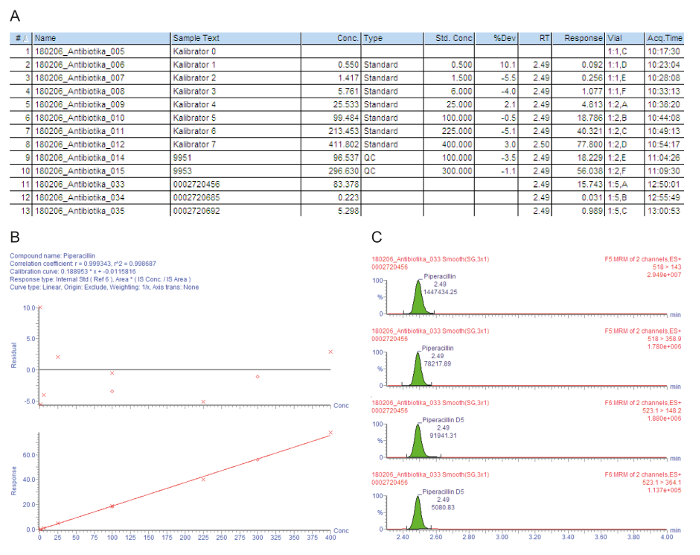
Figure 3 : évaluation de la qualité exemplaire et quantification de l’analyte pipéracilline. Ces panneaux représente l’analyse de données de spectrométrie de masse. (A), ce panneau affiche la liste de l’échantillon, y compris les calibrateurs (Standard, échantillons #1 et #8), contrôles de qualité (QC, échantillons #9 et #10) et le sérum du patient (échantillons #11 - #13). Étalon 0 fait référence à l’essai à blanc sans l’analyte, mais avec l’ajout d’un étalon interne. 9951 représente QC B, 9953 représente QC D. (B), ce panneau montre la courbe d’étalonnage de la pipéracilline. Les déviations de pourcentage de la concentration nominale calibrateur sont données dans le graphique supérieur (y-axe : résiduelle), le graphe du bas représente la gamme d’étalonnage linéaire. (C), ce panneau indique le temps de réaction multiples suivi (MRM) pour la pipéracilline et le correspondant interne standard pipéracilline-D5 pour l’échantillon de sérum de patient #12. Deux transitions de l’ion parent-à-fille sont présentent leurs temps de rétention et les intensités du signal respectif. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Supplémentaire fichier. S’il vous plaît cliquez ici pour télécharger ce fichier.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Dans ce manuscrit, les auteurs rapportent le protocole pour une méthode de basé sur la spectrométrie de masse tandem simple et robuste pour la quantification des antibiotiques fréquemment utilisés à l’ICU19, nommément céfépime, méropénem, ciprofloxacine, la moxifloxacine, linézolide, et pipéracilline14. Une feuille de calcul accompagne le manuscrit pour la préparation des solutions mères antibiotiques, calibrateurs et contrôles de qualité, compte tenu de la pureté des antibiotiques et le poids moléculaire de leurs contre-ions. Étant donné que les concentrations d’antibiotiques sont assez élevées, leur quantification ne doit être aucun défi particulier du point de vue analytique. En conséquence, nous sommes convaincus que le présent protocole est applicable aux diverses plates-formes instrumentales MS. Pour un transfert de méthode, les utilisateurs sont encouragés à quantifier le volume de la colonne extra et Hold-up de volume de leur système de chromatographie et d’adapter le gradient début temps donc16. Au cours de l’installation de la méthode, le système doit également être évalué à reporter et, si nécessaire, un échantillon témoin doit être injecté après les échantillons plus élevés de calibrateur et patient avec de fortes concentrations d’antibiotiques. Les utilisateurs doivent également envisager la possibilité de saturation détecteur qui se produit lorsque trop d’ions entrer dans un spectromètre de masse en tandem. Saturation de détecteur pertinents peut être éliminée avec plus petits volumes d’injection, une dilution d’analyte supérieure durant le nettoyage de l’échantillon, ou un désaccord d’un analyte cible (par exemple, déclassement des réglages de tension optimale).
Contrairement aux autres méthodes, la gamme d’étalonnage permet les deux une quantification des concentrations près de la CMI de pathogènes sensibles, ainsi que les concentrations maximales (cmax) qui sont obtenues avec une administration en bolus. Le plus haut Cmax-valeurs des adultes sont déclarés dans les feuillets d’information professionnelles correspondant dans la base de données de sécurité du médicament FDA comme suit : 163,9 mg/L pour le céfépime20, 112 mg/L pour le Méropénem21, 4,6 mg/L pour la ciprofloxacine22 , 4,1 mg/L pour la moxifloxacine23et 21,2 mg/L pour le linézolide24298 mg/L pour le25de la pipéracilline. Surveillance de la concentration d’antibiotique dans la circulation sanguine du patient permet un ajustement de la dose à la sensibilité des pathogènes impliqués, mais la pharmacocinétique aire sous la courbe peut également être obtenue par le biais de multiples prélèvements sanguins avec la donnée Protocole.
Plusieurs antibiotiques (en particulier le Méropénem bêta-lactame) sont chimiquement instables une fois dissous. L’étape la plus critique dans le présent protocole est, par conséquent, la préparation des solutions mères, étalons et des contrôles de la qualité en vertu des conditions de froid26,27. À cet égard, il est également essentiel de congeler les échantillons de patients aussi rapidement que possible. Bien que le stockage de sérum à-80 ° C est recommandée26, nos expériences de stabilité montrent que les échantillons peut également être conservés jusqu'à 3 jours à-20 ° C sans aucune diminution significative des concentrations d’antibiotiques (même au niveau bac).
Nous vous recommandons d’effectuer un test de pertinence de système avant chaque analyse HPLC-MS/MS des échantillons de patients (p. ex., avec calibrateur 3). En règle générale, un test de pertinence de système est utilisé pour vérifier la reproductibilité du système LC-MS/MS et voir si elle est également suffisante pour l’analyse qui sera effectuée. Ainsi, par exemple, diminution des intensités de signal sont causées par une contamination du cône de balayage de MS, qui, ensuite, exige son nettoyage avec un solvant organique. Pour nettoyer la source MS, une soupape de renvoi peut être introduite après la colonne de chromatographie, mise en scène « exempt d’analyte » des portions de la phase mobile des déchets avant qu’ils atteignent le spectromètre de masse. En revanche, une augmentation globale de la pression peut indiquer colonne colmatage au fil du temps. Pour augmenter la colonne utilisation de longévité d’un filtre precolumn rentable est recommandable. Si la pression continue à être un problème, un débit de 0,4 mL/min peut également servir avec le gradient chromatographique dans le présent protocole.
Une limitation mineure de cette technique est qu’elle nécessite trois étapes manuelles séparées pour échantillon nettoyage, ce qui entraîne un délai total d’environ 30 min. ajouter les étalons internes marqués isotopique à l’agent de précipitation peut sauver certains temps de traitement. Toutefois, cela devrait être effectué uniquement pour taux de débit d’échantillonnage élevé et où les précipitations agent entreposé au froid (par exemple, à-20 ° C), comme les normes internes aussi dégrade in vitro à des températures élevées.
Le protocole décrit a été développé pour l’échantillon de traitement dans des tubes en polypropylène standard 1,5 mL. Un débit plus élevé serait nécessaire pour TDM antibiotique, la procédure peut être améliorée pour le format de plaque multipuits avec inserts de centrifugeuse adéquate ou plaques de filtration avec une tubulure de vide.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Les auteurs n’ont rien à divulguer.
Acknowledgments
Les auteurs remercient Dr Schütze pour son aide à l’établissement de la méthode présentée et Dr. Zoller pour la précieuse contribution en ce qui concerne la gamme d’étalonnage approprié. Les auteurs remercient également le personnel technique de l’installation de spectrométrie de masse.
Materials
| Name | Company | Catalog Number | Comments |
| cefepime hydrochloride | Sigma-Aldrich | 1097636 | USP Reference Standard |
| meropenem trihydrate | Sigma-Aldrich | Y0001252 | EP Reference Standard |
| ciprofloxacin | Sigma-Aldrich | 17850 | |
| moxifloxacin hydrochloride | Sigma-Aldrich | SML1581 | |
| linezolid | Toronto Research Chemicals | L466500 | |
| piperacillin sodium salt | Sigma-Aldrich | 93129 | |
| cefepime-13C12D3 sulfate | Alsachim | C1297 | Isotope labelled internal standard for cefepime |
| meropenem-D6 | Toronto Research Chemicals | M225617 | Isotope labelled internal standard for meropenem |
| ciprofloxacin-D8 | Toronto Research Chemicals | C482501 | Isotope labelled internal standard for ciprofloxacin |
| moxifloxacin-13C1D3 hydrochloride | Toronto Research Chemicals | M745003 | Isotope labelled internal standard for moxifloxacin |
| linezolid-D3 | Toronto Research Chemicals | L466502 | Isotope labelled internal standard for linezolid |
| piperacillin-D5 | Toronto Research Chemicals | P479952 | Isotope labelled internal standard for piperacillin |
| methanol | JT Baker | 8402 | |
| HPLC grade water | JT Baker | 4218 | |
| formic acid | Biosolve | 6914132 | |
| acetic acid | Biosolve | 1070501 | |
| ammonium formate | Sigma-Aldrich | 70221-25G-F | |
| tert-Butyl methyl ether | Merck | 101845 | |
| Fortis 3 μm C8 100 * 2.1 mm | Fortis | F08-020503 | |
| Ti-PEEK-encased Prifilter (2 μm) | Chromsystems | 15011 | |
| 2795 Alliance HPLC system | Waters | 176000491 | |
| Quattro micro API Tandem Quadrupole System | Waters | 720000338 | |
| QuanLynx 4.1 software | Waters | / | Data evaluation software provided by the mass spectrometer manufacturer |
| Supplemental File | Stock Solutions |
References
- Fleischmann, C., et al. Assessment of Global Incidence and Mortality of Hospital-treated Sepsis. Current Estimates and Limitations. American Journal of Respiratory and Critical Care Medicine. 193 (3), 259-272 (2016).
- Dellinger, R. P., et al. Surviving Sepsis Campaign: international guidelines for management of severe sepsis and septic shock, 2012. Intensive Care Medicine. 39 (2), 165-228 (2013).
- Lodise, T. P., Drusano, G. L. Pharmacokinetics and pharmacodynamics: optimal antimicrobial therapy in the intensive care unit. Critical Care Clinics. 27 (1), 1-18 (2011).
- Macedo, R. S., Onita, J. H., Wille, M. P., Furtado, G. H. Pharmacokinetics and pharmacodynamics of antimicrobial drugs in intensive care unit patients. Shock. 39, Suppl 1. 24-28 (2013).
- Petersson, J., Giske, C. G., Eliasson, E. Standard dosing of piperacillin and meropenem fail to achieve adequate plasma concentrations in ICU patients. Acta Anaesthesiologica Scandinavica. 60 (10), 1425-1436 (2016).
- Abdul-Aziz, M. H., Lipman, J., Mouton, J. W., Hope, W. W., Roberts, J. A. Applying pharmacokinetic/pharmacodynamic principles in critically ill patients: optimizing efficacy and reducing resistance development. Seminars in Respiratory and Critical Care Medicine. 36 (1), 136-153 (2015).
- Imani, S., Buscher, H., Marriott, D., Gentili, S., Sandaradura, I. Too much of a good thing: a retrospective study of beta-lactam concentration-toxicity relationships. Journal of Antimicrobial Chemotherapy. 72 (10), 2891-2897 (2017).
- Ventola, C. L. The antibiotic resistance crisis: part 1: causes and threats. Pharmacy & Therapeutics. 40 (4), 277-283 (2015).
- World Health Organization. Global Action Plan on Antimicrobial Resistance. , Available from: http://www.who.int/antimicrobial-resistance/global-action-plan/en (2015).
- Pulcini, C. Antibiotic stewardship: update and perspectives. Clinical Microbiology and Infection. 23 (11), 791-792 (2017).
- Cairns, K. A., et al. The impact of a multidisciplinary antimicrobial stewardship team on the timeliness of antimicrobial therapy in patients with positive blood cultures: a randomized controlled trial. Journal of Antimicrobial Chemotherapy. 71 (11), 3276-3283 (2016).
- Baur, D., et al. Effect of antibiotic stewardship on the incidence of infection and colonisation with antibiotic-resistant bacteria and Clostridium difficile infection: a systematic review and meta-analysis. Lancet Infectious Diseases. 17 (9), 990-1001 (2017).
- Roberts, J. A., Norris, R., Paterson, D. L., Martin, J. H. Therapeutic drug monitoring of antimicrobials. British Journal of Clinical Pharmacology. 73 (1), 27-36 (2012).
- Paal, M., Zoller, M., Schuster, C., Vogeser, M., Schutze, G. Simultaneous quantification of cefepime, meropenem, ciprofloxacin, moxifloxacin, linezolid and piperacillin in human serum using an isotope-dilution HPLC-MS/MS method. Journal of Pharmaceutical and Biomedical Analysis. 152, 102-110 (2018).
- Vogeser, M., Seger, C. Pitfalls associated with the use of liquid chromatography-tandem mass spectrometry in the clinical laboratory. Clinical Chemistry. 56 (8), 1234-1244 (2010).
- United States Pharmacopeia and National Formulary. Chapter <621>. CHROMATOGRAPHY (USP 37-NF 32 S1). , United Book Press, Inc. Baltimore, MD. 6376-6385 (2014).
- Wong, G., Sime, F. B., Lipman, J., Roberts, J. A. How do we use therapeutic drug monitoring to improve outcomes from severe infections in critically ill patients? BMC Infectious Diseases. 14, 288 (2014).
- EUCAST: European Committee on Antimicrobial Susceptibility Testing. Information on clinical breakpoint tables. , Available from: http://www.eucast.org/clinical_breakpoints/ (2017).
- Surveillance der Antibiotika-anwendung und der bakteriellen Resistenzen auf Intensivstationen. SARI information for antibiotic consumption 2016 of all intensive care units participating at SARI. , Available from: http://sari.eu-burden.info/auswertung/down/AD-ZEIT.pdf (2016).
- Cefepime hydrochloride: Highlights of prescribing information. , Available from: https://www.accessdata.fda.gov/drugsatfda_docs/Label/2016/050679s040lbl.pdf (2016).
- Meropenem: Highlights of prescribing information. , Available from: https://www.accessdata.fda.gov/drugsatfda_docs/Label/2008/050706s022lbl.pdf (2006).
- Ciprofloxacin hydrochloride: Highlights of prescribing information. , Available from: https://www.accessdata.fda.gov/drugsatfda_docs/Label/2016/019537s086lbl.pdf (2016).
- Moxifloxacin hydrochloride: Highlights of prescribing information. , Available from: https://www.accessdata.fda.gov/drugsatfda_docs/Label/2010/021277s038lbl.pdf (2010).
- Linezolid: Highlights of prescribing information. , Available from: https://www.accessdata.fda.gov/drugsatfda_docs/Label/2012/021130s028lbl.pdf (2011).
- Piperacillin and Tazobactam: Highlighs of prescribing information. , Available from: https://www.accessdata.fda.gov/drugsatfda_docs/Label/2017/050684s88s89s90_050750s37s38s39lbl.pdf (2017).
- Zander, J., et al. Effects of biobanking conditions on six antibiotic substances in human serum assessed by a novel evaluation protocol. Clinical Chemistry and Laboratory. 54 (2), 265-274 (2016).
- Zander, J., et al. Quantification of piperacillin, tazobactam, cefepime, meropenem, ciprofloxacin and linezolid in serum using an isotope dilution UHPLC-MS/MS method with semi-automated sample preparation. Clinical Chemistry and Laboratory. 53 (5), 781-791 (2015).



