Summary
यहां, हम एक संयोजी दृष्टिकोण वर्तमान उच्च संकल्प microscopy, कंप्यूटेशनल उपकरण का उपयोग कर, और सी एलिगेंस भ्रूण में एकल कोशिका लेबलिंग neurodevelopment के दौरान एकल कोशिका गतिशीलता को समझने के लिए ।
Abstract
कैनोहैब्डिटिस एलिगेंस (ग. एलिगेंस) एकमात्र जीव है जिसमें एक पूरे तंत्रिका तंत्र के सेलुलर मूल को समझने की चुनौती, एकल कोशिका संकल्प के साथ, vivo में मनाया जा सकता है के रूप में बाहर खड़ा है । यहाँ, हम C. एलिगेंस भ्रूणों में न्यूरोडेवलपमेंट की जाँच के लिए एक एकीकृत प्रोटोकॉल प्रस्तुत करते हैं । हमारे प्रोटोकॉल इमेजिंग, lineaging और भ्रूण के विकास में एकल कोशिकाओं के neuroसंरचनात्मक अनुरेखण को जोड़ती है । हम प्राप्त करने के दीर्घकालिक, चार आयामी इमेजिंग C. एलिगेंस भ्रूण के साथ लगभग समदैशिक स्थानिक संकल्प के उपयोग के माध्यम से दोहरे-दृश्य औंधा चयनात्मक विमान रोशनी microscopy (dispim). सूत्रकृमि भ्रूण में नाभिक और न्यूरोनल संरचनाएं imaged और isotropically सभी तीन आयामों में ~ ३३० एनएम के संकल्प के साथ छवियों को उपज के लिए किया जाता है । इन मिनट-दर-मिनट उच्च संकल्प 4D डेटा सेट तो एक सेल और विस्तार के subcellular स्तर पर जीन अभिव्यक्ति और रूपात्मक गतिशीलता के साथ निश्चित सेल-वंशावली पहचान सहसंबंध के लिए विश्लेषण कर रहे हैं । हमारे प्रोटोकॉल वर्णित चरणों में से प्रत्येक के मॉड्यूलर कार्यांवयन सक्षम और embryogenesis, जीन अभिव्यक्ति, या neurodevelopment पर अध्ययन को बढ़ाने के लिए संरचित है ।
Introduction
C. एलिगेंस केवल जीव है जिसमें भ्रूण में हर कोशिका neurodevelopment में मनाया जा सकता है के रूप में बाहर खड़ा है । पूरे सेल-वंशावली ज्ञात और1अपरिवर्तनीय के साथ, और नए उपकरण है कि भ्रूण में एक कोशिकाओं के लेबलिंग और सतत इमेजिंग की अनुमति के विकास के साथ, जीव अब सूत्रकृमि के विकास में विभिंन चरणों की जांच शुरू कर सकते है तंत्रिका सभी कोणों से प्रणाली-सेल जंम; प्रवास और विभेदन; न्यूरोराइट गठन, लक्षित बहिर्वृद्धि और पूकुलेशन; synapse गठन; और कार्यात्मक सर्किट की ट्यूनिंग । Stably व्यक्त संवाददाताओं और प्रतिदीप्ति microscopy के संयोजन से सी एलिगेंस भ्रूण में न्यूरोनल वृद्धि गतिशीलता पर कब्जा, वैज्ञानिक समुदाय के लिए मूल्यवान है.
सी एलिगेंस में विकासात्मक अध्ययन अक्सर अपरिवर्ती कोशिका-वंशावली और सेल के भाग्य का लाभ उठाने इस प्रजाति के नक्शे को बरकरार जीव1के भीतर एकल कोशिका स्तर पर प्रासंगिक समझ बढ़ाने के लिए । ऑटो-lineaging विश्लेषण-starrynite का उपयोग2,3,4 और एसीट्री5,6,7,8 सॉफ्टवेयर-उच्च कंट्रास्ट से लाभ, उच्च संकल्प फ्लोरोसेंट नाभिक की छवियाँ. बेहतर काम करने के लिए, StarryNite/AceTree भी विकास के दौरान imaged भ्रूण की उंमीद के मुताबिक विवश उंमुखीकरण पर निर्भर करता है । Confocal माइक्रोस्कोपी, दो coverslips के बीच संकुचित सी एलिगेंस भ्रूण में किया, एक दशक से अधिक के लिए मानक ऑटो lineaging माइक्रोस्कोपी विधि गया है, क्योंकि यह दोनों उच्च विपरीत प्रदान करता है/ भ्रूण का अभिविन्यास7,8. हम पहले निर्माण और एक उपंयास प्रकाश के उपयोग का वर्णन-चादर आधारित दोहरे देखें उल्टे चयनात्मक विमान रोशनी माइक्रोस्कोप (dispim) जी नमूना इमेजिंग के लिए जैसे सी एलिगेंस भ्रूणोद्भव9,10 , 11 , 12 , 13. प्रकाश शीट microscopy, सामांय में, कम phototoxicity, उच्च गति, और जीने 3d नमूनों14,15के दीर्घकालिक इमेजिंग प्रदान करता है । Dispim विधि, विशेष रूप से, लगभग ३३० एनएम9के लगभग समदैशिक स्थानिक संकल्प के साथ चार आयामी (4d) छवियों का उत्पादन ।
संनाभि माइक्रोस्कोपी के साथ तुलना में, dispim उच्च संकेत करने वाली शोर और गति, अधिक समदैशिक स्थानिक संकल्प प्रदान करता है, और वीवो इमेजिंग16 में लंबी अवधि के लिए अधिक उपयुक्त है । इसलिए हम StarryNite में इनपुट के लिए diSPIM डेटा को अनुकूलित करने के लिए काम/ एक बड़ी बाधा यह है कि diSPIM नमूनों StarryNite के लिए उंमीद झुकाव को अपनाने के लिए eggshell संपीड़न द्वारा आसानी से विवश नहीं किया जा सकता/ मात्रा में कोशिका की स्थिति का यादृच्छिक उंमुखीकरण विश्लेषण किया जा रहा है degrades ऑटो की सटीकता-lineaging ।
हम इसलिए, एक दर्शक-निर्देशित उपयोगकर्ता इंटरफ़ेस जो उपयोगकर्ताओं को पूर्व के दौरान भ्रूण के सटीक 3 डी अभिविन्यास का चयन करने के लिए diSPIM छवियों के प्रसंस्करण, छवि डेटा है कि दोनों गुणवत्ता अनुकूलित और संदर्भ-StarryNite में इनपुट के लिए पता है उपज की अनुमति देता है । /एसीरी। Imaged भ्रूण के उपयोगकर्ता चयन पर, CytoSHOW एक स्वचालित डेटा प्रसंस्करण पाइपलाइन orchestrates । फसली और पृष्ठभूमि-घटाया भ्रूण छवियों प्रत्येक स्थिति, timepoint और देखने के लिए TIFF स्टैक फ़ाइलों के भीतर बच रहे हैं । साइटोशो फिर iteratively कार्यक्रम spimfusion को सह रजिस्टर और संयुक्त रूप से दो पूर्व संसाधित विचार deconvolve, रिचर्डसन-लुसी17,18 एल्गोरिथ्म का उपयोग करने के लिए समदैशिक उच्च संकल्प आयतनी छवियां उपज कॉल । मानक के एक dispim-विशिष्ट सेट starrynite के लिए अनुकूलित किया गया है छवि के दौरान अपने व्यवहार-विभाजन और नाभिक-isotropically संगलित त छवियों में ट्रैकिंग । Fused छवियां और lineaging परिणाम तो AceTree का उपयोग कर संपादित कर रहे हैं, जो उपयोगकर्ताओं की पहचान करने के लिए और स्वत: वंशावली StarryNite द्वारा उत्पंन ट्रेस में किसी भी त्रुटियों को ठीक करने की अनुमति देता है । AceTree भी वंश-वृक्ष और 3D मॉडलिंग की renderings के भ्रूण में ट्रैक नाभिक की वर्तमान कर सकते हैं । हम पाते हैं कि ऑटो-lineaging गति और सटीकता स्पष्ट रूप से isotropically संगलित छवियों का उपयोग कर बढ़ाया जाता है, जब या तो spim कैमरा से कच्चे छवियों की तुलना में. हमारे प्रोटोकॉल, जबकि सी. एलिगेंस आवेदन यहां वर्णित के लिए अनुकूलित, आम तौर पर ऑटो के लिए अनुकूलित किया जा सकता है dispim अंय प्रजातियों या नमूनों के लिए उत्पादित डेटा की lineaging । यदि यह प्रोटोकॉल का इच्छित उपयोग है, तो कृपया ध्यान दें कि starrynite मापदंडों के अतिरिक्त ट्यूनिंग की संभावना नए नमूनों के लिए आवश्यक होगा, के रूप में3,4वर्णित है ।
इस प्रोटोकॉल के सफल कार्यांवयन 4d-isotropic संकल्प के साथ छवियों में परिणाम और कोशिकाओं को ट्रेस करने में सक्षम बनाता है जीव, जबकि एक साथ की पहचान करने और विकासशील सी एलिगेंस भ्रूण में ंयूरॉंस का विश्लेषण । इसके अलावा, कई बाद प्रसंस्करण एल्गोरिदम विलय-हार्डवेयर त्वरण के साथ इन सबसे अधिक समय लेने जा रहा है-हम अब दोनों ठीक subcellular विवरण और सेल-lineages और सेल fates का विश्लेषण कर सकते है अनिवार्य रूप से वास्तविक समय में रहते भ्रूण की । इस नए प्रोटोकॉल की अनुमति देता है सटीक, सूचित हेरफेर और निरीक्षण विकि में भिंनता और संरचनाविकास के परिवीथी अध्ययन के दौरान सेल व्यवहार । इस पांडुलिपि में, हम उंनत प्रोटोकॉल हम सी एलिगेंस भ्रूण के विकास में lineaging और सेल ट्रैकिंग, embryogenesis, जीन अभिव्यक्ति या neurodevelopment के अध्ययन को बढ़ाने के लिए विकसित किया है की एक विस्तृत विवरण प्रस्तुत करते हैं ।
Protocol
1. पाली के साथ diSPIM स्टील इमेजिंग चैंबर कोडांतरण एल-lysine लेपित coverslip
नोट: नीचे दिए गए चरणों का अनुकूलन करने के लिए सभी आवश्यक हैं और StarryNite द्वारा सूत्रकृमि भ्रूणों के वंश विश्लेषण को स्वचालित/ C. एलिगेंस सेल लाइनज की अनुरेखण की आवश्यकता वाले प्रयोगों के लिए कई विकल्प (इस रूप में दर्शाए अनुसार) छोड़े जा सकते हैं ।
- एक साफ आयताकार coverslip के केंद्र में एक छोटा सा आयत (2mm x 5mm) ड्रा (no. १.५, 24 मिमी x ५० mm) एक sharpie (या समान कलम) के साथ ।
- Coverslip पर अचिह्नित पक्ष को फ्लिप और पाली-एल-lysine के 10 μL जगह (कैट । P1524) चिह्नित आयत से अधिक है ।
नोट: 1 मिलीग्राम/एमएल पाली-एल lysine फ़िल्टर्ड पानी (या समकक्ष) में भंग की एक काम एकाग्रता बनाओ । दीर्घकालिक भंडारण के लिए, 5-10 मिलीलीटर aliquots और स्टोर − 20 डिग्री सेल्सियस पर तैयार करते हैं । एक बार thawed, aliquots कमरे के तापमान (23 डिग्री सेल्सियस) में 3 – 4 सप्ताह के लिए संग्रहित किया जा सकता है । - पॉली-एल-lysine को 5 मिनट के लिए coverslip कोट करने के लिए अनुमति दें (चित्र 1a) । पाली-एल-lysine कांच आवरण जहां भ्रूण बढ़ जाएगा फिसल जाता है के लिए प्रयोग किया जाता है, भ्रूण खोल को मजबूती से छड़ी coverslip, यहां तक कि जब उद्देश्यों इमेजिंग माध्यम में डूबे है की अनुमति ।
- स् टील इमेजिंग चैंबर के नीचे के आधे हिस्से में पॉली-एल-लाइसिन कोटेड कोवरलिप रखें ।
- Coverslip के साथ नीचे आधे पर स्टील इमेजिंग चैंबर के शीर्ष आधा प्लेस और चार चैंबर के साथ जुड़े शिकंजा के साथ कस । यह सुनिश्चित करने के लिए पक्ष से नेत्रहीन की जांच करें कि ऊपरी आधा समान रूप से निचली छमाही में बैठा है (चित्र 1b) ।
- 7 के साथ चैंबर भरें-एम९ बफर19के 8 एमएल, एक समपरासारी बफर है जो असामांय परासरणी दबाव के सामने झुकने से प्रारंभिक चरण भ्रूण को रोकने में मदद करता है । 1-कोशिका, और यहां तक कि 2-कोशिका भ्रूण परासरण के प्रति संवेदनशील होते हैं और इस समपरासारी बफर में असामान्य रूप से विकसित नहीं हो सकते । यदि एम९ को पानी से प्रतिस्थापित किया जाए तो भ्रूण को 3-गुना अवस्था में भी गिरफ्तार किया जाता है ।
2. बढ़ते के लिए सी एलिगेंस भ्रूण के नमूनों की तैयारी
नोट: इमेजिंग करने से पहले लगभग 18 घंटे, पांच युवा (अंतिम molt के बाद से 1 दिन) वयस्क और दस लार्वा चरण 4 (L4) C. एलिगेंस को एक सूत्रकृमि वृद्धि माध्यम (ngm) आगर प्लेट में चुना जाता है, जिसे ई. कोलाई स्ट्रेन OP50 के साथ वरीयता दी जाती है । प्लेटिनम वायर उठाता पशु19नुकसान पहुंचाए बिना लार्वा और युवा वयस्क सी एलिगेंस स्थानांतरित करने के लिए उपयोग किया जाता है ।
- एक 1% मिथाइल सेलुलोस तैयार करें (कैट । H7509-25G) एम९ बफर में समाधान ।
नोट: मिथाइल सेल्युलोज गर्म एम९ में तब तक उभारा जाना चाहिए जब तक कि वह घुल न जाए । एक बार तैयार हो जाने पर इस घोल को कमरे के तापमान पर स्टोर किया जा सकता है । - एक अवतल माइक्रोस्कोप स्लाइड के अवसाद में 1% मिथाइल सेलुलोस-इन-एम९ विलयन में ५०० μL जोड़ें । इस चिपचिपा बफर दो अवसरों में इस्तेमाल किया जाएगा: 1) जब फसल की कटाई के वयस्क कीड़े के विच्छेदन से जल्दी भ्रूण और 2) जब धोने देर से चरण भ्रूणों सीधे एक NGM थाली से उठाया ।
नोट: मिथाइल सेलुलोस का प्रयोग भ्रूण को सूक्ष्मदर्शी स्लाइड से चिपके रहने से रोकने के लिए किया जाता है । - इमेजिंग देर से चरण भ्रूण के लिए, ले रखी C. एलिगेंस भ्रूण (अधिमानतः एक एनजीएम प्लेट से युवा उपस्थित वयस्कों के साथ) एक बरौनी उठाओ का उपयोग करके, और अवतल माइक्रोस्कोप स्लाइड पर 1% मिथाइल सेल्युलोज करने के लिए भ्रूण को स्थानांतरित । बरौनी उठाओ बल को कम करने में मदद करता है और इस तरह के हैंडलिंग के दौरान तनाव या भ्रूण को नुकसान को कम । प्रक्रिया एक बरौनी लेने के लिए हार्ट20द्वारा कवर किया जाता है ।
- एक दूसरा बरौनी लेने के साथ (विपरीत हाथ में), धीरे दोनों बरौनी एक साथ नल के लिए मिथाइल सेल्युलोस में भ्रूण को निलंबित ।
- वैकल्पिक: यदि StarryNite के साथ वंश भ्रूणीय कोशिकाओं की योजना बना, एक 1-सेल 4 कोशिका भ्रूण के लिए माउंट चाहिए । ऐसा करने के लिए, पहले एक NGM प्लेट से युवा वयस्कों का चयन करें और उन्हें एक प्लेटिनम वायर पिक का उपयोग करके अवतल माइक्रोस्कोप स्लाइड पर एम९-मिथाइल सेलुलोस समाधान में ले जाएं ।
- वैकल्पिक: सीरिंज सुई (no. 18g x 1 1/2) के तेज सुझावों के साथ, 1-सेल करने के लिए 4-सेल भ्रूण को रिलीज करने के लिए मध्य शरीर पर transversely पशु स्लाइस.
3. मुंह पिपेट: माइक्रोकेशिका पिपेट के साथ कोडांतरण चूषित्र ट्यूब
नोट: हम एक के साथ एक चूषित्र ट्यूब का उपयोग एक हाथ खींचा माइक्रोकेशिका पिपेट ट्यूब के रबर गैसकेट में डाला । इससे हम डिसेक्शन स्लाइड से भ्रूण का ट्रांसफर करने के लिए बफर से भरे इमेजिंग चैंबर में पॉली-एल-लाइसिन-कोटेड सतह पर जा सकते हैं ।
- विस्तारित युक्तियों के साथ दो हिस्सों को बनाने के लिए माइक्रोकेशिका पिपेट को एक खुली लौ पर मैन्युअल रूप से खींचें ।
- माइक्रोकेशिका पिपेट के एक आधे लो और चूषित्र ट्यूब के रबर गैसकेट में कुंद अंत डालने (चित्रा 1 सी) । बाद में उपयोग के लिए माइक्रोकेशिका पिपेट के अन्य आधे को अलग सेट करें (यदि आवश्यक हो) ।
- माइक्रोकेशिका पिपेट के साथ लगे इकट्ठे चूषित्र ट्यूब के साथ, धीरे microcapillary पिपेट की नोक को तोड़ने और एक खोलने के बारे में फिट होगा कि 1 – 2 दो भ्रूण (यहाँ से इस उपकरण पर एक "मुंह पिपेट" कहा जाता है).
4. बढ़ते C. एलिगेंस पाली पर भ्रूण-L-lysine लेपित coverslip
- चूषित्र मुखपत्र के साथ दांतों के बीच धीरे से आयोजित, पूर्व-एम९ बफर के 10 – 15 μl के साथ माइक्रोकेशिका पिपेट को भरें और फिर धीरे से अवतल स्लाइड से केशिका में कई भ्रूणों को चूसें ।
- इस्पात इमेजिंग के लिए भ्रूण स्थानांतरण एम९ बफर से भरा चैंबर, केशिका टिप स्थिति है कि भ्रूण coverslip के केंद्रीय आयत में गिर जाएगी ।
- भ्रूण के लिए चोट से बचने, धीरे से उन्हें एक बरौनी लेने के साथ ले जाएँ या भ्रूण की स्थिति के लिए मुंह पिपेट का उपयोग करें, भ्रूण को ओरिएंट करने के लिए ताकि भ्रूण की लंबी अक्ष coverslip के लंबे अक्ष के लंबवत है (चित्र 1b इनसेट , नीचे पैनल) ।
नोट: इस उंमुखीकरण में भ्रूण स्थिति छवि को स्लाइस की संख्या कम, जिससे प्रकाश की खुराक और डेटा प्रसंस्करण समय को कम करने, जबकि अधिग्रहण की गति में सुधार । - माइक्रोस्कोप स्टेज पर नमूना धारक में स्टील इमेजिंग चैंबर प्लेस (चित्रा 1D).
5. विधानसभा, सॉफ्टवेयर सेटअप और भ्रूण इमेजिंग के लिए लेजर अनुकूलन diSPIM का उपयोग
- कैसे कुमार एट अल10,11 में और http://www.dispim.org पर व्यावसायिक रूप से उपलब्ध भागों से पूरे फाइबर युग्मित dispim इकट्ठा करने के लिए पर कदम दर कदम निर्देश देखें । कैसे diSPIM को इकट्ठा करने के लिए एक वीडियो-प्रोटोकॉल भी एएसआई की वेबसाइट (http://www.asiimaging.com) पर उपलब्ध है ।
नोट: इस प्रोटोकॉल के लिए उपकरण सेटअप कुमार एट अल10,11 dispim, जो इमेजिंग के लिए 40x 0.8 एनए पानी विसर्जन लेंस का उपयोग करता है के समान है । इस प्रोटोकॉल और कुमार एट अल.10,11 में सेटअप के बीच अंतर केवल एक dichroic दर्पण के अलावा (५६० एनएम पर तेज) और एक इमेजिंग बंटवारे डिवाइस (मॉडल A12801-01) पर स्थापित लाल और हरे रंग की bandpass फिल्टर के अंदर है दोनों diSPIM इमेजिंग शस्त्र । छवि बंटवारे प्रकाशिकी के अलावा दो अलग fluorophores से छवियों का एक साथ कब्जा-५६१ एनएम और ४८८ एनएम पराबैंगनीकिरण से उत्तेजित-एक ही कैमरा चिप के दो हिस्सों पर उत्सर्जन बैंड अलग से अनुमति देता है । - उपकरण विधानसभा के बाद, इमेजिंग से पहले diSPIM के ऑप्टिकल संरेखण की जांच करें ।
नोट: उचित रूप से diSPIM देखें https://youtu.be/qnOrg30NNuE संरेखित करने के लिए, और हार्डवेयर जानकारी, http://dispim.org/hardware/objectives और http://www.asiimaging.com के लिए । - ओपन-सोर्स प्लेटफ़ॉर्म माइक्रो-मैनेजर (https://micro-manager.org/)21का उपयोग करें, जिसे उच्च-थ्रूपुट सेलुलर इमेजिंग22के लिए लाइट शीट माइक्रोस्कोप के संचालन के लिए ऑप्टिमाइज़ किया गया है । हम बहु के लिए एएसआई diSPIM प्लगइन का उपयोग की स्थिति अधिग्रहण है, जो23वर्णित के रूप में अप करने के लिए 30 भ्रूण की एक साथ इमेजिंग की अनुमति देता है की सिफारिश ।
- माइक्रो प्रबंधक के साथ खुला, ४८८ एनएम और ~ ७९ μW (०.२५) के लिए ~ १७९ μW (०.५) के लिए लेजर तीव्रता सेट ५६१ एनएम (चित्रा 2a, लाल आयत) के लिए ।
नोट: ये 1-मिनट के अंतराल का उपयोग कर C. एलिगेंस भ्रूणों की दीर्घकालिक इमेजिंग के लिए अनुशंसित सेटिंग्स हैं । लंबी अवधि के दौरान दोहरे रंग इमेजिंग, ५६१ एनएम लेजर छवि नाभिक (Mcherry:: Histone) के लिए प्रयोग किया जाता है जब तक भ्रूण सेम मंच पर हैं, जो बिंदु पर ४८८ एनएम लेजर तो छवि को भी चालू करने के लिए gfp लेबल ंयूरॉंस । इन इमेजिंग शर्तों को कम करने के लिए अनुकूलित कर रहे है phototoxicity और जीवित रहने और भ्रूण के अंडे सेने को सुनिश्चित करते हुए लंबे समय तक (12-14 घंटे) neurodevelopmental और lineaging डेटा के निरंतर अधिग्रहण को सक्षम करने से । - माइक्रो प्रबंधक में, एएसआई diSPIM खिड़की (चित्रा 2b) खोलने के लिए > डिवाइस नियंत्रण > प्राप्ति टैब चुनें । इस टैब की डेटा बचत सेटिंग्स अनुभाग (हरा आयत), वॉल्यूम सेटिंग्स अनुभाग (नीला वर्ग), और स्लाइस सेटिंग्स अनुभाग (नारंगी वर्ग), सुनिश्चित करें कि प्रत्येक पैरामीटर के रूप में सेट है चित्र 2b में दिखाया गया है ।
नोट: हमारी छवि विश्लेषण सॉफ्टवेयर CytoSHOW अंय वैकल्पिक आउटपुट डेटा प्रारूपों के साथ काम करने के लिए अनुकूलित है जैसे कि थोक-concatenated OME-TIFF फ़ाइल श्रृंखला और TIFF-स्टैक फ़ाइल श्रृंखला के रूप में एक निर्यात माइक्रो प्रबंधक में निर्मित समारोह के उपयोग के माध्यम से अधिग्रहण बनाया । सामांयतया, एकल-समय-बिंदु स्टैक मिलान OME-TIFF फ़ाइल डेटा स्वरूप उपयोग किया जाता है क्योंकि यह रीयल-टाइम को देखने और छवि वॉल्यूम के संसाधन के रूप में जैसे ही raw डेटा प्राप्त कर रहे है की अनुमति देता है ।
6. अनुकूलित autofocus पैरामीटर के लिए लंबी अवधि के इमेजिंग सी एलिगेंस भ्रूणों
- लंबे समय के लिए अनुकूलित सेटिंग्स के लिए माइक्रो प्रबंधक autofocus मापदंडों सेट-वंश गुणवत्ता dispim- सी एलिगेंस भ्रूण की इमेजिंग. एएसआई diSPIM विंडो में, ऑटोफोकस टैब क्लिक करें (चित्र 2c) । में सामांय Autofocus विकल्प अनुभाग (काला वर्ग) निर्दिष्ट पैरामीटर ठीक के रूप में दिखाया गया है । ध्यान दें कि Autofocus चैनल (लाल वर्ग) lineaging प्रयोगों में अपने परमाणु चैनल प्रतिदीप्ति चैनल निर्दिष्ट करना चाहिए ।
नोट: यदि अधिकतम ऑफसेट 5 μm से अधिक है, छवियों को ध्यान से बाहर बहाव करते हैं । - क्लिक करें, Plugins ≫ अधिग्रहण उपकरण > पैटर्न ओवरले।
- प्रतिमान ओवरले विंडो में, ग्रिड दिखाएंक्लिक करें ।
- एएसआई diSPIM विंडो में, नेविगेशन टैब क्लिक करें ।
- बीम और पथ A या B के पत्रक के लिए चेक बक्से क्लिक करें फिर लाइवक्लिक करें । छवि अधिग्रहण शुरू होता है । एक लाइव दृश्य विंडो खोलता है । ६.१ से चयनित चैनल पर भ्रूण के चारों ओर एक बॉक्स ड्राइंग द्वारा भ्रूण के ऑटो फोकस विश्लेषण क्षेत्र का चयन करें ।
नोट: हम आमतौर पर इमेजिंग सत्र प्रति 10 भ्रूणों के लिए ४२० समय अंक पर कब्जा । प्रति छवि सत्र कच्चे डेटा आमतौर पर 1.7 टीबी हैं, जबकि deconvolved और StarryNite-प्रसंस्कृत डेटा कर रहे हैं 1.4 टीबी (चरण 9 और 10 देखें). हम छवि अधिग्रहण के लिए बड़ी क्षमता HDD (हमारे वर्तमान प्रणाली पर 18TB), और छवि भंडारण के लिए बादल प्लेटफार्मों का उपयोग करने की सलाह देते हैं । - "प्राप्ति टैब में दीर्घकालिक बहु-आयामी छवि कैप्चर शुरू करने के लिए अधिग्रहण प्रारंभ करें (चित्र 2b) पर क्लिक करे ।
7. खोलने कच्चे माइक्रो-CytoSHOW में प्रबंधक छवियां
- Http://dispimlineage.wormguides.org से सॉफ्टवेयर बंडल डाउनलोड करें ।
नोट: सॉफ्टवेयर बंडल एक. zip फ़ाइल के रूप में डाउनलोड किया जाएगा और करने के लिए निकाले जाने की आवश्यकता होगी "C:\" उपयोग करने से पहले निर्देशिका । संस्थापना के लिए और ब्यौरे http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm पर दिए गए हैं । - सायकशो चल रहा है प्रारंभ करने के लिए फ़ाइल C:\cytoshowextrascontact \
- मेनू फ़ाइल > नई > Dispim मॉनिटर (माइक्रो प्रबंधक)चुनें । जहाँ प्राप्ति timepoint फ़ोल्डर सहेजे गए रूट डेटा सेट फ़ोल्डर की स्थिति जानें । किसी भी timepoint फ़ोल्डर का चयन करें और क्लिक करेंखोलें । बहुआयामी नेविगेशन विंडोज़ ( Dispim मॉनिटर विंडोज़ कहा जाता है) दोनों spima और SPIMB (चित्रा 3a) के लिए स्वचालित रूप से खोल रहे हैं ।
नोट: इन windows रूट डेटा फ़ोल्डर के लिए नए सहेजे गए raw टाइमपॉइंट स्टैक (इवेंट जो एक नमूना अभी भी रिकॉर्ड किया जा रहा है में) की निगरानी करेगा । प्रत्येक नए timepoint के अधिग्रहण के बाद, अलग spim हथियार और नमूना पदों की निगरानी खिड़कियों में से प्रत्येक प्रत्येक भ्रूण के लिए पूरे मल्टीचैनल 4d डेटासेट प्रदर्शित करने के लिए ताज़ा होगा ।
8. साइसिशो के साथ अधिकतम प्रक्षेपण छवियों सृजन
यहां तक कि deconvolution से पहले, कच्चे डेटा जल्दी से नमूना है वैश्विक सुविधाओं के मूल्यांकन के लिए संसाधित किया जा सकता है.
- छवि खिड़की के बाईं ओर पैनल पर Z-MIP बटन पर क्लिक करें (चित्रा 3a, लाल आयत) पूर्ण गहराई और किसी दिए गए स्थान या spim बांह की पूर्ण समय पाठ्यक्रम के माध्यम से अधिकतम तीव्रता अनुमानों बनाने के लिए । एक जेड प्रक्षेपण hyperstack खिड़की दिखाई देगा ।
- Z-प्रोजेक्शन हाइपरस्टैक विंडो में, अधिकतम तीव्रताके लिए प्रोजेक्शन प्रकार सेट करें । निर्दिष्ट करें कि उपयोगकर्ता वरीयता के आधार पर कौन-से चैनल, स्लाइस और समय-बिंदु फ़्रेम संसाधित किए जाएं ।
- पूर्ण होने पर ठीक क्लिक करें ।
- फ़ाइल संवाद विंडो से अधिकतम तीव्रता outputs को बचाने के लिए फ़ोल्डर स्थान का चयन करें, और उसके बाद ठीकक्लिककरें । कुछ समय की अनुमति दें (15 – 20 मिनट, डेटासेट और कंप्यूटर की प्रोसेसिंग शक्ति के आकार के आधार पर) प्रक्षेपण छवियों को उत्पन्न करने के लिए CytoSHOW के लिए.
9. समदैशिक उच्च-रिज़ॉल्यूशन आयतनी डेटा में कक्ष प्रजातियों का विश्लेषण करना
- वैकल्पिक: के साथ कच्चे डेटा CytoSHOW में Dispim मॉनिटर के माध्यम से खुला, बहुभुज चयन उपकरण का चयन करें (चित्रा 3a, काले तीर) और बस के बाहर भ्रूण के पूर्वकाल, पश्च, पृष्ठीय और अधर किनारों पर क्लिक करें (है कि सटीक क्रम में) भ्रूण पर एक "bowtie" पैटर्न उत्पन्न करते हैं । दोनों दृश्यों (SPIM-A और SPIM-B, चित्रा 3a) के लिए करते हैं ।
नोट: इस चयन में भ्रूण केंद्रित है और भ्रूण के अग्रवर्ती अक्ष रिकॉर्ड करता है, जिसमें ब्याज की दीर्घवृत्तीय क्षेत्र (आरओआई) को दर्शाता है । Bowtie पैटर्न cues cytoshow कि उपयोगकर्ता आगे एक उंमुखीकरण है कि starrynite द्वारा विश्लेषण lineaging के लिए इष्टतम है में अंतिम isotropically रूप से संगलित त संस्करणों की एक सटीक रोटेशन निर्दिष्ट करने की योजना/ मामलों में जहां StarryNite lineaging प्रयोगात्मक योजना का हिस्सा नहीं है, अंय चयन उपकरण और आकार छवि प्रसंस्करण के लिए लागत पर लाभ सेट करने के लिए चुना जा सकता है । - यदि एकाधिक भ्रूण एक साथ बहु-स्थिति अधिग्रहण विकल्प का उपयोग करके imaged थे, खुले और सभी भ्रूण के लिए ९.१ कदम प्रदर्शन । यह एक सत्र में सभी भ्रूण के लिए भविष्य के कदम के समानांतर निष्पादन की अनुमति देगा । आप प्रक्रिया करने के लिए इच्छा नहीं है किसी भी भ्रूण के लिए SIMA और SPIMB खिड़कियों को बंद करें ।
- DiSPIM मॉनिटर विंडो के बाईं ओर पैनल पर dispim बटन क्लिक करें (चित्र 3a, पीले रंग में हाइलाइट किया गया) । यह विशिष्ट नियंत्रण के एक उप पैनल से पता चलता है diSPIM प्रसंस्करण ।
- प्रत्येक SPIM बांह के लिए हरे और लाल चैनलों संरेखित करें । क्योंकि उत्सर्जन चैनल splitters के लिए एक ही कैमरे पर एक साथ अलग लाल और हरे रंग की छवियों को पकड़ने के लिए उपयोग किया जाता है, यह नेत्रहीन इन दो शारीरिक रूप से सटे छवि क्षेत्रों के सटीक पिक्सेल पंजीकरण संरेखित करें जब वे मढ़ा रहे है महत्वपूर्ण है । एक ही संरेखण समायोजन का पुनः प्रयोग आमतौर पर कई लगातार इमेजिंग सत्र पर संभव है, लेकिन (के रूप में कदम 9.4.1-9.4.5) की जांच की जानी चाहिए ।
- SPIMA पैनल के साथ शुरू, बाईं ओर (चित्रा 3a, नारंगी तीर, बाएँ पैनल) मुख्यमंत्री स्क्रॉल बार ले जाकर लाल चैनल का चयन करें.
- X-, y-, और z-समायोजक (चित्र 3a, नारंगी वर्ग) का उपयोग करके, लाल चैनल को हरे रंग से मिलाने के लिए खिसकाएं ।
- Dispim बटन (चित्र 3a, पीले रंग में हाइलाइट किया गया) पर क्लिक करें, सबपैनल को बंद करने और अन्य सभी स्थिति विंडो के लिए एक ही पाली के प्रचार को ट्रिगर करने के लिए ।
- "Z" स्क्रॉल पट्टी (चित्र 3a, नीला तीर, बाएँ फलक) और/या "t" स्क्रॉल पट्टी (चित्र 3a, हरा तीर, बाएँ फलक) ले जाकर अन्य फ़्रेम और समय बिंदुओं पर प्रचारित कि सही संरेखण की पुष्टि करें । यदि बहु स्थिति अधिग्रहण प्रदर्शन किया गया था और कई भ्रूण imaged (कदम ५.३) थे, संरेखण भी उन भ्रूण को प्रचारित करना चाहिए था । एक्स-, वाई-, और जेड-समायोजक (चित्रा3 ए, ऑरेंज स्क्वायर, जो सभी भ्रूणों के spima पैनल के लिए एक ही होना चाहिए) के लिए संख्या की जांच भी द्वारा पुष्टि करें ।
- SPIMB diSPIM मॉनिटर विंडो के लिए चरण 9.4.1 – 9.4.4 दोहराएँ (चित्र 3a, दाएँ फलक).
- क्लिक करें "Dispim" बटन और फिर "फ्यूज" बटन (चित्रा 3a, नीला आयत) नामक एक संवाद बॉक्स खोलने के लिए "Deconvolve/फ्यूज dispim रॉ डेटा वॉल्यूम्स" (चित्रा 3b). पैरामीटर्स को चित्र 3bमें दर्शाए अनुसार सेट करें । इन पैरामीटर्स संक्षेप में निंन substeps में संबोधित कर रहे हैं:
- चैनल पर कुंजी पंजीकरण सेट 1 (४८८ एनएम लेजर) या 2 (५६१ एनएम लेजर) । Denser या अधिक सर्वव्यापी संकेत के साथ चैनल का चयन करें । Lineaging प्रयोगों के लिए हमेशा एक सर्वव्यापी परमाणु हिस्टोन प्रतिदीप्ति छवि के लिए इस्तेमाल किया चैनल का चयन करें.
- इनपुट वॉल्यूंस सापेक्ष ओरिएंटेशन को + 1 या-1 पर सेट करें । सही अभिविन्यास सूचकांक एक विशिष्ट diSPIM कैमरा प्लेसमेंट पर निर्भर करता है (चित्रा 4).
नोट: यदि अनिश्चित, दोनों SPIM एक और बी Dispim मॉनिटर विंडोसे एक ही timepoint duplicating द्वारा प्रत्येक विकल्प का परीक्षण, 9.1-9.12 और मनमाने ढंग से एक इनपुट मात्रा के परीक्षण के सापेक्ष उंमुखीकरण का चयन । गलत झुकाव कलाकृतियों के साथ blurry छवियों निकलेगा, जबकि सही झुकाव स्पष्ट छवियों निकलेगा । स्पष्ट छवि पैदावार है कि इनपुट मात्रा सापेक्ष अभिविंयास मूल्य तो दिया dispim उपकरण से भविष्य के सभी डेटा के लिए reused किया जा सकता है । - चुनें कि क्या संगलित खंड एक या बी इनपुट मात्रा के रूप में एक ही उंमुख होना चाहिए (उपयोगकर्ता वरीयता के आधार पर) ।
- "हर खंड के लिए ताजा पंजीकरण" का चयन करें । यह विकल्प नियंत्रण कैसे SpimFusion प्रत्येक timepoint पर प्रत्येक खंड युग्म के लिए पंजीकरण matrices गणना करता है । "ताजा" विकल्प एल्गोरिथ्म प्रत्येक timepoint पर पंजीकरण स्वीकार्य अनुकूलित करने के लिए अनुमति देता है ।
- 10 करने के लिए deconvolution पुनरूक्तियां की संख्या सेट करें । यह संख्या एक समय-कुशल तरीके से वांछित उच्च संकल्प को मज़बूती से प्राप्त करने की आदत है ।
- वैकल्पिक: यदि ऑटो-lineaging वांछित है (अत्यधिक अनुशंसित), की जांच करें स्वतः शुरू StarryNite Fused त संस्करणों की lineaging। यह विकल्प StarryNite स्वचालित रूप से खंड के लिए प्रक्षेपण और imaged संस्करणों में SpimFusion द्वारा उत्पादित कोशिकाओं को ट्रैक करेगा ।
- वैकल्पिक: स्वचालित lineaging में उच्चतम सटीकता के लिए, यह विहित "adl" (एकnterior [x-पश्चिम], Dorsal [y-उत्तर], छोड़ दिया [z-निकट]) अभिविन्यास में isotropically रूप से संगलित त भ्रूण की मात्रा का स्थान लेने के लिए सबसे अच्छा है. चुनें इस विकल्प को इंगित करने के लिए पूर्वावलोकन विकल्प में वॉल्यूम आउटपुट ओरिएंटेशन निर्धारित करें । साइटोशो isotropically संगलित त संस्करणों की एक प्रारंभिक जोड़ी प्रसंस्करण द्वारा जवाब देगा, उपयोगकर्ता बारीकी से निरीक्षण करने के लिए और adl पंजीकरण प्राप्त करने के लिए आवश्यक rotations निर्दिष्ट करने की अनुमति ।
- क्लिक करें, हांएक बार सभी पैरामीटर्स चयनित हैं ।
- आउटपुट निर्देशिका में संसाधित फ़ाइलों को सहेजने के लिए निर्दिष्ट करें । क्लिक करें, ok।
- वैकल्पिक: यदि पूर्वावलोकन में वॉल्यूम आउटपुट ओरिएंटेशन परिभाषित करें विकल्प चयनित किया गया था, तो spm-A विंडो में उस प्रारंभिक टाइमपॉइंट पर t स्क्रॉल पट्टी (चित्र 3a, हरा तीर, बायां पैनल) सेट करें, जिस पर ABa और ABp कक्ष मेटाफ़स पर पहुंच गए हैं । T स्क्रॉल पट्टी को Spm-B विंडो में विकास के बाद के अल्पविराम चरण के लिए सेट करें । यह ADL ओरिएंटेशन निर्दिष्ट करने में सहायता करेगा ।
- वैकल्पिक: तैयार होने पर ठीक क्लिक करें. ऊपर 9.5.7 में पूर्वावलोकन विकल्प चयनित किया गया था, तो केवल दो पूर्वावलोकन वॉल्यूम isotropically रूप से SPM-A और SPIM-B छवि windows के t-स्लाइडर्स द्वारा इंगित टाइमपॉइंट के लिए किया जाएगा । इन दो पूर्वावलोकन timepoints ADL उंमुखीकरण के लिए उत्पादन भ्रूण की मात्रा के सटीक फिर से संगठित करना निर्दिष्ट करने के लिए इस्तेमाल किया जा सकता है, के रूप में नीचे समझाया ।
- नई प्रदर्शित 3Dprojy_ Decon-Fuse_ की स्थिति जानें... । खिड़की. इस पूर्वावलोकन विंडो के समय बिंदु 2 के लिए t स्क्रॉल पट्टी ले जाएं । Z स्लाइडर को तब तक खिसकाएं जब तक कि सीधे भ्रूण के लंबे अक्ष को दिखाई न दिया जाए.
- T स्क्रॉल पट्टी को वापस करने के लिए समय बिंदु 1 3Dprojy_ decon-fuse_... । खिड़की. लाइन चयन टूल चुनें और ईएमएस सेल (अधर-सबसे गोल नाभिक) से एबी-सेल मेटाहासे प्लेटों के तल के माध्यम से एक लाइन चयन ड्रा करें ।
- 3Dprojy_ Decon-Fuse_ पर नारंगी diSPIM पूर्वावलोकन बटन क्लिक करें... । खिड़की. प्रीरीजोलुशन imaged वॉल्यूम के ओरिएंटेशन के लिए ठीक समायोजन पूर्ण dataset संसाधित करने में उपयोग के लिए सहेजा जाएगा ।
- वैकल्पिक: Deconvolve/फ्यूज diSPIM रॉ डेटा वॉल्यूम संवाद, बस के रूप में ऊपर ९.५ कदम में फिर से प्रकट होगा । क्लिक करें, हांचुनने के बिना का चयन करेंवॉल्यूम आउटपुट ओरिएंटेशन में पूर्वावलोकनपूर्वावलोकन विकल्प । पूर्ण डेटा संसाधन चलाने के लिए आउटपुट फ़ोल्डर निर्दिष्ट करें ।
- प्रारंभ टाइमपॉइंट (spima) और प्रक्रिया करने के लिए छवियों की पूर्ण अवधि के टाइमपॉइंट (SPIMB) को समाप्त करने के लिए dispim मॉनिटर windows के t स्क्रॉल पट्टियां (चित्र 3a, हरा तीर, बायां फलक) सेट करें । उसके बाद ठीकक्लिककरें ।
- के रूप में spimfusion की प्रगति, cytoshow खुलता है और एक बहुआयामी खिड़की प्रत्येक भ्रूण के लिए कटा हुआ-4d समदैशिक जुड़े मात्रा दिखा तरोताजा कर देती है, साथ ही साथ दो खिड़कियों घूर्णन-4d अधिकतम-समदैशिक मात्रा की तीव्रता अनुमानों के साथ । इस समय के दौरान, किसी भी साइटोशो विंडो को बाधित या बंद नहीं जब तक समदैशिक संलयन और वंश ट्रैकिंग पूरा कर रहे हैं ।
- वैकल्पिक: ध्यान दें कि एक बार StarryNite छप स्क्रीन दिखाई दिया है और बाद में गायब हो गया, पूरा डेटा प्रसंस्करण पाइपलाइन पूरा कर लिया गया है । इस विंडो को संसाधित करने के दौरान बंद नहीं किया जाना चाहिए या StarryNite बाधित हो जाएगा ।
10. एसिट्री में StarryNite वंशावली ट्रेस श्रृंखला खोलने (वैकल्पिक)
- "AceTree_16BitCompat. jar" प्रदान की अनुकूलित संस्करण खोलें ।
- मेनू फ़ाइल > खोलें फ़ाइल कॉन्फ़िगर करेंचुनें । अपने आउटपुट निर्देशिका को पहले इंगित करने के लिए cytoshow की स्थिति जानें । भ्रूण के लिए Decon_fuse_.. . _ Pos [n] सबफ़ोल्डर [n] खोलें । Aaa_edited. xml का चयन करें और खोलें।
- संपादन ट्रैक को खोलने के लिए एसिट्री मेनू संपादित करें > संपादन उपकरण का उपयोग करना और कक्षों की विंडोज़ को समायोजित करना या हटाना ।
- लाल और हरे रंग की तीव्रता समायोजित करने के लिए आधा छायांकित वृत्त चित्र 5B, लाल वर्ग पर क्लिक करें ।
- वंश दृश्य और संपादन के साथ आगे बढ़ें के रूप में पहले वर्णित5,6,8 (पांडुलिपियों भी हमारे डाउनलोड बंडल में शामिल हैं) ।
Representative Results
हम पहली बार diSPIM अधिग्रहण (वर्गों 1-6) के लिए प्रोटोकॉल के मापदंडों का उपयोग imaged भ्रूण की व्यवहार्यता की पुष्टि की । दस भ्रूण एक साथ 20 डिग्री सेल्सियस पर imaged थे, 2-कोशिका चरण से 2-गुना चरण (७.५ घंटे, ४५१ खंड/भ्रूण) एक मात्रा/ Embryogenesis के दौरान सेल डिवीजनों की निगरानी करने के लिए, हम तनाव BV514, जो हर जगह Mcherryव्यक्त::histone संवाददाता एकीकृत ट्रांसजीन सरणी ujIs11324से constructs इस्तेमाल किया । चित्रा 6 भ्रूण के विकास के इस पहली छमाही के एक imaged भ्रूण के लिए एक समय रेखा से पता चलता है । प्रत्येक छवि imaged भ्रूण के एक एकल दृश्य अधिकतम तीव्रता प्रक्षेपण (7-8 कदम से उत्पादित) का प्रतिनिधित्व करता है । हमने पाया है कि अनुकूलित प्रोटोकॉल भ्रूण के लिए किसी भी detectable phototoxicity प्रेरित नहीं किया, के रूप में सेल डिवीजनों के समय द्वारा मूल्यांकन (नहीं दिखाया गया), अंडे सेने के समय, और समय विकास के मील के पत्थर से संबंधित (चित्रा 6 और संदर्भ1 , 25 , 26) ।
हम तो प्रोटोकॉल लागू करने के लिए भ्रूण के विकास में एकल ंयूरॉंस के बहिर्विकास गतिशीलता का विश्लेषण । हम DCR7692 (olaex4655), एक ट्रांसजेनिक सूत्रकृमि तनाव है कि (DACR2819, pflp-19 (3.6 kb):: Syn21:: gfp:: caax::p 10 3 ' utr) अज्ञात कोशिकाओं का एक सबसेट neuropeptide flp-19 प्रमोटर से . प्रोटोकॉल के चरणों का पालन यहां उल्लिखित, हम निर्धारित किया है कि अज्ञात कोशिकाओं को मोटर ंयूरॉंस RMDDL और RMDDR के अनुरूप, उत्सर्जन नहर सेल के लिए, और दो मांसपेशी कोशिकाओं (चित्रा 7) । हम तो जांच की और rmddl और rmddr ंयूरॉंस के बहिर्वृद्धि गतिशीलता मात्रा निर्धारित । हमने देखा है कि rmddl और rmddr ंयूरॉंस obliquely के रूप में जल्दी के रूप में आकार के रूप में ३६० मिनट के बाद निषेचन, अब सेलुलर धुरी neurite परिणाम के लिए बाद में धुरी का प्रतिनिधित्व करने के साथ (चित्रा 7 और फिल्म S1) । फिजी में "सरल neurite अनुरेखण प्लगइन" का उपयोग और यह isotropically रूप से संगलित संस्करणों के 3 डी पुनर्निर्माण के लिए आवेदन, हम तो rmddl और rmddr न्यूराइटीज के छह भ्रूण के लिए stereotypic आउटग्रोथ मात्रा निर्धारित. हमने निर्धारित किया कि विकास की गतिशीलता RMDDL और भ्रूणों भर RMDDR के लिए टकसाली थे (इस के साथ साथ Rmddr कहा जाता है) । 385-410 मिनट के बाद निषेचन से, RMDDs neurites विस्तारित 6.0 ± 0.5 μm (मतलब ± SEM; द = 12 न्यूलाइट्स) कोशिका पिंड के अग्र (चित्र 7b, ब्, इ) । 415-445 मिनट के बाद निषेचन, दोनों न्यूराइटीज में और प्रकल्पित तंत्रिका अंगूठी ( चित्रा 7Dमें तारांकन) के आसपास का विस्तार । औसत पर, प्रत्येक RMDD neurite विस्तारित ११.० ± ०.६ μm (मतलब ± SEM; द = 12 तंत्रिकाशोथ) कोशिका के शरीर से पूर्व में अपने contralateral समकक्ष की अंगूठी के शीर्ष पर मिलने से पहले (चित्र 7i) । महत्वपूर्ण बात, हमारे प्रतिनिधि परिणाम है कि हम की जांच करने में सक्षम है प्रदर्शन, तुलना और एक पहचान कोशिकाओं के लिए न्यूरोनल विकास सुविधाओं का आकलन हमारे एकीकृत प्रोटोकॉल का उपयोग करके (चित्रा 7 और 8 अंक) ।
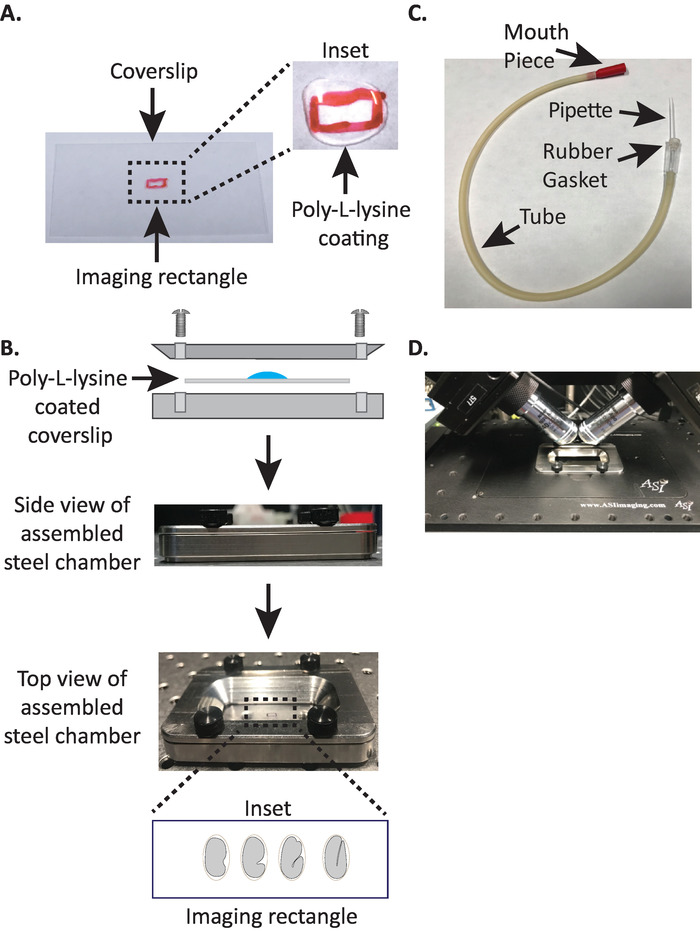
चित्रा 1: नमूना बढ़ते सेटअप diSPIM. (क) पॉली-एल-लाइसिन के साथ कोवरलिप तैयार करना । इनसेट में 10 μL पॉली-एल lysine 5 मिनट के लिए कोवरलिप का लेप किया जाता था । पाली-एल-lysine भ्रूण खोल आयत में coverslip के लिए दृढ़ता से छड़ी की अनुमति देता है । (ख) स्टील इमेजिंग चैंबर और इकट्ठे चैंबर की योजनाबद्ध । इनसेट में प्रतिनिधि भ्रूणों का मुख अग्र-पश्च अक्ष के लंबवत होता है जो सहवर्ध पर दीर्घ अक्ष के लम्बवत् होता है । (ग) माइक्रोकेशिका पिपेट के साथ इकट्ठे चूपनेवाला ट्यूब (घ) स्टील इमेजिंग चैम्बर को डिआईएम 40x उद्देश्यों के अंतर्गत नमूना धारक में रखा गया है । इस आंकड़े का बड़ा संस्करण देखने के लिए कृपया यहां क्लिक करें ।

चित्रा 2: माइक्रो प्रबंधक में लंबी अवधि के diSPIM इमेजिंग सेटअप. (क) फोटोविषाक्तता को कम करते हुए लंबे समय तक प्रतिबिंबन के लिए ऑप्टिमाइज़ किए गए dispim लेज़र पावर पैरामीटर्स (लाल आयत) की सिफारिश की है (जैसा कि सी. एलिगेंस भ्रूणों की उच्च अंडे की दर द्वारा आकलित किया गया है) । ५६१ एनएम लेजर सेट करने के लिए ७९ μW (०.२५) और ४८८ एनएम लेजर के लिए १७९ μW (०.५) । ध्यान दें कि लेजर शक्ति को सॉफ्टवेयर सेटिंग्स के सटीक अंशांकन diSPIM प्रतिष्ठानों के बीच बदलता है । यह सिफारिश की है कि उपयोगकर्ताओं को मापने के लिए और ७९ μW (५६१ एनएम) और १७९ μW (४८८ एनएम) लेजर शक्ति को प्राप्त करने के लिए लेजर शक्ति जांचना । (ख) डेटा की बचत (हरा आयत), वॉल्यूम सेटिंग्स (नीला वर्ग), और स्लाइस सेटिंग्स (नारंगी वर्ग) के लिए dispim पैरामीटर । (ग) सी एलिगेंस भ्रूणोत्पत्ति के दीर्घकालिक इमेजिंग के लिए डिआईएम ऑटोफोकस पैरामीटर (चरण 6-1-6.6 देखें) । इस आंकड़े का बड़ा संस्करण देखने के लिए कृपया यहां क्लिक करें ।

चित्रा 3: CytoSHOW का उपयोग कर छवि दृश्य और डेटा प्रसंस्करण सेटअप । (क) कोशिकाशो द्वारा खोली गई कच्ची dispim छवियां । CytoSHOW दोनों कैमरा पथ (SPIM ए और बी) द्वारा कब्जा कर लिया छवियों को खोलने में सक्षम है । इन कच्चे छवियों Dispim मॉनिटरबुलाया बहुआयामी खिड़कियों में खोला जाता है । Dispim मॉनिटरमें, एक "bowtie पैटर्न" भ्रूण के पूर्वकाल, पश्च, पृष्ठीय और अधर किनारों का चयन करने के लिए उत्पंन होता है (९.१ कदम देखें) । धनुष टाई चयन deconvolution और StarryNite-असिस्टेड lineaging अनुरेखण के लिए भ्रूण अभिविन्यास का संकेत मिलता है. (ख) आइसोट्रोपिक छवियों को उत्पन्न करने के लिए इस्तेमाल किया अनुकूलित मापदंडों. विंडो प्राप्त करते समय Deconvolved में, चरण 9.5.1-9.5.8 में निर्दिष्ट पैरामीटर्स सेट करें । इस आंकड़े का बड़ा संस्करण देखने के लिए कृपया यहां क्लिक करें ।

चित्रा 4: diSPIM कैमरा विंयास । (क) डिआईएम कैमरा प्लेसमेंट और झुकाव की तस्वीर । (ख) Spim A के + ९०-डिग्री घुमाव का चित्रण एकत्र किए गए स्पाइम बी छवियों के मिलान के लिए किया गया । (ग) अभिविंयास सूचकांक के सापेक्ष इनपुट मात्रा + 1 हमारे dispim कैमरा विंयास के आधार पर (देखें कदम 9.5.2) । हम spim B छवि (s) से मेल करने के लिए पंजीकरण से पहले Y-अक्ष के आस-पास SPM (s) + ९० डिग्री घुमाएं । स्केल पट्टियां = 10 μm. छवियां प्रतिनिधिक एकल-दृश्य, अधिकतम तीव्रता के अनुमानों और deconvolution छवियों को लेबल वाले नाभिक (५६१-nm, लाल) और न्यूरॉन्स (४८८-एनएम, ग्रीन) के साथ १.५-fold भ्रूण के हैं । इस आंकड़े का बड़ा संस्करण देखने के लिए कृपया यहां क्लिक करें ।

चित्रा 5: एसिट्री में curation और संपादन सी एलिगेंस भ्रूणीय वंश . (क) हम starrynite वंश निशान को संपादित करने के लिए एसिट्री का उपयोग करें (संदर्भ5,6,8देखें; पांडुलिपियों भी हमारे डाउनलोड बंडल में शामिल हैं) । एसिट्री चरण 10.1-10.2 के पूरा होने पर प्रत्येक नाभिक (हरे आयत) के लिए व्यवस्थित नाम C. एलिगेंस प्रदर्शित करेगा । इस विंडो (क) वंशावली ट्रेस (ABa, नीले रंग में प्रकाश डाला) में प्रत्येक कोशिका के बारे में जानकारी प्रदान करता है (काला आयत) है कि मदद गाइड उपयोगकर्ताओं को जब ट्रैकिंग और वंशावली निशान संपादन । यह अनुशंसित है कि उपयोगकर्ताओं को सत्यापित करने और lineaged कोशिकाओं और उनकी स्थिति की तुलना C. एलिगेंस भ्रूणीय कोशिका वंशावली पहले sulston एट अल.1 द्वारा रिपोर्ट इसके अलावा, यदि उपयोगकर्ताओं में विशिष्ट कोशिकाओं का पता लगाने में रुचि रखते है डेटा श्रृंखला deconvolved (नीचे देखें, B) खोज बार (नारंगी आयत) में C. एलिगेंस व्यवस्थित नाम दर्ज करें. (ख) प्रयोक्ता की विमुक्त डेटा श्रृंखलाएं भी चरण 10.1-10.2 के पूर्ण होने पर स्वत: ही खुलती हैं । यहाँ दिखाया गया है कि नाभिक के साथ एक चार कोशिका चरण भ्रूण की एक isotropically रूप से प्रयुक्त छवि लाल रंग में लेबल. एक नाभिक की ट्रैकिंग के दौरान, उपयोगकर्ताओं को छवि की तीव्रता बदल (लाल वर्ग) और समय और जेड के माध्यम से अपने कुंजीपटल पर तीर कुंजियों का उपयोग कर नेविगेट करना चाहिए (समय-बाएं/ (ग) कुछ कार्यक्षमताओं (बैंगनी आयत) के साथ (ख) में timepoint के 3 डी कार्टून कि rotatable 3 डी दृश्य सक्षम बनाता है । Acetree और इसकी 3 डी कार्यक्षमता के एक सिंहावलोकन के लिए, देखें संदर्भ5,6,8. इस आंकड़े का बड़ा संस्करण देखने के लिए कृपया यहां क्लिक करें ।

चित्रा 6: dispim पर सी एलिगेंस भ्रूण की विकासात्मक गतिशीलता समय पर. शीर्ष पैनल, imaged भ्रूण (तनाव BV514 ujIs11324) में से एक के लिए भ्रूणीय विकास की पहली छमाही दिखा छवियां dispim । भ्रूण लगातार, ७.५ घंटे (20 डिग्री सेल्सियस) के लिए हर मिनट imaged थे । शीर्ष पैनल के पहले दो चित्र 4-और 8-नाभिक के साथ कोशिका भ्रूण (लाल) और ध्रुवीय निकायों की स्थिति (घने लाल क्षेत्रों, नीले तारांकन के साथ चिह्नित) का प्रतिनिधित्व करते हैं । प्रत्येक छवि imaged भ्रूण की एक एकल दृश्य अधिकतम तीव्रता प्रक्षेपण का प्रतिनिधित्व करता है । स्केल पट्टियां = 10 μm । समयरेखा (क्षैतिज पट्टी) C. एलिगेंस भ्रूणों के विकास के मिनट्स पोस्ट फर्टिलाइजेशन (m.p.f.) का प्रतिनिधित्व करती है । हम मान्य है कि हमारे प्रोटोकॉल diSPIM अधिग्रहण के लिए मापदंडों व्यवहार्यता द्वारा मूल्यांकन के रूप में imaged भ्रूण के लिए किसी भी detectable phototoxicity प्रेरित नहीं किया, सेल डिवीजनों के समय, अंडे सेने के समय और विकास के मील के पत्थर के समय (संदर्भ देखें 1 । , 25 , 26). हम ध्यान दें कि विकास के मील के पत्थर के समय हमारे इमेजिंग मापदंडों के साथ भ्रूण भर reproducible था (SEM ± ८.१७४ मिनट के लिए ६.४ घंटे के लंबे इमेजिंग सत्रों; द = 10) । इस आंकड़े का बड़ा संस्करण देखने के लिए कृपया यहां क्लिक करें ।

चित्रा 7 । सेलुलर पहचान और एकल कोशिका के विकास में neurite विकास गतिशीलता के चरित्र चित्रण सी एलिगेंस भ्रूण । BV514 ujIs11324 (lineaging के लिए) और DCR7692 (olaex4655) को पार करके किए गए तनाव के दोहरे रंग इमेजिंग, एक ट्रांसजेनिक सूत्रकृमि तनाव है कि अज्ञात कोशिकाओं का एक सबसेट neuropeptide flp-19 प्रमोटर बंद gfp व्यक्त करता है । (A-H) प्रोटोकॉल के चरणों का पालन यहां उल्लिखित, हम निर्धारित किया है कि अज्ञात कोशिकाओं को मोटर ंयूरॉंस RMDDL और RMDDR (पीले तीर) के अनुरूप, उत्सर्जन नहर सेल (नीला तीर), और दो मांसपेशी कोशिकाओं (सफेद तीर) । (I) फिजी प्लगइन "सिंपल न्यूरोराइट ट्रेसिंग" का उपयोग करके आरडीडीएल और आरएमडीआर न्यूरॉन्स की बहिर्वृद्धि गतिशीलता का परिमाणन और इसे isotropically रूप से इस्तेमाल की गई मात्रा के 3d पुनर्निर्माण में लागू करना । नोट कैसे दोनों rmddl और rmddr शो stereotypic विकास गतिशीलता, प्रत्येक के लिए असिंक्रोनस रूप से विस्तार की कुल लंबाई 11.0 ± 0.6 μm (मतलब ± SEM; n = 12 न्यूलाइट्स) और तंत्रिका अंगूठी के पृष्ठीय शीर्ष पर बैठक (भी फिल्म S1देखें) । इस आंकड़े का बड़ा संस्करण देखने के लिए कृपया यहां क्लिक करें ।

चित्र 8: ग-एलिगेंस भ्रूणों में न्यूरोनल रूपावली के समदैशिक डिस्म प्रतिबिंबों की जांच । AVHL और AVHL ंयूरॉंस के समदैशिक दृश्य (पीले तीर) । Dispim का उपयोग करना, न्यूरोनल morphologies लगभग ३३० एनएम के समदैशिक स्थानिक संकल्प के साथ चार आयामी (4d) छवियों को उपज पर कब्जा कर लिया जा सकता है । DiSPIM लगभग सभी दिशा में समान संकल्प के साथ छवि संस्करणों को घुमाएगी करने के लिए उपयोगकर्ताओं की अनुमति देता है । एक-डी में छवियां एक ही isotropically भ्रूण के लंबे अक्ष के आसपास अलग घुमाव से dispim छवि मात्रा के अधिकतम तीव्रता के अनुमानों हैं । स्केल पट्टियां = 5 μm । इस आंकड़े का बड़ा संस्करण देखने के लिए कृपया यहां क्लिक करें ।
अनुपूरक मूवी S1: C. एलिगेंस भ्रूण २८० से ४३४ मिनट के बाद निषेचन का विकास । तनाव के समदैशिक चलचित्र DCR7692 (olaex4655) DACR2819 के साथ सर्वव्यापी रूप से ujIs113 व्यक्त करना RMDD न्यूराइटीज (चित्रा 7a-D, पीले तीर) लेबलिंग । DACR2819 भ्रूण के विकास के दौरान दो मांसपेशी कोशिकाओं (चित्र 7a-d, सफ़ेद तीर) और उत्सर्जन नलिका कोशिका (चित्र 7A-d, नीला तीर) को भी लेबल कर रहा है (चित्र 7a-d) । स्केल पट्टियां = 10 μm. कृपया इस फ़ाइल को डाउनलोड करने के लिए यहां क्लिक करें ।
Discussion
C. एलिगेंस केवल जीव के रूप में अंतिम स्थिति और प्रत्येक वयस्क न्यूरॉन ज्ञात27की कनेक्टिविटी के साथ बाहर खड़ा है. हालांकि, विकास कार्य सर्किट और नेटवर्क है कि ऊपर सी एलिगेंस बनाता है के संगठन के लिए अग्रणी गतिशीलता अनजान रहते हैं । प्रकाश माइक्रोस्कोपी में अग्रिमों से उभरते अवसरों के आधार पर, अब हम पर कब्जा कर सकते हैं और कोशिका की स्थिति, संरचनाउत्पत्ति, और सी एलिगेंस भ्रूणीय विकास भर में न्यूरोजेनेसिस का विश्लेषण ।
प्रक्रिया है कि हम का वर्णन किया है और है कि हम नियमित रूप से प्रयोगशाला में 4, C. एलिगेंस भ्रूण में सेल-lineaging के लिए लेबल ंयूरॉंस और नाभिक के 4 डी-isotropic छवियों पैदावार का उपयोग करें । इससे भी महत्वपूर्ण बात, हम उच्च संकल्प छवियों के साथ dispim और युग्मित अर्द्ध स्वचालित lineaging क्षमताओं के साथ लंबे समय तक इमेजिंग शर्तों अनुकूलित किया है गति और सी एलिगेंस भ्रूणोत्पत्ति का विश्लेषण करने की परिशुद्धता में सुधार होगा । इस एकीकृत प्रोटोकॉल से उपयोगकर्ताओं को कोशिकाओं की पहचान करने और उन्हें पहचानने में मदद मिलेगी और जल्दी फड़कने की शुरुआत के माध्यम से न्यूरोराइट माइग्रेशन और एक्सॉन पूकुलेशन जैसी तीन आयामी विशेषताओं को मापा जा सकेगा । इस प्रक्रिया को आसानी से एक एएसआई diSPIM प्रणाली के साथ किसी भी सुविधा में अनुकूलित किया जा सकता है, और हम इस प्रोटोकॉल के लिए विशेष रूप से इस प्रणाली की सिफारिश । व्यावसायिक रूप से की पेशकश की अन्य SPIM योगों नमूना चैंबर और ऑप्टिकल संपत्तियों में एएसआई विन्यास से अलग हो सकता है. हालांकि, अन्य प्लेटफार्मों से निर्यात किए गए डेटा को भी हमारी डेटा पाइपलाइन के माध्यम से रखा जा सकता है । इसलिए, lineaging, छवि गुणवत्ता और उपकरण स्थिरता की मांग की परीक्षा में उनके मूल्य का मूल्यांकन, व्यवहार्य है । हालांकि हम सक्रिय रूप से diSPIM का उपयोग करने के लिए नियमित रूप से छवि अंय नमूनों (जैसे droवेलु और zebrafish भ्रूण के रूप में), वर्णित और भ्रूण के व्यापक lineaging विश्लेषण अभी भी वर्तमान में सूत्रकृमि प्रजातियों तक ही सीमित है । बड़े या मोटे नमूनों के लिए, हम स्टेज स्कैनिंग दृष्टिकोणों का उपयोग करना चुनते हैं, जो एक स्थिर प्रकाश पत्रक के माध्यम से नमूनों को स्कैन करते हैं । कुमार एट अल. पहले इस सुधार dispim परिच्छेदन को मोटी नमूनों से उच्च dispim10के लिए अतिरिक्त संशोधनों के बिना गुणवत्ता छवियों उपज का प्रदर्शन किया है ।
प्रोटोकॉल के भीतर महत्वपूर्ण कदम पाली-एल-lysine लेपित coverslip पर बढ़ते सी एलिगेंस भ्रूण, डेटा अधिग्रहण, और डेटा प्रोसेसिंग शामिल हैं । कटाई और बढ़ते C. एलिगेंस कांच पर भ्रूणों coverslip अनुभवहीन उपयोगकर्ताओं के लिए चुनौतीपूर्ण हो सकता है, लेकिन यहाँ हम सीखने की सुविधा के लिए महत्वपूर्ण चरणों का एक विस्तृत प्रोटोकॉल प्रदान करते हैं. यदि लंबी अवधि के इमेजिंग वांछित है, हम सबसे अच्छा परिणाम संचयन चार सेल या पहले भ्रूण से 8-10 युवा वयस्कों28प्राप्त करते हैं । ध्यान दें कि पुराने वयस्कों कम प्रारंभिक चरण भ्रूण के लिए वांछनीय है क्योंकि वे गर्भाशय और अनिषेचित अंडे में बड़े भ्रूण होते हैं । बढ़ते भ्रूण के संबंध में, ऐसे इकट्ठे चूषित्र में रुकावट के रूप में समस्याओं (मुंह पिपेट) या एक भी माइक्रोकेशिका पिपेट में एक खोलने के बड़े उचित बढ़ते और भ्रूण के उंमुखीकरण को रोकने सकता है । इष्टतम इमेजिंग के लिए तैयार करने के लिए, हम प्रकाश चादरें, कैमरा, उद्देश्यों, और autofocus के प्रदर्शन की जांच करने के लिए पूर्व और देर से पहले-twitching भ्रूण पर पूर्व अधिग्रहण परीक्षण प्रदर्शन करते हैं । हम सबसे अच्छा परिणाम प्राप्त जब इन आपरेशनों के सभी परीक्षण कर रहे है और हमारे पूर्व अधिग्रहण परीक्षण के दौरान उच्च गुणवत्ता छवियों उपज । यह विशेष रूप से समदैशिक स्थानिक संकल्प के साथ छवियों को उत्पन्न करने के लिए प्रासंगिक है, जिसके लिए दोनों दृश्यों (उद्देश्यों) से प्राप्त कच्चे चित्र उच्च गुणवत्ता के होने चाहिए । अधिग्रहण के बाद, प्रत्येक दृश्य के लिए अधिग्रहीत संस्करणों समदैशिक छवियों उपज के लिए संसाधित कर रहे हैं । यह इस प्रोटोकॉल में वर्णित के रूप में एक उपयुक्त ग्राफ़िक्स प्रोसेसिंग यूनिट (GPU) कार्ड का उपयोग करने के लिए महत्वपूर्ण है (नीचे देखें) । यह डेटा विश्लेषण करने के लिए समय छोटा, जिस पर isotropically रूप से संगलित छवियाँ जनरेट किया गया है प्रसंस्करण गति में सुधार करता है । यह भी आवश्यक है कि उपयोगकर्ताओं को CytoSHOW के नवीनतम संस्करण चला रहे है और StarryNite ऑटो के लिए हमारे डाउनलोड बंडल के साथ प्रदान की मापदंडों का उपयोग कर रहे है lineaging । यदि उपयोगकर्ताओं को अंय नमूनों के लिए ऑटो lineaging का उपयोग करने में रुचि रखते है (जैसे, zebrafish, droसोफ़िला आदि) तो starrynite में इस्तेमाल किया मानकों को अतिरिक्त अनुकूलन (संदर्भ3,4देखें) की आवश्यकता होगी ।
हालांकि हमारे एकीकृत प्रोटोकॉल पूर्व-हिल भ्रूण में छवियों और lineaging परिणाम प्रदान करता है, उपयोगकर्ताओं को पता होना चाहिए कि पोस्ट-हिल भ्रूण में स्वचालित lineaging वर्तमान में संभव नहीं है: परमाणु पदों में सेकंड के क्रम पर परिवर्तन बाद-हिल भ्रूण, बहुत तेजी से वंश ट्रैकिंग की अनुमति के लिए । तथापि, dispim वास्तव में एक आशाजनक को neurodevelopmental घटनाओं पर कब्जा करने की क्षमता का प्रदर्शन किया है और बाद में कुछ सेल पदों को ट्रैक भ्रूणोद्भव के चरणों twitching23,29। यदि उपयोगकर्ताओं के बाद की जांच में रुचि रखते है-हिल भ्रूण, dispim गति प्रदान करने के लिए आयतनी स्नैपशॉट प्राप्त करने और ऐसे neurite outgrowth के रूप में ठीक neurodevelopmental घटनाओं, ट्रैक तेजी से बढ़ भ्रूण में ।
इस प्रोटोकॉल wormguides एटलस30के सेल द्वारा सेल पूरा करने के लिए मूलभूत होगा, क्योंकि यह उच्च संकल्प समदैशिक छवियों के साथ एक एकीकृत दृष्टिकोण प्रदान करने के लिए की पहचान करने और लेबल ंयूरॉंस के दौरान 3 डी morphologies पर कब्जा होगा embryogenesis के पहले ४३० मिनट । के रूप में यह खड़ा है, प्रोटोटाइप WormGUIDES एटलस विकासशील भ्रूण में कोशिकाओं के परमाणु पदों प्रदान करता है और भ्रूण ंयूरॉंस का एक सबसेट के विकास की गतिशीलता पर कब्जा करना है । इस प्रोटोकॉल WormGUIDES एटलस30में अतिरिक्त विकासशील ंयूरॉंस के एकीकरण के लिए एक महत्वपूर्ण होगा ।
हमारे इंटीग्रेटेड प्रोटोकॉल के तहत सी एलिगेंस भ्रूण में नए जीन एक्सप्रेशन प्रोफाइल को तलाशना भी आसान होगा । ट्रांसजेनिक सी. एलिगेंसमें, बहुत से कोशिका-विशिष्ट प्रमोटरों ने ट्रांजीन अभिव्यक्ति को बहुत अधिक और सामयिक रूप से नियंत्रित किया है । जबकि अधिकांश जीन के अभिव्यक्ति पैटर्न बड़े पैमाने पर किया गया है वयस्क पशु में विशेषता31,३२,३३,३४, लगभग सभी अभी तक विकासशील में विशेषता है (विशेष रूप से स्वर्गीय चरण) भ्रूण । C. एलिगेंस प्रोमोटेरोम कृमि समुदाय के लिए एक उपयोगी संसाधन है जो कोशिका-विशिष्ट ट्रांसजीन अभिव्यक्ति को संचालित करने के साथ-साथ यह भी निर्धारित करेगा कि जीन फलन कोशिका-स्वायत्त या गैर-स्वायत्त है या नहीं । जीनों के आइसोट्रोपिक उच्च संकल्प और गतिशील अभिव्यक्ति पैटर्न पर कब्जा है, और ठीक lineaging के माध्यम से व्यक्त कोशिकाओं की पहचान वैज्ञानिक समुदाय में कई के लिए मूल्यवान हो जाएगा.
भ्रूणोत्पत्ति में दो अंतनमित प्रमुख प्रक्रियाएं, कोशिकीय विभेदन और ऊतक संरचनाजनन शामिल हैं । एक महान सौदा तंत्र और अणुओं है कि सी एलिगेंसके विकास के दौरान विशिष्ट कोशिका प्रकार परिभाषित के बारे में जाना जाता है । हालांकि, थोड़ा कोशिका प्रवास के लिए महत्वपूर्ण तंत्र के बारे में जाना जाता है, सेल आसंजन, और सी एलिगेंस भ्रूण में कोशिका आकार । उदाहरण के लिए, अक्षतंतु पूलिका, synaptogenesis, और न्यूरोनल गतिविधि: C. एलिगेंस अपरिवर्ती कोशिका वंश ज्ञात के साथ, हमारे प्रोटोकॉल हमें आसानी से भ्रूण के सूचीबद्ध 3 डी-माइक्रोएनाटॉमी को पहचानने की सुविधा देता है । आर्डिएल एट अल. पहले सी एलिगेंस भ्रूण23में एक एकल ंयूरॉंस के स्तर पर कैल्शियम यात्रियों पर कब्जा करने के लिए dispim की शक्ति का प्रदर्शन किया है । कई विकास शरीर विज्ञान के अंय पहलुओं इन तरीकों से जांच के लिए परिपक्व हैं ।
अंत में, इस प्रोटोकॉल मोटे तौर पर स्वचालित है और व्यवस्थित समय यह deconvolution छवियों को उत्पन्न करने के लिए लेता है और StarryNite और एसिट्री के माध्यम से सेल-lineaging प्रदर्शन कम कर देता है. इस प्रोटोकॉल में प्रयुक्त सॉफ्टवेयर रणनीतियों बहुत विशिष्ट क्षेत्रों जिसमें हम उंहें यहां प्रदर्शन किया है से दूर जीव विज्ञान के कई सवालों के लिए लागू किया जा सकता है ।
सॉफ्टवेयर संगतता और डाउनलोड का उपयोग पर विवरण
लघु प्रबंधक और diSPIM इमेजिंग के लिए plugins पर जानकारी http://dispim.org/software/micro-manager और https://micro-manager.org/wiki/ASIdiSPIM_Plugin पर उपलब्ध हैं ।
डेटा प्रोसेसिंग पाइपलाइन वर्तमान में एक Windows ऑपरेटिंग सिस्टम की आवश्यकता है । हम सभी आवश्यक डेटा प्रसंस्करण कार्यक्रमों और समर्थन फ़ाइलों की स्थापना को आसान बनाने के लिए एक एकल संग्रह फ़ाइल बंडल है । यह http://dispimlineage.wormguides.org पर डाउनलोड के लिए उपलब्ध है ।
CytoSHOW (http://run.cytoshow.org/) व्यापक रूप से इस्तेमाल किया और खुला स्रोत छवि विश्लेषण मंच, ImageJ (v1) पर आधारित है । जावा स्थापित किया जाना चाहिए और कंप्यूटर पर करने के लिए तारीख CytoSHOW का उपयोग करने के लिए, और CytoSHOW करने के लिए अद्यतन स्वचालित रूप से जावा वेब प्रारंभ के माध्यम से लागू कर रहे हैं । CytoSHOW के कई ImageJ-आधारित कार्यों के रूप में वर्णित है और https://imagej.nih.gov/ij/docs/examples/index.html में सचित्र हैं । CytoSHOW को एएसआई diSPIM, साथ ही अंय इमेजिंग सॉफ्टवेयर है कि झगड़ा उत्पादन बनाता से बहुआयामी कच्चे डेटा प्रदर्शित अनुकूलित किया गया है । सिद्धांत रूप में, अन्य बहु-दृश्य SPIM इमेजिंग सिस्टम इस प्रोटोकॉल अलग माइक्रोस्कोप सिस्टम पर किया जा करने के लिए अनुमति देने के लिए साइटोशो के मामूली संशोधनों के द्वारा समर्थित किया जा सकता है.
SpimFusion में लिखा गया था CUDA/C++ का उपयोग कर Visual Studio २०१३ CUDA टूलकिट v 7.5 के साथ । SpimFusion चल रहा है विशिष्ट कंप्यूटर हार्डवेयर की आवश्यकता है: एक NVIDIA ग्राफिक्स प्रोसेसिंग यूनिट (GPU) कार्ड CUDA १.० या उच्च क्षमता की गणना के साथ और 2 GB ग्राफ़िक्स कार्ड स्मृति की एक ंयूनतम । हमारे प्रोटोकॉल के प्रकाशन के समय, SpimFusion अप्रकाशित (ंयूनतम Guo और हरि श्रॉफ) है, लेकिन ऊपर उल्लेख सॉफ्टवेयर बंडल संग्रह में उपलब्ध है ।
एक विशेष रूप से निर्मित StarryNite के कमांड लाइन चालित संस्करण की आवश्यकता है कि आज़ादी से उपलब्ध MATLAB संकलक रनटाइम स्थापित है, लेकिन वाणिज्यिक MATLAB सॉफ्टवेयर के लिए एक लाइसेंस की आवश्यकता नहीं है । MATLAB संकलक रनटाइम सॉफ्टवेयर बंडल ऊपर उल्लेख संग्रह में शामिल है । इस प्रोटोकॉल में इस्तेमाल के रूप में starrynite के लिए कोड अनिवार्य रूप से संनाभि छवियों6के लिए इस्तेमाल किया है कि से अपरिवर्तित है । हालांकि, StarryNite प्रसंस्करण के लिए इनपुट छवियों के निर्माण में कई परिचालन मामलों और StarryNite परिणामों की हैंडलिंग यहां साइटोशो में तरीकों से संबोधित किया गया है कि एक सतत डेटा प्रोसेसिंग पाइप लाइन के लिए इस्तेमाल किया आइसोट्रोपिक diSPIM वॉल्यूम. इन परिवर्तनों को साइटोशो कोड द्वारा स्वचालित कर रहे है जो इन पूर्व और पोस्ट-प्रोसेसिंग चरणों को संभालता है । कोशिका प्रदर्शन भी एक पूर्व अनुकूलित diSPIM-विशिष्ट टेम्पलेट StarryNite पैरामीटर सेट स्वचालित रूप से imaged डेटा में नाभिक की प्रतिदीप्ति तीव्रता के लिए विभाजन एल्गोरिथ्म ट्यून करने के लिए । अद्वितीय प्रत्येक diSPIM डेटा सेट पर StarryNite द्वारा इस्तेमाल किया पैरामीटर तो उत्पादन छवि और lineaging डेटा के साथ एक फ़ाइल में सहेज कर रहे हैं ।
एसिट्री का एक कस्टम संस्करण है कि 16 बिट छवियों के साथ काम करता है और Java3D प्रतिपादन के साथ संगतता बनाए रखता है सबसे अच्छा इस प्रोटोकॉल के लिए अनुकूल है. यह भी ऊपर उल्लेख सॉफ्टवेयर बंडल संग्रह में शामिल है ।
Disclosures
लेखकों के पास खुलासा करने के लिए कुछ नहीं है ।
Acknowledgments
हम एकीकृत तनाव, ujIs113 के लिए जॉन मरे धंयवाद, lineaging तनाव BV514 उत्पंन; ब्रेंडन हार्वे (NIBIB) प्रोटोकॉल के परीक्षण के साथ मदद के लिए; जॉन डेनियल और गैरी Rondeau (एप्लाइड वैज्ञानिक इंस्ट्रूमेंटेशन) माइक्रो प्रबंधक और diSPIM उपकरण के साथ सहायता के लिए; और एंड्रयू ंयूयार्क और हांक ईडन diSPIM प्रणाली पर उनके महत्वपूर्ण प्रतिक्रिया के लिए । हम भी अल्पसंख्यक संस्थानों कार्यक्रम के लिए अनुसंधान केंद्र और Instituto de Neurobiología जोस डेल Castillo धंयवाद (Universidad de Puerto रीको) एक बैठक और मंच बुद्धिशीलता प्रदान करने के लिए । इस काम के अधिकांश Whitman कार्यक्रम के माध्यम से वुड्स होल में समुद्री जैविक प्रयोगशाला में आयोजित किया गया । यह काम NIH राष्ट्रीय जैव चिकित्सा इमेजिंग और Bioengineering संस्थान के Intramural अनुसंधान कार्यक्रमों और NIH अनुदान नहीं द्वारा समर्थित किया गया । U01-HD075602 और नहीं । R24-OD016474 । मार्क डब्ल्यू Moyle F32 द्वारा समर्थित-NS098616 और Leighton एच Duncan R24-OD016474 के लिए एक विविधता के पूरक द्वारा समर्थित किया गया था ।
Materials
| Name | Company | Catalog Number | Comments |
| Steps 1-4 | |||
| Concavity slides | ThermoFisher Scientific | 1519006 | 5-18mm diameter, 0.6-0.8mm deep, 1.2-1.5mm |
| Dissecting microscope with 10×–50× zoom range | Motic | SMZ-171 | |
| E. coli (OP50) | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Glass coverslips, no. 1.5, 24 × 50 mm | VWR International | 48393-241 | |
| M9 Buffer | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Methyl cellulose | Sigma-Aldrich | H7509-25G | |
| Microcapillary pipette aspirator tube | Sigma-Aldrich | A5177 | |
| Microcapillary pipettes, 0.4-mm i.d | Drummond Scientific | 1-000-800 | |
| Needle, no. 18G x 1 ½ (1.2mm x 40mm) | BD Precision Glide | 305196 | |
| NGM plates | prepared as described by Brenner (1974) | ||
| O-ring for imaging chamber | O-Rings West | M1.5X40 | |
| Pasteur pipette | Corning/Sigma-Aldrich | CLS7095D5X | |
| Platinum wire, 0.5-mm diameter | Sigma-Aldrich | 267201 | |
| Poly-L-lysine | Sigma-Aldrich | P1524 | |
| Stainless steel rectangular chamber (76.0 mm x 50.5 mm) | Applied Scientifics Instrumentations (ASI) | I2450 | |
| Worm Eyelash Pick | Hart, A. C. Behavior. WormBook. (2006). | ||
| Worm Pick | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Name | Company | Catalog Number | Comments |
| Steps 5-6 | |||
| 488 nm long-pass filter | Semrock | LP02-488 RU-2 | |
| 561-nm notch filter | Semrock | NF03-561E-25 | |
| BLP02-561R-25, quantity 2 | Semrock | 561 nm EdgeBasic best-value long-pass edge filter | |
| Control software for bottom camera | Jenoptik | ProgRes CapturePro | |
| diSIPM assembly video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/TAgbr6IrTqw ; http://www.asiimaging.com | |
| diSPIM alignment video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/qnOrg30NNuE | |
| diSPIM imaging PC | Intel | Intel Xeon CPU E5-2630 2.6GHz, 12 cores in total, 64 GB memory, Windows 7 | |
| FF01-525/45-25, quantity 2 | Semrock | 525/45 nm BrightLine single-band bandpass filter | |
| FF555-DI03-25X36, quantity 2 | Semrock | 555 nm edge BrightLine single-edge dichroic beamsplitter | |
| Imaging PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Kumar et al diSPIM Setup | Applied Scientifics Instrumentations (ASI) | Instrument setup for this protocol is identical to Kumar et al 10,11 diSPIM, which makes use of 40x 0.8NA water immersion lenses for imaging. (See steps 5.1 and note) | |
| Micro Manager | Micro-Manager | https://micro-manager.org/ | |
| Modifications to Kumar et al diSPIM Setup (see below) | |||
| Optical table with isolators, 4 feet × 6 feet × 12 inches | TMC | 784-651-02DR and 14-416-34 | |
| Name | Company | Catalog Number | Comments |
| Steps 7-10 | |||
| Analysis PC | Intel | Intel Core i7-8700K CPU 3.70GHz, 6 cores in total, 64 GB memory, Windows 10 | |
| Analysis PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Installation instructions | Software bundle | http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm | |
| Software bundle | Software bundle | http://dispimlineage.wormguides.org |
References
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology. 100 (1), 64-119 (1983).
- Bao, Z., et al. Automated cell lineage tracing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 103 (8), 2707-2712 (2006).
- Santella, A., Du, Z., Bao, Z. A semi-local neighborhood-based framework for probabilistic cell lineage tracing. BMC Bioinformatics. 15, 217 (2014).
- Santella, A., Du, Z., Nowotschin, S., Hadjantonakis, A. K., Bao, Z. A hybrid blob-slice model for accurate and efficient detection of fluorescence labeled nuclei in 3D. BMC Bioinformatics. 11, 580 (2010).
- Boyle, T. J., Bao, Z., Murray, J. I., Araya, C. L., Waterston, R. H. AceTree: a tool for visual analysis of Caenorhabditis elegans embryogenesis. BMC Bioinformatics. 7, 275 (2006).
- Katzman, B., Tang, D., Santella, A., Bao, Z. AceTree: a major update and case study in the long term maintenance of open-source scientific software. BMC Bioinformatics. 19 (1), 121 (2018).
- Murray, J. I., et al. Automated analysis of embryonic gene expression with cellular resolution in C. elegans. Nature Methods. 5 (8), 703-709 (2008).
- Murray, J. I., Bao, Z., Boyle, T. J., Waterston, R. H. The lineaging of fluorescently-labeled Caenorhabditis elegans embryos with StarryNite and AceTree. Nature Protocols. 1 (3), 1468-1476 (2006).
- Wu, Y., et al. Spatially isotropic four-dimensional imaging with dual-view plane illumination microscopy. Nature Biotechnology. 31 (11), 1032-1038 (2013).
- Kumar, A., et al. Using Stage- and Slit-Scanning to Improve Contrast and Optical Sectioning in Dual-View Inverted Light Sheet Microscopy (diSPIM). The Biological Bulletin. 231 (1), 26-39 (2016).
- Kumar, A., et al. Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nature Protocols. 9 (11), 2555-2573 (2014).
- Wu, Y., Christensen, R., Colon-Ramos, D., Shroff, H. Advanced optical imaging techniques for neurodevelopment. Current Opinion in Neurobiology. 23 (6), 1090-1097 (2013).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17708-17713 (2011).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136 (12), 1963-1975 (2009).
- Stelzer, E. H. Light-sheet fluorescence microscopy for quantitative biology. Nature Methods. 12 (1), 23-26 (2015).
- Winter, P. W., Shroff, H. Faster fluorescence microscopy: advances in high speed biological imaging. Current Opinion in Chemical Biology. 20, 46-53 (2014).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. Astronomical Journal. 76 (6), 745-754 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. JOSA. 62 (1), 55-59 (1972).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Hart, A. C. Behavior. WormBook. , (2006).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. Chapter 14, Unit14 20 (2010).
- Gualda, E. J., et al. SPIM-fluid: open source light-sheet based platform for high-throughput imaging. Biomedical Optics Express. 6 (11), 4447-4456 (2015).
- Ardiel, E. L., et al. Visualizing Calcium Flux in Freely Moving Nematode Embryos. Biophysical Journal. 112 (9), 1975-1983 (2017).
- Walton, T., et al. The Bicoid class homeodomain factors ceh-36/OTX and unc-30/PITX cooperate in C. elegans embryonic progenitor cells to regulate robust development. PLoS Genetics. 11 (3), e1005003 (2015).
- Altun, Z. F. WormAtlas. , (2002).
- Wood, W. B. Embryology: In the nematode C. elegans. Cold Spring Harbor Laboratory Press. Chapter 8, 215-241 (1988).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314 (1165), (1986).
- Bao, Z., Murray, J. I. Mounting Caenorhabditis elegans embryos for live imaging of embryogenesis. Cold Spring Harb Protoc. (9), (2011).
- Christensen, R. P., et al. Untwisting the Caenorhabditis elegans embryo. eLife. 4, (2015).
- Santella, A., et al. WormGUIDES: an interactive single cell developmental atlas and tool for collaborative multidimensional data exploration. BMC Bioinformatics. 16, 189 (2015).
- Dupuy, D., et al. A first version of the Caenorhabditis elegans Promoterome. Genome Research. 14 (10B), 2169-2175 (2004).
- Reece-Hoyes, J. S., et al. Insight into transcription factor gene duplication from Caenorhabditis elegans Promoterome-driven expression patterns. BMC Genomics. 8 (27), (2007).
- WormBase. , Available from: https://www.wormbase.org (2019).
- Lee, R. Y. N., et al. WormBase 2017: molting into a new stage. Nucleic Acids Research. 46 (D1), D869-D874 (2018).



