Summary
כאן, אנו מציגים גישה קומבינטורית באמצעות מיקרוסקופ ברזולוציה גבוהה, כלים חישוביים, ותיוג תא יחיד בחיים C. אלגיה העוברים להבין דינמיקה תא בודד במהלך ההתפתחות העצבית.
Abstract
אלנבאבדיוטיס (ג. אלגיה) בולטת כאורגניזם היחיד שבו האתגר של הבנת המקורות הסלולריים של מערכת העצבים כולה ניתן לצפות, עם רזולוציית תא בודדת, בvivo. כאן, אנו מציגים פרוטוקול משולב לבדיקת ההתפתחות העצבית בעוברי המשך. הפרוטוקול שלנו משלב דימות, לשון ומעקב נוירואנטומי של תאים בודדים בפיתוח עוברים. אנו משיגים הדמיה לטווח ארוך, 4-מימדי (4D ) של החיים העוברים עם הרזולוציה המרחבית הכמעט איזוטרופית באמצעות שימוש באמצעות תצוגה כפולה הפוך מיקרוסקופית תאורה סלקטיבית (dispim). גרעינים ומבנים עצביים בעוברי נמטודות הם התמונה התמזגו איזוטרובאלי כדי להניב תמונות עם רזולוציה של ~ 330 ננומטר בכל שלושת הממדים. אלה של דקה אחר דקה ברזולוציה גבוהה 4D הנתונים מנותח לאחר מכן לתיאום זהויות מוחלטות היוחסין של התא עם ביטוי גנים ודינמיקה מורפולוגית בתוך תא יחיד ורמות subcellular של פירוט. הפרוטוקול שלנו מובנה כדי לאפשר יישום מודולרי של כל אחד מהשלבים המתוארים ולשפר את המחקרים על embryogenesis, ביטוי גנטי, או התפתחות עצבית.
Introduction
ג. אלגיה בולטת כאורגניזם היחיד שבו כל תא בעובר ניתן לצפות במהלך התפתחות עצבית. עם כל התא-שושלת היוחסין ידוע וקבוע1, עם פיתוח של כלים חדשים המאפשרים תיוג והדמיה רציפה של תאים בודדים בעוברים, ביולוגים יכולים כעת להתחיל בחינת צעדים שונים בפיתוח של העצבים נמטודות מערכת מכל הזוויות-לידה תאים; הגירה ובידול; היווצרות neurite, ממוקד הצמיחה והפסטולציות; היווצרות סינפסה; וכוונון של מעגלים פונקציונליים. לכידת הדינמיקה העצבית במהלך העובר C. אלגיה , על ידי שילוב של כתבים מבוטא באופן בלתי נשכח ומיקרוסקופ קרינה, הוא בעל ערך לקהילה המדעית.
מחקרים התפתחותיים ב- C. אלגיה ממנף לעתים קרובות את השושלת הקבועה ומפות התאים של המין הזה כדי להגדיל את ההבנה ההקשרית ברמת התא הבודד בתוך האורגניזם השלם1. ניתוח הזדקנות אוטומטי-שימוש ב-starrynite2,3,4 ו-5,6,7,8 תוכנות-יתרונות מניגודיות גבוהה, רזולוציה גבוהה תמונות של גרעיני פלורסנט. כדי לעבוד בצורה אופטימלית, הסטרינייט/מרחרי תלוי גם בכיוון מוגבל צפוי של עוברי התמונה במהלך הפיתוח. מיקרוסקופיה קונפוקלית, מתבצעת ב -C. אלגיה עוברים שנדחסו בין שני שמיכות, היתה שיטת מיקרוסקופ אוטומטי סטנדרטי לינארי במשך יותר מעשור, כי הוא מספק הן חדות גבוהה/רזולוציה גבוהה ומוגבל צפוי התמצאות של העובר7,8. בעבר תיארנו את הבנייה והשימוש של הרומן המבוסס על גיליון האור מבוסס-תצוגה כפולה הפוכה מיקרוסקופ תאורה סלקטיבי (dispim) עבור הדמיה לדוגמה חיה כגון C. אלגיה embryogenesis9,10 , מיכל בן 11 , מיכל בן 12 , 13. מיקרוסקופ האור, באופן כללי, מספק פוטורעילות נמוכה, מהירות גבוהה והדמיה ארוכת טווח של יצורים חיים תלת-ממדיים14,15. השיטה diSPIM, במיוחד, מייצרת 4-מימדי (4D) תמונות עם רזולוציה כמעט איזוטרופי מרחבית של כ 330 ננומטר9.
בהשוואה למיקרוסקופיה קונפוקלית, diSPIM מציעה אות לרעש גבוה יותר ומהירות, יותר רזולוציה איזוטרופית מרחבית, והוא מתאים יותר לטווח ארוך vivo הדמיה 16. לפיכך, עבדנו כדי להתאים את נתוני diSPIM עבור קלט לתוך StarryNite/מרחרי וחקרו אם זה ישפר את ניתוחי הששון. משוכה גדולה היא כי לא ניתן להיות מוגבל בקלות של דגימות של diSPIM על ידי דחיסת קליפת ביצה כדי לאמץ את הכיוונים הצפויים עבור StarryNite/מרחרי. כיוון אקראי של תנוחות תאים באמצעי האחסון המנותח מבזה את הדיוק של ניתוח לינשון אוטומטי.
לפיכך, העסקנו ציטוהצג, ממשק משתמש מונחה מציג המאפשר למשתמשים לבחור כיוון תלת-ממד מדויק של העוברים במהלך עיבוד טרום של תמונות diSPIM, מניב נתונים תמונה כי הוא גם איכות אופטימיזציה ההקשר מודע הקשר לקלט לתוך StarryNite . אני מבין על-ידי בחירת המשתמש של עוברי התמונות, בתזמולן מופיעים צינורות לעיבוד נתונים אוטומטיים. תמונות העובר שנחתכו ומופחתים על רקע נשמרות בתוך קבצי מחסנית TIFF עבור כל מיקום, נקודת זמן ותצוגה. ציטוהצג לאחר מכן שיחות לאחר מכן התוכנית spimfusion כדי שיתוף לרשום ובמשותף לפרק את שתי התצוגות טרום מעובד, באמצעות ריצ'רדסון-לוסי17,18 אלגוריתם לתשואה איזוטרופי ברזולוציה גבוהה נפח תמונות. ערכת פרמטרים ספציפית ל-diSPIM ממוטבת עבור StarryNite כדי למשול בהתנהגותו במהלך פילוח תמונה ומעקב אחר הגרעין בתמונות התמזגו באופן איזוטרואלי. תמונות התמזגו ותוצאות הזדקנות מתקבלות לאחר מכן באמצעות מרחרי, מה שמאפשר למשתמשים לזהות ולתקן שגיאות בעקבות השושלת האוטומטית שנוצרה על ידי StarryNite. מרחרי יכול גם להציג עץ השושלת ואת 3D עיבודים המודל של גרעינים מסומנים העובר. אנו מוצאים כי באופן אוטומטי מהירות ודיוק משופרת במידה ניכרת באמצעות תמונות התמזגו איזוטרובאלי, כאשר לעומת תמונות raw ממצלמת SPIM. הפרוטוקול שלנו, בעוד אופטימיזציה ליישום C. אלגיה המתואר כאן, יכול להיות מותאם בדרך כלל עבור הזדקנות אוטומטית של נתוני dispim המיוצרים עבור מינים אחרים או דגימות. אם זהו השימוש המיועד בפרוטוקול, הינכם מתבקשים לשים לב כי כוונון נוסף של פרמטרי הסטארריניט יהיה נדרש עבור דגימות חדשות, כמתואר3,4.
יישום מוצלח של פרוטוקול זה מביא תמונות עם רזולוציה 4D-isotropic ומאפשר לביולוגים לעקוב אחר שאריות התא, ובמקביל לזהות ולנתח נוירונים בהתפתחות העובר C. אלגיה . יתר על כן, על ידי מיזוג מספר אלגוריתמים לאחר עיבוד-עם האצת חומרה להיות הזמן הארוך ביותר של אלה-אנחנו יכולים עכשיו לנתח הן הפרטים הקטנים בסדר הסלולר ואת התאים לפני הצינוק ואת הגורלות התאים של עוברים חיים בזמן אמת למעשה. פרוטוקול חדש זה מאפשר מניפולציה מדויקת, מושכלת והתבוננות של התנהגות התא במהלך מחקרים המוקד של בידול ומורפולגנזה ב vivo. בכתב יד זה, אנו מציגים הסבר מפורט על הפרוטוקולים המשופרים שפיתחנו לאיתור הזדקנות ומעקב תאים בפיתוח העוברים של C. אלגיה , כדי לשפר את המחקרים של embryogenesis, ביטוי גנטי או התפתחות עצבית.
Protocol
1. הרכבת חדר הדמיה של הפלדה diSPIM עם שמיכות פולי L-ליזין מצופה
הערה: השלבים הבאים נדרשים כדי למטב ולהפוך ניתוח שושלת היוחסין של עוברי נמטודות על ידי starrynite/מרחרי. ניתן להשמיט מספר אפשרויות (המסומנות בהתאם לכאלה) עבור ניסויים הדורשים מעקב אחר שורה של תאים מסוג C. אלגיה .
- לצייר מלבן קטן (2mm x 5mm) במרכז שמיכות מלבני נקי (no. 1.5, 24 מ"מ x 50 מ"מ) עם טוש (או עט דומה).
- הפוך את הכיסויים אל הצד הלא מסומן ומקום 10 μL של פולי ליזין (קטלוג. לא. P1524) מעל המלבן המסומן.
הערה: בצע ריכוז פועל של 1 מ"ג/mL פולי-L-ליזין מומס במים מסוננים (או שווה ערך). לאחסון לטווח ארוך, הכינו 5 – 10 מ ל והחנות ב--20 ° c. ברגע שהוא מופד, ניתן לאחסן את הליבטים בטמפרטורת החדר (23 ° c) במשך 3 – 4 שבועות. - אפשר ל-פולי ליזין להניח את הכיסויים ל -5 דקות (איור 1A). פולי-L-ליזין משמש לציפוי כיסוי זכוכית מכסה שבו העוברים יהיה רכוב, המאפשר העובר קליפת ביצה להיצמד בחוזקה על שמיכות, גם כאשר המטרות שקועים במדיום הדמיה.
- מניחים את שמיכות פולי L-ליזין מצופה במחצית התחתונה של חדר הדמיה פלדה.
- מניחים את החלק העליון של חדר הדמיה פלדה על החצי התחתון עם שמיכות ולהדק עם ארבעת הברגים הקשורים לחדר. בדוק מבחינה חזותית מהצד כדי לוודא שהחצי העליון מוכנס באופן שווה במחצית התחתונה (איור 1B).
- למלא את החדר עם 7-8 מ ל של M9 מאגר19, מאגר isotonic אשר מסייע למנוע עוברי הבמה המוקדמים מתוך להיכנע ללחץ אוסמוטי נורמלי. 1-תא, ואפילו שני תאים עוברים osmotically רגישים ויכולים להתפתח באופן חריג אם לא במאגר זה isotonic. עוברים גם נוטים לעצור בשלב 3-קיפול אם M9 מוחלף במים.
2. הכנת דגימות העובר של C. אלגיה להרכבה
הערה: כ 18 שעות לפני הדמיה, חמישה צעירים (יום אחד מאז מולד הסופי) מבוגרים ו 10 הזחל שלב 4 (L4) C. אלגיה הם להיבחר בינונית צמיחה נמטודות (ngm) לוחית אגר הנזרע עם E. coli זן OP50. מבחר חוטי פלטינה משמשים להזזת זחלים ובוגרים צעירים מבלי לפגוע בבעל החיים19.
- הכינו מתיל תאית 1% (קטלוג...) H7509-25G) פתרון במאגר M9.
הערה: מתיל תאית חייב להיות מעורבב M9 חם עד התפרקה. ברגע שהוכן, ניתן לאחסן פתרון זה בטמפרטורת החדר. - הוסף 500 μL של 1% מתיל תאית-in-M9 פתרון לתוך הדיכאון של שקופית מיקרוסקופ קמור. מאגר צמיגי זה ישמש בשני מקרים: 1) בעת קצירת העוברים המוקדמים על ידי ניתוח של תולעים בוגרות ו -2) כשרוחצים עוברים בשלב מאוחר שנאספו ישירות מצלחת NGM.
הערה: מתיל תאית משמש כדי למנוע מעוברים לדבוק שקופית המיקרוסקופ. - עבור הדמיה העוברים בשלב מאוחר, לבחור הונחו העוברים (רצוי מצלחת NGM עם מבוגרים צעירים נוכח) באמצעות בוחר עפעף, ולהעביר את העוברים אל 1% מתיל תאית על שקופית מיקרוסקופ קמור. לאסוף עפעף עוזר להפחית את הכוח ובכך למזער לחץ או נזק העוברים במהלך הטיפול. ההליך לעשות בוחר עפעף מכוסה על ידי הארט20.
- עם להרים עפעף שני (ביד הנגדית), הקש בעדינות את שני הריסים יחד כדי להשעות את העוברים בתוך מתיל תאית.
- אופציונלי: אם התכנון לתאים עובריים של השושלת עם StarryNite, יש לטעון תא אחד לעוברים ארבעה תאים. כדי לעשות זאת, תחילה בחר מבוגרים צעירים מצלחת NGM ולהעביר אותם לפתרון M9-מתיל תאית בשקופית מיקרוסקופ קמור באמצעות בחירת חוט פלטינה.
- אופציונלי: עם קצות מחודדים של מחטים תת-עוריות (no. 18G x 1 1/2), פורסים את בעלי החיים בגוף באמצע כדי לשחרר תא אחד לעוברי ארבעה תאים.
3. פיפטה בפה: הרכבת שפופרת מעוקר עם פיפטה מיקרוקפילר
הערה: אנו משתמשים בשפופרת משומנת עם יד שלמה של צינור מיקרוקפילר שהוכנס לתוך אטם הגומי של השפופרת. זה מאפשר לנו להעביר עוברים משקופית הניתוח למשטח פולי-L-ליזין-מצופה בחדר ההדמיה מלא מאגר.
- באופן ידני למשוך את הצינורות מיקרונימי מעל להבה פתוחה כדי ליצור שני חצאים עם טיפים מתוחים.
- קחו חצי מהצינור המיקרונימים והכניסו את הקצה הקהה לתוך אטם הגומי של צינור האספירין (איור 1C). הניחו את החצי השני של הצינורות המיקרוקפיציים בצד לשימוש מאוחר יותר (במידת הצורך).
- עם צינור העירוי המורכב מצויד בפיפטה מיקרוקפילר, שבור בעדינות את קצה הפיפטה המיקרונימי וצור פתח שיתאים בערך 1 – 2 עוברים (מכאן בכלי זה נקרא "פיפטה בפה").
4. הרכבה של העוברים באמצעות שמיכות פולי-L-ליזין
- עם הדובר המוחזק בעדינות בין השיניים, למלא מראש את הצנרת microcapillary עם 10 – 15 μL של M9 מאגר ולאחר מכן למצוץ בעדינות מספר עוברים מן השקופית הקמור לתוך הקפילר.
- להעביר את העוברים לחדר הדמיה פלדה מלא עם M9 מאגר, מיקום קצה נימי כך העוברים ייפול לתוך המלבן המרכזי של הכיסויים.
- הימנעות מפגיעה בעוברים, בעדינות להעביר אותם עם בוחר עפעף או להשתמש בפיפטה הפה כדי למקם את העוברים אנכית, כדי לכוון את העוברים כך ציר ארוך של העובר ניצב לציר ארוך של שיבוץ (איור 1B הכניסה , הפאנל התחתון).
הערה: מיקום העובר בכיוון זה ממזער את מספר הפרוסות לתמונה, ובכך מפחית את מינון האור ואת זמן עיבוד הנתונים תוך שיפור מהירות הרכישה. - מניחים את חדר הדמיה פלדה לתוך מחזיק המדגם על מיקרוסקופ הבמה (איור 1D).
5. הרכבה, התקנת תוכנה ואופטימיזציה לייזר להדמיה עובריים באמצעות ה-diSPIM
- לראות הוראות צעד אחר צעד על איך להרכיב את כל הסיבים מצמידים כולו מחלקים זמינים מסחרית ב קומאר et al.10,11 ו ב http://www.dispim.org. פרוטוקול וידאו של כיצד להרכיב את ה-diSPIM זמין גם באתר האינטרנט של אסי (http://www.asiimaging.com).
הערה: הגדרת המכשיר עבור פרוטוקול זה זהה קומאר et al.10,11 dispim, אשר עושה שימוש של 40x 0.8 na המים שטבילה עדשות עבור הדמיה. ההבדל היחיד בין הכיוונון בפרוטוקול זה, קומאר ואח '10,11 היא תוספת של דיקרואיק מראה (פיצול ב 560 ננומטר) ו אדום וירוק מסננים bandpass בתוך התקן פיצול הדמיה (דגם A12801-01) מותקן על שניהם הדמיה של הזרועות. התוספת של התמונה מפצל אופטיקה מאפשר לכידת בו של תמונות משני fluorophores נפרדות – נרגש על ידי 561 ננומטר ו 488 לייזרים nm – על ידי הפרדת להקות פליטה על שני חצאים של אותו שבב מצלמה. - לאחר הרכבת הכלים, בדוק את היישור האופטי של ה-diSPIM לפני הדמיה.
הערה: כדי ליישר כהלכה את ה-diSPIM לראות https://youtu.be/qnOrg30NNuE ועבור מידע חומרה, http://dispim.org/hardware/objectives וhttp://www.asiimaging.com. - השתמש פלטפורמת קוד פתוח מיקרו-מנהל (https://micro-manager.org/)21, אשר כבר אופטימיזציה עבור הפעלת גיליון מיקרוסקופים אור עבור תפוקה גבוהה הדמיה סלולרית22. אנו ממליצים על שימוש בתוסף אסי diSPIM לרכישה מרובת מיקום, המאפשר הדמיה בו של עד 30 עוברים כמתואר23.
- עם מיקרו-מנהל פתוח, להגדיר עוצמות לייזר כדי ~ 179 μW (0.5) עבור 488 nm ו ~ 79 μW (0.25) עבור 561 nm (איור 2A, מלבן אדום).
הערה: אלה הן הגדרות מומלצות להדמיה ארוכת טווח של העוברים באמצעות המשך של דקה אחת. במהלך הדמיה לטווח ארוך בצבע כפול, 561 ננומטר לייזר משמש לגרעיני התמונה (Mcherry:: Histone) עד העוברים נמצאים בשלב השעועית, ובנקודה הזאת 488 ננומטר לייזר הוא לאחר מכן להפעיל גם את התמונה של הנוירונים התוויות gfp. תנאי הדימות הללו ממוטבים לצמצום הרעילות ולהבטחת הישרדות ובקיעה של העוברים תוך הפיכת ממושך (12 – 14 שעות) לרכישה רציפה של נתונים נוירוהתפתחותיים ושינתיים. - ב מיקרו-מנהל, בחר תוספים בתפריט ≫ בקרת התקן ≫ אסי dispim כדי לפתוח את החלון באסי Dispim ( איור 2b). בחר בכרטיסיה רכישה . במקטע הגדרות שמירת נתונים של כרטיסיה זו (מלבן ירוק), המקטע הגדרות אמצעי אחסון (ריבוע כחול) ומקטע הגדרות פרוסה (ריבוע כתום), ודא שכל פרמטר מוגדר כמוצג באיור 2b.
הערה: תוכנת ניתוח התמונה שלנו ציטוהצג מותאם לעבודה עם תבניות אחרות של נתוני פלט אופציונלי כגון סדרת הקבצים OME-TIFF משורשר בצובר ו-TIFF-מחסנית סדרת קבצים שנוצרו לאחר רכישה באמצעות השימוש בפונקציה לייצא מובנה מיקרו מנהל. בדרך כלל, נעשה שימוש בתבנית הנתונים של קובץ OME-TIFF עם מחסנית חד-ממדי, מכיוון שהיא מאפשרת צפייה בזמן אמת ועיבוד של עוצמת התמונה ברגע שהנתונים הגולמיים נרכשים.
6. פרמטרים ממוטבים פוקוס אוטומטי עבור הדמיה לטווח ארוך של C. אלגיה עוברים
- Set מיקרו מנהל הפרמטרים פוקוס אוטומטי להגדרות אופטימיזציה לטווח ארוך שושלת היוחסין באיכות הדמיה של העוברים של C. אלגיה . בחלון ה-ASI, לחץ על הכרטיסייה פוקוס אוטומטי (איור 2c). במקטע אפשרויות פוקוס אוטומטי כללי (ריבוע שחור) לציין פרמטרים בדיוק כפי שמוצג. שים לב כי ערוץ פוקוס אוטומטי (הכיכר האדומה) צריך לציין ערוץ הגרעין שלך ערוץ הזריחה בניסויים הזדקנות.
הערה: אם ההיסט המירבי גדול מ-5 μm, התמונות נוטות להיסחף מחוץ לפוקוס. - לחץ על, תוספים ≫ כלי רכישה > תבנית כיסוי.
- בחלון תבנית שכבת -על, לחץ על הצג רשת.
- בחלון משאבים מסוימים, לחץ על הכרטיסיה ניווט .
- לחץ על תיבות סימון עבור קרן וגליון של נתיב A או B לאחר מכן לחץ על Live. רכישת תמונה מתחילה. נפתח חלון ' תצוגה חיה '. בחר את אזור ניתוח המיקוד האוטומטי של העובר על-ידי ציור תיבה סביב העובר בערוץ שנבחר מ6.1.
הערה: אנחנו בדרך כלל ללכוד 420 נקודות זמן עבור 10 עוברים לכל הפעלה הדמיה. הנתונים הגולמיים לכל הפעלת תמונה הם בדרך כלל 1.7 טרה-בתים, בעוד שנתונים מעובדים באופן שאינו מעובד על-ידי המערכת הם 1.4 טרה-בתים (ראה שלבים 9 ו -10). אנו ממליצים באמצעות דיסק קשיח בקיבולת גבוהה (18TB במערכת הנוכחית שלנו) עבור רכישת תמונה, פלטפורמות ענן עבור אחסון תמונה. - לחץ על התחל רכישה בכרטיסיהרכישה כדי להתחיל לכידת תמונה רב-ממדית לטווח ארוך (איור 2b).
7. פתיחת תמונות מיקרו-מנהל raw ב ציטוהצג
- הורד את צרור התוכנה מhttp://dispimlineage.wormguides.org.
הערה: צרור התוכנה יורדו כקובץ. zip ויהיה צורך לחלץ אותם ל-"C:\" ספריה לפני השימוש. פרטים נוספים לגבי ההתקנה ניתנים ב-http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm. - לחץ לחיצה כפולה על הקובץ C:\tatlltcples\afip.phamautiata כדי להתחיל בהפעלת ציטוהצג.
- בחרו קובץ תפריט ≫ מוניטור ≫ לים חדשים (מיקרו-מנהל). אתר את תיקיית ערכת נתוני הבסיס שבה נשמרו תיקיות של נקודות זמן רכישה. בחר בתיקיית נקודת זמן ולחץ על ' פתח'. חלונות ניווט רב-ממדיים (הנקראים מוניטור windows) נפתחים באופן אוטומטי עבור spima ו-Spimb (איור 3a).
הערה: חלונות אלה מפקחים על תיקיית נתוני הבסיס עבור ערימות שנשמרו של נקודות זמן raw (במקרה שדגימה עדיין מוקלטת). לאחר הרכישה כל נקודת זמן חדשה, כל אחד מהחלונות ניטור הזרועות SPIM ברורים ומשרות לדוגמה ירענן כדי להציג את ערכת הנתונים המלאה של 4D רב-ערוצי עבור כל עובר.
8. יצירת תמונות הקרנה מקסימלית עם ציטוהצג
גם לפני הפירוק, ניתן לעבד במהירות את הנתונים הגולמיים כדי להעריך את התכונות הגלובליות של הדגימה.
- לחץ על לחצן Z-MIP בלוח השמאלי של חלון התמונה (איור 3a, מלבן אדום) כדי להפוך את התחזיות בעוצמה מקסימלית דרך עומק מלא ומהלך במשרה מלאה של מיקום נתון או זרוע spim. יופיע חלון היפרמחסנית של Z .
- בחלון hyperstack של Z , קבע סוג הקרנה לעוצמת Max. ציין אילו ערוצים, פרוסות ומסגרות של נקודות זמן לעיבוד בהתבסס על העדפת המשתמש.
- לחץ על אישור בעת השלמתן.
- בחר את מיקום התיקיה כדי לשמור את פלטי העוצמה המרבי מחלון דו-שיח הקבצים ולאחר מכן לחץ על אישור. אפשר קצת זמן (15 – 20 דקות, בהתאם לגודל של ערכת נתונים וכוח עיבוד של המחשב) עבור ציטוהצג כדי ליצור תמונות הקרנה.
9. ניתוח שיננים של תאים בנתונים נפחי ברזולוציה גבוהה איזוטרופי
- אופציונלי: כאשר הנתונים הגולמיים נפתחים באמצעות צג dispim ב-ציטוהצג, בחרו בכלי בחירת המצולע (איור 3a, חצים שחורים) ולחצו רק מחוץ לקצוות האחוריים של העובר הקדמי, האחורי והגטיים (בסדר המדויק הזה) כדי ל ליצור דפוס "bowtie" על העובר. עשו עבור שתי התצוגות (SPIM-A ו-SPIM-B, איור 3A).
הערה: בחירה זו מציינת את האזור האליפטי של ריבית (ROI) שבו העובר ממורכז ומתעד את הציר האחורי הקדמי של העובר. דפוס עניבת רמזים ציטופי מראים כי המשתמש מתכנן לציין סיבוב מדויק של הכרכים הסופיים האחרונים התמזגו לתוך כיוון האופטימלי לניתוח שורה על-ידי סטאררינייט/מרחרי. במקרים שבהם הזדקנות האזור אינה חלק מהתוכנית הניסיונית, ניתן לבחור בכלי בחירה אחרים ובצורות אחרות כדי להגדיר את ההחזר עבור עיבוד תמונה. - אם עוברים מרובים בו באמצעות אפשרות רכישה מרובת מיקום, לפתוח ולבצע שלב 9.1 עבור כל העוברים. פעולה זו תאפשר ביצוע מקבילי של צעדים עתידיים עבור כל העוברים בהפעלה אחת. סגור את חלונות סימה ו-SPIMB עבור כל העוברים שאין ברצונך לעבד.
- לחץ על לחצן dispim בחלונית הצדדית השמאלית של חלון צג Dispim (איור 3a, מודגש בצהוב). פעולה זו חושפת לוח משנה של פקדים הספציפיים לעיבוד של diSPIM.
- יישר ערוצים ירוקים ואדומים עבור כל זרוע SPIM. מאחר שסוגי מפצלי של ערוצי פליטה משמשים ללכידת תמונות אדומות וירוקות נפרדות בו באותה מצלמה, חשוב ליישר חזותית את רישום הפיקסל המדויק של שני שדות התמונה הסמוכים באופן פיזי כאשר הם מצופים. שימוש חוזר באותם התאמות יישור הוא בדרך כלל אפשרי על הרבה הפעלות הדמיה רצופות, אבל יש לבדוק (כמו בשלבים 9.4.1 – 9.4.5).
- החל בלוח SPIMA, בחר את הערוץ האדום על-ידי הזזת פס הגלילה CM שמאלה (איור 3a, חץ כתום, הלוח השמאלי).
- באמצעות x-, y, ו-z-השיפוט (איור 3A, ריבוע כתום), להזיז את הערוץ האדום כדי להתאים את הירוק.
- לחץ על לחצן Dispim (איור 3a, מודגש בצהוב), כדי לסגור את לוח המשנה ואת התפשטות הגורם המפעיל של אותן המשמרות לכל חלונות המיקום האחרים.
- ודא שיישור נכון הופץ למסגרות ולנקודות זמן אחרות על-ידי הזזת פס הגלילה "z" (איור 3a, חץ כחול, לוח שמאלי) ו/או פס גלילה "t" (איור 3a, החץ הירוק, הלוח השמאלי). אם בוצעה רכישה מרובת מיקום ומספר עוברים הופיעו בתמונות (Step 5.3), היישור צריך גם להיות מופץ לעוברים אלה. אשר על-ידי בדיקת גם המספרים עבור x-, y, ו-z-שפיטה (איור 3A, ריבוע כתום, אשר צריך להיות זהה עבור הפאנל spima של כל העוברים).
- חזור על שלבים 9.4.1 – 9.4.4 עבור חלון צג ה-SPIMB (איור 3A, החלונית הימנית).
- לחץ על לחצן "Dispim" ולאחרמכן את כפתור "מרעום" (איור 3a, מלבן כחול) כדי לפתוח תיבת דו-שיח הנקראת "פירוק/הפתיל נתונים גולמיים של נתוני Raw" (איור 3a). הגדר פרמטרים כמוצג באיור 3B. פרמטרים אלה מטופלות בקצרה בשלבי המשנה הבאים:
- הגדר רישום מפתח בערוץ 1 (488 ננומטר לייזר) או 2 (561 ננומטר לייזר). בחר את הערוץ עם אות צפוף או יותר בכל מקום. עבור ניסויים לינאייג ' תמיד לבחור את הערוץ המשמש לדמות הקרינה הגרעינית בכל מקום.
- הגדר אמצעי אחסון של קלט כיוון יחסי ל-+ 1 או -1. אינדקס האוריינטציה הנכון תלוי במיקומים ספציפיים למצלמה של diSPIM (איור 4).
הערה: אם לא ברור, בדוק כל אפשרות על-ידי שכפול נקודת זמן יחידה מחלון צג SPIM A ו-B Dispim , בעקבות השלבים 9.1 – 9.12 ובחירה שרירותית של אמצעי אחסון של קלט כיוון יחסי לבדיקה. כיוונים שגויים תניב תמונות מטושטשות עם חפצים, בעוד הכיוונים הנכונים תניב תמונות ברורות. לאחר מכן, ניתן לעשות שימוש חוזר באמצעי האחסון היחסיים של הקלט שיוצר את התמונה הנקיה עבור כל הנתונים העתידיים מכלי ה-dispim הנתון. - בחר אם יש לכוון את אמצעי האחסון המונפח באופן זהה לעוצמת הקלט A או B (בהתבסס על העדפת המשתמש).
- בחר "רישום טרי עבור כל אמצעי אחסון". אפשרות זו שולטת באופן שבו SpimFusion מחשב מטריצות רישום עבור כל זוג אמצעי אחסון בכל נקודת זמן. האפשרות "רעננה" מאפשרת לאלגוריתם למטב את הרישום באופן מיטבי בכל נקודת זמן.
- הגדר את מספר האיטראציות לפירוק ל-10. מספר זה נוטה להניב בצורה מהימנה את הרזולוציה הגבוהה הרצויה באופן חסכוני בזמן.
- אופציונלי: אם ברצונך לבצע הזדקנות באופן אוטומטי (מומלץ ביותר), בדוק הפעלה אוטומטית של הפעלת משתני כוננים ממוזגים. אפשרות זו תפעיל את StarryNite אוטומטית כדי לפלח ולעקוב אחר תאים באמצעי הדימות המיוצרים על ידי SpimFusion.
- אופציונלי: לדיוק הגבוה ביותר בהזדקנות שורה אוטומטית, מומלץ למקם מחדש את אמצעי האחסון של העובר האיזוטרובאלי לתוך הקנונית "ADL" (Anterior [x-west], Dorsal [y-North], שמאל [z-בסמוך]) כיוון. בחרו באפשרות ' הגדר כיוון פלט של אמצעי אחסון ' בתצוגה המקדימה כדי לציין בחירה זו. ציטוהצג יגיב על ידי עיבוד זוג ראשוני של אמצעי אחסון התמזגו איזוטרופית, דבר המאפשר למשתמש להתבונן היטב ולציין את הסיבובים הדרושים להשגת רישום ADL.
- לחץ על כן לאחר שכל הפרמטרים ייבחרו.
- ציין את ספריית הפלט שבה יש לשמור את הקבצים המעובדים. לחץ על אישור.
- אופציונלי: אם נבחר כיוון פלט אמצעי האחסון בתצוגה המקדימה , הגדר את פס הגלילה t (איור 3a, החץ הירוק, הלוח השמאלי) בחלון spim-A לנקודת הזמן המוקדמת שבה תאי ABa ו-abp הגיעו למטא-שלב. הגדר את פס הגלילה t בחלון spim-B לשלב ההתפתחות המאוחר יותר בפסיק. פעולה זו תסייע בציון אוריינטציה ADL.
- אופציונלי: לחץ על אישור כאשר מוכן. אם אפשרות התצוגה המקדימה ב9.5.7 לעיל נבחרה, רק שני אמצעי אחסון של תצוגה מקדימה יהיו ממוזגים עבור נקודות הזמן שצוינו על-ידי מחווני ה-t של חלונות התמונה SPIM-A ו-SPIM-B. ניתן להשתמש בשתי נקודות זמן תצוגה מקדימה אלה כדי לציין יישור מדויק של אמצעי אחסון לעובר הפלט לכיוון ADL, כפי שמוסבר להלן.
- אתר את 3DProjY_Decon-Fuse_ שהוצגו לאחרונה.... חלון. הזז את פס הגלילה t לנקודת זמן 2 של חלון תצוגה מקדימה זה. הזז את המחוון Z עד להצגת התצוגה ישירות לאורך הציר הארוך של העובר.
- להעביר את פס הגלילה t בחזרה לנקודת הזמן 1 של 3DProjY_Decon-Fuse_.... חלון. בחרו בכלי בחירת קו וציירו בחירת קו מתא ה-EMS (הגרעין העגול ביותר) דרך מישור לוחיות המטא-מסוף של תא AB.
- לחץ על לחצן כתום diSPIM בתצוגה מקדימה של 3DProjY_Decon-Fuse_.... חלון. ההתאמות הנאות לכיוון של עוצמת התמונה בתצוגה המקדימה תישמר לשימוש בעיבוד ערכת הנתונים המלאה.
- אופציונלי: תיבת הדו של אמצעי אחסון נתונים גולמיים של פירוק/הפתיל מופיעה שוב, בדיוק כמו בשלב 9.5 לעיל. לחץ על כן מבלי לבחור את כיוון הפלט של אמצעי האחסון באפשרות תצוגה מקדימה . ציין את תיקיית הפלט עבור הפעלת עיבוד הנתונים המלאה.
- הגדר את פסי הגלילה t (איור 3a, החץ הירוק, הלוח השמאלי) של החלונות המפקחים על המסך כדי להפעיל את נקודת הזמן ההתחלתית (spima) ונקודת הסיום (spimb) של הטווח המלא של התמונות לעיבוד. לאחר מכן לחץ על אישור.
- כמו SpimFusion מתקדם, ציטוהצג פותח ומרענן חלון רב מימדי המציגה את הנפח חתוך-4D isotropic התמזגו עבור כל עובר, כמו גם שני חלונות עם מסתובב-4D התחזיות מקסימום עוצמה של נפח isotropic. בזמן זה, לא לשבש או לסגור כל חלון ציטוהצג עד עד היתוך איזוטרופי מעקב השושלת הושלמה.
- אופציונלי: שים לב שברגע שמסך הפתיחה של StarryNite הופיע ומאוחר יותר נעלם, צינור עיבוד הנתונים המלא הושלם. אין לסגור חלון זה במהלך העיבוד או בסטררינייט.
10. פתיחת סדרת מעקב של השושלת הסטארריניט באצטרי (אופציה)
- פתח את הגירסה המותאמת אישית של "AceTree_16BitCompat. jar" שסופקו.
- בחר תפריט בקובץ > פתוח את התצורה של קובץ. אתר את ספריית הפלט שצוינה בעבר ל-ציטוהצג. פתח את תיקיית המשנה Decon_Fuse_... _ Pos [n] עבור העובר [n]. בחר aaa_edited. xml ופתח.
- השתמש בתפריט מרחרי ערוך > ערוך כלים כדי לפתוח את מסלול העריכה ולהתאים או למחוק את חלונות התאים.
- לחץ על העיגול החצי מוצלל באיור 5B, בריבוע האדום כדי לכוונן את עוצמות האדום והירוק.
- המשך עם הדמיית השושלת והעריכה כפי שתוארה בעבר5,6,8 (כתבי-יד נכללים גם בצרור ההורדות שלנו).
Representative Results
בתחילה הצלחנו לאמת את יכולת הקיום של העוברים באמצעות הפרמטרים של הפרוטוקול לרכישת diSPIM (סעיפים 1-6). עשרה עוברים בו ב -20 ° c, אחד נפח/עובר/דקה, משלב 2-cell לשלב 2 קיפול (7.5 שעות, 451 כרכים/העובר). כדי לפקח על חטיבות התאים ברחבי הembryogenesis, השתמשנו במתח BV514, אשר מבטא באופן ברור את Mcherry:: העיתונאיhistone בונה ממערך הטרנסגנים המשולב ujIs11324. איור 6 מראה ציר זמן של המחצית הראשונה של ההתפתחות העובריים עבור אחד מעוברי התמונה. כל תמונה מייצגת הקרנה בעוצמה מקסימלית של תצוגה בודדת (המופקת על-ידי שלבים 7-8) של העובר בתמונה. מצאנו כי הפרוטוקולים אופטימיזציה לא לגרום לפוטורעילות כל גילוי לעוברים, כפי שמוערך על ידי תזמון של חטיבות התא (לא מוצג), זמן הבקיעה, והעיתוי הקשור אבני דרך התפתחותית (איור 6 והפניות1 , מיכל בן 25 , 26).
לאחר מכן להחיל את הפרוטוקול כדי לנתח את הדינמיקה התרחבות של נוירונים בודדים בפיתוח עוברים. אנו התמונה DCR7692 (olaex4655), זן נמטודות של הטרנסגניים המבטא gfp מחוץ נוירופפטיד flp-19 היזם בקבוצת משנה של תאים לא מזוהים (DACR2819, החזית-19 (3.6 kb):: Syn21:: gfp:::p 10 3 ' utr) . בעקבות הצעדים של הפרוטוקול המתואר כאן, קבענו כי התאים בלתי מזוהה מתאים נוירונים מוטוריים RMDDL ו RMDDL, לתא תעלת הפרשות, ועל שני תאי שריר (איור 7). לאחר מכן בדקנו לכמת את הדינמיקה מוצלח של rmddl ו rmddl נוירונים. הבחנו כי rmddl ו rmddl הנוירונים הם מעוצבים בצורה מוקדמת כמו 360 דקות הפריה פוסט, עם ציר הסלולר יותר המייצג את הציר הבא עבור neurite מוצלח (איור 7 והסרט S1). באמצעות "neurite פשוטה העקיבה" תוסף בפיג והחלת אותו 3d השחזורים של כרכים התמזגו איזוטרובאלי, אנחנו לאחר מכן כימות הצמיחה סטריוטיפית של rmddl ו rmddl neurites עבור שישה עוברים. קבענו כי הדינמיקה מוצלח הוקלדה עבור rmddl ו rmddl על פני העוברים (להלן המכונה rmdds). מ 385-410 דקות הפריה הפוסט, RMDDs neurites מורחב 6.0 ± 0.5 μm (פירושו ± SEM; n = 12 neurites) הקדמי של גופי התא (איור 7b, C, I). מ 415-445 דקות הפריה פוסט, שני neurites להאריך דורסלי לתוך וסביב טבעת העצב שוערת (כוכבית באיור 7d). בממוצע, כל rmdd neurite extended 11.0 ± 0.6 יקרומטר (ממוצע ± SEM; n = 12 neurites) מגוף התא לפני שפגש באופן סינכרוני את המקביל הטבעתי בקודקוד הטבעת (איור 7i). חשוב מכך, תוצאות הנציג שלנו להפגין כי אנו מסוגלים לבחון, להשוות ולכמת תכונות התפתחותיות עצבי לתאים לזיהוי יחיד באמצעות פרוטוקול משולב שלנו (איור 7 ואיור 8).
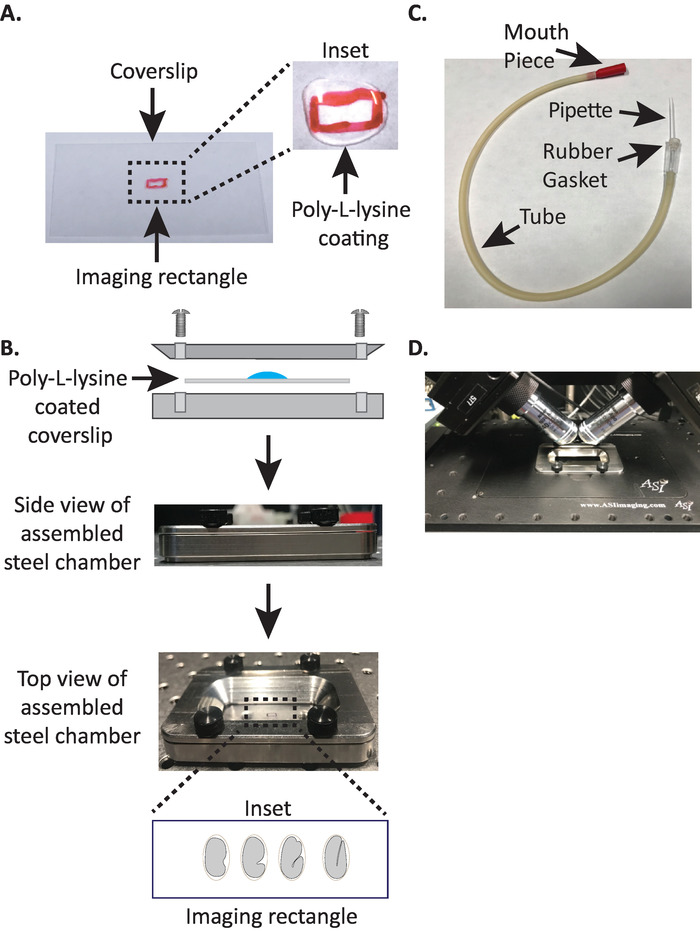
איור 1: הגדרת הרכבה לדוגמה של diSPIM. (א) הכנת שמיכות עם פולי-ל-ליזין. בכניסה, 10 μL של ליזין פולי-L שימש לציפוי הכיסויים במשך 5 דקות. פולי-L-ליזין מאפשר העובר ביצה כדי לתקוע בחוזקה את הכיסויים במלבן. (ב) סכמטי של חדר הדמיה הפלדה והחדר המורכב. בכניסה, עוברים נציגים מכוונים עם הציר האחורי הקדמי בניצב לציר הארוך על הכיסויים. (ג) מורכב שפופרת אספירין עם מיקרוקפילר. (ד) חדר הדמיה פלדה רכוב במחזיק לדוגמה תחת מטרות 40-x לפחות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 2: הגדרת הדמיה diSPIM ארוכת טווח במיקרו-מנהל. (א) מומלץ להפעיל פרמטרים של צריכת חשמל בלייזר (מלבן אדום) הממוטב לדימות ממושך תוך הפחתת רעילות התמונה (כפי שמוערך בשיעור הבקיעה גבוה יותר של עוברי המשך). להגדיר 561 ננומטר לייזר ל 79 μW (0.25) ו 488 לייזר ננומטר ל179 μW (0.5). שים לב שכיול מדויק של הגדרות התוכנה לצריכת חשמל בלייזר משתנה בין התקנות diSPIM. מומלץ למשתמשים למדוד ולכייל את כוח הלייזר כדי להשיג 79 μW (561 nm) ו 179 μW (488 nm) כוח לייזר. (ב) הגדרת פרמטרים עבור שמירת נתונים (מלבן ירוק), הגדרות אמצעי אחסון (ריבוע כחול) והגדרות פרוסה (ריבוע כתום). (ג) dispim פרמטרים פוקוס אוטומטי עבור הדמיה ארוכת טווח של C. אלגיה embryogenesis (ראה שלבים 6.1-6.6). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 3: הדמיית תמונה והתקנת עיבוד נתונים באמצעות ציטוהצג. (א) תמונות Raw שנפתחו על ידי ציטוהצג. ציטוהצג הוא מסוגל לפתוח תמונות שנתפסו על ידי שני נתיבי המצלמה (SPIM A ו-B). תמונות raw אלה נפתחות בחלונות רב-ממדיים שנקראים מוניטור Dispim. במוניטור Dispim, "דפוס עניבת הקשת" נוצר כדי לבחור את הקצוות הקדמי, האחורי, האחוריים והגטיים של העובר (ראה שלב 9.1). בחירות של עניבת פרפר מעידות על כיוון העובר לפירוק ומעקב אחר הזדקנות בסיוע כוכבים. (ב) פרמטרים ממוטבים המשמשים להפקת תמונות איזוטרופיות. במהלך הרכישה בעת רכישת חלון, הגדר את הפרמטרים המצוינים בשלבים 9.5.1-9.5.8. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 4: תצורת מצלמה diSPIM. (א) צילום של מיקומי מצלמה וכיוונים. (ב) תיאור של + 90 מעלות סבבים של Spim A כדי להתאים את Spim B תמונות שנאספו. (ג) אמצעי אחסון לקלט (C) ביחס לכיוון מדד הכיוון + 1 המבוסס על תצורת המצלמה של dispim (ראה שלב 9.5.2). אנחנו מסובבים SPIM תמונה (עם) + 90 מעלות סביב ציר Y לפני ההרשמה להתאים את התמונה SPIM B (s). קנה מידה של סרגלים = 10 μm. תמונות מייצגות תצוגה בודדת, הקרנות בעוצמה מקסימלית ותמונות פירוק של העובר מתקפל 1.5 עם גרעינים עם תוויות (561-nm, אדום) ונוירונים (488-nm, ירוק). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 5: היווצרות ועריכה ג. אלגיה היוחסין העובריים באצטרי. (א) אנו משתמשים במרחרי לערוך את עקבות השושלת של סטארריניט (ראה הפניות5,6,8; כתבי-יד נכללים גם בצרור ההורדות שלנו). מרחרי תציג שמות שיטתית עבור כל גרעין (מלבן ירוק) עם סיום השלבים 10.1-10.2. חלון זה (A) מספק מידע (מלבן שחור) על כל תא בסימן השושלת (ABa, מודגש בכחול) המסייעים למשתמשים להנחות בעת מעקב ועריכה של עקבות שושלת היוחסין. מומלץ למשתמשים לאמת ולהשוות בין תאי שורה ומיקומם לשושלת ה -C. אלגיה שושלת התאים העובריים שדווחה בעבר על ידי sulston et al.1 בנוסף, אם משתמשים מעוניינים לאתר תאים ספציפיים ב שינוי סדרות נתונים (ראה להלן, B) הזינו שם שיטתי של C. אלגיה בסרגל החיפוש (מלבן כתום). (ב) סדרת הנתונים של המשתמש לפירוק נפתחת גם באופן אוטומטי עם סיום השלבים 10.1-10.2. מוצג כאן תמונה התמזגו איזוטרופית של העובר בשלב ארבעה תאים עם גרעינים המסומנים באדום. במהלך מעקב אחר גרעינים, משתמשים צריכים לשנות את עוצמת התמונה (ריבוע אדום) ולנווט בזמן ו-z באמצעות מקשי החצים על המקלדת שלהם (זמן-שמאלה/ימינה, z-up/למטה). (ג) 3d קריקטורה של נקודת הזמן ב (ב) עם פונקציונליות מסוימת (מלבן סגול) המאפשר Rotatable 3d-הדמיה. לקבלת סקירה של מרחרי והפונקציונליות התלת-ממדית שלה, ראה הפניות5,6,8. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 6: הדינמיקה ההתפתחותית של העוברים על הצדדים. הפאנל העליון, תמונות diSPIM המציגה את המחצית הראשונה של התפתחות עובריים עבור אחד העוברים התמונה (זן BV514 ujIs11324). העוברים הועשו ברציפות, כל דקה ל7.5 שעות (בעשרים ° c). שתי התמונות הראשונות של הפאנל העליון מייצגות עוברי 4 ו-8 תאים עם גרעינים (אדום) ועמדות של גופי הקוטב (כדורים אדומים צפופים, מסומנים עם כוכביות כחולות). כל תמונה מייצגת הקרנה בעלת עוצמה מרבית של העובר בתמונה. קנה מידה של סרגלים = 10 μm. ציר הזמן (פס אופקי) מייצג דקות הפריה לאחר ההפריה (m.p.f.) של התפתחות העוברים בתחום הג. אנו מאומתים כי הפרמטרים של הפרוטוקול שלנו עבור רכישת diSPIM לא לגרום לפני התמונות לעבור העוברים התמונה כפי שמוערך על ידי הכדאיות, תזמון חטיבות התא, תזמון של בקיעה ותזמון של אבני דרך התפתחותית (ראה הפניות מיכל בן , מיכל בן 25 , 26). אנו מודעים לכך שהתזמון של אבני הדרך ההתפתחותית מעבר לעוברים עם פרמטרי ההדמיה שלנו (SEM ± 8.174 דקות במשך 6.4 שעות הדמיה של הפעלות ארוכות; n = 10). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

. איור 7 זיהוי תאי ואפיון תא בודד של דינמיקה neurite מוצלח בפיתוח עוברי המשך. הדמיה של צבע כפול של זן שנעשו על ידי חציית BV514 ujIs11324 (עבור הזדקנות) ו DCR7692 (olaex4655), זן נמטודות של טרנסגניים המבטא gfp את המקדם flp הנוירופפטיד -19 בקבוצת משנה של תאים לא מזוהים. (א-ח) בעקבות הצעדים של הפרוטוקול המתואר כאן, קבענו כי התאים בלתי מזוהה מתאים נוירונים מוטוריים RMDDL ו RMDDL (חיצים צהובים), אל תא תעלת הפרשות (חיצים כחולים), ועל שני תאי שריר (חיצים לבנים). (I) הקוונפיקציה של הדינמיקה מוצלח של rmddl ו rmddl הנוירונים באמצעות תוסף פיג'י "neurite מעקב פשוט" והחלת אותו 3d שחזורים של כרכים התמזגו איזוטרופית. שים לב כיצד הפקודה rmddl ו-rmddl מציגות דינמיקה של התרחבות סטריוטיפית, כל הרחבה באופן סינכרוני עבור אורך כולל של 17.7 ± 0.6 μm (ממוצע ± SEM; n = 12 neurites) ומפגש בקודקוד של טבעת העצבים (ראה גם סרט S1). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 8: בחינת תמונות איזוטרופיות של מורפולוגיות עצבי בג עוברים . הדמיית איזוטרופי של AVHL ו-AVHL נוירונים (חיצים צהובים). באמצעות diSPIM, מורפולוגיות עצביים ניתן ללכוד מניב 4-מימדי (4D) תמונות עם רזולוציה מרחבית איזוטרופי של כ 330 ננומטר. ה-diSPIM מאפשר למשתמשים לסובב כמעט אמצעי אחסון של תמונה עם רזולוציה זהה בכל הכיוונים. תמונות ב -D הן הקרנות בעצימות מקסימלית של עוצמת התמונה בעלת אותו מעבר איזוטרופית מסיבובים שונים סביב הציר הארוך של העובר. קנה מידה ברים = 5 μm. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
סרט משלים S1: C. אלגיה עובר פיתוח מ 280 עד 434 דקות הפריה פוסט. הסרט איזוטרופי של זן DCR7692 (olaex4655) הביע ujIs113 באופן מפורש עם תוויות DACR2819 בדלילות rmdd Neurites (איור 7a-D, חיצים צהובים). DACR2819 גם מתייג שני תאי שריר (איור 7a-d, חצים לבנים) ו תא תעלת הפרשות (איור 7a-d, חץ כחול) במהלך התפתחות עובריים (איור 7a-d). קנה מידה ברים = 10 μm. אנא לחץ כאן כדי להוריד קובץ זה.
Discussion
ג. אלגיה בולטת כאורגניזם היחיד עם התפקידים הסופיים והקישוריות של כל תא מבוגר הידוע27. עם זאת, הדינמיקה ההתפתחותית המובילה לארגון מעגלי העבודה והרשתות היוצרת את המערכת מאפשרת להישאר בלתי ידוע. מבוסס על הזדמנויות המתעוררים מתוך התקדמות במיקרוסקופ אור, אנחנו יכולים כעת ללכוד ולנתח את עמדות התא, מורפוגנזה, ו נוירוגנזה ברחבי C. אלגיה התפתחות עובריים.
הנוהל שתיארנו ושאנו שימוש באופן שגרתי במעבדה התשואות 4D-isotropic תמונות של נוירונים וגרעינים שכותרתו עבור הזדקנות התא ב- C. אלגיה עוברים. חשוב מכך, יש לנו אופטימיזציה לטווח ארוך הדמיה התנאים עם diSPIM ומצמידים למחצה אוטומטי יכולות לינארי עם תמונות ברזולוציה גבוהה כדי לשפר את המהירות והדיוק של ניתוח C. אלגיה embryogenesis. פרוטוקול משולב זה יאפשר למשתמשים להמחיש ולזהות תאים ולכמת תכונות תלת ממדיות כגון הגירה neurite ו-אקסון באמצעות תחילתה של העווית המוקדמת. הליך זה יכול להיות מותאם בקלות לתוך כל מתקן עם מערכת הספק אסי diSPIM, ואנו ממליצים על מערכת זו במיוחד עבור פרוטוקול זה. ניסוחים SPIM אחרים המוצעים באופן מסחרי עשויים להיות שונים מתצורת ASI בתכונות החדר והאופטי לדוגמה. עם זאת, נתונים המיוצאים מפלטפורמות אחרות ניתן גם לשים דרך צינור הנתונים שלנו. לכן, הערכת הערך שלהם בהזדקנות, מבחן תובעני של איכות התמונה ויציבות הכלי, הוא אפשרי. למרות שאנו משתמשים באופן פעיל ב-dispim כדי לדמות בקביעות דגימות אחרות (כגון דרוזופילה ועוברי דגים), ניתוח העוברים לפני הזמן המתואר והמקיף של העוברי עדיין מוגבל למינים מסוג נמטודות. לדגימות גדולות או עבות, אנו בוחרים להשתמש בגישות לסריקת במה, הסורקים את הדגימות דרך גיליון אור נייח. קומאר ואח ' הפגינו בעבר את הפרטים המשופרים הללו כדי להניב תמונות באיכות גבוהה מדגימות עבות ללא שינויים נוספים ב-diSPIM10.
השלבים הקריטיים בפרוטוקול כוללים את העוברים הבאים של C. אלגיה באמצעות שמיכות הציפוי פולי ליזין, רכישת נתונים ועיבוד נתונים. קציר והרכבה C. אלגיה עוברים על שמיכות זכוכית יכול להיות מאתגר למשתמשים לא מנוסים, אבל כאן אנו מספקים פרוטוקול מפורט של שלבי מפתח כדי להקל על הלמידה. אם הדמיה ארוכת טווח רצוי, אנו משיגים תוצאות הטוב ביותר קצירת ארבעה תאים או עוברים מוקדם מ 8-10 צעירים מבוגרים28. שימו לב כי המבוגרים הוותיקים פחות רצוי לקצור עוברי הבמה המוקדמים, משום שהם נוטים להכיל עוברים ישנים יותר ברחם ובביצים לא מופרות. לגבי העוברים, בעיות כגון סתימה בתוך מייבש הבית (פיפטה הפה) או גדול מדי של פתיחה בתוך הפיפטה מיקרוקפילר עשוי למנוע הרכבה נכונה וכיוון העוברים. כדי להתכונן לדימות אופטימלי, אנו מבצעים טרום רכישה בדיקות על עוברים מוקדם ומאוחר מתעוות לבדוק את הביצועים של יריעות אור, מצלמות, מטרות, פוקוס אוטומטי. אנו משיגים את התוצאות הטובות ביותר כאשר כל הפעולות הללו נבדקות ומפיקות תמונות באיכות גבוהה במהלך בדיקות טרום הרכישה שלנו. דבר זה רלוונטי במיוחד ליצירת תמונות עם פתרון מרחבי איזוטרופי, שעבורם תמונות raw שנרכשו משתי התצוגות (מטרות) חייבות להיות באיכות גבוהה. לאחר הרכישה, אמצעי האחסון שנרכשו עבור כל תצוגה מעובדים כדי להניב תמונות איזוטרופיות. חשוב להשתמש בכרטיס של יחידת עיבוד גרפית (GPU) מתאימה כמתואר בפרוטוקול זה (ראה להלן). פעולה זו משפרת את מהירות העיבוד שבה נוצרות התמונות האיזוטרומיות, קיצור הזמן לניתוחי נתונים. זה גם הכרחי כי משתמשים מריצים את הגירסה העדכנית ביותר של ציטוהצג והם משתמשים בפרמטרים שסופקו עם צרור ההורדה שלנו עבור הזדקנות אוטומטית StarryNite. אם משתמשים מעוניינים להשתמש באופן אוטומטי לבחירה עבור דגימות אחרות (למשל, zebrafish, דרוזופילה ילה וכו ') אז אופטימיזציה נוספת לפרמטרים המשמשים starrynite יידרש (ראה הפניות3,4).
למרות שהפרוטוקול המשולב שלנו מספק תמונות ותוצאות הזדקנות בעובר מראש, משתמשים צריכים להיות מודעים לכך ששינוי אוטומטי בעובר לאחר העווית אינו אפשרי כרגע: שינויים בתנוחות גרעיניות בסדר השניות ב עובר לאחר העווית, מהר מדי. כדי לאפשר מעקב אחר השושלת עם זאת, הדגים אכן הפגינו יכולת מבטיחה ללכוד אירועים נוירוהתפתחותיים ולעקוב אחר מיקומי תאים בשלבים שלאחר העווית של embryogenesis23,29. אם משתמשים מעוניינים לבחון את העובר לאחר העווית, הפונקציה מספקת את המהירות להשגת תמונות נפחי ולעקוב אחר אירועים נוירוטיביים משובחים, כגון neurite outgrowth, בעוברים במהירות.
פרוטוקול זה יהיה היסוד עבור השלמת תא אחר התא של המדריך לתולעים האטלס30, כפי שהוא יספק גישה משולבת עם ברזולוציה גבוהה איזוטרופי תמונות לזהות וללכוד 3d מורפולוגיות של נוירונים התווית במהלך את 430 הדקות הראשונות של הembryogenesis. כפי שהוא עומד, אב טיפוס האטלס מדריכים מספק עמדות גרעיניות של תאים בעובר המתפתח ומטרתו ללכוד את הדינמיקה ההתפתחותית של תת-ערכה של נוירונים עובריים. פרוטוקול זה יהיה מפתח עבור שילוב של הנוירונים המפתחים נוספים לתוך מדריכי התולעת אטלס30.
הפרוטוקול המשולב שלנו גם יפשט את הביקור בפרופילים חדשים של ביטוי גנים בעובר הג. באופן טרנסגניים, הרבה מיזמים ספציפיים לתאים ולשלוט באופן זמני בביטוי הטרנסגנטי. בעוד דפוסי הביטוי של רוב הגנים התאפיין בהרחבה בחיה למבוגרים31,32,33,34, כמעט כולם עדיין להיות מאופיין בפיתוח (במיוחד העובר בשלב מאוחר). מקדם ה -C. אלגיה מהווה משאב שימושי לקהילת התולעים כדי לכונן ביטוי העברה ספציפי לתאים, כמו גם לקבוע אם פונקציית גן היא אוטונומית או לא אוטונומית. לכידת איזוטרופי דפוסי ביטויים דינמיים ודינאמיים של גנים, ומזהה בדיוק לבטא תאים באמצעות הזדקנות יהיה ערך לרבים בקהילה המדעית.
Embryogenesis כולל שני תהליכים עיקריים שזורים, בידול סלולרי רקמה מורפוגנזה. הרבה ידוע על המנגנונים והמולקולות המגדירים סוגי תאים ברורים במהלך התפתחות הג. עם זאת, מעט ידוע על המנגנונים החשובים להעברת תאים, הדבקה בתאים וצורת תא בעובר C. אלגיה . עם כללי השושלת של ה- C. אלגיה הידועה, הפרוטוקול שלנו מאפשר לנו להבחין בקלות את 3d-מיקרואנטומיה מקוטלגים של העובר במהלך מורפולגנזה ברמות חדשות של פירוט: למשל, האקסון, synaptogenesis, ופעילות עצבית. ארדיאל ואח ' הפגינו בעבר את הכוח של ה-diSPIM ללכוד מעבר סידן ברמה של נוירונים בודדים ב -C. אלגיה עוברים23. היבטים רבים אחרים של הפיזיולוגיה ההתפתחותיים בשלים לחקירה על ידי שיטות אלה.
לבסוף, פרוטוקול זה הוא אוטומטי ברובו באופן שיטתי מפחית את הזמן שלוקח כדי ליצור תמונות לפירוק ולבצע הזדקנות תא באמצעות StarryNite ו מרחרי. את אסטרטגיות התוכנה המשמשות בפרוטוקול זה ניתן להחיל על שאלות רבות של ביולוגיה הרחק מתוך השדות הספציפיים מאוד שבו הדגמנו אותם כאן.
פרטים על תאימות תוכנה וגישת הורדה
מידע על מיקרו-מנהל ותוספים עבור הדמיה של diSPIM זמינים ב http://dispim.org/software/micro-manager ו-https://micro-manager.org/wiki/ASIdiSPIM_Plugin.
צינור עיבוד הנתונים מחייב כעת מערכת הפעלה של Windows. יש לנו ארוזות קובץ ארכיון יחיד כדי לפשט את ההתקנה של כל הדרושים עיבוד נתונים תוכניות ותמיכה קבצים. הוא זמין להורדה ב http://dispimlineage.wormguides.org.
ציטוהצג (http://run.cytoshow.org/) מבוסס על פלטפורמת ניתוח תמונת קוד פתוחה בשימוש נרחב, ImageJ (v1). על java להיות מותקן ומעודכן במחשב לשימוש ב-ציטוהצג, ועדכונים ל-ציטוהצג נפרסים באופן אוטומטי באמצעות הפעלת אתר Java. פונקציות מבוססות ImageJ רבות של ציטוהצג הן כמתואר ומומחשות ב-https://imagej.nih.gov/ij/docs/examples/index.html. ציטוהצג הותאם אישית כדי להציג נתונים גולמיים רב ממדיים מ-ASI diSPIM, כמו גם תוכנות דימות אחרות היוצרות פלט TIFF. בעיקרון, מערכות מרובות תצוגה SPIM הדמיה יכול להיות נתמך על ידי שינויים משניים של ציטוהצג כדי לאפשר פרוטוקול זה להתבצע על מערכות מיקרוסקופ שונים.
SpimFusion נכתב ב CUDA/C++ באמצעות Visual Studio 2013 עם ערכת הכלים של CUDA v 7.5. הפעלת SpimFusion דורש חומרה מחשב ספציפי: יחידת עיבוד גרפיקה של NVIDIA (GPU) כרטיס עם היכולת לחישוב CUDA 1.0 או יותר ומינימום של זיכרון כרטיס גרפי 2 GB. בזמן הפרסום של הפרוטוקול שלנו, SpimFusion הוא לא פורסם (מין גואו ו הארי Shroff) אבל זמין בארכיון צרור התוכנה שהוזכרו לעיל.
גירסה מונחית של שורת פקודה מוכללת של StarryNite מחייבת שהתקנת MATLAB קומפיילר זמן הפנוי הזמינה באופן חופשי, אך אינה דורשת רשיון עבור תוכנת MATLAB מסחרית. זמן הריצה של קומפיילר MATLAB נכלל בארכיון חבילת התוכנה שהוזכר לעיל. הקוד עבור סטארריניט כפי שנעשה בשימוש בפרוטוקול זה הוא ביסודו של דבר ללא שינוי ששימש עבור תמונות מיקוד6. עם זאת, כמה עניינים מבצעיים ביצירת תמונות קלט עבור העיבוד StarryNite והטיפול של התוצאות StarryNite טופלו כאן על ידי שיטות ב ציטוהצג המאפשרים צינור עיבוד נתונים רציפה עבור מחובר איזוטרופי diSPIM אמצעי אחסון. שינויים אלה אוטומטיים באמצעות קוד ציטוהצג המטפל בצעדים אלה לפני ולאחר עיבוד. בנוסף, ציטוהצג עורך את הפרמטר המותאם לפני הגדרת מיטוב של תבנית מותאמת להגדרה באופן אוטומטי כדי לכוונן את האלגוריתם של הפילוח לעוצמת הקרינה הפלואורסצנטית בנתוני התמונה. הפרמטרים הייחודיים המשמשים את סטארריניט בכל ערכת נתונים של diSPIM נשמרים לאחר מכן בקובץ יחד עם תמונת הפלט ונתוני השידור.
גירסה מותאמת אישית של מרחרי הפועלת עם תמונות של 16 סיביות ושומרת על תאימות עם Java3D רינדור מתאימה ביותר לפרוטוקול זה. הוא נכלל גם בארכיון צרור התוכנה שהוזכר לעיל.
Disclosures
. למחברים אין מה לגלות
Acknowledgments
אנו מודים לג מאריי על זן משולב, ujIs113, כדי לייצר זן הזדקנות BV514; ברנדון הארווי (NIBIB) לקבלת עזרה בבדיקת הפרוטוקול; ג'ון דניאלס וגרי רונדו (מיכשור מדעי יישומי) לסיוע בכלי מיקרו-מנהל ומכשיר diSPIM; ואנדרו יורק והאנק עדן על המשוב הקריטי שלהם על מערכת ה-diSPIM. כמו כן, אנו מודים למרכז המחקר לתכנית מוסדות המיעוט ולמכון לנוירוביולוגים חוסה דל קסטיליו (אוניברסינדה דה פוארטו ריקו) למתן מפגש ומערכת סיעור מוחות. הרבה מהעבודה הזאת נערכה במעבדה הביולוגית הימית ביער הול באמצעות תוכנית ויטמן. עבודה זו נתמכת על ידי תוכניות מחקר הפנים של המכון הלאומי NIH של הדמיה ביו-רפואית וביוהנדסה ועל ידי NIH מענק לא. U01-HD075602 ו-No. R24-OD016474. מארק W. Moyle נתמך על ידי F32-NS098616 ו לייטון ה. דאנקן נתמך על ידי תוספת גיוון ל R24-OD016474.
Materials
| Name | Company | Catalog Number | Comments |
| Steps 1-4 | |||
| Concavity slides | ThermoFisher Scientific | 1519006 | 5-18mm diameter, 0.6-0.8mm deep, 1.2-1.5mm |
| Dissecting microscope with 10×–50× zoom range | Motic | SMZ-171 | |
| E. coli (OP50) | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Glass coverslips, no. 1.5, 24 × 50 mm | VWR International | 48393-241 | |
| M9 Buffer | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Methyl cellulose | Sigma-Aldrich | H7509-25G | |
| Microcapillary pipette aspirator tube | Sigma-Aldrich | A5177 | |
| Microcapillary pipettes, 0.4-mm i.d | Drummond Scientific | 1-000-800 | |
| Needle, no. 18G x 1 ½ (1.2mm x 40mm) | BD Precision Glide | 305196 | |
| NGM plates | prepared as described by Brenner (1974) | ||
| O-ring for imaging chamber | O-Rings West | M1.5X40 | |
| Pasteur pipette | Corning/Sigma-Aldrich | CLS7095D5X | |
| Platinum wire, 0.5-mm diameter | Sigma-Aldrich | 267201 | |
| Poly-L-lysine | Sigma-Aldrich | P1524 | |
| Stainless steel rectangular chamber (76.0 mm x 50.5 mm) | Applied Scientifics Instrumentations (ASI) | I2450 | |
| Worm Eyelash Pick | Hart, A. C. Behavior. WormBook. (2006). | ||
| Worm Pick | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Name | Company | Catalog Number | Comments |
| Steps 5-6 | |||
| 488 nm long-pass filter | Semrock | LP02-488 RU-2 | |
| 561-nm notch filter | Semrock | NF03-561E-25 | |
| BLP02-561R-25, quantity 2 | Semrock | 561 nm EdgeBasic best-value long-pass edge filter | |
| Control software for bottom camera | Jenoptik | ProgRes CapturePro | |
| diSIPM assembly video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/TAgbr6IrTqw ; http://www.asiimaging.com | |
| diSPIM alignment video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/qnOrg30NNuE | |
| diSPIM imaging PC | Intel | Intel Xeon CPU E5-2630 2.6GHz, 12 cores in total, 64 GB memory, Windows 7 | |
| FF01-525/45-25, quantity 2 | Semrock | 525/45 nm BrightLine single-band bandpass filter | |
| FF555-DI03-25X36, quantity 2 | Semrock | 555 nm edge BrightLine single-edge dichroic beamsplitter | |
| Imaging PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Kumar et al diSPIM Setup | Applied Scientifics Instrumentations (ASI) | Instrument setup for this protocol is identical to Kumar et al 10,11 diSPIM, which makes use of 40x 0.8NA water immersion lenses for imaging. (See steps 5.1 and note) | |
| Micro Manager | Micro-Manager | https://micro-manager.org/ | |
| Modifications to Kumar et al diSPIM Setup (see below) | |||
| Optical table with isolators, 4 feet × 6 feet × 12 inches | TMC | 784-651-02DR and 14-416-34 | |
| Name | Company | Catalog Number | Comments |
| Steps 7-10 | |||
| Analysis PC | Intel | Intel Core i7-8700K CPU 3.70GHz, 6 cores in total, 64 GB memory, Windows 10 | |
| Analysis PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Installation instructions | Software bundle | http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm | |
| Software bundle | Software bundle | http://dispimlineage.wormguides.org |
References
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology. 100 (1), 64-119 (1983).
- Bao, Z., et al. Automated cell lineage tracing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 103 (8), 2707-2712 (2006).
- Santella, A., Du, Z., Bao, Z. A semi-local neighborhood-based framework for probabilistic cell lineage tracing. BMC Bioinformatics. 15, 217 (2014).
- Santella, A., Du, Z., Nowotschin, S., Hadjantonakis, A. K., Bao, Z. A hybrid blob-slice model for accurate and efficient detection of fluorescence labeled nuclei in 3D. BMC Bioinformatics. 11, 580 (2010).
- Boyle, T. J., Bao, Z., Murray, J. I., Araya, C. L., Waterston, R. H. AceTree: a tool for visual analysis of Caenorhabditis elegans embryogenesis. BMC Bioinformatics. 7, 275 (2006).
- Katzman, B., Tang, D., Santella, A., Bao, Z. AceTree: a major update and case study in the long term maintenance of open-source scientific software. BMC Bioinformatics. 19 (1), 121 (2018).
- Murray, J. I., et al. Automated analysis of embryonic gene expression with cellular resolution in C. elegans. Nature Methods. 5 (8), 703-709 (2008).
- Murray, J. I., Bao, Z., Boyle, T. J., Waterston, R. H. The lineaging of fluorescently-labeled Caenorhabditis elegans embryos with StarryNite and AceTree. Nature Protocols. 1 (3), 1468-1476 (2006).
- Wu, Y., et al. Spatially isotropic four-dimensional imaging with dual-view plane illumination microscopy. Nature Biotechnology. 31 (11), 1032-1038 (2013).
- Kumar, A., et al. Using Stage- and Slit-Scanning to Improve Contrast and Optical Sectioning in Dual-View Inverted Light Sheet Microscopy (diSPIM). The Biological Bulletin. 231 (1), 26-39 (2016).
- Kumar, A., et al. Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nature Protocols. 9 (11), 2555-2573 (2014).
- Wu, Y., Christensen, R., Colon-Ramos, D., Shroff, H. Advanced optical imaging techniques for neurodevelopment. Current Opinion in Neurobiology. 23 (6), 1090-1097 (2013).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17708-17713 (2011).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136 (12), 1963-1975 (2009).
- Stelzer, E. H. Light-sheet fluorescence microscopy for quantitative biology. Nature Methods. 12 (1), 23-26 (2015).
- Winter, P. W., Shroff, H. Faster fluorescence microscopy: advances in high speed biological imaging. Current Opinion in Chemical Biology. 20, 46-53 (2014).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. Astronomical Journal. 76 (6), 745-754 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. JOSA. 62 (1), 55-59 (1972).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Hart, A. C. Behavior. WormBook. , (2006).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. Chapter 14, Unit14 20 (2010).
- Gualda, E. J., et al. SPIM-fluid: open source light-sheet based platform for high-throughput imaging. Biomedical Optics Express. 6 (11), 4447-4456 (2015).
- Ardiel, E. L., et al. Visualizing Calcium Flux in Freely Moving Nematode Embryos. Biophysical Journal. 112 (9), 1975-1983 (2017).
- Walton, T., et al. The Bicoid class homeodomain factors ceh-36/OTX and unc-30/PITX cooperate in C. elegans embryonic progenitor cells to regulate robust development. PLoS Genetics. 11 (3), e1005003 (2015).
- Altun, Z. F. WormAtlas. , (2002).
- Wood, W. B. Embryology: In the nematode C. elegans. Cold Spring Harbor Laboratory Press. Chapter 8, 215-241 (1988).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314 (1165), (1986).
- Bao, Z., Murray, J. I. Mounting Caenorhabditis elegans embryos for live imaging of embryogenesis. Cold Spring Harb Protoc. (9), (2011).
- Christensen, R. P., et al. Untwisting the Caenorhabditis elegans embryo. eLife. 4, (2015).
- Santella, A., et al. WormGUIDES: an interactive single cell developmental atlas and tool for collaborative multidimensional data exploration. BMC Bioinformatics. 16, 189 (2015).
- Dupuy, D., et al. A first version of the Caenorhabditis elegans Promoterome. Genome Research. 14 (10B), 2169-2175 (2004).
- Reece-Hoyes, J. S., et al. Insight into transcription factor gene duplication from Caenorhabditis elegans Promoterome-driven expression patterns. BMC Genomics. 8 (27), (2007).
- WormBase. , Available from: https://www.wormbase.org (2019).
- Lee, R. Y. N., et al. WormBase 2017: molting into a new stage. Nucleic Acids Research. 46 (D1), D869-D874 (2018).



