Summary
ここでは、 elegans胚における高分解能顕微鏡、計算ツール、および単一細胞標識法を用いて、neurodevelopment 中の単一細胞動態を理解するための組合せアプローチを提示する。
Abstract
Caenorhabditis elegans(C. elegans) は、生体内で単一細胞の分解能で、神経系全体の細胞起源を理解するという挑戦が観察できる唯一の有機体として際立っている。ここでは、 elegans胚に neurodevelopment を検査するための統合プロトコールを提示する。我々のプロトコルは、現像胚における単一細胞の画像化、lineaging および neuroanatomical トレースを組み合わせたものである。我々は、二重視野反転選択的平面照明顕微鏡 (diSPIM) の使用を通じて、ほぼ等方性空間分解能を有する生きたelegans胚の長期にわたる4次元 (4d) 画像化を達成する。線虫の胚における核とニューロンの構造は、すべての三次元で ~ 330 nm の分解能で画像を対しに融合して撮像される。次に、この分単位の高解像度4D データセットを分析して、単一細胞および細胞内レベルでの決定的な細胞系列の同一性と遺伝子発現および形態学的ダイナミクスを相関させます。私たちのプロトコルは、記述された各ステップのモジュラー実装を可能にし、胚発生、遺伝子発現、または neurodevelopment に関する研究を強化するように構成しています。
Introduction
Elegansは、胚のすべての細胞が neurodevelopment を通して観察することができる唯一の生物として際立っている。細胞系列全体が既知で不変の1であり、胚の単一細胞の標識および連続画像化を可能にする新しいツールの開発とともに、生物学者は、線虫の神経の発達における異なるステップを検討することができるようになりましたすべての角度からのシステム-細胞誕生;移行と差別化;神経突起の形成、標的化した伸長および手筋;シナプス形成;機能回路のチューニング。Elegans胚におけるニューロン伸長ダイナミクスの捕捉は、レポーター及び蛍光顕微鏡を安定的に発現させることにより、科学界にとって貴重である。
C. elegansにおける発達研究は、しばしば、この種の不変の細胞系譜および細胞運命の地図を利用して、無傷の生物体1内の単一細胞レベルでの文脈的理解を増大させる。自動 lineaging 分析-StarryNite2、3、4 、AceTree5、6、7、8ソフトウェアを使用-高コントラスト、高解像度からの利点蛍光核の画像。最適に働くために、StarryNite/AceTree はまた開発の間にイメージされた胚の予測可能で限られたオリエンテーションによって決まる。共焦点顕微鏡は、2つのカバースリップ間で圧縮されたelegans胚において行われる、高コントラスト/高解像度および予測可能な拘束の両方を提供するので、10年以上にわたって標準のオート lineaging 顕微鏡法である胚の配向は7,8.我々は、以前、 C elegans胚形成などのライブサンプルイメージングのための新規ライトシートベースのデュアルビュー反転選択的平面照明顕微鏡 (diSPIM) の構築と使用について説明しました9,10,11,12,13. ライトシートの顕微鏡検査は、一般に、生きている3d 標本14,15の低い光毒性、高速および長期イメージ投射を提供する。DiSPIM 法は、具体的には、約 330 nm9のほぼ等方性の空間分解能を有する4次元 (4d) 画像を生成する。
共焦点顕微鏡検査と比較されて、diSPIM はより高い信号対雑音および速度を提供し、より等方性の空間分解能、および長期インビボのイメージ投射16のためにより適している。そこで diSPIM データを StarryNite/AceTree に適合させることに取り組み、lineaging 分析を強化するかどうかを検討した。主なハードルは、diSPIM 標本が卵殻圧縮によって容易に StarryNite/AceTree に期待される向きをとることができないことである。解析中のボリューム内のセル位置のランダムな方向は、自動 lineaging 解析の精度を低下させる。
従って私達は CytoSHOW を、ユーザーが diSPIM のイメージの前処理の間に胚の精密な3D オリエンテーションを選択することを可能にし、StarryNite に入力のための質の最大限に活用され、文脈に対応しているイメージデータを降伏させる視聴者ガイド付きユーザー・インタフェースを採用した/AceTree.画像化された胚のユーザ選択に際して、CytoSHOW は自動データ処理パイプラインを指揮する。トリミングされた、背景を減算した胚画像は、各位置、timepoint およびビューのための TIFF スタックファイル内に保存します。次に CytoSHOW は、そのプログラムを SpimFusion して、2つの前処理されたビューを共同登録し、共同 deconvolve して、等方性高解像度の体積画像を生成するためにリチャードソン-ルーシー17、18アルゴリズムを使用します。DiSPIM 固有のパラメータセットは、StarryNite が対し融合画像の画像セグメンテーションと核追跡の際にその動作を制御するために最適化されています。その後、融合イメージと lineaging 結果は AceTree を使用して編集されるため、ユーザーは StarryNite によって生成された自動系統トレース内のエラーを特定して修正することができます。AceTree はまた、胚の中の追跡された核の系譜木と3D モデルのレンダリングを提示することができます。スピムカメラの raw 画像と比較して、対しの融合画像を使用することで、lineaging の速度と精度が著しく向上することがわかります。私たちのプロトコルは、ここで説明するelegansアプリケーション用に最適化されている間、一般的に他の種または標本のために生産 diSPIM データの自動 lineaging に適応することができます。これがプロトコルの使用目的である場合は、3,4に記載されている新しい標本に対して、StarryNite パラメータの追加の調整が必要になる可能性があることに注意してください。
このプロトコルの実装を成功させることは、4D 等方性の分解能を持つ画像になり、生物学者が細胞系譜をトレースし、同時にelegans胚のニューロンを同定および分析することができる。さらに、いくつかの後処理アルゴリズムをマージすることにより、ハードウェアアクセラレーションがこれらの最も時間のかかるものであることから、生きている胚の細胞間の詳細とセルの系譜と細胞の運命の両方を本質的にリアルタイムで分析できるようになりました。この新しいプロトコルは、生体内の分化および形態形成の証拠研究中に、正確で情報に基づいた操作および細胞挙動の観察を可能にする。本稿では、胚発生、遺伝子発現または neurodevelopment の研究を強化するために、 elegans胚の開発における lineaging と細胞追跡のために開発したプロトコルの改良について詳細に説明する。
Protocol
1. ポリ-L-リジンコーティングされたカバースリップで diSPIM スチールイメージングチャンバーを組み立てる
注: 以下のステップはすべて、StarryNite/AceTree による線虫胚の系譜分析を最適化し、自動化するために必要です。Elegansの細胞系譜のトレースを必要とする実験では、いくつかのオプション (そのように示されている) を省略することができます。
- きれいな長方形のカバースリップ (1.5、24 mm x 50 mm) の中央に小さな長方形 (2mm x 5mm) を、油性 (または類似のペン) で描きます。
- マークされていない側にカバースリップを裏返し、ポリ L-リジンの10μ l を置きます (猫.マークされた長方形の上に P1524)。
注: 濾過水 (または同等物) に溶解した 1mg/mL のポリ L-リジンの作業濃度を確認してください。長期保管の場合は、5 ~ 10 mL のアリコートを用意し、−20° c で保管してください。解凍したら、アリコートを室温 (23 ° c) で3〜4週間保存することができます。 - ポリ L-リジンがカバースリップを5分間コーティングすることを許可します (図 1a)。カバースリップは、胚が取り付けられるガラスカバースリップをコーティングするために使用され、目的が画像化媒体に浸漬されている場合でも、受精卵がしっかりと粘着剤にくっつくことを可能にします。
- 鋼鉄イメージの部屋の下半分で塗られるカバースリップを塗ったポリ L リジンを置きなさい。
- スチールイメージングチャンバーの上半分をカバースリップで下半分に置き、チャンバーに関連付けられた4本のネジで締め付けます。側面から目視で、上半分が下半分に均等に収まっていることを確認します (図 1b)。
- 7 ~ 8 mL の M9 バッファー19を使用して、初期段階の胚が異常な浸透圧に屈しないようにする等張性バッファーを入れます。1細胞、さらには2細胞胚も osmotically 感受性であり、この等張バッファーでない場合には異常に発症することがある。また、M9 が水で代用された場合、胚は3段階で停止する傾向があります。
2. マウント用elegans胚サンプルの調製
注: 画像化の約18時間前に、5人の若年 (最終脱皮から1日) の成人および10回の幼虫期 4 (L4) c. elegansを、大腸菌株 OP50 で播種した線虫成長培地 (NGM) 寒天プレートにピックする。プラチナワイヤーピックは、動物19に害を与えずに幼虫と若年成人c. elegansを移動するために使用されます。
- 1% メチルセルロースを準備します (cat.H7509) を M9 バッファーに収めたソリューションです。
注: メチルセルロースは、溶解するまで温かい M9 で撹拌しなければなりません。準備ができたら、この解決は室温で貯えることができる。 - 凹面の顕微鏡のスライドの不況に 1% のメチルセルロース・イン・ M9 の解決の500μ l を加えなさい。この粘性バッファーは、1) NGM から直接採取した後期胚を洗浄するときに、成虫の切開によって初期胚を採取し、2) の2つの機会に使用されます。
注: メチルセルロースは、胚が顕微鏡スライドに固着するのを防ぐために使用されます。 - 後期段階の胚を画像化するために、 elegans胚 (好ましくは若年成人が存在する NGM プレートから) を使用してまつ毛ピックを用い、胚を凹顕微鏡スライド上の 1% メチルセルロースに移動させる。睫毛ピックは、力を減らし、処理中の胚のストレスや損傷を最小限に抑えるのに役立ちます。まつげのピックを作るための手順は、Hart20によって覆われています。
- 2回目のまつげピック (反対側の手) を使用して、両方のまつげを優しくタップして、メチルセルロースの胚を一時停止します。
- 任意: StarryNite を用いた胚細胞の系譜を計画する場合は、1細胞から4細胞胚までを実装する必要があります。これを行うには、まず NGM プレートから若年成人を選択し、白金ワイヤピックを使用して、凹形顕微鏡スライド上の M9-メチルセルロース溶液にそれらを移動させます。
- 任意: 皮下注射針 (18G x 1 1/2) の先鋭化された先端を使用して、中間体で動物の横向をスライスして、1細胞から4細胞胚までを放出する。
3. 口のピペット: microcapillary ピペットで吸引チューブを組み立てる
注: チューブのゴム製ガスケットに手で引っ張られた microcapillary ピペットを挿入した吸引管を使用します。これにより、バッファー充填イメージングチャンバー内のポリ L-リジンコーティングされた表面に解剖スライドから胚を移動することができます。
- 手動で開いた炎の上に microcapillary のピペットを引き、伸張された先端との2つの半分を作成しなさい。
- Microcapillary ピペットの半分を取り、吸引チューブのゴムガスケットにブラントエンドを挿入します (図 1c)。Microcapillary ピペットの残りの半分は、後で使用するために脇に置きます (必要な場合)。
- Microcapillary のピペットを取り付けた組み立てられた吸引管を使用して、microcapillary ピペットの先端を静かに破り、約1〜2個の胚に合う開口部を作成します (ここからこの装置では「口ピペット」と呼ばれています)。
4. カバースリップコーティングされたポリ L リジンにelegans胚を取り付けます
- 吸引器のマウスピースを歯の間に優しく保持した後、microcapillary ピペットを 10 ~ 15 μ l の M9 バッファで事前に充填し、凹状のスライドから毛細血管にそっといくつかの胚を吸引します。
- 胚を M9 バッファで満たされたスチールイメージングチャンバーに移し、胚がカバースリップの中央の矩形に落ちるように毛細血管先端を位置決めする。
- 胚の損傷を避け、ゆっくりとまつげのピックでそれらを動かすか、または口のピペットを使用して胚を垂直に配置し、胚の長軸がカバースリップの長軸に垂直になるように胚を配向させる (図 1bのインセット、下部パネル) があります。
注: 胚をこの方向に配置すると、画像までのスライス数が最小限に抑えられるため、光量とデータ処理時間が短縮され、集録速度が向上します。 - スチールイメージングチャンバーを顕微鏡ステージ上のサンプルホルダに入れます (図 1d)。
5. diSPIM を使用した胚イメージングのためのアセンブリ、ソフトウェアセットアップおよびレーザー最適化
- クマール et al.10,11および http://www.dispim.org で市販部品から繊維結合 diSPIM 全体を組み立てる方法についてのステップバイステップの説明を参照してください。DiSPIM を組み立てる方法のビデオプロトコルは、ASI のウェブサイト (http://www.asiimaging.com) でも入手できます。
注: このプロトコルのための器械の組み立ては diSPIM et al.10,11のイメージ投射のための40〜 0.8 na の水浸レンズを使用すると同一である。このプロトコルのセットアップとクマール et al.10,11の間の唯一の違いは、ダイクロイックミラーミラー (560 nm での分割) と、画像分割装置 (モデル A12801) の内部での赤と緑のバンドパスフィルタの追加です。両方の diSPIM イメージングアーム。画像分割光学の追加により、同じカメラチップの2つの部分に放出バンドを分離することにより、561 nm および 488 nm レーザーで励起された2つの異なる蛍光色素からの画像を同時にキャプチャすることができます。 - インストルメント組み立ての後、イメージング前に diSPIM の光学的アライメントを確認します。
注: diSPIM を適切に位置合わせするには、https://youtu.be/qnOrg30NNuE およびハードウェア情報、http://dispim.org/hardware/objectives、および http://www.asiimaging.com を参照してください。 - ハイスループットセルラーイメージング22のための動作ライトシート顕微鏡用に最適化されたオープンソースプラットフォームマイクロマネージャー (https://micro-manager.org/)21を使用してください。我々は、説明したように30個までの胚の同時画像化を可能にするマルチポジション取得のための ASI diSPIM プラグインの使用を推奨する。
- マイクロマネージャーオープンでは、488 nm と ~ 79 Μ w (0.25) のために179Μ w (0.5) を 561 nm (図2a、赤の長方形) にレーザー強度を設定します。
注: これらは1分間隔を使用してc. elegansの胚の長期イメージ投射のための推薦された設定である。長期間のデュアルカラーイメージングでは、561 nm レーザーを使用して核 (mCherry:: ヒストン) を画像化し、胚がビーン段階になるまで、その時点で 488 nm レーザーがオンになり、GFP 標識ニューロンも画像化されます。これらの撮像条件は、光毒性を最小限に抑え、胚の生存と孵化を確保するために最適化されていますが、長期間 (12-14 時間) 神経発達および lineaging データの継続的な取得を可能にします。 - マイクロマネージャーでは、diSPIM > デバイス >コントロールのメニュープラグインを選択し、asi diSPIMウィンドウを開きます (図 2b)。[取得] タブを選択します。このタブの[データの保存設定] セクション (緑色の四角形)、[ボリューム設定] セクション (青い四角)、および[スライス設定] セクション (オレンジ色の四角) で、各パラメータが図2b に示すように設定されていることを確認します。
注: 当社の画像解析ソフトウェア CytoSHOW は、一括連結青梅 TIFF ファイルシリーズや、マイクロマネージャに組み込まれたエクスポート機能を使用して取得後に作成された TIFF スタックファイルシリーズなどの他のオプションの出力データ形式で動作するように調整されています。通常は、生データが取得されるとすぐに画像ボリュームのリアルタイム表示と処理が可能になるため、シングルタイムポイントスタックの照合青梅-TIFF ファイルデータ形式が使用されます。
6. elegans胚の長期イメージングのための最適化されたオートフォーカスパラメータ
- マイクロマネージャーのオートフォーカスパラメータを、 elegans胚の長期系統品質 diSPIM に最適化された設定に設定します。ASI diSPIM ウィンドウで、オートフォーカスタブをクリックします (図 2c)。一般的なオートフォーカス・オプション・セクション (ブラック・スクエア) では、示されているとおりにパラメータを正確に指定します。オートフォーカスチャンネル(赤の四角) は、lineaging 実験であなたの核チャネル蛍光チャネルを指定する必要があることに注意してください。
注: 最大オフセットが5μ m より大きい場合、画像はフォーカスを外れてドリフトする傾向があります。 - パターンオーバーレイ>プラグイン>取得ツールをクリックします。
- [パターンオーバーレイ] ウィンドウで、[グリッドを表示] をクリックします。
- ASI diSPIMウィンドウで、ナビゲーションタブをクリックします。
- [梁とパス A または B のシートのチェックボックス] をクリックし、[ライブ] をクリックします。画像取得開始ライブビューウィンドウが開きます。6.1 から選択されたチャネル上の胚の周りに箱を描くことによって胚のオートフォーカス分析領域を選択します。
注: 通常、イメージングセッションごとに10個の胚に対して420のタイムポイントをキャプチャします。イメージ・セッションごとの raw データは通常 1.7 TB ですが、deconvolved および StarryNite 処理されるデータは 1.4 TB です (手順9および10を参照)。画像取得には大容量 HDD (現在のシステムでは 18TB)、画像保存にはクラウドプラットフォームを使用することをお勧めします。 - 「取得」タブで「集録を開始」をクリックして、長期間の多次元画像キャプチャを開始します (図 2b)。
7. CytoSHOW で raw マイクロマネージャーイメージを開く
- Http://dispimlineage.wormguides.org からソフトウェアバンドルをダウンロードします。
注: ソフトウェアバンドルは .zip ファイルとしてダウンロードされ、"C:\" に抽出する必要があります。使用前にディレクトリ。インストールの詳細については、http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm で説明します。 - ファイルC:\CytoSHOWextrasForC\CytoSHOW_APP.jnlpをダブルクリックして、CytoSHOW の実行を開始します。
- メニューファイル>新しい≫ diSPIM モニター (マイクロマネージャー)を選択します。取得 timepoint フォルダが保存されたルートデータセットフォルダを探します。任意の timepoint フォルダを選択し、[開く] をクリックします。多次元ナビゲーションウィンドウ ( diSPIM モニタウィンドウと呼ばれます) は、SPIMA と SPIMB の両方で自動的に開かれます (図 3a)。
注: これらのウィンドウは、(標本がまだ記録されている場合に) 新しく保存された raw timepoint スタックのルートデータフォルダを監視します。それぞれの新しい timepoint が取得されると、個別のスピムアームとサンプル位置を監視する各ウィンドウが更新され、各胚に対するマルチチャネル4D データセット全体を表示します。
8. CytoSHOW での最大投影画像の生成
逆畳み込みの前でさえ、生データを迅速に処理して、検体のグローバルな特徴を評価することができます。
- イメージウィンドウの左側のパネル (図3a、赤い長方形) のZ-MIPのボタンをクリックして、特定の位置またはスピムの腕の完全な深さとフルタイムのコースを通じて最大強度の投影を行います。Z 投影 hyperstackウィンドウが表示されます。
- Z 投影 hyperstackウィンドウで、[投影タイプ] を [最大強度] に設定します。ユーザーの設定に基づいて処理するチャネル、スライス、およびタイムポイントフレームを指定します。
- 完了したら [ OK ] をクリックします。
- フォルダの場所を選択して、ファイルダイアログウィンドウから最大強度の出力を保存し、[ OK] をクリックします。CytoSHOW がプロジェクションイメージを生成するには、時間を (15 ~ 20 分程度、データセットのサイズとコンピュータの処理能力に応じて) 許可します。
9. 等方性高解像度体積データにおける細胞系譜の解析
- オプション: raw データを CytoSHOW のdiSPIM モニタを介して開いたまま、ポリゴン選択ツール(図3a、黒色矢印) を選択し、胚の前方、後部、背、腹部の端 (正確な順序) の外側をクリックして、胚の上に「ボウタイ」パターンを生成する。両方のビュー (スピムおよびスピム、図 3a) に対して行います。
注: この選択は、胚の中心となる楕円領域 (ROI) を指定し、胚の前後部軸を記録します。蝶ネクタイパターンキュー CytoSHOW は、ユーザーがさらに StarryNite/AceTree によって lineaging 分析に最適である向きに、最終的な対し融合ボリュームの正確な回転を指定することを計画しています。StarryNite lineaging が実験計画に含まれていない場合、画像処理の ROI を設定するために、他の選択ツールや形状が選択されることがあります。 - 複数の胚がマルチポジション取得オプションを用いて同時に撮像された場合、全ての胚に対してステップ9.1 を開いて実行する。これは1つのセッションのすべての胚のための将来のステップの並行実行を可能にします。あなたが処理したくない任意の胚のためにシマと SPIMB のウィンドウを閉じます。
- DiSPIM モニタウィンドウの左側のパネルにある [ diSPIM ] ボタンをクリックします (図 3a、黄色で強調表示されています)。これにより、diSPIM 処理に固有のコントロールのサブパネルが明らかになります。
- 各スピムアームに緑と赤のチャンネルを合わせます。放出チャネルスプリッタを使用して、同じカメラ上で別個の赤と緑のイメージを同時にキャプチャするため、これらの2つの物理的に隣接するイメージフィールドの正確なピクセル登録を、それらがオーバーレイされるときに視覚的に揃えることが重要です。通常、同じ線形調整の再利用は、多くの連続したイメージングセッションで実行可能ですが、チェックする必要があります (手順9.4.1 – 9.4.5)。
- SPIMA パネルから開始して、 CMスクロールバーを左に移動して赤のチャンネルを選択します (図 3a、オレンジ色の矢印、左パネル)。
- X、y、および z アジャスター (図 3a、オレンジ色の正方形) を使用して、赤のチャンネルを緑に合わせてシフトします。
- [ DiSPIM ] ボタン (図 3a、黄色で強調表示) をクリックしてサブパネルを閉じ、同じシフトの他のすべての位置ウィンドウへの伝播をトリガします。
- "Z" スクロールバー (図 3a、青い矢印、左パネル) および/または "t" スクロールバー (図 3a、緑の矢印、左パネル) を移動することによって、他のフレームおよびタイムポイントに反映された正しいアライメントであることを確認します。複数位置の捕捉が行われ、いくつかの胚が画像化された場合 (ステップ 5.3)、アライメントもそれらの胚に伝播すべきである。また、x、y、および z アジャスターの数値を調べて確認します (図 3a、オレンジ色の正方形は、すべての胚の SPIMA パネルで同じでなければなりません)。
- SPIMB diSPIM モニタウィンドウの手順9.4.1 –9.4.4 を繰り返します (図 3a、右パネル)。
- 「DiSPIM」ボタンをクリックし、次に「ヒューズ」ボタン (図3a、青の長方形) で「Deconvolve/ヒューズ diSPIM 生データボリューム」というダイアログボックスを開きます (図 3b)。図 3bに示すようにパラメータを設定します。これらのパラメーターについては、次のサブステップで簡単に説明します。
- チャンネル 1 (488 nm レーザー) または 2 (561 nm レーザー) のキー登録を設定します。より密度の高い、またはよりユビキタスな信号でチャンネルを選択します。Lineaging 実験のためには、常に普遍的な核ヒストン蛍光を画像するために使用されるチャネルを選択する。
- 入力ボリュームの相対的な向きを + 1 または-1 に設定します。正しい向きのインデックスは、特定の diSPIM のカメラ配置によって異なります (図 4)。
注: 不明な場合は、スピム A と B diSPIM モニター ・ウィンドウの両方から1つの timepoint を複製して各オプションをテストし、次の手順 9.1 ~ 9.12 とテストする入力ボリュームの相対方向を任意に選択します。正しくない向きでは、アーチファクトでぼやけた画像が生成され、正しい向きでは鮮明な画像が得られます。明確なイメージを生成する入力ボリュームの相対方向値は、指定された diSPIM インストゥルメントの将来のすべてのデータに再利用できます。 - 溶融ボリュームを A または B 入力ボリュームと同じ方向に向けるかどうかを選択します (ユーザ設定に基づく)。
- 「すべてのボリュームのための新鮮な登録」を選択します。このオプションは、各 timepoint で各ボリューム・ペアの登録行列を計算する方法を制御します。 SpimFusion「フレッシュ」オプションは、アルゴリズムが各 timepoint で適応的に登録を最適化することを可能にします。
- 逆畳み込み反復の回数を10に設定します。この数値は、所望の高解像度を時間効率のよい方法で確実にもたらす傾向がある。
- オプション: lineaging が必要な場合 (強く推奨)、融合ボリュームの自動起動 StarryNite lineaging を確認します。このオプションは StarryNite を自動的に起動し、SpimFusion によって生成されたイメージボリューム内のセルをセグメント化して追跡します。
- オプション: 自動 lineaging で最高の精度を上げるには、対しに融合した胚のボリュームを、標準の「ADL」 (nterior [x-西]、 Dorsal [y-北]、左 [z-near]) 方向に再配置するのが最適です。[プレビューでボリューム出力の方向を定義] オプションを選択して、この選択を指定します。CytoSHOW は、対し融合ボリュームの初期ペアを処理することによって応答し、ユーザが ADL の登録を達成するために必要な回転を密接に観察し、指定することができます。
- すべてのパラメータを選択したら、[はい] をクリックします。
- 処理されたファイルを保存する出力ディレクトリを指定します。[ OK] をクリックします。
- オプション: 「プレビュー」オプションで「ボリュームの出力方向を定義」が選択されている場合は、スピムウィンドウのtスクロール・バー (図 3a、緑の矢印、左パネル) を、ABa および ABp 細胞が中期に達した初期の timepoint に設定します。スピムウィンドウのtスクロールバーを、開発の後のコンマステージに設定します。これは、ADL の向きを指定するのに役立ちます。
- オプション: 準備ができたら [ OK ] をクリックします。上記の9.5.7 のプレビューオプションが選択されている場合、スピムおよびスピムイメージウィンドウの t スライダーによって示される timepoints に対して、2つのプレビューボリュームのみが対しに融合されます。これらの2つのプレビュー timepoints は、以下に説明するように、ADL の向きに出力胚体積の正確な再配置を指定するために使用することができる。
- 新しく表示された 3DProjY_Decon-Fuse_ を探します。ウィンドウ。Tスクロールバーをこのプレビューウィンドウのタイムポイント2に移動します。[Z] スライダを移動して、胚の長軸を直接下に表示します。
- Tスクロールバーを 3DProjY_Decon-Fuse_ のタイムポイント1に戻します。ウィンドウ。ライン選択ツールを選択し、AB セルの中期プレートの平面を介して EMS セル (腹側の最も丸い核) からライン選択を描画します。
- 3DProjY_Decon-Fuse_ のオレンジ色の diSPIM プレビューボタンをクリックします。ウィンドウ。イメージをプレビューしたボリュームの向きを微調整すると、データセット全体の処理に使用できるように保存されます。
- オプション: 上記の手順9.5 のように、 Deconvolve/Fuse DiSPIM 生データボリュームのダイアログが再び表示されます。[プレビューでボリュームの出力方向を定義する] オプションを選択せずに [はい] をクリックします。完全なデータ処理の実行のための出力フォルダーを指定します。
- DiSPIM モニタウィンドウのtスクロールバー (図 3a、緑の矢印、左パネル) を、開始timepoint (SPIMA) と終了timepoint (SPIMB) を処理する画像の全範囲に設定します。[ Ok] をクリックします。
- SpimFusion が進行するにつれて、CytoSHOW は、各胚についてスライスされ4D 等方性融合体積を示す多次元ウィンドウと、等方性容積の回転4D 最大強度突起を有する2つの窓を開いてリフレッシュする。この間、等方性融着および系統追跡が完了するまで、任意の CytoSHOW ウィンドウを破壊したり閉じたりしないでください。
- オプション: StarryNite スプラッシュ画面が表示され、後で消えた後、完全なデータ処理パイプラインが完了したことに注意してください。このウィンドウは処理中に閉じてはならないか、StarryNite が中断されます。
10. オープニング StarryNite リネージュ・トレース・シリーズ AceTree (オプション)
- 提供されている「AceTree_16BitCompat」のカスタマイズ版を開きます。
- ファイル> 開いている構成ファイルのメニューを選択します。以前に CytoSHOW に指定した出力ディレクトリを探します。Decon_Fuse_ を開きます... 胚のビットセット内 [ n] サブフォルダAaa_editedを選択して開きます。
- AceTree メニューの編集 ツールを使用して、編集トラックを開き、セルウィンドウを調整または削除し > ます。
- 赤と緑の強度を調整するには、半影の円図5b、赤の四角をクリックします。
- 前述の5、6、8 (原稿もダウンロード・バンドルに含まれています) のように、リネージュ・ビジュアライゼーションおよび編集を続行します。
Representative Results
我々はまず、diSPIM 取得のためのプロトコルのパラメータを用いて画像化された胚の生存率を検証した (セクション 1-6)。10個の胚を20° c、1つの容積/胚/分で同時に画像化し、2細胞ステージから2倍ステージ (7.5 時間、451体積/胚) にした。胚発生全体にわたって細胞分裂を監視するために、我々は、遍在mCherryを表現するひずみ BV514 を使用した::ヒストンレポーターは、 ujIs113の統合トランスジーンアレイから構成される。図 6は、画像化された胚の1つについての胚発育のこの前半のタイムラインを示す。各画像は、画像化された胚の単一ビューの最大強度投影 (ステップ7-8 によって生成される) を表す。最適化されたプロトコールは、細胞分裂のタイミング (図示せず)、孵化時間、および発達的マイルストーンに関連するタイミングによって評価されるように、胚に検出可能な光毒性を誘導しなかったことを発見した (図 6および文献1,25,26)。
そして、開発した胚の単一ニューロンの伸長ダイナミクスを解析するプロトコルを適用した。我々は、未同定細胞のサブセットにおいて神経ペプチドflp-19プロモーターから GFP を発現するトランスジェニック線虫株 DCR7692 (olaex4655) を画像化した (DACR2819, Pflp-19 (3.6 KB):: Syn21:: GFP:: CAAX::p 10 3 ' UTR).ここで概説したプロトコルの手順に従って、未知の細胞が RMDDL および RMDDR の運動ニューロン、排泄管細胞、および2つの筋細胞に対応することを決定しました (図 7)。次に、RMDDL および RMDDR ニューロンの伸長ダイナミクスを調べ、定量化しました。我々は、RMDDL および RMDDR ニューロンが、受精後360分の早い時期に斜めに形成されており、より長い細胞軸が神経突起伸長の後続の軸を表すことを観察した (図 7およびムービー S1)。フィジーの「単純神経突起追跡」プラグインを使用して、対し融合体積の3D 再復元にそれを適用し、6つの胚の RMDDL と RMDDR 神経突起の型どおりの伸長を定量化しました。我々は、伸長ダイナミクスが胚全体に RMDDL および RMDDR に対してステレオタイプ化されたことを決定した (ここでは RMDDs と呼ばれる)。受精後385-410 分から、RMDDs 神経突起拡張6.0 ±0.5 μ m (平均± SEM;n = 12 神経突起) 細胞体の前方 (図 7b, C, I)。415-445 分から受精後、両方の神経突起は、推定神経環 (図 7dにおけるアスタリスク) の中およびその周辺に dorsally を拡張する。平均して、各 RMDD 神経突起は11.0 ±0.6 μ m (平均± SEM;n = 12 神経突起) は、リングの頂点で同期的にその対の対応を満たす前に、セルボディから (図 7I)。私たちの代表的な結果は、統合されたプロトコルを使用して、単一の識別可能な細胞のニューロンの発達特徴を検査、比較、定量化できることを示しています (図 7および図 8)。
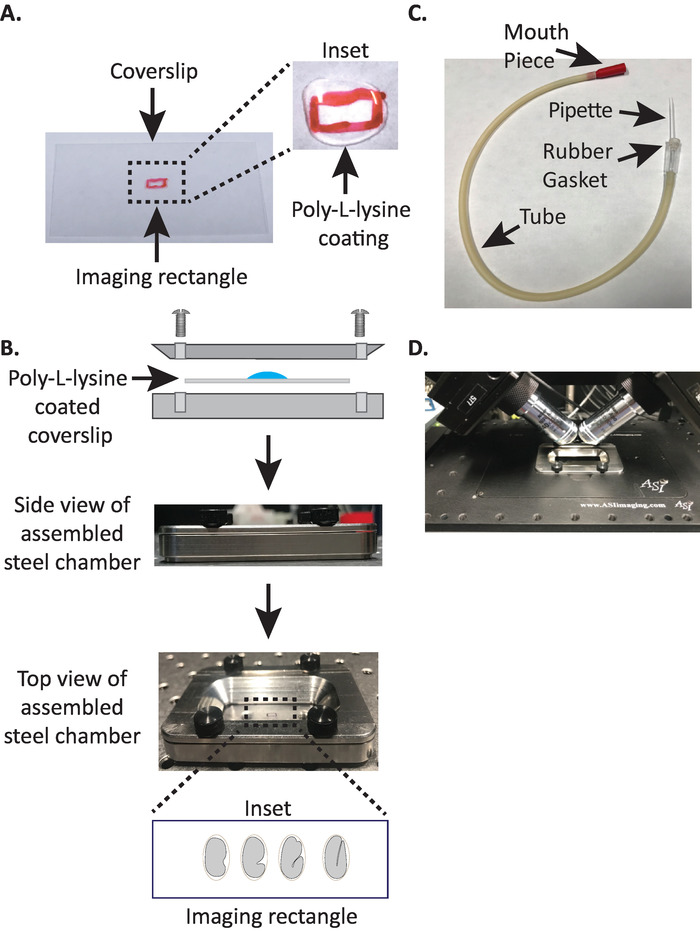
図 1: diSPIM サンプルの取り付け設定。(A) ポリ L リジンをカバースリップの調製.このインセットにおいて、ポリ L リジンの10μ l を5分間カバースリップにコートするために使用した。ポリ L-リジンは胚の卵殻は、長方形のカバースリップにしっかりと固執することができます。(B) 鉄骨イメージングチャンバー及び組立チャンバの概略図。このインセットにおいて、代表的な胚は、カバースリップ上の長軸に直交する前後方軸を向いている。(C) microcapillary ピペットで吸引管を組み立てた。(D) diSPIM 40 倍の目標の下でサンプルホルダーに取り付けられたスチールイメージングチャンバー。この図の大規模なバージョンを表示するには、ここをクリックしてください。

図 2: マイクロマネージャーでの長期 diSPIM イメージングのセットアップ。(A) 光毒性を減少させながら長時間の画像化のために最適化された diSPIM レーザーパワーパラメータ (赤色矩形) を推奨 ( C elegans胚のより高い孵化率によって評価)。561 nm レーザーを79Μ w (0.25) と 488 nm レーザーを179Μ w (0.5) に設定します。レーザーパワーへのソフトウェア設定の正確なキャリブレーションは、diSPIM のインストールによって異なりますのでご注意ください。ユーザーは79Μ w (561 nm) および179Μ w (488 nm) レーザー力を達成するためにレーザー力を測定し、調整することを推薦する。(B) データ保存のためのパラメータを diSPIM (緑の長方形)、音量設定 (青の正方形)、およびスライスの設定 (オレンジ色の正方形)。(C) diSPIM elegans胚発生の長期イメージングのためのオートフォーカスパラメータ (手順 6.1-6.6 を参照)。この図の大規模なバージョンを表示するには、ここをクリックしてください。

図 3: CytoSHOW を使用したイメージの視覚化とデータ処理のセットアップ。(A) CytoSHOW によって開かれた Raw diSPIM 画像。CytoSHOW は、両方のカメラパス (スピム A と B) によってキャプチャされた画像を開くことができます。これらの raw 画像は、 diSPIM モニタと呼ばれる多次元ウィンドウで開きます。DiSPIM モニターでは、胚の前方、後方、背部および腹側縁を選択するために「蝶のパターン」が生成される (ステップ9.1 を参照)。ボウタイ選択は、逆畳み込みおよび StarryNite 支援 lineaging トレースのための胚の向きを示す。(B) 等方性画像を生成するために用いられるパラメータの最適化。Deconvolved ウィンドウを取得中に、ステップ 9.5.1-9.5.8 で指定されたパラメータを設定します。この図の大規模なバージョンを表示するには、ここをクリックしてください。

図 4: diSPIM カメラの構成。(A) diSPIM カメラの配置と向きの写真。(B) 収集されたスピム B 画像に一致するようにスピムの + 90 度の回転の描写。(C) 私たちの diSPIM のカメラ構成に基づいて、オリエンテーションインデックス + 1 に関連する入力ボリューム (ステップ9.5.2 を参照してください)。スピム B 画像と一致するように登録する前に、Y 軸周りの画像 (s) + 90 度のスピムを回転させます。スケールバーは10μ m です。画像は、代表的な単一視野、最大強度の突起、および標識された核 (561-nm、赤) およびニューロン (488-nm、緑色) を有する1.5 倍胚の逆畳み込み画像である。この図の大規模なバージョンを表示するには、ここをクリックしてください。

図 5: AceTree におけるelegans胚系統のキュレーションと編集(A) AceTree を使用して StarryNite の系統トレースを編集します (参照5、6、8、原稿もダウンロードバンドルに含まれています)。AceTree は、手順 10.1-10.2 の完了時に、各核 (緑色の長方形) のelegans体系的な名前を表示します。このウィンドウ (A) は、リネージュ・トレースのトラッキングおよび編集時にユーザーをガイドするのに役立つ、系列追跡の各セルに関する情報 (黒色の四角形) を提供します (青色でハイライト表示されます)。ユーザが、Sulston et al.1によって以前に報告されたelegans胚細胞系統への lineaged 細胞とその位置を確認し、比較することが推奨される。deconvolved データ系列 (下記参照、 B) は、検索バー (オレンジ色の長方形) にelegansの体系的な名前を入力します。(B) ユーザーの deconvolved データ系列も、手順 10.1-10.2 の完了時に自動的に開きます。ここでは、赤色で標識された核を有する4細胞期胚の対し融合画像を示す。原子核の追跡中、ユーザーは画像の強度 (赤の四角) を変更し、キーボードの矢印キー (時間-左/右、z アップ/ダウン) を使用して時間と z をナビゲートする必要があります。(C) timepoint の3d 漫画 (B) で回転可能な3d 視覚化を有効にする特定の機能 (紫色の長方形)。AceTree とその3d 機能の概要については、参照5、6、8を参照してください。この図の大規模なバージョンを表示するには、ここをクリックしてください。

図 6: diSPIM 上のc. Elegans胚の時間発達動態トップパネルは、画像化された胚の1つについての胚発生の前半を示す diSPIM 画像 (株 BV514 ujIs11324) である。胚を連続的に画像化し、毎分7.5 時間 (20 ° c で) にする。トップパネルの最初の2つの画像は4と8細胞の胚の核 (赤) と極体の位置 (濃い赤色の球体、青いアスタリスクでマーク) を表します。各画像は、画像化された胚の単一ビュー最大強度投影を表す。スケールバー = 10 μ m。タイムライン (横棒) は、 elegans胚の発生後の分受精 (m.p.f.) を表す。我々は、diSPIM 獲得のための我々のプロトコルのパラメータが、生存率、細胞分裂のタイミング、孵化のタイミング、および発達マイルストーンのタイミングによって評価されるように、画像化された胚に検出可能な光毒性を誘導しなかったことを検証した (参照1,25,26). 我々は、発達のマイルストーンのタイミングが、我々の画像化パラメータ (6.4 時間長いイメージングセッションのための SEM ±8.174 分) を有する胚間で再現可能であることに留意する。n = 10)。この図の大規模なバージョンを表示するには、ここをクリックしてください。

図 7.Elegans胚の開発における神経突起伸長ダイナミクスの細胞識別と単一細胞の特性解析BV514 ujIs11324 (lineaging 用) と DCR7692 (olaex4655) とを交配して作られた菌株をデュアルカラー画像化することで、未同定細胞のサブセットにおけるニューロペプチドFLP-19プロモーターの GFP を発現するトランスジェニック線虫株である。(A-H)ここで概説したプロトコルの手順に従って、未知の細胞は、運動ニューロン RMDDL および RMDDR (黄色の矢印)、排泄管細胞 (青い矢印)、および2つの筋細胞 (白い矢印) に対応することを決定しました。(I) フィジープラグイン「単純神経突起トレース」を使用して、RMDDL および RMDDR ニューロンの伸長ダイナミクスを定量化し、対し融合ボリュームの3d 復元に適用します。RMDDL と RMDDR の両方が、合計で11.0 ±0.6 μ m (平均± SEM) で同期的に伸びる型どおり伸長ダイナミクスを示していることに注意してください。n = 12 神経突起) と神経環の背尖部での会合 (映画 S1も参照)。この図の大規模なバージョンを表示するには、ここをクリックしてください。

図 8: elegans胚における神経形態の等方性 diSPIM 画像の検討AVHL と AVHR ニューロンの等方性可視化 (黄色矢印).DiSPIM を用いて、神経形態は、約 330 nm の等方性空間分解能を有する4次元 (4D) 画像を得て捕獲することができる。DiSPIM は、ユーザーがすべての方向に同一の解像度で画像ボリュームを仮想的に回転させることができます。A−D内の画像は、胚の長軸を中心とする異なる回転から diSPIM 像体積を融合した同じ対しの最大強度投影である。スケールバー = 5 μ m。この図の大規模なバージョンを表示するには、ここをクリックしてください。
補足の映画 S1: c. elegansの胚は受精の後の434分に280から成長する。DCR7692 (olaex4655) の等方性動画は DACR2819 まばらな標識 RMDD 神経突起でujIs113遍在を発現している (図 7a-D、黄色矢印)。DACR2819 はまた、2つの筋細胞 (図7a −d、白色矢印) および排泄管細胞 (図 7a−d、青色矢印) を胚発生時に標識する (図7a − d)。スケールバー = 10 μ mこのファイルをダウンロードするには、ここをクリックしてください。
Discussion
C. elegansは27の既知の各成体ニューロンの最終的な位置と接続性を持つ唯一の生物として際立っています。しかし、 elegansコネクトームを構成する作業回路やネットワークの組織化につながる発達力学は不明のままである。光顕微鏡の進歩から生じた機会に基づいて、 elegans胚発生における細胞の位置、形態形成、および神経新生を捕捉し、分析することができるようになりました。
我々が研究室で日常的に使用している手順は、 C elegans胚の細胞 lineaging に対する標識ニューロンと核の 4d-等方性画像をもたらす。さらに重要なことは、diSPIM を使用した長期的なイメージング条件を最適化し、半自動 lineaging 機能と高解像度画像を組み合わせて、 C elegansの胚発生を解析する速度と精度を向上させることです。この統合されたプロトコルにより、ユーザーは初期のけいれんの開始を通じて、細胞を可視化して識別し、神経突起の移行や軸索手筋などの三次元特徴を定量化することができます。この手順は、ASI diSPIM システムと任意の施設に容易に適合することができ、我々はこのプロトコルのために特別にこのシステムをお勧めします。商業的に提供される他のスピム製剤は、試料チャンバーおよび光学的特性における ASI 構成とは異なる場合がある。ただし、他のプラットフォームからエクスポートされたデータは、データパイプラインを介して配置することもできます。したがって、lineaging におけるそれらの値の評価は、画像品質および器具安定性の厳しい試験であり、実現可能である。我々は積極的に他の標本 (ショウジョウバエおよびゼブラフィッシュ胚など) をイメージするために diSPIM を使用しているが、胚の記述および包括的な lineaging 分析は、現在でも線虫種に限定されている。より大きいか厚いサンプルのために、私達は静止した軽いシートを通してサンプルをスキャンする段階スキャンのアプローチを使用することを選ぶ。クマール et al. 以前は、diSPIM10に追加の変更を加えることなく厚いサンプルから高品質の画像を生成するために、この改良された diSPIM 断面を実証しました。
プロトコル内の重要なステップは、カバースリップコーティングされたポリ L リジンへのelegans胚の取り付け、データ取得、およびデータ処理を含む。ガラスカバースリップに収穫し、 elegansの胚を取付けることは経験の浅いユーザーに挑戦することができるが、ここで学習を促進するために主要なステップの詳しい議定書を提供する。長期画像化が望まれる場合、我々は8-10 若年成人28から4細胞またはそれ以前の胚を収穫する最良の結果を得る。高齢者は、子宮の古い胚と未受精卵を含む傾向があるので、初期の胚を収穫することはあまり望ましくないことに注意してください。取り付け胚に関しては、組み立てられた吸引器 (口ピペット) の閉塞や microcapillary ピペットでの開口の大きすぎるなどの問題が、胚の適切な取り付けおよび配向を妨げることがある。最適なイメージングのために準備するために、我々は、光シート、カメラ、目標、およびオートフォーカスの性能をチェックするために、早期および後期事前けいれん胚の事前捕捉試験を行います。これらのすべての操作がテストされ、プリ集録テスト中に高品質の画像が得られると、最良の結果を得ることができます。これは、両ビュー (目標) から取得した raw 画像が高品質でなければならない等方性空間分解能を持つ画像を生成する場合に特に重要です。取得後、各ビューについて取得したボリュームは、等方性画像を生成するために処理されます。このプロトコルで説明されているように、適切なグラフィックスプロセッシングユニット (GPU) カードを使用することが重要です (下記参照)。これにより、対しの融合画像が生成される処理速度が向上し、データ解析までの時間を短縮することができます。また、ユーザーが CytoSHOW の最新バージョンを実行しており、StarryNite 自動 lineaging のダウンロードバンドルで提供されているパラメーターを使用していることも不可欠です。他のサンプル (例えば、ゼブラフィッシュ、ショウジョウバエなど) の自動 lineaging を使用することに興味がある場合は、StarryNite で使用されるパラメータに追加の最適化が必要になります (参照3,4を参照)。
私たちの統合されたプロトコルは、画像と lineaging の結果を事前にけいれん胚に提供しますが、ユーザーは、lineaging 後の胚における自動的な変化は現在実現不可能であることに注意すべきである: 核の位置は、秒の順序で過剰にけいれんした胚は、系統追跡を可能にするには速すぎる。しかし、diSPIM は、神経発達のイベントを捕捉し、23,29の胚発生後の段階でいくつかの細胞位置を追跡する有望な能力を実際に実証した。ユーザーがけいれん後の胚を調べることに興味がある場合、diSPIM は容積型のスナップショットを得る速度を提供し、急速に移動する胚における神経突起伸長などの微細な神経発達イベントを追跡します。
このプロトコルは、WormGUIDES アトラス30の細胞ごとの完了のための基礎となり、高解像度等方性画像を有する統合アプローチを提供し、 中に標識されたニューロンの3d 形態を識別し、捕捉する。胚発生の最初の430分。それがあるように、プロトタイプ WormGUIDES アトラスは、開発胚の細胞の核位置を提供し、胚ニューロンのサブセットの発達ダイナミクスを捕捉することを目的としています。このプロトコルは、追加の開発ニューロンを WormGUIDES アトラス30に統合するための鍵となります。
私たちの統合されたプロトコルはまた、 elegans胚における新しい遺伝子発現プロファイルの探索を容易にします。トランスジェニックc. elegansにおいて、多くの細胞特異的プロモーターは、トランスジーン発現を空間的に時間的に制御する。ほとんどの遺伝子の発現パターンは成人動物31、32、33、34で広範囲に特徴付けられているが、ほとんどすべてが開発において特徴付けられることはまだない (特に後期) 胚。Elegans promoterome は、ワームコミュニティにとって細胞特異的なトランスジーン発現を促進し、遺伝子機能が細胞自律的であるか非自律的であるかを判断するために役立つリソースとなっています。等方性高解像度と動的発現パターンの遺伝子を捕捉し、lineaging を介して発現細胞を正確に同定することは、科学界の多くの人にとって貴重なものとなるであろう。
胚発生は、主要なプロセス、細胞分化および組織形態形成の2つの絡み合いを含む。Elegansの開発中に異なる細胞型を定義するメカニズムと分子については、多くのことが知られています。しかし、 elegans胚における細胞遊走、細胞接着、細胞形状にとって重要なメカニズムについては、ほとんど分かっていない。Elegans不変の細胞系統が知られているので、我々のプロトコルは、新しい詳細レベルでの形態形成の間に、例えば軸索手筋、シナプス形成、ニューロン活動といった、カタログ化された microanatomy の胚を容易に識別することを可能にする。Ardiel et al. は以前、 C elegans胚における単一ニューロンのレベルでのカルシウムトランジェントを捕捉するために diSPIM の力を実証した。発達生理学の他の多くの側面は、これらの方法による問い合わせのために熟している。
最後に、このプロトコルは主に自動化されており、逆畳み込み画像を生成し、StarryNite および Acetree を介してセル lineaging を実行するのにかかる時間を体系的に削減します。このプロトコルで使用されるソフトウェア戦略は、我々はここでそれらを実証している非常に特定の分野から遠く離れた生物学の多くの質問に適用することができます。
ソフトウェアの互換性とダウンロードアクセスの詳細
マイクロマネージャーと diSPIM イメージングのプラグインに関する情報は、http://dispim.org/software/micro-manager と https://micro-manager.org/wiki/ASIdiSPIM_Plugin で入手できます。
現在、データ処理パイプラインには Windows オペレーティングシステムが必要です。私たちは、すべての必要なデータ処理プログラムとサポートファイルのインストールを簡素化するために、単一のアーカイブファイルをバンドルしています。Http://dispimlineage.wormguides.org でダウンロードできます。
CytoSHOW (http://run.cytoshow.org/) は、広く使用されているオープンソース画像解析プラットフォーム、ImageJ (v1) に基づいています。CytoSHOW を使用するには java をコンピュータにインストールして最新の状態にする必要があり、CytoSHOW の更新は Java Web Start を介して自動的に展開されます。CytoSHOW の多くの ImageJ ベースの機能は、https://imagej.nih.gov/ij/docs/examples/index.html で説明されており、図示している。CytoSHOW は、ASI diSPIM からの多次元の生データ、および TIFF 出力を作成するその他のイメージングソフトウェアを表示するようにカスタマイズされています。原理的には、他のマルチビュースピムイメージングシステムは、このプロトコルは、異なる顕微鏡システム上で実行することを可能にするために CytoSHOW のマイナーな変更によってサポートすることができます。
SpimFusion は、Visual Studio 2013 を使用した cuda/C++ で CUDA toolkit v 7.5 で書かれました。SpimFusion を実行するには、特定のコンピュータハードウェアが必要です: CUDA 計算機能1.0 以上と最小 2 GB のグラフィックスカードメモリを備えた NVIDIA グラフィックスプロセッシングユニット (GPU) カード。私たちのプロトコルの公開時には、SpimFusion は未発表 (最小の Shroff とハリのない) ですが、上記のソフトウェア・バンドル・アーカイブで利用可能です。
特別に構築されたコマンドライン駆動バージョンの StarryNite では、自由に利用できる MATLAB コンパイラランタイムがインストールされている必要がありますが、商用 MATLAB ソフトウェアのライセンスは必要ありません。MATLAB コンパイラーのランタイムは、前述のソフトウェア・バンドル・アーカイブに含まれています。このプロトコルで使用される StarryNite のコードは、共焦点画像6に使用されるものと本質的に変わりません。しかし、StarryNite 処理のための入力画像の作成および StarryNite 結果の処理のためのいくつかの運用上の問題は、溶融等方性のための連続データ処理パイプラインを可能にする CytoSHOW の方法によってここで対処されている diSPIMボリューム。これらの変更は、これらの前処理および後処理のステップを処理する CytoSHOW コードによって自動化されています。CytoSHOW はまた、事前に最適化された diSPIM 固有のテンプレート StarryNite パラメータセットを編集して、セグメンテーションアルゴリズムを画像データ内の原子核の蛍光強度に自動的に調整します。その後、各 diSPIM データ・セットで StarryNite によって使用される固有のパラメーターが、出力イメージおよび lineaging データとともにファイルに保存されます。
16ビットイメージで動作し、Java3D レンダリングとの互換性を維持する AceTree のカスタムバージョンは、このプロトコルに最も適しています。また、前述のソフトウェア・バンドル・アーカイブにも含まれています。
Disclosures
作者は何も開示することはありません。
Acknowledgments
私たちは、lineaging 株 BV514 を生成するために、統合株、ujIs113 のジョンマレーに感謝します。ブランドン・ハーベイ (NIBIB) プロトコルのテストについてのヘルプ。ジョン・ダニエルズとゲイリー・ロンドー (応用科学機器) マイクロマネージャーと diSPIM 楽器の支援のため;そして、アンドリュー・ヨークとハンク・エデンは、diSPIM システムに関する重要なフィードバックのために。また、会議とブレーンストーミングのプラットフォームを提供するためのセルバンテス文化センター・デ・ Neurobiología ホセ・デル・カスティージョ (ユニベルシダード・デ・プエルトリコ) の研究センターにも感謝しています。この作品の多くは、森の穴にある海洋生物学研究所でホイットマンプログラムを通して行われました。この作品は、NIH 国立生物医学イメージング研究所とバイオエンジニアリングの学内研究プログラム、および NIH 助成金によってサポートされていました。U01-HD075602 とノーR24-OD016474。マーク w. モイルは、F32 ・ NS098616 によってサポートされ、R24-OD016474 の多様性の補足によってサポートされました。
Materials
| Name | Company | Catalog Number | Comments |
| Steps 1-4 | |||
| Concavity slides | ThermoFisher Scientific | 1519006 | 5-18mm diameter, 0.6-0.8mm deep, 1.2-1.5mm |
| Dissecting microscope with 10×–50× zoom range | Motic | SMZ-171 | |
| E. coli (OP50) | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Glass coverslips, no. 1.5, 24 × 50 mm | VWR International | 48393-241 | |
| M9 Buffer | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Methyl cellulose | Sigma-Aldrich | H7509-25G | |
| Microcapillary pipette aspirator tube | Sigma-Aldrich | A5177 | |
| Microcapillary pipettes, 0.4-mm i.d | Drummond Scientific | 1-000-800 | |
| Needle, no. 18G x 1 ½ (1.2mm x 40mm) | BD Precision Glide | 305196 | |
| NGM plates | prepared as described by Brenner (1974) | ||
| O-ring for imaging chamber | O-Rings West | M1.5X40 | |
| Pasteur pipette | Corning/Sigma-Aldrich | CLS7095D5X | |
| Platinum wire, 0.5-mm diameter | Sigma-Aldrich | 267201 | |
| Poly-L-lysine | Sigma-Aldrich | P1524 | |
| Stainless steel rectangular chamber (76.0 mm x 50.5 mm) | Applied Scientifics Instrumentations (ASI) | I2450 | |
| Worm Eyelash Pick | Hart, A. C. Behavior. WormBook. (2006). | ||
| Worm Pick | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Name | Company | Catalog Number | Comments |
| Steps 5-6 | |||
| 488 nm long-pass filter | Semrock | LP02-488 RU-2 | |
| 561-nm notch filter | Semrock | NF03-561E-25 | |
| BLP02-561R-25, quantity 2 | Semrock | 561 nm EdgeBasic best-value long-pass edge filter | |
| Control software for bottom camera | Jenoptik | ProgRes CapturePro | |
| diSIPM assembly video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/TAgbr6IrTqw ; http://www.asiimaging.com | |
| diSPIM alignment video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/qnOrg30NNuE | |
| diSPIM imaging PC | Intel | Intel Xeon CPU E5-2630 2.6GHz, 12 cores in total, 64 GB memory, Windows 7 | |
| FF01-525/45-25, quantity 2 | Semrock | 525/45 nm BrightLine single-band bandpass filter | |
| FF555-DI03-25X36, quantity 2 | Semrock | 555 nm edge BrightLine single-edge dichroic beamsplitter | |
| Imaging PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Kumar et al diSPIM Setup | Applied Scientifics Instrumentations (ASI) | Instrument setup for this protocol is identical to Kumar et al 10,11 diSPIM, which makes use of 40x 0.8NA water immersion lenses for imaging. (See steps 5.1 and note) | |
| Micro Manager | Micro-Manager | https://micro-manager.org/ | |
| Modifications to Kumar et al diSPIM Setup (see below) | |||
| Optical table with isolators, 4 feet × 6 feet × 12 inches | TMC | 784-651-02DR and 14-416-34 | |
| Name | Company | Catalog Number | Comments |
| Steps 7-10 | |||
| Analysis PC | Intel | Intel Core i7-8700K CPU 3.70GHz, 6 cores in total, 64 GB memory, Windows 10 | |
| Analysis PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Installation instructions | Software bundle | http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm | |
| Software bundle | Software bundle | http://dispimlineage.wormguides.org |
References
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology. 100 (1), 64-119 (1983).
- Bao, Z., et al. Automated cell lineage tracing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 103 (8), 2707-2712 (2006).
- Santella, A., Du, Z., Bao, Z. A semi-local neighborhood-based framework for probabilistic cell lineage tracing. BMC Bioinformatics. 15, 217 (2014).
- Santella, A., Du, Z., Nowotschin, S., Hadjantonakis, A. K., Bao, Z. A hybrid blob-slice model for accurate and efficient detection of fluorescence labeled nuclei in 3D. BMC Bioinformatics. 11, 580 (2010).
- Boyle, T. J., Bao, Z., Murray, J. I., Araya, C. L., Waterston, R. H. AceTree: a tool for visual analysis of Caenorhabditis elegans embryogenesis. BMC Bioinformatics. 7, 275 (2006).
- Katzman, B., Tang, D., Santella, A., Bao, Z. AceTree: a major update and case study in the long term maintenance of open-source scientific software. BMC Bioinformatics. 19 (1), 121 (2018).
- Murray, J. I., et al. Automated analysis of embryonic gene expression with cellular resolution in C. elegans. Nature Methods. 5 (8), 703-709 (2008).
- Murray, J. I., Bao, Z., Boyle, T. J., Waterston, R. H. The lineaging of fluorescently-labeled Caenorhabditis elegans embryos with StarryNite and AceTree. Nature Protocols. 1 (3), 1468-1476 (2006).
- Wu, Y., et al. Spatially isotropic four-dimensional imaging with dual-view plane illumination microscopy. Nature Biotechnology. 31 (11), 1032-1038 (2013).
- Kumar, A., et al. Using Stage- and Slit-Scanning to Improve Contrast and Optical Sectioning in Dual-View Inverted Light Sheet Microscopy (diSPIM). The Biological Bulletin. 231 (1), 26-39 (2016).
- Kumar, A., et al. Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nature Protocols. 9 (11), 2555-2573 (2014).
- Wu, Y., Christensen, R., Colon-Ramos, D., Shroff, H. Advanced optical imaging techniques for neurodevelopment. Current Opinion in Neurobiology. 23 (6), 1090-1097 (2013).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17708-17713 (2011).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136 (12), 1963-1975 (2009).
- Stelzer, E. H. Light-sheet fluorescence microscopy for quantitative biology. Nature Methods. 12 (1), 23-26 (2015).
- Winter, P. W., Shroff, H. Faster fluorescence microscopy: advances in high speed biological imaging. Current Opinion in Chemical Biology. 20, 46-53 (2014).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. Astronomical Journal. 76 (6), 745-754 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. JOSA. 62 (1), 55-59 (1972).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Hart, A. C. Behavior. WormBook. , (2006).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. Chapter 14, Unit14 20 (2010).
- Gualda, E. J., et al. SPIM-fluid: open source light-sheet based platform for high-throughput imaging. Biomedical Optics Express. 6 (11), 4447-4456 (2015).
- Ardiel, E. L., et al. Visualizing Calcium Flux in Freely Moving Nematode Embryos. Biophysical Journal. 112 (9), 1975-1983 (2017).
- Walton, T., et al. The Bicoid class homeodomain factors ceh-36/OTX and unc-30/PITX cooperate in C. elegans embryonic progenitor cells to regulate robust development. PLoS Genetics. 11 (3), e1005003 (2015).
- Altun, Z. F. WormAtlas. , (2002).
- Wood, W. B. Embryology: In the nematode C. elegans. Cold Spring Harbor Laboratory Press. Chapter 8, 215-241 (1988).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314 (1165), (1986).
- Bao, Z., Murray, J. I. Mounting Caenorhabditis elegans embryos for live imaging of embryogenesis. Cold Spring Harb Protoc. (9), (2011).
- Christensen, R. P., et al. Untwisting the Caenorhabditis elegans embryo. eLife. 4, (2015).
- Santella, A., et al. WormGUIDES: an interactive single cell developmental atlas and tool for collaborative multidimensional data exploration. BMC Bioinformatics. 16, 189 (2015).
- Dupuy, D., et al. A first version of the Caenorhabditis elegans Promoterome. Genome Research. 14 (10B), 2169-2175 (2004).
- Reece-Hoyes, J. S., et al. Insight into transcription factor gene duplication from Caenorhabditis elegans Promoterome-driven expression patterns. BMC Genomics. 8 (27), (2007).
- WormBase. , Available from: https://www.wormbase.org (2019).
- Lee, R. Y. N., et al. WormBase 2017: molting into a new stage. Nucleic Acids Research. 46 (D1), D869-D874 (2018).



