Summary
Her presenterer vi en Kombinatorisk tilnærming ved hjelp av høyoppløselig mikroskopi, beregningsorientert verktøy, og enkelt celle merkingsteknikker i å leve C. elegans embryo å forstå enkelt celle dynamikk under neurodevelopment.
Abstract
Caenorhabditis elegans (C. elegans) skiller seg ut som den eneste organismen der utfordringen med å forstå mobilnettet opprinnelsen til en hel nervesystemet kan observeres, med én celle oppløsning, in vivo. Her presenterer vi en integrert protokoll for undersøkelse av neurodevelopment i C. elegans embryo. Vår protokoll kombinerer Imaging, lineaging og nevroanatomi sporing av enkeltceller i å utvikle embryo. Vi oppnår langsiktige, fire-dimensjonale (4D) avbildning av levende C. elegans embryo med nesten isotropic romlig oppløsning gjennom bruk av dual-View invertert selektiv Plane belysning mikroskopi (diSPIM). Kjerner og neuronal strukturer i nematode embryo er avbildet og isotropically smeltet for å gi bilder med oppløsning på ~ 330 NM i alle tre dimensjoner. Disse minutt-for-minutt høyoppløselige 4D datasett blir deretter analysert for å relatere definitive celle-avstamning identiteter med genuttrykk og morfologiske dynamikk på single-celle og subcellulære nivåer av detaljer. Vår protokoll er strukturert for å muliggjøre modulær implementering av hver av de beskrevne trinnene og forbedre studier på embryogenesis, genuttrykk eller neurodevelopment.
Introduction
C. elegans skiller seg ut som den eneste organismen der hver celle i fosteret kan observeres gjennom neurodevelopment. Med hele celle-avstamning kjent og invariant1, og med utviklingen av nye verktøy som tillater merking og kontinuerlig bildebehandling av enkeltceller i embryo, biologer kan nå begynne å undersøke ulike trinn i utviklingen av nematode nervøs system fra alle vinkler-celle fødsel; migrasjon og differensiering; neurite formasjon, målrettede utvekst og fasciculation; synapse formasjon; og tuning av funksjonelle kretser. Fange neuronal utvekst dynamikk i C. elegans embryo, ved å kombinere stabilt uttrykt journalister og fluorescens mikroskopi, er verdifullt for det vitenskapelige samfunnet.
Utviklingsstudier i C. elegans ofte utnytter invariant celle-avstamning og celle-skjebne kart av denne arten å øke kontekstuell forståelse på enkelt cellenivå i intakt organisme1. Auto-lineaging analyse-bruke StarryNite2,3,4 og AceTree5,6,7,8 programvare-fordeler fra høy kontrast, høy oppløsning bilder av fluorescerende kjerner. For å fungere optimalt, StarryNite/AceTree også avhengig av forutsigbar begrenset orientering av avbildet embryo under utvikling. Konfokalmikroskopi mikroskopi, gjort i C. elegans embryo komprimert mellom to coverslips, har vært standard Auto-lineaging mikroskopi metode for mer enn et ti år fordi det gir både høy kontrast/høy oppløsning og en forutsigbar begrenset orientering av embryo7,8. Vi har tidligere beskrevet bygging og bruk av en roman lys-ark-baserte dual-View invertert selektiv flyet belysning mikroskop (diSPIM) for Live sample Imaging som C. elegans embryogenesis9,10 , 11 flere , 12 flere , 13. lys-ark mikroskopi, generelt, gir lav Phototoksisitet, høy hastighet, og langsiktig Imaging av levende 3D prøver14,15. Den diSPIM metoden, spesielt, produserer fire-dimensjonale (4D) bilder med nesten isotropic romlig oppløsning på ca 330 NM9.
Sammenlignet med konfokalmikroskopi mikroskopi, tilbyr diSPIM høyere signal-til-støy og hastighet, mer isotropic romlig oppløsning, og er mer egnet for langsiktig in vivo Imaging16. Derfor arbeidet vi med å tilpasse diSPIM data for innspill til StarryNite/AceTree og undersøke om dette ville forsterke lineaging analysene. Et stort hinder er at diSPIM prøver kan ikke være lett begrenset av eggeskall-komprimering for å vedta forventet orientering for StarryNite/AceTree. Random orientering av celle posisjoner i volumet blir analysert forringer nøyaktigheten av Auto-lineaging analyse.
Vi derfor ansatt CytoSHOW, en seer-guidet bruker grenseflate hvilke innrømmer brukernes å velge akkurat 3D orienteringen av embryo i løpet av pre-bearbeiding av diSPIM profilen, gir image data det er begge to kvalitet-optimert og sammenheng-våken for input i StarryNite /AceTree. Ved bruker-valg av avbildet embryo, CytoSHOW appartefakter en automatisert databehandling rørledning. Bilder som trekkes ut og er trukket i bakgrunnen, lagres i TIFF-stack-filer for hver posisjon, timepoint og visning. CytoSHOW så iteratively kaller programmet SpimFusion å co-register og i fellesskap deconvolve de to pre-bearbeidet synspunkter, ved hjelp av Richardson-Lucy17,18 algoritme for å gi isotropic høyoppløselig volum bilder. En diSPIM-spesifikt sett med parametere er optimalisert for StarryNite å styre sin oppførsel under bilde-segmentering og nucleus-sporing i isotropically smeltet bilder. Smeltet bilder og lineaging resultatene blir deretter redigert ved hjelp AceTree, som tillater brukere å identifisere og fikse eventuelle feil i Auto-avstamning spor generert av StarryNite. AceTree kan også kan presentere avstamning-tre og 3D-modellerte gjengivelser av sporede kjerner i fosteret. Vi finner at Auto-lineaging hastighet og nøyaktighet er markert forbedret ved hjelp av isotropically smeltet bilder, sammenlignet med rå bilder fra enten SPIM kamera. Vår protokoll, mens optimalisert for C. elegans programmet beskrevet her, kan være generelt tilpasset for Auto-Lineaging av diSPIM data produsert for andre arter eller prøver. Hvis dette er den tiltenkte bruken av protokollen, Vær oppmerksom på at ytterligere tuning av StarryNite parametrene vil trolig være nødvendig for nye eksemplarer, som beskrevet3,4.
Vellykket gjennomføring av denne protokollen resulterer i bilder med 4D-isotropic oppløsning og lar biologer spore cellelinjene, samtidig identifisere og analysere neurons i utviklingsland C. elegans embryo. Videre, ved å slå sammen flere etterbehandling algoritmer-med hardware akselerasjon er den mest tidkrevende av disse-vi kan nå analysere både fine subcellulære detaljer og celle-linjene og celle-skjebne levende embryo i hovedsak sanntid. Denne nye protokollen gir presis, informert manipulasjon og observasjon av celle atferd under probative studier av differensiering og morphogenesis in vivo. I dette manuskriptet presenterer vi en detaljert forklaring av de forbedrede protokollene vi har utviklet for lineaging og celle sporing i utviklingen av C. elegans embryo, for å forbedre studier av embryogenesis, genuttrykk eller neurodevelopment.
Protocol
1. montering av diSPIM stål Imaging kammer med Poly-L-lysin belagt dekkglass
Merk: trinnene nedenfor er alle nødvendige for å optimalisere og automatisere avstamning analyse av nematode embryo av StarryNite/AceTree. Flere alternativer (angitt av slike) kan utelates for eksperimenter som krever sporing av C. elegans cellelinjene.
- Tegn et lite rektangel (2mm x 5mm) i midten av en ren rektangulær dekkglass (nr. 1,5, 24 mm x 50 mm) med en Sharpie (eller lignende penn).
- Vend dekkglass over til den umerkede siden og plasser 10 μL av Poly-L-lysin (Cat. no. P1524) over det markerte rektangelet.
Merk: Lag en fungerende konsentrasjon på 1 mg/mL Poly-L-lysin oppløst i filtrert vann (eller tilsvarende). For langtidsoppbevaring skal det tilberedes 5 – 10 mL alikvoter og oppbevares ved − 20 ° c. Etter tint kan alikvoter oppbevares ved romtemperatur (23 ° c) i 3 – 4 uker. - La Poly-L-lysin å belegge dekkglass i 5 minutter (figur 1a). Poly-L-lysin brukes til belegg glass deksel slips der embryo vil bli montert, slik at embryo eggeskall å holde fast ved dekkglass, selv når målene er nedsenket i Imaging medium.
- Plasser Poly-L-lysin belagt dekkglass i den nedre halvdelen av stål Imaging kammeret.
- Plasser den øverste halvdelen av stål bilde kammeret på den nedre halvdelen med dekkglass og stram med de fire skruene som er forbundet med kammeret. Kontroller visuelt fra siden for å være sikker på at den øverste halvdelen er jevnt plassert i nedre halvdel (figur 1B).
- Fyll kammeret med 7-8 mL av M9 buffer19, en isoton buffer som bidrar til å hindre tidlig stadium embryo fra succumbing til unormal osmotisk trykk. 1-celle, og selv 2-celle embryo er osmotically følsom og kan utvikle unormalt hvis ikke i denne isoton buffer. Embryo har også en tendens til å arrestere på 3-fold scenen hvis M9 er erstattet med vann.
2. forberedelse av C. elegans embryo prøver for montering
Merk: ca 18 timer før Imaging, fem unge (1 dag siden siste molt) voksne og ti larvestadiet etappe 4 (L4) C. elegans er plukket til en nematode vekstmedium (NGM) agar plate seeded med E. coli stamme OP50. Platinum wire plukker brukes til å flytte larver og unge voksne C. elegans uten å skade dyret19.
- Forbered en 1% methyl cellulose (Cat. no. H7509-25G) løsning i M9 buffer.
Merk: methyl cellulose må røres i varme M9 til den er oppløst. Når forberedt, kan denne løsningen lagres ved romtemperatur. - Tilsett 500 μL av 1% metyl cellulose-i-M9-løsning i depresjonen på et konkav mikroskop lysbilde. Denne tyktflytende bufferen vil bli brukt i to tilfeller: 1) når høsting tidlig embryo av Disseksjon av voksne ormer og 2) når du vasker sent stadium embryo plukket direkte fra en NGM plate.
Merk: metyl cellulose brukes til å hindre embryo fra stikker til mikroskopet lysbildet. - For Imaging sent stadium embryo, plukke lagt C. elegans embryo (fortrinnsvis fra en NGM plate med unge voksne til stede) ved hjelp av en øyenvippe hakke, og flytte embryo til 1% metyl cellulose på den konkave mikroskop lysbildet. Den øyenvippe hakke bidrar til å redusere kraft og dermed minimere stress eller skade på embryo under håndtering. Fremgangsmåten for å gjøre en øyenvippe hakke er dekket av Hart20.
- Med en andre øyenvippe hakke (i motsatt hånd), forsiktig tappe begge øyevipper sammen for å suspendere embryo i metyl cellulose.
- Valgfritt: Hvis du planlegger å avstamning embryonale celler med StarryNite, må man montere 1-celle til 4-celle embryo. For å gjøre dette, velger du først unge voksne fra en NGM plate og flytte dem inn i M9-metyl cellulose løsning på den konkave mikroskop lysbildet ved hjelp av en platina wire hakke.
- Valgfritt: med skjerpet tips av sprøyte nåler (no. 18G x 1 1/2), Slice dyret tvers på midten kroppen å løslate 1-celle til 4-celle embryo.
3. munningen pipette: montering aspirator tube med microcapillary pipette
Merk: vi bruker en aspirator tube med en hånd trukket microcapillary pipette settes inn i gummipakningen av røret. Dette tillater oss å overføre embryo fra disseksjon lysbilde til Poly-L-lysin-belagt overflate i buffer-fylte Imaging kammer.
- Trekk microcapillary pipette manuelt over åpen flamme for å lage to halvdeler med utstrakte spisser.
- Ta halvparten av microcapillary pipette og sett den butte enden inn i gummipakningen på aspirator røret (figur 1C). Sett den andre halvparten av microcapillary pipette til side for bruk senere (hvis nødvendig).
- Med montert aspirator tube utstyrt med microcapillary pipette, forsiktig bryte spissen av microcapillary pipette og skape en åpning som vil passe om 1-2 to embryo (herfra på dette instrumentet kalles en "munn pipette").
4. montering C. elegans embryo på Poly-L-lysin belagt dekkglass
- Med aspirator munnstykket holdt forsiktig mellom tennene, pre-fylle microcapillary pipette med 10-15 μL av M9 buffer og deretter forsiktig suge flere embryo fra den konkave lysbildet i kapillær.
- Overfør embryo til stål bilde kammer fylt med M9 buffer, posisjonering kapillær spissen slik at embryo vil falle inn i den sentrale rektangel av dekkglass.
- Unngå skade på embryo, forsiktig flytte dem med en øyenvippe hakke eller bruke munnen pipette å posisjonere embryo vertikalt, for å orientere embryo slik at den lange aksen av fosteret er vinkelrett på den lange aksen av dekkglass (figur 1B innfelt , nederste panelet).
Merk: posisjonering fosteret i denne orienteringen minimerer antall skiver til bildet, og dermed redusere lys dosering og databehandlingstid samtidig forbedre oppkjøpet hastighet. - Plasser stål bilde kammeret i prøveholderen på mikroskop trinnet (figur 1d).
5. montering, programvareoppsett og laser optimalisering for embryonale Imaging bruker diSPIM
- Se trinn-for-steg instruksjoner om hvordan å montere hele fiber-kombinert diSPIM fra kommersielt tilgjengelige deler i Kumar et al.10,11 og på http://www.dispim.org. En video-protokoll om hvordan du monterer diSPIM er også tilgjengelig på ASI-nettsiden (http://www.asiimaging.com).
Merk: instrument oppsett for denne protokollen er identisk med Kumar et al.10,11 diSPIM, som gjør bruk av 40x 0.8 na vann nedsenking linser for Imaging. Den eneste forskjellen mellom oppsettet i denne protokollen og Kumar et al.10,11 er tillegg av en dichroic speil (splitting på 560 NM) og røde og grønne bånd pass filtre inne i en tenkelig splitting enhet (modell A12801-01) installert på begge diSPIM bilde armer. Tillegg av bildet splitting optikk tillater samtidig fangst av bilder fra to distinkte fluorophores-opphisset av 561 NM og 488 NM lasere-ved å skille utslipps båndene på to halvdeler av samme kamera chip. - Etter at instrumentet er montert, kontrollerer du den optiske justeringen av diSPIM før bildebehandling.
Merk: for å justere diSPIM riktig, se https://youtu.be/qnOrg30NNuE, og for maskinvareinformasjon, http://dispim.org/hardware/objectives og http://www.asiimaging.com. - Bruk åpen kildekode-plattformen Micro-Manager (https://micro-manager.org/)21, som har blitt optimalisert for drifts lys Sheet mikroskop for høy gjennomstrømming Cellular Imaging22. Vi anbefaler bruk av ASI diSPIM-plugin-modulen for oppkjøp med flere posisjoner, som muliggjør samtidig bildebehandling på opptil 30 embryo som beskrevet23.
- Med Micro-Manager åpen, sett laser intensitet til ~ 179 μW (0,5) for 488 NM og ~ 79 μW (0,25) for 561 NM (figur 2a, rødt rektangel).
Merk: Dette er anbefalte innstillinger for lang tids avbildning av C. elegans embryo med 1-minutts intervaller. Under langsiktige dual-Color Imaging, den 561 NM laser brukes til bilde kjerner (mCherry:: histone) inntil embryo er på bønne scenen, noe som peker 488 NM laser er da slå på også bildet GFP-merket neurons. Disse bilde forholdene er optimalisert for å minimere Phototoksisitet og sikre overlevelse og klekking av embryo samtidig som det muliggjør forlenget (12 – 14 timer) kontinuerlig anskaffelse av neurodevelopmental og lineaging data. - I Micro-Manager, Velg menyen Plugins ≫ enhetskontroll > ASI diSPIM å åpne ASI diSPIM vinduet (figur 2b). Velg kategorien Anskaffelse . I denne kategorien data lagring innstillinger seksjon (grønn rektangel), voluminnstillinger seksjon (blå firkant), og Slice Settings seksjon (oransje firkant), sikre at hver parameter er satt som vist i figur 2b.
Merk: vår bildeanalyse programvare CytoSHOW er tilpasset for å arbeide med andre valgfrie output dataformater som bulk-sammensatt OME-TIFF-fil serien og TIFF-stack fil serien opprettet etter oppkjøpet gjennom bruk av en eksport funksjon innebygd i Micro-Manager. Vanligvis er single-time-Point-stack sortert OME-TIFF-fil data format brukes fordi det tillater sanntids visning og behandling av bilde volumet så snart rådata er ervervet.
6. optimaliserte autofokus parametre for lang tids avbildning av C. elegans embryo
- Sett Micro-Manager autofokus parametre til innstillingene optimalisert for lang sikt avstamning-kvalitet diSPIM-Imaging av C. elegans embryo. I ASI diSPIM-vinduet klikker du på kategorien autofokus (figur 2C). I delen generelle alternativer for autofokus (svart firkant) angir du parametere nøyaktig som vist. Legg merke til at autofokus-kanalen (rød firkant) bør spesifisere din kjernefysiske kanal fluorescens kanal i lineaging eksperimenter.
Merk: Hvis maks forskyvningen er større enn 5 μm, vil bildene ha en tendens til å drive ute av fokus. - Klikk, Plugins ≫ oppkjøpet verktøy > mønster overlegg.
- I vinduet Mønsteroverlapping klikker du på Vis rutenett.
- I ASI diSPIM -vinduet klikker du på navigasjon -fanen.
- Klikk avmerkingsbokser for stråle og ark med bane A eller B, og klikk deretter Live. Bildeinnhenting begynner. Et Live View-vindu åpnes. Velg den autofokus analyse regionen av fosteret ved å tegne en boks rundt embryo på den valgte kanalen fra 6,1.
Merk: vi vanligvis fange 420 tid poeng for 10 embryo per Imaging Session. Rådata per bilde økt er vanligvis 1,7 TB, mens deconvolved og StarryNite-behandlede data er 1,4 TB (se trinn 9 og 10). Vi anbefaler at du bruker HDD med stor kapasitet (18 TB på vårt nåværende system) for bildeinnhenting og Sky plattformer for bilde lagring. - Klikk på Start anskaffelse i fanen «Anskaffelse » for å starte langsiktig, flerdimensjonal bilde fangst (figur 2b).
7. åpning rå Micro-Manager bilder i CytoSHOW
- Last ned programvarepakken fra http://dispimlineage.wormguides.org.
Merk: programvaren bunten vil bli lastet ned som en. zip-fil og må pakkes ut til "C:\" katalogen før bruk. Ytterligere detaljer for installasjon er gitt på http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm. - Dobbeltklikk filen C:\CytoSHOWextrasForC\CytoSHOW_APP.jnlp å begynne å kjøre CytoSHOW.
- Velg meny fil > ny ≫ DiSPIM Monitor (Micro-Manager). Finn mappen for rot datasettet der timepoint mapper for anskaffelse ble lagret. Velg en timepoint mappe, og klikk Åpne. Flerdimensjonale navigasjons Vinduer (kalt diSPIM Monitor Windows) åpnes automatisk for både SPIMA og SPIMB (figur 3a).
Merk: disse vinduene vil overvåke rot data mappen for nylig lagrede rå timepoint stabler (i tilfelle at en prøve er fortsatt registreres). Etter hver ny timepoint er ervervet, hver av det Vinduer avlytting det adskilt SPIM armene og eksemplar posisjoner ville fornye å utfoldelse det hel flerkanals 4D datasettet for hver embryo.
8. generere Max projeksjon bilder med CytoSHOW
Selv før deconvolution kan rådata behandles raskt for å vurdere de globale funksjonene til prøven.
- Klikk på Z-MIP- knappen på venstre sidepanel i bildevinduet (figur 3a, rødt rektangel) for å gjøre maksimal intensitet anslag gjennom full dybde og full-time løpet av en gitt posisjon eller SPIM arm. Et Z-projeksjons hyperstack vindu vil vises.
- I vinduet Z-projeksjon hyperstack angir du projeksjons type til Maks intensitet. Angi hvilke kanaler, stykker og tids punkt rammer som skal behandles basert på brukerens preferanse.
- Falle i staver OK når fullført.
- Velge folderen plasseringen å bevare det Max intensiteten resultater fra filen dialogue vindu, og så falle i staver OK. Tillat en viss tid (15 – 20 minutter, avhengig av størrelsen på datasettet og behandlingskraften til maskinen) for CytoSHOW for å generere projeksjons bilder.
9. analysere cellelinjene i isotropic høyoppløselig volumdata
- Valgfritt: med rådata åpen via diSPIM-skjerm i CytoSHOW, velg polygon markeringsverktøyet (figur 3a, svarte piler) og klikk like utenfor embryo fremre, bakre, rygg og ventrale kanter (i den nøyaktige rekkefølgen) for å generere et "Bowtie" mønster over fosteret. Gjør for begge visningene (SPIM-A og SPIM-B, figur 3a).
Merk: Dette valget spesifiserer elliptiske regionen av interesse (ROI) der fosteret er sentrert og registrerer den fremre-bakre aksen av fosteret. Den Bowtie mønsteret stikkord CytoSHOW at brukeren planlegger å ytterligere angi en presis rotasjon av den endelige isotropically smeltet volumer i en orientering som er optimal for lineaging analyser av StarryNite/AceTree. I tilfeller der StarryNite lineaging ikke er en del av den eksperimentelle planen, kan andre Utvalgsverktøy og former velges for å sette AVKASTNINGEN for bildebehandling. - Hvis flere embryo ble avbildet ved hjelp av multi-position oppkjøpet alternativet, åpne og utføre trinn 9,1 for alle embryo. Dette vil tillate parallell kjøring av fremtidige skritt for alle embryo i en sesjon. Lukk SIMA og SPIMB Vinduer for alle embryo du ikke ønsker å behandle.
- Klikk på diSPIM -knappen på venstre sidepanel i diSPIM Monitor vindu (figur 3a, uthevet i gult). Dette avslører en subpanel av kontroller som er spesifikke for diSPIM-behandling.
- Juster grønne og røde kanaler for hver SPIM-arm. Fordi utslipps kanal splitter brukes til å fange distinkte røde og grønne bilder samtidig på samme kamera, er det viktig å visuelt justere nøyaktig piksel registrering av disse to fysisk tilstøtende bildefelt når de er overlappet. Gjenbruk av de samme justerings justeringene er vanligvis gjennomførbart over mange påfølgende bildebehandlings økter, men bør sjekkes (som i trinn 9.4.1 – 9.4.5).
- Fra og med SPIMA-panelet velger du den røde kanalen ved å flytte cm rullefeltet mot venstre (figur 3a, oransje pil, venstre panel).
- Bruk x-, y-og z-justerbar (figur 3a, oransje firkant), og Flytt den røde kanalen slik at den stemmer overens med greenen.
- Klikk diSPIM -knappen (figur 3a, uthevet i gult), for å lukke subpanel og utløse forplantning av de samme skiftene i alle andre stillings Vinduer.
- Bekreft at riktig justering er overført til andre rammer og tids punkt ved å flytte "z"-rullefeltet (figur 3a, blå pil, venstre panel) og/eller "t"-rullefeltet (figur 3a, grønn pil, venstre panel). Hvis multi-posisjon oppkjøpet ble utført og flere embryo ble avbildet (trinn 5,3), bør justeringen har også overført til de embryo. Bekreft ved også å undersøke tallene for x-, y-og z-justerbar (figur 3a, oransje firkant, som skal være den samme for SPIMA-panelet på alle embryo).
- Gjenta trinn 9.4.1 – 9.4.4 for SPIMB diSPIM Monitor vindu (fig. 3a, høyre panel).
- Klikk på "diSPIM"-knappen og deretter på"Fuse"-knappen (figur 3a, blå rektangel) for å åpne en dialogboks kalt "Deconvolve/Fuse DiSPIM RAW data volumer" (figur 3b). Angi parametere som vist i figur 3b. Disse parametrene er kort adressert i følgende substeps:
- Angi nøkkel registrering på kanal 1 (488 NM laser) eller 2 (561 NM laser). Velg kanalen med tettere eller mer allestedsnærværende signal. For lineaging eksperimenter alltid velge den kanalen som brukes til å avbilde allestedsnærværende kjernefysiske histone fluorescens.
- Sett inn datavolumer relativ orientering til + 1 eller-1. Riktig orientering indeksen avhenger av en bestemt diSPIM kamera plasseringer (Figur 4).
Merk: Hvis usikkert, test hvert alternativ ved å duplisere en enkelt timepoint fra både SPIM A og B diSPIM Monitor vindu, følgende trinn 9.1-9.12 og vilkårlig velge en Input volumer relativ orientering for å teste. Feil orientering vil gi uklare bilder med artefakter, mens riktig orientering vil gi klare bilder. Den relative orienterings verdien for inn datavolumer som gir det klare bildet, kan deretter brukes på nytt for alle fremtidige data fra det gitte diSPIM-instrumentet. - Velg om det smeltet volumet skal være det samme som inn datavolumet A eller B (basert på brukerens preferanse).
- Velg "Fresh registrering for hvert volum". Dette alternativet kontrollerer hvordan SpimFusion beregner registrerings matriser for hvert volum par på hver timepoint. "Fresh" alternativet lar algoritmen optimalisere registreringen bearbeides og på hver timepoint.
- Angi antall deconvolution gjentakelser til 10. Dette tallet har en tendens til å pålitelig gi ønsket høy oppløsning på en tid-effektiv måte.
- Valgfritt: Hvis auto-lineaging er ønsket (anbefales), sjekk Auto-Launch StarryNite lineaging av smeltet volumer. Dette alternativet vil lansere StarryNite automatisk å segmentere og spore celler i avbildet volumer produsert av SpimFusion.
- Valgfritt: for høyeste nøyaktighet i automatiserte lineaging, er det best å omplassere isotropically smeltet embryo volumer i den kanoniske "ADL" (Anterior [x-West], Dorsal [y-Nord], venstre [z-nær]) orientering. Velg alternativet Definer volum utskriftsretning i forhåndsvisning for å angi dette valget. CytoSHOW vil svare ved å behandle en innledende par isotropically smeltet volumer, slik at brukeren kan nøye observere og angi rotasjoner som trengs for å oppnå ADL registrering.
- Klikk Ja når alle parametere er valgt.
- Angi output-mappen der de behandlede filene skal lagres. Klikk på OK.
- Valgfritt: Hvis alternativet Definer volum utskriftsretning i forhåndsvisning er valgt, angir du t rullefeltet (figur 3a, grønn pil, venstre panel) i SPIM-A-vinduet til den tidlige timepoint der ABa og ABp-cellene har nådd metafase. Sette det t rullet stang inne det SPIM-B vindu å det siden komma scene av utviklingen. Dette vil hjelpe deg med å angi ADL-orientering.
- Valgfritt: Klikk OK når du er klar. Hvis forhåndsvisningsalternativet i 9.5.7 ovenfor ble valgt, vil bare to forhåndsvisning volumer bli isotropically smeltet for tidspunkter som indikeres av t-glidebryterne i bilde vinduene SPIM-A og SPIM-B. Disse to forhåndsvisning tidspunkter kan brukes til å angi presis omstilling av produksjonen embryo volumer til ADL orientering, som forklart nedenfor.
- Finn den nylig viste 3DProjY_Decon-Fuse_.... Vinduet. Flytt t rullefeltet til tidspunkt punkt 2 i dette forhåndsvisningsvinduet. Flytt glidebryteren Z til visningen rett ned den lange aksen til fosteret vises.
- Bevege det t rullet stang rygg å tid punkt 1 av det 3DProjY_Decon-Fuse_.... Vinduet. Velg linje merkings verktøyet og tegn en linje markering fra EMS-cellen (ventrale-mest runde kjerne) gjennom planet på AB-Cell-metafase plater.
- Klikk på Orange diSPIM preview-knappen på 3DProjY_Decon-Fuse_.... Vinduet. Den fine justeringer i retning av forhåndsvises avbildet volum vil bli lagret for bruk i behandling av hele datasettet.
- Valgfritt: dialogboksen Deconvolve/Fuse DiSPIM RAW data volumer vises på nytt, akkurat som i trinn 9,5 ovenfor. Klikk Ja uten å velge Definer volum utskriftsretning i Forhåndsvisnings alternativet. Angi utdata-mappen for hele databehandlings kjøringen.
- Sett t rullefeltene (figur 3a, grønn pil, venstre panel) av diSPIM Monitor vinduer til Start timepoint (SPIMA) og ending timepoint (SPIMB) av hele span av bilder å behandle. Deretter klikker du OK.
- Som SpimFusion skrider frem, CytoSHOW åpner og oppdaterer et flerdimensjonalt vindu som viser skiver-4D isotropic smeltet volum for hvert embryo, samt to Vinduer med roterende-4D Max-intensitet anslag av isotropic volum. I løpet av denne tiden må du ikke avbryte eller lukke noen av CytoSHOW-vinduet før isotropic fusjon og slekts sporing er fullført.
- Valgfritt: Merk at når StarryNite splash-skjermen har dukket opp og senere forsvant, full data-prosessering rørledningen er fullført. Dette vinduet må ikke lukkes under behandling, eller StarryNite vil bli avbrutt.
10. åpning StarryNite avstamning Trace serien i AceTree (valgfritt)
- Åpne den tilpassede versjonen av "AceTree_16BitCompat. jar" forutsatt.
- Velg meny filen > åpne Konfigurer fil. Finne din produksjon adresseliste tidligere angitt å CytoSHOW. Åpne Decon_Fuse_... _Pos [n] undermappe for embryo [n]. Velg aaa_edited. XML og Åpne.
- Bruk AceTree menyen rediger > Rediger verktøy for å åpne Rediger spor og justere eller slette celler Vinduer.
- Klikk den halv-fargede sirkelen figur 5B, rød firkant for å justere røde og grønne intensitet.
- Fortsett med avstamning visualisering og redigering som tidligere beskrevet5,6,8 (manuskripter er også inkludert i vår Download Bundle).
Representative Results
Vi først validert levedyktigheten til embryo avbildet ved hjelp av protokollen parametere for diSPIM oppkjøp (seksjoner 1-6). Ti embryo ble samtidig avbildet ved 20 ° c, ett volum/embryo/minutt, fra 2-celle scenen til 2-fold scenen (7,5 timer, 451 volumer/embryo). For å overvåke celle divisjoner i hele embryogenesis, brukte vi belastning BV514, som overalt uttrykker mCherry::histone reporter konstruerer fra den integrerte transgene array ujIs11324. Figur 6 viser en tidslinje av denne første halvdelen av embryonale utviklingen for en av de avbildet embryo. Hvert bilde representerer en enkelt-visning maksimal intensitet projeksjon (produsert av trinn 7-8) av avbildet embryo. Vi fant at de optimaliserte protokollene ikke induserer noen synlig Phototoksisitet til embryo, som vurdert av tidspunktet for celle divisjoner (ikke vist), tid for klekking, og timing knyttet til utviklingsmessige milepæler (figur 6 og referanser1 , 25 priser og , 26).
Vi deretter brukt protokollen til å analysere utvekst dynamikk av enkle neurons i å utvikle embryo. Vi avbildet DCR7692 (olaex4655), en transgene nematode stamme som uttrykker GFP av neuropeptide FLP-19 promoter i et delsett av uidentifiserte celler (DACR2819, PFLP-19 (3.6 KB):: Syn21:: GFP:: CAAX::p 10 3 ' UTR) . Etter trinnene i protokollen skissert her, vi fastslått at de uidentifiserte cellene tilsvarer motor neurons RMDDL og RMDDR, til excretory kanalen cellen, og to muskelceller (figur 7). Vi deretter undersøkt og kvantifisert den utvekst dynamikken i RMDDL og RMDDR neurons. Vi observerte at RMDDL og RMDDR neurons er skrått formet så tidlig som 360 minutter etter befruktning, med lengre mobilnettet aksen representerer den påfølgende aksen for neurite utvekst (figur 7 og film S1). Bruke "enkle neurite tracing" plugin i FIJI og bruke den til 3D rekonstruksjoner av isotropically smeltet volumer, vi da kvantifisert den stereotypic utvekst av RMDDL og RMDDR neurites for seks embryo. Vi bestemte at utvekst dynamikk var stereotype for RMDDL og RMDDR på tvers av embryo (heretter kalt RMDDs). Fra 385-410 minutter etter befruktning, RMDDs neurites utvidet 6,0 ± 0,5 μm (gjennomsnittlig ± SEM; n = 12 neurites) fremre celle organer (figur 7b, C, I). Fra 415-445 minutter etter befruktning, både neurites forlenge dorsalt inn og rundt presumptive nerve ringen (stjerne i figur 7d). I gjennomsnitt hver RMDD neurite utvidet 11,0 ± 0,6 μm (gjennomsnittlig ± SEM; n = 12 neurites) fra cellen kroppen før synkront møte sin kontralateral motstykke på toppen av ringen (figur 7I). Viktigere, våre representative resultater viser at vi er i stand til å undersøke, sammenligne og kvantifisere neuronal utviklingsmessige funksjoner for enkelt identifiserbare celler ved hjelp av vår integrerte protokollen (figur 7 og Figur 8).
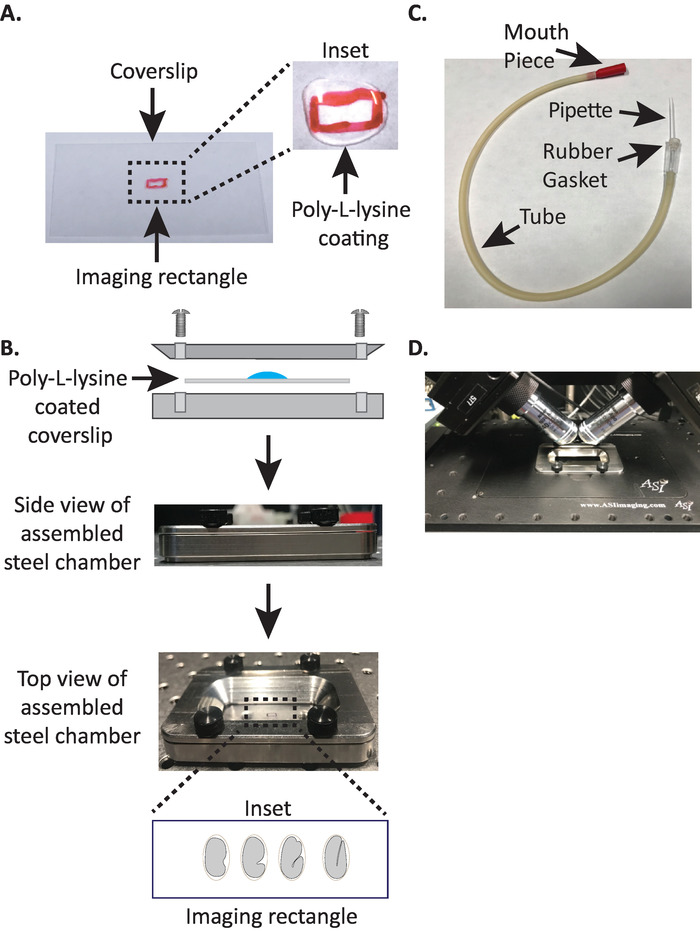
Figur 1: diSPIM for prøve installasjon. (A) utarbeidelse av dekkglass med Poly-L-lysin. I innfelt, 10 μL av Poly-L lysin ble brukt til å belegge dekkglass i 5 minutter. Poly-L-lysin tillater embryo eggeskall å stikke fast til dekkglass i rektangelet. (B) skjematisk av stål bilde kammeret og det sammensatte kammeret. I innfelt, representative embryo er orientert med fremre bakre akse vinkelrett på den lange aksen på dekkglass. (C) montert aspirator rør med microcapillary pipette. (D) Steel Imaging kammer montert i prøve holderen under diSPIM 40x mål. Vennligst klikk her for å se en større versjon av dette tallet.

Figur 2: langsiktig diSPIM Imaging setup i Micro-Manager. (A) anbefalt diSPIM laser kraft parametere (rød rektangel) optimalisert for langvarig bildebehandling samtidig redusere Phototoksisitet (som vurdert av høyere klekking rate på C. elegans embryo). Sett 561 NM laser til 79 μW (0,25) og 488 NM laser til 179 μW (0,5). Vær oppmerksom på at nøyaktig kalibrering av programvareinnstillinger til laser strøm varierer mellom diSPIM-installasjoner. Det anbefales at brukere måle og kalibrere laseren makt for å oppnå 79 μW (561 NM) og 179 μW (488 NM) laser kraft. (B) diSPIM parametere for datalagring (grønt rektangel), voluminnstillinger (blå firkant) og skjære innstillinger (oransje firkant). (C) diSPIM autofokus parametere for lang tids avbildning av C. elegans embryogenesis (se trinn 6.1-6.6). Vennligst klikk her for å se en større versjon av dette tallet.

Figur 3: bilde visualisering og oppsett av databehandling ved hjelp av CytoSHOW. (A) RAW diSPIM bilder åpnet av CytoSHOW. CytoSHOW er i stand til å åpne bilder tatt av både kamera baner (SPIM A og B). Disse rå bildene åpnes i flerdimensjonale Vinduer kalt diSPIM Monitor. I diSPIM Monitor, en "Bowtie mønster" er generert for å velge embryo ' s fremre, bakre, rygg og ventrale kanter (se trinn 9,1). Valg for baug tilknytning angir embryo orientering for deconvolution og StarryNite-assistert lineaging sporing. (B) optimaliserte parametere som brukes til å generere isotropic bilder. Angi parameterne som er angitt i trinn 9.5.1-9.5.8, i Deconvolved mens du henter vinduet. Vennligst klikk her for å se en større versjon av dette tallet.

Figur 4: diSPIM kamera konfigurasjon. (A) fotografi av diSPIM kamera plasseringer og orientering. (B) skildring av + 90-graders ROTASJONER av SPIM A for å matche SPIM B-bilder innsamlet. (C) input volumer i forhold til orientering indeks + 1 basert på vår diSPIM ' s kamera konfigurasjon (se trinn 9.5.2). Vi roterer SPIM et bilde (r) + 90 grader rundt Y-aksen før registreringen for å samsvare med SPIM B-bilde (r). Skala bars = 10 μm. bilder er representative single-View, maksimal intensitet anslag og deconvolution bilder av 1,5-fold embryo med merket kjerner (561-NM, rød) og neurons (488-NM, grønn). Vennligst klikk her for å se en større versjon av dette tallet.

Figur 5: Håndplukking og redigering C. elegans embryonale avstamning i AceTree. (A) vi bruker AceTree å redigere StarryNite ' s avstamning spor (se referanser5,6,8; manuskripter er også inkludert i vår Download Bundle). AceTree vil vise C. elegans systematiske navn for hver kjerne (grønt rektangel) ved fullføring av trinn 10.1-10.2. Dette vinduet (A) gir informasjon (svart rektangel) om hver celle i slekts sporet (ABa, uthevet i blått) som veileder brukerne når de sporer og redigerer slekts sporene. Det anbefales at brukerne verifisere og sammenligne lineaged celler og deres posisjoner til C. elegans embryonale celle avstamning tidligere rapportert av Sulston et al.1 i tillegg, hvis brukerne er interessert i å finne bestemte celler i deconvolved dataserie (se nedenfor, B) Skriv inn C. elegans systematisk navn i søkefeltet (oransje rektangel). (B) brukerens deconvolved dataserie åpnes også automatisk ved fullføring av trinn 10.1-10.2. Vist her en isotropically smeltet bilde av en fire-cellers stadium embryo med kjerner merket i rødt. Under sporing av en kjerner, bør brukerne endre intensiteten i bildet (rød firkant) og Naviger gjennom tid og z ved hjelp av piltastene på tastaturet (tid-venstre/høyre, z-opp/ned). (C) 3D-tegneserie av timepoint i (B) med visse funksjoner (lilla rektangel) som muliggjør Roterbar 3D-visualisering. Hvis du vil ha en oversikt over AceTree og 3D-funksjonaliteten, kan du se referanser5,6,8. Vennligst klikk her for å se en større versjon av dette tallet.

Figur 6: tidsbestemt utviklingsmessige dynamikken i C. elegans embryo på diSPIM. Top panel, diSPIM bilder som viser første halvdel av embryonale utviklingen for en av avbildet embryo (stamme BV514 ujIs11324). Embryo ble avbildet kontinuerlig, hvert minutt for 7,5 timer (ved 20 ° c). De to første bildene av toppen panel representerer 4-og 8-cellers embryo med kjerner (rød) og posisjoner av polare kropper (tette røde kuler, merket med blå stjerner). Hvert bilde representerer en enkelt-visning maksimal intensitet projeksjon av avbildet embryo. Vektstenger = 10 μm. Tidslinjen (horisontal bar) representerer minutter etter befruktning (m.p.f.) av utviklingen av C. elegans embryo. Vi validerte at vår protokoll parametre for diSPIM oppkjøpet ikke indusere noen synlig Phototoksisitet til avbildet embryo som vurderes av levedyktighet, timing av celle divisjoner, tidspunkt for klekking og timing av utviklingsmessige milepæler (se referanser 1 den andre , 25 priser og , 26). vi registrerer at tidspunktet for utviklingsmessige milepæler var reproduserbar over embryo med våre Imaging parametere (SEM ± 8,174 minutter for 6,4 timers lange Imaging økter; n = 10). Vennligst klikk her for å se en større versjon av dette tallet.

Figur 7. Cellular identifisering og enkelt celle karakterisering av neurite utvekst dynamikk i utviklingen av C. elegans embryo. Dual-farge bilde av en stamme gjort ved å krysse BV514 ujIs11324 (for lineaging) og DCR7692 (olaex4655), en transgene NEMATODE stamme som uttrykker GFP av neuropeptide FLP-19 promoter i en undergruppe av uidentifiserte celler. (A-H) Etter trinnene i protokollen skissert her, vi fastslått at de uidentifiserte cellene tilsvarer motor neurons RMDDL og RMDDR (gule piler), til excretory kanalen cellen (blå piler), og to muskelceller (hvite piler). (I) kvantifisering av utvekst DYNAMIKKEN i RMDDL og RMDDR neurons ved å bruke FIJI plugin "enkle neurite tracing" og bruke den til 3D-rekonstruksjoner av isotropically smeltet volumer. Legg merke til hvordan både RMDDL og RMDDR viser stereotypic utvekst dynamikk, hver utvide synkront for en total lengde på 11.0 ± 0,6 μm (gjennomsnitt ± SEM; n = 12 neurites) og møte på rygg toppen av nerve ringen (se også film S1). Vennligst klikk her for å se en større versjon av dette tallet.

Figur 8: undersøkelse av isotropic diSPIM bilder av neuronal morfologier i C. elegans embryo. Isotropic visualisering av AVHL og AVHR neurons (gule piler). Bruk av diSPIM, neuronal morfologier kan fanges gir fire-dimensjonale (4D) bilder med isotropic romlig oppløsning på ca 330 NM. DiSPIM tillater brukere å nesten rotere bilde volumer med identisk oppløsning i alle retninger. Bilder i A-D er anslag for maksimal intensitet av samme isotropically smeltet diSPIM bilde volum fra distinkte rotasjoner rundt embryo lange aksen. Skala bars = 5 μm. Vennligst klikk her for å se en større versjon av dette tallet.
Supplerende Movie S1: C. elegans embryoutvikling fra 280 til 434 minutter etter befruktning. Isotropic film av belastningen DCR7692 (olaex4655) uttrykker UJIS113 overalt med DACR2819 tynt merking RMDD neurites (figur 7a-D, gule piler). DACR2819 også etiketter to muskelceller (figur 7a-d, hvite piler) og excretory kanal celle (figur 7a-d, blå pil) under embryonale utviklingen (figur 7a-d). Skala bars = 10 μm. Vennligst klikk her for å laste ned denne filen.
Discussion
C. elegans skiller seg ut som den eneste organismen med den endelige posisjoner og tilkobling av hver voksen Nevron kjent27. Men den utviklingsmessige dynamikken fører til organisering av arbeider kretser og nettverk som utgjør C. elegans connectome forblir ukjent. Basert på muligheter som kommer ut av fremskritt innen lys mikroskopi, kan vi nå fange opp og analysere celle posisjoner, morphogenesis og neurogenesis gjennom hele C. elegans embryoutvikling.
Prosedyren som vi har beskrevet og at vi rutinemessig bruk i laboratoriet gir 4D-isotropic bilder av merkede neurons og kjerner for celle-lineaging i C. elegans embryo. Enda viktigere, vi har optimalisert langsiktige Imaging forhold med diSPIM og kombinert semi-automatiserte lineaging evner med høyoppløselige bilder for å forbedre hastigheten og presisjonen av å analysere C. elegans embryogenesis. Denne integrert protokollen ville sette i stand brukernes å visualisere og identifisere celler og quantitate tre-dimensjonale vise egenskaper som neurite Migration og axon fasciculation igjennom utbruddet av tidlig beveger. Denne fremgangsmåten kan lett tilpasses alle anlegg med et ASI diSPIM-system, og vi anbefaler dette systemet spesielt for denne protokollen. Andre SPIM-formuleringer som tilbys kommersielt kan avvike fra ASI-konfigurasjonen i prøvekammeret og de optiske egenskapene. Data som eksporteres fra andre plattformer, kan imidlertid også legges gjennom datasamlebåndet. Derfor er vurdering av deres verdi i lineaging, en krevende test av bildekvalitet og instrument stabilitet, gjennomførbart. Selv om vi aktivt bruker diSPIM å regelmessig bilde andre eksemplarer (for eksempel Drosophila og sebrafisk embryo), den beskrevne og omfattende lineaging analyse av embryo er fortsatt for tiden begrenset til nematode arter. For større eller tykke prøver, velger vi å bruke scene-skanning tilnærminger, som skanner prøvene gjennom en stasjonær lys ark. Kumar et al. har tidligere demonstrert denne forbedrede diSPIM-snitting for å gi bilder av høy kvalitet fra tykke prøver uten ytterligere modifikasjoner på diSPIM10.
De kritiske trinnene i protokollen inkluderer montering C. elegans embryo på Poly-L-lysin belagt dekkglass, datainnsamling, og databehandling. Høsting og montering C. elegans embryo på glass dekkglass kan være utfordrende for uerfarne brukere, men her gir vi en detaljert protokoll av viktige trinn for å lette læringen. Hvis langsiktige Imaging er ønsket, får vi best resultat høsting fire-celle eller tidligere embryo fra 8-10 unge voksne28. Merk at gamle voksne er mindre ønskelig å høste tidlig stadium embryo fordi de har en tendens til å inneholde eldre embryo i livmoren og ubefruktede egg. I forhold til montering embryo, problemer som blokkering i den sammensatte aspirator (munn pipette) eller en altfor stor av en åpning i microcapillary pipette kan hindre riktig montering og orientering av embryo. For å forberede for optimal bildebehandling, utfører vi pre-oppkjøpet testing på tidlig og sent pre-rykninger embryo å sjekke ytelsen til lys ark, kameraer, mål og autofokus. Vi får best resultat når alle disse operasjonene er testet og gir bilder av høy kvalitet i løpet av vår pre-oppkjøpet testing. Dette er spesielt relevant for generering av bilder med isotropic romlig oppløsning, der rå bilder ervervet fra begge visningene (målsettinger) må være av høy kvalitet. Etter oppkjøpet blir volumene som er ervervet for hver visning, behandlet for å gi isotropic bilder. Det er viktig å bruke et riktig grafikkort (GPU) som beskrevet i denne protokollen (se nedenfor). Dette forbedrer behandlingshastigheten som isotropically smeltet bilder genereres, forkorte tiden til dataanalyser. Det er også viktig at brukerne kjører den nyeste versjonen av CytoSHOW og bruker parametrene som følger med vår nedlasting bunt for StarryNite Auto-lineaging. Hvis brukere er interessert i å bruke auto-lineaging for andre prøver (f.eks. sebrafisk, Drosophila osv.), vil ytterligere optimering av parametrene som brukes i StarryNite være nødvendig (se referanser3,4).
Selv om våre integrerte protokollen gir bilder og lineaging resultater i pre-rykninger embryo, brukere bør være klar over at automatiserte lineaging i post-rykninger embryo er ikke gjennomførbart: kjernefysiske posisjoner endringer på rekkefølgen av sekunder i post-rykninger embryo, for raskt å tillate avstamning sporing. Imidlertid har diSPIM faktisk vist en lovende evne til å fange neurodevelopmental hendelser og spore noen celle posisjoner i de post-rykninger stadier av embryogenesis23,29. Hvis brukerne er interessert i å undersøke post-rykninger embryo, den diSPIM gir hastigheten for å få volum øyeblikksbilder og spore fine neurodevelopmental hendelser, for eksempel neurite utvekst, i raskt bevegelige embryo.
Denne protokollen vil være grunnleggende for celle-for-celle ferdigstillelse av WormGUIDES Atlas30, som det vil gi en integrert tilnærming med høy oppløsning isotropic bilder for å identifisere og fange 3D morfologier av merkede neurons under de første 430 minuttene av embryogenesis. Som det står, prototypen WormGUIDES Atlas gir kjernefysiske posisjoner av celler i utviklingsland embryo og har som mål å fange den utviklingsmessige dynamikken i en undergruppe av embryonale neurons. Denne protokollen vil være en nøkkel for integrering av ytterligere utvikle neurons i WormGUIDES Atlas30.
Vår integrerte protokoll vil også forenkle utforskningen av nye gen uttrykks profiler i C. elegans embryo. I transgene C. elegans, mange celle-spesifikke arrangører romlig og timelig kontroll transgene uttrykk. Mens uttrykket mønstre av de fleste gener har vært omfattende preget i den voksne dyret31,32,33,34, nesten alle har ennå ikke vært preget i utviklingsland (spesielt sen-Stage) embryo. C. elegans promoterome har vært en nyttig ressurs for ormen samfunnet å kjøre celle-spesifikke transgene uttrykk, samt avgjøre om gen funksjonen er celle-autonome eller ikke-autonome. Fange isotropic høyoppløselig og dynamisk uttrykk mønstre av gener, og presist identifisere uttrykke celler via lineaging vil være verdifullt for mange i det vitenskapelige samfunnet.
Embryogenesis består av to sammenflettede store prosesser, cellulær differensiering og vev morphogenesis. En god del er kjent om mekanismer og molekyler som definerer forskjellige celletyper under utviklingen av C. elegans. Men lite er kjent om mekanismene viktig for celle migrasjon, celle vedheft, og celle form i C. elegans embryo. Med C. elegans invariant celle avstamning kjent, vår protokoll lar oss lett skjelne katalogisert 3D-microanatomy av fosteret under morphogenesis på nye nivåer av detaljer: for eksempel, axon fasciculation, synaptogenesis, og neuronal aktivitet. Ardiel et al. har tidligere demonstrert kraften i diSPIM å fange kalsium transienter på nivå med en enkelt neurons i C. elegans embryo23. Mange andre aspekter av utviklings fysiologi er moden for undersøkelse av disse metodene.
Til slutt, denne protokollen er i stor grad automatisert og systematisk reduserer tiden det tar å generere deconvolution bilder og utføre celle-lineaging via StarryNite og Acetree. Programvaren strategiene som brukes i denne protokollen kan brukes på mange spørsmål om biologi fjernt fra de svært spesifikke felt der vi har vist dem her.
Detaljer om programvare kompatibilitet og nedlasting tilgang
Informasjon om Micro-Manager og plugins for diSPIM Imaging er tilgjengelig på http://dispim.org/software/micro-manager og https://micro-manager.org/wiki/ASIdiSPIM_Plugin.
Pipeline for databehandling krever for øyeblikket et Windows-operativsystem. Vi har samlet en enkelt arkiv fil for å forenkle installasjonen av alle nødvendige data-prosessering programmer og støttefiler. Den er tilgjengelig for nedlasting på http://dispimlineage.wormguides.org.
CytoSHOW (http://run.cytoshow.org/) er basert på mye brukt og åpen kildekode bildeanalyse plattform, ImageJ (v1). Java må være installert og oppdatert på datamaskinen for å bruke CytoSHOW, og oppdateringer til CytoSHOW distribueres automatisk via Java Web Start. Mange ImageJ-baserte funksjoner i CytoSHOW er som beskrevet og illustrert på https://imagej.nih.gov/ij/docs/examples/index.html. CytoSHOW har blitt tilpasset for å vise flerdimensjonale rådata fra ASI diSPIM, samt annen tenkelig programvare som skaper TIFF-utdata. I prinsippet kan andre multi-View SPIM Imaging systemer støttes av mindre modifikasjoner av CytoSHOW å tillate denne protokollen skal utføres på ulike mikroskop systemer.
SpimFusion ble skrevet i CUDA/C++ ved hjelp av Visual Studio 2013 med CUDA Toolkit v 7.5. Running SpimFusion krever spesifikk maskinvare: et NVIDIA-grafikkort (GPU) med CUDA databehandlings funksjon 1,0 eller høyere og minimum 2 GB grafikkort minne. På tidspunktet for utgivelsen av vår protokoll, SpimFusion er upublisert (min Guo og Hari Shroff), men tilgjengelig i programvaren bunt arkivet nevnt ovenfor.
En spesielt bygget kommandere-line kjørt versjon av StarryNite behøver det det fritt anvendelig MATLAB kompilatoren Runtime er installert, bortsett fra er ikke forlange en med lisens for reklamen MATLAB programvare. Den MATLAB Compiler Runtime er inkludert i programvaren bunt arkivet nevnt ovenfor. Koden for StarryNite som brukes i denne protokollen er i hovedsak uendret fra den som brukes for konfokalmikroskopi bilder6. Imidlertid har flere operative saker i etableringen av innspill bilder for StarryNite prosessering og håndtering av StarryNite resultater er adressert her ved metoder i CytoSHOW som muliggjør en kontinuerlig databehandling rørledning for smeltet isotropic diSPIM Volumer. Disse endringene er automatisert av CytoSHOW kode som håndterer disse pre-og post-prosessering trinn. CytoSHOW også redigerer en pre-optimalisert diSPIM-spesifikke mal StarryNite parameter satt til å automatisk tune den segmentering algoritmen til fluorescens intensiteten av kjerner i avbildet data. De unike parametrene som brukes av StarryNite på hvert diSPIM datasett blir deretter lagret i en fil sammen med output image og lineaging data.
En tilpasset versjon av AceTree som fungerer med 16-bits bilder og opprettholder kompatibilitet med Java3D rendering er best egnet for denne protokollen. Det er også inkludert i programvaren bunt arkivet nevnt ovenfor.
Disclosures
Forfatterne har ingenting å avsløre.
Acknowledgments
Vi takker John Murray for integrert belastning, ujIs113, å generere lineaging stamme BV514; Brandon Harvey (NIBIB) for å få hjelp til å teste protokollen; Jon danske og Gary Rondeau (anvendt naturvitenskapelig Instrumentation) for assistanse med mikro--bestyrer og diSPIM apparat; og Andrew York og Hank Eden for deres kritiske tilbakemeldinger på diSPIM systemet. Vi takker også Research Center for minoritets institusjoner programmet og Instituto de Neurobiología Jose del Castillo (Universidad de Puerto Rico) for å gi et møte og brainstorming plattform. Mye av dette arbeidet ble gjennomført ved Marine biologiske laboratorium i Woods Hole gjennom Whitman program. Dette arbeidet ble støttet av intramural Research Programs i NIH National Institute of Biomedical Imaging and bioteknologi og ved NIH Grant nei. U01-HD075602 og nei. R24-OD016474. Markus W. Moyle ble støttet av F32-NS098616 og Leighton H. Duncan ble støttet av et mangfold supplement til R24-OD016474.
Materials
| Name | Company | Catalog Number | Comments |
| Steps 1-4 | |||
| Concavity slides | ThermoFisher Scientific | 1519006 | 5-18mm diameter, 0.6-0.8mm deep, 1.2-1.5mm |
| Dissecting microscope with 10×–50× zoom range | Motic | SMZ-171 | |
| E. coli (OP50) | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Glass coverslips, no. 1.5, 24 × 50 mm | VWR International | 48393-241 | |
| M9 Buffer | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Methyl cellulose | Sigma-Aldrich | H7509-25G | |
| Microcapillary pipette aspirator tube | Sigma-Aldrich | A5177 | |
| Microcapillary pipettes, 0.4-mm i.d | Drummond Scientific | 1-000-800 | |
| Needle, no. 18G x 1 ½ (1.2mm x 40mm) | BD Precision Glide | 305196 | |
| NGM plates | prepared as described by Brenner (1974) | ||
| O-ring for imaging chamber | O-Rings West | M1.5X40 | |
| Pasteur pipette | Corning/Sigma-Aldrich | CLS7095D5X | |
| Platinum wire, 0.5-mm diameter | Sigma-Aldrich | 267201 | |
| Poly-L-lysine | Sigma-Aldrich | P1524 | |
| Stainless steel rectangular chamber (76.0 mm x 50.5 mm) | Applied Scientifics Instrumentations (ASI) | I2450 | |
| Worm Eyelash Pick | Hart, A. C. Behavior. WormBook. (2006). | ||
| Worm Pick | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Name | Company | Catalog Number | Comments |
| Steps 5-6 | |||
| 488 nm long-pass filter | Semrock | LP02-488 RU-2 | |
| 561-nm notch filter | Semrock | NF03-561E-25 | |
| BLP02-561R-25, quantity 2 | Semrock | 561 nm EdgeBasic best-value long-pass edge filter | |
| Control software for bottom camera | Jenoptik | ProgRes CapturePro | |
| diSIPM assembly video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/TAgbr6IrTqw ; http://www.asiimaging.com | |
| diSPIM alignment video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/qnOrg30NNuE | |
| diSPIM imaging PC | Intel | Intel Xeon CPU E5-2630 2.6GHz, 12 cores in total, 64 GB memory, Windows 7 | |
| FF01-525/45-25, quantity 2 | Semrock | 525/45 nm BrightLine single-band bandpass filter | |
| FF555-DI03-25X36, quantity 2 | Semrock | 555 nm edge BrightLine single-edge dichroic beamsplitter | |
| Imaging PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Kumar et al diSPIM Setup | Applied Scientifics Instrumentations (ASI) | Instrument setup for this protocol is identical to Kumar et al 10,11 diSPIM, which makes use of 40x 0.8NA water immersion lenses for imaging. (See steps 5.1 and note) | |
| Micro Manager | Micro-Manager | https://micro-manager.org/ | |
| Modifications to Kumar et al diSPIM Setup (see below) | |||
| Optical table with isolators, 4 feet × 6 feet × 12 inches | TMC | 784-651-02DR and 14-416-34 | |
| Name | Company | Catalog Number | Comments |
| Steps 7-10 | |||
| Analysis PC | Intel | Intel Core i7-8700K CPU 3.70GHz, 6 cores in total, 64 GB memory, Windows 10 | |
| Analysis PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Installation instructions | Software bundle | http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm | |
| Software bundle | Software bundle | http://dispimlineage.wormguides.org |
References
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology. 100 (1), 64-119 (1983).
- Bao, Z., et al. Automated cell lineage tracing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 103 (8), 2707-2712 (2006).
- Santella, A., Du, Z., Bao, Z. A semi-local neighborhood-based framework for probabilistic cell lineage tracing. BMC Bioinformatics. 15, 217 (2014).
- Santella, A., Du, Z., Nowotschin, S., Hadjantonakis, A. K., Bao, Z. A hybrid blob-slice model for accurate and efficient detection of fluorescence labeled nuclei in 3D. BMC Bioinformatics. 11, 580 (2010).
- Boyle, T. J., Bao, Z., Murray, J. I., Araya, C. L., Waterston, R. H. AceTree: a tool for visual analysis of Caenorhabditis elegans embryogenesis. BMC Bioinformatics. 7, 275 (2006).
- Katzman, B., Tang, D., Santella, A., Bao, Z. AceTree: a major update and case study in the long term maintenance of open-source scientific software. BMC Bioinformatics. 19 (1), 121 (2018).
- Murray, J. I., et al. Automated analysis of embryonic gene expression with cellular resolution in C. elegans. Nature Methods. 5 (8), 703-709 (2008).
- Murray, J. I., Bao, Z., Boyle, T. J., Waterston, R. H. The lineaging of fluorescently-labeled Caenorhabditis elegans embryos with StarryNite and AceTree. Nature Protocols. 1 (3), 1468-1476 (2006).
- Wu, Y., et al. Spatially isotropic four-dimensional imaging with dual-view plane illumination microscopy. Nature Biotechnology. 31 (11), 1032-1038 (2013).
- Kumar, A., et al. Using Stage- and Slit-Scanning to Improve Contrast and Optical Sectioning in Dual-View Inverted Light Sheet Microscopy (diSPIM). The Biological Bulletin. 231 (1), 26-39 (2016).
- Kumar, A., et al. Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nature Protocols. 9 (11), 2555-2573 (2014).
- Wu, Y., Christensen, R., Colon-Ramos, D., Shroff, H. Advanced optical imaging techniques for neurodevelopment. Current Opinion in Neurobiology. 23 (6), 1090-1097 (2013).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17708-17713 (2011).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136 (12), 1963-1975 (2009).
- Stelzer, E. H. Light-sheet fluorescence microscopy for quantitative biology. Nature Methods. 12 (1), 23-26 (2015).
- Winter, P. W., Shroff, H. Faster fluorescence microscopy: advances in high speed biological imaging. Current Opinion in Chemical Biology. 20, 46-53 (2014).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. Astronomical Journal. 76 (6), 745-754 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. JOSA. 62 (1), 55-59 (1972).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Hart, A. C. Behavior. WormBook. , (2006).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. Chapter 14, Unit14 20 (2010).
- Gualda, E. J., et al. SPIM-fluid: open source light-sheet based platform for high-throughput imaging. Biomedical Optics Express. 6 (11), 4447-4456 (2015).
- Ardiel, E. L., et al. Visualizing Calcium Flux in Freely Moving Nematode Embryos. Biophysical Journal. 112 (9), 1975-1983 (2017).
- Walton, T., et al. The Bicoid class homeodomain factors ceh-36/OTX and unc-30/PITX cooperate in C. elegans embryonic progenitor cells to regulate robust development. PLoS Genetics. 11 (3), e1005003 (2015).
- Altun, Z. F. WormAtlas. , (2002).
- Wood, W. B. Embryology: In the nematode C. elegans. Cold Spring Harbor Laboratory Press. Chapter 8, 215-241 (1988).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314 (1165), (1986).
- Bao, Z., Murray, J. I. Mounting Caenorhabditis elegans embryos for live imaging of embryogenesis. Cold Spring Harb Protoc. (9), (2011).
- Christensen, R. P., et al. Untwisting the Caenorhabditis elegans embryo. eLife. 4, (2015).
- Santella, A., et al. WormGUIDES: an interactive single cell developmental atlas and tool for collaborative multidimensional data exploration. BMC Bioinformatics. 16, 189 (2015).
- Dupuy, D., et al. A first version of the Caenorhabditis elegans Promoterome. Genome Research. 14 (10B), 2169-2175 (2004).
- Reece-Hoyes, J. S., et al. Insight into transcription factor gene duplication from Caenorhabditis elegans Promoterome-driven expression patterns. BMC Genomics. 8 (27), (2007).
- WormBase. , Available from: https://www.wormbase.org (2019).
- Lee, R. Y. N., et al. WormBase 2017: molting into a new stage. Nucleic Acids Research. 46 (D1), D869-D874 (2018).



