Summary
Her præsenterer vi en kombinatorisk tilgang ved hjælp af høj opløsning mikroskopi, beregningsmæssige værktøjer, og enkelt celle mærkning i levende C. elegans embryoner til at forstå enkelt celle dynamik under neurodevelopment.
Abstract
Caenorhabditis elegans (C. elegans) skiller sig ud som den eneste organisme, hvor udfordringen med at forstå den cellulære oprindelse af et helt nervesystem kan observeres, med en enkelt celle opløsning, in vivo. Her præsenterer vi en integreret protokol til undersøgelse af Neuro udvikling i C. elegans embryoner. Vores protokol kombinerer billeddannelse, lineaging og Neuro anatomisk sporing af enkeltceller i udvikling af embryoner. Vi opnår langsigtede, fire-dimensionelle (4D) billeddannelse af levende C. elegans embryoner med næsten isotropisk rumlig opløsning gennem brug af Dual-View inverteret selektiv plane belysning mikroskopi (dispim). Nuklei og neuronal strukturer i fyrretræsnematoden embryoner er afbildet og isotropisk smeltet til at give billeder med opløsning af ~ 330 nm i alle tre dimensioner. Disse minut-for-minut høj opløsning 4D datasæt analyseres derefter for at korrelere definitive celle-Lineage identiteter med genekspression og morfologiske dynamik på enkelt celle og subcellulære niveauer af detaljer. Vores protokol er struktureret til at muliggøre modulær gennemførelse af hver af de beskrevne trin og forbedre undersøgelser af embryogenesen, genekspression, eller Neuro udvikling.
Introduction
C. elegans skiller sig ud som den eneste organisme, hvor hver celle i embryonet kan observeres gennem neurodevelopment. Med hele celle-Lineage kendt og invariant1, og med udviklingen af nye værktøjer, der tillader mærkning og kontinuerlig billeddannelse af enkeltceller i embryoner, kan biologer nu begynde at undersøge forskellige trin i udviklingen af fyrretræsnematoden nervøs system fra alle vinkler-celle fødsel; migration og differentiering; neurit formation, målrettet udvækst og fasciculation; synapse dannelse; og tuning af funktionelle kredsløb. Opfange neuronal udvækst dynamik i C. elegans embryo, ved at kombinere stabilt udtrykte journalister og Fluorescens mikroskopi, er værdifuld for det videnskabelige samfund.
Udviklingsmæssige undersøgelser i C. elegans ofte udnytte invariant celle-Lineage og celle-skæbne kort af denne art til at forøge kontekstuelle forståelse på enkelt celleniveau inden for intakt organisme1. Auto-lineaging analyse-ved hjælp af starrynite2,3,4 og acetree5,6,7,8 software-fordele ved høj kontrast, høj opløsning billeder af fluorescerende kerner. For at arbejde optimalt, StarryNite/AceTree afhænger også af forudsigelig begrænset orientering af indpakkede embryoner under udvikling. Confokal mikroskopi, udført i C. elegans embryoner komprimeret mellem to dæksedler, har været standard Auto-lineaging mikroskopi metode i mere end et årti, fordi det giver både høj kontrast/høj opløsning og en forudsigelig begrænset fostrets orientering7,8. Vi har tidligere beskrevet opførelsen og brugen af en ny lys-Sheet-baseret Dual-View inverteret selektiv plan belysning mikroskop (dispim) for levende prøve billeder såsom C. elegans embryogenese9,10 , 11 af , 12 ud af , 13. lysplade mikroskopi, generelt, giver lav fototoksicitet, høj hastighed, og langsigtet billeddiagnostik af levende 3D prøver14,15. Metoden diSPIM fremstiller specifikt fire-dimensionelle (4D) billeder med næsten isotropisk rumlig opløsning på ca. 330 nm9.
Sammenlignet med confokal mikroskopi, tilbyder diSPIM højere signal-støj og hastighed, mere isotropisk rumlig opløsning, og er mere velegnet til langsigtet in vivo Imaging16. Vi har derfor arbejdet på at tilpasse diSPIM data til input til StarryNite/AceTree og undersøgt, om dette ville forbedre lineaging analyserne. En større forhindring er, at dispim prøver ikke let kan begrænses af ægshell-kompression til at vedtage forventede retningslinjer for starrynite/acetree. Tilfældig orientering af celle positioner i den diskenhed, der analyseres, forringer nøjagtigheden af Auto-lineaging analyse.
Vi har derfor ansat CytoSHOW, en seer-guidet brugergrænseflade, som giver brugerne mulighed for at vælge præcis 3D orientering af embryoner under forbehandling af diSPIM billeder, giver billeddata, der er både kvalitet-optimeret og kontekst-Aware for input til StarryNite /AceTree. Ved brugervalg af indpakkede embryoner, organiserer CytoSHOW en automatiseret databehandling pipeline. Beskårne og baggrund-subtraede embryo billeder gemmes i TIFF stack-filer for hver position, tidspunkt og visning. Cytoshow derefter iterativt kalder programmet spimfusion at co-registrere og i fællesskab deconvolve de to præ-forarbejdede synspunkter, ved hjælp af Richardson-Lucy17,18 algoritme til at give isotropisk høj opløsning volumetriske billeder. En dispim-specifikke sæt af parametre er blevet optimeret til starrynite at styre sin adfærd under billed-segmentering og Nucleus-tracking i isotropisk smeltet billeder. Fusionerede billeder og lineaging resultater redigeres derefter ved hjælp af AceTree, som giver brugerne mulighed for at identificere og rette eventuelle fejl i Auto-Lineage spor genereret af StarryNite. AceTree kan også præsentere Lineage-træ og 3D modelleret gengivelser af sporede kerner i embryonet. Vi finder, at Auto-lineaging hastighed og nøjagtighed er markant forbedret ved hjælp af isotropisk smeltet billeder, sammenlignet med RAW-billeder fra enten spim kamera. Vores protokol, mens optimeret til C. elegans program beskrevet her, kunne generelt tilpasses til Auto-lineaging af dispim data produceret for andre arter eller enheder. Hvis dette er den tilsigtede anvendelse af protokollen, Bemærk venligst, at yderligere tuning af starrynite parametre vil sandsynligvis være påkrævet for nye enheder, som beskrevet3,4.
Vellykket gennemførelse af denne protokol resulterer i billeder med 4D-isotropisk opløsning og gør det muligt for biologer at spore celle lineages, samtidig med at identificere og analysere neuroner i det udviklende C. elegans embryo. Desuden, ved at fusionere flere efter behandling algoritmer-med hardwareacceleration er den mest tidskrævende af disse-vi kan nu analysere både fine under cellulære detaljer og celle-lineages og celle-skæbner af levende embryoner i stort set realtid. Denne nye protokol giver mulighed for præcis, informeret manipulation og observation af celle adfærd under bevis undersøgelser af differentiering og morfogenesis in vivo. I dette manuskript præsenterer vi en detaljeret forklaring på de forbedrede protokoller, vi har udviklet med henblik på lineaging og celle sporing i udviklingen af C. elegans -embryoner, for at forbedre undersøgelserne af embryogenesen, genekspression eller Neuro udvikling.
Protocol
1. montering af dispim stål Imaging kammer med poly-L-lysin belagt dækglas
Bemærk: nedenstående trin er alle nødvendige for at optimere og automatisere Lineage analyse af fyrretræsnematoden embryoner af StarryNite/acetree. Flere muligheder (angivet som sådan) kan udelades for eksperimenter, der kræver sporing af C. elegans celle lineages.
- Tegn et lille rektangel (2mm x 5 mm) i midten af en ren rektangulær dækseddel (nr. 1,5, 24 mm x 50 mm) med en Sharpie (eller lignende pen).
- Flip dækglas over til den umarkerede side og sted 10 μl poly-L-lysin (kat. no. P1524) over det markerede rektangel.
Bemærk: lav en arbejds koncentration på 1 mg/mL poly-L-lysin opløst i filtreret vand (eller tilsvarende). Ved langtidsopbevaring skal der tilberedes 5 – 10 mL aliquoter og opbevares ved − 20 °C. Efter optøning kan aliquoterne opbevares ved stuetemperatur (23 °C) i 3 – 4 uger. - Tillad poly-L-lysin at pels dækglas i 5 minutter (figur 1a). Poly-L-lysin anvendes til belægning glas dække glider, hvor embryonerne vil blive monteret, så embryo æggeskallen at holde sig fast til coverslip, selv når målene er nedsænket i billedbehandlings mediet.
- Anbring poly-L-lysin belagt dækglas i den nederste halvdel af stål billedbehandlings kammeret.
- Placer den øverste halvdel af stål billedbehandlings kammeret på den nederste halvdel med dækglas og stram med de fire skruer, der er forbundet med kammeret. Kontroller visuelt fra siden for at sikre, at den øverste halvdel er jævnt siddende i den nederste halvdel (figur 1b).
- Fyld kammeret med 7 – 8 mL M9-buffer19, en isotonisk buffer, som hjælper med at forhindre tidlige stadier af fostre i at bukke under for unormalt osmotisk tryk. 1-celle, og selv 2-cellet embryoner er osmotisk følsomme og kan udvikle unormalt, hvis ikke i denne isotoniske buffer. Embryoner har også tendens til at arrestere på 3-fold stadiet, hvis M9 erstattes med vand.
2. klargøring af C. elegans embryo prøver til montering
Bemærk: ca. 18 timer før billeddannelse, fem unge (1 dag siden endelige molt) voksne og ti larve Stage 4 (L4) C. elegans er plukket til en fyrretræsnematoden vækstmedium (NGM) agar plade seedet med E. coli stamme OP50. Platin wire picks bruges til at flytte larver og unge voksne C. elegans uden at skade dyret19.
- Forbered en 1% methylcellulose (kat. nr. H7509-25G) løsning i M9 buffer.
Bemærk: methylcellulose skal omrøres i varm M9, indtil det opløses. Når denne opløsning er forberedt, kan den opbevares ved stuetemperatur. - Tilsæt 500 μL 1% methylcellulose-in-M9-opløsning i depressionen på et konkave mikroskop-dias. Denne viskøse buffer vil blive anvendt i to tilfælde: 1) ved høst af tidlige embryoner ved dissektion af voksne orme og 2) ved vask af sene embryoner plukket direkte fra en NGM-plade.
Bemærk: methylcellulose anvendes til at forhindre, at embryoner klæber til mikroskopet. - Til billeddiagnostik sene embryoner, pick lagt C. elegans embryoner (fortrinsvis fra en NGM plade med unge voksne til stede) ved hjælp af en øjenvipper pick, og flytte embryoner til 1% methylcellulose på konkave mikroskop slide. Øjenvippe plukning hjælper med at reducere kraften og minimerer derved stress eller skader på embryonerne under håndteringen. Proceduren for at gøre en øjenvipper pick er dækket af Hart20.
- Med en anden øjenvipper pick (i den modsatte hånd), forsigtigt tappe begge øjenvipper sammen for at suspendere embryonerne i methylcellulose.
- Valgfrit: Hvis man planlægger at Lineage embryonale celler med StarryNite, skal man montere 1-celle til 4-cellet embryoner. For at gøre dette, skal du først vælge unge voksne fra en NGM plade og flytte dem ind i M9-methyl cellulose opløsning på konkave mikroskop slide ved hjælp af en platin wire pick.
- Valgfri: med spidsede spidser af hypodermiske nåle (nr. 18G x 1 1/2), Skive dyret tværgående i midten af kroppen til at frigive 1-celle til 4-cellet embryoner.
3. mund pipetten: montering af aspiratorrør med mikrokapillar pipette
Bemærk: vi bruger en indsugnings tube med en hånd trukket mikrokapillar pipette indsat i gummipakning af røret. Dette giver os mulighed for at overføre embryoner fra dissektion slide til poly-L-lysin-belagt overflade i buffer-fyldte billedbehandling kammer.
- Træk forsigtigt mikrokapillar pipetten over en åben flamme for at oprette to halvdele med strakte spidser.
- Tag den ene halvdel af mikrokapillar pipetten og Indsæt den stumpe ende i gummipakningen på aspiratorrøret (figur 1c). Indstil den anden halvdel af mikrokapillar pipetten til senere brug (hvis nødvendigt).
- Med den samlede indsugnings tube monteret med mikrokapillar pipette, forsigtigt bryde spidsen af mikrokapillar pipette og skabe en åbning, der vil passe omkring 1 – 2 to embryoner (herfra på dette instrument kaldes en "mund pipette").
4. montering C. elegans embryoner på poly-L-lysin belagt dækglas
- Med indsugnings mundstykket holdes forsigtigt mellem tænderne, præ-fylde mikrokapillar pipette med 10 – 15 μl M9 buffer og derefter forsigtigt suge flere embryoner fra konkave glide ind i kapillar.
- Overfør embryonerne til stål billedbehandlings kammeret fyldt med M9 buffer, positionering kapillar spidsen, således at embryoner vil falde ind i den centrale rektangel af coverslip.
- Undgå skader på embryonerne, forsigtigt flytte dem med en øjenvipper pick eller bruge munden pipette til at placere embryonerne lodret, at orientere embryonerne, således at den lange akse af embryonet er vinkelret på den lange akse af dækglas (figur 1b justering , bundpanel).
Bemærk: positionering af embryonet i denne retning minimerer antallet af skiver til billedet, hvilket reducerer lys dosering og databehandling tid samtidig forbedre erhvervelse hastighed. - Stål billedbehandlings kammeret anbringes i prøveholderen på mikroskop stadiet (figur 1d).
5. montage, software opsætning og laser optimering til embryonale billeddannelse ved hjælp af diSPIM
- Se trin-for-trin instruktioner om, hvordan du samler hele fiber-koblet diSPIM fra kommercielt tilgængelige dele i Kumar et al.10,11 og på http://www.dispim.org. En video-protokol over, hvordan du samler diSPIM er også tilgængelig på ASI hjemmeside (http://www.asiimaging.com).
Bemærk: instrument opsætningen for denne protokol er identisk med Kumar et al.10,11 dispim, som gør brug af 40x 0,8 na vand fordybelse linser til billeddannelse. Den eneste forskel mellem opsætningen i denne protokol og Kumar et al.10,11 er tilføjelsen af et dichroisk spejl (opdeling ved 560 nm) og røde og grønne båndpas filtre i en billedbehandlings spaltning enhed (model A12801-01) installeret på begge diSPIM Imaging arme. Tilføjelsen af billed opdeling optik tillader samtidig opsamling af billeder fra to forskellige fluorophores-ophidset af 561 nm og 488 nm lasere-ved at adskille emission bands på to halvdele af samme kamera chip. - Efter instrumentets montering skal du kontrollere den optiske justering af diSPIM før billedbehandling.
Bemærk: for at justere diSPIM korrekt, se https://youtu.be/qnOrg30NNuE, og for hardware oplysninger, http://dispim.org/hardware/objectives og http://www.asiimaging.com. - Brug open source-platformen Micro-Manager (https://micro-manager.org/)21, som er blevet optimeret til drift af Lysarks mikroskoper til mobil afbildning med høj dataoverførselshastighed22. Vi anbefaler brug af ASI diSPIM plugin til multi-position erhvervelse, som tillader simultan scanning af op til 30 embryoner som beskrevet23.
- Med Micro-Manager åben, indstille laser intensiteter til ~ 179 μW (0,5) for 488 nm og ~ 79 μW (0,25) for 561 nm (figur 2a, rød rektangel).
Bemærk: disse er anbefalede indstillinger for langsigtet billeddannelse af C. elegans embryoner med 1-minutters intervaller. Under langvarig Dual-Color Imaging, 561 nm laser bruges til billed kerner (Mcherry:: Histon) indtil embryoner er på bønne stadiet, på hvilket tidspunkt 488 nm laser er derefter tænde for også at billedet gfp-mærkede neuroner. Disse billeddiagnostiske tilstande er optimeret til minimering af fototoksicitet og sikring af overlevelsen og klækningen af embryonerne, samtidig med at de muliggør langvarig (12 – 14 timer) kontinuerlig erhvervelse af neuroudviklingsmæssige og lineaging data. - I Micro-Manager, skal du vælge Menu Plugins ≫ enhedskontrol > ASI diSPIM at åbne ASI dispim Window (figur 2b). Vælg fanen anskaffelse . I denne fane ' data besparelse indstillinger sektion (grøn rektangel), lydstyrkeindstillinger sektion (blå firkant), og Slice indstillinger sektion (orange firkant), sikre, at hver parameter er indstillet som vist i figur 2b.
Bemærk: vores billedanalyse software CytoSHOW er tilpasset til at arbejde med andre valgfri outputdata formater såsom bulk-sammenkædet OME-TIFF-filserier og TIFF-stack filserier skabt efter erhvervelse gennem brug af en eksport funktion indbygget i Micro-Manager. Typisk, den ugifte-gang-pege-stak indsamles ome-TIFF fil dataformat bruges fordi sig tillader sand-gang gennemsyn og behandling i billed volumen så snart som den rå data er tilegnet.
6. optimerede autofokus parametre for langsigtet billeddannelse af C. elegans embryoner
- Indstil Micro-Manager autofokus parametre til de indstillinger, der er optimeret til langsigtet kvalitet diSPIM-Imaging af C. elegans embryoner. Klik på fanen autofokus (figur 2c) i vinduet ASI dispim. I afsnittet generelle indstillinger for autofokus (sort firkant) skal du angive parametre præcist som vist. Bemærk, at autofocus-kanalen (den røde firkant) skal angive din atomare kanal fluorescens kanal i eksperimenter med lineaging.
Bemærk: Hvis Max offset er større end 5 μm, billeder tendens til at glide ud af fokus. - Klik, Plugins ≫ erhvervelse værktøjer > mønster overlay.
- Klik på Vis gitteri vinduet mønster overlejring.
- Klik på fanen navigation i vinduet ASI dispim .
- Klik på afkrydsningsfelter for bjælke og ark med kurve A eller B, og klik derefter på Live. Billed anskaffelse begynder. Der åbnes et vindue med direkte visning. Vælg et område til analyse af autofokus for embryonet ved at tegne en kasse omkring embryonet på den valgte kanal fra 6,1.
Bemærk: vi henter typisk 420 tidspunkter for 10 embryoner pr. billedbehandlings session. De rå data pr. billede session er typisk 1,7 TB, mens deconvolved og StarryNite-forarbejdede data er 1.4 TB (Se trin 9 og 10). Vi anbefaler, at du bruger HDD med stor kapacitet (18TB på vores nuværende system) til billed anskaffelse og cloudplatforme til billedlagring. - Klik på Start erhvervelse under fanen "anskaffelse " for at påbegynde langsigtet flerdimensionel billedoptagelse (figur 2b).
7. åbning af RAW Micro-Manager billeder i CytoSHOW
- Download softwarepakken fra http://dispimlineage.wormguides.org.
Bemærk: softwarepakken vil blive hentet som en. zip-fil og skal pakkes ud til "C:\" mappe før brug. Yderligere oplysninger om installation er angivet på http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm. - Dobbeltklik på filen C:\cytoshowextrasforc\cytoshow_app.JNLP for at begynde at køre cytoshow.
- Vælg Menufil > ny > dispim Monitor (Micro-Manager). Find mappen med roddatasæt, hvor der blev gemt tidspunkt mapper til anskaffelse. Vælg en tidspunkt mappe, og klik på Åbn. Multidimensionelle navigations vinduer (kaldet Dispim Monitor Windows) åbnes automatisk for både spima og Spimb (figur 3a).
Bemærk: disse Vinduer vil overvåge roddata mappen for nyligt gemte RAW-tidspunkter stakke (i tilfælde af, at en prøve stadig er ved at blive optaget). Efter hvert nyt tidspunkt er anskaffet, vil hver af vinduerne overvåge de distinkte SPIM arme og prøve positioner opdateres for at vise hele multikanal 4D datasæt for hvert embryo.
8. generering af Max projektions billeder med CytoSHOW
Selv før deconvolution, kan de rå data hurtigt behandles til at vurdere prøvens globale funktioner.
- Klik på Z-MIP- knappen i venstre side af billedvinduet (figur 3a, rødt rektangel) for at gøre maksimal intensitet fremskrivninger gennem fuld dybde og fuld tid kursus af en given position eller spim arm. Der vises et Z-projektions-hyperstack- vindue.
- I vinduet Z-projektions-hyperstack skal du indstille projektions typen til maksimal intensitet. Angiv, hvilke kanaler, udsnit og tidspunkt rammer der skal behandles på basis af brugerpræferencer.
- Klik på OK , når du er færdig.
- Vælg mappens placering for at gemme maksimal intensitets output fra fildialog vinduet, og klik derefter på OK. Tillad nogen tid (15 – 20 minutter, afhængigt af størrelsen af datasæt og processorkraft på computeren) for CytoSHOW til at generere projektions billeder.
9. analyse af celle slægter i isotropiske volumetriske data i høj opløsning
- Valgfrit: med de rådata, der åbnes via Dispim Monitor i cytoshow, skal du vælge polygon markeringsværktøjet (figur 3a, sorte pile) og klikke lige uden for fostrets forreste, bageste, dorsale og ventrale kanter (i den nøjagtige rækkefølge) for at skabe et "Bowtie" mønster over embryonet. Gør for begge synspunkter (SPIM-A og SPIM-B, figur 3a).
Bemærk: dette valg specificerer den elliptiske region af interesse (ROI), hvor embryonet er centreret og registrerer den forreste-posterior akse af embryonet. Den Bowtie mønster signaler cytoshow, at brugeren har planer om yderligere at specificere en præcis rotation af den endelige isotropisk smeltet mængder i en retning, der er optimal for lineaging analyser af starrynite/acetree. I tilfælde, hvor StarryNite-lineaging ikke er en del af forsøgsplanen, kan andre markeringsværktøjer og-former vælges til at angive ROI for billedbehandling. - Hvis flere embryoner samtidig er indlagret ved hjælp af multi-position erhvervelse mulighed, åbne og udføre trin 9,1 for alle embryoner. Dette vil tillade parallel udførelse af fremtidige skridt for alle embryoner i en session. Luk SIMA-og SPIMB-vinduerne for de embryoner, du ikke ønsker at behandle.
- Klik på knappen Dispim i panelet til venstre i vinduet dispim Monitor (figur 3a, fremhævet med gult). Dette afslører et underpanel af kontrolelementer, der er specifikke for diSPIM-behandling.
- Juster grønne og røde kanaler for hver SPIM-arm. Da emissions kanal splittere bruges til at indfange tydelige røde og grønne billeder samtidigt på det samme kamera, er det vigtigt visuelt at justere den nøjagtige pixel registrering af disse to fysisk tilstødende billedfelter, når de er overbelastet. Genbrug af de samme justerings justeringer er typisk muligt i mange fortløbende billedbehandlings sessioner, men bør kontrolleres (som i trin 9.4.1 – 9.4.5).
- Start med SPIMA-panelet, Vælg den røde kanal ved at flytte cm scroll bar til venstre (figur 3a, orange pil, venstre panel).
- Brug x-, y-og z-justererne (figur 3a, orange firkant) til at skifte den røde kanal, så den passer til greenen.
- Klik på knappen Dispim (figur 3a, fremhævet med gult) for at lukke under panelet og udløse overførslen af de samme Skift til alle andre positions vinduer.
- Bekræft, at den korrekte justering er overført til andre rammer og tidspunkter ved at flytte rullepanelet "z" (figur 3a, blå pil, venstre panel) og/eller "t"-rullepanel (figur 3a, grøn pil, venstre panel). Hvis multi-position erhvervelse blev udført, og flere embryoner blev indpakket (trin 5,3), bør tilpasningen også har formeret til disse embryoner. Bekræft ved også at undersøge numrene for x-, y-og z-justeringsanordninger (figur 3a, orange firkant, som skal være den samme for spima-panelet af alle embryoner).
- Gentag trin 9.4.1 – 9.4.4 for SPIMB diSPIM Monitor vinduet (figur 3a, højre panel).
- Klik på "Dispim" knappen og derefter på "Fuse" knappen (figur 3a, blå rektangel) for at åbne en dialogboks kaldet "Deconvolve/Fuse Dispim RAW data mængder" (figur 3b). Angiv parametre som vist i figur 3b. Disse parametre behandles kort i følgende under trin:
- Indstil taste registrering på kanal 1 (488 nm laser) eller 2 (561 nm laser). Vælg kanalen med tættere eller mere allestedsnærværende signal. For lineaging eksperimenter altid vælge den kanal, der bruges til at afbilde allestedsnærværende nukleare Histon fluorescens.
- Indstil input volumener relativ retning til + 1 eller-1. Det korrekte orienterings indeks afhænger af en bestemt Dispim's kameraplaceringer (figur 4).
Bemærk: Hvis det er usikkert, skal du teste hver indstilling ved at duplikere et enkelt tidspunkt fra både SPIM A og B Dispim Monitor vindue, følge trin 9.1-9.12 og vilkårligt vælge en input mængder relativ orientering at teste. Forkerte retninger vil give slørede billeder med artefakter, mens korrekte retninger vil give klare billeder. Den relative orienterings værdi for input diskenheder , der giver det klare billede, kan derefter genbruges for alle fremtidige data fra det givne dispim-instrument. - Vælg, om den sammensmeltede diskenhed skal være den samme som A-eller B-indgangs enheden (baseret på brugerpræferencer).
- Vælg "ny registrering for hver diskenhed". Denne indstilling styrer, hvordan SpimFusion beregner registrerings matricer for hvert volumen par på hvert tidspunkt. Indstillingen "Fresh" gør det muligt for algoritmen at optimere registreringen på hvert tidspunkt.
- Indstil antallet af deconvolution iterationer til 10. Dette tal har en tendens til pålideligt at give den ønskede høje opløsning på en tidseffektiv måde.
- Valgfrit: Hvis der ønskes automatisk lineaging (anbefales), skal du markere automatisk start af StarryNite-lineaging af fusionerede enheder. Denne mulighed vil lancere starrynite automatisk at segmentere og spore celler i de afbildet mængder produceret af spimfusion.
- Valgfri: for højeste nøjagtighed i automatiseret lineaging, er det bedst at flytte isotropisk smeltet embryo volumen i den kanoniske "ADL" (ennterior [x-vest], Dorsal [y-nord], venstre [z-Near]) orientering. Vælg indstillingen Definer lydstyrke output i eksempelvisning for at angive dette valg. Cytoshow vil reagere ved at behandle et indledende par af isotropisk smeltet mængder, så brugeren til nøje at observere og specificere rotationer er nødvendige for at opnå ADL registrering.
- Klik på Ja , når alle parametre er valgt.
- Angiv den output mappe , hvor de behandlede filer skal gemmes. Klik på OK.
- Valgfrit: Hvis indstillingen Definer lydstyrke output i eksempelvisning er valgt, skal du indstille t -rullepanelet (figur 3a, den grønne pil, venstre panel) i spim-A-vinduet til det tidlige tidspunkt, hvor ABa-og ABP-cellerne har nået metaphase. Indstil t -rullepanelet i spim-B-vinduet til den senere komma fase i udviklingen. Dette vil hjælpe med at specificere ADL orientering.
- Valgfrit: Klik på OK , når du er klar. Hvis eksempel indstillingen i 9.5.7 ovenfor er valgt, vil kun to forhåndsvisnings volumener være isotropisk smeltet for de tidspunkter, der er angivet af t-skyderne i spim-A-og spim-B-billedvinduerne. Disse to forhåndsvisnings-tidspunkter kan bruges til at specificere nøjagtig justering af output embryoets volumen til ADL-orienteringen som forklaret nedenfor.
- Find den nyligt viste 3DProjY_Decon-Fuse_.... Vinduet. Flyt t -rullepanelet til tidspunkt 2 i dette eksempelvindue. Flyt skyderen Z, indtil visningen direkte ned på fostrets lange akse vises.
- Flyt t rullepanelet tilbage til tidspunkt 1 i 3DProjY_Decon-Fuse_.... Vinduet. Vælg linje markeringsværktøjet, og tegn en linje markering fra EMS-cellen (ventral-den mest runde kerne) gennem planet af AB-Cell knoglemarv-pladerne.
- Klik på knappen orange diSPIM preview på 3DProjY_Decon-Fuse_.... Vinduet. De fine justeringer af retningen af den viste, indpakkede diskenhed gemmes til brug ved behandling af det komplette datasæt.
- Valgfrit: dialogen Deconvolve/Fuse diSPIM RAW data mængder vises igen, ligesom i trin 9,5 ovenfor. Klik på Ja uden at vælge indstillingen Definer output retning for lydstyrke i eksempelvisning . Angiv output mappe for fuld databehandling køre.
- Indstil t -rullepanelerne (figur 3a, den grønne pil, venstre panel) i vinduet dispim Monitor til Start gang får (spima) og sluttids punkt (spimb) for hele spektret af billeder, der skal behandles. Klik derefter på OK.
- Som SpimFusion skrider frem, CytoSHOW åbner og opdaterer et flerdimensionelt vindue, der viser den skiver-4D isotropiske sammensmeltet volumen for hvert embryo, samt to vinduer med roterende-4D Max-intensitet prognoser af isotrope volumen. I dette tidsrum må du ikke afbryde eller lukke et cytoshow-vindue, før isotropisk fusion og Lineage sporing er fuldført.
- Valgfrit: Bemærk, at når StarryNite-velkomstbilledet er dukket op og senere er forsvundet, er den komplette databehandlings pipeline afsluttet. Dette vindue må ikke lukkes under behandlingen, eller StarryNite afbrydes.
10. åbning af StarryNite Lineage-sporings serie i AceTree (valgfri)
- Åbn den tilpassede version af "AceTree_16BitCompat. jar", der er angivet.
- Vælg Menu filen > Åbn Konfigurer fil. Find din output mappe tidligere indikeret til cytoshow. Åbn under mappen Decon_Fuse_... _Pos [n] for embryo [n]. Vælg aaa_edited. XML og Åbn.
- Brug AceTree menu redigere > redigere værktøjer til at åbne Rediger spor og justere eller slette celler vinduer.
- Klik på den halv-skyggelagte cirkel figur 5b, rød firkant for at justere de røde og grønne intensiteter.
- Fortsæt med Lineage visualisering og redigering som tidligere beskrevet5,6,8 (manuskripter er også inkluderet i vores download Bundle).
Representative Results
Vi har først valideret levedygtigheden af embryoner, der er indpakket ved hjælp af protokollens parametre for diSPIM erhvervelse (afsnit 1-6). Ti embryoner blev samtidig indlagret ved 20 °C, et volumen/embryo/minut, fra 2-cellens fase til 2-dobling (7,5 timer, 451 volumen/embryo). For at overvåge celle divisioner i hele embryogenese brugte vi stamme BV514, som allestedsnærværende udtrykker Mcherry::histone reporter konstruterer fra den integrerede transgen matrix ujIs11324. Figur 6 viser en tidslinje for denne første halvdel af fosterudviklingen for en af de indpakkede embryoner. Hvert billede repræsenterer en enkelt visning maksimal intensitet projektion (produceret af trin 7-8) af det afbildet embryon. Vi konstaterede, at de optimerede protokoller ikke fremprovokeret nogen detekterbar fototoksicitet for embryonerne, vurderet ved timing af celle inddelinger (ikke vist), tidspunkt for klækning, og timing relateret til udviklingsmæssige milepæle (figur 6 og referencer1 , 25 stk. , 26).
Vi derefter anvendt protokollen til at analysere udvækst dynamik af enkelt neuroner i udvikling af embryoner. Vi afbildet DCR7692 (olaex4655), en transgene fyrretræsnematoden stamme, der udtrykker gfp off neuropeptid FLP-19 Promoter i en delmængde af uidentificerede celler (DACR2819, PFLP-19 (3.6 KB):: Syn21:: gfp:: caax::p 10 3 ' UTR) . Efter trinene i den protokol, der er skitseret her, besluttede vi, at de uidentificerede celler svarer til motoriske neuroner RMDDL og RMDDR, til den ekskretionsoriske kanal celle og til to muskelceller (figur 7). Derefter undersøgte og kvantificerede vi udvækstdynamikken i RMDDL og RMDDR neuroner. Vi bemærkede, at rmddl og rmddr neuroner er skråt formet så tidligt som 360 minutter efter befrugtning, med den længere cellulære akse, der repræsenterer den efterfølgende akse for neurite udvækst (figur 7 og Movie S1). Ved hjælp af den "simple neurite sporing" plugin i Fiji og anvende det til 3D rekonstruktioner af isotropisk smeltet mængder, vi derefter kvantificeret den stereotypiske udvækst af rmddl og rmddr neurites for seks embryoner. Vi fastslog, at udvækst-dynamikken var stereotyp for rmddl og rmddr på tværs af embryoner (heri kaldet rmdd'er). Fra 385-410 minutter efter befrugtning udvidede RMDDs neurites 6,0 ± 0,5 μm (gennemsnit ± SEM; n = 12 neurites) forreste af celle organerne (figur 7b, C, I). Fra 415-445 minutter efter befrugtning, begge neurites udvide dorsalt i og omkring den formodede nerve ring (stjerne i figur 7d). I gennemsnit udvidede hver RMDD neurite 11,0 ± 0,6 μm (middel ± SEM; n = 12 neurites) fra celle kroppen før synkront at møde sin kontralaterale modstykke i spidsen af ringen (figur 7i). Det er vigtigt, at vores repræsentative resultater viser, at vi er i stand til at undersøge, sammenligne og kvantificere neuronal udviklingsmæssige funktioner for enkelt identificerbare celler ved hjælp af vores integrerede protokol (figur 7 og figur 8).
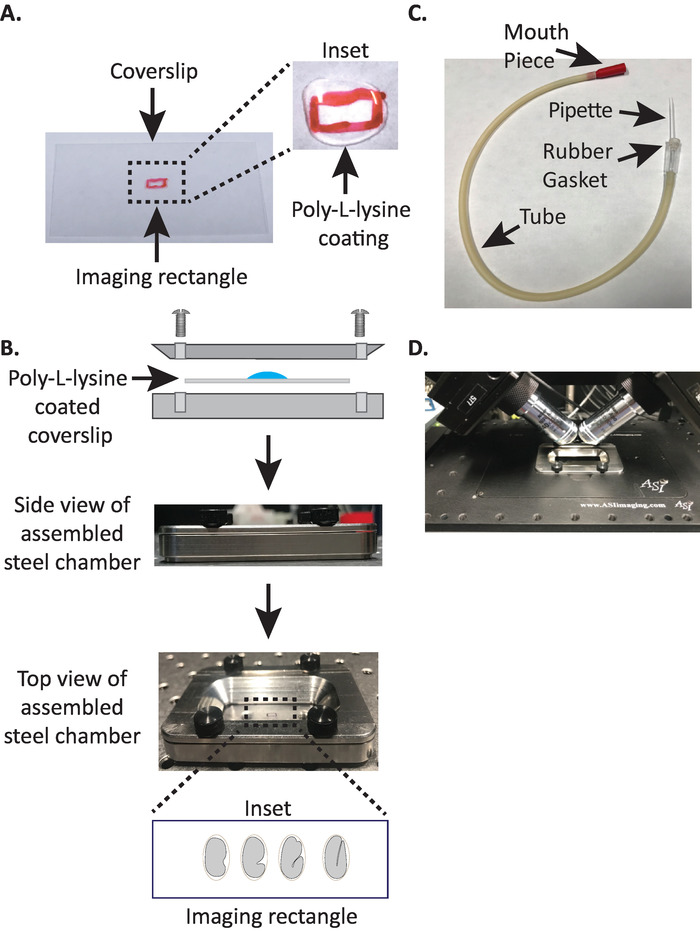
Figur 1: Opsætning af diSPIM-prøvemontering. A) fremstilling af dækglas med poly-L-lysin. I indsættet, 10 μl poly-L lysin blev brugt til at pels dækglas i 5 minutter. Poly-L-lysin tillader embryo ægskallen at holde fast til dækglas i rektanglet. B) skematisk af stål billedbehandlings kammeret og det samlede kammer. I den indsatte, repræsentative embryoner er orienteret med den forreste-posteriort akse vinkelret på den lange akse på coverslip. C) samlet aspiratorrør med mikrokapillar pipette. D) stål billedrum monteret i prøveholderen under dispim 40x-mål. Klik her for at se en større version af dette tal.

Figur 2: Opsætning af langsigtet diSPIM Imaging i Micro-Manager. (A) anbefalede dispim laser effektparametre (rødt rektangel) optimeret til langvarig billeddannelse og samtidig reducere fototoksicitet (vurderet ved højere udklæknings hastighed af C. elegans embryoner). Sæt 561 nm laser til 79 μW (0,25) og 488 nm laser til 179 μW (0,5). Bemærk, at nøjagtig kalibrering af software indstillinger til laser effekt varierer mellem diSPIM-installationer. Det anbefales, at brugerne måler og kalibrerer laser effekten for at opnå 79 μW (561 nm) og 179 μW (488 nm) laser effekt. (B) dispim parametre for databesparelse (grøn rektangel), lydstyrkeindstillinger (blå firkant), og udsnit indstillinger (orange firkant). (C) dispim autofokus parametre for langvarig billeddannelse af C. elegans embryogenese (Se trin 6.1-6.6). Klik her for at se en større version af dette tal.

Figur 3: billed visualisering og databehandlings opsætning ved hjælp af CytoSHOW. (A) RAW-dispim-billeder åbnet af cytoshow. CytoSHOW er i stand til at åbne billeder fanget af begge kamera stier (SPIM A og B). Disse rå billeder åbnes i flerdimensionelle Vinduer kaldet Dispim Monitor. I Dispim Monitorgenereres et "Bowtie mønster" for at udvælge fostrets forreste, bageste, dorsale og ventrale kanter (se trin 9,1). Bow-Tie valg indikerer embryo orientering for deconvolution og StarryNite-Assisted sporing af lineaging. (B) optimerede parametre, der anvendes til at generere isotropiske billeder. Angiv de parametre, der er angivet i trin 9.5.1-9.5.8, i vinduet Deconvolved, mens der anskaffer. Klik her for at se en større version af dette tal.

Figur 4: konfiguration af diSPIM-kamera. (A) fotografi af dispim kameraplaceringer og retningslinjer. (B) skildring af + 90-graders rotationer af spim A til at MATCHE spim B billeder indsamlet. (C) input volumener i forhold til orienterings indeks + 1 baseret på vores dispims kamera konfiguration (Se trin 9.5.2). Vi roterer SPIM et billede (r) + 90 grader omkring Y-aksen før registrering for at matche SPIM B billede (r). Skala bjælker = 10 μm. billeder er repræsentative enkelt-visning, maksimal intensitet fremskrivninger og deconvolution billeder af 1,5-fold embryo med mærkede kerner (561-nm, rød) og neuroner (488-nm, grøn). Klik her for at se en større version af dette tal.

Figur 5: curation og redigering C. elegans embryonale afstamning i acetree. (A) vi bruger acetree til at redigere starrynite Lineage spor (Se referencer5,6,8; manuskripter er også inkluderet i vores download Bundle). AceTree vil vise C. elegans systematiske navne for hver kerne (grøn rektangel) efter afslutning af trin 10.1-10.2. Dette vindue (a) indeholder oplysninger (sort rektangel) om hver celle i Lineage-sporingen (ABa, fremhævet med blåt), der hjælper brugerne, når de sporer og redigerer Lineage-sporene. Det anbefales, at brugerne kontrollerer og sammenligner lineare celler og deres positioner til C. elegans embryonale celle Lineage, der tidligere er indberettet af sulston et al.1 desuden, hvis brugerne er interesseret i at finde bestemte celler i deconvolved dataserie (se nedenfor, B) Indtast C. elegans systematiske navn i søgefeltet (orange rektangel). (B) brugerens deconvolveret dataserie åbnes også automatisk, når trin 10.1-10.2 er afsluttet. Vist her en isotropisk smeltet billede af en fire-cellet fase embryo med kerner mærket med rødt. Under sporing af en kerner, bør brugerne ændre intensiteten af billedet (rød firkant) og navigere gennem tiden og z ved hjælp af piletasterne på deres tastatur (tid-venstre/højre, z-op/ned). (C) 3D-tegneserie af tidspunktet i (b) med visse funktionaliteter (lilla rektangel), der muliggør roterbare 3D-visualisering. Se referencerne5,6,8for at få en oversigt over acetree og dens 3D-funktionalitet. Klik her for at se en større version af dette tal.

Figur 6: tidsindstillet udviklingsdynamik af C. elegans embryoner på dispim. Toppanel, diSPIM billeder, der viser den første halvdel af fosterudvikling for en af de afbildnede embryoner (Strain BV514 ujIs11324). Embryonerne blev kontinuerligt indpakket, hvert minut i 7,5 timer (ved 20 °C). De første to billeder af toppanelet repræsenterer 4-og 8-cellede embryoner med kerner (rød) og positioner af polære kroppe (tætte røde kugler, markeret med blå asterisker). Hvert billede repræsenterer en enkelt visning maksimal intensitet projektion af det afbildet embryon. Skala stænger = 10 μm. Tidslinjen (vandret linje) repræsenterer minutter efter befrugtning (m.p.f.) af udviklingen af C. elegans embryoner. Vi validerede, at vores protokol's parametre for diSPIM erhvervelse ikke inducerede nogen detekterbar fototoksicitet for de indpakkede embryoner som vurderet af levedygtighed, timing af celle divisioner, tidspunkt for klækning og timing af udviklingsmæssige milepæle (Se referencer 1 af , 25 stk. , 26). Vi bemærker, at timingen af udviklingsmæssige milepæle var reproducerbare på tværs af embryoner med vores Imaging parametre (SEM ± 8,174 minutter for 6,4 time lange billedbehandling sessioner; n = 10). Klik her for at se en større version af dette tal.

Figur 7. Cellulære identifikation og enkelt celle karakterisering af neurite udvækst dynamik i udviklingen af C. elegans embryoner. Dual-Color Imaging af en stamme foretaget ved passage BV514 ujIs11324 (for lineaging) og DCR7692 (olaex4655), en transgene fyrretræsnematoden stamme, der udtrykker gfp off Neuro peptid FLP-19 Promoter i en delmængde af uidentificerede celler. (a-H) Efter trinene i den protokol, der er skitseret her, vi fastslået, at de uidentificerede celler svarer til motor neuroner RMDDL og RMDDR (gule pile), at den ekskretionsoriske kanal celle (blå pile), og to muskelceller (hvide pile). (I) kvantificering af udvækstdynamikken i rmddl-og rmddr-neuronerne ved hjælp af Fijis plugin "simpel neurite Tracing" og anvendelse af det på 3D-rekonstruktioner af isotropisk fusionerede volumener. Bemærk, hvordan både rmddl og rmddr viser stereotypisk udvækst-dynamik, der hver især strækker sig synkront for en samlet længde på 11,0 ± 0,6 μm (gennemsnit ± SEM; n = 12 neurites) og møde ved dorsale spids af nerve ring (Se også film S1). Klik her for at se en større version af dette tal.

Figur 8: undersøgelse af isotropic diSPIM billeder af neuronal morfologier i C. elegans embryoner. Isotropisk visualisering af AVHL-og AVHR-neuroner (gule pile). Ved hjælp af diSPIM, neuronal morfologier kan optages giver fire-dimensionelle (4D) billeder med isotrope rumlig opløsning på ca 330 nm. Den diSPIM giver brugerne mulighed for næsten at rotere billed mængder med identisk opløsning i alle retninger. Billeder i A-D er maksimal intensitet fremskrivninger af samme isotropisk smeltet dispim billede volumen fra forskellige rotationer omkring embryoets lange akse. Skala stænger = 5 μm. venligst klik her for at se en større version af dette tal.
Supplerende film S1: C. elegans embryo udvikler sig fra 280 til 434 minutter efter befrugtning. Isotropisk film af stamme DCR7692 (olaex4655) udtrykker ujIs113 allestedsnærværende med DACR2819 tyndt mærknings rmdd Neurites (figur 7a-D, gule pile). DACR2819 også etiketter to muskelceller (figur 7a-d, hvide pile) og excretoriske kanal celle (figur 7a-d, blå pil) under fosterudvikling (figur 7a-d). Skala stænger = 10 μm. venligst klik her for at downloade denne fil.
Discussion
C. elegans skiller sig ud som den eneste organisme med de endelige positioner og konnektivitet af hver voksen neuron kendt27. Men den udviklingsmæssige dynamik, der fører til organisering af de arbejds kredsløb og netværk, der udgør C. elegans connectome forbliver ukendt. Baseret på muligheder fra fremskridt i lys mikroskopi, kan vi nu fange og analysere celle positioner, morfogenesis, og neurogenese hele C. elegans embryonale udvikling.
Den procedure, som vi har beskrevet, og som vi rutinemæssigt bruger i laboratoriet, giver 4D-isotropiske billeder af mærkede neuroner og kerner til celle-lineaging i C. elegans -embryoner. Endnu vigtigere er det, at vi har optimeret langsigtede billeddannelses betingelser med diSPIM og koblede semi-automatiserede lineaging-funktioner med billeder i høj opløsning for at forbedre hastigheden og præcisionen ved at analysere C. elegans embryogenese. Denne integrerede protokol vil gøre det muligt for brugerne at visualisere og identificere celler og kvantificere tredimensionale funktioner såsom neurit migration og Axon fasciculation gennem debut af tidlig trækninger. Denne procedure kan let tilpasses til ethvert anlæg med et ASI diSPIM-system, og vi anbefaler dette system specifikt til denne protokol. Andre SPIM-formuleringer, der udbydes kommercielt, kan afvige fra ASI-konfigurationen i prøvekammeret og de optiske egenskaber. Data, der eksporteres fra andre platforme, kan dog også gennemføres via vores datapipeline. Derfor, vurdering af deres værdi i lineaging, en krævende test af billedkvalitet og instrument stabilitet, er muligt. Selvom vi aktivt bruger diSPIM til regelmæssigt at afbilde andre prøver (som f. eks. Drosophila og zebrafiske embryoner), er den beskrevne og omfattende analyse af embryonerne i øjeblikket stadig begrænset til fyrretræsnematoden-arterne. For større eller tykke prøver vælger vi at bruge trin-scanning tilgange, som scanner prøverne gennem en stationær lys ark. Kumar et al. har tidligere demonstreret denne forbedrede diSPIM skæring til at give billeder af høj kvalitet fra tykke prøver uden yderligere ændringer af diSPIM10.
De kritiske trin i protokollen omfatter montering C. elegans embryoner på poly-L-lysin belagt coverslip, dataopsamling, og databehandling. Høst og montering C. elegans embryoner på glasset dækglas kan være udfordrende for uerfarne brugere, men her giver vi en detaljeret protokol af centrale skridt til at lette læring. Hvis langsigtet billeddannelse ønskes, opnår vi de bedste resultater ved høst af fire-cellet eller tidligere embryoner fra 8-10 unge voksne28. Bemærk, at gamle voksne er mindre ønskeligt at høste tidlige stadier embryoner, fordi de har tendens til at indeholde ældre embryoner i livmoderen og ubefrugtede æg. Med hensyn til montering af embryoner kan problemer som blokering i den samlede indsugnings (mund pipette) eller en for stor åbning i mikrokapillar pipetten forhindre korrekt montering og orientering af embryoner. For at forberede den optimale billeddannelse udfører vi test før anskaffelsen på tidlige og sene præ-twitching-embryoner for at kontrollere ydeevnen af lysark, kameraer, mål og autofokus. Vi opnår de bedste resultater, når alle disse operationer er testet og giver billeder af høj kvalitet under vores pre-Acquisition test. Dette er især relevant for generering af billeder med isotropisk rumlig opløsning, hvor RAW-billeder, der er anskaffet fra begge synspunkter (mål), skal være af høj kvalitet. Efter købet behandles de mængder, der er anskaffet for hver visning, for at give isotropiske billeder. Det er vigtigt at bruge et passende GPU-kort (grafikprocessor) som beskrevet i denne protokol (se nedenfor). Dette forbedrer den behandlingshastighed, hvormed de isotropisk sammensmeltede billeder genereres, hvilket forkorter tiden til dataanalyser. Det er også bydende nødvendigt, at brugerne kører den nyeste version af CytoSHOW og bruger de parametre, der leveres med vores download bundle til StarryNite Auto-lineaging. Hvis brugerne er interesseret i at bruge Auto-lineaging til andre prøver (f. eks. zebrafish, Drosophila osv.), vil der være behov for yderligere optimering af de parametre, der anvendes i starrynite (Se referencer3,4).
Selv om vores integrerede protokol giver billeder og lineaging resultater i præ-twitching embryo, bør brugerne være opmærksomme på, at automatiseret lineaging i post-twitching embryo er i øjeblikket ikke muligt: nukleare positioner ændringer på rækkefølgen af sekunder i efter twitching embryo, for hurtigt at tillade Lineage tracking. Men, den dispim har faktisk vist en lovende evne til at indfange neuroudviklingsmæssige hændelser og spore nogle celle positioner i post-twitching stadier af embryo Genesis23,29. Hvis brugerne er interesseret i at undersøge post-twitching embryo, den diSPIM giver den hastighed for at opnå volumetriske Snapshots og spore fine neuroudviklingsmæssige hændelser, såsom neurite outgrowth, i hurtigt bevægende embryoner.
Denne protokol vil være grundlæggende for celle-for-celle færdiggørelse af WormGUIDES Atlas30, da det vil give en integreret tilgang med høj opløsning isotropiske billeder til at identificere og indfange 3D morfologier af mærkede neuroner under de første 430 minutter af embryogenese. Som det ser ud, prototypen WormGUIDES Atlas giver nukleare positioner af celler i det udviklende embryo og har til formål at indfange den udviklingsmæssige dynamik i en delmængde af embryonale neuroner. Denne protokol vil være nøglen til integration af yderligere udviklings neuroner i WormGUIDES Atlas30.
Vores integrerede protokol vil også forenkle udforskning af nye genekspressions profiler i C. elegans embryo. I transgene C. elegans, mange celle-specifikke initiativtagere rumligt og temporalt kontrol transgen udtryk. Mens udtrykket mønstre af de fleste gener er blevet udførligt karakteriseret i det voksne dyr31,32,33,34, næsten alle er endnu ikke karakteriseret i udviklingslandene (især forsinket) embryo. Den C. elegans promoterome har været en nyttig ressource til ormen samfund til at køre celle-specifikke transgen udtryk, samt afgøre, om gen funktion er celle-autonome eller ikke-autonome. At opfange isotropiske, højopløsnings-og dynamiske udtryks mønstre af gener og præcist at identificere celler via lineaging vil være værdifuldt for mange i forskersamfundet.
Embryogenese består af to sammenflettede store processer, cellulær differentiering og vævs morfogenese. En hel del er kendt om de mekanismer og molekyler, der definerer særskilte celletyper under udviklingen af C. elegans. Men, lidt er kendt om de mekanismer, der er vigtige for celle migration, celle vedhæftning, og celle form i C. elegans embryo. Med C. elegans invariant celle Lineage kendt, vores protokol lader os let skelne katalogiseret 3D-mikroanatomi af embryonet under morfogenese på nye niveauer af detaljer: fx Axon fasciculation, synaptogenesis, og neuronal aktivitet. Ardiel et al. har tidligere demonstreret styrken af diSPIM til at indfange calcium transienter på niveau med en enkelt neuroner i C. elegans embryoner23. Mange andre aspekter af udviklings fysiologi er moden til undersøgelse af disse metoder.
Endelig, denne protokol er stort set automatiseret og systematisk reducerer den tid, det tager at generere deconvolution billeder og udføre celle-lineaging via StarryNite og Acetree. De software strategier, der anvendes i denne protokol, kan anvendes på mange spørgsmål om biologi langt fra de meget specifikke områder, hvor vi har demonstreret dem her.
Oplysninger om software kompatibilitet og download-adgang
Oplysninger om Micro-Manager og plugins til diSPIM Imaging er tilgængelige på http://dispim.org/software/micro-manager og https://micro-manager.org/wiki/ASIdiSPIM_Plugin.
Databehandlings pipelinen kræver i øjeblikket et Windows-operativsystem. Vi har bundtet en enkelt arkivfil for at forenkle installationen af alle nødvendige databehandling programmer og støtte filer. Den kan downloades på http://dispimlineage.wormguides.org.
CytoSHOW (http://run.cytoshow.org/) er baseret på den udbredte og open source billedanalyse platform, ImageJ (v1). Java skal være installeret og opdateret på computeren for at bruge CytoSHOW, og opdateringer til CytoSHOW installeres automatisk via Java Web Start. Mange ImageJ-baserede funktioner i CytoSHOW er som beskrevet og illustreret på https://imagej.nih.gov/ij/docs/examples/index.html. CytoSHOW er blevet tilpasset til at vise flerdimensionelle rå data fra ASI diSPIM, samt anden billedbehandling software, der skaber TIFF-output. I princippet kan andre MultiView SPIM billedsystemer understøttes af mindre ændringer af CytoSHOW, så denne protokol kan udføres på forskellige mikroskop systemer.
SpimFusion blev skrevet i CUDA/C++ med Visual Studio 2013 med CUDA Toolkit v 7.5. Kørsel af SpimFusion kræver specifik computer hardware: et GPU-kort (NVIDIA Graphics Processor Unit) med CUDA-databehandlingskapacitet 1,0 eller højere og mindst 2 GB grafikkorthukommelse. På tidspunktet for offentliggørelsen af vores protokol, SpimFusion er ikke offentliggjort (min Guo og Hari shroff), men findes i software Bundle Arkiv nævnt ovenfor.
En specialbygget kommandolinje drevet version af StarryNite kræver, at den frit tilgængelige MATLAB compiler Runtime er installeret, men kræver ikke en licens til kommerciel MATLAB-software. MATLAB compiler Runtime er inkluderet i software Bundle arkivet nævnt ovenfor. Koden for StarryNite som anvendt i denne protokol er stort set uændret fra den, der anvendes til confokale billeder6. Men, flere operationelle spørgsmål i skabelsen af input billeder til starrynite behandling og håndtering af starrynite resultater er blevet behandlet her ved metoder i cytoshow, der muliggør en kontinuerlig databehandling pipeline for smeltet isotropisk dispim Diskenheder. Disse ændringer er automatiseret af CytoSHOW kode, der håndterer disse præ-og efter behandling trin. CytoSHOW også redigerer en pre-optimeret diSPIM-specifik skabelon StarryNite parameter indstillet til automatisk at tune segmenterings algoritmen til fluorescens intensitet af kerner i de indpakkede data. De unikke parametre, der anvendes af StarryNite på hvert diSPIM datasæt gemmes derefter i en fil sammen med output-billede og line aging data.
En brugerdefineret version af AceTree, der fungerer med 16-bit-billeder og bevarer kompatibiliteten med Java3D-gengivelse, er bedst egnet til denne protokol. Det er også inkluderet i software Bundle Arkiv nævnt ovenfor.
Disclosures
Forfatterne har intet at afsløre.
Acknowledgments
Vi takker John Murray for integreret stamme, ujIs113, til at generere lineaging stamme BV514; Brandon Harvey (NIBIB) for at få hjælp til at teste protokollen; Jon Daniels og Gary Rondeau (Applied Scientific Instrumentation) for hjælp med Micro-Manager og diSPIM instrument; og Andrew York og Hank Eden for deres kritiske feedback på diSPIM systemet. Vi takker også forskningscenteret for mindretals institutioner-programmet og Instituto de Neurobiología Jose del Castillo (Universidad de Puerto Rico) for at levere en møde-og brainstorming-platform. Meget af dette arbejde blev udført på marine biologisk laboratorium i Woods Hole gennem Whitman programmet. Dette arbejde blev støttet af de Intramural forskningsprogrammer af NIH National Institute of Biomedical Imaging and Bioengineering og af NIH Grant No. U01-HD075602 og nej. R24-OD016474. Mark W. Moyle blev støttet af F32-NS098616 og Leighton H. Duncan blev støttet af et mangfoldigheds tillæg til R24-OD016474.
Materials
| Name | Company | Catalog Number | Comments |
| Steps 1-4 | |||
| Concavity slides | ThermoFisher Scientific | 1519006 | 5-18mm diameter, 0.6-0.8mm deep, 1.2-1.5mm |
| Dissecting microscope with 10×–50× zoom range | Motic | SMZ-171 | |
| E. coli (OP50) | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Glass coverslips, no. 1.5, 24 × 50 mm | VWR International | 48393-241 | |
| M9 Buffer | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Methyl cellulose | Sigma-Aldrich | H7509-25G | |
| Microcapillary pipette aspirator tube | Sigma-Aldrich | A5177 | |
| Microcapillary pipettes, 0.4-mm i.d | Drummond Scientific | 1-000-800 | |
| Needle, no. 18G x 1 ½ (1.2mm x 40mm) | BD Precision Glide | 305196 | |
| NGM plates | prepared as described by Brenner (1974) | ||
| O-ring for imaging chamber | O-Rings West | M1.5X40 | |
| Pasteur pipette | Corning/Sigma-Aldrich | CLS7095D5X | |
| Platinum wire, 0.5-mm diameter | Sigma-Aldrich | 267201 | |
| Poly-L-lysine | Sigma-Aldrich | P1524 | |
| Stainless steel rectangular chamber (76.0 mm x 50.5 mm) | Applied Scientifics Instrumentations (ASI) | I2450 | |
| Worm Eyelash Pick | Hart, A. C. Behavior. WormBook. (2006). | ||
| Worm Pick | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Name | Company | Catalog Number | Comments |
| Steps 5-6 | |||
| 488 nm long-pass filter | Semrock | LP02-488 RU-2 | |
| 561-nm notch filter | Semrock | NF03-561E-25 | |
| BLP02-561R-25, quantity 2 | Semrock | 561 nm EdgeBasic best-value long-pass edge filter | |
| Control software for bottom camera | Jenoptik | ProgRes CapturePro | |
| diSIPM assembly video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/TAgbr6IrTqw ; http://www.asiimaging.com | |
| diSPIM alignment video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/qnOrg30NNuE | |
| diSPIM imaging PC | Intel | Intel Xeon CPU E5-2630 2.6GHz, 12 cores in total, 64 GB memory, Windows 7 | |
| FF01-525/45-25, quantity 2 | Semrock | 525/45 nm BrightLine single-band bandpass filter | |
| FF555-DI03-25X36, quantity 2 | Semrock | 555 nm edge BrightLine single-edge dichroic beamsplitter | |
| Imaging PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Kumar et al diSPIM Setup | Applied Scientifics Instrumentations (ASI) | Instrument setup for this protocol is identical to Kumar et al 10,11 diSPIM, which makes use of 40x 0.8NA water immersion lenses for imaging. (See steps 5.1 and note) | |
| Micro Manager | Micro-Manager | https://micro-manager.org/ | |
| Modifications to Kumar et al diSPIM Setup (see below) | |||
| Optical table with isolators, 4 feet × 6 feet × 12 inches | TMC | 784-651-02DR and 14-416-34 | |
| Name | Company | Catalog Number | Comments |
| Steps 7-10 | |||
| Analysis PC | Intel | Intel Core i7-8700K CPU 3.70GHz, 6 cores in total, 64 GB memory, Windows 10 | |
| Analysis PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Installation instructions | Software bundle | http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm | |
| Software bundle | Software bundle | http://dispimlineage.wormguides.org |
References
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology. 100 (1), 64-119 (1983).
- Bao, Z., et al. Automated cell lineage tracing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 103 (8), 2707-2712 (2006).
- Santella, A., Du, Z., Bao, Z. A semi-local neighborhood-based framework for probabilistic cell lineage tracing. BMC Bioinformatics. 15, 217 (2014).
- Santella, A., Du, Z., Nowotschin, S., Hadjantonakis, A. K., Bao, Z. A hybrid blob-slice model for accurate and efficient detection of fluorescence labeled nuclei in 3D. BMC Bioinformatics. 11, 580 (2010).
- Boyle, T. J., Bao, Z., Murray, J. I., Araya, C. L., Waterston, R. H. AceTree: a tool for visual analysis of Caenorhabditis elegans embryogenesis. BMC Bioinformatics. 7, 275 (2006).
- Katzman, B., Tang, D., Santella, A., Bao, Z. AceTree: a major update and case study in the long term maintenance of open-source scientific software. BMC Bioinformatics. 19 (1), 121 (2018).
- Murray, J. I., et al. Automated analysis of embryonic gene expression with cellular resolution in C. elegans. Nature Methods. 5 (8), 703-709 (2008).
- Murray, J. I., Bao, Z., Boyle, T. J., Waterston, R. H. The lineaging of fluorescently-labeled Caenorhabditis elegans embryos with StarryNite and AceTree. Nature Protocols. 1 (3), 1468-1476 (2006).
- Wu, Y., et al. Spatially isotropic four-dimensional imaging with dual-view plane illumination microscopy. Nature Biotechnology. 31 (11), 1032-1038 (2013).
- Kumar, A., et al. Using Stage- and Slit-Scanning to Improve Contrast and Optical Sectioning in Dual-View Inverted Light Sheet Microscopy (diSPIM). The Biological Bulletin. 231 (1), 26-39 (2016).
- Kumar, A., et al. Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nature Protocols. 9 (11), 2555-2573 (2014).
- Wu, Y., Christensen, R., Colon-Ramos, D., Shroff, H. Advanced optical imaging techniques for neurodevelopment. Current Opinion in Neurobiology. 23 (6), 1090-1097 (2013).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17708-17713 (2011).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136 (12), 1963-1975 (2009).
- Stelzer, E. H. Light-sheet fluorescence microscopy for quantitative biology. Nature Methods. 12 (1), 23-26 (2015).
- Winter, P. W., Shroff, H. Faster fluorescence microscopy: advances in high speed biological imaging. Current Opinion in Chemical Biology. 20, 46-53 (2014).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. Astronomical Journal. 76 (6), 745-754 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. JOSA. 62 (1), 55-59 (1972).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Hart, A. C. Behavior. WormBook. , (2006).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. Chapter 14, Unit14 20 (2010).
- Gualda, E. J., et al. SPIM-fluid: open source light-sheet based platform for high-throughput imaging. Biomedical Optics Express. 6 (11), 4447-4456 (2015).
- Ardiel, E. L., et al. Visualizing Calcium Flux in Freely Moving Nematode Embryos. Biophysical Journal. 112 (9), 1975-1983 (2017).
- Walton, T., et al. The Bicoid class homeodomain factors ceh-36/OTX and unc-30/PITX cooperate in C. elegans embryonic progenitor cells to regulate robust development. PLoS Genetics. 11 (3), e1005003 (2015).
- Altun, Z. F. WormAtlas. , (2002).
- Wood, W. B. Embryology: In the nematode C. elegans. Cold Spring Harbor Laboratory Press. Chapter 8, 215-241 (1988).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314 (1165), (1986).
- Bao, Z., Murray, J. I. Mounting Caenorhabditis elegans embryos for live imaging of embryogenesis. Cold Spring Harb Protoc. (9), (2011).
- Christensen, R. P., et al. Untwisting the Caenorhabditis elegans embryo. eLife. 4, (2015).
- Santella, A., et al. WormGUIDES: an interactive single cell developmental atlas and tool for collaborative multidimensional data exploration. BMC Bioinformatics. 16, 189 (2015).
- Dupuy, D., et al. A first version of the Caenorhabditis elegans Promoterome. Genome Research. 14 (10B), 2169-2175 (2004).
- Reece-Hoyes, J. S., et al. Insight into transcription factor gene duplication from Caenorhabditis elegans Promoterome-driven expression patterns. BMC Genomics. 8 (27), (2007).
- WormBase. , Available from: https://www.wormbase.org (2019).
- Lee, R. Y. N., et al. WormBase 2017: molting into a new stage. Nucleic Acids Research. 46 (D1), D869-D874 (2018).



