Summary
Burada, nörogelişme sırasında tek hücreli dinamikleri anlamak için C. elegans embriyoları yaşayan yüksek çözünürlüklü mikroskobik, Hesaplamalı araçlar ve tek hücreli etiketleme kullanarak bir Kombinatoryal yaklaşım sunuyoruz.
Abstract
Halil özgül (C. elegans), tek hücre çözünürlüğü, in vivo ile, tüm sinir sisteminin hücresel kökenini anlamanın zorluk gözlemlenebilir tek organizma olarak öne çıkmaktadır. Burada, C. elegans embriyolarında nörogelişimin incelenmesi için entegre bir protokol sunuyoruz. Protokollerimiz, embriyolar gelişmekte olan tek hücrelerin görüntüleme, lineaging ve nöroanatomik izlemeyi birleştirir. Biz uzun vadeli elde, dört boyutlu (4D) canlı C. elegans embriyo ile neredeyse izotropik uzamsal çözünürlüğü ile çift görünümü ters seçici düzlem aydınlatma Microkopi (dispım) kullanımı ile. Nematod embriyolarında çekirdekler ve nöronal yapılar görüntülenmiş ve her üç boyutta ~ 330 nm çözünürlüğe sahip görüntüleri verim için izotropically erimiş. Bu dakikada dakika yüksek çözünürlüklü 4D veri setleri daha sonra, tek hücreli ve alt hücresel ayrıntı düzeylerinde gen ifadesi ve morfolojik dinamikler ile kesin hücre-satır kimlikleri ilişkilendirilmesi için analiz edilir. Protokollerimiz, açıklanan adımların her birinin modüler olarak uygulanmasını sağlamak ve embriyogenyon, gen ifadesi veya nörogelişim üzerinde çalışmalar geliştirmek için yapılandırılmıştır.
Introduction
C. elegans , embriyo içindeki her hücrenin nörogelişim boyunca gözleneceği tek organizma olarak öne çıkmaktadır. Tüm Cell-Lineage bilinen ve sabit1ile, ve embriyolar tek hücrelerin etiketleme ve sürekli görüntüleme sağlayan yeni araçların geliştirilmesi ile, biyologlar şimdi nematod sinir gelişiminde farklı adımları inceleyerek başlayabilirsiniz tüm açıları-hücre Doğum sistemi; göç ve farklılaşma; nörit oluşumu, hedeflenen büyüme ve fasciculation; sinaps oluşumu; ve fonksiyonel devrelerin ayarlanması. C. elegans embryo 'da nöronal büyüme dinamiklerini yakalamak, belirgin olarak ifade edilen gazetecileri ve floresan mikroskobu ile birleştirerek bilimsel toplum için değerlidir.
C. elegans gelişimsel çalışmalar genellikle sağlam organizma1içinde tek hücreli düzeyde bağlamsal anlayış artırmak için bu türün sabit hücre-soy ve hücre kaderi haritaları kaldıraç. Auto-lineaging Analizi-starrynite2,3,4 ve acetree kullanarak5,6,7,8 yazılım-yüksek kontrast faydaları, yüksek çözünürlüklü Floresan çekirdekleri görüntüleri. Optimal çalışmak için, StarryNite/Asetree de geliştirme sırasında görüntülenmiş embriyo öngörülebilir kısıtlı oryantasyona bağlıdır. İki kapak arasında sıkıştırılmış C. elegans embriyoları ile yapılan Konfokal mikroskopi, yüksek kontrast/yüksek çözünürlük ve öngörülebilir kısıtlı bir güç sağlar, çünkü on yıldan fazla bir süredir standart otomatik lineaging mikroskopisi yöntemi olmuştur. embriyo7,8' in oryantasyonu. Daha önce C. elegans embriyogenez9,10 gibi canlı örnek görüntüleme için yeni bir ışık yapraklık tabanlı çift-görünüm ters seçici düzlem aydınlatma mikroskop (dispım) inşaat ve kullanımı nitelendirdi , 11 ' i , 12 tane , 13. ışık levha mikroskobu, genel olarak, düşük fototoksisite, yüksek hız ve canlı 3D numunelerin uzun vadeli görüntüleme sağlar14,15. Dispım yöntemi, özellikle, yaklaşık 330 nm9yaklaşık izotropik uzamsal çözünürlüğe sahip dört boyutlu (4D) görüntüler üretir.
Konfokal microkopi ile karşılaştırıldığında dispım daha yüksek sinyal-gürültü ve hız, daha izotropik uzamsal çözünürlük sunar ve uzun vadeli In vivo görüntüleme16için daha uygundur. Bu nedenle, Dispım verilerini StarryNite/AceTree içine giriş için uyarlamaya çalışmıştık ve bu da lineaging analizlerini artırıp geliştirmeyeceğini araştırdık. Büyük bir engel Dispım örneklerin kolayca yumurta kabuğu-sıkıştırma tarafından StarryNite/Asetree için beklenen Oryantasyonlar benimsemek için kısıtlı olamaz olmasıdır. Analiz edilen hacimde hücre pozisyonlarının rasgele oryantasyonu, otomatik lineaging analizinin doğruluğunu düşürür.
Bu nedenle, kullanıcıların Dispım görüntülerin ön işleme sırasında embriyolar hassas 3D yönünü seçmek için izin veren bir izleyici destekli Kullanıcı arayüzü, hem kalite-optimize edilmiş görüntü verileri sağlayan ve StarryNite giriş için bağlam-Aware Siteshow istihdam /Asetree. Görüntülenmiş embriyolar Kullanıcı seçimi üzerine, Sitor otomatik bir veri işleme boru hattını düzenler. Kırpılmış ve arka planda açılan embriyo görüntüleri her konum, zaman noktası ve görünüm için TIFF yığın dosyaları içinde kaydedilir. Sitat sonra yinelemeli olarak Co-kayıt ve ortaklaşa iki önceden işlenmiş görünümleri deconvolve için program spimfusion çağırır, Richardson-Lucy17,18 algoritmasını kullanarak Isotropic yüksek çözünürlüklü hacimsel görüntüleri verim. Dispım özgü parametreler kümesi için en iyi duruma getirilmiş starrynite görüntü Segmentation sırasında davranışını yönetmek ve çekirdek izleme izotropically erimiş görüntüler. Fused görüntüler ve lineaging sonuçları sonra asetat kullanarak düzenlenebilir, hangi kullanıcıların tanımlamak ve StarryNite tarafından oluşturulan otomatik Lineage izleme herhangi bir hata düzeltmek sağlar. Asetree aynı zamanda embriyo içinde izlenen çekirdeklerin Lineage-Tree ve 3D modellenmiş render sunabilir. Biz Auto-lineaging hız ve doğruluk belirgin izotropically erimiş görüntüleri kullanarak gelişmiş olduğunu bulmak, ya SPIM kameradan ham görüntülere kıyasla. Bizim protokol, burada açıklanan C. elegans uygulaması için optimize ederken, genellikle diğer türler veya örnekler Için üretilen dispım veri otomatik çizgiyle adapte edilebilir. Bu protokol kullanımı amaçlanan ise, lütfen unutmayın starrynite parametreleri ek ayarlama büyük olasılıkla yeni örnekler için gerekli olacaktır, açıklandığı gibi3,4.
4D-izotropik çözünürlüğe sahip görüntülerde bu protokolün başarılı bir şekilde uygulanması ve biyologlar hücre sırlarını izlemek için sağlar, aynı anda tanımlayan ve gelişmekte olan C. elegans embriyo nöronlar analiz ederken. Dahası, birkaç post-processing algoritmaları birleştirerek-bu en zaman alıcı olan donanım hızlandırma ile-şimdi hem ince hücre altı detayları ve hücre-lineages ve gerçek zamanlı olarak canlı embriyo hücre-kaderlerde analiz edebilirsiniz. Bu yeni protokol, diferansiyel ve morfojeninin in vivo probasyon çalışmaları sırasında hücre davranışlarını hassas, bilgili manipülasyon ve gözlem sağlar. Bu yazıda, C. elegans embriyoları geliştirmede lineaging ve hücre takibi için geliştirdiğimiz gelişmiş protokollerin ayrıntılı bir açıklamasını sunuyoruz, embriyo, gen ifadesi veya nörogelişim çalışmalarını geliştirmek.
Protocol
1. dispim çelik görüntüleme odasının Poly-L-lizin kaplamalı lamel magazini ile montajı
Not: aşağıdaki adımlar, nematod embriyoların Lineage analizini starrynite/acetree tarafından optimize etmek ve otomatikleştirmek için gereklidir. C. elegans hücre sırlarını izleme gerektiren deneyler için birkaç seçenek (böyle belirtildiği gibi) atlanabilir.
- Bir Sharpie (veya benzer kalem) ile temiz bir dikdörtgen lamel magazini (No. 1,5, 24 mm x 50 mm) ortasına küçük bir dikdörtgen (2mm x 5mm) çizin.
- Lamel magazini işaretli olmayan tarafa çevirin ve 10 μL Poly-l-lizin yerleştirin (Cat. No. P1524) işaretli dikdörtgen üzerinde.
Not: 1 mg/mL Poly-L-lizin filtrelenmiş suda (veya eşdeğer) çözünmüş bir çalışma konsantrasyonu yapın. Uzun süreli depolama için 5 – 10 ml plakaya hazırlayın ve − 20 °c ' de saklayın. Çözülmüş bir kez, plakaya 3 – 4 hafta oda sıcaklığında (23 °c) saklanabilir. - Poly-L-lizin lamel magazini 5 dakika (Şekil 1a) için kat izin verin. Poly-L-lizin, embriyo yumurta kabuğunun, hedefleri görüntüleme ortamına batırılmış olsa bile coverslip 'a sıkıca yapışmasına izin veren, embriyoların monte edildiği cam kapak fişlerinin kaplama için kullanılır.
- Poly-L-lizin kaplamalı lamel magazini çelik görüntüleme odasının alt yarısında yerleştirin.
- Çelik görüntüleme odasının üst yarısını lamel magazini ile alt yarısında yerleştirin ve oda ile ilişkili dört vidayı sıkın. Üst yarısını eşit olarak alt yarısında oturduğundan emin olmak için taraftan görsel olarak kontrol edin (Şekil 1B).
- 7 – 8 mL M9 tampon19ile doldurun, erken evre embriyoların anormal osrotik basınca düşmesini önlemeye yardımcı olan bir izotonik tampon. 1 hücreli ve hatta 2 hücreli embriyolar ozmotik açıdan duyarlıdır ve bu izotonik tamponda değilse anormal gelişebilir. Embriyolar da M9 su ile yerine 3 kat aşamada tutuklama eğilimindedir.
2. montaj için C. elegans embriyo örnekleri hazırlanması
Not: yaklaşık 18 saat önce görüntüleme, beş genç (son Molt beri 1 gün) yetişkinler ve on larva Aşama 4 (L4) C. elegans bir nematod büyüme orta (NGM) agar plaka e. coli strain OP50 ile seribaşı için seçilmiş. Platin tel alır hayvan19zarar vermeden larvalar ve Genç Yetişkin C. elegans taşımak için kullanılır.
- % 1 metil selüloz hazırlayın (kedi. H7509-25G) çözüm M9 tampon.
Not: metil selüloz sıcak M9 ' de çözülene kadar karıştırılmalıdır. Bu çözüm, hazırlanan bir kez oda sıcaklığında saklanabilir. - 500 μL% 1 metil selüloz-in-M9 solüsyonu bir konkav mikroskop kaydırağı depresyona ekleyin. Bu viskoz tampon iki kez kullanılacaktır: 1) Yetişkin solucanların diseksiyonu ile erken embriyolar hasat ve 2) geç evre embriyo yıkama zaman doğrudan bir NGM plaka aldı.
Not: metil selüloz, embriyolar mikroskop kaydırağı yapışmasını önlemek için kullanılır. - Görüntüleme geç aşama embriyolar için, bir kirpik Pick kullanarak, (tercihen genç yetişkinler ile bir NGM plaka itibaren) C. elegans embriyoları seçerek, ve embriyo hareket 1% konkav mikroskop slayt üzerinde metil selüloz. Kirpik Pick kuvvet azaltmak ve böylece işleme sırasında embriyo stres veya zarar en aza indirmek yardımcı olur. Bir kirpik pick yapmak için prosedür Hart20ile kaplıdır.
- İkinci bir kirpik Pick (zıt el) ile, metil Selülozda embriyoları askıya almak için her iki kirpik birlikte hafifçe dokunun.
- İsteğe bağlı: Eğer StarryNite ile Lineage embriyonik hücreler için planlama, bir 4 hücreli embriyo 1 hücreli monte edilmelidir. Bunu yapmak için, önce bir NGM plakadan genç yetişkinler seçin ve platin tel çekme kullanarak konkav mikroskop slayt üzerinde M9-metil selüloz çözeltisi içine taşıyın.
- Opsiyonel: hipdermik iğneler (No. 18G x 1 1/2) ile keskinleştirilmiş ipuçları ile, 4 hücreli embriyolar 1 hücreli serbest bırakmak için orta vücutta enine hayvan dilim.
3. Mouth pipet: mikrokapiller pipet ile aspiratör tüpü montajı
Not: tüpün kauçuk contası içine takılan bir el çekti mikrokapiller pipet ile bir aspiratör tüpü kullanıyoruz. Bu da embriyoları tampon dolu görüntüleme odasındaki Poly-L-Lysine kaplı yüzeye diseksiyon slaytından aktarmamızı sağlar.
- Genişletilmiş ipuçları ile iki yarısı oluşturmak için açık bir alev üzerinde mikrokapiller pipet elle çekin.
- Mikrokapiller pipetin bir yarısını alın ve künt ucunu aspiratör tüpünün kauçuk contası içine takın (Şekil 1C). Mikrokapiller pipetin diğer yarısını daha sonra kullanmak üzere bir kenara koyun (gerekirse).
- Mikrokapiller pipet ile donatılmış monte aspiratör tüpü ile, mikro kapiller pipetin ucunu yavaşça kırın ve yaklaşık 1 – 2 iki embriyo sığacak bir açılış oluşturun (buradan Bu aletten "ağız pipet" denir).
4. Poly-L-lizin kaplamalı lamel magazini üzerinde C. elegans embriyoları montajı
- Aspiratör ağızlı dişler arasında yavaşça tutulan ile, 10 – 15 μL M9 tampon ile mikrokapiller pipet önceden doldurun ve sonra yavaşça kapiller içine konkav slayt birkaç embriyo emmek.
- Embriyoları M9 tampon ile dolu çelik görüntüleme odasına aktarın, böylece embriyoların coverslip 'ın merkezi dikdörtgenine düşecek şekilde kapiller ucunu konumlandırın.
- Embriyolar yaralanmasını önlemek, yavaşça bir kirpik Pick ile onları taşımak ya da embriyoları dikey olarak konumlandırmak için ağız pipet kullanın, böylece embriyo uzun ekseni lamel magazini uzun eksenine dik olan embriyolar yönlendirmek için (Şekil 1B Inset , alt panel).
Not: bu yönde embriyonun konumlandırılması, görüntü dilimlerinin sayısını en aza indirir, böylece satın alma hızını iyileştirirken ışık dozajı ve veri işleme süresini azaltır. - Çelik görüntüleme bölmesini mikroskop aşamasında örnek tutucuya yerleştirin (Şekil 1D).
5. Dispım kullanarak embriyonik görüntüleme için montaj, yazılım kurulumu ve Lazer optimizasyonu
- Kumar ve el.10,11 ve http://www.dispim.org adresinde ticari olarak kullanılabilen parçaların tamamına fiber ile bağlanmış dispim 'in nasıl montajına ilişkin adım adım talimatlar konusuna bakın. Dispım montajı için bir video-protokol da ASı Web sitesinde (http://www.asiimaging.com) mevcuttur.
Not: Bu protokol için cihaz kurulumu, görüntüleme için 40X 0.8 NA su daldırma lensleri kullanan kumar ve el.10,11 dispim ile aynıdır. Bu protokoldeki kurulum ile kumar vd.10,11 arasındaki tek fark, bir görüntüleme bölme cihazı (model A12801-01) içinde yüklü bir Dikroik ayna (560 nm 'de bölme) ve kırmızı ve yeşil bant geçiren filtreler eklenmesi diSPIM görüntüleme kolları. Görüntü bölme optik ilavesi iki farklı fluorophores görüntüleri eşzamanlı yakalama sağlar – 561 nm ve 488 nm lazerler tarafından heyecanlı-aynı kamera yongası iki yarısı üzerine emisyon bantları ayırarak. - Enstrüman montajdan sonra, görüntüleme işleminden önce diSPIM optik hizalamasını kontrol edin.
Not: Dispım düzgün hizalamak Için https://youtu.be/qnOrg30NNuE bakın ve donanım bilgileri için http://dispim.org/hardware/objectives ve http://www.asiimaging.com. - Yüksek verim hücresel görüntüleme22Için ışık levha mikroskopları işletim için optimize edilmiş açık kaynak platformu Micro-Manager (https://Micro-Manager.org/)21kullanın. En fazla 30 embriyo gibi eşzamanlı görüntüleme sağlayan çok pozisyonlu edinme için ASI Dispım eklentisi kullanmanızı öneririz23.
- Micro-Manager açık ile, 488 nm için ~ 179 μW (0,5) ve 0,25 Nm için ~ 79 μW (561) için lazer yoğunlukları ayarlayın (Şekil 2a, kırmızı dikdörtgen).
Not: Bunlar, 1 dakikalık aralıklarla C. elegans embriyoları uzun süreli görüntüleme için önerilen ayarlar. Uzun vadeli çift renkli görüntüleme sırasında, 561 nm lazer görüntü çekirdekleri için kullanılır (MCherry:: Histone) embriyo fasulye aşamasında olana kadar, hangi noktada 488 nm lazer sonra da görüntü Gfp etiketli nöronlar açın. Bu görüntüleme koşulları fototoksisite en aza indirmek için optimize edilmiş ve uzun süreli (12 – 14 saat) nörogelişimsel ve lineaging veri sürekli edinme sağlayarak embriyoların hayatta kalma ve hatching sağlanması. - Micro-Manager ' ı seçin menü eklentileri ≫ cihaz kontrolü > ası dispim asi Dispim penceresini açmak için (Şekil 2B). Edinme sekmesini seçin. Bu sekmenin veri kaydetme ayarları bölümünde (yeşil dikdörtgen), birim ayarları bölümü (mavi kare) ve dilim ayarları bölümü (turuncu kare), her parametrenin Şekil 2B 'de gösterildiği gibi ayarlandığından emin olun.
Not: bizim görüntü analiz yazılımı Sitam gibi diğer isteğe bağlı çıkış veri formatları ile çalışmak için adapte edilir toplu birleştirilmiş OME-TIFF dosya serisi ve TIFF-Stack dosya Serisi Mikro-Manager içine yerleşik bir ihracat işlevinin kullanımı ile post-edinme oluşturuldu. Genellikle, tek zamanlı nokta yığını harmanlanmış OME-TIFF dosya veri biçimi, gerçek zamanlı görüntüleme ve ham veri edinilen en kısa sürede görüntü hacmi işleme sağlar çünkü kullanılır.
6. C. elegans embriyoları uzun süreli görüntüleme için optimize edilmiş otofokus parametreleri
- C. elegans embriyoların uzun süreli Lineage kalitesinde dispim-Imaging için optimize edilmiş ayarlara Micro-Manager otofokus parametrelerini ayarlayın. ASı Dispım penceresinde, otomatik netleme sekmesini (Şekil 2C) tıklatın. Genel otofokus seçenekleri bölümünde (siyah kare) parametreleri tam olarak gösterildiği gibi belirtin. Otomatik netleme kanalının (kırmızı kare), nükleer kanal floresan kanalınızı lineaging deneylerinde belirlemesi gerektiğini unutmayın.
Not: maksimum uzaklık 5 μm ' den büyükse, görüntüler odak dışına sürüklenmeye eğilimlidir. - Tıklayın, eklentiler ≫ edinme araçları > desen yerleşimi.
- Desen yerleşimi penceresinde ızgarayı göster'i tıklatın.
- Ası Dispım penceresinde gezinme sekmesini tıklatın.
- A veya B yolu kiriş ve sayfa için onay kutularını tıklatın sonra canlı'ı tıklatın. Görüntü edinme başlar. Canlı Görünüm penceresi açılır. 6,1 gelen seçilen kanalda embriyonun etrafında bir kutu çizerek embriyonun otomatik odaklama analiz bölgesini seçin.
Not: genellikle görüntüleme oturumu başına 10 embriyo için 420 zaman puanı yakalıyoruz. Görüntü oturumu başına ham veri genellikle 1.7 TB, deconvolved ve StarryNite işlenmiş veri 1.4 TB (adımlar 9 ve 10 bakın). Görüntü edinme ve görüntü depolama için bulut platformları için büyük kapasiteli HDD (mevcut sistemimizde 18TB) kullanmanızı öneririz. - Uzun vadeli çok boyutlu görüntü yakalama (Şekil 2B) başlamak Için "edinme sekmesinde edinme Başlat 'ı tıklatın.
7. RAW Micro-Manager görüntülerini Sitat gösterisinde açma
- Http://dispimlineage.wormguides.org sitesinden yazılım paketini indirin.
Not: yazılım paketi bir. zip dosyası olarak indirilecektir ve "C:\" için ayıklanacak gerekir kullanmadan önce dizine kullanın. Kurulum için daha fazla ayrıntı http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm adresinde verilmiştir. - Sitagösterisi çalıştırmayı başlatmak için C:\sitıshowextrasforc\sitıshow_app.jnlp dosyasını çift tıklatın.
- Menü dosyası > Yeni > dispım Monitor (Micro-Manager)seçin. Edinme timepoint klasörlerin kaydedildiği kök veri kümesi klasörünü bulun. Herhangi bir timepoint klasörünü seçin ve Aç'ı tıklatın. Çok boyutlu gezinme pencereleri ( Dispım monitör Windows olarak adlandırılır) spıma ve SPıMB (Şekil 3A) için otomatik olarak açılır.
Not: Bu pencereler, yeni kaydedilmiş ham timepoint yığınları için kök veri klasörünü (bir numune hala kaydediliyor durumunda) izleyecek. Her yeni zaman noktası elde edildikten sonra, her bir embriyo için çok kanallı 4D veri kümesini görüntülemek üzere farklı SPıM kolları ve örnek konumlarını izleyen her pencere yenilenir.
8. sitit ile Max projeksiyon görüntüleri oluşturma
Deconvolution önce bile, ham veri hızlı bir şekilde numunenin küresel özellikleri değerlendirmek için işlenebilir.
- Belirli bir pozisyonun veya SPıM kolunun tam derinliği ve tam zamanlı ders yoluyla maksimum yoğunluk projeksiyonları yapmak için görüntü penceresinin (Şekil 3A, kırmızı dikdörtgen) sol tarafındaki panelinde Z-MIP düğmesini tıklatın. Z-projeksiyon hyperstack penceresi görüntülenir.
- Z-projeksiyon hyperstack penceresinde, yansıtma türünü Maksimum yoğunluğaayarlayın. Hangi kanalları, dilimleri ve zaman noktası çerçevelerini Kullanıcı tercihine göre işleyecek şekilde belirtin.
- Tamamlandığında Tamam 'ı tıklatın.
- Dosya diyalog penceresinden Maksimum yoğunluk çıktılarını kaydetmek için klasör konumunu seçin ve Tamam'ı tıklatın. Bir zaman izin (15 – 20 dakika, veri kümesinin boyutuna bağlı olarak ve bilgisayarın işleme gücü) için Sitongösteri projeksiyon görüntüleri oluşturmak için.
9. izotropik yüksek çözünürlüklü Volumetrik veri hücre sırlarını analiz
- İsteğe bağlı: ham veri Sitokte Dispım monitörü ile açıkken, çokgen seçim aracını (Şekil 3A, siyah oklar) seçin ve embriyonun anterior, posterior, dorsal ve ventral kenarları (Bu kesin sırada) hemen dışında tıklayın Embriyo üzerinde bir "Bowtie" deseni oluşturmak. Her iki görünüm için de (SPıM-A ve SPıM-B, Şekil 3A).
Not: Bu seçim, embriyonun ortalanmış olduğu ve embriyonun anterior-posterior eksenini kaydeden eliptik ilgi (YG) bölgesini belirtir. Papyon desen ipuçları sitashow Kullanıcı daha da son izotropically erimiş hacimlerin kesin bir rotasyon starrynite/asetree tarafından dizme analizleri için optimum bir oryantasyona belirtmek için planlıyor. StarryNite lineaging deneysel planın bir parçası olmadığı durumlarda, diğer seçim araçları ve şekiller görüntü işleme için YG ayarlamak için seçilebilir. - Birden fazla embriyo aynı anda çok pozisyonlu edinme seçeneği kullanılarak görüntülenmiş ise, açık ve tüm embriyo için adım 9,1 gerçekleştirin. Bu, bir oturumda tüm embriyolar için gelecekteki adımların paralel yürütülmesini sağlar. İşlemek istemiyorsanız herhangi bir embriyo için ERI ve SPıMB pencereleri kapatın.
- Dispım monitör penceresinin sol tarafındaki paneldeki dispim düğmesini tıklatın (Şekil 3A, sarı renkte vurgulanır). Bu diSPIM işleme özgü denetimlerin bir alt paneli ortaya çıkarır.
- Her SPıM kolu için yeşil ve kırmızı kanalları hizalayın. Emisyon kanal ayırıcılar aynı kamera aynı anda farklı kırmızı ve yeşil görüntüleri yakalamak için kullanılır, çünkü onlar overlaid zaman bu iki fiziksel bitişik görüntü alanlarının tam piksel kaydını görsel olarak hizalamak önemlidir. Aynı hizalama ayarlamaları genellikle birçok ardışık görüntüleme oturumları üzerinde uygulanabilir, ancak kontrol edilmelidir (Steps 9.4.1 – 9.4.5).
- SPıMA panelinden başlayarak, cm Scroll çubuğunu sola taşıyarak kırmızı kanalı seçin (Şekil 3A, turuncu ok, sol panel).
- X-, y-ve z-ayarlayıcıları kullanarak (Şekil 3A, turuncu kare), kırmızı kanalı yeşil ile eşleşecek şekilde kaydırın.
- Tıklatın Dispim düğmesi (Şekil 3A, sarı vurgulanan), alt paneli kapatmak ve diğer tüm pozisyon Windows aynı vardiyaların tetiklemesi yayma.
- "Z" kaydırma çubuğunu (Şekil 3A, mavi ok, sol panel) ve/veya "t" kaydırma çubuğunu (Şekil 3A, yeşil ok, sol panel) taşıyarak, doğru hizalamanın diğer çerçevelere ve zaman noktalarına yayılmasını onaylayın. Çok pozisyonlu satın alma işlemi yapılırsa ve birkaç embriyo görüntülenmiş (adım 5,3), hizalama da bu embriyo yayılmalıdır. Ayrıca x-, y-ve z-ayarlayıcılar (Şekil 3A, turuncu kare, tüm EMBRIYOLARıN spıma paneli için aynı olmalıdır) numaralarını inceleyerek onaylayın.
- 9.4.1 adımları yineleyin – SPıMB diSPIM monitör penceresi için 9.4.4 (Şekil 3A, sağ panel).
- "Dispim" düğmesine ve ardından "sigorta" düğmesine (Şekil 3A, mavi dikdörtgen) "Deconvolve/Fuse Dispım RAW veri birimleri" (Şekil 3B) adlı bir iletişim kutusunu açmak için tıklayın. Şekil 3B'de gösterildiği gibi parametreleri ayarlayın. Bu parametreler kısa bir süre içinde aşağıdaki alt adımlarda ele alınmıştır:
- Kanal 1 (488 nm lazer) veya 2 (561 nm lazer) anahtar kaydını ayarlayın. Daha yoğun veya daha fazla sinyalli kanalı seçin. Satır boyu deneyler için her zaman her yerde nükleer histon floresans görüntü için kullanılan kanal seçin.
- Giriş birimlerinin göreli yönünü + 1 veya-1 olarak ayarlayın. Doğru oryantasyon indeksi, belirli bir Dispım 'ın kamera yerleşimlerine bağlıdır (Şekil 4).
Not: belirsiz, her iki SPıM A ve B dispım monitör penceresindentek bir zaman noktası çoğaltarak her seçeneği sınayın, adımları 9.1 – 9.12 ve rasgele bir giriş birimleri test etmek için göreli oryantasyonu seçerek. Yanlış oryantasyonlar, yapılarla bulanık görüntüler verirken, doğru Oryantasyonlar net görüntüler verecektir. Açık görüntüyü veren giriş birimleri göreli oryantasyon değeri, sonra verilen dispım enstrümanından gelecekteki tüm veriler için yeniden kullanılabilir. - Erimiş birimin A veya B giriş birimiyle aynı şekilde yönlendirilmiş olup olmadığını seçin (Kullanıcı tercihine göre).
- "Her hacim için taze kayıt" seçeneğini belirleyin. Bu seçenek, her zaman noktasında her birim çifti için Spımfusion 'ın kayıt matrislerini nasıl hesapladığını denetler. "Fresh" seçeneği, algoritmanın her zaman noktasında kaydı adaptif olarak optimize etmesini sağlar.
- Deconvolution yineleme sayısını 10 olarak ayarlayın. Bu sayı, istenen yüksek çözünürlüğü zaman verimli bir şekilde güvenilir bir şekilde verim eğilimindedir.
- İsteğe bağlı: otomatik lineaging isteniyorsa (tavsiye edilir), Fused birimlerin StarryNite lineaging otomatik başlatmakontrol edin. Bu seçenek, Spımfusion tarafından üretilen görüntülenmiş birimlerin hücrelerini segmentlere ayırmak ve izlemek için otomatik olarak StarryNite 'i başlatacaktır.
- İsteğe bağlı: otomatik lineaging en yüksek doğruluk için, en iyi kurallı "adl" (birnterior [x-West], Dorsal [y-Kuzey], sol [z-yakın]) oryantasyona izotropik erimiş embriyo hacimleri yeniden konumlandırmak için. Bu seçimi belirtmek için Önizleme seçeneğinde birim çıkış yönünü tanımla seçeneğini belirleyin. Siteyal, kullanıcının ADL kaydına ulaşmak için gereken rotasyonları yakından gözlemlemek ve belirlemesini sağlayan, izotropik olarak erimiş hacimlerin ilk çiftini işleme alarak yanıt verecektir.
- Tüm parametreler seçildiğinde Evet 'i tıklatın.
- İşlenen dosyaların kaydedileceği çıkış dizinini belirtin. Tamam'ı tıklatın.
- İsteğe bağlı: Önizleme 'de birim çıkış yönünü tanımla seçeneği işaretlenmişse, SPIM-A penceresinde t kaydırma çubuğunu (Şekil 3A, yeşil ok, sol panel) ABA ve ABP hücrelerine metafaz ulaştığı erken zaman noktasına ayarlayın. SPıM-B penceresindeki t kaydırma çubuğunu daha sonraki virgül aşamasına geliştirme olarak ayarlayın. Bu, ADL oryantasyonunu belirtmede yardımcı olacaktır.
- İsteğe bağlı: hazır olduğunda Tamam 'ı tıklatın. Yukarıdaki 9.5.7 Önizleme seçeneği seçilmişse, SPIM-A ve SPIM-B görüntü pencerelerini t-sürgüleri tarafından belirtilen zaman noktaları için yalnızca iki ön izleme birimi izotropik olarak erimiş olacaktır. Bu iki önizleme zaman noktaları, aşağıda açıklandığı gibi, ADL oryantasyonuna çıkış embriyo hacimlerini kesin olarak belirtmek için kullanılabilir.
- Bulmak yeni görüntülenen 3DProjY_Decon-Fuse_.... Pencere. T kaydırma çubuğunu bu önizleme penceresinin saat noktası 2 ' ye taşıyın. Z sürgüsünü doğrudan embryo uzun ekseninin gösterilinceye kadar hareket ettirin.
- T kaydırma çubuğunu geri saat noktası 1 ' ın 3DProjY_Decon-Fuse_.... Pencere. Satır seçimi aracını seçin ve AB hücresi metafaz plakaları düzleminden EMS hücresinden (ventral-en yuvarlak çekirdeği) bir çizgi seçimi çizin.
- Tıklayın Orange diSPIM Önizleme düğmesi 3DProjY_Decon-Fuse_.... Pencere. Önizlenen görüntülenmiş birimin oryantasyonuna yönelik ince ayarlamalar, tam veri kümesinin işlenmesinde kullanılmak üzere kaydedilecektir.
- İsteğe bağlı: Deconvolve/Fuse Dispım RAW veri birimleri iletişim kutusu, yukarıdaki adım 9,5 gibi yeniden görünecektir. Önizleme seçeneği birim çıktı yönünü tanımla seçeneğini belirmeden Evet 'i tıklatın. Tam veri işleme çalıştırmak için çıktı klasörünü belirtin.
- Dispım İzleyicisi pencerelerin t kaydırma çubuklarını (Şekil 3A, yeşil ok, sol panel) Başlangıç timepoint (spıma) ve bitiş timepoint (spımb) için tam span görüntüleri işlemek için ayarlayın. Sonra Tamam' ı tıklatın.
- Spimfusion ilerledikçe, sitroshow açılır ve her embriyo için dilimlenmiş-4d izotropik erimiş hacmi gösteren çok boyutlu bir pencere yeniler, hem de dönen-4d Max-yoğunluğu projeksiyonları izotropik hacim ile iki pencere. Bu süre zarfında, izotropik Fusion ve lineage izleme işlemi tamamlanıncaya kadar herhangi bir sitshow penceresini kesintiye uğratmayın veya kapatın.
- İsteğe bağlı: StarryNite giriş ekranı ortaya çıktığında ve daha sonra kaybolduktan sonra tam veri işleme ardışık düzenini tamamladığını unutmayın. Bu pencere işleme sırasında kapalı olmamalıdır veya StarryNite kesintiye uğrar.
10. Asetree içinde StarryNite soy iz serisi açılış (opsiyonel)
- Sağlanan "AceTree_16BitCompat. jar" özelleştirilmiş sürümünü açın.
- Menü dosya > açık yapılandırma dosyasınıseçin. Çıktı dizininizi daha önce Sitıshow 'da bulun. Embriyo [n] için Decon_Fuse_... _Pos [n] alt klasörünü açın. Seçin aaa_edited. xml ve Açık.
- Düzenleme parça açmak ve ayarlamak veya hücreleri Windows silmek Için asetree menü düzenleme > düzenleme araçlarını kullanın.
- Kırmızı ve yeşil yoğunlukları ayarlamak için yarı gölgeli daire Şekil 5B, kırmızı kare tıklayın.
- Daha önce açıklandığı gibi Lineage görselleştirme ve düzenleme ile devam5,6,8 (el yazmaları da bizim indirme paketinde yer almaktadır).
Representative Results
İlk olarak dispim edinme (bölümler 1-6) için protokolün parametrelerini kullanarak görüntülenmiş embriyolar canlılığı doğrulanmış. On embriyo aynı anda 20 °C, bir hacim/embryo/dakika, 2 hücreli aşamasından 2 kat aşamaya (7,5 saat, 451 hacimler/embriyo) olarak görüntülenmiş. Embriyogenesis boyunca hücre bölümlerini izlemek için, biz her yerde MCherry::histone Reporter yapıları entegre transgene dizi ujIs11324ifade gerginlik BV514, kullanılır. Şekil 6 , görüntülenmiş embriyolar için embriyonik gelişimin ilk yarısının bir zaman çizelgesini gösterir. Her görüntü, görüntülenmiş embriyonun tek görünümlü maksimum yoğunlukta projeksiyon (7-8 adımda üretilir) temsil eder. Biz optimize protokollerin embriyolar için herhangi bir algılanabilir fototoksisite neden vermedi bulundu, Hücre bölünmeleri zamanlaması (gösterilmez) tarafından değerlendirildiği gibi, hatching zaman, ve zamanlama gelişimsel kilometre taşları ile ilgili (Şekil 6 ve referanslar1 , 25 , 26).
Daha sonra embriyolar gelişmekte olan tek nöronların büyüme dinamiklerini analiz etmek için protokol uyguladıysanız. Biz ımaged DCR7692 (olaex4655), bir transgenik nematod gerinim bir alt kümesi içinde neuropeptid FLP-19 Organizatör kapalı Gfp ifade eder (DACR2819, PFLP-19 (3.6 KB):: Syn21:: Gfp:: caax::p 10 3 ' UTR) . Burada özetlenen protokolün adımlarını izleyerek, tanımlanamayan hücrelerin, RMDDL ve RMDDR motor nöronlarına, boşaltım kanal hücresine ve iki kas hücresine karşılık geldiğini tespit ettik (Şekil 7). Daha sonra, RMDDL ve RMDDR nöronların büyüme dinamiklerini inceledi ve değerlendirdik. Biz RMDDL ve RMDDR nöronların oblikik olarak erken 360 dakika gübreleme sonrası olarak şekillendirilir gözlenen, daha uzun hücresel eksen nörit büyüme için sonraki ekseni temsil eden (Şekil 7 ve film S1). "Basit nörit izleme" FIJI eklentisi kullanarak ve izotropically erimiş hacimlerin 3D reinşaatları uygulayarak, daha sonra altı embriyo için RMDDL ve RMDDR nöronların stereotifik büyüme nicelik. Biz büyüme dinamikleri RMDDL ve RMDDR embriyolar (burada RMDDs denilen) boyunca stereotipleşti olduğunu tespit etti. İtibaren 385-410 dakika gübreleme sonrası, RMDDs nöronlar Extended 6,0 ± 0.5 μm (ortalama ± SEM; n = 12 nörit) hücre gövdelerin anterior (şekil 7b, C, ı). İtibaren 415-445 dakika döllenme sonrası, her iki nörosit içine ve varsayımsal sinir halkası etrafında dorsal uzatmak ( Şekil 7dyıldız). Ortalama olarak, her RMDD neurite genişletilmiş 11,0 ± 0,6 μm (ortalama ± SEM; n = 12 nörit) hücre gövdesinde, eşzamanlı olarak halka zirvesindeki kontralateral meslektaşı (Şekil 7i) ile buluşmadan önce. Daha da önemlisi, temsili sonuçlarımız, entegre protokollerimizi (Şekil 7 ve Şekil 8) kullanarak tek tanımlanabilir hücreler için nöronal gelişimsel özellikleri inceleyebilmemizi, karşılaştırabilmemizi ve ölçmemizi mümkün olduğunu göstermektedir.
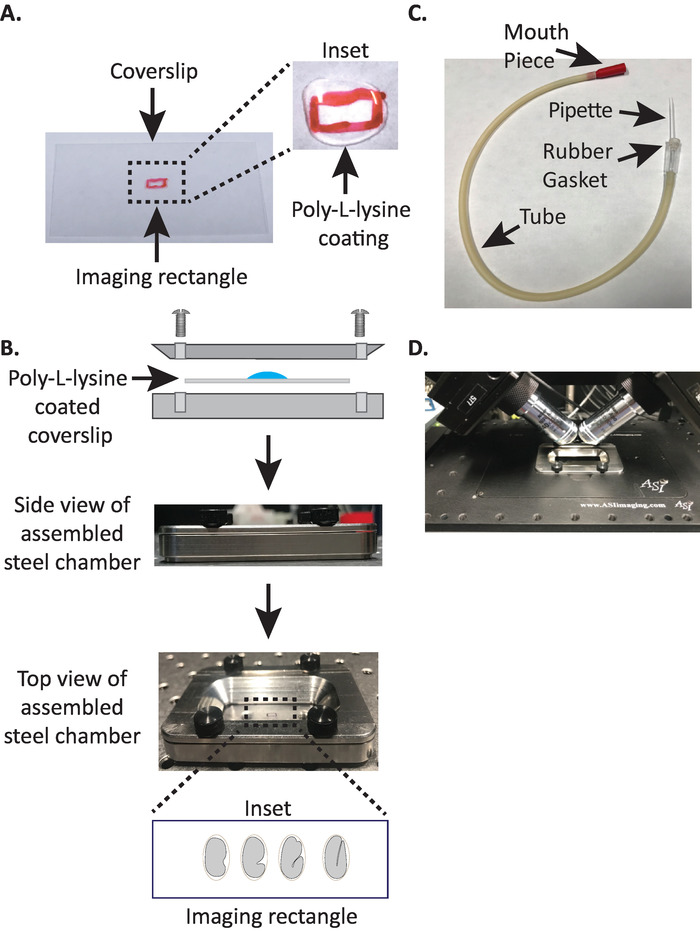
Şekil 1: diSPIM örnek montaj kurulumu. (A) Poly-L-Lysine ile lamel magazini hazırlanması. İnset içinde, 10 μL Poly-l lizin 5 dakika için lamel magazini ceket için kullanıldı. Poly-L-lizin, embriyo yumurta kabuğu dikdörtgen içinde lamel magazini sıkıca sopa sağlar. (B) çelik görüntüleme odasının ve montajlı odanın şematiği. Inset, temsili embriyolar ön-posterior eksen coverslip üzerinde uzun eksen dik ile yönlendirilmiş. (C) mikrokapiller pipet ile monte edilen aspiratör tüpü. (D) dispim 40X hedefleri altında örnek tutucuya monte edilmiş çelik görüntüleme odası. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 2: mikro yöneticisinde uzun süreli diSPIM görüntüleme kurulumu. (A) fotootoksisite azaltarak ( C. elegans embriyoları yüksek hatching oranı ile değerlendirilir) uzun süreli görüntüleme için optimize dispim lazer güç parametreleri (kırmızı dikdörtgen) önerilir. 561 nm lazerle 79 μW (0,25) ve 488 nm lazer ile 179 μW (0,5) arasında ayarlayın. Yazılım ayarlarının lazer gücüne tam olarak kalibre edilmesi diSPIM kurulumları arasında değişir. 79 μW (561 Nm) ve 179 μW (488 Nm) lazer gücü elde etmek için kullanıcıların lazer gücünü ölçmeye ve kalibre etmesi önerilir. (B) veri kaydetme (yeşil dikdörtgen), ses ayarları (mavi kare) ve dilim ayarları (turuncu kare) için dispim parametreleri. (C) c. elegans embriyogenez 'in uzun süreli görüntülenmesi için dispim otofokus parametreleri (bkz. adımlar 6.1-6.6). Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 3: Sitshow kullanarak görüntü görselleştirme ve veri işleme Kurulumu. (A) Sitat tarafından açılan RAW dispim görüntüleri. Sitshow her iki kamera yolu (SPıM A ve B) tarafından yakalanan görüntüleri açmak mümkün. Bu ham görüntüler Dispım monitörüolarak adlandırılan çok boyutlu pencerelerin açılır. Dispim monitörde, bir "Bowtie desen" embriyo ön, posterior, dorsal ve ventral kenarları seçmek için oluşturulur (bkz. Adım 9,1). Bow-tie seçimleri deconvolution ve StarryNite destekli lineaging izleme için embriyo yönünü gösterir. (B) izotropik görüntüler oluşturmak için kullanılan optimum parametreler. İçinde Deconvolved alırken Window, adımları 9.5.1-9.5.8 belirtilen parametreleri ayarlayın. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 4: Dispım kamera yapılandırması. (A) dispim kamera yerleşimlerinin ve oryantasyonların fotoğrafı. (B) SPIM A 'nın + 90 derece rotasyonlarının, toplanan spim B görüntülerine uyacak şekilde tasvir edilmesi. (C) dispım 'ın kamera yapılandırmasına dayalı olarak (bkz. Adım 9.5.2) oryantasyon indeksi + 1 ' e göre birimler girin. Biz spim B görüntü (ler) eşleştirmek için kayıt önce Y ekseni etrafında bir görüntü (ler) + 90 derece döndürmek. Ölçek çubukları = 10 μm. görüntüler temsili tek-görünüm, maksimum yoğunluklu projeksiyonlar ve deconvolution görüntüler 1,5-fold embriyo etiketli çekirdekler (561-Nm, kırmızı) ve nöronlar (488-Nm, yeşil). Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 5: Asetree 'de kürasyon ve düzenleme C. elegans embriyonik soy. (A) biz starrynite 's Lineage izleri düzenlemek için asetree kullanın (referanslar bakın5,6,8; el yazmaları da indirme paketi dahil). AceTree adımları tamamlanması üzerine her çekirdeği (yeşil dikdörtgen) için C. elegans sistematik adlar gösterecektir 10.1-10.2. Bu pencere (A) Lineage izlemedeki her hücre (mavi renkte vurgulanan ABA) hakkında bilgi (siyah dikdörtgen) sağlar ve lineage izlerinin izlenmesi ve düzenlenmesi sırasında kullanıcılara rehberlik eder. Kullanıcıların doğrulamak ve lineaged hücreleri ve konumlarını karşılaştırmak tavsiye edilir C. elegans embriyonik hücre sırık daha önce Sulston ve al.1 tarafından bildirilen Ayrıca, kullanıcılar belirli hücreleri bulma ilgileniyorsanız deconvolved veri serisi (aşağıya bakınız, B) arama çubuğuna C. elegans sistematik adını girin (turuncu dikdörtgen). (B) kullanıcının deconvolved veri serisi de 10.1-10.2 adımları tamamlandıktan sonra otomatik olarak açılır. Burada gösterilen bir izotropically çekirdekten kırmızı etiketli bir dört hücreli aşama embriyo görüntü erimiş. Bir çekirdekler izleme sırasında, kullanıcılar görüntünün yoğunluğunu değiştirmek gerekir (kırmızı kare) ve zaman ve z kendi klavyede ok tuşlarını kullanarak gezinmek (zaman-sol/sağ, z-yukarı/aşağı). (C) döndürülebilir 3D görselleştirme sağlayan belirli işlevleri (mor dikdörtgen) (B) timepoint 3D karikatür. Asetree ve 3D işlevselliğine genel bir bakış için bkz: Referanslar5,6,8. Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 6: diSPIM üzerinde C. elegans embriyoları zaman zaman gelişimsel dinamikleri. Üst panel, diSPIM görüntüler embriyonik gelişim ilk yarısında gösteren bir görüntülenmiş embriyo (strain BV514 ujIs11324). Embriyo, her dakika 7,5 saat (20 °C ' de) sürekli olarak görüntülenmiş. Üst panelin ilk iki görüntüsü, 4-ve 8 hücreli embriyoları çekirdeklerle (kırmızı) ve kutup organları (yoğun kırmızı küreler, mavi yıldız ile işaretlenmiş) pozisyonları ile temsil eder. Her görüntü, görüntülenmiş embriyonun tek görünümlü maksimum yoğunlukta projeksiyon temsil eder. Ölçek çubukları = 10 μm. Zaman çizelgesi (yatay çubuk), C. elegans embriyoları gelişiminin dakika sonrası FERTİLİZASYONU (m.p.f.) temsil eder. Biz diSPIM satın alma için protokolünün parametreleri görüntü embriyolar için herhangi bir algılanabilir fototoksisite teşvik etmedi doğrulanmıştır olarak viability, Hücre bölünmeleri zamanlaması, tarama ve gelişimsel kilometre taşları zamanlaması zamanlaması (referanslar bakın 1 , 25 , 26). biz gelişimsel kilometre taşları zamanlaması bizim görüntüleme parametreleri ile embriyolar arasında tekrarlanabilir olduğunu unutmayın (SEM ± 8,174 dakika 6,4 saat uzun görüntüleme oturumları için; n = 10). Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 7 ' ye. C. elegans embriyoları gelişmekte olan nörit büyüme dinamiklerinin hücresel kimlik ve tek hücreli karakterizasyonu. BV514 ujIs11324 (lineaging için) ve DCR7692 (olaex4655), bir transgenik nematod gerinim geçiş tarafından yapılan bir gerinim çift renkli görüntüleme nörofobi FLP-19 Organizatör kimliği belirsiz hücrelerin bir alt altında Gfp ifade eder. (A-H) Burada özetlenen protokolün adımlarını izleyerek, tanımlanamayan hücrelerin motor nöronlarına RMDDL ve RMDDR (sarı oklar), boşaltım kanalı hücresine (mavi oklar) ve iki kas hücresi (beyaz oklar) karşılık geldiğini tespit ettik. (I) FIJI eklentisi kullanarak rmddl ve RMDDR nöronların büyüme dinamiklerinin miktarını "basit nörit izleme" ve izotropik erimiş hacimlerin 3D reinşaatları için uygulama. Hem RMDDL hem de RMDDR 'nin stereotifik büyüme dinamiklerini nasıl göstermesini unutmayın, her biri toplam uzunluğu 11.0 ± 0.6 μm (ortalama ± SEM; n = 12 nörosit) ve sinir halkası dorsal Apex toplantı (Ayrıca bkz: film S1). Bu figürün daha büyük bir versiyonunu görmek Için lütfen tıklayınız.

Şekil 8: C. elegans embriyolarında nöronal morfolojilerin Izotropik dispim görüntülerinin incelenmesi. AVHL ve AVHR nöronların izotropik görselleştirme (sarı oklar). Dispım kullanarak, nöronal morfolojiler yaklaşık 330 nm izotropik uzamsal çözünürlüğe sahip dört boyutlu (4D) görüntüler elde yakalanabilir. Dispım kullanıcıların neredeyse tüm yönde aynı çözünürlüğe sahip görüntü birimleri döndürmek sağlar. A-D içindeki görüntüler, embriyonun uzun ekseninin çevresindeki farklı rotasyonların aynı Izotropik olarak erimiş dispım görüntü hacminin maksimum yoğunluklu projeksiyonlarına sahiptir. Ölçek çubukları = 5 μm. Bu rakam daha büyük bir sürümünü görüntülemek Için lütfen buraya tıklayın .
Ek film S1: C. elegans embriyo 280 dan 434 dakika içinde fertilizasyon sonrası gelişmekte. Gerilme DCR7692 izotropik film (olaex4655) DACR2819 seyrek RMDD nöronlar etiketleme ile ujIs113 ubiquitously ifade (şekil 7a-D, sarı oklar). DACR2819 Ayrıca iki kas hücresi (şekil 7a-d, beyaz oklar) ve atılım kanal hücresi (şekil 7a-d, mavi ok) embriyonik gelişim sırasında Etiketler (şekil 7a-d). Ölçek çubukları = 10 μm. bu dosyayı indirmek Için lütfen buraya tıklayın .
Discussion
C. elegans son pozisyonlar ve27bilinen her yetişkin nöron bağlantı ile tek organizma olarak öne çıkmaktadır. Ancak, C. elegans konektom 'i yapan çalışma devreleri ve ağlar örgütine giden gelişimsel dinamikler bilinmiyor. Işık mikroskobu gelişmeler gelen fırsatlar dayanarak, şimdi yakalamak ve hücre konumlarını analiz, morfojenler, ve C. elegans embriyonik gelişimi boyunca nörojenez.
Biz tarif ettiğimiz ve rutin olarak laboratuvarda kullandığınız prosedür, C. elegans embriyolarındaki hücre satırı için etiketli nöronların ve çekirdeklerin 4d-izotropik görüntülerini verir. Daha da önemlisi, biz Dispım ile uzun vadeli görüntüleme koşulları optimize edilmiş ve C. elegans embryogenesis analiz hızını ve hassasiyetini artırmak için yüksek çözünürlüklü görüntüleri ile yarı otomatik lineaging yetenekleri birleştiğinde. Bu tümleşik protokol, kullanıcıların hücreleri görselleştirmesine ve tanımlamasına ve erken seğirme başlangıcı ile nörit göçü ve akon fascic Bu yordam, bir ASı Dispım sistemi ile herhangi bir tesis içine kolayca uyarlanabilir ve bu sistem özellikle bu protokol için öneririz. Ticari olarak sunulan diğer SPıM formülasyonları, örnek oda ve optik özellikteki ASı konfigürasyonlarından farklı olabilir. Ancak, diğer platformlardan dışa aktarılan veriler de veri ardışık düzenimize konulabilir. Bu nedenle, lineaging kendi değerinin değerleme, görüntü kalitesi ve enstrüman stabilitesi zorlu bir test, uygulanabilir. Dispım 'ı düzenli olarak diğer numuneler (Drosophila ve zebra balığı embriyoları gibi) için aktif olarak kullansa da, embriyoların açıklanan ve kapsamlı lineaging Analizi halen nematod türlerle sınırlıdır. Daha büyük veya kalın örnekler için, örnekleri sabit bir ışık levha aracılığıyla tarayacak sahne tarama yaklaşımlarını kullanmayı tercih ediyoruz. Kumar ve ark. daha önce Dispım10ek değişiklikler olmadan kalın örneklerden yüksek kaliteli görüntüler elde etmek için bu geliştirilmiş dispım bölümleme göstermiştir.
Protokol içindeki kritik adımlar arasında C. elegans embriyoları poli-L-Lysine kaplamalı coverslip, veri edinme ve veri işleme arasında montaj bulunmaktadır. Hasat ve montaj C. elegans embriyolar cam lamel magazini deneyimsiz kullanıcılar için zor olabilir, ama burada öğrenme kolaylaştırmak için önemli adımlar ayrıntılı bir protokol sağlar. Uzun süreli görüntüleme isteniyorsa, 8-10 genç yetişkinlerden gelen dört hücreli veya daha önceki embriyolar hasat en iyi sonuçları elde28. Onlar rahim ve döllenmeyen yumurtalar eski embriyolar içeren eğilimindedir çünkü eski yetişkinler erken evre embriyo hasat için daha az arzu edilir unutmayın. Embriyo montajı açısından, monte aspiratördeki tıkanma (ağız pipeti) veya mikrokapiller pipetinde çok büyük bir açılış gibi sorunlar embriyoların uygun şekilde takılması ve oryantasyonunu önleyebilir. Optimum görüntüleme hazırlığı için, hafif levhalar, kameralar, hedefler ve otofokus performansını kontrol etmek için erken ve geç saat öncesi embriyolar üzerinde satın alma öncesi testler gerçekleştiriyoruz. Bu işlemlerin tümü test edildiğinde en iyi sonuçları elde ediyoruz ve satın alma öncesi testlerimiz sırasında yüksek kalitede görüntüler kazanıyoruz. Bu özellikle izotropik uzamsal çözünürlüğe sahip görüntüler oluşturmak için geçerlidir, her iki görünümden elde edilen ham görüntülerin (hedefler) yüksek kalitede olması gerekir. Satın aldıktan sonra, her görünüm için alınan birimler izotropik görüntüler elde etmek için işlenir. Bu protokolde açıklandığı gibi uygun bir grafik işleme birimi (GPU) kartı kullanmak önemlidir (aşağıya bakın). Bu, ısotropically erimiş görüntüleri oluşturulur, veri analizleri için zaman kısaltma işlem hızını artırır. Ayrıca, kullanıcıların en son Sitat sürümünü çalıştırmasına ve StarryNite otomatik satırlık için indirme paketimiz ile sağlanan parametreleri kullandığıyla da zorunludur. Kullanıcılar diğer örnekler (örneğin, zebrafish, Drosophila vb.) için otomatik lineaging kullanmayı ilgileniyorsanız, starrynite 'de kullanılan parametrelere ek optimizasyon gerekli olacaktır (bkz. referanslar3,4).
Entegre protokolün ön-seğirme embriyo içinde görüntü ve lineaging sonuçları sağlasa da, kullanıcılar, sonrası seğirme embriyo otomatik lineaging Şu anda mümkün değildir farkında olmalıdır: nükleer pozisyonlar saniye sırasına göre değişir seğirme sonrası embriyo, çok hızlı bir şekilde Lineage izleme izin vermek. Ancak, dispim gerçekten nörogelişimsel olayları yakalamak için umut verici bir yetenek göstermiştir ve embriyogenesis Post-twkaşıntı aşamalarında bazı hücre konumlarını izlemek23,29. Eğer kullanıcılar Post-seğirme embriyo inceleyerek ilgileniyorsanız, diSPIM hacimsel anlık görüntüler elde etmek için hız sağlar ve ince nörogelişimsel olayları izlemek, nörit büyüme gibi, hızla hareket eden embriyolar.
Bu protokol WormGUIDES Atlas30hücre-by-Cell tamamlanması için temel olacak, tanımlamak ve sırasında etiketli nöronların 3D morfolojileri yakalamak için yüksek çözünürlüklü izotropik görüntüleri ile entegre bir yaklaşım sağlayacaktır embriyogenesis ilk 430 dakika. Olduğu gibi, prototip WormGUIDES Atlas gelişmekte olan embriyo hücrelerin nükleer pozisyonlar sağlar ve embriyonik nöronların bir alt kümesinin gelişimsel dinamiklerini yakalamak amaçlamaktadır. Bu protokol WormGUIDES Atlas30içine ek gelişmekte olan nöronların entegrasyonu için bir anahtar olacaktır.
Entegre protokollerimiz C. elegans embryo 'da yeni gen ifade profillerini keşfetmeyi de basitleştirecek. Transjenik C. elegans, birçok hücreye özgü Rehberleri dağınık ve geçici olarak transgen ifade kontrol. Çoğu genlerin ifade desenleri yaygın yetişkin hayvan31,32,33,34, neredeyse tüm henüz gelişmekte olan karakterize edilmelidir karakterize edilmiştir iken (özellikle geç aşama) embriyo. C. elegans promoterome hücre spesifik transgen ifade götürmek için solucan topluluğuna yararlı bir kaynak olmuştur, yanı sıra gen işlevi hücre otonom veya otonom olmayan olup olmadığını belirlemek. Genlerin izotropik yüksek çözünürlüklü ve dinamik ifade desenleri yakalama ve tam olarak lineaging yoluyla ifade hücreleri tanımlayan bilimsel toplumda birçok değerli olacaktır.
Embriyogenesis iki iç içe geçmiş büyük süreçler, hücresel farklılaşma ve doku morfojeninden oluşur. C. elegansgelişimi sırasında farklı hücre türlerini tanımlayan mekanizmalar ve moleküller hakkında büyük bir anlaşma bilinmektedir. Ancak, küçük mekanizma hücre göç, hücre yapışma için önemli ve C. elegans embryo hücre şekli hakkında bilinmektedir. Bilinen C. elegans sabit hücre sırtı ile, bizim protokol bize kolayca detay yeni düzeylerde morfojenler sırasında embriyonun kataloglu 3D-mikroanatomisini ayırt sağlar: Örneğin, Axon fasciculation, synaptogenesis, ve nöronal aktivite. Ardiel ve al. daha önce C. elegans embriyolar23tek bir nöronların düzeyinde kalsiyum geçiciler yakalamak için dispım gücünü göstermiştir. Gelişimsel fizyolojisinin diğer birçok yönü bu yöntemlerle soruşturma için olgunlaşmış durumdadır.
Son olarak, bu protokol büyük ölçüde otomatiktir ve sistematik deconvolution görüntüleri oluşturmak ve StarryNite ve Asetree üzerinden hücre-lineaging gerçekleştirmek için gereken süreyi azaltır. Bu protokolde kullanılan yazılım stratejileri, biyolojinin pek çok soruya, onları burada gösterdiği çok spesifik alanlardan uzak bir şekilde uygulanabilmektedir.
Yazılım uyumluluğu ve indirme erişimi hakkında ayrıntılar
Micro-Manager ve Dispım görüntüleme için eklentiler hakkında bilgi http://dispim.org/software/micro-manager ve https://micro-manager.org/wiki/ASIdiSPIM_Plugin adresinde mevcuttur.
Veri işleme ardışık düzeni şu anda bir Windows işletim sistemi gerektirir. Gerekli tüm veri işleme programlarının ve destek dosyalarının yüklenmesini kolaylaştırmak için tek bir arşiv dosyası paketlemiştir. Http://dispimlineage.wormguides.org adresinde indirilebilir.
Sitshow (http://run.cytoshow.org/) yaygın olarak kullanılan ve açık kaynak görüntü analiz platformu, ımagej (v1) dayanmaktadır. Java 'nın bilgisayarda yüklü olması ve Sitat gösterisi kullanmak için güncel olması ve Sitat güncellemeleri Java Web Start yoluyla otomatik olarak dağıtılmalıdır. Birçok ımagej tabanlı işlevleri Sitshow olarak açıklandığı ve https://imagej.nih.gov/ij/docs/examples/index.html adresinde gösterilmiştir. Sitshow, ASı Dispım 'ın yanı sıra TIFF çıkışı oluşturan diğer görüntüleme yazılımından çok boyutlu ham verileri görüntülemek için özelleştirilmiştir. Prensip olarak, diğer Multi-View SPıM görüntüleme sistemleri bu protokolün farklı mikroskop sistemlerinde gerçekleştirilmesine izin vermek için Sitat 'ın küçük değişiklikleriyle desteklenebilir.
SpimFusion CUDA/C++ CUDA Toolkit v 7.5 ile Visual Studio 2013 kullanarak yazılmıştır. SpimFusion çalışan belirli bir bilgisayar donanımı gerektirir: bir NVıDıA grafik işlemci birimi (GPU) kartı CUDA işlem yeteneği 1,0 veya üzeri ve en az 2 GB grafik kartı belleği. Protokolün yayınlanması sırasında, SpimFusion yayınlanmamış (min Guo ve Hari Shroff) ama yukarıda bahsedilen yazılım paketi arşivinde mevcuttur.
StarryNite özel olarak oluşturulmuş bir komut satırı tabanlı sürümü serbestçe kullanılabilir MATLAB derleyici çalışma zamanı yüklü olduğunu gerektirir, ancak ticari MATLAB yazılımı için bir lisans gerektirmez. MATLAB derleyici çalışma zamanı yukarıda belirtilen yazılım paketi arşivinde bulunur. Bu protokolde kullanılan StarryNite için kod aslında Konfokal görüntüler6için kullanılan değişmeden. Ancak, starrynite işleme ve starrynite sonuçlarının işlenmesi için giriş görüntüleri yaratılmasında birkaç operasyonel konularda burada sitroshow içinde yöntemleri tarafından ele alınmıştır sürekli veri işleme boru hattı için erimiş izotropik dispım etkinleştirmek Birim. Bu değişiklikler, bu pre-ve post-processing adımları işleyen Sitam kodu tarafından otomatiktir. Sitlet Ayrıca, bölümlendirme algoritmasını otomatik olarak görüntülenmiş verilerdeki çekirdeklerin floresan yoğunluğuna ayarlamak için önceden optimize edilmiş Dispım özel şablonu StarryNite parametresini düzenler. Her Dispım veri kümesinde StarryNite tarafından kullanılan benzersiz parametreler sonra çıktı görüntü ve lineaging veri ile birlikte bir dosyaya kaydedilir.
16-bit görüntüler ile çalışır ve Java3D işleme ile uyumluluğu korur Asetree özel bir sürümü en iyi bu protokol için uygundur. Ayrıca yukarıda belirtilen yazılım paketi arşivinde yer almaktadır.
Disclosures
Yazarların ifşa etmesi gereken hiçbir şey yok.
Acknowledgments
Biz entegre strain için John Murray teşekkür ederiz, ujIs113, lineaging gerinim BV514 oluşturmak için; Brandon Harvey (NıBıB) protokolü test etmek için yardım; Jon Daniels ve Gary Rondeau (uygulamalı bilimsel enstrümantasyon) Micro-Manager ve diSPIM enstrüman ile yardım için; ve Andrew York ve Hank eden diSPIM sistemi üzerinde kritik geribildirim için. Ayrıca bir toplantı ve beyin fırtınası platformu sağlamak için azınlık kurumları Araştırma Merkezi programı ve Instituto de Neurobiología Jose del Castillo (Universidad de Puerto Rico) için teşekkür ederiz. Bu çalışmanın çoğu Whitman programı aracılığıyla Woods Hole 'daki deniz biyolojik Laboratuvarı 'nda yapılmıştır. Bu çalışma NıH Ulusal Biyomedikal görüntüleme ve Biyomühendislik Enstitüsü Intramural araştırma programları ve NıH Grant No tarafından destekleniyordu. U01-HD075602 ve hayır. R24-OD016474. Mark W. Moyle, F32-NS098616 ve Leighton H. Duncan tarafından desteklenen bir çeşitlilik Supplement tarafından R24-OD016474 tarafından destekleniyordu.
Materials
| Name | Company | Catalog Number | Comments |
| Steps 1-4 | |||
| Concavity slides | ThermoFisher Scientific | 1519006 | 5-18mm diameter, 0.6-0.8mm deep, 1.2-1.5mm |
| Dissecting microscope with 10×–50× zoom range | Motic | SMZ-171 | |
| E. coli (OP50) | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Glass coverslips, no. 1.5, 24 × 50 mm | VWR International | 48393-241 | |
| M9 Buffer | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Methyl cellulose | Sigma-Aldrich | H7509-25G | |
| Microcapillary pipette aspirator tube | Sigma-Aldrich | A5177 | |
| Microcapillary pipettes, 0.4-mm i.d | Drummond Scientific | 1-000-800 | |
| Needle, no. 18G x 1 ½ (1.2mm x 40mm) | BD Precision Glide | 305196 | |
| NGM plates | prepared as described by Brenner (1974) | ||
| O-ring for imaging chamber | O-Rings West | M1.5X40 | |
| Pasteur pipette | Corning/Sigma-Aldrich | CLS7095D5X | |
| Platinum wire, 0.5-mm diameter | Sigma-Aldrich | 267201 | |
| Poly-L-lysine | Sigma-Aldrich | P1524 | |
| Stainless steel rectangular chamber (76.0 mm x 50.5 mm) | Applied Scientifics Instrumentations (ASI) | I2450 | |
| Worm Eyelash Pick | Hart, A. C. Behavior. WormBook. (2006). | ||
| Worm Pick | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Name | Company | Catalog Number | Comments |
| Steps 5-6 | |||
| 488 nm long-pass filter | Semrock | LP02-488 RU-2 | |
| 561-nm notch filter | Semrock | NF03-561E-25 | |
| BLP02-561R-25, quantity 2 | Semrock | 561 nm EdgeBasic best-value long-pass edge filter | |
| Control software for bottom camera | Jenoptik | ProgRes CapturePro | |
| diSIPM assembly video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/TAgbr6IrTqw ; http://www.asiimaging.com | |
| diSPIM alignment video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/qnOrg30NNuE | |
| diSPIM imaging PC | Intel | Intel Xeon CPU E5-2630 2.6GHz, 12 cores in total, 64 GB memory, Windows 7 | |
| FF01-525/45-25, quantity 2 | Semrock | 525/45 nm BrightLine single-band bandpass filter | |
| FF555-DI03-25X36, quantity 2 | Semrock | 555 nm edge BrightLine single-edge dichroic beamsplitter | |
| Imaging PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Kumar et al diSPIM Setup | Applied Scientifics Instrumentations (ASI) | Instrument setup for this protocol is identical to Kumar et al 10,11 diSPIM, which makes use of 40x 0.8NA water immersion lenses for imaging. (See steps 5.1 and note) | |
| Micro Manager | Micro-Manager | https://micro-manager.org/ | |
| Modifications to Kumar et al diSPIM Setup (see below) | |||
| Optical table with isolators, 4 feet × 6 feet × 12 inches | TMC | 784-651-02DR and 14-416-34 | |
| Name | Company | Catalog Number | Comments |
| Steps 7-10 | |||
| Analysis PC | Intel | Intel Core i7-8700K CPU 3.70GHz, 6 cores in total, 64 GB memory, Windows 10 | |
| Analysis PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Installation instructions | Software bundle | http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm | |
| Software bundle | Software bundle | http://dispimlineage.wormguides.org |
References
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology. 100 (1), 64-119 (1983).
- Bao, Z., et al. Automated cell lineage tracing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 103 (8), 2707-2712 (2006).
- Santella, A., Du, Z., Bao, Z. A semi-local neighborhood-based framework for probabilistic cell lineage tracing. BMC Bioinformatics. 15, 217 (2014).
- Santella, A., Du, Z., Nowotschin, S., Hadjantonakis, A. K., Bao, Z. A hybrid blob-slice model for accurate and efficient detection of fluorescence labeled nuclei in 3D. BMC Bioinformatics. 11, 580 (2010).
- Boyle, T. J., Bao, Z., Murray, J. I., Araya, C. L., Waterston, R. H. AceTree: a tool for visual analysis of Caenorhabditis elegans embryogenesis. BMC Bioinformatics. 7, 275 (2006).
- Katzman, B., Tang, D., Santella, A., Bao, Z. AceTree: a major update and case study in the long term maintenance of open-source scientific software. BMC Bioinformatics. 19 (1), 121 (2018).
- Murray, J. I., et al. Automated analysis of embryonic gene expression with cellular resolution in C. elegans. Nature Methods. 5 (8), 703-709 (2008).
- Murray, J. I., Bao, Z., Boyle, T. J., Waterston, R. H. The lineaging of fluorescently-labeled Caenorhabditis elegans embryos with StarryNite and AceTree. Nature Protocols. 1 (3), 1468-1476 (2006).
- Wu, Y., et al. Spatially isotropic four-dimensional imaging with dual-view plane illumination microscopy. Nature Biotechnology. 31 (11), 1032-1038 (2013).
- Kumar, A., et al. Using Stage- and Slit-Scanning to Improve Contrast and Optical Sectioning in Dual-View Inverted Light Sheet Microscopy (diSPIM). The Biological Bulletin. 231 (1), 26-39 (2016).
- Kumar, A., et al. Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nature Protocols. 9 (11), 2555-2573 (2014).
- Wu, Y., Christensen, R., Colon-Ramos, D., Shroff, H. Advanced optical imaging techniques for neurodevelopment. Current Opinion in Neurobiology. 23 (6), 1090-1097 (2013).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17708-17713 (2011).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136 (12), 1963-1975 (2009).
- Stelzer, E. H. Light-sheet fluorescence microscopy for quantitative biology. Nature Methods. 12 (1), 23-26 (2015).
- Winter, P. W., Shroff, H. Faster fluorescence microscopy: advances in high speed biological imaging. Current Opinion in Chemical Biology. 20, 46-53 (2014).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. Astronomical Journal. 76 (6), 745-754 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. JOSA. 62 (1), 55-59 (1972).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Hart, A. C. Behavior. WormBook. , (2006).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. Chapter 14, Unit14 20 (2010).
- Gualda, E. J., et al. SPIM-fluid: open source light-sheet based platform for high-throughput imaging. Biomedical Optics Express. 6 (11), 4447-4456 (2015).
- Ardiel, E. L., et al. Visualizing Calcium Flux in Freely Moving Nematode Embryos. Biophysical Journal. 112 (9), 1975-1983 (2017).
- Walton, T., et al. The Bicoid class homeodomain factors ceh-36/OTX and unc-30/PITX cooperate in C. elegans embryonic progenitor cells to regulate robust development. PLoS Genetics. 11 (3), e1005003 (2015).
- Altun, Z. F. WormAtlas. , (2002).
- Wood, W. B. Embryology: In the nematode C. elegans. Cold Spring Harbor Laboratory Press. Chapter 8, 215-241 (1988).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314 (1165), (1986).
- Bao, Z., Murray, J. I. Mounting Caenorhabditis elegans embryos for live imaging of embryogenesis. Cold Spring Harb Protoc. (9), (2011).
- Christensen, R. P., et al. Untwisting the Caenorhabditis elegans embryo. eLife. 4, (2015).
- Santella, A., et al. WormGUIDES: an interactive single cell developmental atlas and tool for collaborative multidimensional data exploration. BMC Bioinformatics. 16, 189 (2015).
- Dupuy, D., et al. A first version of the Caenorhabditis elegans Promoterome. Genome Research. 14 (10B), 2169-2175 (2004).
- Reece-Hoyes, J. S., et al. Insight into transcription factor gene duplication from Caenorhabditis elegans Promoterome-driven expression patterns. BMC Genomics. 8 (27), (2007).
- WormBase. , Available from: https://www.wormbase.org (2019).
- Lee, R. Y. N., et al. WormBase 2017: molting into a new stage. Nucleic Acids Research. 46 (D1), D869-D874 (2018).



