Summary
Här presenterar vi en kombinatorisk metod med hög upplöst mikroskopi, beräknings verktyg, och Single-cell märkning i levande C. elegans embryon att förstå encellig dynamik under neurodevelopment.
Abstract
Auktor (C. elegans) sticker ut som den enda organism där utmaningen att förstå cellulära ursprunget till ett helt nerv system kan observeras, med en enda cell upplösning, in vivo. Här presenterar vi ett integrerat protokoll för undersökning av neuroutvecklingssjukdomar i C. elegans embryon. Vårt protokoll kombinerar avbildning, lineaging och neuroanatomisk spårning av enstaka celler i utveckling av embryon. Vi uppnår långsiktig, fyrdimensionell (4D) avbildning av levande C. elegans embryon med nästan ISO tropiskt rumslig upplösning genom användning av Dual-View inverterad selektiv plan belysning Microscopy (dispim). Kärnor och neuronala strukturer i tallvedsnematoden embryon avbildas och isotropically smält för att ge bilder med upplösning på ~ 330 nm i alla tre dimensioner. Dessa minut-för-minut högupplösta 4D data uppsättningar analyseras sedan för att korrelera definitiva cell-härstamning identiteter med gen uttryck och morfologisk dynamik på encelliga och subcellulära nivåer av detaljer. Vårt protokoll är uppbyggt för att möjliggöra modulimplementering av var och en av de beskrivna stegen och förbättra studier av embryogenes, gen uttryck eller neuroutveckling.
Introduction
C. elegans sticker ut som den enda organism där varje cell i embryot kan observeras under hela neurodevelopment. Med hela cellen-härstamning kända och invariant1, och med utvecklingen av nya verktyg som tillåter märkning och kontinuerlig avbildning av enstaka celler i embryon, biologer kan nu börja undersöka olika steg i utvecklingen av tallvedsnematoden nervös systemet från alla vinklar-cell födseln; migration och differentiering. neurite formation, riktad utväxt och fasciympning; synapsen bildas; och tuning av funktionella kretsar. Fånga neuronala utväxt dynamik i C. elegans embryo, genom att kombinera stabilt uttryckta reportrar och fluorescensmikroskopi, är värdefull för det vetenskapliga samfundet.
Utvecklings studier i C. elegans utnyttja ofta invariant cell-härstamning och cell-öde kartor av denna art att öka kontextuella förståelse på encellig nivå inom intakt organism1. Auto-lineaging analys-med starrynite2,3,4 och acetree5,6,7,8 program vara-fördelar från hög kontrast, hög upplösning bilder av fluorescerande kärnor. För att fungera optimalt beror StarryNite/AceTree också på förutsägbar begränsad orientering av avbildade embryon under utveckling. Konfokalmikroskopi, gjort i C. elegans embryon komprimerade mellan två täckglas, har varit standard auto-lineaging mikroskopi metod för mer än ett decennium eftersom det ger både hög kontrast/hög upplösning och en förutsägbar begränsad inriktningen på embryot7,8. Vi beskrev tidigare konstruktion och användning av en ny ljusbladsbaserad Dual-View inverterad selektiv plan belysning Mikroskop (dispim) för levande prov avbildning såsom C. elegans embryogenes9,10 , 11 för att , 12 av de , 13. lätt-blad mikroskopi, i allmänhet, ger låg fototoxicitet, hög hastighet, och långsiktig avbildning av levande 3D-prover14,15. Den diSPIM metoden, specifikt, producerar fyrdimensionella (4D) bilder med nästan ISO tropiskt spatial upplösning på cirka 330 nm9.
Jämfört med konfokalmikroskopi, erbjuder diSPIM högre signal-till-brus och hastighet, mer isotropisk rumslig upplösning, och är mer lämplig för långsiktig in vivo Imaging16. Vi arbetade därför för att anpassa diSPIM data för input till StarryNite/AceTree och undersökte om detta skulle förbättra lineaging analyserna. Ett stort hinder är att diSPIM prover inte lätt kan begränsas av Eggshell-kompression att anta förväntade rikt linjer för StarryNite/AceTree. Slumpmässig orientering av cell positioner i den volym som analyseras försämrar noggrannheten i automatisk lineaging analys.
Vi använde därför CytoSHOW, ett tittarstyrt användar gränssnitt som tillåter användare att välja exakt 3D-orientering av embryon under förbehandling av diSPIM bilder, vilket ger bilddata som är både kvalitetsoptimerade och kontext medveten för input till StarryNite /AceTree. Vid användar val av avbildade embryon samordnar CytoSHOW en automatiserad data bearbetning pipeline. Beskärda och bakgrundssubtraperade embryonala bilder sparas i TIFF-stackfiler för varje position, tids punkt och vy. Cytoshow sedan iterativt anropar programmet spimfusion att samregistrera och gemensamt deconvolve de två pre-bearbetade vyer, med Richardson-Lucy17,18 algoritm för att ge ISO tropiskt högupplösta volymetriska bilder. En diSPIM-specifik uppsättning parametrar har optimerats för StarryNite att styra sitt beteende under bild-segmentering och Nucleus-tracking i isotropically smält bilder. Brända bilder och lineaging resultat sedan redige ras med hjälp av AceTree, som tillåter användare att identifiera och åtgärda eventuella fel i Auto-Lineage spår som genereras av StarryNite. AceTree kan också presentera härstamning träd och 3D modelleras renderingar av spårade kärnor i embryot. Vi tycker att automatisk lineaging hastighet och noggrannhet är markant förstärkt med isotropically smält bilder, jämfört med RAW-bilder från antingen SPIM kamera. Vårt protokoll, medan optimerad för C. elegans program som beskrivs här, kan vara allmänt anpassade för Auto-lineaging av dispim data som produceras för andra arter eller prover. Om detta är den avsedda användningen av protokollet, Observera att ytterligare justering av de starrynite parametrarna kommer sannolikt att krävas för nya exemplar, enligt beskrivningen3,4.
Framgångs rikt genomförande av detta protokoll resulterar i bilder med 4D-isotropisk upplösning och gör det möjligt för biologer att spåra cell linjer, samtidigt identifiera och analysera nerv celler i utvecklingen C. elegans embryo. Dessutom, genom sammanslagning av flera efter bearbetning algoritmer-med hård varu acceleration är den mest tids krävande av dessa-vi kan nu analysera både fina subcellulära detaljer och cell-lineages och cell-öden av levande embryon i huvudsak real tid. Detta nya protokoll möjliggör exakt, informerad manipulation och observation av cellers beteende under probativa studier av differentiering och morfogenes in vivo. I detta manuskript, presenterar vi en detaljerad förklaring av de förbättrade protokoll vi har utvecklat för lineaging och cell spårning i utvecklingen C. elegans embryon, för att förbättra studier av embryogenes, gen uttryck eller neurodevelopment.
Protocol
1. montering av diSPIM stål bild kammaren med Poly-L-Lysin belagd täckslip
Obs: stegen nedan är alla krävs för att optimera och automatisera härstamning analys av tallvedsnematoden embryon av StarryNite/acetree. Flera alternativ (anges av som sådana) kan utelämnas för experiment som kräver spårning av C. elegans cell linjer.
- Rita en liten rektangel (2mm x 5mm) i mitten av en ren rektangulär täckslip (nr 1,5, 24 mm x 50 mm) med en Sharpie (eller liknande penna).
- Vänd täckglasen över till den omärkta sidan och placera 10 μL Poly-L-Lysin (katt. nej. P1524) över den markerade rektangeln.
Anmärkning: gör en arbets koncentration på 1 mg/mL Poly-L-Lysin upplöst i filtrerat vatten (eller motsvarande). För långtids förvaring, Bered 5 – 10 mL-portioner och förvara vid − 20 ° c. Efter upptinat kan Ali kvoten förvaras i rums temperatur (23 ° c) i 3 – 4 veckor. - Låt Poly-L-Lysin att belägga täckglasen i 5 minuter (figur 1a). Poly-L-Lysin används för beläggning glas täckglasen där embryona kommer att monteras, så att embryot äggskal att hålla fast vid täckglasen, även när målen är nedsänkt i Imaging mediet.
- Placera Poly-L-Lysin belagda täckglasen i den nedre halvan av stål bild kammaren.
- Placera den övre halvan av stål bild kammaren på den nedre halvan med täckglasen och dra åt med de fyra skruvarna som är förknippade med kammaren. Kontrol lera visuellt från sidan för att se till att den övre halvan är jämnt sittande i den nedre halvan (figur 1b).
- Fyll kammaren med 7 – 8 mL M9 buffert19, en isotonisk buffert som hjälper till att förhindra att embryon från tidiga stadier duger till onormalt osmotiskt tryck. 1-cells, och även 2-cells embryon är osmotiskt känsliga och kan utvecklas onormalt om inte i denna Isotoniska buffert. Embryon tenderar också att gripa på 3-faldigt stadium om M9 ersätts med vatten.
2. beredning av C. elegans embryoprover för montering
Obs: cirka 18 timmar före avbildning, fem unga (1 dag sedan sista molt) vuxna och tio larv steg 4 (L4) C. elegans plockas till en tallvedsnematoden tillväxt medium (NGM) agar plattan seedade med E. coli stam OP50. Platina tråd plockar används för att flytta larver och unga vuxna C. elegans utan att skada djuret19.
- Förbered en 1% metylcellulosa (katt. nej. H7509-25G) lösning i M9 buffert.
Anmärkning: metylcellulosa måste rörs om i varmt M9 tills den löses upp. När lösningen är färdiglagad kan den förvaras i rums temperatur. - Tillsätt 500 μL 1% metylcellulosa-in-M9-lösning till depressionen hos en konkav Mikroskop bild. Denna viskösa buffert kommer att användas vid två tillfällen: 1) vid skörd av tidiga embryon genom dissektion av vuxna maskar och 2) vid tvättning sent stadium embryon plockas direkt från en NGM tallrik.
Anmärkning: metylcellulosa används för att förhindra att embryon fastnar på Mikroskop glaset. - För avbildning sent skede embryon, plocka som C. elegans embryon (helst från en NGM tallrik med unga vuxna närvarande) med hjälp av en ögonfrans plocka, och flytta embryon till 1% metylcellulosa på den konkava Mikroskop bilden. Frans plockningen hjälper till att reducera kraften och minimerar därmed stress eller skador på embryona under hanteringen. Förfarandet för att göra en ögonfrans plocka täcks av Hart20.
- Med en andra ögonfrans plocka (i motsatt hand), försiktigt knacka båda ögonfransarna tillsammans för att avbryta embryon i metylcellulosa.
- Valfritt: om planerar att härstamning embryonala celler med StarryNite, måste man montera 1-cells till 4-cells embryon. För att göra det, Välj först unga vuxna från en NGM tallrik och flytta dem till M9-metyl cellulosa lösningen på den konkava Mikroskop bilden med hjälp av en platina tråd plocka.
- Tillval: med de slipade spetsar av injektions nålar (nr 18g x 1 1/2), skiva djuret tvären vid mitten kroppen att släppa 1-cell till 4-cells embryon.
3. Munpipetten: montering av sug röret med mikrokapillärpipett
Obs: vi använder ett sug rör med en handdragen mikrokapillärpipett insatt i gummi packningen av röret. Detta gör det möjligt för oss att överföra embryon från dissektion glida till Poly-L-Lysin-belagda ytan i buffertfylld Imaging kammaren.
- Dra manuellt mikrokapillärpipetten över en öppen låga för att skapa två halvor med sträckta spetsar.
- Ta ena halvan av mikrokapillärpipetten och för in den trubbiga änden i sug rörets gummi packning (figur 1c). Ställ den andra halvan av mikrokapillärpipetten åt sidan för användning senare (vid behov).
- Med den monterade Nässug röret försedd med microcapillary pipett, försiktigt bryta spetsen på microcapillary pipetten och skapa en öppning som kommer att passa ca 1-2 två embryon (härifrån på detta instrument kallas en "munpipett").
4. montering C. elegans embryon på Poly-L-Lysin belagda täckglasen
- Med Nässug munstycket hålls försiktigt mellan tänderna, pre-Fyll microcapillary pipetten med 10-15 μl av M9 buffert och sedan försiktigt suga flera embryon från konkava glida in i kapillär.
- Överför embryona till stål Imaging kammaren fylld med M9 buffert, positionering kapillärspetsen så att embryon kommer att falla i den centrala rektangeln av täckglasen.
- Undvika skador på embryon, försiktigt flytta dem med en ögonfrans plocka eller använda munpipetten att placera embryona vertikalt, att orientera embryona så att den långa axeln av embryot är vinkel rät mot den långa axeln av täckglasen (figur 1b infälld , botten panel).
Anmärkning: placering av embryot i denna riktning minimerar antalet skivor till bild, vilket minskar ljus dosering och data behandlings tid samtidigt förbättra förvärvs hastighet. - Placera stål bild kammaren i prov hållaren på Mikroskop stadiet (figur 1d).
5. montering, mjukvaru installation och laser optimering för embryonal avbildning med hjälp av diSPIM
- Se steg-för-steg-instruktioner om hur man monterar hela fiberkopplade diSPIM från kommersiellt tillgängliga delar i Kumar et al.10,11 och vid http://www.dispim.org. En video-protokoll om hur man monterar diSPIM finns också på ASI webbplats (http://www.asiimaging.com).
Anmärkning: instrument inställning för detta protokoll är identisk med Kumar et al.10,11 dispim, som använder sig av 40x 0.8 na vatten nedsänkning linser för avbildning. Den enda skillnaden mellan inställningen i detta protokoll och Kumar et al.10,11 är tillägget av en Dichroic spegel (uppdelning på 560 nm) och röda och gröna bandpass filter inuti en avbildning uppdelning enhet (modell A12801-01) installeras på båda diSPIM Imaging Arms. Tillsatsen av bilddelningsoptik gör det möjligt att samtidigt fånga bilder från två distinkta fluoroforer – upphetsad av 561 nm och 488 nm – genom att separera emissions banden till två halvor av samma kamerachip. - Efter instrument montering, kontrol lera den optiska justeringen av diSPIM före avbildning.
Anmärkning: för att korrekt justera diSPIM se https://youtu.be/qnOrg30NNuE, och för maskin varu information, http://dispim.org/hardware/objectives och http://www.asiimaging.com. - Använd Open-Source Platform Micro-Manager (https://micro-manager.org/)21, som har optimerats för användning av Ljusplåt Mikroskop för hög genom strömning cellulär avbildning22. Vi rekommenderar användning av ASI diSPIM plugin för multi-position förvärv, vilket gör samtidig avbildning av upp till 30 embryon som beskrivs23.
- Med Micro-Manager öppen, ställa in laserintensiteter till ~ 179 μW (0,5) för 488 nm och ~ 79 μW (0,25) för 561 nm (figur 2A, röd rektangel).
Obs: dessa är rekommenderade inställningar för långsiktig avbildning av C. elegans embryon med 1-minutersintervaller. Under lång sikt Dual-Color Imaging, den 561 nm Laser används för att bild kärnor (Mcherry:: Histone) tills embryon är på bönan skede, vid vilken punkt 488 nm lasern är sedan slå på för att också bilden GFP-märkta nerv celler. Dessa bild förhållanden är optimerade för att minimera fototoxicitet och säkerställa överlevnad och kläckning av embryona samtidigt möjliggöra långvarig (12-14 timmar) kontinuerligt förvärv av nerv systemets utveckling och lineaging data. - I Micro-Manager, Välj Meny Plugins ≫ enhets kontroll > ASI DISPIM för att öppna fönstret ASI dispim (figur 2b). Välj fliken Anskaffning . I den här flikens data besparings inställningar avsnitt (grön rektangel), volym inställningar avsnitt (blå fyrkant), och segment inställningar avsnitt (orange kvadrat), se till att varje parameter är inställd som visas i figur 2b.
Obs: vår bild analys program varan CytoSHOW är anpassad för att fungera med andra valfria output data format såsom bulk-sammanforade OME-TIFF-filserier och TIFF-stack fil serien skapas efter förvärvet genom användning av en export funktion inbyggd i Micro-Manager. Typiskt, den enkel-tid-punkt-stack sorterade ome-TIFF arkivera datan formaten är Använd emedan den tillåt verklig-tid betraktande och bearbetningen av bild volym så snart den rå datan är förvärvat.
6. optimerad autofokus parametrar för långsiktig avbildning av C. elegans embryon
- Ställ in Micro-Manager autofokus parametrar till de inställningar som optimerats för långsiktig härstamning-kvalitet diSPIM-Imaging av C. elegans embryon. Klicka på fliken autofokus (figur 2C) i fönstret ASI dispim. I avsnittet allmänna alternativ för autofokus (svart fyrkant) anger du parametrarna exakt som de visas. Observera att Autofokuskanalen (Röda torget) bör specificera din nukleär kanal fluorescenskanal i lineaging experiment.
Obs: om max offset är större än 5 μm, tenderar bilderna att glida ur fokus. - Klicka, Plugins ≫ förvärv verktyg > mönster overlay.
- Klicka på Visa rutnäti fönstret mönster överlägg .
- Klicka på navigeringsfliken i fönstret ASI dispim .
- Klicka på kryss rutor för balk och ark med bana A eller B och klicka sedan på Live. Bild hämtningen inleds. Ett Live View-fönster öppnas. Välj området autofokus analys av embryot genom att rita en ruta runt embryot på den valda kanalen från 6,1.
Obs: vi fångar vanligt vis 420 tid punkter för 10 embryon per bildsession. Rå data per bildsession är vanligt vis 1,7 TB, medan deconvolved och StarryNite-bearbetade data är 1,4 TB (se steg 9 och 10). Vi rekommenderar att du använder hård disk med stor kapacitet (18 TB på vårt nuvarande system) för bild hämtning och molnplattformar för bild lagring. - Klicka på starta förvärv på fliken "förvärv " för att påbörja långsiktig flerdimensionell bild fångst (figur 2b).
7. Öppna RAW Micro-Manager bilder i CytoSHOW
- Ladda ner program varu paketet från http://dispimlineage.wormguides.org.
Obs: mjukvaru paketet kommer att laddas ner som en. zip-fil och måste extraheras till "C:\" Katalog före användning. Ytterligare detaljer för installation ges på http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm. - Dubbelklicka på filen C:\cytoshowextrasforc\cytoshow_app.jnlp för att börja köra cytoshow.
- Välj Meny fil > nya > Dispim Monitor (Micro-Manager). Leta upp mappen med rot data uppsättningen där tidspunktsmapparna för anskaffning sparades. Välj en tidpunkten mapp och klicka på Öppna. Flerdimensionella navigations fönster (kallas Dispim Monitor fönster) öppnas automatiskt för både spima och Spimb (figur 3a).
Obs: dessa fönster kommer att övervaka rotmappen data för nyligen sparade rå tidpunkten stackar (i händelse av att ett exemplar fortfarande registreras). Efter varje ny tidpunkten förvärvas, varje fönster övervakning av distinkta spim armar och prov positioner kommer att uppdatera för att visa hela flerkanals 4D dataset för varje embryo.
8. generera Max projektionsbilder med CytoSHOW
Även före avfaltningen kan rå data snabbt bearbetas för att utvärdera preparatets globala funktioner.
- Klicka på Z-MIP- knappen på den vänstra sido panelen i bild fönstret (figur 3a, röd rektangel) för att göra maximal intensitet prognoser genom hela djup och heltid kurs i en given position eller spim arm. Ett fönster med hyperstack för Z-projektion visas.
- Ställ in projektionstyp till Max-intensiteti fönstret Z-Projection hyperstack . Ange vilka kanaler, segment och tids punkt ramar som ska bearbetas baserat på användar inställningar.
- Klicka på OK när du är klar.
- Välj mappens plats för att spara Max intensitet utdata från dialog rutan fil och klicka sedan på OK. Låt viss tid (15-20 minuter, beroende på storleken på dataset och processor kraft på datorn) för CytoSHOW att generera projektionsbilder.
9. analysera cell linjer i isotropiska högupplösta volymetriska data
- Tillval: med rå data öppna via Dispim Monitor i cytoshow, Välj polygonmarkerings verktyget (figur 3a, svarta pilar) och klicka precis utanför embryots främre, bakre, dorsala och ventrala kanter (i exakt samma ordning) för att skapa ett "Bowtie" mönster över embryot. Gör för båda vyerna (SPIM-A och SPIM-B, figur 3a).
Anmärkning: detta val anger den elliptiska regionen av intresse (ROI) där embryot är centrerad och registrerar den främre-bakre axeln av embryot. Den Bowtie mönster signaler CytoSHOW att användaren planerar att ytterligare specificera en exakt rotation av de slutliga isotropically smält volymer till en orientering som är optimal för lineaging analyser av StarryNite/AceTree. I de fall där StarryNite lineaging inte ingår i försöks planen kan andra markerings verktyg och former väljas för att ställa in ROI för bild behandling. - Om flera embryon samtidigt avbildas med hjälp av alternativet för förvärv av flera positioner, öppna och utför steg 9,1 för alla embryon. Detta kommer att möjliggöra parallella genomförande av framtida åtgärder för alla embryon i en session. Stäng SIMA-och SPIMB-Fönstren för alla embryon som du inte vill bearbeta.
- Klicka på knappen dispim på den vänstra sido panelen i fönstret dispim Monitor (figur 3a, markerad med gult). Detta avslöjar en subpanel av kontroller som är specifika för diSPIM bearbetning.
- Justera gröna och röda kanaler för varje SPIM-arm. Eftersom emissions kanal delare används för att fånga distinkta röda och gröna bilder samtidigt på samma kamera, är det viktigt att visuellt anpassa den exakta pixel registreringen av dessa två fysiskt intilliggande bildfält när de är överliggande. Åter användning av samma justerings justeringar är vanligt vis möjligt under många på varandra följande bildsessioner, men bör kontrol leras (som i steg 9.4.1 – 9.4.5).
- Börja med SPIMA panelen, Välj den röda kanalen genom att flytta cm scroll bar till vänster (figur 3a, orange pil, vänster panel).
- Använd x-, y-och z-justerarna (figur 3a, orange fyrkant) och flytta den röda kanalen så att den matchar greenen.
- Klicka på knappen Dispim (figur 3a, markerad med gult), för att stänga subpanelen och utlösa spridning av samma SKIFT till alla andra positions fönster.
- Bekräfta att korrekt justering sprids till andra bild rutor och tidpunkter genom att flytta "z" rullnings listen (figur 3a, blå pil, vänster panel) och/eller "t" rullnings List (figur 3a, grön pil, vänster panel). Om förvärv av flera positioner utfördes och flera embryon avbildades (steg 5,3), borde anpassningen också ha propagerat för dessa embryon. Bekräfta genom att även undersöka siffrorna för x-, y-och z-justerare (figur 3a, orange fyrkant, som bör vara densamma för spima-panelen för alla embryon).
- Upprepa steg 9.4.1 – 9.4.4 för fönstret SPIMB diSPIM Monitor (figur 3a, höger panel).
- Klicka på "Dispim"-knappen och sedan på "fuse"-knappen (figur 3a, blå rektangel) för att öppna en dialog ruta som heter "Deconvolve/fuse Dispim RAW data volymer" (figur 3b). Ställ in parametrar som visas i figur 3b. Dessa parametrar åtgärdas kortfattat i följande del steg:
- Ange nyckel registrering på kanal 1 (488 nm Laser) eller 2 (561 nm Laser). Välj kanalen med tätare eller mer allestädes närvarande signal. För lineaging experiment alltid välja den kanal som används för att avbilda den allestädes närvarande nukleära histonfluorescens.
- Ange inmatnings volymernas relativa orientering till + 1 eller-1. Rätt orienterings index beror på en specifik diSPIM kamera placeringar (figur 4).
Obs: om osäker, testa varje alternativ genom att duplicera en enda tidpunkten från både spim a och B dispim Monitor fönster, följande steg 9.1-9.12 och godtyckligt välja en input volymer relativ Orientering för att testa. Felaktiga orienteringar ger suddiga bilder med artefakter, medan korrekta orienteringar ger tydliga bilder. Det relativa orienterings värde för indatavolymer som ger den klara bilden kan sedan återanvändas för alla framtida data från det givna dispim-instrumentet. - Välj om den brända volymen ska orienteras på samma som A-eller B-inmatningsvolymen (baserat på användar inställningar).
- Välj "färsk registrering för varje volym". Det här alternativet styr hur SpimFusion beräknar registreringmatriserna för varje volym par vid varje tidpunkt. Alternativet "Fresh" tillåter algoritmen för att optimera registreringen adaptivt vid varje tidpunkt.
- Ange antal avfaltningen iterationer till 10. Detta antal tenderar att tillförlitligt ge önskad hög upplösning på ett tids effektivt sätt.
- Valfritt: om automatisk lineaging önskas (rekommenderas), kontrol lera Auto-Launch StarryNite lineaging av brända volymer. Detta alternativ kommer att starta StarryNite automatiskt för att segmentera och spåra celler i de avbildade volymerna som produceras av SpimFusion.
- Tillval: för högsta noggrannhet i automatiserad lineaging, är det bäst att flytta de isotropically smält embryonala volymerna till Canonical "ADL" (ennterior [x-väst], Dorsal [y-North], vänster [z-Near]) orientering. Välj den definiera volym utdata orientering i förhands gransknings alternativet för att ange detta val. CytoSHOW kommer att reagera genom att bearbeta ett initialt par isotropically smält volymer, så att användaren kan noga observera och specificera de rotationer som behövs för att uppnå ADL registrering.
- Klicka på Ja när alla parametrar har valts.
- Ange utdatakatalogen som du vill spara de bearbetade filerna i. Klicka på OK.
- Valfritt: om Ange volym utmatnings orientering i förhands gransknings alternativet valdes, Ställ in t rullnings listen (figur 3a, grön pil, vänster panel) i spim-A fönstret till den tidiga tidpunkt då ABa och ABp celler har nått metafor. Ställ in t -rullnings listen i spim-B-fönstret till det senare utvecklingsstadiet. Detta kommer att hjälpa till att ange ADL-orientering.
- Valfritt: Klicka på OK när du är klar. Om förhands gransknings alternativet i 9.5.7 ovan valdes, kommer endast två förhands visnings volymer att vara isotropiskt fixerade för de tidpunkter som indikeras av t-reglagen på SPIM-A-och SPIM-B-bildfönstren. Dessa två förhands gransknings tidpunkter kan användas för att ange exakt omjustering av utgående embryovolymer till ADL-orienteringen, vilket förklaras nedan.
- Leta upp den nyligen visade 3DProjY_Decon-Fuse_.... Fönstret. Flytta t -rullnings listen till tids punkt 2 i det här förhands gransknings fönstret. Flytta Z-reglaget tills vyn direkt nedåt på embryots långa axel visas.
- Flytta t rullnings listen tillbaka till tid punkt 1 i 3DProjY_Decon-Fuse_.... Fönstret. Välj linje markerings verktyget och rita ett linje val från EMS-cellen (ventrala-mest runda kärnan) genom planet av AB-cell metafasplattor.
- Klicka på knappen orange diSPIM Preview på 3DProjY_Decon-Fuse_.... Fönstret. De fina justeringarna av orienteringen för den förhandsvisade avbildade volymen sparas för att användas vid bearbetningen av den fullständiga data uppsättningen.
- Tillval: den Deconvolve/fuse diSPIM RAW data volymer dialog rutan visas igen, precis som i steg 9,5 ovan. Klicka på Ja utan att välja den definiera volym utdata orientering i förhands gransknings alternativet. Ange Utdatammappen för den fullständiga data bearbetnings körningen.
- Ställ in t -rullnings listerna (figur 3a, grön pil, vänster panel) i dispim-övervakningsknappen till Start tidpunkten (spima) och ending tidpunkten (spimb) för hela omfånget av bilder som ska bearbetas. Klicka sedan på OK.
- Som SpimFusion fortskrider, öppnar CytoSHOW och fräschar upp ett flerdimensionellt fönster som visar den skivade-4D isotrop smält volym för varje embryo, samt två fönster med roterande-4D Max intensitet projektioner av ISO tropisk volym. Under denna tid, inte störa eller stänga någon cytoshow fönster tills isotropisk fusion och linje spårning är klar.
- Valfritt: Observera att när välkomst skärmen för StarryNite har dykt upp och senare försvann, har den fullständiga pipelinen för data bearbetning slutförts. Detta fönster får inte stängas under bearbetningen eller StarryNite kommer att avbrytas.
10. öppnande av StarryNite linje spårnings serie i AceTree (tillval)
- Öppna den anpassade versionen av "AceTree_16BitCompat. jar" som tillhandahålls.
- Välj Meny filen > Öppna Konfigurera filen. Lokalisera din utgång adress listen tidigare antytt till cytoshow. Öppna mappen Decon_Fuse_... _ POS [n] för embryo [n]. Välj aaa_edited. XML och Öppna.
- Använd menyn AceTree edit > Redigera verktyg för att öppna Redigera spår och Justera eller ta bort celler fönster.
- Klicka på halv skuggade cirkeln Figur 5b, Röda torget för att justera de röda och gröna intensiteter.
- Fortsätt med Lineage visualisering och redigering som tidigare beskrivits5,6,8 (manuskript ingår också i vårt nedladdnings paket).
Representative Results
Vi validerade först lönsamheten för embryon som avbildas med protokollets parametrar för diSPIM-förvärvet (avsnitt 1-6). Tio embryon avbildades samtidigt vid 20 ° c, en volym/embryo/minut, från 2-cellsstadiet till det 2-faldiga stadiet (7,5 timmar, 451 volymer/embryo). För att övervaka cell divisioner hela embryogenesis, vi använde stam BV514, som ubiquitously uttrycker Mcherry::histone reporter konstruktioner från den integrerade Transgene array ujIs11324. Figur 6 visar en tidsplan för den första halvan av embryonal utveckling för ett av de avbildade embryona. Varje bild motsvarar en maximal intensitet projektion (producerad av steg 7-8) av det avbildade embryot. Vi konstaterade att de optimerade protokollen inte inducerade några detekterbara fototoxicitet på embryona, bedömt av tidpunkten för cell delning (visas inte), tid för kläckning och timing i samband med utvecklingsrelaterade mil stolpar (figur 6 och hänvisningar1 , 25 % av den , och 26).
Vi tillämpade sedan protokollet för att analysera uttillväxtdynamiken hos enstaka neuroner i utvecklingen av embryon. Vi avbildade DCR7692 (olaex4655), en transgen tallvedsnematoden stam som uttrycker GFP utanför neuropeptid FLP-19 Promotorn i en delmängd av OIDENTIFIERADE celler (DACR2819, PFLP-19 (3.6 KB):: Syn21:: GFP:: caax::p 10 3 ' utr) . Efter de steg i protokollet som beskrivs här, vi fastställt att de oidentifierade cellerna motsvarar motoriska nerv celler RMDDL och RMDDR, till utsöndringskanalen cellen, och till två muskel celler (figur 7). Vi undersökte och kvantifierade sedan uttillväxtdynamiken hos RMDDL-och RMDDR-neuronerna. Vi observerade att RMDDL och RMDDR neuroner är snett formad så tidigt som 360 minuter efter befruktning, med längre cellulära axeln representerar den efterföljande axeln för neurite utväxt (figur 7 och film S1). Med hjälp av "enkel neurite tracing" plugin i FIJI och tillämpa den på 3D rekonstruktioner av isotropically smält volymer, kvantifierade vi sedan den stereotypiska utväxten av RMDDL och RMDDR neuriter för sex embryon. Vi bestämde att uttillväxtdynamiken var stereotypa för RMDDL och RMDDR över embryon (häri kallade RMDDs). Från 385-410 minuter efter gödsling förlängde RMDDs-neuriterna 6,0 ± 0,5 μm (medelvärde ± SEM; n = 12 neuriter) främre av cell kropparna (figur 7b, C, i). Från 415-445 minuter efter gödsling, båda neuriter förlänga dorsalt i och runt presumtiva nerv ringen (asterisk i figur 7d). I genomsnitt förlängdes varje RMDD neurit 11,0 ± 0,6 μm (medelvärde ± SEM; n = 12 neuriter) från cell kroppen innan synkront möta dess kontralaterala motsvarighet vid spetsen av ringen (figur 7i). Viktigt är att våra representativa resultat visar att vi kan undersöka, jämföra och kvantifiera neuronala utvecklingsfunktioner för enstaka identifierbara celler genom att använda vårt integrerade protokoll (figur 7 och figur 8).
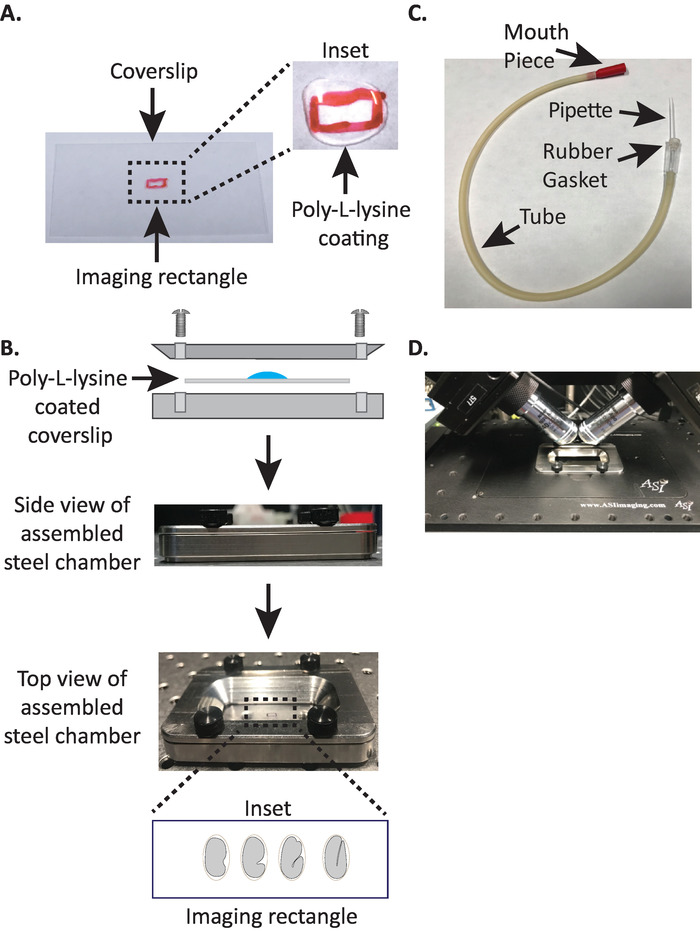
Bild 1: diSPIM installation av prov montering. Aberedning av täckglasöverdrag med Poly-L-Lysin. I det insatte, 10 μL av poly-L Lysin användes för att belägga täckglasen i 5 minuter. Poly-L-Lysin tillåter embryot äggskal att hålla fast vid täckglasen i rektangeln. (B) schema för stål bild kammaren och den monterade kammaren. I det Inset, representativa embryon är orienterade med främre-bakre axeln vinkel rätt mot den långa axeln på täckglasen. (C) monterad Nässug tub med mikrokapillärpipett. D) stål bild kammaren monterad i prov hållaren under dispim 40x-mål. Vänligen klicka här för att se en större version av denna siffra.

Bild 2: långsiktig inställning av diSPIM-avbildning i Micro-Manager. (A) Rekommenderad dispim laser effekt parametrar (röd rektangel) optimerad för långvarig avbildning samtidigt minska fototoxicitet (enligt bedömning av högre kläcknings frekvens av C. elegans embryon). Ställ 561 nm Laser till 79 μW (0,25) och 488 nm Laser till 179 μW (0,5). Observera att exakt kalibrering av program varu inställningar till laser effekt varierar mellan diSPIM-installationer. Det rekommenderas att användare mäter och kalibrerar laser effekten för att uppnå 79 μW (561 nm) och 179 μW (488 nm) Laser effekt. (B) dispim parametrar för data besparing (grön rektangel), volym inställningar (blå kvadrat) och segment inställningar (orange fyrkant). (C) dispim autofokusparametrar för långtids avbildning av C. elegans embryogenes (se steg 6.1-6.6). Vänligen klicka här för att se en större version av denna siffra.

Figur 3: bild visualisering och data behandlings inställning med CytoSHOW. (A) RAW dispim bilder som öppnats av cytoshow. CytoSHOW kan öppna bilder tagna av båda kamera banorna (SPIM A och B). Dessa RAW-bilder öppnas i flerdimensionella fönster som kallas Dispim Monitor. I Dispim Monitor, en "Bowtie mönster" genereras för att välja embryots främre, bakre, dorsala och ventrala kanter (se steg 9,1). Bow-tie val anger embryonal orientering för avfaltning och StarryNite-Assisted lineaging spårning. (B) optimerade parametrar som används för att generera ISO tropiska bilder. Ange de parametrar som anges i steg 9.5.1-9.5.8 i fönstret Deconvolved när du förvärvar . Vänligen klicka här för att se en större version av denna siffra.

Bild 4: diSPIM-kamerans konfiguration. (A) fotografi av dispim kamera placeringar och orienteringar. (B) skildring av + 90-graders rotationer av spim A för att matcha spim B bilder som samlats in. (C) inmatnings volymer i förhållande till orienterings index + 1 baserat på vår dispim kamera konfiguration (se steg 9.5.2). Vi roterar SPIM en bild (s) + 90 grader runt Y-axeln innan registrering för att matcha SPIM B bild (er). Skal streck = 10 μm. bilder är representativa singel-View, maximal intensitet prognoser och avfaltning bilder av 1,5-faldig embryo med märkta kärnor (561-nm, röd) och nerv celler (488-nm, grön). Vänligen klicka här för att se en större version av denna siffra.

Figur 5: Curation och redigering C. elegans embryonal härstamning i acetree. (A) vi använder acetree för att redigera starrynite härstamning spår (se hänvisningar5,6,8; manuskript ingår också i vårt nedladdnings paket). AceTree kommer att visa C. elegans systematiska namn för varje kärna (grön rektangel) efter avslutad steg 10.1-10.2. Det här fönstret (a) innehåller information (svart rektangel) om varje cell i linje spårningen (ABa, markerad med blått) som hjälper användarna att spåra och redigera härstamningsspåren. Det rekommenderas att användare kontrol lera och jämföra lineaged celler och deras positioner till C. elegans embryonala cell härstamning tidigare rapporter ATS av Sulston et al.1 dessutom, om användarna är intresserade av att lokalisera specifika celler i deconvolved data serie (se nedan, B) ange C. elegans systematiskt namn i Sök fältet (orange rektangel). (B) användarens deconvolved data serie öppnas också automatiskt efter avslutad steg 10.1-10.2. Här visas en isotropically smält bild av en fyrcellig etapp embryo med kärnor märkta med rött. Under spårande av en kärna, förbrukaren skulle ändra intensiteten om bild (röd fyrkant) och navigera igenom tid och z Använd ande pilen nyckel på deras klaviatur (tid-lämna/rätt, z-upp/ned). (C) 3D-tecknad av tidpunkten i (B) med vissa funktioner (lila rektangel) som möjliggör vridbar 3D-visualisering. För en översikt av acetree och dess 3D-funktionalitet, se hänvisningar5,6,8. Vänligen klicka här för att se en större version av denna siffra.

Figur 6: fördröjd utvecklingsdynamik av C. elegans embryon på dispim. Översta panelen, diSPIM bilder som visar den första halvan av embryonal utveckling för en av de avbildade embryona (Strain BV514 ujIs11324). Embryon avbildades kontinuerligt, varje minut för 7,5 timmar (vid 20 ° c). De första två bilderna av övre panelen representerar 4-och 8-cells embryon med kärnor (röd) och positioner av polära kroppar (täta röda sfärer, markerade med blå asterisker). Varje bild representerar en maximal intensitet projektion av det avbildade embryot. Skal streck = 10 μm. Tids linjen (horisontell stapel) representerar minuter efter gödsling (m.p.f.) av utvecklingen av C. elegans embryon. Vi har validerat att våra protokollets parametrar för diSPIM-förvärvet inte inducerade fototoxicitet på de avbildade embryona, bedömt genom lönsamhet, tidpunkt för cell delning, tidpunkt för kläckning och tidpunkt för utvecklingsrelaterade mil stolpar (se hänvisningar 1 den första , 25 % av den , 26). Vi noterar att tidpunkten för utvecklings mil stolpar var reproducerbar över embryon med våra Imaging parametrar (SEM ± 8,174 minuter för 6,4 timme långa bildsessioner; n = 10). Vänligen klicka här för att se en större version av denna siffra.

I figur 7. Cellulär identifiering och encellig karakterisering av neurit uttillväxtdynamik vid utveckling av embryon från C. elegans . Dual-Color Imaging av en stam som gjorts genom att korsa BV514 ujIs11324 (för lineaging) och DCR7692 (olaex4655), en transgena tallvedsnematoden stam som uttrycker GFP utanför neuropeptid FLP-19 Promotorn i en delmängd av oidentifierade celler. (a-H) Efter de steg i protokollet som beskrivs här, bestämde vi att de oidentifierade cellerna motsvarar motoriska nerv celler RMDDL och RMDDR (gula pilar), till utsöndringskanalen cellen (blå pilar), och till två muskel celler (vita pilar). (I) kvantifiering av uttillväxtdynamiken i rmddl och rmddr neuroner med hjälp av Fiji plugin "enkel neurite spårning" och tillämpa den på 3D rekonstruktioner av isotropically smält volymer. Observera hur både RMDDL och RMDDR visar stereotypisk uttillväxtdynamik, som var och en sträcker sig synkront för en total längd på 11,0 ± 0,6 μm (medelvärde ± SEM; n = 12 neuriter) och möte vid den dorsala spetsen av nerv ringen (se även film S1). Vänligen klicka här för att se en större version av denna siffra.

Figur 8: undersökning av isotropa diSPIM bilder av neuronala morfologier i C. elegans embryon. Isotropisk visualisering av AVHL-och AVHR-neuroner (gula pilar). Med hjälp av diSPIM kan neuronala morfologier fångas ger fyrdimensionella (4D) bilder med ISO tropiskt spatial upplösning på cirka 330 nm. DiSPIM tillåter användare att virtuellt rotera bild volymer med identisk upplösning i alla riktningar. Bilder i A-D är maximal intensitet projektioner av samma isotropically smält dispim bild volym från olika rotationer runt embryots långa axel. Skal streck = 5 μm. vänligen klicka här för att se en större version av denna siffra.
Kompletterande film S1: C. elegans embryo utvecklas från 280 till 434 minuter post befruktning. Isotropisk film av stam DCR7692 (olaex4655) uttrycker ujIs113 UBIQUITOUSLY med DACR2819 GLEST etikettering rmdd Neuriter (figur 7a-D, gula pilar). DACR2819 också etiketter två muskel celler (figur 7a-d, vita pilar) och utsöndringskanalen cell (figur 7a-d, blå pil) under embryonal utveckling (figur 7a-d). Skal streck = 10 μm. vänligen klicka här för att ladda ner denna fil.
Discussion
C. elegans sticker ut som den enda organismen med de slutliga positionerna och anslutnings möjligheterna för varje vuxen neuron känd27. Utvecklingsdynamik som leder till organisering av de arbets kretsar och nätverk som utgör C. elegans connectome är dock fortfarande okänd. Baserat på möjligheter som uppstår från framsteg inom ljus mikroskopi, kan vi nu fånga och analysera cell positioner, morfogenes, och neurogenes hela C. elegans embryonal utveckling.
Det förfarande som vi har beskrivit och som vi rutinmässigt använder i labbet ger 4D-isotropa bilder av märkta neuroner och kärnor för cell-lineaging i C. elegans embryon. Ännu viktigare, vi har optimerat långsiktiga bild förhållanden med diSPIM och kopplade semi-automatiserad lineaging funktioner med högupplösta bilder för att förbättra hastigheten och precisionen för att analysera C. elegans embryogenes. Detta integrerade protokoll gör det möjligt för användare att visualisera och identifiera celler och kvantifiera tredimensionella funktioner som neurit migration och Axon fasciympning genom uppkomsten av tidiga ryckningar. Detta förfarande kan lätt anpassas till alla anläggningar med ett ASI diSPIM-system, och vi rekommenderar detta system specifikt för detta protokoll. Andra SPIM-formuleringar som erbjuds kommersiellt kan skilja sig från ASI-konfigurationen i prov kammaren och optiska egenskaper. Data som exporteras från andra plattformar kan dock också placeras via vår pipeline för data. Därför är bedömning av deras värde i lineaging, ett krävande test av bild kvalitet och instrument stabilitet, genomförbart. Även om vi aktivt använder diSPIM att regelbundet avbilda andra prover (såsom Drosophila och zebra fisk embryon), den beskrivna och omfattande lineaging analys av embryon är fortfarande för närvarande begränsad till tallvedsnematoden arter. För större eller tjocka prover, väljer vi att använda steg-scanning metoder, som skannar proverna genom en stationär ljusplåt. Kumar et al. har tidigare visat denna förbättrade diSPIM snittning för att ge högkvalitativa bilder från tjocka prover utan ytterligare ändringar i diSPIM10.
De kritiska stegen i protokollet inkluderar montering C. elegans embryon på Poly-L-Lysin belagda täckglasen, data insamling, och data behandling. Skörd och montering C. elegans embryon på glas täckglasen kan vara utmanande för oerfarna användare, men här ger vi ett detaljerat protokoll över viktiga steg för att under lätta inlärning. Om långsiktig avbildning önskas, får vi bäst resultat att skörda fyrcellig eller tidigare embryon från 8-10 unga vuxna28. Observera att gamla vuxna är mindre önskvärt att skörda tidiga embryon eftersom de tenderar att innehålla äldre embryon i livmodern och Obefruktade ägg. När det gäller montering av embryon kan problem som blockering i den sammansatta aspiratorn (munpipett) eller en alltför stor öppning i mikrokapillärpipetten förhindra korrekt montering och orientering av embryon. För att förbereda för optimal avbildning, utför vi pre-förvärv tester på tidiga och sena pre-twitching embryon för att kontrol lera prestandan hos ljus lakan, kameror, mål och autofokus. Vi får bästa resultat när alla dessa åtgärder testas och ger bilder av hög kvalitet under vår pre-Acquisition testning. Detta är särskilt relevant för att skapa bilder med isotropisk rumslig upplösning, för vilka RAW-bilder som erhållits från båda vyerna (mål) måste vara av hög kvalitet. Efter förvärvet bearbetas de volymer som förvärv ATS för varje vy för att ge isotropa bilder. Det är viktigt att använda ett lämpligt grafik processor kort (GPU) enligt beskrivningen i detta protokoll (se nedan). Detta förbättrar bearbetnings hastigheten vid vilken isotropically brända bilder genereras, förkorta tiden till data analyser. Det är också viktigt att användarna kör den senaste versionen av CytoSHOW och använder parametrarna som medföljer vår nedladdnings paket för StarryNite Auto-lineaging. Om användare är intresserade av att använda auto-lineaging för andra prover (t. ex. Zebrafish, Drosophila etc.) kommer ytterligare optimering till de parametrar som används i starrynite krävas (se hänvisningar3,4).
Även om vårt integrerade protokoll ger bilder och lineaging resultat i pre-twitching embryo, användare bör vara medvetna om att automatiserad lineaging i post-twitching embryo är för närvarande inte genomförbart: nukleära positioner ändras på order av sekunder i post-twitching embryo, för snabbt att tillåta härstamning spårning. Men dispim har verkligen visat en lovande förmåga att fånga neuroutvecklande händelser och spåra vissa cell positioner i efter twitching stadier av embryogenes23,29. Om användarna är intresserade av att undersöka efter twitching embryo, diSPIM ger hastigheten för att få volymetriska ögonblicks bilder och spåra fina neuroutvecklande händelser, såsom neurit utväxt, i snabbt rörliga embryon.
Detta protokoll kommer att vara grundläggande för cell-by-cell slutförandet av WormGUIDES Atlas30, eftersom det kommer att ge en integrerad strategi med högupplösta ISO tropiska bilder för att identifiera och fånga 3D - morfologier av märkta nerv celler under första 430 minuter av embryogenes. Som det ser ut, prototypen WormGUIDES Atlas ger nukleära positioner av celler i det växande embryot och syftar till att fånga utvecklingsdynamik av en delmängd av embryonala nerv celler. Detta protokoll kommer att vara en nyckel för integrationen av ytterligare utveckla nerv celler i WormGUIDES Atlas30.
Vårt integrerade protokoll kommer också att förenkla utforskandet av nya gen uttrycks profiler i C. elegans embryo. I transgena C. eleganskontrollerar många cellspecifika initiativtagare rumsligt och tempo rally Transgene uttryck. Medan uttrycks mönstren för de flesta gener har i stor utsträckning karakteriserats i det vuxna djuret31,32,33,34, nästan alla ännu inte har karakteriserats i utvecklingen (särskilt sent stadium) embryo. C. elegans promoterome har varit en användbar resurs för masken samfundet att köra cell-specifika Transgene uttryck, samt avgöra om gen funktion är cell-autonoma eller icke-autonoma. Fånga ISO tropiskt högupplösta och dynamiska uttrycks mönster av gener, och exakt identifiera uttrycka celler via lineaging kommer att vara värdefullt för många i det vetenskapliga samfundet.
Embryogenesis består av två sammanflätade stora processer, cellulär differentiering och vävnad morfogenes. Mycket är känt om mekanismerna och molekylerna som definierar distinkta cell typer under utvecklingen av C. elegans. Emellertid, lite är känt om de mekanismer som är viktiga för cell migration, cell adhesion, och cell form i C. elegans embryo. Med C. elegans invariant cell härstamning känd, vårt protokoll gör att vi lätt urskilja katalogiserade 3D-Microanatomy av embryot under morfogenes på nya nivåer av Detaljer: t. ex., Axon fasciympning, synaptogenesis, och neuronala aktivitet. Ardiel et al. har tidigare visat kraften i diSPIM att fånga kalcium transienter i nivå med en enda nerv celler i C. elegans embryon23. Många andra aspekter av utvecklingsfysiologi är mogna för utredning av dessa metoder.
Slutligen, detta protokoll är till stor del automatiserad och systematiskt minskar den tid det tar att generera deconvolution bilder och utföra cell-lineaging via StarryNite och Acetree. De program varu strategier som används i detta protokoll kan tillämpas på många frågor om biologi långt-kastade från de mycket specifika områden där vi har visat dem här.
Information om Programvarukompatibilitet och nedladdnings åtkomst
Information om Micro-Manager och plugins för diSPIM Imaging finns på http://dispim.org/software/micro-manager och https://micro-manager.org/wiki/ASIdiSPIM_Plugin.
Data-processing pipeline kräver för närvarande ett Windows-operativsystem. Vi har paketerat en enda arkivfil för att förenkla installationen av alla nödvändiga data behandlings program och supportfiler. Den finns att hämta på http://dispimlineage.wormguides.org.
CytoSHOW (http://run.cytoshow.org/) är baserat på den allmänt använda och öppen källkod bild analys plattform, ImageJ (v1). Java måste vara installerat och uppdaterat på datorn för att använda CytoSHOW, och uppdateringar av CytoSHOW distribueras automatiskt via Java Web Start. Många ImageJ-baserade funktioner av CytoSHOW är som beskrivs och illustreras på https://imagej.nih.gov/ij/docs/examples/index.html. CytoSHOW har anpassats för att Visa flerdimensionella rå data från ASI diSPIM, samt andra bild program som skapar TIFF-utdata. I princip kan andra multi-View SPIM Imaging system stödjas av mindre modifieringar av CytoSHOW för att möjliggöra att detta protokoll kan utföras på olika Mikroskop system.
SpimFusion skrevs i CUDA/C++ med Visual Studio 2013 med CUDA Toolkit v 7.5. Kör SpimFusion kräver särskild dator maskin vara: ett NVIDIA grafik processor (GPU) kort med CUDA Compute kapacitet 1,0 eller högre och minst 2 GB grafik kort minne. Vid tidpunkten för offentliggörandet av vårt protokoll, SpimFusion är opublicerade (min Guo och Hari Shroff) men finns i program varan bunt arkiv som nämns ovan.
En specialbyggd kommando rads driven version av StarryNite kräver att den fritt tillgängliga MATLAB compiler runtime är installerad, men kräver ingen licens för kommersiell MATLAB-programvara. MATLAB compiler runtime ingår i program varu bunt arkivet som nämns ovan. Koden för StarryNite som används i detta protokoll är i huvudsak oförändrad från den som används för konfokal bilder6. Men flera operativa frågor i skapandet av ingångs bilder för StarryNite bearbetning och hantering av StarryNite resultat har tagits upp här med metoder i CytoSHOW som möjliggör en kontinuerlig data bearbetning pipeline för smält isotropisk diSPIM Volymer. Dessa ändringar är automatiserade av CytoSHOW kod som hanterar dessa före och efter bearbetning steg. CytoSHOW redigerar också en föroptimerad diSPIM-specifik mall StarryNite parameter inställd för att automatiskt finjustera segmenteringsalgoritmen till fluorescensintensiteten hos kärnor i avbildade data. De unika parametrarna som används av StarryNite på varje diSPIM-datauppsättning sparas sedan i en fil tillsammans med utdatabilden och lineaging-data.
En anpassad version av AceTree som fungerar med 16-bitarsbilder och bibehåller kompatibiliteten med Java3D-rendering lämpar sig bäst för detta protokoll. Det ingår också i program varan bunt arkiv som nämns ovan.
Disclosures
Författarna har inget att avslöja.
Acknowledgments
Vi tackar John Murray för integrerad stam, ujIs113, för att generera lineaging stam BV514; Brandon Harvey (NIBIB) för hjälp med att testa protokollet; Jon Daniels och Gary Rondeau (tillämpad vetenskaplig instrumentering) för hjälp med Micro-Manager och diSPIM instrument; och Andrew York och Hank Eden för deras kritiska återkoppling på diSPIM-systemet. Vi tackar också forsknings centret för minoritets institutioner program och Instituto de Neurobiología Jose del Castillo (Universidad de Puerto Rico) för att tillhandahålla ett möte och brainstorming plattform. Mycket av detta arbete utfördes vid marin biologiska laboratoriet vid Woods Hole genom Whitman-programmet. Detta arbete stöddes av intramural forsknings program av NIH nationella institutet för biomedicinsk Imaging och bio teknik och NIH Grant No. U01-HD075602 och nej. R24-OD016474. Mark W. Moyle stöddes av F32-NS098616 och Leighton H. Duncan stöddes av ett mångfalds tillägg till R24-OD016474.
Materials
| Name | Company | Catalog Number | Comments |
| Steps 1-4 | |||
| Concavity slides | ThermoFisher Scientific | 1519006 | 5-18mm diameter, 0.6-0.8mm deep, 1.2-1.5mm |
| Dissecting microscope with 10×–50× zoom range | Motic | SMZ-171 | |
| E. coli (OP50) | Caenorhabditis Genetics Center (CGC) | OP50 | |
| Glass coverslips, no. 1.5, 24 × 50 mm | VWR International | 48393-241 | |
| M9 Buffer | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Methyl cellulose | Sigma-Aldrich | H7509-25G | |
| Microcapillary pipette aspirator tube | Sigma-Aldrich | A5177 | |
| Microcapillary pipettes, 0.4-mm i.d | Drummond Scientific | 1-000-800 | |
| Needle, no. 18G x 1 ½ (1.2mm x 40mm) | BD Precision Glide | 305196 | |
| NGM plates | prepared as described by Brenner (1974) | ||
| O-ring for imaging chamber | O-Rings West | M1.5X40 | |
| Pasteur pipette | Corning/Sigma-Aldrich | CLS7095D5X | |
| Platinum wire, 0.5-mm diameter | Sigma-Aldrich | 267201 | |
| Poly-L-lysine | Sigma-Aldrich | P1524 | |
| Stainless steel rectangular chamber (76.0 mm x 50.5 mm) | Applied Scientifics Instrumentations (ASI) | I2450 | |
| Worm Eyelash Pick | Hart, A. C. Behavior. WormBook. (2006). | ||
| Worm Pick | Stiernagle, T. Maintenance of C. elegans. WormBook. 1-11, doi:10.1895/wormbook.1.101.1, (2006). | ||
| Name | Company | Catalog Number | Comments |
| Steps 5-6 | |||
| 488 nm long-pass filter | Semrock | LP02-488 RU-2 | |
| 561-nm notch filter | Semrock | NF03-561E-25 | |
| BLP02-561R-25, quantity 2 | Semrock | 561 nm EdgeBasic best-value long-pass edge filter | |
| Control software for bottom camera | Jenoptik | ProgRes CapturePro | |
| diSIPM assembly video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/TAgbr6IrTqw ; http://www.asiimaging.com | |
| diSPIM alignment video | Applied Scientifics Instrumentations (ASI) | https://youtu.be/qnOrg30NNuE | |
| diSPIM imaging PC | Intel | Intel Xeon CPU E5-2630 2.6GHz, 12 cores in total, 64 GB memory, Windows 7 | |
| FF01-525/45-25, quantity 2 | Semrock | 525/45 nm BrightLine single-band bandpass filter | |
| FF555-DI03-25X36, quantity 2 | Semrock | 555 nm edge BrightLine single-edge dichroic beamsplitter | |
| Imaging PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Kumar et al diSPIM Setup | Applied Scientifics Instrumentations (ASI) | Instrument setup for this protocol is identical to Kumar et al 10,11 diSPIM, which makes use of 40x 0.8NA water immersion lenses for imaging. (See steps 5.1 and note) | |
| Micro Manager | Micro-Manager | https://micro-manager.org/ | |
| Modifications to Kumar et al diSPIM Setup (see below) | |||
| Optical table with isolators, 4 feet × 6 feet × 12 inches | TMC | 784-651-02DR and 14-416-34 | |
| Name | Company | Catalog Number | Comments |
| Steps 7-10 | |||
| Analysis PC | Intel | Intel Core i7-8700K CPU 3.70GHz, 6 cores in total, 64 GB memory, Windows 10 | |
| Analysis PC Graphics Card | NVIDIDA | NVIDIA GeForce GTX 1080 Ti graphics cards | |
| Installation instructions | Software bundle | http://dispimlineage.wormguides.org/diSPIMlineaging_InstallationInstructions.htm | |
| Software bundle | Software bundle | http://dispimlineage.wormguides.org |
References
- Sulston, J. E., Schierenberg, E., White, J. G., Thomson, J. N. The embryonic cell lineage of the nematode Caenorhabditis elegans. Developmental Biology. 100 (1), 64-119 (1983).
- Bao, Z., et al. Automated cell lineage tracing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 103 (8), 2707-2712 (2006).
- Santella, A., Du, Z., Bao, Z. A semi-local neighborhood-based framework for probabilistic cell lineage tracing. BMC Bioinformatics. 15, 217 (2014).
- Santella, A., Du, Z., Nowotschin, S., Hadjantonakis, A. K., Bao, Z. A hybrid blob-slice model for accurate and efficient detection of fluorescence labeled nuclei in 3D. BMC Bioinformatics. 11, 580 (2010).
- Boyle, T. J., Bao, Z., Murray, J. I., Araya, C. L., Waterston, R. H. AceTree: a tool for visual analysis of Caenorhabditis elegans embryogenesis. BMC Bioinformatics. 7, 275 (2006).
- Katzman, B., Tang, D., Santella, A., Bao, Z. AceTree: a major update and case study in the long term maintenance of open-source scientific software. BMC Bioinformatics. 19 (1), 121 (2018).
- Murray, J. I., et al. Automated analysis of embryonic gene expression with cellular resolution in C. elegans. Nature Methods. 5 (8), 703-709 (2008).
- Murray, J. I., Bao, Z., Boyle, T. J., Waterston, R. H. The lineaging of fluorescently-labeled Caenorhabditis elegans embryos with StarryNite and AceTree. Nature Protocols. 1 (3), 1468-1476 (2006).
- Wu, Y., et al. Spatially isotropic four-dimensional imaging with dual-view plane illumination microscopy. Nature Biotechnology. 31 (11), 1032-1038 (2013).
- Kumar, A., et al. Using Stage- and Slit-Scanning to Improve Contrast and Optical Sectioning in Dual-View Inverted Light Sheet Microscopy (diSPIM). The Biological Bulletin. 231 (1), 26-39 (2016).
- Kumar, A., et al. Dual-view plane illumination microscopy for rapid and spatially isotropic imaging. Nature Protocols. 9 (11), 2555-2573 (2014).
- Wu, Y., Christensen, R., Colon-Ramos, D., Shroff, H. Advanced optical imaging techniques for neurodevelopment. Current Opinion in Neurobiology. 23 (6), 1090-1097 (2013).
- Wu, Y., et al. Inverted selective plane illumination microscopy (iSPIM) enables coupled cell identity lineaging and neurodevelopmental imaging in Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 108 (43), 17708-17713 (2011).
- Huisken, J., Stainier, D. Y. Selective plane illumination microscopy techniques in developmental biology. Development. 136 (12), 1963-1975 (2009).
- Stelzer, E. H. Light-sheet fluorescence microscopy for quantitative biology. Nature Methods. 12 (1), 23-26 (2015).
- Winter, P. W., Shroff, H. Faster fluorescence microscopy: advances in high speed biological imaging. Current Opinion in Chemical Biology. 20, 46-53 (2014).
- Lucy, L. B. An iterative technique for the rectification of observed distributions. Astronomical Journal. 76 (6), 745-754 (1974).
- Richardson, W. H. Bayesian-Based Iterative Method of Image Restoration. JOSA. 62 (1), 55-59 (1972).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Hart, A. C. Behavior. WormBook. , (2006).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using microManager. Current Protocols in Molecular Biology. Chapter 14, Unit14 20 (2010).
- Gualda, E. J., et al. SPIM-fluid: open source light-sheet based platform for high-throughput imaging. Biomedical Optics Express. 6 (11), 4447-4456 (2015).
- Ardiel, E. L., et al. Visualizing Calcium Flux in Freely Moving Nematode Embryos. Biophysical Journal. 112 (9), 1975-1983 (2017).
- Walton, T., et al. The Bicoid class homeodomain factors ceh-36/OTX and unc-30/PITX cooperate in C. elegans embryonic progenitor cells to regulate robust development. PLoS Genetics. 11 (3), e1005003 (2015).
- Altun, Z. F. WormAtlas. , (2002).
- Wood, W. B. Embryology: In the nematode C. elegans. Cold Spring Harbor Laboratory Press. Chapter 8, 215-241 (1988).
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical Transactions of the Royal Society B: Biological Sciences. 314 (1165), (1986).
- Bao, Z., Murray, J. I. Mounting Caenorhabditis elegans embryos for live imaging of embryogenesis. Cold Spring Harb Protoc. (9), (2011).
- Christensen, R. P., et al. Untwisting the Caenorhabditis elegans embryo. eLife. 4, (2015).
- Santella, A., et al. WormGUIDES: an interactive single cell developmental atlas and tool for collaborative multidimensional data exploration. BMC Bioinformatics. 16, 189 (2015).
- Dupuy, D., et al. A first version of the Caenorhabditis elegans Promoterome. Genome Research. 14 (10B), 2169-2175 (2004).
- Reece-Hoyes, J. S., et al. Insight into transcription factor gene duplication from Caenorhabditis elegans Promoterome-driven expression patterns. BMC Genomics. 8 (27), (2007).
- WormBase. , Available from: https://www.wormbase.org (2019).
- Lee, R. Y. N., et al. WormBase 2017: molting into a new stage. Nucleic Acids Research. 46 (D1), D869-D874 (2018).



