Summary
Présenté ici est un protocole pour une seule cellule, l'épifluorescence technique basée sur la microscopie pour quantifier les taux de pâturage chez les eucaryotes prédateurs aquatiques avec une haute précision et une résolution taxonomique.
Abstract
L'élucidation des interactions trophiques, telles que la prédation et ses effets, est une tâche fréquente pour de nombreux chercheurs en écologie. L'étude des communautés microbiennes a de nombreuses limites, et il est souvent difficile de déterminer les taux de prédateurs, de proies et de prédateurs. Présenté ici est une méthode optimisée basée sur l'ajout de proies fluorescentes étiquetées comme un traceur, ce qui permet une quantitation fiable des taux de pâturage dans les eucaryotes prédateurs aquatiques et l'estimation du transfert de nutriments à des niveaux trophiques plus élevés.
Introduction
Les procaryotes hétérotrophes sont un composant biologique clé dans les systèmes aquatiques et représentent une fraction importante de la biomasse du plancton1,2,3. Les facteurs qui contrôlent leur abondance, leur diversité et leur activité sont essentiels pour comprendre leur rôle dans le cycle biogéochimique (c.-à-d. le sort du carbone organique et d'autres nutriments et le flux d'énergie des procaryotes à des niveaux trophiques plus élevés). Le pâturage protozoaires est l'un de ces facteurs importants. Bacterivory des nanoflagellates et des cils hétérotrophes impose un contrôle descendant fort sur l'abondance procaryotique, la fonction de la communauté, la structure, la diversité, et même la morphologie cellulaire et le taux de croissance de groupes bactériens particuliers4, 5,6. Dans certains systèmes, les protistes sont la principale cause de mortalité bactérienne6,7.
L'approche standard utilisée pour évaluer le bactérioris protozoaire, qui est utilisé depuis un certain temps déjà, implique l'utilisation de bactéries étiquetées fluorescentes (FLB) comme analogues de proie et microscopie épifluorescence. Les taux d'apaisement spécifiques aux cellules peuvent être déterminés en quantifiant le nombre de particules de proies étiquetées dans les vacuoles alimentaires du protistan sur un parcours temporel sélectionné8. Cette approche présente plusieurs avantages. Tracer est ajouté à des échantillons naturels avec des assemblages naturels de prédateurs et de proies. Il y a une manipulation minimale de l'échantillon avant l'incubation, une altération minimale de l'échantillon par le traceur FLB ajouté, et les temps d'incubation sont courts pour s'assurer que les résultats sont solides obtenus dans des conditions proches de l'in situ. Alternativement, dans les environnements où le nombre de protistes bactérioraires ou de zooplancton (p. ex., les systèmes marins extracôtiers), les taux de disparition des FLB ajoutés aux échantillons en faibles quantités (2 % à 3 % de traceur) peuvent être détectés par cytométrie de débit à long terme (12-24 h). expériences d'incubation. Ensuite, les nombres de FLB aux points de départ et d'extrémité (intégrant l'impact de tous les bactérioris) sont quantifiés par cytométrie de flux (pour plus de détails, voir la publication précédente9). Cependant, un tel paramètre ne représente que les taux totaux de bactérioris agrégés qui ne peuvent être directement attribués à des groupes ou espèces de brouteurs de protistan et de zooplancton en particulier.
Dans l'ensemble, il peut être difficile de quantifier avec précision et avec un sens écologique les taux de mortalité bactérienne spécifiques aux espèces protistan ou au morphotype dans l'environnement aquatique. Certains protistes sont des brouteurs sélectifs, et la taille et la forme cellulaire du traceur FLB ajouté peuvent fausser les taux naturels d'ingestion deproies 10,11. En outre, l'activité et le métabolisme du protistan sont très sensibles à la température12; par conséquent, la quantité de traceur FLB ajouté doit être soigneusement manipulée pour chaque type d'échantillon individuel (non seulement en fonction de l'abondance naturelle, la taille et la morphologie des bactéries et des types dominants de bactériivores, mais aussi sur la température). La plupart des études portent sur l'activité de pâturage en vrac du protistan; cependant, le bactériérique d'espèces spécifiques de protistan a souvent une valeur d'information beaucoup plus élevée et peut être préférable. Dans ce cas, la connaissance taxonomique des espèces protistes présentes dans un échantillon et la compréhension de leur comportement sont nécessaires. Par conséquent, des quantités considérables de temps et de travail sont nécessaires pour obtenir des résultats solides sur les taux spécifiques d'espèce de bactériorisattribuable attribuable à un groupe ou une espèce protistan particulier.
Malgré ces difficultés, cette approche demeure l'outil le plus approprié actuellement disponible pour évaluer le bactériivory protistan dans les milieux naturels. Présenté ici est une méthode complète, facile à suivre pour utiliser FLB comme un traceur dans les études d'écologie microbienne aquatique. Tous les aspects problématiques mentionnés de l'approche sont pris en compte et un flux de travail amélioré est décrit, avec deux expériences à partir d'environnements contrastés ainsi que des espèces de ciliate contrastées à titre d'exemples.
La première étude de cas a été menée dans un environnement épilimnétique à partir du réservoir d'eau mésotrophique d'Omov en République tchèque, qui montre des abondances de broussailles et de bactéries comparables à la plupart des plans d'eau douce de surface (cf.5,7). La deuxième étude de cas a été menée dans l'environnement très spécifique à l'intérieur des pièges de la plante carnivore aquatique Utricularia reflexa, qui accueille un nombre extrêmement élevé de deux ciliates mixotrophiques de pâturage (Tetrahymena utriculariae) cellules bactériennes. Les calculs des taux de pâturage spécifiques aux cellules et des stocks permanents bactériens dans les deux types d'échantillons sont présentés. Une série d'interprétations écologiques des résultats est ensuite discutée, et des exemples d'études de suivi possibles sont finalement suggérés.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Collecte d'échantillons
-
Collecte de l'échantillon d'eau du réservoir: première étude de cas (Exp I; système naturel inférieur d'abondance des prédateurs in situ et des proies)
- Recueillir des échantillons d'eau à l'endroit désiré à une profondeur appropriée. Conservez les échantillons dans une glacière à température contrôlée remplie à température in situ (en évitant les chocs de température; il convient de noter que les taux d'utilisation des protistes sont dépendants de la température) pendant le transport au laboratoire.
REMARQUE : Notre échantillonnage a été effectué dans le réservoir méso-eutrophique en forme de canyon (Bohême du Sud, volume 34,5 x 106 m3, profondeur maximale 43 m, temps de rétention moyen 100 jours, dimictique). Le site d'échantillonnage était situé à une profondeur de 30 m, près du barrage. Un échantillon mixte d'une profondeur de 0,5 m a été prélevé par un échantillonneur Friedinger de 2 l. - Continuez à la section 1.2 dès que possible.
- Recueillir des échantillons d'eau à l'endroit désiré à une profondeur appropriée. Conservez les échantillons dans une glacière à température contrôlée remplie à température in situ (en évitant les chocs de température; il convient de noter que les taux d'utilisation des protistes sont dépendants de la température) pendant le transport au laboratoire.
-
Collecte de liquide dans les pièges des plantes carnivores Utricularia reflexa: la deuxième étude de cas (Exp II; un système avec des abondances de prédateurs et de proies élevées, volume d'échantillon extrêmement faible)
- Secouez doucement les plantes sous l'eau, retirez-les du contenant de culture et placez-les sur des matières absorbantes pour absorber l'excès d'eau. Selon la robustesse des plantes et la taille des pièges, choisissez 8-10 individus de plante.
- Divisez chaque pousse en parties à peu près égales en comptant les nœuds de feuilles portant des organes de piégeage. Chaque segment de pousse servira à recueillir des échantillons mixtes représentant des pièges jeunes, d'âge moyen et anciens.
- Fixez un mince capillaire en verre et une fiole Eppendorf pour la collecte d'échantillons à une pompe péristétique. Procéder à l'insertion de la pointe capillaire dans l'ouverture du piège. À l'aide de la pompe à vide, aspirer tout le liquide de chaque piège jusqu'à ce que 900 à 100 l de liquide de piégeage soient recueillis pour chaque catégorie d'âge du piège.
- Utilisez des sous-échantillons triplicate de 200 L du fluide de piégeage mis en commun pour des expériences de pâturage protistan. Traiter immédiatement ceux-ci comme détaillé à la section 3. Préserver les 300 oL restants de l'échantillon pour toutes les autres analyses des composants microbiens vivant dans le fluide, comme détaillé ci-dessous (section 2).
- Passez immédiatement à l'article 2.
2. Fixation des échantillons prélevés
- Exp I et II : fixer des sous-échantillons d'eau/fluide de piégeage pour l'énumération bactérienne (section 4; environ 20 ml et 0,3 ml, respectivement) avec du formaldéhyde pendant au moins 1 h pour obtenir un volume final de 2 % : concentration en volume dans chaque échantillon.
REMARQUE : Manipulez le formaldéhyde exclusivement dans le capot de fumée, et portez des gants en tout temps tout en manipulant des échantillons.
3. Filtration d'échantillon
- Dilution de l'échantillon (Exp I) : aucune dilution n'est nécessaire pour les échantillons d'eau du réservoir. (Exp II) : diluer l'échantillon de fluide de piège 10x-100x avec de l'eau MQ sans particules pour obtenir une distribution appropriée des microbes cibles sur les surfaces filtrantes avant de compter par microscopie épifluorescence.
- Filtrer 1-2 ml (Exp I) de l'eau du réservoir ou 10-30 l (Exp II) du sous-échantillon du fluide de piégeage pour le comptage bactérien sur des filtres noirs de la taille d'un pore de 0,2 m, à l'aide d'un entonnoir de filtration (25 mm de diamètre).
- Tainer les filtres avec dIPI (4',6-diamidino-2'-phenylindole dihydrochlorure, 0.2% concentration finale) pendant 4 min.
REMARQUE : Évitez la contamination de la peau et de la surface de travail et portez des gants. - Placez le filtre avec des microbes concentrés sur une goutte d'huile d'immersion (pour la microscopie fluorescente) sur une lame de microscope. Placez une autre goutte d'huile sur le centre du filtre et recouvrez-la d'une feuille de couverture, en veillant à ce que l'huile soit répartie uniformément.
- À ce stade, traiter de préférence les échantillons immédiatement, ou encore, les conserver au congélateur (-20 oC) pendant plusieurs semaines à plusieurs mois jusqu'à ce qu'une analyse plus approfondie soit effectuée.
4. Recensement des nombres bactériens sur les filtres
- Placez la diapositive sous le microscope épifluorescence (avec le filtre réglé correspondant au DAPI fluorochrome). Placez une grille de comptage de 10 x 10 dans l'une des oculaires. Déplacez la diapositive vers une position aléatoire.
- Quantifier les cellules bactériennes (fluorescence bleue) dans la zone de la grille de comptage (sous le grossissement 1000x). Dans les comptes, inclure les cellules traversant les bords gauche et supérieur de la grille de comptage, tout en excluant celles situées sur les bords droit et inférieur.
- Déplacez-vous vers une autre position aléatoire et répétez l'énumération sur au moins 10-15 grilles de comptage, s'élevant à 500 cellules comptées au total.
- Établir un facteur de conversion pour un microscope et un grossissement donnés, en fonction de la connaissance du rapport de la zone d'une grille à la zone de filtration effective totale du filtre. Ensuite, divisez le nombre total de cellules comptées par le nombre de grilles comptées, ce qui donne un nombre moyen de bactéries par grille.
- Multipliez ce dernier par le facteur de conversion établi et normalisez le nombre résultant par mL d'un échantillon (selon le volume d'échantillon filtré) pour obtenir l'abondance bactérienne totale par mL.
5. Détermination de l'abondance du protistan
- Fixer l'échantillon d'eau (Exp I) ou les sous-échantillons de fluide de piégeage (Exp II) avec soit du glutaraldéhyde (concentration finale de 1 %, plus approprié pour les échantillons contenant de la chlorophylle contenant des particules à traiter quelques jours à quelques semaines après la fixation) ou à l'aide du technique de décoloration formol-thiosulfate spécifiée ci-dessous.
REMARQUE : Les deux techniques de conservation empêchent l'égestion du matériel ingéré des vacuoles alimentaires des protistes8. - En ce qui concerne la technique de décoloration du formol-thiosulfate, ajoutez 100 l/1 l de la solution de Lugol dans 20 mL/200 l du sous-échantillon de fluide d'eau/piège (Exp I/Exp II, respectivement).
- Suivez immédiatement avec l'ajout de 0,5 mL/50 l de formaline tamponnée de borate, puis de 20 l/2 l de thiosulfate de sodium de 3 % (Exp I/Exp II, respectivement).
REMARQUE: Le thiosulphate de sodium décolore la couleur jaune de Lugol pour permettre l'observation des cellules sous le microscope épifluorescence8. - Filtrer un volume connu d'échantillon (selon le nombre de protistes cibles) sur des filtres en polycarbonate noir de la taille d'un pore.
- Estimer le nombre de l'espèce protiste est en comptant au moins 200 cellules sous grossissement 600x en utilisant la grille de comptage (voir ci-dessus).
- Réduire le volume de l'échantillon sous vide dans l'entonnoir de filtration à feu doux à environ 2 ml. Ensuite, relâchez la sous-pression et ajoutez le fluorochrome DAPI (4',6-diamidino-2-phenylindole dihydrochlorure13, 0.2% concentration finale) pendant 2 min.
6. Détermination de la structure communautaire des ciales dans des échantillons de plancton
REMARQUE : Les communautés ciliates dans les habitats d'eau douce sont très diverses14,15,16,18, et leur détermination microscopique est difficile. Trier les groupes de ciliate en guildes fonctionnelles10,14,16,17 permet une analyse plus détaillée des différents groupes de ciliate comme bactérioristes pélagiques.
- Évaluer la structure communautaire du ciliate en combinant les éléments suivants :
- Échantillons tachés de DAPI dans la microscopie épifluorescence (pour localiser les cellules ciliate avec fluorescence lumineuse des macro- et micro-nucléées de différentes tailles et morphologie) combinés avec l'apport de bactéries fluorescentes étiquetées (FLB8; pour plus de détails, voir ci-dessous), le suivi de la capacité des cialiates à se nourrir de bactéries.
- Observation d'échantillon en direct dans certains cas17,19. Pour plus de détails sur les approches et les critères ci-dessus utilisés pour regrouper les ciliens en différentes catégories taxonomiques, voir les publications précédentes16,17.
REMARQUE : Des études citées ont indiqué que parmi les cigales, les espèces omnivores de Stichotrichia (genera Halteria et Pelagohalteria) et D'Oligotrichia (à savoir Rimostrombidium spp.) sont les plus importants consommateurs pélagiques de bacterioplancton dans une grande majorité des habitats d'eau douce10,17,18.
7. Estimation des taux de pâturage du ciliate
- Calculer les taux d'utilisation des cigales sur les bactéries en fonction des changements dans le nombre moyen de traceurs [c.-à-d., FLB8 par ciliate lié au temps d'incubation (5-15 min)] et la quantité de traceur de FLB ajouté, représentant au plus de 5%-15% des bactéries totales.
- Pour comparer les taux d'utilisation entre les différentes espèces de protistan, normalisez les taux d'utilisation en tant que nombre de bactéries par ciliate par heure, avec des calculs basés sur le temps d'incubation réel et la proportion de traceur FLB ajouté.
REMARQUE : Le schéma général de l'application de la méthode FLB pour estimer les taux de bactérioris spécifiques aux cellules ou aux espèces et en vrac dans les échantillons naturels est représenté à la figure 1. - Préparation de FLB à partir de souches bactériennes indigènes à un environnement d'eau douce8
- Sélectionnez une taille appropriée (volume cellulaire moyen et MCV) et une morphologie des bactéries afin qu'elles imitent efficacement les tailles typiques des cellules bactérioplanctoniques/bactériennes dans le système aquatique à l'étude.
REMARQUE : Pour Exp I, un mélange de souches isolées provenant de l'emplacement d'étude du genre Limnohabitans et du Polynucleobacter a été utilisé (c.-à-d. bactérietypique et très abondante dans les lacs et les étangs)20. Pour plus de détails sur la morphologie et la taille des souches, voir les publications précédentes3,17,18,21. - Récoltez les cellules bactériennes de la culture par centrifugation (5 000 x g)pendant 15 min au début de la phase stationnaire et mélangez-les à un rapport numérique qui donne mcV - SD des cellules dans le mélange correspondant au MCV typique des bactéries à l'endroit choisi.
- Suspendre les granulés dans 10 ml de saline tamponnée de phosphate (PBS; pH no 9).
- Ajouter 2 mg de colorant fluoré jaune-vert 5-(4,6-dichlorotriazin-2-yl) aminofluorescein (DTAF, se lie aux protéines) à la suspension cellulaire dans le tampon phosphate-saline, et couver dans un bain d'eau de 60 oC pendant 2 h.
- Après l'incubation, centrifuger les cellules vers le bas, décante la solution DTAF, et laver et centrifuger 3x avec PBS.
- Après le lavage final, re-suspendre les cellules dans20 ml de la mémoire tampon PP i-saline.
- Vortex la suspension FLB et pipette 1,5 mL aliquots en 2 ml cryo-vials, puis garder congelé (à -20 oC) dans PPi-saline tampon jusqu'à l'utilisation.
- Pré-filtre PPi-saline tampon à travers un filtre en polycarbonate de 0,2 m pour une utilisation dans l'étape suivante.
- Pour déterminer la concentration de FLB, transférer un petit aliquot (habituellement 20-40 l) à 2 ml de tampon PP i-saline sans particules, sonicate à 30 W pour plusieurs rafales de 2 s, et filtrez sur un filtre noir en polycarbonate de 0,2 m pour l'énumération par épifluorescence microscopie (1 000X grossissement) sous des paramètres de filtre optique pour DTAF (448 nm/520-540 nm).
- Sélectionnez une taille appropriée (volume cellulaire moyen et MCV) et une morphologie des bactéries afin qu'elles imitent efficacement les tailles typiques des cellules bactérioplanctoniques/bactériennes dans le système aquatique à l'étude.
- Technique de traçage pour l'estimation du bactériivory ciliate
- Pour les expériences de pâturage dans les habitats planctoniques naturels, distribuez des échantillons de 300 ml dans des flacons de 1 L bien rincés et incubez à température in situ pendant 15 min (pour permettre aux protistes de se remettre du choc de manipulation).
- Ajouter les traceurs FLB pour constituer 5%-15% des bactéries totales, avec les quantités ajoutées en fonction de la saison et la température de l'eau.
REMARQUE : Il existe un spectre très large et dépendant de la saison dans les taux d'apitoier spécifiques aux espèces ciliate couvrant plusieurs ordres de grandeur (c.-à-d. de 101-104 bactéries ciliate-1 par heure)10,16, 17,18,22,23,24. - Dans les périodes d'augmentation de l'occurrence des ciales avec des taux d'utilisation élevés (généralement pendant l'été), également exécuter une incubation parallèle avec des ajouts FLB très faible, constituant seulement 2%-4% des bactéries totales pour éviter le chargement excessif de vacuoles de ciliate par traceur FLB (voir exemples de la figure 2).
- Inciler des échantillons de ciliate/plancton avec FLB pendant 5-15 min.
- Il y a deux possibilités pour la fixation d'échantillon pour empêcher l'égestion du matériel ingéré des vacuoles de nourriture des protistes8. Terminer les incubations par l'ajout de 1 % de glutaraldéhyde (concentration finale qui convient mieux aux échantillons contenant des particules contenant de la chlorophylle, comme les algues). Vous pouvez également utiliser 100 l/10 l de la solution de Lugol dans un sous-échantillon de 20 ml/200 l de sous-échantillon de liquide d'eau/piège, suivi immédiatement de l'ajout de 0,5 mL/10 l de formaline tamponnée de borate, puis de 200 l/2 l de thiosulfate de sodium de 3 % (Exp I/Exp II, respectivement).
- Après l'ajout du fixatif, laisser reposer les échantillons pendant au moins 1 h dans l'obscurité à 4 oC afin d'assurer une conservation complète des cellules ciliate.
- Prenez des sous-échantillons naturels de plancton de 4 à 30 mL/10-30 l (Exp I/Exp II, respectivement; le volume dépend de l'abondance du ciliate) et tachent avec dAPI (concentration finale de 0,2 % wt/vol; pour plus de détails, voir l'étape 3.2 ci-dessus).
- Passer à travers des filtres noirs de 1 m et inspecter par microscopie épifluorescence pour compter les ciliates (600x grossissement) et énumérer le nombre de traceurs FLB ingérés (principalement à 1000x grossissement) comme détaillé dans les publications précédentes2,17 . Inspecter les échantillons dans les 7 jours suivant la conservation.
- Pour estimer le nombre total de pâturages spécifiques au protozoaires et aux espèces, multipliez les taux moyens d'utilisation de tous les cigales, ou seulement des espèces de ciliate détectées par l'abondance in situ.
- L'exemple du calcul des taux d'apaisement par cellule à partir des données in situ du réservoir d'eau d'Omov est décrit comme suit :
- Supposons que la concentration bactérienne est de 3,55 x 106 bactéries/mL et traceur FLB ajouté est de 0,25 x 106 FLB/mL, ce qui donne une somme de 3,8 x 106 bactéries/mL de particules bactériennes totales (bactéries naturelles - FLB - 100% des particules de proie) disponibles pour les protistes phagotrophiques dans l'échantillon naturel.
REMARQUE : Les traceurs FLB ajoutés représentent ainsi 6,58 % (un projet de 0,25/0,038) du total des particules bactériennes. Le nombre moyen de FLB par Halteria sp. est de 6,2 FLB en 5 min d'incubations. - Pour normaliser l'utilisation par heure, utilisez le calcul suivant : (6,2 x 12)/(6,58/100) et 1131 bactéries par cellule Halteria/h.
REMARQUE : Pour plus d'exemples de distributions des taux d'apprepat individuels de Halteria sp. détectés sous des températures variables de l'eau, des quantités (en pourcentage) de TRACEur FLB ajouté, et des temps d'incubation différents avec FLB, voir Figure 3.
- Supposons que la concentration bactérienne est de 3,55 x 106 bactéries/mL et traceur FLB ajouté est de 0,25 x 106 FLB/mL, ce qui donne une somme de 3,8 x 106 bactéries/mL de particules bactériennes totales (bactéries naturelles - FLB - 100% des particules de proie) disponibles pour les protistes phagotrophiques dans l'échantillon naturel.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Exemple d'expérience Que j'ai été exécuté dans le réservoir d'eau d'Imov (Bohême du Sud, CZ), qui est un site naturel avec un prédateur naturel plus faible et l'abondance des proies. Des données représentatives sont rapportées pour l'espèce de ciliate omnivore Halteria grandinella, qui est un broussaillage abondant et efficace de particules picoplancton (lt;2 m)10,16,17,18 ,22. La figure 3 montre des parcelles de boîte et de fouet de nombres de FLB par cellule de Halteria sp. du réservoir d'Omov (figure 3A), qui a été recalculée aux taux d'apport bactérien par heure (Figure 3B) dans quatre expériences individuelles menées en avril, mai, août et septembre. Il y avait une grande variabilité dans les taux d'appréétien, en grande partie causée par les différences temporelles dans la température de l'eau.
Il convient de noter que le paramètre Q10 reflète le fait que les processus microbiens fonctionnent environ 2,5 fois plus rapidement avec une augmentation de la température de 10 oC12, qui tient également pour les taux d'uptake de ciliate sur les bactéries. Avec cette règle physiologique à l'esprit, des proportions considérablement différentes de FLB et les temps d'incubation ont été utilisés pour différentes saisons (pour plus de détails, voir Figure 3A). Ainsi, l'effet de température prévu a été compensé, et le réglage expérimental a donné des valeurs moyennes et médianes optimisées des taux d'utilisation approximativement entre 5-10 FLB par cellule ciliate. En général, ces quantités de FLB ingérés sont facilement décomptées (voir des exemples dans la figure 2, deux photographies gauches), générant des estimations précises du traceur (principalement entre 1-15 FLB par ciliate) taux d'admission. Cependant, en raison de la modification flB traceur ajouté (%) et différents moments d'incubation de l'échantillon, les valeurs absolues (exprimées en tant que nombre de bactéries pâturées de ciliate par heure) différaient considérablement (p 'lt; 0.01, test Kruskal-Wallis; suivi du test de comparaison multiple de Dunn, p 'lt; 0.05; voir des exemples dans Figure 3B) parmi les expériences. Les données illustrent également la variabilité naturelle typique des taux absolus de bactériorisdans les populations planctoniques de Halteria grandinella, avec une correspondance étroite de leurs valeurs moyennes et moyennes (figure 3).
En présence de ciliates bactérivorous très efficaces dans les échantillons, tels que les ciliates péririches, ils peuvent devenir fortement « sur-étiquetés » par FLB dans les quantités typiques de traceur de 5%-10% des bactéries totales (voir la photographie de droite à la figure 2). Cela peut fortement limiter la quantification précise du FLB ingéré. Dans de tels cas, il est suggéré d'exécuter des incubations parallèles supplémentaires avec seulement de faibles quantités de FLB représentant seulement 1,5%-3% des bactéries totales. Cependant, généralement, les quantités de traceurs ainsi que les temps d'incubation peuvent être manipulés pour optimiser le nombre de FLB par cellule (Figure 2).
Exemple d'expérience II: Les données d'un système avec de grandes abondances de prédateurs et de proies sont affichées, où seul un volume d'échantillon extrêmement faible est disponible pour estimer expérimentalement les taux de bactériorisation du ciliate Tetrahymena utriculariae 25. C'est un brouteur bactérien modéré vivant en haute abondance exclusivement dans des pièges des usines carnivores d'Utricularia reflexa 26,27. La figure 4 montre des parcelles de boîte et de fouet du nombre de FLB par cellule de T. utriculariae dans différents contextes expérimentaux (Figure 4A,B) qui est recalculée en taux d'utilisation bactérienne par heure (Figure 4 C,D) ) détectés dans les pièges jeunes, matures et anciens. Fait intéressant dans les pièges, des populations porteuses de chloroplaste du ciliate T. utriculariae ont été détectées, tandis que les populations apochloriques de T. utriculariae ont été isolées des pièges et maintenues sur la suspension bactérienne mixte grains de blé dans l'obscurité (pour plus de détails, voir Figure 1 dans une publication précédente26).
Les populations porteuses de chloroplaste vivent dans des pièges éclairés; ainsi, les chloroplastes peuvent fournir une source de carbone organique supplémentaire et de l'oxygène à l'hôte de ciliate. L'une des hypothèses testées était que les populations de ciliat apochlorique paissaient les bactéries beaucoup plus rapidement, car les bactéries représentent la seule source de particules de carbone organique disponible pour les sous-populations isolées cultivées dans les ténèbres du ciliat.
En effet, bien qu'il n'y ait pas eu de différences significatives dans les taux de bactériorisdes des cigales vivant dans des pièges jeunes, matures et anciens d'Utricularia reflexa (Figure 4A,C), les populations apochloriques de T. utriculariae pâlissaient bactéries significativement (p 'lt; 0.01, test Kruskal-Wallis; suivi par le test de comparaison multiple de Dunn, p 'lt; 0.05), environ 3x plus rapide que les ciliates chloroplast-bearing vivant dans les pièges jeunes, matures et vieux (Figure 4C,D). Notez que, encore une fois, les quantités de traceurs ainsi que les temps d'incubation(figure 4A,B, en haut) ont été modifiés pour optimiser le nombre de FLB par cellule (généralement entre 1-15), avec des valeurs moyennes et médianes autour de 5 FLB/ciliate. Ces nombres sont distingués dans les vacuoles de nourriture de ciliate et ont permis le comptage précis de traceur. Cependant, exprimées en nombre absolu de bactéries broutées par heure, les populations de chloroplastes et d'apochloriques broutaient environ 350 et 1 000 bactéries ciliate par heure, respectivement. Cette mise en place expérimentale a apporté de nouvelles perspectives sur les traits métaboliques et physiologiques de deux sous-populations distinctes de la même espèce de ciliate vivant sous des contraintes environnementales étonnamment différentes25,26, 27.
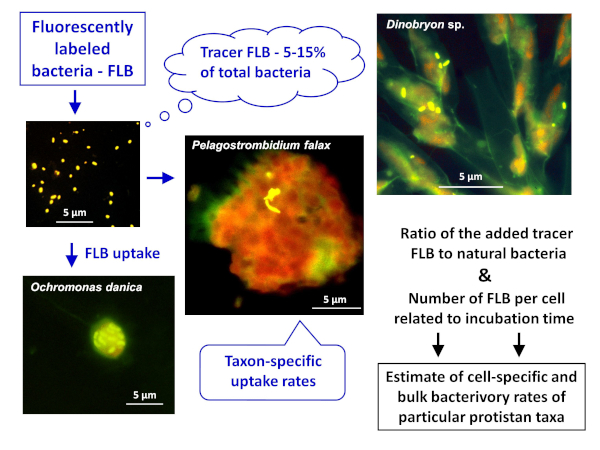
Figure 1 : Flux de travail d'utilisation de bactéries étiquetées fluorescentes (FLB) pour estimer les taux de pâturage spécifiques aux cellules et aux espèces à partir du rapport entre le traceur ingéré FLBet le nombre total de bactéries naturelles dans l'échantillon. Pour plus de détails, consultez la section 7 du protocole. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Exemples de cellules ciliate à partir du plancton d'un étang de poissons eutrophiques. Des exemples sont montrés de l'étang avec FLB countable dans les cellules ciliate (généralement 1-10 traceur FLB par cellule, les deux microphotograps gauches) comparés à un ciliate périssable Pelagovorticella natans (la microphotographie du côté droit). Même pendant une courte période d'incubation de 5 minutes, il est devenu « sur-étiqueté » par le traceur FLB, rendant la quantitation du FLB ingéré inexacte ou presqueimpossible. Dans ce cas, il est suggéré de diminuer le nombre de traceurs à 1,5%-3% du total des bactéries. Cependant, généralement, les quantités de traceurs et les temps d'incubation peuvent être manipulés pour optimiser le nombre de FLB ingérés par cellule. Pour plus de détails, consultez la section 7 du protocole. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3 : Parcelles de box-and-whisker du nombre de FLB par cellule de Halteria sp. du réservoir d'Omov (Exp I) (A), recalculées aux taux d'apport bactérien par heure (B). Les données ont été détectées dans différents milieux saisonniers, représentés par quatre exemples d'avril à septembre. Le haut du panneau A montre des informations sur la température de l'eau, les différents traceurs FLB ajoutés (%), et différents moments d'incubation de l'échantillon. Il convient de noter que ces deux derniers paramètres peuvent être modifiés pour optimiser le nombre de FLB par cellules, avec des valeurs moyennes (ligne complète) et médianes (ligne pointillée) environ entre 5-10 FLB par cellule ciliate (A). Les barres montrent les 25e et 75e percentiles de toutes les données (50-180 cellules inspectées) et les moustaches représentent les 1er et 99e percentiles. (B) Différentes petites lettres indiquent des différences significatives dans les taux de bactériique sp. spécifiques aux cellules au cours de la période étudiée. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : Parcelles de nombre de FLB par cellule. Des parcelles sont montrées des tetrahymena utriculariae chloroplast-bearing de trilaymena des traitements de triplicate des jeunes, mûrs, etvieux pièges d'Utricularia reflexa (Exp II) (A),recalculés aux taux d'apprisectie bactérienne par heure ( C ). Les données ont été comparées aux taux d'utilisation bactérienne des populations apochloriques en double de T. utriculariae (B,D) isolées des pièges, mais maintenues sur suspension bactérienne mixte poussant sur les grains de blé dans l'obscurité. Sur le dessus des panneaux A et B, différents traceurs FLB ajoutés (%) et différents moments d'incubation de l'échantillon sont affichés. Il convient de noter que ces deux derniers paramètres ont été modifiés pour optimiser le nombre de FLB par cellules, avec des valeurs moyennes (ligne complète) et médianes (ligne pointillée) environ entre 5-10 FLB par cellule ciliate (A,B). Les barres montrent les 5e et 95e percentiles de toutes les données (50-100 cellules inspectées), et les moustaches représentent les 1er et 99e percentiles. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Déchiffrer l'interaction trophique dans les systèmes aquatiques est toujours difficile28, en particulier à l'échelle nano-plancton impliquant les protistes et leurs proies, les bactéries. Quand il s'agit de voies d'absorption des nutriments et de quantification, l'application de méthodes utilisées avec succès à des niveaux trophiques plus élevés est moins possible, en raison de la grande complexité des interactions biotiques. Il s'agit, par exemple, d'approches stables d'étiquetage des isotopes. Ce protocole montre les avantages de l'utilisation de la microscopie épifluorescence et des bactéries étiquetées fluorescentes comme traceur pour suivre et semi-quantifier/estimer le flux de carbone (proiebactérienne : divers brouteurs protistan, y compris les brouteurs mixotrophiques29) à la base des réseaux alimentaires microbiens. L'un de ces avantages est la grande précision de l'approche unicellulaire, et l'autre est la résolution sans précédent concernant la structure de la communauté des brouteurs et la distinction entre les différentes guildes fonctionnelles, les espèces (Exp I), et même les sous-populations de la même espèces (Exp II).
Étapes critiques du protocole
Il y a plusieurs étapes critiques dans le protocole, qui peuvent s'assurer que les avantages de la méthodologie sont utilisés à leur plein potentiel. Tout d'abord, une compréhension de base de l'environnement étudié avant le début de l'expérience est toujours bénéfique. Cela comprend le dépistage microscopique de la diversité et de l'abondance des brouteurs potentiels présents, de la taille des proies bactériennes et de la répartition des proies 1) dans la colonne d'eau (p. ex., un profil vertical de l'épilimnion à l'hypolimnion) et 2) dans le cas de réservoirs en forme de canyon, sur le transect d'entrée de barrage. Deuxièmement, une manipulation minutieuse avec des échantillons prélevés assurera des résultats représentatifs. La température est un facteur extrêmement important affectant la plupart des processus microbiens12, y compris les taux de pâturage protiste (figure 3).
Troisièmement, la manipulation de la quantité de traceur ajoutée en fonction de la quantification des cellules bactériennes ou du type de brouteur dans l'échantillon permettra d'éliminer les problèmes de surétiquetage (figure 2). Il convient de noter qu'il existe un spectre très large dans les taux d'accumulation des espèces ciliate (pour plus de détails, voir l'étape 7.2); par conséquent, pour appliquer le protocole de façon appropriée, il est essentiel de connaître au préalable les principales espèces de ciliate avec leurs taux d'adoption dans le temps. Il est fortement conseillé d'exécuter des expériences préliminaires avec différentes quantités de traceurs afin d'éviter un éventuel sous-étiquetage du ciliat (aucun ou trop peu de FLB n'est pris par cellule ciliate, produisant des données statistiquement insalubres) ou un étiquetage excédentaire (apparaît comme un grand nombre de FLB formant des « nuages de FLB condensent » ou des troupeaux dans des vacuoles alimentaires ciliates emballés par les traceurs, limitant ainsi gravement leur quantification précise ; voir l'exemple supérieur à droite à la figure 2). Il convient également de noter que les temps d'incubation avec FLB sont généralement plus courts que 30 min, puisque le temps moyen de digestion du picoplancton par les cigales est d'environ 1,5 h, et la digestion commence (les cellules picoplancton ingérées perd sa forme et sa couleur typiques) environ après 45-60 min30. De même, une dilution et une distribution optimales des échantillons sur le filtre avant l'affichage microscopique doivent être obtenues pour obtenir des résultats précis.
Modifications et dépannage
Les principales étapes, les modifications possibles et les modifications de dépannage de la technique sont illustrées dans la figure 1 et la figure 2. En outre, il convient de noter que dans les cas de fortes concentrations de particules détritales, de cellules phytoplanctoniques ou de leurs colonies en plancton, ces échantillons 1) devraient être dilués en conséquence pour atteindre un stade où les cellules de brouteur saïdpeuvent peuvent être distingué sur la surface du filtre et 2) être soumis à la quantification du contenu des aliments vacuoles.
Limitations
La principale limitation pour l'application réussie de cette méthode réside dans la présence de divers détritus organiques ou de particules inorganiques/organiques abondantes avec des bactéries ou des agrégats attachés en quantités qui empêchent l'affichage d'échantillon clair sous l'épifluorescence microscope et l'estimation précise d'une quantité de traceur ajoutée. Il convient de noter que la technique de traceur présentée fonctionne principalement avec des bactéries libres (c.-à-d. en suspension) qui ne sont pas attachées aux particules. Cependant, sur la base de notre propre expérience et références littéraires (voir les publications précédentes2,4,8,10,16,18,21 ,26), la méthodologie présentée convient à la plupart des milieux aquatiques. Des exemples de deux systèmes naturels et contrastés qui diffèrent en matière de statut trophique, de teneur en détritus et de diversité et de nombres de broussailles sont fournis (figure 3 et figure 4).
Importance de l'approche par rapport aux méthodes existantes
Fait important, en connaissant l'abondance d'un taxon/taxon de bactérioristes et de leurs taux de bactérioris spécifiques à chaque espèce, on peut calculer le taux de bactériorique en vrac du taxon protistan (ou assemblage total de ciliate). Si cette approche est appliquée aux environnements naturels de plancton concomitant pour les flagelats hétérotrophes et les cilagtes (représentant les principaux brzers de bacterioplancton2,6,7), le protistan le temps de rotation induit par le pâturage des populations bactériennes dans un environnement donné peut être estimé16,17,18,22. Ces données sont d'une importance fondamentale pour l'estimation de la dynamique des flux de carbone dans les réseaux alimentaires microbiens.
Applications futures
Il existe d'autres environnements spécifiques dans lesquels cette méthode, avec quelques modifications, peut être utilisée avec succès. Il s'agit notamment des systèmes de boues activées, des écosystèmes rumen, des sédiments aquatiques et des étangs à poissons hypertrophiques17. Cependant, l'application dans ces environnements riches en nutriments et en microbes nécessite des tests préliminaires pour optimiser le protocole concernant la taille, la morphologie et le nombre de TRACEurs FLB qui peuvent imiter la distribution de taille typique et d'autres caractéristiques bactéries proies inhérentes à l'environnement.
À l'heure actuelle, on s'intéresse de plus en plus à la combinaison de cette approche avec l'hybridation in situ de la fluorescence in situ de dépôt de report de reporter catalysé (CARD-FISH), dans laquelle l'identité de la cellule de broussaillage (p. ex. flagellate hétérotrophique) est détectée avec un FISH-probe et le taux d'apprétulité est basé sur la teneur en FLB dans les vacuoles alimentaires de la cellule flagellate sur la même diapositive microscopique31. Une nouvelle approche sophistiquée appelée double hybridation32 est une combinaison de sondes FISH aux niveaux des cellules de prédateurs et des bactéries proies (qui sont également étiquetées spécifiquement par une souche phylogénétique, une sonde FISH spécifique à la lignée bactérienne). L'approche est élégante mais aussi longue et nécessite des compétences spécifiques et l'expérience31,32, tandis que l'application de diverses modifications flB approche d'adoption peut être plus facilement adopté pour une utilisation de routine dans les laboratoires.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Les auteurs n'ont rien à révéler.
Acknowledgments
Cette étude a été soutenue par la Fondation tchèque des sciences dans le cadre de la subvention de recherche 13-00243S et 19-16554S accordée set à K. et D. S., respectivement. Cet article a également été soutenu par le projet "Biomanipulation comme outil pour améliorer la qualité de l'eau des réservoirs de barrages" (No CZ.02.1.01/0.0/0.0/16-025/0007417), financé par le Fonds européen de développement régional, dans le domaine de la recherche, du développement et du développement des programmes opérationnels et l'éducation.
Materials
| Name | Company | Catalog Number | Comments |
| 0.2-µm pore-size filters | SPI supplies, https://www.2spi.com/ | B0225-MB | Black, polycarbonate track etch membrane filters, diameter approprite for filtering apparatus used |
| 5-(4,6-dichlorotriazin-2-yl) aminofluorescein (DTAF) | Any brand | ||
| Automatic pipettes with adjustable volumes | Any brand, various sizes | ||
| Centrifuge | 22 000 x g | ||
| Cryovials | Any brand, 2 mL size | ||
| DAPI (4´,6-Diamidino-2´-phenylindole dihydrochloride) | Any brand | 1 mg ml-1 | |
| Epiflorescence microscope | Magnification from 400 x up to 1000 x | ||
| Filters appropriate for viewing in the DAPI and DTAF range | |||
| Counting grid in one of the oculars | |||
| Filtering apparatus | Usually with a diameter of 25 mm | ||
| Formaldehyde | A brand for microscopy | ||
| Glutaraldehyde | A brand for microscopy | ||
| Immersion oil for microscopy | Specific oil with low fluorescence | ||
| Lugol´s solution | Any brand or see comment | Make an alkaline Lugol' solution as follows: Solution 1 - dissolve 10 g of potassium iodide in 20 ml in MQ water, then add 5 g of iodine. Solution 2 - add 5 g of sodium acetate to 50 ml of MQ water. Add the solution 2 to the solution 1 and thoroughly mix | |
| Methanol stabilized formalin | Any brand available for microscopy purposes | ||
| Microscope slides and cover slips | Any brand produced for microscopy purposes | ||
| MQ water for diluting samples | Any brand |
||
| Phosphate-buffered saline (PBS; pH = 9) | Any brand | 0.05 M Na2HPO4-NaCl solution, adjusted to pH 9 | |
| PPi-saline buffer | Any brand | 0.02 M Na4P2O7-NaCl solution. Add 0.53 g Na4P2O7 to 100 ml of MQ water plus 0.85 g NaCl | |
| Sampling device | Appropriate for obtaining representative sample | e.g. Friedinger sampler for lake plankton | |
| Sodium thiosulfate solution | Any brand | 3% solution is used in the protocol | |
| Sonicator | Any brand | 30 W | |
| Vortex | Any brand allowing thorough mixing of the solutes and samples | ||
| Water bath | Any brand allowing temperature to be maintained at 60 °C |
References
- Azam, F., et al. The ecological role of water-column microbes in the sea. Marine Ecology Progress Series. 10, 257-263 (1983).
- Šimek, K., et al. A finely tuned symphony of factors modulates the microbial food web of a freshwater reservoir in spring. Limnology & Oceanography. 59, 1477-1492 (2014).
- Šimek, K., et al. Bacterial prey food characteristics modulate community growth response of freshwater bacterivorous flagellates. Limnology & Oceanography. 63, 484-502 (2018).
- Šimek, K., et al. Changes in bacterial community composition, dynamics and viral mortality rates associated with enhanced flagellate grazing in a meso-eutrophic reservoir. Applied & Environmental Microbiology. 67, 2723-2733 (2001).
- Jürgens, K., Matz, C. Predation as a shaping force for the phenotypic and genotypic composition of planktonic bacteria. Antonie Van Leeuwenhoek. 81, 413-434 (2002).
- Pernthaler, J. Predation on prokaryotes in the water column and its ecological implications. Nature Reviews Microbiology. 3, 537-546 (2005).
- Berninger, U. B., Finlay, J., Kuuppo-Leinikki, P. Protozoan control of bacterial abundances in freshwaters. Limnology and Oceanography. 36, 139-147 (1991).
- Sherr, E. B., Sherr, B. F. Protistan grazing rates via uptake of fluorescently labeled prey. Handbook of Methods in Aquatic Microbial Ecology. Kemp, P. F., Sherr, B. F., Sherr, E. B., Cole, J. J. , Lewis Publishers. Boca Raton, Florida. 695-701 (1993).
- Vazquez-Dominguez, E., Peters, F., Gasol, J. M., Vaqué, D. Measuring the grazing losses of picoplankton: methodological improvements in the use of fluorescently tracers combined with flow cytometry. Aquatic Microbial Ecology. 20, 119-128 (1999).
- Šimek, K., et al. Ecological role and bacterial grazing of Halteria spp.: Small oligotrichs as dominant pelagic ciliate bacterivores. Aquatic Microbial Ecology. 22, 43-56 (2000).
- Montagnes, D. J. S., et al. Selective feeding behaviour of key free-living protists: avenues for continued study. Aquatic Microbial Ecology. 53, 83-98 (2008).
- Kirchman, D. L. Processes in Microbial Ecology. 2nd Edition. , Oxford University Press. Oxford, UK. (2018).
- Porter, K. G., Feig, Y. S. The use of DAPI for identifying and counting aquatic microflora. Limnology and Oceanography. 25, 943-948 (1980).
- Foissner, W., Berger, H. A user-friendly guide to the ciliates (Protozoa, Ciliophora) commonly used by hydrobiologists as bioindicators in rivers, lakes, and waste waters, with notes on their ecology. Freshwater Biology. 35, 375-482 (1996).
- Foissner, W., Berger, H., Schaumburg, J. Identification and ecology of limnetic plankton ciliates. Informationsberichte des Bayer Landesamtes für Wasserwirtschaft Heft. , 3-99 (1999).
- Šimek, K., et al. Ciliate grazing on picoplankton in a eutrophic reservoir during the summer phytoplankton maximum: a study at the species and community level. Limnology & Oceanography. 40, 1077-1090 (1995).
- Skibbe, O. An improved quantitative protargol stain for ciliates and other planktonic protists. Archiv für. Hydrobiolgie. 130, 339-347 (1994).
- Macek, M., et al. Growth rates of dominant planktonic ciliates in two freshwater bodies of different trophic degree. Journal of Plankton Research. 18, 463-481 (1996).
- Šimek, K., et al. Microbial food webs in hypertrophic fishponds: omnivorous ciliate taxa are major protistan bacterivores. Limnology & Oceanography. , in press (2019).
- Jezbera, J., et al. Major freshwater bacterioplankton groups: Contrasting trends in distribution of Limnohabitans and Polynucleobacter lineages along a pH gradient of 72 habitats. FEMS Microbiology Ecology. 81, 467-479 (2012).
- Kasalický, V., et al. The diversity of the Limnohabitans genus, an important group of freshwater bacterioplankton, by characterization of 35 isolated strains. PLoS One. 8, 58209 (2013).
- Stabell, T. Ciliate bacterivory in epilimnetic waters. Aquatic Microbial Ecology. 10, 265-272 (1996).
- Zingel, P., et al. Ciliates are the dominant grazers on pico- and nanoplankton in a shallow, naturally highly eutrophic lake. Microbial Ecology. 53, 134-142 (2007).
- Bickel, S. L., Tang, K. W., Grossart, H. P. Ciliate epibionts associated with crustacean zooplankton in german lakes: distribution, motility, and bacterivory. Frontiers in Microbiology. 3 (243), (2012).
- Sirová, D., et al. Hunters or gardeners? Linking community structure and function of trap-associated microbes to the nutrient acquisition strategy of a carnivorous plant. Microbiome. 6, 225 (2018).
- Šimek, K., et al. Ecological traits of a zoochlorellae-bearing Tetrahymena sp. (Ciliophora) living in traps of the carnivorous aquatic plant Utricularia reflexa. Journal of Eukaryotic Microbiology. 64, 336-348 (2017).
- Pitsch, G., et al. The green Tetrahymena utriculariae n. sp. (Ciliophora, Oligohymenophorea) with its endosymbiotic algae (Micractinium sp.), living in the feeding traps of a carnivorous aquatic plant. Journal of Eukaryotic Microbiology. 64, 322-335 (2017).
- Nielsen, J. M., Clare, E. L., Hayden, B., Brett, M. T., Kratina, P. Diet tracing in ecology: Method comparison and selection. Methods in Ecology and Evaluation. 9, 278-291 (2018).
- Beisner, B. E., Grossart, H. P., Gasol, J. M. A guide to methods for estimating phago-mixotrophy in nanophytoplankton. Journal of Plankton Research. , 1-13 (2019).
- Dolan, J. D., Šimek, K. Processing of ingested matter in Strombidium sulcatum, a marine ciliate (Oligotrichida). Limnology and Oceanography. 42, 393-397 (1997).
- Massana, R., et al. Grazing rates and functional diversity of uncultured heterotrophic flagellates. The ISME Journal. 3, 588-596 (2009).
- Grujčić, V., et al. Cryptophyta as major freshwater bacterivores in experiments with manipulated bacterial prey. The ISME Journal. 12, 1668-1681 (2018).



