Summary
Um die Wirkung eines Chemokins auf die Makrophagenrekrutierung in vivo zu testen, wurde die gesamte Mount-in-situ-Hybridisierung verwendet, um die ektopische Expression des Chemokins zu erkennen, und Immunstaining wurde verwendet, um Makrophagen zu kennzeichnen. Live-Bildgebung wurde für die Echtzeitbeobachtung der Makrophagenmigration verwendet.
Abstract
Zebrafische sind weit verbreitet in der Grundlagen- und biomedizinischen Forschung verwendet. Viele transgene Zebrafische sind derzeit verfügbar, um verschiedene Arten von Zellen zu kennzeichnen. Aufgrund des transparenten embryonalen Körpers von Zebrafischen ist es für uns bequem, die Wirkung eines Chemokins auf das Verhalten einer bestimmten Art von Zellen in vivo zu untersuchen. Hier haben wir einen Workflow zur Untersuchung der Funktion eines Chemokins zur Makrophagenmigration in vivo bereitgestellt. Wir konstruierten ein gewebespezifisches Überexpressionsplasmid, um IL-34 zu überexpressen und injizierten das Plasmid in transgene Fischembryonen im Einzellstadium, deren Makrophagen speziell durch ein fluoreszierendes Protein gekennzeichnet wurden. Wir verwendeten dann ganze Mount-Fluoreszenz-In-situ-Hybridisierung und Immunostaining, um das Muster der Chemokin-Expression und die Anzahl oder Position von Makrophagen zu erkennen. Die injizierten WT-Embryonen wurden erhöht, um eine stabile transgene Linie zu erzeugen. Schließlich verwendeten wir konfokale Live-Bildgebung, um das Makrophagenverhalten in den stabilen transgenen Fischen direkt zu beobachten, um die Funktion von IL-34 auf Makrophagen in vivo zu untersuchen.
Introduction
Zebrafisch ist ein kleiner tropischer Süßwasserfisch aus Indien. In Bezug auf die Generhaltung haben Zebrafische eine Ähnlichkeit von 87% mit dem Menschlichen1. Es kann uns Einblicke zu verwandten Themen des Menschen geben, indem es die Genregulation, Proteinfunktion und Zellverhalten wie Migration, Proliferation et.al in Zebrafischen untersucht. Zebrafisch-Embryo kann verwendet werden, um die Entwicklung von frühen Embryonen in verschiedenen Stadien nach der Hemmung des Pigments zu beobachten. Inzwischen dauert es nur drei Monate, bis sich Zebrafische zu einer Geschlechtsreife entwickeln, dann kann der Zebrafisch alle 4 Tage Hunderte von Eiern produzieren. Mini-Größe, einfache Zucht, starke Fortpflanzungsfähigkeit, diese Vorteile machen Zebrafischkultur sehr platzsparend, förderlich für großflächige Kultur. Die traditionelle Säugetiermodellmaus hat höhere Wartungskosten als Zebrafische, wodurch der Umfang des Anhebens von Mäusen begrenzt wird. Im Bereich der frühen Embryoentwicklung ist der Mausembryon aufgrund der Eigenschaften der Entwicklung von Mausembryonen im Mutterleib schwer im Leben zu beobachten. Im Gegenteil, Zebrafischembryonen entwickeln sich äußerlich und sind transparent, daher sind sie unter dem Mikroskop leicht zu beobachten. Darüber hinaus ist Zebrafisch sehr einfach, eine Vielzahl von transgenen Linien für die verwandte Genfunktionsforschung zu konstruieren. Derzeit stehen verschiedene transgene Zebrafische zur Verfügung, um verschiedene Zelltypen zu kennzeichnen. Es ist jetzt sehr praktisch, transgene Linien zu konstruieren, um Chemokine an bestimmten Stellen zu überexprimieren und die Chemokinfunktion auf zellzelliges Verhalten bei Zebrafischen zu untersuchen.
Hier haben wir einen Workflow zur Verwendung von Zebrafisch transgene Linie zur Untersuchung der Funktion von IL-34 auf Makrophagen Verhalten in vivo2,3,4,5,6,7. Zunächst konstruierten wir ein leberspezifisches Überexpressionsplasmid des Gens il34 und injizierten das Plasmid in einzellige Tg-Embryos (mpeg1: GFP), die die Makrophagen durch das fluoreszierende Protein GFP spezifisch kennzeichnen. Dann verwendeten wir ganze Mount-Fluoreszenz-In-situ-Hybridisierung und Immunostainierung, um das Muster des il34-Ausdrucks und die Anzahl oder Position von Makrophagen zu erkennen. Die injizierten WT-Embryonen wurden erhöht, um eine stabile transgene Linie zu erzeugen. In diesen Schritten haben wir die Zytokin-produzierende Linie etabliert und validiert und die Auswirkungen, die auf die Makrophagenverteilung zu sehen sind, visuell bewertet. Schließlich haben wir zur Untersuchung des Makrophagenverhaltens als Reaktion auf das Zytokin konfokale Live-Bildgebung verwendet, um die Makrophagenmigration direkt zu beobachten, um die Funktion von il34 auf der Makrophagenmigration in vivo zu bestätigen.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
HINWEIS: Alle Proben wurden mit Phenylthiourea (PTU) Eiwasser behandelt, um Pigment zu hemmen.
1. Erzeugung von Tg (fabp10a:il34) Transgene Konstrukte und Fischinjektion
- Klonen Sie den 2.8 kb fabp10a-Promotor 8 und die IL-34-Kodierungsbereiche (ENSDART00000126460.3) von Zebrafischen in den pTol2-Vektor, um das fabp10a-il34-Konstrukt zu erzeugen. Injizieren Sie die Konstrukte in einZellstadium Tg (mpeg1: GFP) und WT-Fischembryonen zusammen mit der Transposase mRNA. Erhöhen Sie die fabp10a-il34 injizierten WT-Embryonen auf Erwachsene9 und identifizieren Sie den transgenen Gründer durch In-situ-Hybridisierung.
HINWEIS: Die Injektion des Tol2-Konstrukts direkt in ein anderes transgenes Konstrukt könnte problematisch sein, wenn die andere transgene Linie mit demselben Transposonsystem hergestellt wird. Eine allgemeine Praxis wäre es, eine unabhängige transgene Linie zu machen und anschließend die neue Linie mit einer anderen Reporterlinie zu überqueren. Dadurch wird sichergestellt, dass die neue Transgenese keine Auswirkungen auf ein zuvor eingefügtes Transgen hat.
2. Fluoreszierende Whole Mount In Situ Hybridisierung (WISH) kombinieren mit Immunostainierung
- Probenfixierung
- Sammeln Sie Embryonen der transienten Injektion oder stabile TRANSgene IL-34-Linie, die mit Tg (mpeg1: GFP) in gewünschten Stadien kreuzte.
HINWEIS: In diesem Fall wurden die Embryonen bei 4 d nach der Befruchtung (dpf) gesammelt. (Falls erforderlich) entfernen Sie die Chorion mit der Spritze. - Fixieren Sie die Embryonen mit 4% Paraformaldehyd (PFA) über Nacht bei 4 °C oder 2 h bei Raumtemperatur (RT) (ca. 25 °C).
- Waschen Sie die Embryonen mit Phosphat gepufferter Saline plus Tween 20 (PBST) 3x 5 min.
- Dehydrieren Sie die Embryonen separat mit 50% Methanol in PBST (50% Methanol/PBST) und 100% Methanol, jeweils 1x 5 min. Dann zu frischem 100% Methanol wechseln und bei -20 °C (mindestens 2 h) lagern.
HINWEIS: Das Protokoll kann hier angehalten werden.
- Sammeln Sie Embryonen der transienten Injektion oder stabile TRANSgene IL-34-Linie, die mit Tg (mpeg1: GFP) in gewünschten Stadien kreuzte.
- Probehybridisierung (Tag I)
- Rehydrieren Sie die Embryonen in den vorherigen Schritten mit 50% Methanol in PBST (50% Methanol/PBST), dann mit PBST 3x 5 min waschen.
- Verdauen Sie die Embryonen mit Proteinase K in PBST bei RT (Endkonzentration: 10 g/ml; 1:2000 in PBST).
HINWEIS: Die Verdauungszeit hängt vom Embryostadium ab: Weniger als 36 h nach der Befruchtung (hpf), keine Notwendigkeit; 36 hpf-2 dpf Embryo, 3-5 min; 2-3 dpf Embryo, 10 min; 3-4 dpf Embryo, 15 min; 4-5 dpf Embryo, 15-20 min; 5-6 dpf Embryo, 20-27 min; >6 dpf Embryo, 25-30 min bei RT (ca. 25 °C). - Entsorgen Sie die Verdauungslösung und führen Sie die Fixierung erneut mit 4% PFA, für 20 min bei RT durch.
- Waschen Sie die Embryonen mit PBST 2x 10 min.
- Entsorgen Sie den PBST, führen Sie die Vorhybridisierung mit beheiztem Hybridisierungspuffer (HB-Puffer) bei 65 °C für 5 min durch, recyceln Sie den HB-Puffer in das Originalrohr.
- Führen Sie die Vorhybridisierung mit einem neuen beheizten HB-Puffer bei 65 °C mindestens 1 h durch.
- Vorheizen der Sonde9 (für diesen Fall war eine il34 Sonde, 1 ng/ml) bei 65 °C mindestens 10 min. Dann den HB-Puffer in das Originalrohr recyceln. Führen Sie die Hybridisierung mit der vorgeheizten Sonde bei 65 °C über Nacht durch.
- Antikörperbehandlung (Tag II)
- 50% Formamid/2x Saline-Natriumcitrat plus Tween 20 (SSCT), 2x SSCT, 0,2x SSCT bei 65 °C vorheizen.
- Recyceln Sie die Sonde in das Originalrohr und lagern Sie die Sonde bei -20 °C.
- Waschen Sie die Embryonen separat mit 50% Formamid/2x SSCT; 2x SSCT; 0.2x SSCT, 3x 20 min oder 2x 30 min je bei 65 °C.
- Waschen Sie die Embryonen mit PBST 3x 5 min.
- Blockieren Sie die Proben mit 600 l Blockierpuffer (5% gefiltertes fetales Rinderserum (FBS) in PBST) für 1 h bei RT.
- Fügen Sie 400 l Anti-Digoxigenin-HRP-Antikörperlösung (1:1.000-1:2.000 im Sperrpuffer) hinzu und inkubieren Sie die Embryonen über Nacht bei 4 °C. Wenn die Signale schwach sind, verwenden Sie 1:500 Verdünnung des Antikörpers.
- Färbung und Primärantikörperinkubation (Tag III)
- Entfernen Sie den Antikörper; waschen Sie die Embryonen mit PBST, 6x 20 min bei RT.
- Spülen Sie die Probe mit 30 l 1x Plus Ampliflifliflifluent für 5 min bei RT.
- Entsorgen Sie das Verdünnungsmittel durch Pipettieren; verdünnt Fluorophor Tyramid-Lagerlösung (Cyanin 3 Plus Amplification Reagenz (Cy3) oder Cyanin 5 Plus Amplification Reagenz (Cy5), für diesen Fall wurde Cy3 verwendet) 1:50 in 1x Plus Ampliflification Diluent zur Herstellung der Fluorophor Tyramid-Arbeitslösung. Bereiten Sie für jede Probe 50-100 l Arbeitslösung vor.
- Inkubieren Sie die Probe in der Fluorophor-Tyramid-Arbeitslösung für 5-15 min im Dunkeln bei RT. Wenn Signale schwach sind, verlängern Sie die Inkubationszeit auf 30 min.
- Beenden Sie die Reaktion, indem Sie die Arbeitslösung mit PBST ändern und die Signale untersuchen.
- Waschen Sie die Embryonen mit PBST 3x 10 min bei RT.
- Inkubieren Sie die Probe mit primärem Antikörper bei 4 °C über Nacht. Verwenden Sie in diesem Fall den Goat-Anti-GFP-Antikörper als primären Antikörper.
- Sekundäre Antikörperfärbung (Tag IV)
- Waschen Sie die Embryonen mit PBST für 4x 30 min.
- Die Embryonen mit sekundärem Antikörper bei 4 °C über Nacht inkubieren. Verwenden Sie in diesem Fall Alexa 488-Anti-Goat Antikörper als sekundären Antikörper.
- Fotografieren (Tag V)
- Waschen Sie die Embryonen mit PBST 3x 10 min bei RT.
- Lagern Sie die Embryonen in 70% Glycerin in Dunkelheit bei 4 °C über Nacht oder -20 °C länger.
3. Live Imaging
-
Beispielauswahl
HINWEIS: Verwenden Sie das Live-Bild, um direkt zu beobachten, ob Makrophagen von Tg (fabp10a: il34; fabp10a: DsRed; mpeg1: GFP) Fische unter IL-34-Induktion während 3-3.5 dpf in die Leber wandern würden. Hier wird die transgene Linie Tg (fabp10a-DsRed) verwendet, um die Leberregion zu kennzeichnen und sichtbar zu machen, die Lokalisierung der Leber zu erleichtern und festzustellen, ob Makrophagen in die Leber wandern. Verwenden Sie vor der Bildgebung ein Fluoreszenzmikroskop, um die doppelt positiven Embryonen DsRed und GFP auszuwählen. -
Fischbefestigung
- Verwenden Sie ein Metallbad, um 1 ml 1% niedrigschmelzende Agarose auf über 90 °C zu erhitzen, um es vollständig zu schmelzen.
- Nachdem die niedrigschmelzende Agarose auf Körpertemperatur abgekühlt ist, 50 l 0,2% Tricain hinzufügen und Tricain gleichmäßig mit der Agarose vermischen.
- Bewegen Sie die anästhesierten Embryonen auf eine kleine Schale mit einer Abdeckung Gleiten auf dem Boden montiert, entfernen Sie das umgebende Wasser, langsam fallen die niedrig schmelzende Agarose auf die Embryonen, sorgfältig die Position der Fische einstellen, bevor die Agarose verfestigt wird, halten Sie den Leberbereich in der Nähe die Abdeckung gleiten auf der Unterseite der Schale.
- Nachdem die niedrigschmelzende Agarose verfestigt ist, bedecken Sie sie vorsichtig mit einer weiteren Schicht Von Agarose, um sie zu verstärken.
- Legen Sie die Schale auf den konfokalen Mikroskopträgertisch, bedecken Sie den Fisch mit der E2-Lösung10 mit Tricain und beginnen Sie mit der Bildgebung.
-
Softwarebetrieb des Konfokalmikroskops
- Öffnen Sie die SOFTWARE ZEN black 2.3, installieren Sie die lebende Zell-Workbench auf den Mikroskop-Trägertisch.
- Klicken Sie auf Suchen | Inkubation | Temperatur, um die Temperatur auf 29 °C einzustellen.
- Legen Sie die Schale in die Mitte der lebenden Zelle Werkbank, decken Sie den Fisch mit der E2-Lösung10 mit Tricain.
- Klicken Sie auf das Menü Erfassung, wählen Sie den erforderlichen Scanmodus und die Laser im Menü Smart Setup aus, und wählen Sie dann Z-Stack und Positionaus.
- Klicken Sie auf das Menü Experiment-Designer, wählen Sie Multi Block Experimentaktivieren im ersten Block aus, um das Beispiel unter der niedrigen Vergrößerung zu finden, und wechseln Sie dann zur hohen Vergrößerung, lassen Sie den beobachteten Bereich in der Mitte des Gesichtsfelds.
- Legen Sie die Position und die Z-Stack-Informationen fest, wählen Sie die entsprechende Laserintensität, Scanschichten und Bildgeschwindigkeit aus.
- Erstellen Sie einen neuen Block, und wiederholen Sie die obigen Schritte. Legen Sie nach dem Einrichten aller Blöcke die entsprechende Anzahl von Schleifen fest und starten Sie die Aufzeichnung (Abbildung 1).
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Die Schritte im Protokoll der Zebrafische sind in Abbildung 2dargestellt. Zuerst haben wir das pBLK-fabp10a-il34-sv40 Konstrukt generiert, in dem il34 vom promotorium enwerter Bauart "fabp10a" angetrieben wurde (Abbildung 2). Das Konstrukt wurde mikroinjiziert in ein-Zell-Stadium Tg (mpeg1: GFP) Zebrafisch-Embryonen, die Makrophagen mit GFP- und WT-Embryonen kennzeichnen können, die zu Erwachsenen erhoben wurden, um transgene stabile Linie zu erzeugen (Abbildung 2). Die Expression von il34 wurde durch die gesamte Mount-Fluoreszenz-In-situ-Hybridisierung analysiert (Abbildung 2 und Abbildung 3). Makrophagen, die durch GFP gekennzeichnet waren, wurden durch Immunfärbung analysiert (Abbildung 2 und Abbildung 3). Wir verwendeten Live-Bildgebung, um direkt zu beobachten, ob Makrophagen unter der il34-Induktion während 3-3,5 dpf in die Leber wandern würden ( Abbildung2, Abbildung 4, Ergänzender Film 1 und Ergänzender Film 2) .
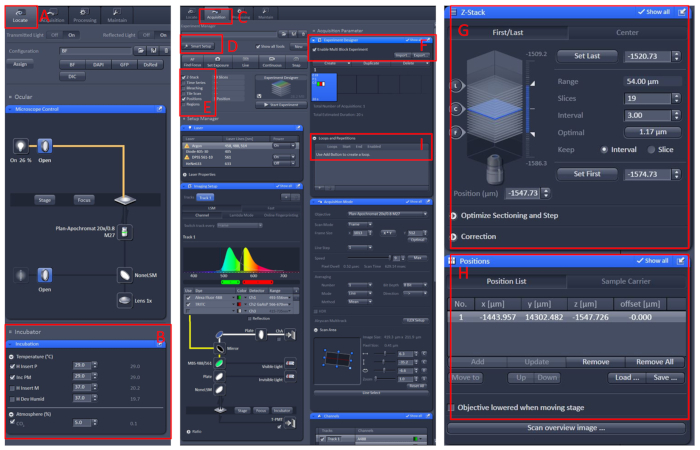
Abbildung 1: Softwarebetrieb des konfokalen Mikroskops Live-Bildgebung. Öffnen Sie die ZEN black 2.3 Software, installieren Sie die lebende Zell-Workbench auf dem Mikroskop-Trägertisch, und klicken Sie dann auf Suchen (A) | Inkubation | Temperatur (B), um die Temperatur auf 29 °C einzustellen. Legen Sie die Schale in die Mitte der lebenden Zelle Werkbank, decken Sie den Fisch mit der E2-Lösung10 mit Tricain. Klicken Sie nach all diesen Ausweise auf das Menü "Erfassung" (C), wählen Sie den erforderlichen Scanmodus und die Laser im Menü Smart Setup (D) aus, und wählen Sie dann Z-Stack und Position (E) aus. Klicken Sie schließlich auf das Menü Experiment Designer (F), wählen Sie im ersten Block das Multiblock-Experiment aktivieren,um die Probe unter der niedrigen Vergrößerung zu finden, und wechseln Sie dann zur hohen Vergrößerung, lassen Sie den beobachteten Bereich im Mitte des Gesichtsfeldes, stellen Sie die Position und Z-Stack (G und H) Informationen, wählen Sie die entsprechende Laserintensität, Scan-Schichten und Bildgebungsgeschwindigkeit. Erstellen Sie einen neuen Block, und wiederholen Sie die obigen Schritte. Nachdem Sie alle Blöcke eingerichtet haben, legen Sie die entsprechende Anzahl von Schleifen (I) fest und starten Sie die Aufnahme. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Ein Workflow zur Untersuchung der Funktion eines Chemokins bei der Makrophagenmigration in vivo. Wir konstruierten ein gewebespezifisches (Leber-) Überexpressionsplasmid, um IL-34 zu überexpressen, und injizierten das Plasmid in transgene Fischembryonen im Einzellstadium, deren Makrophagen speziell durch ein fluoreszierendes Protein gekennzeichnet wurden (Tg: (mpeg1: GFP )). Die injizierten WT-Embryonen wurden erhöht, um eine stabile transgene Linie zu erzeugen. Wir verwendeten dann ganze Mount-Fluoreszenz-In-situ-Hybridisierung und Immunostaining, um das Muster der Genexpression und die Anzahl oder Position der Makrophagen der transientinjizierten Embryonen oder stabilen Linienembryonen (4 dpf) zu erkennen. Schließlich verwendeten wir konfokale Live-Bildgebung, um das Makrophagenverhalten bei den stabilen transgenen Fischen (3-3,5 dpf) direkt zu beobachten, um die Funktion von IL-34 auf Makrophagen in vivo zu untersuchen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Fluoreszierende WISH kombinieren mit Immunostainierung. Diese Zahl wurde von Jiang et al.11geändert. Insgesamt 1,8 nL (30 ng/l) des pBLK-fabp10a-il34-sv40-Konstrukts wurden mikroinjiziert in einzellige Tg(mpeg1: GFP) Zebrafischembryonen. (A) WISH von il34-Expression (rot) und vollwertige Antikörperfärbung der GFP-Expression (grün) in 4 dpf Embryo (6x). Das Ganzkörperbild des Fisches besteht aus zwei getrennten Bildern, die von konfokalen und in Photoshop zusammengenähten Bildern aufgenommen wurden. Eintsets sind eine hohe Vergrößerung (20x) der entsprechenden geschachtelten Bereiche (orange gepunktete Bereiche). (B) Quantitative Analyse der Makrophagenzellzahlen in nicht injizierter und konstruktiv injizierter Embryosleber (im weiß gepunkteten Bereich dargestellt) und Schwanzbereich (ungefähr zwischen dem 13. und 17. Die Daten wurden durch Mann Whitney U Test analysiert, ** p < 0.01 im Vergleich zur Kontrolle. n = 5, 5 für die 4 dpf injizierten und kontrollieren den Fisch. Stäbe: 200 m (weiße Linie); 50 m (gelbe Linie). (C) WISH der il34-Expression und vollwertige Antikörperfärbung der GFP-Expression in 4 dpf stabilen Linienembryon (6x). Das Ganzkörperbild des Fisches besteht aus zwei getrennten Bildern, die von konfokalen und in Photoshop zusammengenähten Bildern aufgenommen wurden. Eintsets sind eine hohe Vergrößerung (20x) der entsprechenden geschachtelten Bereiche (orange gepunktete Bereiche). (D) Quantitative Analyse der Makrophagenzellzahlen in Tg (mpeg1: GFP) und Tg (fabp10a: il34; mpeg1: GFP) EmbryosLeber (im weiß gepunkteten Bereich dargestellt) und Schwanzbereich (ungefähr zwischen dem 13. und 17. Somite, dargestellt zwischen zwei weißen gepunkteten Linien). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Konfokale Live-Bildgebung zur direkten Beobachtung des Makrophagenverhaltens bei den stabilen transgenen Fischen. Diese Zahl wurde von Jiang et al.11geändert. Mikrographien der Live-Bildgebung zeigen den Prozess eines Makrophagens (grün, durch weiße Pfeile beschriftet), das innerhalb von 28 min an der Leber vorbeigeht (rot) in Kontrollfischen (A) und dem Prozess eines Makrophagens (grün, mit weißen Pfeilen beschriftet), das innerhalb von 28 in die Leber (rot) wandert. min in IL-34 überextierbaren Fischen (B). Skalenbalken = 40 m (weiße Linie). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Ergänzender Film 1: Live-Bildgebung, die den Prozess von Makrophagen (grün, mit weißen Pfeilen beschriftet) zeigt, die innerhalb von 2 h in IL-34-Überextierkungsfischen in die Leber (rot) wandern. Skalenbalken = 20 m (weiße Linie). Dieser Film wurde von Jiang et al.11wiederveröffentlicht. Bitte klicken Sie hier, um dieses Video anzuzeigen. (Rechtsklick zum Download.)

Ergänzender Film 2: Live-Bildgebung, die den Prozess von Makrophagen (grün, mit weißen Pfeilen beschriftet) zeigt, die innerhalb von 2 h in Kontrollfischen um die Leber (rot) wandern. Skalenbalken = 20 m (weiße Linie). Dieser Film wurde von Jiang et al.11wiederveröffentlicht. Bitte klicken Sie hier, um dieses Video anzuzeigen. (Rechtsklick zum Download.)
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Das hier beschriebene Protokoll ermöglicht es uns, die Funktion eines Chemokins auf das Verhalten von Macrophagein vivo zu untersuchen, und das Verfahren erfordert einige technische Servierkenntnisse. Zusammenfassend gibt es mehrere kritische Schritte, um Komplikationen im Protokoll zu vermeiden: 1) Wählen Sie eine geeignete transgene Linie, die spezifisches und starkes transgenes Signal zeigt, um die Zelle von Interesse zu kennzeichnen; 2) wählen Sie ein geeignetes Gewebe, das für die Bildgebung und transgene Genüberexpression zugänglich ist; 3) eine empfindliche und spezifische RNA-Sonde zu machen; 4) Wählen Sie ein geeignetes Beobachtungszeitfenster aus, um das Zellverhalten genau zu erfassen.
Bei der Operation der gesamten Mount-Fluoreszenz-In-situ-Hybridisierung in Kombination mit Immunostainierung sollte die RNA-Sonde, die zum Nachweis der Genexpression verwendet wird, empfindlich sein und das Signal muss stark genug sein. Um die Genfunktion auf Zellverhalten zu erfassen, sollte eine Reihe von Zeitpunkten getestet werden. Zum Beispiel, bei der Beobachtung der Wirkung von il34 auf Makrophagen Migration, Obwohl der fabp10a Promotor begann, bei 2-3 dpf auszudrücken, Makrophagen Akkumulation in der Leber war nicht offensichtlich zu dieser Zeit. Erst durch 4 dpf wird die Anreicherung von Makrophagen in der Leber sichtbar. Darüber hinaus wird nach der In-situ-Hybridisierung die Signalintensität der nachfolgenden Immunfärbung beeinflusst. Im Vergleich zu GFP ist DsRed beispielsweise schwierig, die Immunfluoreszenzfärbung nach in situ-Hybridisierung zu färben, wahrscheinlich aufgrund der unterschiedlichen Proteinstrukturen. Im Allgemeinen wäre die Signalintensität der Immunfärbung nach der fluoreszenz-in-situ-Hybridisierung geringer als die der Einzelimmunfärbung.
Im Live-Bildschritt mit dem konfokalen Mikroskop ist es notwendig, die Probe in der Nähe des Bodens der Schale zu halten. Wenn die Embryonen in Agarose schweben, kann der Arbeitsabstand des Objektivs nicht ausreichen, auch die Agarose zwischen dem Objektiv und der Probe würde die Qualität der Bildgebung beeinträchtigen. Außerdem sollte die Anzahl der Proben für die Bildgebung zu jeder Zeit richtig eingestellt werden. Man muss sicherstellen, dass die Zeitspanne zwischen zwei Scans jedes Fisches nicht zu lang wäre, um die Details des Zellverhaltens zu verlieren. Diese Methode eignet sich daher nicht für die Verfolgung von Zellen, die sich schnell in dicken Geweben bewegen.
Zusammenfassend kann dieses Protokoll verwendet werden, um die Funktion von Chemokinen auf das Verhalten einer Vielzahl von Zellen wie Makrophagen, Neutrophile und T-Zellen zu beobachten. Hier verwendeten wir IL-34, einen kürzlich identifizierten Liganden der CSF-1R-Funktion in Chemotaxis6,7, als ektopisch exprimiertes Chemokin, um Makrophagenmigration zu induzieren. Die meisten der bestehenden experimentellen Modelle von Zellchemotaxis basieren auf In-vitro-Zellexperimenten, aber die In-vitro-Experimente sind manchmal zu einfach, um die komplexe Umgebung in vivo zu modellieren. Auch ist es schwierig, die Chemo-Anziehungsfähigkeit in vivo abzubilden, wenn man sich einfach die In-vitro-Situation anschaut. Diese Methode nutzte die spezifischen Vorteile von Zebrafischen für die direkte Zellverhaltensbeobachtung, die für Mäuse schwierig ist. Die aktuelle Methode ermöglichte es uns, die Chemokin-Funktionen innerhalb weniger Tage schnell auf Zellverhalten zu testen und Zebrafische zu einem leistungsfähigen Modell zu machen, um die Molekular- und Zellbiologie zu studieren.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Die Autoren haben nichts zu verraten.
Acknowledgments
Wir danken Dr. Jingrong Peng für die gemeinsame Nutzung der transgenen Tg-Linie (fabp10a: DsRed); Dr. Zilong Wen für die gemeinsame Nutzung der transgenen Tg(mpeg1: GFP) Dr. Koichi Kawakami für die Bereitstellung des pTol2-Vektors. Diese Arbeit wurde von der National Natural Science Foundation of China (31771594), Guangdong Science and Technology Plan Projects (2019A030317001) und den Fundamental Research Funds for the Central Universities (D2191450) unterstützt.
Materials
| Name | Company | Catalog Number | Comments |
| Antibody | |||
| Alexa 488-Anti-Goat antibody | Invitrogen | A11055 | |
| Anti-Digoxigenin-HRP | perkinelmer | NEF832001EA | |
| Goat-Anti-GFP antibody | Abcam | ab6658 | |
| Reagent | |||
| CaCl2· 2H2O | Sigma | 21097 | |
| Cyanine 3 Plus Amplification Reagent | perkinelmer | NEL745001KT | |
| E2 solution | 15 mM NaCl +0.5 mM KCl +1.0 mM MgSO4+150 µM KH2PO4 + 50 µM Na2HPO4 +1.0 mM CaCl2 + 0.7 mM NaHCO3 | ||
| Fetal Bovine Serum (FBS) | Life | 10099-133 | |
| Formamide | Diamond | A100314 | |
| Glycerol | Sigma | V900860 | |
| Heparin sodium | Sigma | H3149 | |
| Hybridization buffer(HB) | 50% formamide+ 5×SSC+9 mM sodium citrate+50 μg/ml heparin sodium+ 500 μg/mL tRNA+ 0.1% Tween20 | ||
| KCl | Sigma | P5405 | |
| KH2PO4 | Sigma | P5655 | |
| Low melting agarose | Sigma | A9414 | |
| Methanol | GHTECH | 1.17112.023 | |
| Methylene blue | Sigma | M9140 | |
| MgSO4 | Sigma | M2643 | |
| Na2HPO4 | Sigma | S5136 | |
| NaCl | Sigma | S5886 | |
| NaHCO3 | Sigma | S5761 | |
| Paraformaldehyde (PFA) | Sigma | 158127 | Suspend 16 g of PFA in 400 ml of 1x PBS, heat at 60 °C to dissolve about 30 min. This solution can be prepared in advance and stored at -4 °C. Caution. Manipulate with mask. |
| 10×PBS | 14.2 g Na2HPO4+80 g NaCl+2 g KCl+ 2.4 g KH2PO4 in 1L ddH2O | ||
| Phenylthiourea (PTU) | Sigma | P7629 | |
| 1×Plus Amplification Diluent | perkinelmer | NEL745001KT | |
| Proteinase K | Fermentas | E00492 | |
| 20×Saline sodium citrate(SSC) | 175.3 g NaCl+ 88.2 g sodium citrate in 1 L ddH2O, PH 7.0 | ||
| Sodium citrate | Sigma | A5040 | |
| Tricaine | Sigma | E10521 | |
| tRNA | Sigma | R6625 | |
| Tween20 | Sigma | P2287 | |
| Plasmid | |||
| pBLK-fabp10a-il34-sv40 | For Tg (fab10a:il34) transgenic line generation | ||
| pBSK-il34 | For il34 probe preparation | ||
| Fish | |||
| Tg (mpeg1: GFP) | Label macrophages with GFP | ||
| Tg (fabp10a: DsRed) | Label liver cells with DsRed | ||
| Tg (fab10a:il34) | Over-expression IL-34 in liver cells |
References
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Wang, Y., et al. IL-34 is a tissue-restricted ligand of CSF1R required for the development of Langerhans cells and microglia. Nature Immunology. 13 (8), 753-760 (2012).
- Lin, H., et al. Discovery of a cytokine and its receptor by functional screening of the extracellular proteome. Science. 320 (5877), 807-811 (2008).
- Wei, S., et al. Functional overlap but differential expression of CSF-1 and IL-34 in their CSF-1 receptor-mediated regulation of myeloid cells. Journal of leukocyte biology. 88 (3), 495-505 (2010).
- Etienne, D., Foucher, S. B. L. P., Norbert Ifrah, P. G. Y. D. IL-34 Induces the Differentiation of Human Monocytes into Immunosuppressive Macrophages. Antagonistic Effects of GM-CSF and IFNc. PLoS One. 8 (2), e56045 (2013).
- Segaliny, A. I., et al. Syndecan-1 regulates the biological activities of interleukin-34. Biochimica et Biophysica Acta. 1853 (5), 1010-1021 (2015).
- Zhou, S. L., et al. miR-28-5p-IL-34-macrophage feedback loop modulates hepatocellular carcinoma metastasis. Hepatology. 63 (5), 1560-1575 (2016).
- Gordon, J. I., et al. Tissue specific expression and developmental regulation of two genes coding for rat fatty acid binding proteins. Journal of Biological Chemistry. 260 (4), 1995-1998 (1985).
- Westerfield, M. The Zebrafish Book: A Guide for the Laboratory Use of Zebrafish (Danio rerio). , University of Oregon. Eugene, OR. (2000).
- Nüsslein-Volhard, C., Dahm, R. Zebrafish: a practical approach. , Oxford University Press. New York. (2002).
- Jiang, Y., Chen, J., Yen, K., Xu, J. Ectopically Expressed IL-34 Can Efficiently Induce Macrophage Migration to the Liver in Zebrafish. Zebrafish. 16 (2), 165-170 (2019).



