Summary
Pour tester l'effet d'une chimiokine sur le recrutement de macrophages in vivo, l'hybridation in situ de montage entière a été employée pour détecter l'expression ectopique de la chimiokine, et l'immunostaining a été employée pour étiqueter des macrophages. L'imagerie en direct a été utilisée pour l'observation en temps réel de la migration des macrophages.
Abstract
Le poisson zèbre est largement utilisé dans la recherche fondamentale et biomédicale. De nombreuses lignées transgéniques de poissons zèbres sont actuellement disponibles pour étiqueter divers types de cellules. En raison du corps embryonnaire transparent du poisson zèbre, il est commode pour nous d'étudier l'effet d'une chimiokine sur le comportement d'un certain type de cellules in vivo. Ici nous avons fourni un workflow pour étudier la fonction d'une chimiokine sur la migration de macrophage in vivo. Nous avons construit un plasmide de surexpression spécifique aux tissus pour surexprimer l'IL-34 et injecté le plasmide dans des embryons de poissons transgéniques à un stade cellulaire dont les macrophages ont été spécifiquement étiquetés par une protéine fluorescente. Nous avons ensuite utilisé l'hybridation in situ fluorescente de montage entier et l'immunostaining pour détecter le modèle de l'expression de chemokine et le nombre ou l'emplacement des macrophages. Les embryons WT injectés ont été élevés pour générer une lignée transgénique stable. Enfin, nous avons utilisé la formation image vivante confocale pour observer directement le comportement de macrophage dans le poisson transgénique stable pour étudier la fonction de l'IL-34 sur des macrophages in vivo.
Introduction
Le poisson zèbre est un petit poisson d'eau douce tropical à os durs originaire de l'Inde. En ce qui concerne la conservation des gènes, le poisson zèbre ont une similitude de 87% à l'homme1. Il peut nous fournir des informations sur des sujets connexes de l'homme en étudiant la régulation des gènes, la fonction des protéines et le comportement cellulaire tels que la migration, la prolifération et.al chez le poisson zèbre. L'embryon de poisson zèbre peut être utilisé pour observer le développement des embryons précoces à différents stades après avoir inhibé le pigment. Pendant ce temps, il ne faut que trois mois pour que le poisson zèbre se développe en maturité sexuelle, puis le poisson zèbre peut produire des centaines d'œufs tous les 4 jours. Mini-taille, reproduction simple, forte capacité de reproduction, ces avantages rendent la culture du poisson zèbre très économe en espace, propice à la culture à grande échelle. La souris modèle mammifère traditionnelle a des coûts d'entretien plus élevés que le poisson zèbre, limitant ainsi l'échelle de l'élevage de souris. Dans l'aspect du développement précoce de l'embryon, l'embryon de souris est difficile à observer en état vivant en raison des caractéristiques du développement de l'embryon de souris dans l'utérus de la mère. Au contraire, les embryons de poissons zèbres se développent à l'extérieur et sont transparents, donc ils sont faciles à observer au microscope. En outre, le poisson zèbre est très facile de construire une variété de lignes transgéniques pour la recherche sur la fonction génique connexe. Actuellement, diverses lignées transgéniques de poisson zèbre sont disponibles pour étiqueter différents types de cellules. Il est très pratique maintenant de construire des lignes transgéniques pour surexprimer les chimiolines dans des endroits spécifiques et d'étudier la fonction de chemokines sur le comportement cellulaire chez le poisson zèbre.
Ici, nous avons fourni un flux de travail pour utiliser la ligne transgénique de poisson zèbre pour étudier la fonction de l'IL-34 sur le comportement de macrophage in vivo2,3,4,5,6,7. Tout d'abord, nous avons construit un plasmide de surexpression spécifique au foie du gène il34 et injecté le plasmide dans un stade à cellule Tg (mpeg1: GFP) embryons de poissons qui ont spécifiquement étiqueté les macrophages par la protéine fluorescente GFP. Ensuite, nous avons utilisé l'hybridation in situ fluorescente de montage entier et l'immunostaining pour détecter le modèle de l'expression il34 et le nombre ou l'emplacement des macrophages. Les embryons WT injectés ont été élevés pour générer une lignée transgénique stable. Dans ces étapes, nous avons établi et validé la ligne de cytokine-productrice et avons évalué visuellement les effets qui peuvent être vus sur la distribution de macrophage. Enfin, pour étudier le comportement de macrophage en réponse à la cytokine, nous avons employé la formation image vivante confocale pour observer directement la migration de macrophage pour confirmer la fonction d'il34 sur la migration de macrophage in vivo.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
REMARQUE: Tous les échantillons ont été traités par l'eau d'oeuf de phénylthiourea (PTU) pour inhiber le pigment.
1. Génération de Tg (fabp10a:il34) Transgenic Constructs and Fish Injection
- Clonez le promoteur 2.8 kb fabp10a 8 et les régions de codage IL-34 (ENSDART00000126460.3) de poissons zèbres dans le vecteur pTol2 pour générer la construction fabp10a-il34. Injecter les constructions dans un stade à cellule Tg (mpeg1: GFP) et des embryons de poissons WT avec l'ARNm transposase. Élever les embryons WT injectés fabp10a-il34 à l'adulte9 et identifier le fondateur transgénique par hybridation in situ.
REMARQUE: L'injection de la construction Tol2 directement dans un autre transgénique pourrait être problématique si l'autre ligne transgénique est faite avec le même système de transposon. Une pratique générale serait de faire une ligne transgénique indépendante et par la suite franchir la nouvelle ligne avec une autre ligne de journaliste. Cela garantit qu'il n'y aura pas d'effets de la nouvelle transgenèse sur un transgène précédemment inséré.
2. Fluorescent Whole Mount In Situ Hybridization (WISH) Combiner avec Immunostaining
- Fixation de l'échantillon
- Recueillir des embryons d'injection transitoire ou de ligne transgénique IL-34 stable qui a traversé avec Tg (mpeg1: GFP) aux stades souhaités.
REMARQUE: Pour ce cas, les embryons ont été rassemblés à 4 d après la fertilisation (dpf). (Si nécessaire) retirer le chorion par seringue. - Fixer les embryons avec 4 % de paraformaldéhyde (PFA) pendant la nuit à 4 oC ou 2 h à température ambiante (RT) (environ 25 oC).
- Laver les embryons avec du phosphate tamponné saline plus Tween 20 (PBST) 3x 5 min.
- Déshydrater les embryons séparément avec 50 % de méthanol en PBST (50 % de méthanol/PBST) et 100 % de méthanol, 1x 5 min chacun. Ensuite, passer au méthanol frais à 100 % et conserver à -20 oC (au moins 2 h).
REMARQUE: Le protocole peut être mis en pause ici.
- Recueillir des embryons d'injection transitoire ou de ligne transgénique IL-34 stable qui a traversé avec Tg (mpeg1: GFP) aux stades souhaités.
- Hybridation des sondes (Jour I)
- Réhydratez les embryons dans les étapes précédentes avec 50 % de méthanol en PBST (50 % de méthanol/PBST), puis lavez-les avec de la PBST 3x 5 min.
- Dig les embryons avec la protéase K en PBST à RT (concentration finale: 10 'g/mL; 1:2000 en PBST).
REMARQUE: Le temps de digestion dépend du stade des embryons : moins de 36 h après la fécondation (hpf), pas besoin; 36 hpf-2 dpf embryon, 3-5 min; 2-3 dpf embryon, 10 min; 3-4 dpf embryon, 15 min; 4-5 dpf embryon, 15-20 min; 5-6 dpf embryon, 20-27 min; Embryon dpf de 25 à 30 min à RT (environ 25 oC). - Jeter la solution de digestion et effectuer la fixation à nouveau avec 4% PFA, pendant 20 min à RT.
- Laver les embryons avec pBST 2x 10 min.
- Jetez la PBST, effectuez la pré-hybridation avec un tampon d'hybridation chauffée (tampon HB) à 65 oC pendant 5 min, recyclez le tampon HB dans le tube d'origine.
- Effectuez la pré-hybridation avec un nouveau tampon HB chauffé à 65 oC au moins 1 h.
- Préchauffer la sonde9 (pour ce cas était une sonde il34, 1 ng/mL) à 65 oC au moins 10 min. Puis recyclez le tampon HB dans le tube d'origine. Effectuez l'hybridation avec la sonde préchauffée à 65 oC pendant la nuit.
- Traitement des anticorps (Jour II)
- Préchauffer le citrate de sodium formamide/2x formamide de 50 % plus Tween 20 (SSCT), 2x SSCT, 0,2x SSCT à 65 oC.
- Recyclez la sonde dans le tube d'origine et entreposez la sonde à -20 oC.
- Laver les embryons séparément avec 50% de formamide/2x SSCT; 2x SSCT; 0,2x SSCT, 3x 20 min ou 2x 30 min chacun à 65 oC.
- Laver les embryons avec PBST 3x 5 min.
- Bloquer les échantillons avec 600 l de tampon de blocage (5% de sérum bovin fœtal filtré (FBS) en PBST) pendant 1 h à RT.
- Ajouter 400 l de solution d'anticorps Anti-digoxigenin-HRP (1:1,000-1:2,000 dans le tampon de blocage) et incuber les embryons à 4 oC pendant la nuit. Si les signaux sont faibles, utilisez 1:500 dilution d'anticorps.
- Coloriage et incubation d'anticorps primaires (Jour III)
- Enlever l'anticorps; laver les embryons avec PBST, 6x 20 min à RT.
- Rincer l'échantillon avec 30 oL de 1x Plus Amplification Diluent pendant 5 min à RT.
- Jeter le diluant en pipetting; diluer Fluorophore Tyramide Stock Solution (Cyanine 3 Plus Amplification Reagent (Cy3) ou Cyanine 5 Plus Amplification Reagent (Cy5), pour ce cas Cy3 a été utilisé) 1:50 dans 1x Plus Amplification Diluent pour faire le Fluorophore Tyramide Solution de travail. Préparer 50 à 100 l de solution de travail pour chaque échantillon.
- Incuber l'échantillon dans la solution de travail Fluorophore Tyramide pendant 5-15 min dans l'obscurité à RT. Si les signaux sont faibles, prolongez le temps d'incubation à 30 min.
- Arrêtez la réaction en modifiant la solution de travail avec PBST et examinez les signaux.
- Laver les embryons avec PBST 3x 10 min à RT.
- Incuber l'échantillon avec un anticorps primaire à 4 oC pendant la nuit. Pour ce cas, utilisez l'anticorps Goat-Anti-GFP comme anticorps primaire.
- Coloration secondaire d'anticorps (Jour IV)
- Laver les embryons avec de la PBST pendant 4x 30 min.
- Incuber les embryons avec un anticorps secondaire à 4 oC pendant la nuit. Pour ce cas, utilisez Alexa 488-Anti-Goat anticorps comme anticorps secondaire.
- Prenez des photos (Jour V)
- Laver les embryons avec PBST 3x 10 min à RT.
- Conserver les embryons dans 70 % de glycérol dans l'obscurité à 4 oC pendant la nuit ou à -20 oC plus longtemps.
3. Imagerie en direct
-
Sélection d'échantillons
REMARQUE: Utilisez l'image en direct pour observer directement si les macrophages de Tg (fabp10a: il34; fabp10a: DsRed; mpeg1: GFP) poissons migreraient dans le foie sous l'induction IL-34 au cours de 3-3.5 dpf. Ici, la ligne transgénique Tg (fabp10a-DsRed)est utilisée pour étiqueter la région du foie et la rendre visible, pour faciliter la localisation du foie et pour déterminer si les macrophages migrent dans le foie. Avant l'imagerie, utilisez un microscope à fluorescence pour sélectionner les embryons Double positif DsRed et GFP. -
Montage de poissons
- Utilisez un bain en métal pour chauffer 1 ml d'agarose fondante de 1 % à plus de 90 oC pour le faire fondre complètement.
- Après que l'agarose fondante faible soit refroidie à la température corporelle, ajouter 50 l de tricaine de 0,2 % et mélanger uniformément la tricaine avec l'agarose.
- Déplacer les embryons anesthésiés vers un petit plat monté avec une glissière de couverture sur le fond, enlever l'eau environnante, laisser tomber lentement l'agarose fondante faible sur les embryons, soigneusement définir la position du poisson avant que l'agarose est solidifiée, garder la zone du foie près de la glissière de couverture sur le fond du plat.
- Après la fusion faible agarose est solidifié, soigneusement le couvrir avec une autre couche d'agarose pour le renforcer.
- Placez le plat sur la table de porteur de microscope confocal, couvrez le poisson avec la solution E210 avec la tricaine et commencez l'imagerie.
-
Fonctionnement logiciel du microscope confocal
- Ouvrez le logiciel ZEN noir 2.3, installez l'établi de cellule vivante sur la table de porteur de microscope.
- Cliquez sur Localiser Incubation (Incubation) Température pour fixer la température à 29 oC.
- Placez le plat au centre de l'établi de cellule vivante, couvrez le poisson avec la solution E210 avec la tricaine.
- Cliquez sur le menu Acquisition, sélectionnez le mode d'analyse et les lasers requis dans le menu Smart Setup, puis sélectionnez Z-Stack et Position.
- Cliquez sur le menu Experiment Designer, sélectionnez Activer l'expérience multibloc, dans le premier bloc, pour trouver l'échantillon sous le grossissement faible, puis passer à l'agrandissement élevé, laisser la zone observée au centre du champ visuel.
- Définir la position et z-Stack informations, sélectionnez l'intensité laser appropriée, les couches de numérisation et la vitesse d'imagerie.
- Créez un nouveau bloc et répétez les étapes ci-dessus. Après la mise en place de tous les blocs, définiz le nombre approprié de boucles et commencez à enregistrer (Figure 1).
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Les étapes du protocole du poisson zèbre sont illustrées à la figure 2. Tout d'abord, nous avons généré la construction pBLK-fabp10a-il34-sv40 dans laquelle il34 a été conduit par le promoteur fabp10a (Figure 2). La construction a été micro-injectée dans un stade à cellule Tg (mpeg1 : GFP) embryons de poissons zèbres qui peuvent étiqueter les macrophages avec des embryons de GFP et de WT qui ont été élevés à des adultes pour générer la ligne stable transgénique (figure 2). L'expression de l'il34 a été analysée par hybridation in situ par fluorescence de monture entière(figure 2 et figure 3). Les macrophages étiquetés par Le PFG ont été analysés par immunostaining(figure 2 et figure 3). Nous avons utilisé l'imagerie en direct pour observer directement si les macrophages migreraient dans le foie sous l'induction il34 pendant 3-3.5 dpf (Figure 2, Figure 4, Film supplémentaire 1 et film supplémentaire 2) .
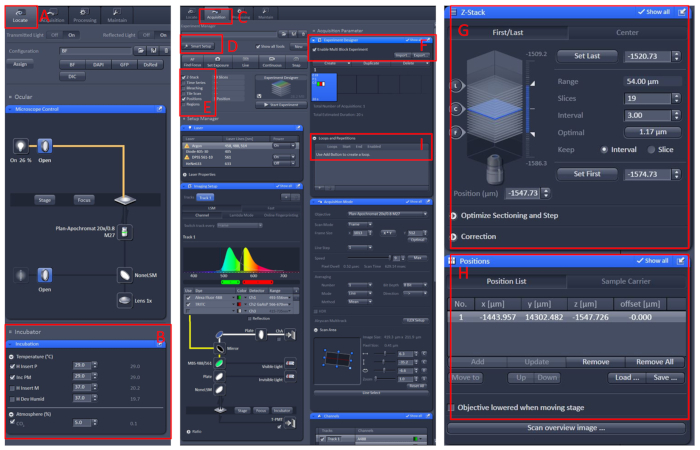
Figure 1 : Fonctionnement logiciel de l'imagerie en direct au microscope confocal. Ouvrez le logiciel ZEN noir 2.3, installez l'établi de cellules vivantes sur la table du porteur du microscope, puis cliquez sur Localiser (A) Incubation (Incubation) Température (B) pour fixer la température à 29 oC. Placez le plat au centre de l'établi de cellule vivante, couvrez le poisson avec la solution E210 avec la tricaine. Après tout cela, cliquez sur le menu Acquisition (C) , sélectionnez le mode d'analyse et les lasers requis dans le menu Smart Setup (D), puis sélectionnez Z-Stack et Position (E). Enfin, cliquez sur le menu Experiment Designer (F) , sélectionnez l'expérience Enable Multi Block, dans le premier bloc, pour trouver l'échantillon sous le grossissement faible, puis passer à la grossissement élevé, laissez la zone observée dans le centre du champ visuel, définir la position et Z-Stack (G et H) informations, sélectionnez l'intensité laser appropriée, les couches de numérisation et la vitesse d'imagerie. Créez un nouveau bloc et répétez les étapes ci-dessus. Après la mise en place de tous les blocs, définir le nombre approprié de boucles (I) et commencer à enregistrer. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Flux de travail pour étudier la fonction d'une chimiokine sur la migration des macrophages in vivo. Nous avons construit un plasmide surexpression spécifique aux tissus (foie) pour surexprimer l'IL-34 et injecté le plasmide dans des embryons de poissons transgéniques à un stade cellulaire dont les macrophages ont été spécifiquement étiquetés par une protéine fluorescente (Tg: (mpeg1: GFP )). Les embryons WT injectés ont été élevés pour générer une lignée transgénique stable. Nous avons ensuite utilisé l'hybridation in situ fluorescente de montage entier et l'immunostaining pour détecter le modèle de l'expression de gène et le nombre ou l'emplacement des macrophages des embryons injectés transitoires ou des embryons de ligne stables (4 dpf). Enfin, nous avons utilisé la formation image vivante confocale pour observer directement le comportement de macrophage dans le poisson transgénique stable (3-3.5 dpf) pour étudier la fonction de l'IL-34 sur des macrophages in vivo. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3 : Le WISH fluorescent se combine avec l'immunostaining. Ce chiffre a été modifié à partir de Jiang et coll.11. Un total de 1,8 nL (30 ng/L) de la construction pBLK-fabp10a-il34-sv40 a été microinjecté dans des embryons de poisson zèbre à un stade à cellule Tg (mpeg1 : GFP). (A) WISH de l'expression il34 (rouge) et de la coloration d'anticorps à monture entière de l'expression GFP (vert) dans 4 embryons dpf (6x). L'image du corps entier du poisson est composé de deux images distinctes prises par confocal et cousu ensemble dans Photoshop. Les insets sont un grossissement élevé (20x) des régions en boîte correspondantes (zones pointillées orange). (B) Analyse quantitative des nombres de cellules macrophages dans le foie des embryons injectés (indiqué dans la zone pointillée blanche) et la région de la queue (environ entre la 13e et la 17e somite, montrée entre deux lignes pointillées blanches). Les données ont été analysées par le test Mann Whitney U, 'p 'lt; 0.01 par rapport au contrôle. n - 5, 5 pour les 4 dpf injectés et contrôler les poissons. Barres : 200 m (ligne blanche); 50 m (ligne jaune). (C) WISH de l'expression il34 et de la coloration d'anticorps à monture entière de l'expression GFP dans l'embryon de ligne stable de 4 dpf (6x). L'image du corps entier du poisson est composé de deux images distinctes prises par confocal et cousu ensemble dans Photoshop. Les insets sont un grossissement élevé (20x) des régions en boîte correspondantes (zones pointillées orange). (D) Analyse quantitative des nombres de cellules macrophages dans Tg (mpeg1: GFP) et Tg (fabp10a: il34; mpeg1: GFP) foie d'embryons (montré dans la zone pointillée blanche) et région de la queue (environ entre le 13e et 17ème somite, montré entre deux lignes pointillées blanches). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : Imagerie vivante confocale pour observer directement le comportement des macrophages chez les poissons transgéniques stables. Ce chiffre a été modifié à partir de Jiang et coll.11. Des micrographies d'imagerie en direct montrent le processus d'un macrophage (vert, étiqueté par des flèches blanches) passant par le foie (rouge) dans les 28 min dans les poissons témoins (A) et le processus d'un macrophage (vert, étiqueté par des flèches blanches) passant dans le foie (rouge) dans les 28 dans il-34 surexprimant le poisson (B). Barres d'échelle de 40 m (ligne blanche). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Film supplémentaire 1: Imagerie en direct montrant le processus de macrophages (vert, étiqueté par des flèches blanches) migrer dans le foie (rouge) dans les 2 h dans IL-34 surexprimant les poissons. Barres d'échelle de 20 m (ligne blanche). Ce film a été réédité de Jiang et al.11. Veuillez cliquer ici pour visionner cette vidéo. (Clic droit pour télécharger.)

Film supplémentaire 2: Imagerie en direct montrant le processus de macrophages (vert, étiqueté par des flèches blanches) errant autour du foie (rouge) dans les 2 h dans les poissons témoins. Barres d'échelle de 20 m (ligne blanche). Ce film a été réédité de Jiang et al.11. Veuillez cliquer ici pour visionner cette vidéo. (Clic droit pour télécharger.)
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Le protocole décrit ici nous permet d'étudier la fonction d'une chimiokine sur le comportement de la macrophagein vivo et la procédure nécessite une certaine expertise technique. En résumé, il y a plusieurs étapes critiques pour éviter des complications dans le protocole : 1) sélectionnez une ligne transgénique appropriée qui montre le signal transgénique spécifique et fort pour étiqueter la cellule d'intérêt ; 2) sélectionner un tissu approprié qui est accessible pour l'imagerie et la surexpression des gènes transgéniques; 3) faire une sonde sensible et spécifique d'ARN ; 4) sélectionnez une fenêtre de temps d'observation appropriée pour capturer avec précision le comportement cellulaire.
Dans le procédé de la fluorescence entière de fluorescence in situ combinée avec l'immunostaining, la sonde d'ARN employée pour détecter l'expression de gène devrait être sensible et le signal doit être assez fort. Afin de capturer la fonction génique sur le comportement cellulaire, une série de points de temps doivent être testés. Par exemple, en observant l'effet de l'il34 sur la migration des macrophages, bien que le promoteur fabp10a a commencé à exprimer à 2-3 dpf, l'accumulation de macrophages dans le foie n'était pas évident à ce moment-là. Ce n'est que par 4 dpf que l'enrichissement du macrophage dans le foie devient apparent. En outre, après l'hybridation in situ, l'intensité de signal de l'immunostaining suivante sera affectée. Par exemple, en comparant avec GFP, DsRed est difficile à colorer dans la coloration d'immunofluorescence après hybridation in situ, probablement en raison des différentes structures protéiques. D'une manière générale, l'intensité du signal de l'immunostaining après l'hybridation in situ de fluorescence de montage entière serait inférieure à celle de l'immunostaining simple.
Dans l'étape d'imagerie en direct avec le microscope confocal, il est nécessaire de garder l'échantillon près du fond du plat. Lorsque les embryons flottent dans l'agarose, la distance de travail de l'objectif peut être insuffisante, aussi, l'agarose entre l'objectif et l'échantillon affecterait la qualité de l'imagerie. En outre, le nombre d'échantillons pour l'imagerie à chaque fois doit être réglé correctement. Il faut s'assurer que le laps de temps entre deux scans de chaque poisson ne serait pas trop long pour perdre les détails du comportement cellulaire. Donc, cette méthode n'est pas adaptée pour le suivi des cellules qui se déplacent rapidement dans les tissus épais.
En conclusion, ce protocole peut être utilisé pour observer la fonction des chimiocines sur le comportement d'une variété de cellules telles que les macrophages, les neutrophiles et les lymphocytes T. Ici, nous avons utilisé IL-34, un ligand récemment identifié de la fonction CSF-1R dans la chimiotaxie6,7, comme une chimiokine exprimée ectopique pour induire la migration des macrophages. La plupart des modèles expérimentaux existants de chimiotaxis cellulaires sont basés sur des expériences cellulaires in vitro, mais les expériences in vitro sont parfois trop simples pour modéliser l'environnement complexe in vivo. En outre, il est difficile d'imaginer la capacité de chimio-attraction in vivo quand il suffit de regarder la situation in vitro. Cette méthode a utilisé les avantages spécifiques du poisson zèbre pour l'observation directe du comportement cellulaire qui est difficile pour les souris. La méthode actuelle nous a permis de tester rapidement les fonctions de chimiokine sur les comportements cellulaires en quelques jours et de faire du poisson zèbre un modèle puissant pour étudier la biologie moléculaire et cellulaire.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Les auteurs n'ont rien à révéler.
Acknowledgments
Nous remercions le Dr Jingrong Peng d'avoir partagé la ligne transgénique Tg (fabp10a: DsRed); Dr. Zilong Wen pour le partage des lignes transgéniques Tg (mpeg1: GFP); Dr. Koichi Kawakami pour fournir le vecteur pTol2. Ces travaux ont été soutenus par la National Natural Science Foundation of China (31771594), les projets du Guangdong Science and Technology Plan (2019A030317001) et les Fonds de recherche fondamentale pour les universités centrales (D2191450).
Materials
| Name | Company | Catalog Number | Comments |
| Antibody | |||
| Alexa 488-Anti-Goat antibody | Invitrogen | A11055 | |
| Anti-Digoxigenin-HRP | perkinelmer | NEF832001EA | |
| Goat-Anti-GFP antibody | Abcam | ab6658 | |
| Reagent | |||
| CaCl2· 2H2O | Sigma | 21097 | |
| Cyanine 3 Plus Amplification Reagent | perkinelmer | NEL745001KT | |
| E2 solution | 15 mM NaCl +0.5 mM KCl +1.0 mM MgSO4+150 µM KH2PO4 + 50 µM Na2HPO4 +1.0 mM CaCl2 + 0.7 mM NaHCO3 | ||
| Fetal Bovine Serum (FBS) | Life | 10099-133 | |
| Formamide | Diamond | A100314 | |
| Glycerol | Sigma | V900860 | |
| Heparin sodium | Sigma | H3149 | |
| Hybridization buffer(HB) | 50% formamide+ 5×SSC+9 mM sodium citrate+50 μg/ml heparin sodium+ 500 μg/mL tRNA+ 0.1% Tween20 | ||
| KCl | Sigma | P5405 | |
| KH2PO4 | Sigma | P5655 | |
| Low melting agarose | Sigma | A9414 | |
| Methanol | GHTECH | 1.17112.023 | |
| Methylene blue | Sigma | M9140 | |
| MgSO4 | Sigma | M2643 | |
| Na2HPO4 | Sigma | S5136 | |
| NaCl | Sigma | S5886 | |
| NaHCO3 | Sigma | S5761 | |
| Paraformaldehyde (PFA) | Sigma | 158127 | Suspend 16 g of PFA in 400 ml of 1x PBS, heat at 60 °C to dissolve about 30 min. This solution can be prepared in advance and stored at -4 °C. Caution. Manipulate with mask. |
| 10×PBS | 14.2 g Na2HPO4+80 g NaCl+2 g KCl+ 2.4 g KH2PO4 in 1L ddH2O | ||
| Phenylthiourea (PTU) | Sigma | P7629 | |
| 1×Plus Amplification Diluent | perkinelmer | NEL745001KT | |
| Proteinase K | Fermentas | E00492 | |
| 20×Saline sodium citrate(SSC) | 175.3 g NaCl+ 88.2 g sodium citrate in 1 L ddH2O, PH 7.0 | ||
| Sodium citrate | Sigma | A5040 | |
| Tricaine | Sigma | E10521 | |
| tRNA | Sigma | R6625 | |
| Tween20 | Sigma | P2287 | |
| Plasmid | |||
| pBLK-fabp10a-il34-sv40 | For Tg (fab10a:il34) transgenic line generation | ||
| pBSK-il34 | For il34 probe preparation | ||
| Fish | |||
| Tg (mpeg1: GFP) | Label macrophages with GFP | ||
| Tg (fabp10a: DsRed) | Label liver cells with DsRed | ||
| Tg (fab10a:il34) | Over-expression IL-34 in liver cells |
References
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Wang, Y., et al. IL-34 is a tissue-restricted ligand of CSF1R required for the development of Langerhans cells and microglia. Nature Immunology. 13 (8), 753-760 (2012).
- Lin, H., et al. Discovery of a cytokine and its receptor by functional screening of the extracellular proteome. Science. 320 (5877), 807-811 (2008).
- Wei, S., et al. Functional overlap but differential expression of CSF-1 and IL-34 in their CSF-1 receptor-mediated regulation of myeloid cells. Journal of leukocyte biology. 88 (3), 495-505 (2010).
- Etienne, D., Foucher, S. B. L. P., Norbert Ifrah, P. G. Y. D. IL-34 Induces the Differentiation of Human Monocytes into Immunosuppressive Macrophages. Antagonistic Effects of GM-CSF and IFNc. PLoS One. 8 (2), e56045 (2013).
- Segaliny, A. I., et al. Syndecan-1 regulates the biological activities of interleukin-34. Biochimica et Biophysica Acta. 1853 (5), 1010-1021 (2015).
- Zhou, S. L., et al. miR-28-5p-IL-34-macrophage feedback loop modulates hepatocellular carcinoma metastasis. Hepatology. 63 (5), 1560-1575 (2016).
- Gordon, J. I., et al. Tissue specific expression and developmental regulation of two genes coding for rat fatty acid binding proteins. Journal of Biological Chemistry. 260 (4), 1995-1998 (1985).
- Westerfield, M. The Zebrafish Book: A Guide for the Laboratory Use of Zebrafish (Danio rerio). , University of Oregon. Eugene, OR. (2000).
- Nüsslein-Volhard, C., Dahm, R. Zebrafish: a practical approach. , Oxford University Press. New York. (2002).
- Jiang, Y., Chen, J., Yen, K., Xu, J. Ectopically Expressed IL-34 Can Efficiently Induce Macrophage Migration to the Liver in Zebrafish. Zebrafish. 16 (2), 165-170 (2019).



