Summary
Questo protocollo descrive un metodo di elettroporazione efficiente per la trasfezione di quattro diverse entità organoidi gastrointestinali con plasmidi più grandi (nella misura di 10 kB). Può essere eseguito entro un giorno e non ha bisogno di una preparazione completa o di speciali tamponi di elettroporazione ad alta intensità di costi.
Abstract
L'elettroporazione è un metodo comune per la trasfezione con diversi tipi di molecole per permeabilizzazione elettrica della membrana plasmatica. Con l'uso crescente di organoidi come metodo di coltura per il materiale primario del paziente negli ultimi anni, sono necessari metodi di trasferimento efficienti dei componenti per l'ingegneria genetica in questo sistema di coltura 3D. Soprattutto per gli organoidi, l'efficienza delle manipolazioni genetiche dipende da una trasfezione di successo. Così, questo protocollo è stato sviluppato per facilitare l'elettroporazione degli organoidi e per dimostrare la sua funzionalità universale in entità diverse. Gli organoidi del cancro umano colorettale, pancreatico, epatico e gastrico sono stati elettropolati con successo con plasmidi piccoli e grandi in confronto. Sulla base dei vettori di codifica GFP, l'efficienza della trasfezione è stata determinata dal FACS. Non è necessaria una preparazione completa delle cellule o buffer di elettroporazione speciali e dispendiosi in termini di costi e il protocollo può essere eseguito entro un giorno.
Introduction
Negli ultimi anni, un nuovo sistema di coltura cellulare 3D, chiamato organoidi, è stato sviluppato per vari tessuti normali e cancerosi. Gli organiidi sono funzionalmente e morfologicamente molto vicini al loro tessuto di origine. Possono essere generati da diverse specie, sono facilmente espandibili, genomicamente stabili e geneticamente modificabili, il che li rende un sistema modello ideale per le indagini genetiche1,2,3. Tecniche di ingegneria genetica come il sistema CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats)/Cas9 consentono manipolazioni diverse. La selezione dei cloni può essere realizzata da condizioni multimediali definite, ad esempio, da WNT ligand ritiro per APC (Adenomatosis Polyposis Coli) cloni knockout4,5. In alternativa, i marcatori di selezione devono essere introdotti mediante la riparazione diretta omologa di un vettore di targeting6,7. A causa del fatto che spesso più di un plasmide deve essere introdotto, una trasfezione efficiente diventa un parametro cruciale. Inoltre, per ridurre gli effetti fuori bersaglio non specifici, è auspicabile un'espressione transitoria dell'endonuclease Cas98.
L'elettroporazione è un metodo relativamente semplice per trasfecare le cellule con DNA, RNA, proteine o altre macromolecole. Per mezzo di impulsi elettrici, la membrana cellulare diventa più permeabile e provoca un aumento dell'assorbimento9. In un protocollo di elettroporazione pubblicato in precedenza di organoidi del colon è stata raggiunta una efficienza del 30 % con una GFP (proteina fluorescente verde) che esprime vettore (7,4 kB) in una procedura di quattro giorni10. Il seguente protocollo è stato sviluppato per facilitare una trasfezione efficiente di organoidi cancerosi o sani con codifica di plasmidi di grandi dimensioni per l'RNA a guida singola (sgRNA) e la sequenza endonuclease Cas9 (ad esempio px458 come vettore con 9,3 kb). L'intero processo di elettroporazione può essere eseguito entro un giorno, senza speciali tamponi di elettroporazione e con efficienze almeno comparabili tra diversi gastrointestinali, vale a dire adenocarcinoma duttale pancreatico (PDAC), cancro colorettale (CRC), colangiocarcinoma (CCC) e organoidi gastrico (GC).
Protocol
L'approvazione etica è stata ottenuta dal comitato etico del TU Dresda (#EK451122014).
1. Cultura organoide e preparativi prima dell'elettroporazione
- Stabilire organoidi per digestione tissutale come descritto in precedenza ed espanderli con il corrispondente mezzo di coltura specifico dell'entità in una matrice seminterrato (panoramica vedi Tabella 1 e Tabella dei materiali)11,12,13,14,15,16,17.
NOTA: Per i campioni di tessuto umano è necessario il consenso informato e l'approvazione dello studio da parte di un comitato etico. - Prewarm piastre di 48 pozze a 37 gradi centigradi per la seedinazione post-elettroporazione.
- Preparare antibiotici basali medi w/o e antibiotici di coltura organoide specifici dell'entità (cfr. tabella 1) compresi 10 - M Y-27632 e 3 CHIR99021.
NOTA: Il ritiro degli antibiotici è importante per ridurre gli effetti tossici. Y-27632 e CHIR99021 migliorare il recupero cellulare10. -
Preparazione degli organoidi (vedere la Figura 1)
- Coltivare 5 pozzetti di organoidi in un campione di 48 pozzetti per elettroporazione in mezzo di coltura.
NOTA: devono essere utilizzati organoidi proliferativi (circa 2-3 giorni dopo l'ultima divisione). - Preparare 230 - L di reagente di dissociazione (vedi Tabella dei materiali) tra cui 10 M Y-27632 per pozzo.
- Togliere il mezzo di coltura da ogni pozzo e dissociare meccanicamente gli organoidi in 230 gradi della miscela di dissociazione preparata. Pool 5 pozzi per campione di elettroporazione in un tubo da 15 mL.
- Mescolare vortice e incubare per 5-15 min a 37 s fino a quando non si verificano cluster di 10-15 cellule. Pertanto, controllare la dissociazione microscopicamente. Interrompere la digestione aggiungendo antibiotici di basale media w/o fino a 10 mL.
NOTA: Questo passaggio è molto critico! L'efficienza dell'elettroporazione sarà ridotta, quando l'incubazione è troppo breve, ma la lunga digestione ridurrà la sopravvivenza. - Centrifuga a 450 x g per 5 min a temperatura ambiente, scartare il supernatante e lavare due volte con 4 mL di buffer di elettroporazione (vedi Tabella dei materiali).
- Coltivare 5 pozzetti di organoidi in un campione di 48 pozzetti per elettroporazione in mezzo di coltura.
2. Elettroporazione
NOTA: il seguente protocollo è sviluppato per gli elettrofaratori in grado di onde quadrate e sequenze di impulsi separati di poring e transfer (vedere Figura 2). Facoltativamente, i valori di impedimento, così come le tensioni, le correnti e le energie trasferite nel campione possono essere misurate come controllo per esperimenti riproducibili.
- Risospendere il pellet organoide in 100 gradi di tampone di elettroporazione (vedi Tabella dei materiali)contenente 30 g di DNA plasmico.
NOTA: La concentrazione del DNA plasmico usato deve superare i 5 g/l per una concentrazione ottimale di sale durante il processo di elettroporazione. Pertanto, si consigliano maxi kit plasmide endofree (vedi Tabella dei materiali)per la preparazione dei vettori. Un totale di 45 g di DNA può essere utilizzato senza effetti citotossici. - Distribuisci l'intera miscela DNA-organoid in una cuvette di elettroporazione con larghezza di 2 mm senza produrre bolle d'aria.
- Impostare i parametri di elettroporazione in base a Fujii et al.10 (vedere la tabella 2, Figura 2).
- Mescolare leggermente le celle senza schiumare toccando la cuvette con un dito. Posizionare la cuvette nella camera della cuvette.
- Premete il tasto dell'elettropuntore e prendere nota del valore di impedance.
NOTA: Un'impedizione tra 30-40 s ha mostrato i migliori risultati. In generale, dovrebbe essere nella gamma tra 30-55 . In caso contrario, controllare i seguenti aspetti: larghezza del divario della cuvette utilizzata, collegamenti via cavo dell'elettroporatore, possibili bolle d'aria, corretta concentrazione di volume e sale della miscela di elettroporazione. - Premere il pulsante Start per eseguire il programma di elettroporazione e controllare i valori di correnti, tensioni ed energie visualizzate.
NOTA: i valori delle tensioni misurate, delle correnti e delle energie devono corrispondere ai parametri di elettroporazione impostati. Per il confronto di esperimenti ripetuti, può essere utile prendere nota di questi dati. - Dopo l'elettroporazione, aggiungere immediatamente 500 - L di antibiotici di coltura media w/o (con CHIR99021 e Y-27632; vedere il passo 1.3). Mescolare pipetting su e giù per dissociare la schiuma bianca.
NOTA: La schiuma bianca appare dopo il processo di elettroporazione e ad essa è collegato un numero significativo di cellule. Quindi, la dissociazione di esso è molto importante per non perdere le cellule. - Trasferire completamente il campione dalla cuvette in un nuovo tubo da 15 mL utilizzando la pipetta appartenente alle cuvette di elettroporazione (vedere Tabella dei materiali). Per ottenere le cellule rimanenti si consiglia di risciacquare nuovamente la cuvette con un mezzo basale.
- Per la rigenerazione delle cellule, incubarle per 40 min a temperatura ambiente.
3. Seeding delle cellule
- Centrifugare le cellule a 450 x g per 5 min a temperatura ambiente e scartare il supernatante.
- Risospendere il pellet in 100 . Incubare per 10 min a 37 gradi centigradi per la polimerizzazione e aggiungere 250 l of culture medium, che viene integrato con Y-27632 e CHIR99021 fino alla successiva divisione degli organoidi coltivati (circa 5-7 giorni).
4. Determinazione dell'efficienza della trasfezione
NOTA: In generale, si raccomanda di elettroporrare un vettore che trasporta un marcatore di fluorescenza come controllo di trasfezione aggiuntivo. A seconda del marcatore scelto e della sua maturazione cromoforo, la fluorescenza sarà visibile entro circa 24-48 h dopo la trasfezione18.
- Controllare la fluorescenza microscopicamente dopo 24-48 h nel controllo della trasfezione (vedere Figura 4B).
-
Scansione fluorescente delle celle attivate (FACS)
- Raccogliere le cellule analogamente al passo 1.4.2-1.4.4 e digerire circa 10-20 min fino a quando non ci sono singole cellule. Aggiungere fino a 10 mL di salina con buffer fosfato (PBS).
- Centrifuga a 450 x g per 5 min a temperatura ambiente e scartare il supernatante.
- Opzionalmente per la discriminazione delle cellule viventi: Risospendere il pellet in 1 mL di salina con tamponamento fosfato (PBS) e aggiungere un anticorpo adatto (vedi Tabella dei materiali)o iodide propidio (PI). Mescolare con molta attenzione solo toccando e incubare per 30 min a temperatura ambiente al buio. Lavare con 10 mL di PBS, centrifugare e scartare il supernatante.
- Risospendere il pellet cellulare in 200 gradi L di PBS e, facoltativamente, filtrare la sospensione attraverso un colino cellulare di 100 m in un tubo FACS.
- Analizzare le celle da una macchina FACS utilizzando una strategia di gating appropriata (vedere Figura 3; Figura 4A) e determinare l'efficienza della trasfezione.
Representative Results
Gli organoidi di quattro diverse entità tumorali (CRC, CCC, PDAC, GC) sono stati elettropotizzati almeno 3 volte utilizzando 30 g di un piccolo plasmide (pCMV-EGFP, 4,2 kb) o un grande plasmide (px458, 9,3 kb). Entrambi i vettori trasportano una cassetta GFP che consente di determinare l'efficienza della trasfezione 48 h dopo l'elettroporazione per citometria di flusso. Per analizzare solo le cellule viventi, colorazione con un anticorpo di vita prima della scansione è stata eseguita. La strategia di gating è illustrata nella Figura 3.
In tutte e quattro le entità organoidi il plasmide di dimensioni 4,2 kB è stato trasincato con maggiore efficienza rispetto a quello più grande (vedere La figura 4). La trasfezione più efficiente del piccolo plasmide è stata raggiunta negli organoidi PDAC con cellule positive GFP 92,1 - 5,2%, mentre il grande plasmide è stato trascurato con un'efficienza del 46,7 % (media - deviazione standard, n - 3). Rispetto agli organoidi tumorali del pancreas, il plasmide più grande è stato trasinfezione in modo più efficiente in organoidi CRC con un'efficienza media di 53,4 x 11,7 %, mentre il piccolo plasmide è stato trasvenuto con un'efficienza media di 84,3 x 5,8 %. L'entità più difficile da trasfecare erano gli organoidi per il cancro gastrico: sia per il grande che per il piccolo plasmide, è stata raggiunta l'efficienza di trasfezione più bassa in questa entità (rispettivamente 32,3 x 12,7 % e 74,1 x 5,5 %). Gli organoidi CCC hanno mostrato un'efficienza media di trasfezione dell'83,0 x del 13,1% per il piccolo plasmide e per il grande plasmide del 39,5 x 10,4 %.
Come prova di concetto, gli organoidi dello stomaco normale umano sono stati elettroporate con una codifica plasmide px458_Conc2 per Cas9, GFP e due sgRNA mirati TP53. Le rotture del doppio filamento indotta da Cas9 sull'esone 8 sono state riparate da un'unione finale non omogenea (NHEJ), con conseguente spostamento di fotogrammi e di conseguenza un knockout del gene (vedi Figura supplementare 1).
Tabella 1: Composizione di supporti basali, miscele di digestione e supporti di coltivazione. Fare clic qui per visualizzare questo file (fare clic con il pulsante destro del mouse per scaricare).

Tabella 2: Impostazioni di elettroporazione secondo Fujii et al.10.

Figura 1: flusso di lavoro di preparazione dell'elettroporazione. In primo luogo, gli organoidi dovrebbero essere dissociati a grappoli di 10-15 cellule e gli antibiotici dovrebbero essere lavati. Dopo l'elettroporrato la schiuma bianca deve essere dissociata. Le cellule possono essere semiate dopo la rigenerazione per 40 min a temperatura ambiente. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: elettroporazione in due fasi. Due impulsi di poring con maggiore tensione und breve durata (175 V e 157.5 V, ciascuno per 5 ms, pausa per 50 ms, decadimento di tensione 10%) portare alla formazione di pori nelle membrane cellulari. I seguenti impulsi di trasferimento forniscono il DNA nelle cellule: cinque impulsi di trasferimento positivi (con 20 V, 12 V, 7.2 V, 4.32 V e 2.592 V, ciascuno per 50 ms, pausa per 50 ms, decadimento della tensione 40%), seguito da cinque impulsi di trasferimento scambiati polarità (con 20 V, 12 V, 7.2 V, 4.32 V e 2.592 V, ciascuno per 50 ms, pausa per 50 ms, decadimento tensione 40%). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Strategia di gating rappresentativa illustrata dagli organoidi CCC. Tutti gli organoidi elettroporati sono stati analizzati dalla citometria di flusso 48 h dopo l'elettroporazione. Le cellule elettroporate senza DNA plasmide sono state usate come controlli negativi. I cancelli sono stati impostati come segue: (A) gating per la forma delle cellule, (B,C) gating per singole cellule (discriminazione del doppiotto), (D) gating per le cellule viventi (macchiate con un anticorpo per le cellule apoptotiche) e (E,F) infine gating per eGFP esprimere le cellule (canale FITC). FSC- dispersione in avanti; SSC - dispersione laterale. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Efficienza dell'elettroporazione di quattro entità organoidi. (A) Vengono mostrati l'analisi FACS (n - 34, deviazione media standard e ogni singolo valore) e (B) confronto visivo al microscopio a fluorescenza. Barra della scala : 1.000 m. BF - campo luminoso; CCC - cholangiocarcinoma; CRC - cancro colorettale; GC - cancro gastrico; PDAC - adenocarcinoma duttale pancreatico. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura supplementare Figura 1: esemplare CRISPR/Cas9-based knockout di TP53 in organoidi stomaco umano normale. Il vettore px458_Conc2 (vedi Tabella dei materiali)è stato clonato combinando il vettore di 2 concatemeri gRNA, un generoso regalo di Bon-Kyoung Koo19, con px45820, con conseguente codifica plasmica per 2 sgRNA, Cas9 e GFP. Due sgRNA mirati TP53 sono stati introdotti nel vettore px458_Conc2 dalla clonazione del cancello d'oro (analogamente ad Andersson-Rolf et al.19). Sono stati elettroponati in organoidi gastrici normali umani (A). I cloni sono stati selezionati dall'amministrazione Nutlin3 (B) e il Knockout TP53 è stato confermato dalla clonazione e sequenziamento TOPO TA degli alleli, qui esemplare mostrato per un clone (C). Gli sgRNA sono sottolineati nel riferimento. Barra della scala : 200 m. BF - campo luminoso. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussion
Questo protocollo fornisce istruzioni dettagliate per un elettroporazione efficiente, rapida e facile da eseguire di diverse entità organoidi. Oltre agli organoidi tumorali presentati da PDAC, CRC, CCC e GC, funziona con successo anche per gli organoidi derivati da tessuti sani. Il protocollo può essere eseguito entro un giorno. Nei protocolli di trasfezione organoide pubblicati l'intera procedura è durata quattro giorni, compresi due giorni di preparazione con diversi tipi di supporti di coltivazione10,21. Nel nostro protocollo non è richiesto alcun pretrattamento speciale. Con il lavaggio con buffer di elettroporazione prima dell'elettroporazione i componenti antibiotici dei supporti sono stati lavati ed è stata raggiunta una regolazione delle concentrazioni saline per ottenere valori di impedimento ottimali. Tuttavia, alcuni aspetti critici dovrebbero essere considerati per un elettroporazione di successo:
Cellule
Nel protocollo di elettroporazione di Fujii et al.10 si raccomanda di dissociare gli organoidi su singole cellule e di filtrarli attraverso un colino cellulare di 20 m. Nelle nostre mani la digestione in singole cellule diminuisce fortemente la sopravvivenza delle cellule. Come suggerito in Merenda et al.21, abbiamo anche dissociato organoidi a cluster di 10-15 cellule e non abbiamo potuto determinare una diminuzione dell'efficienza rispetto alla dissociazione a singola cellula. Dopo l'elettroporazione, è un passo molto importante per dissociare la schiuma bianca, in modo che nessuna cella collegata si perda.
Per la coltura cellulare 2D, è stato dimostrato che un tempo di rigenerazione dopo l'elettroporazione di oltre 10 min fino a 40 min aumenta la sopravvivenza e l'efficienza della trasfezione soprattutto di grandi plasmidi22. Negli esperimenti di prova, lo stesso potrebbe essere documentato per gli organoidi, portando ad una fase di incubazione di 40 min dopo l'elettroporazione in questo protocollo. Al fine di aumentare il recupero dall'elettroporazione, li abbiamo coltivati con l'inibitore della proteina Rho (ROCK) Y-27632 per cinque-sette giorni23. Allo stesso modo, il completamento aggiuntivo di sintetizzata glicogeno chinasi 3 (GSK3) inibitore CHIR99021 è destinato ad aiutare singole cellule a recuperare10.
Impostazioni
Uno dei vantaggi dell'elettroporatore usato è che l'impedimento può essere misurato prima dell'elettroporazione per condizioni ottimali. Secondo il produttore, i valori di impeditto dovrebbero essere 30-55 . Nelle nostre mani, ivalori di impedizione di 30-40 gradi hanno mostrato efficienze ottimali. In un esperimento preliminare, diverse tensioni e valori di lunghezza dell'impulso dell'impulso di poring sono stati variati per trovare la proporzione ottimale di efficienza alla sopravvivenza. In sintesi, potremmo confermare i valori descritti di Fujii et al.10 nelle diverse entità descritte qui.
Dna
L'effetto di diverse quantità di DNA è stato testato in esperimenti preliminari fino a 45 g di DNA per campione. Non è stato possibile rilevare effetti citotossici. L'efficienza della trasfezione è stata aumentata in modo dipendente dalla dose con saturazione > 30 g. Quindi, abbiamo usato 30 g per campione nel protocollo finale, ma ovviamente può essere aumentato (ad esempio, per l'elettroporazione di più plasmidi in parallelo). Inoltre, la purezza e la concentrazione del DNA sembra essere molto importante. Una concentrazione superiore a 5 g/l ha mostrato un'efficienza di trafezione ottimale.
Come previsto, il plasmide da 9,3 kB potrebbe essere trasinfezione con un'efficienza inferiore rispetto al plasmide più piccolo di 4,2 kB (vedere la figura 4). Si prevede che l'uso di plasmidi ancora più grandi di 10 kB riduca ulteriormente l'efficienza. Per le applicazioni future, potrebbe essere interessante testare il DNA dei minicerchi come vettore, poiché questi portatori genici non hanno la spina dorsale batterica di un plasmide che li rende più piccolidi 24. Questo dovrebbe comportare una maggiore efficienza di trasfezione. Inoltre, per le manipolazioni degli organoidi a base CRISPR, un elettroporazione diretta di sgRNA legati a Cas9 come complesso di ribonucleoproteina (RNP) potrebbe essere un'alternativa o un'aggiuntadi 25.
Disclosures
Gli autori non hanno nulla da rivelare.
Acknowledgments
Ringraziamo Juliane Fohgrub, Ann-Christin Meinecke e Max Heiduk per un'eccellente assistenza tecnica. I finanziamenti sono stati erogati da Deutsche Krebshilfe (n. 111350 e 70112925), Sander Stiftung (n. 2014.104.1), Hector Stiftung (n. M65.2) e l'Unione europea (ERC n. 639050).
Materials
| Name | Company | Catalog Number | Comments |
| For establishment and culture medium | |||
| [Leu15] Gastrin | Sigma-Aldrich | G9145 | |
| A83-01 | Tocris Bioscience | 2939 | |
| Advanced DMEM/F-12 | Invitrogen | 12634010 | |
| B27 | Invitrogen | 17504044 | |
| B27 Supplement, minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| CHIR99021 | Stemgent | 04-0004 | |
| Collagenase II | Life Technologies | 17101-015 | |
| Collagenase XI | Sigma-Aldrich | C9407-100MG | |
| Collagenase D | Roche | 11088866001 | |
| Dispase II | Roche | 4942078001 | |
| Dnase I | Sigma-Aldrich | D5319 | |
| D-sorbitol | Roth | 6213.1 | |
| Dithiothreitol | Thermo Scientific | 1859330 | |
| EDTA | Roth | 8040 | |
| Forskolin | Tocris Bioscience | 1099 | |
| Glutamax | Life Technologies | 35050061 | |
| Hepes | Thermo Fisher Scientific | 15630106 | |
| hFGF-10 | Preprotech | 100-26 | |
| KCl | Sigma-Aldrich | P9541 | |
| KH2PO4 | Roth | 3904.2 | |
| Matrigel | Corning | 356231 | basement matrix |
| mEGF | Invitrogen | PMG8043 | |
| N2 | Invitrogen | 17502048 | |
| NaCl | Roth | 3957.1 | |
| Na2HPO4 | Roth | K300.2 | |
| N-Acetyl-L-Cystein | Sigma-Aldrich | A9165 | |
| Nicotinamid | Sigma-Aldrich | N0636 | |
| Noggin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (Hek293-mNoggin-Fc) |
| PBS | Gibco | 14190169 | |
| Penicillin Streptomycin | Life Technologies | 15140122 | |
| Primocin | InvivoGen | ant-pm-1 | |
| Prostaglandin E2 | Tocris Bioscience | 2296 | |
| Recombinant Human HGF | Preprotech | 100-39H | |
| Rspondin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (HA-Rspo1-Fc-293T) |
| SB202190 | Sigma-Aldrich | S7067 | |
| Sucrose | VWR | 27,480,294 | |
| TrypLE Express | Gibco | 12604021 | Dissociation reagent |
| Wnt3A | n.a. | n.a. | Conditioned medium produced from L-Wnt3a cells (from Sylvia Boj) |
| Y-27632 | Sigma-Aldrich | Y0503 | |
| Consumables | |||
| Cell Strainer 100 µm | Falcon | 352360 | |
| 48-well plate | Corning | 3548 | |
| Nepa Electroporation Cuvettes 2mm gap w/pipettes | Nepa Gene Co., Ltd. | EC-002S | |
| Tubes 15 ml | Greiner | 188271 | |
| Tubes 15 ml low binding | Eppendorf | 30122208 | Tubes for FACS preparing |
| Equipment | |||
| Electroporator Nepa21 | Nepa Gene Co., Ltd. | n.a. | |
| EVOS FL Auto | Invitrogen | AMAFD1000 | Fluorescence microscope |
| LSRFortessa | BD Bioscience | 647800E6 | FACS |
| Reagents and plasmids | |||
| Live/dead fixable blue dead cell stain kit | Invitrogen | L34962 | Includes antibody for live/dead staining for FACS analysis |
| EndoFree Plasmid Maxi Kit | Qiagen | 12362 | |
| Nutlin-3 | Selleckchem | S1061/07 | |
| Opti-MEM | Gibco | 31985047 | Electroporation buffer |
| 2 gRNA concatemer vector | AddGene | 84879 | |
| pCMV-EGFP | Nepa Gene Co., Ltd. | n.a. | |
| px458 plasmid | AddGene | 48138 | coding for sgRNA and Cas9 |
| px458_Conc2 plasmid | AddGene | 134449 | px458 plasmid containing 2x U6 promotors for two different sgRNAs |
| sgRNA_hTP53_1a | Eurofins Genomics | n.a. | 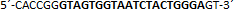 |
| sgRNA_hTP53_1b | Eurofins Genomics | n.a. | 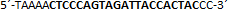 |
| sgRNA_hTP53_2a | Eurofins Genomics | n.a. | 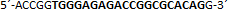 |
| sgRNA_hTP53_2b | Eurofins Genomics | n.a. | 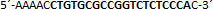 |
References
- Huch, M., et al. Long-term culture of genome-stable bipotent stem cells from adult human liver. Cell. 160 (1-2), 299-312 (2015).
- Werner, K., Weitz, J., Stange, D. E. Organoids as Model Systems for Gastrointestinal Diseases: Tissue Engineering Meets Genetic Engineering. Current Pathobiology Reports. 4 (1), 1-9 (2016).
- Olayanju, A., Jones, L., Greco, K., Goldring, C. E., Ansari, T. Application of porcine gastrointestinal organoid units as a potential in vitro tool for drug discovery and development. Journal of applied toxicology. 39 (1), 4-15 (2019).
- Drost, J., et al. Sequential cancer mutations in cultured human intestinal stem cells. Nature. 521 (7550), 43-47 (2015).
- Matano, M., et al. Modeling colorectal cancer using CRISPR-Cas9-mediated engineering of human intestinal organoids. Nature Medicine. 21 (3), 256-262 (2015).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Drost, J., et al. Use of CRISPR-modified human stem cell organoids to study the origin of mutational signatures in cancer. Science. 358 (6360), 234-238 (2017).
- Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S., Yang, S. H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy Nucleic Acids. 4, e264 (2015).
- Tsong, T. Y. Electroporation of cell membranes. Biophysical journal. 60 (2), 297-306 (1991).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nature Protocols. 10 (10), 1474-1485 (2015).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Bartfeld, S., et al. In vitro expansion of human gastric epithelial stem cells and their responses to bacterial infection. Gastroenterology. 148 (1), 126-136 (2015).
- Boj, S. F., et al. Organoid models of human and mouse ductal pancreatic cancer. Cell. 160 (1-2), 324-338 (2015).
- Broutier, L., et al. Culture and establishment of self-renewing human and mouse adult liver and pancreas 3D organoids and their genetic manipulation. Nature Protocols. 11 (9), 1724-1743 (2016).
- Broutier, L., et al. Human primary liver cancer-derived organoid cultures for disease modeling and drug screening. Nature Medicine. 23 (12), 1424-1435 (2017).
- Hennig, A., et al. CFTR Expression Analysis for Subtyping of Human Pancreatic Cancer Organoids. Stem Cells Int. 2019, 1024614 (2019).
- Seidlitz, T., et al. Human gastric cancer modelling using organoids. Gut. 68 (2), 207-217 (2019).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Current protein & peptide science. 9 (4), 338-369 (2008).
- Andersson-Rolf, A., et al. Simultaneous paralogue knockout using a CRISPR-concatemer in mouse small intestinal organoids. Dev Biol. 420 (2), 271-277 (2016).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8, 2281 (2013).
- Merenda, A., et al. A Protocol for Multiple Gene Knockout in Mouse Small Intestinal Organoids Using a CRISPR-concatemer. Journal of Visualized Experiments. (125), (2017).
- Lesueur, L. L., Mir, L. M., Andre, F. M. Overcoming the Specific Toxicity of Large Plasmids Electrotransfer in Primary Cells In Vitro. Molecular Therapy Nucleic Acids. 5, e291 (2016).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
- Darquet, A. M., Cameron, B., Wils, P., Scherman, D., Crouzet, J. A new DNA vehicle for nonviral gene delivery: supercoiled minicircle. Gene Therapy. 4 (12), 1341-1349 (1997).
- Jacobi, A. M., et al. Simplified CRISPR tools for efficient genome editing and streamlined protocols for their delivery into mammalian cells and mouse zygotes. Methods. 121-122, 16-28 (2017).



