Summary
Este protocolo descreve um método eficiente de eletroporação para a transfecção de quatro diferentes entidades organoides gastrointestinais com plasmídeos maiores (na extensão de 10 kB). Pode ser realizado dentro de um dia e não precisa de extensa preparação ou tampões especiais de eletroporação econômicos.
Abstract
A eletroporação é um método comum para transfecção com diferentes tipos de moléculas por permeabilização elétrica da membrana plasmática. Com o uso crescente de organoides como método de culturingpara material primário do paciente nos últimos anos, estão necessitados métodos eficientes de transferência de componentes para engenharia genética neste sistema de cultura 3D. Especialmente para organoides, a eficiência das manipulações genéticas depende de uma transfecção bem sucedida. Assim, esse protocolo foi desenvolvido para facilitar a eletroporação de organoides e para comprovar sua funcionalidade universal em diferentes entidades. Organoides colorretais, pancreáticos, hepáticos e gástricos foram eletroporados com sucesso com pequenos e grandes plasmídeos em comparação. Com base em vetores de codificação gfp, a eficiência de transfecção foi determinada pela FACS. Não é necessário uma preparação extensiva das células ou tampões especiais de eletroporação intensiva e econômica, e o protocolo pode ser realizado dentro de um dia.
Introduction
Nos últimos anos, um novo sistema de cultura celular 3D, chamado organoides, foi desenvolvido para vários tecidos normais e cancerosos. Organoides são funcionais e morfologicamente muito próximos de seu tecido de origem. Elas podem ser geradas a partir de diferentes espécies, são facilmente expansíveis, genomicamente estáveis e geneticamente modificáveis, o que as torna um sistema modelo ideal para investigações genéticas1,2,3. Técnicas de engenharia genética como o CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats)/Cas9 permitem diversas manipulações. A seleção de clones pode ser realizada por condições de mídia definidas, por exemplo, por meio da WNT ligand withdrawal para aaPC (Adenomatosis Polyposis Coli) clones knockout4,5. Alternativamente, os marcadores de seleção devem ser introduzidos pelo reparo direcionado homologous de um vetor de alvo6,7. Devido ao fato de que muitas vezes mais de um plasmídeo precisa ser introduzido, uma transfecção eficiente torna-se um parâmetro crucial. Além disso, para reduzir efeitos fora do alvo inespecíficos, uma expressão transitória da endonuclocação cas9 é desejável8.
A eletroporação é um método relativamente simples para transfect células com DNA, RNA, proteínas ou outras macromoléculas. Por meio de pulsos elétricos, a membrana celular torna-se mais permeável e causa um aumento da captação9. Em um protocolo de eletroporação publicado anteriormente de organoides de cólon, uma eficiência de 30 % com um vetores expresso de cofic-bac (proteína fluorescente verde) expressando vetor (7,4 kB) foi alcançado em um procedimento de quatro dias10. O seguinte protocolo foi desenvolvido para facilitar uma transfecção eficiente de organoides cancerosos ou saudáveis com grande codificação plasmídeos para RNA guia único (sgRNA) e a sequência cas9 endonuclease (por exemplo, px458 como vetor com 9,3 kb). Todo o processo de eletroporação pode ser realizado dentro de um dia, sem tampões especiais de eletroporação, e com pelo menos eficiências comparáveis entre diferentes organoides gastrointestinais, ou seja, adenocarcinoma ductal pancreático (PDAC), câncer colorretal (CRC), cholangiocarcinoma (CCC) e organoides de câncer gástrico (GC).
Protocol
A aprovação ética foi obtida no comitê ético do TU Dresden (#EK451122014).
1. Cultura organoide e preparações antes da eletroporação
- Estabelecer organoides por digestão tecidual como descrito anteriormente e expandi-los com sua entidade correspondente meio cultural específico em uma matriz de porão (visão geral ver Tabela 1 e Mesa de Materiais)11,12,13,14,15,16,17.
NOTA: Para que as amostras de tecido humano sejam necessárias o consentimento informado e a aprovação do estudo por um comitê ético. - Pré-aqueça 48 placas de poço a 37 °C para semeação pós-eletroporação.
- Prepare antibióticos de meio basal, bem como antibióticos de cultura organoide específica de entidade (ver Tabela 1) incluindo 10 μM Y-27632 e 3 μM CHIR99021.
NOTA: A retirada de antibióticos é importante para reduzir os efeitos tóxicos. Y-27632 e CHIR99021 amenizam a recuperaçãocelular 10. -
Preparação dos organoides (ver Figura 1)
- Cultivar 5 poços de organoides em uma placa de 48 poços por amostra de eletroporação em meio cultural.
NOTA: Organoides proliferativos devem ser usados (cerca de 2-3 dias após a última divisão). - Prepare 230 μL de reagente de dissociação (ver Tabela de Materiais) incluindo 10 μM Y-27632 por poço.
- Remova o meio cultural de cada poço e dissocia os organoides mecanicamente em 230 μL da mistura de dissociação preparada. Pool 5 poços por amostra de eletroporação em um tubo de 15 mL.
- Misture por vórtice e incubar por 5-15 min a 37 °C até que ocorram aglomerados de 10-15 células. Portanto, verifique a dissociação microscopicamente. Pare a digestão adicionando antibióticos de w/o médioba de até 10 mL.
NOTA: Este passo é muito crítico! A eficiência da eletroporação será reduzida, quando a incubação for muito curta, mas a digestão longa reduzirá a sobrevivência. - Centrífuga a 450 x g por 5 min à temperatura ambiente, descarte o supernatante e lave duas vezes com 4 mL de tampão de eletroporação (ver Tabela de Materiais).
- Cultivar 5 poços de organoides em uma placa de 48 poços por amostra de eletroporação em meio cultural.
2. Eletroporação
NOTA: O protocolo a seguir é desenvolvido para eleporantes capazes de ondas quadradas e sequências separadas de porção e pulso de transferência (ver Figura 2). Opcionalmente, os valores de impedância, bem como as tensões, correntes e energias transferidas para a amostra podem ser medidos como controle para experimentos reprodutíveis.
- Resuspender a pelota organoide em 100 μL de tampão de eletroporação (ver Tabela de Materiais) contendo 30 μg de DNA plasmídeo.
NOTA: A concentração do DNA plasmóide usado deve exceder 5 μg/μL para uma concentração de sal ideal durante o processo de eletroporação. Portanto, são recomendados kits de plasmídeo endofree (ver Tabela de Materiais)para a elaboração de vetores. Uma quantidade total de até 45 μg de DNA pode ser usada sem efeitos citotóxicos. - Dispense a mistura completa de DNA-organóide em um cuvette de eletroporação com largura de 2 mm sem produzir bolhas de ar.
- Defina os parâmetros de eletroporação de acordo com Fujii et al.10 (ver Tabela 2, Figura 2).
- Misture as células um pouco sem espumando tocando no cuvette com um dedo. Coloque o cuvette na câmara de Cuvette.
- Pressione o botão Ω do eleporizador e anote o valor da impedância.
NOTA: Uma impedância entre 30-40 Ω mostrou os melhores resultados. Em geral, deve estar no intervalo entre 30-55 Ω. Se este não for o caso, por favor, controle os seguintes aspectos: largura de lacuna do cuvette usado, conexões de cabo do eletroporador, possíveis bolhas de ar, volume correto e concentração de sal da mistura de eletroporação. - Pressione o botão Iniciar para executar o programa de eletroporação e controlar os valores das correntes, tensões e energias exibidas.
NOTA: Os valores das tensões, correntes e energias medidas devem corresponder aos parâmetros de eletroporação definidos. Para a comparação de experimentos repetidos, pode ser útil notar esses dados. - Após a eletroporação, adicione imediatamente 500 μL de antibióticos médios de cultura (com CHIR99021 e Y-27632; veja passo 1.3). Misture pipetting para cima e para baixo para dissociar a espuma branca.
NOTA: A espuma branca aparece após o processo de eletroporação e um número significativo de células é anexado a ela. Então, dissociação é muito importante para não perder células. - Transfira a amostra completamente do cuvette para um novo tubo de 15 mL usando a pipeta pertencente aos cuvetas de eletroporação (ver Tabela de Materiais). A lavagem do cuvette novamente com meio basal é recomendada para obter células remanescentes.
- Para a regeneração das células, incuba-as por 40 min em temperatura ambiente.
3. Semeade das Células
- Centrífuga simê as células a 450 x g por 5 min à temperatura ambiente e descarte o supernatante.
- Resuspender a pelota em 100 μL de matriz do porão e sementes de 20 μL gotas em uma placa pré-aquecida de 48 poços (ver passo 1.2). Incubar por 10 min a 37 °C para polimerização e adicionar 250 μL de meio cultural, que é complementado com Y-27632 e CHIR99021 até a próxima divisão dos organoides cultivados (em torno de 5-7 dias).
4. Determinação da Eficiência transfecção
NOTA: Em geral, recomenda-se eletroporizar um vetor carregando um marcador de fluorescência como controle adicional de transfecção. Dependendo do marcador escolhido e de seu amadurecimento cromosforo, a fluorescência será visível em torno de 24-48 h pós transfecção18.
- Verifique a fluorescência microscopicamente após 24-48 h no controle de transfecção (ver Figura 4B).
-
Varredura de células ativadas fluorescentes (FACS)
- Colher as células análogamente para pisar 1.4.2-1.4.4 e digerir cerca de 10-20 min até que haja células únicas. Adicione até 10 mL de salino tampão de fosfato (PBS).
- Centrífuga a 450 x g por 5 min à temperatura ambiente e descarte o supernatante.
- Opcionalmente para discriminação de células vivas: Resuspender a pelota em 1 mL de sorofia tampão de fosfato (PBS) e adicionar um anticorpo adequado (ver Tabela de Materiais) ou iodeto de propídio (PI). Misture com muito cuidado apenas tocando e incubar por 30 minutos à temperatura ambiente no escuro. Lave com 10 mL de PBS, centrífuga e descarte o supernatante.
- Resuspender a pelota de célula em 200 μL de PBS e filtra opcionalmente a suspensão através de um filtro de célula de 100 μm em um tubo FACS.
- Analisar as células por uma máquina FACS usando uma estratégia adequada de gating (ver Figura 3; Figura 4A) e determinar a eficiência da transfecção.
Representative Results
Organoides de quatro entidades cancerígenas diferentes (CRC, CCC, PDAC, GC) foram eletroporados pelo menos 3 vezes usando 30 μg de um pequeno plasmídeo (pCMV-EGFP, 4,2 kb) ou um grande plasmídeo (px458, 9,3 kb). Ambos os vetores carregam um GFP permitindo a determinação da eficiência de transfecção 48 h após a eletroporação por citometria de fluxo. Para analisar apenas células vivas, manchar com um anticorpo de morte antes da varredura ser realizada. A estratégia de gating é mostrada na Figura 3.
Nas quatro entidades organoides, o plasmídeo de 4,2 kB foi transfecciodo com maior eficiência em relação ao maior (ver Figura 4). A transfecção mais eficiente do pequeno plasmídeo foi alcançada em organoides PDAC com 92,1 ± 5,2 % células positivas gfp, enquanto o grande plasmídeo foi transfeclado com uma eficiência de 46,7 ± 3,7 % (média ± desvio padrão, n = 3). Em comparação com organoides de câncer de pâncreas, o plasmídeo maior foi transfecciodo de forma mais eficiente em organoides CRC com uma eficiência média de 53,4 ± 11,7 %, enquanto o pequeno plasmídeo foi transfeclado com uma eficiência média de 84,3 ± 5,8 %. A entidade mais difícil de transfect era organoides do câncer gástrico: tanto para o grande quanto para o pequeno plasmídeo, a menor eficiência de transfecção foi alcançada nesta entidade (32,3 ± 12,7 % e 74,1 ± 5,5 %, respectivamente). Os organoides ccc apresentaram uma eficiência média de transfecção de 83,0 ± 13,1 % para o pequeno plasmídeo e para o grande plasmídeo 39,5 ± 10,4 % foram obtidos.
Como prova de conceito, os organoides do estômago normal humano foram eletroporados com uma codificação px458_Conc2 plasmóide para Cas9, GFP e dois sgRNAs visando TP53. As quebras de fios duplos induzidas por Cas9 na exon 8 foram reparadas por uma junção final não homologous (NHEJ), resultando em mudanças de quadros e, consequentemente, um nocaute do gene (ver Figura Complementar 1).
Tabela 1: Composição de mídia basal, misturas de digestão e mídia de cultivo. Clique aqui para ver este arquivo (Clique certo para baixar).

Tabela 2: Configurações de eletroporação de acordo com Fujii et al.10.

Figura 1: Fluxo de trabalho de preparação de eletroporação. Primeiro, organoides devem ser dissociados para aglomerados de 10-15 células e antibióticos devem ser lavados. Após a eletroporação, a espuma branca precisa ser dissociada. As células podem ser semeadas após se regenerarem por 40 min à temperatura ambiente. Clique aqui para ver uma versão maior deste valor.

Figura 2: Eletroporação de duas etapas. Dois pulsos de poros com maior tensão sem duração curta (175 V e 157,5 V, cada um para 5 ms, pausa para 50 ms, decomposição de tensão 10%) levar à formação de poros em membranas celulares. Os pulsos de transferência a seguir fornecem o DNA nas células: cinco pulsos de transferência positivos (com 20 V, 12 V, 7,2 V, 4,32 V e 2.592 V, cada um para 50 ms, pausa para 50 ms, decomposição de tensão 40%), seguido por cinco pulsos de transferência trocados de polaridade (com 20 V, 12 V, 7,2 V, 4,32 V e 2.592 V, cada um por 50 ms, pausa para 50 ms, voltage m Clique aqui para ver uma versão maior deste valor.

Figura 3: Estratégia representativa de gating mostrada pelos organoides CCC. Todos os organoides eletroporados foram analisados por citometria de fluxo 48 h após a eletroporação. Células eletroporadas sem DNA plasmídeo foram usadas como controles negativos. Os portões foram definidos como: (A) gating para a forma celular, (B,C) gating para células únicas (dupla discriminação), (D) gating para células vivas (manchado com um anticorpo para células apoptoticas) e(E,F) finalmente gating para eGFP células expressas (canal FITC). FSC = dispersão para a frente; SSC = dispersão lateral. Clique aqui para ver uma versão maior deste valor.

Figura 4: Eficiência de eletroporação de quatro entidades organoides. (A)análise FACS (n = 34, desvio padrão médio e cada valor único são mostrados) e(B) comparação visual por microscópio de fluorescência. Barra de escala = 1.000 μm. BF = campo brilhante; CCC = cholangiocarcinoma; CRC = câncer colorretal; GC = câncer gástrico; PDAC = adenocarcinoma ductal pancreático. Clique aqui para ver uma versão maior deste valor.

Figura Suplementar 1: Nocaute exemplar baseado em CRISPR/Cas9 de TP53 em organoides normais do estômago humano. O vetor px458_Conc2 (ver Tabela de Materiais) foi clonado pela combinação do vetor concatemer de 2 gRNA, um presente generoso de Bon-Kyoung Koo19, com px45820, resultando em uma codificação plasmídea para 2 sgRNAs, Cas9 e GFP. Dois sgRNAs visando TP53 foram introduzidos em px458_Conc2 vetor por clonagem de portão dourado (análogamente a Andersson-Rolf et al.19). 10 μg de DNA plasmídeo foram eletroporados em organoides gástricos normais humanos(A). Os clones foram selecionados pela administração Nutlin3(B)e o nocaute tp53 foi confirmado pela clonagem e sequenciamento do TOPO TA dos alelos, aqui exemplarmostrado para um clone(C). As sgRNAs são sublinhadas na referência. Barra de escala = 200 μm. BF = campo brilhante. Clique aqui para ver uma versão maior deste valor.
Discussion
Este protocolo fornece instruções detalhadas para uma eletroporação eficiente, rápida e fácil de realizar de diferentes entidades organoides. Além dos organoides tumorais apresentados de PDAC, CRC, CCC e GC, funciona com sucesso para organoides derivados de tecido saudável também. O protocolo pode ser realizado dentro de um dia. Em protocolos de transfecção organoide publicados, todo o procedimento durou quatro dias, incluindo dois dias de preparações com diferentes tipos de mídia de cultivo10,21. Em nosso protocolo não é necessário pré-tratamento especial. Ao lavar com tampão de eletroporação antes da eletroporação, os componentes antibióticos da mídia foram lavados e um ajuste das concentrações salinos para valores ideais de impedância foi alcançado. No entanto, alguns aspectos críticos devem ser considerados para uma eletroporação bem sucedida:
Células
No protocolo de eletroporação de Fujii et al.10 é recomendável dissociar organoides para células únicas e filanhê-los através de um filtro de células de 20 μm. Em nossas mãos, a digestão de células únicas diminui fortemente a sobrevivência das células. Como sugerido em Merenda et al.21,também dissociamos organoides para aglomerados de 10-15 células e não conseguimos determinar uma diminuição da eficiência em comparação com a dissociação de células únicas. Após a eletroporação, é um passo muito importante para dissociar a espuma branca, para que nenhuma célula anexa esteja se perdendo.
Para a cultura celular 2D, mostrou-se que um tempo de regeneração após a eletroporação de mais de 10 min até 40 min aumenta a capacidade de sobrevivência e a eficiência da transfecção especialmente de grandes plasmídeos22. Em experimentos de teste, o mesmo poderia ser documentado para organoides, levando a uma etapa de incubação de 40 min após a eletroporação neste protocolo. Para aumentar a recuperação da eletroporação, nós os culturamos com o inibidor de proteína associada à Rho (ROCK) Y-27632 por cinco a sete dias23. Da mesma forma, a suplementação adicional do inibidor de synthase sintetizada 3 (GSK3) de glicogênio é destinada a ajudar células únicas a recuperar10.
Configurações
Uma das vantagens do eletroporator usado é que a impedância pode ser medida antes da eletroporação para condições ideais. De acordo com o fabricante, os valores de impedância devem ser de 30-55 Ω. Em nossas mãos, os valores de impedância de 30-40 Ω mostraram eficiências ideais. Em um experimento preliminar, diferentes tensões e valores de comprimento de pulso do pulso de porção foram variados para encontrar a proporção ideal de eficiência para a sobrevivência. Em resumo, pudemos confirmar os valores descritos de Fujii et al.10 nas diferentes entidades aqui descritas.
Dna
O efeito de diferentes quantidades de DNA foi testado em experimentos preliminares de até 45 μg de DNA por amostra. Nenhum efeito citotóxico pode ser detectado. A eficiência da transfecção foi aumentada de forma dependente de dose com saturação > 30 μg. Então, usamos 30 μg por amostra no protocolo final, mas é claro que pode ser aumentado (por exemplo, para a eletroporação de mais plasmídeos em paralelo). Além disso, a pureza e concentração do DNA parecem ser muito importantes. Uma concentração superior a 5 μg/μL mostrou eficiências de transfecção ideais.
Como esperado, o plasmídeo de 9,3 kB poderia ser transfeclado com uma eficiência menor do que o menor plasmídeo de 4,2 kB (ver Figura 4). O uso de plasmídeos ainda maiores do que 10 kB deve diminuir ainda mais a eficiência. Para aplicações futuras, pode ser interessante testar o DNA do minicírculo como vetor, uma vez que esses portadores de genes não têm a espinha dorsal bacteriana de um plasmídeo que os torna menores24. Isso deve resultar em uma maior eficiência de transfecção. Além disso, para manipulações baseadas em CRISPR de organoides, uma eletroporação direta de sgRNAs ligadas a Cas9 como um complexo de ribonucleoproteína (RNP) pode ser uma alternativa ouadição 25.
Disclosures
Os autores não têm nada para divulgar.
Acknowledgments
Agradecemos a Juliane Fohgrub, Ann-Christin Meinecke e Max Heiduk por excelente assistência técnica. O financiamento foi fornecido pela Deutsche Krebshilfe (nº 111350 e 70112925), Sander Stiftung (Nº 2014.104.1), Hector Stiftung (No M65.2) e pela União Europeia (ERC nº 639050).
Materials
| Name | Company | Catalog Number | Comments |
| For establishment and culture medium | |||
| [Leu15] Gastrin | Sigma-Aldrich | G9145 | |
| A83-01 | Tocris Bioscience | 2939 | |
| Advanced DMEM/F-12 | Invitrogen | 12634010 | |
| B27 | Invitrogen | 17504044 | |
| B27 Supplement, minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| CHIR99021 | Stemgent | 04-0004 | |
| Collagenase II | Life Technologies | 17101-015 | |
| Collagenase XI | Sigma-Aldrich | C9407-100MG | |
| Collagenase D | Roche | 11088866001 | |
| Dispase II | Roche | 4942078001 | |
| Dnase I | Sigma-Aldrich | D5319 | |
| D-sorbitol | Roth | 6213.1 | |
| Dithiothreitol | Thermo Scientific | 1859330 | |
| EDTA | Roth | 8040 | |
| Forskolin | Tocris Bioscience | 1099 | |
| Glutamax | Life Technologies | 35050061 | |
| Hepes | Thermo Fisher Scientific | 15630106 | |
| hFGF-10 | Preprotech | 100-26 | |
| KCl | Sigma-Aldrich | P9541 | |
| KH2PO4 | Roth | 3904.2 | |
| Matrigel | Corning | 356231 | basement matrix |
| mEGF | Invitrogen | PMG8043 | |
| N2 | Invitrogen | 17502048 | |
| NaCl | Roth | 3957.1 | |
| Na2HPO4 | Roth | K300.2 | |
| N-Acetyl-L-Cystein | Sigma-Aldrich | A9165 | |
| Nicotinamid | Sigma-Aldrich | N0636 | |
| Noggin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (Hek293-mNoggin-Fc) |
| PBS | Gibco | 14190169 | |
| Penicillin Streptomycin | Life Technologies | 15140122 | |
| Primocin | InvivoGen | ant-pm-1 | |
| Prostaglandin E2 | Tocris Bioscience | 2296 | |
| Recombinant Human HGF | Preprotech | 100-39H | |
| Rspondin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (HA-Rspo1-Fc-293T) |
| SB202190 | Sigma-Aldrich | S7067 | |
| Sucrose | VWR | 27,480,294 | |
| TrypLE Express | Gibco | 12604021 | Dissociation reagent |
| Wnt3A | n.a. | n.a. | Conditioned medium produced from L-Wnt3a cells (from Sylvia Boj) |
| Y-27632 | Sigma-Aldrich | Y0503 | |
| Consumables | |||
| Cell Strainer 100 µm | Falcon | 352360 | |
| 48-well plate | Corning | 3548 | |
| Nepa Electroporation Cuvettes 2mm gap w/pipettes | Nepa Gene Co., Ltd. | EC-002S | |
| Tubes 15 ml | Greiner | 188271 | |
| Tubes 15 ml low binding | Eppendorf | 30122208 | Tubes for FACS preparing |
| Equipment | |||
| Electroporator Nepa21 | Nepa Gene Co., Ltd. | n.a. | |
| EVOS FL Auto | Invitrogen | AMAFD1000 | Fluorescence microscope |
| LSRFortessa | BD Bioscience | 647800E6 | FACS |
| Reagents and plasmids | |||
| Live/dead fixable blue dead cell stain kit | Invitrogen | L34962 | Includes antibody for live/dead staining for FACS analysis |
| EndoFree Plasmid Maxi Kit | Qiagen | 12362 | |
| Nutlin-3 | Selleckchem | S1061/07 | |
| Opti-MEM | Gibco | 31985047 | Electroporation buffer |
| 2 gRNA concatemer vector | AddGene | 84879 | |
| pCMV-EGFP | Nepa Gene Co., Ltd. | n.a. | |
| px458 plasmid | AddGene | 48138 | coding for sgRNA and Cas9 |
| px458_Conc2 plasmid | AddGene | 134449 | px458 plasmid containing 2x U6 promotors for two different sgRNAs |
| sgRNA_hTP53_1a | Eurofins Genomics | n.a. | 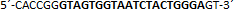 |
| sgRNA_hTP53_1b | Eurofins Genomics | n.a. | 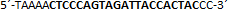 |
| sgRNA_hTP53_2a | Eurofins Genomics | n.a. | 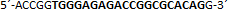 |
| sgRNA_hTP53_2b | Eurofins Genomics | n.a. | 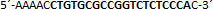 |
References
- Huch, M., et al. Long-term culture of genome-stable bipotent stem cells from adult human liver. Cell. 160 (1-2), 299-312 (2015).
- Werner, K., Weitz, J., Stange, D. E. Organoids as Model Systems for Gastrointestinal Diseases: Tissue Engineering Meets Genetic Engineering. Current Pathobiology Reports. 4 (1), 1-9 (2016).
- Olayanju, A., Jones, L., Greco, K., Goldring, C. E., Ansari, T. Application of porcine gastrointestinal organoid units as a potential in vitro tool for drug discovery and development. Journal of applied toxicology. 39 (1), 4-15 (2019).
- Drost, J., et al. Sequential cancer mutations in cultured human intestinal stem cells. Nature. 521 (7550), 43-47 (2015).
- Matano, M., et al. Modeling colorectal cancer using CRISPR-Cas9-mediated engineering of human intestinal organoids. Nature Medicine. 21 (3), 256-262 (2015).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Drost, J., et al. Use of CRISPR-modified human stem cell organoids to study the origin of mutational signatures in cancer. Science. 358 (6360), 234-238 (2017).
- Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S., Yang, S. H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy Nucleic Acids. 4, e264 (2015).
- Tsong, T. Y. Electroporation of cell membranes. Biophysical journal. 60 (2), 297-306 (1991).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nature Protocols. 10 (10), 1474-1485 (2015).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Bartfeld, S., et al. In vitro expansion of human gastric epithelial stem cells and their responses to bacterial infection. Gastroenterology. 148 (1), 126-136 (2015).
- Boj, S. F., et al. Organoid models of human and mouse ductal pancreatic cancer. Cell. 160 (1-2), 324-338 (2015).
- Broutier, L., et al. Culture and establishment of self-renewing human and mouse adult liver and pancreas 3D organoids and their genetic manipulation. Nature Protocols. 11 (9), 1724-1743 (2016).
- Broutier, L., et al. Human primary liver cancer-derived organoid cultures for disease modeling and drug screening. Nature Medicine. 23 (12), 1424-1435 (2017).
- Hennig, A., et al. CFTR Expression Analysis for Subtyping of Human Pancreatic Cancer Organoids. Stem Cells Int. 2019, 1024614 (2019).
- Seidlitz, T., et al. Human gastric cancer modelling using organoids. Gut. 68 (2), 207-217 (2019).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Current protein & peptide science. 9 (4), 338-369 (2008).
- Andersson-Rolf, A., et al. Simultaneous paralogue knockout using a CRISPR-concatemer in mouse small intestinal organoids. Dev Biol. 420 (2), 271-277 (2016).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8, 2281 (2013).
- Merenda, A., et al. A Protocol for Multiple Gene Knockout in Mouse Small Intestinal Organoids Using a CRISPR-concatemer. Journal of Visualized Experiments. (125), (2017).
- Lesueur, L. L., Mir, L. M., Andre, F. M. Overcoming the Specific Toxicity of Large Plasmids Electrotransfer in Primary Cells In Vitro. Molecular Therapy Nucleic Acids. 5, e291 (2016).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
- Darquet, A. M., Cameron, B., Wils, P., Scherman, D., Crouzet, J. A new DNA vehicle for nonviral gene delivery: supercoiled minicircle. Gene Therapy. 4 (12), 1341-1349 (1997).
- Jacobi, A. M., et al. Simplified CRISPR tools for efficient genome editing and streamlined protocols for their delivery into mammalian cells and mouse zygotes. Methods. 121-122, 16-28 (2017).



