Summary
Este protocolo describe un método de electroporación eficiente para la transfección de cuatro entidades organoides gastrointestinales diferentes con plásmidos más grandes (hasta una extensión de 10 kB). Se puede realizar en el plazo de un día y no necesita una preparación extensa o amortiguadores de electroporación especiales y costosos.
Abstract
La electroporación es un método común para la transfección con diferentes tipos de moléculas por permeabilización eléctrica de la membrana plasmática. Con el uso cada vez mayor de organoides como método de cultivo para el material primario del paciente en los últimos años, se necesitan métodos de transferencia eficientes de componentes para la ingeniería genética en este sistema de cultivo 3D. Especialmente para los organoides, la eficiencia de las manipulaciones genéticas depende de una transfección exitosa. Así, este protocolo fue desarrollado para facilitar la electroporación de organoides y para demostrar su funcionalidad universal en diferentes entidades. Los organoides de cáncer colorrectal, pancreático, hepático y gástrico humano se electroescanearon con éxito con plásmidos pequeños y grandes en comparación. Sobre la base de los vectores de codificación GFP, la eficiencia de la transfección fue determinada por FACS. No se requiere una preparación extensiva de las células o de los amortiguadores de electroporación especiales y costosos, y el protocolo se puede realizar en el plazo de un día.
Introduction
En los últimos años, se desarrolló un nuevo sistema de cultivo celular 3D, llamado organoides, para diversos tejidos normales y cancerosos. Los organoides están funcional y morfológicamente muy cerca de su tejido de origen. Se pueden generar a partir de diferentes especies, son fácilmente ampliables, genómicamente estables y genéticamente modificables, lo que los convierte en un sistema modelo ideal para las investigaciones genéticas1,2,3. Las técnicas de ingeniería genética como el sistema CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats)/Cas9 permiten diversas manipulaciones. La selección de clones se puede realizar mediante condiciones de medios definidas, por ejemplo, mediante la retirada de ligando WNT para APC (Adenomatosis Polyposis Coli) clones nocaut4,5. Alternativamente, los marcadores de selección tienen que ser introducidos por la reparación dirigida homóloga de un vector de segmentación6,7. Debido al hecho de que a menudo más de un plásmido necesita ser introducido, una transfección eficiente se convierte en un parámetro crucial. Además, para reducir los efectos fuera de destino inespecíficos, una expresión transitoria de la endonucleasa Cas9 es deseable8.
La electroporación es un método comparativamente simple para transfectar células con ADN, ARN, proteínas u otras macromoléculas. Por medio de pulsos eléctricos, la membrana celular se vuelve más permeable y causa un aumento de la aceptación9. En un protocolo de electroporación previamente publicado de organoides de colon se alcanzó una eficiencia del 30 % con un vector de expresión de GFP (proteína fluorescente verde) (7,4 kB) en un procedimiento de cuatro días10. El siguiente protocolo fue desarrollado para facilitar una transfección eficiente de organoides cancerosos o sanos con plásmidos grandes codificando para ARN de guía única (sgRNA) y la secuencia de endonucleasa Cas9 (por ejemplo, px458 como vector con 9,3 kb). Todo el proceso de electroporación se puede realizar en un día, sin amortiguadores especiales de electroporación, y con al menos eficiencias comparables entre diferentes organoides gastrointestinales, a saber, adenocarcinoma ductal pancreático (PDAC), cáncer colorrectal (CRC), colangiocarcinoma (CCC) y organoides de cáncer gástrico (GC).
Protocol
La aprobación ética se obtuvo del comité ético de la TU Dresden (#EK451122014).
1. Cultivo organoide o preparaciones antes de la electroporación
- Establecer organoides mediante la digestión tisular como se describió anteriormente y ampliarlos con su correspondiente medio de cultivo específico de la entidad en una matriz de sótano (ver Tabla 1 y Tabla de Materiales)11,12,13,14,15,16,17.
NOTA: Para las muestras de tejido humano es necesario el consentimiento informado y la aprobación del estudio por un comité ético. - Placas de precalentamiento de 48 pocillos a 37oC para la sembración postelectroporación.
- Preparar el medio basal sin antibióticos, así como el medio de cultivo organoide específico de la entidad con antibióticos (ver Tabla 1) incluyendo 10 M Y-27632 y 3 M CHIR99021.
NOTA: La abstinencia de antibióticos es importante para reducir los efectos tóxicos. Y-27632 y CHIR99021 mejorar la recuperación celular10. -
Preparación de los organoides (ver Figura 1)
- Cultivar 5 pozos de organoides en una placa de 48 pocillos por muestra de electroporación en medio de cultivo.
NOTA: Se deben utilizar organoides proliferativos (alrededor de 2-3 días después de la última división). - Preparar 230 l de reactivo de disociación (ver Tabla de Materiales),incluyendo 10 M Y-27632 por pozo.
- Retire el medio de cultivo de cada pocal y disociar los organoides mecánicamente en 230 ml de la mezcla de disociación preparada. Piscina 5 pozos por muestra de electroporación en un tubo de 15 ml.
- Mezclar por vórtice e incubar durante 5-15 min a 37 oC hasta que se produzcan racimos de 10-15 células. Por lo tanto, compruebe la disociación microscópicamente. Detener la digestión añadiendo medio basal sin antibióticos de hasta 10 ml.
NOTA: ¡Este paso es muy crítico! La eficiencia de la electroporación se reducirá, cuando la incubación es demasiado corta, pero la digestión prolongada reducirá la supervivencia. - Centrifugar a 450 x g durante 5 min a temperatura ambiente, desechar el sobrenadante y lavar dos veces con 4 ml de tampón de electroporación (ver Tabla de Materiales).
- Cultivar 5 pozos de organoides en una placa de 48 pocillos por muestra de electroporación en medio de cultivo.
2. Electroporación
NOTA: El siguiente protocolo está desarrollado para electroporadores capaces de ondas cuadradas y secuencias de pulsos de poro y transferencia separadas (ver Figura 2). Opcionalmente, los valores de impedancia, así como los voltajes, corrientes y energías transferidas a la muestra se pueden medir como control para experimentos reproducibles.
- Resuspender el gránudo organoide en 100 l de tampón de electroporación (ver Tabla de Materiales)que contiene 30 g de ADN plásmido.
NOTA: La concentración del ADN plásmido utilizado debe superar los 5 g/l para una concentración óptima de sal durante el proceso de electroporación. Por lo tanto, se recomiendan maxi kits de plásmido endofree (ver Tabla de Materiales)para la preparación de vectores. Se puede utilizar una cantidad total de hasta 45 g de ADN sin efectos citotóxicos. - Dispensar la mezcla completa ADN-organoides en una cubeta de electroporación con un ancho de espacio de 2 mm sin producir burbujas de aire.
- Establezca los parámetros de electroporación de acuerdo con Fujii et al.10 (véase tabla 2, Figura 2).
- Mezclar las células ligeramente sin espuma tocando la cubeta con un dedo. Coloque la cubeta en la cámara de la cubeta.
- Pulse el botón de la tecla de fijación del electroporator y anote el valor de impedancia.
NOTA: Una impedancia entre 30-40o mostró los mejores resultados. En general, debe estar en el rango entre 30-55o. Si este no es el caso, por favor controle los siguientes aspectos: anchura de la cubeta utilizada, conexiones de cable del electroporator, posibles burbujas de aire, volumen correcto y concentración de sal de la mezcla de electroporación. - Pulse el botón Inicio para ejecutar el programa de electroporación y controlar los valores de corrientes, voltajes y energías mostradas.
NOTA: Los valores de voltajes, corrientes y energías medidos deben corresponder a los parámetros de electroporación establecidos. Para la comparación de experimentos repetidos, puede ser útil tener en cuenta estos datos. - Después de la electroporación, agregue inmediatamente 500 ml de medio de cultivo sin antibióticos (con CHIR99021 e Y-27632; ver paso 1.3). Mezclar pipeteando hacia arriba y hacia abajo para disociar la espuma blanca.
NOTA: La espuma blanca aparece después del proceso de electroporación y un número significativo de células se une a ella. Por lo tanto, la disociación de la misma es muy importante para no perder células. - Transfiera la muestra completamente de la cubeta a un nuevo tubo de 15 ml utilizando la pipeta que pertenece a las cubetas de electroporación (ver Tabla de Materiales). Se recomienda volver a envolver la cubeta con un medio basal para obtener las células restantes.
- Para la regeneración de las células, incubarlas durante 40 min a temperatura ambiente.
3. Sembrado de células
- Centrifugar las células a 450 x g durante 5 min a temperatura ambiente y desechar el sobrenadante.
- Resuspenda el gleta en 100 ml de matriz de sótano y la semilla de 20 ol gotas en una placa precalentada de 48 pocillos (ver paso 1.2). Incubar durante 10 min a 37oC para polimerización y añadir 250 oL de medio de cultivo, que se complementa con Y-27632 y CHIR99021 hasta la siguiente división de los organoides cultivados (alrededor de 5-7 días).
4. Determinación de la eficiencia de la transfección
NOTA: En general, se recomienda electroportar un vector que lleva un marcador de fluorescencia como control de transfección adicional. Dependiendo del marcador elegido y su maduración cromófora la fluorescencia será visible dentro de alrededor de 24-48 h después de la transfección18.
- Compruebe la fluorescencia microscópicamente después de 24-48 h en el control de transfección (ver Figura 4B).
-
Escaneo de celdas fluorescentes activadas (FACS)
- Cosecha las células de forma análoga al paso 1.4.2-1.4.4 y digiere alrededor de 10-20 min hasta que haya células individuales. Agregue hasta 10 ml de solución salina con fosfato (PBS).
- Centrifugar a 450 x g durante 5 min a temperatura ambiente y desechar el sobrenadante.
- Opcionalmente para la discriminación de células vivas: Resuspende el pellet en 1 ml de solución salina con fosfato (PBS) y añada un anticuerpo adecuado (ver Tabla de Materiales)o yoduro de propidio (PI). Mezclar con mucho cuidado sólo tocando e incubar durante 30 minutos a temperatura ambiente en la oscuridad. Lavar con 10 ml de PBS, centrifugar y desechar el sobrenadante.
- Resuspenda el gránulo celular en 200 ml de PBS y, opcionalmente, filtre la suspensión a través de un colador de células de 100 m en un tubo FACS.
- Analice las celdas por una máquina FACS utilizando una estrategia de gating apropiada (consulte la figura 3; Figura 4A) y determinar la eficiencia de la transfección.
Representative Results
Los organoides de cuatro entidades cancerosas diferentes (CRC, CCC, PDAC, GC) se electroportaron al menos 3 veces utilizando 30 g de un plásmido pequeño (pCMV-EGFP, 4,2 kb) o un plásmido grande (px458, 9,3 kb). Ambos vectores llevan un casete GFP que permite la determinación de la eficiencia de transfección 48 h después de la electroporación por citometría de flujo. Para analizar sólo las células vivas, se tiñen con un anticuerpo de muerte de por vida antes de la exploración. La estrategia de gating se muestra en la Figura 3.
En las cuatro entidades organoides, el plásmido de 4,2 kB se transfectó con mayor eficiencia en comparación con el más grande (véase la figura 4). El transfecto más eficiente del plásmido pequeño se alcanzó en organoides pDAC con células positivas de 92,1 a 5,2 % de GFP, mientras que el plásmido grande se transfectó con una eficiencia del 46,7 a 3,7 % (media de desviación estándar, n a 3). En comparación con los organoides del cáncer de páncreas, el plásmido más grande se transfectó de manera más eficiente en organoides de la CRC con una eficiencia media del 53,4 al 11,7 %, mientras que el pequeño plásmido se transfectó con una eficiencia media de 84,3 a 5,8 %. La entidad más difícil de transfectar fueron los organoides del cáncer gástrico: tanto para el plásmido grande como para el pequeño, se alcanzó la eficiencia de transfección más baja en esta entidad (32,3 a 12,7 % y 74,1 a 5,5 %, respectivamente). Los organoides CCC mostraron una eficiencia media de transfección de 83,0 a 13,1 % para el plásmido pequeño y para el plásmido grande se obtuvieron un 39,5 % a 10,4 %.
Como prueba de concepto, los organoides humanos normales del estómago fueron electroporados con una codificación de plásmido px458_Conc2 para Cas9, GFP y dos sgRNAs dirigidos a TP53. Las roturas de doble hebra inducidas por Cas9 en el exón 8 fueron reparadas por unión final no homóloga (NHEJ), dando como resultado cambios de marco y, por consiguiente, un nocaut del gen (véase la figura suplementaria 1).
Tabla 1: Composición de medios basales, mezclas de digestión y medios de cultivo. Haga clic aquí para ver este archivo (haga clic con el botón derecho para descargar).

Tabla 2: Ajustes de electroporación según Fujii et al.10.

Figura 1: Flujo de trabajo de preparación de electroporación. En primer lugar, los organoides deben disociarse a grupos de 10-15 células y los antibióticos deben ser lavados. Después de la electroporación, la espuma blanca debe disociarse. Las células se pueden sembrar después de regenerar se gisen durante 40 minutos a temperatura ambiente. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Electroporación en dos pasos. Dos pulsos de ayuno con mayor voltaje y corta duración (175 V y 157,5 V, cada uno para 5 ms, pausa para 50 ms, decaimiento de tensión 10%) conducen a la formación de poros en las membranas celulares. Los siguientes pulsos de transferencia suministran el ADN a las células: cinco pulsos de transferencia positivos (con 20 V, 12 V, 7.2 V, 4.32 V y 2.592 V, cada uno para 50 ms, pausa para 50 ms, decaimiento de tensión 40%), seguido de cinco pulsos de transferencia intercambiados por polaridad (con 20 V, 12 V, 7.2 V, 4.32 V y 2.592 V, cada uno para 50 ms, pausa para 50 ms, tensión dedecafido 40). Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Estrategia representativa de gating mostrada por los organoides de la CCC. Todos los organoides electroportados fueron analizados por citometría de flujo 48 h después de la electroporación. Las células electroporadas sin ADN plásmido se utilizaron como controles negativos. Las puertas se establecieron de la siguiente manera: (A) gating para la forma de la célula, (B,C) gating para células individuales (discriminación doble), (D) gating para células vivas (manchado con un anticuerpo para células apoptoticas) y (E,F) finalmente gating para células emisoras de eGFP (canal FITC). FSC - dispersión hacia adelante; SSC - dispersión lateral. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Eficiencia electroporación de cuatro entidades organoides. (A) Análisis FACS (n a 34, desviación estándar media y cada valor único se muestran) y (B) comparación visual por microscopio de fluorescencia. Barra de escala a 1.000 m. BF - campo brillante; CCC - colangiocarcinoma; CRC - cáncer colorrectal; GC - cáncer gástrico; PDAC - adenocarcinoma ductal pancreático. Haga clic aquí para ver una versión más grande de esta figura.

Figura suplementaria 1: Nocaut ejemplar basado en CRISPR/Cas9 de TP53 en organoides estomacales humanos normales. El vector px458_Conc2 (ver Tabla de Materiales)fue clonado combinando el vector concatemer de 2 gRNA, un generoso regalo de Bon-Kyoung Koo19,con px45820,lo que dio como resultado una codificación plásmido para 2 sgRNAs, Cas9 y GFP. Dos sgRNA dirigidos a TP53 se introdujeron en px458_Conc2 vector mediante clonación de puerta dorada (análogamente a Andersson-Rolf et al.19). Se electroporaron 10 g de ADN plásmido en organoides gástricos normales humanos (A). Los clones fueron seleccionados por la administración Nutlin3 (B) y el nocaut TP53 fue confirmado por la clonación TOPO TA y la secuenciación de los alelos, aquí se muestra ejemplar para un clon (C). Los sgRNA se subrayan en la referencia. Barra de escala de 200 m. BF - campo brillante. Haga clic aquí para ver una versión más grande de esta figura.
Discussion
Este protocolo proporciona instrucciones detalladas para una electroporación eficiente, rápida y fácil de realizar de diferentes entidades organoides. Además de los organoides tumorales presentados de PDAC, CRC, CCC y GC, funciona con éxito para organoides derivados de tejido sano también. El protocolo se puede realizar en el plazo de un día. En los protocolos de transfección organoides publicados todo el procedimiento duró cuatro días incluyendo dos días de preparaciones con diferentes tipos de medios de cultivo10,21. En nuestro protocolo no se requiere pretratamiento especial. Mediante el lavado con tampón de electroporación antes de la electroporación, se lavaron los componentes antibióticos de los medios y se logró un ajuste de las concentraciones salinas para obtener valores de impedancia óptimos. Sin embargo, algunos aspectos críticos deben ser considerados para una electroporación exitosa:
Células
En el protocolo de electroporación de Fujii et al.10 se recomienda disociar organoides a células individuales y filtrarlos a través de un colador celular de 20 m. En nuestras manos la digestión a células individuales disminuye fuertemente la supervivencia de las células. Como se sugiere en Merenda et al.21, también disociamos organoides a grupos de 10-15 células y no pudimos determinar una disminución de la eficiencia en comparación con la disociación de una sola célula. Después de la electroporación, es un paso muy importante para disociar la espuma blanca, de modo que no se pierdan células adheridas.
Para el cultivo celular 2D, se ha demostrado que un tiempo de regeneración después de la electroporación de más de 10 min hasta 40 min aumenta la supervivencia y la eficiencia de transfección especialmente de plásmidos grandes22. En los experimentos de prueba, lo mismo podría documentarse para los organoides, dando lugar a un paso de incubación de 40 minutos después de la electroporación en este protocolo. Con el fin de aumentar la recuperación de la electroporación, los cultivamos con inhibidor de la proteína quinasa asociada a Rho (ROCK) Y-27632 durante cinco a siete días23. Del mismo modo, la suplementación adicional de glucógeno sintasa quinasa 3 (GSK3) inhibidor CHIR99021 está destinado a ayudar a las células individuales a recuperar10.
Configuración
Una de las ventajas del electroporator utilizado es que la impedancia se puede medir antes de la electroporación para condiciones óptimas. Según el fabricante, los valores de impedancia deben ser de 30-55o. En nuestras manos, los valores de impedancia de 30-40o han demostrado una eficiencia óptima. En un experimento preliminar, diferentes voltajes y valores de longitud de pulso del pulso de porto fueron variados para encontrar la proporción óptima de eficiencia a la supervivencia. En resumen, podríamos confirmar los valores descritos de Fujii et al.10 en las diferentes entidades descritas aquí.
Adn
El efecto de diferentes cantidades de ADN se probó en experimentos preliminares de hasta 45 g de ADN por muestra. No se pudieron detectar efectos citotóxicos. La eficiencia de transfección se incrementó de forma dependiente de la dosis con saturación > 30 g. Por lo tanto, usamos 30 g por muestra en el protocolo final, pero por supuesto se puede aumentar (por ejemplo, para la electroporación de más plásmidos en paralelo). Además, la pureza y concentración del ADN parece ser muy importante. Una concentración superior a 5 g/l ha demostrado una eficiencia óptima de transfección.
Como era de esperar, el plásmido de 9,3 kB podría ser transinfectado con una eficiencia menor que el plásmido más pequeño de 4,2 kB (ver Figura 4). Se prevé que el uso de plásmidos aún más grandes de 10 kB disminuya aún más la eficiencia. Para futuras aplicaciones, podría ser interesante probar el ADN de minicírculo como vector, ya que estos portadores de genes carecen de la columna vertebral bacteriana de un plásmido que los hace más pequeños24. Esto debería dar lugar a una mayor eficiencia de transfección. Además, para las manipulaciones basadas en CRISPR de organoides una electroporación directa de los sgRNA unidos a Cas9 como complejo de ribonucleoproteína (RNP) podría ser una alternativa o adición25.
Disclosures
Los autores no tienen nada que revelar.
Acknowledgments
Agradecemos a Juliane Fohgrub, Ann-Christin Meinecke y Max Heiduk por su excelente asistencia técnica. La financiación fue proporcionada por Deutsche Krebshilfe (no 111350 y 70112925), Sander Stiftung (no 2014.104.1), Héctor Stiftung (no M65.2) y la Unión Europea (ERC no 639050).
Materials
| Name | Company | Catalog Number | Comments |
| For establishment and culture medium | |||
| [Leu15] Gastrin | Sigma-Aldrich | G9145 | |
| A83-01 | Tocris Bioscience | 2939 | |
| Advanced DMEM/F-12 | Invitrogen | 12634010 | |
| B27 | Invitrogen | 17504044 | |
| B27 Supplement, minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| CHIR99021 | Stemgent | 04-0004 | |
| Collagenase II | Life Technologies | 17101-015 | |
| Collagenase XI | Sigma-Aldrich | C9407-100MG | |
| Collagenase D | Roche | 11088866001 | |
| Dispase II | Roche | 4942078001 | |
| Dnase I | Sigma-Aldrich | D5319 | |
| D-sorbitol | Roth | 6213.1 | |
| Dithiothreitol | Thermo Scientific | 1859330 | |
| EDTA | Roth | 8040 | |
| Forskolin | Tocris Bioscience | 1099 | |
| Glutamax | Life Technologies | 35050061 | |
| Hepes | Thermo Fisher Scientific | 15630106 | |
| hFGF-10 | Preprotech | 100-26 | |
| KCl | Sigma-Aldrich | P9541 | |
| KH2PO4 | Roth | 3904.2 | |
| Matrigel | Corning | 356231 | basement matrix |
| mEGF | Invitrogen | PMG8043 | |
| N2 | Invitrogen | 17502048 | |
| NaCl | Roth | 3957.1 | |
| Na2HPO4 | Roth | K300.2 | |
| N-Acetyl-L-Cystein | Sigma-Aldrich | A9165 | |
| Nicotinamid | Sigma-Aldrich | N0636 | |
| Noggin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (Hek293-mNoggin-Fc) |
| PBS | Gibco | 14190169 | |
| Penicillin Streptomycin | Life Technologies | 15140122 | |
| Primocin | InvivoGen | ant-pm-1 | |
| Prostaglandin E2 | Tocris Bioscience | 2296 | |
| Recombinant Human HGF | Preprotech | 100-39H | |
| Rspondin | n.a. | n.a. | Conditioned medium produced from HEK293 cells (HA-Rspo1-Fc-293T) |
| SB202190 | Sigma-Aldrich | S7067 | |
| Sucrose | VWR | 27,480,294 | |
| TrypLE Express | Gibco | 12604021 | Dissociation reagent |
| Wnt3A | n.a. | n.a. | Conditioned medium produced from L-Wnt3a cells (from Sylvia Boj) |
| Y-27632 | Sigma-Aldrich | Y0503 | |
| Consumables | |||
| Cell Strainer 100 µm | Falcon | 352360 | |
| 48-well plate | Corning | 3548 | |
| Nepa Electroporation Cuvettes 2mm gap w/pipettes | Nepa Gene Co., Ltd. | EC-002S | |
| Tubes 15 ml | Greiner | 188271 | |
| Tubes 15 ml low binding | Eppendorf | 30122208 | Tubes for FACS preparing |
| Equipment | |||
| Electroporator Nepa21 | Nepa Gene Co., Ltd. | n.a. | |
| EVOS FL Auto | Invitrogen | AMAFD1000 | Fluorescence microscope |
| LSRFortessa | BD Bioscience | 647800E6 | FACS |
| Reagents and plasmids | |||
| Live/dead fixable blue dead cell stain kit | Invitrogen | L34962 | Includes antibody for live/dead staining for FACS analysis |
| EndoFree Plasmid Maxi Kit | Qiagen | 12362 | |
| Nutlin-3 | Selleckchem | S1061/07 | |
| Opti-MEM | Gibco | 31985047 | Electroporation buffer |
| 2 gRNA concatemer vector | AddGene | 84879 | |
| pCMV-EGFP | Nepa Gene Co., Ltd. | n.a. | |
| px458 plasmid | AddGene | 48138 | coding for sgRNA and Cas9 |
| px458_Conc2 plasmid | AddGene | 134449 | px458 plasmid containing 2x U6 promotors for two different sgRNAs |
| sgRNA_hTP53_1a | Eurofins Genomics | n.a. | 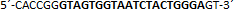 |
| sgRNA_hTP53_1b | Eurofins Genomics | n.a. | 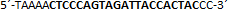 |
| sgRNA_hTP53_2a | Eurofins Genomics | n.a. | 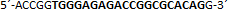 |
| sgRNA_hTP53_2b | Eurofins Genomics | n.a. | 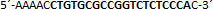 |
References
- Huch, M., et al. Long-term culture of genome-stable bipotent stem cells from adult human liver. Cell. 160 (1-2), 299-312 (2015).
- Werner, K., Weitz, J., Stange, D. E. Organoids as Model Systems for Gastrointestinal Diseases: Tissue Engineering Meets Genetic Engineering. Current Pathobiology Reports. 4 (1), 1-9 (2016).
- Olayanju, A., Jones, L., Greco, K., Goldring, C. E., Ansari, T. Application of porcine gastrointestinal organoid units as a potential in vitro tool for drug discovery and development. Journal of applied toxicology. 39 (1), 4-15 (2019).
- Drost, J., et al. Sequential cancer mutations in cultured human intestinal stem cells. Nature. 521 (7550), 43-47 (2015).
- Matano, M., et al. Modeling colorectal cancer using CRISPR-Cas9-mediated engineering of human intestinal organoids. Nature Medicine. 21 (3), 256-262 (2015).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Drost, J., et al. Use of CRISPR-modified human stem cell organoids to study the origin of mutational signatures in cancer. Science. 358 (6360), 234-238 (2017).
- Zhang, X. H., Tee, L. Y., Wang, X. G., Huang, Q. S., Yang, S. H. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy Nucleic Acids. 4, e264 (2015).
- Tsong, T. Y. Electroporation of cell membranes. Biophysical journal. 60 (2), 297-306 (1991).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nature Protocols. 10 (10), 1474-1485 (2015).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Bartfeld, S., et al. In vitro expansion of human gastric epithelial stem cells and their responses to bacterial infection. Gastroenterology. 148 (1), 126-136 (2015).
- Boj, S. F., et al. Organoid models of human and mouse ductal pancreatic cancer. Cell. 160 (1-2), 324-338 (2015).
- Broutier, L., et al. Culture and establishment of self-renewing human and mouse adult liver and pancreas 3D organoids and their genetic manipulation. Nature Protocols. 11 (9), 1724-1743 (2016).
- Broutier, L., et al. Human primary liver cancer-derived organoid cultures for disease modeling and drug screening. Nature Medicine. 23 (12), 1424-1435 (2017).
- Hennig, A., et al. CFTR Expression Analysis for Subtyping of Human Pancreatic Cancer Organoids. Stem Cells Int. 2019, 1024614 (2019).
- Seidlitz, T., et al. Human gastric cancer modelling using organoids. Gut. 68 (2), 207-217 (2019).
- Stepanenko, O. V., Verkhusha, V. V., Kuznetsova, I. M., Uversky, V. N., Turoverov, K. K. Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes. Current protein & peptide science. 9 (4), 338-369 (2008).
- Andersson-Rolf, A., et al. Simultaneous paralogue knockout using a CRISPR-concatemer in mouse small intestinal organoids. Dev Biol. 420 (2), 271-277 (2016).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8, 2281 (2013).
- Merenda, A., et al. A Protocol for Multiple Gene Knockout in Mouse Small Intestinal Organoids Using a CRISPR-concatemer. Journal of Visualized Experiments. (125), (2017).
- Lesueur, L. L., Mir, L. M., Andre, F. M. Overcoming the Specific Toxicity of Large Plasmids Electrotransfer in Primary Cells In Vitro. Molecular Therapy Nucleic Acids. 5, e291 (2016).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
- Darquet, A. M., Cameron, B., Wils, P., Scherman, D., Crouzet, J. A new DNA vehicle for nonviral gene delivery: supercoiled minicircle. Gene Therapy. 4 (12), 1341-1349 (1997).
- Jacobi, A. M., et al. Simplified CRISPR tools for efficient genome editing and streamlined protocols for their delivery into mammalian cells and mouse zygotes. Methods. 121-122, 16-28 (2017).



