Summary
여기에서는, 우리는 포르말린 고정된 파라핀 내장된 종양 조직 단면도에 있는 면역 세포를 구상하고, 정량화하고, 매핑하기 위한 간단하고 접근가능한 전략을 기술합니다. 이 방법론은 기존의 이미징 및 디지털 분석 기법과 이미징 분석의 다중 분석 기능 및 다중 매개 변수 분석을 확장하는 목적으로 결합합니다.
Abstract
종양 미세 환경 (TME)의 면역 풍경은 암 진행 및 치료에 대한 반응의 결정 인자입니다. 구체적으로, TME에 있는 면역 세포의 조밀도 그리고 위치는 중요한 진단 및 예후 값을 갖는다. TME의 다중 성 프로파일링은 종양 개시 및 진행을 조절하는 수많은 세포 및 분자 네트워크에 대한 우리의 이해를 기하 급수적으로 증가시켰습니다. 그러나, 이들 기술은 세포 또는 세포-세포 상호작용의 공간 조직에 대한 정보를 제공하지 않는다. 단일 세포 기반 의 높은 처리량 기술을 보완하기 위해 조직 섹션에서 면역 세포의 공간 적 분해를 허용하는 저렴하고 접근 가능하며 실행하기 쉬운 멀티플렉싱 기술이 필요합니다. 여기서는 직렬 이미징, 순차적 라벨링 및 이미지 정렬을 통합하여 전체 조직 섹션의 가상 다중 매개 변수 슬라이드를 생성하는 전략을 설명합니다. 가상 슬라이드는 관심 있는 셀 집단을 식별, 정량화 및 매핑할 수 있는 사용자 정의 프로토콜을 사용하여 자동화된 방식으로 분석됩니다. 이미지 분석은, 이 경우 분석 모듈 조직정렬, 저자 및 HISTOmap을 사용하여 수행된다. 우리는 우리가 1개의 임상 견본에 이 전략을 성공적으로 적용한 예를 제시하고, 한정된 조직 견본에서 장악될 수 있는 정보를 최대화하고 전체 조직 단면도에 있는 TME의 편견없는 보기를 제공하.
Introduction
암 발달은 악성 세포와 TME 사이 상호 상호 작용을 관련시키는 다단계 프로세스의 결과입니다. 종양 세포 이외에, TME는 비악성 세포, 기질 세포, 면역 세포 집단 및 세포외 매트릭스(ECM)1로구성된다. 종양 조직의 상이한 세포 및 구조적 구성 요소의 공간 조직및 암과 인접한 비암 세포 사이의 동적 교환은 궁극적으로 종양 진행및 치료에 대한 반응을 조절하여2,,3,,4. 암에서 면역 반응이55,6으로 조절되는 것으로 나타났습니다. 신생물 병변 및 인접한 조직에 침투하는 상이한 면역 세포 집단은 상이한 기능과 관련된 독특한 공간 분포 패턴 및 다양한 활성화 및 분화 상태를 나타낸다(예를 들어, 프로-대 항종양). 이 다른 면역 인구 및 그들의 매개변수는 종양및 기질 구획을 가진 초과 근무를 coevolve.
단 세포 multiomics 프로파일링을 허용하는 기술의 출현은 기하급수적으로 발암과 종양 진행을 통제하는 수많은 세포 및 분자 네트워크의 우리의 이해를 증가시켰습니다. 그러나, 대부분의 단일 세포 기반 고처리량 분석 도구는 조직 파괴 및 단일 세포 분리를 필요로 하며, 그 결과 세포 및 세포 상호 작용의 공간 조직에 대한 정보의손실7. TME에 있는 특정 면역 세포의 위치 그리고 배열은 진단과 예후 가치를 가지고 있기 때문에, 공간 해결책을 허용하는 기술은 단 세포 기지를 둔 면역 프로파일링 기술의 필수적인 보충입니다.
전통적으로, 면역성 화학 (IHC) 및 다중 면역 형광 (mIF)와 같은 화상 진찰 기술은 동시에 구상될 수 있는 몇몇 바이오마커로 제한되었습니다. 이 제한은 종양 침투 면역 세포의 spatiotemporal 역학의 연구 연구를 방해했습니다, 이는 전형적으로 몇몇 자형표 마커에 의해 정의됩니다. 최근 이미징 및 분석 도구의 발전으로 멀티플렉싱의 가능성이 확대되었습니다. 히스토 세포 분석 및 이미징 질량 세포측정과 같은 새로운 항체 기반 라벨링 기술은 각각8,,9,12 및 32 바이오마커를 공간적으로 분리하는 데 사용되어 왔다. 질량 분광법 화상 진찰은, 표지를 요구하지 않는 기술, 단일 조직 단면도10,,11에서동시에 수천개의 바이오마커를 심상할 가능성이 있습니다. 이 기술은 이미 암에 있는 조직 면역 경관을 해부하기위한 큰 잠재력을 보여주었지만, 그들은 매우 정교하고 비싼 장비 및 소프트웨어를 사용하고 연구원의 대다수에 쉽게 접근할 수 없습니다.
대안적으로, 전통적인 IHC 및 mIF의 다중화 능력은 직렬 이미징, 순차적 라벨링 라운드 및 스펙트럼 이미징7,,12,,13,,14,,15,,16의사용을 통해 확장되었다. 이러한 기술은 이미지 분석 소프트웨어를 사용하여 가상 다중 매개 변수 슬라이드로 통합 할 수있는 동일한 또는 직렬 조직 섹션에서 여러 이미지를 생성합니다. 그 결과, 동시에 시각화및 분석할 수 있는 마커의 수가 증가한다.
여기서, 시판시적 시약, 저렴한 현미경 장비 및 사용자 친화적인 소프트웨어를 이용한 조직 멀티플렉스 분석의 합리적 설계를 위한 전략을제안한다(그림 1). 이 방법론은 직렬 이미징, 순차적 멀티플렉스 라벨링, 전체 조직 이미징 및 조직 정렬을 통합하여 조직 섹션에서 면역 세포의 자동 정량화 및 매핑에 사용할 수 있는 가상 다중 매개 변수 슬라이드를 생성합니다. 이 전략을 사용하여 11개의 바이오마커와 자주 사용되는 조직학적 얼룩 2개(헤마톡실린및 에오신(H&E) 및 피크로시리우스 레드(PSR)로 구성된 가상 슬라이드를 만들었습니다. 다중 면역 세포 집단은 조직 히트맵을 사용하여 해결된 다른 조직 구획 및 그들의 공간 분포에서 확인되고, 위치되고, 정량화되었습니다. 이 전략은 제한된 임상 표본에서 얻을 수 있는 정보를 최대화하고 전체 조직, 코어 바늘 생검 및 조직 마이크로어레이를 포함한 포르말린 고정 파라핀 임베디드(FFPE) 보관 조직 샘플에 적용할 수 있습니다. 우리는 TME에 있는 면역 세포 인구의 식별, 정량화 및 지도에 대한 사용자 정의 적인 조사법을 디자인하기 위한 유용한 가이드로 이 방법론을 제안합니다.
Protocol
절제된 B형 간염 바이러스 (HBV)-관련 인간 간세포 암종에서 3개의 직렬 FFPE 단면도는 센터 병원제 드 l'Université de Montréal (CHUM) Hepatopancreatobiliary 암 임상 데이터베이스 및 생물학 견본에서 장악되었습니다 리포지토리 (HBP 바이오 뱅크). 이 조직 은행에 참여하는 환자는 정보에 입각한 동의를 제공했습니다. 이 연구는 기관 윤리위원회 (의정서 번호 09.237)에 의해 승인되고 헬싱키 선언에 따라 수행되었다.
1. 헤마톡실린 및 에오신 (H&E) 염색 프로토콜
참고: H&E 염색은 다음 프로그램을 사용하여 산동 다중 프로그램 로봇 슬라이드 염색기를 사용하여 센터 드 레체르체스 뒤 센터 hospitalier de l'Université de Montréal (CRCHUM)의 분자 병리학 핵심 시설에 의해 수행되었습니다.
- 탈파라핀화를 위해, 자일렌 대체품으로 각각 2.5분 동안 3배의 슬라이드를 담그십시오.
주의: 자일렌 대체품은 인화성, 피부 자극제, 흡입시 유해합니다. - 재수화를 위해 슬라이드를 100% 에탄올 3x에 각각 2.5분 동안 담그십시오. 이중 증류수(ddH 2O)에서21분 동안 세척하여 수분을 보충합니다.
- 헤마톡실린에서 1 분 동안 배양하십시오. ddH2O에서 각각 1 분 동안 3 x를 씻으소서.
- 에신과 함께 5 s용 인큐베이션. 95% 에탄올로 30s를 씻으소서. 100% 에탄올로 1분 동안 2x를 씻으소서.
주의: 에탄올은 인화성 및 눈 자극성입니다. 에신은 눈 자극제입니다. - 탈수의 경우, 자일렌 대체품에 각각 1.5분 동안 3x를 담그고 드시면 됩니다. 슬라이드를 수동으로 마운트합니다.
참고: 프로토콜의 이 부분을 실행하는 데 는 예상 시간이 30분입니다.
2. FFPE 섹션에 대한 멀티플렉스 면역 형광 염색 프로토콜
참고 :이 프로토콜은 로버트슨 외17에서적응되었다 .
- 탈파화 및 수화
참고: IHC 또는 mIF에 의한 FFPE 섹션의 항체 매개 라벨링 전에 파라핀을 제거해야 합니다. 파라핀을 효율적으로 제거하지 못하면 최적이 아닌 염색이 발생합니다.- 4 μm FFPE 조직 섹션 슬라이드를 유리 슬라이드 홀더에 넣습니다. 연기 후드 아래에서 37°C의 미리 따뜻해진 자일렌이 들어 있는 코플린 항아리에 슬라이드를 10분 동안 담그십시오.
주의: 자일렌은 인화성, 피부 자극성, 흡입시 유해합니다. - 2분마다 10초씩 슬라이드를 수동으로 교반합니다.
- 화학 후드에서, 슬라이드를 순차적으로 침지 5 분 각각의 솔루션: 1) 자일렌 : 에탄올 (1:1 v/v); 2) 100% 에탄올; 3) 70% 에탄올; 4) 50% 에탄올; 5) 30% 에탄올; 6) 인산염 완충식염수 (PBS).
참고: 항원 검색을 수행할 준비가 될 때까지 PBS에 슬라이드를 보관합니다. 항상 수분을 공급받은 부분을 유지하십시오. 건조는 비특이적 항체 결합을 일으키고 따라서 높은 배경 염색을 야기할 것이다.
- 4 μm FFPE 조직 섹션 슬라이드를 유리 슬라이드 홀더에 넣습니다. 연기 후드 아래에서 37°C의 미리 따뜻해진 자일렌이 들어 있는 코플린 항아리에 슬라이드를 10분 동안 담그십시오.
- 열 유도 항원 검색
참고 : 항원포르말린 고정시 마스크 될 수 있습니다, 항체 결합을 방지하고 결과적으로 시각화. 항원 마스킹 버퍼 및 절차의 사용은 부분적으로 에피토프의 기본 형성을 재확립하고 항체 인식을 복원합니다. 항원 회수 완충제 및 지속 시간의 유형은 특정 분석 조건(예를 들어, 표적, 항체, 조직 등)에 대해 최적화되어야 한다.- 항원 회수 용액을 함유한 코플린 항아리에 디왁드 슬라이드를 담그십시오(재료 표의레시피).
- 닫힌 코플린 항아리를 수돗물로 전기 압력 밥솥에 넣습니다. 물이 항원 회수 용액과 혼합되지 않도록 수위는 항아리의 절반 높이를 초과해서는 안됩니다.
- 밥솥의 뚜껑과 압력 밸브를 닫습니다. 10 분 동안 고압을 선택하고 시작합니다. 작업이 끝나면 밥솥의 플러그를 뽑고 압력을 풀고 뚜껑을 열고 항아리를 밥솥 내부에 30분 동안 보관하여 슬라이드가 식힙니다.
- 비특이적 바인딩 차단
- 슬라이드가 있는 랙을 PBS로 채워진 코플린 항아리로 옮김. 항원 회수 완충액을 PBS 2x로 각각 5분 동안 헹구는다.
- 소수성 장벽을 만들기 위해 PAP 펜으로 조직 섹션을 둘러싸는다. PBS에서 0.1 M 글리신이 들어있는 코플린 항아리에 슬라이드를 담그십시오. 실온(RT)에서 15분 동안 배양합니다.
참고: 글리신은 항원 회수 중에 생성된 알데히드 군을 포화시다. 이 단은 1 차적인 및 이차 항체를 비구체적으로 묶을 수 있었습니다. - 5 분 동안 PBS로 2x를 세척하여 글리신 용액을 헹구고 슬라이드를 습도 챔버에 넣고 모든 조직 섹션을 덮을 수있는 충분한 차단 솔루션을 추가하십시오. 소수성 장벽이 넘치지 않도록 하십시오. RT에서 30 분 동안 인큐베이션하십시오.
참고: 차단 솔루션의 레시피는 재료 표에서찾을 수 있습니다. 차단 용액은 비특이적 결합 부위를 차단하는 단백질(예를 들어, BSA)을 포함해야 한다. 또한 Triton X-100 또는 Tween 20과 같은 세제를 통합하여 항체와 조직 표적 간의 소수성 상호 작용을 감소시켜 항원 인식을 보다 선택적으로 만들 수 있습니다. 조직이 유래하는 종으로부터 10% 총 혈청을 첨가하면 Fc 수용체를 차단하고 비특이적 항체 결합을 감소시킬 수 있다. 마지막으로, 이차 항체가 제기된 종으로부터 혈청의 10%를 첨가하면 조직 섹션에 대한 이차 항체의 직접적인 비특이적 부착을 최소화할 것이다.
- 면역형광 라벨링
- PBS-Tween(0.1% v/v)으로 각각 5분 동안 2x로 헹구고 슬라이드를 습도 챔버에 다시 놓습니다.
- 차단 용액에서 다시 중단 된 1 차 항체의 칵테일을 추가합니다. 4 °C에서 밤새 배양. 이 연구에 사용되는 1 차 및 이차 항체는 재료 표에나열되어 있습니다.
참고 : 1 차 항체의 칵테일은 다른 종에서 제기 된 항체를 포함해야, 또는 같은 종에서 하지만 다른 아이소 타입의. 본 연구에서 사용된 1차 이차 항체 쌍의 목록은 표 2를참조하십시오. 사용되는 모든 항체의 세부 사항은 재료 표 및 표 2에있습니다. - PBS-Tween(0.1% v/v)으로 3x를 5분 간 헹구고 슬라이드를 다시 습도 챔버에 넣습니다. 어둠 속에서, 이차 항체의 칵테일을 추가하고 RT에서 1 시간 동안 배양하십시오.
참고 : 1 차 항체가 다른 종에서 일 때, 이차 항체는 그들 각각이 1 차적인 항체의 하나에만 결합하고 서로가 아니라 결합하도록 선택되어야 합니다. 이것은 일반적으로 이 종이 1 차적인 항체가 생성된 종에서 다이한 한 동일 종에서 제기된 이차 항체를 사용하여 달성됩니다. 1 차적인 항체가 동일 종에서 올려졌지만 다른 아이소타입이 있는 경우에, 이소타입 특정 이차 항체는 이용되어야 합니다. - PBS-Tween (0.1% v/v)으로 3x를 5분 동안 헹구어 보시고 자합니다. ddH2O. 초과 액체를 제거하고 DAPI를 장착한 마운팅 매체에 장착하십시오. 사용되는 볼륨은 섹션의 크기에 따라 다릅니다. 일반적으로 40 μL은 일반 현미경 슬라이드의 표면을 덮기에 충분합니다.
- 커버 슬라이드를 섹션에 놓고 거품 형성을 피하기 위해 여분의 장착 매체를 부드럽게 짜냅니다. 슬라이드를 어두운 RT에서 20 분 동안 건조시키고 획득 준비가 될 때까지 4 °C에서 보관하십시오.
- 전체 슬라이드 스캐너를 사용하여 모든 채널에 대한 이미지를 수집합니다(재료 표참조).
참고: 항체는 양성 대조군으로 인간 간세포 암종 조직을 사용하여 검증되었다. 각 1차 항체에 대해, 3개의 직렬 섹션은 염색 프로토콜의 나머지 부분에서 변이없이 각각 원발성 항체, 등소형 제어 또는 차단 용액으로 염색되었다. 획득된 이미지는 염색의 특이성을 확립하기 위해 비교되었다. 염색은 1차 항체로 배양된 섹션의 신호가 예상 패턴을 가지고 배경과 쉽게 구별될 때 특이적인 것으로 간주되었다. 1차 항체는 아이소타입에서 높은 배경 신호 또는 라벨링 조직 성분을 부여하고 1차 항체 섹션은 비특이적으로 간주되었다. 프로토콜의 이 부분을 완료하는 데 필요한 예상 시간은 2일입니다. 필수 제어는 다음과 같습니다: (1) 배경 신호에 대한 1차 항체의 비특이적 결합의 기여를 확립하기 위한 아이소타입 제어. 1개의 단면도는 조직 단부에 결석하는 표적을 위해 1 차적인 항체의 동일 이소형 및 기원을 가진 항체로 배양된다는 것을 제외하고는 다른 견본 조직과 같은 방법으로 염색됩니다. 적절한 이소형 조절 항체를 사용할 수 없는 경우, 1차 항체가 제기된 동일한 종으로부터 총 IgG로 대체할 수 있다; (2) 염색의 특이성을 확립하고 배경 신호에 대한 이차 항체의 비특이적 결합의 기여도를 추정하기 위한 1차 항체 제어(즉, 음성 대조군)가 없다. 이 경우, 제어 부는 1 차 항체가 첨가되지 않은 것을 제외하고는 다른 섹션과 동일한 방식으로 염색됩니다. (3) 얼룩이 효과가 있음을 확립하기 위한 포지티브 컨트롤. 이 경우, 염색은 1차 항체에 의해 인식된 마커를 발현하는 것으로 알려진 조직 절편에서 수행된다.
3. 피크로 시리우스 빨간색 (PSR)/빠른 녹색 염색 프로토콜
참고: 이 염색의 목표는 FFPE 조직 단면도에서 섬유질 콜라겐 I 및 III를 시각화하는 것입니다. 이 프로토콜은 세그나니 외18에서적응되었다. 모든 단계는 화학 후드에서 수행됩니다.
- FFPE 섹션에 대한 멀티플렉스 면역형광 염색 프로토콜과 유사한 조직 절편의 탈파카페인 및 재수화를 수행한다(섹션 2.1).
참고: 염색할 섹션이 이전에 면역형 광염 표지에 사용되었고 파라핀이 이미 제거된 경우, 탈파피화-재수화 단계는 마운팅 매체를 제거하는 데 유용하다. DAPI는 이 절차를 사용하여 제거되지 않지만 PSR 염색을 방해하지는 않습니다. - 피크로 시리우스 레드 /빠른 녹색 용액 (재료 표의조리법)을 포함하는 항아리에 슬라이드를 담그고 RT에서 30 분 동안 배양하십시오 (30 분 이상 간세포의 핵이 비특이적 인 염색을 초래함).
- ddH2O (5 딥)에서 슬라이드를 빠르게 씻으십시오. 그런 다음 에탄올 100 % (5 딥)으로 빨리 씻으하십시오. 자일렌 100% 에탄올(1:1 v/v)으로 30s를 세척하십시오. 자일렌에 30s를 씻으소서. 자일렌이 완전히 증발하기 전에 마운팅 매체(재료 표참조)로 장착하십시오(장착에 도움이 됩니다).
참고: 프로토콜의 이 부분을 실행하기 위한 예상 시간은 1시간입니다.
4. 조직 섹션에서 항체의 용출
참고: 순차적 라벨링 어세이에서 조직 섹션을 재사용하려면 1차 및 이차 항체를 완전히 제거해야 합니다. 바운드 항체는 앞서 설명한 바와 같이 제거되었다13.
수조를 56°C로 예열합니다. 스트리핑 버퍼가 들어있는 항아리 안에 섹션을 넣고 (재료 표의레시피), 뚜껑을 닫고 파라핀 필름 테이프로 밀봉하여 흔들리는 동안 누출되지 않도록하십시오.
- 항아리를 수조 안에 넣고 교반으로 30 분 동안 배양하십시오.
- PBS-Tween (0.1 % v / v)와 RT. 린스에서 ddH2O에서 각각 15 분 동안 4 x를 씻으소서.
- 1차 항체의 두 번째 라운드로 단면을 재조사할 준비가 될 때까지 PBS-Tween 또는 물에서 수화된 부분을 유지합니다.
참고: 프로토콜의 이 부분을 실행하기 위한 예상 시간은 2시간입니다. - 항체 용출 절차의 효율을 확인합니다.
참고: 순차적 라벨링 분석에서 항체 용출프로토콜을 사용하기 전에 1차 및 이차 항체의 제거 효율을 검증해야 합니다.- FFPE 단면도에 대한 다중 면역형광 염색 프로토콜에 나타난 바와 같이 주어진 1차 이차 항체 쌍으로 섹션의 염색 및 이미지 획득을 수행한다(섹션 2.1-2.4.6).
- 심상 취득 시, 섹션 4.1-4.3에 표시된 바와 같이 조직 결합 1차 이차 항체 복합체의 용출을 수행합니다.
- 2.4.3 단계에서 사용되는 동일한 이차 항체 및 동일한 조건으로 섹션을 배양한다.
- 2.4.4-2.4.6에 표시된 대로 세척, 장착 및 이미지 수집 단계를 수행합니다.
- 특정 신호가 사라졌는지 여부를 확인하기 위해 스트리핑 전후에 획득한 이미지를 나란히 비교합니다.
참고: 항체 제거 전후의 이미지를 비교하면 용출 절차의 효율성이 검증됩니다. 그러나, 모든 채널에서 배경 신호의 증가뿐만 아니라 DAPI의 확산을 보는 것은 정상이다. 이것은 동일한 조직 섹션에서 실행할 수있는 스트립의 라운드 수를 제한합니다. 스트리핑의 세 라운드는 최대 인 것 같다.
5. 이미지 수집
- 전체 슬라이드 스캐너를 사용하여 이미지를 생성합니다.
- 20x 0.75NA 대물렌즈와 0.3225μm/픽셀의 해상도를 사용합니다.
6. 이미지 분석
참고: 여기에 설명된 메서드는 현재 예제를 참조합니다. 다른 특정 샘플에 맞게 표 1 및 텍스트를 참조하십시오.
- 이미지 분석 소프트웨어의 티슈정렬 모듈을 사용하여 조직 정렬을 수행합니다(이 프로토콜의VIS, 재료 표 참조).
- 이미지 분석 소프트웨어를 열고 조직 정렬 탭을 클릭합니다.
- 파일 |으로 이동하여 슬라이드 트레이에 정렬할 이미지를 가져옵니다. 데이터베이스에서 정렬할 첫 번째 이미지를 선택합니다. [슬라이드 트레이]의 로드 버튼을 클릭하여 티슈정렬 탭으로 돌아가 이미지를 로드합니다. 이미지가 슬라이드 트레이와 작업 공간에 나타납니다.
참고: 관심 있는 스택만 슬라이드 트레이에 로드해야 합니다. - 정렬할 순서대로 모든 이미지에 대해 6.1.2 단계를 반복하여 하나씩 로드합니다. 관심 있는 모든 이미지가 슬라이드 트레이에 로드되면 리본의 워크플로 단계에서 다음을 눌러 이미지를 연결합니다.
- 다음으로 두 번째 이미지를 첫 번째 이미지 위에 드래그 앤 드롭합니다. 이제 첫 번째 및 두 번째 이미지가 연결됩니다. 다른 이미지가 순서대로 하나씩 정렬될 수 있도록 이 단계를 반복합니다. 첫 번째 이미지의 이름이 변경되어 다른 이미지와 연결되었음을 나타냅니다. 동시에 연결된 이미지가 슬라이드 트레이 오른쪽의 작업 공간에 표시됩니다.
- 이 시점에서 자동 정렬, 반자동 정렬 또는 수동 정렬을 사용하여 이미지를 정렬합니다. 자동 정렬을 먼저 시도하는 것이 항상 바람직합니다. 자동 정렬의 경우 리본의 워크플로 단계(3단계)의 다음 단추를 누릅니다.
- 조직의 다른 위치를 탐색하고 다른 이미지의 해당 구조가 이미지의 두 차원에서 동일한 방식으로 배열되어 있는지 육안으로 확인하여 자동 정렬을 검토합니다.
- 자동 정렬 결과가 만족스럽지 않은 경우 연결된 이미지의 상동 조직 특징을 나타내는 핀(이미지당 최소 3핀 사용)을 사용하여 개선합니다. 핀이 연결된 이미지의 상동 위치에 배치되면 사용자는 반자동 정렬 또는 수동 정렬이라는 두 가지 선택 사항을 선택할 수 있습니다. 반자동 정렬의 경우 리본의 현재 핀포인트를 기반으로 단추 자동 정렬을 클릭합니다. 수동 정렬을 위해 리본에 핀 적용 버튼을 클릭합니다.
- 정렬에 만족하면 워크플로 단계의 다음 단추를 클릭하고 데이터베이스에 합성 이미지를 저장합니다.
참고: 11개의 마커에 걸친 6개의 슬라이드와 H&E 및 PSR 이미지를 정렬하는 데 15분이 걸렸습니다.
- 사용자 정의 프로토콜 분석 프로토콜 패키지 1(APP 1, 표 1)을사용하여 조직 검출을 수행한다.
- 리본의 이미지 분석 탭을 클릭하여 소프트웨어의 이미지 분석 모듈을 엽니다.
- 파일 |으로 이동하여 합성(정렬된) 이미지 가져오기 데이터베이스 및 관심 있는 이미지를 선택하고 이미지 분석 탭을 다시 클릭합니다.
- 열기 APP 아이콘을 클릭하여 APP 선택 대화 상자를 열고 사용할 분석 프로토콜 패키지(APP)를 선택합니다. 이 경우 조직 검출을 위해 APP 1을 선택합니다.
- APP 1이 열리면 선택한 조직 위치로 이동하여 미리 보기 버튼을 클릭하여 APP1이 제대로 작동하는지 확인합니다. 결과가 만족스러프면 다음 단계로 이동하십시오.
- APP 1을 실행하고 선택한 APP를 사용하여 이미지를 처리하려면 클릭합니다.
- 파일/내보내기를 클릭하여 분석이 완료되면 데이터(예: 이미지, 측정 등)를 내보냅니다.
참고: APP 1은 조직(ROI 조직)을 고정하는 관심 영역(ROI)을 생성하고 조직의 영역을 계산합니다. - 파일 |으로 이동하여 새로 만든 ROI로 수정된 이미지를 저장합니다. 저장합니다.
참고: 제공된 예에서 조직을 검출하고 APP 1로 ROI를 생성하는 데는 설명된 이미지 분석 스테이션에서 5분이 걸렸습니다. 처리된 조직의 면적은 3.2 cm2였다.
- APP2(표 1)를사용하여 조직 분절을 기질및 자충으로 분절시한다.
참고: APP 2는 미리 정의된 ROI 조직에서 작동합니다. APP 2는 조직을 ROIs 스트로마 및 Parenchyma로 분할합니다.- 리본에서 이미지 분석 탭을 클릭하여 이미지 분석 모듈을 엽니다.
- FILE로 이동하여 ROI 조직이 포함된 이미지를 가져옵니다 | 6.2.7단계에서 저장된 이미지를 데이터베이스로 선택합니다. 이미지 분석 탭으로 돌아가 슬라이드 트레이의 로드 버튼을 클릭하여 이미지를 로드합니다. 이미지가 슬라이드 트레이와 작업 공간에 나타납니다.
- 6.2.3에서와 같이 APP 선택 대화 상자를 사용하여 APP 2를 엽니다.
- 선택한 시야에서 처리하여 APP 2를 미리 봅니다. 결과가 만족스러프면 실행 버튼을 클릭하여 전체 이미지에서 APP 2를 실행합니다. APP 2의 출력으로, ROI 조직은 ROIs Stroma 및 Parenchyma및 그 각각의 영역에서 분할됩니다. 수출 결과는 6.2.6. 6.2.7에서와 같이 수정된 이미지를 저장합니다.
참고: STROMa 및 Parenchyma에서 조직을 APP 2를 사용하여 분할하는 것은 제시된 분석 스테이션에서 4시간 걸렸습니다. 처리된 조직의 면적은 3.2 cm2였다.
- 사용자 정의 프로토콜 APP3(표 1)을사용하여 FoxP3hiCD4+ 셀을 식별하고 정량화합니다.
참고 : APP 3은 미리 정의 된 ROI 스트로마와 Parenchyma에서 작동합니다.- 이미지 분석 모듈을 열고 6.3.1 및 6.3.2에서와 같이 ROIs Stroma 및 Parenchyma가 포함된 이미지를 가져옵니다. 6.2.3에서와 같이 APP 선택 대화 상자를 사용하여 APP 3을 엽니다.
- FoxP3hiCD4+ 셀에서 보강된 선택된 시야에서 APP 3 처리를 미리 봅니다. 결과가 만족스러프면 전체 이미지에서 APP 3을 실행합니다. APP 3의 출력으로서, 모든 개별 FoxP3hiCD4+ 객체가 표지되고 그들의 조직 좌표가 저장됩니다. FOXP3hiCD4+ 개체의 밀도가 ROIs Stroma 및 Parenchyma에서 결정됩니다. 결과를 6.2.6으로 내보냅니다.
- FoxP3hiCD4+ 라벨이 부착된 물체의 조직 열 매핑을 수행합니다.
- 6.2.3에서와 같이 APP 선택 대화 상자를 사용하여 사용자 정의 프로토콜 FoxP3hiCD4+ MAP를 엽니다.
참고 : FoxP3안녕CD4 + MAP밀도 히트 맵을 생성하기위한 FoxP3안녕CD4 + 라벨 객체의 좌표를 사용합니다. APP 3을 사용하여 FoxP3hiCD4+ 라벨이 붙은 물체를 식별하고 계산하는 데는 설명된 이미지 분석 스테이션에서 25분이 걸렸습니다. 처리된 조직의 면적은 3.2 cm2였다. - 실행 버튼을 눌러 FoxP3안녕CD4 + MAP를 실행합니다. 파일 | 을 클릭하여 티슈 히트맵 내보내기 수출 | 작업 영역.
참고 : FoxP3hiCD4 + FoxP3hiCD4 + MAP을 사용하여 레이블이 붙은 객체를 매핑하는 것은 설명 된 이미지 분석 스테이션에서 5 분이 걸렸습니다.
- 6.2.3에서와 같이 APP 선택 대화 상자를 사용하여 사용자 정의 프로토콜 FoxP3hiCD4+ MAP를 엽니다.
- CD8+, CD68+, MPO+, αSMA 및 CD34+ 객체를 사용자 정의 프로토콜 APP 4, APP5, APP6, APP7 및 APP 8을 사용하여각각(표 1)섹션 6.4에서 6.4.3.2로 APP를 로드하는 경우를 식별하고 정량화합니다.
참고: APP 4~ 8은 미리 정의된 ROI 스트로마및 Parenchyma에서 작동합니다.
Representative Results
TME에 관심 있는 셀 모집단을 시각화, 정량화 및 매핑하기 위한 전략 개요
서로 다른 조직 구획(TC)에서 관심 있는 세포 집단(COI)을 정량화하고 공간 조직을 특성화하기 위해 저렴하고 사용하기 쉬운 기술을 통합하고 귀중한 FFPE 임상 표본에서 얻을 수 있는 위치 정보를 극대화하는 워크플로우를 설계했습니다(그림1). 먼저, 직렬 전체 조직 FFPE 절편은 COIs(예를 들어, 면역 세포) 및 TC(예를 들어, 기질 대 자렌치마)의 시각화를 위해 염색되었다(도1,단계 1). 염색될 연속적인 단면도의 수는 연구 질문을 해결하는 데 필요한 관심 있는 세포 또는 조직 특징의 가시화를 허용하는 최소한으로 유지되어야 합니다. 일련 섹션의 수가 작을수록 연속 섹션에 걸쳐 조직 아키텍처유사와 일치가 높아지다. 또한, 멀티플렉싱 기능은19의스트리핑 및 회수 기술을 통해 형광적으로 염색된 단면의 재사용을 통해 확장될 수 있다.
염색 단계가 완료되면 전체 슬라이드 스캐너를 사용하여 이미지를 디지털화했습니다. 직렬 섹션에서 획득한 이미지는 자동화된 방식으로 가상 멀티플렉스 슬라이드에 정렬및 통합되었습니다(그림1,섹션 2). 다음으로, 조직에 대한 ROI를 조직 관련 픽셀(TAP)을 식별하는 사용자 정의 프로토콜로 묘사하였다(도1,단계 3). 그 후, ROI 조직은 추가 ROI로 정의된 TC로 분할되었다. (그림1,4 단계). 다음으로, 사용자 정의 프로토콜은 서로 다른 TC에서 COI를 검출하고 정량화하였다(그림1,단계 5). 마지막으로, COI의 조직 히트맵은 그들의 밀도 및 그들의 조직 좌표에 기초하여 생성되었다(도1,단계 6).
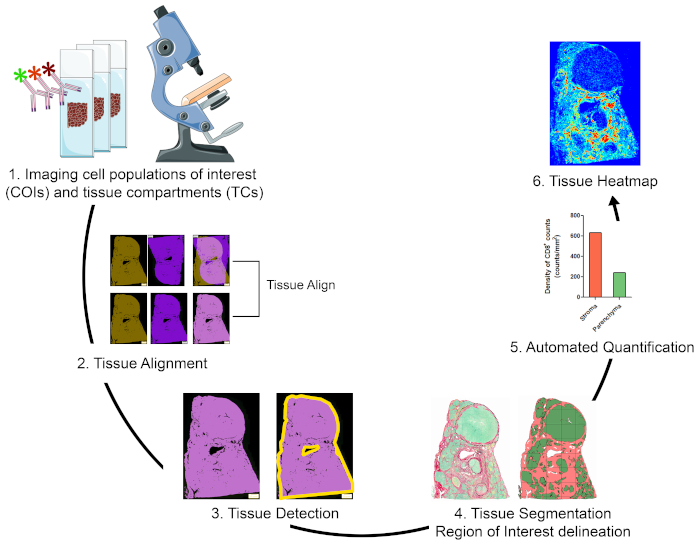
그림 1: TME에서 면역 세포를 시각화, 정량화 및 매핑하기 위한 전략의 개략적 표현. (1)직렬 전체 조직 섹션은 COI 및 TC라벨링을 위해 염색되었다. (2)직렬 섹션에서 획득한 이미지는 Tissuealign 분석 모듈을 사용하여 자동화된 방식으로 연결, 정렬 및 공동 등록되었습니다. 개별 이미지의 고정밀 정렬에서 합성 이미지가 생성되었습니다. (3)사용자 정의 프로토콜은 합성 이미지에서 조직 관련 픽셀(TAP)의 자동 검출을 위해 사용되었다. (4)조직을 ROI로 정의된 TC(예를 들어, 기질 및 실치마)로 분할하였다. (5)사용자 정의 프로토콜은 상이한 TC에서 COI의 자동 검출 및 정량화를 위해 사용되었다.(6)COI의 조직 히트맵이 생성되었다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이미징 COI 및 TC
HBV 관련 간세포 암종을 가진 대상체로부터의 절제된 종양의 3개의 직렬 FFPE 전체 조직 섹션은 도 2A에서와같이 하나 이상의 염색 라운드에서 염색되었다. 제1편은 H&E로 염색하여 조직 구조, 세포 형태, 악성종양의 유형, 종양 등급 및 면역 침윤의 전반적인 평가와 같은 임상적으로 관련있는 파라미터를 결정하기 위해하였다(도 2C). 연속된 섹션 II에서, mIF의 2라운드는 간 자영및 비-자영세포 표지에 사용하였다(도2A). 첫 번째 라운드에서, 정상 및 종양 혈관은 내피 세포의 CD34 염색을 사용하여 시각화되었다. 부가적으로, 상피 세포(간세포 및 담관옥세포)는 시토케라틴 8/18을 사용하여 확인되었고, 섬유성 활성화된 간담세포세포는 알파 평활근 액틴 양성(αSMA+) 세포(αSMA+) 세포로확인되었다(도 2C). 심상 취득 후, 조직 절편을 벗겨내고 대식세포(CD68) 및 근섬유아세포(desmin)에 대한 항체로 재프로브하였다. 종양 면역 침윤을 더 잘 특성화하기 위해, 인접한 직렬 섹션 III는 세포 마커 CD3, CD4, CD8, 지게차 상자 P3(FoxP3) 및 골수페록시다제(MPO)에 대한 2라운드의 mIF를 사용하여 염색하였다. 모든 경우에 DAPI는 핵 카운터스테인으로 사용되었다. 마지막으로, 섹션 III는 PSR 얼룩으로 염색하고 빠른 녹색으로 염색하여 세브릴라 콜라겐을 시각화하고 조직을 기질 및 실치종으로 분할하였다(도2C).
20X 대물 렌즈가 장착된 전체 슬라이드 스캐너를 사용하여 스테인드 섹션을 디지털화하고 가상 슬라이드를 만들었습니다. 3개의 직렬 섹션(그림2B)에서6개의 이미지를 획득하였고, 이후 도 1의개략적 표현에 따라 VIS 소프트웨어를 사용하여 분석된 가상 슬라이드.
이미지 분석
상기 영상 분석은 5단계로 구성되었다: 1) 조직 정렬; 2) 조직 검출; 3) 조직 세분화; 4) COI의 자동 정량화; 및 5) 조직 열 매핑. 이미지 분석을 위한 모든 프로토콜은 이미지 분석 소프트웨어의 Author 모듈을 사용하여 개발되었으며 텍스트에서 APP라고 합니다.
조직 정렬
11개의 마커와 H&E 및 PSR 얼룩에 이르는 3개의 직렬 섹션의 6개의 가상 슬라이드가 이미지 분석 소프트웨어의 티슈정렬 모듈에 로드되었습니다. 다음으로, 이미지는 자동화된 방식으로 연결, 정렬 및 공동 등록되어 개별 이미지의 모든 레이어를 포함하는 11플렉스 플러스 H&E 및 PSR 가상 합성 이미지를 생성했습니다(도2A-C). 정렬은 인접한 직렬 섹션에서 유래한 이미지의 경우 정확했으며, 정렬 시 상동 방식으로 배치되고 배치된 해당 조직 구조를 보여줍니다(도 2C 및 도 S1A). 더욱이, 정렬은 동일한 섹션에서 유래한 이미지에 대한 개별 셀 수준에서 정확하였다(도S1B). 자동 정렬 시간은 정렬할 이미지의 수, 크기, 복잡성 및 유사성에 따라 달라집니다. 위에서 언급 한 6 개의 가상 슬라이드의 정렬은 VIS 스테이션에서 15 분이 걸렸습니다.

그림 2: 직렬 조직 섹션의 염색 및 이미지 정렬. (A)COI 및 TC의 시각화를 위해 세 개의 일련 섹션에서 수행된 얼룩의 요약. 섹션 II 및 III의 경우, 조직을 벗겨내고 항체의 두 번째 칵테일로 재조사하였다. (B)조직 정렬 전후 6개의 개별 전체 조직 이미지 개요(각각 왼쪽 및 오른쪽). 배율 표시줄 = 3,500 μm.(C)정렬된 이미지의 확대보기. 배율 표시줄 = 80 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
조직 검출
이미지가 연결되고 정렬되면 TAP(그림3A)를식별하려고 했습니다. TAP의 자동 검출을 위한 APP를 설계하기 위해(APP 1, 표 1)우리는 조직과 연관되지 않은 픽셀과 TAP를 구별하는 두 가지 특성을 활용했습니다. 첫째, DAPI 신호(blue band)는 조직에만 위치하는 핵으로 제한되며, 이는 모든 DAPI+ 픽셀이 TAP의 하위 집합임을 의미합니다. 둘째, TAP는 조직과 연관되지 않은 픽셀에 비해 녹색 및 노란색 대역에서 더 높은 자가형광 신호를 갖는다. 따라서 간단한 임계값 기술을 사용하여 이러한 채널의 기준선 신호에 따라 TAP를 검출하는 조직 검출용 APP1(표 1)을개발했습니다. 파란색, 녹색 및 노란색 밴드의 임계값을 설정하여 TAP가 임계값 이상의 배경 강도 값을 가지는 반면 조직과 연결되지 않은 픽셀은 아래 값을 가집니다. 조직 검출을 위한 APP 1은 청색, 녹색 및 노란색 채널의 층을 포함하는 이미지 IIA에 적용되었다(그림3A). APP 1의 출력으로, 밝은 녹색 마스크는 TAP의 상단에 배치하고, "조직"이라는 ROI가 묘사되었다 (출력, 그림 3A). 더욱이, 조직의 영역은 정량적 출력 변수로서 결정되었다. APP 1은 조직과 연관되지 않은 픽셀을 ROI 조직에 통합하지 않기 때문에, 이 ROI에 기초한 후속 분석에서 제외되었다(그림3A). MP식별시 APP 1의 정밀도는 그림 3A에나와 있습니다.
TC를 위한 ROI의 조직 세분화 및 묘사
다음으로, 우리는 조직을 기질 대 자충으로 분할하여 ROI 조직 내부의 다른 구획을 정의하기 위해 진행했습니다. 우리는 PSR 염색 된 이미지 (IIIC, 그림 2C)를사용 하 고, 여기서 기질 은 섬유 콜라겐의 증착과 관련 된 영역으로 정의될 수 있다 (빨간 밴드), 피브릴라 콜라겐이 없는 영역으로 실어, 그리고 빠른 녹색 카운터 염색 염료 우세 (녹색 밴드).(그림3B). 우리는 디지털 TC 스트로마와 Parenchyma을 구분하기 위해 APP 2(표 1)를만들었습니다. 이 APP는 미리 정의된 ROI 조직(출력, 그림 3A)에서작동하며 이미지 분석 모듈에 통합된 분류기 도구를 교육하기 위해 대표적인 기질 및 실치마 영역을 사용합니다. 학습된 분류자는 픽셀을 기질 또는 실치마 레이블(연어와 녹색각각 그림 3 Figure 3B)에할당합니다. 픽셀을 분류하면 APP 2는 ROIs Stroma 및 Parenchyma를 정의하는 것을 목표로 하는 형태학적 작업을 실행했습니다(그림3B 및 표 1). 픽셀을 분류하고 각 ROI를 생성하는 APP 2의 성능은 그림 3B에나와 있습니다. 또한 APP 2는 기질과 실치종의 면적을 정량화합니다. 마지막으로, 세분화가 PSR 염색 섹션을 사용하여 수행되더라도 윤곽이 있는 기질 및 실치마 영역은 PSR 이미지에 정렬된 모든 이미지로 전송될 수 있습니다.

그림 3: 조직 검출/세분화 및 각 ROI 생성. (A)이미지 IIA는 TAP를 식별하기 위해 사용되었다(왼쪽 이미지, 스케일 바 = 6,000 μm). 밝은 녹색 마스크는 조직 (출력 1)이라는 ROI를 생성하는 APP 1(표 1)을사용하여 TAP에 할당되었습니다. 오른쪽, inset 은 탭을 감지할 때 APP 1의 정밀도를 보여주는 확대 된 보기를 보여줍니다. 스케일 바 = 350 μm.(B)ROI 조직(출력 1)은 APP 2를 사용하여 기질 및 실치종으로 분할된다. 왼쪽 이미지는 ROI 기질(연어)과 ROI 자렌치마(녹색)로 분할된 ROI 조직의 뷰를 보여줍니다. 배율 막대 = 4,500 μm. 오른쪽에는 ROI 티슈, 원래 PSR 염색(이미지 IIIC) 및 ROI 기질 및 실치마에 대한 인세트의 확대 뷰가 표시됩니다. 배율 막대 = 250 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
COI 자동 정량화
다음으로 ROIs Stroma 및 Parenchyma에서 COI를 식별, 위치 및 정량화했습니다. ApP 3 ~ 8(표 1)은CD4+FoxP3+, CD8+, CD68+, MPO+, αSMA+및 CD34+ 셀을 각각 찾아서 계산하기 위해 만들어졌습니다. APP 3은 CD4+FoxP3+ 셀(이미지 IIIA, 도 2C)을조절 T 셀(Tregs)의 대리 마커로서 찾아서 계산하도록 설계되었습니다. 이 프로토콜은 핵 전사 인자 FoxP3(적색 대역) 및 DNA 표지 염료 DAPI(청색 대역)로부터의 신호의 동국화를 검출한다. 최근 활성화 된 T 세포가 FoxP3를 upregulate, Tregs에 대한 농축을 감안할 때 우리는 밝은 FoxP3 + 세포 (FoxP3hi)만사전 선택하기위한 임계 값을 설정합니다. 다음으로, 미리 선택된 모든 DAPI+FoxP3hi 셀 중에서 밝은 고리 모양의 CD4 신호(녹색 대역)로 둘러싸인 셀만 FoxP3hiCD4+ 셀(분홍색 라벨, 그림 4A)으로분류하였다. ROIs Stroma 및 Parenchyma에서 FoxP3hiCD4+ 셀의 밀도는 APP 3의 정량적 출력 변수로 결정되었다(도4A).
유사하게, APP 4~ 6은 CD8+, CD68+및 MPO+ 셀의 검출을 위해 설계되었다. 이러한 APP는 COI를 감지하고 정량화하기 위한 동일한 기준 설계를 공유합니다. 구체적으로, COI는 특정 세포 집단 바이오마커로부터의 신호 강도에 기초하여 식별되고, 이어서 몇몇 후처리 형태학적 단계는 개별 세포를 묘사하기 위해 실행된다(표1). 개별 세포 또는 COI는 등록된 표지, 계수 및 그들의 조직 좌표입니다. APP 4 를 4~ 6은 또한 ROIs Stroma 및 Parenchyma에서 COI의 밀도를 결정한다(그림4B-D).
우리의 DAPI 염색의 질은 Apps 3에 6에 핵 분할을 통합하기에 충분하지 않았습니다, 그래서 우리는 개별적으로 표지된 객체가 개별적인 세포이다는 것을 보장할 수 없습니다. 이러한 이유로, 우리는 표지된객체/mm2의 수에 있는 세포의 밀도를 표현했습니다(그림 4). 그러나, 세포 응집체는 APP 3 에 6에 내장된 후처리 단계에서 개별 세포로 성공적으로 분리되었고, 광범위한 육안 검사는 대부분의 표지된 객체가 단일 세포에 대응하는 것으로 나타났다.
αSMA+ 및 CD34+ 영역을 검출하기 위해 각각 ApP 7과 8을 개발했습니다(표1). 두 APP 모두 임계값에 따라 특정 신호를 감지하고 ROIs Stroma 및 Parenchyma에서 양면적의백분율을 결정합니다(그림 4E-F).
가상 멀티플렉스 슬라이드를 생성하는 가장 흥미로운 가능성 중 하나는 공동 지역화 식의 분석입니다. 우리는 αSMA와 desmin, 간에서 myofibroblasts에 의해 공동 발현된 2개의 마커 사이 동분화를 검출하기 위하여 APP 10를 생성했습니다. APP 10은 αSMA, desmin 및 αSMA 플러스 데스민에 대해 양수 픽셀을 찾기 위한 임계값을사용합니다(표 1). 정량 출력 변수로서, APP 10은 αSMA+ 영역, desmin+ 영역 및 이들 두 마커의 동국화 발현 영역을 결정한다(그림S3).

그림 4: TCs 기질 및 실치종에서 COI의 식별 및 정량화. (A–F) 프로토콜 3, 4, 5, 6, 7 및 8을 사용하여 ROIs Stroma 및 Parenchyma에서 CD4+FoxP3+, CD8+, CD68+, MPO+, αSMA+및 CD34+ COI의 자동 감지 및 정량화(표1). 왼쪽에는 원본 이미지, 중간에 처리된 이미지, 오른쪽에는 정량화가 표시됩니다. 그림 4A-D의경우 배율 막대 = 40 μm입니다. 그림 4E 및 F의경우 배율 표시줄 = 350 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
TCs Stroma 및 Parenchyma에서 COIs를 정량화하는 대안으로서, 우리는 1-4(그림5A, H,및 I)라는다른 악성 결절에서 면역 세포의 밀도를 결정하였다. 각 작은혹에 대한 ROI는 그림 5A에표시된 대로 수동으로 설명되었습니다. 특유의 조직 면역 서명은 각 결절을 특징으로 하며, TME의 본질적인 이질성을 더욱 드러냅니다.
티슈 히트맵
위에서 언급한 바와 같이, APP 3-8은 개별적으로 표지된 모든 개체의 조직 좌표를 저장한다. 이 기능을 사용하면 지정된 세포 집단의 고밀도 영역이 핫 스팟(빨간색)으로 표시되고 밀도가 상대적으로 낮은 영역이 콜드 스팟(진한 파란색)으로 표시되는 조직 맵을 자동으로 생성할 수 있습니다. 중간 밀도 값은 그림 5에표시된 색상 축척에 따라 색상이 할당됩니다. 조직 히트맵은 이미지를 50 μm 직경의 원으로 나누고 원 내부의 주어진 COI의 상대밀도에 따라 색상을 할당하는 APP에 의해 생성되었습니다. 도 5B-G에도시된 바와 같이, TME 내의 상이한 COI의 위치 패턴 및 강도 분포는 매우 다양했다. 더욱이, 개별 적인 작은혹의 수준에서, 조직 지역에 있는 다른 인구의 배열은 유일했습니다(그림 S2A-C). 이 기술의 힘의 예를 제공하고 동일한 결절에 있는 다른 인구에서 다른 인구에서 핫스팟의 공간 조직을 구상하기 위하여, 개별 적인 세포 모형에서 핫스팟은 결절 2의 윤곽선에 수동으로 추출되고 매핑되었습니다(그림 S2, 그림 D및 그림 E).

그림 5: TME에서 COIs의 조직 히트맵. (A)작은혹 1, 2, 3 및 4의 위치를 보여주는 피크로시리우스 적색 염색. (B–G) CD4+FoxP3+, CD8+, CD68+, MPO+, CD34+및 αSMA+ COI용 티슈 히트맵. 진한 파란색은 상대적인 낮은 밀도를 나타내고 빨간색은 상대적인 고밀도를 나타냅니다. 중간 밀도 값은 표시된 색상 축척에 따라 색상이 할당됩니다. (H와 I)결절 1, 2 및 3 + 4에서 COI의 정량화는 세포 유형 당 및 결절 당 각각 조직됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

보충 그림 S1: 조직 정렬의 유효성 검사. (A)CD34 염색(빨간색)은 섹션 II(입력 1)에서 수행되며 CD34 마스크를 녹색으로 생성하는 데 사용됩니다(출력 1). 녹색 마스크(출력 1)는 정렬된 직렬 섹션 I(입력 2)으로부터 H&E 이미지상에 중첩된다. 병합 이미지는 혈관 구조의 완벽한 대응을 보여줍니다. 배율 막대 = 50 μm. (B)DAPI, CD4 및 FoxP3(입력 1)의 병합을 나타내는 이미지 IIIA는 CD4+FoxP3+ 셀에 대한 라벨을 생성하는데 사용되었다(마젠타에서 출력 1). 출력 1 라벨은 정렬된 이미지 IIIB(입력 2)로 전송되었으며 병합 이미지에서 FoxP3/DAPI 및 CD4/CD3 쌍 간의 완벽한 대응을 보여줍니다. 배율 막대 = 15 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

보조 그림 S2: 티슈 히트맵의 확대보기. (A–C) CD4+FoxP3+, CD8+, CD68+, 그리고 MPO+ 세포를 위한 조직 히트맵 1-4. 축하 1, 2 및 3 + 4의 스케일 바는 각각 1,500 μm, 700 μm 및 500 μm을 나타냅니다. (D)검정색 실선이 있는 결절 2의 윤곽선. (E)CD4+FoxP3+, CD8+, CD68+및 결절 2의 MPO+ 셀에 대한 핫스팟을 추출하여 D에정의된 결절 2 윤곽선에 함께 매핑했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

보조 그림 S3: 공동 지역화 분석. (A)왼쪽과 가운데에는 αSMA 라벨이 녹색이고 데스민 라벨이 빨간색으로 표시됩니다. 오른쪽은 노란색αSMA/desmin 이중 양성 영역입니다. (B)αSMA+ 영역, 데스민 + 영역 및 αSMA/desmin 이중 양성 영역의 정량화. 배율 막대 = 150 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 앱 | 목적 | 분류 | 분류 | 사후 처리 단계 | 출력 변수 |
| 메서드 | 기능 | ||||
| (픽셀 값) | |||||
| 1 | 조직 검출 | 임계값 | 채널 DAPI (150) | o 3개 채널에 대해 동지역화된 임계값이 있는 오브젝트 레이블 지정 | o ROI 조직 |
| 채널 FITC/A488 (120) | o 양수 오브젝트 5픽셀 닫기 | o 조직 영역 | |||
| 채널 TRITC/A568 (40) | o ROI 조직 만들기 | ||||
| 2 | 조직 세분화 | 결정 포리스트 | RGB-R 중앙값 | o 구멍 채우기 | o ROI 스트로마 |
| RGB-G 중앙값 | o ROI 스트로마 만들기 | o 스트로마 지역 | |||
| RGB-B 중앙값 | o ROI Parenchyma 만들기 | o ROI Parenchyma | |||
| IHS-S 중앙값 | o 자렌치마 지역 | ||||
| H&E 에오신 중앙값 | |||||
| 3 | CD4+ FoxP3+ 셀을 찾고 정량화하려면 | 임계값 | 채널 DAPI (>600) | o FITC/A488 신호로 둘러싸인 DAPI 및 Cy5/A647의 공동 지역화로 객체 레이블 지정 | o ROIs 스트로마 및 Parenchyma에 있는 CD4+FoxP3+ 세포의 개수 그리고 조밀도 |
| 채널 FITC/A488 폴리 스무딩 (>850) | o 7 μm2보다 작은 물체 지우기 | o 개별 CD4+FoxP3+ 셀의 좌표 | |||
| 채널 Cy5/A647(>800) | |||||
| 4 | CD8+ 셀을 찾고 정량화하려면 | 임계값 | 채널 DAPI (&1200) | o 15 μm2보다 작은 양물체 지우기 | o ROIs 스트로마 및 자영에서 CD8+ 셀의 개수 및 밀도 |
| 채널 Cy5/A647 중앙값(>80) | o 양수 오브젝트 닫기 2픽셀 | o 개별 셀의 좌표 | |||
| o 개체 분리 | |||||
| 5 | CD68+ 셀을 찾고 정량화하려면 | 임계값 | 채널 FITC/A488 (>200) | o 20 μm2보다 작은 양물체 지우기 | o ROIs 스트로마 및 Parenchyma에 있는 CD68+ 세포의 개수 그리고 조밀도 |
| o 양수 객체 3픽셀 넓어지다 | o 개별 CD68+ 셀의 좌표 | ||||
| o 개체 분리 | |||||
| 6 | MPO+ 셀을 찾고 정량화하려면 | 임계값 | 채널 DAPI (>400) | o 5 μm2보다 작은 물체 지우기 | o ROIs Stroma 및 Parenchyma에서 MPO+ 셀의 개수 및 밀도. |
| 채널 TRITC/A568 (900-4000) | o 3픽셀 양수 오브젝트 팽창 | o 개별 MPO+ 셀의 좌표. | |||
| o 개체 분리 | |||||
| 7 | αSMA+ 영역을 찾아 정량화하려면 | 임계값 | 채널 TRITC/CF568 (>1050) | o 25 μm2보다 작은 양물체 지우기 | o ROIs 스트로마 및 자렌치마지역의 αSMA+ 영역의 개수 및 밀도 |
| o 3픽셀 양수 오브젝트 팽창 | o αSMA+ 픽셀의 좌표 | ||||
| 8 | CD34+ 영역을 찾고 정량화하려면 | 임계값 | 채널 DAPI (&5000) | o 25 μm2보다 작은 양물체 지우기 | o ROIs 스트로마 및 Parenchyma에서 CD34+ 영역의 개수 및 밀도 |
| 채널 Cy5/A647 중앙값(>120) | o 3픽셀 양수 오브젝트 팽창 | o CD34+ 픽셀좌표 | |||
| 9 | 주어진 세포 집단에 대한 티슈 히트맵 만들기 | 오브젝트 히트맵 | 오브젝트 히트맵 | o 히트맵 | |
| 드로잉 반경 50 μm | --- | ||||
| 10 | αSMA와 데스민 간의 공동 지역화 정량화 | 임계값 | 채널 TRITC (CF568) (>1050) | o TRITC(CF568)의 임계값을 초과하는 객체에 레이블 지정 | o αSMA 및 Desmin의 공동 국부화 식 정량화 |
| 채널 Cy5 (A647) (>1000) | o Cy5(A647)에 대한 임계값을 초과하는 객체에 레이블 지정 | ||||
| o TRITC(CF568) 및 Cy5(A647)에 대한 임계값 이상의 동지역화가 있는 객체 레이블 지정 | |||||
| o 25 μm2보다 작은 양물체 지우기 |
표 1: 이미지 분석에 사용되는 APP 설계에 사용되는 일반 매개변수입니다. 이 표에 지정된 매개변수는 이 분석에 사용된 이미지의 고유한 특성(예: 배경, 아티팩트 등)으로 조정되며 다른 이미지에는 적용되지 않을 수 있습니다. 언급한 후처리 단계는 이 연구에서 분석된 특정 이미지에 대해 정의되었기 때문에 의도적으로 상세하지 않습니다. 사용자는 분석할 이미지에 맞게 APP를 사용자 지정해야 합니다.
| 섹션/염색 | 1 차 항체 | 이차 항체 |
| 섹션 II/1st 스테인링 | 마우스 IgG2a 안티 인간 αSMA 마우스 IgG1 안티 인간 CD34 토끼 안티 인간 사이토 케라틴 8/18 |
염소 안티 마우스 IgG2a CF568 쥐 안티 마우스 IgG1 A647 당나귀 안티 토끼 A488 |
| 섹션 II/2nd 스테인링 | 토끼 안티 휴먼 데스민 마우스 안티 인간 CD68 |
당나귀 안티 토끼 A647 당나귀 안티 마우스 DyLight 755 |
| 섹션 III/1st 스테인링 | 마우스 안티 인간 CD4 토끼 안티 인간 폭스P3 염소 안티 인간 MPO |
당나귀 안티 마우스 A488 당나귀 안티 토끼 A647 당나귀 안티 염소 A568 |
| 섹션 III/2nd 스테인링 | 토끼 안티 인간 CD3 마우스 안티 인간 CD8 |
당나귀 안티 마우스 DyLight 755 당나귀 안티 토끼 A647 |
표 2: mIF를 위한 1 차 이차 항체 쌍.
Discussion
조직 단면도에 있는 면역 세포의 공간 해결책을 허용하는 간단하고, 접근가능하고, 실행하기 쉬운 기술은 암과 그밖 면역학 무질서에 있는 면역 풍경을 지도로 하기 위하여 필요합니다. 여기서, 우리는,이미징분석12,,13,17,,19의다중화 능력 및 다차원 평가를 확장하기 위해 널리 이용 가능한 라벨링 및 디지털 분석 기법을 통합하는 전략을 설명한다. 서로 다른 마커에 대한 세 개의 직렬 섹션의 염색과 스트리핑 및 재생 기술을 통해 섹션을 재사용하면 H&E 및 PSR 얼룩 외에도 11개의 매개 변수를 시각화할 수 있었습니다. 이 섹션에서 6 개의 이미지는 조직 정렬 모듈을 사용하여 자동화 된 방식으로 정렬되었습니다. 정렬은 동일한 섹션에서 시작된 이미지에 대한 개별 셀 수준에서 정확했으며 인접 섹션에서 발생하는 이미지에 대해 매우 일치합니다. 가상 멀티플렉싱을 통해 한 섹션에서 시각화된 마커가 다른 연속 섹션에서 시각화된 마커와 공간적으로 어떻게 관련되는지 확인할 수 있었습니다. 일부 얼룩은 COI라고 표시되어 있지만 다른 얼룩은 TC에 레이블을 지정하여 다른 TC에서 COI를 정량화할 수 있었습니다. COI의 자동 정량화를 위한 소프트웨어 도구의 사용은 이미지 처리를 크게 단순화하고 가속화시켰습니다. 더욱이, 디지털 분석은 TME의 편견 표현의 결과로, 선택된 시야 대신 전체 조직 단면도에 적용되었습니다. 더욱이, COI의 조직 좌표가 등록되었기 때문에, 조직 히트맵을 생성할 수 있었다.
이 프로토콜에는 문제 해결이 필요할 수 있는 여러 영역이 있습니다. 첫째, 가난한 항원 검색은 mIF의 질에 영향을 미칠 수 있으므로 항원 검색 버퍼 및 지속 시간의 유형은 사용되는 특정 분석/바이오마커 조건에 맞게 최적화되어야 합니다. 둘째, 사용되는 차단 용액의 유형은 1 차 및 이차 항체의 조직 / 항원 / 종에 적응해야합니다. 우리의 손에, 조직이 차단 된 Fc 수용체로부터 오는 종으로부터 10 % 총 혈청을 첨가하여 비특이적 항체 결합을 감소시켰다. 이차 항체가 제기된 종으로부터 혈청의 10%를 첨가하면 조직 섹션에 대한 이차 항체의 직접적인 비특이적 부착을 최소화할 것이다. 셋째, 적절한 양성 및 음성 대조군을 이용한 1차 및 2차 항체의 특이성의 검증이 필수적이다. 넷째, 일부 채널에서 자가형광증가 및 1차 항체 스트리핑 시 DAPI의 확산도 일반적이다. 향상된 자기 형광을 해결하기 위하여는, 우리는 특정 신호가 배경의 적어도 5 배 의 강렬 값이 있던 1 차/이 차 항체 쌍을 이용했습니다. 마지막으로, 몇몇 높은 친화도 항체는 정규 스트리핑 절차로 용출될 수 없습니다. 이 경우, 우리는 라벨의 마지막 라운드에서 이러한 항체를 사용하는 것이 좋습니다. 사용자는 관심 있는 항체에 대한 최적의 구성을 찾기 위해 상이한 염색 서열을 시도해야 할 수도 있다. 라벨링의 두 번째 또는 세 번째 라운드로 진행하기 전에 스트립의 효율을 확인해야합니다.
이 전략의 주요 한계 그리고 도전은 관심의 마커를 위한 1 차및 이차 형광 항체의 적당한 조합을 찾아내는 것입니다. 동시에 사용될 수 있는 다른 종 또는 다른 등방형으로 올려진 1 차적인 항체를 찾아내는 것은 상업적으로 유효한 무슨에 의해 제한됩니다. 대부분의 전체 슬라이드 스캐너에는 최대 5개의 채널을 이미징할 수 있는 램프와 필터가 장착되어 있으며, 올바른 종과 우측 형광단의 이차 항체가 항상 사용할 수 있는 것은 아닙니다. 우리는 직렬 염색 및 순차적 라벨링을 사용하여 이러한 한계를 부분적으로 극복했습니다. 몇몇 항체 조합은 관심있는 마커를 위한 최적 조합에 도착하기 위하여 시험될 필요가 있을 수 있습니다. 또 다른 제한은 DAPI 염색의 품질입니다, 스트리핑 및 reprobing항상 핵 세분화를 수행 할 수 없습니다 때문에.
조직 정렬 모듈은 최소한의 교육과 사용자의 프로그래밍 기술이 필요하지 않습니다. 이 소프트웨어는 이론적으로 이미지의 무제한 정렬을 할 수 있습니다. 그러나 정확한 정렬은 더 조직학적으로 일치하는 더 가까운 단면이 더 정확하게 정렬되는 섹션의 관련성에 따라 달라집니다. 우리는 APP를 생성하기 위해 VIS의 작성모듈을 사용했습니다. APP를 만들 때 이미지 분석에 대한 기본 지식이 필요하지만 다른 이미지 분석 소프트웨어를 사용할 때도 마찬가지입니다. 다른 이미지 분석 소프트웨어와 비교하여 VIS의 독특한 장점은 서로 다른 방법(예: IF, 히스토화학, IHC)을 사용하여 제조된 섹션의 이미지 자동 정렬을 포함합니다. 이를 통해 가상 멀티플렉싱을 사용하여 여러 관심 마커에 대한 동지역화 연구가 가능합니다. 또한 APP의 유연하고 사용자 친화적인 디자인은 사용자별 사용자 지정을 가능하게 합니다. 자동화된 정량화 및 매핑, 전체 조직 섹션 처리 가능성, 시간 절약 및 육안 검사에 의한 수동 계산에 비해 바이어스 감소.
이 전략은 암과 자기 면역의 맥락에서 조직 면역학을 위한 아주 유용한 연구 공구입니다 그러나 임상 사용을 위해 유효하지 않은 남아 있습니다. 추가적인 표준화 및 검증을 통해, 향후 여러 응용 분야에 사용될 수 있다(예를 들어, 암에서면역경관을 매핑하여 면역치료제에 대한 반응을 예측하고 모니터링하기 위해). 또한 병리학적 평가와 예후 바이오마커를 결합하기 위해 상이한 염증성 조건(예를 들어, 염증성 장 질환)에 적응될 수 있다.
이 프로토콜의 주요 중요한 단계는 라벨링의 효율성/특이성 및 의도된 용도 또는 바이오마커를 위해 설계된 APP의 견고성입니다. 따라서 특히 새 APP를 설계할 때 육안으로 정기적인 검증이 필수적입니다. 같은 섹션에 스트립 및 reprobing 또는 얼룩의 다른 유형의 여러 라운드의 효율적인 사용은 중요 한 구성 요소 이며 조직 또는 섹션 특정 될 수 있습니다. 대규모 배치 분석을 진행하기 전에 이러한 프로세스의 효율성을 확인하는 것이 중요합니다.
요약하면, 우리는 귀중한 임상 조직 샘플에서 얻을 수있는 정량적 및 공간 적 정보를 극대화하는 전략을 제공합니다. 이 방법론을 구현하는 데 필요한 리소스, 장비 및 지식은 널리 액세스할 수 있습니다. 우리는 TME에 있는 면역 세포 인구를 확인, 정량화 및 매핑하는 것을 겨냥한 계획 적인 계획 보기를 위한 유용한 가이드로 이 방법론을 제안합니다.
Disclosures
저자는 이해 상충을 선언하지 않습니다.
Acknowledgments
우리는 연구 참가자에게 감사드립니다. 우리는 조직 견본 및 모든 관련임상 정보의 복구를 위한 HBP biobank의 조정자 루이스 Rousseau, 감사합니다. 우리는 우수한 기술 지원을 위해 Visiopharm에서 CRCHUM및 마이클 퍼쉬에서 분자 병리학 및 세포 이미징 핵심 시설을 인정합니다. 자금 조달: 이 연구 결과는 캐나다 간 재단, Fonds de recherche du Québec-Santé (FRQS) 에이즈 및 감염증 네트워크 (Réseau SIDA-MI) 및 C형 간염에 캐나다 네트워크 (CanHepC)에서 보조금에 의해 지원되었습니다. CanHepC는 캐나다 보건 연구 기관 (CIHR) (NHC-142832)와 캐나다의 공중 보건국에서 공동 이니셔티브에 의해 투자됩니다. M.F.M.은 몬트리올 대학교, 부르스 가브리엘 마르퀴스, 그리고 FRQS로부터 펠로우십을 받았습니다. T.F.는 CIHR과 CanHepC로부터 박사 펠로우십을 받았습니다. S.T.는 몬트리올 대학교의 간및 췌장 종양학 수술에서 Roger-Des-Groseillers 의자를 보유하고 있습니다.
작성자 기여: M.F.M.은 데이터를 설계, 수행 및 분석했습니다. T.F. 디자인 된 실험. A.C-B. 기술 적인 지침을 제공합니다. G.S.는 연구 대상자의 모든 병리학 적 평가를 수행하고 모든 병리학 적 측면에 대한 입력을 제공했습니다. L.M.은 H&E 염색, 최적화 및 이미지 수집을 수행했습니다. M.N.A.는 PSR 얼룩을 수행하고 귀중한 기술적 입력을 제공했습니다. N.B.는 이미지 분석에 기여했습니다. S.T.는 HBP 바이오뱅크의 주요 조사자이며 바이오뱅크의 전반적인 운영을 감독하고 있습니다. 그는 또한 프로젝트의 모든 측면과 임상적 의미에 대한 귀중한 의견을 제공했습니다. M.F.M, T.F., 및 N.H.S.는 연구를 개념화하고 설계했습니다. N.H.S.는 작업을 감독하고 자금을 확보했습니다. M.F.M., T.F., A.C-B, N.H.S.는 원고를 썼습니다. 모든 저자는 원고를 검토하고 승인했습니다.
Materials
| Name | Company | Catalog Number | Comments |
| Antigen Retrieval Solution: Sodium Citrate Buffer (10 mM Sodium Citrate, 0.05% v/v Tween 20, pH 6.0) | |||
| Blocking Solution: 1 % BSA, 10 % filtered human serum, 10 % filtered donkey serum, 0.1 % Tween 20, and 0.3% Triton in PBS | |||
| Bovine serum albumin (BSA) | Multicell | 800-095-EG | |
| Coplin jars (EASYDIP SLIDE STAINING SYSTEM) | Newcomersupply | 5300KIT | |
| Cover slides | Fisherbrand | 12-545E 22*50 | |
| Direct Red 80 | Sigma Aldrich | 365548 | |
| Donkey Serum | Sigma Aldrich | D9663 | |
| Ethanol 100% | |||
| Electric pressure cooker | Salton | ||
| Eosin | Leica Biosystems | 3801600 | CAUTION, eye irritation |
| Fast Green FCF | Sigma Aldrich | F7252 | CAUTION, harmful by inhalation, ingestion and skin absortion |
| FFPE section (4μm) slides | |||
| Glycine 0,1 M in PBS | |||
| Hematoxylin Stain Solution, Gil 1. Formulation, Regular Strength | Ricca Chemical Company | 3535-32 | |
| Holder (EasyDip Staining Jar Holder) | Newcomersupply | 5300RK | |
| Human Serum | Gemini | 22210 | |
| Humidity chamber | Millipore Sigma | Z670138-1EA | |
| Pap pen | abcam | ab2601 | |
| PBS | |||
| PBS-Tween 20 (0.1% v/v) | |||
| Permount Mounting Media | Fisher Chemical | SP15-500 | |
| Picric Acid 1.3 % | Sigma Aldrich | P6744 | CAUTION, skin and eye irritation |
| Picro-Sirius Red/Fast Green solution: Fast Green 0.1 % w/v + Sirius Red 0.2 % w/v in 1,3 % picric acid solution | |||
| Primary Antibody Anti-αSMA | Mouse IgG2a 1A4 | Sigma A2547 | Dilution 1/100 |
| Primary Antibody Anti-CD34 | Mouse IgG1 HPCA1/763 | Novus Biologicals NBP2-44568 | Dilution 1/250 |
| Primary Antibody Anti-Cytokeratin 8/18 | Rabbit EP17/EP30 | Agilent IR09461-2 | Ready to use |
| Primary Antibody Anti-CD68 | Mouse KP1 | Abcam ab955 | Dilution 1/200 |
| Primary Antibody Anti-Desmin | Rabbit Polyclonal | Invitrogen PA5-16705 | Dilution 1/200 |
| Primary Antibody Anti-CD4 | Mouse N1UG0 | Affymetrix 14-2444 | Dilution 1/250 |
| Primary Antibody Anti-FoxP3 | Rabbit 1054C | R & D MAB8214 | Dilution 1/100 |
| Primary Antibody Anti-MPO | Goat Polyclonal | R & D Systems AF3667 | Dilution 1/250 |
| Primary Antibody Anti-CD3 | Rabbit SP7 | Abcam ab16669 | Dilution 1/200 |
| Primary Antibody Anti-CD8 | Mouse C8/144B | Invitrogen 14-0085-80 | Dilution 1/200 |
| Secondary Antibody Donkey anti-mouse A488 | Polyclonal | Invitrogen A-21202 | Dilution 1/500 |
| Secondary Antibody Donkey anti-Rabbit A488 | Polyclonal | Invitrogen A-21206 | Dilution 1/500 |
| Secondary Antibody Donkey anti-goat A568 | Polyclonal | Invitrogen A-11057 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit A647 | Polyclonal | Invitrogen A-31573 | Dilution 1/500 |
| Secondary Antibody Rat anti-mouse IgG1 A647 | RMG1-1 | Biolegend 406618 | Dilution 1/500 |
| Secondary Antibody Goat anti-mouse IgG2a CF568 | Polyclonal | Sigma Aldrich SAB4600315 | Dilution 1/500 |
| Secondary Antibody Donkey anti-mouse DyLight 755 | Polyclonal | Invitrogen SA5-10171 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit DyLight 755 | Polyclonal | Invitrogen SA5-10043 | Dilution 1/500 |
| SDS | BioShop | SDS001,500 | CAUTION, oral skin and eye toxicity |
| Shandon multi-program robotic slide stainer | LabX | 11384903 | |
| Shandon Xylene Substitute, | Thermo Fisher Scientific | CA89413-336 | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Shaking water bath | |||
| SlowFade Gold antifade reagent with DAPI | Invitrogen | S36938 | |
| Sodium Citrate Dihydrate | Millipore Sigma | 1545801 | CAUTION, eye irritation |
| Stripping Buffer: mix 20 ml 10% w/v SDS with 12.5 ml 0.5 M Tris-HCl (pH 6.8), and 67.5 ml ultra-pure water. Under a fume hood, add 800 uL of 2-mercapto ethanol (114,4 mM final concentration) | |||
| Triton X-100 | Sigma Aldrich | T8787-50ML | |
| Tris-HCl | BioShop | 77-86-1 | |
| Tween 20 | Fisher Scientific | BP337-500 | |
| VIS Software | Visiopharm | ||
| Whole slide scanner Olympus BX61VS | Olympus | Microscope: Olympus Slide Scanner BX61VS, 5 slides scanner, motorized stage, autofocus. Camera: Lightsource: Xcite-120. Filters: BrightLine® Sedat filter set (# LED-DA/FI/TR/Cy5-4X4M-B-000, Semrock) | |
| Xylene | Sigma Aldrich | 214736-4L | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Xylene : Ethanol solution (1:1 v/v) | |||
| 2-mercaptoethanol | Sigma | M6250 | CAUTION, harmful by ingestion, inhalation, fatal if sking absortion. Eye irritation. Use fume hood |
References
- Greten, F. R., Grivennikov, S. I. Inflammation and Cancer:Triggers, Mechanisms, and Consequences. Immunity. 51 (1), 27-41 (2019).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Binnewies, M., et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nature Medicine. 24 (5), 541-550 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathology: an official journal of the United States and Canadian Academy of Pathology, Inc. 31 (2), 214-234 (2018).
- Bindea, G., et al. Spatiotemporal dynamics of intratumoral immune cells reveal the immune landscape in human cancer. Immunity. 39 (4), 782-795 (2013).
- Galon, J., et al. Towards the introduction of the 'Immunoscore' in the classification of malignant tumours. The Journal of Pathology. 232 (2), 199-209 (2014).
- Finotello, F., Eduati, F. Multi-Omics Profiling of the Tumor Microenvironment: Paving the Way to Precision Immuno-Oncology. Frontiers in Oncology. 8, 430 (2018).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: a method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Porta Siegel, T., et al. Mass Spectrometry Imaging and Integration with Other Imaging Modalities for Greater Molecular Understanding of Biological Tissues. Molecular Imaging and Biology : MIB: the official publication of the Academy of Molecular Imaging. 20 (6), 888-901 (2018).
- Buchberger, A. R., DeLaney, K., Johnson, J., Li, L. Mass Spectrometry Imaging: A Review of Emerging Advancements and Future Insights. Analytical Chemistry. 90 (1), 240-265 (2018).
- Pirici, D., et al. Antibody elution method for multiple immunohistochemistry on primary antibodies raised in the same species and of the same subtype. Journal of Histochemistry and Cytochemistry. 57 (6), 567-575 (2009).
- Gendusa, R., Scalia, C. R., Buscone, S., Cattoretti, G. Elution of High-affinity (>10-9 KD) Antibodies from Tissue Sections: Clues to the Molecular Mechanism and Use in Sequential Immunostaining. Journal of Histochemistry and Cytochemistry. 62 (7), 519-531 (2014).
- van der Loos, C. M. Multiple immunoenzyme staining: methods and visualizations for the observation with spectral imaging. Journal of Histochemistry and Cytochemistry. 56 (4), 313-328 (2008).
- Stack, E. C., Wang, C., Roman, K. A., Hoyt, C. C. Multiplexed immunohistochemistry, imaging, and quantitation: a review, with an assessment of Tyramide signal amplification, multispectral imaging and multiplex analysis. Methods. 70 (1), 46-58 (2014).
- Toth, Z. E., Mezey, E. Simultaneous visualization of multiple antigens with tyramide signal amplification using antibodies from the same species. Journal of Histochemistry and Cytochemistry. 55 (6), 545-554 (2007).
- Robertson, D., Savage, K., Reis-Filho, J. S., Isacke, C. M. Multiple immunofluorescence labeling of formalin-fixed paraffin-embedded (FFPE) tissue. BMC Cell Biology. 9, 13 (2008).
- Segnani, C., et al. Histochemical Detection of Collagen Fibers by Sirius Red/Fast Green Is More Sensitive than van Gieson or Sirius Red Alone in Normal and Inflamed Rat Colon. PloS One. 10 (12), 0144630 (2015).
- Bolognesi, M. M., et al. Multiplex Staining by Sequential Immunostaining and Antibody Removal on Routine Tissue Sections. Journal of Histochemistry and Cytochemistry. 65 (8), 431-444 (2017).



