Summary
Her beskriver vi en enkel og tilgængelig strategi for visualisering, kvantificering og kortlægning af immunceller i formalin-faste paraffin-indlejrede tumorvævsektioner. Denne metode kombinerer eksisterende billedbehandlings- og digitale analyseteknikker med det formål at udvide multipleksingkapaciteten og multiparameteranalysen af billeddiagnostiske analyser.
Abstract
Immunlandskabet i tumormikromiljøet (TME) er en afgørende faktor i kræftprogression og respons på terapi. Specifikt har tætheden og placeringen af immunceller i TME vigtige diagnostiske og prognostiske værdier. Multiomic profilering af TME har eksponentielt øget vores forståelse af de mange cellulære og molekylære netværk, der regulerer tumor indledning og progression. Disse teknikker indeholder dog ikke oplysninger om den rumlige organisation af celler eller cellecelleinteraktioner. Overkommelige, tilgængelige og nemme at udføre multiplexing teknikker, der tillader rumlig opløsning af immunceller i væv sektioner er nødvendige for at supplere encellede high-throughput teknologier. Her beskriver vi en strategi, der integrerer seriel billedbehandling, sekventiel mærkning og billedjustering for at generere virtuelle multiparameterdias af hele vævssektioner. Virtuelle slides analyseres efterfølgende på en automatiseret måde ved hjælp af brugerdefinerede protokoller, der muliggør identifikation, kvantificering og kortlægning af cellepopulationer af interesse. Billedet analyse er gjort, i dette tilfælde ved hjælp af analysemoduler Tissuealign, Forfatter, og HISTOmap. Vi præsenterer et eksempel, hvor vi anvendte denne strategi med succes til en klinisk prøve, maksimere de oplysninger, der kan opnås fra begrænsede vævsprøver og giver et upartisk billede af TME i hele vævssektionen.
Introduction
Kræft udvikling er resultatet af en multistep proces, der involverer gensidige interaktioner mellem maligne celler og TME. Andre end tumorceller, TME er sammensat af nonmaligne celler, stromale celler, immuncellepopulationer, og ekstracellulære matrix (ECM)1. Den rumlige organisation af de forskellige cellulære og strukturelle komponenter i tumorvævet og den dynamiske udveksling mellem kræft og tilstødende ikke-kræftceller i sidste ende modulere tumor progression og respons på terapi2,3,4. Det har vist sig, at immunresponset i kræft er spatiotemporally reguleret5,6. Forskellige immuncellepopulationer, der infiltrerer den neoplastiske læsion og det tilstødende væv, udviser karakteristiske rumlige fordelingsmønstre og forskellige aktiverings- og differentieringstilstande forbundet med forskellige funktioner (f.eks. pro- versus antitumor). Disse forskellige immunpopulationer og deres parametre skaber overarbejde med tumoren og de stromale rum.
Fremkomsten af teknologier, der tillader encellede multiomics profilering har eksponentielt øget vores forståelse af de mange cellulære og molekylære netværk, der regulerer carcinogenese og tumor progression. De fleste analyseværktøjer med høj gennemløb kræver imidlertid vævsforstyrrelser og isolation af en enkelt celle, hvilket resulterer i tab af oplysninger om den rumlige organisation af celler og cellecelleinteraktioner7. Da placeringen og placeringen af specifikke immunceller i TME har diagnostisk og prognostisk værdi, er teknologier, der muliggør rumlig opløsning, et vigtigt supplement til encellede immunprofileringsteknikker.
Traditionelt har billeddannelsesteknikker som immunhistokemi (IHC) og multiplex immunfluorescens (mIF) været begrænset til et lille antal biomarkører, der kan visualiseres samtidigt. Denne begrænsning har hæmmet studiet af spatiotemporal dynamik tumor-infiltrerende immunceller, som typisk er defineret af flere fænotypiske markører. De seneste fremskridt inden for billedbehandling og analytiske værktøjer har udvidet mulighederne for multipleksing. Nye antistofbaserede mærkningsteknologier som histocytometri og billeddannelse af massecytometri er blevet anvendt til rumligt at adskille op til henholdsvis 12 og 32 biomarkører, henholdsvis 8,9. Massespektrometri imaging, en teknik, der ikke kræver mærkning, har potentiale til at billedet tusindvis af biomarkører samtidigt i et enkelt væv afsnit10,11. Selv om disse teknikker allerede har vist et stort potentiale for at dissekere vævsimmunlandskabet i kræft, bruger de meget sofistikeret og dyrt udstyr og software og er ikke umiddelbart tilgængelige for de fleste forskere.
Alternativt er multipleksing evnen til traditionelle IHC og mIF blevet udvidet ved hjælp af seriel billeddannelse, sekventielle runder af mærkning, og spektral billeddannelse7,12,13,14,15,16. Disse teknikker genererer flere billeder fra samme eller fra seriel væv sektioner, der kan konsolideres i virtuelle multiparameter dias ved hjælp af billedanalyse software. Derfor øges antallet af markører, der kan visualiseres og analyseres samtidigt.
Her foreslår vi en strategi for rationel tæring af vævsmultiplex-analyser ved hjælp af kommercielt tilgængelige reagenser, kopiudstyr til overkommelige beløb og brugervenlig software (figur 1). Denne metode integrerer seriel billeddannelse, sekventiel multiplex mærkning, hele væv simaging, og væv tilpasning til at generere virtuelle multiparameter dias, der kan bruges til automatiseret kvantificering og kortlægning af immunceller i væv sektioner. Ved hjælp af denne strategi, skabte vi en virtuel dias bestående af 11 biomarkører plus to hyppigt anvendte histologiske pletter: hematoxylin og eosin (H & E) og picrosirius rød (PSR). Flere immuncellepopulationer blev identificeret, placeret og kvantificeret i forskellige vævsrum, og deres rumlige fordeling blev løst ved hjælp af vævsheatmaps. Denne strategi maksimerer de oplysninger, der kan opnås fra begrænsede kliniske prøver og gælder for formalin-faste paraffin-indlejrede (FFPE) arkiverede vævsprøver, herunder hele væv, kernenålbiopsier, og vævsmikroarrays. Vi foreslår denne metode som en nyttig vejledning til at designe brugerdefinerede analyser til identifikation, kvantificering og kortlægning af immuncellepopulationer i TME.
Protocol
Tre serielle FFPE sektioner fra resected hepatitis B-virus (HBV)-associeret humant hepatocellulært karcinom blev fremstillet fra Centre hospitalier de l'Université de Montréal (CHUM) Hepatopancreatobiliary Cancer Clinical Database and Biological Specimen Repository (HBP Biobank). Patienter, der deltog i denne vævsbank, gav informeret samtykke. Denne undersøgelse blev godkendt af den institutionelle etiske komité (protokol nummer 09.237) og udført i overensstemmelse med Helsingfors-erklæringen.
1. Hæmatoxylin og eosin (H & E) farvning protokol
BEMÆRK: H&E-farvningen blev udført af centren Centre de Recherches du Centre hospitalier de l'Université de Montréal (CRCHUM) ved hjælp af Shandon-multiprogramrobotdiaspletten ved hjælp af følgende program.
- For deparaffinization, nedsænke dias 3x i 2,5 min hver i xylen erstatning.
FORSIGTIG: Xylenerstatninger er brandfarlige, hudirriterende stoffer og skadelige ved indånding. - For rehydrering nedsænkes objektglas i 100% ethanol 3x i 2,5 min. hver. Vask i 1 minut i dobbelt destilleret vand (ddH2O) for at rehydrere.
- Inkuber i 1 min i hematoxylin. Der vaskes 3x i 1 min hver i ddH2O.
- Inkuber i 5 s med eosin. Vask 30 s med 95% ethanol. Der vaskes 2x i 1 minut med 100% ethanol.
FORSIGTIG: Ethanol er brandfarligt og et øjenirriterende stof. Eosin er et øjenirriterende. - For dehydrering, nedsænkes 3x i 1,5 min hver i xylen erstatning. Monter slides manuelt.
BEMÆRK: Den anslåede tid til at udføre denne del af protokollen er 30 min.
2. Multiplex immunofluorescens farvning protokol for FFPE sektioner
BEMÆRK: Denne protokol er tilpasset fra Robertson et al.17.
- Deparaffinisering og rehydrering
BEMÆRK: Før iHC eller mIF har foretaget antistofmedieret mærkning af FFPE-sektioner, skal paraffinen fjernes. Undladelse af effektivt at fjerne paraffin resulterer i suboptimal farvning.- Placer 4 μm FFPE vævssektionsglidere i glasglasholdere. Under røgemhætten nedsænkes rutsjebanerne i en Coplin-krukke med 37°C forvarmet xylen i 10 min.
FORSIGTIG: Xylen er brandfarlig, hudirriterende og skadelig ved indånding. - Ryst diasene manuelt i 10 s hver 2 min. Gentag 1x i frisk xylen i yderligere 5 min.
- I den kemiske hætte nedsænkes objektglassene sekventialt i 5 min. i hver af følgende opløsninger: 1) xylen : ethanol (1:1 v/v); 2) 100% ethanol; 3) 70% ethanol; 4) 50% ethanol; 5) 30% ethanol; 6) fosfat-buffered saltvand (PBS).
BEMÆRK: Hold objektglassene i PBS, indtil de er klar til at udføre antigenudtagningen. Hold de afvoksbehandlede sektioner hydreret på alle tidspunkter. Udtørring vil forårsage uspecifik antistof binding og derfor høj baggrund farvning.
- Placer 4 μm FFPE vævssektionsglidere i glasglasholdere. Under røgemhætten nedsænkes rutsjebanerne i en Coplin-krukke med 37°C forvarmet xylen i 10 min.
- Udtagning af varmeinduceret antigen
BEMÆRK: Antigener kan maskeres ved formalinfiksering, hvilket forhindrer antistofbinding og dermed visualisering. Brugen af antigen afmaskering buffere og procedurer delvist genetablere den indfødte kropsbygning af epitoper og dermed genskaber antistof anerkendelse. Typen af antigenudtagningsbuffer og varighed skal optimeres til de specifikke analysebetingelser (f.eks. mål, antistof, væv osv.).- Fordyb afvoksningsglas i en Coplin krukke indeholdende antigenhentningsopløsningen (opskrift i Materialetabel).
- Placer lukket Coplin krukke i en elektrisk trykkoger med vand fra hanen. Vandstanden må ikke overstige halvdelen af glassets højde, så vandet ikke blandes med antigenudtagningsopløsningen.
- Luk låget og kogerens trykventil. Vælg højt tryk i 10 min og start. Når du er færdig, tag stikket ud af komfuret, slip trykket, åbn låget, og hold glasset inde i komfuret i 30 min, så diasene afkøles.
- Blokering af uspecifik binding
- Overfør stativet med rutsjebanerne til en Coplin krukke fyldt med PBS. Antigenudtagningsbufferen skylles af med PBS 2x i 5 minutter hver.
- Omkransvævsektioner med en PAP pen til at skabe en hydrofob barriere. Der nedsaett glassene i en Coplin-krukke med 0,1 M glycin i PBS. Inkuber i 15 minutter ved stuetemperatur (RT).
BEMÆRK: Glycin mætter aldehydgrupperne, der dannes under antigenudtagningen. Disse grupper kan binde primære og sekundære antistoffer uspecifikt. - Glycinopløsningen skylles af ved at vaske 2x med PBS i 5 min. Placer objektglassene i et fugtighedskammer, og tilsæt tilstrækkelig blokerende opløsning til at dække alle vævssektionerne. Undgå at flyde over den hydrofobe barriere. Inkuber i 30 min på RT.
BEMÆRK: Opskriften på blokeringsopløsningen findes i materialetabellen. Blokeringsopløsningen skal indeholde et protein (f.eks. BSA) til blokering af uspecifikke bindingssteder. Det kan også indarbejde rengøringsmidler som Triton X-100 eller Tween 20, der reducerer hydrofobe interaktioner mellem antistoffer og væv mål, hvilket gør antigen anerkendelse mere selektiv. Tilsætning af 10% total serum fra de arter, hvor vævet kommer fra, ville blokere Fc-receptorer og dermed reducere uspecifik antistofbinding. Endelig ville tilsætning af 10% serum fra arten, som de sekundære antistoffer blev rejst i, minimere direkte uspecifik fastgørelse af sekundære antistoffer til vævssektionen.
- Mærkning af immunfluorescens
- Skyl med PBS-Tween (0,1% v/v) 2x i 5 min hver og læg gliderne tilbage i fugtighedskammeret.
- Tilsæt cocktail af primære antistoffer resuspended i blokerende opløsning. Inkuber natten over ved 4 °C. Primære og sekundære antistoffer, der anvendes til denne undersøgelse, er opført i Materialetabel.
BEMÆRK: Cocktailen af primære antistoffer skal indeholde antistoffer, der opdrættes i forskellige arter, eller fra samme art, men af forskellige isotyper. For en liste over de primære sekundære antistofpar, der anvendes i denne undersøgelse, henvises til tabel 2. Nærmere oplysninger om alle anvendte antistoffer findes i materialetabellen og tabel 2. - Skyl med PBS-Tween (0,1% v/v) 3x i 5 min og placer gliderne tilbage i fugtighedskammeret. I mørke, tilsæt cocktail af sekundære antistoffer og inkubere i 1 time på RT.
BEMÆRK: Når de primære antistoffer er fra forskellige arter, skal de sekundære antistoffer vælges, så hver af dem kun binder sig til et af de primære antistoffer og ikke til hinanden. Dette opnås almindeligvis ved at anvende sekundære antistoffer, der alle er opdrættet i samme art, så længe denne art adskiller sig fra de arter, hvor de primære antistoffer blev genereret. I tilfælde, hvor de primære antistoffer blev opdrættet hos samme art, men har forskellige isotyper, bør der anvendes isotypespecifikke sekundære antistoffer. - Skyl med PBS-Tween (0,1% v/v) 3x i 5 min. Skyl med ddH2O. Fjern overskydende væske, og monter i monteringsmedier med DAPI. Den anvendte diskenhed afhænger af sektionens størrelse. Normalt 40 μL er nok til at dække overfladen af en regelmæssig mikroskopi dias.
- Placer dækslet på sektionen og pres forsigtigt de overskydende monteringsmedier ud, så bobledannelse undgås. Lad objektglassene tørre i 20 min ved RT i mørke og opbevares ved 4 °C, indtil de er klar til erhvervelse.
- Hent billeder til alle kanaler ved hjælp af hele diasscanneren (se Materialetabel).
BEMÆRK: Antistofferne blev valideret ved hjælp af humant hepatocellulært karcinomvæv som en positiv kontrol. For hvert primært antistof blev der farves tre serielle sektioner med enten primærantistof, isotypekontrol eller kun blokerende opløsning uden variation i resten af farvningsprotokollen. De erhvervede billeder blev sammenlignet for at fastslå specificiteten af farvning. Farvningen blev anset for specifik, når signalet i afsnittet inkuberet med primært antistof havde det forventede mønster og let kunne skelnes fra baggrunden. Primære antistoffer, der giver et højt baggrundssignal eller mærkningsvævskomponenter i isotypen, og ingen primære antistofsektioner blev betragtet som uspecifikke. Den anslåede tid for at udfylde denne del af protokollen er 2 dage. De nødvendige kontroller omfatter: (1) Isotype-kontrol for at fastslå, at den ikke-specifikke binding af det primære antistof bidrager til baggrundssignalet. Den ene sektion farves på samme måde som det andet prøvevæv, bortset fra at det inkuberes med et antistof med samme isotype og oprindelse af det primære antistof, men specifikt for et mål, der ikke er til stede i vævssektionen. Hvis det relevante isotypekontrolantistof ikke er tilgængeligt, kan det erstattes af total IgG fra de samme arter, hvor det primære antistof er blevet fremrejst. (2) Ingen primær antistofkontrol (dvs. negativ kontrol) til at fastslå farvningens specificitet og til at anslå uspecifik binding af sekundære antistoffer ser ud til baggrundssignalet. I dette tilfælde farves kontrolsektionen på samme måde som de andre afsnit, bortset fra at der ikke tilsættes noget primært antistof. (3) Positiv kontrol for at fastslå, at farvningen virker. I dette tilfælde udføres farvningen på et vævsafsnit, der er kendt for at udtrykke den markør, der genkendes af det primære antistof.
3. Picro-sirius rød (PSR)/hurtig grøn farvning protokol
BEMÆRK: Målet med denne farvning er at visualisere fibrillar kollagener I og III i FFPE væv sektioner. Denne protokol er tilpasset fra Segnani et al.18. Alle trin udføres i en kemisk hætte.
- Deparaffinisering og rehydrering af vævssektioner svarende til multipleximmunofluorescensfarvningsprotokollen for FFPE-afsnittene (punkt 2.1).
BEMÆRK: Hvis den sektion, der skal farves, tidligere har været anvendt til immunfluorescensmærkning, og paraffinen allerede er fjernet, er deparaffiniserings-rehydreringstrinnenene nyttige til at fjerne monteringsmediet. DAPI fjernes ikke ved hjælp af denne procedure, men det forstyrrer ikke uden om psr-farvningen. - Fordyb objektglassene i en krukke, der indeholder picro-sirius rød/hurtig grøn opløsning (opskrift i Materialetabel)og inkuberi i 30 min på RT (mere end 30 min resulterer i uspecifik farvning af kernerne af hepatocytter).
- Vask glider hurtigt i ddH2O (5 dips). Derefter vaskes hurtigt i ethanol 100% (5 dips). Vaskes i 30 s i xylen-100% ethanol (1:1 v/v). Vask i 30 s i xylen. Monter med monteringsmedier (se Materialetabel),før xylen er helt fordampet (dette hjælper med monteringen).
BEMÆRK: Den anslåede tid for udførelse af denne del af protokollen er 1 time.
4. Eluering af antistoffer fra vævssektioner
BEMÆRK: For at genbruge vævssektioner i sekventielle mærkningsanalyser er det nødvendigt at fjerne primære og sekundære antistoffer fuldstændigt. Bundne antistoffer blev fjernet som tidligere beskrevet13.
Forvarm et vandbad til 56 °C. Sæt sektionerne inde i en krukke, der indeholder stripping buffer (opskrift i Tabel af materialer),lukke låget, og forsegle det med paraffin film tape for at forhindre utætte under omrystning.
- Sæt krukken inde i vandbad og inkuber i 30 min med agitation.
- Der vaskes 4x i 15 minutter hver i ddH2O ved RT. Skyl med PBS-Tween (0,1% v/v).
- Hold sektionerne hydreret i PBS-Tween eller vand, indtil de er klar til at omsonde sektionen med anden runde af primære antistoffer.
BEMÆRK: Den anslåede tid for udførelse af denne del af protokollen er 2 timer. - Kontroller effektiviteten af antistofelueringsproceduren.
BEMÆRK: Før protokollen for antistofeluering anvendes i en sekventiel mærkningsanalyse, skal effektiviteten af fjernelsen af primære og sekundære antistoffer verificeres.- Opnå farvning og billedanskaffelse af et afsnit med et givet primærsekundært antistofpar af interesse som angivet i multiplex-immunofluorescensfarvningsprotokollen for FFPE-sektioner (punkt 2.1-2.4.6).
- Ved billederhvervelse udføres eluering af vævsbundne primære sekundære antistofkomplekser som angivet i punkt 4.1-4.3.
- Inkubersektionen med samme sekundære antistof og samme betingelser, der anvendes i trin 2.4.3.
- Udfør vaske-, monterings- og billedanskaffelsestrin som angivet i 2.4.4-2.4.6.
- Sammenlign side om side billeder erhvervet før og efter stripning for at fastslå, om det specifikke signal er forsvundet.
BEMÆRK: Sammenligning af billeder før og efter fjernelse af antistoffer vil validere effektiviteten af elueringsproceduren. Det er dog normalt at se en stigning i baggrundssignalet i alle kanaler, samt udbredelse af DAPI. Dette begrænser antallet af runder af stripning, der kan udføres på samme væv sektion. Tre runder af stripning synes at være det maksimale.
5. Erhvervelse af billeder
- Generer billeder ved hjælp af en hel slidescanner.
- Brug en objektiv linse på 20x 0,75NA og en opløsning på 0,3225 μm/pixel.
6. Billedanalyse
BEMÆRK: Den metode, der er skitseret her, henviser til det aktuelle eksempel. Der henvises til tabel 1 og teksten for at tilpasse den til andre specifikke prøver.
- Udfør vævsjustering ved hjælp af modulet Tissualign i billedanalysesoftwaren (VIS i denne protokol, se Materialetabel).
- Åbn billedanalysesoftwaren, og klik på fanen Tissuealign.
- Importer de billeder, der skal justeres i slidebakken, ved at gå til Fil | databasen, og vælg det første billede, der skal justeres. Gå tilbage til fanen Tissuealign, og indlæs billedet ved at klikke på knappen Indlæs i slidebakken. Billedet vises i slidebakken og i arbejdsområdet.
BEMÆRK: Kun den stak af interesse bør lægges i diasbakken. - Gentag trin 6.1.2 for alle billederne i den rækkefølge, der skal justeres, og indlæs dem en efter en. Når alle de billeder af interesse er indlæst på slide bakken fortsætte med at forbinde billederne ved at trykke på Næste i Trin i arbejdsprocessen på båndet.
- Træk og slip derefter det andet billede oven på det første billede. Det første og det andet billede er nu sammenkædet. Gentag dette trin for de andre billeder, der skal justeres, én efter én, på en ordentlig måde. Navnet på det første billede ændres, hvilket indikerer, at det er blevet sammenkædet med de andre billeder. Samtidig vises de sammenkædede billeder i arbejdsområdet til højre for slidebakken.
- På dette tidspunkt skal du justere billederne enten ved hjælp af automatisk justering, halvautomatisk justering eller manuel justering. Det er altid at foretrække at prøve automatisk justering først. Tryk på knappen Næste i arbejdsprocestrinnene (trin 3) på båndet for at få automatisk justering.
- Gennemgå den automatiske justering ved at navigere i forskellige placeringer af vævet og visuelt kontrollere, at de tilsvarende strukturer i forskellige billeder er arrangeret på samme måde i de to dimensioner af billedet.
- Hvis resultatet af den automatiske justering ikke er tilfredsstillende, skal du forbedre den ved hjælp af stifter (brug mindst tre ben pr. billede), der angiver homologe vævsfunktioner i de sammenkædede billeder. Når stifterne er placeret på homologe steder i de sammenkædede billeder, har brugeren to muligheder: halvautomatisk justering eller manuel justering. For halvautomatisk justering klik på knappen Juster automatisk baseret på de aktuelle peger på båndet. Hvis du vil justere manuelt, skal du klikke på knappen Anvend pins på båndet.
- Når du er tilfreds med justeringen, skal du klikke på knappen Næste i arbejdsprocestrinnene og gemme det sammensatte billede i databasen.
BEMÆRK: Tilpasning seks dias spænder over 11 markører plus H & E og PSR billeder tog 15 min i den præsenterede analyse.
- Udfør vævsdetektering ved hjælp af den brugerdefinerede protokolanalysisprotokolpakke 1 (APP 1, tabel 1).
- Åbn billedanalysemodulet for softwaren ved at klikke på fanen Billedanalyse på båndet.
- Importer det sammensatte (justerede) billede ved at gå til Fil | Database og vælge billedet af interesse og klikke tilbage fanen Billedanalyse.
- Åbn dialogboksen APP-valg ved at klikke på ikonet Åbn APP, og vælg, hvilken APP (Analysis Protocol Package), der skal bruges. I dette tilfælde skal du vælge APP 1 til vævsdetektering.
- Når APP 1 er åbnet, skal du bekræfte, at APP1 fungerer korrekt ved at gå til en valgt vævsplacering og klikke på knappen Eksempel. Hvis resultaterne er tilfredsstillende, skal du gå videre til næste trin.
- Klik her for at køre APP 1 og behandle billedet ved hjælp af den valgte APP.
- Eksporter dataene (f.eks. billeder, målinger osv.), når analysen udføres, ved at klikke på Filer/eksportér.
BEMÆRK: APP 1 skaber en region af interesse (ROI) delineating vævet (ROI Væv) og beregner det område af vævet. - Gem det ændrede billede med det nyoprettede investeringsafkast ved at gå til Fil | Gem.
BEMÆRK: Detektering af vævet og oprettelse af et investeringsafkast med APP 1 i det medfølgende eksempel tog 5 min i den beskrevne billedanalysestation. Det behandlede vævsareal var 3,2 cm2.
- Segmentering af væv i Stroma og Parenkym ved hjælp af APP 2 (Tabel 1).
BEMÆRK: APP 2 virker på det foruddefinerede ROI-væv. APP 2 segmenterer vævet i ROIs Stroma og Parenchyma.- Åbn modulet Billedanalyse ved at klikke på fanen Billedanalyse på båndet.
- Importer det billede, der indeholder roi-vævet, ved at gå til Fil | database og vælge det billede, der blev gemt i trin 6.2.7. Gå tilbage til fanen Billedanalyse, og indlæs billedet ved at klikke på knappen Indlæs i slidebakken. Billedet vises i slidebakken og i arbejdsområdet.
- Åbn APP 2 ved hjælp af app-valgdialogboksen som i 6.2.3.
- Få vist APP 2 ved at behandle i et markeret visningsfelt. Hvis resultaterne er tilfredsstillende, skal du køre APP 2 på det fulde billede ved at klikke på knappen Kør. Da produktionen af APP 2, er ROI væv segmenteret i ROIs Stroma og Parenchyma og deres respektive områder bestemmes. Eksportresultater som i 6.2.6. Gem det ændrede billede som i 6.2.7.
BEMÆRK: Segmentering af vævet i Stroma og Parenchyma ved hjælp af APP 2 tog 4 timer i den præsenterede analysestation. Det behandlede vævsareal var 3,2 cm2.
- Identificer og kvantificer FoxP3hiCD4+ celler ved hjælp af den brugerdefinerede protokol APP 3 (Tabel 1).
BEMÆRK: APP 3 virker på de foruddefinerede ROIs Stroma og Parenchyma.- Åbn billedanalysemodulet, og importer det billede, der indeholder ROIs Stroma og Parenchyma, som i 6.3.1 og 6.3.2. Åbn APP 3 ved hjælp af appvalgdialogboksen som i 6.2.3.
- Få vist APP 3-behandling i et markeret synsfelt, der er beriget med FoxP3hiCD4+-celler. Hvis resultaterne er tilfredsstillende, skal du køre APP 3 på det fulde billede. Som produktionen af APP 3, alle de individuelle FoxP3hiCD4 + objekter vil blive mærket og deres væv koordinater gemt. Tætheder af FoxP3hiCD4 + objekter i ROIs Stroma og Parenchyma vil blive fastlagt. Resultaterne eksporteres som i 6.2.6.
- Udfør vævsvarmekortlægning af FoxP3hiCD4+ mærkede objekter.
- Åbn den brugerdefinerede protokol FoxP3hiCD4+ MAP ved hjælp af appvalgsdialogboksen som i 6.2.3.
BEMÆRK: FoxP3hiCD4+ MAP bruger koordinaterne for FoxP3hiCD4+ mærkede objekter til generering af tæthedsvarmekort. Identifikation og optælling FoxP3hiCD4 + mærkede objekter ved hjælp af APP 3 tog 25 min i billedet analyse station beskrevet. Det behandlede vævsareal var 3,2 cm2. - Kør FoxP3hiCD4+ MAP ved at trykke på knappen Kør. Eksporter vævsheatmap ved at klikke på Filer | Eksport | arbejdsområde.
BEMÆRK: Kortlægning FoxP3hiCD4 + mærkede objekter ved hjælp af FoxP3hiCD4 + MAP tog 5 min i billedet analyse station beskrevet.
- Åbn den brugerdefinerede protokol FoxP3hiCD4+ MAP ved hjælp af appvalgsdialogboksen som i 6.2.3.
- Identificer og kvantificer CD8+, CD68+, MPO+, αSMA og CD34 + objekter ved hjælp af de brugerdefinerede protokoller APP 4, APP5, APP6, APP7 og APP 8 (tabel 1) som udført i henholdsvis punkt 6.4 til 6.4.3.2 indlæsning af app af interesse i hvert enkelt tilfælde.
BEMÆRK: APPs 4 til 8 arbejde på den foruddefinerede ROIs Stroma og Parenchyma.
Representative Results
Oversigt over strategien for visualisering, kvantificering og kortlægning af cellepopulationer af interesse i TME
For at kvantificere cellepopulationer af interesse (COI'er) i forskellige vævsrum (TC'er) og for at karakterisere deres rumlige organisation, har vi designet en arbejdsgang, der integrerer prismæssigt overkommelige og nemme at bruge teknikker og maksimerer de positionsoplysninger, der kan fås ved henvendelse til dyrebare FFPE kliniske prøver (Figur 1). For det første blev ffpe-sektionerne for seriel hele væv farvestoffer til visualisering af COI'er (f.eks. immunceller) og tcs (f.eks. stroma versus parenkym)(figur 1,trin 1). Antallet af på hinanden følgende sektioner, der skal farves, bør holdes på et minimum, der gør det muligt at visualisering af de celler af interesse eller væv funktioner er nødvendige for at løse forskningsspørgsmålet. Jo mindre antallet af serielle sektioner, jo højere væv arkitektur lighed og konkordans på tværs af sammenhængende sektioner. Desuden kan multipleksing kapacitet udvides gennem genbrug af fluorescerende farvede sektioner gennem stripning og reprobing teknikker19.
Når farvningtrinnene var udført, blev en hel diasscanner brugt til at digitalisere billederne. Billeder erhvervet fra serielle sektioner blev justeret og konsolideret i en virtuel multiplex dias i en automatiseret måde (Figur 1, afsnit 2). Dernæst blev et investeringsafkast for vævet afgrænset med en brugerdefineret protokol, der identificerede vævsrelaterede pixel (TOP' er)(figur 1, trin 3). Efterfølgende blev roi-vævet opdelt i tcs defineret som yderligere ROI'er. (Figur 1, trin 4). Dernæst registreres e-pc'er, der er identificeret og kvantificerede coi'er, i forskellige overvågningsenheder (figur 1,trin 5). Endelig blev der genereret vævsheatmaps af COI'er på grundlag af deres tætheder og deres vævskoordinater (figur 1,trin 6).
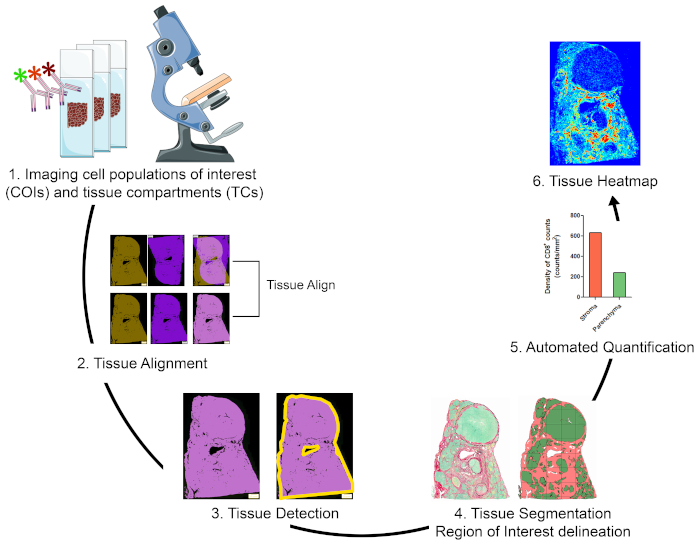
Figur 1: Skematisk repræsentation af strategien for visualisering, kvantificering og kortlægning af immunceller i TME. (1)Seriel hele væv sektioner blev farves til mærkning cois og TCs. Farves hele væv sektioner blev digitaliseret ved hjælp af en hel dias scanner. (2) Billeder erhvervet fra serielle sektioner blev forbundet, justeret, og coregistered på en automatiseret måde ved hjælp af en Tissuealign analyse modul. Der blev genereret et sammensat billede ud fra højpræcisionsjusteringen af de enkelte billeder. (3) Der blev anvendt en brugerdefineret protokol til automatiseret påvisning af vævsrelaterede pixel (TOP'er) i det sammensatte billede. (4) Vævet blev opdelt i tcs (f.eks stroma og parenkym) defineret som ROI'er. (5)Der blev anvendt brugerdefinerede protokoller til automatiseret detektion og6kvantificering af COI'er i forskellige OVERVÅGNINGS'er. Klik her for at se en større version af dette tal.
Billedbehandlings-COI'er og tcs
Tre serielle FFPE hele væv sektioner af resected tumor fra et emne med HBV-associerede hepatocellulære karcinom blev plettet i en eller flere runder af farvning som i figur 2A. Afsnit I blev plettet med H & E for at vise væv arkitektur, celle morfologi, og at bestemme klinisk relevante parametre såsom type malignitet, tumor kvalitet, og den samlede vurdering af immun infiltration (Figur 2C). I sammenhængende afsnit II blev der anvendt to runder mIF til mærkning af leverparenkymale og ikke-parenkymale celler (figur 2A). I første runde blev normale og tumorkar visualiseret ved hjælp af CD34-farvning af endotelceller. Derudover blev epitelceller (hepatocytter og cholangiocytter) identificeret ved hjælp af cytokeratin 8/18, og fibrogene aktiverede leverceller blev identificeret som alfaglatte muskelactinpositive (αSMA+) celler (Figur 2C). Efter billederhvervelse blev vævssektioner strippet og omprobed med antistoffer mod makrofager (CD68) og myofibroblaster (desmin). For bedre at karakterisere tumor immun infiltrere, tilstødende seriel afsnit III blev plettet ved hjælp af to runder af mIF for cellulære markører CD3, CD4, CD8, gaffelhoved boks P3 (FoxP3), og myeloperoxidase (MPO). I alle tilfælde blev DAPI brugt som en nuklear modplet. Endelig blev afsnit III plettet med PSR-plet og tællerefarvet med hurtig grøn for at visualisere fibrillarkollagen og segmentere vævet i stroma og parenkym (Figur 2C).
En hel slide scanner udstyret med en 20X objektiv blev brugt til at digitalisere farvede sektioner og til at skabe virtuelle dias. Seks billeder blev erhvervet fra de tre serielle sektioner (Figur 2B) og de virtuelle dias efterfølgende analyseret ved hjælp af VIS-softwaren i henhold til den skematiske repræsentation i figur 1.
Analyse af billeder
Billedanalysen omfattede fem trin: 1) vævsjustering; 2) vævsdetektering; 3) vævsegmentering; 4) automatiseret kvantificering af COI'er og 5) vævsvarme kortlægning. Alle protokoller til billedanalyse blev udviklet ved hjælp af forfattermodulet i billedanalysesoftwaren og omtales i teksten som APP.
Justering af væv
Seks virtuelle dias fra tre serielle sektioner, der spænder over 11 markører plus H & E og PSR pletter, blev indlæst i Tissualign modul af billedet analyse software. Dernæst blev billederne forbundet, justeret og coregistered på en automatiseret måde, hvilket genererede en 11-plex plus H & E og PSR virtuelle sammensatte billede, der indeholder alle lagaf de enkelte billeder (Figur 2A-C). Tilpasningen var nøjagtig for billeder fra tilstødende serielle sektioner, der viser tilsvarende vævsstrukturer placeret og arrangeret på en homolog måde ved justering (figur 2C og figur S1A). Desuden var justeringen præcis på det individuelle celleniveau for billeder, der stammer fra samme sektion (figur S1B). Tiden for automatisk justering afhænger af antallet, størrelsen, kompleksiteten og ligheden mellem de billeder, der skal justeres. Tilpasningen af de ovennævnte seks virtuelle dias tog 15 min i vores VIS station.

Figur 2: Farvning af seriel vævsektioner og billedjustering. (A) Sammenfatning af farvninger udført på tre serielle sektioner til visualisering af COI'er og TC'er. Tal i parentes angiver billedbetegnelse. For afsnit II og III blev væv strippet og omproberet med en anden cocktail af antistoffer. (B) Oversigt over seks individuelle hele vævsbilleder før og efter vævsjustering (henholdsvis venstre og højre). Skalabjælke = 3.500 μm. (C) Zoomet visning af justerede billeder. Skalabar = 80 μm. Klik her for at se en større version af dette tal.
Registrering af væv
Når billederne var forbundet og justeret, vi forsøgte at identificere De TAPs (Figur 3A). For at designe en APP til automatiseret påvisning af TOP 'er (APP 1, Tabel 1)benyttede vi os af to egenskaber, der adskiller TOP'er fra pixel, der ikke er forbundet med væv. For det første er DAPI-signalet (blåt bånd) begrænset til kernerne, som udelukkende er placeret i vævet, hvilket betyder, at alle DAPI+ pixels er en delmængde af TOP'er. For det andet har TOP'er højere autofluorescenssignal i de grønne og gule bånd sammenlignet med pixel, der ikke er forbundet med vævet. Derfor udviklede vi APP 1 til vævsdetektering (tabel 1), som registrerer TOP'erne baseret på basissignal i disse kanaler ved hjælp af enkle tærskelteknikker. Der blev angivet tærskler for de blå, grønne og gule bånd, så TA'erne havde baggrundsintensitetsværdier over tærsklerne, mens pixel, der ikke var forbundet med vævet, havde værdier nedenfor. APP 1 til vævsdetektering blev anvendt på billede IIA, som indeholder lag i de blå, grønne og gule kanaler (Figur 3A). Som output af APP 1, en lys grøn maske blev fastsat på toppen af TAP'er, og en ROI kaldet "Væv" blev afgrænset (output, Figur 3A). Desuden blev området af vævet bestemt som en kvantitativ output variabel. Da APP 1 ikke inkorporerer de pixels, der ikke er forbundet med vævet, i roivævet, blev de udelukket fra efterfølgende analyse baseret på dette investeringsafkast (figur 3A). Præcisionen af APP 1 til identifikation af TOP'er er vist i figur 3A.
Vævssegmentering og afgrænsning af INVESTERINGSAFKAST for TC'er
Dernæst fortsatte vi med at definere forskellige rum inde i ROI væv ved at segmentere vævet i stroma versus parenkym. Vi brugte PSR-plettet billede (IIIC, Figur 2C),hvor stroma kan defineres som det område, der er forbundet med aflejring af fibrillar collagens (rødt bånd), parenkym som det område, hvor fibrillar kollagener er fraværende, og den hurtige grønne counterstaining farvestof hersker (grønt bånd) (Figur 3B). Vi har oprettet APP 2 (Tabel 1) for digitalt at afgrænse TCs Stroma og Parenchyma. Denne APP virker på det foruddefinerede ROI-væv (output, figur 3A)og bruger repræsentative stroma- og parenkymområder til træning af klassificeringsværktøjet, der er integreret i billedanalysemodulet. Den uddannede klassificering tildeler pixels til enten en stroma eller en parenkym etiket (laks og grøn, henholdsvis figur 3B). Ved klassificering af pixels udførte APP 2 morfologiske operationer med henblik på at definere ROI Stroma og Parenchyma (figur 3B og tabel 1). APP 2's ydeevne ved klassificering af pixels og generering af de respektive ROI'er er vist i figur 3B. Derudover kvantificerer APP 2 stroma- og parenkymområdet. Endelig, selv om segmenteringen sker ved hjælp af PSR farves sektion, kan de skitserede stroma og parenkym regioner overføres til ethvert billede justeret til PSR billedet.

Figur 3: Automatiseret vævsdetektering/segmentering og generering af respektive ROI'er. (A) Billed-IIA blev brugt til at identificere TA'erne (venstre billede, skalalinjen = 6.000 μm). En lys grøn maske blev tildelt tOP'erne ved hjælp af APP 1 (Tabel 1),der genererer et investeringsafkast kaldet Tissue (output 1). Højre, indsat viser zoomet visning viser præcisionen af APP 1 på detektering TAPs. Skalabjælke = 350 μm. (B) ROI-væv (output 1) segmenteres i stroma og parenkym ved hjælp af APP 2. Billedet til venstre viser en visning af ROI Væv segmenteret i ROI stroma (laks) og ROI parenkym (grøn). Skalabar = 4.500 μm. Til højre, zoomet visninger af indsat for ROI Tissue, den oprindelige PSR farvning (billede IIIC), og ROIs stroma og parenkym. Skalabjælke = 250 μm. Klik her for at se en større version af dette tal.
Automatiseret kvantificering af COI'er
Dernæst fortsatte vi med at identificere, lokalisere og kvantificere COI'er i ROIs Stroma og Parenchyma. AOP'er 3 til 8 (Tabel 1) blev oprettet for at finde og tælle følgende COI'er: CD4+FoxP3+, CD8+, CD68+, MPO+, αSMA+, og CD34+ celler. APP 3 er designet til at lokalisere og tælle CD4+FoxP3+ celler (billede IIIA, figur 2C) som surrogatmarkører for regulerende T-celler (Tregs). Denne protokol registrerer samlokalisering af signalet fra den nukleare transskriptionfaktor FoxP3 (rødt bånd) og DNA-mærkningsfarvestoffet DAPI (blåt bånd). I betragtning af at nyligt aktiverede T-celler upregulate FoxP3, at berige for Tregs vi fastsætte tærskler for preselecting kun lyse FoxP3 + celler (FoxP3hi). Dernæst, ud af alle forudvalgte DAPI + FoxP3hi celler, kun dem, der var omgivet af lyse ringformede CD4 signaler (grønt bånd) blev mærket og tælles som FoxP3hiCD4 + celler (pink label, Figur 4A). Tætheden af FoxP3hiCD4+ celler i ROIs Stroma og Parenchyma blev bestemt som kvantitative outputvariabler af APP 3 (Figur 4A).
På samme måde blev AOP'er 4 til 6 designet til påvisning af CD8+-, CD68+- og MPO+-celler. Disse ApP'er har samme basisdesign til påvisning og kvantificering af COI'er. Specifikt identificeres COI'er baseret på signalintensiteten fra den specifikke cellepopulationsbiomarkør, og derefter udføres flere morfologiske trin efter forarbejdning for at afgrænse individuelle celler (tabel 1). De enkelte celler eller coi'er mærkes, tælles, og deres vævskoordinater registreres. AOP'er 4 til 6 bestemmer også tætheden af COI'erne i ROI'erne Stroma og Parenkym (figur 4B-D).
Kvaliteten af vores DAPI-farvning var ikke god nok til at integrere kernersegmentering i AOP'er 3 til 6, så vi kan ikke sikre, at alle individuelt mærkede objekter er individuelle celler. Af denne grund udtrykte vi cellernes massefylde i optællinger af mærkede objekter/mm2 (Figur 4). Celleaggregater blev dog med succes opdelt i individuelle celler i efterbehandlingstrinnene, der er indbygget i AOP'er 3 til 6, og omfattende visuel inspektion viste, at de fleste mærkede objekter svarede til enkelte celler.
Til detektering af αSMA+ og CD34+-området udviklede vi Henholdsvis POP'er 7 og 8 (tabel 1). Begge AOP'er registrerer det specifikke signal baseret på tærskler og bestemmer procentdelen af positive områder i ROI Stroma og Parenchyma (figur 4E-F).
En af de mest interessante muligheder for at generere virtuelle multiplex dias er analysen af colocalization udtryk. Vi genererede APP 10 for at opdage samlokalisering mellem αSMA og desmin, to markører udtrykt af myofibroblaster i leveren. APP 10 anvender grænseværdier for at finde pixel, der er positive for αSMA, desmin og αSMA plus desmin (tabel 1). Som kvantitative outputvariabler bestemmer APP 10 αSMA+-området, desmin+-området og området med samlokaliseret udtryk for disse to markører (figur S3).

Figur 4: Identifikation og kvantificering af COI'er i tcerne stroma og parenkym. (A–F) Automatiseret detektion og kvantificering af CD4+FoxP3+, CD8+, CD68+, MPO+, αSMA+, og CD34+ COI'er i ROIs Stroma og Parenchyma ved hjælp af henholdsvis protokol 3, 4, 5, 6, 7 og 8 (tabel 1). Vist til venstre er de originale billeder, i midten de behandlede billeder, og til højre kvantificeringer. For figur 4A-Dskalerestilsen = 40 μm. For figur 4E og Fskalabar = 350 μm. Klik her for at se en større version af dette tal.
Som et alternativ til kvantificering af COI'er i TCs Stroma og Parenchyma, vi bestemmes tætheden af immunceller i de forskellige maligne knuder navngivet 1 til 4(Figur 5A, H, og I). Investeringsafkastet for hver knuder blev manuelt afgrænset som angivet i figur 5A. Karakteristiske væv immun signaturer karakteriseret hver knud, yderligere afslører den iboende heterogenitet af TME.
Væv Heatmaps
Som nævnt ovenfor opbevarer AOP'er 3 til 8 vævskoordinaterne for hver enkelt mærket genstand. Denne funktion gør det muligt at automatisere den automatiserede generation af vævskort, hvor områder med høj tæthed af en given cellepopulation vises som hotspots (rød) og områder med relativt lav tæthed som kolde pletter (mørkeblå). Mellemliggende tæthedsværdier tildeles farver i henhold til den farveskala, der er vist i figur 5. Vævsheatmaps blev genereret af AOP'er, der delte billederne i cirkler med en diameter på 50 μm og tildelte en farve i henhold til den relative tæthed af en given COI inde i cirklen. Som det fremgår af figur 5B-G, var positioneringsmønstrene og intensitetsfordelingen af de forskellige COI'er i TME ret varierede. Desuden var placeringen af forskellige populationer i vævsområdet på niveau med individuelle knuder enestående (figur S2A-C). For at give et eksempel på styrken ved denne teknik og for at visualisere den rumlige organisering af hotspots fra forskellige populationer i samme knud blev hotspots fra de enkelte celletyper manuelt udvundet og kortlagt sammen på omridset af knud2 (Figur S2, Figur Dog Figur E).

Figur 5: Vævsvarmekort over COI'er i TME. (A) Picrosirius Rød farvning med angivelse af nodules 1, 2, 3 og 4. (B–G) Vævsheatmaps til henholdsvis CD4+FoxP3+, CD8+, CD68+, MPO+, CD34+ og αSMA+COI'er. Mørkeblå angiver relativ lav tæthed, og rød angiver relativ høj tæthed. Mellemliggende tæthedsværdier tildeles farver i henhold til den viste farveskala. (H og I) Kvantificering af COI'er i knuder 1, 2 og 3 + 4 organiseret pr. celletype og pr. nodule. Klik her for at se en større version af dette tal.

Supplerende figur S1: Validering af vævsjustering. (A) CD34 farvning (med rødt) udført på afsnit II (indgang 1) bruges til at generere en CD34 maske i grøn (udgang 1). Den grønne maske (output 1) er overlejret på H&E-billedet fra den justerede seriesektion I (input 2). Den fusionerebilledet viser perfekt korrespondance af vaskulære strukturer. Skalabar = 50 μm. (B) Billede IIIA, der viser sammenfletningen af DAPI, CD4 og FoxP3 (input 1), blev brugt til at generere en etiket til CD4+FoxP3+-celler (output 1 i magenta). Output 1-mærket blev overført til justeret billede IIIB (input 2) og viser perfekt overensstemmelse mellem parrene FoxP3/DAPI og CD4/CD3 i flettebilledet. Skalabar = 15 μm. Klik her for at se en større version af dette tal.

Supplerende figur S2: Zoomet udsyn til vævsvarmekort. (A–C) Vævsheatmaps til CD4+FoxP3+, CD8+, CD68+ og MPO+-celler i knuder 1-4. Skalastænger ne i knuder 1, 2 og 3 + 4 repræsenterer henholdsvis 1.500 μm, 700 μm og 500 μm. (D) Kontur af knud2 med sort fast linje. (E) Hotspots for CD4+FoxP3+, CD8+, CD68+, og MPO+ celler i nodule 2 blev udtrukket og kortlagt sammen på knuden 2,som er defineret i D. Klik her for at se en større version af dette tal.

Supplerende figur S3: Kolokaliseringsanalyse. (A) Til venstre og i midten er der billeder af αSMA-mærket i henholdsvis grøn og desminetiket med rødt. Til højre ses et αSMA/desmin dobbelt positivt område i gult. (B) Kvantificering af αSMA+-området, desmin + område og αSMA/desmin dobbeltpositivt område. Skalabar = 150 μm. Klik her for at se en større version af dette tal.
| App | Formål | Klassificering | Klassificering | Trin til efterbehandling | Outputvariabler |
| Metode | Funktioner | ||||
| (pixelværdi) | |||||
| 1 | Detektering af væv | Tærskel | Kanal DAPI (150) | o Mærk objekter med colocalized over tærskelværdier for de 3 kanaler | o ROI Væv |
| Kanal FITC/A488 (120) | o Luk positivt objekt 5 pixel | o Vævsområde | |||
| Kanal TRITC/A568 (40) | o Opret ROI-væv | ||||
| 2 | Vævssegmentering | Beslutning Skov | RGB-R median | o Fyld huller | o ROI Stroma |
| RGB-G median | o Opret ROI Stroma | o Stroma-området | |||
| RGB-B median | o Opret ROI Parenchyma | o ROI Parenchyma | |||
| IHS-S median | o Parenkyma-området | ||||
| H&E Eosin median | |||||
| 3 | Sådan finder og kvantificeres CD4+ FoxP3+-celler | Tærskel | Kanal DAPI (>600) | o Mærk objekter med samlokalisering af DAPI og Cy5/A647, omgivet af FITC/A488-signal | o Tællinger og tæthed af CD4+FoxP3+ celler i ROIs Stroma og Parenchyma |
| Kanal FITC/A488 poly udjævning (>850) | o Ryd objekter, der er mindre end 7 μm2 | o Koordinater for individuelle CD4+FoxP3+ celler | |||
| Kanal Cy5/A647(>800) | |||||
| 4 | Sådan finder og kvantificeres CD8+-celler | Tærskel | Kanal DAPI (<1200) | o Klare positive objekter, der er mindre end 15 μm2 | o Tællinger og tæthed af CD8+ celler i ROIs Stroma og Parenchyma |
| Kanal Cy5/A647 median (>80) | o Luk positive objekter 2 pixel | o Koordinater for de enkelte celler | |||
| o Adskil objekter | |||||
| 5 | Sådan finder og kvantificeres CD68+-celler | Tærskel | Kanal FITC/A488 (>200) | o Klare positive objekter, der er mindre end 20 μm2 | o Tælling er og tæthed af CD68+ celler i ROIs Stroma og Parenchyma |
| o Spil positive objekter 3 pixel | o Koordinater for individuelle CD68+ celler | ||||
| o Adskil objekter | |||||
| 6 | Sådan lokaliseres og kvantificeres MPO+-celler | Tærskel | Kanal DAPI (>400) | o Ryd objekter, der er mindre end 5 μm2 | o Tællinger og tæthed af MPO+ celler i ROIs Stroma og Parenchyma. |
| Kanal TRITC/A568 (900-4000) | o Spil 3 pixels positive objekter | o Koordinater for de enkelte MPO+-celler. | |||
| o Adskil objekter | |||||
| 7 | Sådan lokaliseres og kvantificeres αSMA+-området | Tærskel | Kanal TRITC/CF568 (>1050) | o Klare positive objekter, der er mindre end 25 μm2 | o Tællinger og tæthed af αSMA+ område i ROIs Stroma og Parenchyma |
| o Spil 3 pixels positive objekter | o Koordinater for αSMA+ pixels | ||||
| 8 | Sådan finder og kvantificeres CD34+-området | Tærskel | Kanal DAPI (<5000) | o Klare positive objekter, der er mindre end 25 μm2 | o Tællinger og tæthed af CD34+ område i ROIs Stroma og Parenchyma |
| Kanal Cy5/A647 median (>120) | o Spil 3 pixels positive objekter | o Koordinater for CD34+ pixels | |||
| 9 | Opret vævsheatmaps for en given cellepopulation | Objektheatmap | Objektheatmap | o Heatmap | |
| Tegningsradius 50 μm | --- | ||||
| 10 | Kvantificer samlokalisering mellem αSMA og Desmin | Tærskel | Kanal TRITC (CF568) (>1050) | o Mærk objekter med ovenstående tærskelværdier for TRITC (CF568) | o Kvantificer samlokaliseret udtryk for αSMA og Desmin |
| Kanal Cy5 (A647) (>1000) | o Mærk objekter med ovenstående tærskelværdier for Cy5 (A647) | ||||
| o Mærke objekter med samlokalisering af værdier over tærskelværdier for TRITC (CF568) og Cy5 (A647) | |||||
| o Klare positive objekter, der er mindre end 25 μm2 |
Tabel 1: Generelle parametre, der anvendes til design af APP'er, der anvendes til billedanalyse. De parametre, der er angivet i denne tabel, justeres til de unikke egenskaber ved de billeder, der anvendes i denne analyse (f.eks. baggrund, artefakter osv.), og gælder muligvis ikke for andre billeder. Da de nævnte efterbehandlingstrin blev defineret for de specifikke billeder, der analyseres i denne undersøgelse, er de bevidst ikke detaljerede. Brugeren skal tilpasse APPs til de billeder, der skal analyseres.
| Sektion/farvning | Primært antistof | Sekundært antistof |
| Sektion II/1st Farvning | Mus IgG2a anti-menneskelige αSMA Mus IgG1 anti-menneskelige CD34 Kanin anti-menneskelige Cytokeratin 8/18 |
Ged anti-mus IgG2a CF568 Rotte anti-mus IgG1 A647 Æsel anti-kanin A488 |
| Afsnit II/2: Farvningnd | Kanin anti-menneskelige Desmin Mus anti-menneskelige CD68 |
Æsel anti-kanin A647 Æsel anti-mus DyLight 755 |
| Sektion III/1st Farvning | Mus anti-menneskelige CD4 Kanin anti-menneskelige FoxP3 Ged anti-menneskelige MPO |
Æsel anti-mus A488 Æsel anti-kanin A647 Æsel anti-ged A568 |
| SektionIII/2: Farvning | Kanin anti-menneskelige CD3 Mus anti-menneskelige CD8 |
Æsel anti-mus DyLight 755 Æsel anti-kanin A647 |
Tabel 2: Paraf mIF i primær sekundært antistof.
Discussion
Enkel, tilgængelig og nem at udføre multiplexing teknikker, der tillader rumlig opløsning af immunceller i væv sektioner er nødvendige for at kortlægge immunlandskabet i kræft og andre immunologiske lidelser. Her beskriver vi en strategi, der integrerer bredt tilgængelige mærknings- og digitale analyseteknikker for at udvide multiplexing-kapaciteten og flerdimensional vurdering af billeddiagtorer12,13,17,19. Farvning af tre serielle sektioner til forskellige markører, og genbrug af sektioner gennem stripning og reprobing teknikker, gjorde det muligt for os at visualisere 11 parametre ud over H & E og PSR pletter. Seks billeder fra disse sektioner blev justeret på en automatiseret måde ved hjælp af væv justering modul. Justeringen var præcis på det individuelle celleniveau for billeder, der stammer fra samme sektion og meget konkordans for billeder, der stammer fra tilstødende sektioner. Virtuel multipleksing gjorde det muligt for os at bestemme, hvordan markører visualiseret i en sektion relatererumsligt til markører visualiseret i en anden sammenhængende sektion. Mens nogle af de farvninger mærket COI'er, andre mærket TCs, giver os mulighed for at kvantificere cois i de forskellige tcs. Brugen af softwareværktøjer til automatiseret kvantificering af COI'er forenklede og fremskyndede i høj grad behandlingen af billeder. Desuden blev der anvendt digital analyse på hele vævssektioner i stedet for udvalgte synsfelter, hvilket resulterede i en upartisk repræsentation af TME. Da vævkoordinaterne for COI'er blev registreret, var det desuden muligt at generere vævsheatmaps.
Der er flere områder i denne protokol, hvor der kan være behov for fejlfinding. For det første kan dårlig antigenudtagning påvirke kvaliteten af mIF, og typen af antigenudtagningsbuffer og varighed bør derfor optimeres for de specifikke assay-/biomarkørbetingelser, der anvendes. For det andet bør den anvendte type blokeringsopløsning tilpasses væv/antigen/arter af primære og sekundære antistoffer. I vores hænder, tilsætning af 10% samlede serum fra de arter, hvor vævet kommer fra blokerede Fc receptorer, og dermed reduceret uspecifik antistof bindende. Tilsætning af 10% serum fra arten de sekundære antistoffer blev rejst i ville minimere direkte uspecifik fastgørelse af sekundære antistoffer til vævssektionen. For det tredje er det vigtigt at validere de primære og sekundære antistoffers specificitet ved hjælp af de korrekte positive og negative kontroller. For det fjerde er øget autofluorescens i nogle kanaler og diffusion af DAPI ved primær antistofstripning også almindelig. For at løse den forbedrede autofluorescens brugte vi primære/sekundære antistofpar, hvor det specifikke signal havde intensitetsværdier mindst 5x baggrundsværdien. Endelig kan nogle højaffinitet antistoffer ikke elueres med regelmæssige stripping procedurer. I dette tilfælde anbefaler vi at bruge sådanne antistoffer i den sidste runde af mærkning. Brugeren kan have til at prøve forskellige farvning sekvenser for at finde den optimale konfiguration for antistoffer af interesse. Effektiviteten af stripningbør bekræftes, før der går videre til en anden eller tredje runde af mærkningen.
Den største begrænsning og udfordring ved denne strategi er at finde de rigtige kombinationer af primære og sekundære fluorescerende antistoffer til markører af interesse. At finde primære antistoffer, der er opdrættet i forskellige arter eller med forskellige isotyper, og som kan anvendes samtidigt, begrænses af, hvad der er kommercielt tilgængeligt. De fleste hele diasscannere er udstyret med lamper og filtre, der tillader billeddannelse maksimalt fem kanaler, og sekundære antistoffer i den rigtige art og højre fluorophore er ikke altid tilgængelige. Vi overvandt delvist disse begrænsninger ved hjælp af serielle farvninger og sekventiel mærkning. Det kan være nødvendigt at teste flere antistofkombinationer for at nå frem til den bedste kombination for de markører, der er interessante. En anden begrænsning er kvaliteten af DAPI farvning, fordi stripning og reprobing ikke altid tillader at udføre kerner segmentering.
Vævet tilpasse modul kræver minimal uddannelse og ingen programmering færdigheder fra brugerne. Softwaren teoretisk tillader tilpasning af et ubegrænset antal billeder. Men præcis tilpasning afhænger af beslægtede sektioner, hvor tættere sektioner, der er mere histologisk konkordans er mere præcist afstemt. Vi brugte author-modulet i VIS til at generere ApPs. Grundlæggende viden om billedanalyse er nødvendig for at skabe APPs, men det er også tilfældet, når du bruger andre billedanalyse software. De unikke fordele ved VIS i forhold til andre billedanalysesoftware omfatter automatiseret justering af billeder fra sektioner, der er udarbejdet ved hjælp af forskellige metoder (f.eks. Dette giver mulighed for colocalization undersøgelser af flere markører af interesse ved hjælp af virtuelle multiplexing. Desuden giver det fleksible og brugervenlige design af AOP'er mulighed for brugerspecifik tilpasning. Automatiseret kvantificering og kortlægning, og muligheden for behandling af hele vævsektioner, sparer tid og reducerer bias sammenlignet med manuel optælling ved visuel inspektion.
Denne strategi er et meget nyttigt forskningsværktøj til vævimmunologi i forbindelse med kræft og autoimmunitet, men forbliver ikke valideret til klinisk brug. Med yderligere standardisering og validering, kan det bruges i fremtiden til flere anvendelser (f.eks at kortlægge immunlandskabet i kræft til at forudsige og overvåge reaktionen på immunterapeutiske midler). Det kan også tilpasses forskellige inflammatoriske tilstande (f.eks. inflammatorisk tarmsygdom) for at kombinere patologisk evaluering med prognostiske biomarkører.
De vigtigste kritiske trin i denne protokol er mærkningens effektivitet/specificitet og de konstruerede AOP'ers robusthed til den tilsigtede anvendelse eller biomarkør. Derfor er regelmæssig validering ved visuel inspektion, især ved at designe en ny APP, afgørende. Effektiv brug af flere runder af stripning og reprobing eller forskellige typer af pletter på samme sektion er kritiske komponenter og kan være væv eller afsnit specifikke. Det er vigtigt at kontrollere effektiviteten af sådanne processer, før du fortsætter med en stor batchanalyse.
Sammenfattende giver vi en strategi, der maksimerer de kvantitative og rumlige oplysninger, der kan opnås fra værdifulde kliniske vævsprøver. De ressourcer, det udstyr og den viden, der er nødvendig for at gennemføre denne metode, er bredt tilgængelige. Vi foreslår denne metode som en nyttig vejledning til planlægning af analyser, der har til formål at identificere, kvantificere og kortlægge immuncellepopulationer i TME.
Disclosures
Forfatterne erklærer ingen interessekonflikter.
Acknowledgments
Vi takker studiedeltageren. Vi takker Louise Rousseau, koordinator for HBP-biobanken for genvinding af vævsprøverne og alle tilknyttede kliniske oplysninger. Vi anerkender den molekylære patologi og celle imaging kernefaciliteter på CRCHUM og Michael Persch fra Visiopharm for fremragende teknisk bistand. Finansiering: Denne undersøgelse blev støttet af tilskud fra Canadian Liver Foundation, Fonds de recherche du Québec-Santé (FRQS) AIDS and Infectious Disease Network (Réseau SIDA-MI) og Canadian Network on Hepatitis C (CanHepC). CanHepC er finansieret af et fælles initiativ fra den canadiske Institutes of Health Research (CIHR) (NHC-142832) og Public Health Agency of Canada. M.F.M. modtog stipendier fra Université de Montréal, Bourse Gabriel Marquis og FRQS. T.F. modtog ph.d.-stipendier fra CIHR og CanHepC. ST holder Roger-Des-Groseillers stol i hepatobiliary og pancreas onkologisk kirurgi, Université de Montréal.
Forfatterbidrag: M.F.M. designet, udført eksperimenter, og analyserede data. T.F. designede eksperimenter. A.C-B. teknisk vejledning. G.S. udførte alle de patologiske vurderinger af forsøgspersonen og gav input til alle de patologiske aspekter. L.M. udførte H&E-farvning, optimeret og udført billedanskaffelse. M.N.A. udførte PSR-pletten og leverede værdifulde tekniske input. N.B. bidrog til billedanalysen. S.T. er hovedinvestigator for HBP-biobanken og er ansvarlig for at føre tilsyn med biobankens samlede drift. Han gav også uvurderlige input om alle aspekter af projektet og dets kliniske konsekvenser. M.F.M. T.F., og N.H.S. konceptualiseret og designet undersøgelsen. N.H.S. overvågede arbejdet og opnåede finansiering. M.F.M., T.F., A.C-B og N.H.S. skrev manuskriptet. Alle forfattere gennemgik og godkendte manuskriptet.
Materials
| Name | Company | Catalog Number | Comments |
| Antigen Retrieval Solution: Sodium Citrate Buffer (10 mM Sodium Citrate, 0.05% v/v Tween 20, pH 6.0) | |||
| Blocking Solution: 1 % BSA, 10 % filtered human serum, 10 % filtered donkey serum, 0.1 % Tween 20, and 0.3% Triton in PBS | |||
| Bovine serum albumin (BSA) | Multicell | 800-095-EG | |
| Coplin jars (EASYDIP SLIDE STAINING SYSTEM) | Newcomersupply | 5300KIT | |
| Cover slides | Fisherbrand | 12-545E 22*50 | |
| Direct Red 80 | Sigma Aldrich | 365548 | |
| Donkey Serum | Sigma Aldrich | D9663 | |
| Ethanol 100% | |||
| Electric pressure cooker | Salton | ||
| Eosin | Leica Biosystems | 3801600 | CAUTION, eye irritation |
| Fast Green FCF | Sigma Aldrich | F7252 | CAUTION, harmful by inhalation, ingestion and skin absortion |
| FFPE section (4μm) slides | |||
| Glycine 0,1 M in PBS | |||
| Hematoxylin Stain Solution, Gil 1. Formulation, Regular Strength | Ricca Chemical Company | 3535-32 | |
| Holder (EasyDip Staining Jar Holder) | Newcomersupply | 5300RK | |
| Human Serum | Gemini | 22210 | |
| Humidity chamber | Millipore Sigma | Z670138-1EA | |
| Pap pen | abcam | ab2601 | |
| PBS | |||
| PBS-Tween 20 (0.1% v/v) | |||
| Permount Mounting Media | Fisher Chemical | SP15-500 | |
| Picric Acid 1.3 % | Sigma Aldrich | P6744 | CAUTION, skin and eye irritation |
| Picro-Sirius Red/Fast Green solution: Fast Green 0.1 % w/v + Sirius Red 0.2 % w/v in 1,3 % picric acid solution | |||
| Primary Antibody Anti-αSMA | Mouse IgG2a 1A4 | Sigma A2547 | Dilution 1/100 |
| Primary Antibody Anti-CD34 | Mouse IgG1 HPCA1/763 | Novus Biologicals NBP2-44568 | Dilution 1/250 |
| Primary Antibody Anti-Cytokeratin 8/18 | Rabbit EP17/EP30 | Agilent IR09461-2 | Ready to use |
| Primary Antibody Anti-CD68 | Mouse KP1 | Abcam ab955 | Dilution 1/200 |
| Primary Antibody Anti-Desmin | Rabbit Polyclonal | Invitrogen PA5-16705 | Dilution 1/200 |
| Primary Antibody Anti-CD4 | Mouse N1UG0 | Affymetrix 14-2444 | Dilution 1/250 |
| Primary Antibody Anti-FoxP3 | Rabbit 1054C | R & D MAB8214 | Dilution 1/100 |
| Primary Antibody Anti-MPO | Goat Polyclonal | R & D Systems AF3667 | Dilution 1/250 |
| Primary Antibody Anti-CD3 | Rabbit SP7 | Abcam ab16669 | Dilution 1/200 |
| Primary Antibody Anti-CD8 | Mouse C8/144B | Invitrogen 14-0085-80 | Dilution 1/200 |
| Secondary Antibody Donkey anti-mouse A488 | Polyclonal | Invitrogen A-21202 | Dilution 1/500 |
| Secondary Antibody Donkey anti-Rabbit A488 | Polyclonal | Invitrogen A-21206 | Dilution 1/500 |
| Secondary Antibody Donkey anti-goat A568 | Polyclonal | Invitrogen A-11057 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit A647 | Polyclonal | Invitrogen A-31573 | Dilution 1/500 |
| Secondary Antibody Rat anti-mouse IgG1 A647 | RMG1-1 | Biolegend 406618 | Dilution 1/500 |
| Secondary Antibody Goat anti-mouse IgG2a CF568 | Polyclonal | Sigma Aldrich SAB4600315 | Dilution 1/500 |
| Secondary Antibody Donkey anti-mouse DyLight 755 | Polyclonal | Invitrogen SA5-10171 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit DyLight 755 | Polyclonal | Invitrogen SA5-10043 | Dilution 1/500 |
| SDS | BioShop | SDS001,500 | CAUTION, oral skin and eye toxicity |
| Shandon multi-program robotic slide stainer | LabX | 11384903 | |
| Shandon Xylene Substitute, | Thermo Fisher Scientific | CA89413-336 | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Shaking water bath | |||
| SlowFade Gold antifade reagent with DAPI | Invitrogen | S36938 | |
| Sodium Citrate Dihydrate | Millipore Sigma | 1545801 | CAUTION, eye irritation |
| Stripping Buffer: mix 20 ml 10% w/v SDS with 12.5 ml 0.5 M Tris-HCl (pH 6.8), and 67.5 ml ultra-pure water. Under a fume hood, add 800 uL of 2-mercapto ethanol (114,4 mM final concentration) | |||
| Triton X-100 | Sigma Aldrich | T8787-50ML | |
| Tris-HCl | BioShop | 77-86-1 | |
| Tween 20 | Fisher Scientific | BP337-500 | |
| VIS Software | Visiopharm | ||
| Whole slide scanner Olympus BX61VS | Olympus | Microscope: Olympus Slide Scanner BX61VS, 5 slides scanner, motorized stage, autofocus. Camera: Lightsource: Xcite-120. Filters: BrightLine® Sedat filter set (# LED-DA/FI/TR/Cy5-4X4M-B-000, Semrock) | |
| Xylene | Sigma Aldrich | 214736-4L | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Xylene : Ethanol solution (1:1 v/v) | |||
| 2-mercaptoethanol | Sigma | M6250 | CAUTION, harmful by ingestion, inhalation, fatal if sking absortion. Eye irritation. Use fume hood |
References
- Greten, F. R., Grivennikov, S. I. Inflammation and Cancer:Triggers, Mechanisms, and Consequences. Immunity. 51 (1), 27-41 (2019).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Binnewies, M., et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nature Medicine. 24 (5), 541-550 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathology: an official journal of the United States and Canadian Academy of Pathology, Inc. 31 (2), 214-234 (2018).
- Bindea, G., et al. Spatiotemporal dynamics of intratumoral immune cells reveal the immune landscape in human cancer. Immunity. 39 (4), 782-795 (2013).
- Galon, J., et al. Towards the introduction of the 'Immunoscore' in the classification of malignant tumours. The Journal of Pathology. 232 (2), 199-209 (2014).
- Finotello, F., Eduati, F. Multi-Omics Profiling of the Tumor Microenvironment: Paving the Way to Precision Immuno-Oncology. Frontiers in Oncology. 8, 430 (2018).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: a method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Porta Siegel, T., et al. Mass Spectrometry Imaging and Integration with Other Imaging Modalities for Greater Molecular Understanding of Biological Tissues. Molecular Imaging and Biology : MIB: the official publication of the Academy of Molecular Imaging. 20 (6), 888-901 (2018).
- Buchberger, A. R., DeLaney, K., Johnson, J., Li, L. Mass Spectrometry Imaging: A Review of Emerging Advancements and Future Insights. Analytical Chemistry. 90 (1), 240-265 (2018).
- Pirici, D., et al. Antibody elution method for multiple immunohistochemistry on primary antibodies raised in the same species and of the same subtype. Journal of Histochemistry and Cytochemistry. 57 (6), 567-575 (2009).
- Gendusa, R., Scalia, C. R., Buscone, S., Cattoretti, G. Elution of High-affinity (>10-9 KD) Antibodies from Tissue Sections: Clues to the Molecular Mechanism and Use in Sequential Immunostaining. Journal of Histochemistry and Cytochemistry. 62 (7), 519-531 (2014).
- van der Loos, C. M. Multiple immunoenzyme staining: methods and visualizations for the observation with spectral imaging. Journal of Histochemistry and Cytochemistry. 56 (4), 313-328 (2008).
- Stack, E. C., Wang, C., Roman, K. A., Hoyt, C. C. Multiplexed immunohistochemistry, imaging, and quantitation: a review, with an assessment of Tyramide signal amplification, multispectral imaging and multiplex analysis. Methods. 70 (1), 46-58 (2014).
- Toth, Z. E., Mezey, E. Simultaneous visualization of multiple antigens with tyramide signal amplification using antibodies from the same species. Journal of Histochemistry and Cytochemistry. 55 (6), 545-554 (2007).
- Robertson, D., Savage, K., Reis-Filho, J. S., Isacke, C. M. Multiple immunofluorescence labeling of formalin-fixed paraffin-embedded (FFPE) tissue. BMC Cell Biology. 9, 13 (2008).
- Segnani, C., et al. Histochemical Detection of Collagen Fibers by Sirius Red/Fast Green Is More Sensitive than van Gieson or Sirius Red Alone in Normal and Inflamed Rat Colon. PloS One. 10 (12), 0144630 (2015).
- Bolognesi, M. M., et al. Multiplex Staining by Sequential Immunostaining and Antibody Removal on Routine Tissue Sections. Journal of Histochemistry and Cytochemistry. 65 (8), 431-444 (2017).



