Summary
هنا، نصف استراتيجية بسيطة ويمكن الوصول إليها لتصور وقياس ورسم خرائط الخلايا المناعية في أقسام أنسجة الورم المدمجة البارافين. تجمع هذه المنهجية بين تقنيات التصوير والتحليل الرقمي الحالية بهدف توسيع قدرة تعدد الإرسال وتحليل المعلمات المتعددة لفحوصات التصوير.
Abstract
المشهد المناعي للبيئة الدقيقة للورم (TME) هو عامل حاسم في تطور السرطان والاستجابة للعلاج. على وجه التحديد ، فإن كثافة وموقع الخلايا المناعية في TME لها قيم تشخيصية وتشخيصية مهمة. وقد أدى التنميط متعدد المجالات للTME إلى زيادة فهمنا للعديد من الشبكات الخلوية والجزيئية التي تنظم بدء الورم وتطوره. ومع ذلك، لا توفر هذه التقنيات معلومات حول التنظيم المكاني للخلايا أو تفاعلات الخلية الخلية. هناك حاجة إلى تقنيات متعددة السهولة يمكن الحصول عليها بأسعار معقولة ومتاحة وسهلة التنفيذ تسمح بالاستبانة المكانية للخلايا المناعية في أقسام الأنسجة لاستكمال تقنيات الإنتاجية العالية القائمة على الخلايا الواحدة. هنا ، نحن نصف استراتيجية تدمج التصوير التسلسلي ، والوسم التسلسلي ، ومحاذاة الصورة لتوليد شرائح متعددة المعلمات الظاهرية من أقسام الأنسجة بأكملها. يتم تحليل الشرائح الظاهرية لاحقًا بطريقة تلقائية باستخدام بروتوكولات محددة من قبل المستخدم تمكن من تحديد مجموعات الخلايا ذات الأهمية وتقديرها الكمي وتعيينها. يتم تحليل الصورة ، في هذه الحالة باستخدام وحدات التحليل من حيث الأنسجة والمؤلف وHISTOmap. نقدم مثالاً حيث طبقنا هذه الاستراتيجية بنجاح على عينة سريرية واحدة، وتعظيم المعلومات التي يمكن الحصول عليها من عينات الأنسجة المحدودة وتوفير وجهة نظر غير متحيزة من TME في قسم الأنسجة بأكمله.
Introduction
تطور السرطان هو نتيجة لعملية متعددة الخطوات تنطوي على تفاعلات متبادلة بين الخلايا الخبيثة وTME. بخلاف الخلايا السرطانية ، يتكون TME من خلايا غير خبيثة ، وخلايا سترومال ، وتجمعات الخلايا المناعية ، ومصفوفة خارج الخلية (ECM)1. التنظيم المكاني للمكونات الخلوية والهيكلية المختلفة للأنسجة الورم والتبادل الديناميكي بين السرطان والخلايا المجاورة غير السرطانية في نهاية المطاف تعديل تطور الورم والاستجابة للعلاج2،3،4. وقد ثبت أن الاستجابة المناعية في السرطان هو تنظيم spatiotemporally5,6. مجموعات مختلفة من الخلايا المناعية التي تتسلل إلى آفة البلاستيك النيولي والأنسجة المجاورة تظهر أنماط توزيع مكانية مميزة وحالات تنشيط وتمايز متنوعة مرتبطة بوظائف مختلفة (على سبيل المثال، الموالية مقابل المضادة للورم). هذه الفئات المختلفة المناعية ومعلماتها تتطور العمل الإضافي مع الورم والمقصورات سترومال.
وقد أدى ظهور التقنيات التي تسمح بتنميط متعدد الخلايا المفردة إلى زيادة فهمنا للعديد من الشبكات الخلوية والجزيئية التي تنظم التسرطن وتطور الورم. ومع ذلك، تتطلب معظم الأدوات التحليلية عالية الإنتاجية القائمة على الخلية الواحدة تعطيل الأنسجة وعزل الخلية الواحدة، مما يؤدي إلى فقدان المعلومات حول التنظيم المكاني للخلايا والتفاعلات بين الخليةوالخلية 7. لأن موقع وترتيب خلايا مناعية محددة في TME لها قيمة تشخيصية وتشخيصية، والتكنولوجيات التي تسمح الاستبانة المكانية هي تكملة أساسية لتقنيات التنميط المناعي الواحدة القائمة على الخلية.
تقليديا، تقنيات التصوير مثل الكيمياء المناعية (IHC) والفلورالمناعة متعددة (mIF) وقد اقتصرت على عدد قليل من المؤشرات الحيوية التي يمكن تصورها في وقت واحد. وقد أعاق هذا القيد دراسة الديناميات الصدغية للخلايا المناعية المتسللة للورم ، والتي يتم تعريفها عادة من خلال العديد من العلامات النفية. وقد أدت التطورات الأخيرة في أدوات التصوير والتحليل إلى توسيع إمكانيات تعدد الإرسال. وقد استخدمت تقنيات جديدة القائمة على الأجسام المضادة وضع العلامات مثل القياس الهسهي والقياس الخلوي الشامل التصويري لفصل مكان مكاني تصل إلى 12 و 32 المؤشرات الحيوية، على التوالي8،9. التصوير الطيفي الشامل، وهي تقنية لا تتطلب وضع العلامات، لديه القدرة على صورة الآلاف من المؤشرات الحيوية في وقت واحد في قسم نسيج واحد10،11. على الرغم من أن هذه التقنيات قد أظهرت بالفعل إمكانات كبيرة لتشريح المناظر الطبيعية المناعية النسيج في السرطان، فإنها تستخدم معدات وبرامج متطورة للغاية ومكلفة وغير متاحة بسهولة لغالبية الباحثين.
بدلا من ذلك، تم توسيع قدرة تعدد الإمكانات من IHC التقليدية وmIF من خلال استخدام التصوير التسلسلي، وجولات متتابعة من وضع العلامات، والتصوير الطيفي7،12،1313،14،15،16. هذه التقنيات تولد صورا متعددة من نفس أو من أقسام الأنسجة التسلسلية التي يمكن دمجها في الشرائح الظاهرية متعددة المعلمات باستخدام برامج تحليل الصور. ونتيجة لذلك، يزداد عدد العلامات التي يمكن تصورها وتحليلها في وقت واحد.
هنا ، نقترح استراتيجية للتصميم العقلاني لمقالات متعددة الأنسجة باستخدام الكواشف المتاحة تجاريًا ، ومعدات المجهر بأسعار معقولة ، والبرامج سهلة الاستخدام(الشكل 1). تدمج هذه المنهجية التصوير التسلسلي، ووضع العلامات المتسلسلة متعددة، وتصوير الأنسجة بأكملها، ومحاذاة الأنسجة لتوليد شرائح متعددة المعلمات الظاهرية التي يمكن استخدامها للقياس الكمي الآلي ورسم خرائط الخلايا المناعية في أقسام الأنسجة. باستخدام هذه الاستراتيجية، أنشأنا شريحة واحدة افتراضية تتألف من 11 علامة حيوية بالإضافة إلى اثنين من البقع النسيجية المستخدمة بشكل متكرر: الهيماتوكسيلين والإيوسين (H & E) وpicrosirius الأحمر (PSR). تم تحديد مجموعات متعددة من الخلايا المناعية، وتحديد موقعها، وقياسها كميا في مقصورات الأنسجة المختلفة وحل توزيعها المكاني باستخدام خرائط حرارة الأنسجة. هذه الاستراتيجية تعظيم المعلومات التي يمكن الحصول عليها من عينات سريرية محدودة وينطبق على الفورماين ثابتة جزءا لا يتجزأ من عينات الأنسجة المؤرشفة (FFPE) ، بما في ذلك الأنسجة الكاملة ، خزعات إبرة الأساسية ، والخلايا الدقيقة الأنسجة. نقترح هذه المنهجية كدليل مفيد لتصميم المقالات المخصصة لتحديد وتحديد كمية ورسم خرائط مجموعات الخلايا المناعية في TME.
Protocol
تم الحصول على ثلاثة أقسام متسلسلة من فيروس التهاب الكبد B (HBV) المرتبط بسرطان الكبد الخلوي البشري المرتبط به من مستشفى مركز مستشفى جامعة مونتريال (CHUM) قاعدة بيانات سريرية للسرطان الكبدي وعينات بيولوجية مستودع (HBP Biobank). وقدم المرضى المشاركون في هذا البنك النسيجي موافقة مستنيرة. وقد وافقت لجنة الأخلاقيات المؤسسية على هذه الدراسة (البروتوكول رقم 09.237) وأُجريها وفقاً لإعلان هلسنكي.
1. هيماتوكسيلين وإيوسين (H & E) بروتوكول تلطيخ
ملاحظة: تم تنفيذ تلطيخ H & E من قبل المرفق الأساسي لعلم الأمراض الجزيئية التابع لمركز Recherches du Centre hospitalier de l'Université de Montréal (CRCHUM) باستخدام أداة تلطيخ الشريحة الروبوتية متعددة البرامج من شاندون باستخدام البرنامج التالي.
- لdeparaffinization، تزج الشرائح 3x لمدة 2.5 دقيقة لكل منهما في بديل xylene.
تنبيه: بدائل Xylene قابلة للاشتعال، والمهيجات الجلدية، والضارة إذا استنشاقها. - للإماهة، اغمر الشرائح في الإيثانول 3x 100٪ لمدة 2.5 دقيقة لكل منها. يغسل لمدة دقيقة واحدة في الماء المقطر المزدوج (ddH2O) لترطيب.
- احتضان لمدة 1 دقيقة في الهيماتوكسيلين. اغسل 3x لمدة دقيقة واحدة لكل منها في ddH2O.
- احتضان لمدة 5 s مع الإيوسين. اغسل 30 s مع 95% الإيثانول. اغسل 2x لمدة دقيقة واحدة مع الإيثانول بنسبة 100٪.
تنبيه: الإيثانول قابل للاشتعال ومهيج العين. إيوسين هو مهيج العين. - للجفاف، اغمر 3x لمدة 1.5 دقيقة لكل منها في بديل الزيلين. تحميل الشرائح يدويا.
ملاحظة: الوقت المقدر لتنفيذ هذا الجزء من البروتوكول هو 30 دقيقة.
2. متعدد الفلور المناعي تلطيخ بروتوكول لأقسام FFPE
ملاحظة: تم تكييف هذا البروتوكول من روبرتسون وآخرون17.
- إزالة البارافين ة والإماهة
ملاحظة: قبل وضع العلامات بوساطة الأجسام المضادة من أقسام FFPE بواسطة IHC أو mIF، يجب إزالة البارافين. الفشل في إزالة فعالة البارافين النتائج في تلطيخ دون المستوى الأمثل.- ضع 4 ميكرون شرائح قسم أنسجة FFPE في أصحاب شرائح الزجاج. تحت غطاء الدخان، اغمر الشرائح في جرة كوبلين تحتوي على 37 درجة مئوية قبل الدفء xylene لمدة 10 دقيقة.
تنبيه: الزلين قابل للاشتعال، وهو مهيج للبشرة، وضار إذا تم استنشاقه. - تهيّج الشرائح يدويًا مقابل 10 ق كل 2 دقيقة. كرر 1x في xylene الطازج ة لمدة 5 دقيقة أخرى.
- في غطاء محرك السيارة الكيميائي، تزج الشرائح بالتتابع لمدة 5 دقيقة في كل من الحلول التالية: 1) xylene: الإيثانول (1:1 v/v)؛ 2) 100٪ الإيثانول؛ 3) 70٪ الإيثانول؛ 4) 50٪ الإيثانول؛ 5) 30٪ الإيثانول؛ 6) الفوسفات المخزنة مؤقتا المالحة (PBS).
ملاحظة: الاحتفاظ بالشرائح في PBS حتى تكون جاهزة لتنفيذ استرداد المستضد. الحفاظ على أقسام مزيل الشعر رطب في جميع الأوقات. سوف يسبب تجفيف خارج غير محددة الأجسام المضادة ملزمة وبالتالي تلطيخ خلفية عالية.
- ضع 4 ميكرون شرائح قسم أنسجة FFPE في أصحاب شرائح الزجاج. تحت غطاء الدخان، اغمر الشرائح في جرة كوبلين تحتوي على 37 درجة مئوية قبل الدفء xylene لمدة 10 دقيقة.
- استرجاع المستضد الناجم عن الحرارة
ملاحظة: يمكن ملثمين المستضدات عند التثبيت الرسمي، ومنع ربط الأجسام المضادة وبالتالي التصور. استخدام antigen كشف المخازن المؤقتة والإجراءات إعادة جزئيا التوافق الأصلي من epitopes وبالتالي استعادة التعرف على الأجسام المضادة. يجب تحسين نوع عازل استرجاع المستضد والمدة لظروف الفحص المحددة (على سبيل المثال، الهدف، الأجسام المضادة، الأنسجة، إلخ).- تزج الشرائح المشمع في جرة كوبلين التي تحتوي على حل استرجاع مستضد (وصفة في جدول المواد).
- ضع جرة كوبلين المغلقة في طنجرة ضغط كهربائي مع مياه الصنبور. يجب ألا يتجاوز مستوى الماء نصف ارتفاع الجرة بحيث لا يختلط الماء بمحلول استرجاع المستضد.
- أغلق الغطاء وصمام الضغط للطنجرة. حدد الضغط العالي لمدة 10 دقيقة وابدأ. عند الانتهاء من ذلك، افصل الطباخ، والإفراج عن الضغط، وفتح الغطاء، والحفاظ على جرة داخل طنجرة لمدة 30 دقيقة، والسماح للشرائح لتبرد.
- حظر الربط غير المحدد
- نقل الرف مع الشرائح إلى جرة كوبلين مليئة برنامج تلفزيوني. شطف قبالة العازلة استرجاع المستضد مع PBS 2x لمدة 5 دقيقة لكل منهما.
- تطويق أقسام الأنسجة مع قلم PAP لإنشاء حاجز كاره للماء. تزج الشرائح في جرة كوبلين تحتوي على 0.1 M الجليسين في برنامج تلفزيوني. احتضان لمدة 15 دقيقة في درجة حرارة الغرفة (RT).
ملاحظة: الجليسين تشبع مجموعات ألدهيد ولدت أثناء استرجاع المستضد. يمكن لهذه المجموعات ربط الأجسام المضادة الأولية والثانوية بشكل غير محدد. - شطف قبالة محلول الجليسين عن طريق غسل 2x مع برنامج تلفزيوني لمدة 5 دقيقة. ضع الشرائح في غرفة الرطوبة وإضافة ما يكفي من محلول الحجب لتغطية جميع أقسام الأنسجة. تجنب تجاوز الحاجز الكاره للماء. احتضان لمدة 30 دقيقة في RT.
ملاحظة: يمكن العثور على وصفة لحل حظر في جدول المواد. يجب أن يحتوي محلول الحظر على بروتين (على سبيل المثال، BSA) لمنع مواقع الربط غير المحددة. ويمكن أن تتضمن أيضا المنظفات مثل تريتون X-100 أو Tween 20 التي تقلل من التفاعلات الكارهة للماء بين الأجسام المضادة وأهداف الأنسجة، مما يجعل التعرف على المستضد أكثر انتقائية. إضافة 10٪ مجموع المصل من الأنواع حيث يأتي من الأنسجة من شأنه أن يمنع مستقبلات Fc، وبالتالي تقليل غير محددة ربط الأجسام المضادة. وأخيرا، إضافة 10٪ من المصل من الأنواع التي أثيرت فيها الأجسام المضادة الثانوية من شأنها أن تقلل من التعلق المباشر غير المحدد للأجسام المضادة الثانوية إلى قسم الأنسجة.
- وضع العلامات المناعية للفلورات
- شطف مع PBS-Tween (0.1٪ v/v) 2x لمدة 5 دقيقة لكل منهما ووضع الشرائح مرة أخرى في غرفة الرطوبة.
- أضف مزيج الأجسام المضادة الأولية التي أعيد تعليقها في محلول الحظر. احتضان بين عشية وضحاها في 4 °C. يتم سرد الأجسام المضادة الأولية والثانوية المستخدمة في هذه الدراسة في جدول المواد.
ملاحظة: يجب أن يحتوي مزيج الأجسام المضادة الأولية إما على أجسام مضادة أثيرت في أنواع مختلفة، أو من نفس النوع ولكن من أنماط isotypes مختلفة. للحصول على قائمة بأزواج الأجسام المضادة الأولية الثانوية المستخدمة في هذه الدراسة راجع الجدول 2. تفاصيل جميع الأجسام المضادة المستخدمة موجودة في جدول المواد والجدول 2. - شطف مع PBS-Tween (0.1٪ v/v) 3x لمدة 5 دقيقة ووضع الشرائح مرة أخرى في غرفة الرطوبة. في الظلام، أضف مزيج الأجسام المضادة الثانوية واحتضنت لمدة 1 ساعة في RT.
ملاحظة: عندما تكون الأجسام المضادة الأولية من أنواع مختلفة، يجب اختيار الأجسام المضادة الثانوية بحيث يرتبط كل منها فقط بأحد الأجسام المضادة الأساسية وليس ببعضها البعض. ويتحقق ذلك عادة باستخدام الأجسام المضادة الثانوية التي تربى جميعها في نفس الأنواع طالما أن هذا النوع يختلف عن الأنواع التي تم فيها توليد الأجسام المضادة الأولية. في الحالات التي أثيرت فيها الأجسام المضادة الأولية في نفس الأنواع ولكن لها أنواع isotypes مختلفة ، يجب استخدام الأجسام المضادة الثانوية الخاصة بالأيزوtype. - شطف مع برنامج تلفزيوني-Tween (0.1٪ v/v) 3x لمدة 5 دقيقة لكل منهما. شطف مع ddH2O. إزالة السائل الزائد وجبل في تصاعد وسائل الإعلام مع DAPI. يعتمد حجم الصوت المستخدم على حجم المقطع. عادة 40 ميكرولتر يكفي لتغطية سطح شريحة المجهر العادية.
- ضع شريحة الغطاء على القسم واعصر بلطف الوسائط الزائدة المتصاعدة التي تتجنب تشكيل الفقاعة. دع الشرائح تجف لمدة 20 دقيقة في RT في الظلام وتخزينها عند 4 درجات مئوية حتى تصبح جاهزة للاقتناء.
- الحصول على صور لجميع القنوات باستخدام الماسح الضوئي الشريحة بأكمله (انظر جدول المواد).
ملاحظة: تم التحقق من صحة الأجسام المضادة باستخدام أنسجة سرطان الكبد الخلوي البشري كتحكم إيجابي. لكل الأجسام المضادة الأولية، كانت ملطخة ثلاثة أقسام المسلسل إما الأجسام المضادة الأولية، والتحكم isotype، أو منع الحل فقط على التوالي مع أي اختلاف في بقية بروتوكول تلطيخ. تمت مقارنة الصور المكتسبة لتحديد خصوصية تلطيخ. واعتبر تلطيخ محددة عندما إشارة في القسم المحتضن مع الأجسام المضادة الأولية كان النمط المتوقع وكان يمكن تمييزها بسهولة من الخلفية. الأجسام المضادة الأولية إعطاء إشارة خلفية عالية أو وضع العلامات مكونات الأنسجة في isotype وعدم وجود أقسام الأجسام المضادة الأولية تعتبر غير محددة. الوقت المقدر لإكمال هذا الجزء من البروتوكول هو 2 أيام. وتشمل الضوابط المطلوبة: (1) التحكم في النمط الإيزوتايب لتحديد مساهمة الربط غير المحدد للجسم المضاد الأساسي في إشارة الخلفية. يتم تلطيخ أحد الأقسام بنفس الطريقة التي تلطخت بها أنسجة العينة الأخرى باستثناء أنها مُضاَضَلة بجسم مضاد بنفس النمط الأوزوووووووووواني وأصل الأجسام المضادة الأولية ولكنها محددة لهدف غائب في قسم الأنسجة. إذا لم يتوفر الأجسام المضادة المناسبة للتحكم في النمط الإزوتايب ، يمكن استبداله بإجمالي IgG من نفس النوع الذي تم فيه تربية الأجسام المضادة الأولية؛ (2) لا يوجد تحكم أولي في الأجسام المضادة (أي التحكم السلبي) لتحديد خصوصية تلطيخ وتقدير مساهمة الربط غير المحدد للأجسام المضادة الثانوية في إشارة الخلفية. في هذه الحالة ، يتم تلطيخ قسم التحكم بنفس الطريقة مثل الأقسام الأخرى باستثناء عدم إضافة أي جسم مضاد أساسي ؛ (3) السيطرة الإيجابية لإثبات أن يعمل تلطيخ. في هذه الحالة ، يتم إجراء تلطيخ على قسم الأنسجة التي من المعروف أن تعبر عن علامة معترف بها من قبل الأجسام المضادة الأولية.
3. Picro سيريوس الأحمر (PSR) / بروتوكول تلطيخ الأخضر السريع
ملاحظة: الهدف من هذا تلطيخ هو تصور الكولاجين الفيبريلار الأول والثالث في أقسام الأنسجة FFPE. تم تكييف هذا البروتوكول من Segnani وآخرون18. يتم تنفيذ جميع الخطوات في غطاء محرك السيارة الكيميائية.
- إجراء deparaffinization والإماهة من أقسام الأنسجة مماثلة لبروتوكول تلطيخ الفلور المناعي متعددة لأقسام FFPE (القسم 2.1).
ملاحظة: إذا كان المقطع الذي سيتم تلطيخه قد تم استخدامه مسبقًا لوضع علامات الفلور المناعة وتم إزالة البارافين بالفعل، فإن خطوات إزالة الإماهة والإماهة مفيدة لإزالة الوسائط المتصاعدة. DAPI لا تتم إزالة باستخدام هذا الإجراء ولكن لا تتداخل بشكل ملحوظ مع تلطيخ PSR. - تزج الشرائح في جرة تحتوي على الحل الأحمر / الأخضر سريع picro سيريوس (وصفة في جدول المواد)واحتضان لمدة 30 دقيقة في RT (أكثر من 30 دقيقة النتائج في تلطيخ غير محددة من نواة الكبد).
- غسل الشرائح بسرعة في ddH2O (5 الانخفاضات). ثم يغسل بسرعة في الإيثانول 100٪ (5 الانخفاضات). يغسل لمدة 30 s في الزيلين-100% الإيثانول (1:1 v/v). يغسل لمدة 30 s في الزيلين. جبل مع تصاعد وسائل الإعلام (انظر جدول المواد)قبل أن يتبخر تماما xylene (وهذا يساعد مع تصاعد).
ملاحظة: الوقت المقدر لتنفيذ هذا الجزء من البروتوكول هو 1 ساعة.
4. الإلوتي للأجسام المضادة من أقسام الأنسجة
ملاحظة: من أجل إعادة استخدام أقسام الأنسجة في المقالات الوسم متتابعة، مطلوب الإزالة الكاملة للأجسام المضادة الأولية والثانوية. تم تجريد الأجسام المضادة ملزمة كما وصفها سابقا13.
سخني حمامًا مائيًا إلى 56 درجة مئوية. وضع المقاطع داخل جرة تحتوي على تجريد العازلة (وصفة في جدول المواد)،وإغلاق الغطاء، وختم مع شريط الفيلم البارافين لمنع تسرب أثناء الهز.
- وضع جرة داخل حمام الماء واحتضان لمدة 30 دقيقة مع الانفعالات.
- اغسل 4x لمدة 15 دقيقة لكل منها في ddH2O في RT. شطف مع PBS-Tween (0.1٪ v/v).
- الحفاظ على أقسام رطب في برنامج تلفزيوني-Tween أو الماء حتى تكون جاهزة لإعادة التحقيق في القسم مع الجولة الثانية من الأجسام المضادة الأولية.
ملاحظة: الوقت المقدر لتنفيذ هذا الجزء من البروتوكول هو 2 ساعة. - التحقق من كفاءة الإجراء اللاوطي الأجسام المضادة.
ملاحظة: قبل استخدام بروتوكول elution الأجسام المضادة في وصف الوسم متتابعة، ينبغي التحقق من كفاءة إزالة الأجسام المضادة الأولية والثانوية.- إجراء تلطيخ وصورة اقتناء مقطع مع زوج معين من الأجسام المضادة الأولية الثانوية من الفائدة كما هو مبين في بروتوكول تلطيخ الفلور المناعي متعددة لأقسام FFPE (الأقسام 2.1-2.4.6).
- عند الحصول على الصورة، قم بإجراء الأنسجة من الأنسجة المرتبطة بمجمعات الأجسام المضادة الأولية الثانوية كما هو مبين في الأقسام 4.1-4.3.
- احتضان القسم مع نفس الأجسام المضادة الثانوية ونفس الشروط المستخدمة في الخطوة 2.4.3.
- تنفيذ خطوات الغسيل والتركيب واقتناء الصور كما هو مبين في 2.4.4-2.4.6.
- قارن الصور جنبًا إلى جنب التي تم الحصول عليها قبل وبعد التجريد من أجل تحديد ما إذا كانت الإشارة المحددة قد اختفت أم لا.
ملاحظة: سوف مقارنة الصور قبل وبعد إزالة الأجسام المضادة التحقق من كفاءة الإجراء elution. ومع ذلك ، فمن الطبيعي أن نرى زيادة في إشارة الخلفية في جميع القنوات ، فضلا عن نشر DAPI. وهذا يحد من عدد جولات من تجريد التي يمكن تنفيذها على نفس القسم الأنسجة. ثلاث جولات من التجريد يبدو أن الحد الأقصى.
5- الحصول على الصور
- إنشاء صور باستخدام ماسح ضوئي شرائح كامل.
- استخدم عدسة موضوعية 20x 0.75NA ودقة 0.3225 ميكرومتر/بكسل.
6. تحليل الصور
ملاحظة: الأسلوب الموضح هنا يشير إلى المثال الحالي. يرجى الرجوع إلى الجدول 1 والنص للتكيف مع عينات محددة أخرى.
- تنفيذ محاذاة الأنسجة باستخدام وحدة Tissualign من برنامج تحليل الصور (VIS في هذا البروتوكول ، انظر جدول المواد).
- افتح برنامج تحليل الصور وانقر على علامة التبويب منديل.
- استيراد الصور التي سيتم محاذاتها في علبة الشرائح عن طريق الانتقال إلى ملف | قاعدة البيانات وتحديد الصورة الأولى التي سيتم محاذاتها. العودة إلى علامة التبويب المنديل وتحميل الصورة عن طريق النقر على زر التحميل في علبة الشرائح. ستظهر الصورة في علبة الشرائح وفي مساحة العمل.
ملاحظة: يجب تحميل مكدس الاهتمامات فقط في علبة الشرائح. - كرر الخطوة 6.1.2 لجميع الصور في ترتيب لتكون محاذاة، تحميلها واحدا تلو الآخر. بمجرد تحميل جميع الصور ذات الأهمية على درج الشريحة قم بالمتابعة لربط الصور عن طريق الضغط التالي في خطوات سير العمل في الشريط.
- بعد ذلك، اسحب الصورة الثانية وأسقطها أعلى الصورة الأولى. يتم الآن ربط الصور الأولى والثانية. كرر هذه الخطوة لمحاذاة الصور الأخرى، واحدة تلو الأخرى، بطريقة منظمة. سيتغير اسم الصورة الأولى، مما يشير إلى أنه تم ربطها بالصور الأخرى. في نفس الوقت، سيتم عرض الصور المرتبطة في مساحة العمل على يمين علبة الشرائح.
- عند هذه النقطة، قم بمحاذاة الصور إما باستخدام المحاذاة التلقائية أو المحاذاة نصف التلقائية أو المحاذاة اليدوية. من الأفضل دائمًا تجربة المحاذاة التلقائية أولاً. للمحاذاة التلقائية اضغط على الزر التالي في خطوات سير العمل (الخطوة 3) في الشريط.
- مراجعة المحاذاة التلقائية عن طريق التنقل في مواقع مختلفة من الأنسجة والتحقق بصريا من أن يتم ترتيب الهياكل المقابلة في صور مختلفة بنفس الطريقة في بُعدي الصورة.
- إذا كانت نتيجة المحاذاة التلقائية غير مرضية، قم بتحسينها باستخدام دبابيس (استخدم ما لا يقل عن ثلاثة دبابيس لكل صورة) مما يشير إلى ميزات الأنسجة المتجانسة في الصور المرتبطة. بمجرد وضع الدبابيس في مواقع متجانسة في الصور المرتبطة ، يكون لدى المستخدم خياران: المحاذاة نصف التلقائية أو المحاذاة اليدوية. للمحاذاة نصف التلقائي انقر على زر محاذاة تلقائية استناداً إلى الأبعاد الحالية في الشريط. للمحاذاة اليدوية، انقر فوق الزر تطبيق دبابيس على الشريط.
- عندما راض عن المحاذاة انقر على الزر التالي في خطوات سير العمل وحفظ الصورة المركبة في قاعدة البيانات.
ملاحظة: استغرق محاذاة ست شرائح تمتد 11 علامة بالإضافة إلى صور H & E و PSR 15 دقيقة في التحليل المقدم.
- إجراء الكشف عن الأنسجة باستخدام حزمة بروتوكول تحليل بروتوكول المعرفة من قبل المستخدم 1 (APP 1، الجدول 1).
- افتح وحدة تحليل الصور للبرنامج بالنقر فوق علامة التبويب تحليل الصور في الشريط.
- استيراد الصورة المركبة (المحاذاة) عن طريق الانتقال إلى ملف | قاعدة البيانات وتحديد صورة الاهتمام والنقر مرة أخرى علامة التبويب تحليل الصور.
- افتح مربع حوار تحديد APP بالنقر على رمز APP المفتوح وحدد حزمة بروتوكول التحليل (APP) التي سيتم استخدامها. في هذه الحالة حدد APP 1 للكشف عن الأنسجة.
- بمجرد فتح APP 1 ، تأكد من أن APP1 يعمل بشكل صحيح من خلال الانتقال إلى موقع نسيج محدد والنقر على زر المعاينة. إذا كانت النتائج مرضية، انتقل إلى الخطوة التالية.
- انقر لتشغيل APP 1 ومعالجة الصورة باستخدام APP المحدد.
- تصدير البيانات (على سبيل المثال، الصور والقياسات، وما إلى ذلك) عندما يتم التحليل عن طريق النقر فوق الملف / التصدير.
ملاحظة: يقوم APP 1 بإنشاء منطقة ذات أهمية (ROI) تحدد النسيج (أنسجة العائد على الاستثمار) ويحسب مساحة الأنسجة. - حفظ الصورة المعدلة مع العائد على الاستثمار التي تم إنشاؤها حديثا عن طريق الذهاب إلى ملف | حفظ.
ملاحظة: استغرق الكشف عن الأنسجة وإنشاء عائد الاستثمار مع APP 1 في المثال المقدم 5 دقيقة في محطة تحليل الصور الموصوفة. وكانت مساحة الأنسجة المعالجة 3.2 سم2.
- إجراء تجزئة الأنسجة في ستروما وParenchyma باستخدام APP 2(الجدول 1).
ملاحظة: يعمل APP 2 على أنسجة ROI المحددة مسبقًا. APP 2 شرائح الأنسجة في روما روما وParenchyma.- افتح وحدة تحليل الصور بالنقر فوق علامة التبويب تحليل الصور في الشريط.
- استيراد الصورة التي تحتوي على أنسجة عائد الاستثمار عن طريق الذهاب إلى ملف | قاعدة البيانات وتحديد الصورة المحفوظة في الخطوة 6.2.7. العودة إلى علامة التبويب تحليل الصور وتحميل الصورة عن طريق النقر على زر التحميل في علبة الشرائح. ستظهر الصورة في علبة الشرائح وفي مساحة العمل.
- افتح APP 2 باستخدام مربع حوار تحديد APP كما هو الحال في 6.2.3.
- معاينة APP 2 عن طريق المعالجة في حقل عرض محدد. إذا كانت النتائج مرضية، قم بتشغيل APP 2 على الصورة الكاملة بالنقر على زر التشغيل. كما إخراج APP 2، يتم تقسيم الأنسجة العائد على الاستثمار في روما رهويس وParenchyma والمناطق الخاصة بهم تحديد. نتائج التصدير كما في 6.2.6. حفظ الصورة المعدلة كما هو الحال في 6.2.7.
ملاحظة: استغرق تقسيم الأنسجة في ستروما وParenchyma باستخدام APP 2 4 ساعة في محطة التحليل المقدمة. وكانت مساحة الأنسجة المعالجة 3.2 سم2.
- تحديد وقياس FoxP3مرحباCD4 + الخلايا باستخدام البروتوكول المعرفة من قبل المستخدم APP 3(الجدول 1).
ملاحظة: يعمل APP 3 على ROIs Stroma وParenchyma المحددين مسبقًا.- افتح وحدة تحليل الصور وتستورد الصورة التي تحتوي على ROIs Stroma و Parenchyma كما هو الحال في 6.3.1 و 6.3.2. افتح APP 3 باستخدام مربع حوار تحديد APP كما هو الحال في 6.2.3.
- معاينة APP 3 معالجة في مجال محدد من الرؤية المخصب في FoxP3مرحباCD4 + الخلايا. إذا كانت النتائج مرضية، قم بتشغيل APP 3 على الصورة الكاملة. كما الإخراج من APP 3، سيتم تسمية جميع الكائنات FoxP3مرحباCD4+ الفردية وإحداثيات الأنسجة الخاصة بهم المخزنة. سيتم تحديد كثافات من الكائنات FoxP3مرحباCD4 + في ROIs ستروما وParenchyma. تصدير النتائج كما في 6.2.6.
- تنفيذ رسم خرائط حرارة الأنسجة من FoxP3مرحباCD4 + الكائنات المسماة.
- افتح البروتوكول المعرفة من قبل المستخدم FoxP3مرحباCD4 + MAP باستخدام حوار تحديد APP كما هو الحال في 6.2.3.
ملاحظة: FoxP3مرحباCD4 + MAP يستخدم إحداثيات FoxP3مرحباCD4 + الكائنات الموسومة لتوليد خرائط الحرارة كثافة. استغرق تحديد والعد FoxP3مرحباCD4 + الكائنات المسماة باستخدام APP 3 25 دقيقة في محطة تحليل الصور الموصوفة. وكانت مساحة الأنسجة المعالجة 3.2 سم2. - تشغيل FoxP3مرحباCD4 + MAP عن طريق الضغط على زر تشغيل. تصدير خريطة حرارة الأنسجة عن طريق النقر على ملف | التصدير | منطقة العمل.
ملاحظة: استغرق رسم خرائط FoxP3مرحباCD4 + الكائنات المسماة باستخدام FoxP3مرحباCD4 + MAP 5 دقيقة في محطة تحليل الصور الموصوفة.
- افتح البروتوكول المعرفة من قبل المستخدم FoxP3مرحباCD4 + MAP باستخدام حوار تحديد APP كما هو الحال في 6.2.3.
- تحديد وقياس CD8+، CD68+، MPO+، αSMA، و CD34 + الكائنات باستخدام البروتوكولات المعرفة من قبل المستخدم APP 4 و APP5 و APP6 و APP7 و APP 8، على التوالي(الجدول 1)كما هو الحال في القسم 6.4 إلى 6.4.3.2 تحميل APP من الفائدة في كل حالة.
ملاحظة: APPs 4 إلى 8 العمل على ROIs ستروما محددة مسبقا وParenchyma.
Representative Results
نظرة عامة على استراتيجية تصور مجموعات الخلايا ذات الأهمية في TME وقياسها كمياً ورسم خرائط لها
لتحديد مجموعات الخلايا ذات الأهمية (COIs) في مقصورات الأنسجة المختلفة (TCs) وتوصيف تنظيمها المكاني ، قمنا بتصميم سير العمل الذي يدمج تقنيات بأسعار معقولة وسهلة الاستخدام ويزيد من المعلومات الموضعية التي يمكن الحصول عليها من العينات السريرية الثمينة FFPE(الشكل 1). أولاً، كانت أقسام الأنسجة التسلسلية الكاملة في الأنسجة ملطّفة لتصور COIs (على سبيل المثال، الخلايا المناعية) وTCs (على سبيل المثال، ستروما مقابل parenchyma)(الشكل 1،الخطوة 1). يجب إبقاء عدد الأقسام المتتالية التي سيتم تلطيخها إلى الحد الأدنى الذي يسمح بتصور الخلايا ذات الأهمية أو ميزات الأنسجة اللازمة لمعالجة سؤال البحث. كلما كان عدد المقاطع التسلسلية أصغر ، كلما ارتفع تشابه بنية الأنسجة والتوافق عبر المقاطع المتجاورة. وبالإضافة إلى ذلك، يمكن توسيع قدرة تعدد الإرسال من خلال إعادة استخدام المقاطع الملطخة الفلورسنت من خلال تقنيات التجريد وإعادة التحقيق19.
بمجرد الانتهاء من خطوات تلطيخ، تم استخدام ماسح ضوئي كامل للشرائح لرقمنة الصور. تم محاذاة الصور التي تم الحصول عليها من المقاطع التسلسلية ودمجها في شريحة متعددة افتراضية بطريقة تلقائية(الشكل 1، القسم 2). بعد ذلك، تم تحديد عائد الاستثمار للأنسجة ببروتوكول معرف من قبل المستخدم يحدد البيكسلات المرتبطة بالأنسجة (TAPs)(الشكل 1، الخطوة 3). وفي وقت لاحق، تم تقسيم أنسجة عائد الاستثمار إلى TCs التي تم تعريفها على أنها ROIs إضافية. (الشكل1،الخطوة 4). بعد ذلك، تم الكشف عن بروتوكولات المعرفة من قبل المستخدم وقياسها كميًا في مراكز اتصال مختلفة(الشكل 1،الخطوة 5). وأخيراً، تم إنشاء خرائط حرارة الأنسجة من COIs على أساس كثافاتها وإحداثيات الأنسجة(الشكل 1، الخطوة 6).
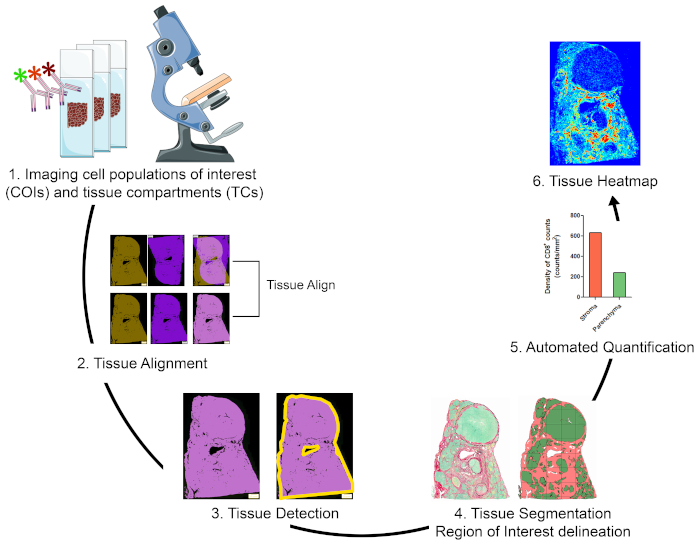
الشكل 1: التمثيل التخطيطي لاستراتيجية تصور الخلايا المناعية وقياسها كمياً ورسم خرائطلها في الـ TME. (1)تم تلطيخ أقسام الأنسجة الكاملة التسلسلية لتسمية COIs و TCs. تم رقمنة أقسام الأنسجة الكاملة الملطخة باستخدام ماسح ضوئي كامل للشرائح. (2)تم ربط الصور التي تم الحصول عليها من الأقسام التسلسلية، ومحاذاتها، والمشاركة في التسجيل بطريقة آلية باستخدام وحدة تحليل الأنسجة. تم إنشاء صورة مركبة من محاذاة عالية الدقة للصور الفردية. (3)تم استخدام بروتوكول معرف من قبل المستخدم للكشف الآلي عن البيكسلات المرتبطة بالأنسجة (TAPs) في الصورة المركبة. (4)تم تقسيم الأنسجة إلى TCs (على سبيل المثال، ستروما وparenchyma) تعرف باسم ROIs. (5)تم استخدام بروتوكولات المعرفة من قبل المستخدم للكشف الآلي وقياس كموية COIs في مختلف TCs.(6)تم إنشاء خرائط حرارة الأنسجة من COIs. يرجى الضغط هنا لعرض نسخة أكبر من هذا الرقم.
COIs التصوير وTCs
ثلاثة أقسام الأنسجة الكاملة FFPE المسلسل من الورم المنقسم من موضوع مع سرطان الكبد المرتبطة بفيروس التهاب الكبد BBV كانت ملطخة في واحدة أو أكثر من جولات تلطيخ كما هو الحال في الشكل 2A. القسم الأول كان ملطخا مع H & E لإظهار بنية الأنسجة، مورفولوجيا الخلايا، وتحديد المعلمات ذات الصلة سريريا مثل نوع من الأورام الخبيثة، ودرجة الورم، والتقييم العام للتسلل المناعي(الشكل 2C). في القسم الثاني المتجاورة ، تم استخدام جولتين من mIF لتسمية خلايا الكبد parenchymal وغير parenchymal(الشكل 2A). في الجولة الأولى ، تم تصور الأوعية الطبيعية والورم باستخدام تلطيخ CD34 للخلايا الانهوفية. بالإضافة إلى ذلك ، تم تحديد الخلايا الظهارية (خلايا الكبد والخلايا الكلانجيوية) باستخدام السيتوكيراتين 8/18 ، وتم تحديد خلايا ستيلات الكبد المنشطة الليفية كخلايا ألفا العضلات الملساء الإيجابية (αSMA +) الخلايا(الشكل 2C). بعد الحصول على الصورة، تم تجريد أقسام الأنسجة وإعادة التحقيق مع الأجسام المضادة ضد الضامة (CD68)، وmyofibroblasts (desmin). لتوصيف أفضل تسلل المناعة الورم، وملطخة القسم التسلسلي المجاورة الثالث باستخدام جولتين من mIF للعلامات الخلوية CD3، CD4، CD8، مربع رأس الشوكة P3 (FoxP3)، وmyeloperoxidase (MPO). في جميع الحالات تم استخدام DAPI كبقع مضادة نووية. وأخيرا، كان ملطخا القسم الثالث مع وصمة عار PSR ومضادة مع الأخضر السريع لتصور الكولاجين الفيبريلار وتقسيم الأنسجة إلى ستروما وparenchyma(الشكل 2C).
تم استخدام ماسح شرائح كامل مجهز بعدسة موضوعية 20X لرقمنة المقاطع الملطخة وإنشاء شرائح افتراضية. تم الحصول على ست صور من الأقسام التسلسلية الثلاثة(الشكل 2B)والشرائح الافتراضية التي تم تحليلها لاحقًا باستخدام برنامج VIS وفقًا للتمثيل التخطيطي في الشكل 1.
تحليل الصور
وشمل تحليل الصورة خمس خطوات: 1) محاذاة الأنسجة؛ 1) محاذاة الأنسجة؛ 1) محاذاة الأنسجة؛ 1) محاذاة الأنسجة؛ 1 2) الكشف عن الأنسجة؛ 3) تجزئة الأنسجة؛ 4) القياس الكمي الآلي لثاني أكسيد الكربون؛ و 5) رسم خرائط حرارة الأنسجة. تم تطوير جميع بروتوكولات تحليل الصور باستخدام وحدة المؤلف لبرنامج تحليل الصور ويشار إليها في النص باسم APP.
محاذاة الأنسجة
تم تحميل ست شرائح افتراضية من ثلاثة أقسام تسلسلية ، تغطي 11 علامة بالإضافة إلى بقع H & E و PSR ، في وحدة Tissualign من برنامج تحليل الصور. بعد ذلك ، تم ربط الصور ومحاذاتها وتسجيلها بشكل تلقائي ، مما أدى إلى إنشاء صورة مركبة افتراضية من 11 plex بالإضافة إلى H & E و PSR ، تحتوي على جميع طبقات الصور الفردية(الأشكال 2A-C). كانت المحاذاة دقيقة في حالة الصور الناشئة من الأقسام التسلسلية المجاورة ، والتي تظهر هياكل الأنسجة المقابلة المتمركزة ومرتبة بطريقة متجانسة عند المحاذاة(الشكل 2C والشكل S1A). وعلاوة على ذلك، كانت المحاذاة دقيقة على مستوى الخلية الفردية للصور الناشئة من نفس القسم(الشكل S1B). يعتمد وقت المحاذاة التلقائية على عدد الصور التي سيتم محاذاتها وحجمها وتعقيدها وتشابهها. استغرق محاذاة الشرائح الافتراضية الست المذكورة أعلاه 15 دقيقة في محطة VIS الخاصة بنا.

الشكل 2: تلطيخ أقسام الأنسجة التسلسلية ومحاذاة الصورة. (أ)ملخص البقع القيام به على ثلاثة أقسام المسلسل لتصور COIs و TCs. الأرقام بين قوسين تشير إلى تسمية الصورة. بالنسبة للقسمين الثاني والثالث، تم تجريد الأنسجة وإعادة فحصها مع كوكتيل ثان ٍ من الأجسام المضادة. (ب)نظرة عامة على ست صور فردية للأنسجة الكاملة قبل وبعد محاذاة الأنسجة (يسار ويمين، على التوالي). شريط مقياس = 3500 ميكرومتر.(C)عرض التكبير من الصور المحاذاة. شريط مقياس = 80 ميكرون. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الكشف عن الأنسجة
بمجرد ربط الصور ومحاذاتها ، سعينا إلى تحديد TAPs(الشكل 3A). لتصميم APP للكشف الآلي من TAPs (APP 1، الجدول 1)،استفدنا من اثنين من الخصائص التي تميز TAPs من بكسل غير المرتبطة الأنسجة. أولاً، تقتصر إشارة DAPI (النطاق الأزرق) على النوى، التي تقع حصريًا في الأنسجة، مما يعني أن جميع وحدات بكسل DAPI + هي مجموعة فرعية من TAPs. ثانياً، لدى TAPs إشارة أعلى للفلور الفلورفية في النطاقات الخضراء والصفراء مقارنة بالبيكسلات غير المرتبطة بالأنسجة. وبالتالي، قمنا بتطوير APP 1 للكشف عن الأنسجة(الجدول 1)،الذي يكتشف TAPs على أساس إشارة خط الأساس في هذه القنوات باستخدام تقنيات العتبات البسيطة. تم تعيين عتبات النطاقات الزرقاء والخضراء والصفراء بحيث يكون TAPs قيم كثافة الخلفية فوق العتبات ، في حين أن وحدات البكسل غير المرتبطة بالأنسجة لها قيم أدناه. تم تطبيق APP 1 للكشف عن الأنسجة على IIA الصورة، التي تحتوي على طبقات في القنوات الزرقاء والخضراء والصفراء(الشكل 3A). كما تم وضع مخرجات APP 1 ، قناع أخضر مشرق على رأس TAPs ، وتم تحديد عائد الاستثمار يسمى "الأنسجة" (الإخراج ، الشكل 3A). وعلاوة على ذلك، تم تحديد منطقة الأنسجة كمتغير الناتج الكمي. لأن APP 1 لا يتضمن وحدات البكسل غير المرتبطة بالأنسجة في أنسجة عائد الاستثمار ، فقد تم استبعادها من التحليل اللاحق استنادًا إلى عائد الاستثمار هذا(الشكل 3A). وترد دقة APP 1 في تحديد TAPs في الشكل 3A.
تجزئة الأنسجة وترسيم ROIs للمراكز الاستراتيجية
بعد ذلك ، شرعنا في تحديد مقصورات مختلفة داخل أنسجة عائد الاستثمار عن طريق تقسيم الأنسجة إلى ستروما مقابل parenchyma. استخدمنا صورة PSR الملطخة (IIIC، الشكل 2C)،حيث يمكن تعريف ستروما كالمنطقة المرتبطة بترسب الكولاجين الفيبريلار (النطاق الأحمر)، وparenchyma كمنطقة حيث الكولاجين الفيبريلار غائبة، وصبغة مضادة خضراء سريعة يسود (الشريط الأخضر)(الشكل 3B). أنشأنا APP 2(الجدول 1)لتحديد رقميا TCs ستروما وParenchyma. يعمل هذا التطبيق على أنسجة ROI المحددة مسبقًا (الإخراج ، الشكل 3A)ويستخدم مناطق ستروما وparenchyma التمثيلية لتدريب أداة المصنف المدمجة في وحدة تحليل الصور. المصنف المدرب ة يعيّن البيكسلات إلى إما ستروما أو علامة parenchyma (السلمون والأخضر، على التوالي، الشكل 3B). عند تصنيف وحدات البكسل، نفذ APP 2 عمليات مورفولوجية تهدف إلى تعريف ROIs Stroma و Parenchyma(الشكل 3B والجدول 1). يظهر أداء APP 2 في تصنيف وحدات البكسل وتوليد ROIs ذات الصلة في الشكل 3B. بالإضافة إلى ذلك، APP 2 يحدد مساحة ستروما وparenchyma. وأخيراً، على الرغم من أن التقسيم يتم باستخدام قسم PSR الملون، يمكن نقل مناطق ستروما وparenchyma الموضحة إلى أي صورة تتماشى مع صورة PSR.

الشكل 3: الكشف الآلي عن الأنسجة/تجزئتها وتوليد الأقراص ذات الصلة. (أ)تم استخدام صورة IIA لتحديد TAPs (الصورة اليسرى، شريط المقياس = 6000 ميكرومتر). تم تعيين قناع أخضر مشرق إلى TAPs باستخدام APP 1(الجدول 1)توليد عائد استثمار يسمى الأنسجة (الإخراج 1). اليمين، يعرض inset طريقة عرض مكبرة مما يدل على دقة APP 1 في الكشف عن TAPs. شريط مقياس = 350 ميكرون(ب)يتم تقسيم أنسجة العائد على الاستثمار (الإخراج 1) إلى ستروما وparenchyma باستخدام APP 2. تظهر الصورة على اليسار منظرًا لأنسجة عائد الاستثمار المجزأة إلى ستروما عائد الاستثمار (السلمون) وعائد الاستثمار parenchyma (أخضر). شريط المقياس = 4500 ميكرومتر. على اليمين ، تم تكبير طرق عرض inset لأنسجة ROI ، وتلطيخ PSR الأصلي (صورة IIIC) ، وROIs stroma وparenchyma. شريط المقياس = 250 ميكرومتر. يرجى الضغط هنا لعرض نسخة أكبر من هذا الرقم.
القياس الكمي الآلي لثاني أكسيد الكربون
بعد ذلك ، شرعنا في تحديد وتحديد وتحديد وقياس COIs في ROIs Stroma و Parenchyma. تم إنشاء APPs من 3 إلى 8(الجدول 1)لتحديد موقع وإحصاء COIs التالية: CD4+ FoxP3+، CD8+، CD68+، MPO+، αSMA+، وخلايا CD34+ ، على التوالي. تم تصميم APP 3 لتحديد موقع وإحصاء خلايا CD4+ FoxP3 +(صورة IIIA، الشكل 2C)كعلامات بديلة للخلايا T التنظيمية (Tregs). يكتشف هذا البروتوكول التوطين المشترك للإشارة من عامل النسخ النووي FoxP3 (النطاق الأحمر) وصبغة الحمض النووي DAPI (النطاق الأزرق). وبالنظر إلى أن تنشيط مؤخرا الخلايا T upregulate FoxP3 ، لإثراء لTregs وضعنا عتبات لاختيار مسبق فقط خلايا FoxP3 + مشرق (FoxP3مرحبا). بعد ذلك ، من بين جميع الخلايا DAPI + FoxP3المحددة مسبقًا ، تم وضع علامة على خلايا DAPI + FoxP3 المحددة مسبقًا ، فقط تلك التي كانت محاطة بإشارات CD4 على شكل حلقة ساطعة (النطاق الأخضر) وتحسب باسم FoxP3hiCD4 + الخلايا (التسمية الوردية ، الشكل 4A). تم تحديد كثافة FoxP3مرحباCD4 + الخلايا في روما ستروما وParenchyma كمية النواتج متغيرات APP 3(الشكل 4A).
وبالمثل، تم تصميم APPs 4-6 للكشف عن خلايا CD8+، CD68+، وMPO+ الخلايا. وتشترك هذه الAPPs في نفس التصميم الأساسي للكشف عن ثاني أكسيد الكربون وقياسه كمياً. على وجه التحديد، يتم تحديد COIs استنادًا إلى شدة الإشارة من المؤشرات الحيوية لأعداد الخلايا المحددة، ثم يتم تنفيذ العديد من الخطوات المورفولوجية بعد المعالجة لترسيم الخلايا الفردية(الجدول 1). يتم وضع علامات على الخلايا الفردية أو COIs وحسابها وتسجيل إحداثيات الأنسجة الخاصة بها. APPs 4 إلى 6 أيضا تحديد كثافة COIs في روما روا سروما وParenchyma(الشكل 4B-D).
لم تكن جودة تلطيخ DAPI لدينا جيدة بما يكفي لدمج تجزئة النوى في APPs 3 إلى 6 ، لذلك لا يمكننا التأكد من أن جميع الكائنات المسماة بشكل فردي هي خلايا فردية. لهذا السبب، أعربنا عن كثافة الخلايا في أعداد الكائنات المسماة / مم2 (الشكل 4). ومع ذلك، تم فصل مجاميع الخلايا بنجاح إلى خلايا فردية في خطوات ما بعد المعالجة المضمنة في APPs 3 إلى 6، وأظهر الفحص البصري المكثف أن معظم الكائنات المسماة تتوافق مع خلايا مفردة.
للكشف عن مساحة αSMA+ و CD34+ ، قمنا بتطوير APPs 7 و 8 على التوالي(الجدول 1). كلا APPs الكشف عن إشارة محددة على أساس عتبات وتحديد النسبة المئوية للمنطقة الإيجابية في روما روا وParenchyma(الشكل 4E -F).
واحدة من الاحتمالات الأكثر إثارة للاهتمام لتوليد الشرائح متعددة الظاهري هو تحليل التعبير colocalization. قمنا بإنشاء APP 10 للكشف عن التوطين المشترك بين αSMA وdesmin ، وهما علامات شارك في التعبير عنها الخلايا العضلية في الكبد. يستخدم APP 10 عتبات للعثور على وحدات بكسل إيجابية لαSMA وdesmin و αSMA بالإضافة إلى desmin(الجدول 1). كمتغيرات الناتج الكمي، يحدد APP 10 منطقة αSMA+ ومنطقة desmin+ ومنطقة التعبير المشترك لهذين المؤشرين(الشكل S3).

الشكل 4: تحديد وقياس كموية الكواب في الـ TCs stroma وparenchyma. (أ-واو) الكشف الآلي والتقدير الكمي لـ CD4+FoxP3+، CD8+، CD68+، MPO+، αSMA+، و CD34+ COIs في ROIs Stroma و Parenchyma باستخدام البروتوكولات 3 و 4 و 5 و 6 و 7 و 8 على التوالي(الجدول 1). تظهر على اليسار هي الصور الأصلية ، في منتصف الصور المعالجة ، وعلى اليمين التحديدالكمي. للأرقام 4A-D،شريط مقياس = 40 ميكرومتر. للحصول على الشكلين 4E و F، شريط مقياس = 350 ميكرومتر. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
كبديل لقياس COIs في الـ TCs Stroma و Parenchyma ، حددنا كثافة الخلايا المناعية في العقيدات الخبيثة المختلفة المسماة من 1 إلى 4(الشكل 5A، H، وأنا). تم تحديد عائد الاستثمار لكل عقيدات يدويًا كما هو مبين في الشكل 5A. تميزت التوقيعات المناعية المميزة للأنسجة بكل عقيدات ، مما يكشف عن التغاير الجوهري للTME.
خرائط حرارة الأنسجة
كما ذكر أعلاه، APPs 3 إلى 8 تخزين إحداثيات الأنسجة من كل كائن وصفت على حدة. تسمح هذه الميزة بتوليد خرائط الأنسجة آليًا حيث يتم عرض مناطق ذات كثافة عالية من مجموعة خلايا معينة كنقاط ساخنة (حمراء) ، ومناطق ذات كثافة منخفضة نسبيًا كبقع باردة (أزرق داكن). يتم تعيين قيم الكثافة المتوسطة بألوان وفقًا لمقياس اللون المبين في الشكل 5. تم إنشاء خرائط حرارة الأنسجة بواسطة APPs التي قسمت الصور إلى دوائر قطرها 50 ميكرومتر وتعيين لون وفقًا للكثافة النسبية لثاني أكسيد الكربون المعطى داخل الدائرة. كما هو معروض في الشكل 5B-G، كانت أنماط تحديد المواقع وتوزيع الكثافة لمختلف ثاني أكسيد الكربون في TME متنوعة تمامًا. وعلاوة على ذلك، على مستوى العقيدات الفردية، كان ترتيب مختلف التجمعات السكانية في منطقة الأنسجة فريداً(الشكل S2A-C). ولتقديم مثال على قوة هذه التقنية وتصور التنظيم المكاني للنقاط الساخنة من مجموعات سكانية مختلفة في نفس العقيدات، تم استخراج النقاط الساخنة من أنواع الخلايا الفردية يدويًا وتعيينها معًا على المخطط التفصيلي للعقيدات 2(الشكل S2والشكل Dوالشكل E).

الشكل 5: خرائط حرارة الأنسجة لثاني أكسيد الكربون في TME. (أ)بيكروسيوسيو الأحمر تلطيخ تبين موقع العقيدات 1 و 2 و 3 و 4. (ب-ز) خرائط حرارة الأنسجة لـ CD4+ FoxP3+، CD8+، CD68+، MPO+، CD34+، وαSMA+ COIs، على التوالي. يشير اللون الأزرق الداكن إلى كثافة منخفضة نسبية، ويشير اللون الأحمر إلى الكثافة العالية النسبية. يتم تعيين قيم الكثافة المتوسطة بألوان وفقًا لمقياس اللون المعروض. (H و I)القياس الكمي لثاني أكسيد الكربون في العقيدات 1 و 2 و 3 + 4 منظمة لكل نوع خلية وكل عقيدة، على التوالي. يرجى الضغط هنا لعرض نسخة أكبر من هذا الرقم.

الرقم التكميلي S1: التحقق من محاذاة الأنسجة. (أ)يستخدم تلطيخ CD34 (باللون الأحمر) على القسم الثاني (الإدخال 1) لتوليد قناع CD34 باللون الأخضر (الإخراج 1). يتم تراكب القناع الأخضر (الإخراج 1) على صورة H & E من القسم التسلسلي المنحاز I (الإدخال 2). تظهر صورة الدمج مراسلات مثالية لهياكل الأوعية الدموية. شريط المقياس = 50 ميكرومتر. (ب)تم استخدام صورة IIIA التي تعرض دمج DAPI و CD4 و FoxP3 (الإدخال 1) لإنشاء تسمية لخلايا CD4+ FoxP3 +(الإخراج 1 باللون الأرجواني). تم نقل ملصق الإخراج 1 إلى صورة متوافقة IIIB (الإدخال 2) ويظهر مراسلات مثالية بين أزواج FoxP3/DAPI، و CD4/CD3 في صورة الدمج. شريط المقياس = 15 ميكرومتر. يرجى الضغط هنا لعرض نسخة أكبر من هذا الرقم.

الرقم التكميلي S2: عرض مكبر لخرائط حرارة الأنسجة. (أ-ج) خرائط حرارة الأنسجة لخلايا CD4+FoxP3+، CD8+، CD68+، وخلايا MPO+ في العقيدات 1-4. وتمثل أشرطة المقياس في العقيدات 1 و2 و3 + 4 500 1 ميكرومتر و700 ميكرومتر و500 ميكرومتر على التوالي. (D)مخطط العقيدات 2 مع خط صلب أسود. (E)تم استخراج النقاط الساخنة لـ CD4+FoxP3+، CD8+، CD68+، وخلايا MPO+ في العقيدات 2 وتعيينها معًا على مخطط العقيدات 2 المحدد في D. يرجى الضغط هنا لعرض نسخة أكبر من هذا الرقم.

الرقم التكميلي S3: تحليل التعريب المشترك. (أ)على اليسار والوسط هي صور تسمية αSMA في تسمية خضراء وdesmin باللون الأحمر على التوالي. على اليمين هو αSMA / desmin منطقة إيجابية مزدوجة باللون الأصفر. (ب)القياس الكمي لمنطقة αSMA + ، desmin + المنطقة ، وαSMA / desmin منطقة إيجابية مزدوجة. شريط المقياس = 150 ميكرومتر. يرجى الضغط هنا لعرض نسخة أكبر من هذا الرقم.
| التطبيق | الغرض | تصنيف | تصنيف | خطوات ما بعد المعالجة | متغيرات الإخراج |
| الاسلوب | ميزات | ||||
| (قيمة البكسل) | |||||
| 1 | الكشف عن الأنسجة | عتبه | قناة DAPI (150) | o تسمية الكائنات ذات القيم المترجمة فوق العتبة للقنوات 3 | س الأنسجة العائد على الاستثمار |
| قناة FITC/A488 (120) | س إغلاق الكائن الإيجابي 5 بكسل | س منطقة الأنسجة | |||
| قناة TRITC/A568 (40) | س إنشاء أنسجة العائد على الاستثمار | ||||
| 2 | تجزئة الأنسجة | قرار الغابات | RGB-R المتوسط | س ملء الثقوب | o عائد الاستثمار ستروما |
| RGB-G المتوسط | س إنشاء عائد الاستثمار ستروما | س منطقة ستروما | |||
| متوسط RGB-B | س إنشاء عائد الاستثمار Parenchyma | o عائد الاستثمار Parenchyma | |||
| IHS-S المتوسط | س منطقة Parenchyma | ||||
| H & E إيوسين المتوسط | |||||
| 3 | لتحديد موقع وقياس خلايا CD4+ FoxP3+ | عتبه | قناة DAPI (> 600) | o كائنات التسمية مع توطين مشترك من DAPI و Cy5/A647، محاطة بإشارة FITC/A488 | س التهم وكثافة خلايا CD4 + FoxP3 + في ROIs ستروما وParenchyma |
| قناة FITC/A488 بولي تنعيم (> 850) | o مسح الكائنات أصغر من 7 ميكرون2 | س إحداثيات خلايا CD4 + FoxP3 + الفردية | |||
| قناة Cy5/A647 (>800) | |||||
| 4 | لتحديد موقع خلايا CD8+ وقياسها | عتبه | قناة DAPI (<1200) | o مسح الأجسام الإيجابية أصغر من 15 ميكرومتر2 | س التهم وكثافة خلايا CD8 + في ROIs ستروما وParenchyma |
| قناة Cy5/A647 المتوسط (>80) | س إغلاق الكائنات الإيجابية 2 بكسل | س إحداثيات الخلايا الفردية | |||
| o كائنات منفصلة | |||||
| 5 | لتحديد موقع خلايا CD68+ وقياسها | عتبه | قناة FITC/A488 (>200) | o مسح الأجسام الإيجابية أصغر من 20 ميكرومتر2 | س التهم وكثافة خلايا CD68 + في ROIs ستروما وParenchyma |
| س Dilate الكائنات الإيجابية 3 بكسل | س إحداثيات خلايا CD68 + الفردية | ||||
| o كائنات منفصلة | |||||
| 6 | لتحديد موقع خلايا MPO+ وقياسها | عتبه | قناة DAPI (> 400) | o مسح الكائنات أصغر من 5 ميكرومتر2 | س التهم وكثافة الخلايا MPO + في ROIs ستروما وParenchyma. |
| قناة TRITC/A568 (900-4000) | س Dilate 3 بكسل الكائنات الإيجابية | س إحداثيات خلايا MPO + الفردية. | |||
| o كائنات منفصلة | |||||
| 7 | لتحديد موقع وقياس مساحة αSMA+ | عتبه | قناة TRITC/CF568 (> 1050) | o مسح الأجسام الإيجابية أصغر من 25 ميكرومتر2 | س عدد وكثافة αSMA + المنطقة في ROIs ستروما وParenchyma |
| س Dilate 3 بكسل الكائنات الإيجابية | س إحداثيات αSMA + بكسل | ||||
| 8 | لتحديد موقع منطقة CD34+ وقياسها | عتبه | قناة DAPI (<5000) | o مسح الأجسام الإيجابية أصغر من 25 ميكرومتر2 | س التهم وكثافة منطقة CD34 + في ROIs ستروما وParenchyma |
| قناة Cy5/A647 المتوسط (> 120) | س Dilate 3 بكسل الكائنات الإيجابية | س إحداثيات بكسل CD34+ | |||
| 9 | إنشاء خرائط حرارة الأنسجة لسلسلة خلايا معينة | خريطة حرارة الكائن | خريطة حرارة الكائن | س هيت ماب | |
| رسم نصف قطر 50 ميكرومتر | --- | ||||
| 10 | تحديد التوطين المشترك بين αSMA وDesmin | عتبه | قناة TRITC (CF568) (> 1050) | o تسمية الكائنات ذات قيم العتبة الأعلى لـ TRITC (CF568) | o تحديد التعبير المترجمة المشتركة لـ αSMA وDesmin |
| قناة Cy5 (A647) (> 1000) | o تسمية الكائنات ذات قيم العتبة الأعلى لـ Cy5 (A647) | ||||
| o تسمية الكائنات ذات التعريب المشترك لقيم العتبة فوق لـ TRITC (CF568) و Cy5 (A647) | |||||
| o مسح الأجسام الإيجابية أصغر من 25 ميكرومتر2 |
الجدول 1: البارامترات العامة المستخدمة في تصميم الأدوات المستخدمة لتحليل الصور. يتم تعديل المعلمات المحددة في هذا الجدول وفقًا للخصائص الفريدة للصور المستخدمة في هذا التحليل (على سبيل المثال، الخلفية والقطع الأثرية وما إلى ذلك) وقد لا تنطبق على الصور الأخرى. نظرًا لأن خطوات ما بعد المعالجة المذكورة تم تحديدها للصور المحددة التي تم تحليلها في هذه الدراسة ، فهي غير مفصلة عن عمد. يجب على المستخدم تخصيص APPs للصور التي سيتم تحليلها.
| قسم / تلطيخ | الأجسام المضادة الأولية | الأجسام المضادة الثانوية |
| القسم الثاني/1st تلطيخ | الماوس IgG2a المضادة للإنسان αSMA الماوس IgG1 المضادة للإنسان CD34 أرنب مضاد للإنسان سايتوكراتين 8/18 |
الماعز المضادة للفأرة IgG2a CF568 الجرذ المضادة للفأر IgG1 A647 الحمار المضادة للأرنب A488 |
| القسم الثاني/2تلطيخ | أرنب مضاد للإنسان ديسمين الماوس المضادة للإنسان CD68 |
الحمار المضادة للأرنب A647 الحمار المضادة للفأرة DyLight 755 |
| القسم الثالث/1st تلطيخ | الماوس المضادة للإنسان CD4 أرنب مضاد للإنسان FoxP3 الماعز المضادة للإنسان MPO |
الحمار المضادة للفأرA488 الحمار المضادة للأرنب A647 الحمار المضادة للماعز A568 |
| القسم الثالث/2تلطيخ | أرنب مضاد للالإنسان CD3 الماوس المضادة للإنسان CD8 |
الحمار المضادة للفأرة DyLight 755 الحمار المضادة للأرنب A647 |
الجدول 2: أزواج الأجسام المضادة الأولية الثانوية لـ mIF.
Discussion
هناك حاجة بسيطة وسهلة الوصول إليها لتنفيذ تقنيات متعددة الشينغ التي تسمح بالاستبانة المكانية للخلايا المناعية في أقسام الأنسجة لرسم خريطة للمشهد المناعي في السرطان والاضطرابات المناعية الأخرى. هنا، ونحن نصف استراتيجية تدمج العلامات المتاحة على نطاق واسع وتقنيات التحليل الرقمي لتوسيع قدرة تعدد النصائح والتقييم متعدد الأبعاد من المقالات التصوير12،13،17،19. تلطيخ ثلاثة أقسام المسلسل لعلامات مختلفة ، وإعادة استخدام المقاطع من خلال تقنيات التجريد وإعادة التحقيق ، مكنتنا من تصور 11 المعلمات بالإضافة إلى بقع H & E و PSR. تم محاذاة ست صور من هذه الأقسام بطريقة آلية باستخدام وحدة محاذاة الأنسجة. كانت المحاذاة دقيقة على مستوى الخلية الفردية للصور الناشئة من نفس القسم ومتوافقة للغاية للصور الناشئة من الأقسام المجاورة. متعددة الظاهري مكنتنا من تحديد كيفية علامات تصور في أحد الأقسام تتصل مكانيا إلى علامات تصور في قسم آخر متجاورة. وفي حين أن بعض البقع وصفت بثاني أكسيد الكربون، فإن البعض الآخر وصف بـ TCs، مما يسمح لنا بتحديد كمي لثاني أكسيد الكربون في مختلف اللجان. وقد أدى استخدام أدوات برمجية للقياس الكمي الآلي لـ COIs إلى تبسيط وتسريع معالجة الصور إلى حد كبير. وعلاوة على ذلك، تم تطبيق التحليل الرقمي على أقسام الأنسجة بأكملها بدلاً من مجالات نظر مختارة، مما أدى إلى تمثيل غير متحيز للإدارة المتكاملة للأنسجة. وعلاوة على ذلك، ونظرا ً لأن إحداثيات الأنسجة في COIs تم تسجيلها، كان من الممكن توليد خرائط حرارة الأنسجة.
هناك عدة مناطق في هذا البروتوكول حيث قد تكون هناك حاجة لاستكشاف الأخطاء وإصلاحها. أولاً، يمكن أن يؤثر استرداد المستضد الضعيف على جودة الـ mIF، وبالتالي يجب تحسين نوع عازل استرجاع المستضد والمدة لظروف الفحص/العلامات الحيوية المحددة المستخدمة. ثانياً، ينبغي تكييف نوع محلول الحجب المستخدم مع الأنسجة/المستضد/الأنواع من الأجسام المضادة الأولية والثانوية. في أيدينا، إضافة 10٪ مجموع المصل من الأنواع حيث النسيج يأتي من مستقبلات FC منعت، وبالتالي انخفاض غير محدد ربط الأجسام المضادة. إضافة 10٪ من المصل من الأنواع التي أثيرت فيها الأجسام المضادة الثانوية من شأنها أن تقلل من التعلق المباشر غير المحدد للأجسام المضادة الثانوية إلى قسم الأنسجة. ثالثاً، من الضروري التحقق من خصوصية الأجسام المضادة الأولية والثانوية باستخدام الضوابط الإيجابية والسلبية المناسبة. رابعا، زيادة الفلورات في بعض القنوات ونشر DAPI على تجريد الأجسام المضادة الأولية هي أيضا شائعة. لمعالجة الفلور التّيعز المُحسّن، استخدمنا أزواج الأجسام المضادة الأولية/الثانوية حيث كانت الإشارة المحددة تحتوي على قيم كثافة لا تقل عن 5 x تلك التي تحتوي عليها الخلفية. وأخيرا، لا يمكن توصف بعض الأجسام المضادة تقارب عالية مع إجراءات تجريد العادية. في هذه الحالة، نوصي باستخدام مثل هذه الأجسام المضادة في الجولة الأخيرة من وضع العلامات. قد يضطر المستخدم إلى محاولة تسلسل تلطيخ مختلف للعثور على التكوين الأمثل للأجسام المضادة ذات الاهتمام. وينبغي تأكيد كفاءة التجريد قبل الشروع في جولة ثانية أو ثالثة من وضع العلامات.
القيد الرئيسي والتحدي الذي تواجهه هذه الاستراتيجية هو العثور على التوليفات الصحيحة من الأجسام المضادة الفلورية الأولية والثانوية لعلامات الاهتمام. العثور على الأجسام المضادة الأولية التي أثيرت في أنواع مختلفة أو مع isotypes المختلفة التي يمكن استخدامها في وقت واحد محدودة بما هو متاح تجاريا. معظم الماسحات الضوئية الشريحة كلها مجهزة المصابيح والمرشحات التي تسمح التصوير بحد أقصى خمس قنوات، والأجسام المضادة الثانوية في الأنواع الصحيحة والفلوروفور الحق ليست متاحة دائما. لقد تغلبنا جزئيا على هذه القيود باستخدام البقع التسلسلية والعلامات المتسلسلة. قد تحتاج العديد من تركيبات الأجسام المضادة إلى اختبارها للوصول إلى أفضل تركيبة لعلامات الاهتمام. القيد الآخر هو جودة تلطيخ DAPI ، لأن تجريد وإعادة التحقيق قد لا تسمح دائمًا بإجراء تجزئة النوى.
وحدة محاذاة الأنسجة يتطلب الحد الأدنى من التدريب وليس مهارات البرمجة من المستخدمين. البرنامج يسمح نظريا محاذاة عدد غير محدود من الصور. ومع ذلك، تعتمد المحاذاة الدقيقة على صلة المقاطع، حيث يتم محاذاة الأقسام الأقرب التي تكون أكثر توافقًا من الناحية النسيجية بشكل أكثر دقة. استخدمنا وحدة المؤلف من VIS لتوليد APPs. هناك حاجة إلى المعرفة الأساسية لتحليل الصور لإنشاء APPs ، ولكن هذا هو الحال بنفس القدر عند استخدام أي برنامج آخر لتحليل الصور. وتشمل المزايا الفريدة لـ VIS بالمقارنة مع برامج تحليل الصور الأخرى المحاذاة الآلية للصور من الأقسام المعدة باستخدام أساليب مختلفة (على سبيل المثال، IF، كيمياء النسيج، IHC). وهذا يسمح الدراسات توطين مشترك من علامات متعددة من الفائدة باستخدام multixing الظاهري. وعلاوة على ذلك، فإن التصميم المرن والسهل الاستعمال للAPPs يسمح بالتخصيص الخاص بالمستخدم. القياس الكمي الآلي ورسم الخرائط ، وإمكانية معالجة أقسام الأنسجة بأكملها ، يوفر الوقت ويقلل من التحيز مقارنة بالعد اليدوي عن طريق الفحص البصري.
هذه الاستراتيجية هي أداة بحثية مفيدة للغاية لمناعة الأنسجة في سياق السرطان والمناعة الذاتية ولكنها لا تزال غير مصدقة للاستخدام السريري. مع توحيد إضافية والتحقق من صحة, ويمكن استخدامه في المستقبل لتطبيقات متعددة (على سبيل المثال, لرسم خريطة المشهد المناعي في السرطان للتنبؤ ورصد الاستجابة لعوامل العلاج المناعي). كما يمكن تكييفه مع الحالات الالتهابية المختلفة (على سبيل المثال، مرض الأمعاء الالتهابي) للجمع بين التقييم المرضي والمؤشرات الحيوية التكهنية.
وتتمثل الخطوات الحاسمة الرئيسية في هذا البروتوكول في كفاءة/خصوصية وضع العلامات ومتانة الإجراءات المصممة للاستخدام المقصود أو العلامة البيولوجية. وبالتالي ، فإن التحقق من الصحة المنتظم عن طريق الفحص البصري ، وخاصة عند تصميم APP جديد ، أمر ضروري. الاستخدام الفعال لجولات متعددة من تجريد وإعادة التحقيق أو أنواع مختلفة من البقع على نفس القسم هي مكونات حاسمة ويمكن أن تكون أنسجة أو قسم محدد. التحقق من كفاءة هذه العمليات قبل الشروع في تحليل دفعة كبيرة أمر بالغ الأهمية.
باختصار، نحن نقدم استراتيجية تزيد من المعلومات الكمية والمكانية التي يمكن الحصول عليها من عينات الأنسجة السريرية القيمة. ويمكن الوصول على نطاق واسع إلى الموارد والمعدات والمعارف اللازمة لتنفيذ هذه المنهجية. ونحن نقترح هذه المنهجية كدليل مفيد لتخطيط المقالات التي تهدف إلى تحديد مجموعات الخلايا المناعية وقياسها كمياً ورسم خرائط لها في منطقة TME.
Disclosures
ويعلن صاحبا البلاغ عدم وجود تضارب في المصالح.
Acknowledgments
نشكر المشارك في الدراسة. نشكر لويز روسو، منسقة البنك الحيوي HBP لاسترداد عينات الأنسجة وجميع المعلومات السريرية المرتبطة بها. نحن نعترف بعلم الأمراض الجزيئية ومرافق التصوير الخلوي الأساسية في CRCHUM ومايكل بيرش من Visiopharm للحصول على مساعدة تقنية ممتازة. التمويل: تم دعم هذه الدراسة من خلال منح مقدمة من مؤسسة الكبد الكندية، ومؤسسة التنمية الاقتصادية في كيبيك-سانتي (FRQS) شبكة الإيدز والأمراض المعدية (Réseau SIDA-MI)، والشبكة الكندية لالتهاب الكبد C (CanHepC). يتم تمويل CanHepC من خلال مبادرة مشتركة من المعاهد الكندية للبحوث الصحية (NHC-142832) ووكالة الصحة العامة الكندية. حصل M.F.M. على زمالات من جامعة مونتريال، وبورصة غابرييل ماركيز، وFRQS. حصل T.F. على زمالات الدكتوراه من CIHR و CanHepC. S.T. يحمل كرسي روجر-ديس غروسيليرس في جراحة الأورام الكبدية والبنكرياس، جامعة مونتريال.
مساهمات المؤلف: M.F.M. تم تصميمها وتجربتها وتحليلها وتحليل البيانات. T.F. صممت التجارب. أ. سي بي. قدمت التوجيه التقني. أجرى G.S. جميع التقييم المرضي لموضوع الدراسة وقدم مدخلات حول جميع الجوانب المرضية. قامت L.M. بتلطيخ H & E وتحسينها وتنفيذها. M.N.A. أداء وصمة عار PSR وقدمت مدخلات تقنية قيمة. ساهم N.B. في تحليل الصورة. S.T. هو المحقق الرئيسي للبنك الحيوي HBP وهو مسؤول عن الإشراف على التشغيل العام للبنك الحيوي. كما قدم مدخلات لا تقدر بثمن في جميع جوانب المشروع وآثاره السريرية. M.F.M، T.F. ، وN.H.S. تصور وتصميم الدراسة. وأشرف ت. ه. س. على العمل وحصل على تمويل. كتب م.ف.م، وT.F.، و A.C-B، ون.ه.س. المخطوطة. قام جميع المؤلفين بمراجعة المخطوطة والموافقة عليها.
Materials
| Name | Company | Catalog Number | Comments |
| Antigen Retrieval Solution: Sodium Citrate Buffer (10 mM Sodium Citrate, 0.05% v/v Tween 20, pH 6.0) | |||
| Blocking Solution: 1 % BSA, 10 % filtered human serum, 10 % filtered donkey serum, 0.1 % Tween 20, and 0.3% Triton in PBS | |||
| Bovine serum albumin (BSA) | Multicell | 800-095-EG | |
| Coplin jars (EASYDIP SLIDE STAINING SYSTEM) | Newcomersupply | 5300KIT | |
| Cover slides | Fisherbrand | 12-545E 22*50 | |
| Direct Red 80 | Sigma Aldrich | 365548 | |
| Donkey Serum | Sigma Aldrich | D9663 | |
| Ethanol 100% | |||
| Electric pressure cooker | Salton | ||
| Eosin | Leica Biosystems | 3801600 | CAUTION, eye irritation |
| Fast Green FCF | Sigma Aldrich | F7252 | CAUTION, harmful by inhalation, ingestion and skin absortion |
| FFPE section (4μm) slides | |||
| Glycine 0,1 M in PBS | |||
| Hematoxylin Stain Solution, Gil 1. Formulation, Regular Strength | Ricca Chemical Company | 3535-32 | |
| Holder (EasyDip Staining Jar Holder) | Newcomersupply | 5300RK | |
| Human Serum | Gemini | 22210 | |
| Humidity chamber | Millipore Sigma | Z670138-1EA | |
| Pap pen | abcam | ab2601 | |
| PBS | |||
| PBS-Tween 20 (0.1% v/v) | |||
| Permount Mounting Media | Fisher Chemical | SP15-500 | |
| Picric Acid 1.3 % | Sigma Aldrich | P6744 | CAUTION, skin and eye irritation |
| Picro-Sirius Red/Fast Green solution: Fast Green 0.1 % w/v + Sirius Red 0.2 % w/v in 1,3 % picric acid solution | |||
| Primary Antibody Anti-αSMA | Mouse IgG2a 1A4 | Sigma A2547 | Dilution 1/100 |
| Primary Antibody Anti-CD34 | Mouse IgG1 HPCA1/763 | Novus Biologicals NBP2-44568 | Dilution 1/250 |
| Primary Antibody Anti-Cytokeratin 8/18 | Rabbit EP17/EP30 | Agilent IR09461-2 | Ready to use |
| Primary Antibody Anti-CD68 | Mouse KP1 | Abcam ab955 | Dilution 1/200 |
| Primary Antibody Anti-Desmin | Rabbit Polyclonal | Invitrogen PA5-16705 | Dilution 1/200 |
| Primary Antibody Anti-CD4 | Mouse N1UG0 | Affymetrix 14-2444 | Dilution 1/250 |
| Primary Antibody Anti-FoxP3 | Rabbit 1054C | R & D MAB8214 | Dilution 1/100 |
| Primary Antibody Anti-MPO | Goat Polyclonal | R & D Systems AF3667 | Dilution 1/250 |
| Primary Antibody Anti-CD3 | Rabbit SP7 | Abcam ab16669 | Dilution 1/200 |
| Primary Antibody Anti-CD8 | Mouse C8/144B | Invitrogen 14-0085-80 | Dilution 1/200 |
| Secondary Antibody Donkey anti-mouse A488 | Polyclonal | Invitrogen A-21202 | Dilution 1/500 |
| Secondary Antibody Donkey anti-Rabbit A488 | Polyclonal | Invitrogen A-21206 | Dilution 1/500 |
| Secondary Antibody Donkey anti-goat A568 | Polyclonal | Invitrogen A-11057 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit A647 | Polyclonal | Invitrogen A-31573 | Dilution 1/500 |
| Secondary Antibody Rat anti-mouse IgG1 A647 | RMG1-1 | Biolegend 406618 | Dilution 1/500 |
| Secondary Antibody Goat anti-mouse IgG2a CF568 | Polyclonal | Sigma Aldrich SAB4600315 | Dilution 1/500 |
| Secondary Antibody Donkey anti-mouse DyLight 755 | Polyclonal | Invitrogen SA5-10171 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit DyLight 755 | Polyclonal | Invitrogen SA5-10043 | Dilution 1/500 |
| SDS | BioShop | SDS001,500 | CAUTION, oral skin and eye toxicity |
| Shandon multi-program robotic slide stainer | LabX | 11384903 | |
| Shandon Xylene Substitute, | Thermo Fisher Scientific | CA89413-336 | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Shaking water bath | |||
| SlowFade Gold antifade reagent with DAPI | Invitrogen | S36938 | |
| Sodium Citrate Dihydrate | Millipore Sigma | 1545801 | CAUTION, eye irritation |
| Stripping Buffer: mix 20 ml 10% w/v SDS with 12.5 ml 0.5 M Tris-HCl (pH 6.8), and 67.5 ml ultra-pure water. Under a fume hood, add 800 uL of 2-mercapto ethanol (114,4 mM final concentration) | |||
| Triton X-100 | Sigma Aldrich | T8787-50ML | |
| Tris-HCl | BioShop | 77-86-1 | |
| Tween 20 | Fisher Scientific | BP337-500 | |
| VIS Software | Visiopharm | ||
| Whole slide scanner Olympus BX61VS | Olympus | Microscope: Olympus Slide Scanner BX61VS, 5 slides scanner, motorized stage, autofocus. Camera: Lightsource: Xcite-120. Filters: BrightLine® Sedat filter set (# LED-DA/FI/TR/Cy5-4X4M-B-000, Semrock) | |
| Xylene | Sigma Aldrich | 214736-4L | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Xylene : Ethanol solution (1:1 v/v) | |||
| 2-mercaptoethanol | Sigma | M6250 | CAUTION, harmful by ingestion, inhalation, fatal if sking absortion. Eye irritation. Use fume hood |
References
- Greten, F. R., Grivennikov, S. I. Inflammation and Cancer:Triggers, Mechanisms, and Consequences. Immunity. 51 (1), 27-41 (2019).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Binnewies, M., et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nature Medicine. 24 (5), 541-550 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathology: an official journal of the United States and Canadian Academy of Pathology, Inc. 31 (2), 214-234 (2018).
- Bindea, G., et al. Spatiotemporal dynamics of intratumoral immune cells reveal the immune landscape in human cancer. Immunity. 39 (4), 782-795 (2013).
- Galon, J., et al. Towards the introduction of the 'Immunoscore' in the classification of malignant tumours. The Journal of Pathology. 232 (2), 199-209 (2014).
- Finotello, F., Eduati, F. Multi-Omics Profiling of the Tumor Microenvironment: Paving the Way to Precision Immuno-Oncology. Frontiers in Oncology. 8, 430 (2018).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: a method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Porta Siegel, T., et al. Mass Spectrometry Imaging and Integration with Other Imaging Modalities for Greater Molecular Understanding of Biological Tissues. Molecular Imaging and Biology : MIB: the official publication of the Academy of Molecular Imaging. 20 (6), 888-901 (2018).
- Buchberger, A. R., DeLaney, K., Johnson, J., Li, L. Mass Spectrometry Imaging: A Review of Emerging Advancements and Future Insights. Analytical Chemistry. 90 (1), 240-265 (2018).
- Pirici, D., et al. Antibody elution method for multiple immunohistochemistry on primary antibodies raised in the same species and of the same subtype. Journal of Histochemistry and Cytochemistry. 57 (6), 567-575 (2009).
- Gendusa, R., Scalia, C. R., Buscone, S., Cattoretti, G. Elution of High-affinity (>10-9 KD) Antibodies from Tissue Sections: Clues to the Molecular Mechanism and Use in Sequential Immunostaining. Journal of Histochemistry and Cytochemistry. 62 (7), 519-531 (2014).
- van der Loos, C. M. Multiple immunoenzyme staining: methods and visualizations for the observation with spectral imaging. Journal of Histochemistry and Cytochemistry. 56 (4), 313-328 (2008).
- Stack, E. C., Wang, C., Roman, K. A., Hoyt, C. C. Multiplexed immunohistochemistry, imaging, and quantitation: a review, with an assessment of Tyramide signal amplification, multispectral imaging and multiplex analysis. Methods. 70 (1), 46-58 (2014).
- Toth, Z. E., Mezey, E. Simultaneous visualization of multiple antigens with tyramide signal amplification using antibodies from the same species. Journal of Histochemistry and Cytochemistry. 55 (6), 545-554 (2007).
- Robertson, D., Savage, K., Reis-Filho, J. S., Isacke, C. M. Multiple immunofluorescence labeling of formalin-fixed paraffin-embedded (FFPE) tissue. BMC Cell Biology. 9, 13 (2008).
- Segnani, C., et al. Histochemical Detection of Collagen Fibers by Sirius Red/Fast Green Is More Sensitive than van Gieson or Sirius Red Alone in Normal and Inflamed Rat Colon. PloS One. 10 (12), 0144630 (2015).
- Bolognesi, M. M., et al. Multiplex Staining by Sequential Immunostaining and Antibody Removal on Routine Tissue Sections. Journal of Histochemistry and Cytochemistry. 65 (8), 431-444 (2017).



