ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Den sætteplante oversvømmelse assay letter hurtig screening af vilde tomat tiltrædelser for modstanden mod Pseudomonas syringae bakterie. Denne analyse, der anvendes i forbindelse med sætteplante bakterievækst assay, kan hjælpe med yderligere at karakterisere den underliggende resistens over for bakterien, og kan bruges til at screene kortlægning populationer til at bestemme det genetiske grundlag for resistens.
Abstract
Tomat er en agronomisk vigtig afgrøde, der kan være inficeret med Pseudomonas syringae, en Gram-negativ bakterie, resulterer i bakteriel plet sygdom. Dentomat-P. syringae pv. tomat patosystem er almindeligt anvendt til at dissekere det genetiske grundlag for plante medfødte reaktioner og sygdomsresistens. Mens sygdommen blev forvaltet med succes i mange årtier gennem indførelsen af Pto / Prf genklyngen fra Solanum pimpinellifolium til dyrket tomat, race 1 stammer af P. syringae har udviklet sig til at overvinde resistens overrakt af Pto / Prf genklyngen og forekomme over hele verden.
Vilde tomatarter er vigtige reservoirer af naturlig mangfoldighed i patogengenkendelse, fordi de udviklede sig i forskellige miljøer med forskellige patogentryk. I typiske skærme for sygdomsresistens i vilde tomat, voksne planter anvendes, som kan begrænse antallet af planter, der kan screenes på grund af deres udvidede vækst tid og større vækst plads krav. Vi udviklede en metode til at screene 10-dages gamle tomatplanter for modstand, hvilket minimerer plantevækst tid og vækst kammer plads, giver mulighed for en hurtig omsætning af planter, og giver mulighed for store stikprøvestørrelser, der skal testes. Frøplanter resultater af overlevelse eller død kan behandles som diskrete fænotyper eller på en modstand skala defineret ved mængden af ny vækst i overlevende planter efter oversvømmelse. Denne metode er blevet optimeret til at screene 10-dages-gamle tomat planter for modstand mod to P. syringae stammer og kan nemt tilpasses til andre P. syringae stammer.
Introduction
Pseudomonas syringae er en Gram-negativ patogen bakterie, der inficerer en bred vifte af plante værter. Bakterier ind i værtsplanten gennem stomata eller fysiske sår og formere sig i apoplast1. Planter har udviklet en todelt immunrespons for at beskytte mod infektion med bakterielle patogener. Det første niveau forekommer på plantecelleoverfladen, hvor mønstergenkendelsesreceptorer på plantecellemembranen opfatter stærkt bevarede patogenrelaterede molekylære mønstre (PAMP'er) i en proces kaldet PAMP-udløst immunitet (PTI)2. Under denne proces, værtsplanten upregulates forsvar respons veje, herunder aflejring af callose til cellevæggen, lukning af stomata, produktion af reaktive ilt arter, og induktion af patogenese-relaterede gener.
Bakterier kan overvinde PTI ved at udnytte en type III sekresion system til at levere proteiner, kaldet effektorer, direkte ind i plantecellen3. Effektorproteiner er normalt rettet mod komponenter i PTI og fremmer patogenvirulens4. Det andet niveau af planteimmunitet opstår i plantecellen ved anerkendelse af effektorproteiner. Denne anerkendelse er afhængig af resistens gener, som indkode nukleotid-bindende site leucin-rige gentage indeholdende receptorer (NLRs). NLR'er er i stand til enten at genkende effektorer direkte eller genkende deres aktivitet på et virulensmål eller lokkedue5. De udløser derefter en sekundær immunrespons i en proces kaldet effector-udløst immunitet (ETI), som ofte er forbundet med en overfølsom respons (HR), en form for lokaliseret celledød på infektionsstedet6. I modsætning til gen-for-genresistens forbundet med ETI, planter kan udvise kvantitativ delvis resistens, som er afhængig af bidraget fra flere gener7.
P. syringae pv. tomat (Pst) er årsagssammenhæng af bakteriel plet på tomat og er en vedvarende landbrugsproblem. Fremherskende stammer i feltet har typisk været Pst race 0 stammer, der udtrykker den ene eller begge af type III effektorer AvrPto og AvrPtoB. DC3000 (PstDC3000) er en repræsentativ race 0 stamme og en model patogen, der kan forårsage bakteriel plet i tomat. For at bekæmpe bakteriel plet sygdom, opdrættere introgressed Pto [P. syringae pv. tomat]/ Prf [Pto resistens og fenthion følsomhed] genklynge fra den vilde tomatarter Solanum pimpinellifolium i moderne sorter8,9. Pto-genet koder et serine-threoninproteinkinase, der sammen med Prf NLR giver resistens over for PstDC3000 via anerkendelse af effektorerne AvrPto og AvrPtoB10,11,12,13,14. Denne modstand er imidlertid ineffektiv over for nye race 1-stammer, hvilket giver mulighed for deres hurtige og aggressive spredning i de seneste år15,16. Race 1 stammer unddrage anerkendelse af Pto / Prf klynge, fordi AvrPto er enten tabt eller muteret i disse stammer, og AvrPtoB synes at akkumulere minimalt15,17,18.
Vilde tomatpopulationer er vigtige reservoirer af naturlig variation for pst-resistens og er tidligere blevet anvendt til at identificere potentielresistens loci19,20,21. Men, nuværende skærme for patogenmodstand udnytte 4-5-ugergamle voksne planter20,21. Derfor er de begrænset af væksttid, vækstkammer plads, og relativt små stikprøvestørrelser. For at løse begrænsningerne af konventionelle tilgange, vi udviklet en høj-throughput tomat P. syringae modstand assay ved hjælp af 10-dages gamle tomat planter22. Denne tilgang giver flere fordele i forhold til at bruge voksne planter: nemlig kortere væksttid, reducerede pladskrav og højere gennemløb. Desuden har vi vist, at denne fremgangsmåde trofast opsummerer sygdomsresistens fænotyper observeret i voksne planter22.
I den kimvarmeanalyse, der er beskrevet i denne protokol, dyrkes tomatplanter på petriskåle af sterile Murashige- og Skoog-medier (MS) i 10 dage og oversvømmes derefter med et inokulum, der indeholder de pågældende bakterier og et overfladeaktivt stof. Efter oversvømmelser kan planter vurderes kvantitativt for sygdomsresistens via bakterievækstanalyser. Derudover kan sætteplante overlevelse eller død fungere som en diskret modstand eller sygdom fænotype 7-14 dage efter oversvømmelse. Denne tilgang tilbyder en høj-throughput alternativ til screening et stort antal vilde tomat tiltrædelser for resistens over for Pst race 1 stammer, såsom Pst stamme T1 (PstT1), og kan nemt tilpasses til andre bakterielle stammer af interesse.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Forberedelse og anvendelse af biosikkerhedsskabe
- Tør biosikkerhedsskabet af med 70 % ethanol.
- Luk rammen og tænd for ultraviolet lys i biosikkerhedsskabet i 15 min.
- Efter 15 minutter skal det ultraviolette lys slukkes i biosikkerhedsskabet. Løft rammen og tænd for blæseren i 15 min.
- Tør alle genstande, der skal bruges i biosikkerhedsskabet, af med 70 % ethanol, før varerne lægges i det steriliserede kabinet.
- Rene handsker eller bare hænder med 70 % ethanol, før du arbejder i biosikkerhedsskabet.
- Arbejde i midten af biosikkerhedsskabet, væk fra blæseren.
- Brug uåbnede flasker autoclaved steril10 mM MgCl2 og ultrapure H2O til eksperimenter. Sæt flasker i biosikkerhedsskabet, og åbn dem kun i det steriliserede biosikkerhedsskab, ikke på bordpladen.
- Brug dedikerede glaspipetter og pipettespidser til arbejde i det steriliserede biosikkerhedsskab. Sørg for, at disse kun åbnes i biosikkerhedsskabet, aldrig på bordpladen.
- Efter brug af biosikkerhedsskabet skal alt affald (undtagen blegemiddelaffald) autoklaves, og overfladen tørres af med 70 % ethanol.
2. Fremstilling af plantemedier
- Der vejes og opløses 0,5 x MS basalsalte i ultrarent H2O. Afvejes 0,8% bacto agar og tilsættes derefter til opløst 0,5 x MS.
- Autoklave og lad mediet køle af i 50 °C vandbad i 1 time før hælde eller pipettere.
- For at sikre, at pladerne ikke overfyldes, markeres polystyren engangssterile 100 x 25 mm plader til et fyldniveau på 40 ml. Mediet hældes i 100 x 25 mm sterile plader i et steriliseret biosikkerhedsskab.
3. Fremstilling af plantematerialer og vækstbetingelser
- Læg tomatfrø i et 2,2 ml mikrocentrifugerør og tilsæt 2,0 ml 50% blegemiddelopløsning.
- Rock røret på en rocker i 25 min.
- Efter 25 min fjernes frøene fra vippen, og blegemiddelopløsningen fjernes med en pipette i det sterile biosikkerhedsskab. Sørg for, at alt blegemiddel er fjernet.
- Der tilsættes 2 ml sterilt ultrarent H2O for at vaske frøene. Inverter røret 5x.
- Fjern væsken fra røret med en pipette.
- Gentag trin 3.3-3.5 for at vaske frøene 4x mere.
- Der tilsættes 2 ml sterilt ultrarent H2O, og frøene hældes i en tom steril petriskål.
- Flammepincet i ethanol og lad det køle af før overførsel og jævnafstand frø på 100 x 25 mm plader, der indeholder 0,5x MS + 0,8% agar medier.
- Overfør 5-7 frø i en linje over midten af en plade og forsegle kanterne af pladerne med kirurgisk tape (1,25 cm x 9,1 m).
- Stratificere de steriliserede frø ved 4 °C i mørke i mindst 3 dage for at synkronisere spiring. Sørg for, at pladerne er stablet fladt og med billedsiden nedad, så frøene ikke går over på pladen.
- Orienterpladerne lodret, så rødderne vokser ned langs pladens overflade, med linjen af frø orienteret vandret, når de overføres til vækstkammeret.
BEMÆRK: Indstil vækstkammeret til 22 °C og giv 16 timers lys ved en lysintensitet på ~200-220 μE meter-2 s-1 og 8 timers mørke. - Før oversvømmelser, vokse planter i 10 dage i vækstkammeret på hvilket tidspunkt planter typisk vises fuldt frem og ekspanderede cotyledons og nye første sande blade (Figur 1).

Figur 1: Udviklingsstadiet af typiske 10 dage gamle tomatplanter. Rio Grande-PtoR tomatfrø blev steriliseret, belagt og stratificeret i mindst 3 dage i mørke ved 4 °C. Frøplanterne blev dyrket på 0,5 x MS-plader i 10 dage ved 22 °C, før de blev oversvømmet. Typisk, på 10 dage cotyledons er fuldt udvidet, og de første sande blade er begyndt at dukke op. Klik her for at se en større version af dette tal.
4. Forberedelse af King's B23 (KB) medier
- Fyld bægerglas med 500 ml ultraren H2O og rør på en rørplade.
- 20 g bacto peptone, 1,5 g vandfri K2HPO4og 12,5 ml glycerol i et bægerglas med ultrarent H2O.
- Den opløste blanding hældes i en 1 L gradueret cylinder, og der bringes op til et 1 L sidste volumen med ultrarent H2O.
- Hæld bouillon tilbage i bægerglasset og rør indtil blandet.
- 7,5 g bacto agar vejes i to 500 ml glasflasker, og der tilsættes 500 ml KB bouillon fra trin 4.4 til hver flaske. Autoklave i 20 min.
- Fjern flaskerne fra autoklaven og hvirvle forsigtigt at distribuere agar.
- Flaskerne overføres til et vandbad på 50 °C i 1 time.
- Efter 1 time overføres flasken til biosikkerhedsskabet og under aseptiske forhold, der tilsættes 1.600 μL steril1 M MgSO4og passende antibiotika til mediet.
BEMÆRK: For rifampicinresistente stammer PstDC3000 og PstT1 anvendes rifampicin opløst i dimethylformamid ved en endelig koncentration på 50 μg/ml. Brug cycloheximid opløst i ethanol ved en endelig koncentration på 50 μg/ml for at forhindre svampevækst på pladerne. - Hvirvle mediet forsigtigt at blande og derefter hælde til at dække bunden af pladerne.
- Pladerne skal størkne mindst 1 time, før de opbevares på hovedet ved 4 °C.
5. Vedligeholdelse af bakteriestammer og dyrkningsbetingelser
- Hold en glycerolbestand fra en enkelt bakteriekoloni som 1 ml mættet bakteriekultur og 333 μL steril 80% glycerol ved -80 °C.
- Plasterbakterier (dvs. pstT1) fra en glycerolbestand til KB agar med passende antibiotika (afsnit 4).
- Lad bakterierne komme sig i 2 dage ved 28 °C, før de stinrer friske bakterier på selektiv KB agar ved hjælp af en flad, steril tandstikker.
- Streak friske bakterier fra glycerol lager på passende selektiv KB agar ved hjælp af en flad, steril tandstikker.
BEMÆRK: Sørg for, at det lappede glycerollager ikke er mere end 2 uger gammelt. - For PstDC3000 inkuberes KB-pladen ved 28 °C i 24 timer, før der blev brugt bakterier i oversvømmelsesforsøget.
- For PstT1 inkuberes KB-pladen ved 28 °C i 48 timer, før der blev brugt bakterier i oversvømmelsesforsøget.
6. Forberedelse af PstT1 i nokulum
- Aseptisk resuspenderbakterierne i sterile 10 mM MgCl2 til en optisk massefylde ved 600 nm (OD600)på 0,1 eller ca. 5 x 107 kolonidannende enheder (CFU)/mL).
- Udfør serielle fortyndinger med steril 10 mM MgCl2-opløsning i biosikkerhedsskabet. For PstT1 skal du bruge et spektrofotometer til at lave inoculum med en startkoncentration på OD600 = 0,1.
- For PstT1 fremstilles en 1/10 fortynding fra den første resuspension ved OD600 = 0,1 for at opnå en seriel fortynding ved en koncentration af OD600 = 0,01.
- Brug seriefortyndingen ved OD600 = 0,01 fra trin 6.3 til at foretage en 3/4-fortynding for at opnå en endelig OD600 = 0,0075.
- Der forekommer 1/10 fortynding af ikke-ionisk organisk organisk tefsiliciummetcopolymer C13H34O4Si3 (dvs. overfladeaktivt stof) i 10 mM MgCl2 og vortex for 15 s. Tilsæt 1/10 lager af overfladeaktivt stof til den sidste serielle fortynding (OD600 = 0,0075) til en endelig koncentration på 0,015% og hvirvle godt at blande.
7. Forberedelse af PstDC3000 inoculum
- Aseptisk resuspenderbakterier i steril10 mM MgCl2 til en optisk massefylde ved 600 nm (OD600)på 0,1 (ca. 5 x 107 CFU/ml).
- Udfør serielle fortyndinger med steril 10 mM MgCl2-opløsning i biosikkerhedsskabet. For PstDC3000 skal du bruge et spektrofotometer til at lave inoculum med en startkoncentration på OD600 = 0,1.
- For PstDC3000 skal der laves en 1/10 fortynding fra den første resuspension ved OD600 = 0,1 for at opnå en seriel fortynding ved en koncentration af OD600 = 0,01.
- Brug seriefortyndingen ved OD600 = 0,01 fra trin 3 til at foretage en 1/2-fortynding for at opnå en endelig OD600 = 0,005.
- Der foresættes 1/10 fortynding af overfladeaktivt stof i 10 mM MgCl2 og vortex i 15 s. Der tilsættes 1/10-lageret af overfladeaktivt stof til den sidste seriefortynding (OD600 = 0,005) til en endelig koncentration på 0,015% og hvirvles godt til mix.
8. Tomat sætteplante oversvømmelse metode
- Tag pladerne med de 10 dage gamle kimplanter ud af vækstkammeret og sat i biosikkerhedsskabet for at forberede pladerne til oversvømmelse.
- Fjern det kirurgiske bånd fra to plader.
- Indstil en timer til 3 min. Mål 6 ml endelig inokulum(PstT1 OD600 = 0,0075 [afsnit 6] eller PstDC3000 OD600 = 0,005 [afsnit 7]) og overfør 6 ml inokulum til hver plade med de 10 dage gamle planter.
- Skub forsigtigt frøplanterne ned i inoculum med en steril pipettespids. Start timeren.
- Hold en plade i hver hånd. Vip forsiden af pladen ned for at akkumulere inokulum og hovedsagelig nedsænke cotyledons og blade af kimplanter.
- Swish side til side 5-7x og derefter tip pladerne tilbage til at dække rødderne og hele pladen.
- Vip pladerne ned igen for at nedsænke cotyledons og blade, og gentag i alt 3 min.
- Hæld inoculum fra pladerne, sæt pladerne ned på en flad overflade og hæld derefter eventuelle resterende ioculum af en anden gang.
- Ompak pladerne med kirurgisk tape og gentag trin 8.2-8.8 for eventuelle resterende plader.
- Inkuberpladerne igen i vækstkammeret (se trin 3.11 BEMÆRK), efter at alle plader er blevet oversvømmet.
- Fænotype efter 7-10 dage for PstDC3000 eller 10-14 dage for PstT1 (afsnit 11). Hvis bakterievækstanalyser udføres, indsamles bladvæv efter 4 dage (afsnit 9 og 10) og derefter fænotype (afsnit 11). Alternativt kan du udføre fænotypiske analyser og bakterievækstanalyser på separate sæt af planter.
9. Overflade sterilisering af cotyledons for bakterievækst assay
- Fire dage efter oversvømmelse og re-inkubering af planter i vækstkammeret (afsnit 8), fjerne pladerne med tomat planter fra vækstkammeret.
- Nummerer de enkelte frøplanter på pladens bundørt, hvor sætteplanten fastgøres til pladen for hver genotype.
- Mærk sterile 1,5 ml mikrocentrifugerør med de enkelte sættetal, og brug rene pincet til at tabe en 3 mm steril borosilikatperle i hvert rør til brug med en perlebeder. (Se NOTE i trin 10.1).
- Pipette 200 μL på 10 mM MgCl2 i hvert rør og lukke rør.
- Forbered 70% ethanol og hæld 100 ml i et rent bægerglas. Hæld 100 ml sterilt ultrarent H2O i et separat, rent bægerglas.
- Rengør lige finpunktspincet i rustfrit stål med savtakkede spidser med ethanol. Åbn pladen en smule for at tillade aseptisk fjernelse af en cotyledon med de rene pincet.
- Stil smedefolien i bunden af cotyledonen for at fjerne et blad og slip ned i bægerglasset med 70% ethanol for overfladesterilisere i 10 s. Skyl cotyledonen i ultraren H2O for 10 s.
- Placer cotyledon på en køkkenrulle og blot tør med sarte videnskab klude.
- Individuelt vejer hver cotyledon efter overflade sterilisation og blotting, og registrere vægten.
- Cotyledonen anbringes i et tidligere forberedt 1,5 ml mikrocentrifugerør (fra trin 9.3 og 9.4) mærket med den tilsvarende genotype og det tilsvarende individuelle nummer.
- Pladerne forsegles igen med sterilt tape, og planterne i vækstkammeret igen (se trin 3.11 BEMÆRK).
10. Bakteriel vækst assay
- Ved hjælp af prøver fra trin 9.10 homogeniseres vævet ved hjælp af perlebederen i 10 mM MgCl2 i 1-2 min. Hvis vævet ikke er tilstrækkeligt udblødt, homogeniseres igen.
BEMÆRK: Mange producenter producerer perle beater homogenisatorer. Antallet og typen af perler, samt homogenisering tid og hastighed (hvis programmerbare) bør optimeres for hver type homogenisator. Sørg for, at prøverne ikke overophedes under homogeniseringen. - Der tilsættes 800 μL på 10 mM MgCl2 til hvert rør, der indeholder udblødt væv fra trin 10.1, og der vendes flere gange for at blande.
- Der fremstilles seriefortyndinger for hver prøve i 10 mM MgCl2 i 96 brøndplade (100, 10-1, 10-2, 10-3, 10-4, 10-5) ved hjælp af en multikanalpipette (Figur 2A).
- 5 μL fra hver fortyndingsserie med en multikanalpipette på en KB-agarplade (150 mm x 15 mm) med cycloheximid og passende valg for den relevante bakteriestamme (se trin 4.8 BEMÆRK). Lad pladerne tørre helt.
- Pladen inkuberes på hovedet ved 28 °C i 36 timer, og der udvisualisersderefterkolonierne på pladerne ved hjælp af et dissekeret mikroskop for at afgøre, om kolonierne er store nok til at tælle.
BEMÆRK: Hvis kolonierne ikke er store nok, skal pladerne geninkuberes igen, og koloniernes størrelse kontrolleres igen med få timers mellemrum. Typisk kolonierne er tællelige ~ 36-48 h efter inkubation.

Figur 2: Seriefortyndinger til kimning bakterievækst analyser. (A) Macerated bladvæv fra inficerede planter fortyndes før kolonitælling. Fortyndinger udføres i en 96 brøndplade (100 er ufortyndet). Typisk er fortyndinger lavet fra 10-1 til 10-5. (B) Fortynding af plating foringer til bakteriekolonitællinger. I alt 5 μL af hver kolonne i fortyndingsserien er belagt, fra de fleste fortyndede til mest koncentrerede. Når kolonierne er helt tørrede, inkuberes pladen ved 28 °C i 36-48 timer. Kolonier tælles under et 10x dissekeringsmikroskop. Klik her for at se en større version af dette tal.
- Tæl kolonierne under et dissekeret mikroskop, før de smelter sammen (Figur 2B). Kolonierne tælles fra fortyndingsseriens plader med færre end 100 kolonier.
- Efter at have opnået kolonital (Figur 2B) normaliseres tællingerne til 0,01 g væv for planter og konverteres til at logføre bakterievækst (tabel 1).
BEMÆRK: Den gennemsnitlige masse af en Moneymaker-PtoS cotyledon er 0,01 g og bestemmes empirisk22.
| Genotype1 Kolonne A | Vævsvægt (g) Kolonne B | Antal kolonier på et sted kolonne C | Fortyndingsfaktor for spot2 Kolonne D | Justeret antal kolonier3 Kolonne E | Fortyndingsfaktor for seriefortynding kolonne F | Antal kolonier i kolonne G (cfu/0,01 g)4 | Gennemsnit # af kolonier (cfu/0,01 g) Kolonne H | Gennemsnitlig logvækst (cfu/0,01 g (log10)) Kolonne I |
| Eksempel 1 | 0.004 g | 10 | 200 | beregnet som: (C2 x 0,01 g) / B2 = 25 | 1000 | beregnet som: (D2 x E2 x F2) = 5000000 | gennemsnit for prøve 1 til sidste prøve: (dvs. gennemsnit G1:G3) = 7000000 | log over gennemsnittet dvs. log(H2) = 6,85 |
| Eksempel 2 | 0.003 g | 15 | 200 | 50 | 1000 | 10000000 | ||
| Eksempel 3 | 0.002 g | 6 | 200 | 30 | 1000 | 6000000 | ||
| kr. Data vist for 3 prøver | ||||||||
| kr. Baseret på plettering 5 μL x 200 for 1 ml | ||||||||
| kr. Cotyledons er for små til kerne, så koloni tæller blev normaliseret til 0,01 g væv baseret på den gennemsnitlige masse af enMoneyMaker-PtoS cotyledon (data ikke vist) | ||||||||
| kr. Justeret pr. ml baseret på volumenbelagt | ||||||||
Tabel 1: Stikprøveberegninger for vækstanalyse af planter. Prøveberegninger viser, hvordan man normaliserer bakterietal og bestemme log bakterievækst.
- For vilde tiltrædelser og andre linjer med komplekse genetiske baggrunde korrelerer niveauet af bakterievækst i de enkelte planter med deres fænotype som beskrevet i afsnit 11.
11. Fænobiologi for modstand
- Fjern pladerne fra vækstkammeret og fænotype individuelle planter til død (på grund af sygdom) eller overlevelse (på grund af resistens) efter 7-14 dage.
- Fænotype planter inficeret med en meget virulent stamme som PstDC3000 tidligere, på 7-10 dage efter oversvømmelse vaccination.
- Fænotype planter inficeret med PstT1 på 10-14 dage efter oversvømmelse inokulering.
- Bestemme et pointsystem baseret på det observerede område af resistensfænotyper. Optag binære fænotyper for sorter, isogene linjer og vilde tiltrædelser med konsekvente, stærke til mellemliggende resistensfænotyper (Figur 4A, 4B).
- Hvis sætteplante viser ny vækst fra den apikale meristem inden for tidsrammen for fænostoling, tælle det som en overlevelse. Hvis sætteplanten har en brun apiisk meristem og ikke viser nogen ny, grøn vegetativ vækst, tælle det som en død (Figur 3).

Figur 3: Skematisk repræsentation af en tomatsætteplante. Forskellige dele af en tomat sætteplante er afbildet, herunder hypocotyl, cotyledons, epicotyl, skyde apical meristem, og sande blade. Klik her for at se en større version af dette tal.
- Registrer fænotyper på et sygdomsspektrum for populationer, såsom F2-kortlægningspopulationer, med en bred vifte af resistensfænotyper (figur 4C).
- Nøje overvåge kimplanter for udseendet af sygdomssymptomer og død for at identificere det relevante vindue til fænotypebestemmelse.

Figur 4: Skematisk repræsentation af forventede fænotyper til kimresistens og død i forskellige genetiske baggrunde. (A) Frøplanter af Rio Grande-PtoR og den næsten isogene sort Rio Grande-PtoS vises 7 dage efter oversvømmelse med PstDC3000 (OD600 = 0,005) + 0,015% overfladeaktivt stof. Rio Grande-PtoR udviser konsekvent modstand, og Rio Grande-PtoS udviser konsekvent følsomhed over for infektion med PstDC3000. Disse linjer giver anledning til diskrete og binære fænotyper. (B) Frøplanter af en vild tiltrædelse, såsom Solanum neorickii LA1329, vises 10 dage efter oversvømmelse med PstT1 (OD600 = 0,0075) + 0,015% overfladeaktivt stof. Kimplanter viser fænotypiske variabilitet, men blev registreret som binære fænotyper. Mængden af fænotypisk variabilitet og fænotypebestemmelsesmetoden (binær modstand eller modstandsspektrum) vil afhænge af den specifikke tiltrædelsestest. (C)Kortlægning af populationer, der genereres ved udpassage af vilde tiltrædelser til modtagelige sorter, kan vise et bredere spektrum af fænotyper i F2-adskillelsespopulationer. I dette tilfælde kan det være mest hensigtsmæssigt at registrere sætteplantefænotyper på et spektrum. Meget modtagelige planter fra en kortlægning befolkning kan fænoskrives til døden så tidligt som dag 7, når oversvømmet med PstT1, og typisk viser en brun apilogisk meristem, nej til meget lidt udvidelse af epicotyl, og ingen nye, grønne vegetativ vækst. Den apikale meristem af modtagelige planter kan forblive grøn eller meget lys brun i mere tid, og der kan være en vis forlængelse af epicotyl og meget lidt vegetativ vækst, som bliver brun og anholdelser af dag 10. Individuelle planter kan fænoskrives for resistens baseret på mængden af nye og igangværende vegetativ vækst på dag 14. Kimplanter kan derefter grupperes baseret på de fænotyper, der er beskrevet ovenfor, i forskellige kategorier af resistens såsom svag, medium eller stærk modstand. Klik her for at se en større version af dette tal.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Påvisning af PtoR-medieretimmunitet i sorter og isogene linjer ved hjælp af sætteplantemodstandsanalysen
Figur 5 viser repræsentative resultater for Moneymaker-PtoR og Moneymaker-PtoS kultivarer 7-10 dage efter oversvømmelse med PstDC3000. Før infektion, 10-dages gamle planter vises fuldt opstået og udvidet cotyledons og nye første sande blade. Frøplanterne blev oversvømmet med 10 mM MgCl2 + 0,015% overfladeaktivt stof som et negativt kontrol (data ikke vist) og PstDC3000 med en optisk tæthed på 0,005 + 0,015% overfladeaktivt stof. Frøplanterne blev fænotype7-10 dage efter oversvømmelse (Figur 5). Individuelle kimplanter fra genotypisk homogene linjer, såsomMoneymaker-PtoR og Moneymaker-PtoS giver meget konsekvent og binære fænotyper i sætteplante oversvømmelse assay. NårMoneymaker-PtoR, som bærer Pto / Prf genklynge (n = 5), blev behandlet med PstDC3000 ved den optimale koncentration af OD600 = 0,005, modstand på grund af PtoR-medieretimmunitet var stærk og blev karakteriseret ved nye, grønne vegetativ vækst i alle individer22. Nær-isogeneMoneymaker-PtoS planter (n = 5), som ikke kan genkende PstDC3000 effektorer AvrPto eller AvrPtoB, døde hurtigt inden for 7 dage efter oversvømmelser og karakteristisk havde brun apical meristems, bakteriel plet, chlorose, og ingen tegn på nye, grønne vegetativ vækst (Figur 5).

Figur 5: Fænotypisk karakterisering af resistens- eller sygdomssymptomer 7-10 dage efter infektion i en sort. Moneymaker-PtoR og Moneymaker-PtoS tomat planter blev dyrket på 0,5 x MS plader i 10 dage, før de blev oversvømmet med P. syringae pv. Tomat DC3000 (OD600 = 0,005) + 0,015% overfladeaktivt stof. Moneymaker-PtoR planter overlevede (n = 5) og Moneymaker-PtoS planter (n = 5) døde. Antallet af overlevende frøplanter for hver genotype ud af det samlede antal testede er vist. Skalabar = 1 cm. Klik her for at se en større version af dette tal.
Fænotypisk screening af vilde tiltrædelser ved hjælp af kimmodstandsanalysen
Figur 6 viser repræsentative resultater for frøplanter af modtagelige og resistente tiltrædelser 10-14 dage efter oversvømmelse med PstT1. Modtagelige tiltrædelser omfatter RG-PtoR, S. pimpinellifolium LA1375, og S. pimpinellifolium LA1606, og resistente tiltrædelser omfatter S. neorickii LA1329. Ti-dages gamle planter blev oversvømmet med 10 mM MgCl2 + 0,015% overfladeaktivt stof som en negativ kontrol, og PstT1 ved en optisk tæthed på 0,0075 + 0,015% overfladeaktivt stof. De planter blev fænotyped mindst 10 dage efter oversvømmelser, som PstT1-inficerede planter døde langsommere end PstDC3000-inficerede planter. Mock-podede planter var grønne, sunde og aktivt voksende. Denne kontrol er vigtig for at sikre, at tiltrædelserne ikke er følsomme over for koncentrationen af overfladeaktive stoffer, og for at sikre, at der ikke er nogen bakteriel kontaminering. Modtagelige tiltrædelser (Rio Grande-PtoR [n = 7], S. pimpinellifolium LA1375 [n = 7], og S. pimpinellifolium LA1606 [n = 5]) var døde, havde brun apical meristems, og manglede ny vækst 10-14 dage efter vaccination med PstT1. I modsætning hertil viste to S. neorickii LA1329 (n = 3) kimplanter et højt niveau af ny, grøn vækst og overlevede infektion med PstT1 (Figur 6). Tre LA1329 frøplanter spirede ikke. Typisk blev 5-7 personer screenet for hver tiltrædelse i en primær skærm for at bestemme forekomsten af resistens i befolkningen. Når en mere genetisk kompleks vilde tiltrædelse, såsom LA1329, er oversvømmet med PstT1, modstanden fænotyper vise lidt mere variation blandt de enkelte planter, sammenlignet med Moneymaker-PtoR behandlet med PstDC3000. Modstandsfænotyperne var dog normalt mindre variable end dem, der blev set i F2-kortlægningspopulationer. Således blev der anvendt binære fænosningkriterier for LA1329.

Figur 6: Fænotypisk karakterisering af resistens- eller sygdomssymptomer 10-14 dage efter infektion i vilde tiltrædelser. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 og S. neorickii LA1329 tomatplanter blev dyrket på 0,5 x MS plader i 10 dage, og derefter oversvømmet med PstT1 (OD600 = 0,0075) + 0,015% overfladeaktive stoffer. Antallet af overlevende planter for hver vild tiltrædelse ud af det samlede antal testede er vist. Skalabar = 1 cm. Klik her for at se en større version af dette tal.
Kvantitativ vurdering af bakterievækst ved hjælp af kimen oversvømmelse assay
For at bekræfte, at den observerede resistens i LA1329 til PstT1 resulterede i lavere bakterievækst, bakterievækst assays blev udført i tomat planter. Niveauet af PstT1 vækst i Moneymaker-PtoS og S. neorickii LA1329 blev bestemt 4 dage efter infektion. Moneymaker-PtoS er en næsten isogenlinje med konsekvent modtagelighed blandt de enkelte planter. Vilde tiltrædelser som S. neorickii LA1329 er ofte mere genetisk komplekse. LA1329 viser ca. 60 % modstandsdygtighed over for PstT1 i befolkningen22. Fordi kimplanter kan droppe deres cotyledons efter infektion, en kimplante blev dyrket på hver plade til at korrelere bakterievækst i den høstede cotyledon med samlede kimplanter overlevelse eller død som bestemmes fænotypisk mindst 10 dage efter oversvømmelse. Bakterieten tal på dag 4 for hver sætteplante blev normaliseret til 0,01 g af væv og konverteret til log vækst (CFU/0,01 g (log10)). Log vækst for fænotypisk resistente LA1329 planter (LA1329RES) eller fænotypisk modtagelige planter (LA1329SUS) blev separat samlet og sammenlignet med hinanden og den modtagelige sort Moneymaker-PtoS . For eksempel var der en 1,7 log forskel i bakterievækst mellem LA1329RES (log 6.3) og LA1329SUS (log 8.0), og en 1,6 log forskel mellem LA1329RES (log 6.3) og Moneymaker-PtoS (log 7.9) (Figur 7). Derfor korreleret fænotypisk resistens med kvantitativ resistens i sætteplantningsanalyserne.

Figur 7: Resistente Solanum neorickii LA1329 planter understøtter lavere bakterievækst end Moneymaker-PtoS eller modtagelige S. neorickii LA1329. Bakterietal blev bestemt 4 dage efter podning fra S. neorickii LA1329 (n = 14) og Moneymaker-PtoS (n = 10) kimplanter inficeret med PstT1 og normalisering blev udført til 0,01 g væv. For LA1329 blev de to fænotypiske grupper, modtagelige (SUS) eller resistente (RES), observeret og talt separat. Over bjælken * = statistisk signifikant forskel bestemt af en en-faktor analyse af variansen. Der blev anvendt en generel lineær modelprocedure (p < 0,001) efterfulgt af en flere sammenligninger af midler ved hjælp af Tukey's post hoc-test. Fejllinjer = standardfejl. Tallet angiver ét repræsentativt eksperiment. Klik her for at se en større version af dette tal.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
En protokol for oversvømmelse inokulering med PstDC3000 eller Pst T1 optimerettil at opdage resistens over for disse bakterielle stammer i tomat planter er beskrevet. Der er flere kritiske parametre for optimale resultater i sætteplanteresistensanalysen, herunder bakteriekoncentration og overfladeaktivt stofkoncentration, som empirisk blev bestemt22. For PstDC3000 blev den optiske tæthed optimeret til at opnå fuldstændig overlevelse på en resistent sort, der indeholder Pto/Prf-klyngen og fuldstændig død på en modtagelig kultivar, der manglede Pto/Prf-klyngen 22. For en stamme som PstT1, hvor der ikke er nogen kendte resistente sorter, den optiske tæthed blev optimeret til at være den lavest mulige for konsekvent og komplet plantedød22. Uppalapati et al.24 designet en tomat sætteplante assay at undersøge patogenesen af PstDC3000 og virulens funktion af koroninans. I denne virulens assay, infektioner blev udført ved hjælp af bakterier koncentreret til en OD600 af 0,124, 20x højere end den optiske tæthed af stammer, der anvendes i vores modstand assay. Anerkendelse af PstDC3000 effektorer AvrPto og AvrPtoB i tomatplanter bærer Pto / Prf genklyngen resulterer i ETI og en makroskopisk HR22. I forbindelse med et stærkt immunrespons som ETI blev der anvendt en lavere baktier til PstDC3000 for at undgå overvældende genetisk resistens fra Pto/Prf-genklyngen 22. Desuden tyder disse resultater på, at en høj bakteriekoncentration kan overvælde svagere immunrespons såsom PTI eller kvantitativ delvis resistens, hvor flere gener bidrager til den samlede fænotype. Overfladeaktivt stof er nødvendigt for bakterierne til at overholde bladets overflade; høje koncentrationer kan dog forårsage klorose i bladet22. Vi har tidligere testet en række overfladeaktive koncentrationer for empirisk bestemme den ideelle koncentration i 10-dages-gamle tomat planter22. Ved test af nye arter, der kan variere i deres følsomhed over for overfladeaktivt stof, bør koncentrationen af overfladeaktive stoffer optimeres til at identificere en koncentration, der ikke forårsager skade eller chlorosis, hvis der ikke er bakterier. Passende analyse betingelser vil kræve optimering af en overfladeaktivt stof koncentration, der ikke forårsager skade, og en bakteriel koncentration, der forårsager sygdom i alle modtagelige kontroller.
Yderligere kritiske parametre for succes i sætteplante oversvømmelse assay omfatter brug af planter på specifikke udviklingsstadier (10-dages gamle planter) (Figur 1),opretholde stabile vækstkammer betingelser (lysintensitet på omkring 200 μE m-2 s-1,konstant temperatur på 22 °C, 16 h lys) og udføre forsøg i en steril biosikkerhed kabinet. Medievolumen over 45 ml eller under 35 ml kan påvirke den konsekvente død af modtagelige kontroller, da volumen kan påvirke det omgivende mikromiljø af frøplanterne på pladen. For eksempel kan forskelle i relativ luftfugtighed inde i de forseglede plader påvirke bakteriernes infektivitet og planternes evne til at overleve infektionen. Steril teknik er kritisk, fordi kontaminering på pladerne kan forvirre kilden til død eller modtagelighed i planter. Hertil kommer, fordi plante-patogen interaktioner er påvirket af døgnrytmen ur24,25,26, anbefales det, at planterne blive smittet på et ensartet tidspunkt af dagen.
Pst er et bladpatogen, der fortrinsvis koloniserer luftdelene af tomatplanter, herunder cotyledons24 (Figur 3). Derfor, kvalitativfægtning i sætteplante oversvømmelse assay fokuserer på vækst og sygdom symptomer i luften dele af sætteplante, og væv til bakterievækst assay er udtaget fra cotyledons til kvantitativ analyse. Efter oversvømmelsesvaccination kan frøplanter dø inden for 7-10 dage efter inokulering med PstDC3000 eller 10-14 dage efter vaccination med PstT1, som beskrevet i afsnit 11. Sætteplante død er visualiseret af en brun apikale meristem, anholdt epicotyl forlængelse, og / eller anholdt vegetativ vækst. Hvis der anvendes forskellige bakteriestammer, skal timingen empires. Desuden bør udviklingen af sygdom på kontrolplanter overvåges dagligt efter oversvømmelse, indtil der kan identificeres en ensartet tidsramme fra sygdomssymptomernes indtræden til kimdød. Afhængigt af de genotyper og behandlinger, der anvendes i oversvømmelsesanalysen, kan sætteplantefænotyper registreres som binære fænotyper eller på et sygdomsspektrum (figur 4). Et bredere spektrum af fænotyper kan observeres, når oversvømmelsesinokulerende F2-kortlægning af populationer fra vilde tomattiltiltrædelser krydses til modtagelige sorter (figur 4C). Det kan være bedst at fætre populationer på et sygdomsspektrum, afhængigt af hvor hurtigt sætteplanten dør, og graden af ny vegetativ vækst og forgrening (figur 4C). Den sætteplante oversvømmelse assay kan også bruges i forbindelse med sætteplante bakterievækst assay at kvantitativt vurdere niveauer af bakterievækst forbundet med kvalitative fænotyper i de enkelte planter (Figur 7). Meget store reduktioner (dvs. ~ log 3) i bakterievækst eller stærk resistens i resistente planter af en vild tiltrædelse i forhold til en modtagelig kultivar tyder på, at det underliggende genetiske grundlag for resistens kan skyldes ETI22. Mindre reduktioner i bakterievækst (dvs. ~log 1.7), som observeret i LA1329 frøplanter, kan skyldes bidraget fra svagere resistens fra kvantitative træk loci og / eller PTI. Således kan sætteplante vækst analyse være et vigtigt redskab i yderligere at karakterisere modstand i vilde tomat linjer.
Typisk er genetiske skærme blevet udført på fire- til fem uger gamle voksne tomatplanter for at identificere det genetiske grundlag for P. syringae resistens i vilde tiltrædelser20,21. Voksne tomatplanter kræver meget længere væksttider, kræver mere plads i vækstkammeret, og er meget større planter, hvilket betyder, at normalt få individer screenes for hver linje. Den sætteplante oversvømmelse assay giver en kraftfuld, alternativ tilgang i identifikationen af P. syringae modstand i vilde tomat tiltrædelser. Screening på sætteplanstadiet gør det muligt at afprøve en stor stikprøvestørrelse, som kan være særlig fordelagtig med hensyn til påvisning af resistens i genetisk komplekse populationer. Reducerede krav til vækstkammerplads og væksttid letter en tilgang med høj gennemløb og hurtig påvisning af naturlig resistens i vilde tiltrædelser til nye patogener. Endvidere er P. syringae modstand, der blev identificeret på sætteplantestadiet i denne analyse ikke begrænset til udviklingsfasen. S. neorickii LA1329 og S. habrochaites LA1253 blev oprindeligt identificeret på sætteplantestadiet og også vise resistens over for PstT1 i voksne planter som tidligere beskrevet22.
Den sætteplante oversvømmelse assay er en alsidig protokol, der kan ændres og optimeres til at opdage vært modstand mod andre P. syringae stammer. Det kunne potentielt anvendes yderligere i forbindelse med forskellige bakterielle patogener af tomat, såsom Xanthomonas arter. Denne metode vil fremskynde søgningen efter nye kilder til sygdomsresistens over for bakterielle patogener.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Forfatterne har intet at afsløre.
Acknowledgments
Vi takker Jamie Calma for at teste effekten af medievolumen på sygdom eller resistens resultater. Vi takker Dr. Maël Baudin og Dr. Karl J. Scheiber fra Lewis Lab for at give konstruktive kommentarer og forslag til manuskriptet. Forskning i planteimmunitet i Lewis laboratoriet blev støttet af USDA ARS 2030-21000-046-00D og 2030-21000-050-00D (JDL), og NSF Direktoratet for BiologiskE Videnskaber IOS-1557661 (JDL).
Materials
| Name | Company | Catalog Number | Comments |
| 3M Tape Micropore 1/2" x 10 YD CS 240 (1.25 cm x 9.1 m) | VWR International | 56222-182 | |
| 3mm borosilicate glass beads | Friedrich & Dimmock | GB3000B | |
| Bacto peptone | BD | 211677 | |
| Bacto agar | BD | 214010 | |
| Biophotometer Plus | Eppendorf | E952000006 | |
| Biosafety cabinet, class II type A2 | |||
| BRAND Disposable Plastic Cuvettes, Polystyrene | VWR International | 47744-642 | |
| Chenille Kraft Flat Wood Toothpicks | VWR International | 500029-808 | |
| cycloheximide | Research Products International | C81040-5.0 | |
| Dibasic potassium phosphate anhydrous, ACS grade | Fisher Scientific | P288-500 | |
| Dimethylformamide | |||
| Dissecting microscope (Magnification of at least 10x) | |||
| Ethanol - 190 Proof | |||
| Falcon polystyrene 96 well microplates, flat-bottom | Fisher Scientific | 08-772-3 | |
| Glass Alcohol Burner Wick | Fisher Scientific | S41898A / No. W-125 | |
| Glass Alcohol Burners | Fisher Scientific | S41898 / No. BO125 | |
| Glycerol ACS reagent | VWR International | EMGX0185-5 | |
| Kimberly-Clark™ Kimtech Science™ Kimwipes™ Delicate Task Wipers | Fisher Scientific | 06-666-A | |
| Magnesium chloride, ACS grade | VWR International | 97061-356 | |
| Magnesium sulfate heptahydrate, ACS grade | VWR International | 97062-130 | |
| Microcentrifuge tubes, 1.5 mL | |||
| Microcentrifuge tubes, 2.2 mL | |||
| Mini Beadbeater-96, 115 volt | Bio Spec Products Inc. | 1001 | |
| Murashige & Skoog, Basal Salts | Caisson Laboratories, Inc. | MSP01-50LT | |
| Pipet-Lite XLS LTS 8-CH Pipet 20-200uL | Rainin | L8-200XLS | |
| Pipet-Lite XLS LTS 8-CH Pipet 2-20uL | Rainin | L8-20XLS | |
| Polystyrene 100mm x 25mm sterile petri dish | VWR International | 89107-632 | |
| Polystyrene 150mm x 15mm sterile petri dish | Fisher Scientific | FB08-757-14 | |
| Polystyrene 150x15mm sterile petri dish | Fisher Scientific | 08-757-148 | |
| Pure Bright Germicidal Ultra Bleach 5.7% Available Chlorine (defined as 100% bleach) | Staples | 1013131 | |
| Rifampicin | Gold Biotechnology | R-120-25 | |
| Silwet L-77 (non-ionic organosilicone surfactant co-polymer C13H34O4Si3 surfactant) | Fisher Scientific | NCO138454 | |
| Tips LTS 20 μL 960/10 GPS-L10 | Rainin | 17005091 | |
| Tips LTS 250 μL 960/10 GPS-L250 | Rainin | 17005093 | |
| VWR dissecting forceps fine tip, 4.5" | VWR International | 82027-386 |
References
- Underwood, W., Melotto, M., He, S. Y. Role of plant stomata in bacterial invasion. Cell Microbiology. 9 (7), 1621-1629 (2007).
- Zipfel, C. Early molecular events in PAMP-triggered immunity. Current Opinion in Plant Biology. 12 (4), 414-420 (2009).
- Galan, J. E., Wolf-Watz, H. Protein delivery into eukaryotic cells by type III secretion machines. Nature. 444 (7119), 567-573 (2006).
- Lewis, J. D., Desveaux, D., Guttman, D. S. The targeting of plant cellular systems by injected type III effector proteins. Seminars in Cell and Developmental Biology. 20 (9), 1055-1063 (2009).
- Schreiber, K. J., Baudin, M., Hassan, J. A., Lewis, J. D. Die another day: molecular mechanisms of effector-triggered immunity elicited by type III secreted effector proteins. Seminars in Cell and Developmental Biology. 56, 124-133 (2016).
- Heath, M. C. Hypersensitive response-related death. Plant Molecular Biology. 44 (3), 321-334 (2000).
- Boyd, L. A., Ridout, C., O'Sullivan, D. M., Leach, J. E., Leung, H. Plant-pathogen interactions: disease resistance in modern agriculture. Trends in Genetics. 29 (4), 233-240 (2013).
- Pitblado, R. E., MacNeill, B. H. Genetic basis of resistance to Pseudomonas syringae pv. tomato in field tomatoes. Canadian Journal of Plant Pathology. 5 (4), 251-255 (1983).
- Pedley, K. F., Martin, G. B. Molecular basis of Pto-mediated resistance to bacterial speck disease in tomato. Annual Reviews of Phytopathology. 41, 215-243 (2003).
- Ronald, P. C., Salmeron, J. M., Carland, F. M., Mehta, A. Y., Staskawicz, B. J. The cloned avirulence gene AvrPto induces disease resistance in tomato cultivars containing the Pto resistance gene. Journal of Bacteriology. 174 (5), 1604-1611 (1992).
- Martin, G. B., et al. Map-based cloning of a protein kinase gene conferring disease resistance in tomato. Science. 262 (5138), 1432-1436 (1993).
- Salmeron, J. M., Barker, S. J., Carland, F. M., Mehta, A. Y., Staskawicz, B. J. Tomato mutants altered in bacterial disease resistance provide evidence for a new locus controlling pathogen recognition. Plant Cell. 6 (4), 511-520 (1994).
- Salmeron, J. M., et al. Tomato Prf is a member of the leucine-rich repeat class of plant disease resistance genes and lies embedded within the Pto kinase gene cluster. Cell. 86 (1), 123-133 (1996).
- Scofield, S. R., et al. Molecular basis of gene-for-gene specificity in bacterial speck disease of tomato. Science. 274 (5295), 2063-2065 (1996).
- Kunkeaw, S., Tan, S., Coaker, G. Molecular and evolutionary analyses of Pseudomonas syringae pv. tomato race 1. Molecular Plant-Microbe Interactions. 23 (4), 415-424 (2010).
- Cai, R., et al. The plant pathogen Pseudomonas syringae pv. tomato is genetically monomorphic and under strong selection to evade tomato immunity. PLoS Pathogens. 7 (8), 1002130 (2011).
- Almeida, N. F., et al. A draft genome sequence of Pseudomonas syringae pv. tomato T1 reveals a type III effector repertoire significantly divergent from that of Pseudomonas syringae pv. tomato DC3000. Molecular Plant-Microbe Interactions. 22 (1), 52-62 (2009).
- Lin, N. C., Abramovitch, R. B., Kim, Y. J., Martin, G. B. Diverse AvrPtoB homologs from several Pseudomonas syringae pathovars elicit Pto-dependent resistance and have similar virulence activities. Applied and Environmental Microbiology. 72 (1), 702-712 (2006).
- Rose, L. E., Langley, C. H., Bernal, A. J., Michelmore, R. W. Natural variation in the Pto pathogen resistance gene within species of wild tomato (Lycopersicon). I. Functional analysis of Pto alleles. Genetics. 171 (1), 345-357 (2005).
- Thapa, S. P., Miyao, E. M., Davis, R. M., Coaker, G. Identification of QTLs controlling resistance to Pseudomonas syringae pv. tomato race 1 strains from the wild tomato Solanum habrochaites LA1777. Theoretical and Applied Genetics. 128 (4), 681-692 (2015).
- Bao, Z. L., et al. Identification of a candidate gene in Solanum habrochaites for resistance to a race 1 strain of Pseudomonas syringae pv. tomato. Plant Genome. 8 (3), 1-15 (2015).
- Hassan, J. A., Zhou, Y. J., Lewis, J. D. A rapid seedling resistance assay identifies wild tomato lines that are resistant to Pseudomonas syringae pv. tomato race 1. Molecular Plant-Microbe Interactions. 30 (9), 701-709 (2017).
- King, E. O., Ward, M. K., Raney, D. E. Two simple media for the demonstration of pyocyanin and fluorescin. Journal of Laboratory and Clinical Medicine. 44 (2), 301-307 (1954).
- Uppalapati, S. R., et al. Pathogenicity of Pseudomonas syringae pv. tomato on tomato seedlings: phenotypic and gene expression analyses of the virulence function of coronatine. Molecular Plant-Microbe Interactions. 21 (4), 383-395 (2008).
- Bhardwaj, V., Meier, S., Petersen, L. N., Ingle, R. A., Roden, L. C. Defence responses of Arabidopsis thaliana to infection by Pseudomonas syringae are regulated by the circadian clock. PLoS One. 6 (10), 26968 (2011).
- Lu, H., McClung, C. R., Zhang, C. Tick tock: circadian regulation of plant innate immunity. Annual Review of Phytopathology. 55, 287-311 (2017).
- Wang, W., et al. Timing of plant immune responses by a central circadian regulator. Nature. 470 (7332), 110-114 (2011).
Tags
Bioengineering Pseudomonas syringae tomat sygdomsresistens skærm bakterievækst fænotyperErratum
Formal Correction: Erratum: High-Throughput Identification of Resistance to Pseudomonas syringae pv. Tomato in Tomato using Seedling Flood Assay
Posted by JoVE Editors on 10/18/2023.
Citeable Link.
An erratum was issued for: High-Throughput Identification of Resistance to Pseudomonas syringae pv. Tomato in Tomato using Seedling Flood Assay. The Introduction, Protocol, Representative Results and Discussion sections were updated.
The last paragraph of the Introduction section was updated from:
In the seedling flood assay described in this protocol, tomato seedlings are grown on Petri dishes of sterile Murashige and Skoog (MS) media for 10 days and then are flooded with an inoculum containing the bacteria of interest and a surfactant. Following flooding, seedlings can be quantitatively evaluated for disease resistance via bacterial growth assays. Additionally, seedling survival or death can act as a discrete resistance or disease phenotype 7–14 days after flooding. This approach offers a high-throughput alternative for screening large numbers of wild tomato accessions for resistance to Pst race 1 strains, such as Pst strain T1 (PstT1), and can easily be adapted to other bacterial strains of interest.
to:
In the seedling flood assay described in this protocol, tomato seedlings are grown on Petri dishes of sterile Murashige and Skoog (MS) media for 10 days and then are flooded with an inoculum containing the bacteria of interest and a surfactant. Following flooding, seedlings can be quantitatively evaluated for disease resistance via bacterial growth assays. Additionally, seedling survival or death can act as a discrete resistance or disease phenotype 7–14 days after flooding. This approach offers a high-throughput alternative for screening large numbers of wild tomato accessions for resistance to Pst race 1 strains, such as Pst strain 19 (Pst19), and can easily be adapted to other bacterial strains of interest.
Step 4.8 of the Protocol section was updated from:
- After 1 h, transfer the bottle to the biosafety cabinet and under aseptic conditions, add 1,600 µL of sterile 1 M MgSO4, and appropriate antibiotics to the media.
NOTE: For rifampicin resistant strains PstDC3000 and PstT1, use rifampicin dissolved in dimethylformamide at a final concentration of 50 µg/mL. Use cycloheximide dissolved in ethanol at a final concentration of 50 µg/mL to prevent fungal growth on the plates.
to:
- After 1 h, transfer the bottle to the biosafety cabinet and under aseptic conditions, add 1,600 µL of sterile 1 M MgSO4, and appropriate antibiotics to the media.
NOTE: For rifampicin resistant strains PstDC3000 and Pst19, use rifampicin dissolved in dimethylformamide at a final concentration of 50 µg/mL. Use cycloheximide dissolved in ethanol at a final concentration of 50 µg/mL to prevent fungal growth on the plates.
Step 5.2 of the Protocol section was updated from:
- Patch bacteria (i.e., PstT1) from a glycerol stock onto KB agar with appropriate antibiotics (section 4).
to:
- Patch bacteria (i.e., Pst19) from a glycerol stock onto KB agar with appropriate antibiotics (section 4).
Step 5.6 of the Protocol section was updated from:
- For PstT1, incubate the KB plate at 28 ˚C for 48 h prior to using bacteria in the flood experiment.
to:
- For Pst19, incubate the KB plate at 28 ˚C for 48 h prior to using bacteria in the flood experiment.
Step 6 of the Protocol section was updated from:
6. Preparation of PstT1 inoculum
to
6. Preparation of Pst19 inoculum
Step 6.2 of the Protocol section was updated from:
- Perform serial dilutions using sterile 10 mM MgCl2 solution in the biosafety cabinet. For PstT1, use a spectrophotometer to make inoculum with a starting concentration of OD600 = 0.1.
to:
- Perform serial dilutions using sterile 10 mM MgCl2 solution in the biosafety cabinet. For Pst19, use a spectrophotometer to make inoculum with a starting concentration of OD600 = 0.1.
Step 6.3 of the Protocol section was updated from:
- For PstT1, make a 1/10 dilution from the initial resuspension at OD600 = 0.1 to obtain a serial dilution at a concentration of OD600 = 0.01.
to:
- For Pst19, make a 1/10 dilution from the initial resuspension at OD600 = 0.1 to obtain a serial dilution at a concentration of OD600 = 0.01.
Step 8.3 of the Protocol section was updated from:
- Set a timer for 3 min. Measure 6 mL of final inoculum (PstT1 OD600 = 0.0075 [section 6] or PstDC3000 OD600 = 0.005 [section 7]) and transfer 6 mL of inoculum to each plate with the 10-day-old seedlings.
to:
- Set a timer for 3 min. Measure 6 mL of final inoculum (Pst19 OD600 = 0.0075 [section 6] or PstDC3000 OD600 = 0.005 [section 7]) and transfer 6 mL of inoculum to each plate with the 10-day-old seedlings.
Step 8.11 of the Protocol section was updated from:
- Phenotype after 7–10 days for PstDC3000 or 10–14 days for PstT1 (section 11). If carrying out bacterial growth assays, collect leaf tissue after 4 days (sections 9 and 10) and then phenotype (section 11). Alternatively, perform phenotypic analysis and bacterial growth assays on separate sets of plants.
to:
- Phenotype after 7–10 days for PstDC3000 or 10–14 days for Pst19 (section 11). If carrying out bacterial growth assays, collect leaf tissue after 4 days (sections 9 and 10) and then phenotype (section 11). Alternatively, perform phenotypic analysis and bacterial growth assays on separate sets of plants.
Step 10.7 of the Protocol section was updated from:
- After obtaining colony counts (Figure 2B), normalize the counts to 0.01 g of tissue for seedlings and convert to log bacterial growth (Table 1).
NOTE: The average mass of one Moneymaker-PtoS cotyledon is 0.01 g and is empirically determined22.
| Genotype1 Column A | Tissue Weight (g) Column B | # of Colonies in a spot Column C | Dilution factor for spot2 Column D | Adjusted # of Colonies3 Column E | Dilution factor for serial dilution Column F | Total # of Colonies Column G (cfu/0.01 g)4 | Average # of Colonies (cfu/0.01 g) Column H | Average Log Growth (cfu/0.01 g (log10)) Column I |
| Sample 1 | 0.004 g | 10 | 200 | calculated as: (C2 x 0.01 g) / B2 = 25 | 1000 | calculated as: (D2 x E2 x F2) = 5000000 | average for sample 1 through last sample: (ie. average G1:G3) = 7000000 | log of average ie. log(H2) = 6.85 |
| Sample 2 | 0.003 g | 15 | 200 | 50 | 1000 | 10000000 | ||
| Sample 3 | 0.002 g | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1Data shown for 3 samples | ||||||||
| 2Based on plating 5 µL x 200 for 1 mL | ||||||||
| 3Cotyledons are too small to core so colony counts were normalized to 0.01 g of tissue based on the average mass of one MoneyMaker-PtoS cotyledon (data not shown) | ||||||||
| 4Adjusted per mL based on volume plated | ||||||||
Table 1: Sample calculations for seedling bacterial growth assay. Sample calculations demonstrate how to normalize bacterial counts and determine log bacterial growth.
to:
- After obtaining colony counts (Figure 2B), normalize the counts to 0.1 g of tissue for seedlings and convert to log bacterial growth (Table 1).
NOTE: The average mass of one Moneymaker-PtoS cotyledon is 0.1 g and is empirically determined22.
| Genotype1 Column A | Tissue Weight (g) Column B | # of Colonies in a spot Column C | Dilution factor for spot2 Column D | Adjusted # of Colonies3 Column E | Dilution factor for serial dilution Column F | Total # of Colonies Column G (cfu/0.01 g)4 | Average # of Colonies (cfu/0.01 g) Column H | Average Log Growth (cfu/0.1 g (log10)) Column I |
| Sample 1 | 0.04 g | 10 | 200 | calculated as: (C2 x 0.1 g) / B2 = 25 | 1000 | calculated as: (D2 x E2 x F2) = 5000000 | average for sample 1 through last sample: (ie. average G1:G3) = 7000000 | log of average ie. log(H2) = 6.85 |
| Sample 2 | 0.03 g | 15 | 200 | 50 | 1000 | 10000000 | ||
| Sample 3 | 0.02 g | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1Data shown for 3 samples | ||||||||
| 2Based on plating 5 µL x 200 for 1 mL | ||||||||
| 3Cotyledons are too small to core so colony counts were normalized to 0.1 g of tissue based on the average mass of one MoneyMaker-PtoS cotyledon (data not shown) | ||||||||
| 4Adjusted per mL based on volume plated | ||||||||
Table 1: Sample calculations for seedling bacterial growth assay. Sample calculations demonstrate how to normalize bacterial counts and determine log bacterial growth.
Step 11.3 of the Protocol section was updated from:
- Phenotype plants infected with PstT1 at 10–14 days after flood inoculation.
to:
- Phenotype plants infected with Pst19 at 10–14 days after flood inoculation.
Figure 4 in the Protocol section was updated from:
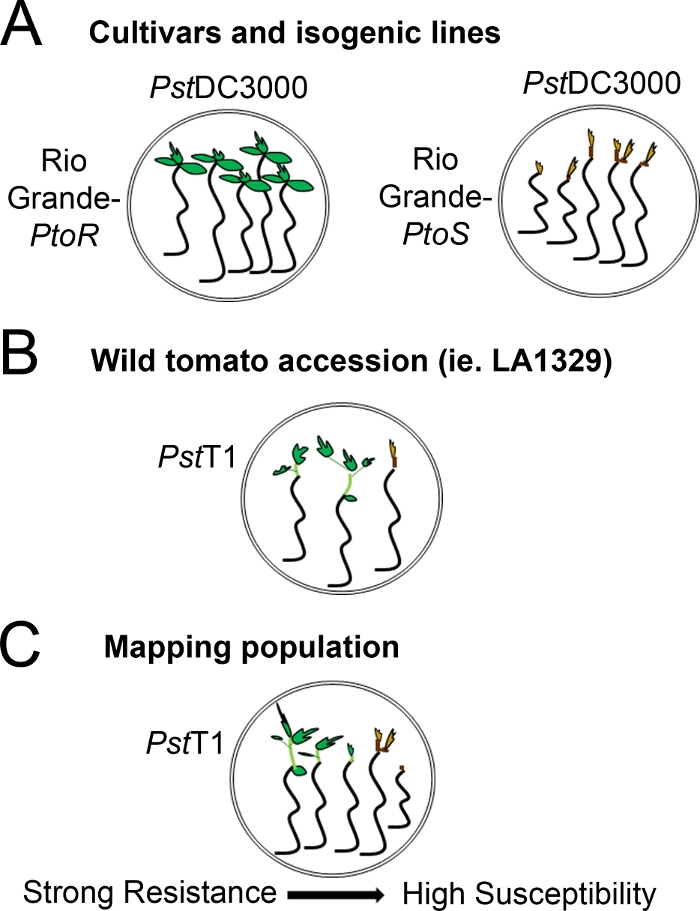
Figure 4: Schematic representation of expected phenotypes for seedling resistance and death in various genetic backgrounds. (A) Seedlings of Rio Grande-PtoR and the near-isogenic cultivar Rio Grande-PtoS are displayed 7 days after flooding with PstDC3000 (OD600 = 0.005) + 0.015% surfactant. Rio Grande-PtoR displays consistent resistance, and Rio Grande-PtoS displays consistent susceptibility to infection with PstDC3000. These lines give rise to discrete and binary phenotypes. (B) Seedlings of a wild accession, such as Solanum neorickii LA1329, are shown 10 days after flooding with PstT1 (OD600 = 0.0075) + 0.015% surfactant. Seedlings display phenotypic variability but were recorded as binary phenotypes. The amount of phenotypic variability and the method of phenotyping (binary resistance or resistance spectrum) will depend on the particular accession tested. (C) Mapping populations generated by outcrossing wild accessions to susceptible cultivars may display a wider spectrum of phenotypes in F2 segregating populations. In this case, it may be most appropriate to record seedling phenotypes on a spectrum. Highly susceptible seedlings from a mapping population may be phenotyped for death as early as day 7 when flooded with PstT1, and typically show a brown apical meristem, no to very little extension of the epicotyl, and no new, green vegetative growth. The apical meristem of susceptible seedlings may stay green or very light brown for more time, and there may be some extension of the epicotyl and very little vegetative growth, which turns brown and arrests by day 10. Individual seedlings can be phenotyped for resistance based on the amount of new and ongoing vegetative growth by day 14. Seedlings can then be grouped based on the phenotypes described above into different categories of resistance such as weak, medium, or strong resistance. Please click here to view a larger version of this figure.
to:
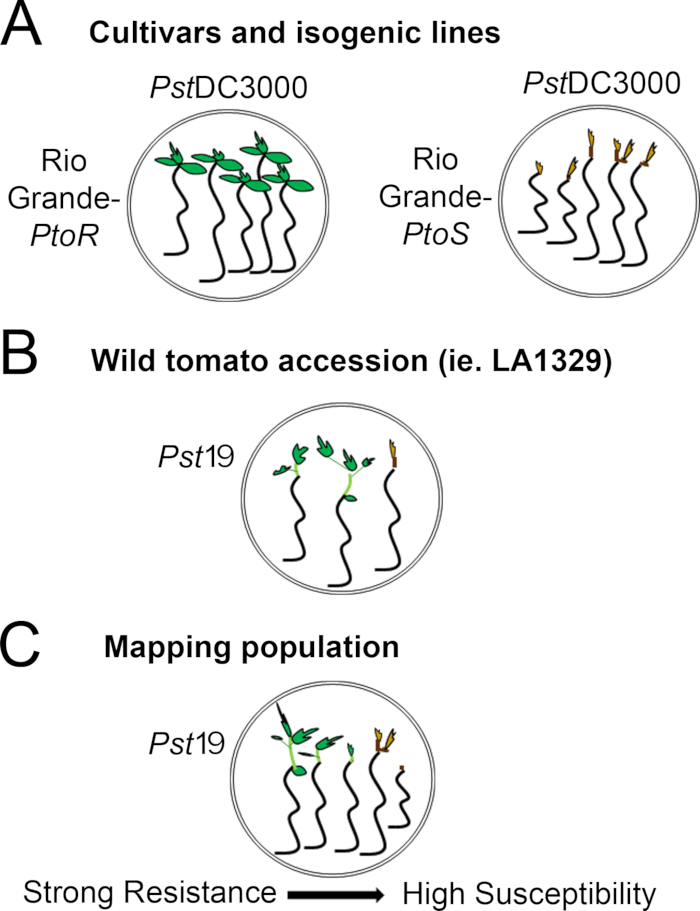
Figure 4: Schematic representation of expected phenotypes for seedling resistance and death in various genetic backgrounds. (A) Seedlings of Rio Grande-PtoR and the near-isogenic cultivar Rio Grande-PtoS are displayed 7 days after flooding with PstDC3000 (OD600 = 0.005) + 0.015% surfactant. Rio Grande-PtoR displays consistent resistance, and Rio Grande-PtoS displays consistent susceptibility to infection with PstDC3000. These lines give rise to discrete and binary phenotypes. (B) Seedlings of a wild accession, such as Solanum neorickii LA1329, are shown 10 days after flooding with Pst19 (OD600 = 0.0075) + 0.015% surfactant. Seedlings display phenotypic variability but were recorded as binary phenotypes. The amount of phenotypic variability and the method of phenotyping (binary resistance or resistance spectrum) will depend on the particular accession tested. (C) Mapping populations generated by outcrossing wild accessions to susceptible cultivars may display a wider spectrum of phenotypes in F2 segregating populations. In this case, it may be most appropriate to record seedling phenotypes on a spectrum. Highly susceptible seedlings from a mapping population may be phenotyped for death as early as day 7 when flooded with Pst19, and typically show a brown apical meristem, no to very little extension of the epicotyl, and no new, green vegetative growth. The apical meristem of susceptible seedlings may stay green or very light brown for more time, and there may be some extension of the epicotyl and very little vegetative growth, which turns brown and arrests by day 10. Individual seedlings can be phenotyped for resistance based on the amount of new and ongoing vegetative growth by day 14. Seedlings can then be grouped based on the phenotypes described above into different categories of resistance such as weak, medium, or strong resistance. Please click here to view a larger version of this figure.
The second paragraph of the Representative Results section was updated from:
Phenotypic screening of wild accessions using the seedling resistance assay
Figure 6 shows representative results for seedlings of susceptible and resistant accessions 10–14 days after flooding with PstT1. Susceptible accessions include RG-PtoR, S. pimpinellifolium LA1375, and S. pimpinellifolium LA1606, and resistant accessions include S. neorickii LA1329. Ten-day-old seedlings were flooded with 10 mM MgCl2 + 0.015% surfactant as a negative control, and PstT1 at an optical density of 0.0075 + 0.015% surfactant. The seedlings were phenotyped at least 10 days after flooding, as PstT1-infected seedlings died more slowly than PstDC3000-infected seedlings. Mock-inoculated seedlings were green, healthy, and actively growing. This control is important to ensure that the accessions are not sensitive to the concentration of surfactant, and to ensure there is no bacterial contamination. Susceptible accessions (Rio Grande-PtoR [n = 7], S. pimpinellifolium LA1375 [n = 7], and S. pimpinellifolium LA1606 [n = 5]) were dead, had brown apical meristems, and lacked new growth 10–14 days after inoculation with PstT1. In contrast, two S. neorickii LA1329 (n = 3) seedlings displayed a high level of new, green growth and survived infection with PstT1 (Figure 6). Three LA1329 seedlings did not germinate. Typically, 5–7 individuals were screened for each accession in a primary screen to determine the prevalence of resistance in the population. When a more genetically complex wild accession, such as LA1329, is flooded with PstT1, the resistance phenotypes display slightly more variability among individual seedlings, compared to Moneymaker-PtoR treated with PstDC3000. However, the resistance phenotypes were usually less variable than those seen in F2 mapping populations. Thus, binary phenotyping criteria was used for LA1329.
to:
Phenotypic screening of wild accessions using the seedling resistance assay
Figure 6 shows representative results for seedlings of susceptible and resistant accessions 10–14 days after flooding with Pst19. Susceptible accessions include RG-PtoR, S. pimpinellifolium LA1375, and S. pimpinellifolium LA1606, and resistant accessions include S. neorickii LA1329. Ten-day-old seedlings were flooded with 10 mM MgCl2 + 0.015% surfactant as a negative control, and Pst19 at an optical density of 0.0075 + 0.015% surfactant. The seedlings were phenotyped at least 10 days after flooding, as Pst19-infected seedlings died more slowly than PstDC3000-infected seedlings. Mock-inoculated seedlings were green, healthy, and actively growing. This control is important to ensure that the accessions are not sensitive to the concentration of surfactant, and to ensure there is no bacterial contamination. Susceptible accessions (Rio Grande-PtoR [n = 7], S. pimpinellifolium LA1375 [n = 7], and S. pimpinellifolium LA1606 [n = 5]) were dead, had brown apical meristems, and lacked new growth 10–14 days after inoculation with Pst19. In contrast, two S. neorickii LA1329 (n = 3) seedlings displayed a high level of new, green growth and survived infection with Pst19 (Figure 6). Three LA1329 seedlings did not germinate. Typically, 5–7 individuals were screened for each accession in a primary screen to determine the prevalence of resistance in the population. When a more genetically complex wild accession, such as LA1329, is flooded with Pst19, the resistance phenotypes display slightly more variability among individual seedlings, compared to Moneymaker-PtoR treated with PstDC3000. However, the resistance phenotypes were usually less variable than those seen in F2 mapping populations. Thus, binary phenotyping criteria was used for LA1329.
Figure 6 in the Representative Results section was updated from:
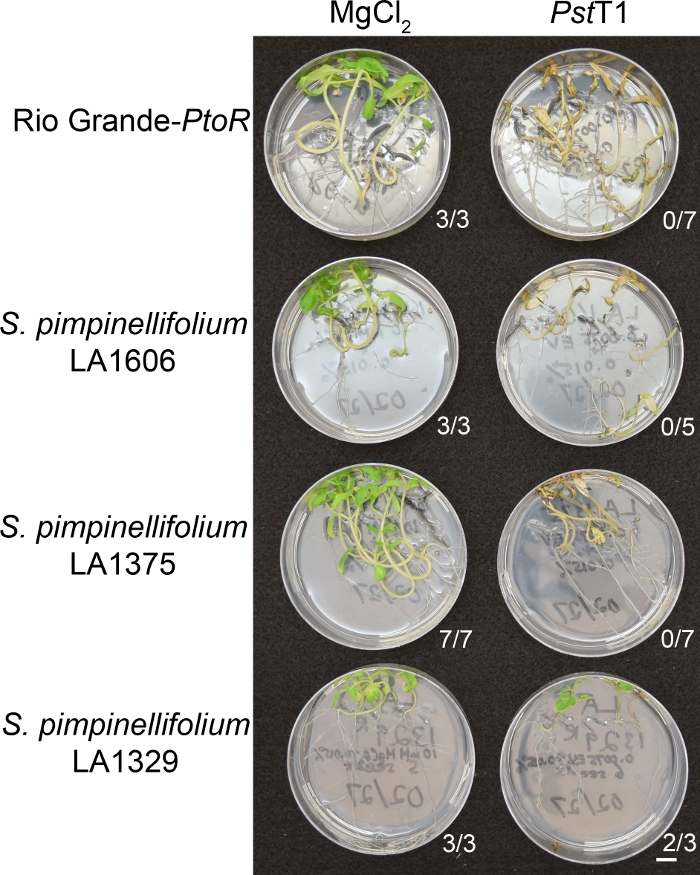
Figure 6: Phenotypic characterization of resistance or disease symptoms 10–14 days post-infection in wild accessions. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 and S. neorickii LA1329 tomato seedlings were grown on 0.5x MS plates for 10 days, and then flooded with PstT1 (OD600 = 0.0075) + 0.015% surfactant. The number of surviving seedlings for each wild accession out of the total number tested is shown. Scale bar = 1 cm. Please click here to view a larger version of this figure.
to:
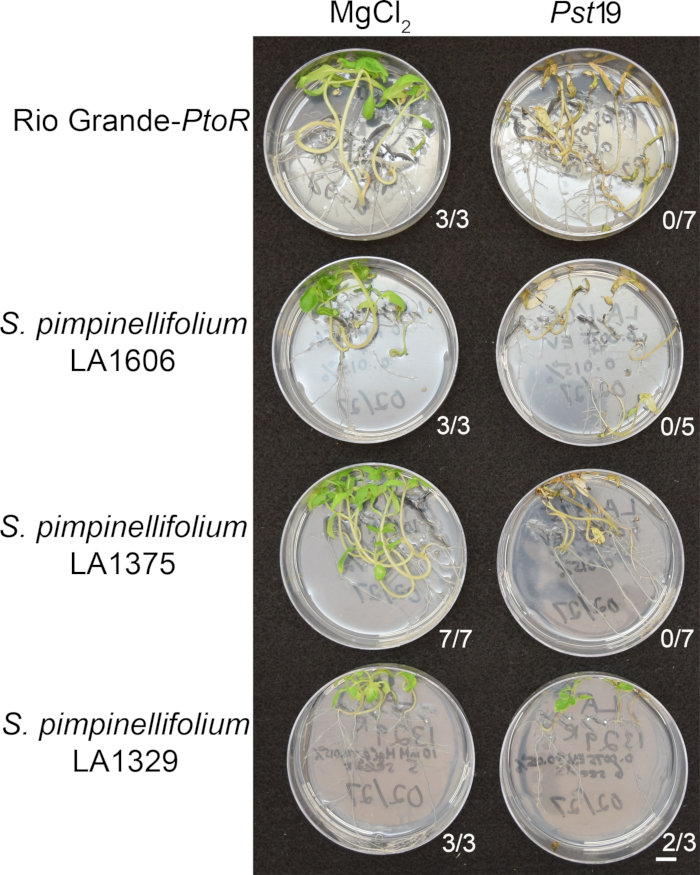
Figure 6: Phenotypic characterization of resistance or disease symptoms 10–14 days post-infection in wild accessions. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 and S. neorickii LA1329 tomato seedlings were grown on 0.5x MS plates for 10 days, and then flooded with Pst19 (OD600 = 0.0075) + 0.015% surfactant. The number of surviving seedlings for each wild accession out of the total number tested is shown. Scale bar = 1 cm. Please click here to view a larger version of this figure.
The third paragraph of the Representative Results section was updated from:
Quantitative assessment of bacterial growth using the seedling flood assay
To confirm that the observed resistance in LA1329 to PstT1 resulted in lower bacterial growth, bacterial growth assays were carried out in tomato seedlings. The level of PstT1 growth in Moneymaker-PtoS and S. neorickii LA1329 was determined 4 days post-infection. Moneymaker-PtoS is a near-isogenic line with consistent susceptibility among individual seedlings. Wild accessions such as S. neorickii LA1329 are often more genetically complex. LA1329 displays approximately 60% resistance to PstT1 across the population22. Because seedlings may drop their cotyledons after infection, one seedling was grown on each plate to correlate bacterial growth in the harvested cotyledon with overall seedling survival or death as determined phenotypically at least 10 days after flooding. The bacterial counts on day 4 for each seedling were normalized to 0.01 g of tissue and converted to log growth (CFU/0.01 g(log10)). Log growth for phenotypically resistant LA1329 seedlings (LA1329RES) or phenotypically susceptible seedlings (LA1329SUS) were separately pooled and compared to each other and the susceptible cultivar Moneymaker-PtoS. For example, there was a 1.7 log difference in bacterial growth between LA1329RES (log 6.3) and LA1329SUS (log 8.0), and a 1.6 log difference between LA1329RES (log 6.3) and Moneymaker-PtoS (log 7.9) (Figure 7). Therefore, phenotypic resistance correlated with quantitative resistance in the seedling assays.
to:
Quantitative assessment of bacterial growth using the seedling flood assay
To confirm that the observed resistance in LA1329 to Pst19 resulted in lower bacterial growth, bacterial growth assays were carried out in tomato seedlings. The level of Pst19 growth in Moneymaker-PtoS and S. neorickii LA1329 was determined 4 days post-infection. Moneymaker-PtoS is a near-isogenic line with consistent susceptibility among individual seedlings. Wild accessions such as S. neorickii LA1329 are often more genetically complex. LA1329 displays approximately 60% resistance to Pst19 across the population22. Because seedlings may drop their cotyledons after infection, one seedling was grown on each plate to correlate bacterial growth in the harvested cotyledon with overall seedling survival or death as determined phenotypically at least 10 days after flooding. The bacterial counts on day 4 for each seedling were normalized to 0.01 g of tissue and converted to log growth (CFU/0.01 g(log10)). Log growth for phenotypically resistant LA1329 seedlings (LA1329RES) or phenotypically susceptible seedlings (LA1329SUS) were separately pooled and compared to each other and the susceptible cultivar Moneymaker-PtoS. For example, there was a 1.7 log difference in bacterial growth between LA1329RES (log 6.3) and LA1329SUS (log 8.0), and a 1.6 log difference between LA1329RES (log 6.3) and Moneymaker-PtoS (log 7.9) (Figure 7). Therefore, phenotypic resistance correlated with quantitative resistance in the seedling assays.
Figure 7 in the Representative Results section was updated from:
x
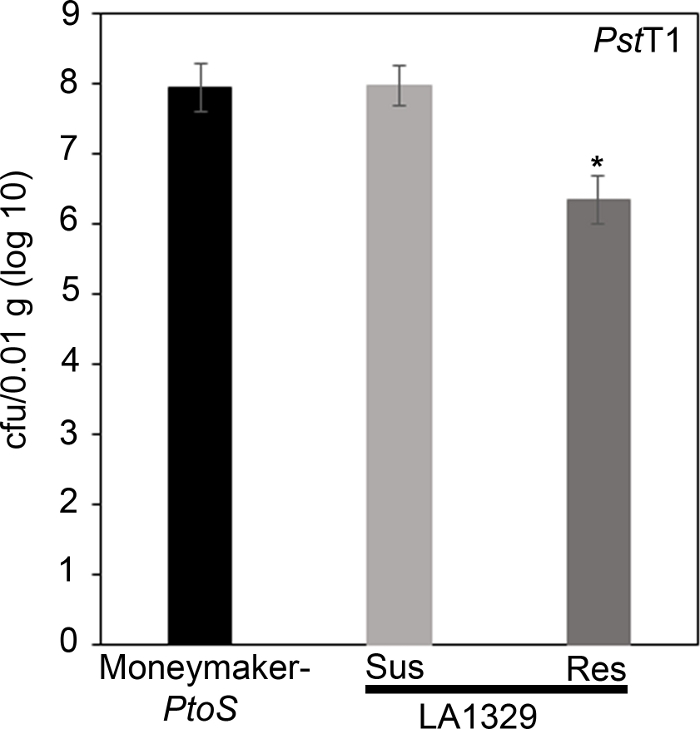
Figure 7: Resistant Solanum neorickii LA1329 seedlings support lower bacterial growth than Moneymaker-PtoS or susceptible S. neorickii LA1329. Bacterial counts were determined 4 days post-inoculation from S. neorickii LA1329 (n = 14) and Moneymaker-PtoS (n = 10) seedlings infected with PstT1 and normalization was performed to 0.01 g of tissue. For LA1329, the two phenotypic groups, susceptible (SUS) or resistant (RES), were observed and counted separately. Above the bar * = statistically significant difference determined by a one-factor analysis of variance. A general linear model procedure (p < 0.001) followed by a multiple comparison of means using Tukey's post hoc test was used. Error bars = standard error. The figure indicates one representative experiment. Please click here to view a larger version of this figure.
x
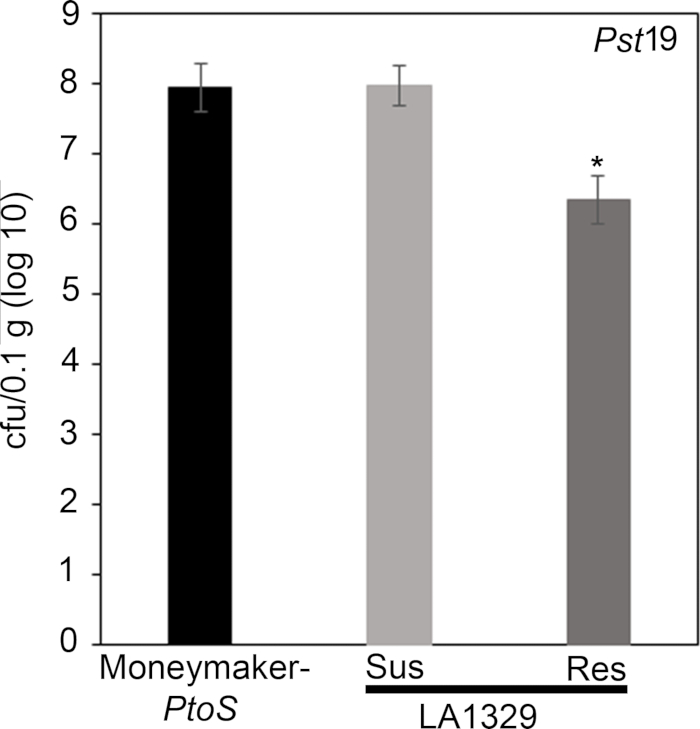
Figure 7: Resistant Solanum neorickii LA1329 seedlings support lower bacterial growth than Moneymaker-PtoS or susceptible S. neorickii LA1329. Bacterial counts were determined 4 days post-inoculation from S. neorickii LA1329 (n = 14) and Moneymaker-PtoS (n = 10) seedlings infected with Pst19 and normalization was performed to 0.1 g of tissue. For LA1329, the two phenotypic groups, susceptible (SUS) or resistant (RES), were observed and counted separately. Above the bar * = statistically significant difference determined by a one-factor analysis of variance. A general linear model procedure (p < 0.001) followed by a multiple comparison of means using Tukey's post hoc test was used. Error bars = standard error. The figure indicates one representative experiment. Please click here to view a larger version of this figure.
The first paragraph of the Discussion section was updated from:
A protocol for flood inoculation with PstDC3000 or PstT1 optimized to detect resistance to these bacterial strains in tomato seedlings is described. There are several critical parameters for optimal results in the seedling resistance assay, including bacterial concentration and surfactant concentration, which were empirically determined22. For PstDC3000, the optical density was optimized to achieve complete survival on a resistant cultivar containing the Pto/Prf cluster and complete death on a susceptible cultivar lacking the Pto/Prf cluster22. For a strain such as PstT1, where there are no known resistant varieties, the optical density was optimized to be the lowest possible for consistent and complete plant death22. Uppalapati et al.24 designed a tomato seedling assay to investigate the pathogenesis of PstDC3000 and the virulence function of coronatine. In this virulence assay, infections were performed using bacteria concentrated to an OD600 of 0.124, 20x higher than the optical density of strains used in our resistance assay. Recognition of PstDC3000 effectors AvrPto and AvrPtoB in tomato seedlings carrying the Pto/Prf gene cluster results in ETI and a macroscopic HR22. In the context of a strong immune response such as ETI, a lower bacterial titer was used for PstDC3000 to avoid overwhelming genetic resistance from the Pto/Prf gene cluster22. In addition, these results suggest that a high bacterial concentration could overwhelm weaker immune responses such as PTI or quantitative partial resistance, where multiple genes contribute to the overall phenotype. Surfactant is necessary for the bacteria to adhere to the leaf surface; however, high concentrations can cause chlorosis of the leaf22. We previously tested a range of surfactant concentrations to empirically determine the ideal concentration in 10-day-old tomato seedlings22. When testing new species that may differ in their sensitivity to surfactant, the surfactant concentration should be optimized to identify a concentration that does not cause damage or chlorosis in the absence of bacteria. Appropriate assay conditions will require optimization of a surfactant concentration that does not cause damage, and a bacterial concentration that causes disease in all susceptible controls.
to:
A protocol for flood inoculation with PstDC3000 or Pst19 optimized to detect resistance to these bacterial strains in tomato seedlings is described. There are several critical parameters for optimal results in the seedling resistance assay, including bacterial concentration and surfactant concentration, which were empirically determined22. For PstDC3000, the optical density was optimized to achieve complete survival on a resistant cultivar containing the Pto/Prf cluster and complete death on a susceptible cultivar lacking the Pto/Prf cluster22. For a strain such as Pst19, where there are no known resistant varieties, the optical density was optimized to be the lowest possible for consistent and complete plant death22. Uppalapati et al.24 designed a tomato seedling assay to investigate the pathogenesis of PstDC3000 and the virulence function of coronatine. In this virulence assay, infections were performed using bacteria concentrated to an OD600 of 0.124, 20x higher than the optical density of strains used in our resistance assay. Recognition of PstDC3000 effectors AvrPto and AvrPtoB in tomato seedlings carrying the Pto/Prf gene cluster results in ETI and a macroscopic HR22. In the context of a strong immune response such as ETI, a lower bacterial titer was used for PstDC3000 to avoid overwhelming genetic resistance from the Pto/Prf gene cluster22. In addition, these results suggest that a high bacterial concentration could overwhelm weaker immune responses such as PTI or quantitative partial resistance, where multiple genes contribute to the overall phenotype. Surfactant is necessary for the bacteria to adhere to the leaf surface; however, high concentrations can cause chlorosis of the leaf22. We previously tested a range of surfactant concentrations to empirically determine the ideal concentration in 10-day-old tomato seedlings22. When testing new species that may differ in their sensitivity to surfactant, the surfactant concentration should be optimized to identify a concentration that does not cause damage or chlorosis in the absence of bacteria. Appropriate assay conditions will require optimization of a surfactant concentration that does not cause damage, and a bacterial concentration that causes disease in all susceptible controls.
The third paragraph of the Discussion section was updated from:
Pst is a foliar pathogen that preferentially colonizes the aerial parts of tomato seedlings, including the cotyledons24 (Figure 3). Therefore, qualitative phenotyping in the seedling flood assay focuses on growth and disease symptoms in aerial portions of the seedling, and tissue for the bacterial growth assay is sampled from the cotyledons for quantitative analysis. After flood inoculation, seedlings may die within 7–10 days after inoculation with PstDC3000 or 10–14 days after inoculation with PstT1, as discussed in section 11. Seedling death is visualized by a brown apical meristem, arrested epicotyl elongation, and/or arrested vegetative growth. If different bacterial strains are used, the timing will have to be empirically determined. In addition, the progression of disease on control plants should be monitored daily after flooding until a consistent time frame from the onset of disease symptoms to seedling death can be identified. Depending on the genotypes and treatments used in the flood assay, seedling phenotypes can be recorded as binary phenotypes or on a disease spectrum (Figure 4). A broader spectrum of phenotypes may be observed when flood inoculating F2 mapping populations from wild tomato accessions crossed to susceptible cultivars (Figure 4C). It may be best to phenotype segregating populations on a disease spectrum depending on how quickly the seedling dies and the degree of new vegetative growth and branching (Figure 4C). The seedling flood assay can also be used in conjunction with the seedling bacterial growth assay to quantitatively assess levels of bacterial growth associated with qualitative phenotypes in individual seedlings (Figure 7). Very large reductions (i.e., ~log 3) in bacterial growth or strong resistance in resistant seedlings of a wild accession compared to a susceptible cultivar suggest that the underlying genetic basis of resistance may be due to ETI22. Smaller reductions in bacterial growth (i.e., ~log 1.7), as observed in LA1329 seedlings, may be due to the contribution of weaker resistance from quantitative trait loci and/or PTI. Thus, the seedling growth assay can be an important tool in further characterizing resistance in wild tomato lines.
to:
Pst is a foliar pathogen that preferentially colonizes the aerial parts of tomato seedlings, including the cotyledons24 (Figure 3). Therefore, qualitative phenotyping in the seedling flood assay focuses on growth and disease symptoms in aerial portions of the seedling, and tissue for the bacterial growth assay is sampled from the cotyledons for quantitative analysis. After flood inoculation, seedlings may die within 7–10 days after inoculation with PstDC3000 or 10–14 days after inoculation with Pst19, as discussed in section 11. Seedling death is visualized by a brown apical meristem, arrested epicotyl elongation, and/or arrested vegetative growth. If different bacterial strains are used, the timing will have to be empirically determined. In addition, the progression of disease on control plants should be monitored daily after flooding until a consistent time frame from the onset of disease symptoms to seedling death can be identified. Depending on the genotypes and treatments used in the flood assay, seedling phenotypes can be recorded as binary phenotypes or on a disease spectrum (Figure 4). A broader spectrum of phenotypes may be observed when flood inoculating F2 mapping populations from wild tomato accessions crossed to susceptible cultivars (Figure 4C). It may be best to phenotype segregating populations on a disease spectrum depending on how quickly the seedling dies and the degree of new vegetative growth and branching (Figure 4C). The seedling flood assay can also be used in conjunction with the seedling bacterial growth assay to quantitatively assess levels of bacterial growth associated with qualitative phenotypes in individual seedlings (Figure 7). Very large reductions (i.e., ~log 3) in bacterial growth or strong resistance in resistant seedlings of a wild accession compared to a susceptible cultivar suggest that the underlying genetic basis of resistance may be due to ETI22. Smaller reductions in bacterial growth (i.e., ~log 1.7), as observed in LA1329 seedlings, may be due to the contribution of weaker resistance from quantitative trait loci and/or PTI. Thus, the seedling growth assay can be an important tool in further characterizing resistance in wild tomato lines.
The fourth paragraph of the Discussion section was updated from:
Typically, genetic screens have been performed on four- to five-week-old adult tomato plants to identify the genetic basis of P. syringae resistance in wild accessions20,21. Adult tomato plants require much longer growth times, require more space in the growth chamber, and are much larger plants, which means that usually few individuals are screened for each line. The seedling flood assay provides a powerful, alternative approach in the identification of P. syringae resistance in wild tomato accessions. Screening at the seedling stage permits a large sample size to be tested which can be particularly advantageous in detecting resistance in genetically complex populations. Reduced growth chamber space requirements and growth time facilitate a high-throughput approach and rapid detection of natural resistance in wild accessions to emerging pathogens. Furthermore, P. syringae resistance that was identified at the seedling stage in this assay is not restricted to the developmental stage. S. neorickii LA1329 and S. habrochaites LA1253 were initially identified at the seedling stage and also display resistance to PstT1 in adult plants as previously described22.
to:
Typically, genetic screens have been performed on four- to five-week-old adult tomato plants to identify the genetic basis of P. syringae resistance in wild accessions20,21. Adult tomato plants require much longer growth times, require more space in the growth chamber, and are much larger plants, which means that usually few individuals are screened for each line. The seedling flood assay provides a powerful, alternative approach in the identification of P. syringae resistance in wild tomato accessions. Screening at the seedling stage permits a large sample size to be tested which can be particularly advantageous in detecting resistance in genetically complex populations. Reduced growth chamber space requirements and growth time facilitate a high-throughput approach and rapid detection of natural resistance in wild accessions to emerging pathogens. Furthermore, P. syringae resistance that was identified at the seedling stage in this assay is not restricted to the developmental stage. S. neorickii LA1329 and S. habrochaites LA1253 were initially identified at the seedling stage and also display resistance to Pst19 in adult plants as previously described22.



