ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
अंकुर बाढ़ परख स्यूडोमोनास सिरिंगजीवाणु के प्रतिरोध के लिए जंगली टमाटर परिग्रहण की तेजी से स्क्रीनिंग की सुविधा प्रदान करता है । यह परख, अंकुर जीवाणु विकास परख के साथ संयोजन के रूप में इस्तेमाल किया, आगे जीवाणु के लिए अंतर्निहित प्रतिरोध की विशेषता में सहायता कर सकते हैं, और प्रतिरोध के आनुवंशिक आधार का निर्धारण करने के लिए मानचित्रण आबादी स्क्रीन करने के लिए इस्तेमाल किया जा सकता है ।
Abstract
टमाटर एक कृषि विज्ञानी महत्वपूर्ण फसल है जिसे ग्राम-नकारात्मक जीवाणु स्यूडोमोनास सिरिंगेसे संक्रमित किया जा सकता है, जिसके परिणामस्वरूप बैक्टीरियल स्पेक रोग होता है। टमाटर-पी सिरिंगपीवी। टमाटर रोगप्रणाली व्यापक रूप से पौधे जन्मजात प्रतिक्रियाओं और रोग प्रतिरोध के आनुवंशिक आधार को विच्छेदन करने के लिए प्रयोग किया जाता है। जबकि रोग सफलतापूर्वक खेती टमाटर में Solanum पिंपिनालीफोलियम से Pto/Prf जीन क्लस्टर की शुरूआत के माध्यम से कई दशकों के लिए प्रबंधित किया गया था, पी सिरिंगे की दौड़ 1 उपभेदों Pto द्वारा प्रदत्त प्रतिरोध को दूर करने के लिए विकसित किया है/
जंगली टमाटर प्रजातियां रोगजनक मान्यता में प्राकृतिक विविधता के महत्वपूर्ण जलाशय हैं, क्योंकि वे विभिन्न रोगजनक दबावों के साथ विविध वातावरण में विकसित हुए। जंगली टमाटर में रोग प्रतिरोध के लिए विशिष्ट स्क्रीन में, वयस्क पौधों का उपयोग किया जाता है, जो पौधों की संख्या को सीमित कर सकते हैं जिन्हें उनके विस्तारित विकास समय और अधिक विकास स्थान आवश्यकताओं के कारण जांच की जा सकती है। हमने प्रतिरोध के लिए 10 दिन पुरानी टमाटर की पौध को स्क्रीन करने के लिए एक विधि विकसित की है, जो पौधों के विकास के समय और विकास कक्ष अंतरिक्ष को कम करती है, पौधों के तेजी से कारोबार की अनुमति देती है, और बड़े नमूना आकारों का परीक्षण करने की अनुमति देती है। जीवित रहने या मृत्यु के अंकुर परिणामों को असतत फेनोटाइप के रूप में या बाढ़ के बाद जीवित रोपण में नए विकास की मात्रा द्वारा परिभाषित प्रतिरोध पैमाने पर माना जा सकता है। इस विधि को दो पी सिरिंग उपभेदों के प्रतिरोध के लिए 10 दिन पुरानी टमाटर रोपण स्क्रीन करने के लिए अनुकूलित किया गया है और आसानी से अन्य पी सिरिंग उपभेदों के लिए अनुकूलित किया जा सकता है।
Introduction
स्यूडोमोनास सिरिंगे एक ग्राम-नकारात्मक रोगजनक जीवाणु है जो पौधे के मेजबानों की एक विस्तृत श्रृंखला को संक्रमित करता है। बैक्टीरिया स्टोमेटा या शारीरिक घावों के माध्यम से मेजबान पौधे में प्रवेश करते हैं और एपोप्ट1में पैदा होते हैं । पौधों ने बैक्टीरियल रोगजनकों द्वारा संक्रमण से बचाने के लिए दो-स्तरीय प्रतिरक्षा प्रतिक्रिया विकसित की है। पहला स्तर पौधे की कोशिका की सतह पर होता है, जहां पौधे की कोशिका झिल्ली पर पैटर्न मान्यता रिसेप्टर्स पीएमपी-ट्रिगर प्रतिरक्षा (पीटीआई)2नामक प्रक्रिया में अत्यधिक संरक्षित रोगजनक-संबद्ध आणविक पैटर्न (पीम्प्स) का अनुभव करते हैं। इस प्रक्रिया के दौरान, मेजबान संयंत्र रक्षा प्रतिक्रिया रास्तों को अपनियंत्रित करता है, जिसमें सेल की दीवार पर कैलोज का जमाव, स्टोमेटा को बंद करना, प्रतिक्रियाशील ऑक्सीजन प्रजातियों का उत्पादन और रोगजनन से संबंधित जीन शामिल करना शामिल है ।
बैक्टीरिया प्रोटीन देने के लिए एक प्रकार III स्राव प्रणाली का उपयोग करके पीटीआई को दूर कर सकते हैं, जिसे प्रभावक कहा जाता है, सीधे संयंत्र सेल3में । प्रभावक प्रोटीन आमतौर पर पीटीआई के घटकों को लक्षित करते हैं और रोगजनक उग्रता को बढ़ावा देते हैं4. प्रभावक प्रोटीन की मान्यता पर पौधे की प्रतिरक्षा का दूसरा स्तर पौधे की कोशिका के भीतर होता है। यह मान्यता प्रतिरोध जीन पर निर्भर करती है, जो न्यूक्लियोटाइड-बाध्यकारी साइट ल्यूसिन-रिच रिपीट को एन्कोड करती है जिसमें रिसेप्टर्स (एनएलआर) होते हैं। एनएलआर या तो प्रभावकों को सीधे पहचानने या उग्रता लक्ष्य या अपवित्र5पर उनकी गतिविधि को पहचानने में सक्षम हैं । फिर वे प्रभावक-ट्रिगर प्रतिरक्षा (ईटीआई) नामक प्रक्रिया में एक माध्यमिक प्रतिरक्षा प्रतिक्रिया को ट्रिगर करते हैं, जो अक्सर एक अतिसंवेदनशील प्रतिक्रिया (एचआर) से जुड़ा होता है, जो संक्रमण6की साइट पर स्थानीयकृत कोशिका मृत्यु का एक रूप है। ईटीआई से जुड़े जीन-फॉर-जीन प्रतिरोध के विपरीत, पौधे मात्रात्मक आंशिक प्रतिरोध प्रदर्शित कर सकते हैं, जो कई जीन7के योगदान पर निर्भर है ।
पी सिरिंगे पीवी टमाटर (पीएसटी)टमाटर पर बैक्टीरियल स्पेक का कारण एजेंट है और यह लगातार कृषि समस्या है। क्षेत्र में प्रमुख उपभेदों आम तौर पर पीएसटी रेस 0 उपभेदों कि या तो या दोनों प्रकार III प्रभावकों AvrPto और AvrPtoB व्यक्त किया गया है । DC3000(पीएसटीDC3000) एक प्रतिनिधि दौड़ 0 तनाव और एक मॉडल रोगजनक है कि टमाटर में बैक्टीरियल धब्बा पैदा कर सकता है । बैक्टीरियल स्पेक रोग का मुकाबला करने के लिए, प्रजनकों ने पीटीओ [पी सिरिंगपीवी टमाटर]/Prf [Pto प्रतिरोध और फेनथिऑन संवेदनशीलता]जीन क्लस्टर को जंगली टमाटर प्रजाति सोलनम पिंपिनेलीफोलियम से आधुनिक खेती8,9में प्रवेश किया । Pto जीन एक सेरीन-थ्रेनोइन प्रोटीन किनेस को एन्कोड करता है जो पीआरएफ एनएलआर के साथ मिलकर, प्रभावकों AvrPto और AvrPtoB10,11,12,13,14की मान्यता के माध्यम से पीएसटीDC3000 के लिए प्रतिरोध प्रदान करता है। हालांकि, यह प्रतिरोध उभरते रेस 1 उपभेदों के खिलाफ अप्रभावी है, जो हाल के वर्षोंमें 15,16में उनके तेजी से और आक्रामक प्रसार की अनुमति देता है । रेस 1 उपभेदों को Pto/Prf क्लस्टर द्वारा मान्यता से बचने, क्योंकि AvrPto या तो खो दिया है या इन उपभेदों में उत्परिवर्तित है, और AvrPtoB कम सेकम 15,17,18जमा प्रतीत होता है ।
जंगली टमाटर की आबादी पीएसटी प्रतिरोध के लिए प्राकृतिक भिन्नता के महत्वपूर्ण जलाशय हैं और पहले इसका उपयोग संभावित प्रतिरोध लोकी19,20,21की पहचान करने के लिए किया गया है । हालांकि, रोगजनक प्रतिरोध के लिए वर्तमान स्क्रीन 4-5 सप्ताह पुराने वयस्क पौधों20,21का उपयोग करते हैं। इसलिए, वे विकास समय, विकास कक्ष अंतरिक्ष, और अपेक्षाकृत छोटे नमूना आकार से सीमित हैं। पारंपरिक दृष्टिकोणों की सीमाओं को संबोधित करने के लिए, हमने 10 दिन पुराने टमाटर रोपण22का उपयोग करके एक उच्च-थ्रूपुट टमाटर पी सिरिंग प्रतिरोध परख विकसित की। यह दृष्टिकोण वयस्क पौधों का उपयोग करने पर कई फायदे प्रदान करता है: अर्थात्, कम विकास समय, कम स्थान आवश्यकताएं, और उच्च थ्रूपुट। इसके अलावा, हमने दिखा दिया है कि यह दृष्टिकोण ईमानदारी से वयस्क पौधों22में मनाए गए रोग प्रतिरोध फेनोटाइप को फिर से दोहराता है।
इस प्रोटोकॉल में वर्णित अंकुर बाढ़ परख में, टमाटर रोपण बाँझ मुराशिगे और Skoog (एमएस) मीडिया के पेट्री व्यंजन पर 10 दिनों के लिए उगाया जाता है और फिर ब्याज के बैक्टीरिया और एक सर्फेक्टन सरफेस्ट युक्त एक inoculum के साथ बाढ़ आ जाती है । बाढ़ के बाद, रोपण मात्रात्मक बैक्टीरियल विकास परख के माध्यम से रोग प्रतिरोध के लिए मूल्यांकन किया जा सकता है । इसके अतिरिक्त, अंकुर अस्तित्व या मृत्यु बाढ़ के 7-14 दिनों के बाद एक असतत प्रतिरोध या रोग फेनोटाइप के रूप में कार्य कर सकती है। यह दृष्टिकोण पीएसटी रेस 1 उपभेदों, जैसे पीएसटी स्ट्रेन टी 1(पीएसटीटी 1) के प्रतिरोध के लिए बड़ी संख्या में जंगली टमाटर परिग्रहण की स्क्रीनिंग के लिए एक उच्च-थ्रूपुट विकल्प प्रदान करता है, और आसानी से ब्याज के अन्य बैक्टीरियल उपभेदों के अनुकूल किया जा सकता है।
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. जैव सुरक्षा कैबिनेट की तैयारी और उपयोग
- 70% इथेनॉल के साथ बायोसेफ्टी कैबिनेट को मिटा दें।
- सैश बंद करें और 15 मिन के लिए बायोसेफ्टी कैबिनेट में पराबैंगनी प्रकाश चालू करें।
- 15 मिन के बाद बायोसेफ्टी कैबिनेट में पराबैंगनी प्रकाश बंद कर दें। सैश उठाएं और 15 मिन के लिए ब्लोअर चालू करें।
- निष्फल कैबिनेट में आइटम डालने से पहले 70% इथेनॉल के साथ बायोसेफ्टी कैबिनेट में उपयोग किए जाने वाले सभी वस्तुओं को मिटा दें।
- बायोसेफ्टी कैबिनेट में काम करने से पहले 70% इथेनॉल के साथ साफ दस्ताने या नंगे हाथ।
- ब्लोअर से दूर, जैव सुरक्षा कैबिनेट के केंद्र में काम करते हैं।
- प्रयोगों के लिए ऑटोक्लेव्ड बाँझ 10 एमएम एमजीसीएल2 और अल्ट्राप्योर एच2ओ की खुली बोतलों का उपयोग करें। बायोसेफ्टी कैबिनेट में बोतलें रखें और केवल उन्हें निष्फल बायोसेफ्टी कैबिनेट में खोलें, बेंचटॉप पर नहीं ।
- निष्फल जैव सुरक्षा कैबिनेट में काम के लिए समर्पित ग्लास पिपेट और पिपेट युक्तियों का उपयोग करें। सुनिश्चित करें कि ये केवल जैव सुरक्षा कैबिनेट में खोले जाते हैं, कभी बेंचटॉप पर नहीं।
- बायोसेफ्टी कैबिनेट के उपयोग के बाद, सभी अपशिष्ट (ब्लीच कचरे को छोड़कर) को स्वचालित करें और 70% इथेनॉल के साथ सतह को मिटा दें।
2. प्लांट मीडिया की तैयारी
- बाहर वजन और अल्ट्राप्योर एच2ओ में 0.5 x एमएस बेसल लवण भंग 0.8% bacto आगर वजन और फिर भंग 0.5x एमएस में जोड़ें।
- ऑटोक्लेव और मीडिया को डालने या पाइपिंग से पहले 1 घंटे के लिए 50 डिग्री सेल्सियस पानी स्नान में ठंडा करने की अनुमति दें।
- यह सुनिश्चित करने के लिए कि प्लेटें अधिक न भर ें, मार्क पॉलीस्टीरिन डिस्पोजेबल बाँझ 100 x 25 मिमी प्लेटें 40 मीटर के भरने के स्तर तक। एक निष्फल जैव सुरक्षा कैबिनेट में 100 x 25 मिमी बाँझ प्लेटों में मीडिया डालो।
3. पौधे सामग्री और विकास की स्थिति की तैयारी
- टमाटर के बीजों को 2.2 मिलीएम माइक्रोसेंट्रोफ्यूज ट्यूब में रखें और 50% ब्लीच समाधान के 2.0 मिलील जोड़ें।
- 25 मिन के लिए एक झूली कुरसी पर ट्यूब रॉक ।
- 25 किमी के बाद, रॉकर से बीज निकालें और बाँझ जैव सुरक्षा कैबिनेट में एक पिपेट के साथ ब्लीच समाधान हटा दें। सुनिश्चित करें कि सभी ब्लीच हटा दिया गया है।
- बीज धोने के लिए बाँझ अल्ट्राप्योर एच2ओ का 2 एमएल जोड़ें। ट्यूब 5x उलटा।
- एक पिपेट के साथ ट्यूब से तरल निकालें।
- बीज 4x अधिक धोने के लिए चरण 3.3-3.5 दोहराएं।
- बाँझ अल्ट्राप्योर एच2ओ के 2 mL जोड़ें और एक खाली बाँझ पेट्री पकवान में बीज डालना ।
- लौ इथेनॉल में संदंश और 100 x 25 मिमी प्लेटों पर बीज स्थानांतरित करने और समान रूप से अंतर करने से पहले ठंडा करने की अनुमति देती है जिसमें 0.5 x एमएस + 0.8% आगर मीडिया होता है।
- एक प्लेट के बीच में एक लाइन में 5-7 बीज स्थानांतरित करें और सर्जिकल टेप (1.25 सेमी x 9.1 मीटर) के साथ प्लेटों के किनारों को सील करें।
- अंकुरण को सिंक्रोनाइज़ करने के लिए कम से कम 3 दिनों के लिए अंधेरे में 4 डिग्री सेल्सियस पर बंध्याकरण बीजों को स्तरित करें। सुनिश्चित करें कि प्लेटें फ्लैट खड़ी हैं और सामना कर रही हैं, ताकि बीज प्लेट पर शिफ्ट न हों।
- लंबवत प्लेटों को उन्मुख करें ताकि जड़ें प्लेट की सतह के साथ नीचे बढ़ जाएंगी, बीजों की रेखा क्षैतिज रूप से उन्मुख होगी, जब विकास कक्ष में स्थानांतरित हो जाएगी।
नोट: विकास कक्ष को 22 डिग्री सेल्सियस तक सेट करें और ~ 200-220 माइक्रोन मीटर-2 एस-1 और 8 घंटे के अंधेरे की हल्की तीव्रता पर 16 घंटे प्रकाश प्रदान करें। - बाढ़ से पहले, विकास कक्ष में 10 दिनों के लिए रोपण बढ़ें जिस पर बिंदु रोपण आम तौर पर पूरी तरह से उभरा और विस्तारित कोटाइलेडन और उभरती हुई पहली सच्ची पत्तियों(चित्रा 1)प्रदर्शित करते हैं।

चित्रा 1: ठेठ 10 दिन पुराने टमाटर रोपण के विकास के चरण । रियो ग्रांडे-टॉर टमाटर के बीजों को 4 डिग्री सेल्सियस पर अंधेरे में कम से कम 3 दिनों के लिए निष्फल, चढ़ाया और स्तरीकृत किया गया। पौध भर जाने से पहले 22 डिग्री सेल्सियस पर 10 दिनों के लिए 0.5 x एमएस प्लेटों पर उगाई गई थी। आमतौर पर, 10 दिनों में कोटाइलेडन पूरी तरह से विस्तारित होते हैं, और पहली सच्ची पत्तियां उभरने लगी हैं। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
4. राजा के बी23 (केबी) मीडिया की तैयारी
- अल्ट्राप्योर एच2ओ के 500 मीटर के साथ बीकर भरें और एक हलचल प्लेट पर हलचल।
- 20 ग्राम बैक्टो पेप्टोन, 1.5 ग्राम हाइड्रोस के2एचपीओ4और अल्ट्राप्यूरी एच2ओ के साथ बीकर में ग्लाइसेरोल का 12.5 ग्राम भंग करें।
- भंग मिश्रण को 1 एल स्नातक सिलेंडर में डालें और अल्ट्रापुरेएच2ओ के साथ 1 एल अंतिम मात्रा तक लाएं।
- शोरबा को वापस बीकर में डालें और मिश्रित होने तक हिलाएं।
- दो 500 मीटर ग्लास बोतलों में 7.5 ग्राम बैक्टो एगर का वजन करें और प्रत्येक बोतल में चरण 4.4 से केबी शोरबा का 500 मीटर जोड़ें। 20 मिन के लिए ऑटोक्लेव।
- ऑटोक्लेव से बोतलें निकालें और आगर वितरित करने के लिए धीरे-धीरे चक्कर लगाएं।
- बोतलों को 1 घंटे के लिए 50 डिग्री सेल्सियस पानी स्नान में स्थानांतरित करें।
- 1 घंटे के बाद, बोतल को बायोसेफ्टी कैबिनेट में और असेप्टिक स्थितियों के तहत स्थानांतरित करें, बाँझ 1 एम एमजीएसओ4के 1,600 माइक्रोन जोड़ें, और मीडिया को उपयुक्त एंटीबायोटिक दवाएं जोड़ें।
नोट: रिफाम्पिसिन प्रतिरोधी उपभेदों के लिए पीएसटीDC3000 और पीएसटीT1, 50 μg/mL की अंतिम एकाग्रता पर डाइमिथाइलफॉर्ममाइड में भंग रिफाम्पिसिन का उपयोग करें। प्लेटों पर फंगल विकास को रोकने के लिए 50 μg/mL की अंतिम एकाग्रता पर इथेनॉल में भंग साइक्लोहेक्सिमाइड का उपयोग करें। - मीडिया को धीरे से मिलाने के लिए भंवर और फिर प्लेटों के नीचे कवर करने के लिए डालना ।
- प्लेटों को 4 डिग्री सेल्सियस पर उल्टा स्टोर करने से पहले जमना के लिए कम से कम 1 घंटे की अनुमति दें।
5. बैक्टीरियल उपभेदों और संस्कृति की स्थिति का रखरखाव
- बैक्टीरिया की एकल कॉलोनी से एक ग्लाइसेरोल स्टॉक को संतृप्त जीवाणु संस्कृति के 1 मिलील और बाँझ के 333 माइक्रोन 80% ग्लाइसेरोल -80 डिग्री सेल्सियस पर बनाए रखें।
- पैच बैक्टीरिया (यानी, पीएसटीT1) उचित एंटीबायोटिक दवाओं (धारा 4) के साथ KB आगर पर एक ग्लाइसेरोल स्टॉक से ।
- बैक्टीरिया को एक फ्लैट, बाँझ टूथपिक का उपयोग करके चयनात्मक केबी आगर पर ताजा बैक्टीरिया को लकीर देने से पहले 28 डिग्री सेल्सियस पर 2 दिनों के लिए ठीक होने दें।
- एक फ्लैट, बाँझ टूथपिक का उपयोग करउचित चयनात्मक KB आगर पर ग्लाइसेरोल स्टॉक से लकीर ताजा बैक्टीरिया ।
नोट: सुनिश्चित करें कि समझौता ग्लाइसेरोल स्टॉक 2 सप्ताह से अधिक पुराना नहीं है। - पीएसटीDC3000 के लिए, बाढ़ प्रयोग में बैक्टीरिया का उपयोग करने से पहले 24 घंटे के लिए 28 डिग्री सेल्सियस पर केबी प्लेट इनक्यूबेट करें ।
- पीएसटीटी 1 के लिए, बाढ़ प्रयोग में बैक्टीरिया का उपयोग करने से पहले 48 घंटे के लिए 28 डिग्री सेल्सियस पर केबी प्लेट को इनक्यूबेट करें।
6. पीएसटीटी 1 इनोकुलम की तैयारी
- एसेप्टिक रूप से बाँझ 10 एनएम एमजीसीएल2 में बैक्टीरिया को 0.1 के 600 एनएम (ओडी600)या लगभग 5 x 107 कॉलोनी बनाने वाली इकाइयों (सीएफयू) /एमएल) पर ऑप्टिकल घनत्व में फिर से निलंबित करें।
- जैव सुरक्षा कैबिनेट में बाँझ 10 mM MgCl2 समाधान का उपयोग कर धारावाहिक कमजोर होजाता है। पीएसटीटी 1 के लिए, ओडी600 = 0.1 की शुरुआती एकाग्रता के साथ इनोक्यूलम बनाने के लिए स्पेक्ट्रोफोटोमीटर का उपयोग करें।
- पीएसटीटी 1 के लिए, ओडी600 = 0.1 पर प्रारंभिक पुनर्निलंबन से 1/10 कमजोर पड़ने के लिए ओडी600 = 0.01 की एकाग्रता पर एक धारावाहिक कमजोर पड़ने प्राप्त करने के लिए।
- 6.3 चरण से ओडी600 = 0.01 पर धारावाहिक कमजोर पड़ने का उपयोग करना, अंतिम ओडी600 = 0.0075 प्राप्त करने के लिए 3/4 कमजोर पड़ने का काम करें।
- नॉन-आयनिक ऑर्गेनोसिलिकॉन सरफेसटैंट कोपॉलिमर सी13एच34ओ4एसआई3 (यानी, 1/10 कमजोर पड़ना बनाओ 10 mM MgCl2 में surfactant) और 15 एस के लिए भंवर । पिछले धारावाहिक कमजोर पड़ने (OD ६०० = ०.००७५) के लिए सर्फेक्टेंट के 1/10 स्टॉक ०.०१५% की अंतिम एकाग्रता के लिए जोड़ें और अच्छी तरह से मिश्रण करने के लिए भंवर ।
7. पीएसटीDC3000 इनोकुलम की तैयारी
- एसेप्टिक रूप से बाँझ 10 एनएम एमजीसीएल2 में बैक्टीरिया को ०.१ (लगभग 5 x 107 सीएफयू/एमएल) के ६०० एनएम (ओडी६००)पर ऑप्टिकल घनत्व में फिर से निलंबित करें ।
- जैव सुरक्षा कैबिनेट में बाँझ 10 mM MgCl2 समाधान का उपयोग कर धारावाहिक कमजोर होजाता है। पीएसटीDC3000 के लिए, ओडी600 = 0.1 की शुरुआती एकाग्रता के साथ इनोक्यूलम बनाने के लिए स्पेक्ट्रोफोटोमीटर का उपयोग करें।
- पीएसटीDC3000 के लिए, ओडी600 = 0.1 पर प्रारंभिक पुनर्निलंबन से 1/10 कमजोर पड़ने के लिए OD600 = 0.01 की एकाग्रता पर एक धारावाहिक कमजोर पड़ने प्राप्त करने के लिए।
- 3 चरण से ओडी600 = 0.01 पर धारावाहिक कमजोर पड़ने का उपयोग करना, अंतिम ओडी600 = 0.005 प्राप्त करने के लिए 1/2 कमजोर पड़ने का काम करें।
- 10 एमएम एमजीसीएल2 में सर्फेक्टेंट का 1/10 कमजोर पड़ना और 15 एस के लिए भंवर करें। 0.015% की अंतिम एकाग्रता के लिए अंतिम धारावाहिक कमजोर पड़ने (ओडी600 = 0.005) में सर्फेक्टेंट का 1/10 स्टॉक जोड़ें और मिश्रण करने के लिए अच्छी तरह से भंवर करें।
8. टमाटर अंकुर बाढ़ विधि
- 10 दिन पुरानी पौध के साथ प्लेटों को विकास कक्ष से बाहर ले जाएं और बाढ़ के लिए प्लेटें तैयार करने के लिए बायोसेफ्टी कैबिनेट में डाल दें ।
- सर्जिकल टेप को दो प्लेटों से हटा दें।
- 3 मिन के लिए एक टाइमर सेट करें। अंतिम इनोकुलम के 6 mL को मापें(पीएसटीटी 1 ओडी600 = 0.0075 [धारा 6] या पीएसटीDC3000 OD600 = 0.005 [धारा 7]) और 10 दिन पुरानी अंकुर के साथ प्रत्येक प्लेट में इनोकुलम के 6 mL स्थानांतरित करें।
- धीरे-धीरे रोपण को एक बाँझ पिपेट टिप के साथ इनोक्यूलम में नीचे धकेलें। टाइमर शुरू करें।
- प्रत्येक हाथ में एक प्लेट पकड़ो। प्लेट के सामने को इनोक्यूलम जमा करने के लिए झुकाएं और मुख्य रूप से रोपण के कोटाइलेडन और पत्तियों को जलमग्न कर दें।
- बेंत की ओर 5-7x पक्ष और फिर जड़ों और पूरी थाली को कवर करने के लिए प्लेटों को वापस टिप।
- प्लेटों को फिर से झुकाएं ताकि कोटाइलेडन और पत्तियों को जलमग्न किया जा सके, और कुल 3 मिन के लिए दोहराएं।
- प्लेटों से इनोकुलम डालो, प्लेटों को एक सपाट सतह पर सेट करें और फिर दूसरी बार किसी भी अवशिष्ट इनोकुलम को बंद करें।
- सर्जिकल टेप के साथ प्लेटों को फिर से लपेटें और किसी भी शेष प्लेटों के लिए 8.2-8.8 चरणदोहराएं।
- सभी प्लेटों में बाढ़ आने के बाद विकास कक्ष में प्लेटों को फिर से इनक्यूबेट करें (चरण ३.११ नोट देखें) ।
- पीएसटीDC3000 के लिए 7-10 दिनों के बाद फेनोटाइप या पीएसटीटी 1 (धारा 11) के लिए 10-14 दिन। यदि बैक्टीरियल विकास परखों को बाहर ले जाने, 4 दिनों के बाद पत्ती ऊतक इकट्ठा (वर्ग9 और 10) और फिर फेनोटाइप (धारा 11) । वैकल्पिक रूप से, पौधों के अलग-अलग सेट पर फेनोटाइपिक विश्लेषण और बैक्टीरियल विकास परख करें।
9. बैक्टीरियल विकास परख के लिए cotyledons की सतह नसबंदी
- बाढ़ के चार दिन बाद और विकास कक्ष (धारा 8) में रोपण को फिर से इनक्यूबेटिंग करने के चार दिन बाद, टमाटर के रोपण के साथ प्लेटों को विकास कक्ष से हटा दें।
- प्लेट के नीचे बाहरी पर व्यक्तिगत रोपण की संख्या जहां रोपण प्रत्येक जीनोटाइप के लिए प्लेट को देता है।
- लेबल बाँझ 1.5 mL माइक्रोसेंटरिफ्यूज ट्यूब ों को व्यक्तिगत अंकुर संख्या के साथ लेबल करें और एक बीड बीटर के साथ उपयोग के लिए प्रत्येक ट्यूब में एक 3 मिमी बाँझ बोरोसिलिकेट बीड को छोड़ने के लिए स्वच्छ संदंश का उपयोग करें। (चरण 10.1 में नोट देखें।)
- प्रत्येक ट्यूब और बंद ट्यूबों में 10 एमएम एमजीसीएल2 का पिपेट 200 माइक्रोन।
- 70% इथेनॉल तैयार करें और 100 मीटर को एक साफ बीकर में डालें। बाँझ अल्ट्राप्योर एच2ओ के 100 मीटर एक अलग, साफ बीकर में डालो।
- इथेनॉल के साथ नर्टेड युक्तियों के साथ साफ स्टेनलेस स्टील सीधे ठीक बिंदु संदंश। साफ संदंश के साथ एक कोटायलेडोन के एसेप्टिक हटाने की अनुमति देने के लिए प्लेट को थोड़ा खोलें।
- कोटिलेडसन के आधार पर पेटीओल को चुटकी लें एक पत्ते को हटाने के लिए और 70% इथेनॉल के साथ बीकर में छोड़ने के लिए सतह के लिए 10 एस के लिए बंध्याकरण। 10 एस के लिए अल्ट्राप्योर एच2ओ में कोटाइलेडन कुल्ला करें।
- कोटायलेडन को पेपर तौलिया पर रखें और नाजुक विज्ञान पोंछे के साथ सूखी दाग करें।
- व्यक्तिगत रूप से सतह नसबंदी और दाग के बाद प्रत्येक cotyledon वजन, और वजन रिकॉर्ड ।
- कोटायलेडन को पहले से तैयार 1.5 मिलीएम माइक्रोसेंरिफ्यूज ट्यूब (चरण 9.3 और 9.4 से) में रखें, जो इसी जीनोटाइप और व्यक्तिगत संख्या के साथ लेबल किया गया था।
- बाँझ टेप के साथ प्लेटों को फिर से सील करें और विकास कक्ष में रोपण को फिर से इनक्यूबेट करें (चरण 3.11 नोट देखें)।
10. बैक्टीरियल विकास परख
- चरण 9.10 से नमूनों का उपयोग करके, 1-2 मिन के लिए 10 एमएम एमजीसीएल2 में बीड बीटर का उपयोग करके ऊतक को समरूप करें। यदि ऊतक को पर्याप्त रूप से मैकेरेटेड नहीं किया जाता है, तो फिर से समरूपता।
नोट: कई निर्माता मनका बीटर होमोजेनेज़र का उत्पादन करते हैं। मोतियों की संख्या और प्रकार, साथ ही समरूपता समय और गति (यदि प्रोग्राम करने योग्य) को प्रत्येक प्रकार के समरूपता के लिए अनुकूलित किया जाना चाहिए। सुनिश्चित करें कि नमूने समरूपता के दौरान अधिक गरम न करें। - चरण 10.1 से मैसेरेटेड ऊतक युक्त प्रत्येक ट्यूब में 10 एमएम एमजीसीएल2 के 800 माइक्रोन जोड़ें और मिश्रण करने के लिए कई बार उलटा।
- मल्टीचैनल पिपेट(चित्रा 2ए)का उपयोग करके 96 अच्छी प्लेट (100,10-1, 10-2,10-3,10-4,10-5)में 10 एमएम एमजीसीएल2 में प्रत्येक नमूने के लिए धारावाहिक कमजोरियां तैयार करें।
- साइक्लोहेक्सिमाइड के साथ केबी एगर प्लेट (150 मिमी x 15 मिमी) पर मल्टीचैनल पिपेट का उपयोग करके प्रत्येक कमजोर पड़ने वाली श्रृंखला से 5 माइक्रोन (चक्रव्यूह के बैक्टीरियल तनाव के लिए उपयुक्त चयन (चरण 4.8 नोट देखें)। प्लेटों को पूरी तरह से सूखने दें।
- प्लेट को 36 घंटे के लिए 28 डिग्री सेल्सियस पर उल्टा इनक्यूबेट करें, फिर कल्पना करें(चित्रा 2बी)प्लेटों पर उपनिवेशों को विच्छेदन माइक्रोस्कोप का उपयोग करके यह निर्धारित करने के लिए कि क्या उपनिवेश ों की गणना करने के लिए पर्याप्त बड़ी है।
नोट: यदि कॉलोनियां काफी बड़ी नहीं हैं, तो प्लेटों को फिर से इनक्यूबेट करें और हर कुछ घंटों में कॉलोनियों के आकार की जांच करें। आमतौर पर, उपनिवेशों को ऊष्मायन के बाद ~ 36-48 एच गिना जाता है।

चित्रा 2: बैक्टीरियल विकास परख रोपण के लिए धारावाहिक कमजोरी । (A)संक्रमित पौधों से मैसेरेटेड पत्ती ऊतक कॉलोनी गिनती से पहले पतला हो जाता है। कमजोर पड़ने से 96 अच्छी प्लेट में प्रदर्शन किया जाता है (100 पतला नहीं होता)। आमतौर पर, कमजोर पड़ने 10-1 से 10-5 तक बनाए जाते हैं। (ख)बैक्टीरियल कॉलोनी मायने रखता है के लिए चढ़ाना कमजोर । कमजोर पड़ने की श्रृंखला के प्रत्येक स्तंभ के कुल 5 μL चढ़ाया जाता है, सबसे पतला से सबसे केंद्रित करने के लिए । कॉलोनियों के पूरी तरह सूख जाने के बाद, प्लेट को 36-48 घंटे के लिए 28 डिग्री सेल्सियस पर इनक्यूबेटेड किया जाता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
- विलय से पहले एक विच्छेदन माइक्रोस्कोप के तहत कालोनियों की गिनती करें(चित्रा 2बी)। 100 से कम कॉलोनियों के साथ कमजोर पड़ने वाली सीरीज प्लेटों से कॉलोनियों की गिनती करें।
- कॉलोनी की गिनती(चित्रा 2बी)प्राप्त करने के बाद, रोपण के लिए 0.01 ग्राम ऊतक ों की गिनती को सामान्य करें और बैक्टीरियल विकास(तालिका 1)लॉग इन करने के लिए परिवर्तित करें।
नोट: एक मनीमेकर का औसत द्रव्यमान -PtoS cotyledon 0.01 ग्राम है और अनुभवजन्य रूप से निर्धारित22है।
| जीनोटाइप1 कॉलम ए | टिशू वेट (जी) कॉलम बी | # एक जगह कॉलम सी में कालोनियों की | स्पॉट2 कॉलम डी के लिए कमजोर पड़ने का कारक | समायोजित # कालोनियों के3 कॉलम ई | धारावाहिक कमजोर पड़ने कॉलम एफ के लिए कमजोर पड़ने का कारक | कालोनियों कॉलम जी के कुल # (cfu/0.01 ग्राम)4 | कालोनियों का औसत # (cfu/0.01 ग्राम) कॉलम एच | औसत लॉग ग्रोथ (cfu/0.01 ग्राम (लॉग10))कॉलम I |
| नमूना 1 | 0.004 ग्राम | 10 | 200 | के रूप में गणना: (C2 x 0.01 ग्राम) / B2 = 25 | 1000 | के रूप में गणना: (D2 x E2 x F2) = 5000000 | पिछले नमूने के माध्यम से नमूना 1 के लिए औसत: (यानी औसत G1:G3) = 7000000 | औसत यानी का लॉग। लॉग (एच 2) = 6.85 |
| नमूना 2 | 0.003 ग्राम | 15 | 200 | 50 | 1000 | 10000000 | ||
| नमूना 3 | 0.002 ग्राम | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1 3 नमूनों के लिए दिखाया गया डेटा | ||||||||
| 2 चढ़ाना के आधार पर 1 मीटर के लिए 5 μL x 200 | ||||||||
| 3 कोटायलेडन कोर करने के लिए बहुत छोटे हैं इसलिए कॉलोनी की गिनती को एक मनीमेकर के औसत द्रव्यमान के आधार पर 0.01 ग्राम ऊतक तक सामान्यीकृत किया गया था -पीसीओएस कोटाइलेडन (डेटा नहीं दिखाया गया) | ||||||||
| 4 प्लेटेड वॉल्यूम के आधार पर प्रति एमएमएल समायोजित | ||||||||
तालिका 1: बैक्टीरियल विकास परख को अंकुरित करने के लिए नमूना गणना। नमूना गणना यह प्रदर्शित करती है कि बैक्टीरियल की गिनती को सामान्य कैसे किया जाए और लॉग बैक्टीरियल विकास निर्धारित किया जाए।
- जटिल आनुवंशिक पृष्ठभूमि के साथ जंगली परिग्रहण और अन्य लाइनों के लिए, धारा 11 में वर्णित उनके फेनोटाइप के साथ व्यक्तिगत रोपण में जीवाणु वृद्धि के स्तर को सहसंबंधित करें।
11. प्रतिरोध के लिए फेनोटाइपिंग
- 7-14 दिनों के बाद मृत्यु (रोग के कारण) या अस्तित्व (प्रतिरोध के कारण) के लिए विकास कक्ष और फेनोटाइप व्यक्तिगत रोपण से प्लेटों को हटा दें।
- बाढ़ टीका के 7-10 दिनों बाद पीएसटीDC3000 पहले जैसे अत्यधिक उग्र तनाव से संक्रमित फेनोटाइप पौधे।
- बाढ़ टीका के बाद 10-14 दिनों में पीएसटीटी1 से संक्रमित फेनोटाइप पौधे।
- मनाए गए प्रतिरोध फेनोटाइप की सीमा के आधार पर एक स्कोरिंग सिस्टम निर्धारित करें। खेती, आइसोजेनिक लाइनों, और लगातार, मध्यवर्ती प्रतिरोध फेनोटाइप(चित्र4ए, 4B)के लिए मजबूत के साथ जंगली परिग्रहण के लिए बाइनरी फेनोटाइप रिकॉर्ड करें।
- यदि रोपण फेनोटाइपिंग के लिए समय सीमा के भीतर एपिकल मेरिस्टेम से नए विकास को प्रदर्शित करता है, तो इसे अस्तित्व के रूप में गिनें। यदि रोपण में भूरे रंग का एक ात्मक मेरिस्टेम होता है और कोई नया, हरे वनस्पति विकास को प्रदर्शित करता है, तो इसे मृत्यु(चित्र ा 3)के रूप में गिनें।

चित्रा 3: टमाटर की अंकुर का योजनाबद्ध प्रतिनिधित्व। टमाटर के अंकुर के विभिन्न हिस्सों को चित्रित किया गया है, जिसमें हाइपोकोटाइल, कोटाइलेडन, एपिकोटाइल, शूट एपिकल मेरिस्टेम और सच्ची पत्तियां शामिल हैं। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
- आबादी के लिए रोग स्पेक्ट्रम पर फेनोटाइप रिकॉर्ड करें, जैसे कि F2 मानचित्रण आबादी, प्रतिरोध फेनोटाइप(चित्रा 4सी)की एक विस्तृत श्रृंखला के साथ।
- फेनोटाइपिंग के लिए उपयुक्त खिड़की की पहचान करने के लिए रोग के लक्षणों और मौत की उपस्थिति के लिए रोपण की सावधानीपूर्वक निगरानी करें।

चित्रा 4: विभिन्न आनुवंशिक पृष्ठभूमि में रोपण प्रतिरोध और मृत्यु के लिए अपेक्षित फेनोटाइप का योजनाबद्ध प्रतिनिधित्व। (A)रियो ग्रांडे-टॉर और निकट आइसोजेनिक खेती रियो ग्रांडे के रोपण-PtoS पीएसटीDC3000 (OD600 = 0.005) + 0.015% सरफेसटैंट के साथ बाढ़ के 7 दिन बाद प्रदर्शित किए जाते हैं। रियो ग्रांडे-पीटीओआर लगातार प्रतिरोध प्रदर्शित करता है, और रियो ग्रांडे-पीटीओपीएस पीएसटीDC3000 के साथ संक्रमण के लिए लगातार संवेदनशीलता प्रदर्शित करता है। ये रेखाएं असतत और बाइनरी फेनोटाइप को जन्म देती हैं। (ख) सोलनम नियोरिकी LA1329 जैसे जंगली राज्यारोहण के रोपण को पीएसटीटी 1 (ओडी600 = 0.0075) + 0.015% सर्फेक्टेंट के साथ बाढ़ के 10 दिन बाद दिखाया गया है। रोपण फेनोटाइपिक परिवर्तनशीलता प्रदर्शित करते हैं लेकिन बाइनरी फेनोटाइप के रूप में दर्ज किए गए थे। फेनोटाइपिक परिवर्तनशीलता की मात्रा और फेनोटाइपिंग (बाइनरी रेजिस्टेंस या प्रतिरोध स्पेक्ट्रम) की विधि परीक्षण किए गए विशेष परिग्रहण पर निर्भर करेगी। (ग)अतिसंवेदनशील खेती के लिए जंगली परिग्रहण को मात देकर उत्पन्न आबादी का मानचित्रण F2 अलग आबादी में फेनोटाइप का एक व्यापक स्पेक्ट्रम प्रदर्शित कर सकता है । इस मामले में, स्पेक्ट्रम पर रोपण फेनोटाइप रिकॉर्ड करना सबसे उपयुक्त हो सकता है। एक मानचित्रण आबादी से अत्यधिक अतिसंवेदनशील रोपण 7 दिन के रूप में मौत के लिए फेनोटाइप किया जा सकता है जब पीएसटीT1 के साथ बाढ़ आ गई, और आम तौर पर एक भूरे रंग के एपिकल मेरिस्टेम, नहीं एपिकोटाइल के बहुत कम विस्तार के लिए, और कोई नया, हरे वनस्पति विकास दिखाओ । अतिसंवेदनशील रोपण का एपिकल मेरिस्टेम अधिक समय तक हरा या बहुत हल्का भूरा रह सकता है, और एपिकोटाइल और बहुत कम वनस्पति विकास का कुछ विस्तार हो सकता है, जो 10 दिन तक भूरे और गिरफ्तारियों को बदल देता है। 14 दिन तक नए और चल रहे वनस्पति विकास की मात्रा के आधार पर प्रतिरोध के लिए व्यक्तिगत रोपण को फेनोटाइप किया जा सकता है। रोपण तो कमजोर, मध्यम, या मजबूत प्रतिरोध के रूप में प्रतिरोध की विभिन्न श्रेणियों में ऊपर वर्णित फेनोटाइप के आधार पर समूहीकृत किया जा सकता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
रोपण प्रतिरोध परख का उपयोग कर खेती और आइसोजेनिक लाइनों में टॉर-मध्यस्थीयप्रतिरक्षा का पता लगाना
चित्रा 5 मनीमेकर के लिए प्रतिनिधि परिणाम दिखाता है-PtoR और मनीमेकर- पीएसटी DC3000 के साथ बाढ़ के 7-10 दिन बादPtoS cultivars। संक्रमण से पहले, 10 दिन पुरानी रोपण पूरी तरह से उभरा और विस्तारित cotyledons और उभरते पहले सच्ची पत्तियों प्रदर्शित किया । रोपण में 10 एम एम एमजीसीएल2 +0.015% सर्फेक्टेंट को नकारात्मक नियंत्रण (डेटा नहीं दिखाया गया) और पीएसटीDC3000 के ऑप्टिकल घनत्व पर 0.005 + 0.015% सरफेसटैंट के रूप में बाढ़ आ गई थी। रोपण को बाढ़ के 7-10 दिन बाद फेनोटाइप किया गया था(चित्रा 5)। मनीमेकर-टॉर और मनीमेकर जैसी जेनोआमतौर पर समरूप रेखाओं से व्यक्तिगत रोपण-PtoS रोपण बाढ़ परख में अत्यधिक सुसंगत और बाइनरी फेनोटाइप देते हैं। जबमनीमेकर-PtoR,जो Pto/Prf जीन क्लस्टर (एन = 5) किया जाता है, पीटीएसDC3000 के साथ ओडी६०० = ०.००५ की इष्टतम एकाग्रता पर इलाज किया गया था, PtoRके कारण प्रतिरोध-मध्यस्थता प्रतिरक्षा मजबूत था और सभी व्यक्तियों में नए, हरे वनस्पति वृद्धि द्वारा टाइप किया गया था22। निकट-आइसोजेनिक मनीमेकर-PtoS रोपण (एन = 5), जो पीएसटीDC3000 प्रभावकों AvrPto या AvrPtoB को पहचान नहीं सकता है, बाढ़ के बाद 7 दिनों के भीतर जल्दी से मर गया और विशेषता ब्राउन एपिकल मेरिस्टेम, बैक्टीरियल स्पेक, क्लोरोसिस, और नए, हरे वनस्पति विकास(चित्र 5)के कोई संकेत नहीं था।

चित्रा 5: प्रतिरोध या रोग के लक्षणों का फेनोटाइपिक लक्षण वर्णन 7-10 दिन के बाद एक खेती में संक्रमण । मनीमेकर-टॉर और मनीमेकर- पी. सिरिंगपीवी से भर जाने से पहले 10 दिन तक 0.5x एमएस प्लेटों परPtoS टमाटर की पौध उगाई गई थी। टमाटर DC3000 (OD600 = 0.005) + 0.015% surfactant। मनीमेकर-टॉर रोपण बच गया (एन = 5) और मनीमेकर-PtoS रोपण (n = 5) की मृत्यु हो गई। परीक्षण की गई कुल संख्या में से प्रत्येक जीनोटाइप के लिए जीवित रोपण की संख्या दिखाई गई है। स्केल बार = 1 सेमी. कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
अंकुर प्रतिरोध परख का उपयोग कर जंगली परिग्रहण की फेनोटाइपिक स्क्रीनिंग
चित्रा 6 पीएसटीT1 के साथ बाढ़ के बाद 10-14 दिनों के अतिसंवेदनशील और प्रतिरोधी परिग्रहण के रोपण के लिए प्रतिनिधि परिणाम दिखाता है । अतिसंवेदनशील परिग्रहण मेंआरजी-टॉर, एस पिंपिनेलीफोलियम LA1375, और एस पिंपिनेल्लीफोलियम LA1606 शामिल हैं, और प्रतिरोधी परिग्रहण में एस नियोरिकी LA1329 शामिल हैं। दस दिन पुरानी रोपण में नकारात्मक नियंत्रण के रूप में 10 एमएम एमजीसीएल2 + 0.015% सर्फेक्टेंट और 0.0075 + 0.015% सरफेक्ट के ऑप्टिकल घनत्व पर 10 एम एम एमजीसीएल 2 + 0.015% सर्फेक्टेंट की भरमार हो गई थी। रोपण को बाढ़ के कम से 10 दिन बाद फेनोटाइप किया गया था, क्योंकि पीएसटीटी1-संक्रमित रोपण पीएसटीDC3000-संक्रमित रोपण की तुलना में अधिक धीरे से मर गया । नकली टीका रोपण हरे, स्वस्थ और सक्रिय रूप से बढ़ रहे थे। यह नियंत्रण यह सुनिश्चित करने के लिए महत्वपूर्ण है कि परिग्रहण सर्फेक्टेंट की एकाग्रता के प्रति संवेदनशील नहीं हैं, और यह सुनिश्चित करने के लिए कि कोई जीवाणु संदूषण नहीं है। अतिसंवेदनशील परिग्रहण (रियोग्रांडे-टॉर [एन = 7], एस पिंपिनेलीफोलियम LA1375 [n = 7], और एस पिंपिनेलिफोलियम LA1606 [n = 5]) मर चुके थे, भूरे रंग के एपिकल मेरिस्टेम थे, और पीएसटीटी 1 के साथ टीका लगाने के 10-14 दिनों के बाद नई वृद्धि का अभाव था। इसके विपरीत, दो एस neorickii LA1329 (n = 3) रोपण नए, हरे रंग के विकास के एक उच्च स्तर का प्रदर्शन किया और पीएसटीT1(चित्रा 6)के साथ संक्रमण बच गया । तीन LA1329 रोपण अंकुरित नहीं हुए । आमतौर पर, जनसंख्या में प्रतिरोध की व्यापकता निर्धारित करने के लिए प्राथमिक स्क्रीन में प्रत्येक राज्यारोहण के लिए 5-7 व्यक्तियों की जांच की गई थी। जब LA1329 जैसे अधिक आनुवंशिक रूप से जटिल जंगली परिग्रहण, पीएसटीटी 1 से भर जाता है, तो प्रतिरोध फेनोटाइप व्यक्तिगत रोपण के बीच थोड़ा अधिक परिवर्तनशीलता प्रदर्शित करता है, मनीमेकर-PtoR की तुलना में पीएसटीDC3000 के साथ इलाज किया जाता है। हालांकि, प्रतिरोध फेनोटाइप आमतौर पर F2 मानचित्रण आबादी में देखे गए लोगों की तुलना में कम परिवर्तनशील थे। इस प्रकार, LA1329 के लिए बाइनरी फेनोटाइपिंग मानदंड ों का उपयोग किया गया था।

चित्रा 6: प्रतिरोध या रोग के लक्षणों का फेनोटाइपिक लक्षण वर्णन जंगली परिग्रहण में संक्रमण के बाद 10-14 दिन। रियो ग्रांडे-टॉर, एस पिंपिनेलीफोलियम LA1606, एस पिंपिनेलीफोलियम LA1375 और एस neorickii LA1329 टमाटर रोपण 10 दिनों के लिए 0.5x एमएस प्लेटों पर उगाए गए थे, और फिर पीएसटीटी 1 (ओडी600 = 0.0075) + 0.015% सरेक्टन के साथ बाढ़ आ गई। परीक्षण की गई कुल संख्या में से प्रत्येक जंगली राज्यारोहण के लिए जीवित रोपण की संख्या दिखाई गई है। स्केल बार = 1 सेमी. कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
अंकुर बाढ़ परख का उपयोग कर जीवाणु विकास का मात्रात्मक आकलन
इस बात की पुष्टि करने के लिए कि LA1329 में पीएसटीटी1 में मनाया प्रतिरोध कम जीवाणु वृद्धि के परिणामस्वरूप, टमाटर रोपण में बैक्टीरियल विकास परख किया गया । मनीमेकर-पेटोस और एस नेरिकि लाई एल1329 में पीएसटीटी1 वृद्धि का स्तर संक्रमण के बाद 4 दिन निर्धारित किया गया था। मनीमेकर-पीटीओ एक निकट-आइसोजेनिक रेखा है जिसमें व्यक्तिगत रोपण के बीच लगातार संवेदनशीलता होती है। एस नियोरिकी LA1329 जैसे जंगली परिग्रहण अक्सर अधिक आनुवंशिक रूप से जटिल होते हैं। LA1329 जनसंख्या22भर में पीएसटीटी 1 के लिए लगभग 60% प्रतिरोध प्रदर्शित करता है . क्योंकि रोपण संक्रमण के बाद अपने cotyledons छोड़ सकते हैं, एक अंकुर प्रत्येक थाली पर उगाया गया था समग्र अंकुर अस्तित्व या मौत के रूप में निर्धारित phenotypical के रूप में निर्धारित करने के लिए काटा cotyledon में जीवाणु विकास सहसंबंधित के रूप में निर्धारित करने के लिए बाढ़ के बाद कम से 10 दिन । प्रत्येक अंकुर के लिए 4 दिन पर जीवाणु गिनती ऊतक के ०.०१ ग्राम करने के लिए सामान्यीकृत किया गया और लॉग विकास (CFU/0.01 ग्राम (लॉग10)में परिवर्तित) । फेनोआमतौर पर प्रतिरोधी LA1329 रोपण (LA1329RES)या phenotypically अतिसंवेदनशील रोपण (LA1329SUS)के लिए लॉग ग्रोथ अलग से जमा और एक दूसरे और अतिसंवेदनशील खेती मनीमेकर-PtoS की तुलना में थे । उदाहरण के लिए, LA1329RES (लॉग 6.3) और LA1329SUS (लॉग 8.0) के बीच बैक्टीरियल विकास में 1.7 लॉग अंतर था, और LA1329आरईएस (लॉग 6.3) और मनीमेकर-PtoS (7.9) (चित्रा 7)के बीच 1.6 लॉग अंतर था। इसलिए, फेनोटाइपिक प्रतिरोध अंकुर परखों में मात्रात्मक प्रतिरोध के साथ सहसंबद्ध है।

चित्रा 7: प्रतिरोधी सोलनम नियोरिकी LA1329 रोपण मनीमेकर-टॉस या अतिसंवेदनशील एस नियोरिकी LA1329 की तुलना में कम बैक्टीरियल विकास का समर्थन करते हैं। बैक्टीरियल काउंट एस नियोरिकी LA1329 (n = 14) और मनीमेकर-PtoS (n = 10) पीएसटीT1 से संक्रमित रोपण और सामान्यीकरण से टीका के बाद 4 दिन निर्धारित किए गए थे। LA1329 के लिए, दो फेनोटाइपिक समूह, अतिसंवेदनशील (SUS) या प्रतिरोधी (आरईएस), मनाया और अलग से गिना गया । बार के ऊपर * = सांख्यिकीय महत्वपूर्ण अंतर विचरण के एक कारक विश्लेषण द्वारा निर्धारित। एक सामान्य रैखिक मॉडल प्रक्रिया (पी एंड एलटी; 0.001) जिसके बाद तुकी के पोस्ट हॉक परीक्षण का उपयोग करने वाले साधनों की एक ाधिक तुलना की गई थी। त्रुटि सलाखों = मानक त्रुटि। यह आंकड़ा एक प्रतिनिधि प्रयोग को इंगित करता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
Subscription Required. Please recommend JoVE to your librarian.
Discussion
टमाटर के रोपण में इन जीवाणु उपभेदों के प्रतिरोध का पता लगाने के लिए अनुकूलित पीएसटीDC3000 या पीएसटीT1 के साथ बाढ़ टीका के लिए एक प्रोटोकॉल वर्णित है। रोपण प्रतिरोध परख में इष्टतम परिणामों के लिए कई महत्वपूर्ण पैरामीटर हैं, जिनमें बैक्टीरियल एकाग्रता और सर्फेक्टेंट एकाग्रता शामिल है, जो अनुभवजन्य रूप से22निर्धारित किए गए थे। पीएसटीDC3000 के लिए, ऑप्टिकल घनत्व को एक प्रतिरोधी पुलिया पर पूर्ण अस्तित्व प्राप्त करने के लिए अनुकूलित किया गया था जिसमें Pto/Prf क्लस्टर शामिल है और Pto/Prf क्लस्टर22की कमी वाली अतिसंवेदनशील खेती पर पूर्ण मृत्यु हुई थी । पीएसटीटी 1 जैसे तनाव के लिए, जहां कोई ज्ञात प्रतिरोधी किस्में नहीं हैं, ऑप्टिकल घनत्व को सुसंगत और पूर्ण पौधों की मृत्यु22के लिए सबसे कम संभव होने के लिए अनुकूलित किया गया था। उपापालापति एट अल24 ने पीएसटीDC3000 के रोगजनन और कोरोटिन के उग्रता समारोह की जांच करने के लिए टमाटर की अंकुर परख तैयार की । इस उग्रता परख में, हमारे प्रतिरोध परख में उपयोग किए जाने वाले उपभेदों के ऑप्टिकल घनत्व से 20x अधिक 0.124के ओडी600 पर केंद्रित बैक्टीरिया का उपयोग करके संक्रमण किए गए थे। पीएसटी DC3000 प्रभावकों AvrPto और AvrPtoB की मान्यता टोमैटो रोपण में Pto/Prf जीन क्लस्टर ले जाने के परिणामस्वरूप ईटीआई और एक स्थूल एचआर22। ईटीआई जैसी मजबूत प्रतिरक्षा प्रतिक्रिया के संदर्भ में, पीएसटीDC3000 के लिए एक कम बैक्टीरियल टिटर का उपयोग Pto/Prf जीन क्लस्टर22से भारी आनुवंशिक प्रतिरोध से बचने के लिए किया गया था । इसके अलावा, इन परिणामों से पता चलता है कि एक उच्च जीवाणु एकाग्रता पीटीआई या मात्रात्मक आंशिक प्रतिरोध जैसी कमजोर प्रतिरक्षा प्रतिक्रियाओं को अभिभूत कर सकती है, जहां कई जीन समग्र फेनोटाइप में योगदान देते हैं। बैक्टीरिया के लिए पत्ते की सतह का पालन करने के लिए सर्फेक्टेंट आवश्यक है; हालांकि, उच्च सांद्रता पत्ती22के क्लोरोसिस पैदा कर सकता है . हमने पहले 10 दिन पुराने टमाटर के रोपण22में आदर्श एकाग्रता निर्धारित करने के लिए सर्फेक्टेंट सांद्रता की एक श्रृंखला का परीक्षण किया था। जब नई प्रजातियों का परीक्षण किया जा सकता है जो सर्फेक्टेंट के प्रति उनकी संवेदनशीलता में भिन्न हो सकती हैं, तो एक एकाग्रता की पहचान करने के लिए सर्फेक्टेंट एकाग्रता को अनुकूलित किया जाना चाहिए जो बैक्टीरिया की अनुपस्थिति में नुकसान या क्लोरोसिस का कारण नहीं बनता है। उचित परख की स्थिति के लिए एक सर्फेक्टेंट एकाग्रता के अनुकूलन की आवश्यकता होगी जो नुकसान का कारण नहीं बनती है, और एक जीवाणु एकाग्रता जो सभी अतिसंवेदनशील नियंत्रणों में बीमारी का कारण बनती है।
रोपण बाढ़ परख में सफलता के लिए अतिरिक्त महत्वपूर्ण मापदंडों में विशिष्ट विकासात्मक चरणों (10 दिन पुरानी रोपण)(चित्रा 1)पर रोपण का उपयोग करना, स्थिर विकास कक्ष की स्थिति (लगभग 200 माइक्रोई मीटर-2 एस-1की हल्की तीव्रता, 22 डिग्री सेल्सियस का निरंतर तापमान, 16 घंटे का प्रकाश) और एक बाँझ जैव सुरक्षा कैबिनेट में प्रयोग करना शामिल है। 45 मीटर से ऊपर या 35 मीटर से नीचे मीडिया की मात्रा अतिसंवेदनशील नियंत्रण ों की लगातार मौत को प्रभावित कर सकती है, क्योंकि मात्रा प्लेट पर रोपण के आसपास के माइक्रोएनवायरमेंट को प्रभावित कर सकती है। उदाहरण के लिए, सील प्लेटों के अंदर सापेक्ष आर्द्रता में अंतर बैक्टीरिया की संक्रमणता और पौधों के संक्रमण से बचने की क्षमता को प्रभावित कर सकता है। बाँझ तकनीक महत्वपूर्ण है, क्योंकि प्लेटों पर संदूषण रोपण में मृत्यु या संवेदनशीलता के स्रोत को चकित कर सकता है। इसके अलावा, क्योंकि पौधे-रोगजनक बातचीत सर्कैडियन घड़ी24,25,26से प्रभावित होती है, यह सिफारिश की जाती है कि पौधे दिन के लगातार समय पर संक्रमित हों।
पीएसटी एक फोलियार रोगजनक है जो अधिमानतः टमाटर के रोपण के हवाई हिस्सों को उपनिवेश करता है, जिसमें कोटाइलेडन24 (चित्रा 3)शामिल है। इसलिए, अंकुर बाढ़ परख में गुणात्मक फेनोटाइपिंग अंकुर के हवाई भागों में विकास और रोग के लक्षणों पर केंद्रित है, और बैक्टीरियल विकास परख के लिए ऊतक मात्रात्मक विश्लेषण के लिए कोटाइलेडन से नमूना है। बाढ़ टीका के बाद, रोपण पीएसटीDC3000 या 10-14 दिनों के साथ टीका के बाद 7-10 दिनों के भीतर मर सकता है PstT1 के साथ टीका के बाद, के रूप में धारा 11 में चर्चा की । अंकुर मौत एक भूरे रंग के apical meristem द्वारा कल्पना की है, गिरफ्तार epicotyl विस्तार, और/ यदि विभिन्न जीवाणु उपभेदों का उपयोग किया जाता है, तो समय को अनुभवजन्य रूप से निर्धारित करना होगा। इसके अलावा, नियंत्रण पौधों पर रोग की प्रगति बाढ़ के बाद दैनिक निगरानी की जानी चाहिए जब तक रोग के लक्षणों की शुरुआत से एक सुसंगत समय सीमा से रोपण मौत की पहचान की जा सकती है । बाढ़ परख में उपयोग किए जाने वाले जीनोटाइप और उपचारों के आधार पर, रोपण फेनोटाइप को बाइनरी फेनोटाइप के रूप में या रोग स्पेक्ट्रम(चित्रा 4)पर दर्ज किया जा सकता है। जब बाढ़ जंगली टमाटर परिग्रहण से F2 मानचित्रण आबादी को अतिसंवेदनशील खेती(चित्रा 4सी)को पार कर गया तो फेनोटाइप का एक व्यापक स्पेक्ट्रम देखा जा सकता है। यह एक रोग स्पेक्ट्रम पर आबादी को अलग करने के लिए सबसे अच्छा हो सकता है कितनी जल्दी अंकुर मर जाता है और नए वनस्पति विकास और शाखाओं की डिग्री(चित्रा 4सी)के आधार पर । अंकुर बाढ़ परख का उपयोग अंकुर जीवाणु विकास परख के साथ मिलकर व्यक्तिगत रोपण(चित्रा 7)में गुणात्मक फेनोटाइप से जुड़े जीवाणु विकास के स्तर का मात्रात्मक आकलन करने के लिए भी किया जा सकता है। बहुत बड़ी कटौती (यानी, ~ लॉग 3) जीवाणु विकास में या एक अतिसंवेदनशील खेती की तुलना में एक जंगली परिग्रहण के प्रतिरोधी रोपण में मजबूत प्रतिरोध का सुझाव है कि प्रतिरोध के अंतर्निहित आनुवंशिक आधार ईटीआई22के कारण हो सकता है । बैक्टीरियल विकास में छोटी कटौती (यानी, ~ लॉग 1.7), जैसा कि LA1329 रोपण में मनाया गया है, मात्रात्मक विशेषता लोकी और/या पीटीआई से कमजोर प्रतिरोध के योगदान के कारण हो सकता है। इस प्रकार, रोपण विकास परख जंगली टमाटर लाइनों में आगे प्रतिरोध की विशेषता में एक महत्वपूर्ण उपकरण हो सकता है ।
आमतौर पर, आनुवंशिक स्क्रीन जंगली परिग्रहण20,21में पी सिरिंग प्रतिरोध के आनुवंशिक आधार की पहचान करने के लिए चार से पांच सप्ताह पुराने वयस्क टमाटर के पौधों पर प्रदर्शन किया गया है । वयस्क टमाटर के पौधों को बहुत लंबे समय तक विकास के समय की आवश्यकता होती है, विकास कक्ष में अधिक स्थान की आवश्यकता होती है, और बहुत बड़े पौधे होते हैं, जिसका अर्थ है कि आमतौर पर प्रत्येक पंक्ति के लिए कुछ व्यक्तियों की जांच की जाती है। अंकुर बाढ़ परख जंगली टमाटर परिग्रहण में पी सिरिंग प्रतिरोध की पहचान में एक शक्तिशाली, वैकल्पिक दृष्टिकोण प्रदान करता है । अंकुर चरण में स्क्रीनिंग एक बड़े नमूना आकार का परीक्षण करने की अनुमति देती है जो आनुवंशिक रूप से जटिल आबादी में प्रतिरोध का पता लगाने में विशेष रूप से लाभप्रद हो सकता है । कम विकास कक्ष अंतरिक्ष आवश्यकताओं और विकास के समय एक उच्च थ्रूपुट दृष्टिकोण और उभरते रोगजनकों के लिए जंगली परिग्रहण में प्राकृतिक प्रतिरोध का तेजी से पता लगाने की सुविधा । इसके अलावा, पी सिरिंग प्रतिरोध है कि इस परख में अंकुर चरण में पहचान की थी विकास के स्तर तक ही सीमित नहीं है । एस नियोरिकी LA1329 और एस habrochaites LA1253 शुरू में अंकुर चरण में पहचान की गई थी और वयस्क पौधों में पीएसटीटी 1 के प्रतिरोध को भी प्रदर्शित किया गया था जैसा कि पहले22वर्णित था।
अंकुर बाढ़ परख एक बहुमुखी प्रोटोकॉल है जिसे अन्य पी सिरिंग उपभेदों के मेजबान प्रतिरोध का पता लगाने के लिए संशोधित और अनुकूलित किया जा सकता है। यह संभावित रूप से टमाटर के विभिन्न जीवाणु रोगजनकों के संदर्भ में लागू किया जा सकता है, जैसे Xanthomonas प्रजातियों। इस विधि से बैक्टीरियल रोगजनकों के रोग प्रतिरोध के नए स्रोतों की खोज में तेजी आएगी।
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
लेखकों के पास खुलासा करने के लिए कुछ नहीं है ।
Acknowledgments
हम रोग या प्रतिरोध परिणामों पर मीडिया की मात्रा के प्रभाव का परीक्षण करने के लिए जेमी Calma शुक्रिया अदा करते हैं । हम पांडुलिपि पर रचनात्मक टिप्पणियां और सुझाव प्रदान करने के लिए लुईस लैब से डॉ Maël Baudin और डॉ कार्ल जे Scheiber शुक्रिया अदा करते हैं । लुईस प्रयोगशाला में पौधों की प्रतिरक्षा पर अनुसंधान USDA ARS 2030-21000-046-00D और 2030-21000-050-00D (JDL), और एनएसएफ जैविक विज्ञान IOS के लिए निदेशालय-1557661 (जेडीएल) द्वारा समर्थित था ।
Materials
| Name | Company | Catalog Number | Comments |
| 3M Tape Micropore 1/2" x 10 YD CS 240 (1.25 cm x 9.1 m) | VWR International | 56222-182 | |
| 3mm borosilicate glass beads | Friedrich & Dimmock | GB3000B | |
| Bacto peptone | BD | 211677 | |
| Bacto agar | BD | 214010 | |
| Biophotometer Plus | Eppendorf | E952000006 | |
| Biosafety cabinet, class II type A2 | |||
| BRAND Disposable Plastic Cuvettes, Polystyrene | VWR International | 47744-642 | |
| Chenille Kraft Flat Wood Toothpicks | VWR International | 500029-808 | |
| cycloheximide | Research Products International | C81040-5.0 | |
| Dibasic potassium phosphate anhydrous, ACS grade | Fisher Scientific | P288-500 | |
| Dimethylformamide | |||
| Dissecting microscope (Magnification of at least 10x) | |||
| Ethanol - 190 Proof | |||
| Falcon polystyrene 96 well microplates, flat-bottom | Fisher Scientific | 08-772-3 | |
| Glass Alcohol Burner Wick | Fisher Scientific | S41898A / No. W-125 | |
| Glass Alcohol Burners | Fisher Scientific | S41898 / No. BO125 | |
| Glycerol ACS reagent | VWR International | EMGX0185-5 | |
| Kimberly-Clark™ Kimtech Science™ Kimwipes™ Delicate Task Wipers | Fisher Scientific | 06-666-A | |
| Magnesium chloride, ACS grade | VWR International | 97061-356 | |
| Magnesium sulfate heptahydrate, ACS grade | VWR International | 97062-130 | |
| Microcentrifuge tubes, 1.5 mL | |||
| Microcentrifuge tubes, 2.2 mL | |||
| Mini Beadbeater-96, 115 volt | Bio Spec Products Inc. | 1001 | |
| Murashige & Skoog, Basal Salts | Caisson Laboratories, Inc. | MSP01-50LT | |
| Pipet-Lite XLS LTS 8-CH Pipet 20-200uL | Rainin | L8-200XLS | |
| Pipet-Lite XLS LTS 8-CH Pipet 2-20uL | Rainin | L8-20XLS | |
| Polystyrene 100mm x 25mm sterile petri dish | VWR International | 89107-632 | |
| Polystyrene 150mm x 15mm sterile petri dish | Fisher Scientific | FB08-757-14 | |
| Polystyrene 150x15mm sterile petri dish | Fisher Scientific | 08-757-148 | |
| Pure Bright Germicidal Ultra Bleach 5.7% Available Chlorine (defined as 100% bleach) | Staples | 1013131 | |
| Rifampicin | Gold Biotechnology | R-120-25 | |
| Silwet L-77 (non-ionic organosilicone surfactant co-polymer C13H34O4Si3 surfactant) | Fisher Scientific | NCO138454 | |
| Tips LTS 20 μL 960/10 GPS-L10 | Rainin | 17005091 | |
| Tips LTS 250 μL 960/10 GPS-L250 | Rainin | 17005093 | |
| VWR dissecting forceps fine tip, 4.5" | VWR International | 82027-386 |
References
- Underwood, W., Melotto, M., He, S. Y. Role of plant stomata in bacterial invasion. Cell Microbiology. 9 (7), 1621-1629 (2007).
- Zipfel, C. Early molecular events in PAMP-triggered immunity. Current Opinion in Plant Biology. 12 (4), 414-420 (2009).
- Galan, J. E., Wolf-Watz, H. Protein delivery into eukaryotic cells by type III secretion machines. Nature. 444 (7119), 567-573 (2006).
- Lewis, J. D., Desveaux, D., Guttman, D. S. The targeting of plant cellular systems by injected type III effector proteins. Seminars in Cell and Developmental Biology. 20 (9), 1055-1063 (2009).
- Schreiber, K. J., Baudin, M., Hassan, J. A., Lewis, J. D. Die another day: molecular mechanisms of effector-triggered immunity elicited by type III secreted effector proteins. Seminars in Cell and Developmental Biology. 56, 124-133 (2016).
- Heath, M. C. Hypersensitive response-related death. Plant Molecular Biology. 44 (3), 321-334 (2000).
- Boyd, L. A., Ridout, C., O'Sullivan, D. M., Leach, J. E., Leung, H. Plant-pathogen interactions: disease resistance in modern agriculture. Trends in Genetics. 29 (4), 233-240 (2013).
- Pitblado, R. E., MacNeill, B. H. Genetic basis of resistance to Pseudomonas syringae pv. tomato in field tomatoes. Canadian Journal of Plant Pathology. 5 (4), 251-255 (1983).
- Pedley, K. F., Martin, G. B. Molecular basis of Pto-mediated resistance to bacterial speck disease in tomato. Annual Reviews of Phytopathology. 41, 215-243 (2003).
- Ronald, P. C., Salmeron, J. M., Carland, F. M., Mehta, A. Y., Staskawicz, B. J. The cloned avirulence gene AvrPto induces disease resistance in tomato cultivars containing the Pto resistance gene. Journal of Bacteriology. 174 (5), 1604-1611 (1992).
- Martin, G. B., et al. Map-based cloning of a protein kinase gene conferring disease resistance in tomato. Science. 262 (5138), 1432-1436 (1993).
- Salmeron, J. M., Barker, S. J., Carland, F. M., Mehta, A. Y., Staskawicz, B. J. Tomato mutants altered in bacterial disease resistance provide evidence for a new locus controlling pathogen recognition. Plant Cell. 6 (4), 511-520 (1994).
- Salmeron, J. M., et al. Tomato Prf is a member of the leucine-rich repeat class of plant disease resistance genes and lies embedded within the Pto kinase gene cluster. Cell. 86 (1), 123-133 (1996).
- Scofield, S. R., et al. Molecular basis of gene-for-gene specificity in bacterial speck disease of tomato. Science. 274 (5295), 2063-2065 (1996).
- Kunkeaw, S., Tan, S., Coaker, G. Molecular and evolutionary analyses of Pseudomonas syringae pv. tomato race 1. Molecular Plant-Microbe Interactions. 23 (4), 415-424 (2010).
- Cai, R., et al. The plant pathogen Pseudomonas syringae pv. tomato is genetically monomorphic and under strong selection to evade tomato immunity. PLoS Pathogens. 7 (8), 1002130 (2011).
- Almeida, N. F., et al. A draft genome sequence of Pseudomonas syringae pv. tomato T1 reveals a type III effector repertoire significantly divergent from that of Pseudomonas syringae pv. tomato DC3000. Molecular Plant-Microbe Interactions. 22 (1), 52-62 (2009).
- Lin, N. C., Abramovitch, R. B., Kim, Y. J., Martin, G. B. Diverse AvrPtoB homologs from several Pseudomonas syringae pathovars elicit Pto-dependent resistance and have similar virulence activities. Applied and Environmental Microbiology. 72 (1), 702-712 (2006).
- Rose, L. E., Langley, C. H., Bernal, A. J., Michelmore, R. W. Natural variation in the Pto pathogen resistance gene within species of wild tomato (Lycopersicon). I. Functional analysis of Pto alleles. Genetics. 171 (1), 345-357 (2005).
- Thapa, S. P., Miyao, E. M., Davis, R. M., Coaker, G. Identification of QTLs controlling resistance to Pseudomonas syringae pv. tomato race 1 strains from the wild tomato Solanum habrochaites LA1777. Theoretical and Applied Genetics. 128 (4), 681-692 (2015).
- Bao, Z. L., et al. Identification of a candidate gene in Solanum habrochaites for resistance to a race 1 strain of Pseudomonas syringae pv. tomato. Plant Genome. 8 (3), 1-15 (2015).
- Hassan, J. A., Zhou, Y. J., Lewis, J. D. A rapid seedling resistance assay identifies wild tomato lines that are resistant to Pseudomonas syringae pv. tomato race 1. Molecular Plant-Microbe Interactions. 30 (9), 701-709 (2017).
- King, E. O., Ward, M. K., Raney, D. E. Two simple media for the demonstration of pyocyanin and fluorescin. Journal of Laboratory and Clinical Medicine. 44 (2), 301-307 (1954).
- Uppalapati, S. R., et al. Pathogenicity of Pseudomonas syringae pv. tomato on tomato seedlings: phenotypic and gene expression analyses of the virulence function of coronatine. Molecular Plant-Microbe Interactions. 21 (4), 383-395 (2008).
- Bhardwaj, V., Meier, S., Petersen, L. N., Ingle, R. A., Roden, L. C. Defence responses of Arabidopsis thaliana to infection by Pseudomonas syringae are regulated by the circadian clock. PLoS One. 6 (10), 26968 (2011).
- Lu, H., McClung, C. R., Zhang, C. Tick tock: circadian regulation of plant innate immunity. Annual Review of Phytopathology. 55, 287-311 (2017).
- Wang, W., et al. Timing of plant immune responses by a central circadian regulator. Nature. 470 (7332), 110-114 (2011).
Tags
बायोइंजीनियरिंग इश्यू 157 स्यूडोमोनास सिरिंग,टमाटर रोग प्रतिरोध स्क्रीन बैक्टीरियल ग्रोथ फेनोटाइपErratum
Formal Correction: Erratum: High-Throughput Identification of Resistance to Pseudomonas syringae pv. Tomato in Tomato using Seedling Flood Assay
Posted by JoVE Editors on 10/18/2023.
Citeable Link.
An erratum was issued for: High-Throughput Identification of Resistance to Pseudomonas syringae pv. Tomato in Tomato using Seedling Flood Assay. The Introduction, Protocol, Representative Results and Discussion sections were updated.
The last paragraph of the Introduction section was updated from:
In the seedling flood assay described in this protocol, tomato seedlings are grown on Petri dishes of sterile Murashige and Skoog (MS) media for 10 days and then are flooded with an inoculum containing the bacteria of interest and a surfactant. Following flooding, seedlings can be quantitatively evaluated for disease resistance via bacterial growth assays. Additionally, seedling survival or death can act as a discrete resistance or disease phenotype 7–14 days after flooding. This approach offers a high-throughput alternative for screening large numbers of wild tomato accessions for resistance to Pst race 1 strains, such as Pst strain T1 (PstT1), and can easily be adapted to other bacterial strains of interest.
to:
In the seedling flood assay described in this protocol, tomato seedlings are grown on Petri dishes of sterile Murashige and Skoog (MS) media for 10 days and then are flooded with an inoculum containing the bacteria of interest and a surfactant. Following flooding, seedlings can be quantitatively evaluated for disease resistance via bacterial growth assays. Additionally, seedling survival or death can act as a discrete resistance or disease phenotype 7–14 days after flooding. This approach offers a high-throughput alternative for screening large numbers of wild tomato accessions for resistance to Pst race 1 strains, such as Pst strain 19 (Pst19), and can easily be adapted to other bacterial strains of interest.
Step 4.8 of the Protocol section was updated from:
- After 1 h, transfer the bottle to the biosafety cabinet and under aseptic conditions, add 1,600 µL of sterile 1 M MgSO4, and appropriate antibiotics to the media.
NOTE: For rifampicin resistant strains PstDC3000 and PstT1, use rifampicin dissolved in dimethylformamide at a final concentration of 50 µg/mL. Use cycloheximide dissolved in ethanol at a final concentration of 50 µg/mL to prevent fungal growth on the plates.
to:
- After 1 h, transfer the bottle to the biosafety cabinet and under aseptic conditions, add 1,600 µL of sterile 1 M MgSO4, and appropriate antibiotics to the media.
NOTE: For rifampicin resistant strains PstDC3000 and Pst19, use rifampicin dissolved in dimethylformamide at a final concentration of 50 µg/mL. Use cycloheximide dissolved in ethanol at a final concentration of 50 µg/mL to prevent fungal growth on the plates.
Step 5.2 of the Protocol section was updated from:
- Patch bacteria (i.e., PstT1) from a glycerol stock onto KB agar with appropriate antibiotics (section 4).
to:
- Patch bacteria (i.e., Pst19) from a glycerol stock onto KB agar with appropriate antibiotics (section 4).
Step 5.6 of the Protocol section was updated from:
- For PstT1, incubate the KB plate at 28 ˚C for 48 h prior to using bacteria in the flood experiment.
to:
- For Pst19, incubate the KB plate at 28 ˚C for 48 h prior to using bacteria in the flood experiment.
Step 6 of the Protocol section was updated from:
6. Preparation of PstT1 inoculum
to
6. Preparation of Pst19 inoculum
Step 6.2 of the Protocol section was updated from:
- Perform serial dilutions using sterile 10 mM MgCl2 solution in the biosafety cabinet. For PstT1, use a spectrophotometer to make inoculum with a starting concentration of OD600 = 0.1.
to:
- Perform serial dilutions using sterile 10 mM MgCl2 solution in the biosafety cabinet. For Pst19, use a spectrophotometer to make inoculum with a starting concentration of OD600 = 0.1.
Step 6.3 of the Protocol section was updated from:
- For PstT1, make a 1/10 dilution from the initial resuspension at OD600 = 0.1 to obtain a serial dilution at a concentration of OD600 = 0.01.
to:
- For Pst19, make a 1/10 dilution from the initial resuspension at OD600 = 0.1 to obtain a serial dilution at a concentration of OD600 = 0.01.
Step 8.3 of the Protocol section was updated from:
- Set a timer for 3 min. Measure 6 mL of final inoculum (PstT1 OD600 = 0.0075 [section 6] or PstDC3000 OD600 = 0.005 [section 7]) and transfer 6 mL of inoculum to each plate with the 10-day-old seedlings.
to:
- Set a timer for 3 min. Measure 6 mL of final inoculum (Pst19 OD600 = 0.0075 [section 6] or PstDC3000 OD600 = 0.005 [section 7]) and transfer 6 mL of inoculum to each plate with the 10-day-old seedlings.
Step 8.11 of the Protocol section was updated from:
- Phenotype after 7–10 days for PstDC3000 or 10–14 days for PstT1 (section 11). If carrying out bacterial growth assays, collect leaf tissue after 4 days (sections 9 and 10) and then phenotype (section 11). Alternatively, perform phenotypic analysis and bacterial growth assays on separate sets of plants.
to:
- Phenotype after 7–10 days for PstDC3000 or 10–14 days for Pst19 (section 11). If carrying out bacterial growth assays, collect leaf tissue after 4 days (sections 9 and 10) and then phenotype (section 11). Alternatively, perform phenotypic analysis and bacterial growth assays on separate sets of plants.
Step 10.7 of the Protocol section was updated from:
- After obtaining colony counts (Figure 2B), normalize the counts to 0.01 g of tissue for seedlings and convert to log bacterial growth (Table 1).
NOTE: The average mass of one Moneymaker-PtoS cotyledon is 0.01 g and is empirically determined22.
| Genotype1 Column A | Tissue Weight (g) Column B | # of Colonies in a spot Column C | Dilution factor for spot2 Column D | Adjusted # of Colonies3 Column E | Dilution factor for serial dilution Column F | Total # of Colonies Column G (cfu/0.01 g)4 | Average # of Colonies (cfu/0.01 g) Column H | Average Log Growth (cfu/0.01 g (log10)) Column I |
| Sample 1 | 0.004 g | 10 | 200 | calculated as: (C2 x 0.01 g) / B2 = 25 | 1000 | calculated as: (D2 x E2 x F2) = 5000000 | average for sample 1 through last sample: (ie. average G1:G3) = 7000000 | log of average ie. log(H2) = 6.85 |
| Sample 2 | 0.003 g | 15 | 200 | 50 | 1000 | 10000000 | ||
| Sample 3 | 0.002 g | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1Data shown for 3 samples | ||||||||
| 2Based on plating 5 µL x 200 for 1 mL | ||||||||
| 3Cotyledons are too small to core so colony counts were normalized to 0.01 g of tissue based on the average mass of one MoneyMaker-PtoS cotyledon (data not shown) | ||||||||
| 4Adjusted per mL based on volume plated | ||||||||
Table 1: Sample calculations for seedling bacterial growth assay. Sample calculations demonstrate how to normalize bacterial counts and determine log bacterial growth.
to:
- After obtaining colony counts (Figure 2B), normalize the counts to 0.1 g of tissue for seedlings and convert to log bacterial growth (Table 1).
NOTE: The average mass of one Moneymaker-PtoS cotyledon is 0.1 g and is empirically determined22.
| Genotype1 Column A | Tissue Weight (g) Column B | # of Colonies in a spot Column C | Dilution factor for spot2 Column D | Adjusted # of Colonies3 Column E | Dilution factor for serial dilution Column F | Total # of Colonies Column G (cfu/0.01 g)4 | Average # of Colonies (cfu/0.01 g) Column H | Average Log Growth (cfu/0.1 g (log10)) Column I |
| Sample 1 | 0.04 g | 10 | 200 | calculated as: (C2 x 0.1 g) / B2 = 25 | 1000 | calculated as: (D2 x E2 x F2) = 5000000 | average for sample 1 through last sample: (ie. average G1:G3) = 7000000 | log of average ie. log(H2) = 6.85 |
| Sample 2 | 0.03 g | 15 | 200 | 50 | 1000 | 10000000 | ||
| Sample 3 | 0.02 g | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1Data shown for 3 samples | ||||||||
| 2Based on plating 5 µL x 200 for 1 mL | ||||||||
| 3Cotyledons are too small to core so colony counts were normalized to 0.1 g of tissue based on the average mass of one MoneyMaker-PtoS cotyledon (data not shown) | ||||||||
| 4Adjusted per mL based on volume plated | ||||||||
Table 1: Sample calculations for seedling bacterial growth assay. Sample calculations demonstrate how to normalize bacterial counts and determine log bacterial growth.
Step 11.3 of the Protocol section was updated from:
- Phenotype plants infected with PstT1 at 10–14 days after flood inoculation.
to:
- Phenotype plants infected with Pst19 at 10–14 days after flood inoculation.
Figure 4 in the Protocol section was updated from:
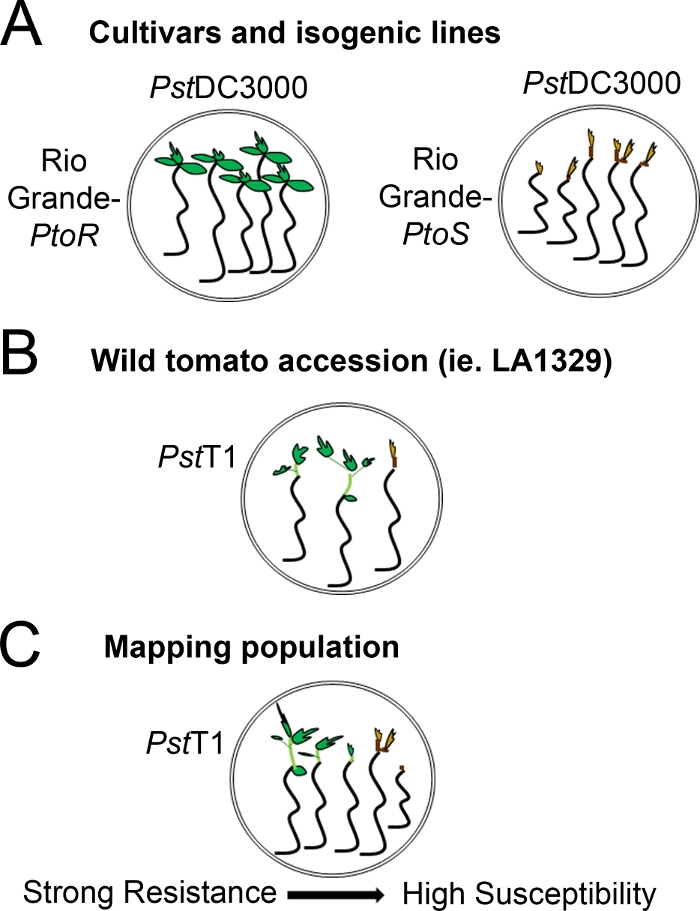
Figure 4: Schematic representation of expected phenotypes for seedling resistance and death in various genetic backgrounds. (A) Seedlings of Rio Grande-PtoR and the near-isogenic cultivar Rio Grande-PtoS are displayed 7 days after flooding with PstDC3000 (OD600 = 0.005) + 0.015% surfactant. Rio Grande-PtoR displays consistent resistance, and Rio Grande-PtoS displays consistent susceptibility to infection with PstDC3000. These lines give rise to discrete and binary phenotypes. (B) Seedlings of a wild accession, such as Solanum neorickii LA1329, are shown 10 days after flooding with PstT1 (OD600 = 0.0075) + 0.015% surfactant. Seedlings display phenotypic variability but were recorded as binary phenotypes. The amount of phenotypic variability and the method of phenotyping (binary resistance or resistance spectrum) will depend on the particular accession tested. (C) Mapping populations generated by outcrossing wild accessions to susceptible cultivars may display a wider spectrum of phenotypes in F2 segregating populations. In this case, it may be most appropriate to record seedling phenotypes on a spectrum. Highly susceptible seedlings from a mapping population may be phenotyped for death as early as day 7 when flooded with PstT1, and typically show a brown apical meristem, no to very little extension of the epicotyl, and no new, green vegetative growth. The apical meristem of susceptible seedlings may stay green or very light brown for more time, and there may be some extension of the epicotyl and very little vegetative growth, which turns brown and arrests by day 10. Individual seedlings can be phenotyped for resistance based on the amount of new and ongoing vegetative growth by day 14. Seedlings can then be grouped based on the phenotypes described above into different categories of resistance such as weak, medium, or strong resistance. Please click here to view a larger version of this figure.
to:
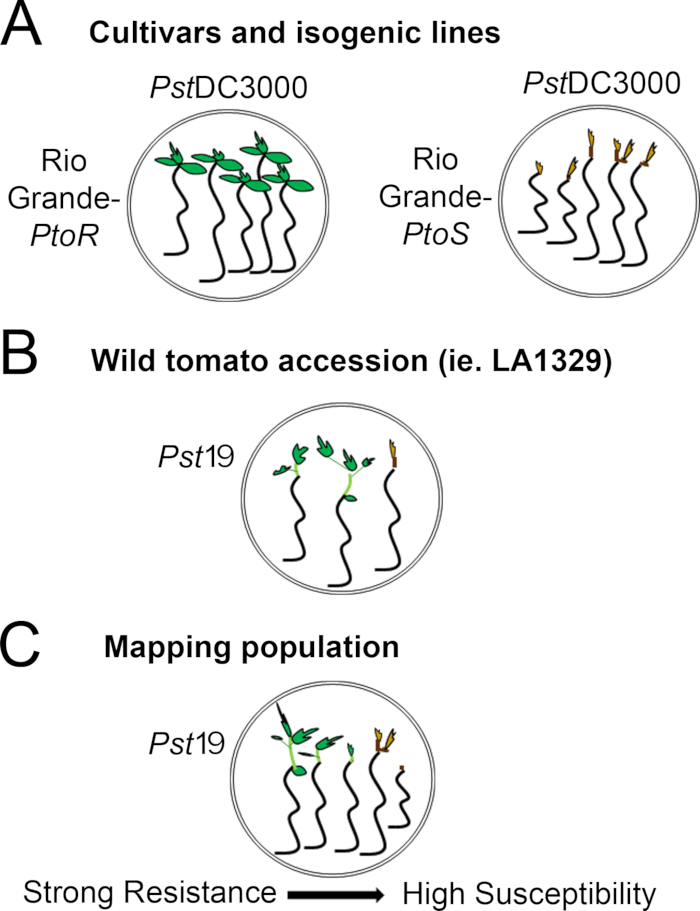
Figure 4: Schematic representation of expected phenotypes for seedling resistance and death in various genetic backgrounds. (A) Seedlings of Rio Grande-PtoR and the near-isogenic cultivar Rio Grande-PtoS are displayed 7 days after flooding with PstDC3000 (OD600 = 0.005) + 0.015% surfactant. Rio Grande-PtoR displays consistent resistance, and Rio Grande-PtoS displays consistent susceptibility to infection with PstDC3000. These lines give rise to discrete and binary phenotypes. (B) Seedlings of a wild accession, such as Solanum neorickii LA1329, are shown 10 days after flooding with Pst19 (OD600 = 0.0075) + 0.015% surfactant. Seedlings display phenotypic variability but were recorded as binary phenotypes. The amount of phenotypic variability and the method of phenotyping (binary resistance or resistance spectrum) will depend on the particular accession tested. (C) Mapping populations generated by outcrossing wild accessions to susceptible cultivars may display a wider spectrum of phenotypes in F2 segregating populations. In this case, it may be most appropriate to record seedling phenotypes on a spectrum. Highly susceptible seedlings from a mapping population may be phenotyped for death as early as day 7 when flooded with Pst19, and typically show a brown apical meristem, no to very little extension of the epicotyl, and no new, green vegetative growth. The apical meristem of susceptible seedlings may stay green or very light brown for more time, and there may be some extension of the epicotyl and very little vegetative growth, which turns brown and arrests by day 10. Individual seedlings can be phenotyped for resistance based on the amount of new and ongoing vegetative growth by day 14. Seedlings can then be grouped based on the phenotypes described above into different categories of resistance such as weak, medium, or strong resistance. Please click here to view a larger version of this figure.
The second paragraph of the Representative Results section was updated from:
Phenotypic screening of wild accessions using the seedling resistance assay
Figure 6 shows representative results for seedlings of susceptible and resistant accessions 10–14 days after flooding with PstT1. Susceptible accessions include RG-PtoR, S. pimpinellifolium LA1375, and S. pimpinellifolium LA1606, and resistant accessions include S. neorickii LA1329. Ten-day-old seedlings were flooded with 10 mM MgCl2 + 0.015% surfactant as a negative control, and PstT1 at an optical density of 0.0075 + 0.015% surfactant. The seedlings were phenotyped at least 10 days after flooding, as PstT1-infected seedlings died more slowly than PstDC3000-infected seedlings. Mock-inoculated seedlings were green, healthy, and actively growing. This control is important to ensure that the accessions are not sensitive to the concentration of surfactant, and to ensure there is no bacterial contamination. Susceptible accessions (Rio Grande-PtoR [n = 7], S. pimpinellifolium LA1375 [n = 7], and S. pimpinellifolium LA1606 [n = 5]) were dead, had brown apical meristems, and lacked new growth 10–14 days after inoculation with PstT1. In contrast, two S. neorickii LA1329 (n = 3) seedlings displayed a high level of new, green growth and survived infection with PstT1 (Figure 6). Three LA1329 seedlings did not germinate. Typically, 5–7 individuals were screened for each accession in a primary screen to determine the prevalence of resistance in the population. When a more genetically complex wild accession, such as LA1329, is flooded with PstT1, the resistance phenotypes display slightly more variability among individual seedlings, compared to Moneymaker-PtoR treated with PstDC3000. However, the resistance phenotypes were usually less variable than those seen in F2 mapping populations. Thus, binary phenotyping criteria was used for LA1329.
to:
Phenotypic screening of wild accessions using the seedling resistance assay
Figure 6 shows representative results for seedlings of susceptible and resistant accessions 10–14 days after flooding with Pst19. Susceptible accessions include RG-PtoR, S. pimpinellifolium LA1375, and S. pimpinellifolium LA1606, and resistant accessions include S. neorickii LA1329. Ten-day-old seedlings were flooded with 10 mM MgCl2 + 0.015% surfactant as a negative control, and Pst19 at an optical density of 0.0075 + 0.015% surfactant. The seedlings were phenotyped at least 10 days after flooding, as Pst19-infected seedlings died more slowly than PstDC3000-infected seedlings. Mock-inoculated seedlings were green, healthy, and actively growing. This control is important to ensure that the accessions are not sensitive to the concentration of surfactant, and to ensure there is no bacterial contamination. Susceptible accessions (Rio Grande-PtoR [n = 7], S. pimpinellifolium LA1375 [n = 7], and S. pimpinellifolium LA1606 [n = 5]) were dead, had brown apical meristems, and lacked new growth 10–14 days after inoculation with Pst19. In contrast, two S. neorickii LA1329 (n = 3) seedlings displayed a high level of new, green growth and survived infection with Pst19 (Figure 6). Three LA1329 seedlings did not germinate. Typically, 5–7 individuals were screened for each accession in a primary screen to determine the prevalence of resistance in the population. When a more genetically complex wild accession, such as LA1329, is flooded with Pst19, the resistance phenotypes display slightly more variability among individual seedlings, compared to Moneymaker-PtoR treated with PstDC3000. However, the resistance phenotypes were usually less variable than those seen in F2 mapping populations. Thus, binary phenotyping criteria was used for LA1329.
Figure 6 in the Representative Results section was updated from:
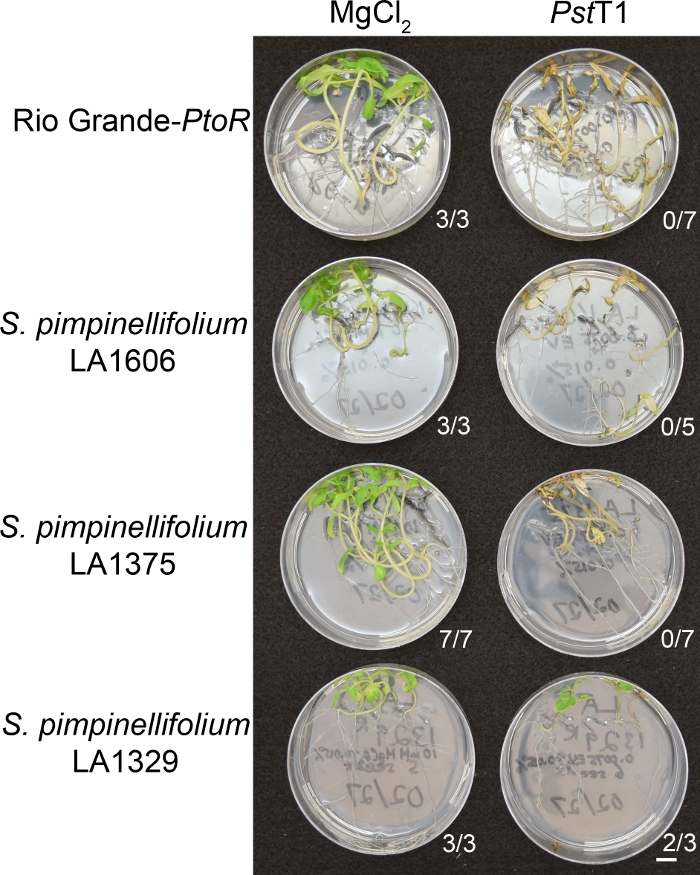
Figure 6: Phenotypic characterization of resistance or disease symptoms 10–14 days post-infection in wild accessions. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 and S. neorickii LA1329 tomato seedlings were grown on 0.5x MS plates for 10 days, and then flooded with PstT1 (OD600 = 0.0075) + 0.015% surfactant. The number of surviving seedlings for each wild accession out of the total number tested is shown. Scale bar = 1 cm. Please click here to view a larger version of this figure.
to:
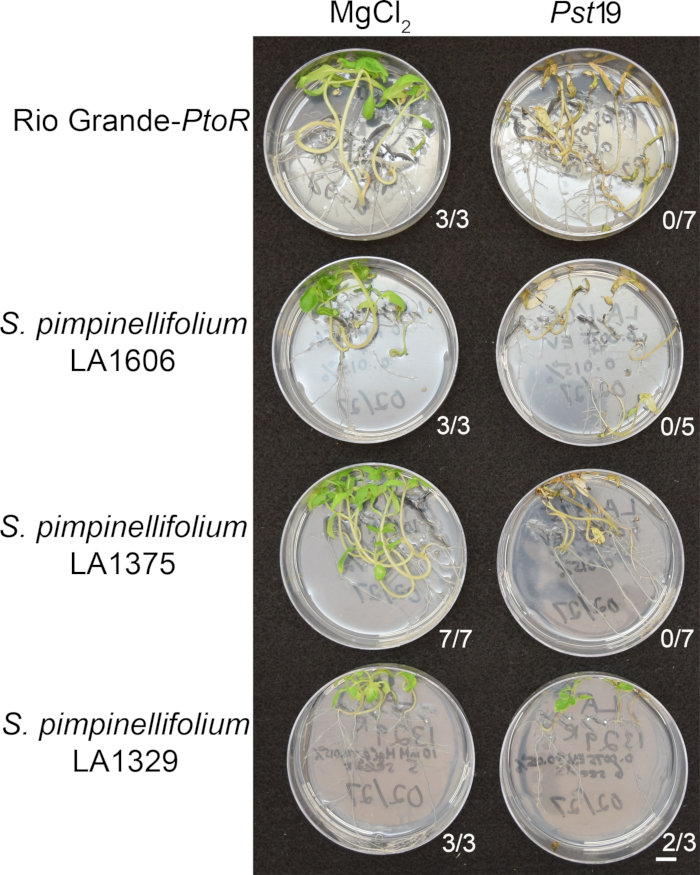
Figure 6: Phenotypic characterization of resistance or disease symptoms 10–14 days post-infection in wild accessions. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 and S. neorickii LA1329 tomato seedlings were grown on 0.5x MS plates for 10 days, and then flooded with Pst19 (OD600 = 0.0075) + 0.015% surfactant. The number of surviving seedlings for each wild accession out of the total number tested is shown. Scale bar = 1 cm. Please click here to view a larger version of this figure.
The third paragraph of the Representative Results section was updated from:
Quantitative assessment of bacterial growth using the seedling flood assay
To confirm that the observed resistance in LA1329 to PstT1 resulted in lower bacterial growth, bacterial growth assays were carried out in tomato seedlings. The level of PstT1 growth in Moneymaker-PtoS and S. neorickii LA1329 was determined 4 days post-infection. Moneymaker-PtoS is a near-isogenic line with consistent susceptibility among individual seedlings. Wild accessions such as S. neorickii LA1329 are often more genetically complex. LA1329 displays approximately 60% resistance to PstT1 across the population22. Because seedlings may drop their cotyledons after infection, one seedling was grown on each plate to correlate bacterial growth in the harvested cotyledon with overall seedling survival or death as determined phenotypically at least 10 days after flooding. The bacterial counts on day 4 for each seedling were normalized to 0.01 g of tissue and converted to log growth (CFU/0.01 g(log10)). Log growth for phenotypically resistant LA1329 seedlings (LA1329RES) or phenotypically susceptible seedlings (LA1329SUS) were separately pooled and compared to each other and the susceptible cultivar Moneymaker-PtoS. For example, there was a 1.7 log difference in bacterial growth between LA1329RES (log 6.3) and LA1329SUS (log 8.0), and a 1.6 log difference between LA1329RES (log 6.3) and Moneymaker-PtoS (log 7.9) (Figure 7). Therefore, phenotypic resistance correlated with quantitative resistance in the seedling assays.
to:
Quantitative assessment of bacterial growth using the seedling flood assay
To confirm that the observed resistance in LA1329 to Pst19 resulted in lower bacterial growth, bacterial growth assays were carried out in tomato seedlings. The level of Pst19 growth in Moneymaker-PtoS and S. neorickii LA1329 was determined 4 days post-infection. Moneymaker-PtoS is a near-isogenic line with consistent susceptibility among individual seedlings. Wild accessions such as S. neorickii LA1329 are often more genetically complex. LA1329 displays approximately 60% resistance to Pst19 across the population22. Because seedlings may drop their cotyledons after infection, one seedling was grown on each plate to correlate bacterial growth in the harvested cotyledon with overall seedling survival or death as determined phenotypically at least 10 days after flooding. The bacterial counts on day 4 for each seedling were normalized to 0.01 g of tissue and converted to log growth (CFU/0.01 g(log10)). Log growth for phenotypically resistant LA1329 seedlings (LA1329RES) or phenotypically susceptible seedlings (LA1329SUS) were separately pooled and compared to each other and the susceptible cultivar Moneymaker-PtoS. For example, there was a 1.7 log difference in bacterial growth between LA1329RES (log 6.3) and LA1329SUS (log 8.0), and a 1.6 log difference between LA1329RES (log 6.3) and Moneymaker-PtoS (log 7.9) (Figure 7). Therefore, phenotypic resistance correlated with quantitative resistance in the seedling assays.
Figure 7 in the Representative Results section was updated from:
x
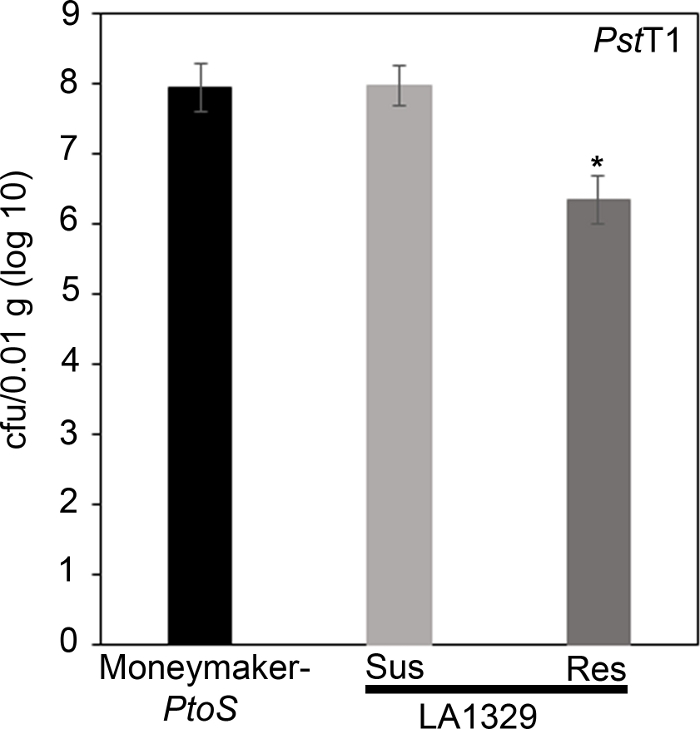
Figure 7: Resistant Solanum neorickii LA1329 seedlings support lower bacterial growth than Moneymaker-PtoS or susceptible S. neorickii LA1329. Bacterial counts were determined 4 days post-inoculation from S. neorickii LA1329 (n = 14) and Moneymaker-PtoS (n = 10) seedlings infected with PstT1 and normalization was performed to 0.01 g of tissue. For LA1329, the two phenotypic groups, susceptible (SUS) or resistant (RES), were observed and counted separately. Above the bar * = statistically significant difference determined by a one-factor analysis of variance. A general linear model procedure (p < 0.001) followed by a multiple comparison of means using Tukey's post hoc test was used. Error bars = standard error. The figure indicates one representative experiment. Please click here to view a larger version of this figure.
x
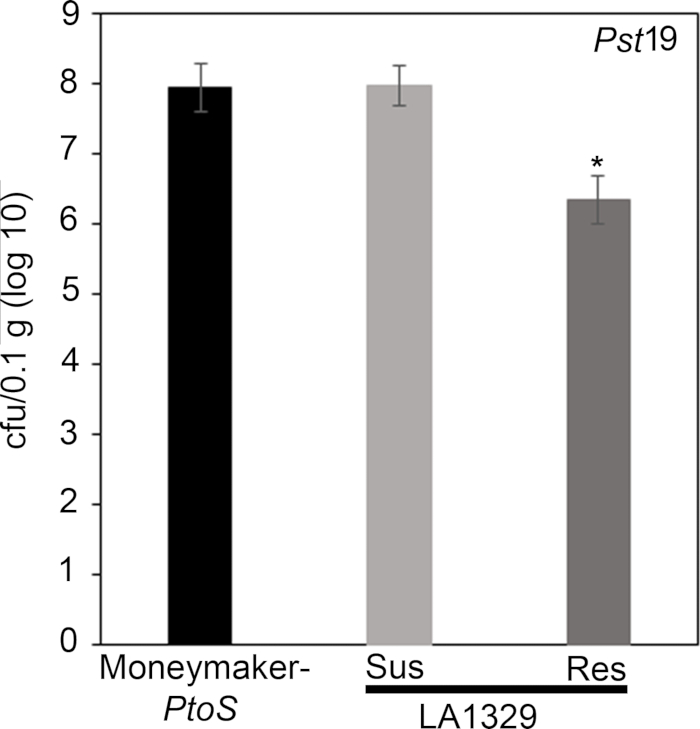
Figure 7: Resistant Solanum neorickii LA1329 seedlings support lower bacterial growth than Moneymaker-PtoS or susceptible S. neorickii LA1329. Bacterial counts were determined 4 days post-inoculation from S. neorickii LA1329 (n = 14) and Moneymaker-PtoS (n = 10) seedlings infected with Pst19 and normalization was performed to 0.1 g of tissue. For LA1329, the two phenotypic groups, susceptible (SUS) or resistant (RES), were observed and counted separately. Above the bar * = statistically significant difference determined by a one-factor analysis of variance. A general linear model procedure (p < 0.001) followed by a multiple comparison of means using Tukey's post hoc test was used. Error bars = standard error. The figure indicates one representative experiment. Please click here to view a larger version of this figure.
The first paragraph of the Discussion section was updated from:
A protocol for flood inoculation with PstDC3000 or PstT1 optimized to detect resistance to these bacterial strains in tomato seedlings is described. There are several critical parameters for optimal results in the seedling resistance assay, including bacterial concentration and surfactant concentration, which were empirically determined22. For PstDC3000, the optical density was optimized to achieve complete survival on a resistant cultivar containing the Pto/Prf cluster and complete death on a susceptible cultivar lacking the Pto/Prf cluster22. For a strain such as PstT1, where there are no known resistant varieties, the optical density was optimized to be the lowest possible for consistent and complete plant death22. Uppalapati et al.24 designed a tomato seedling assay to investigate the pathogenesis of PstDC3000 and the virulence function of coronatine. In this virulence assay, infections were performed using bacteria concentrated to an OD600 of 0.124, 20x higher than the optical density of strains used in our resistance assay. Recognition of PstDC3000 effectors AvrPto and AvrPtoB in tomato seedlings carrying the Pto/Prf gene cluster results in ETI and a macroscopic HR22. In the context of a strong immune response such as ETI, a lower bacterial titer was used for PstDC3000 to avoid overwhelming genetic resistance from the Pto/Prf gene cluster22. In addition, these results suggest that a high bacterial concentration could overwhelm weaker immune responses such as PTI or quantitative partial resistance, where multiple genes contribute to the overall phenotype. Surfactant is necessary for the bacteria to adhere to the leaf surface; however, high concentrations can cause chlorosis of the leaf22. We previously tested a range of surfactant concentrations to empirically determine the ideal concentration in 10-day-old tomato seedlings22. When testing new species that may differ in their sensitivity to surfactant, the surfactant concentration should be optimized to identify a concentration that does not cause damage or chlorosis in the absence of bacteria. Appropriate assay conditions will require optimization of a surfactant concentration that does not cause damage, and a bacterial concentration that causes disease in all susceptible controls.
to:
A protocol for flood inoculation with PstDC3000 or Pst19 optimized to detect resistance to these bacterial strains in tomato seedlings is described. There are several critical parameters for optimal results in the seedling resistance assay, including bacterial concentration and surfactant concentration, which were empirically determined22. For PstDC3000, the optical density was optimized to achieve complete survival on a resistant cultivar containing the Pto/Prf cluster and complete death on a susceptible cultivar lacking the Pto/Prf cluster22. For a strain such as Pst19, where there are no known resistant varieties, the optical density was optimized to be the lowest possible for consistent and complete plant death22. Uppalapati et al.24 designed a tomato seedling assay to investigate the pathogenesis of PstDC3000 and the virulence function of coronatine. In this virulence assay, infections were performed using bacteria concentrated to an OD600 of 0.124, 20x higher than the optical density of strains used in our resistance assay. Recognition of PstDC3000 effectors AvrPto and AvrPtoB in tomato seedlings carrying the Pto/Prf gene cluster results in ETI and a macroscopic HR22. In the context of a strong immune response such as ETI, a lower bacterial titer was used for PstDC3000 to avoid overwhelming genetic resistance from the Pto/Prf gene cluster22. In addition, these results suggest that a high bacterial concentration could overwhelm weaker immune responses such as PTI or quantitative partial resistance, where multiple genes contribute to the overall phenotype. Surfactant is necessary for the bacteria to adhere to the leaf surface; however, high concentrations can cause chlorosis of the leaf22. We previously tested a range of surfactant concentrations to empirically determine the ideal concentration in 10-day-old tomato seedlings22. When testing new species that may differ in their sensitivity to surfactant, the surfactant concentration should be optimized to identify a concentration that does not cause damage or chlorosis in the absence of bacteria. Appropriate assay conditions will require optimization of a surfactant concentration that does not cause damage, and a bacterial concentration that causes disease in all susceptible controls.
The third paragraph of the Discussion section was updated from:
Pst is a foliar pathogen that preferentially colonizes the aerial parts of tomato seedlings, including the cotyledons24 (Figure 3). Therefore, qualitative phenotyping in the seedling flood assay focuses on growth and disease symptoms in aerial portions of the seedling, and tissue for the bacterial growth assay is sampled from the cotyledons for quantitative analysis. After flood inoculation, seedlings may die within 7–10 days after inoculation with PstDC3000 or 10–14 days after inoculation with PstT1, as discussed in section 11. Seedling death is visualized by a brown apical meristem, arrested epicotyl elongation, and/or arrested vegetative growth. If different bacterial strains are used, the timing will have to be empirically determined. In addition, the progression of disease on control plants should be monitored daily after flooding until a consistent time frame from the onset of disease symptoms to seedling death can be identified. Depending on the genotypes and treatments used in the flood assay, seedling phenotypes can be recorded as binary phenotypes or on a disease spectrum (Figure 4). A broader spectrum of phenotypes may be observed when flood inoculating F2 mapping populations from wild tomato accessions crossed to susceptible cultivars (Figure 4C). It may be best to phenotype segregating populations on a disease spectrum depending on how quickly the seedling dies and the degree of new vegetative growth and branching (Figure 4C). The seedling flood assay can also be used in conjunction with the seedling bacterial growth assay to quantitatively assess levels of bacterial growth associated with qualitative phenotypes in individual seedlings (Figure 7). Very large reductions (i.e., ~log 3) in bacterial growth or strong resistance in resistant seedlings of a wild accession compared to a susceptible cultivar suggest that the underlying genetic basis of resistance may be due to ETI22. Smaller reductions in bacterial growth (i.e., ~log 1.7), as observed in LA1329 seedlings, may be due to the contribution of weaker resistance from quantitative trait loci and/or PTI. Thus, the seedling growth assay can be an important tool in further characterizing resistance in wild tomato lines.
to:
Pst is a foliar pathogen that preferentially colonizes the aerial parts of tomato seedlings, including the cotyledons24 (Figure 3). Therefore, qualitative phenotyping in the seedling flood assay focuses on growth and disease symptoms in aerial portions of the seedling, and tissue for the bacterial growth assay is sampled from the cotyledons for quantitative analysis. After flood inoculation, seedlings may die within 7–10 days after inoculation with PstDC3000 or 10–14 days after inoculation with Pst19, as discussed in section 11. Seedling death is visualized by a brown apical meristem, arrested epicotyl elongation, and/or arrested vegetative growth. If different bacterial strains are used, the timing will have to be empirically determined. In addition, the progression of disease on control plants should be monitored daily after flooding until a consistent time frame from the onset of disease symptoms to seedling death can be identified. Depending on the genotypes and treatments used in the flood assay, seedling phenotypes can be recorded as binary phenotypes or on a disease spectrum (Figure 4). A broader spectrum of phenotypes may be observed when flood inoculating F2 mapping populations from wild tomato accessions crossed to susceptible cultivars (Figure 4C). It may be best to phenotype segregating populations on a disease spectrum depending on how quickly the seedling dies and the degree of new vegetative growth and branching (Figure 4C). The seedling flood assay can also be used in conjunction with the seedling bacterial growth assay to quantitatively assess levels of bacterial growth associated with qualitative phenotypes in individual seedlings (Figure 7). Very large reductions (i.e., ~log 3) in bacterial growth or strong resistance in resistant seedlings of a wild accession compared to a susceptible cultivar suggest that the underlying genetic basis of resistance may be due to ETI22. Smaller reductions in bacterial growth (i.e., ~log 1.7), as observed in LA1329 seedlings, may be due to the contribution of weaker resistance from quantitative trait loci and/or PTI. Thus, the seedling growth assay can be an important tool in further characterizing resistance in wild tomato lines.
The fourth paragraph of the Discussion section was updated from:
Typically, genetic screens have been performed on four- to five-week-old adult tomato plants to identify the genetic basis of P. syringae resistance in wild accessions20,21. Adult tomato plants require much longer growth times, require more space in the growth chamber, and are much larger plants, which means that usually few individuals are screened for each line. The seedling flood assay provides a powerful, alternative approach in the identification of P. syringae resistance in wild tomato accessions. Screening at the seedling stage permits a large sample size to be tested which can be particularly advantageous in detecting resistance in genetically complex populations. Reduced growth chamber space requirements and growth time facilitate a high-throughput approach and rapid detection of natural resistance in wild accessions to emerging pathogens. Furthermore, P. syringae resistance that was identified at the seedling stage in this assay is not restricted to the developmental stage. S. neorickii LA1329 and S. habrochaites LA1253 were initially identified at the seedling stage and also display resistance to PstT1 in adult plants as previously described22.
to:
Typically, genetic screens have been performed on four- to five-week-old adult tomato plants to identify the genetic basis of P. syringae resistance in wild accessions20,21. Adult tomato plants require much longer growth times, require more space in the growth chamber, and are much larger plants, which means that usually few individuals are screened for each line. The seedling flood assay provides a powerful, alternative approach in the identification of P. syringae resistance in wild tomato accessions. Screening at the seedling stage permits a large sample size to be tested which can be particularly advantageous in detecting resistance in genetically complex populations. Reduced growth chamber space requirements and growth time facilitate a high-throughput approach and rapid detection of natural resistance in wild accessions to emerging pathogens. Furthermore, P. syringae resistance that was identified at the seedling stage in this assay is not restricted to the developmental stage. S. neorickii LA1329 and S. habrochaites LA1253 were initially identified at the seedling stage and also display resistance to Pst19 in adult plants as previously described22.



