ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Frøplanteflomanalysen letter rask screening av villtomattiltredelse for motstanden mot Pseudomonas syringae bakterien. Denne analysen, som brukes sammen med frøplante bakteriell vekst analyse, kan bidra til ytterligere karakterisere den underliggende motstanden mot bakterien, og kan brukes til å screene kartlegging populasjoner for å bestemme det genetiske grunnlaget for resistens.
Abstract
Tomat er en agronomically viktig avling som kan bli smittet av Pseudomonas syringae, en Gram-negativ bakterie, noe som resulterer i bakteriell flekksykdom. Tomat-P. syringae pv. tomat patosystem er mye brukt til å dissekere det genetiske grunnlaget for plantemedfødte responser og sykdomsresistens. Mens sykdommen ble vellykket forvaltet i mange tiår gjennom innføringen av Pto / Prf genklyngen fra Solanum pimpinellifolium til dyrket tomat, rase 1 stammer av P. syringae har utviklet seg til å overvinne motstand overdratt av Pto / Prf genklyngen og forekommer over hele verden.
Ville tomatarter er viktige reservoarer av naturlig mangfold i patogenanerkjennelse, fordi de utviklet seg i ulike miljøer med ulike patogentrykk. I typiske skjermer for sykdomsresistens i vill tomat brukes voksne planter, noe som kan begrense antall planter som kan screenes på grunn av utvidet veksttid og større vekstplasskrav. Vi utviklet en metode for å screene 10-dagers gamle tomatfrøplanter for motstand, noe som minimerer planteveksttid og vekstkammerplass, gjør det mulig å teste store utvalgsstørrelser. Frøplanteutfall av overlevelse eller død kan behandles som diskrete fenotyper eller på en motstandsskala definert av mengden ny vekst i overlevende frøplanter etter flom. Denne metoden er optimalisert for å screene 10-dagers gamle tomatfrøplanter for motstand mot to P. syringae stammer og kan lett tilpasses andre P. syringae stammer.
Introduction
Pseudomonas syringae er en Gram-negativ patogen bakterie som infiserer et bredt spekter av planteverter. Bakterier kommer inn i vertsanlegget gjennom stomata eller fysiske sår og sprer seg i apoplast1. Planter har utviklet en todelt immunrespons for å beskytte mot infeksjon av bakterielle patogener. Det første nivået oppstår på plantecelleoverflaten, hvor mønstergjenkjenningsreseptorer på plantecellemembranen oppfatter svært bevarte patogenetilhørende molekylære mønstre (PAMPs) i en prosess som kalles PAMP-utløst immunitet (PTI)2. I løpet av denne prosessen regulerer vertsanlegget forsvarsresponsveier, inkludert avsetning av callose til celleveggen, lukking av stomata, produksjon av reaktive oksygenarter og induksjon av patogeneserelaterte gener.
Bakterier kan overvinne PTI ved å bruke en type III sekresjonssystem for å levere proteiner, kalt effektorer, direkte inn i plantecellen3. Effector proteiner vanligvis målrette komponenter av PTI og fremme patogen virulence4. Den andre delen av planteimmunitet oppstår i plantecellen ved anerkjennelse av effektorproteinene. Denne anerkjennelsen er avhengig av resistensgener, som koder nukleotidbindende sted leucine-rik repetisjon som inneholder reseptorer (NLRs). NLRs er i stand til enten å gjenkjenne effektorer direkte eller gjenkjenne sin aktivitet på et virulensmål eller lokkedue5. De utløser deretter en sekundær immunrespons i en prosess som kalles effektorutløst immunitet (ETI), som ofte er forbundet med en overfølsom respons (HR), en form for lokalisert celledød på infeksjonsstedet6. I motsetning til gen-for-genresistens forbundet med ETI, kan planter vise kvantitativ delvis motstand, som er avhengig av bidrag fra flere gener7.
P. syringae pv. tomat (PST) er årsaksmiddelet av bakteriell flekk på tomat og er et vedvarende landbruksproblem. Dominerende stammer i feltet har vanligvis vært Pst rase 0 stammer som uttrykker enten eller begge av typen III effektorer AvrPto og AvrPtoB. DC3000 (PstDC3000) er en representativ rase 0 belastning og en modell patogen som kan forårsake bakteriell flekk i tomat. For å bekjempe bakteriell flekksykdom, gikk oppdrettere inn i Pto [P. syringae pv. tomat]/ Prf [Pto motstand og fenthion følsomhet] genklynge fra de ville tomatartene Solanum pimpinellifolium i moderne sorter8,9. Pto genet koder en serin-threoninprotein kinase som, sammen med Prf NLR, gir motstand mot PSTDC3000 via anerkjennelse av effektorene AvrPto og AvrPtoB10,11,12,13,14. Denne motstanden er imidlertid ineffektiv mot nye løp 1 stammer, slik at for deres raske og aggressive spredning de siste årene15,16. Race 1 stammer unngå anerkjennelse av Pto / Prf klyngen, fordi AvrPto er enten tapt eller mutert i disse stammene, og AvrPtoB ser ut til å samle minimalt15,17,18.
Villtomatpopulasjoner er viktige reservoarer av naturlig variasjon for PST-resistens og har tidligere blitt brukt til å identifisere potensiell motstand loci19,20,21. Dagens skjermer for patogenresistens benytter imidlertid 4–5 uker gamle voksne planter20,21. Derfor er de begrenset av veksttid, vekstkammerplass og relativt små utvalgsstørrelser. For å løse begrensningene av konvensjonelle tilnærminger, utviklet vi en høy gjennomstrømning tomat P. syringae motstand analyse ved hjelp av 10-dagers gamle tomat frøplanter22. Denne tilnærmingen gir flere fordeler ved bruk av voksne planter: nemlig kortere veksttid, reduserte romkrav og høyere gjennomstrømning. Videre har vi vist at denne tilnærmingen trofast rekapitulerer sykdomsresistens fenotyper observert i voksne planter22.
I frøplanteflomanalysen beskrevet i denne protokollen dyrkes tomatfrøplanter på Petri-retter av sterile Murashige og Skoog (MS) medier i 10 dager og blir deretter oversvømmet med et inokulum som inneholder bakterier av interesse og et overflateaktivt middel. Etter flom kan frøplanter evalueres kvantitativt for sykdomsresistens via bakteriell vekstanalyser. I tillegg kan frøplante overlevelse eller død fungere som en diskret resistens eller sykdom fenotype 7-14 dager etter flom. Denne tilnærmingen tilbyr et høygjennomstrømningsalternativ for screening av et stort antall villtomattiltredelseer for motstand mot PST-løp 1 stammer, for eksempel PST-stamme T1 (PstT1), og kan enkelt tilpasses andre bakterielle stammer av interesse.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Tilberedning og bruk av biosikkerhetsskap
- Tørk av biosikkerhetsskapet med 70% etanol.
- Lukk rammen og slå på det ultrafiolette lyset i biosikkerhetsskapet i 15 min.
- Etter 15 minutter, slå av det ultrafiolette lyset i biosikkerhetsskapet. Løft rammen og slå på blåseren i 15 min.
- Tørk av alle gjenstander som skal brukes i biosikkerhetsskapet med 70% etanol før du setter gjenstandene inn i det steriliserte kabinettet.
- Rengjør hansker eller bare hender med 70% etanol før du arbeider i biosikkerhetsskapet.
- Arbeid i midten av biosikkerhetsskapet, vekk fra blåseren.
- Bruk uåpnede flasker med autoklet steril 10 mM MgCl2 og ultrapure H2O for eksperimenter. Sett flasker i biosikkerhetsskapet og bare åpne dem i det steriliserte biosikkerhetsskapet, ikke på benkeplaten.
- Bruk dedikerte glasspipetter og pipettespisser for arbeid i det steriliserte biosikkerhetsskapet. Sørg for at disse kun åpnes i biosikkerhetsskapet, aldri på benkeplaten.
- Etter bruk av biosikkerhetsskapet, autoklavalt avfall (unntatt blekemiddelavfall) og tørk ned overflaten med 70% etanol.
2. Utarbeidelse av plantemedier
- Veie ut og oppløse 0,5x MS basal salter i ultrapure H2O. Veie ut 0,8% bacto agar og deretter legge til oppløst 0,5x MS.
- Autoklav og la mediet avkjøles i 50 °C vannbad i 1 t før helling eller pipettering.
- For å sikre at platene ikke er overfylt, markerpolystyren disponibel steril 100 x 25 mm plater til et fyllnivå på 40 ml. Hell media i 100 x 25 mm sterile plater i et sterilisert biosikkerhetsskap.
3. Utarbeidelse av plantematerialer og vekstforhold
- Legg tomatfrø i et 2,2 ml mikrocentrifugerør og tilsett 2,0 ml 50% blekemiddeloppløsning.
- Rock røret på en rocker i 25 min.
- Etter 25 minutter, fjern frøene fra rockeren og fjern blekemiddeloppløsningen med en pipette i det sterile biosikkerhetsskapet. Kontroller at alt blekemiddelet er fjernet.
- Tilsett 2 ml steril ultraren H2O for å vaske frøene. Vend røret 5x.
- Fjern væsken fra røret med en pipette.
- Gjenta trinn 3,3–3,5 for å vaske frøene 4x mer.
- Tilsett 2 ml steril ultraren H2O og hell frøene i en tom steril petriskål.
- Flammetang i etanol og la avkjøles før overføring og jevnt avstandfrø på 100 x 25 mm plater som inneholder 0,5x MS + 0,8% agar media.
- Overfør 5–7 frø i en linje over midten av en plate og forsegle kantene på platene med kirurgisk tape (1,25 cm x 9,1 m).
- Stratifiser de steriliserte frøene ved 4 °C i mørket i minst 3 dager for å synkronisere spiring. Sørg for at platene er stablet flatt og med forsiden opp, slik at frøene ikke skifter på platen.
- Vertikalt orientere platene slik at røttene vil vokse ned langs overflaten av platen, med linjen av frø orientert horisontalt, når du overfører til vekstkammeret.
MERK: Sett vekstkammeret til 22 °C og gi 16 timer lys med en lysintensitet på ~200–220 μE meter-2 s-1 og 8 timer mørke. - Før flom, vokse frøplanter i 10 dager i vekstkammeret på hvilket tidspunkt frøplanter vanligvis vises fullt dukket opp og utvidet cotyledons og nye første sanne blader (Figur 1).

Figur 1: Utviklingsstadium av typiske 10-dagers tomatfrøplanter. Rio Grande-PtoR tomatfrø ble sterilisert, belagt, og stratifisert i minst 3 dager i mørket ved 4 °C. Plantene ble dyrket på 0,5x MS plater i 10 dager ved 22 ° C før de ble oversvømmet. Vanligvis, på 10 dager cotyledons er fullt utvidet, og de første sanne bladene begynner å dukke opp. Vennligst klikk her for å se en større versjon av denne figuren.
4. Forberedelse av Kings B23 (KB)-medier
- Fyll begeret med 500 ml ultraren H2O og rør på en røreplate.
- Oppløs 20 g bacto peptone, 1,5 g vannfri K2HPO4og 12,5 ml glyserol i et beger med ultraren H2O.
- Hell den oppløste blandingen i en 1 L gradert sylinder og ta opp til en 1 L endelig volum med ultraren H2O.
- Hell buljongen tilbake i begeret og rør til blandet.
- Vei ut 7,5 g bacto agar i to 500 ml glassflasker og tilsett 500 ml KB buljong fra trinn 4.4 i hver flaske. Autoklav i 20 min.
- Fjern flaskene fra autoklaven og virvler forsiktig for å distribuere agaren.
- Overfør flaskene til et 50 °C vannbad i 1 t.
- Etter 1 time, overføre flasken til biosafety kabinettet og under aseptiske forhold, tilsett 1600 μL steril 1 M MgSO4, og passende antibiotika til media.
MERK: For rifampicinresistente stammer PSTDC3000 og PSTT1, bruk rifampicin oppløst i dimetylformamid ved en endelig konsentrasjon på 50 μg/ml. Bruk cykloheximid oppløst i etanol ved en endelig konsentrasjon på 50 μg/ml for å forhindre soppvekst på platene. - Virvle mediet forsiktig for å blande og hell deretter for å dekke bunnen av platene.
- La platene stivne minst 1 time til platene må stivne størks en opp ned ved 4 °C.
5. Vedlikehold av bakteriestammer og kulturforhold
- Opprettholde en glyserol lager fra enkelt koloni av bakterier som 1 ml mettet bakteriell kultur og 333 μL steril 80% glyserol ved -80 °C.
- Patch bakterier (dvs. PSTT1) fra en glyserol lager på KB agar med passende antibiotika (avsnitt 4).
- La bakteriene komme seg i 2 dager ved 28 °C før du streker friske bakterier på selektiv KB agar ved hjelp av en flat, steril tannpirker.
- Strekk ferske bakterier fra glyserolbestanden på passende selektiv KB agar ved hjelp av en flat, steril tannpirker.
MERK: Kontroller at den lappede glyserolbestanden ikke er mer enn 2 uker gammel. - For PSTDC3000, inkuber KB-platen ved 28 °C i 24 timer før du bruker bakterier i flomeksperimentet.
- For PSTT1, inkuber KB-platen ved 28 °C i 48 timer før du bruker bakterier i flomeksperimentet.
6. Utarbeidelse av PSTT1 inokulum
- Aseptisk resuspendere bakteriene i sterile 10 mM MgCl2 til en optisk tetthet ved 600 nm (OD600) på 0,1, eller ca 5 x 107 kolonidannende enheter (CFU)/ml).
- Utfør serielle fortynninger ved hjelp av steril oppløsning på 10 mM MgCl2 i biosikkerhetsskapet. For PSTT1, bruk et spektrotometer til å lage inoculum med en startkonsentrasjon på OD600 = 0,1.
- For PSTT1, lag en 1/10 fortynning fra den første resuspensjonen ved OD600 = 0,1 for å oppnå en seriell fortynning med en konsentrasjon på OD600 = 0,01.
- Ved hjelp av seriefortynning ved OD600 = 0,01 fra trinn 6.3, må du lage en 3/4 fortynning for å få en endelig OD600 = 0,0075.
- Lag en 1/10 fortynning av ikke-ionisk organosilikon overflateaktivt kopolymer C13H34O4Si3 (dvs. overflateaktivt middel) i 10 mM MgCl2 og vortex for 15 s. Legg til 1/10 lager av overflateaktivt middel til siste seriell fortynning (OD600 = 0,0075) til en endelig konsentrasjon på 0,015% og virvle godt å blande.
7. Utarbeidelse av PSTDC3000 inokulum
- Aseptisk resuspenderbakterier i steril 10 mM MgCl2 til en optisk tetthet ved 600 nm (OD600) på 0,1 (ca. 5 x 107 CFU/ml).
- Utfør serielle fortynninger ved hjelp av steril oppløsning på 10 mM MgCl2 i biosikkerhetsskapet. For PSTDC3000, bruk et spektrotometer til å lage innonometer med en startkonsentrasjon på OD600 = 0,1.
- For PSTDC3000, lag en 1/10 fortynning fra den første resuspensjonen ved OD600 = 0,1 for å få en seriell fortynning med en konsentrasjon på OD600 = 0,01.
- Ved hjelp av seriefortynning ved OD600 = 0,01 fra trinn 3, må du lage en 1/2 fortynning for å få en endelig OD600 = 0,005.
- Lag en 1/10 fortynning av overflateaktivt middel i 10 mM MgCl2 og vortex for 15 s. Legg 1/10 lager av overflateaktivt middel til siste seriell fortynning (OD600 = 0,005) til en endelig konsentrasjon på 0,015% og virvle godt å blande.
8. Tomat frøplante flom metode
- Ta platene med de 10-dagers gamle frøplanter ut av vekstkammeret og sett i biosikkerhetsskapet for å forberede platene for flom.
- Fjern kirurgisk tape fra to plater.
- Still inn en timer for 3 min. Mål 6 ml endelig inoculum (PSTT1 OD600 = 0,0075 [avsnitt 6] eller PSTDC3000 OD600 = 0,005 [avsnitt 7]) og overfør 6 ml inokulat til hver plate med de 10-dagers gamle frøplanter.
- Skyv plantene forsiktig ned i inokulumet med en steril pipettespiss. Start tidtakeren.
- Hold en tallerken i hver hånd. Vipp forsiden av platen ned for å samle inokulum og hovedsakelig senke cotyledons og blader av plantene.
- Swish side til side 5-7x og deretter spissen platene tilbake for å dekke røttene og hele platen.
- Vipp platene ned igjen for å senke cotyledons og blader, og gjenta i totalt 3 min.
- Hell inokulumet av platene, sett platene ned på en flat overflate og hell deretter av eventuelle gjenværende inokulering en gang til.
- Pakk platene med kirurgisk tape og gjenta trinn 8,2–8,8 for eventuelle gjenværende plater.
- Inkuber platene i vekstkammeret igjen (se trinn 3.11 OBS) etter at alle platene er oversvømt.
- Fenotype etter 7–10 dager for PSTDC3000 eller 10–14 dager for PSTT1 (avsnitt 11). Hvis du utfører bakteriell vekst analyser, samle bladvev etter 4 dager (avsnitt 9 og 10) og deretter fenotype (§ 11). Alternativt kan du utføre fenotypisk analyse og bakteriell vekstanalyser på separate sett med planter.
9. Overflatesterilisering av kotyledoner for bakteriell vekstanalyse
- Fire dager etter flom og re-inkubere plantene i vekstkammeret (seksjon 8), fjern platene med tomatfrøplanter fra vekstkammeret.
- Nummerer de enkelte frøplanter på bunnen av platen der frøplanten festes til platen for hver genotype.
- Merk sterile 1,5 ml mikrocentrifugerør med de enkelte frøplantenumrene og bruk rene tang til å slippe en 3 mm steril borosilikatperle i hvert rør for bruk med perlevisp. (Se MERKNAD i trinn 10.1.)
- Pipette 200 μL av 10 mM MgCl2 i hvert rør og lukk rørene.
- Forbered 70% etanol og hell 100 ml i et rent beger. Hell 100 ml steril ultraren H2O i et eget, rent beger.
- Rengjør rette finpunktpiner i rustfritt stål med taggete spisser med etanol. Åpne platen litt for å tillate aseptisk fjerning av en kotyledon med de rene tang.
- Klyp petiole ved foten av cotyledon for å fjerne et blad og slippe inn i begeret med 70% etanol for å overflatesterilisere i 10 s. Skyll cotyledon i ultraren H2O for 10 s.
- Plasser cotyledon på et papirhåndkle og blot tørr med delikate vitenskapsservietter.
- Individuelt veie hver cotyledon etter overflate sterilisering og blotting, og registrere vekten.
- Plasser cotyledon i et tidligere forberedt 1,5 ml mikrosentrifugerør (fra trinn 9,3 og 9,4) merket med tilsvarende genotype og individuelt nummer.
- Forsegle platene med steril tape og inkuber plantene i vekstkammeret igjen (se trinn 3.11 MERK).
10. Bakteriell vekstanalyse
- Ved hjelp av prøver fra trinn 9.10, homogenisere vevet ved hjelp av perlevispen i 10 mM MgCl2 i 1–2 min. Hvis vevet ikke er tilstrekkelig macerated, homogenisere igjen.
MERK: Mange produsenter produserer perlevisper homogenisatorer. Antall og type perler, samt homogeniseringstid og hastighet (hvis programmerbar) bør optimaliseres for hver type homogenisator. Sørg for at prøvene ikke overopphetes under homogenisering. - Tilsett 800 μL av 10 mM MgCl2 i hvert rør som inneholder macerated vev fra trinn 10.1 og inverter flere ganger for å blande.
- Forbered seriefortynninger for hver prøve i 10 mM MgCl2 i 96 brønnplate (100,10-1,10-2,10-3,10-4, 10-5) ved hjelp av en flerkanals pipette (figur 2A).
- Pipette 5 μL fra hver fortynningsserie med en flerkanals pipette på en KB agarplate (150 mm x 15 mm) med cykloheksamid og passende valg for bakteriell interessestamme (se trinn 4.8 MERK). La platene tørke helt.
- Inkuber platen opp ned ved 28 °C i 36 timer, og visualiserderetterkoloniene på platene ved hjelp av et dissekerende mikroskop for å avgjøre om koloniene er store nok til å telle.
MERK: Hvis koloniene ikke er store nok, inkuberplatene på nytt og kontroller størrelsen på koloniene med noen få timer. Vanligvis er koloniene countable ~ 36-48 timer etter inkubasjon.

Figur 2: Seriefortynninger for frøplantebakterievekstanalyser. (A) Macerated bladvev fra infiserte planter fortynnes før kolonitelling. Fortynninger utføres i en 96 brønnplate (100 er ufortynnet). Fortynninger er vanligvis laget av 10-1 til 10-5. (B) Plating fortynninger for bakteriellkoloniteller. Totalt 5 μL av hver kolonne i fortynningsserien er belagt, fra de fleste fortynnes til mest konsentrerte. Etter at koloniene er fullstendig tørket, inkuberes platen ved 28 °C i 36–48 timer. Kolonier telles under et 10x dissekerende mikroskop. Vennligst klikk her for å se en større versjon av denne figuren.
- Tell koloniene under et dissekerende mikroskop før de fusjonerer (figur 2B). Tell koloniene fra fortynningsserienplater med færre enn 100 kolonier.
- Etter å ha fått kolonitellinger (Figur 2B), normaliserer tellingene til 0,01 g vev for frøplanter og konverterer til logbakteriell vekst (Tabell 1).
MERK: Den gjennomsnittlige massen av en Moneymaker-PtoS cotyledon er 0,01 g og bestemmes empirisk22.
| Genotype1 Kolonne A | Vevvekt (g) kolonne B | # av kolonier på et sted Kolonne C | Fortynningsfaktor for punkt2 kolonne D | Justert # av kolonier3 kolonne E | Fortynningsfaktor for seriell fortynning kolonne F | Totalt # av kolonier Kolonne G (cfu/0.01 g)4 | Gjennomsnittlig # av kolonier (cfu/0.01 g) Kolonne H | Gjennomsnittlig loggvekst (cfu/0.01 g (logg10)) Kolonne I |
| Eksempel 1 | 0.004 g (0.004) | 10 | 200 | beregnet som: (C2 x 0,01 g) / B2 = 25 | 1000 | beregnet som: (D2 x E2 x F2) = 5000000 | gjennomsnitt for eksempel 1 til siste utvalg: (dvs. gjennomsnittlig G1:G3) = 7000000 | logg over gjennomsnittlig dvs. log(H2) = 6,85 |
| Eksempel 2 | 0.003 g (0.003) | 15 | 200 | 50 | 1000 | 10000000 | ||
| Eksempel 3 | 0.002 g (0.002) | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1.1.1 Data vist for 3 eksempler | ||||||||
| 2. Basert på plating 5 μL x 200 for 1 ml | ||||||||
| 3.3 Til og Cotyledons er for små til kjernen, slik at koloniantallet ble normalisert til 0,01 g vev basert på den gjennomsnittlige massen av en MoneyMaker-PtoS cotyledon (data ikke vist) | ||||||||
| 4. Justert per ml basert på volumbelagt | ||||||||
Tabell 1: Eksempelberegninger for frøplantebakterievekstanalyse. Eksempelberegninger viser hvordan man normaliserer bakterieantall og bestemmer log bakteriell vekst.
- For ville tiltredelseer og andre linjer med kompleks genetisk bakgrunn korrelerer nivået av bakteriell vekst i individuelle frøplanter med fenotypen som beskrevet i § 11.
11. Fenotyping for motstand
- Fjern platene fra vekstkammeret og fenotype individuelle frøplanter for død (på grunn av sykdom) eller overlevelse (på grunn av motstand) etter 7-14 dager.
- Fenotypeplanter smittet med en svært virulent stamme som PSTDC3000 tidligere, på 7-10 dager etter flom inokulasjon.
- Fenotypeplanter infisert med PSTT1 på 10–14 dager etter flominokulasjon.
- Bestem et poengsystem basert på omfanget av resistens fenotyper observert. Registrere binære fenotyper for sorter, isogene linjer og ville tiltredelseer med konsistente, sterke til mellomliggende resistens fenotyper (Figur 4A, 4B).
- Hvis frøplanten viser ny vekst fra den apikale meristem innen tidsrammen for fenotyping, telle det som en overlevelse. Hvis frøplanten har en brun apical meristem og viser ingen ny, grønn vegetativ vekst, telle det som en død (Figur 3).

Figur 3: Skjematisk representasjon av en tomatfrøplante. Ulike deler av en tomatfrøplante er avbildet, inkludert hypokotylen, kotyledoner, epicotyl, skyt apical meristem og sanne blader. Vennligst klikk her for å se en større versjon av denne figuren.
- Registrere fenotyper på et sykdomsspekter for populasjoner, for eksempel F2 kartleggingpopulasjoner, med et bredt spekter av resistens fenotyper (figur 4C).
- Nøye overvåke frøplanter for utseendet av sykdomssymptomer og død for å identifisere riktig vindu for fenotyping.

Figur 4: Skjematisk representasjon av forventede fenotyper for frømotresistens og død i ulike genetiske bakgrunner. (A) Frøplanter av Rio Grande-PtoR og den nesten isogene kultivarRio Grande-PtoS vises 7 dager etter flom med PSTDC3000 (OD600 = 0,005) + 0,015% overflateaktivt middel. Rio Grande-PtoR viser konsistent motstand, og Rio Grande-PtoS viser konsekvent mottakelighet for infeksjon med PSTDC3000. Disse linjene gir opphav til diskrete og binære fenotyper. (B) Frøplanter av en vill tiltredelse, for eksempel Solanum neorickii LA1329, vises 10 dager etter flom med PSTT1 (OD600 = 0,0075) + 0,015% overflateaktivt middel. Frøplanter viser fenotypisk variabilitet, men ble registrert som binære fenotyper. Mengden av fenotypisk variabilitet og metoden for fenotyping (binær motstand eller motstandspektrum) vil avhenge av den spesielle tiltredelse testet. (C) Kartlegging populasjoner generert ved utkryssende ville tiltredelseer til utsatte sorter kan vise et bredere spekter av fenotyper i F2 segregerende populasjoner. I dette tilfellet kan det være mest hensiktsmessig å registrere frøfenotyper på et spektrum. Svært utsatte frøplanter fra en kartlegging stredelser kan være fenoskrevet for død så tidlig som dag 7 når oversvømmet med PstT1, og vanligvis viser en brun apical meristem, nei til svært liten forlengelse av epicotyl, og ingen ny, grønn vegetativ vekst. Den apical meristem av mottakelige frøplanter kan forbli grønn eller svært lys brun for mer tid, og det kan være noen forlengelse av epicotyl og svært lite vegetativ vekst, som blir brun og arrestasjoner av dag 10. Individuelle frøplanter kan fenoskrevet for motstand basert på mengden ny og pågående vegetativ vekst etter dag 14. Frøplanter kan deretter grupperes basert på fenotypene som er beskrevet ovenfor i forskjellige kategorier av resistens som svak, middels eller sterk motstand. Vennligst klikk her for å se en større versjon av denne figuren.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Påvisning av PtoR-mediertimmunitet i sorter og isogene linjer ved hjelp av frømotstandsanalysen
Figur 5 viser representative resultater for Moneymaker-PtoR og Moneymaker-PtoS kultivarer 7-10 dager etter flom med PSTDC3000. Før infeksjon, 10-dagers gamle frøplanter vises fullt dukket opp og utvidet cotyledons og nye første sanne blader. Plantene ble oversvømmet med 10 mM MgCl2 + 0,015% overflateaktivt middel som en negativ kontroll (data ikke vist) og PSTDC3000 med en optisk tetthet på 0,005 + 0,015% overflateaktivt middel. Plantene ble fenoskrevet 7-10 dager etter flom (Figur 5). Individuelle frøplanter fra genotypisk homogene linjer, som Moneymaker-PtoR og Moneymaker-PtoS gir svært konsekvente og binære fenotyper i frøflomanalysen. Når Moneymaker-PtoR, som bærer Pto / Prf genklyngen (n = 5), ble behandlet med PstDC3000 ved optimal konsentrasjon av OD600 = 0,005, var motstanden på grunn av PtoR-mediertimmunitet sterk og ble preget av ny, grønn vegetativ vekst hos alle individer22. Nær-isogenic Moneymaker-PtoS frøplanter (n = 5), som ikke kan gjenkjenne PSTDC3000 effektorer AvrPto eller AvrPtoB, døde raskt innen 7 dager etter flom og karakteristisk hadde brune apical meristems, bakteriell flekk, klorose, og ingen tegn på ny, grønn vegetativ vekst (Figur 5).

Figur 5: Fenotypisk karakterisering av resistens- eller sykdomssymptomer 7–10 dager etter infeksjon i en sort. Moneymaker-PtoR og Moneymaker-PtoS tomatfrøplanter ble dyrket på 0,5 x MS-plater i 10 dager før de ble oversvømmet med P. syringae pv. Tomat DC3000 (OD600 = 0,005) + 0,015 % overflateaktivt middel. Moneymaker-PtoR frøplanter overlevde (n = 5) og Moneymaker-PtoS frøplanter (n = 5) døde. Antall overlevende frøplanter for hver genotype ut av det totale antallet som er testet, vises. Skala bar = 1 cm. Vennligst klikk her for å vise en større versjon av denne figuren.
Phenotypic screening av ville tiltredelseer ved hjelp av frøplantemotstandsanalysen
Figur 6 viser representative resultater for frøplanter av mottakelige og resistente tiltredelser 10–14 dager etter flom med PSTT1. Mottakelige tiltredelseer inkluderer RG-PtoR, S. pimpinellifolium LA1375 og S. pimpinellifolium LA1606, og resistente tiltredelseer inkluderer S. neorickii LA1329. Ti-dagers gamle frøplanter ble oversvømmet med 10 mM MgCl2 + 0,015% overflateaktivt middel som en negativ kontroll, og PSTT1 med en optisk tetthet på 0,0075 + 0,015% overflateaktivt middel. Plantene ble fenoskrevet minst 10 dager etter flom, som PSTT1-infiserte frøplanter døde saktere enn PSTDC3000-infiserte frøplanter. Mock-inokulerte frøplanter var grønne, sunne og aktivt voksende. Denne kontrollen er viktig for å sikre at tiltredelsesegenskapene ikke er følsomme for konsentrasjonen av overflateaktivt middel, og for å sikre at det ikke er bakteriell forurensning. Mottakelige tiltredelseer (Rio Grande-PtoR [n = 7], S. pimpinellifolium LA1375 [n = 7], og S. pimpinellifolium LA1606 [n = 5]) var døde, hadde brune apical meristems, og manglet ny vekst 10-14 dager etter inokulasjon med PSTT1. I motsetning, to S. neorickii LA1329 (n = 3) frøplanter viste et høyt nivå av ny, grønn vekst og overlevde infeksjon med PSTT1 (Figur 6). Tre LA1329 frøplanter spiret ikke. Vanligvis ble 5–7 personer screenet for hver tiltredelse i en primærskjerm for å bestemme utbredelsen av resistens i befolkningen. Når en mer genetisk kompleks vill tiltredelse, som LA1329, er oversvømmet med PstT1, motstand fenotyper viser litt mer variasjon blant individuelle frøplanter, sammenlignet med Moneymaker-PtoR behandlet med PSTDC3000. Resistensfenotypene var imidlertid vanligvis mindre variable enn de som ble sett i F2-kartpopulasjoner. Dermed ble binære fenotypingkriterier brukt for LA1329.

Figur 6: Fenotypisk karakterisering av resistens- eller sykdomssymptomer 10–14 dager etter infeksjon i ville tiltredelseer. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 og S. neorickii LA1329 tomat frøplanter ble dyrket på 0,5 x MS plater i 10 dager, og deretter oversvømmet med PstT1 (OD600 = 0,0075) + 0,015% overflateaktivtmiddel. Antall overlevende frøplanter for hver vill tiltredelse ut av det totale antallet testet vises. Skala bar = 1 cm. Vennligst klikk her for å vise en større versjon av denne figuren.
Kvantitativ vurdering av bakteriell vekst ved hjelp av frøflomanalysen
For å bekrefte at den observerte motstanden i LA1329 til PstT1 resulterte i lavere bakteriell vekst, ble bakteriell vekstanalyser utført i tomatfrøplanter. Nivået på PSTT1 vekst i Moneymaker-PtoS og S. neorickii LA1329 ble bestemt 4 dager etter infeksjon. Moneymaker-PtoS er en nesten isogene linje med konsekvent mottakelighet blant individuelle frøplanter. Ville tiltredelseer som S. neorickii LA1329 er ofte mer genetisk komplekse. LA1329 viser ca 60% motstand mot PstT1 over befolkningen22. Fordi frøplanter kan slippe sine kotyledoner etter infeksjon, ble en frøplante dyrket på hver tallerken for å korrelere bakteriell vekst i høstet kotyledon med total frøplante overlevelse eller død som bestemt fenotypisk minst 10 dager etter flom. Bakterietellingen på dag 4 for hver frøplante ble normalisert til 0,01 g vev og omdannet til loggvekst (CFU/0,01 g(logg10)). Loggvekst for fenotypisk resistente LA1329 frøplanter (LA1329RES) eller fenotypisk utsatte frøplanter (LA1329SUS)ble separat samlet og sammenlignet med hverandre og den følsomme cultivar Moneymaker-PtoS. For eksempel var det en 1,7 loggforskjell i bakteriell vekst mellom LA1329RES (log 6.3) og LA1329SUS (log 8.0), og en 1,6 loggforskjell mellom LA1329RES (log 6.3) og Moneymaker-PtoS (log 7.9) (Figur 7). Derfor korrelerte fenotypisk motstand med kvantitativ motstand i frøplanteassier.

Figur 7: Resistente Solanum neorickii LA1329 frøplanter støtter lavere bakteriell vekst enn Moneymaker-PtoS eller utsatt S. neorickii LA1329. Bakterieantall ble bestemt 4 dager etter inokulasjon fra S. neorickii LA1329 (n = 14) og Moneymaker-PtoS (n = 10) frøplanter infisert med PSTT1 og normalisering ble utført til 0,01 g vev. For LA1329 ble de to fenotyske gruppene, utsatte (SUS) eller resistente (RES), observert og talt separat. Over baren * = statistisk signifikant forskjell bestemt av en enfaktoranalyse av varians. En generell lineær modellprosedyre (p < 0,001) etterfulgt av en flere sammenligning av midler ved hjelp av Tukeys post hoc-test ble brukt. Feilfelt = standardfeil. Figuren indikerer ett representativt eksperiment. Vennligst klikk her for å se en større versjon av denne figuren.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
En protokoll for flom inokulasjon med PSTDC3000 eller PstT1 optimalisert for å oppdage motstand mot disse bakterielle stammer i tomat frøplanter er beskrevet. Det er flere kritiske parametere for optimale resultater i frøplanteresistensanalysen, inkludert bakteriell konsentrasjon og overflateaktivt konsentrasjon, som empirisk ble bestemt22. For PSTDC3000 ble den optiske tettheten optimalisert for å oppnå fullstendig overlevelse på en motstandsdyktig sort som inneholder Pto/Prf-klyngen og fullstendig død på en utsatt kultivar som mangler Pto/Prf-klyngen 22. For en stamme som PstT1, hvor det ikke er noen kjente resistente varianter, ble den optiske tettheten optimalisert for å være lavest mulig for konsekvent og fullstendig plantedød22. Uppalapati et al.24 designet en tomat frøplante analyse for å undersøke patogenesen av PSTDC3000 og virulence funksjon av koronontin. I denne virulensanalysen ble infeksjoner utført ved hjelp av bakterier konsentrert til en OD600 på 0,124, 20x høyere enn den optiske tettheten av stammer som brukes i vår resistensanalyse. Anerkjennelse av PSTDC3000 effektorer AvrPto og AvrPtoB i tomatfrøplanter som bærer Pto / Prf genklyngen resulterer i ETI og en makroskopisk HR22. I sammenheng med en sterk immunrespons som ETI, ble en lavere bakteriell titer brukt for PSTDC3000 for å unngå overveldende genetisk motstand fra Pto / Prf genklyngen22. I tillegg tyder disse resultatene på at en høy bakteriell konsentrasjon kan overvelde svakere immunresponser som PTI eller kvantitativ delvis motstand, hvor flere gener bidrar til den totale fenotypen. Overflateaktivt middel er nødvendig for at bakteriene skal holde seg til bladoverflaten; Imidlertid kan høye konsentrasjoner forårsake klorose i bladet22. Vi har tidligere testet en rekke overflateaktive konsentrasjoner for å empirisk bestemme den ideelle konsentrasjonen i 10-dagers gamle tomatfrøplanter22. Ved testing av nye arter som kan variere i deres følsomhet overfor overflateaktivt middel, bør overflateaktivt konsentrasjon optimaliseres for å identifisere en konsentrasjon som ikke forårsaker skade eller klorose i fravær av bakterier. Passende analyseforhold vil kreve optimalisering av en overflateaktivt konsentrasjon som ikke forårsaker skade, og en bakteriell konsentrasjon som forårsaker sykdom i alle utsatte kontroller.
Ytterligere kritiske parametere for suksess i frøflomanalysen inkluderer bruk av frøplanter i spesifikke utviklingsstadier (10-dagers gamle frøplanter) (Figur 1), opprettholde stabil vekstkammerforhold (lysintensitet på ca 200 μE m-2 s-1, konstant temperatur på 22 °C, 16 timer lys) og utføre eksperimenter i et sterilt biosikkerhetsskap. Medievolum over 45 ml eller under 35 ml kan påvirke konsistent død av mottakelige kontroller, fordi volumet kan påvirke det omkringliggende mikromiljøet til plantene på platen. For eksempel kan forskjeller i relativ fuktighet inne i de forseglede platene påvirke infeksiviteten til bakteriene og plantenes evne til å overleve infeksjon. Steril teknikk er kritisk, fordi forurensning på platene kan forvirre kilden til død eller mottakelighet i frøplanter. I tillegg, fordi plantepatogen interaksjoner påvirkes av circadian klokke24,25,26, anbefales det at plantene bli smittet på en konsekvent tid på dagen.
PST er et foliar patogen som fortrinnsvis koloniserer antennedelene av tomatfrøplanter, inkludert kotyledonene24 (Figur 3). Derfor fokuserer kvalitativ fenotyping i frøflomanalysen på vekst- og sykdomssymptomer i antennedeler av frøplanten, og vev for bakteriell vekstanalyse er samplet fra kotyledoner for kvantitativ analyse. Etter flominokulasjon kan frøplanter dø innen 7–10 dager etter inokulasjon med PSTDC3000 eller 10–14 dager etter inokulasjon med PSTT1, som omtalt i avsnitt 11. Frøplante død er visualisert av en brun apical meristem, arrestert epicotyl forlengelse, og / eller arrestert vegetativ vekst. Hvis ulike bakterielle stammer brukes, må timingen bestemmes empirisk. I tillegg bør sykdomsprogresjonen på kontrollplanter overvåkes daglig etter flom til en konsekvent tidsramme fra sykdomssymptomer til frøplantedød kan identifiseres. Avhengig av genotyper og behandlinger som brukes i flomanalysen, kan frøplantefenotyper registreres som binære fenotyper eller på et sykdomsspekter (Figur 4). Et bredere spekter av fenotyper kan observeres når flom inokulerer F2 kartlegging populasjoner fra vill tomat tiltredelse krysset til utsatte sorter (Figur 4C). Det kan være best å fenotype segregerende populasjoner på et sykdomsspekter avhengig av hvor raskt frøplanten dør og graden av ny vegetativ vekst og forgrening (Figur 4C). Frøplanteflomanalysen kan også brukes sammen med siverende bakteriell vekstanalyse for å kvantitativt vurdere nivåer av bakteriell vekst forbundet med kvalitative fenotyper i individuelle frøplanter (figur 7). Svært store reduksjoner (dvs. ~ log 3) i bakteriell vekst eller sterk motstand i resistente frøplanter av en vill tiltredelse sammenlignet med en utsatt sorter tyder på at det underliggende genetiske grunnlaget for resistens kan skyldes ETI22. Mindre reduksjoner i bakteriell vekst (dvs. ~log 1.7), som observert i LA1329 frøplanter, kan skyldes bidrag et svakere motstand fra kvantitativ ei trafiloci og/eller PTI. Dermed kan frøplantevekstanalysen være et viktig verktøy for ytterligere karakterisering av motstand i ville tomatlinjer.
Vanligvis har genetiske skjermer blitt utført på fire- til fem uker gamle voksne tomatplanter for å identifisere det genetiske grunnlaget for P. syringae motstand i ville tiltredelseer20,21. Voksne tomatplanter krever mye lengre veksttider, krever mer plass i vekstkammeret, og er mye større planter, noe som betyr at vanligvis få individer blir screenet for hver linje. Frøplanteflomanalysen gir en kraftig, alternativ tilnærming i identifiseringen av P. syringae motstand i vill tomat tiltredelse. Screening på frøplantestadiet tillater en stor prøvestørrelse som kan testes som kan være spesielt fordelaktig i å oppdage motstand i genetisk komplekse populasjoner. Reduserte vekstkammerromkrav og veksttid letter en høy gjennomstrømningstilnærming og rask påvisning av naturlig motstand i ville tiltredelseer til nye patogener. Videre er P. syringae motstand som ble identifisert på frøplantestadiet i denne analysen ikke begrenset til utviklingsstadiet. S. neorickii LA1329 og S. habrochaites LA1253 ble opprinnelig identifisert på frøplantestadiet og viser også motstand mot PstT1 i voksne planter som tidligere beskrevet22.
Frøplanteflomanalysen er en allsidig protokoll som kan endres og optimaliseres for å oppdage vertsmotstand mot andre P. syringae stammer. Det kan potensielt brukes videre i sammenheng med ulike bakterielle patogener av tomat, som Xanthomonas arter. Denne metoden vil fremskynde søket etter nye kilder til sykdomsresistens mot bakterielle patogener.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Forfatterne har ingenting å avsløre.
Acknowledgments
Vi takker Jamie Calma for å teste effekten av medievolum på sykdom eller resistensutfall. Vi takker Dr. Maël Baudin og Dr. Karl J. Scheiber fra Lewis Lab for å gi konstruktive kommentarer og forslag til manuskriptet. Forskning på planteimmunitet i Lewis-laboratoriet ble støttet av USDA ARS 2030-21000-046-00D og 2030-21000-050-00D (JDL), og NSF Direktoratet for biologiske vitenskaper IOS-1557661 (JDL).
Materials
| Name | Company | Catalog Number | Comments |
| 3M Tape Micropore 1/2" x 10 YD CS 240 (1.25 cm x 9.1 m) | VWR International | 56222-182 | |
| 3mm borosilicate glass beads | Friedrich & Dimmock | GB3000B | |
| Bacto peptone | BD | 211677 | |
| Bacto agar | BD | 214010 | |
| Biophotometer Plus | Eppendorf | E952000006 | |
| Biosafety cabinet, class II type A2 | |||
| BRAND Disposable Plastic Cuvettes, Polystyrene | VWR International | 47744-642 | |
| Chenille Kraft Flat Wood Toothpicks | VWR International | 500029-808 | |
| cycloheximide | Research Products International | C81040-5.0 | |
| Dibasic potassium phosphate anhydrous, ACS grade | Fisher Scientific | P288-500 | |
| Dimethylformamide | |||
| Dissecting microscope (Magnification of at least 10x) | |||
| Ethanol - 190 Proof | |||
| Falcon polystyrene 96 well microplates, flat-bottom | Fisher Scientific | 08-772-3 | |
| Glass Alcohol Burner Wick | Fisher Scientific | S41898A / No. W-125 | |
| Glass Alcohol Burners | Fisher Scientific | S41898 / No. BO125 | |
| Glycerol ACS reagent | VWR International | EMGX0185-5 | |
| Kimberly-Clark™ Kimtech Science™ Kimwipes™ Delicate Task Wipers | Fisher Scientific | 06-666-A | |
| Magnesium chloride, ACS grade | VWR International | 97061-356 | |
| Magnesium sulfate heptahydrate, ACS grade | VWR International | 97062-130 | |
| Microcentrifuge tubes, 1.5 mL | |||
| Microcentrifuge tubes, 2.2 mL | |||
| Mini Beadbeater-96, 115 volt | Bio Spec Products Inc. | 1001 | |
| Murashige & Skoog, Basal Salts | Caisson Laboratories, Inc. | MSP01-50LT | |
| Pipet-Lite XLS LTS 8-CH Pipet 20-200uL | Rainin | L8-200XLS | |
| Pipet-Lite XLS LTS 8-CH Pipet 2-20uL | Rainin | L8-20XLS | |
| Polystyrene 100mm x 25mm sterile petri dish | VWR International | 89107-632 | |
| Polystyrene 150mm x 15mm sterile petri dish | Fisher Scientific | FB08-757-14 | |
| Polystyrene 150x15mm sterile petri dish | Fisher Scientific | 08-757-148 | |
| Pure Bright Germicidal Ultra Bleach 5.7% Available Chlorine (defined as 100% bleach) | Staples | 1013131 | |
| Rifampicin | Gold Biotechnology | R-120-25 | |
| Silwet L-77 (non-ionic organosilicone surfactant co-polymer C13H34O4Si3 surfactant) | Fisher Scientific | NCO138454 | |
| Tips LTS 20 μL 960/10 GPS-L10 | Rainin | 17005091 | |
| Tips LTS 250 μL 960/10 GPS-L250 | Rainin | 17005093 | |
| VWR dissecting forceps fine tip, 4.5" | VWR International | 82027-386 |
References
- Underwood, W., Melotto, M., He, S. Y. Role of plant stomata in bacterial invasion. Cell Microbiology. 9 (7), 1621-1629 (2007).
- Zipfel, C. Early molecular events in PAMP-triggered immunity. Current Opinion in Plant Biology. 12 (4), 414-420 (2009).
- Galan, J. E., Wolf-Watz, H. Protein delivery into eukaryotic cells by type III secretion machines. Nature. 444 (7119), 567-573 (2006).
- Lewis, J. D., Desveaux, D., Guttman, D. S. The targeting of plant cellular systems by injected type III effector proteins. Seminars in Cell and Developmental Biology. 20 (9), 1055-1063 (2009).
- Schreiber, K. J., Baudin, M., Hassan, J. A., Lewis, J. D. Die another day: molecular mechanisms of effector-triggered immunity elicited by type III secreted effector proteins. Seminars in Cell and Developmental Biology. 56, 124-133 (2016).
- Heath, M. C. Hypersensitive response-related death. Plant Molecular Biology. 44 (3), 321-334 (2000).
- Boyd, L. A., Ridout, C., O'Sullivan, D. M., Leach, J. E., Leung, H. Plant-pathogen interactions: disease resistance in modern agriculture. Trends in Genetics. 29 (4), 233-240 (2013).
- Pitblado, R. E., MacNeill, B. H. Genetic basis of resistance to Pseudomonas syringae pv. tomato in field tomatoes. Canadian Journal of Plant Pathology. 5 (4), 251-255 (1983).
- Pedley, K. F., Martin, G. B. Molecular basis of Pto-mediated resistance to bacterial speck disease in tomato. Annual Reviews of Phytopathology. 41, 215-243 (2003).
- Ronald, P. C., Salmeron, J. M., Carland, F. M., Mehta, A. Y., Staskawicz, B. J. The cloned avirulence gene AvrPto induces disease resistance in tomato cultivars containing the Pto resistance gene. Journal of Bacteriology. 174 (5), 1604-1611 (1992).
- Martin, G. B., et al. Map-based cloning of a protein kinase gene conferring disease resistance in tomato. Science. 262 (5138), 1432-1436 (1993).
- Salmeron, J. M., Barker, S. J., Carland, F. M., Mehta, A. Y., Staskawicz, B. J. Tomato mutants altered in bacterial disease resistance provide evidence for a new locus controlling pathogen recognition. Plant Cell. 6 (4), 511-520 (1994).
- Salmeron, J. M., et al. Tomato Prf is a member of the leucine-rich repeat class of plant disease resistance genes and lies embedded within the Pto kinase gene cluster. Cell. 86 (1), 123-133 (1996).
- Scofield, S. R., et al. Molecular basis of gene-for-gene specificity in bacterial speck disease of tomato. Science. 274 (5295), 2063-2065 (1996).
- Kunkeaw, S., Tan, S., Coaker, G. Molecular and evolutionary analyses of Pseudomonas syringae pv. tomato race 1. Molecular Plant-Microbe Interactions. 23 (4), 415-424 (2010).
- Cai, R., et al. The plant pathogen Pseudomonas syringae pv. tomato is genetically monomorphic and under strong selection to evade tomato immunity. PLoS Pathogens. 7 (8), 1002130 (2011).
- Almeida, N. F., et al. A draft genome sequence of Pseudomonas syringae pv. tomato T1 reveals a type III effector repertoire significantly divergent from that of Pseudomonas syringae pv. tomato DC3000. Molecular Plant-Microbe Interactions. 22 (1), 52-62 (2009).
- Lin, N. C., Abramovitch, R. B., Kim, Y. J., Martin, G. B. Diverse AvrPtoB homologs from several Pseudomonas syringae pathovars elicit Pto-dependent resistance and have similar virulence activities. Applied and Environmental Microbiology. 72 (1), 702-712 (2006).
- Rose, L. E., Langley, C. H., Bernal, A. J., Michelmore, R. W. Natural variation in the Pto pathogen resistance gene within species of wild tomato (Lycopersicon). I. Functional analysis of Pto alleles. Genetics. 171 (1), 345-357 (2005).
- Thapa, S. P., Miyao, E. M., Davis, R. M., Coaker, G. Identification of QTLs controlling resistance to Pseudomonas syringae pv. tomato race 1 strains from the wild tomato Solanum habrochaites LA1777. Theoretical and Applied Genetics. 128 (4), 681-692 (2015).
- Bao, Z. L., et al. Identification of a candidate gene in Solanum habrochaites for resistance to a race 1 strain of Pseudomonas syringae pv. tomato. Plant Genome. 8 (3), 1-15 (2015).
- Hassan, J. A., Zhou, Y. J., Lewis, J. D. A rapid seedling resistance assay identifies wild tomato lines that are resistant to Pseudomonas syringae pv. tomato race 1. Molecular Plant-Microbe Interactions. 30 (9), 701-709 (2017).
- King, E. O., Ward, M. K., Raney, D. E. Two simple media for the demonstration of pyocyanin and fluorescin. Journal of Laboratory and Clinical Medicine. 44 (2), 301-307 (1954).
- Uppalapati, S. R., et al. Pathogenicity of Pseudomonas syringae pv. tomato on tomato seedlings: phenotypic and gene expression analyses of the virulence function of coronatine. Molecular Plant-Microbe Interactions. 21 (4), 383-395 (2008).
- Bhardwaj, V., Meier, S., Petersen, L. N., Ingle, R. A., Roden, L. C. Defence responses of Arabidopsis thaliana to infection by Pseudomonas syringae are regulated by the circadian clock. PLoS One. 6 (10), 26968 (2011).
- Lu, H., McClung, C. R., Zhang, C. Tick tock: circadian regulation of plant innate immunity. Annual Review of Phytopathology. 55, 287-311 (2017).
- Wang, W., et al. Timing of plant immune responses by a central circadian regulator. Nature. 470 (7332), 110-114 (2011).
Tags
Bioengineering Utgave 157 Pseudomonas syringae tomat sykdomsresistens skjerm bakteriell vekst fenotyperErratum
Formal Correction: Erratum: High-Throughput Identification of Resistance to Pseudomonas syringae pv. Tomato in Tomato using Seedling Flood Assay
Posted by JoVE Editors on 10/18/2023.
Citeable Link.
An erratum was issued for: High-Throughput Identification of Resistance to Pseudomonas syringae pv. Tomato in Tomato using Seedling Flood Assay. The Introduction, Protocol, Representative Results and Discussion sections were updated.
The last paragraph of the Introduction section was updated from:
In the seedling flood assay described in this protocol, tomato seedlings are grown on Petri dishes of sterile Murashige and Skoog (MS) media for 10 days and then are flooded with an inoculum containing the bacteria of interest and a surfactant. Following flooding, seedlings can be quantitatively evaluated for disease resistance via bacterial growth assays. Additionally, seedling survival or death can act as a discrete resistance or disease phenotype 7–14 days after flooding. This approach offers a high-throughput alternative for screening large numbers of wild tomato accessions for resistance to Pst race 1 strains, such as Pst strain T1 (PstT1), and can easily be adapted to other bacterial strains of interest.
to:
In the seedling flood assay described in this protocol, tomato seedlings are grown on Petri dishes of sterile Murashige and Skoog (MS) media for 10 days and then are flooded with an inoculum containing the bacteria of interest and a surfactant. Following flooding, seedlings can be quantitatively evaluated for disease resistance via bacterial growth assays. Additionally, seedling survival or death can act as a discrete resistance or disease phenotype 7–14 days after flooding. This approach offers a high-throughput alternative for screening large numbers of wild tomato accessions for resistance to Pst race 1 strains, such as Pst strain 19 (Pst19), and can easily be adapted to other bacterial strains of interest.
Step 4.8 of the Protocol section was updated from:
- After 1 h, transfer the bottle to the biosafety cabinet and under aseptic conditions, add 1,600 µL of sterile 1 M MgSO4, and appropriate antibiotics to the media.
NOTE: For rifampicin resistant strains PstDC3000 and PstT1, use rifampicin dissolved in dimethylformamide at a final concentration of 50 µg/mL. Use cycloheximide dissolved in ethanol at a final concentration of 50 µg/mL to prevent fungal growth on the plates.
to:
- After 1 h, transfer the bottle to the biosafety cabinet and under aseptic conditions, add 1,600 µL of sterile 1 M MgSO4, and appropriate antibiotics to the media.
NOTE: For rifampicin resistant strains PstDC3000 and Pst19, use rifampicin dissolved in dimethylformamide at a final concentration of 50 µg/mL. Use cycloheximide dissolved in ethanol at a final concentration of 50 µg/mL to prevent fungal growth on the plates.
Step 5.2 of the Protocol section was updated from:
- Patch bacteria (i.e., PstT1) from a glycerol stock onto KB agar with appropriate antibiotics (section 4).
to:
- Patch bacteria (i.e., Pst19) from a glycerol stock onto KB agar with appropriate antibiotics (section 4).
Step 5.6 of the Protocol section was updated from:
- For PstT1, incubate the KB plate at 28 ˚C for 48 h prior to using bacteria in the flood experiment.
to:
- For Pst19, incubate the KB plate at 28 ˚C for 48 h prior to using bacteria in the flood experiment.
Step 6 of the Protocol section was updated from:
6. Preparation of PstT1 inoculum
to
6. Preparation of Pst19 inoculum
Step 6.2 of the Protocol section was updated from:
- Perform serial dilutions using sterile 10 mM MgCl2 solution in the biosafety cabinet. For PstT1, use a spectrophotometer to make inoculum with a starting concentration of OD600 = 0.1.
to:
- Perform serial dilutions using sterile 10 mM MgCl2 solution in the biosafety cabinet. For Pst19, use a spectrophotometer to make inoculum with a starting concentration of OD600 = 0.1.
Step 6.3 of the Protocol section was updated from:
- For PstT1, make a 1/10 dilution from the initial resuspension at OD600 = 0.1 to obtain a serial dilution at a concentration of OD600 = 0.01.
to:
- For Pst19, make a 1/10 dilution from the initial resuspension at OD600 = 0.1 to obtain a serial dilution at a concentration of OD600 = 0.01.
Step 8.3 of the Protocol section was updated from:
- Set a timer for 3 min. Measure 6 mL of final inoculum (PstT1 OD600 = 0.0075 [section 6] or PstDC3000 OD600 = 0.005 [section 7]) and transfer 6 mL of inoculum to each plate with the 10-day-old seedlings.
to:
- Set a timer for 3 min. Measure 6 mL of final inoculum (Pst19 OD600 = 0.0075 [section 6] or PstDC3000 OD600 = 0.005 [section 7]) and transfer 6 mL of inoculum to each plate with the 10-day-old seedlings.
Step 8.11 of the Protocol section was updated from:
- Phenotype after 7–10 days for PstDC3000 or 10–14 days for PstT1 (section 11). If carrying out bacterial growth assays, collect leaf tissue after 4 days (sections 9 and 10) and then phenotype (section 11). Alternatively, perform phenotypic analysis and bacterial growth assays on separate sets of plants.
to:
- Phenotype after 7–10 days for PstDC3000 or 10–14 days for Pst19 (section 11). If carrying out bacterial growth assays, collect leaf tissue after 4 days (sections 9 and 10) and then phenotype (section 11). Alternatively, perform phenotypic analysis and bacterial growth assays on separate sets of plants.
Step 10.7 of the Protocol section was updated from:
- After obtaining colony counts (Figure 2B), normalize the counts to 0.01 g of tissue for seedlings and convert to log bacterial growth (Table 1).
NOTE: The average mass of one Moneymaker-PtoS cotyledon is 0.01 g and is empirically determined22.
| Genotype1 Column A | Tissue Weight (g) Column B | # of Colonies in a spot Column C | Dilution factor for spot2 Column D | Adjusted # of Colonies3 Column E | Dilution factor for serial dilution Column F | Total # of Colonies Column G (cfu/0.01 g)4 | Average # of Colonies (cfu/0.01 g) Column H | Average Log Growth (cfu/0.01 g (log10)) Column I |
| Sample 1 | 0.004 g | 10 | 200 | calculated as: (C2 x 0.01 g) / B2 = 25 | 1000 | calculated as: (D2 x E2 x F2) = 5000000 | average for sample 1 through last sample: (ie. average G1:G3) = 7000000 | log of average ie. log(H2) = 6.85 |
| Sample 2 | 0.003 g | 15 | 200 | 50 | 1000 | 10000000 | ||
| Sample 3 | 0.002 g | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1Data shown for 3 samples | ||||||||
| 2Based on plating 5 µL x 200 for 1 mL | ||||||||
| 3Cotyledons are too small to core so colony counts were normalized to 0.01 g of tissue based on the average mass of one MoneyMaker-PtoS cotyledon (data not shown) | ||||||||
| 4Adjusted per mL based on volume plated | ||||||||
Table 1: Sample calculations for seedling bacterial growth assay. Sample calculations demonstrate how to normalize bacterial counts and determine log bacterial growth.
to:
- After obtaining colony counts (Figure 2B), normalize the counts to 0.1 g of tissue for seedlings and convert to log bacterial growth (Table 1).
NOTE: The average mass of one Moneymaker-PtoS cotyledon is 0.1 g and is empirically determined22.
| Genotype1 Column A | Tissue Weight (g) Column B | # of Colonies in a spot Column C | Dilution factor for spot2 Column D | Adjusted # of Colonies3 Column E | Dilution factor for serial dilution Column F | Total # of Colonies Column G (cfu/0.01 g)4 | Average # of Colonies (cfu/0.01 g) Column H | Average Log Growth (cfu/0.1 g (log10)) Column I |
| Sample 1 | 0.04 g | 10 | 200 | calculated as: (C2 x 0.1 g) / B2 = 25 | 1000 | calculated as: (D2 x E2 x F2) = 5000000 | average for sample 1 through last sample: (ie. average G1:G3) = 7000000 | log of average ie. log(H2) = 6.85 |
| Sample 2 | 0.03 g | 15 | 200 | 50 | 1000 | 10000000 | ||
| Sample 3 | 0.02 g | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1Data shown for 3 samples | ||||||||
| 2Based on plating 5 µL x 200 for 1 mL | ||||||||
| 3Cotyledons are too small to core so colony counts were normalized to 0.1 g of tissue based on the average mass of one MoneyMaker-PtoS cotyledon (data not shown) | ||||||||
| 4Adjusted per mL based on volume plated | ||||||||
Table 1: Sample calculations for seedling bacterial growth assay. Sample calculations demonstrate how to normalize bacterial counts and determine log bacterial growth.
Step 11.3 of the Protocol section was updated from:
- Phenotype plants infected with PstT1 at 10–14 days after flood inoculation.
to:
- Phenotype plants infected with Pst19 at 10–14 days after flood inoculation.
Figure 4 in the Protocol section was updated from:
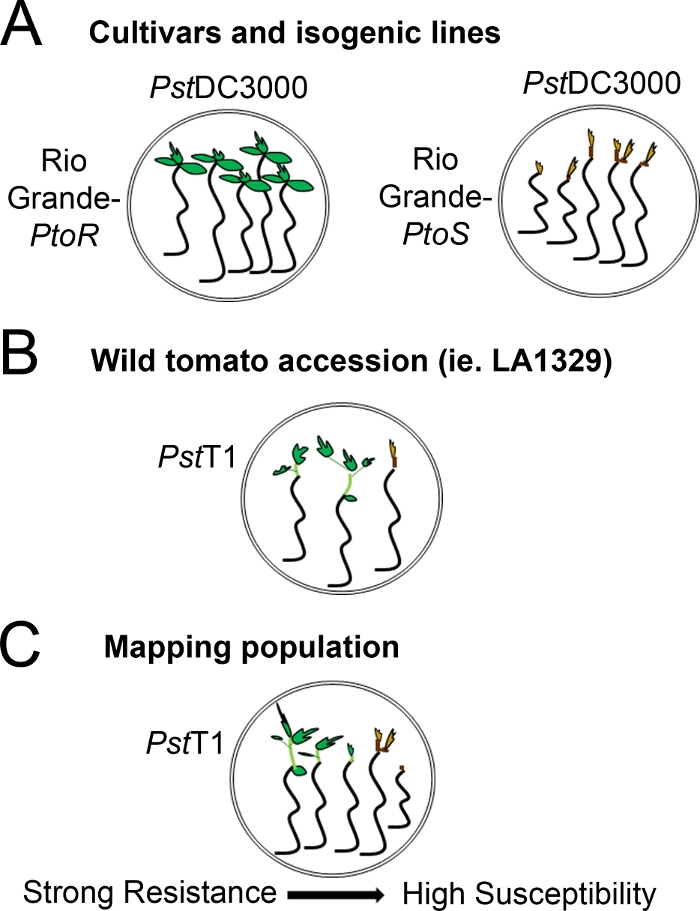
Figure 4: Schematic representation of expected phenotypes for seedling resistance and death in various genetic backgrounds. (A) Seedlings of Rio Grande-PtoR and the near-isogenic cultivar Rio Grande-PtoS are displayed 7 days after flooding with PstDC3000 (OD600 = 0.005) + 0.015% surfactant. Rio Grande-PtoR displays consistent resistance, and Rio Grande-PtoS displays consistent susceptibility to infection with PstDC3000. These lines give rise to discrete and binary phenotypes. (B) Seedlings of a wild accession, such as Solanum neorickii LA1329, are shown 10 days after flooding with PstT1 (OD600 = 0.0075) + 0.015% surfactant. Seedlings display phenotypic variability but were recorded as binary phenotypes. The amount of phenotypic variability and the method of phenotyping (binary resistance or resistance spectrum) will depend on the particular accession tested. (C) Mapping populations generated by outcrossing wild accessions to susceptible cultivars may display a wider spectrum of phenotypes in F2 segregating populations. In this case, it may be most appropriate to record seedling phenotypes on a spectrum. Highly susceptible seedlings from a mapping population may be phenotyped for death as early as day 7 when flooded with PstT1, and typically show a brown apical meristem, no to very little extension of the epicotyl, and no new, green vegetative growth. The apical meristem of susceptible seedlings may stay green or very light brown for more time, and there may be some extension of the epicotyl and very little vegetative growth, which turns brown and arrests by day 10. Individual seedlings can be phenotyped for resistance based on the amount of new and ongoing vegetative growth by day 14. Seedlings can then be grouped based on the phenotypes described above into different categories of resistance such as weak, medium, or strong resistance. Please click here to view a larger version of this figure.
to:
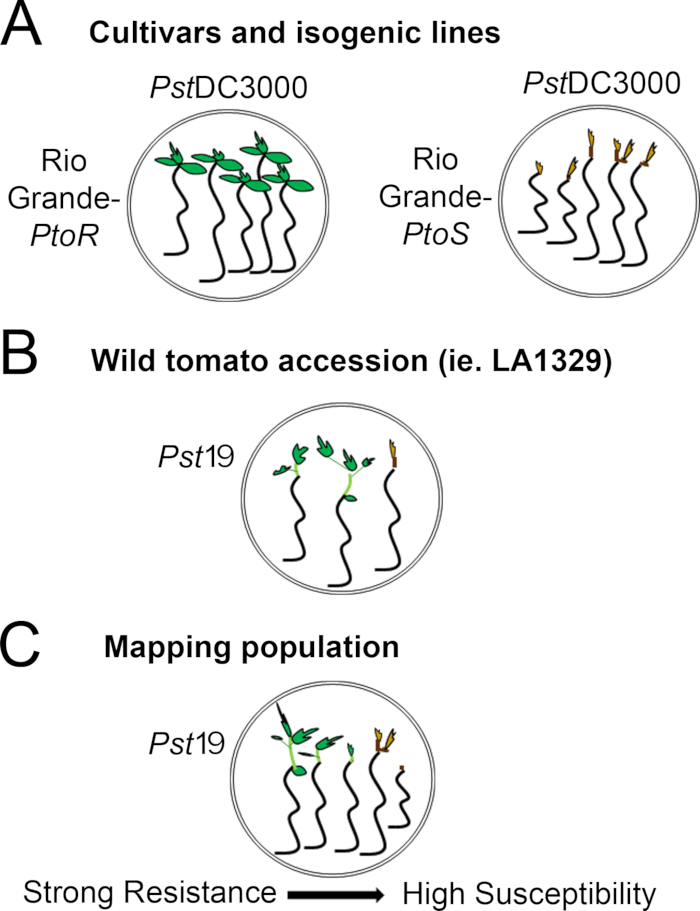
Figure 4: Schematic representation of expected phenotypes for seedling resistance and death in various genetic backgrounds. (A) Seedlings of Rio Grande-PtoR and the near-isogenic cultivar Rio Grande-PtoS are displayed 7 days after flooding with PstDC3000 (OD600 = 0.005) + 0.015% surfactant. Rio Grande-PtoR displays consistent resistance, and Rio Grande-PtoS displays consistent susceptibility to infection with PstDC3000. These lines give rise to discrete and binary phenotypes. (B) Seedlings of a wild accession, such as Solanum neorickii LA1329, are shown 10 days after flooding with Pst19 (OD600 = 0.0075) + 0.015% surfactant. Seedlings display phenotypic variability but were recorded as binary phenotypes. The amount of phenotypic variability and the method of phenotyping (binary resistance or resistance spectrum) will depend on the particular accession tested. (C) Mapping populations generated by outcrossing wild accessions to susceptible cultivars may display a wider spectrum of phenotypes in F2 segregating populations. In this case, it may be most appropriate to record seedling phenotypes on a spectrum. Highly susceptible seedlings from a mapping population may be phenotyped for death as early as day 7 when flooded with Pst19, and typically show a brown apical meristem, no to very little extension of the epicotyl, and no new, green vegetative growth. The apical meristem of susceptible seedlings may stay green or very light brown for more time, and there may be some extension of the epicotyl and very little vegetative growth, which turns brown and arrests by day 10. Individual seedlings can be phenotyped for resistance based on the amount of new and ongoing vegetative growth by day 14. Seedlings can then be grouped based on the phenotypes described above into different categories of resistance such as weak, medium, or strong resistance. Please click here to view a larger version of this figure.
The second paragraph of the Representative Results section was updated from:
Phenotypic screening of wild accessions using the seedling resistance assay
Figure 6 shows representative results for seedlings of susceptible and resistant accessions 10–14 days after flooding with PstT1. Susceptible accessions include RG-PtoR, S. pimpinellifolium LA1375, and S. pimpinellifolium LA1606, and resistant accessions include S. neorickii LA1329. Ten-day-old seedlings were flooded with 10 mM MgCl2 + 0.015% surfactant as a negative control, and PstT1 at an optical density of 0.0075 + 0.015% surfactant. The seedlings were phenotyped at least 10 days after flooding, as PstT1-infected seedlings died more slowly than PstDC3000-infected seedlings. Mock-inoculated seedlings were green, healthy, and actively growing. This control is important to ensure that the accessions are not sensitive to the concentration of surfactant, and to ensure there is no bacterial contamination. Susceptible accessions (Rio Grande-PtoR [n = 7], S. pimpinellifolium LA1375 [n = 7], and S. pimpinellifolium LA1606 [n = 5]) were dead, had brown apical meristems, and lacked new growth 10–14 days after inoculation with PstT1. In contrast, two S. neorickii LA1329 (n = 3) seedlings displayed a high level of new, green growth and survived infection with PstT1 (Figure 6). Three LA1329 seedlings did not germinate. Typically, 5–7 individuals were screened for each accession in a primary screen to determine the prevalence of resistance in the population. When a more genetically complex wild accession, such as LA1329, is flooded with PstT1, the resistance phenotypes display slightly more variability among individual seedlings, compared to Moneymaker-PtoR treated with PstDC3000. However, the resistance phenotypes were usually less variable than those seen in F2 mapping populations. Thus, binary phenotyping criteria was used for LA1329.
to:
Phenotypic screening of wild accessions using the seedling resistance assay
Figure 6 shows representative results for seedlings of susceptible and resistant accessions 10–14 days after flooding with Pst19. Susceptible accessions include RG-PtoR, S. pimpinellifolium LA1375, and S. pimpinellifolium LA1606, and resistant accessions include S. neorickii LA1329. Ten-day-old seedlings were flooded with 10 mM MgCl2 + 0.015% surfactant as a negative control, and Pst19 at an optical density of 0.0075 + 0.015% surfactant. The seedlings were phenotyped at least 10 days after flooding, as Pst19-infected seedlings died more slowly than PstDC3000-infected seedlings. Mock-inoculated seedlings were green, healthy, and actively growing. This control is important to ensure that the accessions are not sensitive to the concentration of surfactant, and to ensure there is no bacterial contamination. Susceptible accessions (Rio Grande-PtoR [n = 7], S. pimpinellifolium LA1375 [n = 7], and S. pimpinellifolium LA1606 [n = 5]) were dead, had brown apical meristems, and lacked new growth 10–14 days after inoculation with Pst19. In contrast, two S. neorickii LA1329 (n = 3) seedlings displayed a high level of new, green growth and survived infection with Pst19 (Figure 6). Three LA1329 seedlings did not germinate. Typically, 5–7 individuals were screened for each accession in a primary screen to determine the prevalence of resistance in the population. When a more genetically complex wild accession, such as LA1329, is flooded with Pst19, the resistance phenotypes display slightly more variability among individual seedlings, compared to Moneymaker-PtoR treated with PstDC3000. However, the resistance phenotypes were usually less variable than those seen in F2 mapping populations. Thus, binary phenotyping criteria was used for LA1329.
Figure 6 in the Representative Results section was updated from:
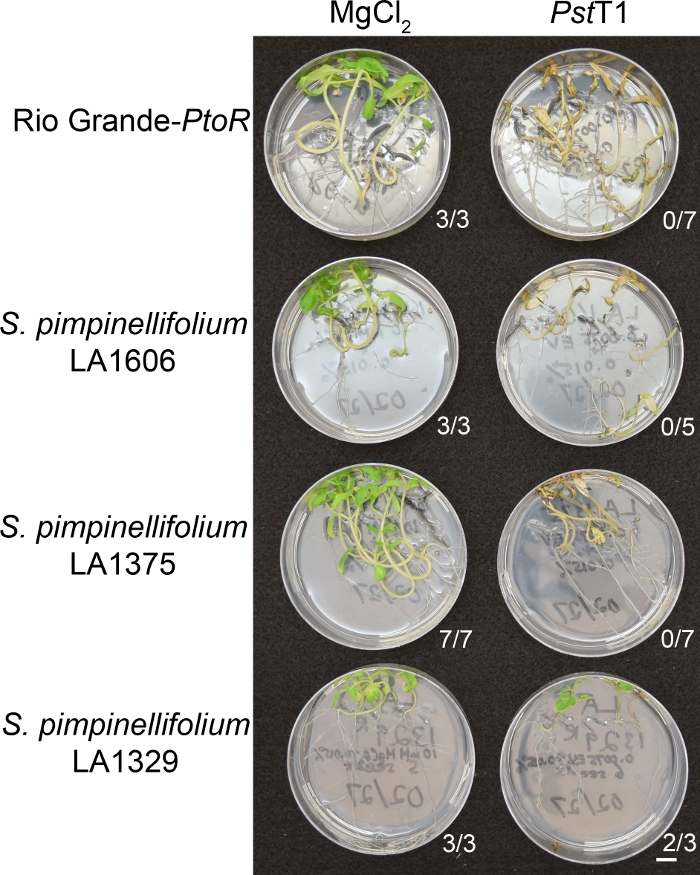
Figure 6: Phenotypic characterization of resistance or disease symptoms 10–14 days post-infection in wild accessions. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 and S. neorickii LA1329 tomato seedlings were grown on 0.5x MS plates for 10 days, and then flooded with PstT1 (OD600 = 0.0075) + 0.015% surfactant. The number of surviving seedlings for each wild accession out of the total number tested is shown. Scale bar = 1 cm. Please click here to view a larger version of this figure.
to:
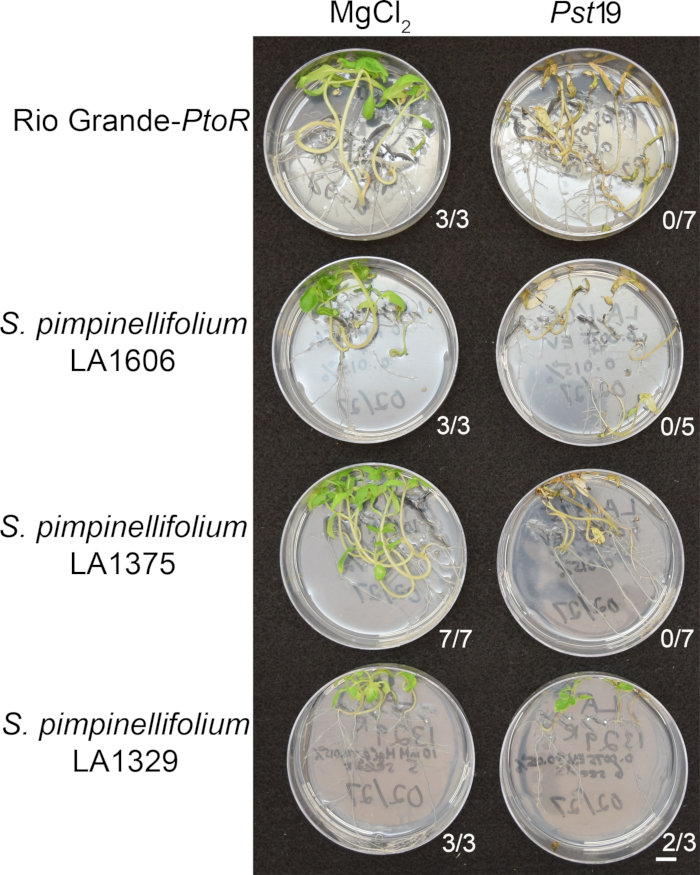
Figure 6: Phenotypic characterization of resistance or disease symptoms 10–14 days post-infection in wild accessions. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 and S. neorickii LA1329 tomato seedlings were grown on 0.5x MS plates for 10 days, and then flooded with Pst19 (OD600 = 0.0075) + 0.015% surfactant. The number of surviving seedlings for each wild accession out of the total number tested is shown. Scale bar = 1 cm. Please click here to view a larger version of this figure.
The third paragraph of the Representative Results section was updated from:
Quantitative assessment of bacterial growth using the seedling flood assay
To confirm that the observed resistance in LA1329 to PstT1 resulted in lower bacterial growth, bacterial growth assays were carried out in tomato seedlings. The level of PstT1 growth in Moneymaker-PtoS and S. neorickii LA1329 was determined 4 days post-infection. Moneymaker-PtoS is a near-isogenic line with consistent susceptibility among individual seedlings. Wild accessions such as S. neorickii LA1329 are often more genetically complex. LA1329 displays approximately 60% resistance to PstT1 across the population22. Because seedlings may drop their cotyledons after infection, one seedling was grown on each plate to correlate bacterial growth in the harvested cotyledon with overall seedling survival or death as determined phenotypically at least 10 days after flooding. The bacterial counts on day 4 for each seedling were normalized to 0.01 g of tissue and converted to log growth (CFU/0.01 g(log10)). Log growth for phenotypically resistant LA1329 seedlings (LA1329RES) or phenotypically susceptible seedlings (LA1329SUS) were separately pooled and compared to each other and the susceptible cultivar Moneymaker-PtoS. For example, there was a 1.7 log difference in bacterial growth between LA1329RES (log 6.3) and LA1329SUS (log 8.0), and a 1.6 log difference between LA1329RES (log 6.3) and Moneymaker-PtoS (log 7.9) (Figure 7). Therefore, phenotypic resistance correlated with quantitative resistance in the seedling assays.
to:
Quantitative assessment of bacterial growth using the seedling flood assay
To confirm that the observed resistance in LA1329 to Pst19 resulted in lower bacterial growth, bacterial growth assays were carried out in tomato seedlings. The level of Pst19 growth in Moneymaker-PtoS and S. neorickii LA1329 was determined 4 days post-infection. Moneymaker-PtoS is a near-isogenic line with consistent susceptibility among individual seedlings. Wild accessions such as S. neorickii LA1329 are often more genetically complex. LA1329 displays approximately 60% resistance to Pst19 across the population22. Because seedlings may drop their cotyledons after infection, one seedling was grown on each plate to correlate bacterial growth in the harvested cotyledon with overall seedling survival or death as determined phenotypically at least 10 days after flooding. The bacterial counts on day 4 for each seedling were normalized to 0.01 g of tissue and converted to log growth (CFU/0.01 g(log10)). Log growth for phenotypically resistant LA1329 seedlings (LA1329RES) or phenotypically susceptible seedlings (LA1329SUS) were separately pooled and compared to each other and the susceptible cultivar Moneymaker-PtoS. For example, there was a 1.7 log difference in bacterial growth between LA1329RES (log 6.3) and LA1329SUS (log 8.0), and a 1.6 log difference between LA1329RES (log 6.3) and Moneymaker-PtoS (log 7.9) (Figure 7). Therefore, phenotypic resistance correlated with quantitative resistance in the seedling assays.
Figure 7 in the Representative Results section was updated from:
x
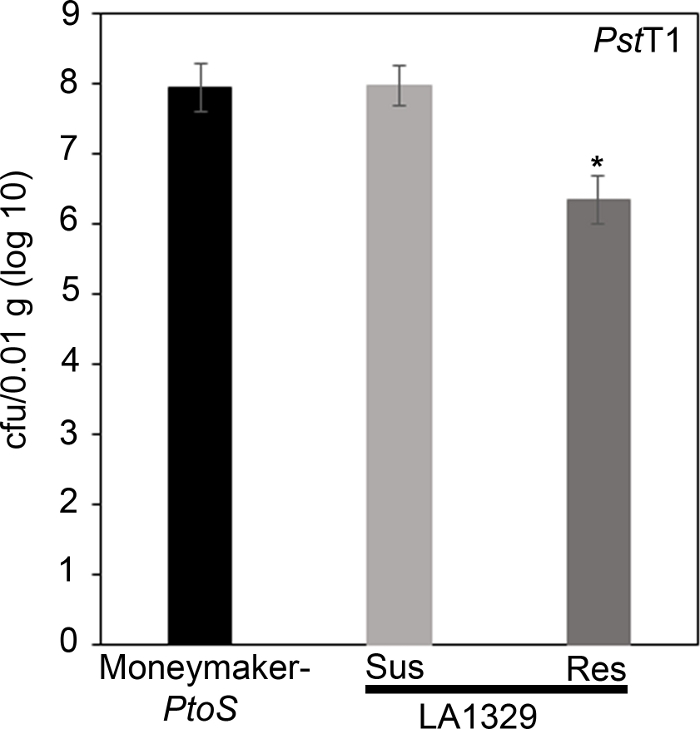
Figure 7: Resistant Solanum neorickii LA1329 seedlings support lower bacterial growth than Moneymaker-PtoS or susceptible S. neorickii LA1329. Bacterial counts were determined 4 days post-inoculation from S. neorickii LA1329 (n = 14) and Moneymaker-PtoS (n = 10) seedlings infected with PstT1 and normalization was performed to 0.01 g of tissue. For LA1329, the two phenotypic groups, susceptible (SUS) or resistant (RES), were observed and counted separately. Above the bar * = statistically significant difference determined by a one-factor analysis of variance. A general linear model procedure (p < 0.001) followed by a multiple comparison of means using Tukey's post hoc test was used. Error bars = standard error. The figure indicates one representative experiment. Please click here to view a larger version of this figure.
x
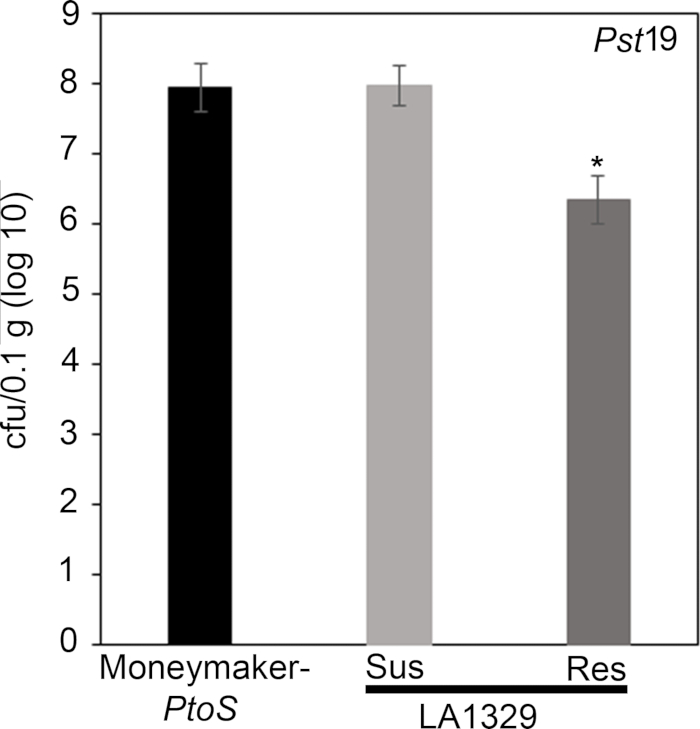
Figure 7: Resistant Solanum neorickii LA1329 seedlings support lower bacterial growth than Moneymaker-PtoS or susceptible S. neorickii LA1329. Bacterial counts were determined 4 days post-inoculation from S. neorickii LA1329 (n = 14) and Moneymaker-PtoS (n = 10) seedlings infected with Pst19 and normalization was performed to 0.1 g of tissue. For LA1329, the two phenotypic groups, susceptible (SUS) or resistant (RES), were observed and counted separately. Above the bar * = statistically significant difference determined by a one-factor analysis of variance. A general linear model procedure (p < 0.001) followed by a multiple comparison of means using Tukey's post hoc test was used. Error bars = standard error. The figure indicates one representative experiment. Please click here to view a larger version of this figure.
The first paragraph of the Discussion section was updated from:
A protocol for flood inoculation with PstDC3000 or PstT1 optimized to detect resistance to these bacterial strains in tomato seedlings is described. There are several critical parameters for optimal results in the seedling resistance assay, including bacterial concentration and surfactant concentration, which were empirically determined22. For PstDC3000, the optical density was optimized to achieve complete survival on a resistant cultivar containing the Pto/Prf cluster and complete death on a susceptible cultivar lacking the Pto/Prf cluster22. For a strain such as PstT1, where there are no known resistant varieties, the optical density was optimized to be the lowest possible for consistent and complete plant death22. Uppalapati et al.24 designed a tomato seedling assay to investigate the pathogenesis of PstDC3000 and the virulence function of coronatine. In this virulence assay, infections were performed using bacteria concentrated to an OD600 of 0.124, 20x higher than the optical density of strains used in our resistance assay. Recognition of PstDC3000 effectors AvrPto and AvrPtoB in tomato seedlings carrying the Pto/Prf gene cluster results in ETI and a macroscopic HR22. In the context of a strong immune response such as ETI, a lower bacterial titer was used for PstDC3000 to avoid overwhelming genetic resistance from the Pto/Prf gene cluster22. In addition, these results suggest that a high bacterial concentration could overwhelm weaker immune responses such as PTI or quantitative partial resistance, where multiple genes contribute to the overall phenotype. Surfactant is necessary for the bacteria to adhere to the leaf surface; however, high concentrations can cause chlorosis of the leaf22. We previously tested a range of surfactant concentrations to empirically determine the ideal concentration in 10-day-old tomato seedlings22. When testing new species that may differ in their sensitivity to surfactant, the surfactant concentration should be optimized to identify a concentration that does not cause damage or chlorosis in the absence of bacteria. Appropriate assay conditions will require optimization of a surfactant concentration that does not cause damage, and a bacterial concentration that causes disease in all susceptible controls.
to:
A protocol for flood inoculation with PstDC3000 or Pst19 optimized to detect resistance to these bacterial strains in tomato seedlings is described. There are several critical parameters for optimal results in the seedling resistance assay, including bacterial concentration and surfactant concentration, which were empirically determined22. For PstDC3000, the optical density was optimized to achieve complete survival on a resistant cultivar containing the Pto/Prf cluster and complete death on a susceptible cultivar lacking the Pto/Prf cluster22. For a strain such as Pst19, where there are no known resistant varieties, the optical density was optimized to be the lowest possible for consistent and complete plant death22. Uppalapati et al.24 designed a tomato seedling assay to investigate the pathogenesis of PstDC3000 and the virulence function of coronatine. In this virulence assay, infections were performed using bacteria concentrated to an OD600 of 0.124, 20x higher than the optical density of strains used in our resistance assay. Recognition of PstDC3000 effectors AvrPto and AvrPtoB in tomato seedlings carrying the Pto/Prf gene cluster results in ETI and a macroscopic HR22. In the context of a strong immune response such as ETI, a lower bacterial titer was used for PstDC3000 to avoid overwhelming genetic resistance from the Pto/Prf gene cluster22. In addition, these results suggest that a high bacterial concentration could overwhelm weaker immune responses such as PTI or quantitative partial resistance, where multiple genes contribute to the overall phenotype. Surfactant is necessary for the bacteria to adhere to the leaf surface; however, high concentrations can cause chlorosis of the leaf22. We previously tested a range of surfactant concentrations to empirically determine the ideal concentration in 10-day-old tomato seedlings22. When testing new species that may differ in their sensitivity to surfactant, the surfactant concentration should be optimized to identify a concentration that does not cause damage or chlorosis in the absence of bacteria. Appropriate assay conditions will require optimization of a surfactant concentration that does not cause damage, and a bacterial concentration that causes disease in all susceptible controls.
The third paragraph of the Discussion section was updated from:
Pst is a foliar pathogen that preferentially colonizes the aerial parts of tomato seedlings, including the cotyledons24 (Figure 3). Therefore, qualitative phenotyping in the seedling flood assay focuses on growth and disease symptoms in aerial portions of the seedling, and tissue for the bacterial growth assay is sampled from the cotyledons for quantitative analysis. After flood inoculation, seedlings may die within 7–10 days after inoculation with PstDC3000 or 10–14 days after inoculation with PstT1, as discussed in section 11. Seedling death is visualized by a brown apical meristem, arrested epicotyl elongation, and/or arrested vegetative growth. If different bacterial strains are used, the timing will have to be empirically determined. In addition, the progression of disease on control plants should be monitored daily after flooding until a consistent time frame from the onset of disease symptoms to seedling death can be identified. Depending on the genotypes and treatments used in the flood assay, seedling phenotypes can be recorded as binary phenotypes or on a disease spectrum (Figure 4). A broader spectrum of phenotypes may be observed when flood inoculating F2 mapping populations from wild tomato accessions crossed to susceptible cultivars (Figure 4C). It may be best to phenotype segregating populations on a disease spectrum depending on how quickly the seedling dies and the degree of new vegetative growth and branching (Figure 4C). The seedling flood assay can also be used in conjunction with the seedling bacterial growth assay to quantitatively assess levels of bacterial growth associated with qualitative phenotypes in individual seedlings (Figure 7). Very large reductions (i.e., ~log 3) in bacterial growth or strong resistance in resistant seedlings of a wild accession compared to a susceptible cultivar suggest that the underlying genetic basis of resistance may be due to ETI22. Smaller reductions in bacterial growth (i.e., ~log 1.7), as observed in LA1329 seedlings, may be due to the contribution of weaker resistance from quantitative trait loci and/or PTI. Thus, the seedling growth assay can be an important tool in further characterizing resistance in wild tomato lines.
to:
Pst is a foliar pathogen that preferentially colonizes the aerial parts of tomato seedlings, including the cotyledons24 (Figure 3). Therefore, qualitative phenotyping in the seedling flood assay focuses on growth and disease symptoms in aerial portions of the seedling, and tissue for the bacterial growth assay is sampled from the cotyledons for quantitative analysis. After flood inoculation, seedlings may die within 7–10 days after inoculation with PstDC3000 or 10–14 days after inoculation with Pst19, as discussed in section 11. Seedling death is visualized by a brown apical meristem, arrested epicotyl elongation, and/or arrested vegetative growth. If different bacterial strains are used, the timing will have to be empirically determined. In addition, the progression of disease on control plants should be monitored daily after flooding until a consistent time frame from the onset of disease symptoms to seedling death can be identified. Depending on the genotypes and treatments used in the flood assay, seedling phenotypes can be recorded as binary phenotypes or on a disease spectrum (Figure 4). A broader spectrum of phenotypes may be observed when flood inoculating F2 mapping populations from wild tomato accessions crossed to susceptible cultivars (Figure 4C). It may be best to phenotype segregating populations on a disease spectrum depending on how quickly the seedling dies and the degree of new vegetative growth and branching (Figure 4C). The seedling flood assay can also be used in conjunction with the seedling bacterial growth assay to quantitatively assess levels of bacterial growth associated with qualitative phenotypes in individual seedlings (Figure 7). Very large reductions (i.e., ~log 3) in bacterial growth or strong resistance in resistant seedlings of a wild accession compared to a susceptible cultivar suggest that the underlying genetic basis of resistance may be due to ETI22. Smaller reductions in bacterial growth (i.e., ~log 1.7), as observed in LA1329 seedlings, may be due to the contribution of weaker resistance from quantitative trait loci and/or PTI. Thus, the seedling growth assay can be an important tool in further characterizing resistance in wild tomato lines.
The fourth paragraph of the Discussion section was updated from:
Typically, genetic screens have been performed on four- to five-week-old adult tomato plants to identify the genetic basis of P. syringae resistance in wild accessions20,21. Adult tomato plants require much longer growth times, require more space in the growth chamber, and are much larger plants, which means that usually few individuals are screened for each line. The seedling flood assay provides a powerful, alternative approach in the identification of P. syringae resistance in wild tomato accessions. Screening at the seedling stage permits a large sample size to be tested which can be particularly advantageous in detecting resistance in genetically complex populations. Reduced growth chamber space requirements and growth time facilitate a high-throughput approach and rapid detection of natural resistance in wild accessions to emerging pathogens. Furthermore, P. syringae resistance that was identified at the seedling stage in this assay is not restricted to the developmental stage. S. neorickii LA1329 and S. habrochaites LA1253 were initially identified at the seedling stage and also display resistance to PstT1 in adult plants as previously described22.
to:
Typically, genetic screens have been performed on four- to five-week-old adult tomato plants to identify the genetic basis of P. syringae resistance in wild accessions20,21. Adult tomato plants require much longer growth times, require more space in the growth chamber, and are much larger plants, which means that usually few individuals are screened for each line. The seedling flood assay provides a powerful, alternative approach in the identification of P. syringae resistance in wild tomato accessions. Screening at the seedling stage permits a large sample size to be tested which can be particularly advantageous in detecting resistance in genetically complex populations. Reduced growth chamber space requirements and growth time facilitate a high-throughput approach and rapid detection of natural resistance in wild accessions to emerging pathogens. Furthermore, P. syringae resistance that was identified at the seedling stage in this assay is not restricted to the developmental stage. S. neorickii LA1329 and S. habrochaites LA1253 were initially identified at the seedling stage and also display resistance to Pst19 in adult plants as previously described22.



