ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
El ensayo de inundación de plántulas facilita el cribado rápido de las adhesiones de tomate silvestre para la resistencia a la bacteria pseudomonas de las siestas. Este ensayo, utilizado junto con el ensayo de crecimiento bacteriano de plántulas, puede ayudar a caracterizar aún más la resistencia subyacente a la bacteria, y se puede utilizar para examinar las poblaciones de mapeo para determinar la base genética de la resistencia.
Abstract
El tomate es un cultivo de importancia agronómica que puede ser infectado por Pseudomonas syringae,una bacteria Gram-negativa, lo que resulta en la enfermedad bacteriana de la mota. El tomateP. syringae pv. el pathosistema de tomate se utiliza ampliamente para diseccionar la base genética de las respuestas innatas de las plantas y la resistencia a las enfermedades. Mientras que la enfermedad se manejó con éxito durante muchas décadas a través de la introducción del grupo de genes Pto/Prf de Solanum pimpinellifolium en tomate cultivado, las cepas de la raza 1 de P. syringae han evolucionado para superar la resistencia conferida por el grupo de genes Pto/Prf y se producen en todo el mundo.
Las especies de tomate sin efecto son importantes reservorios de diversidad natural en el reconocimiento de patógenos, ya que evolucionaron en diversos ambientes con diferentes presiones de patógenos. En las pantallas típicas para la resistencia a las enfermedades en el tomate silvestre, se utilizan plantas adultas, que pueden limitar el número de plantas que se pueden examinar debido a su mayor tiempo de crecimiento y mayores requisitos de espacio de crecimiento. Desarrollamos un método para detectar resistencia a las plántulas de tomate de 10 días de edad, lo que minimiza el tiempo de crecimiento de las plantas y el espacio de la cámara de crecimiento, permite una rápida rotación de plantas y permite probar grandes tamaños de muestra. Los resultados de supervivencia o muerte pueden tratarse como fenotipos discretos o en una escala de resistencia definida por la cantidad de nuevo crecimiento en las plántulas supervivientes después de las inundaciones. Este método se ha optimizado para examinar las plántulas de tomate de 10 días de edad para la resistencia a dos cepas de P. syringae y se puede adaptar fácilmente a otras cepas de P. syringae.
Introduction
Pseudomonas syringae es una bacteria patógena Gram-negativa que infecta una amplia gama de huéspedes de plantas. Las bacterias entran en la planta huésped a través de los estomas o heridas físicas y proliferan en el apoplast1. Las plantas han desarrollado una respuesta inmune de dos niveles para proteger contra la infección por patógenos bacterianos. El primer nivel se produce en la superficie de la célula vegetal, donde los receptores de reconocimiento de patrones en la membrana celular de la planta perciben patrones moleculares asociados a patógenos altamente conservados (PamP) en un proceso llamado inmunidad desencadenada por PAMP (PTI)2. Durante este proceso, la planta anfitriona regula las vías de respuesta de defensa, incluyendo la deposición de calisa a la pared celular, el cierre de estomas, la producción de especies reactivas de oxígeno e la inducción de genes relacionados con la patogénesis.
Las bacterias pueden superar la PTI mediante la utilización de un sistema de secreción de tipo III para suministrar proteínas, llamadas efectores, directamente en la célula de la planta3. Las proteínas efectoras comúnmente se dirigen a los componentes de la PTI y promueven la virulencia de patógenos4. El segundo nivel de inmunidad de la planta se produce dentro de la célula vegetal tras el reconocimiento de las proteínas efectoras. Este reconocimiento depende de los genes de resistencia, que codifican la repetición rica en leucina (NRM) del sitio de unión a nucleótidos. Las NLE son capaces de reconocer a los efectores directamente o reconocer su actividad en un objetivo de virulencia o señuelo5. A continuación, desencadenan una respuesta inmune secundaria en un proceso llamado inmunidad desencadenada por el efector (ETI), que a menudo se asocia con una respuesta hipersensible (HR), una forma de muerte celular localizada en el sitio de la infección6. A diferencia de la resistencia gen-por-genes asociada con la ETI, las plantas pueden exhibir resistencia parcial cuantitativa, que depende de la contribución de múltiples genes7.
P. syringae pv. el tomate (Pst) es el agente causal de la mota bacteriana en el tomate y es un problema agrícola persistente. Las cepas predominantes en el campo han sido típicamente Pst raza 0 cepas que expresan uno o ambos de los efectores tipo III AvrPto y AvrPtoB. DC3000 (PstDC3000) es una cepa representativa de raza 0 y un patógeno modelo que puede causar mota bacteriana en el tomate. Para combatir la enfermedad bacteriana de la mota, los criadores introgresieron el grupo de genes Pto [P. syringae pv. tomate]/Prf [Resistencia al pto y sensibilidad al fentionamiento] de las especies de tomates silvestres Solanum pimpinellifolium en cultivares modernos8,9. El gen Pto codifica una proteína quinasa de serina-talonina que, junto con el Prf NLR, confiere resistencia a PstDC3000 mediante el reconocimiento de los efectores AvrPto y AvrPtoB10,11,12,13,14. Sin embargo, esta resistencia es ineficaz contra las cepas emergentes de la raza 1, permitiendo su rápida y agresiva propagación en los últimos años15,16. Las cepas de la raza 1 evaden el reconocimiento por parte del clúster Pto/Prf, ya que AvrPto se pierde o muta en estas cepas, y AvrPtoB parece acumularse mínimamente15,17,18.
Las poblaciones de tomate silvestre son importantes reservorios de variación natural para la resistencia Pst y se han utilizado previamente para identificar la resistencia potencial loci19,20,21. Sin embargo, las pantallas actuales para la resistencia a patógenos utilizan plantas adultas de 4 a 5 semanas de edad20,21. Por lo tanto, están limitados por el tiempo de crecimiento, el espacio de la cámara de crecimiento y los tamaños de muestra relativamente pequeños. Para hacer frente a las limitaciones de los enfoques convencionales, desarrollamos un ensayo de resistencia a las siringas de tomate de alto rendimiento utilizando plántulas de tomate de 10 días de edad22. Este enfoque ofrece varias ventajas sobre el uso de plantas adultas: a saber, un menor tiempo de crecimiento, menores requisitos de espacio y un mayor rendimiento. Además, hemos demostrado que este enfoque recapitula fielmente los fenotipos de resistencia a la enfermedad observados en plantas adultas22.
En el ensayo de inundación de plántulas descrito en este protocolo, las plántulas de tomate se cultivan en platos Petri de medios estériles de Murashige y Skoog (MS) durante 10 días y luego se inundan con un inóculo que contiene las bacterias de interés y un tensioactivo. Después de las inundaciones, las plántulas pueden ser evaluadas cuantitativamente para la resistencia a la enfermedad a través de ensayos de crecimiento bacteriano. Además, la supervivencia de la plántula o la muerte pueden actuar como una resistencia discreta o fenotipo de enfermedad 7-14 días después de la inundación. Este enfoque ofrece una alternativa de alto rendimiento para el cribado de un gran número de adhesiones de tomate silvestre para la resistencia a las cepas de la raza Pst 1, como la cepa Pst T1 (PstT1), y se puede adaptar fácilmente a otras cepas bacterianas de interés.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Preparación y uso del gabinete de bioseguridad
- Limpie el gabinete de bioseguridad con un 70% de etanol.
- Cierre la faja y encienda la luz ultravioleta en el gabinete de bioseguridad durante 15 minutos.
- Después de 15 minutos, apague la luz ultravioleta en el gabinete de bioseguridad. Levante la faja y encienda el soplador durante 15 minutos.
- Limpie todos los artículos que se utilizarán en el gabinete de bioseguridad con un 70% de etanol antes de colocar los artículos en el armario esterilizado.
- Limpie los guantes o las manos desnudas con 70% de etanol antes de trabajar en el gabinete de bioseguridad.
- Trabajar en el centro del gabinete de bioseguridad, lejos del soplador.
- Utilice botellas sin abrir de autoclave estéril 10 mM MgCl2 y ultrapura H2O para experimentos. Ponga las botellas en el gabinete de bioseguridad y sólo ábralas en el gabinete de bioseguridad esterilizado, no en la mesa de trabajo.
- Utilice pipetas de vidrio y puntas de pipeta dedicadas para trabajar en el gabinete de bioseguridad esterilizado. Asegúrese de que solo se abren en el armario de bioseguridad, nunca en la mesa de trabajo.
- Después del uso del gabinete de bioseguridad, autoclave todos los residuos (excepto los residuos de lejía) y limpiar la superficie con 70% de etanol.
2. Preparación de medios vegetales
- Pesar y disolver 0,5x Sales basales MS en ultrapuro H2O. Pesar 0.8% bacto agar y luego añadir a disuelto 0.5x Em.
- Autoclave y dejar que los medios se enfríen en un baño de agua de 50oC durante 1 h antes de verter o entubar.
- Para asegurarse de que las placas no estén sobrecargadas, marque las placas estériles desechables de poliestireno de 100 x 25 mm a un nivel de llenado de 40 ml. Vierta los medios en placas estériles de 100 x 25 mm en un gabinete de bioseguridad esterilizado.
3. Preparación de materiales vegetales y condiciones de crecimiento
- Coloque las semillas de tomate en un tubo de microcentrífuga de 2,2 ml y agregue 2,0 ml de solución de lejía al 50%.
- Mece el tubo en un balancín durante 25 minutos.
- Después de 25 minutos, retire las semillas del balancín y retire la solución de lejía con una pipeta en el gabinete de bioseguridad estéril. Asegúrese de que se retira toda la lejía.
- Añadir 2 ml de Ultrapure H2O estéril para lavar las semillas. Invertir el tubo 5x.
- Retire el líquido del tubo con una pipeta.
- Repita los pasos 3.3–3.5 para lavar las semillas 4 veces más.
- Añadir 2 ml de ultrapuro estéril H2O y verter las semillas en un plato estéril vacío de Petri.
- La llama fórceps en etanol y permite enfriar antes de transferir y espaciar uniformemente las semillas en placas de 100 x 25 mm que contienen 0,5x MS + 0,8% de medios de agar.
- Transfiera de 5 a 7 semillas en una línea a través de la mitad de una placa y selle los bordes de las placas con cinta quirúrgica (1,25 cm x 9,1 m).
- Estratificar las semillas esterilizadas a 4oC en la oscuridad durante al menos 3 días para sincronizar la germinación. Asegúrese de que las placas estén apiladas planas y boca arriba, para que las semillas no se desplacen en la placa.
- Orientar verticalmente las placas para que las raíces crezcan a lo largo de la superficie de la placa, con la línea de semillas orientada horizontalmente, al transferira a la cámara de crecimiento.
NOTA: Ajuste la cámara de crecimiento a 22 oC y proporcione 16 h de luz a una intensidad de luz de 200–220 oE de metro-2 s-1 y 8 h de oscuridad. - Antes de las inundaciones, las plántulas crecen durante 10 días en la cámara de crecimiento, momento en el que las plántulas suelen mostrar sedes completamente emergidas y ampliadas cotiledonas y las primeras hojas verdaderas emergentes(Figura 1).

Figura 1: Etapa de desarrollo de las plántulas de tomate típicas de 10 días de edad. Las semillas de tomate Rio Grande-PtoR fueron esterilizadas, chapadas y estratificadas durante al menos 3 días en la oscuridad a 4oC. Las plántulas fueron cultivadas en placas 0.5x MS durante 10 días a 22 oC antes de ser inundadas. Típicamente, a los 10 días los cotiledones están completamente expandidos, y las primeras hojas verdaderas están empezando a emerger. Haga clic aquí para ver una versión más grande de esta figura.
4. Preparación de los medios del Rey B23 (KB)
- Llene el vaso de precipitados con 500 ml de Ultrapure H2O y revuelva en una placa de agitación.
- Disolver completamente 20 g de peptona, 1,5 g de Anhidro K2HPO4y 12,5 ml de glicerol en un vaso de precipitados con Ultrapure H2O.
- Vierta la mezcla disuelta en un cilindro graduado de 1 L y lleve hasta un volumen final de 1 L con Ultrapure H2O.
- Vierta el caldo de nuevo en el vaso de precipitados y revuelva hasta que se mezcle.
- Pesar 7,5 g de agar bacto en dos botellas de vidrio de 500 ml y añadir 500 ml de caldo KB del paso 4.4 en cada botella. Autoclave durante 20 min.
- Retire las botellas del autoclave y gire suavemente para distribuir el agar.
- Transfiera las botellas a un baño de agua de 50oC durante 1 h.
- Después de 1 h, transfiera el frasco al gabinete de bioseguridad y en condiciones asépticas, agregue 1.600 ml de 1 M MgSO4estéril y los antibióticos adecuados a los medios.
NOTA: Para cepas resistentes a rifampicina PstDC3000 y PstT1, utilice rifampicina disuelta en dimetilformamida a una concentración final de 50 g/ml. Utilice cicloheximida disuelta en etanol a una concentración final de 50 g/ml para evitar el crecimiento de hongos en las placas. - Gire el soporte suavemente para mezclar y luego vierta para cubrir la parte inferior de las placas.
- Dejar al menos 1 h para que las placas se solidifiquen antes de almacenarlas boca abajo a 4 oC.
5. Mantenimiento de cepas bacterianas y condiciones de cultivo
- Mantener una población de glicerol de una sola colonia de bacterias como 1 ml de cultivo bacteriano saturado y 333 ml de glicerol estéril al 80% de glicerol a -80 oC.
- Parchear las bacterias (es decir, PstT1) de una acción de glicerol en el agar KB con antibióticos apropiados (sección 4).
- Permita que las bacterias se recuperen durante 2 días a 28 oC antes de rayar bacterias frescas en el agar KB selectivo usando un palillo plano y estéril.
- Rasga las bacterias frescas de la culata de glicerol en un agar KB selectivo apropiado usando un palillo de dientes plano y estéril.
NOTA: Asegúrese de que el stock de glicerol parcheado no tenga más de 2 semanas. - Para PstDC3000, incubar la placa KB a 28 oC durante 24 horas antes de usar bacterias en el experimento de inundación.
- Para PstT1, incubar la placa KB a 28 oC durante 48 horas antes de usar bacterias en el experimento de inundación.
6. Preparación del inóculo PstT1
- Resuspenda asépticamente la bacteria en mgCl2 estéril de 10 mM a una densidad óptica a 600 nm (OD600) de 0,1, o aproximadamente 5 x 107 unidades formadoras de colonias (CFU)/ml).
- Realice diluciones en serie utilizando una solución estéril de 10 mM MgCl2 en el gabinete de bioseguridad. Para PstT1, utilice un espectrofotómetro para hacer inóculo con una concentración inicial de OD600 a 0,1.
- Para PstT1, haga una dilución de 1/10 a partir de la resuspensión inicial en OD600 a 0,1 para obtener una dilución en serie a una concentración de600 o00 a 0,01.
- Usando la dilución en serie a OD600 a 0,01 del paso 6.3, realice una dilución de 3/4 para obtener una DA OD finalde 600 a 0,0075.
- Hacer una dilución 1/10 de copolímero tensioactivo organosioactivo no iónico C13H34O4Si3 (es decir, surfactante) en 10 mM MgCl2 y vórtice para 15 s. Añadir el stock 1/10 de surfactante a la última dilución en serie (OD600 a 0,0075) a una concentración final de 0,015% y agitar bien.
7. Preparación del inóculo PstDC3000
- Resuspenda asépticamente las bacterias en mgCl2 estériles de 10 mM a una densidad óptica a 600 nm(600OD) de 0,1 (aproximadamente 5 x 107 CFU/ml).
- Realice diluciones en serie utilizando una solución estéril de 10 mM MgCl2 en el gabinete de bioseguridad. Para PstDC3000, utilice un espectrofotómetro para hacer inóculo con una concentración inicial de OD600 a 0,1.
- Para PstDC3000, realice una dilución de 1/10 a partir de la resuspensión inicial en OD600 a 0,1 para obtener una dilución en serie a una concentración de600 o00 a 0,01.
- Usando la dilución en serie a OD600 a 0,01 del paso 3, realice una dilución de 1/2 para obtener una OD finalde 600 a 0,005.
- Hacer una dilución de 1/10 de surfactante en 10 mM MgCl2 y vórtice durante 15 s. Añadir el stock 1/10 de surfactante a la última dilución en serie (OD600 a 0,005) a una concentración final de 0,015% y girar bien para mezclar.
8. Método de inundación de plántulas de tomate
- Saque las placas con las plántulas de 10 días de la cámara de crecimiento y colóquelas en el armario de bioseguridad para preparar las placas para la inundación.
- Retire la cinta quirúrgica de dos placas.
- Ajuste un temporizador para 3 min. Medir 6 ml de inóculo final (PstT1 OD600 a 0.0075 [sección 6] o PstDC3000 OD600 a 0.005 [sección 7]) y transferir 6 ml de inóculo a cada placa con las plántulas de 10 días de edad.
- Empuje suavemente las plántulas hacia abajo en el inóculo con una punta de pipeta estéril. Encienda el temporizador.
- Sostenga un plato en cada mano. Incline la parte delantera de la placa hacia abajo para acumular inóculo y sumergir principalmente los cotiledons y hojas de las plántulas.
- Swish de lado a lado 5-7x y luego inclinar las placas hacia atrás para cubrir las raíces y toda la placa.
- Incline las placas hacia abajo de nuevo para sumergir los cotiledons y hojas, y repita durante un total de 3 minutos.
- Vierta el inóculo de las placas, ponga las placas sobre una superficie plana y luego vierta cualquier inóculo residual una segunda vez.
- Vuelva a envolver las placas con cinta quirúrgica y repita los pasos 8.2–8.8 para las placas restantes.
- Vuelva a incubar las placas en la cámara de crecimiento (ver paso 3.11 NOTA) después de que todas las placas se hayan inundado.
- Fenotipo después de 7-10 días para PstDC3000 o 10–14 días para PstT1 (sección 11). Si lleva a cabo ensayos de crecimiento bacteriano, recoja el tejido de la hoja después de 4 días (secciones 9 y 10) y luego el fenotipo (sección 11). Alternativamente, realizar análisis fenotípicos y ensayos de crecimiento bacteriano en conjuntos separados de plantas.
9. Esterilización superficial de cotiledones para el ensayo de crecimiento bacteriano
- Cuatro días después de inundar y volver a incubar las plántulas en la cámara de crecimiento (sección 8), retire las placas con las plántulas de tomate de la cámara de crecimiento.
- Numerar las plántulas individuales en el exterior inferior de la placa donde la plántula se une a la placa para cada genotipo.
- Etiquete tubos de microcentrífuga estériles de 1,5 ml con los números de plántulas individuales y utilice fórceps limpios para soltar un cordón de borosilicato estéril de 3 mm en cada tubo para su uso con un batidor de perlas. (Véase NOTA en el paso 10.1.)
- Pipetear 200 ml de 10 mM MgCl2 en cada tubo y tubos de cierre.
- Preparar 70% de etanol y verter 100 ml en un vaso de precipitados limpio. Vierta 100 ml de Ultrapure H2O estéril en un vaso de precipitados separado y limpio.
- Fórceps rectos de acero inoxidable con puntas serradas con etanol. Abra ligeramente la placa para permitir la extracción aséptica de un cotilería con los fórceps limpios.
- Pellizcar el pecíolo en la base del cotiledón para eliminar una hoja y caer en el vaso de precipitados con 70% de etanol para esterilizar en superficie durante 10 s. Enjuagar el cotiledón en Ultrapure H2O durante 10 s.
- Coloque el cotilería sobre una toalla de papel y seque con delicadas toallitas científicas.
- Pesar individualmente cada cotilete después de la esterilización y hincha de la superficie, y registrar el peso.
- Coloque el cotilerín en un tubo de microcentrífuga previamente preparado de 1,5 ml (a partir de los pasos 9.3 y 9.4) etiquetado con el genotipo y el número individual correspondientes.
- Vuelva a sellar las placas con cinta estéril y vuelva a incubar las plántulas en la cámara de crecimiento (ver paso 3.11 NOTA).
10. Ensayo de crecimiento bacteriano
- Usando muestras del paso 9.10, homogeneizar el tejido usando el batidor de cuentas en 10 mM MgCl2 durante 1-2 min. Si el tejido no está adecuadamente macerado, homogeneizar de nuevo.
NOTA: Muchos fabricantes producen homogeneizadores de batidores de perlas. El número y tipo de perlas, así como el tiempo y la velocidad de homogeneización (si se programan) deben optimizarse para cada tipo de homogeneizador. Asegúrese de que las muestras no se sobrecalienten durante la homogeneización. - Añadir 800 ml de 10 mM MgCl2 a cada tubo que contenga tejido macerado del paso 10.1 e invertir varias veces para mezclar.
- Preparar diluciones en serie para cada muestra en 10 mM MgCl2 en placa de pozo 96 (100, 10-1, 10-2, 10-3, 10-4, 10-5) utilizando una pipeta multicanal(Figura 2A).
- Pipeta de 5 ml de cada serie de dilución utilizando una pipeta multicanal en una placa de agar KB (150 mm x 15 mm) con cicloheximida y selección adecuada para la cepa bacteriana de interés (ver paso 4.8 NOTA). Deje que las placas se sequen por completo.
- Incubar la placa boca abajo a 28 oC durante 36 h, luego visualizar(Figura 2B) las colonias en las placas utilizando un microscopio de disección para determinar si las colonias son lo suficientemente grandes como para contar.
NOTA: Si las colonias no son lo suficientemente grandes, vuelva a incubar las placas y vuelva a comprobar el tamaño de las colonias cada pocas horas. Típicamente, las colonias son contables de 36 a 48 h después de la incubación.

Figura 2: Diluciones en serie para ensayos de crecimiento bacteriano de plántulas. (A) El tejido de hojas maceradas de las plantas infectadas se diluye antes del recuento de colonias. Las diluciones se realizan en una placa de 96 pocillos (100 no está diluido). Típicamente, las diluciones se hacen de 10-1 a 10-5. (B) Diluciones de chapado para recuentos de colonias bacterianas. Se chapa un total de 5 l de cada columna de la serie de dilución, desde la más diluja hasta la más concentrada. Después de que las colonias se hayan secado completamente, la placa se incuba a 28 oC durante 36-48 h. Las colonias se cuentan bajo un microscopio de disección de 10x. Haga clic aquí para ver una versión más grande de esta figura.
- Cuente las colonias bajo un microscopio de disección antes de fusionarse(Figura 2B). Cuente las colonias de las placas de la serie de dilución con menos de 100 colonias.
- Después de obtener recuentos de colonias(Figura 2B),normalice los recuentos a 0,01 g de tejido para plántulas y conviértalo en crecimiento bacteriano log (Tabla 1).
NOTA: La masa media de un cotiledón Moneymaker-PtoS es de 0,01 g y se determina empíricamente22.
| Genotipo1 Columna A | Peso tisú (g) Columna B | N.o de colonias en un lugar Columna C | Factor de dilución para el punto2 Columna D | Ajustado n.o de colonias3 Columna E | Factor de dilución para dilución en serie Columna F | Número total de Colonias Columna G (cfu/0.01 g)4 | Número promedio de colonias (cfu/0.01 g) Columna H | Crecimiento medio del registro (cfu/0,01 g (log10)) Columna I |
| Muestra 1 | 0.004 g | 10 | 200 | calculado según: (C2 x 0,01 g) / B2 a 25 | 1000 | calculado según: (D2 x E2 x F2) a 5000000 | promedio para la muestra 1 hasta la última muestra: (es decir, promedio G1:G3) a 7000000 | registro de promedio, es decir. log(H2) á 6,85 |
| Muestra 2 | 0.003 g | 15 | 200 | 50 | 1000 | 10000000 | ||
| Muestra 3 | 0.002 g | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1 Datos mostrados para 3 muestras | ||||||||
| 2 Basado en chapado de 5 l x 200 para 1 ml | ||||||||
| 3 Los cotiledones son demasiado pequeños para el núcleo, por lo que los recuentos de colonias se normalizaron a 0,01 g de tejido en función de la masa media de un cotiledo MoneyMaker-PtoS (datos no mostrados) | ||||||||
| 4 Ajustado por ml en función del volumen chapado | ||||||||
Tabla 1: Cálculos de muestra para el ensayo de crecimiento bacteriano de plántulas. Los cálculos de muestras demuestran cómo normalizar los recuentos bacterianos y determinar el crecimiento bacteriano del tronco.
- Para las adhesiones silvestres y otras líneas con orígenes genéticos complejos, correlacionar el nivel de crecimiento bacteriano en plántulas individuales con su fenotipo como se describe en la sección 11.
11. Fenotipado para resistencia
- Retire las placas de la cámara de crecimiento y las plántulas individuales de fenotipo para la muerte (debido a la enfermedad) o la supervivencia (debido a la resistencia) después de 7-14 días.
- Plantas de fenotipo infectadas con una cepa altamente virulenta como PstDC3000 antes, a los 7-10 días después de la inoculación de inundación.
- Plantas de fenotipo infectadas con PstT1 a los 10-14 días después de la inoculación de inundación.
- Determinar un sistema de puntuación basado en el rango de fenotipos de resistencia observado. Registrar fenotipos binarios para cultivares, líneas isogénicas y adhesiones silvestres con fenotipos consistentes, fuertes a de resistencia intermedia(Figura 4A, 4B).
- Si la plántula muestra un nuevo crecimiento del meristem apical dentro del período de tiempo para el fenotipado, cuente como una supervivencia. Si la plántula tiene un meristem apical marrón y no muestra un nuevo crecimiento vegetativo verde, cuente como una muerte(Figura 3).

Figura 3: Representación esquemática de una plántula de tomate. Se representan diferentes partes de una plántula de tomate, incluyendo el hipocoal, cotiledons, epicotyl, meristem apical de brote, y hojas verdaderas. Haga clic aquí para ver una versión más grande de esta figura.
- Registrar fenotipos en un espectro de enfermedades para poblaciones, como las poblaciones de mapeo F2, con una amplia gama de fenotipos de resistencia(Figura 4C).
- Supervise cuidadosamente las plántulas para la aparición de los síntomas de la enfermedad y la muerte para identificar la ventana apropiada para el fenotipado.

Figura 4: Representación esquemática de fenotipos esperados para la resistencia a la plántula y la muerte en diversos orígenes genéticos. (A) Las plántulas de Rio Grande-PtoR y el cultivar casi isogénico Rio Grande-PtoS se muestran 7 días después de la inundación con PstDC3000 (OD600 a 0.005) + 0.015% tensioactivo. Rio Grande-PtoR muestra una resistencia constante, y Rio Grande-PtoS muestra la susceptibilidad constante a la infección con PstDC3000. Estas líneas dan lugar a fenotipos discretos y binarios. (B) Las plántulas de una adhesión silvestre, como Solanum neorickii LA1329, se muestran 10 días después de la inundación con PstT1 (OD600 a 0,0075) + 0,015% tensioactivo. Las plántulas muestran variabilidad fenotípica, pero se registraron como fenotipos binarios. La cantidad de variabilidad fenotípica y el método de fenotipado (espectro de resistencia binaria o resistencia) dependerán de la adhesión concreta probada. (C) La cartografía de las poblaciones generadas por la salida a las adhesiones silvestres a cultivares susceptibles puede mostrar un espectro más amplio de fenotipos en las poblaciones segregadas de F2. En este caso, puede ser más apropiado registrar fenotipos de plántulas en un espectro. Las plántulas altamente susceptibles de una población cartografiante pueden ser fenotipo para la muerte tan pronto como el día 7 cuando se inundan con PstT1, y por lo general muestran un meristem apical marrón, no a muy poca extensión del epicotilo, y no hay un nuevo crecimiento vegetativo verde. El meristem apical de las plántulas susceptibles puede permanecer verde o marrón muy claro durante más tiempo, y puede haber alguna extensión del epicotilo y muy poco crecimiento vegetativo, que se vuelve marrón y detenciones para el día 10. Las plántulas individuales pueden ser fenototipadas para la resistencia basada en la cantidad de crecimiento vegetativo nuevo y continuo para el día 14. Las plántulas se pueden agrupar en función de los fenotipos descritos anteriormente en diferentes categorías de resistencia, como resistencia débil, media o fuerte. Haga clic aquí para ver una versión más grande de esta figura.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Detección de inmunidad mediada por PtoRen cultivares y líneas isogénicas utilizando el ensayo de resistencia a la plántula
La Figura 5 muestra resultados representativos para los cultivares Moneymaker-PtoR y Moneymaker-PtoS 7-10 días después de la inundación con PstDC3000. Antes de la infección, las plántulas de 10 días de edad mostraron cedías completamente y ampliaron los cotiledón y las primeras hojas verdaderas emergentes. Las plántulas se inundaron con 10 mM MgCl2 + 0.015% tensioactivo como un control negativo (datos no mostrados) y PstDC3000 a una densidad óptica de 0.005 + 0.015% tensioactivo. Las plántulas fueron fenotografiadas 7-10 días después de la inundación(Figura 5). Las plántulas individuales de líneas genotípicamente homogéneas, como Moneymaker-PtoR y Moneymaker-PtoS dan fenotipos binarios y altamente consistentes en el ensayo de inundación de plántulas. Cuando Moneymaker-PtoR, que lleva el grupo de genes Pto/Prf (n 5), fue tratado con PstDC3000 a la concentración óptima de OD600 a 0,005, la resistencia debida a la inmunidad mediada por PtoRfue fuerte y fue tipificada por un nuevo crecimiento vegetativo verde en todos los individuos22. Las plántulas deDotos-PtoS (n.o 5), que no pueden reconocer a los efectores PstDC3000 AvrPto o AvrPtoB, murieron rápidamente dentro de los 7 días después de la inundación y característicamente tenían meristems apicales marrones, motas bacterianas, clorosis y sin signos de nuevo crecimiento vegetativo verde (Figura 5).

Figura 5: Caracterización fenotípica de la resistencia o síntomas de la enfermedad 7-10 días después de la infección en un cultivar. Las plántulas de tomatePtoS se cultivaron en placas de 0,5x MS durante 10 días antes de ser inundadas con P. syringae pv. Tomate DC3000 (OD600 a 0,005) + 0,015% tensioactivo. Las plántulas de losPtoR sobrevivieron (n.o 5) y las plántulas de LaPtoS (n.o 5) murieron. Se muestra el número de plántulas supervivientes para cada genotipo del número total analizado. Barra de escala de 1 cm. Haga clic aquí para ver una versión más grande de esta figura.
Examen fenotípico de adhesiones salvajes utilizando el ensayo de resistencia a las plántulas
La Figura 6 muestra resultados representativos para plántulas de adhesiones susceptibles y resistentes 10-14 días después de la inundación con PstT1. Las adhesiones susceptibles incluyen RG-PtoR, S. pimpinellifolium LA1375, y S. pimpinellifolium LA1606, y las adhesiones resistentes incluyen S. neorickii LA1329. Las plántulas de diez días de edad se inundaron con 10 mM MgCl2 + 0.015% surfactante como un control negativo, y PstT1 a una densidad óptica de 0.0075 + 0.015% surfactante. Las plántulas fueron fenotótipo al menos 10 días después de la inundación, como las plántulas infectadas por PstT1 murieron más lentamente que las plántulas infectadas por PstDC3000. Las plántulas inoculadas simuladas eran verdes, saludables y en crecimiento activo. Este control es importante para garantizar que las adhesiones no sean sensibles a la concentración de tensioactivos y para garantizar que no haya contaminación bacteriana. Las adhesiones susceptibles (Rio Grande-PtoR [n.o 7], S. pimpinellifolium LA1375 [n.o 7], y S. pimpinellifolium LA1606 [n 5]) estaban muertas, tenían meristems apicales marrones y carecían de un nuevo crecimiento de 10 a 14 días después de la inoculación con PstT1. Por el contrario, dos plántulas S. neorickii LA1329 (n.o 3) mostraron un alto nivel de crecimiento verde nuevo y sobrevivieron a la infección con PstT1(Figura 6). Tres plántulas LA1329 no germinaron. Típicamente, 5-7 individuos fueron examinados para cada adhesión en una pantalla primaria para determinar la prevalencia de resistencia en la población. Cuando una adhesión silvestre más compleja genéticamente, como LA1329, se inunda con PstT1, los fenotipos de resistencia muestran ligeramente más variabilidad entre las plántulas individuales, en comparación con Moneymaker-PtoR tratado con PstDC3000. Sin embargo, los fenotipos de resistencia eran generalmente menos variables que los observados en las poblaciones de mapeo F2. Por lo tanto, se utilizaron criterios de fenotipado binario para LA1329.

Figura 6: Caracterización fenotípica de los síntomas de la resistencia o de la enfermedad 10-14 días después de la infección en las adhesiones silvestres. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 y S. neorickii LA1329 plántulas de tomate fueron cultivadas en placas 0.5x MS durante 10 días, y luego inundadas con PstT1 (OD600 a 0.0075) + 0.015% tensioactivo. Se muestra el número de plántulas supervivientes para cada adhesión silvestre del número total de pruebas. Barra de escala de 1 cm. Haga clic aquí para ver una versión más grande de esta figura.
Evaluación cuantitativa del crecimiento bacteriano mediante el ensayo de inundación de plántulas
Para confirmar que la resistencia observada en LA1329 a PstT1 dio lugar a un menor crecimiento bacteriano, se llevaron a cabo ensayos de crecimiento bacteriano en plántulas de tomate. El nivel de crecimiento de PstT1 en Moneymaker-PtoS y S. neorickii LA1329 se determinó 4 días después de la infección. Moneymaker-PtoS es una línea casi isogénica con susceptibilidad constante entre las plántulas individuales. Las adhesiones salvajes como S. neorickii LA1329 son a menudo más complejas genéticamente. LA1329 muestra aproximadamente 60% de resistencia a PstT1 en toda la población22. Debido a que las plántulas pueden caer sus cotiledones después de la infección, se cultiva una plántula en cada plato para correlacionar el crecimiento bacteriano en el cotilerín cosechado con la supervivencia total de la plántula o la muerte como se determina fenotípicamente al menos 10 días después de la inundación. Los recuentos bacterianos del día 4 para cada plántula se normalizaron a 0,01 g de tejido y se convirtieron en crecimiento tronco (CFU/0.01 g(log10)). El crecimiento de troncos para plántulas LA1329 resistentes a fenotípicamente (LA1329RES)o plántulas fenotípicamente susceptibles (LA1329SUS)se agruparon por separado y se compararon entre sí y con el cultivar susceptible Moneymaker-PtoS. Por ejemplo, hubo una diferencia de 1,7 registros en el crecimiento bacteriano entre LA1329RES (log 6.3) y LA1329SUS (log 8.0), y una diferencia de registro de 1,6 entre LA1329RES (log 6.3) y Moneymaker-PtoS (log 7.9) (Figura 7). Por lo tanto, resistencia fenotípica correlacionada con la resistencia cuantitativa en los ensayos de plántulas.

Figura 7: Las plántulas resistentes Solanum neorickii LA1329 apoyan un crecimiento bacteriano más bajo que Moneymaker-PtoS o susceptible S. neorickii LA1329. Los recuentos bacterianos se determinaron 4 días después de la inoculación de S. neorickii LA1329 (n.o 14) y De moneymaker-PtoS (n a 10) plántulas infectadas con PstT1 y la normalización se realizó a 0,01 g de tejido. Para LA1329, los dos grupos fenotípicos, susceptibles (SUS) o resistentes (RES), se observaron y contaron por separado. Por encima de la barra * - diferencia estadísticamente significativa determinada por un análisis de un factor de varianza. Se utilizó un procedimiento de modelo lineal general (p < 0.001) seguido de una comparación múltiple de medios utilizando la prueba post hoc de Tukey. Barras de error: error estándar. La figura indica un experimento representativo. Haga clic aquí para ver una versión más grande de esta figura.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Se describe un protocolo para la inoculación por inundación con PstDC3000 o PstT1 optimizado para detectar resistencia a estas cepas bacterianas en plántulas de tomate. Existen varios parámetros críticos para obtener resultados óptimos en el ensayo de resistencia a la plántula, incluida la concentración bacteriana y la concentración de surfactantes, que se determinaron empíricamente22. Para PstDC3000, la densidad óptica fue optimizada para lograr una supervivencia completa en un cultivar resistente que contiene el racimo Pto/Prf y completar la muerte en un cultivar susceptible que carece del cúmulo22de Pto/Prf. Para una cepa como PstT1, donde no hay variedades resistentes conocidas, la densidad óptica se optimizó para ser la más baja posible para la muerte constante y completa de la planta22. 24 diseñaron un ensayo de plántulas de tomate para investigar la patogénesis de PstDC3000 y la función de virulencia de la coronatina. En este ensayo de virulencia, las infecciones se realizaron utilizando bacterias concentradas en una DO600 de 0,124,20 veces mayor que la densidad óptica de las cepas utilizadas en nuestro ensayo de resistencia. El reconocimiento de los efectores PstDC3000 AvrPto y AvrPtoB en plántulas de tomate que transportan el clúster de genes Pto/Prf da como resultado ETI y un HR22macroscópico. En el contexto de una fuerte respuesta inmune como la ETI, se utilizó un titer bacteriano inferior para PstDC3000 para evitar una resistencia genética abrumadora del grupo22del gen Pto/Prf. Además, estos resultados sugieren que una alta concentración bacteriana podría abrumar a las respuestas inmunitarias más débiles, como la PTI o la resistencia parcial cuantitativa, donde múltiples genes contribuyen al fenotipo general. Surfactante es necesario para que las bacterias se adhieran a la superficie de la hoja; sin embargo, las altas concentraciones pueden causar clorosis de la hoja22. Anteriormente probamos una gama de concentraciones de surfactantes para determinar empíricamente la concentración ideal en plántulas de tomate de 10 días de edad22. Al probar nuevas especies que pueden diferir en su sensibilidad a los tensioactivos, la concentración de surfactantes debe optimizarse para identificar una concentración que no cause daños o clorosis en ausencia de bacterias. Las condiciones adecuadas del ensayo requerirán la optimización de una concentración de surfactantes que no causa daño, y una concentración bacteriana que causa enfermedad en todos los controles susceptibles.
Los parámetros críticos adicionales para el éxito en el ensayo de inundación de plántulas incluyen el uso de plántulas en etapas específicas de desarrollo (plántulas de 10 días de edad)(Figura 1),el mantenimiento de condiciones estables de la cámara de crecimiento (intensidad de luz de aproximadamente 200 e m-2 s-1, temperatura constante de 22 oC, 16 h de luz) y la realización de experimentos en un armario de bioseguridad estéril. El volumen de los medios por encima de 45 ml o por debajo de 35 ml puede afectar a la muerte constante de los controles susceptibles, ya que el volumen puede afectar al microambiente circundante de las plántulas en la placa. Por ejemplo, las diferencias en la humedad relativa dentro de las placas selladas podrían afectar la infectividad de las bacterias y la capacidad de las plantas para sobrevivir a la infección. La técnica estéril es crítica, porque la contaminación en las placas puede confundir la fuente de muerte o susceptibilidad en las plántulas. Además, debido a que las interacciones planta-patógeno se ven afectadas por el reloj circadiano24,25,26, se recomienda que las plantas se infecten en un momento constante del día.
Pst es un patógeno foliar que coloniza preferentemente las partes aéreas de las plántulas de tomate, incluyendo los cotiledons24 (Figura 3). Por lo tanto, el fenotipado cualitativo en el ensayo de inundación de plántulas se centra en el crecimiento y los síntomas de la enfermedad en las porciones aéreas de la plántula, y el tejido para el ensayo de crecimiento bacteriano se muestrea a partir de los cotiledones para el análisis cuantitativo. Después de la inoculación por inundación, las plántulas pueden morir dentro de los 7-10 días después de la inoculación con PstDC3000 o 10-14 días después de la inoculación con PstT1, como se describe en la sección 11. La muerte por plántula es visualizada por un meristem apical marrón, una elongación epicotil arrestada y/o un crecimiento vegetativo detenido. Si se utilizan diferentes cepas bacterianas, el tiempo tendrá que ser determinado empíricamente. Además, la progresión de la enfermedad en las plantas de control debe controlarse diariamente después de las inundaciones hasta que se pueda identificar un plazo constante desde la aparición de los síntomas de la enfermedad hasta la muerte por plántulas. Dependiendo de los genotipos y tratamientos utilizados en el ensayo de inundación, los fenotipos de plántulas pueden registrarse como fenotipos binarios o en un espectro de enfermedades(Figura 4). Se puede observar un espectro más amplio de fenotipos cuando se inocular nifiquen poblaciones cartografiantes F2 de adhesiones de tomate silvestre sacadas a cultivares susceptibles(Figura 4C). Puede ser mejor que el fenotipo que segrega a las poblaciones en un espectro de enfermedades dependiendo de la rapidez con la que muere la plántula y el grado de nuevo crecimiento vegetativo y ramificación(Figura 4C). El ensayo de inundación de plántulas también se puede utilizar junto con el ensayo de crecimiento bacteriano de plántulas para evaluar cuantitativamente los niveles de crecimiento bacteriano asociados con fenotipos cualitativos en plántulas individuales(Figura 7). Reducciones muy grandes (es decir, el registro 3) en el crecimiento bacteriano o una fuerte resistencia en plántulas resistentes de una adhesión silvestre en comparación con un cultivar susceptible sugieren que la base genética subyacente de la resistencia puede deberse a la ETI22. Las reducciones más pequeñas en el crecimiento bacteriano (es decir, el registro 1.7), como se observa en las plántulas LA1329, pueden deberse a la contribución de una resistencia más débil de los loci de rasgo cuantitativo y/o PTI. Por lo tanto, el ensayo de crecimiento de la plántula puede ser una herramienta importante para caracterizar aún más la resistencia en las líneas de tomate silvestre.
Típicamente, se han realizado análisis genéticos en plantas de tomate adulto de cuatro a cinco semanas de edad para identificar la base genética de la resistencia de P. siringas en las adhesiones silvestres20,21. Las plantas de tomate adultas requieren tiempos de crecimiento mucho más largos, requieren más espacio en la cámara de crecimiento, y son plantas mucho más grandes, lo que significa que por lo general pocos individuos son examinados para cada línea. El ensayo de inundación de plántulas proporciona un enfoque potente y alternativo en la identificación de la resistencia de las sísiras de P. en las adhesiones de tomate silvestre. El cribado en la fase de plántula permite probar un gran tamaño de muestra que puede ser particularmente ventajoso en la detección de resistencia en poblaciones genéticamente complejas. La reducción de los requisitos de espacio de la cámara de crecimiento y el tiempo de crecimiento facilitan un enfoque de alto rendimiento y una rápida detección de resistencia natural en las adhesiones silvestres a patógenos emergentes. Además, la resistencia de P. syringae que se identificó en la etapa de plántula en este ensayo no se limita a la etapa de desarrollo. S. neorickii LA1329 y S. habrochaites LA1253 fueron identificados inicialmente en la etapa de plántula y también muestran resistencia a PstT1 en plantas adultas como se describió anteriormente22.
El ensayo de inundación de plántulas es un protocolo versátil que se puede modificar y optimizar para detectar la resistencia del huésped a otras cepas de P. syringae. Potencialmente podría aplicarse en el contexto de diferentes patógenos bacterianos del tomate, como las especies de Xanthomonas. Este método acelerará la búsqueda de nuevas fuentes de resistencia a enfermedades a patógenos bacterianos.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Los autores no tienen nada que revelar.
Acknowledgments
Agradecemos a Jamie Calma por probar el efecto del volumen de los medios en los resultados de enfermedades o resistencia. Agradecemos al Dr. Maél Baudin y al Dr. Karl J. Scheiber del Laboratorio Lewis por proporcionar comentarios constructivos y sugerencias sobre el manuscrito. La investigación sobre la inmunidad vegetal en el laboratorio Lewis fue apoyada por el USDA ARS 2030-21000-046-00D y 2030-21000-050-00D (JDL), y la Dirección de Ciencias Biológicas de la NSF IOS-1557661 (JDL).
Materials
| Name | Company | Catalog Number | Comments |
| 3M Tape Micropore 1/2" x 10 YD CS 240 (1.25 cm x 9.1 m) | VWR International | 56222-182 | |
| 3mm borosilicate glass beads | Friedrich & Dimmock | GB3000B | |
| Bacto peptone | BD | 211677 | |
| Bacto agar | BD | 214010 | |
| Biophotometer Plus | Eppendorf | E952000006 | |
| Biosafety cabinet, class II type A2 | |||
| BRAND Disposable Plastic Cuvettes, Polystyrene | VWR International | 47744-642 | |
| Chenille Kraft Flat Wood Toothpicks | VWR International | 500029-808 | |
| cycloheximide | Research Products International | C81040-5.0 | |
| Dibasic potassium phosphate anhydrous, ACS grade | Fisher Scientific | P288-500 | |
| Dimethylformamide | |||
| Dissecting microscope (Magnification of at least 10x) | |||
| Ethanol - 190 Proof | |||
| Falcon polystyrene 96 well microplates, flat-bottom | Fisher Scientific | 08-772-3 | |
| Glass Alcohol Burner Wick | Fisher Scientific | S41898A / No. W-125 | |
| Glass Alcohol Burners | Fisher Scientific | S41898 / No. BO125 | |
| Glycerol ACS reagent | VWR International | EMGX0185-5 | |
| Kimberly-Clark™ Kimtech Science™ Kimwipes™ Delicate Task Wipers | Fisher Scientific | 06-666-A | |
| Magnesium chloride, ACS grade | VWR International | 97061-356 | |
| Magnesium sulfate heptahydrate, ACS grade | VWR International | 97062-130 | |
| Microcentrifuge tubes, 1.5 mL | |||
| Microcentrifuge tubes, 2.2 mL | |||
| Mini Beadbeater-96, 115 volt | Bio Spec Products Inc. | 1001 | |
| Murashige & Skoog, Basal Salts | Caisson Laboratories, Inc. | MSP01-50LT | |
| Pipet-Lite XLS LTS 8-CH Pipet 20-200uL | Rainin | L8-200XLS | |
| Pipet-Lite XLS LTS 8-CH Pipet 2-20uL | Rainin | L8-20XLS | |
| Polystyrene 100mm x 25mm sterile petri dish | VWR International | 89107-632 | |
| Polystyrene 150mm x 15mm sterile petri dish | Fisher Scientific | FB08-757-14 | |
| Polystyrene 150x15mm sterile petri dish | Fisher Scientific | 08-757-148 | |
| Pure Bright Germicidal Ultra Bleach 5.7% Available Chlorine (defined as 100% bleach) | Staples | 1013131 | |
| Rifampicin | Gold Biotechnology | R-120-25 | |
| Silwet L-77 (non-ionic organosilicone surfactant co-polymer C13H34O4Si3 surfactant) | Fisher Scientific | NCO138454 | |
| Tips LTS 20 μL 960/10 GPS-L10 | Rainin | 17005091 | |
| Tips LTS 250 μL 960/10 GPS-L250 | Rainin | 17005093 | |
| VWR dissecting forceps fine tip, 4.5" | VWR International | 82027-386 |
References
- Underwood, W., Melotto, M., He, S. Y. Role of plant stomata in bacterial invasion. Cell Microbiology. 9 (7), 1621-1629 (2007).
- Zipfel, C. Early molecular events in PAMP-triggered immunity. Current Opinion in Plant Biology. 12 (4), 414-420 (2009).
- Galan, J. E., Wolf-Watz, H. Protein delivery into eukaryotic cells by type III secretion machines. Nature. 444 (7119), 567-573 (2006).
- Lewis, J. D., Desveaux, D., Guttman, D. S. The targeting of plant cellular systems by injected type III effector proteins. Seminars in Cell and Developmental Biology. 20 (9), 1055-1063 (2009).
- Schreiber, K. J., Baudin, M., Hassan, J. A., Lewis, J. D. Die another day: molecular mechanisms of effector-triggered immunity elicited by type III secreted effector proteins. Seminars in Cell and Developmental Biology. 56, 124-133 (2016).
- Heath, M. C. Hypersensitive response-related death. Plant Molecular Biology. 44 (3), 321-334 (2000).
- Boyd, L. A., Ridout, C., O'Sullivan, D. M., Leach, J. E., Leung, H. Plant-pathogen interactions: disease resistance in modern agriculture. Trends in Genetics. 29 (4), 233-240 (2013).
- Pitblado, R. E., MacNeill, B. H. Genetic basis of resistance to Pseudomonas syringae pv. tomato in field tomatoes. Canadian Journal of Plant Pathology. 5 (4), 251-255 (1983).
- Pedley, K. F., Martin, G. B. Molecular basis of Pto-mediated resistance to bacterial speck disease in tomato. Annual Reviews of Phytopathology. 41, 215-243 (2003).
- Ronald, P. C., Salmeron, J. M., Carland, F. M., Mehta, A. Y., Staskawicz, B. J. The cloned avirulence gene AvrPto induces disease resistance in tomato cultivars containing the Pto resistance gene. Journal of Bacteriology. 174 (5), 1604-1611 (1992).
- Martin, G. B., et al. Map-based cloning of a protein kinase gene conferring disease resistance in tomato. Science. 262 (5138), 1432-1436 (1993).
- Salmeron, J. M., Barker, S. J., Carland, F. M., Mehta, A. Y., Staskawicz, B. J. Tomato mutants altered in bacterial disease resistance provide evidence for a new locus controlling pathogen recognition. Plant Cell. 6 (4), 511-520 (1994).
- Salmeron, J. M., et al. Tomato Prf is a member of the leucine-rich repeat class of plant disease resistance genes and lies embedded within the Pto kinase gene cluster. Cell. 86 (1), 123-133 (1996).
- Scofield, S. R., et al. Molecular basis of gene-for-gene specificity in bacterial speck disease of tomato. Science. 274 (5295), 2063-2065 (1996).
- Kunkeaw, S., Tan, S., Coaker, G. Molecular and evolutionary analyses of Pseudomonas syringae pv. tomato race 1. Molecular Plant-Microbe Interactions. 23 (4), 415-424 (2010).
- Cai, R., et al. The plant pathogen Pseudomonas syringae pv. tomato is genetically monomorphic and under strong selection to evade tomato immunity. PLoS Pathogens. 7 (8), 1002130 (2011).
- Almeida, N. F., et al. A draft genome sequence of Pseudomonas syringae pv. tomato T1 reveals a type III effector repertoire significantly divergent from that of Pseudomonas syringae pv. tomato DC3000. Molecular Plant-Microbe Interactions. 22 (1), 52-62 (2009).
- Lin, N. C., Abramovitch, R. B., Kim, Y. J., Martin, G. B. Diverse AvrPtoB homologs from several Pseudomonas syringae pathovars elicit Pto-dependent resistance and have similar virulence activities. Applied and Environmental Microbiology. 72 (1), 702-712 (2006).
- Rose, L. E., Langley, C. H., Bernal, A. J., Michelmore, R. W. Natural variation in the Pto pathogen resistance gene within species of wild tomato (Lycopersicon). I. Functional analysis of Pto alleles. Genetics. 171 (1), 345-357 (2005).
- Thapa, S. P., Miyao, E. M., Davis, R. M., Coaker, G. Identification of QTLs controlling resistance to Pseudomonas syringae pv. tomato race 1 strains from the wild tomato Solanum habrochaites LA1777. Theoretical and Applied Genetics. 128 (4), 681-692 (2015).
- Bao, Z. L., et al. Identification of a candidate gene in Solanum habrochaites for resistance to a race 1 strain of Pseudomonas syringae pv. tomato. Plant Genome. 8 (3), 1-15 (2015).
- Hassan, J. A., Zhou, Y. J., Lewis, J. D. A rapid seedling resistance assay identifies wild tomato lines that are resistant to Pseudomonas syringae pv. tomato race 1. Molecular Plant-Microbe Interactions. 30 (9), 701-709 (2017).
- King, E. O., Ward, M. K., Raney, D. E. Two simple media for the demonstration of pyocyanin and fluorescin. Journal of Laboratory and Clinical Medicine. 44 (2), 301-307 (1954).
- Uppalapati, S. R., et al. Pathogenicity of Pseudomonas syringae pv. tomato on tomato seedlings: phenotypic and gene expression analyses of the virulence function of coronatine. Molecular Plant-Microbe Interactions. 21 (4), 383-395 (2008).
- Bhardwaj, V., Meier, S., Petersen, L. N., Ingle, R. A., Roden, L. C. Defence responses of Arabidopsis thaliana to infection by Pseudomonas syringae are regulated by the circadian clock. PLoS One. 6 (10), 26968 (2011).
- Lu, H., McClung, C. R., Zhang, C. Tick tock: circadian regulation of plant innate immunity. Annual Review of Phytopathology. 55, 287-311 (2017).
- Wang, W., et al. Timing of plant immune responses by a central circadian regulator. Nature. 470 (7332), 110-114 (2011).
Tags
Bioingeniería Número 157 Pseudomonas syringae tomate resistencia a enfermedades pantalla crecimiento bacteriano fenotiposErratum
Formal Correction: Erratum: High-Throughput Identification of Resistance to Pseudomonas syringae pv. Tomato in Tomato using Seedling Flood Assay
Posted by JoVE Editors on 10/18/2023.
Citeable Link.
An erratum was issued for: High-Throughput Identification of Resistance to Pseudomonas syringae pv. Tomato in Tomato using Seedling Flood Assay. The Introduction, Protocol, Representative Results and Discussion sections were updated.
The last paragraph of the Introduction section was updated from:
In the seedling flood assay described in this protocol, tomato seedlings are grown on Petri dishes of sterile Murashige and Skoog (MS) media for 10 days and then are flooded with an inoculum containing the bacteria of interest and a surfactant. Following flooding, seedlings can be quantitatively evaluated for disease resistance via bacterial growth assays. Additionally, seedling survival or death can act as a discrete resistance or disease phenotype 7–14 days after flooding. This approach offers a high-throughput alternative for screening large numbers of wild tomato accessions for resistance to Pst race 1 strains, such as Pst strain T1 (PstT1), and can easily be adapted to other bacterial strains of interest.
to:
In the seedling flood assay described in this protocol, tomato seedlings are grown on Petri dishes of sterile Murashige and Skoog (MS) media for 10 days and then are flooded with an inoculum containing the bacteria of interest and a surfactant. Following flooding, seedlings can be quantitatively evaluated for disease resistance via bacterial growth assays. Additionally, seedling survival or death can act as a discrete resistance or disease phenotype 7–14 days after flooding. This approach offers a high-throughput alternative for screening large numbers of wild tomato accessions for resistance to Pst race 1 strains, such as Pst strain 19 (Pst19), and can easily be adapted to other bacterial strains of interest.
Step 4.8 of the Protocol section was updated from:
- After 1 h, transfer the bottle to the biosafety cabinet and under aseptic conditions, add 1,600 µL of sterile 1 M MgSO4, and appropriate antibiotics to the media.
NOTE: For rifampicin resistant strains PstDC3000 and PstT1, use rifampicin dissolved in dimethylformamide at a final concentration of 50 µg/mL. Use cycloheximide dissolved in ethanol at a final concentration of 50 µg/mL to prevent fungal growth on the plates.
to:
- After 1 h, transfer the bottle to the biosafety cabinet and under aseptic conditions, add 1,600 µL of sterile 1 M MgSO4, and appropriate antibiotics to the media.
NOTE: For rifampicin resistant strains PstDC3000 and Pst19, use rifampicin dissolved in dimethylformamide at a final concentration of 50 µg/mL. Use cycloheximide dissolved in ethanol at a final concentration of 50 µg/mL to prevent fungal growth on the plates.
Step 5.2 of the Protocol section was updated from:
- Patch bacteria (i.e., PstT1) from a glycerol stock onto KB agar with appropriate antibiotics (section 4).
to:
- Patch bacteria (i.e., Pst19) from a glycerol stock onto KB agar with appropriate antibiotics (section 4).
Step 5.6 of the Protocol section was updated from:
- For PstT1, incubate the KB plate at 28 ˚C for 48 h prior to using bacteria in the flood experiment.
to:
- For Pst19, incubate the KB plate at 28 ˚C for 48 h prior to using bacteria in the flood experiment.
Step 6 of the Protocol section was updated from:
6. Preparation of PstT1 inoculum
to
6. Preparation of Pst19 inoculum
Step 6.2 of the Protocol section was updated from:
- Perform serial dilutions using sterile 10 mM MgCl2 solution in the biosafety cabinet. For PstT1, use a spectrophotometer to make inoculum with a starting concentration of OD600 = 0.1.
to:
- Perform serial dilutions using sterile 10 mM MgCl2 solution in the biosafety cabinet. For Pst19, use a spectrophotometer to make inoculum with a starting concentration of OD600 = 0.1.
Step 6.3 of the Protocol section was updated from:
- For PstT1, make a 1/10 dilution from the initial resuspension at OD600 = 0.1 to obtain a serial dilution at a concentration of OD600 = 0.01.
to:
- For Pst19, make a 1/10 dilution from the initial resuspension at OD600 = 0.1 to obtain a serial dilution at a concentration of OD600 = 0.01.
Step 8.3 of the Protocol section was updated from:
- Set a timer for 3 min. Measure 6 mL of final inoculum (PstT1 OD600 = 0.0075 [section 6] or PstDC3000 OD600 = 0.005 [section 7]) and transfer 6 mL of inoculum to each plate with the 10-day-old seedlings.
to:
- Set a timer for 3 min. Measure 6 mL of final inoculum (Pst19 OD600 = 0.0075 [section 6] or PstDC3000 OD600 = 0.005 [section 7]) and transfer 6 mL of inoculum to each plate with the 10-day-old seedlings.
Step 8.11 of the Protocol section was updated from:
- Phenotype after 7–10 days for PstDC3000 or 10–14 days for PstT1 (section 11). If carrying out bacterial growth assays, collect leaf tissue after 4 days (sections 9 and 10) and then phenotype (section 11). Alternatively, perform phenotypic analysis and bacterial growth assays on separate sets of plants.
to:
- Phenotype after 7–10 days for PstDC3000 or 10–14 days for Pst19 (section 11). If carrying out bacterial growth assays, collect leaf tissue after 4 days (sections 9 and 10) and then phenotype (section 11). Alternatively, perform phenotypic analysis and bacterial growth assays on separate sets of plants.
Step 10.7 of the Protocol section was updated from:
- After obtaining colony counts (Figure 2B), normalize the counts to 0.01 g of tissue for seedlings and convert to log bacterial growth (Table 1).
NOTE: The average mass of one Moneymaker-PtoS cotyledon is 0.01 g and is empirically determined22.
| Genotype1 Column A | Tissue Weight (g) Column B | # of Colonies in a spot Column C | Dilution factor for spot2 Column D | Adjusted # of Colonies3 Column E | Dilution factor for serial dilution Column F | Total # of Colonies Column G (cfu/0.01 g)4 | Average # of Colonies (cfu/0.01 g) Column H | Average Log Growth (cfu/0.01 g (log10)) Column I |
| Sample 1 | 0.004 g | 10 | 200 | calculated as: (C2 x 0.01 g) / B2 = 25 | 1000 | calculated as: (D2 x E2 x F2) = 5000000 | average for sample 1 through last sample: (ie. average G1:G3) = 7000000 | log of average ie. log(H2) = 6.85 |
| Sample 2 | 0.003 g | 15 | 200 | 50 | 1000 | 10000000 | ||
| Sample 3 | 0.002 g | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1Data shown for 3 samples | ||||||||
| 2Based on plating 5 µL x 200 for 1 mL | ||||||||
| 3Cotyledons are too small to core so colony counts were normalized to 0.01 g of tissue based on the average mass of one MoneyMaker-PtoS cotyledon (data not shown) | ||||||||
| 4Adjusted per mL based on volume plated | ||||||||
Table 1: Sample calculations for seedling bacterial growth assay. Sample calculations demonstrate how to normalize bacterial counts and determine log bacterial growth.
to:
- After obtaining colony counts (Figure 2B), normalize the counts to 0.1 g of tissue for seedlings and convert to log bacterial growth (Table 1).
NOTE: The average mass of one Moneymaker-PtoS cotyledon is 0.1 g and is empirically determined22.
| Genotype1 Column A | Tissue Weight (g) Column B | # of Colonies in a spot Column C | Dilution factor for spot2 Column D | Adjusted # of Colonies3 Column E | Dilution factor for serial dilution Column F | Total # of Colonies Column G (cfu/0.01 g)4 | Average # of Colonies (cfu/0.01 g) Column H | Average Log Growth (cfu/0.1 g (log10)) Column I |
| Sample 1 | 0.04 g | 10 | 200 | calculated as: (C2 x 0.1 g) / B2 = 25 | 1000 | calculated as: (D2 x E2 x F2) = 5000000 | average for sample 1 through last sample: (ie. average G1:G3) = 7000000 | log of average ie. log(H2) = 6.85 |
| Sample 2 | 0.03 g | 15 | 200 | 50 | 1000 | 10000000 | ||
| Sample 3 | 0.02 g | 6 | 200 | 30 | 1000 | 6000000 | ||
| 1Data shown for 3 samples | ||||||||
| 2Based on plating 5 µL x 200 for 1 mL | ||||||||
| 3Cotyledons are too small to core so colony counts were normalized to 0.1 g of tissue based on the average mass of one MoneyMaker-PtoS cotyledon (data not shown) | ||||||||
| 4Adjusted per mL based on volume plated | ||||||||
Table 1: Sample calculations for seedling bacterial growth assay. Sample calculations demonstrate how to normalize bacterial counts and determine log bacterial growth.
Step 11.3 of the Protocol section was updated from:
- Phenotype plants infected with PstT1 at 10–14 days after flood inoculation.
to:
- Phenotype plants infected with Pst19 at 10–14 days after flood inoculation.
Figure 4 in the Protocol section was updated from:
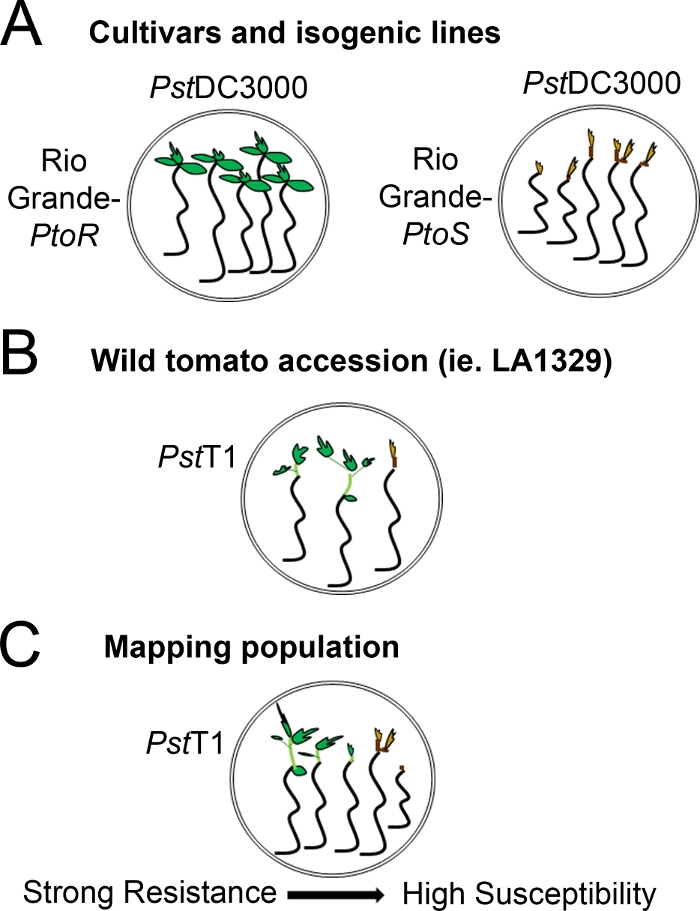
Figure 4: Schematic representation of expected phenotypes for seedling resistance and death in various genetic backgrounds. (A) Seedlings of Rio Grande-PtoR and the near-isogenic cultivar Rio Grande-PtoS are displayed 7 days after flooding with PstDC3000 (OD600 = 0.005) + 0.015% surfactant. Rio Grande-PtoR displays consistent resistance, and Rio Grande-PtoS displays consistent susceptibility to infection with PstDC3000. These lines give rise to discrete and binary phenotypes. (B) Seedlings of a wild accession, such as Solanum neorickii LA1329, are shown 10 days after flooding with PstT1 (OD600 = 0.0075) + 0.015% surfactant. Seedlings display phenotypic variability but were recorded as binary phenotypes. The amount of phenotypic variability and the method of phenotyping (binary resistance or resistance spectrum) will depend on the particular accession tested. (C) Mapping populations generated by outcrossing wild accessions to susceptible cultivars may display a wider spectrum of phenotypes in F2 segregating populations. In this case, it may be most appropriate to record seedling phenotypes on a spectrum. Highly susceptible seedlings from a mapping population may be phenotyped for death as early as day 7 when flooded with PstT1, and typically show a brown apical meristem, no to very little extension of the epicotyl, and no new, green vegetative growth. The apical meristem of susceptible seedlings may stay green or very light brown for more time, and there may be some extension of the epicotyl and very little vegetative growth, which turns brown and arrests by day 10. Individual seedlings can be phenotyped for resistance based on the amount of new and ongoing vegetative growth by day 14. Seedlings can then be grouped based on the phenotypes described above into different categories of resistance such as weak, medium, or strong resistance. Please click here to view a larger version of this figure.
to:
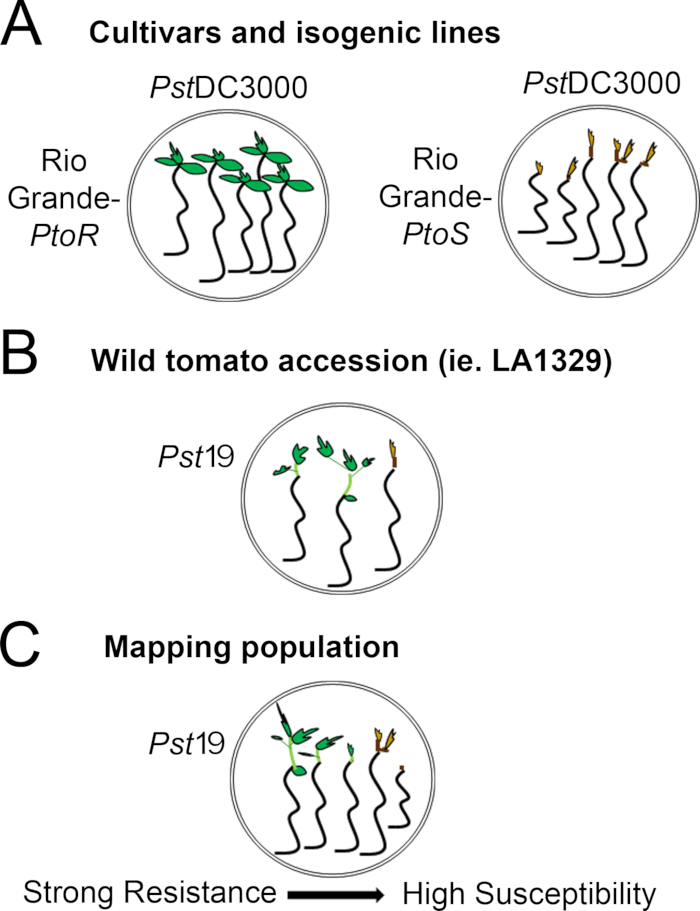
Figure 4: Schematic representation of expected phenotypes for seedling resistance and death in various genetic backgrounds. (A) Seedlings of Rio Grande-PtoR and the near-isogenic cultivar Rio Grande-PtoS are displayed 7 days after flooding with PstDC3000 (OD600 = 0.005) + 0.015% surfactant. Rio Grande-PtoR displays consistent resistance, and Rio Grande-PtoS displays consistent susceptibility to infection with PstDC3000. These lines give rise to discrete and binary phenotypes. (B) Seedlings of a wild accession, such as Solanum neorickii LA1329, are shown 10 days after flooding with Pst19 (OD600 = 0.0075) + 0.015% surfactant. Seedlings display phenotypic variability but were recorded as binary phenotypes. The amount of phenotypic variability and the method of phenotyping (binary resistance or resistance spectrum) will depend on the particular accession tested. (C) Mapping populations generated by outcrossing wild accessions to susceptible cultivars may display a wider spectrum of phenotypes in F2 segregating populations. In this case, it may be most appropriate to record seedling phenotypes on a spectrum. Highly susceptible seedlings from a mapping population may be phenotyped for death as early as day 7 when flooded with Pst19, and typically show a brown apical meristem, no to very little extension of the epicotyl, and no new, green vegetative growth. The apical meristem of susceptible seedlings may stay green or very light brown for more time, and there may be some extension of the epicotyl and very little vegetative growth, which turns brown and arrests by day 10. Individual seedlings can be phenotyped for resistance based on the amount of new and ongoing vegetative growth by day 14. Seedlings can then be grouped based on the phenotypes described above into different categories of resistance such as weak, medium, or strong resistance. Please click here to view a larger version of this figure.
The second paragraph of the Representative Results section was updated from:
Phenotypic screening of wild accessions using the seedling resistance assay
Figure 6 shows representative results for seedlings of susceptible and resistant accessions 10–14 days after flooding with PstT1. Susceptible accessions include RG-PtoR, S. pimpinellifolium LA1375, and S. pimpinellifolium LA1606, and resistant accessions include S. neorickii LA1329. Ten-day-old seedlings were flooded with 10 mM MgCl2 + 0.015% surfactant as a negative control, and PstT1 at an optical density of 0.0075 + 0.015% surfactant. The seedlings were phenotyped at least 10 days after flooding, as PstT1-infected seedlings died more slowly than PstDC3000-infected seedlings. Mock-inoculated seedlings were green, healthy, and actively growing. This control is important to ensure that the accessions are not sensitive to the concentration of surfactant, and to ensure there is no bacterial contamination. Susceptible accessions (Rio Grande-PtoR [n = 7], S. pimpinellifolium LA1375 [n = 7], and S. pimpinellifolium LA1606 [n = 5]) were dead, had brown apical meristems, and lacked new growth 10–14 days after inoculation with PstT1. In contrast, two S. neorickii LA1329 (n = 3) seedlings displayed a high level of new, green growth and survived infection with PstT1 (Figure 6). Three LA1329 seedlings did not germinate. Typically, 5–7 individuals were screened for each accession in a primary screen to determine the prevalence of resistance in the population. When a more genetically complex wild accession, such as LA1329, is flooded with PstT1, the resistance phenotypes display slightly more variability among individual seedlings, compared to Moneymaker-PtoR treated with PstDC3000. However, the resistance phenotypes were usually less variable than those seen in F2 mapping populations. Thus, binary phenotyping criteria was used for LA1329.
to:
Phenotypic screening of wild accessions using the seedling resistance assay
Figure 6 shows representative results for seedlings of susceptible and resistant accessions 10–14 days after flooding with Pst19. Susceptible accessions include RG-PtoR, S. pimpinellifolium LA1375, and S. pimpinellifolium LA1606, and resistant accessions include S. neorickii LA1329. Ten-day-old seedlings were flooded with 10 mM MgCl2 + 0.015% surfactant as a negative control, and Pst19 at an optical density of 0.0075 + 0.015% surfactant. The seedlings were phenotyped at least 10 days after flooding, as Pst19-infected seedlings died more slowly than PstDC3000-infected seedlings. Mock-inoculated seedlings were green, healthy, and actively growing. This control is important to ensure that the accessions are not sensitive to the concentration of surfactant, and to ensure there is no bacterial contamination. Susceptible accessions (Rio Grande-PtoR [n = 7], S. pimpinellifolium LA1375 [n = 7], and S. pimpinellifolium LA1606 [n = 5]) were dead, had brown apical meristems, and lacked new growth 10–14 days after inoculation with Pst19. In contrast, two S. neorickii LA1329 (n = 3) seedlings displayed a high level of new, green growth and survived infection with Pst19 (Figure 6). Three LA1329 seedlings did not germinate. Typically, 5–7 individuals were screened for each accession in a primary screen to determine the prevalence of resistance in the population. When a more genetically complex wild accession, such as LA1329, is flooded with Pst19, the resistance phenotypes display slightly more variability among individual seedlings, compared to Moneymaker-PtoR treated with PstDC3000. However, the resistance phenotypes were usually less variable than those seen in F2 mapping populations. Thus, binary phenotyping criteria was used for LA1329.
Figure 6 in the Representative Results section was updated from:
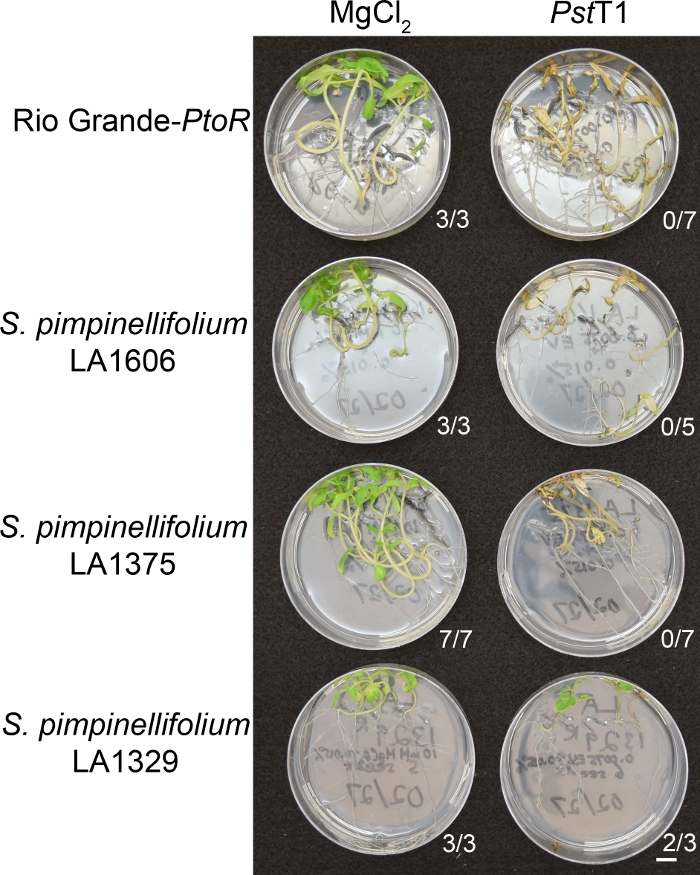
Figure 6: Phenotypic characterization of resistance or disease symptoms 10–14 days post-infection in wild accessions. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 and S. neorickii LA1329 tomato seedlings were grown on 0.5x MS plates for 10 days, and then flooded with PstT1 (OD600 = 0.0075) + 0.015% surfactant. The number of surviving seedlings for each wild accession out of the total number tested is shown. Scale bar = 1 cm. Please click here to view a larger version of this figure.
to:
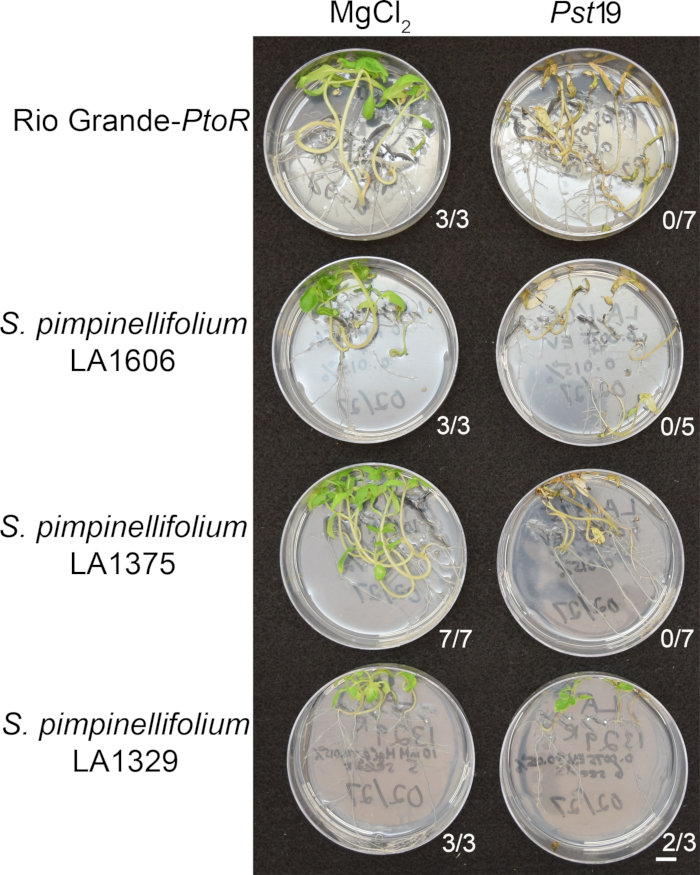
Figure 6: Phenotypic characterization of resistance or disease symptoms 10–14 days post-infection in wild accessions. Rio Grande-PtoR, S. pimpinellifolium LA1606, S. pimpinellifolium LA1375 and S. neorickii LA1329 tomato seedlings were grown on 0.5x MS plates for 10 days, and then flooded with Pst19 (OD600 = 0.0075) + 0.015% surfactant. The number of surviving seedlings for each wild accession out of the total number tested is shown. Scale bar = 1 cm. Please click here to view a larger version of this figure.
The third paragraph of the Representative Results section was updated from:
Quantitative assessment of bacterial growth using the seedling flood assay
To confirm that the observed resistance in LA1329 to PstT1 resulted in lower bacterial growth, bacterial growth assays were carried out in tomato seedlings. The level of PstT1 growth in Moneymaker-PtoS and S. neorickii LA1329 was determined 4 days post-infection. Moneymaker-PtoS is a near-isogenic line with consistent susceptibility among individual seedlings. Wild accessions such as S. neorickii LA1329 are often more genetically complex. LA1329 displays approximately 60% resistance to PstT1 across the population22. Because seedlings may drop their cotyledons after infection, one seedling was grown on each plate to correlate bacterial growth in the harvested cotyledon with overall seedling survival or death as determined phenotypically at least 10 days after flooding. The bacterial counts on day 4 for each seedling were normalized to 0.01 g of tissue and converted to log growth (CFU/0.01 g(log10)). Log growth for phenotypically resistant LA1329 seedlings (LA1329RES) or phenotypically susceptible seedlings (LA1329SUS) were separately pooled and compared to each other and the susceptible cultivar Moneymaker-PtoS. For example, there was a 1.7 log difference in bacterial growth between LA1329RES (log 6.3) and LA1329SUS (log 8.0), and a 1.6 log difference between LA1329RES (log 6.3) and Moneymaker-PtoS (log 7.9) (Figure 7). Therefore, phenotypic resistance correlated with quantitative resistance in the seedling assays.
to:
Quantitative assessment of bacterial growth using the seedling flood assay
To confirm that the observed resistance in LA1329 to Pst19 resulted in lower bacterial growth, bacterial growth assays were carried out in tomato seedlings. The level of Pst19 growth in Moneymaker-PtoS and S. neorickii LA1329 was determined 4 days post-infection. Moneymaker-PtoS is a near-isogenic line with consistent susceptibility among individual seedlings. Wild accessions such as S. neorickii LA1329 are often more genetically complex. LA1329 displays approximately 60% resistance to Pst19 across the population22. Because seedlings may drop their cotyledons after infection, one seedling was grown on each plate to correlate bacterial growth in the harvested cotyledon with overall seedling survival or death as determined phenotypically at least 10 days after flooding. The bacterial counts on day 4 for each seedling were normalized to 0.01 g of tissue and converted to log growth (CFU/0.01 g(log10)). Log growth for phenotypically resistant LA1329 seedlings (LA1329RES) or phenotypically susceptible seedlings (LA1329SUS) were separately pooled and compared to each other and the susceptible cultivar Moneymaker-PtoS. For example, there was a 1.7 log difference in bacterial growth between LA1329RES (log 6.3) and LA1329SUS (log 8.0), and a 1.6 log difference between LA1329RES (log 6.3) and Moneymaker-PtoS (log 7.9) (Figure 7). Therefore, phenotypic resistance correlated with quantitative resistance in the seedling assays.
Figure 7 in the Representative Results section was updated from:
x
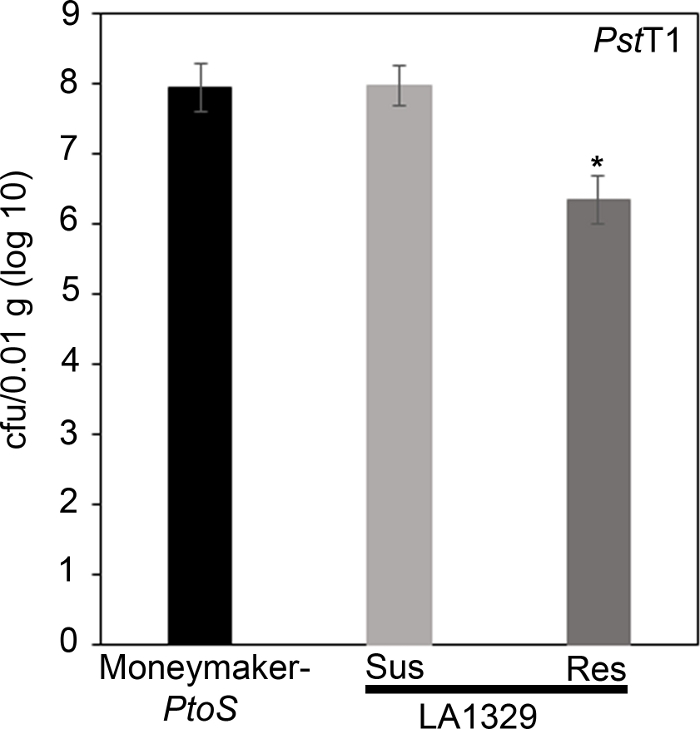
Figure 7: Resistant Solanum neorickii LA1329 seedlings support lower bacterial growth than Moneymaker-PtoS or susceptible S. neorickii LA1329. Bacterial counts were determined 4 days post-inoculation from S. neorickii LA1329 (n = 14) and Moneymaker-PtoS (n = 10) seedlings infected with PstT1 and normalization was performed to 0.01 g of tissue. For LA1329, the two phenotypic groups, susceptible (SUS) or resistant (RES), were observed and counted separately. Above the bar * = statistically significant difference determined by a one-factor analysis of variance. A general linear model procedure (p < 0.001) followed by a multiple comparison of means using Tukey's post hoc test was used. Error bars = standard error. The figure indicates one representative experiment. Please click here to view a larger version of this figure.
x
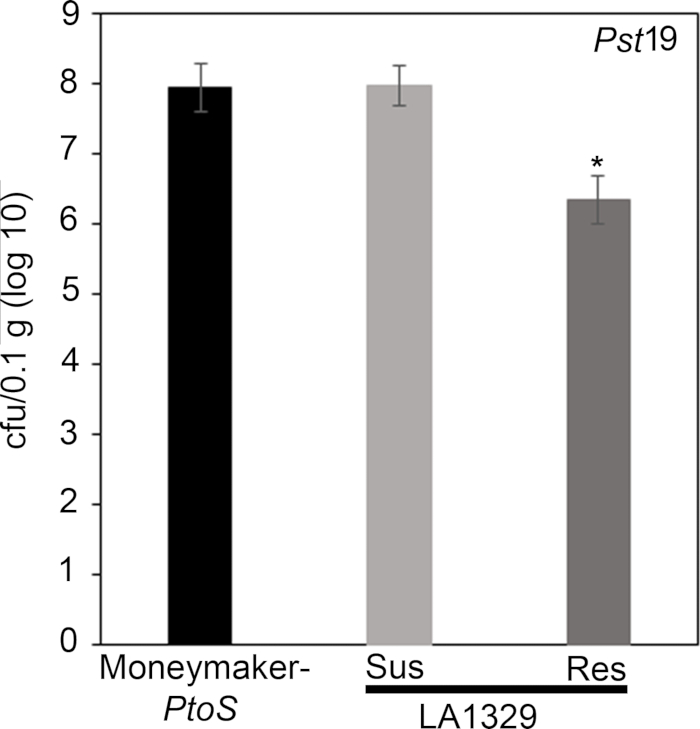
Figure 7: Resistant Solanum neorickii LA1329 seedlings support lower bacterial growth than Moneymaker-PtoS or susceptible S. neorickii LA1329. Bacterial counts were determined 4 days post-inoculation from S. neorickii LA1329 (n = 14) and Moneymaker-PtoS (n = 10) seedlings infected with Pst19 and normalization was performed to 0.1 g of tissue. For LA1329, the two phenotypic groups, susceptible (SUS) or resistant (RES), were observed and counted separately. Above the bar * = statistically significant difference determined by a one-factor analysis of variance. A general linear model procedure (p < 0.001) followed by a multiple comparison of means using Tukey's post hoc test was used. Error bars = standard error. The figure indicates one representative experiment. Please click here to view a larger version of this figure.
The first paragraph of the Discussion section was updated from:
A protocol for flood inoculation with PstDC3000 or PstT1 optimized to detect resistance to these bacterial strains in tomato seedlings is described. There are several critical parameters for optimal results in the seedling resistance assay, including bacterial concentration and surfactant concentration, which were empirically determined22. For PstDC3000, the optical density was optimized to achieve complete survival on a resistant cultivar containing the Pto/Prf cluster and complete death on a susceptible cultivar lacking the Pto/Prf cluster22. For a strain such as PstT1, where there are no known resistant varieties, the optical density was optimized to be the lowest possible for consistent and complete plant death22. Uppalapati et al.24 designed a tomato seedling assay to investigate the pathogenesis of PstDC3000 and the virulence function of coronatine. In this virulence assay, infections were performed using bacteria concentrated to an OD600 of 0.124, 20x higher than the optical density of strains used in our resistance assay. Recognition of PstDC3000 effectors AvrPto and AvrPtoB in tomato seedlings carrying the Pto/Prf gene cluster results in ETI and a macroscopic HR22. In the context of a strong immune response such as ETI, a lower bacterial titer was used for PstDC3000 to avoid overwhelming genetic resistance from the Pto/Prf gene cluster22. In addition, these results suggest that a high bacterial concentration could overwhelm weaker immune responses such as PTI or quantitative partial resistance, where multiple genes contribute to the overall phenotype. Surfactant is necessary for the bacteria to adhere to the leaf surface; however, high concentrations can cause chlorosis of the leaf22. We previously tested a range of surfactant concentrations to empirically determine the ideal concentration in 10-day-old tomato seedlings22. When testing new species that may differ in their sensitivity to surfactant, the surfactant concentration should be optimized to identify a concentration that does not cause damage or chlorosis in the absence of bacteria. Appropriate assay conditions will require optimization of a surfactant concentration that does not cause damage, and a bacterial concentration that causes disease in all susceptible controls.
to:
A protocol for flood inoculation with PstDC3000 or Pst19 optimized to detect resistance to these bacterial strains in tomato seedlings is described. There are several critical parameters for optimal results in the seedling resistance assay, including bacterial concentration and surfactant concentration, which were empirically determined22. For PstDC3000, the optical density was optimized to achieve complete survival on a resistant cultivar containing the Pto/Prf cluster and complete death on a susceptible cultivar lacking the Pto/Prf cluster22. For a strain such as Pst19, where there are no known resistant varieties, the optical density was optimized to be the lowest possible for consistent and complete plant death22. Uppalapati et al.24 designed a tomato seedling assay to investigate the pathogenesis of PstDC3000 and the virulence function of coronatine. In this virulence assay, infections were performed using bacteria concentrated to an OD600 of 0.124, 20x higher than the optical density of strains used in our resistance assay. Recognition of PstDC3000 effectors AvrPto and AvrPtoB in tomato seedlings carrying the Pto/Prf gene cluster results in ETI and a macroscopic HR22. In the context of a strong immune response such as ETI, a lower bacterial titer was used for PstDC3000 to avoid overwhelming genetic resistance from the Pto/Prf gene cluster22. In addition, these results suggest that a high bacterial concentration could overwhelm weaker immune responses such as PTI or quantitative partial resistance, where multiple genes contribute to the overall phenotype. Surfactant is necessary for the bacteria to adhere to the leaf surface; however, high concentrations can cause chlorosis of the leaf22. We previously tested a range of surfactant concentrations to empirically determine the ideal concentration in 10-day-old tomato seedlings22. When testing new species that may differ in their sensitivity to surfactant, the surfactant concentration should be optimized to identify a concentration that does not cause damage or chlorosis in the absence of bacteria. Appropriate assay conditions will require optimization of a surfactant concentration that does not cause damage, and a bacterial concentration that causes disease in all susceptible controls.
The third paragraph of the Discussion section was updated from:
Pst is a foliar pathogen that preferentially colonizes the aerial parts of tomato seedlings, including the cotyledons24 (Figure 3). Therefore, qualitative phenotyping in the seedling flood assay focuses on growth and disease symptoms in aerial portions of the seedling, and tissue for the bacterial growth assay is sampled from the cotyledons for quantitative analysis. After flood inoculation, seedlings may die within 7–10 days after inoculation with PstDC3000 or 10–14 days after inoculation with PstT1, as discussed in section 11. Seedling death is visualized by a brown apical meristem, arrested epicotyl elongation, and/or arrested vegetative growth. If different bacterial strains are used, the timing will have to be empirically determined. In addition, the progression of disease on control plants should be monitored daily after flooding until a consistent time frame from the onset of disease symptoms to seedling death can be identified. Depending on the genotypes and treatments used in the flood assay, seedling phenotypes can be recorded as binary phenotypes or on a disease spectrum (Figure 4). A broader spectrum of phenotypes may be observed when flood inoculating F2 mapping populations from wild tomato accessions crossed to susceptible cultivars (Figure 4C). It may be best to phenotype segregating populations on a disease spectrum depending on how quickly the seedling dies and the degree of new vegetative growth and branching (Figure 4C). The seedling flood assay can also be used in conjunction with the seedling bacterial growth assay to quantitatively assess levels of bacterial growth associated with qualitative phenotypes in individual seedlings (Figure 7). Very large reductions (i.e., ~log 3) in bacterial growth or strong resistance in resistant seedlings of a wild accession compared to a susceptible cultivar suggest that the underlying genetic basis of resistance may be due to ETI22. Smaller reductions in bacterial growth (i.e., ~log 1.7), as observed in LA1329 seedlings, may be due to the contribution of weaker resistance from quantitative trait loci and/or PTI. Thus, the seedling growth assay can be an important tool in further characterizing resistance in wild tomato lines.
to:
Pst is a foliar pathogen that preferentially colonizes the aerial parts of tomato seedlings, including the cotyledons24 (Figure 3). Therefore, qualitative phenotyping in the seedling flood assay focuses on growth and disease symptoms in aerial portions of the seedling, and tissue for the bacterial growth assay is sampled from the cotyledons for quantitative analysis. After flood inoculation, seedlings may die within 7–10 days after inoculation with PstDC3000 or 10–14 days after inoculation with Pst19, as discussed in section 11. Seedling death is visualized by a brown apical meristem, arrested epicotyl elongation, and/or arrested vegetative growth. If different bacterial strains are used, the timing will have to be empirically determined. In addition, the progression of disease on control plants should be monitored daily after flooding until a consistent time frame from the onset of disease symptoms to seedling death can be identified. Depending on the genotypes and treatments used in the flood assay, seedling phenotypes can be recorded as binary phenotypes or on a disease spectrum (Figure 4). A broader spectrum of phenotypes may be observed when flood inoculating F2 mapping populations from wild tomato accessions crossed to susceptible cultivars (Figure 4C). It may be best to phenotype segregating populations on a disease spectrum depending on how quickly the seedling dies and the degree of new vegetative growth and branching (Figure 4C). The seedling flood assay can also be used in conjunction with the seedling bacterial growth assay to quantitatively assess levels of bacterial growth associated with qualitative phenotypes in individual seedlings (Figure 7). Very large reductions (i.e., ~log 3) in bacterial growth or strong resistance in resistant seedlings of a wild accession compared to a susceptible cultivar suggest that the underlying genetic basis of resistance may be due to ETI22. Smaller reductions in bacterial growth (i.e., ~log 1.7), as observed in LA1329 seedlings, may be due to the contribution of weaker resistance from quantitative trait loci and/or PTI. Thus, the seedling growth assay can be an important tool in further characterizing resistance in wild tomato lines.
The fourth paragraph of the Discussion section was updated from:
Typically, genetic screens have been performed on four- to five-week-old adult tomato plants to identify the genetic basis of P. syringae resistance in wild accessions20,21. Adult tomato plants require much longer growth times, require more space in the growth chamber, and are much larger plants, which means that usually few individuals are screened for each line. The seedling flood assay provides a powerful, alternative approach in the identification of P. syringae resistance in wild tomato accessions. Screening at the seedling stage permits a large sample size to be tested which can be particularly advantageous in detecting resistance in genetically complex populations. Reduced growth chamber space requirements and growth time facilitate a high-throughput approach and rapid detection of natural resistance in wild accessions to emerging pathogens. Furthermore, P. syringae resistance that was identified at the seedling stage in this assay is not restricted to the developmental stage. S. neorickii LA1329 and S. habrochaites LA1253 were initially identified at the seedling stage and also display resistance to PstT1 in adult plants as previously described22.
to:
Typically, genetic screens have been performed on four- to five-week-old adult tomato plants to identify the genetic basis of P. syringae resistance in wild accessions20,21. Adult tomato plants require much longer growth times, require more space in the growth chamber, and are much larger plants, which means that usually few individuals are screened for each line. The seedling flood assay provides a powerful, alternative approach in the identification of P. syringae resistance in wild tomato accessions. Screening at the seedling stage permits a large sample size to be tested which can be particularly advantageous in detecting resistance in genetically complex populations. Reduced growth chamber space requirements and growth time facilitate a high-throughput approach and rapid detection of natural resistance in wild accessions to emerging pathogens. Furthermore, P. syringae resistance that was identified at the seedling stage in this assay is not restricted to the developmental stage. S. neorickii LA1329 and S. habrochaites LA1253 were initially identified at the seedling stage and also display resistance to Pst19 in adult plants as previously described22.



