ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Qui è descritto un metodo di litografia a nanosfera per la fabbricazione parallela di guide d'onda in modalità zero, che sono array di nanoperture in una microscopia in vetro rivestita di metallo coverlip per l'imaging di singole molecole a concentrazioni nano-micromolari di fluorofori. Il metodo sfrutta l'autoassemicolo del cristallo colloidale per creare un modello di guida d'onda.
Abstract
La fluorescenza a singola molecola, la fluorescenza di fondo da substrati etichettati in soluzione spesso limita la concentrazione di fluoroforo a intervalli pico- a nanomolari, diversi ordini di grandezza inferiori a molte concentrazioni fisiologiche di ligando. Le nanostrutture ottiche chiamate guide d'onda a modalità zero (ZWW), che hanno aperture di diametro di 100−200 nm fabbricate in un sottile metallo conduttore come l'alluminio o l'oro, consentono l'imaging di singole molecole a concentrazioni micromolari di fluorofori limitando l'eccitazione della luce visibile ai volumi efficaci dello zeptoliter. Tuttavia, la necessità di apparecchiature di nanofabbricazione costose e specializzate ha precluso l'uso diffuso di ZWW. Tipicamente, nanostrutture come gli ZMW sono ottenute scrivendo direttamente usando la litografia a fascio di elettroni, che è sequenziale e lenta. Qui, colloidale, o nanosfera, la litografia è usata come strategia alternativa per creare maschere su scala nanometrica per la fabbricazione di guide d'onda. La presente relazione descrive l'approccio in dettaglio, con considerazioni pratiche per ogni fase. Il metodo consente di creare migliaia di ZW in alluminio o oro in parallelo, con diametri e profondità finali della guida d'onda di 100−200 nm. Sono necessarie solo attrezzature da laboratorio comuni e un evaporatore termico per la deposizione di metalli. Rendendo gli ZMW più accessibili alla comunità biochimica, questo metodo può facilitare lo studio dei processi molecolari a concentrazioni e velocità cellulari.
Introduction
Tecniche a singola molecola come il trasferimento di energia di risonanza a fluorescenza a singola molecola (smFRET) o la spettroscopia di correlazione a fluorescenza a singola molecola (FCS) sono potenti strumenti per la biofisica molecolare, consentendo lo studio di movimenti dinamici, conformazioni e interazioni di singole biomolecole in processi come la trascrizione1,2,3,traduzione4,5,6e molti altri7. Per smFRET, la microscopia tirf (Total Internal Reflection Fluorescence) è un metodo comune perché molte molecole legate possono essere seguite nel tempo e l'onda evanescente generata dal TIR è limitata a una regione di 100−200 nm adiacente al coverslip8. Tuttavia, anche con questa limitazione del volume di eccitazione, i fluorofori di interesse devono ancora essere diluiti in intervalli di pM o nM al fine di rilevare segnali di singola molecola al di sopra della fluorecenza di fondo9. Poiché le costanti michaelis-menten degli enzimi cellulari sono tipicamente nell'intervallo da μM a mM10, le reazioni biochimiche negli studi su singole molecole sono di solito molto più lente di quelle nella cellula. Ad esempio, la sintesi proteica avviene a 15−20 aminoacidi al secondo in E. coli11,12,mentre la maggior parte dei ribosomi procariotici negli esperimenti smFRET si traducono in 0,1−1 aminoacido al secondo13. Nella sintesi proteica, le strutture cristalline e lo smFRET sui ribosomi in stallo hanno mostrato che gli RRNA di trasferimento (tRNA) oscillano tra stati "ibridi" e "classici" prima della traslocazione tRNA-mRNAfase 14,15. Tuttavia, quando erano presenti concentrazioni fisiologiche del fattore GTPasi di traslocazione, EF-G, è stata osservata una conformazione diversa, intermedia tra gli stati ibridi e classici, in smFRET6. Studiare processi molecolari dinamici a tassi e concentrazioni simili a quelli della cellula è importante, ma rimane una sfida tecnica.
Una strategia per aumentare la concentrazione del substrato fluorescente è l'uso di aperture di lunghezza d'onda sub-visibile a base di metallo, chiamate guide d'onda in modalità zero (ZMW), per generare campi di eccitazione confinati che eccitano selettivamente le biomolecole localizzate all'interno delle aperture16 (Figura 1). Le aperture hanno in genere un diametro di 100−200 nm e una profondità di 100−150 nm17. Al di sopra di una lunghezza d'onda di taglio correlata alla dimensione e alla forma dei pozzi (λc ≈ 2,3 volte il diametro per le guide d'onda circolari con acqua come mezzo dielettrico18), non sono consentiti modi di propagazione nella guida d'onda, da cui il termine guide d'onda in modalità zero. Tuttavia, un campo elettromagnetico oscillante, direttamente un'onda evanescente, in decomposizione esponenziale in intensità tunnela ancora a breve distanza nella guida d'onda18,19. Sebbene simili alle onde evanescenti TIR, le onde evanescenti ZMW hanno una costante di decadimento più breve, con conseguente regione di eccitazione effettiva di 10−30 nm all'interno della guida d'onda. A concentrazioni micromolari di ligandi etichettati fluorescentmente, solo una o poche molecole sono simultaneamente presenti all'interno della regione di eccitazione. Questa limitazione del volume di eccitazione e la conseguente riduzione della fluorescenza di fondo consentono l'imaging a fluorescenza di singole molecole a concentrazioni biologicamente rilevanti. Questo è stato applicato a molti sistemi20,tra cui misurazioni FCS di diffusione di singole proteine21,misurazioni FRET a singola molecola di interazioni ligando-proteina22 a bassa affinità e proteina-proteina23,e misurazioni spettro-elettrochimiche di singoli eventi di turnovermolecolare 24.
Gli ZWW sono stati prodotti modellando direttamente uno strato metallico utilizzando la fresatura difasci ionici 25,26 o litografia a fascio di elettroni (EBL) seguita dall'incisione al plasma16,27. Questi metodi di litografia senza maschera creano guide d'onda in serie e in genere richiedono l'accesso a strutture specializzate di nanofabbricazione, impedendo l'adozione diffusa della tecnologia ZMW. Un altro metodo, il lift-off della litografia nanoimprint ultravioletta28, utilizza uno stampo scorrevole al quarzo per premere un modello ZMW inverso su un film di resistenza come un timbro. Sebbene questo metodo sia più snello, richiede ancora EBL per la fabbricazione dello stampo al quarzo. Questo articolo presenta il protocollo per un metodo di fabbricazione basato su modelli semplice ed economico che non richiede la fresatura EBL o fasci di ioni e si basa sull'imballaggio ravvicinato di nanosfere per formare una maschera litografica.
La nanosfera o litografia "naturale", proposta per la prima volta nel 1982 da Deckman e Dunsmuir29,30, utilizza l'auto-assemblaggio di particelle colloidali monodisperse, che vanno da decine di nanometri a decine di micrometri31,per creare modelli per la creazione di superfici attraverso l'incisione e/o la deposizione di materiali. Gli array periodici estesi bidimensionali (2D) o tridimensionali (3D) di particelle colloidali, denominati cristalli colloidali, sono caratterizzati da una luminosa iridescenza dallo scattering e dalla diffrazione32. Sebbene meno ampiamente utilizzata del fascio di elettroni o della fotolitografia, questa metodologia di mascheramento è semplice, a basso costo e facilmente ridimensionabile per creare dimensioni delle funzionalità inferiori a 100 nm.
Dirigere l'auto-assemblaggio delle particelle colloidali determina il successo dell'uso dei cristalli colloidali come maschere per la creazione di superfici. Se le dimensioni e la forma delle particelle sono omogenee, le particelle colloidali possono essere facilmente auto-assemblate con imballaggio esagonale, guidate dall'esaurimentoentropico 33. L'evaporazione dell'acqua dopo il rivestimento a goccia è un percorso efficace per sedimentare le particelle colloidali, anche se altri metodi includono il dip-coating34,il rivestimento di spin35,la deposizione elettroforetica36e il consolidamento in un'interfaccia aria-acqua37. Il protocollo presentato di seguito si basa sul metodo di sedimentazione dell'evaporazione, che è stato il più semplice da implementare. Gli interstizi triangolari tra perline di polistirene imballate a distanza ravvicinata formano aperture in cui placcare un metallo sacrificale, formando pali(Figura 2 e Figura supplementare 1). Breve ricottura delle perline prima che questo passaggio adatti la forma e il diametro di questi pali. Le perline vengono rimosse, uno strato metallico finale viene depositato intorno ai pali e quindi i pali vengono rimossi. Dopo che i due passaggi di deposizione metallica sulla nanomaschera colloidale, rimozione dei post intermedi e modifica della chimica delle superfici per la passivazione e il tethering, gli array ZMW sono pronti per l'uso per l'imaging a singola molecola. Una caratterizzazione più estesa delle proprietà ottiche ZMW dopo la fabbricazione può essere trovata in un articolo38 di accompagnamento. Oltre a un evaporatore termico per la deposizione di vapore dei metalli, non sono necessari strumenti specializzati.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
NOTA: tutti i passaggi possono essere completati nello spazio generale del laboratorio.
1. Pulizia delle labbra con coperture in vetro
- Per fornire una superficie pulita per la deposizione evaporativa di particelle colloidali, posizionare 24 x 30 mm di rivestimenti in vetro borosilicato ottico (spessore 0,16−0,19 mm) all'interno degli inserti scanalati di un barattolo di colorazione in vetro coplin per la pulizia.
NOTA: Assicurarsi che i copriparsi siano in posizione verticale e siano ben separati in modo che tutte le superfici siano chiaramente esposte durante il processo di pulizia. - Versare abbastanza acetone nel barattolo di colorazione per coprire le copertine, posizionare il coperchio e sonicare per 10 minuti a 40 °C.
- Versare l'acetone e sciacquare le copertine riempiendo il barattolo di colorazione con H2O distillato e versando l'acqua. Ripeti altre 2 volte.
- Ripetere ancora una volta la sonicazione dell'acetone (passaggi 1.2 e 1.3).
- Versare abbastanza KOH da 200 mM nel barattolo per coprire le coverlips e sonicare, coperto, per 20 min a 40 °C.
NOTA: Il KOH intasa leggermente il vetro. - Risciacquare le coverlips con H distillato2O 6 volte.
- Aggiungere l'etanolo per coprire i copricapo, aggiungere il coperchio e sonicare per 10 minuti a 40 °C.
- Risciacquare le coverlips con H distillato2O 3 volte.
- Raccogliere ogni coverlip sul bordo con forcep delicate e asciugare i copricapo con gas N2. Toccare solo i bordi del coverslip. Posizionare ciascuna delle coverlip essiccate e pulite in una singola piastra di Petri pulita.
2. Deposizione evaporativa di perline di polistirolo
- Per creare la maschera di cristallo colloidale per l'array ZMW, centrifugare 50 μL di 1 μm di diametro, perline di polistirolo non funzionalizzate (2,5% w/v in acqua) a 15.000 x g,25 °C per 5 min.
NOTA: Prima di pipeggiare le perline, la soluzione stock deve essere brevemente vortice nel caso in cui le perline si siano depositate sul fondo della bottiglia. - Scartare il supernatante, lasciando meno acqua rimasta possibile.
NOTA: L'acqua residua può cambiare le proprietà di evaporazione della resopensione dell'etanolo39,quindi è accettabile la rimozione di una piccola quantità di perline per rimuovere tutta l'acqua. - Rimossare le perline dal passo 2.2 in 50 μL di 1:400 TritonX-100:etanolo solvente. Pipettare su e giù più volte per mescolare accuratamente le perline con il solvente.
NOTA: TritonX-100:etanolo solvente deve essere sigillato con pellicola di paraffina dopo l'uso e preparato fresco una volta al mese. Le perline tendono ad aderire ai lati di un recipiente di plastica, come un tubo di microcentrifugo, quindi pipetta lungo i lati per garantire che tutte le perline vengano rimescolate. - Per impostare una camera di umidità per la deposizione, posizionare 6 piastre di Petri, ognuna con un copripasto, su una panca in linea con coperchi lasciati leggermente socchiusa. In ogni piatto, spostare il coverslip nella regione aperta in modo che i copriparsi siano esposti all'ambiente quando l'umidità aumenta nel passaggio successivo.
- Posizionare un igrometro e un piccolo ventilatore elettrico centrato dietro le piastre di Petri.
- Registrare l'umidità relativa iniziale (RH) in laboratorio. Riempire un becher da 200 mL con 150−200 mL di ~75 °C di acqua e posizionarlo dietro il ventilatore.
- Accendere il ventilatore e coprire le piastre di Petri, il ventilatore, il becher e l'igrometro con un contenitore di stoccaggio in plastica rovesciato e trasparente (66 cm x 46 cm x 38 cm).
- Lasciare che l'RH nella camera sali al 70−75%, che in genere richiede 5−10 min.
NOTA: Se il laboratorio ambientale RH è basso (inferiore a ~50%), lasciare che la camera raggiunga un RH più alto, ma non superiore all'80%, per compensare la perdita di umidità durante la deposizione (vedi sotto). - Quando l'RH raggiunge il 70−75%, registrare la RH e sollevare leggermente il contenitore di plastica per posizionare rapidamente le coperture sulle piastre di Petri, il che impedisce l'inumuramento esortibile dei copripavimenti.
NOTA: La temperatura nella camera sarà leggermente più calda della temperatura ambiente, tipicamente 25−26 °C, a causa dell'umidificazione. Se l'umidità è visibile sui coprispasmi, le superfici di vetro sono troppo bagnate. Una scatola portaoggetti commerciale potrebbe semplificare questa parte del protocollo. - Lascia che l'RH nella camera continui a salire all'85%. A quel punto, registrare l'RH nella camera di umidità e pipettare 5 μL della sospensione del tallone al centro di ogni coverlip.
- Chiudere la camera e le piastre di Petri dopo ogni deposizione per ridurre al minimo la perdita di umidità. Mira a finire tutte e 6 le deposizioni entro 2 minuti.
- Registrare l'RH nella camera dopo la deposizione.
NOTA: La deposizione rh dopo la deposizione aiuterà a misurare la velocità con cui l'umidità è stata persa durante la deposizione, che dipende dalle condizioni del laboratorio ambientale. Per una tipica corsa di successo, la camera inizierà all'85% di RH prima della deposizione e terminerà al 70−75% di RH dopo la deposizione. - Lasciare che le goccioline di perline si diffondano e si asciughino per 5 minuti.
NOTA: Se i cristalli colloidali hanno molti fori o regioni multistrato, la camera era probabilmente troppo umida o asciutta, rispettivamente. Regolare l'umidità relativa alla quale chiudere le piastre di Petri e iniziare le deposizioni (vedi la sezione risultati per ulteriori discussioni sull'ottimizzazione).
3. Ricottura delle perline per ridurre le dimensioni dei pori nel modello di cristallo colloidale
- Per fornire una superficie di temperatura uniforme per la ricottura delle perline di polistirolo, che restringe gli interstizi tra perline e arrotonda gli angoli degli interstizi, posizionare una piastra di alluminio piatta e fresata sopra una piastra calda in ceramica standard.
- Impostare la temperatura della piastra calda su 107 °C, la temperatura di transizione del vetro del polistirolo40.
NOTA: Per ottenere una temperatura stabile e accurata, una sonda termocopia è stata tenuta in un foro largo 2−3 mm e profondo 4−5 mm nella piastra di alluminio. - Posizionare un coverslip contenente il modello di perline sulla piastra di alluminio caldo e ricottura per 20 s (vedere la sezione discussione per la spiegazione del tempo di fusione).
- Dopo il riscaldamento, rimuovere il coverslip dalla piastra di alluminio e posizionarlo prontamente su un'altra superficie in alluminio a temperatura ambiente per raffreddarlo.
NOTA: È utile che i copripavimenti pendino leggermente sopra il bordo della piastra o mulino canali poco profondi (vedi il video di accompagnamento) nella piastra per facilitare il ritiro dei copripavimenti.
4. Nanofabbricazione di guide d'onda in modalità zero in alluminio utilizzando il modello di cristallo colloidale
- Utilizzando la deposizione evaporativa a fascio termico o elettrone, depositare 300 nm di rame a 2 Å/s sul modello di cristallo colloidale per generare pali negli interstizi tra le perline.
- Rimuovere il metallo in eccesso sopra le perline premendo delicatamente la superficie con del nastro adesivo. Sbucciare lentamente il nastro per estrarre il metallo.
NOTA: Alcune piccole macchie di metallo in eccesso riflettente possono rimanere dopo l'estrazione del nastro, e queste possono spesso essere rimosse da un flusso di gas N2. Se dopo l'accostamento del nastro rimangono macchie sostanziali di metallo in eccesso riflettente, provare a immergere i modelli in toluene per 2 ore per sciogliere parzialmente le perline di polistirolo. Lavare le coverlips con acqua distillata, asciugare con N2e ripetere l'estratto del nastro. L'ammollo aggiuntivo non deve sciogliere completamente le perline, poiché le perline aiutano a proteggere i pali dai danni durante l'esercista del nastro. - Per sciogliere le perline di polistirolo, posizionare i modelli di perline in toluene e immergersi durante la notte.
ATTENZIONE: I fumi di toluene possono essere tossici. Lavora con il toluene sotto un cappuccio ben ventilato e indossa dispositivi di protezione individuale, tra cui guanti, occhiali di sicurezza e un camice da laboratorio. Il toluene deve essere conservato in armadi ventilati designati per liquidi infiammabili. - Dopo l'incubazione del toluene, sciacquare i modelli una volta con cloroformio e due volte con etanolo. Maneggiare accuratamente le coperture a questo punto perché i delicati pali metallici alti 200−300 nm sono ora esposti. Asciugare i modelli con N2 e rimuovere polimeri e contaminanti residui in un detergente al plasma di ossigeno per 30 minuti.
ATTENZIONE: I fumi cloroformi possono essere tossici. Lavorare con il cloroformio sotto un cappuccio ben ventilato e indossare dispositivi di protezione individuale, tra cui guanti, occhiali di sicurezza e un camice da laboratorio. Il cloroformio deve essere conservato in armadi ventilati lontano da altri solventi infiammabili. - Utilizzando la deposizione evaporativa a fascio termico o elettronico, depositare 3 nm di uno strato di adesione al titanio a 1 Å/s seguito da 100−150 nm di alluminio a 4 Å/s intorno e sopra i pali di rame.
NOTA: Si può usare rivestimenti più spessi per ottenere guide più profonde e una migliore attenuazione della fluorescenza di fondo, ma questo diminuisce anche la resa dopo aver esposto e dissolto i post nella fase successiva (vedi la sezione discussione). - Per sciogliere i pali metallici, immergere le copertine in incisione di rame (a base di acido citrico; Tavola dei materiali) per 2 ore.
ATTENZIONE: L'incisione metallica può causare ustioni alla pelle. Lavorare con le incisioni sotto una cappa ben ventilata e indossare dispositivi di protezione. Lavarsi accuratamente le mani dopo la manipolazione. L'incisione metallica deve essere conservata in armadi ventilati designati per liquidi corrosivi. - Risciacquare i copricapo con acqua distillata, asciugare con N2e lucidare delicatamente la superficie del rivestimento metallico con carta lente per esporre eventuali pali che sono ancora coperti di rivestimento. Riposizionare le coverlips in incisione di rame per altre 2 ore, quindi risciacquare di nuovo con acqua distillata e asciugare con N2.
NOTA: le diapositive ZMW devono essere conservate in piastre di Petri coperte e pulite per mantenerle libere da contaminanti.
5. Nanofabbricazione di guide d'onda in modalità zero oro utilizzando il modello di cristallo colloidale
NOTA: Il metodo per fabbricare ZW d'oro (Figura supplementare 1), che rispecchia il protocollo per fabbricare ZW in alluminio, è fornito in questa sezione.
- Utilizzando la deposizione evaporativa a fascio termico o elettronico, depositare 3 nm di uno strato di adesione al titanio a 1 Å/s seguito da 300 nm di alluminio a 4 Å/s.
- Rimuovere il metallo in eccesso sopra le perline premendo delicatamente la superficie con del nastro adesivo. Sbucciare lentamente il nastro per estrarre il metallo.
- Per sciogliere le perline di polistirolo, posizionare i modelli di perline in toluene e immergersi durante la notte.
- Dopo l'incubazione del toluene, sciacquare i modelli una volta con cloroformio e due volte con etanolo. Asciugare i modelli con N2 e rimuovere i contaminanti polimerici residui in un detergente al plasma di ossigeno per 30 minuti.
- Utilizzando la deposizione evaporativa del fascio termico o elettronico, depositare 100−150 nm di oro a 5 Å/s intorno e sopra i pali di alluminio.
- Per sciogliere i pali metallici, immergere le coperture in incisione in alluminio (a base di acido fosforico; Tavola dei materiali) per 1 h.
- Risciacquare i copricapo con acqua distillata, asciugare con N2e lucidare delicatamente la superficie del rivestimento metallico con carta lente per esporre eventuali pali che sono ancora coperti di rivestimento. Riposizionare i copricapo in incisione in alluminio per 1 h, quindi risciacquare nuovamente con acqua distillata e asciugare con N2.
NOTA: le diapositive ZMW devono essere conservate in piastre di Petri coperte e pulite.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
L'auto-assemblaggio delle particelle colloidali di polistirolo attraverso la sedimentazione evaporativa (fasi 2.1−2.13) può produrre una serie di risultati poiché richiede il controllo del tasso di evaporazione del solvente. Tuttavia, poiché le deposizioni sono veloci (10−15 minuti per round), la procedura può essere rapidamente ottimizzata per diverse condizioni di laboratorio ambientale. La figura 3A mostra un modello colloidale ben formato dopo la deposizione e l'evaporazione. Macroscopicamente, la regione delle perline è circolare, con bordi definiti da un anello opaco a più perline. Le regioni traslucide, ma non bianche nell'immagine, sono le aree monostrato desiderate. La figura 3B mostra un modello colloidale confezionato in un ambiente esente da umidità (80% RH quando le piastre di Petri erano chiuse). Questi modelli tendono a non avere un confine circolare pulito e hanno viticci multistrato che si estendono verso l'esterno. La deposizione è accettabile e può essere utilizzata nelle fasi successive, ma i fori nel reticolo riducono il numero di aree di array ZMW utilizzabili per l'imaging di singole molecole. La figura 3C mostra un modello colloidale confezionato in un ambiente esente da asciutto (65% rh quando le piastre di Petri erano chiuse). Questi modelli sono di solito di diametro inferiore rispetto ai modelli ideali e ben distribuiti. La deposizione può essere utilizzata, ma le regioni bianche multistrato, che strisciano verso l'interno, riducono l'area utilizzabile per l'imaging. Pertanto, non si consiglia di eseguire più di 6 deposizioni alla volta poiché le deposizioni verso la fine del processo si verificheranno a un'umidità inferiore man mano che la camera viene aperta e chiusa. La figura 3D mostra il modello arcobaleno prodotto dalla diffrazione della luce riflessa dal cristallo di polistirolo. Questo modello può essere utilizzato per confermare il successo e la qualità dell'imballaggio del cristallo a occhio. La figura 3E,F mostra immagini di microscopia a forza atomica (AFM) di modelli colloidali ben imballati. I difetti tra i grani derivano dall'inceppamento durante la sedimentazione evaporativa41e grani distinti possono essere visti con un obiettivo 10x. Pertanto, l'esame delle deposizioni colloidi con un microscopio a luce a bassa potenza può anche essere utilizzato per valutare l'imballaggio.
Dopo la deposizione di rame sui modelli colloidali ricotti (fase 4.1), il modello di diffrazione arcobaleno dovrebbe essere ancora visibile e migliorato dal rivestimento metallico riflettente delle cime delle perline(figura 4A,B). I modelli perdono il modello riflettente di diffrazione arcobaleno dopo l'estrapolazione del nastro scozzese (passaggio 4.2) che rimuove il rame in eccesso (Figura 4C). La figura 4D,E mostra le immagini AFM di un tipico campo di pali di rame dopo la deposizione metallica. I difetti tra i grani di cristallo colloidali nella figura 3E sono visibili nelle immagini del palo di rame come regioni più grandi di rame. L'analisi delle immagini AFM mostra che, per uno spessore di deposizione di rame di 300 nm, l'eventuale rame registra in media 255 nm(Figura 4F)in altezza e 121 nm di diametro (Figura 4G).
La deposizione del rivestimento in alluminio (fase 4.5) dove si trovavano le perline e sopra i pali di rame, e la successiva dissoluzione dei pali (passaggi 4.6 e 4.7) si traducono nelle ZMW in alluminio mostrate nella figura 5A−C. I difetti tra i grani di cristallo colloidale sono visibili come aperture più grandi (Figura 5B). La distanza media tra i centri ZMW nella figura 5C è di 559 nm, coerente con la spaziatura impostata dalla geometria esagonale di imballaggio ravvicinato delle perline da 1 μm ( Utilizzando modelli  di polistirolo che sono stati ricottura per 20 s si traduce in guide d'onda che hanno in media 118 nm di diametro (Figura 5D,E), coerenti con i diametri del palo e sufficientemente piccole per tagliare la propagazione della luce visibile. Un profilo di altezza di una guida d'onda della Figura 5D mostra anche che è profondo ~ 120 nm.
di polistirolo che sono stati ricottura per 20 s si traduce in guide d'onda che hanno in media 118 nm di diametro (Figura 5D,E), coerenti con i diametri del palo e sufficientemente piccole per tagliare la propagazione della luce visibile. Un profilo di altezza di una guida d'onda della Figura 5D mostra anche che è profondo ~ 120 nm.
Fret a singola molecola è stato eseguito negli ZWW per verificare la funzionalità (Figura 6A). Un campo tipico degli ZW per l'imaging è mostrato nella figura 6B, che contiene guide d'onda >3000 in un campo visivo di 40 x 80 μm. Gli ZW sono stati passivati per la prima volta utilizzando protocolli descrittiin precedenza 42,43. In breve, gli ZW in alluminio sono stati passivati con poli (acido vinylfosfonico) per rivestire il rivestimento in alluminio seguito da glicole polietilenglicole (PEG) terminato in metossi con PEG con terminazione biotina per rivestire i fondi di vetro degli ZWW. Gli ZW d'oro possono essere passivati con PEG derivati dal tiolo per rivestire il rivestimento in oro seguito da un trattamento PEG simile per i fondi di vetro. Le camere di flusso, ~20 μL di volume, sono state quindi costruite per l'imaging a singolamolecola 44. L'imaging FRET a singola molecola dei duplex del DNA è stato eseguito come descritto inprecedenza 38. In breve, 100 pM−1 nM di cianina-3/cianina-5 (Cy3/Cy5), duplex di DNA biotinilati (lunghezza di 33 coppie di basi) sono stati incubati per 10 minuti in canali di flusso funzionalizzati con streptavidina (incubazione di 5 minuti, soluzione di 0,5 mg/mL). La concentrazione di macromolecole etichettate può essere titolazione per ottenere ~20% di carico delle guide d'onda con una molecola, portando al <5% di guide d'onda caricate con più di una molecola, basate sul carico distribuito di Poisson (la maggior parte delle guide d'onda, ~75%, non avràmolecole) 45. Il DNA non legato è stato lavato via con tampone duplex privo di nucleasi seguito da tampone di illuminazione (0,3% [w/v] glucosio, 300 μg/mL glucosio ossidasi, 120 μg/mL catalasi e 1,5 mM Trolox [6-idrossi-2,5,7,8-tetrametil-crominano-2-acido carbossilico]). I duplex di DNA non biotinilati con etichetta Cy5 (lunghezza di 33 coppie di basi) erano presenti nel buffer di illuminazione a 0, 50, 100 e 500 nM come fluorofori di fondo in soluzione. Tracce fret a singola molecola dalle molecole di DNA duplex Cy3/Cy5 immobilizzate sono state registrate con un microscopio TIRF su misura regolato in base alle condizioni di epi-fluorescenza. I filmati sono stati registrati con un obiettivo di immersione dell'olio 100x ad apertura numerica (NA) 100x alternato con eccitazione alternata a 532 nm e 640 nm (esposizione a 100 ms) e uno splitter spettrale a doppia vista per registrare contemporaneamente l'emissione Cy3 e Cy5 su una fotocamera ad accoppiamento di carica (EMCCD) che moltiplica gli elettroni. Tracce fret a singola molecola con candeggine a passo singolo nel canale Cy5 sono state rilevabili a tutte le concentrazioni di Cy5 ambientale testato (Figura 6C−F). In confronto, singole molecole sarebbero rilevabili solo nell'illuminazione TIRF con concentrazioni di fluoroforo da pM a bassa soluzione nM46.

Figura 1: Schema delle guide d'onda in modalità zero. Diagramma della matrice ZMW con diagramma di sezione trasversale espanso di un singolo ZMW a destra. Singoli enzimi di interesse taggati fluorescentmente (ribosoma marrone con cerchio rosso per rappresentare il colorante fluorescente) chimicamente immobilizzati (tramite l'mRNA in questo esempio) sul fondo di vetro degli ZWW (tipicamente funzionalizzati con biotinylated-PEG) possono essere immagini con una tipica configurazione di microscopia a epifluorescenza a base laser. La luce di eccitazione di 532 nm (frecce verdi) si riflette al confine vetro-metallo a causa delle piccole dimensioni dell'apertura (diametro 100−200 nm), ma un'onda evanescente non propagante che decade esponenzialmente in intensità è presente all'interno dello ZMW. Ciò si traduce in una profondità di illuminazione effettiva di 10−30 nm (ombreggiatura verde in apertura). Vengono aggiunti singoli ligandi fluorescenti (tRNA blu con cerchi verdi come tag fluorescenti) a concentrazioni da nM a μM. Un singolo ligando che si diffonde nell'apertura e interagisce con l'enzima viene immaginato senza fluorescenza di fondo proibitiva. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Schema del metodo di templating colloidale sviluppato per fabbricare array ZMW in alluminio. Le perle di polistirolo, di 1 μm di diametro, vengono depositate e autoassemblate su una coverlip di vetro pulita, come descritto nella sezione 2 del protocollo. Le perline vengono quindi ricotte per ridurre le dimensioni dei pori (sezione 3), seguite dalla deposizione di rame e dalla dissoluzione delle perline nel toluene. L'alluminio viene depositato intorno e sopra i pali di rame, che vengono quindi selettivamente incisi per lasciare dietro di sé una serie esagonale di nanoperture (sezione 4). Per gli ultimi tre passaggi, le viste di sezione trasversale vengono fornite a destra delle viste del piano per mostrare le larghezze e le altezze dei pali di rame e degli ZW in alluminio. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Risultati rappresentativi della deposizione evaporativa dei colloidi. (A) Esempio di deposizione colloide ottimale. (B) Esempio di deposizione colloide accettabile in cui le condizioni erano più umide (80% RH) che ideali. I fori nel monostrato di cristallo sono evidenti. (C) Esempio di deposizione colloide accettabile in cui le condizioni erano più secche (65% DI RH) che ottimali. Le regioni monostrato sono leggermente traslucide mentre le aree multistrato sono bianche e opache (perimetro e striature verso l'interno). (D) Un cristallo colloidale illuminato con luce bianca per evidenziare la diffrazione arcobaleno dai cristalli. (E) Immagine AFM (sonda di maschiatura AFM in aria) di un monostrato di perline di polistirene imballate esagonalmente da una deposizione colloide di successo (barra di scala = 10 μm). (F) Immagine AFM espansa di perline imballate (barra di scala = 2 μm). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Immagini macroscopiche e microscopiche dei modelli ZMW dopo la deposizione di rame. (A) Immagine di diapositive dopo evaporazione fisica del rame sopra i modelli di perline. (B) Schema di diffrazione arcobaleno da modelli di perline dopo deposizione di rame. (C) Immagine di un modello (a destra) dopo un tirante del nastro per rimuovere il rame in eccesso e il nastro (a sinistra). (D) Immagine AFM dei pali di rame dopo l'estrazione del nastro e la completa dissoluzione delle perline di polistirolo (barra di scala = 5 μm). (E) Immagine AFM ad ingrandimento più elevato del pannello D (barra di scala = 2 μm). (F) Istogramma delle altezze del palo di rame (definito come misurazione dell'altezza massima all'interno di ciascun palo), n = 534. (G) Istogramma dei diametri del rame post Feret, n = 201. Il diametro di Feret è la distanza massima tra due linee parallele tangenti al limite del palo (quantificata in ImageJ47). Per identificare le particelle da esaminare, è stata utilizzata una soglia a metà strada tra la parte superiore del rivestimento e la superficie inferiore del vetro. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Immagini macroscopiche e microscopiche di ZW in alluminio. (A) Immagine di diapositive dopo deposizione fisica evaporativa di 150 nm di alluminio intorno e sopra i pali di rame. (B) Immagine AFM di ZW in alluminio dopo la post dissoluzione (barra di scala = 5 μm). (C) Immagine di ingrandimento più elevata del pannello B (barra di scala = 0,2 μm). (D) Profilo di profondità tipico di un singolo ZMW dal pannello C. Profilo tratto dalla linea verde tracciata nel pannello C. (E) Istogramma dei diametri ZMW Feret, n = 240. I diametri feret sono stati misurati come nella figura 4. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: Imaging FRET a singola molecola in ZWW. (A) Schema (non in scala) di imaging FRET a singola molecola di Cy3, duplex di DNA etichettati Cy5 in ZMW con duplex etichettati Cy5 sullo sfondo. (B) Campo di esempio degli ZW sotto illuminazione a luce bianca (barra di scala = 10 μm). (C−F) Registrazioni FRET a singola molecola di duplex di DNA immobilizzati negli ZW in presenza di duplex etichettati 0 (C),50 (D),100 (E)e 500 nM (F) Cy5 in soluzione. Per ogni concentrazione, il pannello superiore mostra l'intensità della fluorescenza Cy3 (verde) e Cy5 (rosso) sotto l'illuminazione laser di 532 nm (imaging FRET), il pannello centrale mostra l'intensità di fluorescenza Cy5 sotto l'illuminazione laser a 640 nm (eccitazione ad accettore diretto), e il pannello più basso mostra l'efficienza FRET (  ) calcolata dalle intensità di fluorescenza Cy3 (ID) e Cy5 (IA). Durante l'imaging, la lunghezza d'onda di eccitazione alternata tra 532 e 640 nm ogni 100 ms.
) calcolata dalle intensità di fluorescenza Cy3 (ID) e Cy5 (IA). Durante l'imaging, la lunghezza d'onda di eccitazione alternata tra 532 e 640 nm ogni 100 ms.
Figura supplementare 1: Schema del metodo di templating colloidale sviluppato per fabbricare array ZMW oro. Il protocollo per fabbricare l'array ZMW oro è analogo al protocollo per fabbricare l'array ZMW in alluminio (Figura 2). Invece di depositare rame sopra le perline di polistirolo, l'alluminio viene depositato. Dopo aver dissolto le perline in toluene, l'oro viene depositato al posto dell'alluminio sopra i pali. I pali in alluminio vengono quindi incisi selettivamente per lasciare dietro di sé l'array ZMW in oro. Per gli ultimi tre passaggi, le viste trasversali sono fornite a destra delle viste del piano per mostrare le larghezze e le altezze dei pali in alluminio e degli ZW d'oro. Fare clic qui per scaricare questo file.
Figura supplementare 2: Modellazione a elementi finiti della propagazione del campo elettromagnetico in ZWW. (A−D) Sezioni trasversali della grandezza del vettore di Poynting mediato nel tempo (W/m 2 )attraversouna guida d'onda fatta da un modello nonnnealed (A,C) e un modello ricotto (B,D). Le onde piane elettromagnetiche polarizzate linearmente (1 W distribuito sull'area della diapositiva) alle lunghezze d'onda elencate nella figura (400 nm o 1.000 nm) sono state proiettate sulla superficie inferiore, e la modalità più bassa (fondamentale), che ha il numero d'onda più basso, è stata calcolata utilizzando il software di modellazione(Table of Materials)con il metodo degli elementi finiti per risolvere le equazioni di Maxwell e le condizioni limite appropriate. Si presume che i confini delle guide d'onda siano conduttori elettrici perfetti, che sono ben approssimati da pareti in alluminio o oro. La sezione trasversale del modello di guida d'onda nonnnea è stata determinata dall'impacchettamento esagonale di cerchi di diametro di 1 μm, e le punte a tre punte dalla forma triangolare risultante sono state ritagliate a ~ 60 nm di larghezza per modellare un'apertura fisica realistica. La sezione trasversale dei modelli ricottura era approssimata come un cerchio di 130 nm di diametro. Entrambe le guide d'onda avevano una profondità di 130 nm, simile alla profondità del rivestimento dopo la fabbricazione. (E,F) Oltre alle lunghezze d'onda di eccitazione di 400 nm e 1.000 nm, i modelli sono stati risolti a 100 lunghezze d'onda di eccitazione uniformemente distanziate tra 400 nm e 1.000 nm, e l'indice di modalità effettiva (definito  come , dove k z è il numero d'onda nella guida d'onda, che è diminuito a causa della restrizione nel piano trasversale, e k è il numero d'onda della luce di eccitazione nel vuoto) è stato tracciato contro la lunghezza d'onda di eccitazione per le guide d'onda triangolari (E) e circolari (F). Per lunghezze d'onda più corte, le modalità più alte sono eccitate e l'indice di modalità effettiva aumenta (l'indice di modalità effettiva massima è 1, che è il caso limite in cui l'onda piana elettromagnetica viaggia illimitata nella dimensione trasversale). Le lunghezze d'onda di taglio effettive delle guide d'onda sono state stimate come la lunghezza d'onda alla quale l'indice di modalità effettiva scende a 0. Si noti che la guida circolare λcutoff = 221 nm dalla modellazione di elementi finiti (F) è coerente con la previsione teorica della lunghezza d'onda limite di una guida d'onda circolare (λcutoff,analytical = 1.7d = 221 nm, dove d è il diametro della guida d'onda). Clicca qui per scaricare questo file.
come , dove k z è il numero d'onda nella guida d'onda, che è diminuito a causa della restrizione nel piano trasversale, e k è il numero d'onda della luce di eccitazione nel vuoto) è stato tracciato contro la lunghezza d'onda di eccitazione per le guide d'onda triangolari (E) e circolari (F). Per lunghezze d'onda più corte, le modalità più alte sono eccitate e l'indice di modalità effettiva aumenta (l'indice di modalità effettiva massima è 1, che è il caso limite in cui l'onda piana elettromagnetica viaggia illimitata nella dimensione trasversale). Le lunghezze d'onda di taglio effettive delle guide d'onda sono state stimate come la lunghezza d'onda alla quale l'indice di modalità effettiva scende a 0. Si noti che la guida circolare λcutoff = 221 nm dalla modellazione di elementi finiti (F) è coerente con la previsione teorica della lunghezza d'onda limite di una guida d'onda circolare (λcutoff,analytical = 1.7d = 221 nm, dove d è il diametro della guida d'onda). Clicca qui per scaricare questo file.
Figura supplementare 3: Risultati rappresentativi della fabbricazione Au ZMW. (A) Immagine macroscopica degli array ZMW in oro. (B−D) Immagini AFM di pali di alluminio da un modello di perline che non è stato ricotto (B),un modello che è stato ricotto a 107 ° C per 20 s (C)e un modello che è stato ricotto a 107 ° C per 25 s (D). (E) Immagine AFM di ZMW d'oro dopo la dissoluzione dei pali di alluminio. (F) Immagine AFM ad ingrandimento più elevato del pannello E.(G) Profilo di profondità tipico di una ZMW oro. Profilo tratto dalla linea verde tracciata nel pannello F (barra di scala = 1 μm in B, C, De F; 5 μm in E). Clicca qui per scaricare questo file.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Per l'auto-assemblaggio colloidale (sezione del protocollo 2), l'uso di etanolo piuttosto che di acqua come solvente di sospensione accelera il processo di evaporazione in modo che i modelli siano pronti in 2−3 minuti dopo la deposizione piuttosto che 1−2 h come nei metodiprecedenti 48,49. Il protocollo di sedimentazione evaporativa qui presentato è anche più semplice dei precedenti protocolli di sedimentazione che richiedono il controllo dell'inclinazione superficiale, della temperatura e del volumed'aria al di sopra della sospensione 49,50,51. La frazione di volume delle particelle utilizzata in questo protocollo è del 2,4%, superiore allo 0,2−0,5% utilizzato nei precedenti metodi di sedimentazione48, che rimescolavano i colloidi nelle miscele acqua-glicerolo per tempi di sedimentazione molto più lunghi. Tuttavia, la qualità delle deposizioni è robusta ai cambiamenti nella frazione del volume delle particelle, con studi passati che hanno scoperto che può essere variata tra 2−10%49,50,51. Le dimensioni dei grani dei cristalli colloidali ottenuti in una deposizione riuscita da questo protocollo sono di 20−30 μm di diametro, più grandi dei grani dei precedenti metodi di sedimentazione (tipicamente diverse centinaia di nanometri didiametro) 48,49. Macroscopicamente, le aree di circa 2 cm di diametro del monostrato colloidale sono anche paragonabili alle aree di 1 cm prodotte con metodiprecedenti 49. Le grandi dimensioni dei modelli di cristallo colloidale prodotti in questo metodo consentono anche di fare 3−5 camere di flusso separate44, ognuna larga circa 3−4 mm, su ogni diapositiva ZMW. Pertanto, più esperimenti indipendenti a singola molecola possono essere eseguiti su ogni diapositiva.
Ricottura dei modelli di perline colloidali (sezione 3 del protocollo) dopo l'auto-assemblaggio è un passo semplice, ma cruciale per ridurre sufficientemente la fluorescenza di fondo con gli ZWW. Come mostra la Figura Supplementare 2, la lunghezza d'onda effettiva del taglio per una guida d'onda con la sezione triangolare da un modello nonnnealed è di 894 nm. In confronto, la lunghezza d'onda effettiva del taglio per una guida d'onda circolare di 130 nm di diametro da un modello ricotto è di 221 nm, come determinato sia analiticamente (1,7 volte il diametro dellaguida 18) che numericamente. L'uso di perline più piccole per la deposizione potrebbe anche ridurre le dimensioni dei pori modello, ma le guide d'onda sarebbero quindi distanziate più vicine a 200 nm, che si aggira intorno al limite di diffrazione della luce visibile. Inoltre, le guide d'onda resteranno triangolari nella sezione trasversale, il che porta alla propagazione della potenza nonsimmetrica attraverso la guida d'onda (Figura supplementare 2A−D). Uno svantaggio della fase di ricottura è che la variabilità nel tempo di fusione può introdurre fluttuazioni nel diametro della guida d'onda, quindi tempi precisi aiutano a ridurre al minimo la variazione tra i lotti. Gli interstizi iniziano a chiudersi a tempi di ricottura superiori a 25 s, e i diametri post non diminuiscono molto tra 20−25 s(Figura supplementare 3B−D). Un rapido test per la chiusura dell'interstizio è quello di verificare se i modelli ricottura producono ancora un motivo di diffrazione arcobaleno quando illuminati con luce e visti ad angolo. In caso meno, la maggior parte degli interstizi probabilmente si è chiusa. La relazione tra il tempo di ricottura e i tipici diametri dei pori è stata presentata all'iniziodel 38.
Dopo aver raggiunto la dimensione dei pori desiderata durante la fase di ricottura, il rame viene depositato (passaggio 4.1) sui modelli per creare un'ombra della maschera. È importante utilizzare la deposizione della linea di vista, con il metallo che si avvicina al modello il più perpendicolarmente possibile. Pertanto, aumentare la distanza tra il campione e la fonte metallica e garantire che la piastra che tiene i modelli non gira, come avviene automaticamente in alcune macchine per la deposizione di vapore, aiietterà a ridurre al minimo la deposizione laterale di metallo sul substrato di polistirolo. Tuttavia, una certa deposizione laterale è inevitabile, il che riduce le dimensioni del foro interstiziale e quindi la sezione post-sezione come più metallo viene depositato52. Ciò si traduce in pali metallici piramidali piuttosto che in struttureprismatiche 52.
Poiché i pali di rame sono probabilmente piramidali piuttosto che a forma di prisma, la deposizione di alluminio (fase 4.5) sopra i pali copre anche alcuni dei lati inclinati, bloccando l'accessibilità dell'incisione di rame per alcuni dei pali. Pertanto, il gradino di lucidatura della carta dell'obiettivo (fase 4.7 o 5.7) è stato aggiunto dopo il primo ammollo in inciso per interrompere meccanicamente eventuali pali di rame ancora ricoperti di alluminio. Depositare più rame per creare pali più alti rende anche i pali più suscettibili alle interruzioni meccaniche durante la lucidatura della carta dell'obiettivo. Tuttavia, più di 500 nm di rame non dovrebbero essere depositati poiché l'obiettivo della deposizione è quello di proiettare il foro interstiziale alla linea mediana di 500 nm delle perline da 1 μm.
Un'altra potenziale difficoltà è la rimozione involontaria del rivestimento in alluminio durante la lucidatura della carta dell'obiettivo (fase 4.7 o 5.7). Si è scoperto che la perdita di rivestimento in alluminio durante la lucidatura è diventata più frequente dopo l'aggiunta della fase di ricottura, probabilmente a causa dell'aumento dei residui di polistirolo, che possono interferire con l'aderenza dell'alluminio al vetro (sezione 3 del protocollo). Tuttavia, l'immersione notturna del toluene (passaggio 4.3 o 5.3) dopo che il tirante del nastro ha risolto questo problema. Nell'immagine AFM nella Figura 4E, alcuni anelli residui di polistirolo possono essere visti tra i pali, ma il rivestimento in alluminio resistette ancora a più buff nel passaggio 4.7. Se la perdita del rivestimento in alluminio rimane un problema dopo l'immersione notturna del toluene, è possibile aggiungere un lavaggio RCA-1 (clean-1 standard), un lavaggio piranha o un'ulteriore pulizia del plasma di ossigeno al passaggio 4.4 o 5.4. Questi passaggi di lavaggio possono essere aggiunti anche dopo la fase finale dell'incisione (passaggio 4.7 o 5.7) e prima della passivazione al fine di pulire ulteriormente gli ZWW.
Le prestazioni degli ZWW negli esperimenti FRET a singola molecola erano simili a quella degli ZW fabbricati con EBL. In uno studioprecedente 53 che eseguiva fret a singola molecola su DNA a singolo filamento etichettato Cy3 con proteina caricatore di erlicase di DNA etichettata Cy5 in soluzione (la stessa disposizione donatore-accettore di quella nella figura 6A),gli eventi FRET erano chiaramente percepibili su sfondo Cy5 da 100 nM, erano meno chiari (segnale di traccia accettore inferiore al rumore) a 1 μM e non percepibili a 10 μM. Notiamo che uno studio precedente con ZMW commerciali ha riportato segnali di accettore FRET a singola molecola a concentrazioni di fondo fino a 1 mM54, superiore a quelli che abbiamo raggiunto e altri studi precedenti42,53 con ZMW fabbricati internamente. Un'ulteriore discussione sulle prestazioni da segnale a sfondo tra gli ZMW è fornita in Jamiolkowski etal. L'interazione non specifica del campione fluorescente con le superfici ZMW53 è una sfida comune che limita l'accesso a concentrazioni più elevate, specialmente se la specie fluorescente diffusiva in soluzione è una grande macromolecola. Gli studi con ZMW su sistemi biochimici complessi come la traduzione hanno tipicamente limitato le concentrazioni di substrato fluorescente libero a 100−250 nM55,56,57,58. Indipendentemente dall'applicazione prevista degli ZWW, l'ottimizzazione dei metodi di passivazione per diversi sistemi sarà probabilmente necessaria per mantenere un segnale accettabile al rumore ad alte concentrazioni.
Nel complesso, il metodo qui presentato non richiede competenze o attrezzature specializzate, consente la fabbricazione parallela di molti modelli contemporaneamente e può essere adattato per fabbricare ZMW in metalli diversi. In questo lavoro, rame e alluminio sono stati sostituiti rispettivamente con alluminio e oro per fabbricare ZMW d'oro(Figura supplementare 3). Questo è vantaggioso per i laboratori che utilizzano metodi di passivazione dell'oro piuttosto che dell'alluminio. Inoltre, gli ZWW in oro hanno dimostrato di migliorare le emissioni per i fluorofori che assorbono nella regione rossa dello spettro visibile, mentre gli ZWW in alluminio migliorano le emissioni per i fluorofori che assorbono nella regione verde59. In futuro, l'intensità del segnale fluorescente da ZW fabbricati con questo metodo potrebbe essere migliorata incisa nel vetro sotto il rivestimento metallico ZMW utilizzando HF16,26,60. Questo porta le biomolecole immobilizzate più lontano dalle pareti metalliche, che possono dissetare i fluorofori61. Inoltre, c'è un'intensità massima di illuminazione di eccitazione sotto l'ingresso dell'apertura, e questo è stato sfruttato in precedenza per migliorare l'emissione disingole molecole 26,60.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Gli autori non hanno nulla da rivelare.
Acknowledgments
Questo lavoro è stato supportato dalle sovvenzioni NIH R01GM080376, R35GM118139 e NSF Center for Engineering MechanoBiology CMMI: da 15-48571 a Y.E.G., e da una borsa di studio NRSA pre-dottorato NIAID da F30AI114187 a R.M.J.
Materials
| Name | Company | Catalog Number | Comments |
| 1. Glass Coverslip Cleaning | |||
| Acetone | Sigma | 32201 | 1 L |
| Coplin glass staining jar | Fisher Scientific | 08-817 | Staining jar with 8 grooves and molded glass cover |
| Coverslips | VWR | 48404-467 | 24 mm x 30 mm (No.1½, Rectangular) |
| Ethanol | Sigma | E7023 | 1 L |
| KOH | Sigma | 30603 | Potassium hydroxide |
| Petri dishes | Fisher Scientific | R80115TS | 100 mm diameter, 15 mm deep |
| Sonicator | Branson | Z245143 | Tabletop ultrasonic cleaner, 5510 |
| 2. Evaporative Deposition of Polystyrene Beads | |||
| Clear storage container | Fisher Scientific | 50-110-8222 | 26 x 18 x 15 in. |
| Desk fan | O2Cool | FD05001A | Any small desk (~5 in.) fan will work |
| Glass beaker | Fisher Scientific | 02-555-25B | 250 mL |
| Humidity meter | Fisher Scientific | 11-661-19 | |
| Microcentrifuge tubes | Fisher Scientific | 21-402-903 | 1.5 mL |
| Polystyrene microspheres | Polysciences | 18602-15 | 1.00 µm diameter, non-functionalized |
| Triton X-100 deturgent | Sigma | X100 | 100 mL |
| 3. Bead Annealing for Reducing Pore Size in the Colloidal Crystal Template | |||
| Aluminum plate | Fisher Scientific | AA11062RY | Customized in-house to 14 cm x 14 cm |
| Ceramic hotplate | Fisher Scientific | HP88857100 | 13 x 8.2 x 3.8 in. |
| Temperature controller | McMaster-Carr | 38615K71 | Read temperature with thermocouple probe |
| Thermocouple probe | McMaster-Carr | 9251T93 | Type K, surface probe |
| 4/5. Nanofabrication of Zero Mode Waveguides Using the Colloidal Crystal Template | |||
| Aluminum etchant | Transene | Type A | |
| Aluminum pellets | Kurt J. Lesker | EVMAL40QXHB | For electron beam evaporation |
| Chloroform | Sigma | 288306 | 1 L |
| Copper etchant | Transene | 49-1 | |
| Copper pellets | Kurt J. Lesker | EVMCU40QXQA | For electron beam evaporation |
| Gold pellets | Kurt J. Lesker | EVMAUXX40G | For electron beam evaporation |
| Lens paper | Thorlabs | MC-5 | |
| Plasma cleaner | Harrick Plasma | PDC-32G | |
| Scotch tape | Staples | MMM119 | |
| Thin film deposition system | Kurt J. Lesker | PVD-75 | Tabletop thermal evaporation system will also work |
| Titanium pellets | Kurt J. Lesker | EVMTI45QXQA | For electron beam evaporation |
| Toluene | Sigma | 244511 | 1 L |
| Representative Results | |||
| COMSOL Multiphysics Modeling Software | COMSOL, Inc. | ||
| Dual View spectral splitter | Photometrics, Inc. |
References
- Kapanidis, A. N., et al. Initial transcription by RNA polymerase proceeds through a DNA-scrunching mechanism. Science. 314 (5802), 1144-1147 (2006).
- Santoso, Y., et al. Conformational transitions in DNA polymerase I revealed by single-molecule FRET. Proceedings of the National Academy of Sciences of the United States of America. 107 (2), 715-720 (2010).
- Herbert, K. M., Greenleaf, W. J., Block, S. M. Single-molecule studies of RNA polymerase: motoring along. Annual Review of Biochemistry. 77, 149-176 (2008).
- Chen, C., et al. Dynamics of translation by single ribosomes through mRNA secondary structures. Nature Structural & Molecular Biology. 20 (5), 582-588 (2013).
- Chen, C., et al. Single-molecule fluorescence measurements of ribosomal translocation dynamics. Molecular Cell. 42 (3), 367-377 (2011).
- Jamiolkowski, R. M., Chen, C., Cooperman, B. S., Goldman, Y. E. tRNA Fluctuations Observed on Stalled Ribosomes Are Suppressed during Ongoing Protein Synthesis. Biophysical Journal. 113 (11), 2326-2335 (2017).
- Myong, S., Stevens, B. C., Ha, T. Bridging Conformational Dynamics and Function Using Single-Molecule Spectroscopy. Structure. 14 (4), 633-643 (2006).
- Martin-Fernandez, M. L., Tynan, C. J., Webb, S. E. A 'pocket guide' to total internal reflection fluorescence. Journal of Microscopy. 252 (1), 16-22 (2013).
- Holzmeister, P., Acuna, G. P., Grohmann, D., Tinnefeld, P. Breaking the concentration limit of optical single-molecule detection. Chemical Society Reviews. 43 (4), 1014-1028 (2014).
- Scheer, M., et al. BRENDA, the enzyme information system in 2011. Nucleic Acids Research. 39, 670-676 (2011).
- Kudva, R., et al. Protein translocation across the inner membrane of Gram-negative bacteria: the Sec and Tat dependent protein transport pathways. Research in Microbiology. 164 (6), 505-534 (2013).
- Talkad, V., Schneider, E., Kennell, D. Evidence for variable rates of ribosome movement in Escherichia coli. Journal of Molecular Biology. 104 (1), 299-303 (1976).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Dunkle, J. A., et al. Structures of the bacterial ribosome in classical and hybrid states of tRNA binding. Science. 332 (6032), 981-984 (2011).
- Kim, H. D., Puglisi, J. D., Chu, S. Fluctuations of transfer RNAs between classical and hybrid states. Biophysical Journal. 93 (10), 3575-3582 (2007).
- Levene, M. J., et al. Zero-mode waveguides for single-molecule analysis at high concentrations. Science. 299 (5607), 682-686 (2003).
- Zhu, P., Craighead, H. G. Zero-mode waveguides for single-molecule analysis. Annual Review of Biophysics. 41, 269-293 (2012).
- Pollack, G. L., Stump, D. R. Electromagnetism. , Addison-Wesley. Boston, MA. (2002).
- Jackson, J. D. Classical electrodynamics. Third edition. , Wiley. New York, NY. (1999).
- Crouch, G. M., Han, D., Bohn, P. W. Zero-mode waveguide nanophotonic structures for single molecule characterization. Journal of Physics D: Applied Physics. 51 (19), 193001 (2018).
- Wenger, J., et al. Dual-color fluorescence cross-correlation spectroscopy in a single nanoaperture: towards rapid multicomponent screening at high concentrations. Optics Express. 14 (25), 12206-12216 (2006).
- Goldschen-Ohm, M. P., et al. Structure and dynamics underlying elementary ligand binding events in human pacemaking channels. eLife. 5, 20797 (2016).
- Miyake, T., et al. Real-Time Imaging of Single-Molecule Fluorescence with a Zero-Mode Waveguide for the Analysis of Protein-Protein Interaction. Analytical Chemistry. 80 (15), 6018-6022 (2008).
- Zhao, J., Branagan, S. P., Bohn, P. W. Single-Molecule Enzyme Dynamics of Monomeric Sarcosine Oxidase in a Gold-Based Zero-Mode Waveguide. Applied Spectroscopy. 66 (2), 163-169 (2012).
- Fore, S., Yuen, Y., Hesselink, L., Huser, T. Pulsed-interleaved excitation FRET measurements on single duplex DNA molecules inside C-shaped nanoapertures. Nano Letters. 7 (6), 1749-1756 (2007).
- Rigneault, H., et al. Enhancement of single-molecule fluorescence detection in subwavelength apertures. Physical Review Letters. 95 (11), 117401 (2005).
- Foquet, M., et al. Improved fabrication of zero-mode waveguides for single-molecule detection. Journal of Applied Physics. 103 (3), 034301 (2008).
- Wada, J., et al. Fabrication of Zero-Mode Waveguide by Ultraviolet Nanoimprint Lithography Lift-Off Process. Japanese Journal of Applied Physics. 50 (6), 06 (2011).
- Fischer, U. C., Zingsheim, H. P. Submicroscopic pattern replication with visible light. Journal of Vacuum Science and Technology. 19 (4), 881-885 (1981).
- Deckman, H. W., Dunsmuir, J. H. Natural lithography. Applied Physics Letters. 41 (4), 377-379 (1982).
- Li, B., Zhou, D., Han, Y. Assembly and phase transitions of colloidal crystals. Nature Reviews Materials. 1 (2), 15011 (2016).
- Bohn, J. J., Tikhonov, A., Asher, S. A. Colloidal crystal growth monitored by Bragg diffraction interference fringes. Journal of Colloid and Interface Science. 350 (2), 381-386 (2010).
- Dimitrov, A. S., Nagayama, K. Continuous Convective Assembling of Fine Particles into Two-Dimensional Arrays on Solid Surfaces. Langmuir. 12 (5), 1303-1311 (1996).
- Pisco, M., et al. Nanosphere lithography for optical fiber tip nanoprobes. Light: Science & Applications. 6 (5), 16229 (2017).
- Chandramohan, A., et al. Model for large-area monolayer coverage of polystyrene nanospheres by spin coating. Scientific Reports. 7, 40888 (2017).
- Besra, L., Liu, M. A review on fundamentals and applications of electrophoretic deposition (EPD). Progress in Materials Science. 52 (1), 1-61 (2007).
- Yu, J., et al. Preparation of High-Quality Colloidal Mask for Nanosphere Lithography by a Combination of Air/Water Interface Self-Assembly and Solvent Vapor Annealing. Langmuir. 28 (34), 12681-12689 (2012).
- Jamiolkowski, R. M., et al. Nanoaperture fabrication via colloidal lithography for single molecule fluorescence analysis. PLoS ONE. 14 (10), 0222964 (2019).
- Innocenzi, P., et al. Evaporation of Ethanol and Ethanol-Water Mixtures Studied by Time-Resolved Infrared Spectroscopy. The Journal of Physical Chemistry A. 112 (29), 6512-6516 (2008).
- Rieger, J. The glass transition temperature of polystyrene. Journal of Thermal Analysis. 46 (3), 965-972 (1996).
- Donev, A., Torquato, S., Stillinger, F. H., Connelly, R. Jamming in hard sphere and disk packings. Journal of Applied Physics. 95 (3), 989-999 (2004).
- Kinz-Thompson, C. D., et al. Robustly Passivated, Gold Nanoaperture Arrays for Single-Molecule Fluorescence Microscopy. ACS Nano. 7 (9), 8158-8166 (2013).
- Korlach, J., et al. Selective aluminum passivation for targeted immobilization of single DNA polymerase molecules in zero-mode waveguide nanostructures. Proceedings of the National Academy of Sciences of the United States of America. 105 (4), 1176-1181 (2008).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. Journal of Visualized Experiments. (86), e50549 (2014).
- Plénat, T., Yoshizawa, S., Fourmy, D. DNA-Guided Delivery of Single Molecules into Zero-Mode Waveguides. ACS Applied Materials & Interfaces. 9 (36), 30561-30566 (2017).
- Kudalkar, E. M., Davis, T. N., Asbury, C. L. Single-Molecule Total Internal Reflection Fluorescence Microscopy. Cold Spring Harbor protocols. 2016 (5), 077800 (2016).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Hoogenboom, J. P., Derks, D., Vergeer, P., Blaaderen, A. v Stacking faults in colloidal crystals grown by sedimentation. The Journal of Chemical Physics. 117 (24), 11320-11328 (2002).
- Micheletto, R., Fukuda, H., Ohtsu, M. A Simple Method for the Production of a Two-Dimensional, Ordered Array of Small Latex Particles. Langmuir. 11 (9), 3333-3336 (1995).
- Denkov, N., et al. Mechanism of formation of two-dimensional crystals from latex particles on substrates. Langmuir. 8 (12), 3183-3190 (1992).
- Okubo, T. Convectional, sedimentation and drying dissipative patterns of colloidal crystals of poly(methyl methacrylate) spheres on a watch glass. Colloid and Polymer Science. 286 (11), 1307-1315 (2008).
- Ye, S., Routzahn, A. L., Carroll, R. L. Fabrication of 3D Metal Dot Arrays by Geometrically Structured Dynamic Shadowing Lithography. Langmuir. 27 (22), 13806-13812 (2011).
- Zhao, Y., et al. Dark-Field Illumination on Zero-Mode Waveguide/Microfluidic Hybrid Chip Reveals T4 Replisomal Protein Interactions. Nano Letters. 14 (4), 1952-1960 (2014).
- Goldschen-Ohm, M. P., White, D. S., Klenchin, V. A., Chanda, B., Goldsmith, R. H. Observing Single-Molecule Dynamics at Millimolar Concentrations. Angewandte Chemie International Edition. 56 (9), 2399-2402 (2017).
- Noriega, T. R., Chen, J., Walter, P., Puglisi, J. D. Real-time observation of signal recognition particle binding to actively translating ribosomes. eLife. 3, 04418 (2014).
- Uemura, S., et al. Real-time tRNA transit on single translating ribosomes at codon resolution. Nature. 464 (7291), 1012-1017 (2010).
- Choi, J., Puglisi, J. D. Three tRNAs on the ribosome slow translation elongation. Proceedings of the National Academy of Sciences of the United States of America. 114 (52), 13691-13696 (2017).
- Eid, J., et al. Real-Time DNA Sequencing from Single Polymerase Molecules. Science. 323 (5910), 133-138 (2009).
- Martin, W. E., Srijanto, B. R., Collier, C. P., Vosch, T., Richards, C. I. A Comparison of Single-Molecule Emission in Aluminum and Gold Zero-Mode Waveguides. The Journal of Physical Chemistry A. 120 (34), 6719-6727 (2016).
- Wenger, J., et al. Single molecule fluorescence in rectangular nano-apertures. Optics Express. 13 (18), 7035-7044 (2005).
- Pineda, A. C., Ronis, D. Fluorescence quenching in molecules near rough metal surfaces. The Journal of Chemical Physics. 83 (10), 5330-5337 (1985).
Tags
Bioingegneria Numero 159 guide d'onda in modalità zero nano-apertura fluorescenza a singola molecola litografia nanosfera cristallo colloidale auto-assemblaggioErratum
Formal Correction: Erratum: Fabrication of Zero Mode Waveguides for High Concentration Single Molecule Microscopy
Posted by JoVE Editors on 08/10/2021.
Citeable Link.
An erratum was issued for: Fabrication of Zero Mode Waveguides for High Concentration Single Molecule Microscopy. A figure was updated.
Figure 3 was updated from:
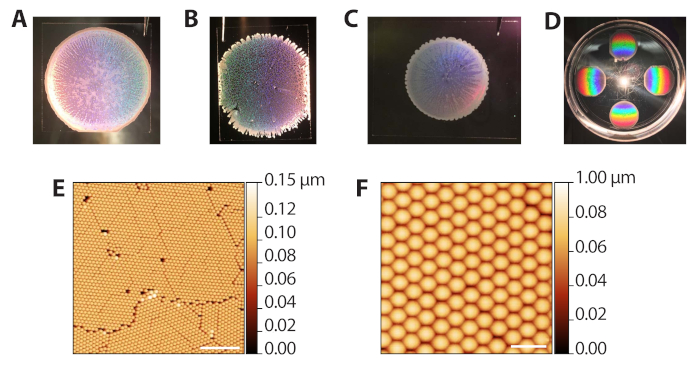
Figure 3: Representative results from evaporative deposition of colloids. (A) Example of optimal colloid deposition. (B) Example of an acceptable colloid deposition in which conditions were more humid (80% RH) than ideal. Holes in the crystal monolayer are apparent. (C) Example of an acceptable colloid deposition in which conditions were drier (65% RH) than optimal. The monolayer regions are slightly translucent while multilayered areas are white and opaque (perimeter and streaks inward). (D) A colloidal crystal illuminated with white light to highlight the rainbow diffraction from the crystals. (E) AFM image (tapping probe AFM in air) of a monolayer of hexagonally packed polystyrene beads from a successful colloid deposition (scale bar = 10 µm). (F) Expanded AFM image of packed beads (scale bar = 2 µm). Please click here to view a larger version of this figure.
to:
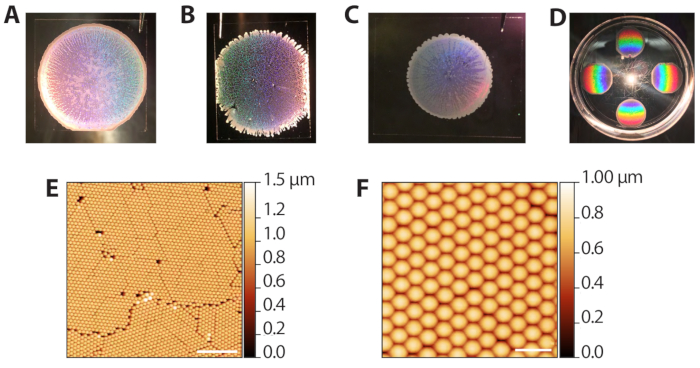
Figure 3: Representative results from evaporative deposition of colloids. (A) Example of optimal colloid deposition. (B) Example of an acceptable colloid deposition in which conditions were more humid (80% RH) than ideal. Holes in the crystal monolayer are apparent. (C) Example of an acceptable colloid deposition in which conditions were drier (65% RH) than optimal. The monolayer regions are slightly translucent while multilayered areas are white and opaque (perimeter and streaks inward). (D) A colloidal crystal illuminated with white light to highlight the rainbow diffraction from the crystals. (E) AFM image (tapping probe AFM in air) of a monolayer of hexagonally packed polystyrene beads from a successful colloid deposition (scale bar = 10 µm). (F) Expanded AFM image of packed beads (scale bar = 2 µm). Please click here to view a larger version of this figure.



