ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Clonal ekspansjon er et sentralt trekk ved antigenspesifikk T-cellerespons. Cellesyklusen til antigenresponserende T-celler har imidlertid blitt dårlig undersøkt, blant annet på grunn av tekniske begrensninger. Vi beskriver en strømningscytometrisk metode for å analysere clonally ekspanderende antigenspesifikke CD8 T-celler i milt og lymfeknuter av vaksinerte mus.
Abstract
Cellesyklusen til antigenspesifikke T-celler in vivo har blitt undersøkt ved hjelp av noen få metoder, som alle har noen begrensninger. Bromodeoxyuridine (BrdU) markerer celler som er i eller nylig fullført S-fase, og karboksyfluorescein succinimidyl ester (CFSE) oppdager datterceller etter divisjon. Disse fargestoffene tillater imidlertid ikke identifisering av cellesyklusfasen på analysetidspunktet. En alternativ tilnærming er å utnytte Ki67, en markør som er sterkt uttrykt av celler i alle faser av cellesyklusen bortsett fra passiv fase G0. Dessverre tillater Ki67 ikke ytterligere differensiering, da det ikke skiller celler i S-fase som er forpliktet til mitose fra de i G1 som kan forbli i denne fasen, fortsett å sykle eller flytte inn i G0.
Her beskriver vi en strømningscytometrisk metode for å fange et "øyeblikksbilde" av T-celler i forskjellige cellesyklusfaser i musens sekundære lymfoidorganer. Metoden kombinerer Ki67 og DNA-farging med store histokompatibilitetskompleks (MHC)-peptid-multimer farging og en innovativ gating strategi, slik at vi kan skille mellom antigenspesifikke CD8 T-celler i G0, i G1 og i S-G2/ M-faser av cellesyklusen i milten og drenering av lymfeknuter av mus etter vaksinasjon med virale vektorer som bærer modellen antigen gag av humant immunsviktvirus (HIV)-1.
Kritiske trinn i metoden var valget av DNA-fargestoffet og gatingstrategien for å øke analysefølsomheten og inkludere svært aktiverte / proliferaterende antigenspesifikke T-celler som ville ha blitt savnet av dagens analysekriterier. DNA-fargestoffet, Hoechst 33342, gjorde det mulig for oss å få en diskriminering av G0/ G1 og G2/ M DNA-toppene, samtidig som vi bevarte membran- og intracellulær farging. Metoden har et stort potensial for å øke kunnskapen om T-cellerespons in vivo og forbedre immunovervåkingsanalysen.
Introduction
Naive T-celler gjennomgår klonisk ekspansjon og differensiering ved antigenpriming. Differensierte T-celler viser effektorfunksjoner som er avgjørende for antigenclearance og for vedlikehold av antigenspesifikk minne, som er nøkkelen for langvarig beskyttelse. I løpet av de første trinnene i den primære responsen er naiv T-celleinteraksjon med antigen-presenting celler (APCer) innenfor spesialiserte nisjer i lymfoide organer avgjørende for å indusere den enorme T-celleproliferasjonen som karakteriserer klonet ekspansjonsfase1,2,3. T celle-APC interaksjon er fint regulert av konsentrasjon og utholdenhet av antigen, kostimulatoriske signaler, og løselige faktorer (cytokiner og kjemokiner) som påvirker mengden og kvaliteten på T-celle klonet avkom4,5,6,7.
Til tross for intensive studier av T-celleklonal ekspansjon, er det fortsatt ikke kjent om antigenprimerte T-celler fullfører hele cellesyklusen på stedet for antigengjenkjenning, eller om de migrerer til andre organer under cellesyklusprogresjon. Denne mangelen på kunnskap skyldes egenskapene til tilgjengelige verktøy for cellesyklusanalyse. Disse inkluderer monoklonale antistoffer (mAbs) som er spesifikke for atommarkøren, Ki67 og cellefarger som enten identifiserer celler som har gjennomgått S-fasen av cellesyklusen (f.eks. Bromodeoxyuridine (BrdU)) eller diskriminerer blant datterceller og deres forfedre (f.eks. karboksyfluorescein succinimidyl ester (CFSE)).
Cellemerkingsfarger, for eksempel CFSE og BrdU, tillater imidlertid ikke fastsettelse av om celler som finnes i et bestemt organ, spredte seg lokalt eller heller migrert til dette nettstedet etter divisjon8,9. Videre er det intranukleære proteinet, Ki67, bare i stand til å skille celler i G0 (Ki67-negative celler) fra de i en hvilken som helst annen cellesyklusfase (Ki67-positive celler). Dermed skiller Ki67-analysen ikke celler i aktiv spredning (dvs. i S, G2eller M) fra de i G1, som enten raskt kan gå videre til divisjon eller bli i lange perioder i G1 eller gå tilbake til passivence10,11.
Her beskriver vi en ny strømningscytometrisk metode for cellesyklusanalyse av antigenspesifikke CD8 T-celler12 fra milten og lymfeknuter (LNer) av vaksinerte mus (figur 1). Metoden utnytter en kombinasjon av Ki67 og DNA-farging som tidligere ble brukt til å analysere cellesyklusen til musebenmarg (BM) hematopoietiske celler13,14. Her brukte vi vellykket Ki67 pluss DNA-farging, sammen med den nylig publiserte innovative gatingstrategien12, til analysen av CD8 T celleklonal ekspansjon. Vi var i stand til å klart diskriminere mellom antigenspesifikke CD8 T-celler i G0, i G1, og i S-G2/ M-faser i milten og drenering av LNer av vaksinerte mus.
Protocol
Mus ble plassert på Plaisant Animal Facility, og arbeidet ble utført under det italienske helsedepartementets autorisasjonsnummer 1065/2015-PR. Protokollen fulgte retningslinjene for dyrepleie i henhold til nasjonale og internasjonale lover og retningslinjer (UE-direktiv 2010/63/UE; Italiensk lovgivende dekret 26/2014).
1. Fremstilling av middels og fargingsoppløsning
- Forbered komplett medium: Roswell Park Memorial Institute (RPMI) medium med 2 mM glutamin, 100 U / ml penicillin / streptomycin, 50 μM beta-mercaptoethanol og 10% volum / volum (v / v) av foster bovint serum (FBS)
- Klargjør fargingsbuffer: Fosfatbufret saltvann uten Ca2+/Mg2+ (PBS) med 1% vekt/volum (m/v) bovint serumalbumin (BSA) og 2 mM etylendiamintetraacetisk syredisodiumsalt (EDTA)
2. Musebehandling
- Prime 7-8-ukers, kvinnelige Balb / c mus ved intramuskulær (i.m.) injeksjon i quadriceps av humant immunsviktvirus (HIV)-1-gag-expressing-sjimpanse adenoviral vektor (ChAd3-gag) med en dose på 107 virale partikler.
- Ved 1-4 måneder etter grunning, øke når musene ved i.m. injeksjon av HIV-1-gag-uttrykke modifisert vaccinia Ankara virus (MVA-gag) med en dose på 106 plakk-forming enheter.
- På dag 3 etter boost, ofre de forsterkede musene ved cervical dislokasjon, og analyser dem parallelt med ubehandlede mus.
- Høst LN-ene som drenerer quadriceps (iliac, popliteal og inguinal) og miltene fra forsterkede og ubehandlede mus. Videre samler du BM fra de to bakbenene fra ubehandlede mus, og bruker denne BM for strømningscytometerinnstillinger og som positiv kontroll for cellesyklusanalyse (Figur 2).
MERK: Generer ChAd3-gag- og MVA-gag-vektorer som beskrevet tidligere12,15,16,17.
3. Isolering av drenering av LN-, milt- og BM-celler
- Isolering av milt- og LN-celler
- Plasser 5 ml komplett medium i hvert av to 15 ml rør, og hold dem på is, klar for organer som skal samles.
- Ofre en voksen mus ved cervical dislokasjon.
- Plasser musen på ryggen, og steriliser hudoverflaten med 70% v / v etanol.
- For å samle inguinale LNer, lag et ~ 1 cm langsgående snitt på magen med saks, og strekk snittet med tangene.
- Visualiser inguinale LNer på den indre overflaten av huden, og høst dem med tang. Plasser de inguinale LN-ene i et av de to 15 ml rørene som er utarbeidet i trinn 3.1.1.
- For å samle milten, lag et peritonealt snitt med saks og fjern milten. Etter å ha kuttet det omkringliggende bindevevet, plasser milten i det andre 15 ml-røret som er fremstilt i trinn 3.1.1.
- For å samle iliac LNer, flytt tarmene til side og visualiser iliac LNer nær den dårligere vena cava, og samle dem deretter ved hjelp av tang. Plasser iliac-LN-ene i samme rør som inneholder inguinale LNer.
MERK: For å få nok LN-celler til farging (se avsnitt 4), er det ofte nødvendig å samle popliteal, inguinal og iliac LNer fra en mus. Disse LN-ene drenerer alle quadriceps (stedet for i.m. vaksinasjon). Denne protokollen bruker bare ett 15 ml rør med samlede LNer. - For å samle popliteale LNer, ta tak i huden på bakbenene og trekk den forsiktig nedover for å avdekke musklene. Sett deretter tang mellom musklene under kneleddet, og samle popliteal LNs. Plasser popliteale LNer i samme rør som inneholder inguinale og iliac LNer.
MERK: Se merknad etter 3.1.7. - Plasser milten i en 70 μm cellesil innenfor en 60 mm kulturrett fylt med 5 ml komplett medium. Bruk et 5 ml sprøytestempel til å mose orgelet forsiktig til det er fullstendig oppsamlert.
- Fjern silen, og overfør cellefjæringen til et rent 15 ml rør.
- Tilsett 5 ml komplett medium til kulturretten, og vask forsiktig parabolen og silen for å sikre at alle celler er gjenopprettet. Basseng med resten av miltcellefjæringen inn i 15 ml-røret.
- For de samlede inguinale, iliac og popliteale LN-ene, klargjør du en enkeltcellet suspensjon etter en lignende prosedyre som den som brukes i trinn 3.1.9 til 3.1.11 for milten.
- Sentrifuger celler ved 400 × g i 10 min ved 4 °C. Kast supernatanten, og resuspender cellepellets i PBS.
- Tell cellene med et Neubauer-kammer ved hjelp av rød blodcelle lysisbuffer og 0,04% v / v trypan blå i PBS.
- Isolering av BM-celler
- Plasser 5 ml komplett medium i et 15 ml rør, og hold det på is, klar for innsamling av bakben.
- Ofre en voksen mus ved cervical dislokasjon.
- Steriliser hudoverflaten med 70 % v/v etanol.
- Lag et tverrsnitt på den ventrale huden med saks, ta godt tak i huden på begge sider av kuttet, og trekk forsiktig nedover for å avdekke musklene i bakbenene.
- For å eliminere huden fra baksiden av bakbenene, hold musen i en liggende stilling, plasser klemmen under kneet, og trekk oppover for å eksponere musklene.
- Klipp beinene i de to ekstremitetene på ett bakben: bekkenet / hofteleddet og ankelen.
- Overfør begge bakbenene til 15 ml-røret som er klargjort i trinn 3.2.1. Hold røret på is.
- Ta bakbenene fra 15 ml-røret og overfør dem til silkepapir. Klipp bakbenene like under kneleddet for å fjerne tibia. Disseker lårbenet og tibia fra de omkringliggende musklene, fjern overflødig vev ved hjelp av saks og vått vevspapiret.
- Klipp beinendene med saks for å eksponere den indre margskaftet. Sett tibia og lårben inn i BM ekstraksjonsrøret (se tilberedning i 3.2.9.1-3.2.9.218), med den bredeste enden nederst.
- Klipp en 200 μL pipettespiss på linjen like over enden av spissen og ved 100 μL-linjen.
- Plasser den midterste delen i den øvre, større delen av spissen, og legg denne i et 1,5 ml mikrofugerør.
- Spinn BM ekstraksjonsrøret på 800 × g i 1 min.
- Kast benet, og resuspender pelletsen kraftig i 1 ml komplett medium for å fjerne klynger. Filtrer celleopphenget gjennom et 70 μm filter plassert på toppen av et 15 ml rør.
- Vask BM ekstraksjonsrøret to ganger med 1 ml komplett medium hver gang. Filtrer gjennom et 70 μm-filter, og slå sammen volumet med resten av celleopphenget oppnådd i trinn 3.2.11.
MERK: Et enkelt 15 ml rør vil inneholde celler fra begge bakbenene på en mus. - Sentrifuger celler ved 400 × g i 10 min ved 4 °C. Kast supernatanten, og bruk cellepelleten på nytt i PBS.
- Tell cellene med et Neubauer-kammer ved hjelp av rød blodcelle lysisbuffer og 0,04% v / v trypan blå i PBS.
4. Farging av milt-, LN- og BM-celler
- Del celleprøver som skal farges i 3 undergrupper: celleprøver for kompensasjon, inkludert BM-celler fra ubehandlede mus som bare skal farges med Hoechst 33342 (heretter referert til som Hoechst) og miltceller fra ubehandlede mus som skal brukes til å forberede en død / levende celleblanding for dødcellefargekompensasjon; positiv kontroll for cellesyklusanalyse, bestående av en BM-prøve fra ubehandlede mus; eksperimentelle prøver som inneholder milt- og LN-prøver fra ubehandlede og vaksinerte mus.
MERK: Kontroller at det er nok milt- og LN-celler til analyse av tilstrekkelig antall gag-spesifikke CD8 T-celler. Det er ofte nødvendig å bruke samlede miltceller og samlede LN-celler fra 3 vaksinerte mus og flekke to eller flere identiske prøver av samlede celler, som hver inneholder 3 × 106 celler. Slå sammen identiske prøver på trinnet av Hoechst farging. På samme måte, flekk samlet miltceller og LN celler fra 3 ubehandlede mus, og slå sammen identiske prøver på slutten. Sett til side en uoppdaget prøve av miltceller fra en ubehandlet mus som skal brukes til instrument- og kompensasjonsoppsett. - Forbered død / levende celleblanding for dødcellefargekompensasjon (denne blandingen av celler vil bare bli farget med dødcellefargen).
- Varm opp et vannbad ved 65 °C.
- Ta en aliquot av miltceller (~3 × 106).
- Overfør cellefjæringen til et mikrofugerør, plasser den i vannbadet ved 65 °C i 5 minutter, og plasser den deretter umiddelbart på is i 10 minutter.
- Bland de varme-drepte cellene med levende miltceller (~ 3 × 106) i et forhold på 1:1, og overfør halvparten av blandingen til en 96 godt rund bunnplate (~ 3 × 106 celler / brønn for dødcelleknusingskontrollen).
- Dødcellefarging av eksperimentelle prøver, positiv kontroll for cellesyklusanalyse og død/levende celleblanding
- Overfør milt, LN, BM-celler (3 × 106 celler/brønn), og død/levende celleblanding (avsnitt 4.2) i 96-brønns rundbunnsplate, i henhold til fargingsskjemaet (trinn 4.1), og sentrifuge ved 400 × g i 3 min ved 4 °C.
- Resuspend hver cellepellet i 50 μL død cellefarge fortynnet i PBS, og resuspend ved pipettering opp og ned 3 ganger umiddelbart.
- Inkuber i 30 min ved 4 °C, beskyttet mot lys.
- Vask celler 2 ganger med fargebuffer; første gang med 200 μL og andre gang med 250 μL. For hver vask sentrifuger platen ved 400 × g i 3 min ved 4 °C.
- Kast supernatanten, og bruk cellepelleten på nytt i 20 μL PBS.
- Membrancellefarging med større histokompatibilitetskompleks (MHC)-peptid multimers og mAbs.
- Ta hensyn til de nødvendige volumene i henhold til fargingsskjemaet (Flow cytometer-innstillinger, tabell 1), klargjør følgende reagenser:
- Fortynn mAb 2,4 G2 i fargingsbufferen i henhold til riktig fortynning (se Materialtabell); For hver prøve som skal farges, bruk 10 μL av denne fortynning.
MERK: 2,4 G2 mAb blokkerer ikke-antigenspesifikk binding av immunglobuliner til FcγIII- og FcγII-reseptorene. - Fortynn H-2k(d) AMQMLKETI allophycocyanin (APC)-merket tetramer (Tetr-gag) i fargingsbufferen for å oppnå riktig fortynning (se Materialfortegnelse); For hver prøve som skal farges, bruk 20 μL av denne fortynning.
- Forbered antistoffblandingen ved å fortynne mAbs i fargingsbufferen i henhold til riktig fortynning (se Materialfortegnelse) som tidligere er bestemt i titreringseksperimenter; For hver prøve som skal farges, bruk 20 μL av denne antistoffblandingen.
MERK: Her, anti-CD3e peridinin klorofyllprotein (PerCP-Cy5.5) (klone 145-2C11), anti-CD8a strålende ultrafiolett (BUV805) (klone 53-6.7), og anti-CD62L fycoerythrin cyanine7 (PECy7) (klone MEL-14) ble brukt.
- Fortynn mAb 2,4 G2 i fargingsbufferen i henhold til riktig fortynning (se Materialtabell); For hver prøve som skal farges, bruk 10 μL av denne fortynning.
- Tilsett 10 μL av den tidligere fortynnede 2,4 G2 mAb (trinn 4.4.1.1), og inkuber i 10 min ved 4 °C, beskyttet mot lys.
- Tilsett 20 μL av den tidligere fortynnede Tetr-gag APC (trinn 4.4.1.2) og 10 μL H-2k(d) AMQMLKETI phycoerythrin (PE) pentamer (pent-gag). Inkuber i 15 min ved 4 °C, beskyttet mot lys.
- Tilsett 20 μL av den tidligere tilberedte antistoffblandingen (trinn 4.4.1.3), og inkuber 15 min ved 4 °C, beskyttet mot lys.
MERK: Derfor er sluttvolumet 80 μL per brønn (trinn 4.3.5, trinn 4.4.2 til 4.4.4). - Vask celler med 200 μL fargebuffer. Sentrifuge ved 400 × g i 5 min ved 4 °C.
- Resuspend cellepellet i 250 μL farging buffer, og overføre celle suspensjon til 5 ml rør. Tilsett 1 ml fargingsbuffer til røret, og sentrifuger ved 400 × g i 5 min ved 4 °C.
- Ta aliquot av BM-celler (3 × 106 celler) (se liste over celleprøver, avsnitt 4.1) som skal brukes til å kompensere Hoechst-kanalen (Hoechst 33342 er begeistret av en ultrafiolett laser (flow cytometer innstillinger (Tabell 2)), og overfør cellefjæringen til et 5 ml rør. Tilsett 1 ml fargingsbuffer til røret, og sentrifuger 400 × g i 5 min ved 4 °C.
- Ta hensyn til de nødvendige volumene i henhold til fargingsskjemaet (Flow cytometer-innstillinger, tabell 1), klargjør følgende reagenser:
5. Fiksering / permeabilisering
- Forbered fersk fikserings-/permeabiliseringsbuffer ved å fortynne 1 del fikserings-/permeabiliseringskonsentrat med 3 deler fikserings-/permeabiliseringsfortynning, i henhold til produsentens instruksjoner.
- Kast supernatanten og pulsvirvel prøvene for å fullstendig disaggregate pelletsen.
- Tilsett 1 ml av den nylagde fikserings-/permeabiliseringsbufferen i hvert rør, inkludert et rør med unstained miltceller (3 x 106, se liste over celleprøver, avsnitt 4.1) og virvel.
- Inkuber i 16 timer ved 4 °C.
MERK: Protokollen kan settes på pause her.
6. Intracellulær farging
- Ki67 flekker
- Forbered fersk permeabiliseringsbuffer 1x ved å fortynne permeabiliseringsbuffer 10x med destillert vann, i henhold til produsentens instruksjoner. Før bruk må permeabiliseringsbufferen 1x filtreres gjennom et 0,45 μm filter for å eliminere aggregater.
- Fortynn mAb Ki67 fluorescein isothiocyanate (FITC) (klone SolA15) i permeabiliseringsbuffer 1x (se Materialtabell), som tidligere bestemt i titreringseksperimenter (sluttvolum på 100 μL per prøve).
- Tilsett 3 ml permeabiliseringsbuffer 1x til hvert rør, og sentrifuger ved 400 × g i 5 minutter ved romtemperatur (RT).
- Kast supernatanten og gjenta trinn 6.1.3.
- Kast supernatanten, og resuspend cellepellet i 100 μL tidligere fortynnet mAb Ki67 FITC (trinn 6.1.2).
- Inkuber i 30 min på RT, beskyttet mot lys.
- Vask celler 2 ganger med 4 ml permeabiliseringsbuffer 1x. For hver vask sentrifuge ved 400 × g i 5 min ved RT.
- Resuspend cellepellet i PBS vurderer følgende volumer: 350 μL PBS for prøvene som skal anskaffes direkte ved strømningscytometeret; 250 μL PBS for at prøvene skal inkuberes med Hoechst kort tid før strømningscytometri (pkt. 6.2).
- DNA-farging
- Tilsett 250 μL 4 μg/ml Hoechst i PBS i hver prøve (den endelige konsentrasjonen av Hoechst er 2 μg/ml).
MERK: I tilfelle to eller flere identiske prøver på 250 μL i PBS ble utarbeidet, slå dem sammen på dette trinnet, og legg til like store mengder 4 μg/ml Hoechst-oppløsning i PBS (den endelige konsentrasjonen av Hoechst er 2 μg/ml). Antall celler påvirker DNA-fargingstrinnet sterkt. Bruk samme cellenummer i hvert utvalg. Vær oppmerksom på at selv et litt redusert cellenummer (f.eks. på grunn av celletap i tidligere vasketrinn) resulterer i høyere Hoechst-binding til DNA og høyere Hoechst-intensitet. - Inkuber i 15 min på RT, beskyttet mot lys.
- Sentrifuger prøvene ved 400 × g i 5 min ved RT.
- Resuspend cellepellet i 350 μL PBS.
- Tilsett 250 μL 4 μg/ml Hoechst i PBS i hver prøve (den endelige konsentrasjonen av Hoechst er 2 μg/ml).
7. Utarbeidelse av kompensasjonsperleprøver
- Forbered 5 μL av antistoffet ved å fortynne mAb i fargingsbufferen på riktig måte.
MERK: For hver fluorokromkonjugert mAb som brukes i forsøket, klargjør den tilsvarende kompensasjonsperleprøven. - Vortex Negativ kontroll og Anti-Rat/Hamster Ig,κ Comp Perler før bruk.
- For hver prøve, introdusere en dråpe (~ 20 μL) av Negative Control CompBeads og en dråpe Anti-Rat / Hamster Ig, k CompBeads.
- Tilsett 5 μL av det forvannede antistoffet (trinn 7.1) på røret, og pipet opp og ned.
- Inkuber i 15 min ved 4 °C, beskyttet mot lys.
- Vask prøver med 2 ml fargebuffer. Sentrifuge ved 400 × g i 5 min ved 4 °C.
- Kast supernatanten, og bruk pelletsen på nytt ved å tilsette 500 μL PBS til hvert rør og virvel.
8. Instrument- og kompensasjonsoppsett og eksperimentelt prøveanskaffelse ved strømningscytometeret
MERK: Se innstillingene for strømningscytometer (tabell 2) for cytometerkonfigurasjonen.
- Generelt instrument- og kompensasjonsoppsett
- Åpne programvaren for eksempelanskaffelse (se Tabell over materialer), og opprett et nytt eksperiment ved å klikke Nytt eksperiment i arbeidsområdebånddelen og velge Nytt tomt eksperiment.
- Dobbeltklikk på det opprettede eksperimentet for å åpne det.
- I vinduet Cytometer Settings klikker du på Parametere og velger alle kanalene (f.eks. PE, APC, etc.) som brukes i fargepanelet, inkludert Parameterne Forward Scatter (FSC) og Side Scatter (SSC).
- Velg lineær skala som en Hoechst-parameter ved å fjerne merket for loggskalaen, og kontroller bredden (W) på spenningspulsen for FCS, SSC og Hoechst.
MERK: Alle parametrene vises som standard i logaritmisk skala (loggskala), bortsett fra FSC og SSC som er i lineær skala. Alle parametrene analyseres av spenningspulsens område (A) og høyde (H). - I det globale regnearketoppretter du en prikktegning med FSC-A på x-aksen og SSC-A på y-aksen.
- Kjør det ubelagte milten ved å klikke Hent data på instrumentbordet for anskaffelse.
- Angi de riktige FSC- og SSC-innstillingene for å visualisere cellene ved å endre spenningsverdiene i Parametere -delen, og opprett en port for å merke alle cellene som vises i prikkplottet FSC-A/SSC-A ved å klikke Polygon Gate på verktøylinjen for arbeidsområdet i det globale regnearket.
- Vis de inngjerdede cellene i histogrammer med hver fluorescensparameter på x-aksen.
- Kjør uoppdagede og fullt fargede miltprøver for å justere fluorescensdetektoren (PMT) for å ha en klar separasjon mellom negative og positive signaler fra de fargede cellene for hver fluorescensparameter.
- Hvis du vil utføre kompensasjonsoppsett, klikker du Eksperimenter på båndet i arbeidsområdet, og under Kompensasjonsoppsett velger du Opprett kompensasjonskontroller. Fjern merket for Inkluder unstained Control Tube/Well , og klikk OK.
MERK: Denne operasjonen vil resultere i opprettelsen av et eksemplar kalt Kompensasjonskontroller og et normalt regneark som inneholder flere ark som tilsvarer hver valgte parameter. - Kjør et utvalg av kompensasjonsperler (se avsnitt 7); angi de riktige FSC- og SSC-innstillingene for å visualisere perlene ved å endre spenningsverdiene og anskaffelsesterskelen på 5000 på FSC-parametere i Cytometer-vinduet.
- Juster P1-porten på perlepopulasjonen, og kontroller at de positive og negative toppene begge er synlige på x-aksen. Gjenta denne operasjonen for hvert eksempel på kompensasjonsperler, og registrer til slutt hver eksempelfil ved å klikke Registrer data på anskaffelsesinstrumentbordet (registrer minst 5 000 hendelser for hvert eksempel).
- For hver registrerte perleprøve setter du P2- og P3-portene på henholdsvis de positive og negative toppene.
- Kjør celleprøvene for kompensasjon (se trinn 4.2 og 4.4.7, og avsnitt 5 og 6). Endre FSC- og SSC-spenningene og terskelverdien for å visualisere cellene, justere P1-porten og til slutt registrere hver prøvefil (registrere minst 10 000 hendelser). Sett P2- og P3-portene på henholdsvis de positive og negative toppene.
MERK: For kompensasjon av Hoechst-kanalen, bruk G0/G1 som negativ topp (P3) og G2/M som den positive (P2). - Klikk Eksperiment i delen på båndet i arbeidsområdet, og velg Beregn kompensasjonunder Kompensasjonsoppsett .
- Gi den opprettede kompensasjonsinnstillingen et navn, koble til og lagre den i gjeldende eksperiment.
- Eksperimentell prøveanskaffelse
- Åpne et eksemplar ved å klikke Nytt eksemplar på verktøylinjen i webleseren, og opprett gatingstrategien i det globale regnearket.
MERK: Gatingstrategien for prøveanskaffelse ligner på utvalgsanalysen, beskrevet i figur 3 og avsnitt 9. - Vis all hendelsespopulasjon i et histogram med CD3-A på x-aksen. Opprett en intervallport for å merke bare CD3+-cellene.
- På instrumentbordetfor anskaffelse velger du lagringsport som Alle hendelser for LN-prøver, og enten Alle hendelser eller CD3+ celler for miltprøver.
- Kjør de eksperimentelle prøvene med lav hastighet, og ta til slutt opp alle filene som sørger for å samle minst 100-200 antigenspesifikke CD8 T-celler for hver prøve fra de vaksinerte musene.
MERK: Filstørrelsen på eksperimentelle prøver er vanligvis stor (30-120 MB), spesielt når frekvensen av antigenspesifikke CD8 T-celler er lav. Derfor må et høyt antall hendelser (> 1 ×10 6) samles inn for å registrere minst 100-200 antigenspesifikke CD8 T-celler. Store filer kan redusere den påfølgende dataanalyseprosessen. Oppkjøpet av bare CD3+ celler i miltprøver (se trinn 8.2.2 ovenfor) er nyttig for å holde filstørrelsen mindre. - Kjør og registrer den positive kontrollen for cellesyklusanalyse, det vil si BM-prøve fra ubehandlede mus.
- Åpne et eksemplar ved å klikke Nytt eksemplar på verktøylinjen i webleseren, og opprett gatingstrategien i det globale regnearket.
9. Dataanalyse
- Åpne programvaren (se Tabell over materialer), og opprett forskjellige grupper som tilsvarer de forskjellige organene som skal analyseres, ved å klikke Opprett gruppe i arbeidsområdebånddelen (dvs. opprette gruppe "a-LNer"; "b-milt"; "c-BM").
MERK: Nyopprettede grupper vises i gruppelisten, mens kompensasjonsgruppen genereres automatisk av programvaren. - Åpne Vinduet Endre gruppe ved å dobbeltklikke gruppenavnet og kontrollere at de nyopprettede gruppene er synkronisert. Hvis ikke, setter du en hake i funksjonen Synkronisert.
- Dra hver FCS-fil i den tilsvarende gruppen.
- Opprett gatingstrategien som begynner med "a-LNs"-gruppen.
- Dobbeltklikk på den fullt fargede prøven i gruppen for å åpne grafvinduet; x- og y-aksen er merket som i fcs-filene (se innstillinger for flytcytometer, tabell 2).
- Vis de totale hendelsene som er oppnådd for denne prøven i et punktplott med DNA-A på x-aksen og DNA-W på y-aksen.
- Velg bare enkeltcellepopulasjonen ved å klikke Rektangel i delen for trykkingsverktøyet i diagramvinduet.
MERK: Enkeltceller har DNA-A-verdier som følger: 2N (lav): mellom 2N og 4N (middels), eller lik 4N (høy), mens DNA-W-verdier er identiske for dem alle (trinn 1 i figur 3). - Dobbeltklikk midt i den rektangulære porten for å vise enkeltceller i en prikkplott med FSC-A-parameteren på x-aksen og død cellefargen på y-aksen.
- Velg bare den aktive cellepopulasjonen ved å klikke på Polygon i delen for trykkingsverktøyet i diagramvinduet. Levende celler er negative for dødcellefargen (trinn 2 i figur 3).
- Dobbeltklikk midt i den mangekantede porten for å vise cellene i en prikktegning med FSC-A-parameteren på x-aksen og SSC-A-parameteren på y-aksen.
- Klikk Rektangel, og opprett en "avslappet" port for å inkludere alle de enkle levende cellene i diagrammet12 (trinn 3 i figur 3).
- Dobbeltklikk midt i den "avslappede" porten for å vise cellene i en prikkplott med CD3 på x-aksen og CD8 på y-aksen.
- Velg CELLENE CD3+CD8+ ved å klikke på Polygon (trinn 4 i figur 3).
- Dobbeltklikk midt på CD3+CD8+ porten for å vise cellene i en prikkplott med Tetr-gag på x-aksen og Pent-gag på y-aksen.
- Merk de antigenspesifikke CD8 T-cellene (positivt for både Tetr-gag og Pent-gag) ved å klikke på Polygon (trinn 5 i figur 3).
- Dobbeltklikk midt i den gag-spesifikke porten for å vise cellene i et punktplott med DNA-A på x-aksen og Ki67 på y-aksen (figur 4).
- Merk cellene i de forskjellige cellesyklusfasene ved å klikke på Quad i gatingverktøydelen av grafvinduet.
MERK: Celle i G0 fase er Ki67neg-DNA lave celler (nederst til venstre kvadrant); celler i G1 er Ki67pos-DNA lav (øvre venstre kvadrant); celler i S-G2/M er Ki67pos-DNA mellomliggende/høy (kvadrant øverst til høyre) (Figur 4). - Kopier gatingstrategien som er opprettet i ett utvalg, til den tilsvarende gruppen for å bruke portene på alle eksemplene i gruppen.
- Gjenta trinn 9,5 til 9,18 for A-LN-gruppen.
- Kontroller at alle portene passer for hver prøve av "b-milten"-gruppen. For å analysere cellesyklusen blant BM-cellene (positiv kontroll), klikk i midten av den "avslappede" porten for å vise cellene i en prikkplott med DNA-A på x-aksen og Ki67 på y-aksen.
- Kontroller at alle portene passer for hvert utvalg av de 3 gruppene (dvs. for celler fra milt, LN og BM).
MERK: Encellet populasjonsport (trinn 9.7) og Quad gate for cellesyklus (trinn 9.17) kan ha forskjellige portkoordinater i forskjellige prøver, hovedsakelig på grunn av de mulige små forskjellene i Hoechst-fargeintensiteten mellom prøver (avsnitt 6.2). Derfor kan det være nødvendig å endre populasjonsporten enkeltcellet og fire portene for cellesyklus i hvert utvalg. Dette gjøres på følgende måte: Dobbeltklikk på gruppenavnet, og fjern synkroniseringen fra gruppeegenskapene. Denne operasjonen tillater endring av portene i ett utvalg uten å endre de samme portene i alle de andre eksemplene i gruppen. Når synkroniseringen er fjernet, endrer du portene der det er nødvendig. - Hvis du vil visualisere resultatene fra denne analysen, klikker du Oppsettredigering i delen for arbeidsområdebånd for å åpne den. Dra hver port for gatingstrategien i eksempelruten til oppsettredigeringsprogrammet, og plasser plottene i henhold til sekvensen av gating-strategien. Hvis det er nødvendig, endrer du diagramtypen ved å dobbeltklikke på det tilsvarende plottet i oppsettet og velge riktig type i Graph Definition-vinduet.
- Klikk på gruppen og gjenta etter funksjoner på layoutbåndet for å visualisere resultatene oppnådd i hvert organ, og sammenlign forskjellige prøver.
Representative Results
Cellesyklusfasene til celler fra milt,LNer og BM av Balb/c-mus ble analysert ved hjelp av fluorescerende DNA-fargestoff, Hoechst og en anti-Ki67 mAb, i henhold til protokollen oppsummert i figur 1. Denne fargingen tillot differensiering av celler i følgende faser av cellesyklusen: G0 (Ki67neg, med 2N DNA definert som DNAlow), G1 (Ki67pos, DNAlow) og S-G2/M (Ki67pos, med et DNA-innhold som består av mellom 2N og 4N, eller lik 4N DNA definert som DNAintermediate/high).
Vi utførte først cellesyklusanalyse av BM-celler for å reprodusere tidligere publiserte resultater13,14 og deretter analyserte cellene av interesse, det vil si CD8 T-celler. Figur 2 viser et typisk eksempel på cellesyklusanalyse av BM-celler (Figur 2A). Protokollen ga en lav variasjonskoeffisient (CV) av G0/G1 og G2/M DNA-topper, noe som indikerer den utmerkede kvaliteten på DNA-fargingen (Figur 2B, som viser et eksempel med CV < 2,5; CV var alltid < 5 i alle forsøkene).
Deretter brukte vi den samme protokollen på antigenspesifikke CD8 T-celler fra vaksinerte mus. BALB/c mus ble vaksinert mot antigen gag av HIV-1 ved å bruke Chad3-gag for grunning og MVA-gag for boosting, begge konstruert for å bære HIV-1 gag. På dag (d) 3 etter boost analyserte vi frekvensen av gag-spesifikke CD8 T-celler fra milten og drenering av LNer. Vi benyttet oss av den nylig definerte gatingstrategien for T-celler i den tidlige fasen av immunresponsen, som i motsetning til den konvensjonelle strategien er egnet for å oppdage høyt aktiverte antigenresponserende CD8 T-celler12. Vi utførte den nye strategien i fem påfølgende trinn. I trinn 1 ekskluderte vi dobler eller aggregater ved DNA-A / -W gate, og i trinn 2 identifiserte vi levende celler ved utelukkelse av dødcellemarkør. I trinn 3 identifiserte vi interessepopulasjonen ved hjelp av en ikke-konvensjonell "avslappet" FSC-A / SSC-A-port (figur 3A) i stedet for den kanoniske smale lymfocyttporten12. Etter å ha kneblet på CD3+CD8+ celler (trinn 4 i figur 3A), identifiserte vi gag-spesifikke CD8 T-celler ved hjelp av to forskjellige MHC-multimere, det vil si Pent-gag og Tetr-gag (trinn 5 i figur 3A). Vi brukte to multimere i stedet for en for å forbedre følsomheten til gag-spesifikk CD8 T celledeteksjon hos vaksinerte mus, uten å øke fargingsbakgrunnen hos ubehandlede mus (Figur 3B og C, trinn 5). Dermed utmerket vi ubehandlede mus (henholdsvis 0,00% og 0,00% antigenspesifikke CD8 T-celler i LNer og milt) fra vaksinerte mus (henholdsvis 0,46% og 0,29% antigenspesifikke CD8 T-celler i LNer og milt, henholdsvis figur 3B og C).
Spesielt tillot protokollen oss å ha en ekstremt lav bakgrunn i den antigenspesifikke CD8 T-celleporten til LNer og milt av ubehandlede mus (vanligvis 0,00% og maksimalt 0,02%). Sammenligningen av gag-spesifikke og ikke gag-spesifikke FSC-A / SSC-A-tomter viste at de gag-spesifikke cellene hadde høy SSC-A og FSC-A (Figur 3D), som bekrefter behovet for å bruke en "avslappet" FSC-A / SSC-A-port for å fange disse cellene. Deretter evaluerte vi prosentandelene av gag-spesifikke CD8 T-celler i ulike cellesyklusfaser (figur 4A). Vi fant at gag-spesifikke CD8 T-celler i milten og enda mer i drenerings-LN-ene inneholdt en høy andel celler i S-G2/ M-faser på dag 3 etter boost (henholdsvis 18,60% og 33,52%).
Videre fant vi ut at gag-spesifikke CD8 T-celler i S-G2/ M-faser hadde høye FSC-A og SSC-A, når de ble lagt over på de totale CD8 T-cellene fra samme organ (Figur 4B). CD62L-uttrykk ved gag-spesifikke CD8 T-celler var lavt, som forventet for aktiverte T-celler, bortsett fra noen få celler i G0 i LN-ene (figur 4C). Til sammen bekreftet disse resultatene at den "avslappede" porten (trinn 3 i figur 3A, B og C) var nødvendig for å inkludere alle de proliferaterende antigenspesifikke CD8 T-cellene12. Protokollen var ekstremt verdifull for en "snapshot" evaluering av cellesyklusfaser av antigenspesifikke CD8 T-celler på analysetidspunktet og av CD62L-uttrykk av celler i forskjellige cellesyklusfaser.

Figur 1: Skjema for protokollen for cellesyklusanalyse av antigenspesifikke CD8 T-celler. Klikk her for å se en større versjon av denne figuren.

Figur 2: Cellesyklusanalyse av BM-celler. BM-celler fra ubehandlede Balb/c-mus ble farget og analysert ved strømningscytometri. (A) Eksempel på gating strategi. Vi gated på enkeltceller i DNA-A / W plottet (venstre) og deretter på levende celler ved død celle fargestoff ekskludering (midten). Deretter ble en "avslappet" FSC-A / SSC-A-port brukt til alle BM-celler (høyre). (B) Eksempel på cellesyklusanalyse av BM-celler (venstre). Vi brukte en kombinasjon av Ki67 og DNA-farging for å identifisere celler i følgende faser av cellesyklusen: G0 (nederst til venstre kvadrant, Ki67neg-DNAlow-celler), G1 (øverst til venstre kvadrant, Ki67pos-DNAlow), S-G2/M (øverste høyre kvadrant, Ki67pos-DNAintermediate/high). Fluorescens Minus En (FMO) kontroll av Ki67 mAb (midten) og DNA histogram (høyre) vises. I DNA-histogramplottet tilsvarer venstre og høyre porter henholdsvis G0/G1 og G2/M DNA-toppen, og tallene representerer variasjonskoeffisientene (CV) for hver topp. I alle de andre plottene representerer tallene celleprosenter i de angitte portene. Figuren viser 1 representativt eksperiment av 5. I hvert eksperiment analyserte vi samlede BM-celler fra 3 mus. Klikk her for å se en større versjon av denne figuren.

Figur 3: Analyse av antigenspesifikke CD8 T-celler fra LNer og milt. Balb/c mus ble primet intramuskulært (i.m.) med Chad3-gag og forsterket i.m. med MVA-gag. På dag 3 etter boost ble drenering av LN- og miltceller fra vaksinerte og ubehandlede kontrollmus farget og analysert av strømningscytometri. (A) Ordningen med gating strategien i fem trinn for å identifisere enkeltceller (Trinn 1); levende celler (trinn 2); lymfocytter (trinn 3); CD8 T-celler (trinn 4); og gag-spesifikke celler (trinn 5). (B-C) Eksempel på plott: analyse av celler fra (B) LNer og (C) milt av ubehandlede (topp) og vaksinerte (nederste) mus. Vi identifiserte enkeltceller på DNA-A/ -W-plottet i trinn 1. Så, i trinn 2, valgte vi levende celler etter utelukkelse av død cellefarge. I trinn 3 brukte vi en ikke-kanonisk "avslappet" port for lymfocytter. I trinn 4 identifiserte vi CD8 T-celler ved deres doble uttrykk for CD3 og CD8. Vi identifiserte deretter gag-spesifikke celler og ikke gag-spesifikke i trinn 5, basert på deres evne til å binde fluorokrom-merket H-2kd-gag-Pentamer (Pent-gag) og H-2kd-gag-Tetramer (Tetr-gag), eller ikke, henholdsvis. (D) FSC-A/SSC-A-profiler av gag-spesifikke (blå) og ikke gag-spesifikke (grå) celler etter gating som beskrevet ovenfor. Tall representerer celleprosenter i de angitte portene. Figuren viser 1 representativt eksperiment av 5. I hvert eksperiment analyserte vi samlet milt og samlet LN-celler fra 3 vaksinerte mus og 3 ubehandlede mus. Klikk her for å se en større versjon av denne figuren.

Figur 4: Cellesyklusanalyse av antigenspesifikke CD8 T-celler. Mus ble vaksinert som i figur 3 og cellesyklusanalyse av gag-spesifikke celler ble utført på dag 3 etter boost, etter gating i 5 trinn som i figur 3. (A) Eksempel på cellesyklusanalyse av gag-spesifikke CD8 T-celler fra LNer (øverst) og milt (bunn) av vaksinerte mus. Cellesyklusfaser ble identifisert som i figur 2B. Panelene representerer celler i G0, i G1og i S-G2/M (venstre) og Fluorescence Minus One (FMO) kontroll av Ki67 mAb (høyre). Tall representerer celleprosenter i de angitte portene. (B) FSC-A/SSC-A-punktplott som viser gag-spesifikke CD8 T-celler i S-G2/M-faser (i rødt) lagt over på total CD3+CD8+ T-celler (i grått) fra LNer (øverst) og milt (nederst) av vaksinerte mus. (C) Forskjøvet histogrammer som viser CD62L-uttrykk ved gag-spesifikke CD8 T-celler i G0 (grønn), i G1 (blå) og i S-G2/M (rød) fra LNer (øverst) og milt (nederst) av vaksinerte mus. Y-aksene angir normalisert antall hendelser. Figuren viser 1 representativt eksempel av 5 uavhengige eksperimenter med totalt 15 mus. Klikk her for å se en større versjon av denne figuren.
Tilleggsmateriale: Innstillinger for flytcytometer. Klikk her for å laste ned denne filen.
Discussion
Selv om T-celleklonal ekspansjon har blitt intensivt studert, er noen aspekter fortsatt ukjente, hovedsakelig fordi verktøyene som er tilgjengelige for å undersøke det er få og har sine egne ulemper. Fra dette perspektivet setter vi opp en svært følsom strømningscytometrisk metode for å analysere cellesyklusen til antigenspesifikke CD8 T-celler tidlig etter vaksinering i en musemodell. Protokollen er basert på en kombinasjon av Ki67 og DNA-farging, som tidligere ble brukt til å analysere cellesyklusen til BM hematopoietiske celler hos mus13,14. For å tilpasse protokollen til antigenspesifikke CD8 T-celler, måtte vi vurdere noen kritiske problemer, inkludert valg av DNA-fargestoff, de riktige forholdene for å oppnå sammenlignbar DNA-farging på tvers av forskjellige prøver, og gatingstrategien for dataanalyse.
Mange fargestoffer er tilgjengelige for DNA-farging, inkludert propidiumjodid og 7-aminoactinomycin D; vi valgte Hoechst fordi den var kompatibel med membranfarging og den milde fikserings- / permeabiliseringsprotokollen som kreves for Ki67-farging. Samtidig tillot farging med Hoechst oss å få et DNA-histogram av utmerket kvalitet, det vil si at G0/ G1 og G2/ M DNA-toppene hadde en mye lavere variasjonskoeffisient (CV) enn DNA-topper som vanligvis oppnås med andre DNA-fargestoffer, for eksempel DRAQ519. Faktisk kan Hoechst flekke DNA selv i levende celler20.
Noen strategier ble brukt for å unngå svingningene i Hoechst-intensiteten i forskjellige prøver av samme eksperiment. Hoechst farging ble utført like før prøveanskaffelse på strømningscytometeret for å minimere nedgangen i fargeintensiteten i løpet av tiden. For de som er interessert i å reprodusere protokollen i store eksperimenter med mange prøver, anbefaler vi å utføre Hoechst-farging på noen få prøver om gangen. En annen ulempe er at Hoechst-intensiteten kan bli sterkt påvirket av cellenummer under inkubasjon med fargestoffet. Av denne grunn anbefaler vi på det sterkeste alltid å bruke samme antall celler og samme volum per prøve for DNA-farging. Hvis et høyt antall celler kreves for oppkjøp ved strømningscytometeret, anbefaler vi å forberede to eller flere identiske prøver og deretter slå dem sammen like før Hoechst-fargingstrinnet.
Et sentralt punkt i protokollen er gatingstrategien for dataanalyse. Vi publiserte nylig en ny strategi for T-celleanalyse i tidlige tider av immunresponsen, noe som tillot oss å øke følsomheten for påvisning av antigenspesifikke T-celler12. Vi brukte denne strategien på dataene som vises her som følger. Først utelukket vi cellemengder i DNA-A/W-plottet. For det andre, etter å ha stirret ut døde celler, brukte vi en ganske stor lymfocyttport i FSC / SSC-tomten ("avslappet port"). Med denne strategien var vi i stand til å inkludere høyt aktiverte antigenspesifikke CD8 T-celler i S-G2/ M som vanligvis går glipp av dagens gatingstrategier, da disse cellene har høy FSC-A og SSC-A. Oppsummert representerer dataanalysen en kritisk del av metoden, noe som er viktig for å oppnå en sensitiv deteksjon av aktiverte / proliferaterende antigenspesifikke T-celler.
Metoden forhindrer muligheten for manglende kritiske T-celledata i tidlige faser av immunresponsen og åpner nye perspektiver for T-celleimmunoovervåking. En fremtidig forbedring kan være å inkludere farging for fosfo-histone 3 som vil tillate differensiering mellom G2 og M21. En nåværende begrensning er at celler må fikses og gjennomsyres for å flekke for atommarkøren Ki67. Celler kan derfor ikke brukes til andre typer analyser, for eksempel sortering og etterfølgende funksjonsanalyse. Videre forstyrrer DNA-fargestoffer, inkludert Hoechst, vanligvis den genomiske DNA-analysen og er ikke egnet for denne typen evaluering. Identifisering av membranmarkører som korrelerer med forskjellige cellesyklusfaser, og som kan farges på levende celler, kan overvinne denne begrensningen. Avslutningsvis har metoden et stort potensial for evaluering av aktiverte/prolifererende T-celler i flere sammenhenger som vaksinasjon, infeksjon, immunmediert sykdom og immunterapi.
Disclosures
A. Folgori og S. Capone er ansatte i Reithera Srl. A. Nikosia er utnevnt oppfinner på patentsøknad WO 2005071093 (A3) "Chimpanzee adenovirus vaksine bærere." De andre forfatterne har ingenting å avsløre.
Acknowledgments
Dette arbeidet ble støttet av Reithera, av MIUR-prosjektet 2017K55HLC_006, og med 5 × 1000 stipend fra Associazione Italiana Ricerca sul Cancro (AIRC). Følgende tetramer ble innhentet gjennom NIH Tetramer Facility: APC-konjugert H-2K (d) HIV gag 197-205 AMQMLKETI.
Materials
| Name | Company | Catalog Number | Comments |
| 1-200 μL universal fit bulk packed pipet tips | Corning | CLS4866-1000EA | |
| 2.4G2 anti-FcγR mAb | BD | 553141 | 10 μg/ml final concentration |
| 5 ml syringe plunger | BD Emerald | 307733 | |
| 15 ml conical tubes | MercK Millipore | SBHA025SB | |
| 60 mm TC-treated Cell Culture Dish | Falcon | 353002 | |
| 70 μm cell strainer | Falcon | 352097 | |
| 96-well Clear Round Bottom TC-treated Culture Microplate | Falcon | 353077 | |
| Anti-Rat/Hamster Ig,k/Negative Control Compensation Particles | BD- Bioscience | 552845 | |
| Beta-mercaptoethanol | Sigma | M3148 | |
| Bovine Serum Albumin | Sigma | A07030 | |
| BUV805 Rat Anti-Mouse CD8a | BD- Bioscience | 564920 | 4 μg/ml final concentration |
| Dulbecco's Phosphate Buffer Saline w/o Calcium w/o Magnesium | Euroclone | ECB4004L | |
| Eppendorf Safe-Lock Tubes, 1.5 mL | Eppendorf | 30120159 | |
| Ethanol | Sigma | 34852-1L-M | |
| Ethylenediaminetetraacetic Acid Disodium Salt solution (EDTA) | Sigma | E7889 | |
| Fetal Bovine Serum | Corning | 35-079-CV | |
| Filcon, Sterile, Syringe-Type 70 μm | Falcon | 352350 | |
| Fixable Viability Dye eFluor 780 | eBioscience | 65-0865-14 | 1:1000 final concentration |
| Foxp3 / Transcription Factor Staining Buffer Set | eBioscience | 00-5523-00 | This Set contains fixation/permeabilization concentrate and diluent, and permeabilization buffer 10x |
| H-2k(d) AMQMLKETI allophycocyanin (APC)-labelled tetramer | provided by NIH Tetramer Core Facility | 6 μg/ml final concentration | |
| H-2k(d) AMQMLKETI phycoerythrine (PE) labelled pentamer | Proimmune | F176-2A-E - 176 | 10 μL / sample |
| Hoechst 33342, Trihydrochloride, Trihydrate - 10 mg/mL Solution in Water | ThermoFisher | H3570 | |
| Ki-67 Monoclonal Antibody (SolA15), FITC | eBioscience | 11-5698-82 | 5 μg/ml final concentration |
| L-Glutamine 100X (200 mM) | Euroclone | ECB3000D | |
| Millex-HA Filters 0,45 µm | BD | 340606 | |
| Penicillin/Streptomycin 100X | Euroclone | ECB3001D | |
| PE/Cyanine7 anti-mouse CD62L Antibody | Biolegend | 104418 | 0.2 μg/ml final concentration |
| PerCP-Cy™5.5 Hamster Anti-Mouse CD3e | BD- Bioscience | 551163 | 4.4 μg/ml final concentration |
| Red Blood Cell Lysis Buffer | Sigma | R7757 | |
| Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352058 | |
| RPMI 1640 Medium without L-Glutamine with Phenol Red | Euroclone | ECB9006L | |
| Software package for analyzing flow cytometry data | FlowJo | v.10 | |
| Software for acquisition of samples at flowcytometer | BD FACSDiva | v 6.2 | |
| Trypan Blue Solution | Euroclone | ECM0990D |
References
- Castellino, F., et al. Chemokines enhance immunity by guiding naive CD8+ T cells to sites of CD4+ T cell-dendritic cell interaction. Nature. 440 (7086), 890-895 (2006).
- Zhang, N., Bevan, M. J. CD8(+) T cells: foot soldiers of the immune system. Immunity. 35 (2), 161-168 (2011).
- Bajénoff, M., et al. Highways, byways and breadcrumbs: directing lymphocyte traffic in the lymph node. Trends Immunology. 28 (8), 346-352 (2007).
- Bevan, M. J., Fink, P. J. The CD8 response on autopilot. Nature Immunology. 2 (5), 381-382 (2001).
- Van Stipdonk, M. J., Lemmens, E. E., Schoenberger, S. P. Naïve CTLs require a single brief period of antigenic stimulation for clonal expansion and differentiation. Nature Immunology. 2 (5), 423-429 (2001).
- Kaech, S. M., Wherry, E. J., Ahmed, R. Effector and memory T-cell differentiation: implications for vaccine development. Nature Review Immunology. 2 (4), 251-262 (2002).
- Beverley, P. C. Primer: making sense of T-cell memory. Nature Clinical Practice Rheumatology. 4 (1), 43-49 (2008).
- Parretta, E., et al. CD8 cell division maintaining cytotoxic memory occurs predominantly in the bone marrow. Journal of Immunology. 174 (12), 7654-7664 (2005).
- Di Rosa, F. Maintenance of memory T cells in the bone marrow: survival or homeostatic proliferation. Nature Review Immunology. 16 (4), 271 (2016).
- Di Rosa, F. Two niches in the bone marrow: a hypothesis on life-long T cell memory. Trends in Immunology. 37 (8), 503-512 (2016).
- Di Rosa, F. Commentary: Memory CD8(+) T cells colocalize with IL-7(+) stromal cells in bone marrow and rest in terms of proliferation and transcription. Frontiers in Immunology. 7, 102 (2016).
- Simonetti, S., et al. Antigen-specific CD8 T cells in cell cycle circulate in the blood after vaccination. Scandinavian Journal of Immunology. 89 (2), 12735 (2019).
- Wilson, A., et al. c-Myc controls the balance between hematopoietic stem cell self-renewal and differentiation. Genes & Development. 18 (22), 2747-2763 (2004).
- Hirche, C., et al. Systemic virus infections differentially modulate cell cycle state and functionality of long-term hematopoietic stem cells in vivo. Cell Report. 19 (11), 2345-2356 (2017).
- Colloca, S., et al. Vaccine vectors derived from a large collection of simian adenoviruses induce potent cellular immunity across multiple species. Science Translational Medicine. 4 (115), (2012).
- Di Lullo, G., et al. Marker gene swapping facilitates recombinant Modified Vaccinia Virus Ankara production by host-range selection. Journal of Virological Methods. 156 (1-2), 37-43 (2009).
- Di Lullo, G., et al. The combination of marker gene swapping and fluorescence-activated cell sorting improves the efficiency of recombinant modified vaccinia virus Ankara vaccine production for human use. Journal of Virological Methods. 163 (2), 195-204 (2010).
- Mouse phenotype. , Available from: https://www.mousephenotype.org/data/secondaryproject/3i (2020).
- Yoon, H., Kim, T. S., Braciale, T. J. The cell cycle time of CD8+ T cells responding in vivo is controlled by the type of antigenic stimulus. PLoS One. 5 (11), 15423 (2010).
- Pauklin, S., Vallier, L. The cell-cycle state of stem cells determines cell fate propensity. Cell. 155 (1), 135-147 (2013).
- Vignon, C., et al. Flow cytometric quantification of all phases of the cell cycle and apoptosis in a two-color fluorescence plot. PLoS One. 8 (7), 68425 (2013).
Tags
Immunologi og infeksjon utgave 167 antigenspesifikke CD8 T-celler cellesyklus Ki67 DNA-fargestoff strømningscytometri milt lymfeknuter musErratum
Formal Correction: Erratum: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice
Posted by JoVE Editors on 11/03/2021.
Citeable Link.
An erratum was issued for: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice. The Authors section and a figure were updated.
The authors section was updated from:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
* These authors contributed equally
To:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2,6, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
6IRCCS, Neuromed
* These authors contributed equally
Figure 1 was updated from:
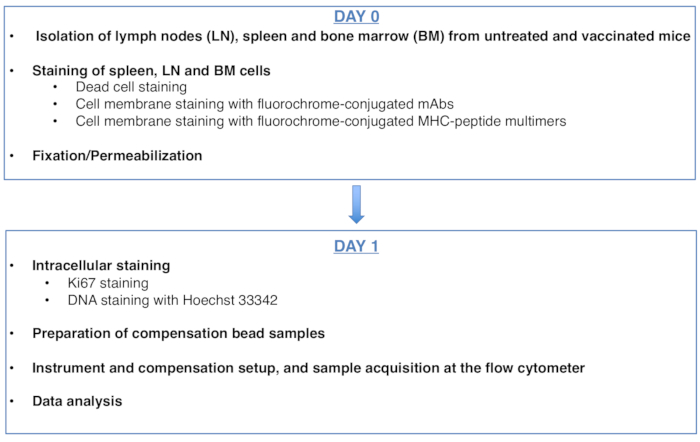
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
To:
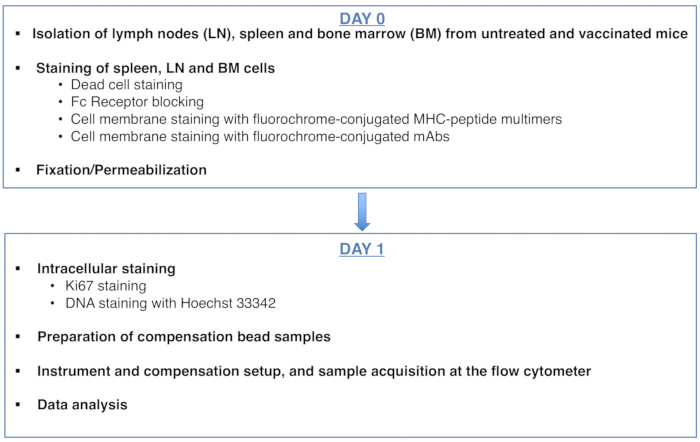
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.



