ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Dette dokumentet beskriver protokollene for prøvepreparering, datareduksjon og dataanalyse i NSE-studier (neutron spin echo) av lipidmembraner. Fornuftig deuteriummerking av lipider gir tilgang til forskjellige membrandynamikker på mesoskopisk lengde og tidsskalaer, over hvilke vitale biologiske prosesser oppstår.
Abstract
Lipid bilayers danner hovedmatrisen av cellemembraner og er den primære plattformen for næringsutveksling, proteinmembraninteraksjoner og viral spirende, blant andre vitale cellulære prosesser. For effektiv biologisk aktivitet bør cellemembraner være stive nok til å opprettholde cellens og dens roms integritet, men likevel flytende nok til at membrankomponenter, som proteiner og funksjonelle domener, kan spre seg og samhandle. Denne delikate balansen mellom elastiske og flytende membranegenskaper, og deres innvirkning på biologisk funksjon, nødvendiggjør en bedre forståelse av kollektiv membrandynamikk over mesoskopisk lengde og tidsskalaer av viktige biologiske prosesser, for eksempel membrandeformasjoner og proteinbindingshendelser. Blant teknikkene som effektivt kan sondere dette dynamiske området er nøytron spinn ekko (NSE) spektroskopi. Kombinert med deuteriummerking kan NSE brukes til å få direkte tilgang til bøying og tykkelsessvingninger samt mesoskopisk dynamikk i utvalgte membranfunksjoner. Dette dokumentet gir en kort beskrivelse av NSE-teknikken og skisserer prosedyrene for å utføre NSE-eksperimenter på liposomale membraner, inkludert detaljer om prøvepreparerings- og deuterasjonsordninger, sammen med instruksjoner for datainnsamling og reduksjon. Artikkelen introduserer også dataanalysemetoder som brukes til å trekke ut viktige membranparametere, for eksempel bøyestivhetsmodulus, områdekompressibility modulus og in-plane viskositet. For å illustrere den biologiske betydningen av NSE-studier diskuteres utvalgte eksempler på membranfenomener undersøkt av NSE, nemlig effekten av tilsetningsstoffer på membranbøyende stivhet, virkningen av domenedannelse på membransvingninger og den dynamiske signaturen av membranproteininteraksjoner.
Introduction
Forståelsen av cellemembraner og deres funksjon har utviklet seg bemerkelsesverdig de siste tiårene. Det tidligere synet på cellemembraner som passive lipid-bilayere som definerer cellegrenser og husmembranproteiner1, har gradvis forvandlet seg til en dynamisk modell der lipidbilayere spiller en viktig rolle i å regulere vitale biologiske prosesser, inkludert cellulær signalering, molekylær utveksling og proteinfunksjon - for å nevne noen2,3,4,5,6. Denne erkjennelsen av at cellemembraner er svært dynamiske, som stadig gjennomgår ombygging og molekylær omfordeling, har oppfordret til vitenskapelige undersøkelser utover likevektsstrukturer av membraner7,8,9. Følgelig er flere tilnærminger utviklet for å studere de ulike dynamiske modusene i biologiske og bioinspirerte lipidmembraner. Til dags dato har de fleste av disse studiene primært fokusert på diffusive molekylære bevegelser10,11,12,13 og makroskopiske formsvingninger14,15,16, og etterlater et betydelig gap i å forstå mellomliggende membrandynamikk, det vil si kollektive svingninger i lipidsamlinger som består av få 10-100-tallet lipidmolekyler. Disse dynamikkene forekommer over lengdeskalaer på få tiere til få 100 Å og over tid skalaer av sub-ns til få hundre ns (se figur 1), referert til her som mesoskopiske skalaer. Det er faktisk på disse skalaene at viktig biologisk aktivitet foregår påmembrannivå 17. Dette inkluderer viral spirende18, kanal gating19, og membran-protein interaksjoner20. Det er også viktig å påpeke at energilandskapet til membranproteiner21,22 viser at konformasjonsendringer i proteiner - nødvendig for deres regulatoriske rolle - skjer over ns-tidsskalaene23 av kollektive membransvingninger, og understreker ytterligere betydningen av mesoskopisk dynamikk i cellemembranens biologiske funksjon og deres bioinspirerte analoger20. Dette papiret fokuserer på de to primære mesoskopiske dynamiske modusene i lipidmembraner, nemlig bøyningssvingninger og tykkelsessvingninger.
Hovedutfordringen med å undersøke disse svingningsmodusene direkte er vanskeligheten med samtidig tilgang til deres romlige og tidsmessige skalaer ved hjelp av standard spektroskopimetoder. Den andre utfordringen er at direkte kontaktteknikker kan påvirke de samme svingningene de er ment å måle16. Dette forverres ytterligere av sammensetningen og strukturell kompleksiteten til biologiske membraner24,25, noe som resulterer i ikke-homogene membranegenskaper, inkludert lipiddomenedannelse26,27,28,29,30 og membran asymmetri31,32,33- krever selektive sonder for å forstå dynamikken i forskjellige membranegenskaper. Heldigvis kan disse utfordringene overvinnes med ikke-invasive nøytronspektroskopimetoder, for eksempel nøytronspinnekko (NSE), som i seg selv får tilgang til ønsket lengde og tidsskalaer, og ytterligere muliggjør studier av selektive membranfunksjoner uten å endre deres fysisk-kjemiske miljø34. Faktisk har NSE-spektroskopi de siste årene utviklet seg til en unik og kraftig sonde av kollektiv membrandynamikk35. Resultater fra NSE-studier på lipidmembraner har gitt ny innsikt imekaniske 36,37 og viskoelastiske38,39 egenskaper til lipidmembraner og har kastet nytt lys over deres potensielle rolle i biologisk funksjon40,41.
NSE spektroskopi teknikken er basert på en interferometrisk instrument design, først foreslått av Mezei42, ved hjelp av en rekke spin-flippers og magnetiske spoler for å kontrollere precession av nøytron spinn som nøytroner traversere instrumentet. Designet hviler på magnetisk speiling av magnetfeltelementene med hensyn til prøveposisjonen (Figur 1A). Dette innebærer at i fravær av energiutveksling mellom nøytron og prøven, utfører nøytronet samme antall spinnpresesjoner, i motsatte retninger, i første og andre halvdel av instrumentet (legg merke til π-flipper mellom de to precession spolene). Som et resultat forblir nøytronens endelige spinntilstand uendret i forhold til den opprinnelige tilstanden - et fenomen referert til som spin-ekko (se gjennomsiktig nøytron i figur 1A). Men når nøytronen samhandler energisk med prøven, endrer energiutvekslingen antall spinnpresesjoner i andre halvdel av instrumentet, noe som fører til en annen endelig spinntilstand (se figur 1A). Dette oppdages eksperimentelt som tap i polarisering, som vist senere i dette papiret. Hvis du vil ha mer informasjon om NSE-teknikken, henvises leseren til dedikerte tekniske papirer42,43,44,45.
Her presenteres en forenklet beskrivelse for å gi et grovt estimat av lengden og tidsskalaene som er tilgjengelige for NSE. Lengdeskalaene bestemmes av rekkevidden av oppnåelige bølgevektoroverføringer, Q = 4π sin θ/λ, hvor 2θ er spredningsvinkelen og λ er nøytronbølgelengden. Man kan se at Q er satt av bølgelengdeområdet og rotasjonsgraden til den andre armen på spektrometeret (se figur 1A). Et typisk Q-område på NSE-spektrometre er ~0,02-2 Å-146,47og opptil 0,01-4 Å-1 med nylige oppgraderinger48,49, tilsvarende romlige vekter på ~ 1-600 Å. På den annen side beregnes den tilgjengelige tidsskalaen fra den totale presesjonsvinkelen (eller fasen) som er oppnådd av nøytronet i de magnetiske precession-spolene, og er funnet å være50:  . I dette uttrykket er t Fourier-tiden definert som
. I dette uttrykket er t Fourier-tiden definert som  , hvor er
, hvor er  nøytron gyromagnetisk forhold,
nøytron gyromagnetisk forhold,  er spolelengden, og
er spolelengden, og  er styrken til spolens magnetfelt. Det er verdt å påpeke at Fourier-tiden er en mengde som er strengt avhengig av instrumentgeometrien, magnetfeltstyrken og nøytronbølgelengden. For eksempel, ved hjelp av nøytroner av bølgelengde
er styrken til spolens magnetfelt. Det er verdt å påpeke at Fourier-tiden er en mengde som er strengt avhengig av instrumentgeometrien, magnetfeltstyrken og nøytronbølgelengden. For eksempel, ved hjelp av nøytroner av bølgelengde  = 8 Å og instrumentinnstillinger på
= 8 Å og instrumentinnstillinger på  = 1,2 m og
= 1,2 m og  = 0,4 T, beregnes Fourier-tiden til å være t ~ 50 ns. Eksperimentelt er Fourier-tiden innstilt ved å endre strømmen i precession-spolene (dvs. magnetisk feltstyrke) eller ved hjelp av forskjellige nøytronbølgelengder, noe som resulterer i typiske NSE-tidsskalaer på ~ 1 ps til 100 ns. Nylige oppgraderinger i NSE-spektrometre har imidlertid gitt tilgang til lengre Fourier-ganger, opp til ~ 400 ns på J-NSE-Phoenix spektrometeret på Heinz Maier-Leibnitz Zentrum51 og SNS-NSE-spektrometeret på Oak Ridge National Lab48, og opptil ~ 1000 ns på IN15 NSE-spektrometeret ved Institut Laue-Langevin (ILL)49.
= 0,4 T, beregnes Fourier-tiden til å være t ~ 50 ns. Eksperimentelt er Fourier-tiden innstilt ved å endre strømmen i precession-spolene (dvs. magnetisk feltstyrke) eller ved hjelp av forskjellige nøytronbølgelengder, noe som resulterer i typiske NSE-tidsskalaer på ~ 1 ps til 100 ns. Nylige oppgraderinger i NSE-spektrometre har imidlertid gitt tilgang til lengre Fourier-ganger, opp til ~ 400 ns på J-NSE-Phoenix spektrometeret på Heinz Maier-Leibnitz Zentrum51 og SNS-NSE-spektrometeret på Oak Ridge National Lab48, og opptil ~ 1000 ns på IN15 NSE-spektrometeret ved Institut Laue-Langevin (ILL)49.
Foruten direkte tilgang til lengden og tidsskalaen til membrandynamikk, har NSE de iboende egenskapene til nøytronisotopfølsomhet52. Spesielt resulterer nøytronenes evne til å samhandle annerledes med isotoper av hydrogen, det mest tallrike elementet i biologiske systemer, i en annen nøytronspredningslengdetetthet,34 eller NSLD (tilsvarende den optiske indeksen for brytning50), når protium erstattes av deuterium. Dette aktiverer en tilnærming kjent som kontrastvariasjon, som vanligvis brukes til å fremheve bestemte membranfunksjoner eller skjule andre – sistnevnte scenario kalles kontrastsamsvar. En hyppig påføring av kontrastvariasjon/matching er utskifting av vann (NSLD = -0,56 × 10-6 Å-2) med tungtvann eller D2O (NSLD = 6.. 4 × 10-6 Å-2) for å forsterke nøytronsignalet fra protierte lipidmembraner (NSLD ~ 0 × 10-6 Å-2). Denne tilnærmingen er svært effektiv i studier av membranstruktur fordi penetrasjonen av D2O i membranens hodegrupperegion tillater nøyaktig bestemmelse av membrantykkelsene (se figur 2A, venstre panel) og av plasseringen av forskjellige lipidundergrupper når mer sofistikerte modeller påføres53,54. Dette dokumentet fremhever noen eksempler på bruk av kontrastvariasjon for studier av kollektiv dynamikk i biomimetiske membraner og utvalgte membranfunksjoner.
Her illustreres effektiviteten av NSE i å gi unik innsikt i dynamiske og funksjonelle membranegenskaper gjennom konkrete eksempler på NSE-studier på modell og biologisk relevante lipidmembransystemer med vekt på mesoskala dynamikk i frittstående membraner, i form av liposomale suspensjoner. For NSE-målinger av in-plane membrandynamikk henvises leseren til dedikerte publikasjoner om beiteforekomst nøytron spin-echo spektroskopi (GINSES)55,56 og andre studier av justerte multilamellarmembran stabler57,58,59,60.
For enkelhets skyld fremhever dette dokumentet tre forskjellige ordninger for membrandeuterasjon illustrert på en godt studert domeneforming, eller fase separering, lipid bilayer system av 1,2-dimyristoyl-sn-glysero-3-fosfocholine (DMPC) og 1,2-distearoyl-sn-glysero-3-fosfocholine (DSPC) blandinger61,62. De to lipidene er preget av en uoverensstemmelse i deres hydrokarbonkjedelengde (14 karboner/hale i DMPC vs. 18 karboner/hale i DSPC) og deres overgangstemperatur for gelvæske (Tm, DMPC = 23 °C vs. Tm, DSPC = 55 °C). Dette resulterer i lateral faseseparasjon i DMPC: DSPC-membraner ved temperaturer mellom øvre og nedre overgangstemperaturer i blandingen63. Deuterasjonsordningene som vurderes her, velges for å demonstrere de forskjellige dynamiske modusene som er tilgjengelige i NSE-målinger på liposomale membraner, nemlig bøyesvingninger, tykkelsessvingninger og selektive bøynings- / tykkelsessvingninger i laterale domener. Alle lipidsammensetninger rapporteres for DMPC:DSPC-bilayers fremstilt ved en molefraksjon på 70:30, ved hjelp av kommersielt tilgjengelige protierte og perdeutererte varianter av DMPC og DSPC. Alle prøveprepareringstrinn er basert på 4 ml liposomal suspensjon, i D2O, med en lipidkonsentrasjon på 50 mg / ml, for en total lipidmasse på Mtot = 200 mg per prøve.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Deuteration ordningen som kreves for eksperimentet
- For bøyevariasjonsmålinger, lag fullt protierte liposomer i D2O (D 99,9%) eller D2O-buffer (f.eks. fosfatbuffer fremstilt med D2O i stedet forH2O). Bruk fullt protert DMPC (C36H72NO8P) og DSPC (C44H88NO8P) med
 133,4 mg, der XDMPC og XDSPC er molefraksjonene av DMPC og DSPC, her satt til henholdsvis 0,7 og 0,3, og MwDMPC og MwDSPC er molarvektene gitt av henholdsvis 677,9 g/mol og 790,1 g/mol. Tilsvarende mDSPC = 66,6 mg. Denne deuteringsordningen øker spredningskontrasten mellom membranen (NSLD ~ 0 × 10-6 Å-2) og den deutererte bufferen (NSLD ~ 6,4 × 10-6 Å-2) og forsterker signalet fra membranunduleringer (se figur 2A venstre panel).
133,4 mg, der XDMPC og XDSPC er molefraksjonene av DMPC og DSPC, her satt til henholdsvis 0,7 og 0,3, og MwDMPC og MwDSPC er molarvektene gitt av henholdsvis 677,9 g/mol og 790,1 g/mol. Tilsvarende mDSPC = 66,6 mg. Denne deuteringsordningen øker spredningskontrasten mellom membranen (NSLD ~ 0 × 10-6 Å-2) og den deutererte bufferen (NSLD ~ 6,4 × 10-6 Å-2) og forsterker signalet fra membranunduleringer (se figur 2A venstre panel). - For å måle bøyedynamikken til utvalgte laterale membranfunksjoner, for eksempel matrisedynamikk i faseseparerende DMPC:DSPC-membraner, bruke protiated DMPC (C36H72NO8P) og deuterated, DSPC-d83 (C44H5NO8PD83, Mw 873.7 g/mol), slik at mDMPC = 128,8 mg og mDSPC-d83 = 71,2 mg. Dette deuteringsskjemaet minimerer spredningen fra de uønskede DSPC-rike domenene, noe som muliggjør selektive målinger av bøyesvingninger fra den DMPC-rike matrisen (se figur 2B i midten).
MERK: For å finne den optimale lipiddeutering som kreves for en bestemt kontrastmatchingsordning, kan du bruke tilgjengelige nettbaserte SLD-kalkulatorer (scattering length tetthet), for eksempel den som er utviklet av NIST Center for Neutron Research64. Disse nettbaserte grensesnittene er utstyrt med brukervennlige verktøy for enkel beregning av SLD av lipider med ulike grader av deuterasjon, samt lipidblandinger. - For NSE-målinger av gjennomsnittlige membrantykkelsessvingninger (uten lateral kontrast), bruk hale-deutererte varianter av de bestanddelene lipider, dvs. DMPC-d54 (C36H18NO8PD54, 732,3 g/mol) og DSPC-d70 (C44H18NO8PD70, 860,1 g/mol)35,38, slik at mDMPC-d54 = 133,0 mg og mDSPC-d70 = 67,0 mg. Dette kontrastskjemaet (figur 2A, høyre panel) forsterker spredningssignalet fra lipidhodegruppene (NSLD ~ 4,5 × 10-6 Å-2) ved å kontrasttilpasse halegruppen (NSLD ~ 6,4 × 10-6 Å-2) til den deutererte bufferen som muliggjør deteksjon av svingninger i membrantykkelsen.
- For tykkelsessvingningsstudier av utvalgte membranrom, F.eks. DMPC-rik matrise, bruk den samme strategien som er beskrevet i trinn 1.2 ved å erstatte protierte DMPC-lipider med sine tail-deuterated analoger, det vil si DMPC-d54, slik at DSPC-rike domener er kontrast-matchet med den deutererte bufferen og det primære spredningssignalet er fra hodegruppeområdet til tail-deuterated DMPC-rich.
2. Fremstilling av lipidfjæring for ekstrudering
- Beregn massen av hver bestanddel i prøven, avhengig av prøvesammensetning. Som en tommelfingerregel, for prøver med flere molekylære komponenter, er massen av en komponent gitt av sin molarmasse, Mwi, vektet av sin molefraksjon, Xi, og normalisert over alle komponenter slik at:
 hvorM-tot er den totale massen, satt her til 200 mg. Se eksemplet ovenfor for DMPC-DSPC lipid-bilayers med forskjellige deuterasjonsordninger.
hvorM-tot er den totale massen, satt her til 200 mg. Se eksemplet ovenfor for DMPC-DSPC lipid-bilayers med forskjellige deuterasjonsordninger. - Bruk en digital halvmikrobalanse, vei de beregnede massene av lipider (og andre prøvebestanddeler, for eksempel proteiner, nanopartikler, etc.) og legg dem til et hetteglass eller rundbunnsflaske - husk å veie hetteglasset eller kolben på forhånd. Tilsett 1 ml oppløsningsvæske for å løse opp de veide komponentene ved å blande manuelt inne i en hette. For rene lipidprøver, bruk kloroform eller etanol. For prøver med ekstra, ikke-lipidkomponenter (f.eks. nanopartikler), velg et vanlig løsningsmiddel som sprer alle komponenter.
- For små lipidmengder (<10 mg), lag en lagerløsning og pipette det nødvendige volumet i blandingen.
MERK: Ikke tilsett store mengder løsemiddel, da det vil redusere tørketrinnet for løsningsmidlet som er beskrevet nedenfor, betydelig.
- For små lipidmengder (<10 mg), lag en lagerløsning og pipette det nødvendige volumet i blandingen.
- Tørk lipidløsningen inne i en hette ved å forsiktig strømme en inert gass (f.eks. nitrogen, argon) i hetteglasset mens du sakte roterer hetteglasset i en vinkel. Hold hetteglassene i vippet posisjon for å lage en tynn film av tørket lipid på hetteglassveggene, noe som vil tillate jevn tørking. Plasser hetteglasset periodisk i et vannbad ved 35 °C for å omgå fordampningsmediert kjøling, noe som vil redusere fordampningen av løsningsmidlet.
- Plasser hetteglassene over natten i en vakuumovn ved ~35 °C for å fjerne det gjenværende løsningsmidlet helt. For umettede lipider, tøm vakuumet med en inert gass for å minimere oksidasjon.
- For å sikre fullstendig fjerning av løsemiddel, vei hetteglasset etter lipidtørking og bekreft at det ikke er overflødig masse utover de målte materialmengdene. Gjør dette ved å trekke massen av hetteglasset fra den målte massen etter tørking. Hvis det er overflødig masse, tørk prøven under vakuum i ytterligere 6 timer. Gjenta denne prosessen etter behov.
- Hydrater lipidfilmen med 4 ml D2O eller D2O-buffer for å oppnå en lipidkonsentrasjon på 50 mg/ml. For lipider med høye overgangstemperaturer, for eksempel DMPC-DSPC-blandinger, varme bufferen til over overgangstemperaturen (60 °C) for å sikre jevn blanding.
MERK: Siden NSE-eksperimenter krever relativt store prøvevolumer (~4 ml), bør du vurdere å hydrere prøven ved hjelp av halvparten av den nødvendige bufferen, det vil si 2 ml, for å minimere antall profiler per prøve (se avsnitt 3). I dette tilfellet legger du til den gjenværende halvdelen av bufferpostekstruderingen. Legg merke til at kapasiteten til sprøyter som brukes i ekstrudering er begrenset til 1 ml. Dermed vil fuktighet med 4 ml buffer kreve fire sett med ekstrudering. - Virvelblanding den hydrerte lipidoppløsningen til lipidfilmen er fullstendig oppløst og ikke lenger er synlig på veggene i hetteglasset. På dette stadiet danner de hydrerte lipidene multilamellar vesicles og mikron størrelse multilamellar stabler og suspensjonen vises melkehvit.
- For å lette brudd på lipidstabelene og for å redusere multilamellaritet, utfør fem fryse-/tinesykluser ved å plassere hetteglasset med hydrert lipidoppløsning i en lab-fryseenhet (helst -80 °C fryser) til det er helt frosset og deretter overføre hetteglasset til et 35 °C vannbad til lipidoppløsningen er fullstendig tint. Virvel den tinte løsningen til homogen. Gjenta fire ganger til.
MERK: Alternativt kan et tørt isbad tilberedes for rask frysing ved å kombinere aceton og tørris.
3. Ekstrudering av den hydrerte lipidoppløsningen
- Monter ekstruderoppsettet ved hjelp av en polykarbonatmembran mellom to membranstøtter og legg til to papirfiltre på hver side for å gi ekstra støtte. Bruk en polykarbonatmembran med porestørrelse som samsvarer med målet liposomal størrelse (vanlige porestørrelser for NSE-eksperimenter er 50 nm og 100 nm - vanligvis tillater 100 nm-diameter liposomer mindre begrensede membransvingninger, men mindre 50 nm liposomer kan brukes til krumningsstudier). Påse at polykarbonatmembranen er helt strukket før du fullfører monteringen og strammer det eksterne ekstruderhuset.
- Hydrater polykarbonatmembranen ved å passere ~ 0,3 ml D2O eller D2O-buffer et par ganger gjennom membranenheten ved hjelp av lufttette glasssprøyter. Bruk den samme bufferen som brukes i prøvepreparering. La den stå i minst 10 minutter, og sug deretter bufferen helt ut før du introduserer prøven.
- Fyll en 1 ml gasstett sprøyte med den tilberedte lipidoppløsningen og sett den inn i den ene enden av ekstruderapparatet. Sett deretter en tom sprøyte inn i motsatt ende. Når sprøytene er koblet til ekstruderenheten, plasserer du den i ekstruderblokken.
- Hvis forhøyede temperaturer er nødvendig for ekstrudering, som ved mettede lipider med høye overgangstemperaturer (f.eks. DSPC, Tm = 55 °C), forvarm ekstrudervarmeblokken over lipid overgangstemperaturen (f.eks. 60 °C), ved å plassere varmeblokken på en kokeplate eller ved å bruke et sirkulasjonsbad som vist i figur 3A.
MERK: Dette trinnet er avgjørende for å sikre homogen blanding av lipider og for å unngå å utøve ekstremt trykk under ekstrudering, noe som kan ødelegge polykarbonatmembranen. For lipidprøver med lave overgangstemperaturer (<25 °C), utfør ekstrudering ved romtemperatur. - For å ekstrudere lipidløsningen, fest ekstrudersettet til en programmerbar sprøytepumpe med en aluminiums-/stålramme som vist i figur 3A. For temperaturkontrollerte profiler, legg til en spesialbygd ekstruderbase med en væskekanal og fest til et sirkulerende vannbad.
- Programmer sprøytepumpen til å utføre 15-20 ekstruderingssykluser i henhold til produsentens bruksanvisning. Når den ekstruderes, endres fargen på lipidoppløsningen fra melkehvit til gjennomsiktig opalblå (Figur 3B,C), noe som indikerer en endelig liposomal størrelse som er mindre enn bølgelengden til synlig lys, som forventet. Følg trinnene nedenfor for den typen sprøytepumpe som er vist i figur 3A.
- Begynn med å justere pumpeinnstillingene. Hold nede Hastighet-knappen og angi ekstruderingshastigheten (50,99 ml/t), trykk deretter på Diameter-knappen og angi sprøytediameteren (4,606 mm). Bruk pil opp under hvert siffer på skjermen for å endre sifferverdien.
- Plasser ekstrudersettet med prøvesprøyten til høyre (se figur 3A). Trykk på Trekk ut-knappen til uttrekkslyset slås på. Trykk på Start og vent til prøven dispenseres i den venstre (tomme) sprøyten.
- Trykk på Stopp-knappen like før prøven (høyre) sprøyten er helt tom. Registrer det dispenserte volumet og bruk det til å programmere ekstruderingssyklusen. Hold nede Rate-knappen til fase 1 (PH:01) vises på skjermen. Trykk på Volum-knappen for å angi det dispenserte volumet som ble registrert tidligere. I denne fasen må du sørge for at uttrekkslyset er av – dette dispenserer prøven i riktig retning.
- Trykk på Rate-knappen igjen og bruk pil lengst til høyre for å få tilgang til fase 2 (PH:02). Trykk på Volum for å angi samme verdi for det dispenserte volumet som ble registrert tidligere. I denne fasen trykker du på Trekk ut-knappen til uttrekkslyset er på – dette dispenserer prøven til venstre.
- For å gjenta denne syklusen, trykk på Rate-knappen igjen og bruk pil lengst til høyre for å få tilgang til fase 3 (PH:03). Trykk på Volum-knappen til LP:SE vises på skjermen og sett den til 20. Dette er antall løkker eller repetisjoner som pumpen vil utføre. Til slutt trykker du på Hastighet-knappen, tilgangsfase 4 (PH: 04) og trykker på Volum-knappen for å komme til Stopp-funksjonen. Pumpen er nå satt opp for automatisert ekstrudering.
- Trykk på Start for å starte ekstruderingssyklusen.
- Tøm sprøyten som inneholder den ekstruderte lipidfjæringen i et rent hetteglass og forbered lagring eller målinger. For lipidprøver med høy smeltetemperatur, oppbevar prøven over væskefaseovergangen til den måles. Ellers må du oppbevare prøver ved romtemperatur.
- Ikke frys ekstruderte prøver, da frysing vil føre til at vesiklene brister (suspensjonen blir melkehvit igjen).
4. NSE-målinger for utvalg(er) og reduksjon av innsamlede data
- Før NSE-eksperimentet karakteriserer du den ekstruderte liposomale prøven fra trinn 3.7 ved hjelp av tilgjengelige metoder for å sikre tilstrekkelig prøvekvalitet. En liste over potensielle kariseriseringsmetoder som kan brukes til å vurdere kvaliteten på liposomale suspensjoner for NSE-eksperimenter, for eksempel størrelsesfordeling, multilamellaritet, lateral membranstruktur, er inkludert i diskusjonsdelen.
- Bestem Q-området og tilsvarende instrumentinnstillinger som kreves for eksperimentet. For bøying av stivhetsmålinger av lipidbilayers, bruk et Q-område på ~(0,04 - 0,2) Å-1. For studier av membrantykkelsessvingninger, bruk et Q-område på ~(0,04 - 0,2) Å-1 som tilsvarer membrantykkelsen35,66,67.
MERK: Diskuter det eksperimentelle oppsettet med instrumentforskeren før eksperimentstart. Som nevnt tidligere er SANS-karakterisering av prøven nødvendig, spesielt hvis tidligere informasjon om spredningssignalet ikke er tilgjengelig, som i selektivt deutererte membraner. Alternativt kan du kjøre statiske (også kjent som diffraksjon) målinger over et begrenset Q-område på NSE-instrumentet, med forbehold om at slike målinger tar mye lengre tid sammenlignet med SANS. - Bruk en sprøyte eller en overføringspipette til å laste de ekstruderte liposomale suspensjonene i de angitte prøvecellene som er tilgjengelige ved NSE-bjelker. Vær oppmerksom på at standard NSE-prøveceller har tykkelser på 1, 2, 3 og 4 mm. Velg celletykkelsen på en slik måte at du optimaliserer spredningssignalet samtidig som det usammenhengende bakgrunnssignalet holdes til en rimelig intensitet.
MERK: Som en tommelfingerregel, bruk prøveceller med 1 eller 2 mm banelengde for protierte liposomer i deutert buffer - tykkere celler kan resultere i flere spredningseffekter som er vanskelige å rette opp for. For liposomer med høyere nivåer av deuterasjoner (f.eks. halekontrasttilpassede liposomer eller asymemetriske liposomer med enkle protierte brosjyrer), bør du vurdere å bruke en tykkere prøvecelle (f.eks. 3 eller 4 mm banelengde) for å forbedre tellestatistikken hvis prøven er tilgjengelig i større mengder - noen ganger kan dette være kostnadsforbudende. - Klargjør en identisk eksempelcelle for bufferen. Bruk samme buffer som i liposomal suspensjon. Målinger på bufferen er nødvendige for intensitetsrealisering og bakgrunnskorrigering (BKG).
- Plasser eksempelcellen(e) i prøveholderen på NSE-spektrometeret, programmer målingene og samle inn ekkodata. Rådfør deg med instrumentforskeren om programmering av målingene hvis en førstegangs NSE-bruker.
- Utfør ytterligere to sett med målinger som er nødvendige for datareduksjonen: Mål for oppløsning (R) og overføring (T).
- Utfør oppløsningsmåling (R) på en elastisk spredningsreferanse (f.eks. karbon) – som skal kjøres under de samme innstillingene. det vil si samme wavevector og Fourier ganger som prøve- og buffermålingene.
- Utfør overføringsmålinger (T) på prøven og bufferen for å beregne intensiteten til den overførte nøytronstrålen (se trinn 4.9. nedenfor). Overføringen beregnes som forholdet mellom nøytrontall fra prøven eller bufferen delt på nøytronantallet for en åpen stråle (dvs. med en tom prøveposisjon).
- Bruk den dedikerte datareduksjonsprogramvaren for NSE-spektrometeret som målingene utføres på for å redusere de innsamlede dataene.
MERK: Ulike spektrometre kan bruke annen programvare eller brukergrensesnitt. Nedenfor er et eksempel på reduksjon av NSE-data ved hjelp av dataanalyse- og visualiseringsmiljøet (DAVE)68 programvare spesielt skrevet for NSE spektrometeret ved NIST Center for Neutron Research.- Åpne DAVE-programvaren og velg Reduser NSE-data fra datareduksjonsmenyen. Flere popup-vinduer vises.
- Last opp datafilene over forskjellige Q-verdier ved hjelp av Åpne .echo-filer fra filmenyen. Disse filene tilsvarer rå datafiler med spin echo signaler og har filtypen .echo i filnavnet. Når filopplastingen er fullført, vises filene under de tilgjengelige datasettene.
- Høyreklikk på den valgte filen og merk den i henhold til målingen den tilsvarer; eksempel, celle (for tom celle eller buffer) eller oppløsning.
- Grupper detektor-pixlene i 2 x 2 for å forbedre signal-til-støy-forholdet ved hjelp av datasettfanen. Bruk den samme binning på alle filer. det vil si Oppløsning, Celle og Eksempel.
- Inspiser dataene over alle pikselgrupper og masker dem med dårlige signaler (se figur 4B) ved å trykke på m-tasten på tastaturet. Trykk ENTER for å få tilgang til et popup-vindu for å bruke den samme masken på alle Fire ganger eller senere Fire ganger. Dette kan også brukes på enkeltpiksler når som helst under datareduksjon. Maskerte bildepunkter blir grønne.
- Forsikre deg om at de innsamlede dataene er i form av et ekkosignal, det vil si cosinusfunksjon når det gjelder fasestrømmen, over hver detektorpiksel (se figur 4A).
MERK: Fasestrømmen er proporsjonal med presesjonsvinkelen til nøytronspinningen; Derfor er det vanlig å representere fasestrømmen som en fasevinkel som vist i figur 4A. For målinger på pulserende kilder brukes ekstra tid på flyberegninger på dataene for å oppnå ekkosignalene som en funksjon av hendelse nøytronbølgelengde i en nøytronpuls. - Begynn med å tilpasse oppløsningsfilen. Velg en oppløsningsfil fra listen over opplastede filer, og høyreklikk filen. Velg Tilpass operasjoner: Tilpass ekko (oppløsning) fra hurtigmenyen.
- Kontroller at signalenes anfall gir en rekke tilpasningsparametere, inkludert parameteren A, som kreves i trinn 4.8. Tilpassingene utføres automatisk ved hjelp av følgende uttrykk.

Her er ζ perioden for ekkosignalet (dvs. cosinusfunksjon i figur 4A), σ er bredden på den gaussiske konvolutten bestemt av gjennomsnittlig bølgelengde og bølgelengde spredning av hendelsen nøytronstråle, Φc er fasestrømmen, og Φ0 er ekkopunktet som avhenger av feltbanen som oppleves av nøytroner50. Fysisk informasjon om prøven er kodet i amplitude, A, av cosinusfunksjonen i ligningen (1).
MERK: Bredden på den gaussiske konvolutten er basert på verdier som er forhåndsbestemt av instrumentforskeren og bør ikke endres. De andre paramters er variabler som er montert på det spesifikke ekkosignalet over hver piksel. - Inspiser tilpasningsresultatene ved å klikke på hver piksel for å vise de resulterende tilpasningsparametrene, kvaliteten på passformen og det gjennomsnittlige kvadratiske avviket for passformen. Hvis du vil undersøke feilen som er knyttet til hver monteringsparameter over hele detektoren, velger du Bildealternativer og velger deretter interesseparameteren for tilpasning. Dette vil generere et kart med verdien av tilpasningsparamteren over hver piksel. Høyreklikk på detektorbildet. Det vises et popup-vindu som viser et feilfelttilordning for den valgte tilpasningsparameteren.
- Hvis passformen over en bestemt piksel er utilfredsstillende (f.eks. montere parameetere med store feilfelt), må du montere signalet på nytt over den bestemte pikselen. Merk bildepunktet, trykk kategorien Tilpassing , og trykk deretter Tilpass bildepunkt. Angi nye startparametere for fasen (Φ0) og perioden (ζ) i kategorien Montering for å få en mer tilfredsstillende passform.
MERK: Det er nyttig å plotte den monterte fasen som en funksjon av Fourier Time. Dette gjør du ved å gå til hovedtegningsvinduet og velge Tilpass fase v. Fourier Time. Denne tomten skal være jevn og kontinuerlig. Inspiser diskontinuiteter i dette plottet, og monter pikslene de tilsvarer, på nytt.
- Reduser eksempel- eller cellefilen ved å velge den tilsvarende filen fra listen over opplastede og merkede filer.
- Inspiser alle piksler og masker de med dårlig statistikk som beskrevet i trinn 4.7.5.
- Høyreklikk filen, og velg Tilpass operasjoner: Importfaser (eksempel, Celle). Dette importerer fasene og den brukte masken fra oppløsningsfilen.
- Monter ekkosignalene ved hjelp av samme prosedyre som er beskrevet tidligere for løsningsfilen (trinn 4.7.8-4.7.10). Når du tilpasser eksempel- og cellefilene, må du ikke endre verdiene for punktum- og ekkofasepunktet som importeres fra oppløsningstilpassingene. Disse parametrene avhenger av instrumentelle innstillinger og bør ikke variere med prøver.
- Før du går videre til datareduksjon, skriv inn strålesenteret for alle datafiler. Velg datafilen, gå til kategorien Generelt og skriv inn X- og Y-strålesenterverdier. Disse verdiene registreres under eksperimentet.
- Når tilpassingene til prøve-, celle- og oppløsningsfilene er fullført, beregner du den normaliserte mellomliggende spredningsfunksjonen som skal brukes senere i dataanalyse og tolkning. For å gjøre det, høyreklikk på eksempelfilen som skal reduseres fra listen over monterte filer, og velg Beregn I(Q) fra hurtigmenyen. Det vises et vindu med inngangsvalg for filene Oppløsning og Celle (dvs. buffer) og antall Q-buer (se trinn 4.9). Når du har skrevet inn all nødvendig informasjon, trykker du på OK-knappen. Resultatene vises i et nytt vindu.
MERK: Datareduksjonen utføres i henhold til følgende ligning for å oppnå den normaliserte mellomliggende spredningsfunksjonen69.
der t er Fourier-tid, N opp og Nned er nøytronantallet i ikke-spin-flip- og spin-flip-konfigurasjonene (målt med π/2-flippers av og π-flipper av og på, henholdsvis), og hevet skrift, BKG og R, tilsvarer henholdsvis bakgrunns- og oppløsningsmålingene, som definert i trinn 4.4 og 4.6. Legg merke til at strålepolariseringen , og dermed endres i spinntilstanden på grunn av energiutveksling mellom nøytronet og prøven oppdages som en dråpe i polariseringen (fra enhet).
, og dermed endres i spinntilstanden på grunn av energiutveksling mellom nøytronet og prøven oppdages som en dråpe i polariseringen (fra enhet).
- Til slutt grupperer du detektorpikslene i Q-buersom vist i figur 4B for å oppnå Q-avhengighetentil den normaliserte mellomliggende spredningsfunksjonen, S(Q,t) / S(Q,0). Dette kalles teknisk sett data binning og bør gjøres med omhu, det vil si med tanke på tellestatistikken fra utvalget og det forventede standardavviket for dataene over de grupperte bildepunktene.
- For sterkt spredte prøver, del detektoren i flere Q-buer samtidig som du opprettholder rimelige feilfelt på den resulterende mellomliggende spredningsfunksjonen, S(Q,t) / S(Q,0). Dette gir flere Q-datapunkter og er viktig for dataanalyseprosedyren beskrevet nedenfor. Vær oppmerksom på at for svakt spredning av prøver resulterer overdreven binning i dårlige forfallssignaler, det vil si store feilfelt på S(Q,t) / S(Q,0), noe som kan føre til store usikkerheter.
5. Dataanalyse og tolkning
- Tilpass de normaliserte mellomliggende spredningsfunksjonene S(Q,t) / S(Q,0), hentet fra datareduksjonen ovenfor til en strukket eksponentiell funksjon med en strekkeksponent på 2/370.

MERK: Et eksempel på disse anfallene finnes i figur 5B. Fits of S(Q,t) / S(Q,0) til ligning (3) gir Q-avhengige avslapningshastigheter Γ(Q). - Plott Γ(Q) som en funksjon av Q og passer til en passende modell for å trekke ut relevante membranparametere.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
NSE-studier som får tilgang til bøyesvingninger utføres vanligvis over et Q-område på ~ (0,04 - 0,2) Å-1. Dette Q-området tilsvarer mellomliggende lengdeskalaer mellom membrantykkelsen og liposomal radius, hvor bøyedynamikk dominerer. Måling over et utvidet Q-område kan gi tilgang til flere dynamiske moduser, inkludert liposomal diffusjon og intramembrane dynamikk. Hvis du vil ha mer informasjon om krysset i membrandynamikken som NSE har tilgang til, kan du se disse relevante publikasjonene25,71. Det er viktig å understreke at NSE-signaler er proporsjonale med:  , hvor jegcoh og jeginc er henholdsvis den sammenhengende og usammenhengende spredningsintensiteten fra prøven. Derfor anbefales det å forberede NSE liposomale prøver i deutererte buffere (dvs. buffere utarbeidet med D2O i stedet for H2O) for å minimere det usammenhengende spredningssignalet, hovedsakelig bidratt av hydrogeninnholdet i prøven. I noen tilfeller kan imidlertid mellomliggende deuteringsordninger (dvs. bruk av blandinger av D2O og H2O) være nødvendig for å oppnå optimale kontrastforhold. Vanligvis utføres NSE-målinger av membranbøyende svingninger på fullt protierte liposomer i deutert buffer, referert til som fullt kontrasterte liposomer i figur 5. Denne deuterasjonsordningen resulterer i en stor NSLD-forskjell mellom membrankjernen (~ 0 × 10-6 Å-2) og dets deutererte væskemiljø (~ 6,4 × 10-6 Å-2), noe som forbedrer spredningssignalet fra liposomale membraner betydelig og forbedrer målestatistikken for bøyedynamikk. Denne kontrastordningen (figur 2A venstre panel) brukes ofte i studier av bøyningsstivhet av lipidmembraner med enkelt38,72 og flere39,66 lipidkomponenter og i studier av membranmykning/stivning ved biologiske inneslutninger (f.eks. kolesterol, legemiddelmolekyler, peptider/proteiner)36,37,73,74,75og syntetiske tilsetningsstoffer (f.eks. nanopartikler)76,77.
, hvor jegcoh og jeginc er henholdsvis den sammenhengende og usammenhengende spredningsintensiteten fra prøven. Derfor anbefales det å forberede NSE liposomale prøver i deutererte buffere (dvs. buffere utarbeidet med D2O i stedet for H2O) for å minimere det usammenhengende spredningssignalet, hovedsakelig bidratt av hydrogeninnholdet i prøven. I noen tilfeller kan imidlertid mellomliggende deuteringsordninger (dvs. bruk av blandinger av D2O og H2O) være nødvendig for å oppnå optimale kontrastforhold. Vanligvis utføres NSE-målinger av membranbøyende svingninger på fullt protierte liposomer i deutert buffer, referert til som fullt kontrasterte liposomer i figur 5. Denne deuterasjonsordningen resulterer i en stor NSLD-forskjell mellom membrankjernen (~ 0 × 10-6 Å-2) og dets deutererte væskemiljø (~ 6,4 × 10-6 Å-2), noe som forbedrer spredningssignalet fra liposomale membraner betydelig og forbedrer målestatistikken for bøyedynamikk. Denne kontrastordningen (figur 2A venstre panel) brukes ofte i studier av bøyningsstivhet av lipidmembraner med enkelt38,72 og flere39,66 lipidkomponenter og i studier av membranmykning/stivning ved biologiske inneslutninger (f.eks. kolesterol, legemiddelmolekyler, peptider/proteiner)36,37,73,74,75og syntetiske tilsetningsstoffer (f.eks. nanopartikler)76,77.
Målinger av bøyesvingninger resulterer i avslapningshastigheter som følger en Q3-avhengighet, som spådd av Zilman og Granek for termisk bølgende elastiske tynne ark70. En raffinert form for denne Q-avhengigheten er hentet fra teoretiske korreksjoner av Watson og Brown78, som tar hensyn til effekten av intermonolayerfriksjon foreslått av Seifert og Langer79. Ved i tillegg å definere det nøytrale planet til å være i grensesnittet mellom de hydrofile hodegruppene og membranens hydrofobe haler, kan bøyeavslappingsratene deretter monteres på følgende uttrykk38.

der ηbuff er bufferviskositeten, er kBT den termiske energien, κ og er bøyestivheten til den målte membranen (eller av den kontrasterte delen av membranen i selektivt deuterte systemer). Denne typen måling muliggjør direkte beregning av membran elastiske egenskaper i form av bøyestivhet modulus. Legg merke til at κ trekkes ut fra hellingen til den lineære tilpasningen til Γ vs. Q3, som vist i figur 5C.
På den annen side viser NSE-målinger av svingninger i membrantykkelse avvik fra Q3-avhengigheten i Γ(Q) rundt Q-verdier som tilsvarer membrantykkelsen (se figur 2 i ref.66). For å isolere tykkelsesvariasjonssignalet kan man dele Γ(Q) med Q3, som vist i figur 5D. De resulterende dataene viser at overflødig dynamikk på grunn av tykkelsessvingninger følger en Lorentzian-funksjon i Q, som nylig bekreftet i grovkornede molekylære dynamikk (MD)simuleringer 67. For å passe til den observerte overskytende dynamikken utviklet Nagao et al.38 et uttrykk basert på det teoretiske rammeverket for membransvingninger av Bingham et al.80 som følger.

I dette uttrykket er Q0 den høyeste Q-verdiensom tilsvarer membrantykkelsen (som kan oppnås uavhengig av SANS-målinger), μ er in-plane membranviskositeten, AL er området per lipid (målt med SANS / SAXS), og KA er området compressibility modulus. Forutsatt at KA kan beregnes fra κ ved hjelp av polymerbørstemodellen, reduseres dette uttrykket til en passformparameter, nemlig membranviskositeten μ, og presenterer en ny tilnærming for å måle membranviskositet uten behov for fluorescensmerking eller partikkeltethering / sporing13. Forutsetningen er at i henhold til deformasjonsmodeller av elastiske tynne ark81, κ og KA er gjensidig avhengige slik at: ,  hvor tm er den mekaniske (eller deformerbare) membrantykkelsen og β er en konstant som beskriver interleafletkobling. Forutsetningen er at β = 12 for fullt koblede brosjyrer, β = 48 for helt usammenhengende brosjyrer, og β = 24 for mellomliggende koblede brosjyrer. Sistnevnte kalles polymerbørstemodellen81 og har vist seg å gjelde i enkeltkomponent- og binærvæskelipidmembraner39. Dette må imidlertid nærmer seg med forsiktighet. For eksempel nylige simuleringer av Doktorova et al. 82 viste at for at polymerbørstemodellen skal holde i umettede lipidmembraner som inneholder kolesterol, må et modifisert uttrykk for den mekaniske membrantykkelsen brukes. Ideelt sett, hvis en uavhengig måling av KA er mulig, for eksempel ved hjelp av mikropipetteaspirasjon83, vil det å kombinere KA-resultater med NSE-bøyestivhetsmålinger gi en unik mulighet til å undersøke interleafletkobling i modell- og biologiske membraner - et langvarig spørsmål i membranbiofysikk og strukturell biologi. Når verdiene til KA er validert, kan de brukes i ligning 5 for å oppnå mesoskopisk membranviskositet.
hvor tm er den mekaniske (eller deformerbare) membrantykkelsen og β er en konstant som beskriver interleafletkobling. Forutsetningen er at β = 12 for fullt koblede brosjyrer, β = 48 for helt usammenhengende brosjyrer, og β = 24 for mellomliggende koblede brosjyrer. Sistnevnte kalles polymerbørstemodellen81 og har vist seg å gjelde i enkeltkomponent- og binærvæskelipidmembraner39. Dette må imidlertid nærmer seg med forsiktighet. For eksempel nylige simuleringer av Doktorova et al. 82 viste at for at polymerbørstemodellen skal holde i umettede lipidmembraner som inneholder kolesterol, må et modifisert uttrykk for den mekaniske membrantykkelsen brukes. Ideelt sett, hvis en uavhengig måling av KA er mulig, for eksempel ved hjelp av mikropipetteaspirasjon83, vil det å kombinere KA-resultater med NSE-bøyestivhetsmålinger gi en unik mulighet til å undersøke interleafletkobling i modell- og biologiske membraner - et langvarig spørsmål i membranbiofysikk og strukturell biologi. Når verdiene til KA er validert, kan de brukes i ligning 5 for å oppnå mesoskopisk membranviskositet.

Figur 1: NSE instrumentdesign og synergistisk overlapping med lengde/tidsskalaer av mesoskopisk membrandynamikk. (A) Skjematisk for de ulike magnetiske elementene i et NSE-instrument, som brukes til å manipulere spinnet av nøytroner som krysser instrumentet fra venstre til høyre. Det markerte nøytronet indikerer endring i spinnorientering (eller polariseringstap) på grunn av energiutveksling mellom nøytronet og prøven, mens det gjennomsiktige nøytronet representerer spin-echo, det vil si ingen endring i nøytronspinningen på grunn av null energiutveksling. Den grå pilen indikerer muligheten for å rotere den andre armen på spektrometeret for å få tilgang til større spredningsvinkler. (B) Billedlig representasjon av hierarkisk dynamikk i lipidmembraner, som viser ulike dynamiske moduser som strekker seg over flere lengde- og tidsskalaer. Det skyggelagte området representerer lengden og tidsskalaene som NSE har tilgang til, som overlapper med mesoskalaene av kollektive membransvingninger, nemlig bøyning og tykkelsessvingninger. Klikk her for å se en større versjon av denne figuren.

Figur 2: Eksempler på mulige deuteringsordninger i NSE-eksperimenter på lipidmembraner. (A) Venstre: Fullt kontrasterte membraner, for eksempel protierte membraner i deuterert buffer, som viser NSLD-profilen langs normalen til membranoverflaten. Forskjellen i NSLD mellom haleområdet (~0 × 10-2 Å-2) og hodegruppeområdet (~4,5 × 10-6 Å-2) i membranen skyldes hodegruppehydrering med deutert buffer. Høyre: Halekontrasttilpassede membraner slik at hydrokarbonhaleområdet i membranen har samme NSLD som bufferen, som vist i den tilsvarende NSLD-profilen langs membranens normale. (B) Domenedannende membraner med to nøytronkontrastordninger der domenene (midten) eller matrisen (til venstre) er kontrasttilpasset bufferen, noe som muliggjør selektive studier av henholdsvis matrise- eller domenedynamikk. Dette tallet er endret fra Nickels et al., JACS 201541. (C) Asymmetriske membraner fremstilt av cyclodextrin utveksling mellom protiated og deuterated lipid vesicles, noe som resulterer i deutering av en membran brosjyre mens du holder den andre brosjyren protiated. Dette gjør det mulig å studere bøyedynamikken til det protierte pakningsvedlegget og gir innsikt i den mekaniske koblingen mellom motstridende brosjyrer i asymmetriske membraner. Denne figuren er endret fra Rickeard et al., Nanoscale 202040. Klikk her for å se en større versjon av denne figuren.

Figur 3: Illustrasjon av oppsettet for automatisert ekstrudering av liposomer. (A) Spesialbygd automatisert ekstruder ved hjelp av en sprøytepumpe, et miniekstrudersett og en aluminiums-/stålramme for å muliggjøre sykliske profiler. (B) og (C) viser forskjellen i visuelt utseende av lipid suspensjoner før (melkehvit) og etter (gjennomsiktig opalblå) ekstrudering. Dette skyldes den første dannelsen av mikron-størrelse lipid stabler eller gigantiske vesicles som er på størrelse med, eller større enn, bølgelengden av synlig lys. Etter ekstrudering vil suspensjonen bestå av nanoskopiske vesicles (~ 100 nm), som er mindre enn bølgelengden av synlig lys, noe som gir en gjennomsiktig suspensjon. Klikk her for å se en større versjon av denne figuren.

Figur 4: Representative data fra NSE-eksperimenter på liposomale suspensjoner. (A) Eksempel på ekkosignal over en enkelt detektorpiksel (merket piksel i panel B), som viser passer til ekkosignalet ved hjelp av ligning (1), med en illustrasjon av de forskjellige parametrene som kreves i ekkotilpasningen. Vær oppmerksom på at ekkosignalet er plottet inn som en funksjon av fasevinkelen i stedet for fasestrømmen som beskrevet i trinn 4.7 i protokollen. (B) NSE-detektorbilde som viser variasjonen i nøytronantall per piksel. Bildet viser også eliminerte detektorpiksler (grønn) på grunn av dårlige ekkosignaler. Binning av detektorpikslene i Q-buer (også kjent som Debye-Scherrer-ringer) gir Q-avhengigheten til den mellomliggende spredningsfunksjonen, som trengs for å analysere og tolke NSE-data. Denne figuren ble endret fra Ashkar, J. Appl. Phys. 202050. Klikk her for å se en større versjon av denne figuren.

Figur 5: Representative resultater fra NSE-eksperimenter på liposomale suspensjoner med ulike deuterasjonsordninger. (A) Spredningsgeometri av en nøytron som samhandler med et liposom, som viser spredningsvinkelen, 2θog wavevector-overføringen,  . (B) Mellomliggende spredningsfunksjoner, S(Q,t) / S(Q,0), viser forfall som en funksjon av Fourier-tiden. Passformen til de målte forfallene til en strukket eksponentiell funksjon gitt ved ligning 3 gir avslapningsraten, Γ(Q). (C) For fullstendig protierte liposomer i deutert buffer følger Γ(Q) en Q3-avhengighet, typisk for bøyedynamikk. Den lineære passformen til de oppnådde dataene til en Zilman-Granek-modell gir den bøyende stivhetsmodulus av membranen. (D) For tail deuterated liposomer observeres overflødig dynamikk i tillegg til bøyesvingninger og er mest uttalt ved Q-verdier som tilsvarer membrantykkelsen. Montering av overflødig dynamikk til en lorentzisk funksjon (ligning 5) tillater utvinning av membranviskositeten. Datasett ble samlet inn på NSE-spektrometeret ved NIST. Klikk her for å se en større versjon av denne figuren.
. (B) Mellomliggende spredningsfunksjoner, S(Q,t) / S(Q,0), viser forfall som en funksjon av Fourier-tiden. Passformen til de målte forfallene til en strukket eksponentiell funksjon gitt ved ligning 3 gir avslapningsraten, Γ(Q). (C) For fullstendig protierte liposomer i deutert buffer følger Γ(Q) en Q3-avhengighet, typisk for bøyedynamikk. Den lineære passformen til de oppnådde dataene til en Zilman-Granek-modell gir den bøyende stivhetsmodulus av membranen. (D) For tail deuterated liposomer observeres overflødig dynamikk i tillegg til bøyesvingninger og er mest uttalt ved Q-verdier som tilsvarer membrantykkelsen. Montering av overflødig dynamikk til en lorentzisk funksjon (ligning 5) tillater utvinning av membranviskositeten. Datasett ble samlet inn på NSE-spektrometeret ved NIST. Klikk her for å se en større versjon av denne figuren.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
NSE er en kraftig og unik teknikk for å måle mesoskopisk dynamikk i lipidmembraner under ulike forhold. Den effektive utnyttelsen av NSE avhenger av prøvekvalitet, nøytronkontrast og utvalget av tilgjengelig dynamikk som kan undersøkes for en gitt prøve. Dermed kreves det flere kritiske trinn for å utføre vellykkede NSE-eksperimenter og samle inn data av høy kvalitet. Et viktig skritt for å sikre effektiv bruk av nøytronstråletid under et NSE-eksperiment er å karakterisere liposomale suspensjoner med laboratoriebaserte metoder før NSE-eksperimentet. For eksmappel kan størrelsesfordelingen (eller diffusjonskonstanten) av ekstruderte liposomer bestemmes av dynamisk lysspredning (DLS), lett tilgjengelig i individuelle laboratorier eller i delte fasiliteter84. Kryo-elektronmikroskopi er en annen karismaeriseringsmetode som nylig ble validert på liposomale prøver, der mikroskopibilder med høy oppløsning på kryokrotomerte deler av liposomal suspensjon effektivt kan brukes til å undersøke liposomal unilamellarity65, domenedannelse85,86eller inkorporering av tilsetningsstoffer som nanopartikler76 og proteiner87. Alternativt kan småvinklet røntgenspredning (SAXS) brukes til å karakterisere membranstrukturen88, vurdere liposomal multilamellaritet65, eller vurdere effekten av tilsetningsstoffer på membranstrukturelle egenskaper89. Foruten disse lab-baserte teknikkene, er det sterkt tilrådelig at NSE-målinger på liposomale prøver er forbundet med strukturelle studier ved hjelp av småvinklet nøytronspredning (SANS)54,90. SANS er et utmerket supplement til NSE, ikke bare for å skaffe strukturell membraninformasjon, men også for å undersøke intensiteten av nøytronspredningssignalet fra prøven, bekrefte kontrastordningen og gjøre et informert valg om Q-området som NSE-målingene skal utføres over. Derfor anbefales det at NSE-brukere ber om SANS-stråletid ved søknad om NSE-eksperimenter.
NSE lider imidlertid av prøvebegrensninger i studier av biologiske membraner. En av de viktigste begrensende faktorene for slike eksperimenter er standard mengde prøve som kreves for NSE-målinger (2-4 ml) og de høye prøvekonsentrasjonene som utgjør 100-200 mg membranmateriale (lipider og proteiner) for å oppnå data av høy kvalitet. I mange tilfeller er produksjonen av slike mengder biologisk materiale ikke mulig eller er kostnadsforbudende. I slike scenarier er det mulig å redusere konsentrasjonen til 20-25 mg / ml, men dette vil kreve minst en 4 ganger økning i oppkjøpstiden for å oppnå sammenlignbar statistikk til prøver med 50 mg / ml konsentrasjoner. Disse strenge kravene til prøvevolum og konsentrasjon kan lindres med neste generasjon NSE-spektrometre på høyere fluks nøytronkilder, for eksempel den andre målstasjonen ved Oak Ridge National Lab og European Spallation Source. En annen kritisk begrensning i å utføre NSE-eksperimenter på lipidmembraner som krever selektive deuteringsordninger, er mangelen på kommersiell tilgjengelighet av noen deutererte varianter av lipidmolekyler eller deres ublu priser, hvis tilgjengelig. I noen tilfeller kan disse begrensningene omgås ved å be om syntese av deutererte lipider (eller kolesterol, proteiner) gjennom brukerdeuteringsanlegg, for eksempel biodeuteringslaboratoriet ved Oak Ridge National Lab, det nasjonale deuteringsanlegget ved ANSTO, eller deuterasjonsanlegget ved ISIS Neutron og Muon Source. Tilgang til disse fasilitetene og deres syntesefunksjoner er tilgjengelig gjennom innsendte brukerforslag som er fagfellevurdert basert på den vitenskapelige verdien av den foreslåtte materialsyntesen og dens tiltenkte bruk i isotopfølsomme studier.
Til tross for disse begrensningene har anvendelsen av NSE-spektroskopi i studier av membranmekanikk ført til bestemmelse av bøyestivhetsmoduli av membraner med ulike grader av kompleksitet, fra enkeltkomponent lipidmembraner35,38 til multikomponent biomimetiske membraner41,66,91, som alle har avansert vår forståelse av lipidmembranenes dynamiske natur. For eksempel har NSE bøyestivhetsmålinger av lipidmembraner med forskjellige molekylære enheter, for eksempel lipider av forskjellige acylkjedelengder og kjedemetning38,72,92, gitt viktig informasjon om rollen som molekylær kjemi i membranmekanikk. Når disse målingene kombineres med strukturell informasjon, for eksempel membrantykkelse eller molekylær pakking93, begynner de å gi nye perspektiver på gjensidig avhengighet mellom membranstruktur og dynamikk og hvordan de påvirker membranfunksjonen. De mesoskopiske skalaene til NSE posisjonerer det unikt for slike grunnleggende undersøkelser av struktur-eiendomsrelasjoner, mest relevante på lengden av molekylære forsamlinger. Dette emnet ble nylig utforsket i to NSE-studier på kolesterolrike lipidmembraner36 og i binære lipidmembraner med hydrofob uoverensstemmelse mellom de to lipidkomponentene39. Begge studiene fant sterke bevis på at membranmekanikk skalerer med området per lipid, og bekrefter konklusjonene fra en nylig atomisk MD-simulering av Doktorova et al.82. Disse funnene understreker lipidmembranenes selvmonterte natur og gir et samlende bilde av molekylær pakking som en nøkkelparameter for å definere membrandynamikk og funksjonelle egenskaper.
Andre anvendelser av NSE innebærer studier av membranens mekaniske respons på små tilsetningsstoffer, inkludert biologiske molekyler som kolesterol36,37, trehalose92og melittin73,94eller uorganiske tilsetningsstoffer som nanopartikler for legemiddelleveringsapplikasjoner76. NSE har også blitt brukt til å forstå hvordan membranmekanikk reagerer på endringer i miljøet, inkludert temperatur92, pH74, og tilstedeværelsen av trengsel makromolekyler96. Slike studier bidrar til en bedre forståelse av faktorene som påvirker mykgjøring eller stivning av lipidmembraner, under biologiske forhold relatert til helse og sykdom, og i kontrollerte omgivelser for terapeutiske applikasjoner. Spesielt har NSE-målinger også blitt brukt til å sondere effekten av antimikrobielle peptider påmembrandynamikk 73,94,95. Ytterligere eksempler på NSE-applikasjoner på biomembraner inkluderer studier av dynamikken i flate membranstrukturer, kalt thylakoider, som huser fotosyntetiske maskineri i cyanobakterielle celler97,98.
Man kan også benytte selektiv lipiddeutering i NSE-studier for å undersøke dynamikken i spesifikke membranfunksjoner som er relevante for biologisk funksjon. For eksempel brukte Nickels et al. selektiv lipiddeutering i domenedannende lipidmembraner for å generere lateral kontrast i membranen, som tidligere illustrert av Heberle et al.28. Dette deuteringsskjemaet aktiverte uavhengige målinger av bøyestivheten til lipiddomenene og verts lipidmatrisen41 (se figur 2B). Funnene bekreftet at de to membranrommene har tydelig bøyestivhetsmoduli, noe som kan være en kjøremekanisme for domenedannelse i cellulære membraner. I en nyere studie brukte Rickeard et al. cyclodextrinutveksling mellom protierte og deutererte liposomer for å oppnå asymmetriske liposomer med isotopisk merkede brosjyrer40 (Figur 2C). Deres ende liposomer hadde et protiated brosjyre og et deutert brosjyre som er kontrast tilpasset bufferen, muliggjør studier av individuell brosjyredynamikk og gir en første direkte eksperimentell redegjørelse for effekten av asymmetri og brosjyrekobling på membranbøyende svingninger.
Selektiv membrandeutering har også blitt brukt i NSE-studier av membrantykkelsessvingninger, en lang anslått dynamisk modus i lipidmembraner99 som bare nylig ble observert med ankomsten av NSE-spektroskopi35,100. Disse målingene bruker hale-deutererte membraner for å forsterke signalet fra membranhodegruppeområdene og løse tykkelsesvariasjonssignalet. Denne typen NSE-eksperimenter er relativt nylig, men det har blitt effektivt brukt til å forstå gjensidig avhengighet av membran elastiske og viskøse egenskaper38, for å utforske skalering av bøyningsstivhet og viskositet med molekylær pakking i blandede lipidmembraner39, og for å undersøke de lokale effektene av kolesterol på membranviskositet36. Et annet område av biologisk betydning der denne dynamiske modusen kan ha vidtrekkende implikasjoner er mesoskopiske membranproteininteraksjoner95. Det er kjent at funksjonen til membranproteiner er tett knyttet til hydrofob matching mellom proteinet og vertsmembranen. Dermed kan variasjoner i membrantykkelse, på grunn av tykkelsesvariasjoner, fungere som en regulatorisk mekanisme for funksjonen av membranproteiner. NSE er ekstremt godt egnet for slike studier, da det direkte kan undersøke effekten av proteinbinding og innsetting på membrantykkelsessvingninger. Nylige NSE-målinger fra vår gruppe (upublisert) tyder på at transmembran proteininnsetting kan undertrykke svingninger i membrantykkelsen betydelig og kan utgjøre en potensiell mekanisme for regulering av signalhendelser. Dette er et presserende, men underutviklet forskningsområde der NSE kan ha betydelig innvirkning på forståelsen av membranenes dynamiske respons på proteinbinding og innsetting på lengde og tidsskalaer av viktige biologiske funksjoner formidlet av interaksjoner mellom proteiner og cellemembraner.
Oppsummert har NSE utviklet seg de siste årene som et kraftig verktøy for å forhøre membrandynamikk over romlige og tidsmessige skalaer av vitale biologiske funksjoner. Teknikken får raskt utbredt interesse, og potensialet i å svare på viktige spørsmål i membranfunksjonen blir godt anerkjent. Kontrastvariasjonsevnen i NSE har posisjonert den som en unik tilnærming for å måle mesoskopiske membranegenskaper som ellers ville være utfordrende å få tak i. En annen betydelig fordel med NSE fremfor tradisjonelle spektroskopimetoder i studier av membrandynamikk er overlappingen med lengden og tidsskalaene som er tilgjengelige med MD-simuleringer, noe som gjør det mulig for synergistiske eksperimentelle / beregningsstudier å få en molekylær nivåforståelse av de forskjellige molekylære komponentene som utgjør membraner. Til tross for løftet er det fortsatt noen begrensninger i bruken av NSE i biologiske membranstudier, inkludert kravet til store prøvevolumer, vanskeligheten med selektiv deutering i biologiske systemer, og den relativt lave nøytronfluksen på NSE-spektrometre, noe som resulterer i lengre måletider og begrenset stråletidstilgjengelighet. Imidlertid kan disse manglene overvinnes i nær fremtid med konstant utvikling i nøytronkilder og instrumentering sammen med fremskritt i deuterasjonsanlegg.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Forfatterne erklærer ingen interessekonflikter og har ingenting å avsløre.
Acknowledgments
R. Ashkar takker M. Nagao, L.-R. Stingaciu og P. Zolnierczuk for mange nyttige diskusjoner og for deres hyppige hjelp med NSE-eksperimenter på deres respektive bjelkelinjer. Forfatterne erkjenner bruken av nøytron spinn ekkospektrometre ved NIST og ORNL. NSE-spektrometeret ved NIST støttes av Center for High Resolution Neutron Scattering, et partnerskap mellom National Institute of Standards and Technology og National Science Foundation under avtalenr. DMR-1508249. NSE-spektrometeret ved ORNLs Spallation Neutron Source støttes av Scientific User Facilities Division, Office of Basic Energy Sciences, US Department of Energy. Oak Ridge National Laboratory administreres av UT-Battelle, LLC under US DOE Contract No. DE-AC05-00OR22725.
Materials
| Name | Company | Catalog Number | Comments |
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
References
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. Ø, Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes--a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich's model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a "modeling manifesto". Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D. Neutrons in Soft Matter. Imae, T., Kanaya, T., Furusaka, M., Torikai, N. , Wiley. ch6 147-182 (2011).
- Pynn, R. Neutron Spin Echo. Mezei, F., Pappas, C., Gutberlet, T. , Springer. Berlin Heidelberg. 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , Oxford University Press. (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- Neutron activation and scattering calculator. , Available from: https://www.ncnr.nist.gov/resources/activation/ (2021).
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , Walter de Gruyter, Inc. 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles - as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O'Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -R., O'Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).
Tags
Biologi utgave 171 kollektiv lipiddynamikk bøyemodulus områdekompresensitet tykkelsessvingninger membranviskositet selektiv deutering liposompreparatErratum
Formal Correction: Erratum: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions
Posted by JoVE Editors on 08/06/2021.
Citeable Link.
An erratum was issued for: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions. The Introduction, Protocol, and Representative Results sections have been updated.
In the Introduction, the fith pargraph was updated from:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 2 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
to:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 0 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
In the Protocol, step 1.1 was updated from:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 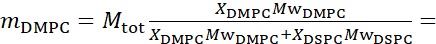 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
to:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 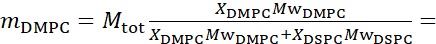 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
In the Representative Results, the fist pagargaph was updted from:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 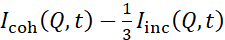 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
to:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 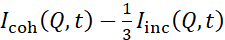 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
In the Representative Reults, Figure 2 was updated from:
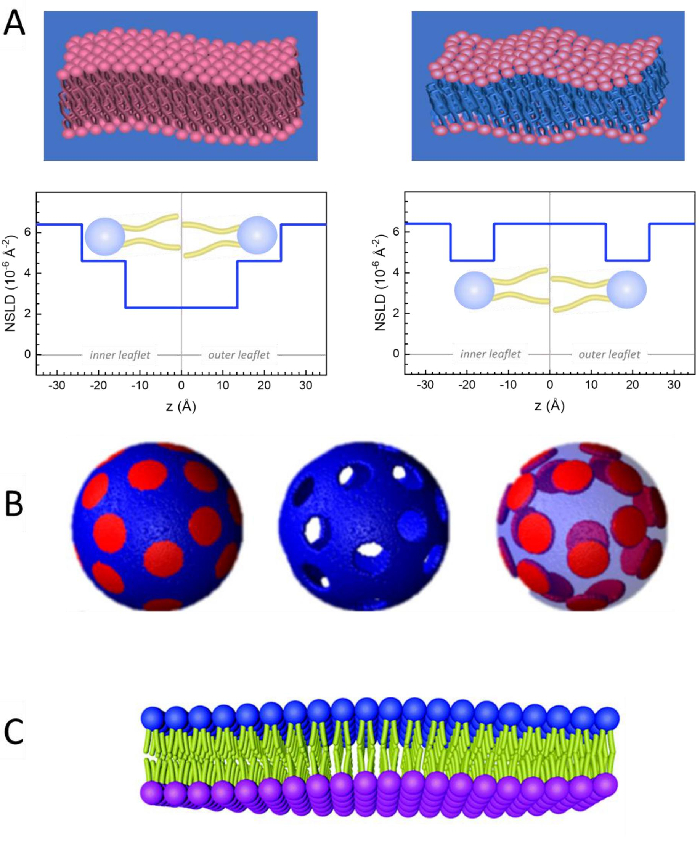
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the headgroup (~2 × 10-2 Å-2) and tail region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
to:
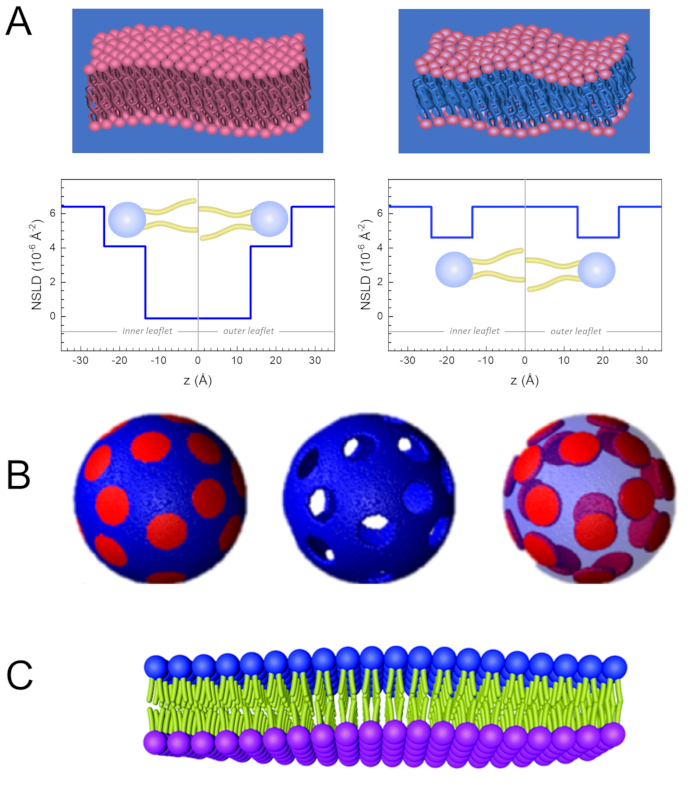
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the tail region (~0 × 10-2 Å-2) and headgroup region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.



 133,4 mg, der XDMPC og XDSPC er molefraksjonene av DMPC og DSPC, her satt til henholdsvis 0,7 og 0,3, og MwDMPC og MwDSPC er molarvektene gitt av henholdsvis 677,9 g/mol og 790,1 g/mol. Tilsvarende mDSPC = 66,6 mg. Denne deuteringsordningen øker spredningskontrasten mellom membranen (NSLD ~ 0 × 10-6 Å-2) og den deutererte bufferen (NSLD ~ 6,4 × 10-6 Å-2) og forsterker signalet fra membranunduleringer (se figur 2A venstre panel).
133,4 mg, der XDMPC og XDSPC er molefraksjonene av DMPC og DSPC, her satt til henholdsvis 0,7 og 0,3, og MwDMPC og MwDSPC er molarvektene gitt av henholdsvis 677,9 g/mol og 790,1 g/mol. Tilsvarende mDSPC = 66,6 mg. Denne deuteringsordningen øker spredningskontrasten mellom membranen (NSLD ~ 0 × 10-6 Å-2) og den deutererte bufferen (NSLD ~ 6,4 × 10-6 Å-2) og forsterker signalet fra membranunduleringer (se figur 2A venstre panel). hvorM-tot er den totale massen, satt her til 200 mg. Se eksemplet ovenfor for DMPC-DSPC lipid-bilayers med forskjellige deuterasjonsordninger.
hvorM-tot er den totale massen, satt her til 200 mg. Se eksemplet ovenfor for DMPC-DSPC lipid-bilayers med forskjellige deuterasjonsordninger.

 , og dermed endres i spinntilstanden på grunn av energiutveksling mellom nøytronet og prøven oppdages som en dråpe i polariseringen (fra enhet).
, og dermed endres i spinntilstanden på grunn av energiutveksling mellom nøytronet og prøven oppdages som en dråpe i polariseringen (fra enhet).
