Summary
פרוטוקול זה מתאר שיטה לא פולשנית לזיהוי יעיל של תאי S-phase למחקרי מיקרוסקופיה במורד הזרם, כגון מדידת גיוס חלבונים לתיקון דנ"א באמצעות הקרנת מיקרו-קרינה בלייזר.
Abstract
תיקון נזקי DNA שומר על השלמות הגנטית של תאים בסביבה תגובתית מאוד. תאים עשויים לצבור סוגים שונים של נזק DNA בשל מקורות אנדוגניים ואקסוגניים כגון פעילויות מטבוליות או קרינת UV. ללא תיקון DNA, הקוד הגנטי של התא הופך לפגום, מערער את המבנים והפונקציות של חלבונים ועלול לגרום למחלות.
הבנת הדינמיקה spatiotemporal של מסלולי תיקון DNA שונים בשלבי מחזור תאים שונים היא חיונית בתחום של תיקון נזק DNA. טכניקות מיקרוסקופיה פלואורסצנטיות נוכחיות מספקות כלים נהדרים למדידת קינטיקה של גיוס חלבונים שונים לאחר אינדוקציה נזק DNA. סינתזת DNA במהלך שלב S של מחזור התא היא נקודה מוזרה בגורל התא לגבי תיקון DNA. הוא מספק חלון ייחודי כדי לסנן את הגנום כולו לטעויות. יחד עם זאת, שגיאות סינתזת DNA גם להוות איום על שלמות ה- DNA כי הוא לא נתקל בתאים שאינם מתחלקים. לכן, תהליכי תיקון DNA שונים באופן משמעותי בשלב S בהשוואה לשלבים אחרים של מחזור התא, והבדלים אלה מובנים היטב.
הפרוטוקול הבא מתאר את הכנת קווי התא ואת מדידת הדינמיקה של חלבוני תיקון DNA בשלב S באתרי נזק לדנ"א הנגרמים באופן מקומי, באמצעות מיקרוסקופ קונפוקלי סורק לייזר המצויד בקו לייזר של 405 ננומטר. PCNA מתויג (עם mPlum) משמש כסמן מחזור תא בשילוב עם חלבון תיקון תווית AcGFP של עניין (כלומר, EXO1b) כדי למדוד את גיוס נזקי ה- DNA בשלב S.
Introduction
מספר מסלולי תיקון DNA התפתחו כדי לטפל בסוגים שונים של נגעי DNA שיכולים להתעורר בתאים, שכולם מוסדרים מאוד הן במרחב והן בזמן. אחת התקופות הפגיעות ביותר של מחזור התא היא שלב S, כאשר מתרחשת סינתזת DNA. בעוד שהתפשטות היא בסיסית לחיים, היא גם מספקת אתגר גדול. תאים צריכים להבטיח שכפול נאמן של הגנום שלהם כדי למנוע מוטציות שיועברו לדורות הבאים. כתוצאה מכך, ההתפשטות מספקת נקודת התערבות טיפולית שהופעלה לפיתוח גישות טיפוליות בתחום האונקולוגיה.
לכל הטכניקות העיקריות המשמשות לחקר גיוס חלבונים נגעי DNA יש את נקודות החוזק והמגבלות שלהם. מיקרו-הקרנה יש רזולוציה מרחבית וטמפורלית טובה יותר1 מאשר רוב השיטות החלופיות כמו הדמיה חיסונית של מוקדים מייננת הנגרמת על ידי קרינה (IRIF), כרומטין-אימונופרציפיטציה (ChIP), או פירוק ביוכימי. עם זאת, מיקרו-הקרנה משתליבה את החוסן של הטכניקות הנ"ל שיכולות לדגום מספר גדול של תאים בו זמנית.
כדי לחקור תיקון DNA בשלב S, יש להיות מסוגל להבחין בין תאי פאזה S באוכלוסיית תרבית תאים אסינכרונית. קיימות שיטות ידועות רבות לטיפול בכך, הכוללות סינכרון תאים או הדמיה של שלבי מחזור התא השונים. עם זאת, שתי הגישות מציגות אתגרים משמעותיים וחפצים אפשריים. שיטות סנכרון כימיות בשימוש נרחב כדי להעשיר תאים בשלב S מוקדם (למשל, בלוק תימידין כפול, aphidicolin, וטיפול hydroxyurea) להשיג סנכרון באמצעות אינדוקציה של מתח שכפול ובסופו של דבר נזק DNA עצמו. זה מגביל את השימוש בשיטות אלה כדי ללמוד תהליכי תיקון DNA בשלבS 2. סנכרון באמצעות רעב בסרום ושחרור חל רק על מספר מוגבל של קווי תאים, בעיקר למעט קווי תאים סרטניים המסתמכים פחות על גורמי גדילה להתקדמות מחזור התא בהשוואה לקווי תאים שאינם שעברו שינוי. מערכת מחוון מחזור תא אוביקוויטין פלואורסצנטין (FUCCI) היא כלי שימושי במיוחד לחקר מחזור התא, אך יש לה מגבלה בסיסית בעת הבחנה בין S ו- G2 שלבים מחזור התא3.
כאן הוא מוצג כי באמצעות PCNA מתויג פלואורסצנטית כסמן לא פולשני עבור שלב S מגביל את החסרונות של שיטות סינכרון מחזור תאים כימיים, תוך מתן אפשרות יותר ספציפיות וגמישות מאשר מערכת FUCCI. כסמן יחיד, לא רק PCNA יכול להדגיש תאי S-פאזה באוכלוסייה אסינכרונית, אבל זה יכול גם להראות את ההתקדמות המדויקת של תאים בתוך שלב S (כלומר, מוקדם, באמצע, או מאוחר S-שלב)4. רמות ביטוי נמוכות של PCNA אקסוגני, מתויג מבטיח הפרעה מינימלית הן התקדמות מחזור התא ותהליכי תיקון DNA. חשוב לציין, PCNA משמש גם כבקרה פנימית עבור אינדוקציה נזק DNA תקין כפי שהוא מעורב בתיקון של כמה נגעים DNA והוא מגויס לאתרי נזק DNA המושרההמקומית 1,4.
הניסויים המוצגים כאן מדגימים כיצד למדוד את דינמיקת הגיוס של EXO1b בשלב S וכיצד זה מושפע ממעכב PARP מבוסס היטב, olaparib. פעילות נוקלאז EXO1b רלוונטית למגוון רחב של מסלולי תיקון DNA כולל תיקון אי התאמה (MMR), תיקון כריתת נוקלאוטיד (NER) ותיקון הפסקה כפולה (DSB). בשלב S, EXO1b ממלא תפקיד מרכזי רקומבינציה הומולוגית (HR) באמצעות היווצרות של 3 ' ssDNA overhangs במהלך כריתת DNA5. EXO1b כבר מעורב עוד יותר בשכפול DNA עם תפקידים בהפעלת נקודת ביקורת כדי להפעיל מחדש מזלגות DNA תקוע, כמו גם הסרת פריימר והבשלת שבר Okazaki בגדיל הפיגור במהלך עקירת גדיל בשכפול5. גיוס EXO1b לאתרי DNA פגומים מוסדר על ידי האינטראקציה הישירה עם פולי (ADP-ריבוז) (PAR)6,7. בשל ההשלכות הספציפיות הרבות של מחזור התא של EXO1b, זוהי בחירה מצוינת עבור מחקרי גיוס ספציפיים S-phase באמצעות PCNA.
Protocol
1. טיפוח תאים אנושיים שמקורם באוסטאוסרקומה (U-2 OS)
הערה: תאי מערכת ההפעלה U-2 הם אידיאליים עבור מחקרים אלה שכן יש להם מורפולוגיה שטוחה, גרעין גדול וחיבור חזק למספר משטחים, כולל זכוכית. ניתן להשתמש גם בקווי תאים אחרים בעלי מאפיינים דומים.
- לטיפוח קווי תאי מערכת ההפעלה U-2, יש להשתמש במדיום 5A של McCoy בתוספת סרום בקר עוברי 10% (FBS) ואנטיביוטיקה (100 פניצילין U/mL ו-100 מיקרוגרם/מ"ל סטרפטומיצין). תאי דגירה ב 37 °C (5 °F) באטמוספירה לחה המכילה 5% CO2. למחקרי מיקרוסקופיה, לשמור על תרבית התאים בצלחת 10 ס"מ כדי לספק ספירת תאים מספקת.
- כאשר תאים מתקרבים ל-90% השפעה (כ-7 x 106 תאים/10 ס"מ צלחת), מפצלים את התאים.
- יש לשטוף תאים באמצעות PBS כדי לשטוף מעכבי טריפסין הכלולים בסרום.
- הוסף מ"ל אחד של טריפסין-EDTA וודא ששכבת התא מכוסה באופן שווה.
- דגירה ב 37 °C (50 °F) עד שכבת התא הוא הרים את הצלחת (כ 6 דקות).
- Resuspend התאים טריפסין בסרום המכיל מדיה כדי לנטרל את הטריפסין ולהוסיף 1/10th של הנפח (~ 0.7 x 106 תאים) לתוך צלחת חדשה 10 ס"מ המכיל 10 מ"ל של מדיום צמיחה נוסף.
- לפני הניסויים, בדוק באופן שגרתי תאים לזיהום מיקופלסמה באמצעות ערכת זיהוי מיקופלסמה אוניברסלית בעקבות המלצת היצרן.
2. זיהום רטרו-ויראלי
הערה: עבור אמצעי בטיחות BSL-2 ובזמן עבודה עם וירוסים רקומביננטיים, אנא עיין ב: הנחיות NIH, סעיף III-D-3: וירוסים רקומביננטיים בתרבית הרקמות.
- זרע 4 x 106 HEK293T תאים כדי להשיג ~ 60% השפעה בתוך 24 שעות לאחר ציפוי לתוך צלחת תרבות 10 ס"מ.
- לטיפוח HEK293T אנא בצע את שלבי הטיפוח של מערכת ההפעלה U-2 המתוארים ב- 1.1-1.3 של פרוטוקול זה. עבור HEK293T תחליף 5A של מק'קוי בינוני עבור DMEM. הקפד תמיד לשטוף בעדינות תאי HEK293T כפי שהם מתחברים צלחות תרבית רקמות חלש.
- Transfect HEK293T תאים באמצעות ריאגנט transfection מבוסס שומנים עבור אריזה ויראלית של plasmids.
- עבור וקטורים רטרו-ויראליים, שלבו 1.5 מיקרוגרם של VSV-G (Addgene #8454) ו-1.5 מיקרוגרם של pUMVC (Addgene #8449) וקטורים לאריזה יחד עם 3 מיקרוגרם של הווקטור המכיל את גן העניין (בעמוד שדרה וקטורי רטרו-ויראלי עם התנגדות לטיהור) ל-250 μL של מדיה מופחתת בסרום Opti-MEM בצינור מיקרוצנטריפוגה. הוסיפו 1 μL של P3000 ריאגנט עבור כל מיקרוגרם של DNA הוסיף לתערובת Opti-MEM / DNA (במקרה זה 6 μL) ולערבב בעדינות על ידי הקשה. אין מערבולת או פיפטה למעלה ולמטה.
- בצינור microcentrifuge אחר, לשלב 2 μL לכל DNA מיקרוגרם (במקרה זה 12 μL) של ריאגנט טרנספקטיון עם 250 μL של Opti-MEM מופחת מדיה בסרום.
- מערבבים את שתי התערובות (500 μL בשילוב, לא מערבולת, רק לערבב על ידי הקשה עדינה) ולתת לו לדגור במשך 15 דקות בטמפרטורת החדר.
- בזהירות, מוסיפים את התערובת טיפה תפילה לתאי HEK293T זרעים מבלי לנתק את התאים. מערבבים את הצלחות בעדינות.
- זיהום ויראלי כדי ליצור קווי תאים יציבים.
- הסר את הווירוס המכיל supernatant מתאי HEK293T 72 שעות לאחר transfection. לסנן בזהירות את הפתרון עם מסנן 0.45 מיקרומטר כדי להסיר פסולת תאים ותאים מנותקים. לחלופין, להוסיף 8 מיקרוגרם / mL polybrene supernatant ויראלי כדי להקל על זיהום ויראלי.
- הוסף וירוס המכיל supernatant לתאי מערכת ההפעלה U-2 ב~ 50% השפעה בצלחת 10 ס"מ (~ 3 x 106 תאים). זרע את תאי מערכת ההפעלה U-2 יום קודם לכן.
- להדביק במשך 6-16 שעות לפני הסרת והשלכת supernatant המכיל וירוס.
הערה: כדי להשיג את הכמות הרצויה של ביטוי יתר עבור הגן של עניין, לדגור סדרה של דילול ויראלי במשך פרק זמן קבוע. בדוק את רמות הביטוי של transgene בכל קו תא שהוקם לאחרונה עם כתם מערבי השוואתו לרמות אנדוגני. - אפשר לתאים לבחור בנוכחות אנטיביוטיקה מתאימה (במשך 3-4 ימים במקרה של puromycin בריכוז הסופי של 2 מיקרוגרם / מ"ל) ולאמת את הביטוי של חלבון פלואורסצנטי מתויג גן עניין תחת מיקרוסקופ.
- חזור על שלבים אלה כדי ליצור קווי תאים עם תוויות כפולות. בניסויים שהוצגו כאן mPlum-PCNA בא לידי ביטוי מווקטור רטרו-ויראלי (pBABE) בשילוב עם EXO1B-AcGFP, שבא לידי ביטוי גם מווקטור רטרו-ויראלי (pRetroQ-AcGFP1-N1).
3. הכנת תאים להקרנה מיקרו
- תאי ציפוי: 24 שעות לפני הניסוי, צלחת סך של 8.0 x 104 תאים לתוך נפח בין 500 μL-1 מ"ל של מדיה (עבור בערך 70% השפעה) על ארבעה שעון כיסוי מצויד היטב עם תחתית זכוכית borosilicate מס '1.5 המספק תוצאות אידיאליות עבור מיקרוסקופיה קונפוקלית הגדלה גבוהה ומיקרו הקרנה מיקרו לייזר. השפעה גבוהה יותר על התאים מאפשרת יותר תאים הנמדדים בשדה ראייה יחיד (FOV); עם זאת שקופיות במפגש מלא יציגו אי סדרים במחזור התא.
- מדיית הדמיה: שעה לפני מיקרו-הקרנה, להחליף מדיום צמיחה רגיל עבור פלואורו-Brite DMEM בתוספת 10% FBS, 100 פניצילין U /mL ו 100 מיקרוגרם / מ"ל סטרפטומיצין, 15 mM HEPES (pH = 7.4) ו 1 mM נתרן פירובט. מדיית הדמיה זו מסייעת למקסם את יחס האות לרעש המאפשר זיהוי של פלואורסצנטיות עמומה מאוד. מאז הוא מכיל HEPES, זה גם מייצב pH בהיעדר 5% CO2 אטמוספרה.
- יש למרוח טיפול נוסף לפני ההדמיה בשלב זה. בניסויים שהוצגו כאן, תאים טופלו מראש שעה לפני הדמיה עם אוולפריב (מעכב PARP, בריכוז הסופי של 1 מיקרומטר) או בקרת רכב (DMSO)1,8,9.
4. הכנת המיקרוסקופ ובחירת תאי פאזה S להדמיה.
- השתמש במערכת קונפוקלית עם מאפיינים דומים לזה של המערכת המתוארת כאן לקבלת התוצאות הטובות ביותר. הניסויים שהוצגו כאן בוצעו באמצעות מיקרוסקופ קונפוקלי המותקן על מעמד מיקרוסקופ הפוך (ראה טבלת חומרים).
הערה: המיקרוסקופ המשמש כאן היה מצויד במודול לייזר FRAP 50mm 405 ננומטר, ויעד תוכנית-apochromat 60x 1.4 NA. לראש הסריקה הקונפוקללי היו שתי אפשרויות סורק: סורק גלוואנו (ברזולוציה גבוהה) וסורק מהדהד (להדמיה במהירות גבוהה).- הצג שחזור פלואורסצנטיות לאחר צילום הלייזר (FRAP) לדגימה באמצעות מכשיר XY galvano הנשלט על ידי תוכנה. השתמש בקו לייזר של 488 ננומטר כדי לרגש את AcGFP וקו לייזר של 561 ננומטר או 594 ננומטר כדי לרגש את mPlum.
הערה: שילוב המסנן הבא נותן תוצאות אופטימליות: באמצעות מסנן מעבר באורך 560 ננומטר, נורית פליטה עם אורך גל נמוך מ- 560 ננומטר הועברה דרך מסנן פליטה של 525/50 ננומטר עבור AcGFP, בעוד שאור פליטה עם אורך גל גבוה מ- 560 ננומטר הועבר דרך מסנן פליטה של 595/50 ננומטר עבור mPlum. כל ערכת מסננים מתאימה (למשל, FITC/TRITC, GFP/mCherry, FITC/TxRed) המבטיעה שימוש בפלואורסצנטיות מינימלית.
- הצג שחזור פלואורסצנטיות לאחר צילום הלייזר (FRAP) לדגימה באמצעות מכשיר XY galvano הנשלט על ידי תוכנה. השתמש בקו לייזר של 488 ננומטר כדי לרגש את AcGFP וקו לייזר של 561 ננומטר או 594 ננומטר כדי לרגש את mPlum.
- הפעל את התא הסביבתי ואת רכיבי המיקרוסקופ.
- הפעל את החימום (שלב, אובייקטיבי ותא סביבתי במידת האפשר), אספקת CO2 ואת ווסת הלחות לפחות 4 שעות לפני תחילת הניסוי כדי להבטיח שיווי משקל תרמי לרכישת תמונה יציבה.
- אתחל מקורות אור יחד עם קווי הלייזר לפחות שעה אחת לפני העברת התאים למיקרוסקופ.
- בחר תאי S-פאזה באוכלוסייה אסינכרונית באמצעות PCNA מתויג פלואורסצנטי כסמן. בצע זאת על-ידי ביצוע השלבים שלהלן.
- חפש את תבנית הה לוקליזציה הייחודית של PCNA שתויג על-ידי mPlum בשלב S, מה שהופך את הזיהוי של שלב מחזור תא זה לאפשרי. ל- PCNA יש התפלגות הומוגנית לחלוטין בגרעין בשלבי G1 ו- G2 של מחזור התא, תוך אי הכללה מהגרעין. ב- S-phase, PCNA יוצר מוקדים במיקום של replisomes בגרעין. איור 1 מציג את הדפוסים השונים של מוקדי PCNA לאורך S-phase, מה שמאפשר אפילו להבדיל בין שלב S מוקדם, בינוני ומאוחר.
- חפש דרך העין כדי לבחור FOV שיש לו מספיק תאי S-פאזה עבור מיקרו-הקרנה. תאי מערכת ההפעלה U-2 האסינכרוניים בדרך כלל יש 30-40% מהאוכלוסייה שלהם בשלב S.
- נסו להימנע מקיצוניות ברמות הביטוי (תאים בהירים ועמומים כאחד) הן עבור PCNA והן עבור חלבון העניין (POI), במקרה זה EXO1b-AcGFP, אשר יכול להוביל חפצים ניסיוניים.
- בעת מציאת FOV מתאים, נסה להימנע מסריקה של השדה במשך זמן רב כדי למזער את ההלבנה ונזקי DNA לא רצויים.
- הגדר את אזור העניין הרצוי (ROI) להקרנה זעירה. באמצעות התוכנה המשויכת (ראה טבלת חומרים), הגדר את ה- ROI הרצוי על-ידי הוספת קווים בינאריים תחילה (הגדר את מספר השורות והמרווח הרצוי). לחץ על בינאריולאחר מכן לחץ על הוסף | שורה | מעגל אליפסה כדי לצייר את מספר הקווים הרצוי.
- המר קווים בינאריים אלה ל- ROIs ולבסוף המר ROIs אלה ל- ROIs לגירוי. כדי לעשות זאת, לחץ תחילה על ROIולאחר מכן לחץ על העבר בינארי ל- ROIולאחר מכן לחץ באמצעות לחצן העכבר הימני על כל אחד מההשערות ובחר השתמש כהבגת גירוי: S1. מקם קווים אלה ב- FOV כדי לעבור דרך גרעין התאים. נעשה שימוש ב- ROIs באורך של 1024 פיקסלים שהשתרעו על פני ה- FOV כולו לאורך הפרוטוקול.
5. הקרנה מיקרו עבור כתמי אימונופלואורסצנטיות או הדמיה לשגות זמן.
- קביעת הגדרות מיקרו-הקרנה אופטימליות.
- לפני הקרנה מיקרו של התאים, לקחת תמונה ברזולוציה גבוהה יותר של FOV כדי לזהות מוקדי PCNA לניתוח מאוחר יותר. במקום סריקה רציפה, הקלט בו זמנית את שני הערוצים האופטיים המשמשים (ירוק ואדום), כדי למנוע תנועת תאים בין סריקה בשני אורכי הגל. לרזולוציה נכונה של המוקדים השתמש לפחות 1024 x 1024 פיקסלים / רזולוציית שדה עם זום 1x (גודל פיקסל 0.29 מיקרומטר במערכת ההדמיה המשמשת כאן), עם מהירות סריקה של 1/8 מסגרת /s (4.85 מיקרו-פיקסל/ פיקסל) עם ממוצע של פי 2. לאחר שפרמטרים אלה מוגדרים ב- A1 LFOV Compact GUI ובחלונות אזור הסריקה A1 LFOV, לחץ על לחצן לכידה כדי להקליט את ה- FOV.
הערה: חשוב לשמור על אותו גודל פיקסלים לאורך כל הניסויים כדי להבטיח תוצאות דומות. - כדי להגדיר את המיקרו-הקרנה, פתח את הכרטיסיה גירוי ND בתוכנת ההדמיה כדי לגשת ללוח הזמנים (A1 LFOV / Galvano Device). זה משתמש סורקי galvano כדי לרכוש סדרה של תמונות טרום גירוי, לעורר (באמצעות LUN-F 50 mW 405 ננומטר FRAP לייזר), ולאחר מכן לרכוש סדרה של תמונות לאחר גירוי שוב באמצעות סורקי galvano. הגדר תחילה שלושה שלבים בחלון לוח הזמנים של הזמן. בעמודה Acq/Stim בחר | רכישה | להלבנה רכישה עבור שלושת השלבים בהתאמה. עבור שלב ההלבנה, הגדר את S1 כ- ROI.
הערה: בניסוי שהוצג כאן, לא נרכשו תמונות בשלב הגירוי. - בחלון Galvano XY, הגדר את הגורמים העיקריים עבור הקרנה מיקרו: 405 ננומטר תפוקת כוח לייזר, זמן השתהות (איטרציה היא 1 כברירת מחדל במערכת זו). בניסויים שהוצגו כאן, תאים היו מוקרן עם לייזר FRAP 405 ננומטר (50 mW בקצה הסיבים) ב 100% תפוקת כוח עם זמן התעכבות 1000-3000 מיקרו.
הערה: מכיוון שזמן השתהות הלייזר הוא על בסיס פיקסל, כל עוד גודל הפיקסלים נשאר זהה, הקשר בין זמן ההשתהות לצפיפות החשמל יהיה דומה בין FOVs שונים. איור 2A מציג את השימוש בחלבונים ספציפיים לתגובה לנזק לדנ"א (DDR) (FBXL10 עבור DSBs ו-NTHL1 לנזקי בסיס חמצוניים) כדי למטב את הגדרות צריכת הלייזר עבור אינדוקציה ספציפית של נזקים. קווי תאים יציבים אלה נוצרו עם זיהום ויראלי בעקבות סעיף 2 של הפרוטוקול.
- לפני הקרנה מיקרו של התאים, לקחת תמונה ברזולוציה גבוהה יותר של FOV כדי לזהות מוקדי PCNA לניתוח מאוחר יותר. במקום סריקה רציפה, הקלט בו זמנית את שני הערוצים האופטיים המשמשים (ירוק ואדום), כדי למנוע תנועת תאים בין סריקה בשני אורכי הגל. לרזולוציה נכונה של המוקדים השתמש לפחות 1024 x 1024 פיקסלים / רזולוציית שדה עם זום 1x (גודל פיקסל 0.29 מיקרומטר במערכת ההדמיה המשמשת כאן), עם מהירות סריקה של 1/8 מסגרת /s (4.85 מיקרו-פיקסל/ פיקסל) עם ממוצע של פי 2. לאחר שפרמטרים אלה מוגדרים ב- A1 LFOV Compact GUI ובחלונות אזור הסריקה A1 LFOV, לחץ על לחצן לכידה כדי להקליט את ה- FOV.
- הדמיית מעידת זמן.
- הגדר הדמיית זמן לשגות עבור חלון הזמן הרצוי ומרווחי זמן באמצעות לוח הזמנים, A1 LFOV Compact GUI וחלונות אזור הסריקה A1 LFOV. בניסויים שהוצגו כאן, הגיוס של EXO1b ו- PCNA היה בתמונה במשך 12 דקות, סורק את ה- FOV כל 5 שניות, ב 1024 x 1024 פיקסלים / שדה, באמצעות זום 1x (וכתוצאה מכך 0.29 מיקרומטר פיקסל גודל במערכת ההדמיה המשמשת כאן) עם 0.35 מסגרת / מהירות סריקה (1.45 μs /) פיקסל ללא ממוצע כדי להפחית את ההלבנה תמונה.
- מטב את צריכת הלייזר %, השג והקזז הגדרות כדי להפחית את ההלבנה של תמונות במהלך ההדמיה בחלון A1 LFOV Compact GUI. אם שואפים למדוד הן POI והן PCNA, השתמש בסריקה סימולטנית במקום בסריקה רציפה כדי למנוע תנועת תאים בין סריקת השדה עבור שני הפלורופורים הנפרדים.
- נעשה שימוש במערכת ההדמיה עם ההגדרות הבאות. עבור קו הלייזר 488 ננומטר (20 mW): 7% כוח לייזר, רווח: 45 (גלאי GaAsP) עם היסט של 2, עבור קו לייזר 561 ננומטר (20 mW): 4% כוח לייזר, להשיג 40 (גלאי GaAsP) עם היסט של 2.
- בהתאם לקינטיקה של החלבון, להאריך או לקצר את המרווח בין תמונות או את משך הזמן הכולל לשגות. בחלון לוח זמנים, הגדר את מרווח הזמן ומשך הזמן הרצויים עבור שורת הרכישה של השלב השלישי.
- לחץ על הפעל כעת כדי לבצע את המיקרו-הקרנה ואת הדמיית ההפוגה שלאחר מכן.
- בסוף הזמן לשגות הדמיה, לשמור את ROIs גירוי כתמונות נפרדות, אשר יהיה סיוע שימושי לזהות את הקואורדינטות של מיקרו הקרנה בכל תוכנה במורד הזרם המשמש לניתוח.
- כתמי אימונופלואורסצנטיות.
הערה: שלב 5.1.3 ואיור 2A מדגים את השימוש בחלבוני תיקון דנ"א ידועים כדי להעריך את סוגי נגעי ה-DNA שהוכנסו על ידי הקרנה זעירה. נגעי DNA מסוימים ניתן לזהות גם באמצעות נוגדנים ספציפיים לאחר תיקון התאים. ניתן גם לזהות את הגיוס של POI על ידי זיהוי נוגדנים של חלבון אנדוגני. ההדמיה של γH2A.X לבדיקת DSBs מודגמת להלן(איור 2B). איור 3 מציג את העקביות של לוקליזציה וגיוס PCNA לאורך מחזור התא עבור PCNA מתויג אנדוגני ואקסוגני.- לאחר שלב 5.1.3, קח תמונה אחת בלבד לאחר הקרנה זעירה כדי להבטיח אירוע FRAP תקין המבוסס על גיוס mPlum-PCNA. שים לב לקואורדינטות המדויקות של ה- FOV כדי למצוא את השדה מאוחר יותר לאחר תיוג האימונופלואורסצנטי.
- הוציאו את תא תרבית התאים מהמיקרוסקופ ותאי הדגירה ב-37 °C (5 °F) באטמוספירה לחה המכילה 5% CO2 למשך 5-10 דקות.
הערה: Paraformaldehyde (PFA) הוא רעיל, ועבודה צריכה להיעשות באזור מאוורר היטב או מכסה המנוע אדים. כל הכביסה והדגירה הבאות ייעשו עם נפחים של 0.5 מ"ל במגלשת התא 4 הבאר. לאחר זמן הדגירה, לשטוף את התאים עם 0.5 מ"ל של PBS (137 מ"מ NaCl, 2.7 mM KCl, 8m Na2HPO4, ו 2 mM KH2PO4) ולתקן עם 0.5 מ"ל של 4% PFA ב PBS במשך 10 דקות בטמפרטורת החדר (RT). - לשטוף את התאים פעם אחת עם PBS, ולאחר מכן לשטוף אותם עם 50 mM NH4Cl כדי להרוות PFA שיורית.
- פרמזביל להפוך את התאים במשך 15 דקות ב RT עם 0.1% טריטון X-100 ב PBS.
- חסום את הדגימות למשך שעה אחת עם מאגר חסימה (5% FBS, 3% BSA, 0.05% טריטון X-100 ב- PBS).
- הסר את פתרון החסימה והוסף את הנוגדן הראשי מדולל (anti-γH2A.X, 1:2000) בחסימת מאגר למשך שעה אחת ב- RT.
- לשטוף את בארות עם חוצץ חסימה 3 x 10 דקות.
- הוסף נוגדן משני מדולל (אנטי עכבר Alexa 488 פלוס מצומד, 1:2000) בחסימת מאגר עבור 1 שעה ב RT.
- לשטוף את בארות עם חוצץ חסימה 3 x 10 דקות.
- תטען את הגרעין עם פתרון DAPI של 1 מיקרוגרם/מ"ל ב-PBS למשך 15 דקות.
- לשטוף את התאים פעם אחת עם PBS. ההדמיה יכולה להתבצע ישירות ב- PBS או בפתרון PBS עם ריאגנטים נגד מפייד (למשל, AFR3) כדי להפחית את ההלבנה.
6. ניתוח גיוס
הערה: איור 4A מציג תמונות מייצגות של גיוס Exo1b ו- PCNA בנוכחות DMSO או olaparib. איור 4B מציג תמונה מייצגת לניתוח נתונים. ערכי פלואורסצנטיות ממוצעים חושבו על ידי מדידת עוצמות AcGFP ממוצעות באמצעות מלבן לאורך מסלול הלייזר המודגש על ידי mPlum-PCNA (A, מלבנים צהובים) על פני נקודות זמן שונות באמצעות פיג'י. PCNA יכול לשמש בקרה פנימית כדי להדגיש הקרנה מוצלחת לאורך קואורדינטות ROI. באופן דומה, ערכי פלואורסצנטיות ממוצעים של AcGFP חושבו גם עבור אזורים שלא ניזוקו בגרעין (B, מלבנים כחולים). עוצמת אות הרקע נמדדה באזורים לא מאוכלסים (C, מלבנים אדומים) והופחתה מערכי הפלואורסצנטי הממוצעים (איור A ו- B). לפיכך, יחידת הפלואורסצנט הממוצעת היחסית (RFU) עבור כל נקודת איסוף נתונים חושבה על ידי המשוואה RFU = (A − C)/ (B − C)8,9. ערכי RFU המתקבלים של האזור המוקרן מיקרו מנורמל לערכי RFU לפני הקרנה מיקרו.
- להגדרת האזור A של האתר מיקרו-מוקרן, לא לכלול אזורים נוקלאולר, מוקדי שכפול, ואזורים גרעיניים לא סדירים של התא מן המדידה. החזק מקש Shift בין ציור שני ROIs בפיג'י כדי לקבץ שני אזורים נפרדים כאחד.
הערה: גיוס חלבונים ישתנה בין גנים שונים ותנאי הקרנה; לכן, גודל אזור A חייב להיקבע בנפרד. לאחר אופן קביעת רוחב הפיקסלים של אזור A, עליו להישאר קבוע עבור כל גיוס השוואתי. בניסויים שהוצגו כאן, נעשה שימוש במלבנים ברוחב 7 פיקסלים. - אל תכלול תאים שעברו במהלך הסרטונים המוקלטים מניתוח. כדי לכלול תאים ניידים מאוד, הניתוח המתואר חייב להתבצע פריים אחר פריים.
- כדי לדמיין את פרופיל הגיוס, התווה את ערכי RFU מנורמלים כנגד הזמן באמצעות תוכנה סטטיסטית.
- חשב את ההפרש בנקודת זמן מצוינת בין טיפול DMSO ו- olaparib (n = 31) באמצעות מבחן מאן-ויטני.
Representative Results
תאים מטפלים בכל סוג של נגע DNA באופן ספציפי שתלוי גם באיזה שלב מחזור התא הם נמצאים. לדוגמה, לאחר הקרנה מיקרו, הפסקות כפולות גדילים (DSB) יעובדו על ידי צירוף קצה לא הומולוגי (NHEJ) או HR בהתאם לשלב מחזור התא. נוקלאזים הפועלים באופן הנרחב ביותר במהלך שלבי S ו- G2 של מחזור התא יוצרים overhangs DNA החיוניים למשאבי אנוש תקינים. כדי לקדם את ההערכה של תאים בשלב S, PCNA הועסק כסמן מחזור תא בצבע יחיד. איור 1A מציג את פרופיל הלוקאליזציה של mPlum-PCNA במהלך התקדמות מחזור התא. PCNA יש התפלגות הומוגנית לחלוטין בגרעין בשלב G1 ו- G2 (תוך הכללה בעיקר מן הגרעין). בשלב S, PCNA לוקליזציה לאתרים של שכפול DNA, אשר ניתן לדמיין כתמים בהירים בגרעין. בתאי שלב S המוקדמים, הכתמים קטנים יחסית ומחולקים באופן שווה לאורך גרעין התא. מתקדמים לתוך שלב S באמצע, כתמים הופכים מטושטשים לוקליזציה יותר לכיוון ההיקף של הגרעין ואת הגרעין. בשלב S המאוחר, הכתמים מצטמקים במספרים אך הופכים גדולים יותר ויותר ככל ש-PCNA מתרכז באתרי שכפול מאוחרים(איור 1B). חשוב לציין, ביטוי PCNA אקסוגני מעמוד השדרה הווקטורי pBABE היה פחות מהרמות האנדוגניות, אך הספיק לזיהוי על ידי מיקרוסקופיה אשר ממזערת חפצים פוטנציאליים בהתקדמות מחזור התא ו- DDR. איור 1C מראה את מידת ביטוי יתר של PCNA בהשוואה לרמות אנדוגניות. שים לב כי הרצועה המתאימה ל- mPlum-PCNA נודדת לאט יותר בשל גודלה הגדול יותר.
אנו שואפים להציג DSBs במהלך הקרנה מיקרו לחקור את הגיוס תלוי PARP1/2 של EXO1b נגעים אלה בשלב S. איור 2A מראה כי מינונים נמוכים של אנרגיה (1000 μs זמן להתעכב) אינם גורמים לגיוס של EGFP-FBXL10, מגיב DSB (רכיב של מתחם FRUCC 8), בעוד זה היה מספיק כדי לגרום לגיוס של NTHL1-mCherry, חלבון מסלול תיקון כריתה בסיסית (BER), גיוס לאתרים של נזק DNA חמצוני10,11,12. בזמן 6000 μs להתעכב, הן EGFP-FBXL10 והן NTHL1-mCherry לגייס, הדגמת תפוקת לייזר שמייצרת הן נגעים חמצוניים ו- DSBs. חיזוק תוצאות אלה, איור 2B מראה כתמים אימונופלואורסצנטיים נגד γH2A.X (סמן DSB), אשר ברור יותר בעת שימוש במינונים גבוהים יותר אנרגיה. PCNA משמש הן סמן מחזור התא וסמן עבור הקרנה מיקרו מוצלחת, כפי שהוא מגייס כראוי עם שתי הגדרות זמן התעכבות לייזר. חשוב לציין, PCNA פלואורסצנטי אקסוגני ו/או אנדוגני מתויג בחלבון יכול לשמש לתפקוד כתב זה כפי שהם מתנהגים באופן דומה (איור 3). אנדוגנית מתויג PCNA תוכנן על ידי החדרת mRuby במסגרת עם האקסון הראשון לתוך אלל אחד של לוקוס PCNA13 (קו התא היה מתנה סוג של יורג מנספלד).
איור 4A ואיור 4C מציג את הגיוס של EXO1b מתויג ACGFP בתאי פאזה S. EXO1b מגיע לרמה המרבית של הצטברות באתרי הקרנה זעירה סביב 1 דקה ולאחר מכן לאט לאט מתחיל להתנתק נגעי ה- DNA לאחר מכן. העשרות באתרי מיקרו-הקרנה מוזכרות על ידי יחידת פלואורסצנטיות יחסית > 1 בגרף. בנוכחות olaparib, הצטברות של EXO1b בפס הלייזר בדקה אחת היא פחותה באופן משמעותי בהשוואה לשליטה ברכב. תוצאות אלה הן בהסכמה עם הספרות6,7. איור 4B מדגים אזורים מייצגים לכימות (אזורים A, B ו- C) כמתואר בנקודה 6 בפרוטוקול. איור 4D מציג את רמות הביטוי הדומות של EXO1b אנדוגני ו-EXO1b-AcGFP אקסוגני בתאים המשמשים להקרנה זעירה.
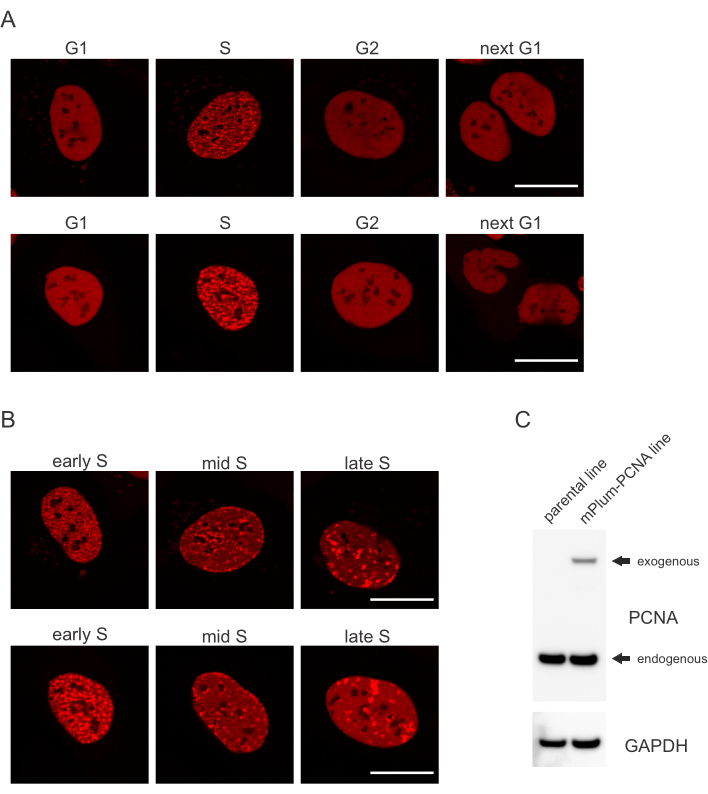
איור 1: תבנית לוקליזציה של PCNA. (A)תמונות מראות דפוס לוקליזציה של PCNA משולב ביציבות אקסוגני לאורך מחזור התא בתאי מערכת ההפעלה U-2. (B)תמונות מראות דפוסי מוקדי PCNA בשלבים שונים של שלב S (מוקדם, אמצע ומאוחר) בתאי מערכת ההפעלה U-2. (C)כתם מערבי המציג רמות אנדוגניות ואקסוגניות של PCNA בתאי מערכת ההפעלה U-2 המשמשים להדמיה. סרגל קנה המידה מייצג 20 מיקרומטר. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 2: אינדוקציה של DSBs באמצעות תפוקת כוח לייזר ממוטבת. (A)ניתן למטב את הגדרות הלייזר כדי לגרום לצורות שונות של נזק לדנ"א. תאי מערכת ההפעלה U-2 מבטאים ביציבות הן EGFP-FBXL10 והן NTHL1-mCherry שימשו לזיהוי DSBs ואתרים של נגעים חמצוניים, בהתאמה. הקרנה מיקרו עם קו לייזר 405 ננומטר בוצעה על תאי מערכת ההפעלה U-2 אסינכרוניים עם 1000 μs או 3000 μs זמן השתהות. סרגל קנה מידה מייצג 20 מיקרומטר. (B) כתמים אימונופלואורסצנטיים נגד γH2A.X נעשה על תאי אפיתל פיגמנט רשתית אנושית (hTERT RPE-1) לאחר PCNA אנדוגני מתויג mRuby. התאים תוקנו ועובדו 5 דקות לאחר הקרנה זעירה עם 1000 מיקרו או 3000 μs זמן השתהות. סרגל קנה המידה מייצג 20 מיקרומטר. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 3: גיוס דומה של mRuby-PCNA אנדוגני ו- mPlum-PCNA אקסוגני לאתרי מיקרו-הקרנה ב 1000 μs או 3000 μs זמן התעכבות לייזר. הן אנדוגני אקסוגני מתויג PCNA טופס מוקדי שכפול במהלך שלב S. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 4: גיוס תלוי PARP1/2 של EXO1b בשלב S. תאי מערכת ההפעלה U-2 מבטאים ביציבות EXO1b-AcGFP ו- mPlum-PCNA היו מיקרו-הקרנה עם קו לייזר FRAP 405 ננומטר באמצעות זמן השתהות 3000 μs. (A)תמונות מייצגות של תאים מיקרו-מוקרן בנקודות הזמן שצוינו לאחר טיפול מקדים עם בקרת רכב (DMSO) או olaparib (1 μM). סרגל קנה המידה מייצג 20 מיקרומטר. (B) תמונות מייצגות של אזורים מוגדרים של אזורי A, B ו- C לניתוח הגיוס. סרגל קנה המידה מייצג 20 מיקרומטר. (C)דינמיקת גיוס נזקי דנ"א נתפסה על ידי הדמיה של תאים חיים. ערכי פלואורסצנטיות ממוצעים יחסית ותמונות נרכשו כל 5 s במשך 12 דקות. עבור כל תנאי, ≥30 תאים הוערכו. ערכי פלואורסצנטיות יחסיים ממוצעים (קווים שחורים מלאים) ושגיאה סטנדרטית (טווח המומחז על-ידי אזור מוצלל) התווו כנגד הזמן. קו מקווקו מציג ערכי גיוס בדקה אחת לאחר הקרנה זעירה. ההבדל בין טיפול DMSO (n = 32) לבין טיפול olaparib (n = 31) חושב באמצעות מבחן מאן-ויטני. אסטריקס מציינת עמ<0.0001. (D)כתם מערבי משווה את רמות הביטוי של EXO1b אנדוגני ו EXO1b-AcGFP אקסוגני בתאים המשמשים להקרנה מיקרו. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Discussion
שלבים קריטיים ופתרון בעיות/שינויים פוטנציאליים בפרוטוקול
כלי תרבית רקמות נכון עבור הקרנה מיקרו הוא קריטי להצלחה. רוב מערכות ההדמיה ברזולוציה גבוהה מותאמות לעובי זכוכית כיסוי של 0.17 מ"מ. שימוש בתאי הדמיה בעובי גבוה או נמוך יותר או בתאים העשויים מפולימרים מפלסטיק (שאינם מותאמים להדמיה של 405 ננומטר), יכולים להפחית באופן משמעותי את איכות התמונה. בעת שימוש במשטחי זכוכית, ודא כי הם תרבית רקמות מטופלים כדי לשפר את הידבקות התא. אם הם אינם מטופלים בתרבית הרקמות, יהיה צורך לצפות תאים אלה, למשל, בפולי-די-ליסין לפני זריעת התאים. כאשר ציפוי תאים לתוך שעון כיסוי התא, צפיפות התא האידיאלית היא בעלת חשיבות עליונה כדי למנוע אי סדרים במחזור התא ומתח נוסף לתאים. שיווי משקל תרמי נכון של רכיבי המיקרוסקופ לפני הניסויים כדי לשמור על טמפרטורה יציבה חיוני הן לשמירה על המיקוד לאורך כל הזמן הדמיה לשגות והוא גם הכרחי כדי להבטיח DDR הומוגני לאורך זמן ודגימות.
זה קריטי כי תאים נמצאים במצב בריא לפני מיקרו הקרנה כדי להפחית נתונים חפצניים. אם לתאים יש מורפולוגיה לא סדירה לאחר זיהום/בחירה, אפשר לתאים להתקדם במעברים מרובים עד שהמורפולוגיה תחזור לשגרה. ודא תמיד כי קווי התאים המשמשים נקיים מזיהום mycoplasma. בין ההשפעות השליליות הרבות של זיהום mycoplasma, זה גם גורם נזק DNA לתאים המארחים יכול להשפיע על מסלולי DDR שלהם14,15. הדרך הרגישה ביותר לזהות mycoplasma בתרבות התא היא באמצעות PCR (לעומת זיהוי עם DAPI או Hoechst).
ביטוי יתר אופטימלי של חלבון התיקון של עניין צריך להיות דומה לרמות אנדוגני, עם זאת, גבוה מספיק לגילוי. המקדם המשמש על הווקטורים הנגיפיים, טיטר ויראלי במהלך זיהום, ואת אורך זמן ההדבקה ניתן להתאים את כל לרמות הביטוי האידיאליות. לקבלת תוצאות עקביות, בודד שיבוטים של תאים בודדים כדי להבטיח רמות ביטוי הומוגניות ומורפולוגיה רגילה של תאים. מומלץ להשתמש במבנים וקטוריים שאינם מגזמים ב- PCNA מתויג ברמות אנדוגניות גבוהות יותר עבור מחזור תאים תקין ותפקוד סמן תיקון DNA. אפילו רמות נמוכות של ביטוי יתר של PCNA מספיקות כדי להפלות תאים של פאזה S. וקטורים רטרו-ויראליים pBABE שימשו בהצלחה למטרה זו (Addgene #1764, #1765, #1766, #1767). PCNA ניתן לתייג עם כל אדום מונומרי(למשל, mPlum, mCherry, mRuby, וכו ') או חלבונים פלואורסצנטיים ירוקים מונומריים (למשל, mEGFP, AcGFP, mWasabi, mNeonGreen, mEmerald, וכו ') אשר לאחר מכן ניתן לשלב עם POI מתויג לסירוגין. להבעשת יתר של POI מתויג פלואורסצנטי יש כמה מגבלות ושיקולים. תגי פלורסנט עלולים לשבש את תפקוד החלבון הרגיל ואת לוקליזציה. לכן, יש לקחת בחשבון את מיקום התג (N או C-terminal). השתמש תמיד בחלבונים פלואורסצנטיים מונומריים, כמו אוליגומריזציה של וריאנטים שאינם מונומריים יכול להשפיע על הפונקציה של POI.
יש לקבוע את הגדרות הלייזר עבור כל מערכת הדמיה, שכן רכיבים רבים של הנתיב האופטי ישפיעו על הכוח הממשי המועבר לתאים. הקרנה מיקרו-הקרנה לייזר יכול לגרום מספר סוגים של נגעים DNA בהתאם אורך גל העירור, תפוקת הכוח של לייזר FRAP ואם כל סוכנים רגישים מראש (כמו ברומודוקסיורידין או Hoechst) שימשו. לייזרים 405 ננומטר יכול לגרום נזק DNA חמצוני, שברים בודדים וכפולתקועים 16,17. באמצעות הגדרות פלט לייזר גבוהות יותר, כמות ה- DSBs גדלה. בפרוטוקול זה שיטות טרום רגישות לא נוצלו, אך טכניקות אלה מכוסות מאוד בספרות ומכוסות מחדש בדיון שלהלן. לדעתנו, הדרך הטובה ביותר לבדוק אם הנגע הרצוי נוצר היא על ידי בדיקה לגיוס של גנים ספציפיים למסלול נזקי DNA ידועים. גיוס של NTHL1 או OGG1, רכיבים של מסלול BER, מציע את האינדוקציה של בסיסי DNA מחומצן10,11,17,18,19, בעוד FBXL10 או XRCC5 מצביעים על נוכחות של DSBs8,20,21. גיוס של XRCC1 יכול להצביע הן על נוכחות של בסיסי DNA מחומצנים והן הפסקות בודדות תקועות (SSB)22,23. XPC (כלומר, RAD4) הוא אינדיקטור טוב של NER המסיר את תוספות ה- DNA המגושמים הנוצרות על ידי אור אולטרה סגול (UV)17,24. מכיוון שגיוס חלבונים אקסוגניים עלול לגרום לאי סדרים מסוימים, כתמים אימונופלואורסצנטיים של חלבונים או סמנים לתיקון דנ"א אנדוגני (כמו γH2A.X להפסקות כפולות) יכולים לאשר את נוכחותם של נגעים ספציפיים ב- DNA. לחלופין, נוגדנים שהועלו נגד סוגים מסוימים של נגעי DNA יכולים לשמש גם. כדי לכוונן את כוח הלייזר המועבר, ניתן לשנות הן את זמן ההשתהות והן את כוח הלייזר.
בעזרת מודלים מתמטיים, ניתן לבצע ניתוח קינטי מפורט שיכול לספק תובנות חשובות על תכונות הגיוס של POI (למשל, תרומה של תחומים כריכי DNA מרובים, רגישות לאירועי איתות שונים וכו '). ניתן לשלב הערכת גיוס אוטומטית ומעקב אחר תאים כדי ליצור זרימות עבודה חזקות 1,25.
יתרונות ומגבלות של רגישות מוקדמת DNA
רגישות מוקדמת של DNA לפני מיקרו-הקרנה הוא כלי נפוץ לתיקון DNAגיוס חלבון 16,17. רגישות DNA לפני מיקרו הקרנה משאירה אותו רגיש יותר DSBs. שתי השיטות הנפוצות ביותר עבור DNA טרום רגישות הם טרום טיפול בתאים עם ברומודוקסיורידין (BrdU) או צבע Hoechst. עבור מערכות שאינן מסוגלות מיקרו-הקרנה בכוחות לייזר גבוהים, שיטות אלה עשויות להיות נחוצות לזירוז נגעי DNA כמו DSBs. בנוסף, בהיעדר גלאי אור מועבר או אות פלואורסצנטי המדגיש את גרעין התא (לדוגמה, כאשר לומדים את הגיוס של חלבוני תיקון DNA אנדוגני לא מתויג), Hoechst פועל הן ככלי רגיש מראש וכתם גרעיני פלואורסצנטי. עם זאת, DNA מראש רגישות יכול להציג סיבוכים משמעותיים. BrdU (בשימוש בריכוז סופי של 10 מיקרומטר) יש להוסיף לתאים 24 שעות (או זמן שווה ערך למחזור תא מלא בקו התא המשמש) כדי לשלב כראוי ב- DNA ויכול לגרום להפרעות מחזור התא26. Hoechst 33342 (בשימוש בריכוז סופי של 1 מיקרוגרם / מ"ל) הוא ציטוטוקסי לאחר תקופות דגירה ארוכות, אך דורש מספיק זמן כדי להרוות את הגרעין עם הצבע. לכן, זה צריך להיות מיושם רק 15-20 דקות לפני מיקרו הקרנה; אחרת, נתוני הגיוס לא יהיו עקביים. התאים מוכתמים בדרך זו לא ניתן לשמור בתרבות במשך יותר מכמה שעות27,28. הקפד לא להשתמש Hoechst 33358, אשר אינו חדיר תא כמו צבע Hoechst 33342. רגישות מוקדמת יכולה גם להכניס שונות מיותרת בין ניסויים והופכת את הניסוי לרגיש עוד יותר להבדלים בצפיפות התאים (שכן זה ישפיע על כמות הצבע / התא המשולב).
יתרונות ומגבלות של מיקרוסקופיה קונפוקלית
מהירות ההדמיה של מיקרוסקופיה קונפוקלית יכולה להיות מוגבלת בהשוואה למיקרוסקופיה רחבת שדה. עם זאת, מיקרוסקופ קונפוקל המצויד בסורק מהדהד יכול לשפר מאוד את מהירות ההדמיה (במחיר הרזולוציה) מתקרב למהירויות של מיקרוסקופיה של דיסק מסתובב. שלוש תכונות הופכות את מערכת הקונפוקלית A1R HD25 לבחירה מצוינת עבור הפרוטוקול המוצג כאן. ראשית, FOV 25 מ"מ של המערכת מאפשר תמונה בין 15-20 תאים בשדה סרוק יחיד (לעומת 5-10 תאים בכיוונונים רגילים), הגבלת מספר הרכישות הדרושות כדי לקבל מספיק תאים לניתוח סטטיסטי. שנית, מודול FRAP ושני ראשי סריקה מאפשרים לדמיין ולהקרין את התאים בו זמנית, לא רק ברצף. לבסוף, הגמישות שיש הן סורקי מהדהד והן סורק galvano מספק את היכולת לעבור בקלות בין הדמיה ברזולוציה גבוהה-זמנית עם מהירות יוצאת דופן אשר ממזערת מרווה של פלואורופורים, הדמיה ברזולוציה מרחבית גבוהה המשתמשת במהירויות סריקה איטיות יותר כדי לייצר תמונות עם יחס אות לרעש גבוה יותר. בעוד שהמערכת המשומשת אפשרה את הגמישות הנ"ל, להידמות לתצורות מיקרוסקופ קונפוקליות זמינות יותר, רק סורק גלוונו שימש בניסויים שהוצגו (הן עבור מיקרו-הקרנה והן עבור הדמיה עוקבת).
יתרונות ומגבלות של הקרנה מיקרו
בעוד מיקרו-הקרנה מספקת רזולוציה מרחבית וטמפורלית ללא תחרות, זה לא ללא מגבלות. נזק לדנ"א על ידי הקרנת מיקרו לייזר מקובצים מאוד לחלקים מסוימים של הגרעין בהשוואה לסוכנים מזיקים טבעיים. לכן, תגובת הכרומטין עקב הקרנה מיקרו עשוי להיות שונה לעומת נזק מופץ הומוגני. בנוסף, מיקרו-הקרנה גוזלת זמן ויכולה להתבצע רק על כמה עשרות תאים, בעוד ששיטות ביוכימיות גדולות המבוססות על אוכלוסייה (שבר כרומטין, אימונופרציפיטציה, ChIP) יכולות לספק חוסן מוגבר על ידי לימוד אלפי תאים בכל פעם. אימות תצפיות שנעשו על ידי הקרנה מיקרו עם טכניקות ביוכימיות מסורתיות היא אסטרטגיה יעילה למסקנות אמינות. למרות הקרנה מיקרו בו זמנית של תאים רבים ב- FOV מסוים אפשרי, מערכת ההדמיה תצטרך יותר זמן כדי לבצע את המשימה. לכן, מדידת הדינמיקה של חלבונים המגייסים במהירות רבה נגעי DNA מגבילה את מספר ROIs אפשרי עבור מיקרו הקרנה בשימוש בו זמנית. במערכת ההדמיה המשמשת לפרוטוקול זה, הקרנה זעירה של ROI יחיד באורך 1024 פיקסלים אורכת 1032 אלפיות שניה תוך שימוש בזמן השתהות של 1000 מיקרו ו- 3088 אלפיות שניים באמצעות זמן השתהות של 3000 מיקרו μs כדי להשלים. שימוש בקווים מרובים של ROIs יגדיל באופן משמעותי את הזמן הדרוש כדי לסיים הקרנה מיקרו (למשל, 7 x 1024 פיקסלים ארוך ROI לוקח 14402 אלפיות שני באמצעות 1000 μs זמן השתהות ו 21598 אלפיות שני באמצעות זמן השתהות 3000 מיקרו). הפעם הוא אבד מרכישת תמונה ויש לקחת בחשבון. בעת הדמיה של אירועי גיוס מהירים, השתמש בהחזר ההבשלה הקצר ביותר האפשרי ורק מיקרו-הקרנה תא אחד בכל פעם.
יתרונות ומגבלות על-פני שיטות סינכרון
עבור מחקרים ספציפיים למחזור התא, השיטות הקיימות כוללות סינכרון של תאים לשלבי מחזור תאים ספציפיים או באמצעות כתבים פלואורסצנטיים כדי לזהות את שלב מחזור התא הספציפי של התא. עם זאת, כל אחת משיטות אלה מספקת אתגרים ומגבלות משלה.
מערכת FUCCI3 (הסתמכות על חלבון פלואורסצנטי מתויג צורות חתוכים של CDT1 ו Geminin) הוא כלי שימושי במיוחד עבור מחקרי מחזור התא אבל יש מגבלות כשמדובר להבדיל בין S ו- G2 שלבים של מחזור התא. רמות Geminin כבר גבוהות משלב S באמצע ולהישאר גבוה עד שלב M, מה שהופך שלבים אלה קשה להפריד. שימוש במערכת FUCCI פירושו גם כי שני ערוצים אופטיים של המיקרוסקופ לא ניתן להשתמש עבור הדמיה POI.
קווי תאים שאינם סרטניים יכולים להיות מסונכרנים לתוך G0 על ידי הסרת גורמי גדילה שנמצאו בסרום (רעב בסרום) הגורמים נזק DNA קטן או ללא נזק לתאים. עם זאת, רוב קווי התא הסרטני ימשיכו להתקדם חלקית במחזור התאים גם ללא כמויות נאותות של סרום במדיה שלהם. בנוסף, תאים מתחילים חלקית לאבד סינכרון על ידי סוף G1, שלב S מוקדם. בנוסף לרעב בסרום, ישנן שיטות כימיות רבות להשגת סנכרון מחזור התא. הידרוקסיאוריה, אפידיקולין, ותימידין בלוקים הם שיטות לעצור שכפול DNA כדי לסנכרן תאים לשלב S מוקדם. בעוד שיטות אלה הן זולות ופשוטות, הם מציגים מתח שכפול אשר גורם נזק DNA. מעכבי שכפול DNA אלה הוכחו לגרום זרחן של H2A. X, סמן ידוע של DSBs2,29. שיטת השימוש ב- PCNA מתויג כסמן לתאי S-phase מפחיתה את הפוטנציאל לחפצים הנגרמים על ידי סנכרון כימי וניתן להחיל אותה על מגוון רחב של קווי תאים בהשוואה לרעב בסרום.
מסקנה
נזק לדנ"א הוא כוח מניע למחלות גנטיות שבהן נגעים מוטגניים יכולים להוביל לשינוי ממאיר של תאים. מיקוד מכונות סינתזת DNA היא אסטרטגיה טיפולית בסיסית בטיפול במחלות היפרפרוliferative כמו סרטן. על מנת לטפל במחלות אלה בצורה ממוקדת יותר, אנו זקוקים להבנה טובה יותר של החלבונים המתקנים נגעי DNA. הפרוטוקול המתואר כאן מסייע למחקרים מבוססי הקרנה זעירה בשלב S על ידי מזעור האתגרים המוצגים בשיטות סנכרון מסורתיות כדי להפחית חפצים אפשריים ולהגדיל את יכולת הרבייה של הניסויים.
Disclosures
המחברים מציינים כי פרסום העבודה המוצגת היה בחסות תאגיד ניקון. המחברים מצהירים כי אין אינטרסים מתחרים קיימים.
Acknowledgments
המחברים מודים למ. פגאנו על תמיכתו המתמשכת, כמו גם לד' סימונצ'י, א. מרציו וג'י טאנג על הביקורת הביקורתית שלהם על כתב היד. ב. מיוואטני-מינטר מודה לר' מיוואטני וב. מינטר על תמיכתם המתמשכת. ג. רונה מודה ל-ק. רונאן ג'ורה וג' רונה על תמיכתם המתמשכת.
Materials
| Name | Company | Catalog Number | Comments |
| Ammonium chloride | Sigma-Aldrich | A9434-500G | For quenching formaldehyde |
| Anti-EXO1 Rabbit Polyclonal Antibody | Proteintech | 16253-1-AP | primary antibody |
| Anti-phospho-Histone H2A.X (Ser139) Antibody, clone JBW301 | Millipore | 05-636 | primary antibody |
| Bovine Serum Albumin | Sigma-Aldrich | 3117332001 | BSA for blocking |
| BrdU (5-Bromo-2'-deoxyuridine) | Merck | 19-160 | pre-sensitizing agent |
| Citifluor™ Mountant Solution AFR3 | Electron Microscopy Sciences | 17973-10 | antifade containing PBS solution for imaging |
| DAPI | Sigma-Aldrich | D9542-1MG | nucleic acid stain |
| DMEM Medium | Thermo Fisher Scientific | 10569010 | Cell culture medium for HEK293T cells |
| DMSO | Sigma-Aldrich | D2650-100ML | Vehichle control and dissolution solvent |
| EGFP-FBXL10 | Addgene | #126542 | viral expression vector for EGFP-FBXL10 |
| EXO1b-AcGFP (in pRetroQ) | custom cloning | na | EXO1b cDNA was cloned in the NheI, BamHI sites of pRetroQ-AcGFP1-N1 vector. |
| Fetal Bovine Serum | Gibco | 16140071 | Media supplement |
| FluoroBrite DMEM | Thermo Fisher Scientific | A1896701 | Phenol red free medium for microscopy |
| Goat anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 488 | Thermo Fisher Scientific | A32723 | secondary antibody |
| HEK293T cells | ATCC | ATCC CRL-3216 | Cell line for viral packaging |
| HEPES | Sigma-Aldrich | H0887-100ML | Buffering agent to supplement live cell imaging medium |
| Hoechst 33342 | Thermo Fisher Scientific | H3570 | pre-sensitizing agent |
| Lipofectamine 3000 | Thermo Fisher Scientific | L3000015 | Transfection reagent |
| McCoy’s 5A (Modified) Medium | Life Technologies | 16600-108 | Cell culture medium for U-2 OS cells |
| mCherry-PCNA | Addgene | #55117 | non-viral PCNA construct suitable for cell cycle marker |
| mPlum-PCNA | Addgene | #55994 | non-viral PCNA construct suitable for cell cycle marker |
| mPlum-PCNA (in pBABE) | custom cloning | na | mPlum-PCNA cDNA was cloned from Addgene #55994 in the BamHI, SalI sites of pBABE (puro) |
| Nikon A1R-HD25 Confocal Scanhead and Controller | Nikon | na | confocal imaging system |
| Nikon LUN4 laser unit | Nikon | na | excitation system |
| Nikon LUN-F 50 mW 405 nm FRAP laser unit | Nikon | na | FRAP laser unit |
| Nikon NIS Elements Confocal Controller Software | Nikon | na | Confocal controlling software |
| Nikon Ti2-E Inverted Microscope | Nikon | na | inverted epifluorescent microscope base |
| Nikon Ti2-LAPP Modular Illumination System | Nikon | na | illumination system |
| NTHL1-mCherry (in pRetroQ) | custom cloning | na | NTHL1 cDNA was cloned in the NheI, SalI sites of pRetroQ-mCherry-N1 vector. |
| Nunc Lab-Tek II Chambered Coverglass (4 well) | Thermo Fisher Scientific | 155382PK | Live cell microscopy cell culture chamber |
| Olaparib | Selleck Chemicals | S1060 | PARP inhibitor |
| Opti-MEM reduced serum media | Thermo Fisher Scientific | 31985062 | Dilution medium for transient transfection |
| Paraformaldehyde aqueous solution (32%) | Thermo Fisher Scientific | 50-980-494 | Fixative |
| pBABE (hygro) | Addgene | #1765 | retroviral expression vector (for low expression levels) |
| pBABE (neo) | Addgene | #1767 | retroviral expression vector (for low expression levels) |
| pBABE (puro) | Addgene | #1764 | retroviral expression vector (for low expression levels) |
| pBABE (zeo) | Addgene | #1766 | retroviral expression vector (for low expression levels) |
| PCNA Antibody (PC10) | Santa Cruz | sc-56 | primary antibody |
| Penicillin-Streptomycin-Glutamine (100x) | Gibco | 10378016 | Media supplement |
| polybrene | Sigma-Aldrich | TR-1003 | Increase viral infection efficiency |
| pRetroQ-AcGFP-C1 | Takara | 632506 | retroviral expression vector |
| pRetroQ-AcGFP-N1 | Takara | 632505 | retroviral expression vector |
| pRetroQ-mCherry-C1 | Takara | 632567 | retroviral expression vector |
| pRetroQ-mCherry-N1 | Takara | 632568 | retroviral expression vector |
| pUMVC | Addgene | #8449 | Viral packaging vector |
| Sodium-pyruvate | Thermo Fisher Scientific | 11360070 | Supplement for live cell imaging medium |
| Triton X-100 aqueous solution (10%) | Sigma-Aldrich | 11332481001 | Dilute in PBS for cell permeabilization buffer |
| Trypsin-EDTA Solution 10X | Sigma-Aldrich | 59418C-100ML | Dilute in PBS to split cells |
| U-2 OS Cells | ATCC | HTB-96 | Optimal cell line for microscopy experiments |
| Universal Mycoplasma Detection Kit | ATCC | 30-1012K | PCR based Mycoplasma detection kit |
| VSV-G | Addgene | #8454 | Viral protein envelope vector |
References
- Aleksandrov, R., et al. Protein dynamics in complex DNA lesions. Molecular Cell. 69 (6), 1046-1061 (2018).
- Darzynkiewicz, Z., Halicka, H. D., Zhao, H., Podhorecka, M. Cell synchronization by inhibitors of DNA replication induces replication stress and DNA damage response: Analysis by flow cytometry. Methods in Molecular Biology. 761, 85-96 (2011).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Herce, H. D., Rajan, M., Lattig-Tunnemann, G., Fillies, M., Cardoso, M. C. A novel cell permeable DNA replication and repair marker. Nucleus. 5 (6), 590-600 (2014).
- Keijzers, G., et al. Human exonuclease 1 (EXO1) regulatory functions in dna replication with putative roles in cancer. International Journal of Molecular Sciences. 20 (1), (2018).
- Cheruiyot, A., et al. Poly(ADP-ribose)-binding promotes Exo1 damage recruitment and suppresses its nuclease activities. DNA Repair (Amsterdam). 35, 106-115 (2015).
- Zhang, F., Shi, J., Chen, S. H., Bian, C., Yu, X. The PIN domain of EXO1 recognizes poly(ADP-ribose) in DNA damage response. Nucleic Acids Research. 43 (22), 10782-10794 (2015).
- Rona, G., et al. PARP1-dependent recruitment of the FBXL10-RNF68-RNF2 ubiquitin ligase to sites of DNA damage controls H2A.Z loading. elife. 7, (2018).
- Young, L. M., et al. TIMELESS forms a complex with PARP1 distinct from its complex with TIPIN and plays a role in the dna damage response. Cell Reports. 13 (3), 451-459 (2015).
- Kong, X., et al. Laser microirradiation to study in vivo cellular responses to simple and complex dna damage. Journal of Visualized Experiments. (131), e56213 (2018).
- Kong, X., et al. Condensin I recruitment to base damage-enriched DNA lesions is modulated by PARP1. PLoS One. 6 (8), 23548 (2011).
- Lan, L., et al. Novel method for site-specific induction of oxidative DNA damage reveals differences in recruitment of repair proteins to heterochromatin and euchromatin. Nucleic Acids Research. 42 (4), 2330-2345 (2014).
- Zerjatke, T., et al. Quantitative cell cycle analysis based on an endogenous all-in-one reporter for cell tracking and classification. Cell Reports. 19 (9), 1953-1966 (2017).
- Ji, Y., Karbaschi, M., Cooke, M. S. Mycoplasma infection of cultured cells induces oxidative stress and attenuates cellular base excision repair activity. Mutation Research. 845, 403054 (2019).
- Sun, G., et al. Mycoplasma pneumoniae infection induces reactive oxygen species and DNA damage in A549 human lung carcinoma cells. Infection and Immunity. 76 (10), 4405-4413 (2008).
- Gassman, N. R., Wilson, S. H. Micro-irradiation tools to visualize base excision repair and single-strand break repair. DNA Repair (Amsterdam). 31, 52-63 (2015).
- Muster, B., Rapp, A., Cardoso, M. C. Systematic analysis of DNA damage induction and DNA repair pathway activation by continuous wave visible light laser micro-irradiation. AIMS Genetics. 4 (1), 47-68 (2017).
- Ikeda, S., et al. Purification and characterization of human NTH1, a homolog of Escherichia coli endonuclease III. Direct identification of Lys-212 as the active nucleophilic residue. Journal of Biological Chemistry. 273 (34), 21585-21593 (1998).
- Rosenquist, T. A., Zharkov, D. O., Grollman, A. P. Cloning and characterization of a mammalian 8-oxoguanine DNA glycosylase. Proceedings of the National Academy of Science U. S. A. 94 (14), 7429-7434 (1997).
- Reid, D. A., et al. Organization and dynamics of the nonhomologous end-joining machinery during DNA double-strand break repair. Proceedings of the National Academy of Science U. S. A. 112 (20), 2575-2584 (2015).
- Taccioli, G. E., et al. Ku80: product of the XRCC5 gene and its role in DNA repair and V(D)J recombination. Science. 265 (5177), 1442-1445 (1994).
- Marsin, S., et al. Role of XRCC1 in the coordination and stimulation of oxidative DNA damage repair initiated by the DNA glycosylase hOGG1. Journal of Biological Chemistry. 278 (45), 44068-44074 (2003).
- Thompson, L. H., Brookman, K. W., Jones, N. J., Allen, S. A., Carrano, A. V. Molecular cloning of the human XRCC1 gene, which corrects defective DNA strand break repair and sister chromatid exchange. Molecular and Cell Biology. 10 (12), 6160-6171 (1990).
- Scharer, O. D. Nucleotide excision repair in eukaryotes. Cold Spring Harbor Perspective Biology. 5 (10), 012609 (2013).
- Oeck, S., et al. High-throughput evaluation of protein migration and localization after laser micro-irradiation. Science Reports. 9 (1), 3148 (2019).
- Mistrik, M., et al. Cells and stripes: A novel quantitative photo-manipulation technique. Science Reports. 6, 19567 (2016).
- Durand, R. E., Olive, P. L. Cytotoxicity, mutagenicity and dna damage by hoechst 33342. Journal of Histochemistry and Cytochemistry. 30 (2), 111-116 (1982).
- Tobey, R. A., Oishi, N., Crissman, H. A. Cell cycle synchronization: reversible induction of G2 synchrony in cultured rodent and human diploid fibroblasts. Proceedings of the National Academy of Science U. S. A. 87 (13), 5104-5108 (1990).
- Podhorecka, M., Skladanowski, A., Bozko, P. H2AX phosphorylation: Its role in DNA damage response and cancer therapy. Journal of Nucleic Acids. 2010, (2010).



