Summary
Gli endosomi di riciclaggio fanno parte della rete tubulare endosomiale. Qui presentiamo un metodo per quantificare le dinamiche del riciclaggio degli endosomi utilizzando GFP-STX13 come marcatore di organelli.
Abstract
Gli endosomi di riciclaggio (RE) sono organelli tubolari-vescicolari generati da endosomi precoci / di selezione in tutti i tipi di cellule. Questi organelli svolgono un ruolo chiave nella biogenesi dei melanosomi, un organello correlato al lisosoma prodotto dai melanociti. Le RE trasportano il carico specifico dei melanociti ai melanosomi prematuri durante la loro formazione. Il blocco nella generazione di RE, osservato in diversi mutanti della sindrome di Hermansky-Pudlak, provoca ipopigmentazione di pelle, capelli e occhi. Pertanto, studiare la dinamica (riferirsi a numero e lunghezza) delle RE è utile per comprendere la funzione di questi organelli in condizioni normali e di malattia. In questo studio, miriamo a misurare la dinamica RE utilizzando uno SNARE STX13 residente.
Introduction
La biosintesi dei pigmenti di melanina si verifica nei melanosomi, un organello correlato al lisosoma specifico per i melanociti (LRO) che coesiste con i lisosomi convenzionali. Il sistema endocitico svolge un ruolo chiave nella biogenesi dei melanosomi, necessari per il colore della pelle e la fotoprotezione contro le radiazioni ionizzanti1,2,3. Durante questo processo, gli enzimi di sintesi della melanina vengono smistati su endosomi precoci / di selezione e quindi trasportati a melanosomi prematuri attraverso endosomi tubulari o vescicolari chiamati endosomi di riciclaggio (RE)4,5,6,7,8,9,10. Il targeting e la fusione di questi organelli regolano la maturazione dei melanosomi pigmentati completamente funzionali7,11,12,13,14. Difetti nella formazione di questi organelli o nella selezione del carico a questi organelli causano albinismo oculocutaneo e altri fenotipi clinici, osservati nella sindrome di Hermansky-Pudlak15,16.
Qui descriviamo una semplice tecnica basata sulla microscopia per studiare e analizzare i RE. In questo metodo, abbiamo sfruttato una proteina transmembrana, Qa-SNARE Syntaxin (STX)13 che risiede sul riciclaggio degli endosomi17 e cicli tra endosomi di selezione e melanosomi nei melanociti12,18. Inoltre, la delezione del dominio normativo non strutturato N-terminale (vale a dire SynN o STX13Δ129) consente allo SNARE di rimanere bloccato nei melanosomi, che misura la via di traffico in avanti verso il melanosoma12. Abbiamo utilizzato un noto marcatore endosomiale di riciclaggio Rab GTPasi (Rab)11 nei nostri studi14,19. L'imaging a fluorescenza delle proteine GFP-STX13WT, GFP-STX13Δ129, mCherry-Rab11 e TYRP1 nei melanociti wild type seguito dalla quantificazione della loro localizzazione relativa fornirà la natura e la dinamica delle RE oltre al loro targeting ai melanosomi. Pertanto, questa è una tecnica semplice che può essere utilizzata per visualizzare e misurare la dinamica delle RE nei melanociti.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Il protocollo prevede la semina dei melanociti seguita dalla trasfezione dei plasmidi. Ulteriori passaggi includono la fissazione, l'immunocolorazione, l'imaging e l'analisi delle cellule per misurare la lunghezza e il numero di RE. La descrizione dettagliata del protocollo è riportata di seguito.
1. Semina di melanociti di topo su coverslip pretrattati
- Rivestire le coperture di vetro in una capsula di Petri (cioè 4 - 5 in una parabola da 35 mm) con mezzo a matrice di membrana basale (1:20 in mezzo RPMI completo: RPMI + FBS inattivato al calore 10% + 1x glutammina + 1x miscela antibiotica) e asciugarlo nella cappa di coltura tissutale per 15 min. Lavare le coverslip una volta con 1x PBS prima dell'uso.
- Mantenere i melanociti di topo wild type (melan-Ink4a-Arf-1 da C57BL/6J, a/a, Topi Ink4a-Arf-/- , descritti in20 e disponibili presso The Welcome Trust Functional Genomics Cell Bank) in una capsula di Petri (cioè una capsula da 35 o 60 mm) integrata con un mezzo RPMI completo.
- Lavare le cellule due volte con 1x PBS e aggiungere 0,5 o 1 mL di soluzione di tripsina-EDTA (0,25%) per le cellule tripsinizzanti. Incubare le cellule a 37 °C per 2 - 5 minuti per il distacco dalla capsula di Petri.
- Aggiungere 1 - 2 ml di mezzo RPMI completo, sospendere e quindi trasferire le celle in un tubo centrifugo.
- Centrifugare la sospensione cellulare a 4 °C, 376 x g per 5 minuti e quindi risospesere il pellet in 1x PBS.
- Ripetere la fase di centrifugazione e risospescere le celle in 1 mL di supporto RPMI completo.
- Seminare le cellule sulla membrana basale con coperture medie al 50-60% di confluenza (circa 6 x 105 cellule su una parabola di coltura cellulare di 35 mm contenente 4 - 5 coverslips). Aggiungere sempre 200 nM di PMA (aggiungere 5 μL di stock di lavoro 40 μM phorbol 12-myristate 13-acetato) alla sospensione cellulare placcata in mezzo RPMI completo.
- Dopo la semina, incubare la piastra a 37 °C per 12-24 ore.
2. Trasfezione di cellule con i plasmidi STX13
- Utilizzare i seguenti reagenti: pEGFP-C1-STX13WT e pEGFP-C1-STX13Δ129 (descritto in12). mCherry-Rab11, è stato un gentile dono di Graça Raposo, Institut Curie, Parigi (descritto nel riferimento19). Anticorpo Anti-TYRP1 di ATCC (TA99).
- Dopo 12 - 24 ore di semina, trasfettare le cellule con plasmidi utilizzando un reagente di trasfezione a base lipidica. Per un piatto da 35 mm, prelevare 5 μL del reagente di trasfezione in 250 μL di mezzo OPTI-MEM in un tubo microcentrifuga e prelevare circa 200 ng di ciascun plasmide in 250 μL di OPTI-MEM.
- Incubare le provette contenenti DNA e il reagente di trasfezione per 5 min. Miscelare senza pipettaggio ripetuto (il volume totale sarà di circa 500 μL). Incubare per 30 minuti a temperatura ambiente. Eseguire la picchiettatura manuale del tubo ogni 10 minuti per 30 minuti a RT.
- Durante l'incubazione, lavare le cellule due volte con 1x PBS, una volta con OPTI-MEM e quindi aggiungere 1 mL di OPTI-MEM alle cellule.
- Dopo 30 minuti di incubazione, aggiungere la miscela reagente-DNA di trasfezione alle cellule in modo goccioso coprendo il piatto.
- Incubare le cellule a 37 °C per 6 ore. Aspirare il mezzo OPTI-MEM con reagente di trasfezione e aggiungere un mezzo RPMI completo integrato con 200 nM di PMA.
- Incubare le cellule a 37 °C per 48 ore.
3. Fissazione delle cellule
NOTA: La seguente procedura viene eseguita al di fuori del cappuccio di coltura del tessuto.
- Dopo 48 ore di trasfezione, lavare le cellule due volte con 1x PBS e quindi fissare le cellule con il 3% di formaldeide (preparata al momento in 1x PBS) per 30 min.
- Dopo la fissazione, lavare le celle due volte con 1x PBS e conservare le coverslip in 1x PBS fino a nuovo utilizzo. In alternativa, le celle possono essere montate su vetrini (vedi sotto) o conservate a 4 °C.
4. Immunocolorazione delle cellule
- Preparare una camera umida: posizionare il pezzo tagliato con pellicola di paraffina su carta da filtro umida in una capsula di Petri, coperta con un foglio di alluminio.
- Preparare 25 μL di soluzione anticorpale primaria (0,2% di saponina in 1x PBS, 0,1% BSA in 1x PBS e 0,02% di sodio azide in 1x PBS). Aggiungere l'anticorpo ad una diluizione di 1:200. Aggiungere questa soluzione come goccia su un film di paraffina nella camera umida.
- Sollevare con attenzione il coperchio con una pinza, capovolgerlo su questa goccia di soluzione di colorazione anticorpale primaria e quindi coprire il coperchio della camera umida. Incubare a temperatura ambiente per 30 min.
- Allo stesso modo, preparare la soluzione anticorpale secondaria ad una diluizione di 1:500 e posizionarla su un film di paraffina accanto al coperchio nella camera umida. Per colorare il nucleo, aggiungere DAPI (da 1:20.000 a 1:30.000) alla soluzione.
- Usando una pinza, raccogliere con attenzione il coverslip dalla soluzione anticorpale primaria e immergerlo tre volte in 1x PBS (in un becher di vetro).
- Toccare il coperchio su carta velina per rimuovere il PBS in eccesso sul coperchio. Posizionarlo su una soluzione di colorazione anticorpale secondaria nella camera umida e non esporlo alla luce a causa della presenza di anticorpi marcati fluorescentemente nella soluzione.
- Incubare nuovamente il coverslip per 30 minuti a temperatura ambiente. Si prega di tenere sempre una nota sul lato del coverslip che ha le celle in questi passaggi.
- Dopo l'incubazione, prelevare con cura il coverslip dalla soluzione anticorpale secondaria e quindi immergerlo tre volte in 1x PBS. Inoltre, toccare il coperchio su carta velina per rimuovere il PBS in eccesso sul coperchio.
- Posizionare 12 μL di reagente di montaggio Fluoromount-G su un vetrino e posizionare con attenzione il coperchio colorato (rivolto verso il vetro) sul reagente di montaggio. Capovolgere il vetrino su carta velina e quindi premere delicatamente.
5. Microscopia a fluorescenza delle cellule
- Immagina le cellule colorate sotto i filtri a campo luminoso (BF) e a fluorescenza (IF) utilizzando un microscopio a fluorescenza invertita dotato di una telecamera CCD che utilizza un obiettivo apocromatico 60x (olio) o qualsiasi altro microscopio con una configurazione simile.
6. Quantificazione della sovrapposizione tra le proteine localizzate RE e i melanosomi:
NOTA: Per la quantificazione del coefficiente di sovrapposizione di Mander tra le proteine si seguono i seguenti passaggi (scaricabili gratuitamente dal link: https://imagej.net/software/fiji/). Utilizzare l'immagine TIFF con più canali.
- Aprite l'immagine raw. Vai all'opzione Immagine, seleziona Colore | Dividere i canali e utilizzare i due canali per l'analisi.
- Apri il plugin JACoP nell'opzione Plugin.
- Impostare la soglia per entrambi i canali in modo che tutti i punti luminosi siano selezionati e lo sfondo venga eliminato.
- Vai all'opzione Analisi, seleziona i coefficienti M1 e M2 per ottenere il coefficiente di sovrapposizione di Mander.
- Premi l'opzione Analizza nel plugin JACoP e vedi il risultato che visualizza il coefficiente di sovrapposizione di Mander.
7. Quantificazione del numero tubolare e della lunghezza degli endosomi riciclati:
NOTA: I seguenti passaggi sono seguiti per la quantificazione del numero e della lunghezza dei tubuli utilizzando il software Fiji.
- Apri l'opzione Vai all'immagine raw, seleziona Colore | Dividere i canali e utilizzare il canale desiderato per l'analisi.
- Vai di nuovo a Immagine , seleziona Tipo | Converti in immagine a 8 bit.
- Quindi vai su Plugin, seleziona Analizza | Tubosità. Impostare il valore sigma su 0,1075 . Premere OK.
- Vai di nuovo a Immagine , seleziona Tipo | Converti in immagine a 8 bit.
- Vai a | immagine Regolare | Soglia (Utilizzare gli stessi valori di soglia per tutte le immagini. Le immagini dovrebbero avere un'intensità approssimativamente uguale).
- Vai a | processo | binario Converti in maschera.
- Vai a Processo, seleziona Binario e quindi seleziona Scheletra.
- Andare su Analizza, selezionare Ossatura e scegliere Analizza ossatura. Nel risultato e nell'output | selezionare (a) Calcola il percorso più corto più grande | (b) mostra informazioni dettagliate | (c) visualizza lo scheletro etichettato. Premere OK.
NOTA: il risultato verrà aperto nel formato tabulato. La colonna di Lunghezza media del ramo mostra la lunghezza di tutti i diversi tubuli nella cella selezionata (scala impostata per ottenere valori in micrometri). - Per ottenere il numero di tubuli nella cella, andare su Analizza e selezionare l'opzione Analizza particella . Premere OK.
NOTA: nei risultati ottenuti, la colonna conteggio mostra il numero di tubuli in quella particolare cella. - Salva i dati e analizzali.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Quantificazione della localizzazione mutante STX13Δ129 ai melanosomi
La microscopia a immunofluorescenza di STX13 in melanociti wild type di topo ha mostrato GFP-STX13WT localizzato come strutture vescicolari e tubulari e GFP-STX13Δ129 localizzato come strutture ad anello oltre alla superficie cellulare (Figura 1A). Inoltre, il GFP-STX13Δ129 ad anello intracellulare ha mostrato colocalizzazione con la proteina melanosoma TYRP1 (Figura 1A) e melanosomi con immagini a campo luminoso (dati non mostrati)12. Come mostrato in precedenza, una coorte di GFP-STX13WT sovraespressa è osservata nei melanosomi12. Per misurare la localizzazione relativa di GFP-STX13WT e GFP-STX13Δ129 ai melanosomi, abbiamo usato Fiji e analizzato con il plugin JACoP. Il coefficiente di sovrapposizione di Mander misurato (MOC) tra GFP-STX13Δ129 con TYRP1 è circa 1,5 volte superiore rispetto a GFP-STX13WT con TYRP1 (Figura 1B). È interessante notare che TYRP1 ha mostrato valori MOC 2,9 volte più alti con GFP-STX13Δ129 rispetto a GFP-STX13WT (Figura 1B). Questi dati indicano che la localizzazione di GFP-STX13Δ129 in melanosomi è relativamente più alta rispetto a GFP-STX13WT a uno stato stazionario.
Quantificazione della localizzazione STX13WT agli endosomi di riciclo
La microscopia a immunofluorescenza di GFP-STX13WT ha mostrato colocalizzazione con proteina endosomiale rab11 nota di riciclaggio (espressa come mCherry-Rab11) (Figura 2A,B). Il MOC misurato tra GFP-STX13WT con mCherry-Rab11 è circa 1,4 volte superiore rispetto a mCherry-Rab11 con GFP-STX13WT (Figura 2B). Per misurare il numero e la lunghezza dei tubuli endosomiali GFP-STX13WT-positivi, abbiamo utilizzato il software Fiji come descritto nella sezione protocollo. mCherry-Rab11 è usato come controllo positivo negli esperimenti (Figura 2). I melanociti trasfettati con GFP-STX13WT hanno mostrato un numero maggiore di tubuli per cellula rispetto alle cellule che esprimono mCherry-Rab11 (Figura 2C grafico in alto, confronta la barra A con la barra B). Tuttavia, i numeri dei tubuli sono ridotti in caso di co-espressione di GFP-STX13WT e mCherry-Rab11 nelle celle (Figura 2C grafico superiore, confronta la barra A con C e la barra B con D). È interessante notare che la lunghezza media dei tubuli (μm) sia per GFP-STX13WT che per mCherry-Rab11 è paragonabile tra loro nelle cellule che si esprimono individualmente o insieme (Figura 2C grafico in basso). Insieme, questi dati suggeriscono che GFP-STX13WT si localizza in RE come simile a Rab11.
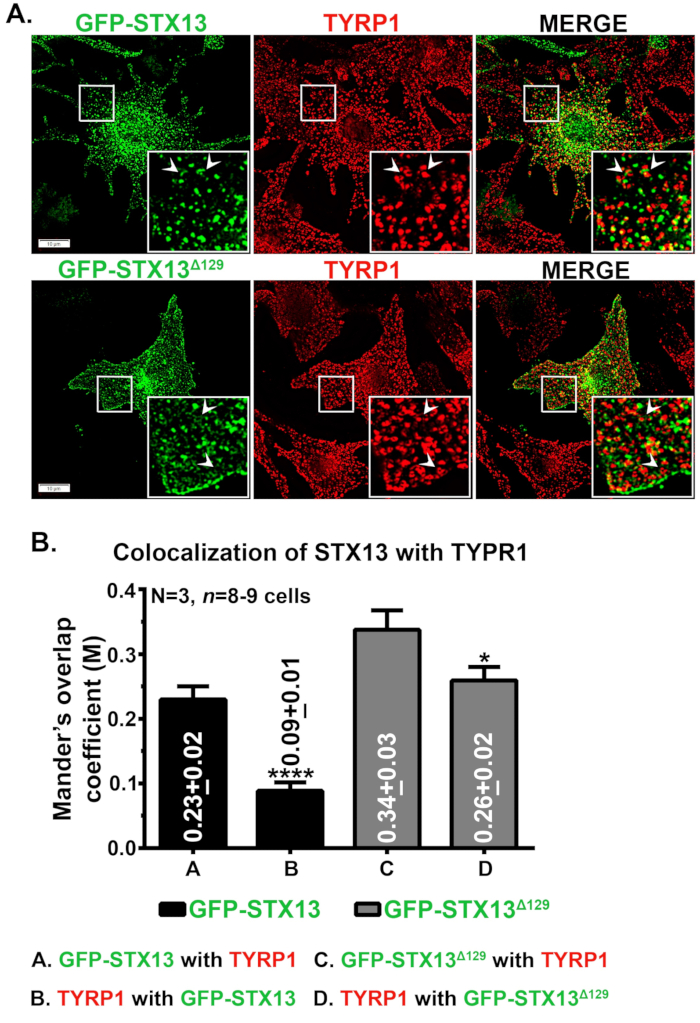
Figura 1: Localizzazione di GFP-STX13WT e GFP-STX13Δ129 in melanosomi in melanociti wild type. (A) I melanociti Melan-Ink4a-Arf-1 sono stati trasfettati con GFP-STX13WT e GFP-STX13Δ129. Le cellule sono state fissate, colorate con anticorpi anti-TYRP1 e poi analizzate al microscopio a fluorescenza. Gli inserti sono ingranditi delle aree bianche in scatola. Le frecce puntano alla localizzazione di GFP-STX13WTto RE e GFP-STX13Δ129 in melanosomi. Barre di scala, 10 μm. (B) Quantificazione della colocalizzazione tra STX13 e TYRP1. Il coefficiente di sovrapposizione di Mander (M) tra GFP-STX13WT o GFP-STX13Δ129 con TYRP1 e viceversa è rappresentato (media ± S.E.M.) separatamente nel grafico. N=3. *p ≤0,05 e ****p ≤0,0001. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Localizzazione di GFP-STX13WT per il riciclaggio di endosomi in melanociti wild type. (A) I melanociti Melan-Ink4a-Arf-1 sono stati trasfettati con GFP-STX13WT e mCherry-Rab11. Le cellule sono state fissate e analizzate al microscopio a fluorescenza. Gli inserti sono ingranditi delle aree bianche in scatola. Le frecce indicano la localizzazione di GFP-STX13WT in compartimenti mCherry-Rab11-positivi. Barre di scala, 10 μm. (B) Quantificazione della colocalizzazione tra STX13 e Rab11. Il coefficiente di sovrapposizione di Mander (M) tra GFP-STX13WT con mCherry-Rab11 e viceversa è rappresentato (media ± S.E.M.) separatamente nella trama. N=3. p ≤0.001. C. Quantificazione del numero e della lunghezza (in μm) delle RE STX13- o Rab11-positive. Il numero medio di tubuli/cellule e la lunghezza media dei tubuli di GFP-STX13WT e mCherry-Rab11 sono rappresentati (media ± S.E.M.) separatamente nel grafico. N=3. Si noti che le cellule sono trasfettate con GFP-STX13WT e mCherry-Rab11 in (C) e (D). Fare clic qui per visualizzare una versione più grande di questa figura.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Gli endosomi di riciclaggio sono una coorte di organelli endocitici e mediano il riciclaggio del carico sulla superficie cellulare in tutti i tipi di cellule21,22,23,24,25. In tipi di cellule specializzate come i melanociti, questi organelli deviano in parte la loro rotta di traffico verso i melanosomi per la loro biogenesi3,16,26. Inoltre, i RE sono generati dopo lo smistamento del carico su endosomi precoci / smistatori e morfologicamente appaiono come strutture tubolari-vescicolari. La loro dinamica (numero e lunghezza) dipende da diversi fattori cellulari, tra cui le proteine motorie21,22,23,24,25,27,28. Pochi studi hanno utilizzato Rab11 come marcatore per etichettare le RE nei melanociti14,19. Mentre nei non melanociti, STX13 e KIF13A sono stati utilizzati in aggiunta a Rab11 per visualizzare e caratterizzare REs13,17,19. In questo studio, abbiamo usato STX13 come marcatore per etichettare le RE nei melanociti seguito da un confronto con i tubuli Rab11-positivi (Figura 2). È interessante notare che la delezione N-terminale in STX13 (GFP-STX13Δ129) provoca una cattiva localizzazione dello SNARE in melanosomi (Figura 1). Pertanto, la localizzazione di GFP-STX13WT e GFP-STX13Δ129 nei melanociti visualizza rispettivamente i RE e i melanosomi. Queste proteine possono essere utilizzate come rispettivi marcatori di organelli per gli studi di localizzazione dello stato stazionario.
Gli studi hanno dimostrato che diverse proteine come TYRP1, TYR (tirosinasi), VAMP7, OCA2, Rab32/38 localizzano ai melanosomi oltre ad altri organelli intracellulari3,29. I nostri studi hanno dimostrato che il mutante N-terminale cancellato STX13 (GFP-STX13Δ129) si localizza nei melanosomi e nella membrana plasmatica. Ipotizziamo che GFP-STX13Δ129 possa essere utilizzato come reporter per studiare il traffico da RE alla superficie cellulare e LRO. Al contrario, GFP-STX13WT localizza in RE come simile a Rab11. I nostri studi hanno dimostrato che GFP-STX13WT potrebbe anche essere utilizzato per marcare IRE nei melanociti. Prevediamo che GFP-STX13WT possa essere un marcatore RE migliore di Rab11 poiché la sovraespressione di Rab11 altera la dinamica endosomiale. Complessivamente, GFP-STX13WT agisce come un potenziale marcatore RE per studiare le loro dinamiche in condizioni di stato stazionario.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Gli autori dichiarano di non avere alcun conflitto di interessi.
Acknowledgments
Questo lavoro è stato sostenuto dal Dipartimento di Biotecnologie (BT/PR32489/BRB/10/1786/2019 a SRGS); Consiglio di ricerca scientifica e ingegneristica (CRG/2019/000281 alla SRGS); DBT-NBACD (BT/HRD-NBA-NWB/38/2019-20 a SRGS) e programma di partnership IISc-DBT (a SRGS). L'infrastruttura del dipartimento era supportata da DST-FIST, DBT e UGC. AMB è stato supportato da DBT-JRF (DBT/2015/IISc/NJ-02).
Materials
| Name | Company | Catalog Number | Comments |
| anti-TYRP1 antibody (TA99) | ATCC | HB-8704 | |
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Lipofectamine 2000 | ThermoFisher Scientific | 11668-500 | |
| Matrigel matrix | BD Biosciences | 356231 | |
| OPTI-MEM | ThermoFisher Scientific | 022600-050 | |
| Phorbol 12-myristate 13-acetate | Sigma-Aldrich | P8139 | |
| RPMI Medium 1640 | ThermoFisher Scientific | 31800-022 |
References
- Dell'Angelica, E. C. The building BLOC(k)s of lysosomes and related organelles. Current Opinion in Cell Biology. 16 (4), 458-464 (2004).
- Raposo, G., Marks, M. S. Melanosomes--dark organelles enlighten endosomal membrane transport. Nature Reviews in Molecular Cell Biology. 8 (10), 786-797 (2007).
- Ohbayashi, N., Fukuda, M. Recent advances in understanding the molecular basis of melanogenesis in melanocytes. F1000Research. 9, F1000 Faculty Rev-608 (2020).
- Theos, A. C., et al. Functions of adaptor protein (AP)-3 and AP-1 in tyrosinase sorting from endosomes to melanosomes. Molecular Biology of the Cell. 16 (11), 5356-5372 (2005).
- Di Pietro, S. M., et al. BLOC-1 interacts with BLOC-2 and the AP-3 complex to facilitate protein trafficking on endosomes. Molecular Biology of the Cell. 17 (9), 4027-4038 (2006).
- Setty, S. R., et al. BLOC-1 is required for cargo-specific sorting from vacuolar early endosomes toward lysosome-related organelles. Molecular Biology of the Cell. 18 (3), 768-780 (2007).
- Delevoye, C., et al. AP-1 and KIF13A coordinate endosomal sorting and positioning during melanosome biogenesis. Journal of Cell Biology. 187 (2), 247-264 (2009).
- Bultema, J. J., Ambrosio, A. L., Burek, C. L., Di Pietro, S. M. BLOC-2, AP-3, and AP-1 proteins function in concert with Rab38 and Rab32 proteins to mediate protein trafficking to lysosome-related organelles. Journal of Biological Chemistry. 287 (23), 19550-19563 (2012).
- Sitaram, A., et al. Differential recognition of a dileucine-based sorting signal by AP-1 and AP-3 reveals a requirement for both BLOC-1 and AP-3 in delivery of OCA2 to melanosomes. Molecular Biology of the Cell. 23 (16), 3178-3192 (2012).
- Nag, S., et al. Rab4A organizes endosomal domains for sorting cargo to lysosome-related organelles. Journal of Cell Science. 131 (18), (2018).
- Dennis, M. K., et al. BLOC-2 targets recycling endosomal tubules to melanosomes for cargo delivery. Journal of Cell Biology. 209 (4), 563-577 (2015).
- Jani, R. A., Purushothaman, L. K., Rani, S., Bergam, P., Setty, S. R. STX13 regulates cargo delivery from recycling endosomes during melanosome biogenesis. Journal Cell Science. 128 (17), 3263-3276 (2015).
- Shakya, S., et al. Rab22A recruits BLOC-1 and BLOC-2 to promote the biogenesis of recycling endosomes. EMBO Reports. 19 (12), 45918 (2018).
- Bowman, S. L., et al. A BLOC-1-AP-3 super-complex sorts a cis-SNARE complex into endosome-derived tubular transport carriers. Journal of Cell Biology. 220 (7), 202005173 (2021).
- Wei, M. L. Hermansky-Pudlak syndrome: a disease of protein trafficking and organelle function. Pigment Cell Research. 19 (1), 19-42 (2006).
- Bowman, S. L., Bi-Karchin, J., Le, L., Marks, M. S. The road to lysosome-related organelles: Insights from Hermansky-Pudlak syndrome and other rare diseases. Traffic. 20 (6), 404-435 (2019).
- Prekeris, R., Klumperman, J., Chen, Y. A., Scheller, R. H. Syntaxin 13 mediates cycling of plasma membrane proteins via tubulovesicular recycling endosomes. Journal of Cell Biology. 143 (4), 957-971 (1998).
- Mahanty, S., et al. Rab9A is required for delivery of cargo from recycling endosomes to melanosomes. Pigment Cell Melanoma Research. 29 (1), 43-59 (2016).
- Delevoye, C., et al. Recycling endosome tubule morphogenesis from sorting endosomes requires the kinesin motor KIF13A. Cell Reports. 6 (3), 445-454 (2014).
- Ha, L., et al. ARF functions as a melanoma tumor suppressor by inducing p53-independent senescence. Proceedings of the National Academy of Science U. S. A. 104 (26), 10968-10973 (2007).
- Soldati, T., Schliwa, M. Powering membrane traffic in endocytosis and recycling. Nature Reviews Molecular Cell Biology. 7 (12), 897-908 (2006).
- Grant, B. D., Donaldson, J. G. Pathways and mechanisms of endocytic recycling. Nature Reviews Molecular Cell Biology. 10 (9), 597-608 (2009).
- Hsu, V. W., Prekeris, R. Transport at the recycling endosome. Current Opinion in Cell Biology. 22 (4), 528-534 (2010).
- Taguchi, T. Emerging roles of recycling endosomes. Journal of Biochemistry. 153 (6), 505-510 (2013).
- Goldenring, J. R. Recycling endosomes. Current Opinion in Cell Biology. 35, 117-122 (2015).
- Delevoye, C., Marks, M. S., Raposo, G. Lysosome-related organelles as functional adaptations of the endolysosomal system. Current Opinion in Cell Biology. 59, 147-158 (2019).
- Hsu, V. W., Bai, M., Li, J. Getting active: protein sorting in endocytic recycling. Nature Reviews in Molecular Cell Biology. 13 (5), 323-328 (2012).
- Desfougeres, Y., D'Agostino, M., Mayer, A. A modular tethering complex for endosomal recycling. Nature Cell Biology. 17 (5), 540-541 (2015).
- Le, L., Sires-Campos, J., Raposo, G., Delevoye, C., Marks, M. S. Melanosome biogenesis in the pigmentation of mammalian skin. Integrated Computational Biology. 61 (4), 1517-1545 (2021).



