Overview
Quelle: Hannah L. Cebull1, Arvin H. Soepriatna1, John J. Boyle2 und Craig J. Goergen1
1 Weldon School of Biomedical Engineering, Purdue University, West Lafayette, Indiana
2 Mechanical Engineering & Materials Science, Washington University in St. Louis, St Louis, Missouri
Das mechanische Verhalten von Weichgeweben wie Blutgefäßen, Haut, Sehnen und anderen Organen wird stark durch ihre Zusammensetzung von Elastin und Kollagen beeinflusst, die Elastizität und Festigkeit bieten. Die Faserausrichtung dieser Proteine hängt von der Art des Weichgewebes ab und kann von einer einzigen bevorzugten Richtung bis hin zu komplizierten netzgebundenen Netzwerken reichen, die sich in krankem Gewebe verändern können. Daher verhalten sich Weichgewebe oft anisotrop auf Zell- und Organebene, was eine notwendigkeit dreidimensionale Charakterisierung erzeugt. Die Entwicklung einer Methode zur zuverlässigen Abschätzung von Dehnungsfeldern innerhalb komplexer biologischer Gewebe oder Strukturen ist wichtig, um Krankheiten mechanisch zu charakterisieren und zu verstehen. Dehnung stellt dar, wie Sichweich im Laufe der Zeit relativ verformt, und kann mathematisch durch verschiedene Schätzungen beschrieben werden.
Durch das Erfassen von Bilddaten im Laufe der Zeit können Verformungen und Dehnungen geschätzt werden. Alle medizinischen Bildgebungsmodalitäten enthalten jedoch eine gewisse Menge an Rauschen, was die Schwierigkeit erhöht, die in vivo-Stämme genau zu schätzen. Die hier beschriebene Technik bewindet diese Probleme erfolgreich, indem eine DDE-Methode (Direct Deformation Estimation) verwendet wird, um räumlich variierende 3D-Dehnungsfelder aus volumetrischen Bilddaten zu berechnen.
Zu den aktuellen Methoden zur Dehnungsschätzung gehören die digitale Bildkorrelation (DIC) und die digitale Volumenkorrelation. Leider kann DIC die Belastung von einer 2D-Ebene nur genau abschätzen, was die Anwendung dieser Methode stark einschränkt. Obwohl sie nützlich sind, haben 2D-Methoden wie DIC Schwierigkeiten, Dehnungen in Regionen zu quantifizieren, die einer 3D-Verformung unterzogen werden. Dies liegt daran, dass a-of-plane Bewegung Verformungsfehler erzeugt. Die digitale Volumenkorrelation ist eine anwendbarere Methode, die die anfänglichen Volumendaten in Regionen unterteilt und den ähnlichsten Bereich des verformten Volumens findet, wodurch Fehler a-of-plane reduziert werden. Diese Methode erweist sich jedoch als geräuschempfindlich und erfordert Annahmen über die mechanischen Eigenschaften des Materials.
Die hier gezeigte Technik beseitigt diese Probleme mit einer DDE-Methode und macht sie somit sehr nützlich bei der Analyse medizinischer Bildgebungsdaten. Darüber hinaus ist es robust bis hoch oder lokalisiert. Hier beschreiben wir die Erfassung von gated, volumetrischen 4D-Ultraschalldaten, deren Umwandlung in ein analyzierbares Format und die Verwendung eines benutzerdefinierten Matlab-Codes zur Abschätzung von 3D-Verformungen und entsprechenden Green-Lagrange-Stämmen, ein Parameter, der große Verformungen besser beschreibt. Der Green-Lagrange Dehnungstensor wird in vielen 3D-Dehnungsschätzungsmethoden implementiert, da er es ermöglicht, F aus einem Least Squares Fit (LSF) der Verschiebungen zu berechnen. Die folgende Gleichung stellt den Grün-Lagrange-Dehnungstensor Edar, wobei F und I den Verformungsgradienten bzw. den Identitätstensor zweiter Ordnung darstellen.
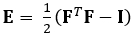 (1)
(1)
Principles
4D-Ultraschall ist ein dynamisches Volumen, das mit einem linear übersetzenden Motor an einem Ultraschallwandler erfasst wird, der die Erfassung von sequenziellen Herz- und Atemschlepren-Videoschleifen über einen Interessenbereich ermöglicht. Diese Methode ist nützlich für die Visualisierung komplexer Strukturen wie das Herz, wo Hypertrophie oder Infarkt einzigartige Geometrien verursacht, oder Aortenaneurysmen, bei denen asymmetrische Gefäßausdehnung und -sektion oft in gewundenen Gefäßen auftreten. Darüber hinaus können 4D-Daten hochauflösende räumliche und zeitliche Informationen liefern, was auch für die kardiovaskuläre Bildgebung wichtig ist.
Die Auf 4D-Ultraschalldaten angewendete DDE-Methode ist anderen Methoden überlegen, da sie eine nicht starre Bildregistrierung verwendet. Verformunggradiententensoren werden traditionell anhand von Verschiebungsfeldern nach der digitalen Volumenkorrelation geschätzt. Im Gegensatz dazu schätzt die DDE-Methode verformungsgradienten Tensoren während der Volumenregistrierung an sich, indem sie eine Verzugsfunktion optimiert, die sorgfältig so gewählt wird, dass sie direkt analog zum Verformungstensor ist. Die Verwarrfunktion hängt sowohl von der räumlichen Position als auch vom Warping-Parameter ab (p):
 (2)
(2)
Die ersten drei Elemente dieser Funktion stellen den Verformungsgradiententensor Fdar, der es ermöglicht, die Berechnung der Verformung direkt in die Verformungsfunktion einzubauen. Diese Verformungsmethode hat sich bewährt, um die Genauigkeit und Präzision der Dehnungsschätzung im Vergleich zu ähnlichen früheren Techniken zu erhöhen, da sie große oder lokalisierte Verformungen ermöglicht, die häufig in Weichgeweben zu finden sind.
Subscription Required. Please recommend JoVE to your librarian.
Procedure
1. 4D-Ultraschall-Setup
- Verwenden Sie bei der Verwendung der Imaging-Software einen Laptop, der mathematische Computersoftware ausführt, um den 4D-Erfassungsprozess zu automatisieren. Schließen Sie den Laptop mit diesem benutzerdefinierten Code über den USB-Anschluss an das Ultraschallsystem an. Beachten Sie, dass die Bildverarbeitungssoftware über eine 4D-Ultraschallfunktion verfügt, die in die Software integriert ist.
- Nach dem Einschalten des Ultraschallsystems richten Sie die physiologische Überwachungseinheit ein und stellen Sie gleichzeitig sicher, dass die Herzfrequenz- und Temperaturtasten eingeschaltet sind. Initialisieren Sie die am Wandlerhalter befestigte 3D-Motorstufe.
- Verwenden Sie den entsprechenden Bühnen- und Ultraschallwandler für die Bildaufnahme. Stellen Sie sicher, dass alle richtigen Verbindungen hergestellt werden.
- Fahren Sie mit der Anästhesisierung fort und bereiten Sie das Tier auf die Bildgebung vor. Fügen Sie den Augen eine ophthalmologische Salbe hinzu, um eine Hornhautaustrocknung zu verhindern, die Pfoten an den Bühnenelektroden zu befestigen und eine geschmierte rektale Temperatursonde einzufügen. Pelz im Interessenbereich mit einer Enthaarungscreme entfernen.
- Stellen Sie sicher, dass enthaart Creme abgeschlossen ist entfernt. Dann eine großzügige Menge des erwärmten Ultraschall-Transducing-Gels auf das Tier auftragen. Dies ist besonders wichtig, um eine gute Verbindung über die gesamte Interessensregion für die 4D-Bildgebung zu schaffen.
2. 4D-Ultraschall-Erfassung
- Beginnen Sie eine neue Studie über das Ultraschallsystem und öffnen Sie das Bildgebungsfenster im B-Modus (Helligkeitsmodus). Senken Sie den Messumformer auf das Tier und lokalisieren Sie den Interessenbereich mit den x- und y-Achsenknöpfen auf der Bühne, um sicherzustellen, dass die Atemfrequenz nicht wesentlich abnimmt. Überwachen Sie dies am unteren Bildschirmrand.
- Positionieren Sie den Messumformer in der Mitte der Interessenregion. Von dort aus, ungefähr die Entfernung für den Wandler benötigt, um nach oben und unten zu bewegen, so dass die gesamte Region von Interesse enthalten ist.
- Geben Sie die ungefähren Abmessungen in den Computersoftwarecode ein, einschließlich einer Schrittgröße, die in der Regel 0,08 mm für die Abdominalaortenaneurysm-Bildgebung beträgt. Beginnen Sie mit dem Ausführen des Codes, nachdem Sie sichergestellt haben, dass das Herz und die Atemfrequenzen des Tieres stabil sind. Dies ist wichtig, um Fehler beim Rekonstruieren von Bildern zu reduzieren.
- Exportieren Sie die Daten nach Abschluss der Bildaufnahme als XML-Rohdateien.
3. 4D-Ultraschall-Datenkonvertierung
- Geben Sie die UN-XML-Dateien in software ein, die die Daten in das richtige Format für die 3D-Fleckenanalyse konvertieren kann. Hier verwenden wir Matlab, um XML-Dateien in MAT-Dateien zu konvertieren. Das vollständige Matlab-Skript finden Sie hier.
- Für eine ordnungsgemäße Konvertierung müssen auch die Anzahl der Frames, die Schrittgröße und die gewünschte Ausgabeauflösung eingegeben werden.
- Importieren Sie nach dem erneuten Sampling der Matrix in der Ebene die neue MAT-Datei in den 3D-Stammanalysecode.
4. 3D-Dehnungscodeanalyse
- Beginnen Sie mit der Analyse, indem Sie die importierte MAT-Datei ordnungsgemäß anpassen. Beispielsweise muss das Bildvolumen möglicherweise neu skaliert werden, um die Berechnungszeit zu reduzieren.
- Geben Sie den zu analysierenden Bereich ein, und bestimmen Sie die entsprechende Netzvorlage, um die Bilddaten entweder als einfache Felder oder manuell ausgewählte Polygone zu segmentieren. Die Boxgröße der Bereiche und der Abstand zwischen den Mittelpunkten müssen möglicherweise für jeden Datensatz geändert werden. Die optimalen Zahlen, die für die Boxgröße ausgewählt werden, entsprechen der Reihenfolge der Pixel des nachverfolgten Features, die durch Betrachten der Anzahl der Pixel in zwei Dimensionen in einem Segment angenähert werden können. Der Abstand der Felder bestimmt die Auflösung der Dehnungsfelder. Mehr Boxen erhöhen die Auflösung, können aber auch die Rechenzeit erheblich erhöhen.
- Beginnen Sie, die Jakobianer und Gradienten iterativ innerhalb jeder dieser Regionen zu berechnen. Wenden Sie nach Abschluss der Vorberechnung die Verwarbfunktion an.
- Berechnen Sie den Verformungsgradiententensor. Berechnen Sie zuerst die Dehnung und dann die Eigenwerte und Eigenvektoren mit der Methode zur Schätzung der direkten Verformung.
- Zeichnen Sie diese Ergebnisse in den gewünschten Ebenen mit einer Technik wie der Farbzuordnung einer Schnittebene, um das Dehnungsfeld über Ihrem Interessenbereich darzustellen.
Dreidimensionale Dehnungsbildgebung wird verwendet, um die Verformung von Weichteilen im Laufe der Zeit zu schätzen und Krankheiten zu verstehen. Das mechanische Verhalten von Weichteilen wie Haut, Blutgefäßen, Sehnen und anderen Organen wird stark durch ihre extrazelluläre Zusammensetzung beeinflusst, die sich durch Alterung und Krankheit verändern kann. Innerhalb komplexer biologischer Gewebe ist es wichtig, diese Veränderungen zu charakterisieren, die die mechanischen und funktionellen Eigenschaften eines Organs erheblich beeinflussen können.
Quantitative Dehnungszuordnung verwendet volumetrische Bilddaten und eine methode der direkten Verformung, um die räumlich variierenden dreidimensionalen Dehnungsfelder zu berechnen. In diesem Video werden die Prinzipien der Dehnungszuordnung veranschaulicht, gezeigt, wie quantitative Dehnungszuordnung zur Schätzung von Stammfeldern innerhalb komplexer biologischer Gewebe verwendet wird, und andere Anwendungen diskutieren.
Biologische Gewebe werden stark durch die Zusammensetzung und Ausrichtung von Elastin und Kollagen beeinflusst. Das Protein Elastin ist ein hochelastischer Bestandteil von Geweben, die sich ständig dehnen und zusammenziehen, wie Blutgefäße und die Lunge. Kollagen ist das am häufigsten vorkommende Protein im Körper und wird aus einzelnen dreifach-helischen Polymeren zusammengesetzt, die in größeren Fasern gebündelt werden, die Geweben von der Haut bis zu den Knochen strukturelle Integrität verleihen.
Die Ausrichtung dieser Proteine reicht von ausgerichteten Fasern bis hin zu faserigen Netznetzen, was sich auf die mechanischen Eigenschaften des Gewebes auswirkt. Dehnung ist ein Maß für die relative Verformung von Weichteilen im Laufe der Zeit und kann verwendet werden, um Verletzungen und Krankheiten zu visualisieren. Es wird beschrieben und anhand mathematischer Schätzungen abgebildet.
Um Dehnungen in komplexen Organen wie dem Herzen abzubilden, können vierdimensionale Ultraschalldaten verwendet werden, die hohe Auflösungs-, räumliche und zeitliche Informationen liefern. Anschließend wird die Methode zur Direkten Verformungsschätzung (DDE) auf die Daten angewendet. Ein Code wird verwendet, um die 3D-Verformung und die entsprechenden Green-Lagrange-Stämme mit der folgenden Gleichung zu schätzen.
Der Green-Lagrange-Dehnungssor hängt vom Verformungsgradiententensor und dem Identitätstensor zweiter Ordnung ab. Verformunggradiententensoren werden traditionell aus Verschiebungsfeldern geschätzt. Bei der DDE-Methode wird eine Verzugsfunktion so optimiert, dass sie direkt analog zum Verformungstensor ist. Die Verwarrfunktion hängt sowohl von der räumlichen Position als auch vom Warping-Parameter ab. Die Berechnung der Verformung wird direkt in die Verformungsfunktion integriert. Die ersten neun Elemente stellen den Verformungsgradiententensor dar.
Diese Methode wird verwendet, um sowohl große als auch lokalisierte Verformungen in Weichgeweben zu schätzen. Nun, da wir die Prinzipien der Dehnungszuordnung verstehen, sehen wir nun, wie die Dehnungszuordnung durchgeführt wird, um Aortenaneurysmen bei Mäusen zu erkennen.
Um mit der Einrichtung zu beginnen, öffnen Sie die Vivo 2100 Software und schließen Sie den Laptop an das Ultraschallsystem an. Stellen Sie sicher, dass die physiologische Überwachungseinheit eingeschaltet ist, um Herzfrequenz und Temperatur zu messen. Dann initialisieren Sie die 3D-Motorstufe.
Installieren Sie den Ultraschallwandler und stellen Sie sicher, dass alle richtigen Anschlüsse hergestellt werden. Als nächstes befeuchten Sie das Tier, das mit 3% Isofluran in einer Klopfkammer abgebildet wird. Sobald die Maus betäut ist, bewegen Sie sie auf die erhitzte Stufe und sichern Sie einen Nasenkegel, um 1-2% Isofluran zu liefern. Tragen Sie die ophthalmologische Salbe auf die Augen auf und sichern Sie die Pfoten an den Bühnenelektroden, um die Atmung und Herzfrequenz des Tieres zu überwachen. Setzen Sie dann eine rektale Temperatursonde ein. Tragen Sie Enthaarungscreme auf, um das Haar aus dem Interessenbereich zu entfernen, und wenden Sie dann eine großzügige Menge warmes Ultraschallgel auf den enthaarten Bereich an.
Um die Bildaufnahme zu starten, öffnen Sie zunächst das Bildgebungsfenster und wählen Sie den B-Modus aus. Senken Sie dann den Messumformer auf das Tier und verwenden Sie die x- und y-Achsenknöpfe auf der Bühne, um den Interessenbereich zu lokalisieren. Überwachen Sie die Atemfrequenz, um sicherzustellen, dass sie nicht wesentlich abnimmt. Positionieren Sie den Messumformer in der Mitte der Interessenregion. Dann nähern Sie sich der Entfernung erforderlich, um die gesamte Region von Interesse zu decken.
Geben Sie diese Dimensionen in den MATLAB-Code ein und wählen Sie eine Schrittgröße von 0,08 Millimetern aus. Stellen Sie sicher, dass herz- und atemsicher das Herz und die Atemfrequenz des Tieres stabil sind, und führen Sie dann den MATLAB-Code aus.
Exportieren Sie die Daten nach der Bildaufnahme als XML-Rohdateien und konvertieren Sie sie in MAT-Dateien. Stellen Sie sicher, dass Sie die Anzahl der Frames, die Schrittgröße und die Ausgabeauflösung eingeben. Anschließend wird die Matrix in durchgangsebene erneut abgetastet.
Importieren Sie die neue MAT-Datei in den 3D-Stammanalysecode. Es kann erforderlich sein, die Datei neu zu skalieren, um die Berechnungszeit zu reduzieren. Geben Sie dann den zu analysierenden Bereich ein. Approximate die Anzahl der Pixel in einem zweidimensionalen Segment des verfolgten Features, und wählen Sie die Netzvorlage entweder als einfaches Feld oder manuell ausgewählte Polygone aus. Wählen Sie die optimale Pixelzahl für die Netzgröße aus. Berechnen Sie die Jakobianer und die Gradienten. Wiederholen Sie diesen Vorgang für jede Region. Wenden Sie dann die Verwarrfunktion an.
Als Nächstes bestimmen Sie mithilfe von kartesischen Verformungen, die aus DDE berechnet werden, die Eigenwerte und Eigenvektoren der Verformung. Wählen Sie dann die Slices aus, für die Sie Dehnungswerte darstellen möchten, indem Sie durch die Ansichten der langen Achse, der Sortierachse und der koronalen Achsen blättern.
Drücken Sie Verteiler für die Analyse auswählen. Verwenden Sie dann den Cursor, um Markierungen entlang der Aortenwand zu platzieren, einschließlich des Thrombus, Aneurysmus und gesunder Teile der Aorta. Wiederholen Sie dies für alle Ansichten. Verwenden Sie schließlich die Farbzuordnung, um die Ergebnisse des Stammfelds über den Interessenbereich zu zeichnen.
Werfen wir einen genauen Blick auf das Beispiel eines angiotensin II-induzierten suprarenal enzierenden Abdominalaortenaneurysms, das von einer Maus erworben wurde. Zunächst werden mehrere Kurzachs-EKG-Gated-Kilohertz-Visualisierungsschleifen in einer bestimmten Schrittgröße entlang der Aorta erhalten und zu 4D-Daten kombiniert.
Nach der 3D-Dehnungsberechnung mit einer optimierten Verzugsfunktion wird das 3D-Slice-Visualisierungsdiagramm der Infrarot-Aorta erhalten. Die Farbkarte der wichtigsten grünen Dehnung wird überlagert, um Regionen heterogener Aortenwanddehnung enmittiert zu markieren. Darüber hinaus zeigen lange Achsen- und Kurzachsenansichten heterogene räumliche Dehnungsunterschiede, insbesondere wenn ein Thrombus vorhanden ist.
Entsprechende Dehnungsdiagramme zeigen höhere Dehnungswerte in gesunden Regionen der Aorta in der langen Achse, während die aneurysmale Region eine verringerte Belastung in der kurzen Achse zeigt.
Die genaue quantitative Dehnungsvisualisierung mittels direkter Verformungsschätzung ist ein nützliches Werkzeug, das in verschiedenen biomedizinischen Anwendungen eingesetzt wird.
Zum Beispiel kann der Herzstamm quantifiziert werden. Während des Herzzyklus erfährt das Myokard eine 3D-Deformation. Die Quantifizierung der Dehnung in drei Dimensionen ist integraler Bestandteil der zuverlässigen Charakterisierung der Dynamik dieses Gewebes im Laufe der Zeit. Dies ist nützlich bei der Verfolgung der Krankheitsprogression in Tiermodellen.
Eine weitere Anwendung ist in der Charakterisierung von Darmgewebe. In vivo Bildgebung des Darms ist aufgrund der Auswirkungen von umgebenden Strukturen eine Herausforderung. Die Berechnung der Belastung aus Bildern der Darmfibrose könnte jedoch besonders nützlich sein, um problematische Bereiche, die einen chirurgischen Eingriff erfordern, frühzeitig zu erkennen.
In einem viel kleineren Maßstab wird diese DDE-Methode auch auf zelluläre Ebene angewendet, indem bildgebende Techniken mit höherer Auflösung wie konfokale Mikroskopie verwendet werden. Es dient zum Beispiel bei der Charakterisierung der extrazellulären Matrix, um zu verstehen, wie Zellen unter mechanischen Veränderungen kommunizieren.
Sie haben gerade JoVeTs Einführung in die quantitative Dehnungsvisualisierung beobachtet. Sie sollten nun verstehen, wie sie dreidimensionale Stämme in biologischen Geweben messen und wie diese bei der Früherkennung von Krankheiten eingesetzt werden. Danke fürs Zuschauen!
Subscription Required. Please recommend JoVE to your librarian.
Results
Mit dem oben beschriebenen Verfahren wurde 4D-Ultraschall eines Angiotensin-II-induzierten suprarenalen sezierenden Abdominalaortenaneurysms (AAA) einer Maus erfasst. Entlang der Aorta wurden mehrere Kurzachs-EKV-Videoschleifen erfasst und zu 4D-Daten kombiniert, wie in Abbildung 1dargestellt. Diese Daten wurden dann mit einem benutzerdefinierten Code in eine MAT-Datei konvertiert, die dann in einem 3D-Stammberechnungscode mit einer Verzugsfunktion analysiert wurde. Nach der Optimierung der Parameter des Codes für einen bestimmten Datensatz wurde eine repräsentative Langachsansicht mit entsprechenden Dehnungswerten sowie ein 3D-Slice-Visualisierungsdiagramm mit einer überlagerten Dehnungsfarbkarte erstellt (Abbildung 2). Diese DDE-Technik und Dehnungsdaten unterstreichen die heterogenen räumlichen Variationen der Dehnung, insbesondere wenn ein Thrombus vorhanden ist. Diese Ergebnisse können dann mit der Gefäßstruktur korreliert werden, um die Beziehung zwischen In-vivo-Deformation und Aneurysmzusammensetzung zu bestimmen.

Abbildung 1: EKG-Gated Kilohertz Visualisierung (EKV) Schleifen der Aorta werden von manuell eingegebenen Start- und Endpositionen nach einer Schrittgröße von 0,2 mm erfasst.

Abbildung 2: 4D-Hochfrequenz-Ultraschalldaten eines murin enzierenden abdominalen Aortenaneurysms, das bei Systole (A) dargestellt wird, wobei die Hauptdehnungsfelder geschätzt und überlagert (B) (Scalebar = 5 mm) sind. Lang- und Kurzachsenansichten, die sowohl aneurysmale als auch gesunde Regionen darstellen, entsprechen dem Hauptstamm über einen Herzzyklus (Systole: t= 0,4) (C, D). Diese Daten zeigen relativ hohe Dehnungsgrade in gesunden Regionen und reduzierte Dehnungswerte innerhalb des sezierenden Aneurysmus.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
Die lokalisierte mechanische In-vivo-Charakterisierung ist ein wichtiger Bestandteil des Verständnisses des Wachstums und der Umgestaltung biologischer Gewebe. Im Vergleich zu bestehenden Ansätzen verwendet das hier beschriebene Dehnungsquantifizierungsverfahren eine verbesserte Methode zur genauen Berechnung der 3D-Dehnung durch optimale Verzug des unverformten Bildes vor der Kreuzkorrelation. Bei dieser Methode werden keine wesentlichen Annahmen zur Bestimmung von Stämmen innerhalb von Gewebevolumina verwendet. Leider ist die Dehnungsschätzung nur bis zu einer Kernelgröße von 15x15x15 Voxeln zuverlässig, wenn Ultraschalldaten verwendet werden, was darauf hindeutet, dass dieser DDE-Ansatz möglicherweise keine subtilen Merkmale innerhalb eines Dehnungsfeldes erkennt. Trotz dieser Einschränkung bleibt es ein wichtiges Werkzeug für die Untersuchung mechanischer Reaktionen, die Diagnose von Pathologie und die Verbesserung von Krankheitsmodellen.
Viele Forschungsbereiche jenseits von Aortenaneurysmen können von diesem Dehnungsmesswerkzeug profitieren. Mit dieser Methode kann auch der Herzstamm leicht quantifiziert werden. Da das Myokard während des Herzzyklus einer 3D-Deformation unterzogen wird, ist die Drei-Dimensionen-Quantifizierung der Belastung integraler Bestandteil der zuverlässigen Charakterisierung der Dynamik dieses Gewebes. Zuverlässige Dehnungsdaten sind besonders wichtig, wenn sie das Krankheitsverlauf in Tiermodellen verfolgen.
3D-Dehnungsanalyse kann auch auf Darm-Ultraschall-Bildgebung angewendet werden. Mechanische Charakterisierung von Darmgewebe wird am häufigsten in vitrodurchgeführt. Dies ist jedoch nicht immer eine echte Darstellung des tatsächlichen Verhaltens des Darms in vivo aufgrund von Auswirkungen von umgebenden Strukturen. Als Beispiel für die klinisch übersetzung dieses Ansatzes könnte die Berechnung des Stammes aus Bildern der Darmfibrose aufgrund eines abnormalen Luminaldrucks eine Früherkennung problematischer Bereiche ermöglichen, die einen chirurgischen Eingriff erfordern.
Abgesehen von den größeren Anwendungen kann diese Methode auch auf die zelluläre Ebene angewendet werden, indem bildgebende Techniken mit höherer Auflösung verwendet werden, z. B. die konfokale Mikroskopie. Die Charakterisierung der extrazellulären Matrix ist wichtig, um zu verstehen, wie Zellen kommunizieren. Es wurde viel über die biochemische Charakterisierung geforscht, aber das Verständnis, wie Kommunikation durch mechanische Reaktionen durchgeführt werden kann, erfordert ein Verständnis von Verformung und Dehnung. Massenstamm ist nicht vorteilhaft, weil es keine Möglichkeit gibt, den Ursprung der Verformungsänderung zu bestimmen. Die Anwendung eines hochauflösenden DDE-Ansatzes könnte direkt zeigen, wie die extrazelluläre Matrix auf mechanische Veränderungen reagiert.
Bestätigungen
Wir möchten John Boyle, Guy Genin und Stavros Thomopoulos für den Beitrag des Benutzercodes Von DDE Matlab danken, der in der Lage ist, die Lagrange-Green-Sorte direkt abzuschätzen.
Subscription Required. Please recommend JoVE to your librarian.




