Overview
Fonte: Tonya J. Webb1
1 Dipartimento di Microbiologia e Immunologia, University of Maryland School of Medicine e Marlene and Stewart Greenebaum Comprehensive Cancer Center, Baltimora, Maryland 21201
ELISPOT è un test standardizzato e riproducibile utilizzato per rilevare le risposte immunitarie cellulari. Il test utilizza un metodo basato su un saggio immunoassorbente enzimatico (ELISA) per rilevare le risposte immunitarie a singola cellula che possono essere visualizzate da macchie, da cui il nome ELISPOT. ELISPOT è stato descritto per la prima volta nel 1983, da Czerkinsky, come un metodo per enumerare il numero di ibridomi a cellule B che producono immunoglobuline antigene-specifiche (1). Lo stesso gruppo ha ulteriormente sviluppato il test per misurare la frequenza dei linfociti T che producono citochine. Ora ELISPOT è diventato un gold standard per misurare l'immunità delle cellule T antigene-specifiche negli studi clinici e nei candidati vaccini. Ad esempio, dopo la vaccinazione o durante un'infezione, le plasmacellule e le cellule B della memoria secernono anticorpi che forniscono protezione. Tipicamente, queste risposte delle cellule B sono valutate misurando i titoli sierici di anticorpi antigene-specifici. Tuttavia, questo tipo di analisi, tipicamente misurata da ELISA, potrebbe non includere le cellule B di memoria, che possono essere presenti anche in assenza di livelli sierici di anticorpi rilevabili. Inoltre, è stato ben stabilito che le cellule B della memoria circolanti sono importanti per la risposta anticorpale rapida e protettiva osservata dopo la ricon esposizione dei patogeni, quindi è fondamentale essere in grado di rilevare queste cellule. Pertanto, per valutare chiaramente le risposte delle cellule B della memoria antigene-specifica, devono essere utilizzati sia ELISA che ELISPOT (2).
Il test ELISPOT utilizza una piastra contenente pozzetti rivestiti di membrana rivestiti di anticorpi al fine di catturare le proteine secrete di interesse. Quindi, la piastra viene caricata con cellule e stimoli per indurre la produzione di proteine. Le proteine secrete vengono catturate dagli anticorpi rivestiti sulla superficie. Dopo un adeguato tempo di incubazione, le cellule vengono rimosse e la molecola secreta viene rilevata utilizzando un anticorpo biotinilato specifico per un epitopo diverso, rispetto all'anticorpo di cattura. Successivamente, viene aggiunta la streptavicina perossidasi, seguita dall'aggiunta di un substrato che consente il rilevamento delle macchie (Figura 1). Il punto di forza di questo test è che consente di quantificare il numero di cellule che producono la proteina di interesse. È importante sottolineare che si può valutare se ci sono cambiamenti nel numero totale di cellule che producono una proteina specifica o se le singole cellule all'interno di una popolazione producono più proteine. Inoltre, può fornire informazioni sulla cinetica e può essere utilizzato per valutare l'attivazione immunitaria complessiva (stimolazione mitogena) rispetto alle risposte antigene-specifiche (simulazione dell'antigene). Il test ELISPOT consentirà di rilevare una cellula attivata tra 300.000 cellule a seguito di attivazione mitogenica o antigene-specifica.
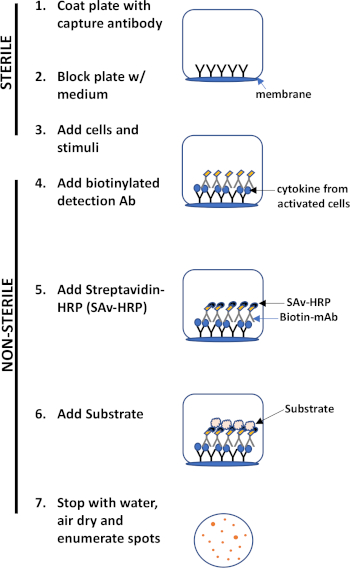
Figura 1: Panoramica del protocollo ELISPOT.
I principali vantaggi di questo test sono il suo- a. Semplicità: il protocollo è relativamente semplice e diretto. Non richiede competenze tecniche, b. Sensibilità- permette il rilevamento di cellule immunitarie a livello di singola cellula e richiede pochissime cellule rispetto ad altri metodi come la citometria a flusso, c. Funzionalità: fornisce dati quantitativi relativi alla funzione immunitaria.
Questo esercizio di laboratorio dimostra il protocollo ELISPOT per il rilevamento di splenociti secernenti IFN-γ, ma come accennato in precedenza questo test può anche essere utilizzato per valutare la secrezione di anticorpi da parte delle cellule B (3).
Procedure
1. Configurazione
Tamponi e reagenti
- Soluzione salina sterile tamponata con fosfato (PBS) senza calcio o magnesio
- Tampone di rivestimento: PBS sterile o tampone carbonaato
- Diluente di dosaggio- 10% siero bovino fetale (FBS) in PBS
- Terreno di coltura cellulare - RPMI 1640 con 10% FBS, penicillina / streptomicina e L-glutammina
- Tampone di lavaggio - PBS contenente 0,05% Tween20
- Acqua doppia distillata (ddH2O)
- Substrato di rilevamento- 100 mg AEC (3-ammino-9-etil-carbazolo) in 10 mL DMF (N,N, Dimetilformammide).
Attrezzatura
- Cappa a flusso laminare
- Incubatore umidificato (impostato a 37°C, 5% CO2)
- Lettore ELISPOT automatizzato o microscopio sezionante
Materiali
- Piastre ELISPOT
- Serbatoi sterili e non sterili
- Pipettors e punte
- Pipette sierologiche sterili
- Tubi sterili conici in polipropilene
- Due bottiglie di spremi per il lavaggio delle piastre
Reagenti specifici per il saggio
- Cellule- cellule primarie o linee cellulari (qui sono stati utilizzati splenociti di topi C57BL/6)
- Stimolanti: mitogeno o antigene (qui, phorbol 12-myristate 13-acetato (PMA, 50 ng / mL) e ionomicina (1 μM) sono stati usati)
- Anticorpo primario - anticorpo anti-citochina biotinilato (diluito a 2 μg/mL nel diluente di saggio)
- Anticorpo secondario- streptavidina-rafano perossidasi (SAv-HRP)
2. Procedura
Rivestimento
- Mantenendo le condizioni sterili e all'interno di una cappa a flusso laminare, diluire l'anticorpo di cattura anti-citochine purificato fino a una concentrazione finale di 0,5-4,0 μg/mL in tampone di rivestimento sterile. (Nota: per IFN- γ e IL-6 utilizzare 5 μg/mL).
- Trasferire la soluzione anticorpale di cattura, 100 μL/bene, nella piastra ELISPOT.
- Coprire la piastra con un coperchio della piastra e sigillarla per evitare l'evaporazione.
- Incubare la piastra durante la notte a 4°C.
Inceppamento
- Il giorno dopo, scopri la piastra ELISPOT nella cappa a flusso laminare. Invertire rapidamente la piastra su salviette sterili per rimuovere la soluzione anticorpale di cattura da ciascun pozzo.
- Quindi, aggiungere 200 μL di terreno di coltura cellulare a ciascun pozzo. Questo passaggio bloccherà il legame non specifico durante il test.
- Sostituire il coperchio della piastra e incubare la piastra per 2 ore a 37°C.
Placcatura e attivazione delle cellule
- Mentre la piastra è in incubazione, preparare una soluzione mitogena 2X contenente 50 ng/mL PMA e 1 μM di ionomicina in terreno di coltura cellulare.
- Quindi, preparare le sospensioni cellulari target a una concentrazione di stock di 2 x 106 cellule / mL.
- Al termine dell'incubazione, rimuovere il terreno di coltura cellulare da ciascun pozzo invertendo rapidamente la piastra su salviette sterili all'interno della cappa a flusso laminare.
- Quindi, generare una diluizione seriale 2X della soluzione di sospensione della cella di riserva. Per fare ciò, aggiungere prima 200 μL della soluzione stock di sospensione cellulare preparata nei pozzi nella fila superiore della piastra ELISPOT.
- Quindi, aggiungere 100 μL di terreno di coltura cellulare semplice alle successive cinque righe della piastra sotto le file contenenti soluzione stock cellulare.
- Successivamente, eseguire una diluizione seriale 2X pipettando 100 μL della sospensione cellulare dalla fila superiore nella riga direttamente sottostante. Garantire una corretta miscelazione pipettando delicatamente questa soluzione su e giù per garantire una distribuzione uniforme delle cellule.
- Ripetere questa procedura per le restanti quattro righe.
- Lascia la sesta riga solo con il mezzo culturale. Servirà come controllo sperimentale.
- Quindi, aggiungere 100 μL della soluzione mitogena preparata ai pozzi sperimentali delle prime cinque file della piastra. Nei pozzi di controllo e nella sesta fila, aggiungere 100 μL del mezzo di coltura cellulare senza mitogeno.
- Sostituire il coperchio della piastra e incubare la piastra a 37° C, 5% CO2 in un incubatore per 20-48 ore. (Nota: 20-24 ore sono in genere sufficienti per rilevare IL-2 e TNF-α, mentre 48 ore sono ottimali per IL-4 e IFN-γ).
Scoperta
Anticorpo primario
- Preparare l'anticorpo anti-citochine biotinilato per rilevare una concentrazione di 2 μg/mL nel diluente di saggio.
- Preparare 20-25 ml di tampone di lavaggio in questo momento mescolando lo 0,05% di Tween-20 in PBS.
- Al termine dell'incubazione, scapitolare la piastra e invertirla rapidamente su un lavandino per rimuovere tutto il liquido dai pozzetti. (Nota: dopo questo punto la piastra non deve più essere mantenuta sterile).
- Quindi, lavare la piastra aggiungendo ~ 200 μL di tampone di lavaggio a ciascun pozzo. Espellere questo liquido invertendo rapidamente e facendo scorrere la piastra su un lavandino. Ripeti questo processo per un totale di cinque lavaggi.
- Quindi, aggiungere 100 μL della soluzione anticorpale anti-citochina biotinilata diluita a ciascun pozzo. Incubare a temperatura ambiente per 2 ore a temperatura ambiente o durante la notte a 4°C.
Anticorpo secondario
- Al termine dell'incubazione, espellere l'anticorpo di rilevamento invertendo e facendo scorrere la piastra sul lavandino.
- Come prima, lavare la piastra 5 volte con un tampone di lavaggio da ~ 200 μL, espellendo il liquido tra ogni lavaggio.
- Aggiungere quindi 100 μL di soluzione diluita di streptavitina-rafano perossidasi a ciascun pozzo (diluito alla sua concentrazione ottimale predeterminata nel diluente di saggio).
- Sostituire il coperchio della piastra e incubare a temperatura ambiente per 1,5-2 ore a 37°C.
Substrato
- Dopo l'incubazione, non più di 15 minuti prima dell'uso, attivare prima la soluzione di substrato AEC secondo le istruzioni del produttore.
- Quindi, scartare il contenuto dei pozzi e lavare la piastra cinque volte con tampone di lavaggio, come prima.
- Quindi, aggiungere immediatamente 100 μL di soluzione di substrato AEC preparata in ciascun pozzet.
- Incubare la piastra a temperatura ambiente per ~ 10-20 minuti mentre si monitora lo sviluppo dello spot.
- Interrompere la reazione risciacquando la piastra con acqua e facendo scorrere la piastra sul lavandino.
- Tamponare il piatto su carta assorbente e lasciare asciugare il piatto all'aria durante la notte o fino a quando non è completamente asciutto. La rimozione del vassoio di plastica sotto la piastra faciliterà l'asciugatura.
3. Acquisizione e analisi dei dati
- Dopo l'essiccazione, i punti sono pronti per essere contati con un lettore automatico di lastre. Qui viene utilizzato il lettore CTL Immunospot, ma questo protocollo può essere adattato per qualsiasi lettore.
- Prima accendi lo strumento, poi il computer. Quindi, apri il programma CTL e fai clic su "conteggio scansioni".
- Spingere "espellere" affinché il vassoio si estenda dalla macchina. Quindi, rimuovere l'adattatore in plastica e allineare la riga "A" sulla piastra e sull'adattatore ELISPOT.
- Scegliere un nome e una posizione per il file da salvare e caricare la piastra sul vassoio.
- Quindi, fare clic su "carica" sul software e chiudere la porta sul lato della macchina.
- Premi "start- after counting". Assicurarsi che il file venga salvato, quindi aprire il software di controllo qualità "QC" per analizzare i dati e contare il numero di punti.
Note:
- Il numero minimo di cellule dovrebbe essere determinato in esperimenti preliminari. Il numero ottimale di punti è ~ 50 / bene. Se vengono caricate troppe celle, sarà difficile rilevare punti distinti. Inoltre, le cellule si sovrapporranno e potrebbero non formare un monostrato sulla membrana, quindi il livello di rilevamento potrebbe essere ridotto.
- Quando si ottimizza l'esperimento, considerare il livello di espressione previsto della proteina bersaglio. Minore è l'espressione, maggiore è il numero di celle richieste per pozzo.
- A differenza di ELISA, è meglio lavare a mano il piatto piuttosto che usare una lavapiatti. Le piastre ELISPOT sono più delicate e si dovrebbe evitare di forare la membrana PVDF.
- Si dovrebbe limitare il movimento della piastra durante il periodo di incubazione in quanto può causare macchie alle macchie.
- Le lastre devono essere conservate al buio, poiché l'esposizione alla luce diretta provoca lo sbiadimento delle macchie.
Il test Enzyme-linked Immunospot, o ELISPOT, è un metodo per analizzare la risposta immunitaria a un agente patogeno o a un danno cellulare. Permette di quantificazione dell'attivazione di diverse cellule immunitarie rilevando proteine specifiche che secernono. Ad esempio, ELISPOT è comunemente usato per misurare le risposte delle cellule T in caso di esposizione a un antigene estraneo rilevando citochine secrete.
Per un test ELISPOT basato su citochine, il processo inizia con il rivestimento di una micropiastra ELISPOT con un anticorpo di cattura, che è specifico per la citochina bersaglio. Dopo il rivestimento anticorpale, le cellule T vengono aggiunte ai pozzi e stimolate da un agente esterno, come ad esempio l'anticorpo anti-CD3. Le cellule quindi secernono la citochina bersaglio, che viene immediatamente immobilizzata dall'anticorpo di cattura. Poiché la proteina viene catturata istantaneamente dopo la secrezione da cellule vive, senza diluizione o degradazione, questo test ha un'elevata precisione. Dopo che la citochina bersaglio è stata immobilizzata, viene aggiunto un anticorpo di rilevamento, che si lega anche alla citochina catturata.
La tecnica ELISPOT può anche essere utilizzata per quantificare le cellule B della memoria dopo un'infezione o una vaccinazione analizzando la loro produzione di anticorpi specifici. In un ELISPOT a base di anticorpi, viene utilizzato un antigene specifico al posto di un anticorpo per la fase di cattura, in cui l'antigene sarà legato alla piastra, o nella fase di rilevamento, in cui l'antigene rileva l'anticorpo bersaglio post-cattura. In tutte le varianti del processo, per le cellule T o B, l'anticorpo o l'antigene di rilevamento è biotinilato, il che gli consente di legarsi a un enzima di rilevamento coniugato con streptavidina, come la perossidasi di rafano. Quindi, con l'aggiunta del substrato della perossidasi, AEC, viene prodotto un precipitato scuro e insolubile. Questo precipitato segna la posizione della proteina catturata e ogni cellula secretoria si traduce in un punto visibile, che può essere quantificato utilizzando un lettore ELISPOT o un microscopio. La dimensione delle macchie è una stima relativa della quantità di proteine secrete da ciascuna cellula. Questo test può rilevare le risposte immunitarie da singole cellule, anche in sottopopolazioni relativamente piccole di cellule secretorie, rendendolo utile per studiare le risposte immunitarie a livello cellulare.
In questo video, imparerai come eseguire un test ELISPOT e quindi quantificare le macchie che rappresentano le cellule secretorie.
Durante l'esperimento, garantire condizioni sterili lavorando in un cappuccio a flusso laminare e indossando guanti.
Tutti i calcoli in questo protocollo si basano sul volume necessario per una piastra da 96 pozzi.
In primo luogo, diluire l'anticorpo di cattura anti-citochine. Per fare questo, trasferire 10 millilitri di tampone in un tubo conico sterile da 15 millilitri. Quindi, utilizzare una pipetta per aggiungere 10 microlitri di un milligrammo per millilitro di anticorpo monoclonale al tampone per creare una soluzione con una concentrazione finale di un microgrammo per millilitro. Quindi, versare la soluzione anticorpale di cattura in un serbatoio sterile e, utilizzando una pipetta multicanale, distribuire 100 microlitri in ciascun pozzetti di una piastra ELISPOT da 96 pozzetti.
Coprire la piastra con un coperchio della piastra, sigillarla per evitare l'evaporazione e incubare durante la notte a quattro gradi Celsius. Il giorno dopo, scopri la piastra ELISPOT nella cappa a flusso laminare. Invertire rapidamente la piastra su salviette sterili per rimuovere la soluzione anticorpale di cattura da ciascun pozzo. Quindi, utilizzare una pipetta multicanale per aggiungere 200 microlitri di terreno di coltura cellulare a ciascun pozzo. Questo passaggio bloccherà il legame non specifico durante il test. Sostituire il coperchio della piastra e incubare in un incubatore a 37 gradi Celsius per due ore.
Mentre la piastra è in incubazione, preparare una soluzione mitogena 2X aggiungendo un microlitro di PMA e 20 microlitri di ionomicina a 10 millilitri di terreno di coltura cellulare per ottenere una concentrazione finale di 15 nanogrammi per millilitro PMA e una ionomicina micromolare.
Anche le sospensioni cellulari di splenociti di topo devono essere preparate in questo momento in un cappuccio sterile. Utilizzando un microscopio e un emocitometro, misurare la concentrazione di cellule e regolare il volume totale fino a raggiungere una concentrazione di riserva di due milioni di cellule per millilitro.
Al termine dell'incubazione, invertire rapidamente la piastra su salviette sterili per rimuovere il terreno di coltura cellulare da ciascun pozzo. Quindi, aggiungere 200 microlitri della soluzione di sospensione cellulare preparata ai pozzi nella fila superiore della piastra ELISPOT. Impostare l'esperimento in triplice copia, in modo che ogni tipo di cella testato venga placcato in un insieme di tre colonne raggruppate. Al di sotto di questo, aggiungere 100 microlitri di terreno di coltura cellulare semplice alle successive cinque file della piastra, sotto le file contenenti soluzione stock cellulare.
Quindi, eseguire una diluizione seriale pipettando 100 microlitri della sospensione cellulare dalla fila superiore nella fila direttamente sotto, pipettando delicatamente la soluzione su e giù per distribuire uniformemente le celle. Ripetere questo processo per le righe rimanenti, spostando 100 microlitri dalla riga precedente alla riga sottostante ad ogni passaggio, continuando fino a quando la quinta riga non è stata diluita in serie. Lasciare la sesta riga con il solo terreno di coltura cellulare, per fungere da controllo. Per stimolare le cellule nei pozzi sperimentali della piastra, aggiungere 100 microlitri della soluzione mitogena preparata alle sospensioni cellulari in ciascun pozzo di file da una a cinque. Assicurati di lasciare la sesta fila, che servirà come controllo, non stimolata. Sostituire il coperchio e incubare la piastra a 37 gradi Celsius e 5% di CO2 per 24-48 ore.
Preparare l'anticorpo anti-citochina biotinilato diluito. In primo luogo, preparare 50 millilitri di diluente di saggio aggiungendo 5 millilitri di siero bovino fetale al 10% a 45 millilitri di PBS. Quindi, diluire l'anticorpo rilevante a una concentrazione di 2 microgrammi per millilitro nel diluente di saggio. Inoltre, preparare da 20 a 25 millilitri di tampone di lavaggio in questo momento, mescolando lo 0,05% di Tween-20 e PBS.
Al termine dell'incubazione, scollebare la piastra e invertirla rapidamente per rimuovere tutto il liquido dai pozzi. Lavare la piastra aggiungendo circa 200 microlitri di tampone di lavaggio a ciascun pozzo. Espellere questo liquido invertendo rapidamente e facendo scorrere la piastra su un lavandino. Ripeti questo processo altre quattro volte per un totale di cinque lavaggi. Quindi, aggiungere 100 microlitri della soluzione anticorpale di rilevamento diluita a ciascun pozzo, sostituire il coperchio e incubare a temperatura ambiente per due ore. Dopo l'incubazione, espellere la soluzione anticorpale di rilevamento dai pozzi della piastra invertendo e facendo scorrere la piastra sul lavandino.
Come prima, lavare la piastra cinque volte con tampone di lavaggio, espellendo il liquido tra ogni lavaggio. Dopo il lavaggio finale, preparare la soluzione di streptavitina-rafano perossidasi diluendola secondo le istruzioni del produttore. Successivamente, con i pozzi della piastra vuoti, aggiungere 100 microlitri di soluzione diluita di streptavitina-rafano perossidasi a ciascun pozzo. Rimettere il coperchio sul piatto e incubare a temperatura ambiente per due ore.
Dopo l'incubazione, non più di 15 minuti prima dell'uso, attivare la soluzione di substrato AEC prefabbrica. Scartare il contenuto dei pozzi e lavare la piastra cinque volte con tampone di lavaggio, come prima. Quindi, aggiungere immediatamente 100 microlitri di soluzione di substrato AEC preparata in ciascun pozzet. Lasciare la piastra a temperatura ambiente per svilupparsi per circa 10-20 minuti, monitorando lo sviluppo spot. Queste macchie appariranno come piccoli cerchi oscurati sulla superficie dei pozzi. Quindi, interrompere la reazione risciacquando la piastra con acqua e facendola scorrere sul lavandino. Asciugare la piastra su carta assorbente e lasciare asciugare all'aria durante la notte o fino a completa asciugatura. La rimozione del vassoio di plastica sotto la piastra faciliterà l'asciugatura. Dopo l'essiccazione, i punti sono pronti per essere contati con un lettore automatico di lastre.
Qui viene utilizzato il lettore CTL ImmunoSpot, ma questo protocollo può essere adattato per qualsiasi lettore. Quindi, apri il programma CTL e fai clic su Scan Count. Spingere l'espulsione per far estendere il vassoio dalla macchina. Quindi, rimuovere l'adattatore di plastica e allineare la riga A sulla piastra ELISPOT e sull'adattatore. Scegliere un nome e una posizione per il file da salvare e caricare la piastra e l'adattatore sul vassoio. Fare clic su Carica sul software e chiudere lo sportello sul lato della macchina. Quindi, premere Avvia dopo il conteggio. Assicurarsi che il file venga salvato e quindi aprire il software QC di controllo qualità per analizzare i dati e contare il numero di punti. Esportare questi dati come file Excel. Una volta completata l'analisi, fate clic su Espelli (Eject) per recuperare la piastra.
In questo esperimento, le cellule di topi selvatici e portatori di tumore sono state placcate e analizzate per la gamma IFN. Si noti che il numero di macchie diminuisce con la diminuzione della concentrazione cellulare. In genere, i dati ELISPOT vengono presentati come il numero di conteggi spot per numero di celle placcate. In questo esempio, il numero di punti è stato visualizzato in un grafico a barre, con ogni rispettiva concentrazione cellulare elencata sull'asse x. Si noti che il numero di punti indica il numero di cellule attivate per numero totale di cellule in una determinata popolazione.
Subscription Required. Please recommend JoVE to your librarian.
Results
In questo test ELISPOT, i leucociti splenici di topi wildtype e portatori di tumore sono stati analizzati per la γ IFN. La Figura 2 A mostra l'immagine visiva del risultato del test. I numeri nel colore verde indicano il numero di macchie per pozzo (TNTC indica "troppo numerosi per contare"). Si noti che il numero di macchie diminuisce con la diminuzione della concentrazione cellulare.

Figura 2A: Diminuzione delle risposte immunitarie nei topi portatori di tumore. Fare clic qui per visualizzare una versione più grande di questa figura.
In genere, i dati ELISPOT vengono presentati come il numero di conteggi spot per numero di celle placcate. Nella Figura 2 B il numero di punti è visualizzato in un grafico a barre, con ogni rispettiva concentrazione cellulare elencata sull'asse X. Ai fini della grafica, 150 è stato utilizzato per indicare il numero massimo di punti. Il numero di leucociti splenici murini che producono γ IFN negli animali portatori di tumore è inferiore a quelli selvatici.

Figura 2B: Diminuzione delle risposte immunitarie nei topi portatori di tumore. Gli splenociti sono stati prelevati da topi di controllo C57BL/6 (wildtype) e portatori di tumore e stimolati con PMA/ionomicina per 48 ore. I saggi ELISPOT sono stati utilizzati per quantificare il numero di leucociti splenici produttori di IFN-γ. (A) Rappresentazione visiva e (B) grafica dei dati. TNTC indica troppo numerosi per contare. Ai fini della grafica, 150 è stato utilizzato per indicare il numero massimo di punti. I numeri verdi indicano il numero di punti contati per pozzo. I numeri rossi indicano i pozzi di riferimento utilizzati per determinare quali punti erano celle e quali punti erano detriti, artefatti o effetti di bordo e devono essere esclusi dall'analisi.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
Il test ELISPOT consente di valutare l'attivazione delle cellule immunitarie determinando il numero di cellule che secernono un analita specifico. La dimensione e l'intensità delle macchie forniscono informazioni sulla quantità di analita prodotta da ciascuna cellula. Il protocollo sopra descritto ha dettagliato il rilevamento di una singola citochina. Tuttavia, i recenti sviluppi hanno migliorato l'utilità di questo test. Attualmente, è possibile utilizzare coloranti fluorescenti per rilevare più analiti all'interno di un pozzo. Ciò consente l'individuazione di diverse sottopopolazioni di cellule che secernono uno o entrambi gli analiti.
Subscription Required. Please recommend JoVE to your librarian.
References
- Czerkinsky, C. C., Nilsson, L. A., Nygren, H., Ouchterlony, O., & Tarkowski, A. A solid-phase enzyme-linked immunospot (ELISPOT) assay for enumeration of specific antibody-secreting cells. Journal of Immunological Methods, 65 (1), 109-121(1983).
- Wahid, R., Simon, J. K., Picking, W. L., Kotloff, K. L., Levine, M. M., & Sztein, M. B. Shigella antigen-specific B memory cells are associated with decreased disease severity in subjects challenged with wild-type Shigella flexneri 2a. Clinical Immunology, 148 (1), 35-43 (2013).
- Roberts, T. J., Lin, Y., Spence, P. M., Van Kaer, L., & Brutkiewicz, R. R. CD1d1-dependent control of the magnitude of an acute antiviral immune response. The Journal of Immunology, 172, 3454-3461 (2004).




