Overview
מקור: אווה בוקובסקה-פאניבנד1, עדדה אנדרסון1, רולף לוד1
1 המחלקה למדעים קליניים לונד, החטיבה לרפואת זיהומים, המרכז הביו-רפואי, אוניברסיטת לונד, 221 00 לונד, שוודיה
כדור הארץ הוא בית גידול למיליוני מינים חיידקיים, שלכל אחד מהם מאפיינים ספציפיים. זיהוי של מינים חיידקיים נמצא בשימוש נרחב באקולוגיה מיקרוביאלית כדי לקבוע את המגוון הביולוגי של דגימות סביבתיות ומיקרוביולוגיה רפואית לאבחון חולים נגועים. חיידקים יכולים להיות מסווגים בשיטות מיקרוביולוגיות קונבנציונליות, כגון מיקרוסקופיה, צמיחה על מדיה ספציפית, בדיקות ביוכימיות וסרולוגיות, ובדיקות רגישות לאנטיביוטיקה. בעשורים האחרונים, שיטות מיקרוביולוגיה מולקולרית חוללו מהפכה בזיהוי חיידקים. שיטה פופולרית היא ריצוף גנים RNA ריבוזומלי (rRNA) 16S. שיטה זו היא לא רק מהירה ומדויקת יותר מאשר שיטות קונבנציונליות, אלא גם מאפשר זיהוי של זנים שקשה לגדול בתנאי מעבדה. יתר על כן, הבחנה של זנים ברמה המולקולרית מאפשרת אפליה בין חיידקים זהים פנוטיפיים (1-4).
16S rRNA מצטרף לתסביך של 19 חלבונים ליצירת תת-unit של ריבוזום חיידקי (5). הוא מקודד על ידי הגן 16S rRNA, אשר קיים ושמובטח מאוד בכל החיידקים בשל תפקידו החיוני בהרכבת ריבוזום; עם זאת, הוא מכיל גם אזורים משתנים אשר עשוי לשמש טביעות אצבעות עבור מינים מסוימים. תכונות אלה הפכו את גן 16S rRNA לשבר גנטי אידיאלי לשימוש בזיהוי, השוואה וסיווג פילוגנטי של חיידקים (6).
ריצוף גנים rRNA 16S מבוסס על תגובת שרשרת פולימראז (PCR) (7-8) ואחריו ריצוף DNA (9). PCR היא שיטת ביולוגיה מולקולרית המשמשת להגברת שברי DNA ספציפיים באמצעות סדרה של מחזורים הכוללים:
i) דנטורציה של תבנית DNA כפולה תקועה
ii) חישול פריימרים (אוליגונוקלאוטידים קצרים) המשלימים לתבנית
iii) הרחבת פריימרים על ידי אנזים פולימראז DNA, אשר סינתזה גדיל DNA חדש
מבט כולל סכמטי על פעולת השירות מוצג באיור 1.
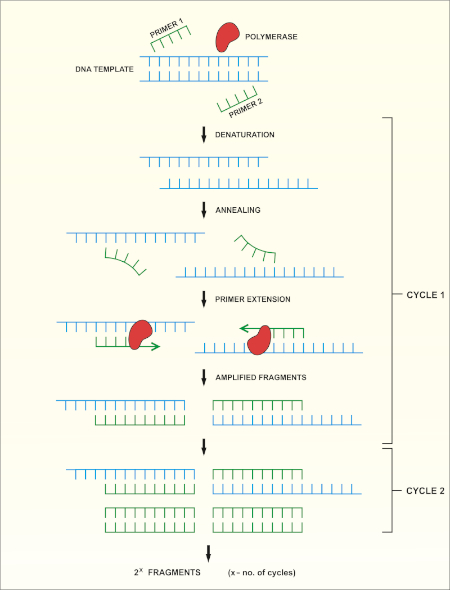
איור 1: מבט כולל סכמטי על תגובת ה- PCR. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
ישנם מספר גורמים החשובים לתגובת PCR מוצלחת, שאחד מהם הוא איכות תבנית ה- DNA. בידוד של DNA כרומוזומלי מחיידקים יכול להתבצע באמצעות פרוטוקולים סטנדרטיים או ערכות מסחריות. יש לנקוט בזהירות מיוחדת כדי להשיג DNA כי הוא ללא מזהמים שיכולים לעכב את תגובת PCR.
אזורים שמורים בגן 16S rRNA מאפשרים תכנון של זוגות פריימר אוניברסליים (אחד קדימה ואחד הפוך) שיכולים להיקשר ולהגביר את אזור היעד בכל מין חיידקי. אזור היעד עשוי להשתנות בגודלו. בעוד שזוגות פריימר מסוימים יכולים להגביר את רוב הגן 16S rRNA, אחרים מגבירים רק חלקים ממנו. דוגמאות של פריימרים נפוצים מוצגות בטבלה 1 ואתרי האיגוד שלהם מתוארים באיור 2.
| שם פריימר | רצף (5'→3') | קדימה/הפוך | הפניה |
| 8F b) | AGAGTTTGATCCTGGCTCAG | קדימה | -1 |
| 27F | AGAGTTTGATCMTGGCTCAG | קדימה | -10 |
| 515F | GTGCCAGCMGCCGCGGTAA | קדימה | -11 |
| 911R | GCCCCCGTCAATTCMTTTGA | הפוך | -12 |
| 1391R | GACGGGCGGTGTGTRCA | הפוך | -11 |
| 1492R | GGTTACCTTGTTACGACTT | הפוך | -11 |
טבלה 1: דוגמאות של אוליגונוקלאוטידים סטנדרטיים המשמשים להגברה של 16S rRNA גנים a).
א) ניתן להעריך את האורכים הצפויים של מוצר ה- PCR שנוצר באמצעות שילובי פריימר שונים על-ידי חישוב המרחק בין אתרי האיגוד עבור הפוריימר קדימה לבין הפריימר ההפוך (ראה איור 2), למשל גודל מוצר ה- PCR באמצעות זוג פריימר 8F-1492R הוא ~ 1500 bp, ולזוג פריימר 27F-911R ~ 900 bp.
ב) הידוע גם בשם fD1

איור 2: דמות מייצגת של רצף rRNA 16S ואתרי איגוד הפריימר. אזורים שמורים נצבעים באפור ואזורים משתנים מלאים בקווים אלכסוניים. כדי לאפשר את הרזולוציה הגבוהה ביותר, פריימר 8F ו- 1492R (שם המבוסס על מיקום על רצף rRNA) משמשים להגברת הרצף כולו, ומאפשרים רצף של מספר אזורים משתנים של הגן. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
תנאי הרכיבה על אופניים עבור PCR (כלומר הטמפרטורה והזמן הנדרשים עבור ה- DNA להיות denatured, חישול עם פריימרים, מסונתז) תלויים בסוג של פולימראז המשמש ואת המאפיינים של פריימרים. מומלץ לעקוב אחר הנחיות היצרן עבור פולימראז מסוים.
לאחר השלמת תוכנית ה- PCR, המוצרים מנותחים על ידי אלקטרופורזה ג'ל אגרוז. PCR מוצלח מניב רצועה אחת בגודל הצפוי. יש לטהר את המוצר לפני הרצף כדי להסיר פריימרים שיורית, deoxyribonucleotides, פולימראז, חוצץ אשר היו נוכחים בתגובת PCR. שברי הדנ"א המטוהרים נשלחים בדרך כלל לרצף לשירותי רצף מסחריים; עם זאת, מוסדות מסוימים מבצעים ריצוף DNA במתקני הליבה שלהם.
רצף הדנ"א נוצר באופן אוטומטי מכרומטוגרמה DNA על ידי מחשב ויש לבדוק אותו בקפידה לאיכות, שכן לעתים יש צורך בעריכה ידנית. לאחר שלב זה, רצף הגנים מושווה לרצפים שהופקדו במסד הנתונים של 16S rRNA. אזורי הדמיון מזוהים, והרצפים הדומים ביותר מועברים.
Procedure
1. ערוך
- בעת טיפול במיקרואורגניזמים, יש לעקוב אחר פרקטיקה מיקרוביולוגית טובה. כל המיקרואורגניזמים, במיוחד דגימות לא ידועות, צריכים להיות מטופלים כאל פתוגנים פוטנציאליים. בצע טכניקה אספטית כדי למנוע זיהום הדגימות, החוקרים, או המעבדה. יש לשטוף ידיים לפני ואחרי הטיפול בחיידקים, להשתמש בכפפות וללבוש ביגוד מגן.
- בצע הערכת סיכונים עבור הפרוטוקול הניסיוני לבידוד DNA גנומי וטיהור מוצר PCR. ריאגנטים מסוימים עלולים להזיק!
- תרבות טהורה חיונית לרצף 16S rRNA. לפני שתמשיכו לבידוד הדנ"א הגנומי, ודאו שהחומר ההתחלתי טהור לחלוטין. זה יכול להיעשות על ידי ציפוי פסים כדי לבודד מושבות בודדות. אלה יכולים לגדול עוד יותר פסים על צלחות בנפרד, או מרק, במידת הצורך.
- ציוד מעבדה נדרש:
- רוכב אופניים תרמי עבור פי.סי.אר. הפונקציה של רוכב האופניים התרמי היא להעלות ולהוריד את הטמפרטורה על פי תוכנית מוגדרת. בעת יצירת התוכנית תתבקש להזין את ערכי הטמפרטורה והשעה עבור כל שלב PCR, כמו גם מספר מחזורים כולל.
- מערכת אלקטרופורזה ג'ל אגרוז. הוא משמש להפרדת שברי DNA בהתבסס על גודלם ומטעןם. בפרוטוקול זה, אלקטרופורזה ג'ל אגרוז ישמש כדי לדמיין את האיכות של DNA גנומי מבודד ומוצרי PCR.
2. פרוטוקול
הערה: הפרוטוקול המודגם חל על ריצוף גנים 16S rRNA מתרבית חיידקים טהורה. זה לא חל על מחקרים מטגנומיים.
-
פולחן חיידקים לבידוד של DNA גנומי (gDNA).
- הגדל את המיקרואורגניזם שלך במדיום מתאים. ניתן להשתמש הן במדיה נוזלית והן במדיה מוצקה בשלב זה. בחר תנאים שמניבים את הצמיחה הטובה ביותר. בעת תכנון הניסוי, יש לזכור כי חיידקים הגדלים באיטיות עשויים להזדקק למספר ימים כדי להגיע לשלב הצמיחה המאוחרת/ נייחת. בפרוטוקול זה, Bacillus subtilis 168 גדל מרק ליסוגניה (LB) לילה בחממה רועדת להגדיר ב 200 סל"ד, 37 °C.
-
בידוד של GDNA.
- אם חיידקים גדלו על מדיום מוצק, לגרד כמה תאים באמצעות לולאה סטרילית resuspend אותם 1 מ"ל של מים מזוקקים
- אם חיידקים גדלו במדיום נוזלי, השתמש כ 1.5 מ"ל של תרבות לילה.
- גלולה את התאים על ידי צנטריפוגה (דקה אחת, 12,000 - 16,000 × גרם), להסיר את supernatant, ולהשתמש בתאים לבידוד gDNA באמצעות ערכה מסחרית או פרוטוקולים סטנדרטיים [למשל CTAB הכולל הכנת DNA (13) או מיצוי פנול-כלורופורם (14)]. כאן, ערכה מסחרית שימשה כדי לבודד gDNA מ 1.5 מ"ל של B. subtilis 168 תרבות לילה, OD600 = 1.5.
הערה 1: עבור חיידקים גרם שליליים מסוימים ניתן להשמיט שלב זה ולהחליף על ידי שחרור פשוט של DNA מהתאים על ידי רותחים. כדור חיידקי resuspend במים מזוקקים ודגרה בבלוק חימום להגדיר על 100 °C (10 דקות).
הערה 2: קשה לשבש תאי חיידקים חיוביים גרם. לכן מומלץ לבחור שיטת בידוד gDNA או ערכה המוקדשת לבידוד מקבוצת חיידקים זו.
-
בדיקת איכות gDNA.
- בדוק את האיכות של gDNA מבודד על ידי אלקטרופורזה ג'ל אגרוז. ראשית, לערבב 5 μL של gDNA מבודד עם 1 μL של צבע הטעינה (6x), לטעון את המדגם על ג'ל אגרוז 0.8% המכיל ריאגנט מכתים DNA.
- לטעון תקן מסה מולקולרית ולהפעיל את האלקטרופורזה עד חזית הצבע מגיע לתחתית הג'ל.
- לאחר השלמת האלקטרופורזה, דמיינו את הג'ל על טרנסילומינטור מתאים (אור UV או כחול). gDNA מופיע כפס מולקולרי גבוה ועבה (מעל 10 kb). דוגמה לבדיקת האיכות של gDNA מוצגת באיור 3.
- אם ה- gDNA עובר את בקרת האיכות (כלומר,הרצועה המולקולרית הגבוהה קיימת ויש מעט מאוד מריחה של ה- gDNA), לדלל את ה- gDNA שלך באופן סדרתי על ידי תיוג ראשון 3 צינורות microcentrifuge כדלקמן: "10x", "100x" ו -"1000x".
- פיפטה 90 μL של מים מזוקקים סטריליים לתוך כל אחד 3 צינורות.
- קח 10 μL של פתרון gDNA ולהוסיף אותו לצינור מסומן "10x".
- הזרימו את כל הנפח(כלומר 100 μL) למעלה ולמטה ביסודיות כדי להבטיח שהפתרון מעורבב באופן אחיד. לאחר מכן, לקחת 10 μL של הפתרון מן הצינור הזה ולהעביר אותו לצינור מסומן "100x".
- לערבב כמתואר לפני ולחזור על אותו הליך על ידי העברת 10 μL של הפתרון מצינור "100x" לצינור "1000x". דילולים אלה ישמשו כתבנית בתגובת ה- PCR.

איור 3: אלקטרופורזה ג'ל אגרוז של gDNA מבודד Bacillus subtilis. נתיב 1: M - סמן מסה מולקולרית (מלמעלה למטה: 10000 bp, 8000 bp, 6000 bp, 5000 bp, 4000 bp, 3500 bp, 3000 bp, 2500 bp, 2000 bp, 1500 bp, 1000 bp). נתיב 2: gDNA - DNA גנומי מבודד מבצילוס סובטיליס. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
-
הגברה של גן 16S rRNA על ידי PCR.
הערה: פרוטוקול PCR להלן מותאם לפולימראז DNA מסוים וזוג פריימר 8F - 1492R (ראה טבלה 1). אופטימיזציה של הפרוטוקול נדרשת עבור כל זוג פולימראז ופריימר.- להפשיר את כל הריאגנטים על קרח.
- הכן את תערובת המאסטר של PCR כפי שמוצג בטבלה 2. מאז פולימראז DNA פעיל בטמפרטורת החדר, הגדרת התגובה חייבת להתבצע על קרח, כלומר צינורות PCR ואת רכיבי התגובה צריך להישמר על קרח כל הזמן. הכן תגובה אחת לכל דגימת gDNA ותגובה אחת לשליטה שלילית. שליטה שלילית היא תערובת PCR ללא תבנית gDNA ומשמשת כדי להבטיח כי הרכיבים האחרים של התגובה אינם מזוהמים.
הערה: במקרה של דגימות מרובות, תערובת מאסטר מוכנה בדרך כלל. תמהיל ראשי הוא פתרון המכיל את כל רכיבי התגובה למעט התבנית. זה עוזר להשמיט צנרת חוזרת, למנוע שגיאת pipetting, ומבטיח עקביות גבוהה בין הדגימות. כדי להכין תערובת ראשית, הכפל את עוצמת הקול של כל רכיב (למעט תבנית ה- DNA) במספר הדגימות שנבדקו. מערבבים את כל הרכיבים בצינור microcentrifuge וצינור את כל הנפח למעלה ולמטה מספר פעמים. - Aliquot 49 μL של תערובת מאסטר לתוך צינורות PCR בודדים.
- הוסף תבנית μL אחת לצינורות עם תערובת ראשית. לשליטה שלילית להוסיף 1 μL של מים סטריליים. כדי להבטיח כי הרכיבים מעורבים היטב, בעדינות pipet את התערובת למעלה ולמטה ~ 10 פעמים עם pipette להגדיר 30-50μL.
- הגדר את מחשב ה- PCR עם התוכנית המוצגת בטבלה 3.
- שים את הצינורות במכונת PCR ולהתחיל את התוכנית.
- לאחר השלמת התוכנית, לבחון את איכות המוצר PCR שלך על ידי אלקטרופורזה ג'ל אגרוז.
- תגובת PCR מוצלחת באמצעות צמד פריימר 8F-1492R מניבה רצועה אחת של כ- 1.5 kb (איור 4). אם קיימים רצועות אחרות (כלומר מוצרים לא מוגדרים), מיטוב תוכנית ה- PCR על-ידי התאמת טמפרטורת ה- annealing. אם קיימת רצועה אחת בגודל צפוי, המשך לשלב הבא. כאן, תגובת PCR עם תבנית gDNA מדוללת פי 100 הניבה את המוצר הטוב ביותר מכיוון שהיה לה רצועה חדה בגודל הצפוי וחסרה מוצרים לא יפים. לפיכך הוא נבחר להיות מטוהר ונשלח לרצף.
- לפני הרצף, יש לנקות את המוצר משיורי פריימרים, deoxyribonucleotides, פולימראז, חוצץ אשר היו נוכחים בתגובת PCR. ניתן לבודד את מוצרי ה- PCR באמצעות ערכת טיהור PCR מסחרית. תגובת ה- PCR נטענת בעמודה המכילה מטריצת איגוד DNA. מוצר ה- PCR מאוגד לעמודה, בעוד שרכיבים אחרים זורמים דרך העמודה. לאחר מכן, העמוד נשטף באמצעות חיץ כביסה, ולבסוף, הדנ"א נחוץ במאגר הבחירה. ודא שמאגר ההבעה בתוספת הערכה תואם לרצף.
- שלח את מוצר ה- PCR המטוהר לרצף דנ"א. פעל בהתאם להנחיות להגשת דגימות ריצוף במתקן הרצף שנבחר. לקבלת כיסוי הרצף הטוב ביותר, השתמש בפריימרים להגברת PCR (זהים לנעשה בהם שימוש בסעיף 2.4.1) כפריימרים רצפים. כאן, פריימרים 8F ו 1492R שימשו לרצף את מוצר PCR.
| רכיב | ריכוז סופי | עוצמת קול לכל תגובה | עוצמת קול לתגובות x (עירוב ראשי) |
| מאגר תגובה 5x | 1x | 10 μL | 10 μL × x |
| 10 mM dNTPs | 200 מיקרומטר | 1 μL | 1 μL × x |
| 10 מיקרומטר פריימר 8F | 0.5 מיקרומטר | 2.5 μL | 2.5 μL × x |
| 10 מיקרומטר פריימר 1492R | 0.5 מיקרומטר | 2.5 μL | 2.5 μL × x |
| פוציון פולימראז | יחידה אחת | 0.5 μL | 0.5 μL × x |
| DNA של תבנית * | - | 1 μL | - |
| ddH2O | - | 32.5 μL | 32.5 μL × x |
| סה"כ אמצעי אחסון | 50 μL | 49 μL × x |
טבלה 2: רכיבי תגובת פי.סי.אר. * השתמש ב-gDNA 10x, 100x או 1000x מדולל מהשלב 2.3.
| צעד | טמפרטורה | זמן | מחזורים |
| denaturation ראשונית | 98°C | 30 שניות | |
| דנטורציה | 98°C | 10 שניות | 25-30 |
| חישול | 60°C | 30 שניות | |
| סיומת | 72°C | 45 שניות | |
| הארכה סופית | 72°C | 7 דקות | |
| אחז | 4°C | ∞ |
טבלה 3: תוכנית PCR להגברת הגן 16S rRNA.

איור 4: אלקטרופורזה ג'ל אגרוז של מוצרי PCR מוגבר באמצעות פריימרים 8F ו 1492R ו- gDNA כתבנית. דגימת ה-gDNA מ-B. subtilis (ראו איור 3) דולדה 10, 100 ו-1000 פעמים כדי לבדוק את התוצאה הטובה ביותר. נתיב 1: M - סמן מסה מולקולרית (מלמעלה למטה: 10000 bp, 8000 bp, 6000 bp, 5000 bp, 4000 bp, 3500 bp, 3000 bp, 2500 bp, 2000 bp, 1500 bp, 1000bp, 750 bp, 500 bp, 250 bp). נתיב 2: תגובת PCR עם תבנית מדוללת פי 10. נתיב 3: תגובת PCR עם תבנית מדוללת 100x. נתיב 4: תגובת PCR עם תבנית מדוללת 1000x. נתיב 5: (C-) - שליטה שלילית (תגובה ללא תבנית ה- DNA). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
3. ניתוח נתונים ותוצאות
הערה: מוצר PCR הוא רצף באמצעות קדימה (כאן 8F) ואת הפריימרים ההפוכים (כאן 1492R). לכן, נוצרות שתי קבוצות של רצף נתונים, אחת עבור הפורוורד ואחת עבור פריימר הפוך. עבור כל רצף נוצרים לפחות שני סוגים של קובץ: i) קובץ טקסט המכיל את רצף ה- DNA ו- ii) כרומטוגרמה של DNA, המציגה את איכות ריצת הרצף.
- עבור הפריימר הקדמי, פתח את הכרומטוגרמה, ובחן בקפידה את הרצף. כרומטוגרמה אידיאלית לרצף איכות צריכה להיות פסגות מרווחות באופן שווה ואותות רקע מעטים או ללא אותות רקע (איור 5A).
- אם הכרומטוגרמה אינה באיכות גבוהה, יש למחוק את הרצף, או לשנות את קובץ טקסט הרצף בהתאם לאפשרויות הבאות:
- נוכחות של פסגות כפולות ברחבי הכרומטוגרמה מצביעה על נוכחות של תבניות DNA מרובות. זה יכול להיות המקרה אם התרבות החיידקית לא הייתה טהורה. יש לבטל רצף כזה (איור 5B).
- כרומטוגרמה מעורפלת עשויה לנבוע מנוכחות של פסגות צבעוניות שונות באותו מיקום. אחת השגיאות הנפוצות ביותר היא נוכחות של שתי פסגות צבעוניות שונות באותו מיקום והקצאה לא נכונה של הבסיסים על ידי תוכנת הרצף (איור 5C). תקן באופן ידני את כל הנוקלאוטידים שהוקצו באופן שגוי וערוך אותם בקובץ הטקסט.
- כרומטוגרמה ברזולוציה נמוכה עלולה לגרום ל"פסגות רחבות" שלעתים קרובות גורמות לספירה שגויה של הנוקלאוטידים באזורים אלה (איור 5D). שגיאה זו קשה לתיקון, ולכן אין להתייחס לאי-התאמות אפשריות בשלב היישור הנוסף כאמינים.
- איכות קריאה ירודה של כרומטוגרמה ונוכחות של פסגות מרובות נראית בדרך כלל בקצוות של 5 ' ו -3 'של הרצף. תוכנות רצפים מסוימות מסירות קטעים באיכות נמוכה אלה באופן אוטומטי (איור 5E), והנוקלאוטידים אינם כלולים בקובץ הטקסט. אם הרצף שלך לא נחתך באופן אוטומטי, קבע את הקטעים באיכות נמוכה (למשל אות חלש, פסגות חופפות, אובדן רזולוציה) בקצוות והסר את הבסיסים המתאימים מקובץ הטקסט.

איור 5: דוגמאות לפתרון בעיות ברצף דנ"א. A) דוגמה לרצף כרומטוגרמה איכותי (פסגות חד-משמעיות בעלות מרווחים שווים). B) רצף איכות ירודה המתרחש בדרך כלל בתחילת הכרומטוגרמה. אזור האזור האפור נחשב לאיכות נמוכה והוסר באופן אוטומטי על-ידי תוכנת הרצף. ניתן לקצץ בסיסים נוספים באופן ידני. ג) נוכחות של פסגות כפולות (המצוינות על ידי חצים). נוקלאוטיד המצוין על ידי החץ האדום נקרא על ידי הרצף כ- "T" (פסגה אדומה), אך הפסגה הכחולה חזקה יותר, והיא יכולה להתפרש גם כ- "C". ד) פסגות חופפות מצביעות על זיהום DNA (כלומר יותר מתבנית אחת). ה) אובדן רזולוציה ומכונה "פסגות רחבות" (המסומנות במלבן) המונעות שיחות בסיס אמינות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- חזור על 3.1 ו-3.2 עבור הפריימר ההפוך.
- לבסוף, להרכיב את הרצפים קדימה והפוך לתוך רצף רציף אחד. ריצת רצף טובה מניבה רצף של עד 1100 bp. בהתחשב בכך שמוצר ה- PCR הוא באורך ~ 1500 bp, הרצפים המתקבלים באמצעות פריימרים קדמיים והופוכים צריכים לחפוף חלקית.
- מזג את שני הרצפים באמצעות תוכנית ההרכבה של רצף ה- DNA, למשל כלי חופשי כגון CAP3 (http://doua.prabi.fr/software/cap3) (15).
- הוסף את שני הרצפים בתבנית FASTA לתיבה שצוינה. לחץ על לחצן "שלח" והמתן לחזרת התוצאות.
- כדי להציג את הרצף המורכב הקש "Contigs" בכרטיסיית התוצאה. כדי להציג את פרטי היישור, הקש על "פרטי ההרכבה".
הערה 1: אם תוכנת CAP3 משמשת להרכבה contig אין צורך להמיר את רצף פריימר הפוך לתוך הפוך משלים; עם זאת, ייתכן שיהיה צורך בשלב זה אם נעשה שימוש בתוכנית אחרת.
הערה 2: תבנית FASTA היא תבנית מבוססת טקסט המייצגת את רצף הנוקלאוטידים. השורה הראשונה (שורת התיאור) בקובץ FASTA מתחילה בסימן ">" ואחריו השם או המזהה הייחודי של הרצף. לאחר שורת התיאור נמצא רצף הנוקלאוטידים. הדבק את הרצפים בתבנית הבאה:
>sequence_frw_primer
הדבק את הרצף מקובץ הטקסט כאן
>sequence_rvs_primer
הדבק את הרצף מקובץ הטקסט כאן
- ערוך חיפוש במסד נתונים על-ידי ביקור באתר האינטרנט עבור כלי חיפוש היישור המקומי הבסיסי (BLAST; https://blast.ncbi.nlm.nih.gov/Blast.cgi).
- בחר בכלי "Nucleotide BLAST" כדי להשוות את הרצף למסד הנתונים.
- הזן את הרצף שלך (הרצף שהורכב ב- 3.5) בתיבת הטקסט "רצף שאילתות", ולאחר מכן בחר את מסד הנתונים "רצפי rRNA 16S (חיידקים וארצ'יה)" בתפריט הגלילה למטה.
- לחץ על לחצן "BLAST" בתחתית העמוד. הרצפים הדומים ביותר יוחזרו. תוצאת BLAST לדוגמה מוצגת באיור 6. בניסוי שהוצג הלהיט העליון הוא B. subtilis זן 168, מראה 100% זהות עם הרצף זמין במסד הנתונים BLAST.
- אם הלהיט העליון אינו מציג 100% זהות, עבור ליישור ובדוק אם יש אי-התאמות. בעת לחיצה על הלהיט העליון, תופנה לפרטי היישור. נוקלאוטידים מיושרים יצטרפו לקווים אנכיים קצרים בעוד לנוקלאוטידים לא תואמים יש פער ביניהם. חזור לכרומטוגרמה שקיבלת מחברת הרצף ושנה את הרצף שוב תוך התמקדות באזור שאינו תואם. תקן את הרצף אם נמצאו שגיאות נוספות. הפעל שוב את BLAST באמצעות הרצף המתוקן.

איור 6: דוגמה לתוצאה של נוקלאוטיד BLAST. רצף גנים rRNA 16S מתרבות טהורה של B. subtilis 168 שימש כרצף שאילתות. הלהיט העליון מראה 100% זהות (הדגיש) לזן B. subtilis 168, כצפוי. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
כדור הארץ הוא ביתם של מיליוני מינים חיידקיים, כל אחד עם מאפיינים ייחודיים. זיהוי מינים אלה הוא קריטי בהערכת דגימות סביבתיות. רופאים גם צריכים להבחין בין מינים חיידקיים שונים כדי לאבחן חולים נגועים.
כדי לזהות חיידקים, ניתן להשתמש במגוון טכניקות, כולל תצפית מיקרוסקופית של מורפולוגיה או צמיחה באמצעי תקשורת ספציפי כדי לבחון את המורפולוגיה של המושבה. ניתוח גנטי, טכניקה נוספת לזיהוי חיידקים גדלה בפופולריות בשנים האחרונות, בין היתר בשל ריצוף גנים ריבוזומלי RNA 16S.
ריבוזום חיידקי הוא קומפלקס RNA חלבון המורכב משני תתי-units. תא ה- subunit של 30S, הקטן יותר משני יחידות המשנה הללו, מכיל 16S rRNA, המקודד על ידי הגן rRNA 16S הכלול בדנ"א הגנומי. אזורים ספציפיים של rRNA 16S נשמרים מאוד, בשל תפקידם החיוני בהרכבה ריבוזום. בעוד שאזורים אחרים, פחות קריטיים לתפקוד, עשויים להשתנות בין מיני חיידקים. האזורים המשתנים ב- 16S rRNA, יכולים לשמש טביעות אצבע מולקולריות ייחודיות למינים חיידקיים, ומאפשרים לנו להבחין בין זנים זהים פנוטיפיים.
לאחר קבלת דגימת איכות של gDNA, PCR של גן קידוד rRNA 16S יכול להתחיל. PCR היא שיטה נפוצה בביולוגיה מולקולרית, המורכבת ממחזורים של דנטורציה של תבנית ה- DNA הכפולה, חישול של זוגות פריימר אוניברסליים, אשר מגבירים אזורים שמורים מאוד של הגן, ואת הרחבת פריימרים על ידי DNA פולימראז. בעוד שחלק מהפריימרים מגבירים את רוב הגן של קידוד rRNA 16S, אחרים רק מגבירים שברים ממנו. לאחר PCR, ניתן לנתח את המוצרים באמצעות אלקטרופורזה ג'ל אגרוז. אם ההגברה הצליחה, הג'ל צריך להכיל רצועה אחת בגודל צפוי, בהתאם לזוג הפריימרים המשמש, עד 1500bp, האורך המשוער של הגן 16S rRNA.
לאחר טיהור ורצף, ניתן להזין את הרצפים המתקבלים למסד הנתונים BLAST, שם ניתן להשוותם לרצפי rRNA של 16S. כמו מסד נתונים זה מחזיר התאמות המבוססות על הדמיון הגבוה ביותר, זה מאפשר אישור של זהות החיידקים של עניין. בסרטון וידאו זה, תוכלו לצפות ריצוף גנים 16S rRNA, כולל PCR, ניתוח רצף DNA ועריכה, הרכבה רצף וחיפוש מסד נתונים.
בעת טיפול במיקרואורגניזמים, חיוני לעקוב אחר פרקטיקה מיקרוביולוגית טובה, כולל שימוש בטכניקה אספטית ולבוש ציוד מגן אישי מתאים. לאחר ביצוע הערכת סיכונים מתאימה עבור מיקרואורגניזם או מדגם סביבתי של עניין, להשיג תרבות בדיקה. בדוגמה זו, נעשה שימוש בתרבות טהורה של Bacillus subtilis.
כדי להתחיל, לגדל את המיקרואורגניזם שלך על מדיום מתאים בתנאים המתאימים. בדוגמה זו, Bacillus subtilis 168 גדל מרק LB לילה באינקובטור רועד להגדיר 200 סל"ד ב 37 מעלות צלזיוס. לאחר מכן, השתמש בערכה זמינה מסחרית כדי לבודד DNA גנומי או gDNA מ 1.5 מיליליטר של B. subtilis תרבות הלילה.
כדי לבדוק את איכות הדנ"א המבודד, מערבבים תחילה חמישה מיקרוליטרים של ה-gDNA המבודד עם מיקרוליטר אחד של צבע טעינת ג'ל DNA. לאחר מכן, לטעון את המדגם על ג'ל אגרוז 0.8%, המכיל DNA מכתים ריאגנט, כגון SYBR בטוח או EtBr. לאחר מכן, לטעון תקן מסה מולקולרית קילו-בסיס אחד על הג'ל, ולהפעיל את האלקטרופורזה עד הצבע הקדמי הוא כ 0.5 ס"מ מתחתית הג'ל. לאחר השלמת אלקטרופורזה ג'ל, לדמיין את הג'ל על transilluminator אור כחול. ה- gDNA צריך להופיע כפס עבה, מעל 10 קילו בסיס בגודל ויש להם מריחה מינימלית.
לאחר מכן, כדי ליצור דילול סדרתי של gDNA, סמן שלושה צינורות microcentrifuge כמו 10X, 100X, ו 1000X. לאחר מכן, השתמש פיפטה כדי לחלק 90 microliters של מים מזוקקים סטריליים לתוך כל הצינורות. לאחר מכן, הוסף 10 מיקרוליטרים של פתרון gDNA לצינור 10X. Pipette את כל עוצמת הקול למעלה ולמטה כדי להבטיח את הפתרון מעורבב ביסודיות. לאחר מכן, להסיר 10 microliters של הפתרון מן הצינור 10X ולהעביר את זה לצינור 100X. ערבבו את הפתרון כפי שתואר קודם לכן. לבסוף, להעביר 10 microliters של הפתרון בצינור 100X, לצינור 1000X.
כדי להתחיל את פרוטוקול PCR, להפשיר את ריאגנטים הדרושים על קרח. לאחר מכן, הכינו את תערובת המאסטר של PCR. מאז פולימראז DNA פעיל בטמפרטורת החדר, התגובה להגדיר חייב להתרחש על קרח. Aliquot 49 microliters של תערובת האב לתוך כל אחד צינורות PCR. לאחר מכן, הוסף מיקרוליטר אחד של תבנית לכל אחד מהצינורות הניסיוניים ומיקרוליטר אחד של מים סטריליים לצינור הבקרה השלילי, צנרת למעלה ולמטה לערבב. לאחר מכן, הגדר את מחשב ה- PCR בהתאם לתוכנית המתוארת בטבלה. מניחים את הצינורות לתוך התרמוציקלר ולהתחיל את התוכנית.
לאחר השלמת התוכנית, לבחון את איכות המוצר שלך באמצעות אלקטרופורזה ג'ל agarose, כפי שהודגם בעבר. תגובה מוצלחת באמצעות הפרוטוקול המתואר אמורה להניב רצועה אחת של כ-1.5 קילו-בסיס. בדוגמה זו, המדגם המכיל 100X מדולל gDNA הניב את המוצר באיכות הגבוהה ביותר. לאחר מכן, לטהר את המוצר PCR הטוב ביותר, במקרה זה, 100X gDNA, עם ערכה זמינה מסחרית. כעת ניתן לשלוח את מוצר ה- PCR לרצף.
בדוגמה זו, מוצר PCR הוא רצף באמצעות פריימרים קדימה והפוך. לפיכך, נוצרות שתי ערכות נתונים, שכל אחת מהן מכילה רצף דנ"א וכרום דנ"א: אחת עבור הפריימר הקדמי והשנייה עבור הפריימר ההפוך. ראשית, לבחון את הכרומטוגרמה שנוצרת מכל פריימר. כרומטוגרמה אידיאלית הייתה צריכה להיות פסגות מרווחות באופן שווה עם מעט מאוד אותות רקע.
אם הכרומטוגרמה מציגה פסגות כפולות, ייתכן שתבניות DNA מרובות היו קיימות במוצרי ה- PCR ויש למחוק את הרצף. אם הכרומטוגרמה הכילה פסגות של צבעים שונים באותו מיקום, סביר להניח שתוכנת הרצף קראה לא נכון נוקלאוטידים. ניתן לזהות ולתקן שגיאה זו באופן ידני בקובץ הטקסט. נוכחותם של פסגות רחבות בכרומטוגרמה מצביעה על אובדן רזולוציה, מה שגורם לספירה שגויה של הנוקלאוטידים באזורים הקשורים. שגיאה זו קשה לתיקון ויש להתייחס לאי-התאמות בכל אחד מהצעדים הבאים כלא אמינים. איכות קריאה ירודה של כרומטוגרמה, המצוינת על ידי נוכחות של פסגות מרובות, מתרחשת בדרך כלל בחמשת הקצוות העיקריים ושלושת הקצוות העיקריים של הרצף. תוכניות רצף מסוימות מסירות מקטעים באיכות נמוכה אלה באופן אוטומטי. אם הרצף שלך לא נחתך באופן אוטומטי, זהה את הקטעים באיכות נמוכה והסר את הבסיסים המתאימים שלהם מקובץ הטקסט.
השתמש בתוכנית הרכבה DNA כדי להרכיב את שני רצפי פריימר לרצף רציף אחד. זכור, רצפים המתקבלים באמצעות פריימרים קדמיים והופכים צריכים לחפוף חלקית. בתוכנית ההרכבה של ה- DNA, הכנס את שני הרצפים בתבנית FASTA לתיבה המתאימה. לאחר מכן, לחץ על לחצן שלח והמתן עד שהתוכנית תחזיר את התוצאות.
כדי להציג את הרצף המורכב, לחץ על Contigs בכרטיסיה תוצאות. לאחר מכן, כדי להציג את פרטי היישור, בחר פרטי הרכבה. נווט אל אתר האינטרנט עבור כלי חיפוש היישור המקומי הבסיסי, או BLAST, ובחר בכלי BLAST נוקלאוטיד כדי להשוות את הרצף שלך למסד הנתונים. הזן את הרצף בתיבת הטקסט של רצף השאילתות ובחר את מסד הנתונים המתאים בתפריט הגלילה למטה. לבסוף, לחץ על לחצן BLAST בתחתית הדף והמתן שהכלי יחזיר את הרצפים הדומים ביותר ממסד הנתונים.
בדוגמה זו, הלהיט העליון הוא B. subtilis זן 168, המציג 100% זהות עם הרצף במסד הנתונים BLAST. אם הלהיט העליון אינו מציג 100% זהות למין או לזן הצפויים שלך, לחץ על הרצף המתאים ביותר לשאילתה שלך כדי לראות את פרטי היישור. לנוקלאוטידים מיושרים יצטרפו קווים אנכיים קצרים ולנוקלאוטידים לא תואמים יהיו פערים ביניהם. התמקדות באזורים שלא הותרו, ערוך את הרצף וחזור על חיפוש BLAST אם תרצה בכך.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
זיהוי מינים חיידקיים חשוב לחוקרים שונים, כמו גם עבור אלה בתחום הבריאות. רצף rRNA 16S שימש בתחילה על ידי החוקרים כדי לקבוע יחסים פילוגנטיים בין חיידקים. עם הזמן, זה יושם במחקרים metagenomic כדי לקבוע את המגוון הביולוגי של דגימות סביבתיות במעבדות קליניות כשיטה לזהות פתוגנים פוטנציאליים. היא מאפשרת זיהוי מהיר ומדויק של חיידקים הנמצאים בדגימות קליניות, מקלה על אבחון מוקדם יותר וטיפול מהיר יותר בחולים.
Subscription Required. Please recommend JoVE to your librarian.
References
- Weisburg, W.G., Barns, S.M., Pelletier, D.A. and Lane D.J. 16S ribosomal DNA amplification for phylogenetic study. J Bacteriol. 173 (2): 697-703. (1991)
- Drancourt, M., Bollet, C., Carlioz, A., Martelin, R., Gayral, J.P., Raoult D. 16S ribosomal DNA sequence analysis of a large collection of environmental and clinical unidentifiable bacterial isolates. J Clin Microbiol. 38 (10):3623-3630. (2000)
- Woo, P.C., Lau, S.K., Teng, J.L., Tse, H., Yuen, K.Y. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clin Microbiol Infect. 14 (10):908-934. (2008)
- Tang, Y.W., Ellis, N.M., Hopkins, M.K., Smith, D.H., Dodge, D.E., Persing, D.H. Comparison of phenotypic and genotypic techniques for identification of unusual aerobic pathogenic gram-negative bacilli. J Clin Microbiol. 36 (12):3674-3679. (1998)
- Tsiboli, P., Herfurth, E., Choli, T. Purification and characterization of the 30S ribosomal proteins from the bacterium Thermus thermophilus. Eur J Biochem. 226 (1):169-177. (1994)
- Woese, C.R. Bacterial evolution. Microbiol Rev. 51 (2):221-271. (1987)
- Bartlett, J.M., Stirling, D. A short history of the polymerase chain reaction. Methods Mol Biol. 226:3-6. (2003)
- Wilson, K.H., Blitchington, R.B., Greene, R.C. Amplification of bacterial 16S ribosomal DNA with polymerase chain reaction. J Clin Microbiol. 28 (9):1942-1946. (1990)
- Shendure, J., Balasubramanian, S., Church, G.M., Gilbert, W., Rogers, J., Schloss, J.A., Waterston, R.H. (2017) DNA sequencing at 40: past, present and future. Nature. 550:345-353.
- Lane, D.J. 16S/23S rRNA sequencing. (1991) In Nucleic acid techniques in bacterial systematics. (Goodfellow, M. and Stackebrandt, E., eds.) p.115-175. Wiley and Sons, Chichester, United Kingdom.
- Turner, S., Pryer, K.M., Miao, V.P., Palmer, J.D. (1999) Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis. J Eukaryot Microbiol. 46:327-338.
- Fredricks, D.N., Relman, D.A. (1998) Improved amplification of microbial DNA from blood cultures by removal of the PCR inhibitor sodium polyanetholesulfonate. J Clin Microbiol. 36:2810-2816.
- Wilson, K. Preparation of genomic DNA from bacteria. (2001) Curr Protoc Mol Biol. Chapter 2:Unit 2.4.
- Wright, M. H., Adelskov, J., Greene, A.C. (2017) Bacterial DNA extraction using individual enzymes and phenol/chloroform separation. J Microbiol Biol Educ. 18:18.2.48.
- Huang, X., Madan, A. (1999). CAP3: A DNA sequence assembly program. Genome Res. 9:868-877.




