Overview
Fonte: Alexander S. Gold1, Tonya M. Colpitts1
1 Dipartimento di Microbiologia, Boston University School of Medicine, National Emerging Infections Diseases Laboratories, Boston, MA
Scoperta per la prima volta da Lederberg e Tatum nel 1946, la coniugazione è una forma di trasferimento genico orizzontale tra batteri che si basa sul contatto fisico diretto tra due cellule batteriche (1). A differenza di altre forme di trasferimento genico, come la trasformazione o la trasduzione, la coniugazione è un processo naturale in cui il DNA viene secreto da una cellula donatrice a una cellula ricevente in modo unidirezionale. Questa direzionalità e la capacità di questo processo di aumentare la diversità genetica dei batteri ha dato alla coniugazione la reputazione di una forma di "accoppiamento" batterico, che si ritiene abbia contribuito notevolmente al recente aumento dei batteri resistenti agli antibiotici (2, 3). Utilizzando pressioni selettive, ad esempio l'uso di antibiotici, la coniugazione è stata manipolata per l'uso in laboratorio, rendendola un potente strumento per il trasferimento genico orizzontale tra batteri e, in alcuni casi, da batteri a lieviti, cellule vegetali e animali (4). Oltre alle applicazioni in laboratorio, il trasferimento genico batterio-eucariota mediante coniugazione è un'entusiasmante via di trasferimento del DNA con una moltitudine di possibili applicazioni biotecniche e implicazioni naturali (5).
Si pensa che la coniugazione funzioni con un "meccanismo a due fasi" (6). In primo luogo, prima che qualsiasi DNA possa essere trasferito, la cellula donatrice deve entrare in contatto diretto cellula a cellula con il ricevente. Questo processo è stato caratterizzato meglio nei batteri gram-negativi, il più studiato dei quali è Escherichia coli. Il contatto cellula-cellula è stabilito dalla presenza di una complessa rete di filamenti extracellulari sul donatore nota come sex pilus, un elemento coniugativo codificato dal gene trasferibile noto come fattore F (fertilità) (7, 8). Oltre a stabilire un contatto tra donatore e ricevente, diverse proteine vengono trasportate attraverso il pilus sessuale al citoplasma ricevente, formando un condotto del sistema di secrezione di tipo IV (T4SS) tra le due cellule, una struttura necessaria per la seconda fase di coniugazione, il trasferimento del DNA (6). Combinando questa funzione del pilus sessuale con la replicazione del cerchio rotolante del DNA, la cellula donatrice è in grado di trasferire il DNA sotto forma di un elemento trasponibile, come un plasmide o un trasposone, al ricevente mediante un modello "shoot and pump" (6). In questo caso, lo "shooting" è il trasporto della proteina pilota, con DNA collegato, da parte del T4SS nella cellula ricevente, e il "pumping" è il trasporto attivo del DNA al ricevente, un processo dipendente dal T4SS e catalizzato dall'accoppiamento delle proteine (6). Il macchinario utilizzato in questo processo è composto da un'origine di sequenza di trasferimento (oriT), che deve essere fornita dal DNA nei geni cis e trans, che codificano una relaxasi, un complesso di formazione di coppie di accoppiamenti e una proteina di accoppiamento di tipo IV, e possono essere presenti in cis o trans (9). Questa relaxasi fende il sito nic all'interno della sequenza oriT e si attacca covalentemente all'estremità 5' del filamento trasferito per produrre il relaxosoma, un complesso DNA-relaxasi a singolo filamento con altre proteine ausiliarie (9). Una volta formato, il relaxosoma si collega al complesso di formazione della coppia di accoppiamento, tramite la proteina di accoppiamento di tipo IV, che consente il trasferimento del complesso ssDNA-relaxasi nelle cellule riceventi da parte del T4SS (10). Una volta nel citoplasma del ricevente, il DNA può integrarsi nel genoma del ricevente o esistere separatamente sotto forma di plasmide, entrambi i quali consentono l'espressione dei suoi geni.
In questo esperimento, il ceppo di donatore di coniugazione ampiamente utilizzato E. coli WM3064 è stato utilizzato per trasferire il gene che codifica per la resistenza all'ampicillina al ceppo ricevente E. coli J53. Mentre entrambi i ceppi dei batteri gram-negativi erano resistenti alla tetraciclina, solo il ceppo donatore WM3064 aveva il gene per la resistenza all'ampicillina, codificato per nel vettore navetta pWD2-oriT, ed era auxotrofico all'acido diaminopimetico (DAP) (11-13). Questo esperimento consisteva in due fasi principali, la preparazione di ceppi donatore e ricevente, seguita dal trasferimento del gene di resistenza all'ampicillina dal donatore al ricevente mediante coniugazione (Figura 1).
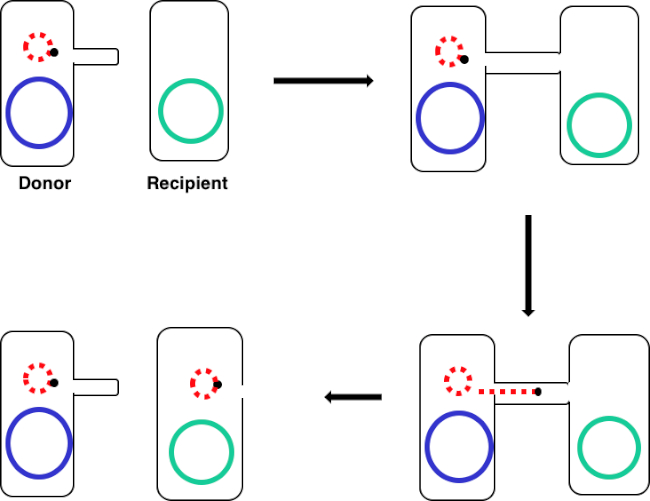
Figura 1: Schema di coniugazione. Questo schema mostra il successo del trasferimento di un plasmide, solo un esempio di elemento di DNA trasponibile, da una cellula donatrice a una cellula ricevente usando la coniugazione. Al contatto con la cellula ricevente da parte della cellula donatrice attraverso il pilus sessuale, il plasmide si replica mediante replicazione del cerchio rotolante, si muove attraverso il complesso multiproteico unendo le due cellule e forma un nuovo plasmide a lunghezza intera nella cellula ricevente.
Incubando una miscela di cellule donatrici e riceventi, quindi placcando successivamente queste cellule in presenza di tetraciclina e DAP, ciò ha permesso il trasferimento riuscito del gene di resistenza all'ampicillina. Successivamente, le cellule placcate cresciute da questa miscela in presenza di tetraciclina e ampicillina, hanno rimosso tutte le cellule donatrici a causa della mancanza di DAP e di eventuali cellule riceventi che potrebbero non aver acquisito il gene di resistenza all'ampicillina, producendo batteri del ceppo J53 strettamente riceventi che hanno acquisito resistenza all'ampicillina (Figura 2). Una volta effettuato, il successo del trasferimento del gene di resistenza all'ampicillina è stato confermato dalla PCR. Poiché la coniugazione ha avuto successo, il ceppo J53 di E. coli conteneva pWD2-oriT ed era resistente all'ampicillina, e il gene che codifica per questa resistenza è rilevabile dalla PCR. Tuttavia, in caso di insuccestamento non ci sarebbe stato alcun rilevamento del gene di resistenza all'ampicillina e l'ampicillina avrebbe comunque funzionato come un antibiotico efficace contro il ceppo J53.
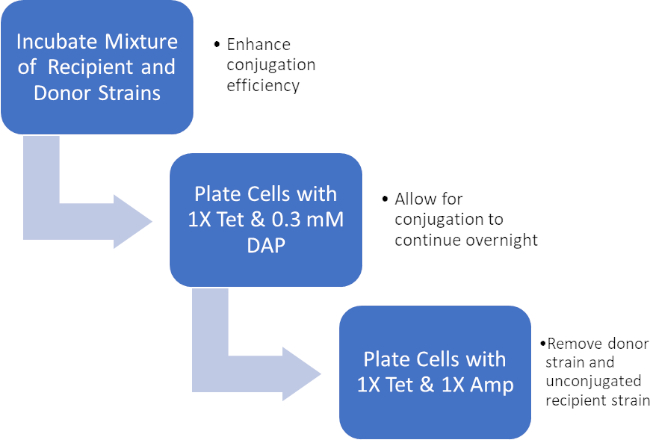
Figura 2: Schema del protocollo. Questo schema mostra una panoramica del protocollo presentato.
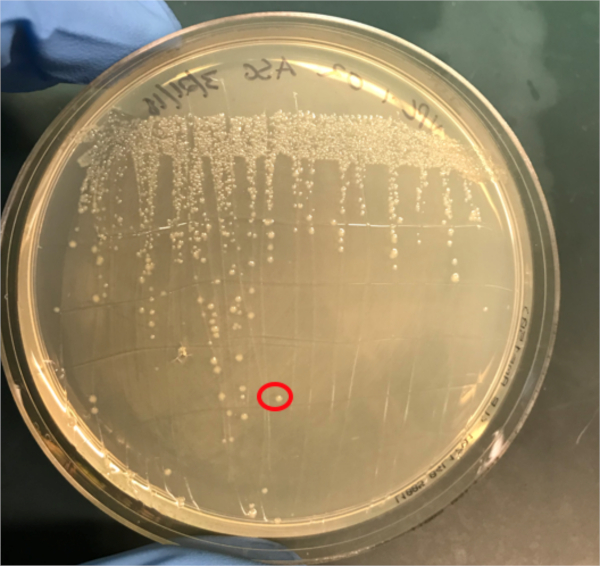
Figura 3A: La conferma del successo della coniugazione mediante PCR. A) Le scorte congelatrici dei campioni di controllo coniugati e negativi sono state strisciate su piastre di agar e una colonia è stata selezionata (rossa) per l'isolamento del DNA.
Procedure
1. Configurazione
- Autoclave circa 1L di Luria-Bertani medium (LB). Questo LB sterile sarà utilizzato per produrre circa 5 ml di LB contenente 0,3 mM di acido diaminopimetico (DAP).
- Raccogliere le seguenti piastre: piastre di agar LB con 1X Tet e 0,3 mM DAP, piastre lb agar con 1X Tet solo e piastre LB agar con solo 1X Amp/Tet.
- Assicurarsi che un po 'di glicerolo e una scatola di punte di pipette di plastica pre-sterilizzate siano a portata di mano.
- Prima di iniziare qualsiasi lavoro che coinvolga i microbi, sterilizzare lo spazio di lavoro con il 70% di etanolo. Indossare sempre i dispositivi di protezione individuale necessari, tra cui un camice da laboratorio e guanti.
- Una volta terminato, sterilizzare tutte le superfici e i guanti con etanolo al 70% e lavarsi le mani.
2. Preparazione del ceppo del donatore e del ricevente
- Preparare colture batteriche da 5 ml dei ceppi donatori e riceventi e coltivarle durante la notte a 37 °C con aerazione e agitazione a 220 giri/min. Il ceppo donatore deve essere coltivato in LB con 0,3 mM DAP.
- Girare verso il basso 1 mL di entrambe le colture (~ 3000 rpm per 5 minuti) e lavare le celle con PBS.
- Spese di sodio delle cellule in 500 μL di soluzione salina tamponata con fosfato (PBS).
3. Coniugazione
- Combinare 50 μL di cellule riceventi con 50 μL di cellule donatrici in un tubo di microcentrifuga e mescolare mediante pipettaggio.
- Incubare la miscela cellulare per 1 ora a 37 °C. Ciò migliorerà l'efficienza della coniugazione prima e durante la fase successiva della placcatura.
- Pipettare 100 μL della miscela cellulare su una piastra di agar con 1X Tet e 0,3 mM DAP. Non stendere sul piatto.
- Pipettare 100 μL di coltura cellulare ricevente su una piastra di agar con 1X Tet e 0,3 mM DAP. Non stendere sul piatto.
- Incubare entrambe le piastre durante la notte a 37 °C.
- Raschiare la miscela cellulare di coniugazione e la coltura cellulare ricevente con un raschietto cellulare sterile. Trasferire le cellule in un tubo microcentrifuga sterile e risospentare le cellule in 1 mL di PBS.
- Ruotare le celle e ruotarle delicatamente verso il basso (~ 3000 giri / min per 5 minuti).
- Risuspendare le cellule in 1 mL di PBS.
- Placcare la miscela di cellule di reazione di coniugazione su una piastra di agar LB con solo 1X Amp/Tet. Su questa piastra, solo i batteri riceventi che hanno ottenuto con successo il gene di resistenza Amp tramite coniugazione dovrebbero crescere.
- Placcare le cellule riceventi su una piastra di agar LB con solo 1X Tet. Su questa piastra, solo i batteri riceventi non coniugati, che non hanno il gene di resistenza Amp, dovrebbero crescere.
- Incubare le piastre durante la notte a 37 °C.
- Prelevare singole colonie da entrambe le piastre e utilizzarle per coltivare colture notturne in 5 ml di mezzi (37 °C con aerazione a 220 giri/min).
4. Isolamento del DNA
- Isolare il DNA dalle colture precedentemente preparate utilizzando 4,5 ml del volume totale di coltura mediante DNA miniprep.
- Per fare questo, eluire il DNA usando 35 μL di acqua priva di nucleasi.
- Il DNA puro genererà un rapporto di assorbanza (A260/280)di circa 1,8.
- Utilizzare i restanti 0,5 ml di ciascuna coltura per preparare le scorte di glicerolo facendo una miscela 1: 1 di coltura batterica e glicerolo al 100%.
- Conservare le scorte congelatrici a -80 °C.
5. Conferma della coniugazione del trasferimento plasmidiale mediante PCR
- Preparare due master mix PCR, ciascuno con un diverso set di primer avanti e indietro, uno mirato a un segmento di 500 coppie di basi all'interno del gene di resistenza all'ampicillina e l'altro mirato a un segmento all'interno di un gene di pulizia.
- I primer genetici di housekeeping sono stati progettati per amplificare un segmento di DNA all'interno del gene batterico che codifica per la DNA girasi B (14).
- I seguenti volumi di reagenti sono stati utilizzati per preparare 90 μL di ciascuna miscela master:
7,5 μL di primer avanti da 10 μM
7,5 μL di primer inverso da 10 μM
75 μL di 2X PCR Master Mix
- Preparare le seguenti sei reazioni PCR utilizzando una miscela master da 15 μL, 10 ng di DNA modello e acqua priva di nucleasi fino a un volume finale di 25 μL.
Reazione coniugante DNA e ampicillina primer
DNA di reazione coniugale e primer per la pulizia
Primer di controllo negativo del DNA e dell'ampicillina
DNA di controllo negativo e primer per le pulizie
Senza primer dna e ampicillina
Nessun DNA e primer per le pulizie - Trasferire queste reazioni a una macchina PCR con il blocco preriscaldato a 98 C e iniziare il termociclico nelle seguenti condizioni:
98 °C per 30 secondi
25-35 cicli di 98 °C per 5-10 secondi, 45-72 °C per 10-30 secondi e 72 °C per 15-30 secondi per kb
72 °C per 5-10 minuti
Tenere a 4 °C - Caricare tutte e sei le reazioni PCR su un gel di acarosio all'1% ed eseguire a ~ 150V per circa 20 minuti.
- Visualizza il prodotto PC utilizzando un illuminatore UV.
Le cellule batteriche, come E. coli, sono in grado di trasferire informazioni genetiche da cellula a cellula. La coniugazione differisce da altri meccanismi di trasferimento del DNA, come la trasduzione o la trasformazione, in quanto richiede il contatto fisico tra le cellule.
Per procedere, la coniugazione richiede una cellula donatrice che esprime il fattore fertilità, o F, e una cellula ricevente senza di essa, una cellula F meno. Il processo richiede due passaggi. Il primo è la creazione di un contatto diretto cellula-cellula. Per fare questo, la cellula donatrice genera una struttura filamentosa extracellulare chiamata pilus sessuale. Si chiama così poiché la coniugazione è una forma di accoppiamento per batteri che si riproducono asessualmente, ma va notato che non è vera riproduzione sessuale in quanto non vengono scambiati gameti e non si forma prole.
Il secondo passo è la consegna del DNA alla cellula ricevente. Dopo che il pilus sessuale stabilisce il contatto tra due cellule, viene costruito un condotto chiamato sistema di secrezione di tipo IV che consente il trasferimento del DNA. La cellula donatrice inizia quindi a replicare il DNA extracromosomiale che verrà trasferito selezionato in base alla presenza di un elemento genetico noto come OriT o origine del trasferimento. Un'estremità del DNA appena replicato viene infilata nel condotto attraverso il legame con la proteina del DNA. Man mano che il DNA viene ulteriormente replicato, viene pompato attraverso il canale, facilitato da un complesso di proteine codificate da geni situati vicino all'OriT. Una volta che il DNA è completamente trasferito, formerà un plasmide cromosomico extra o potrebbe integrarsi nel cromosoma della cellula ricevente. Qualunque sia l'endpoint del DNA trasferito, i geni che codifica saranno quindi espressi. Questa espressione genica può essere utilizzata per confermare il successo della coniugazione.
Ad esempio, si consideri uno scenario in cui il ceppo donatore esprime resistenza all'ampicillina e la trasmette nel DNA coniugato al batterio ricevente, ma il ceppo ricevente ha anche un gene di resistenza alle tetracicline non presente nel donatore. In questo caso, quando le cellule sono placcate su supporti LB contenenti sia tetraciclina che ampicillina, le colonie dovrebbero crescere solo da batteri coniugati con successo, che esprimeranno entrambi i fenotipi di resistenza. Per confermare ulteriormente il successo della coniugazione, il DNA plasmidico di queste colonie può essere raccolto e quindi una sezione di DNA specifica per il plasmide trasferito può essere amplificata utilizzando la reazione a catena della polimerasi o PCR. Quando il prodotto PCR viene eseguito su un gel di elettroforesi insieme a una scala di dimensioni standard, un frammento di PCR di dimensioni note dovrebbe essere visibile sul gel, confermando ulteriormente il successo della coniugazione. In questo esperimento, un plasmide sarà utilizzato per trasferire il gene di resistenza all'ampicillina tramite coniugazione da un ceppo donatore a un ceppo ricevente resistente alle tetracicline. Dopo questo, per confermare la coniugazione, la miscela di coniugazione verrà incubata su una piastra contenente entrambi gli antibiotici lasciando solo i batteri trasformati. Infine, il successo della coniugazione sarà ulteriormente confermato con la PCR.
Prima di iniziare la procedura, indossare i dispositivi di protezione individuale appropriati, tra cui un cappotto da laboratorio e guanti. Quindi, sterilizzare lo spazio di lavoro utilizzando il 70% di etanolo per pulire la superficie.
In questa procedura, il gene di resistenza all'ampicillina verrà trasferito dal ceppo WM3064 di E. coli al ceppo J53 di E. coli tramite coniugazione. Il ceppo donatore WM3064 è resistente alla tetraciclina e all'ampicillina e richiede acido diaminopimelico, o DAP, per crescere. Il ceppo ricevente J53 è resistente solo alla tetraciclina e non richiede DAP per crescere. Ciò significa che le cellule coniugate con successo dovrebbero essere resistenti alla tetraciclina e all'ampicillina e possono crescere senza DAP.
Preparare la coltura del ceppo del donatore inoculando cinque millilitri di LB contenenti 0,3 millimoli di DAP con uno scarto del ceppo di glicerolo congelato del donatore. Quindi, preparare il ceppo ricevente inoculando cinque millilitri di brodo LB senza DAP con uno scarto del ceppo ricevente congelato. Coltiva queste colture durante la notte a 37 gradi Celsius con aerazione e agitazione a 220 RPM in un incubatore vibrante. Una volta che le colture sono cresciute fino a un OD 600 di due, rimuovere un millilitro di coltura da ciascuna e posizionarlo in due nuovi tubi microcentrifuga separati da 1,5 millilitro. Quindi, centrifugare queste aliquote a 3000 RPM per cinque minuti per pellettare le cellule batteriche. Scartare il surnatante e lavare ogni pellet con 250 microlitri di 1X PBS. Centrifugare nuovamente i campioni e, dopo aver scartato il surnatante, risuscidere ogni pellet in 500 microlitri di PBS.
Per iniziare la procedura di coniugazione, combinare prima 50 microlitri di cellule riceventi con 50 microlitri di cellule donatrici in un tubo microcentrifuga da 1,5 millilitri e mescolare pipettando su e giù delicatamente. Successivamente, pipettare 100 microlitri della coltura cellulare ricevente su un'altra piastra tetraciclina 1X contenente DAP. Quindi, preparare il controllo negativo pipettando 100 microlitri della coltura cellulare ricevente solo su una piastra di agar non selettiva contenente DAP. Quindi, incubare le piastre di coniugazione e controllo negativo durante la notte a 37 gradi Celsius.
Il giorno dopo, prendi un raschietto sterile e raccogli le cellule dalla piastra di coniugazione raccogliendo colonie. Quindi, trasferire le colonie in un tubo microcentrifuga sterile da 1,5 millilitro contenente un millilitro di 1X PBS. Ripetere questo processo per raccogliere le cellule riceventi dall'altra piastra.
Dopo questo, vortice i campioni da mescolare. Dopo la miscelazione, trasferire i tubi in una centrifuga per pellettare delicatamente le celle. Scartare il surnatante, quindi lavare i pellet cellulari in un millilitro di PBS e vorticare i tubi per ricaspendare le cellule. Pellet le celle nuovamente centrifugando. Scartare nuovamente il surnatante e risuscimpere entrambi i pellet cellulari in un millilitro di PBS. Ora, utilizzando una punta di pipetta sterile, piastra 100 microlitri della miscela di cellule di reazione di coniugazione su una piastra di agar LB senza DAP contenente 1X tetraciclina e 1X ampicillina. Ripetere il metodo di placcatura utilizzando 100 microlitri di una diluizione dieci volte della stessa miscela cellulare in PBS su un'altra piastra di agar LB senza DAP contenente 1X tetraciclina e 1X ampicillina.
Infine, pipettare 100 microlitri della miscela di celle di controllo negativo su una singola piastra di agar LB con solo tetraciclina 1X. Dopo l'incubazione notturna a 37 gradi Celsius, le colonie dovrebbero essere visibili. Usando una punta di pipetta sterile, prelevare una singola colonia dalla piastra di reazione di coniugazione e aggiungerla a un tubo contenente cinque millilitri di mezzi LB selettivi contenenti entrambi gli antibiotici. Quindi, ripetere l'isolamento della colonia selezionando una singola colonia dalla piastra cellulare ricevente. Coltiva queste colture durante la notte a 37 gradi Celsius con aerazione a 220 RPM.
Il giorno dopo, pulire il piano di lavoro con etanolo al 70% e rimuovere le piastre dall'incubatrice. Usa un kit di preparazione del DNA mini per isolare il DNA da 4. 5 millilitri di ogni coltura secondo le istruzioni del produttore. Dopo aver completato la mini preparazione del DNA, eluire il DNA utilizzando 35 microlitri di acqua priva di nucleasi. Infine, utilizzare lo 0 rimanente. 5 millilitri di ogni coltura per preparare un millilitro di scorte di glicerolo aggiungendo 0,5 millilitri di glicerolo al 100% per una diluizione uno-a-uno. Posizionare queste aliquote a meno 80 gradi Celsius per la conservazione fino a quando necessario.
Per confermare il successo della coniugazione mediante PCR, preparare prima un mix master PCR aggiungendo 75 microlitri di mix master PCR 2X a un tubo microcentrifuga. Quindi, aggiungere 7,5 microlitri ciascuno di un primer in avanti da 10 micromolari e un primer inverso da 10 micromolari progettato per amplificare il gene di resistenza all'ampicillina dal plasmide. Successivamente, preparare un secondo mix master PCR aggiungendo 75 microlitri di mix master PCR 2X a un tubo microcentrifuga e quindi aggiungendo 7,5 microlitri ciascuno di un primer in avanti da 10 micromolari e 10 primer inverso micromolare progettato per amplificare un gene di pulizia, in questo caso DNA girasi B.
Ora, aggiungi 15 microlitri del primo mix master a un tubo PCR e poi aggiungi 10 nanogrammi, circa due microlitri del DNA sperimentale modello allo stesso tubo. Portare la reazione fino a un volume finale di 25 microlitri con acqua priva di nucleasi. Ripetere questi passaggi per produrre le restanti cinque reazioni, in modo che i tubi contengano i componenti mostrati qui. Ora, trasferisci queste reazioni su un termociclatore con il blocco preriscaldati a 98 gradi Celsius e quindi avvia il programma. Dopo il completamento della PCR, rimuovere i tubi dalla macchina. Quindi, caricare due microlitri di ogni reazione mescolati con due microlitri di colorante di carico e quattro microlitri di un marcatore di peso molecolare in pozzeli consecutivi di un gel di agarose all'1%. Impostare il gel in modo che funzioni a 150 volt per 20 minuti. Infine, visualizza il gel usando un illuminatore UV.
In questo esperimento, il successo del trasferimento del gene di resistenza all'ampicillina tramite coniugazione è stato confermato tramite PCR. Qui, una banda di circa 500 coppie di basi dovrebbe essere osservata nel pozzo contenente il DNA coniugato e i primer di ampicillina, ben due in questo esempio. Un gene di pulizia, DNA gyrase B, è stato caricato nei pozzi tre e cinque con DNA coniugato e DNA cellulare ricevente, rispettivamente. Le bande osservate in questi pozzette agiscono come un controllo positivo per garantire che il modello di DNA fosse presente e che la PCR abbia avuto successo. Le bande non devono essere osservate nel pozzo contenente la reazione per il DNA della cellula ricevente e la coppia di primer dell'ampicillina, ben quattro in questo esempio, perché le cellule riceventi non sono resistenti all'ampicillina. Inoltre, nessuna banda dovrebbe essere osservata nelle reazioni prive di DNA modello, pozzi sei e sette qui. Se queste condizioni sono soddisfatte, ciò confermerà il successo del trasferimento del gene di resistenza all'ampicillina, conferendo resistenza all'ampicillina dal ceppo WM3064 di E. coli al ceppo J53 di E. coli.
Subscription Required. Please recommend JoVE to your librarian.
Results
Se la coniugazione ha avuto successo, un prodotto PCR a banda di 500 coppie di basi sarà osservato nel pozzo in cui è stata caricata la reazione PCR 1 (Ben #2 in Figura 3B), mentre non si osserveranno bande nel pozzo in cui è stata caricata la reazione PCR 3 (Ben #4 in Figura 3B). La presenza di questa banda conferma il successo del trasferimento del gene di resistenza all'ampicillina, conferendo così resistenza all'ampicillina al ceppo J53 di E. coli.

Figura 3B: La conferma del successo della coniugazione mediante PCR. B) L'analisi PCR è stata effettuata utilizzando DNA isolato dalla colonia selezionata. Il contenuto di ciascun pozzo è il seguente: 1) Scala del DNA, 2) DNA di coniugazione e primer di ampicillina, 3) DNA di coniugazione e primer di pulizia, 4) Primer di DNA e ampicillina di controllo negativo, 5) DNA di controllo negativo e primer di pulizia, 6) Nessun primer di DNA e ampicillina e 7) Nessun primer di controllo del DNA e negativo. La presenza di un prodotto PCR a banda base ~500 dalla reazione PCR 1 (ben 2) e la mancanza di questo prodotto dalla reazione PCR 3 (bene 4), conferma il successo della coniugazione.
Subscription Required. Please recommend JoVE to your librarian.
Applications and Summary
La coniugazione è un processo naturale di trasferimento genico orizzontale che si basa sul contatto diretto cellula-cellula di una cellula donatrice e di una cellula ricevente. Questo processo è condiviso tra tutti i tipi di batteri ed è stato determinante nell'evoluzione batterica, in particolare la resistenza agli antibiotici. In laboratorio, la coniugazione può essere utilizzata come metodo efficace di trasferimento genico che è molto meno dirompente rispetto ad altre tecniche. Al di fuori del laboratorio, la capacità di trasferire il DNA dai batteri agli eucarioti attraverso la coniugazione offre una nuova entusiasmante strada di terapia genica e la comprensione delle implicazioni di questi trasferimenti genici naturali, ad esempio la relazione tra infezione batterica e cancro, è un'area di ricerca in rapida amerlica.
Subscription Required. Please recommend JoVE to your librarian.
References
- Lederberg J, Tatum, E.L. Gene recombination in Escherichia coli Nature. 1946;158:558.
- Holmes R.K. J, M.G. Genetics: Exchange of Genetic Information. 4th Edition ed. Baron S, editor. Galveston, TX: University of Texas Medical Branch at Galveston; 1996.
- Cruz F, Davies, J. Horizontal gene transfer and the origin of species: lessons from bacteria. Trends in Microbiology. 2000;8:128-33.
- Llosa M, Cruz, F. Bacterial conjugation: a potential tool for genomic engineering. Ressearch in Microbiology. 2005;156:1-6.
- Lacroix B, Citovsky, V. Transfer of DNA from Bacteria to Eukaryotes. mBio. 2016;7(4):1-9.
- Llosa M, et al. Bacterial conjugation: a two-step mechanism for
- DNA transport. Molecular Microbiology. 2002;45:1-8.
- Grohmann E, Muth, G., Espinosa, M. Conjugative Plasmid Transfer in Gram-Positive Bacteria. Microbiology and Molecular Biology Reviews. 2003;67:277-301.
- Firth N, Ippen-Ihler, K, Skurray, RA. Structure and function of the F factor and mechanism of conjugation. Escherichia coli and salmonella: cellular and molecular biology. 1996;2:2377-401.
- Smillie C, Garcillan-Barcia MP, Francia MV, Rocha EPC, De La Cruz F. Mobility of Plasmids. Microbiology and Molecular Biology Reviews. 2010;74(3):434-52.
- Cascales E. Definition of a Bacterial Type IV Secretion Pathway for a DNA Substrate. 2004;304(5674):1170-3.
- Wang P, Yu Z, Li B, Cai X, Zeng Z, Chen X, et al. Development of an efficient conjugation-based genetic manipulation system for Pseudoalteromonas. Microbial Cell Factories. 2015;14(1):11.
- Yi H, Cho YJ, Yong D, Chun J. Genome Sequence of Escherichia coli J53, a Reference Strain for Genetic Studies. Journal of Bacteriology. 2012;194(14):3742-3.
- Baumann RLB, E. H.; Wiseman, J. S.; Vaal, M.; Nichols, J. S. Inhibition of Escherichia coli Growth and Diaminopimelic Acid Epimerase by 3-Chlorodiaminopimelic Acid. Antimicrobial Agents and Chemotherapy 1988;32:1119-23.
- Rocha D, Santos, CS, Pacheco LG. Bacterial reference genes for gene expression studies by RT-qPCR: survey and analysis. Antonie Van Leeuwenhoek. 2015;108:685-93.




